样品处理
选择黑龙江省海林市横道河子镇(A, N 44°48′44″,E 129°02′04″)森林地区于2020年5月21日至8月22日(夏季节)捕获鼠类82只,作为研究样本。共采集样品22份软性肌病(MR),16大鼠半岛(AP),15黑线姬鼠,11西伯利亚Tamias sibiricus (TS),10Sciurus vulgaris (SV)和8大仓鼠(CT)。共有六个来自同一物种的样本被混合。
将粪便颗粒样品均质,用PBS按1:10的比例稀释,制成悬浮液,并进行涡流充分混合。然后,样品在4°C下以2000 rpm离心10分钟。随后,将上清液转移到新鲜试管中,离心10分钟,以完全去除细胞碎片、细菌细胞和其他杂质。上清液经0.22 μm注射器过滤器(Jet, Guangzhou, China)过滤浓缩。滤液在SW55Ti转子中用Beckman超离心机以45000 rpm的转速离心2小时。沉淀物在PBS中重新悬浮,通过0.22 μm注射器过滤器。然后将样品保存在- 80℃下,直到后续分析。
病毒宏基因组学分析
按照制造商的建议,使用NEBNext®Ultra™DNA文库准备试剂盒for Illumina (NEB, USA)生成测序文库。在每个样本的属性序列中添加索引码。简单地说,通过超声将DNA样本破碎至300 bp大小,然后对DNA片段进行末端抛光,a尾,并与全长适配器连接,用于Illumina测序,以帮助进一步的PCR扩增。
最后,纯化PCR产物(AMPure XP系统),使用Agilent2100生物分析仪分析文库的大小分布,并使用实时PCR定量。根据制造商的说明,使用cBot聚类生成系统对索引编码样本进行聚类。聚类生成后,在Illumina HiSeq2500平台上对文库制剂进行测序,并生成配对端读。
物种注释和丰度分析
采用Prinseq软件(版本0.20.4)评估样本数据质量,筛选低质量和重复序列,以啮齿动物基因组作为参考基因组。使用Bowtie2软件去除宿主DNA序列,使用Mira (v4.0.2)软件进行序列拼接和组装。使用BLAST +软件包中的BLASTX和BLASTN工具在本地NCBI病毒数据库中搜索组装和未组装序列,获得病毒注释。采用2019.2.1遗传版本对新标注病毒的ORF进行预测。
粪便上清在BHK-21细胞中的传代
BHK-21用于繁殖野生小鼠的粪便悬浮液,并根据先前发表的方法检测病毒[11,12].简单地说,将均质粪便颗粒的上清液通过0.22 μ m注射器过滤器过滤,并接种到BHK21细胞上,然后孵育2小时以允许病毒吸附。加入新鲜培养基后,每天在37℃下孵育细胞,直到感染后7天发生CPE。感染细胞盲传3 ~ 6次,直至出现CPE。cpe阳性BHK-21细胞的上清进一步接种于BHK-21细胞以检测感染性病毒的存在。
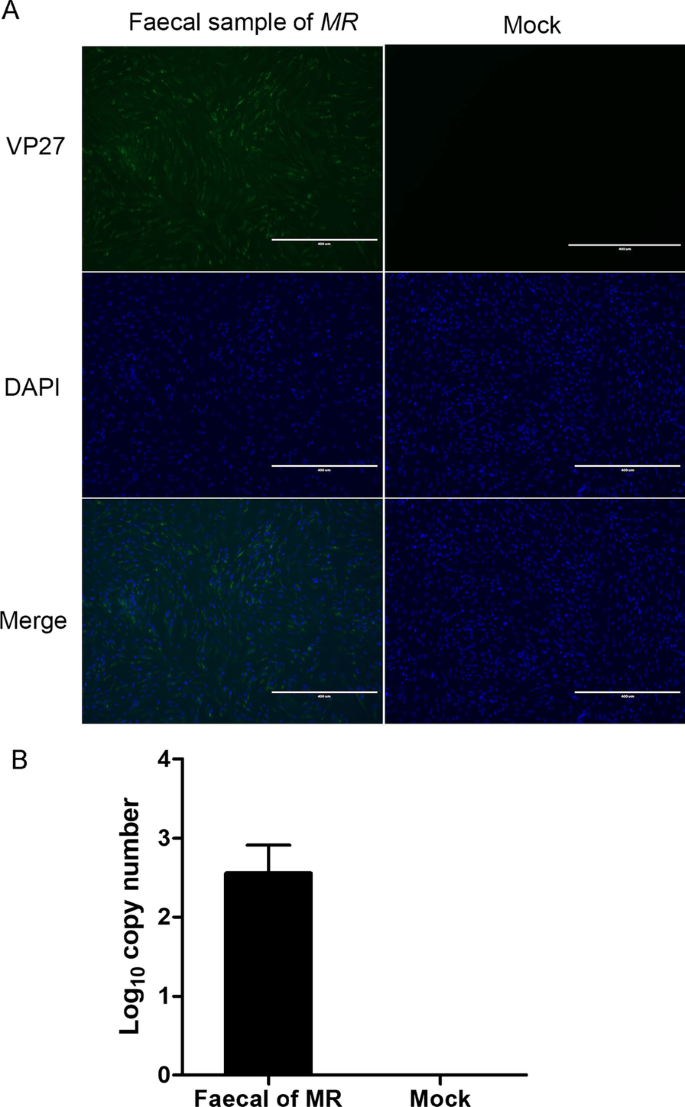
星状病毒在cpe阳性细胞中的检测
cpe阳性细胞上清接种BHK-21细胞,培养36 h,采用实时荧光定量反转录PCR (RT-qPCR)、免疫荧光检测(IFA)分析接种细胞中星状病毒的存在情况。在RT-qPCR分析中,根据组装的contig序列设计了特异性扩增星状病毒靶基因的引物。
系统发育分析
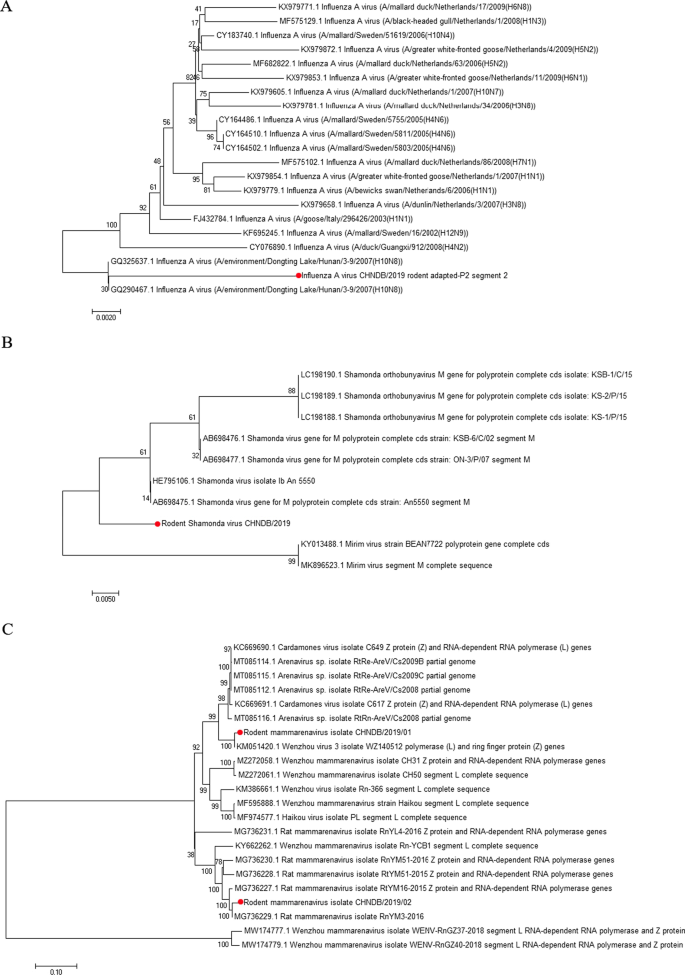
与本研究病毒相似度较高的参考毒株序列从NCBI下载。基于近邻连接最大复合似然法,利用MEGA软件构建了1000个自举重复的系统发育树,分析了系统发育关系。
统计分析
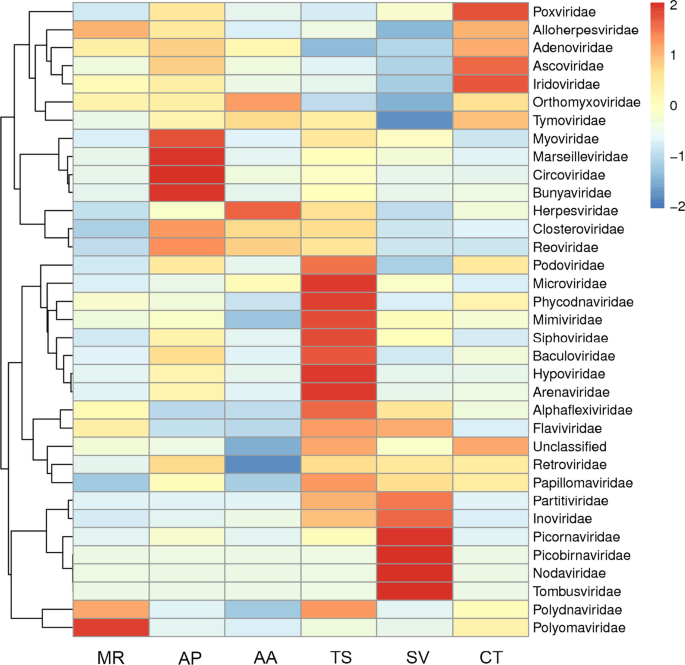
利用MetaStat软件对3个样品中丰度最高的10个分类序列标签进行分析。这些差异被认为具有统计学意义P-value小于0.05。