摘要
背景
慢性乙型肝炎病毒(HBV)感染已被证明与肝细胞癌(HCC)的发展高度相关。
目标
本研究的目的是探讨HBV前s区准种与HCC发展的关系,并利用HBV前s区准种建立HCC诊断模型。
方法
共纳入104例慢性乙型肝炎(CHB)患者和117例hbv相关HCC患者。采用下一代测序(NGS)对HBV前s区进行测序,计算核苷酸熵进行准种评价。使用稀疏逻辑回归(SLR)预测HCC的发展,并使用受试者工作特征曲线评估预测性能。
结果
HBV preS1、preS2区和多个核苷酸点的熵值在CHB和HCC患者中存在显著差异。采用SLR对HCC/CHB组进行分类,训练数据中受试者工作特征曲线(AUC)下的平均面积为0.883,测试数据为0.795。该预测模型也由香港的一个完全独立的数据集验证。所选的10个核苷酸位点在CHB和HCC患者之间显示出明显的熵值差异。HBV准种还对试验数据中AUC值大于0.6的HBeAg、HBVDNA、碱性磷酸酶(Alkaline phosphatase, ALP) 3个临床参数进行了分类。
结论
通过NGS和SLR,我们的研究验证了HBV前s区核苷酸熵与HCC发展之间的关系,这可以促进对HCC进展机制的理解。
简介
肝细胞癌(HCC)是最常见的原发性肝癌类型。慢性HBV感染患者发生HCC的风险增加,特别是慢性肝病和肝硬化患者[1,2,3.].
HBV群体以准种的形式存在,由宿主中大量的遗传异种变体组成[4].由于HBV复制中的逆转录酶缺乏校对活性,HBV复制错误率远高于其他DNA病毒。因此,在HBV准种长期感染期间,可以观察到各种突变[5].一些突变可以作为预测hbv相关HCC发展和进展的潜在病毒标记物。HBV整合位点、HBV基因型、基础核心启动子突变、前核突变和前s缺失都与HCC的发生有关[5,6,7,8,9,10,11,12,13,14,15,16].
HBV的双链DNA (dsDNA)基因组包含四个重叠的开放阅读框。由preS1和preS2片段组成的preS区域(核苷酸2854-155)与聚合酶基因的一个区域重叠。在慢性HBV感染患者中,自然发生或治疗诱导的带有前S/S基因组区突变的HBV变异的选择和出现是经常发生的事件。特别是,前S/S区域的特定突变可能诱导在肝细胞内质网(ER)中积累的包膜蛋白的不平衡生产,潜在地激活ER应激信号通路,从而诱导氧化DNA损伤和基因组不稳定[17].因此,前s区可能对HCC的发展有更多的临床意义。
下一代测序(NGS)技术的发展极大地加速了基因组研究。NGS可同时对大量病毒准种进行测序,具有较高的敏感性和特异性[18NGS已广泛应用于HBV低频耐药的探索[19,20.].在HBV研究中,NGS与以往测序技术的比较研究显示了NGS的优势[21,22].近年来,人们利用NGS研究了preS突变与HCC之间的关系[16,23,24,25].在研究中[16,23],作者主要运用统计学检验来分析preS突变与HCC的关系。我们的团队[25]也研究了前s缺失与HCC的相关性,并使用支持向量机(SVM)对已识别的相关前s缺失的预测结果进行检验。我们的团队还应用HBV基因组的单词模式来定义HCC患者之间的距离、HBV基因型的异质性以及HBV preS区单词模式与HCC的相关性[25].其他研究人员也报道了前s缺失的预测作用[6,7,11,26,27,28]在肝癌发展中的作用。虽然HBV前s准种与HCC之间的关系已在有限的研究中进行了探讨[16,29,应该进行更深入的调查。
在这项工作中,我们基于前s区NGS研究了HCC与HBV准种之间的关系。探讨了慢性乙型肝炎(CHB)和HCC患者s前区准种。利用稀疏逻辑回归(SLR)等最先进的统计学习方法,我们建立了HBV准种的HCC/CHB预测模型。我们还分析了促成这些关联的职位。研究了HBV准种与临床参数的相关性。
材料与方法
数据来源和参与者
本研究遵循了个体预后或诊断多变量预测模型透明报告(TRIPOD)报告[30.](附加文件5:表S1)。中国上海东方肝胆外科医院于2011年3月至2012年5月期间入组HCC患者。hbv相关HCC患者符合以下标准:(1)血清乙型肝炎病毒表面抗原(HBsAg)阳性至少6个月;(2) HBV DNA水平> 1000 IU/ml;(3)经手术和组织病理学检查证实的HCC特征。排除标准包括丙型肝炎病毒或人类免疫缺陷病毒合并感染、肝移植史、自身免疫性肝病、转移性肝癌、其他恶性肿瘤、药物相关性肝病、酒精性肝炎和入组前诊断的其他原因的慢性肝病。符合标准的CHB患者包括:(1)血清HBsAg阳性至少6个月;(2)连续或反复血清谷丙转氨酶(ALT)升高(非因HBV感染而高于参考值上限2倍)或经肝活检证实为慢性病毒性肝炎特征;(3) HBV DNA水平> 1000 IU/ml。排除标准包括肝细胞癌、恶性肿瘤或其他严重疾病。 This study was approved by The Ethics Committee of the Eastern Hepatobiliary Hospital (EHBHKY2015-01–004). Serum samples were collected from all patients before hepatectomy. Totally, 104 CHB samples and 117 HCC samples were amplified and sequenced successfully, with 63 CHB patients (CHB group) and 46 HBV-related HCC patients (HCC group) in the training set (Shanghai dataset), and 41 CHB and 71 HCC samples in the test set (Shanghai dataset). For the HCC patients, we also collected their clinical examination data.
前s区HBV DNA提取及Illumina测序
使用QIAamp DNA Mini试剂盒(QIAGEN GmbH, Hilden, Germany)从200 μl血清样本中提取HBV基因组,用100 μl蒸馏水洗脱。使用Phanta Super-Fidelity DNA聚合酶(Vazyme Biotech, Piscataway, New Jersey, USA)和一对引物:5 ' -CGCCTCATTYTKYGGGTCA-3 '(正向,核苷酸2801-2819)和5 ' - tcckgaactggagccac -3 '(反向,核苷酸62 - 79)扩增preS区。前s区PCR扩增物用agcourt AMPure XP珠(Beckman Coulter, Beverly, Massachusetts)纯化,用Qubit dsDNA HS检测试剂盒(Invitrogen, Carlsbad, CA, USA)定量。使用TruSeq DNA PCR-免费样品制备试剂盒(Illumina, San Diego, CA, USA)制备preS区域PCR产物文库,并在MiSeq测序仪(Illumina, San Diego, CA, USA)上运行,根据Illumina协议进行配对测序。最后,利用MiSeq控制软件对荧光信号进行分析,并以FASTQ格式转换为序列数据。
序列读取映射和基因分型
使用在线工具fastqc (http://fastqc)对原始读数进行质量评估www.bioinformatics.babraham.ac.uk项目/ fastqc /),平均呼叫质量分数低于20的读取被丢弃。经过质量过滤和适配器移除后,配对的端读与FLASH连接,v1.2.10 [31].合并前s区序列用HBV STAR软件进行基因分型,如先前报道[32],并对GenBank数据库中23个HBV参考基因组进行基因分型(登录号:X02763、X51970、AF090842、D00329、AB073846、AB602818、X04615、AY123041、AB014381、X65259、M32138、X85254、X75657、AB032431、X69798、AB036910、AF223965、AF160501、AB064310、AF405706、AY090454、AY090457、AY090460)。每个样本的基因型定义为A - H 8种类型中最常见的基因型。
外部验证
该数据集包括32例与hbv相关的HCC患者和32例无HCC的CHB患者(香港数据集),患者于2007年7月至2012年12月在香港香港大学玛丽医院肝炎及肝脏诊所入组[16].采集血清样本并测序。更多关于患者登记和HBV测序的细节可在[16].除了深度测序中使用的Illumina MiSeq平台外,所有其他平台和工具都与我们生成数据时使用的不同。我们从研究人员那里得到了数据。16],并使用BLAST将合并的reads (fasta格式)映射到HBV参考基因组。根据映射结果,过滤掉带有插入、删除和翻转的读操作。如果一个样本的正常读数百分比小于20,我们就删除该样本。最后,我们获得了26例HCC和23例CHB患者的数据。该序列包含589个核苷酸,其中457个核苷酸与我们测序的片段重叠。对于这个数据集,我们只考虑了与我们数据集中相同的457个位置。
数据预处理和预测器
对准种进行测序后,收集了前s区1 ~ 61位、前s区附近2820 ~ 3215位等457个位点的点突变数据。我们计算了每个位置核苷酸的频率。为了描述每个位置的突变复杂度,我们将频率数据转换为香农熵,定义为\(H = - \sum\nolimits_{i} {p_{i}}\ log p_{i}\),\(\sum\nolimits_{i} {p_{i}} = 1\)在哪里\(i \in \{A,C,G,T\}\)而且p我是它的频率,(x\log (x) = 0\)当x= 0。前s区所有457个核苷酸位置的熵被用作HCC诊断的预测因子。
模型开发和验证
我们应用稀疏逻辑回归(SLR)对HCC/CHB组与准种之间的关系进行建模。单反是加名词\(\lambda \左\| {\beta_{1}} \右\|\)到原逻辑回归模型,其中β\ (\ \)是变量的系数向量。该模型可以同时进行分类和变量选择。通过调优参数\λ(\ \)的稀疏形式β\ (\ \)与所选变量相对应的非零项。本研究的自变量包括457个体位的熵数据,反应变量表示属于CHB或HCC组的患者。我们的目标是建立457个部位与CHB/HCC组之间的关联模型。我们应用K-fold交叉验证(CV)来选择参数\λ(\ \)这样β\ (\ \)是那些在最高精度的一个标准差内实现精度的最罕见的。然后将所选训练数据应用于拟合模型\λ(\ \)到测试集看预测性能。我们直接在R包' glmnet '中实现了函数:glmnet() [16]通过设置alpha = 1,这是一个平衡贡献的参数\(\左\| {\beta_{1}} \右\|\)而且\(\左\| {\beta_{2}} \右\|\).当= 1时\(\左\| {\beta_{2}} \右\|\)术语对模型没有贡献,选择的变量较少,但分类精度相同。在我们的实验中,我们使用了四个标准来评估模型的性能:准确性、ROC曲线下面积(AUC)、敏感性和特异性。
HCC患者临床参数与准种的相关性
对于分类临床参数和非正态分布的定量参数,我们采用了SLR,如上所述。对于服从正态分布的参数,我们应用了稀疏偏最小二乘回归(SPLS),这是一种寻找所有自变量的组合,以便与响应变量最相关的方法。这里,我们也强加了l1得到了系数的稀疏解。我们采用了[34]并直接使用R包' spls ' [34].为了选择潜在成分(组合)的数量κ和软阈值η来确定系数的零项,我们还使用CV来调整参数。我们首先固定η和变化κ来选择最佳的κ,然后固定κ来选择最佳的η。
结果
在本节中,我们介绍了我们关于前s区核苷酸点熵与HCC发展之间关系的主要发现。我们的主要目的是利用前s区核苷酸点熵对CHB/HCC患者进行分类或建立HCC预测模型,同时寻找一些有助于HCC发展的重要点突变。由于临床指标更容易获得,我们也探讨了前s区点突变与临床指标之间的关系。
前s区参与者和核苷酸熵
CHB和HCC患者的基线信息(上海数据集)汇总于表中1.在训练组和测试组中,HCC患者的肝功能更差,年龄更大,血清HBV DNA水平更低。
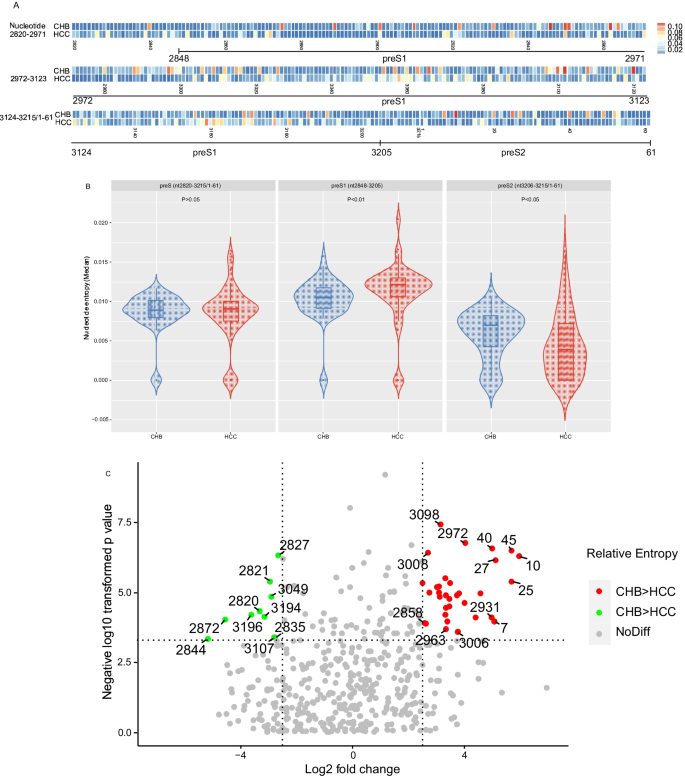
计算preS区核苷酸熵,其熵分布如图所示。1A. CHB患者前s区的熵中位数为0.0087(0.0074-0.0092),低于HCC患者的0.0090(0.0076-0.01001)。preS区各核苷酸点的熵没有显著差异(图2)。1B).分别比较preS1和preS2的核苷酸点熵,HCC患者preS1区的核苷酸点熵明显高于CHB患者。而在preS2区,HCC患者与CHB患者的趋势相反(图2)。1B).此外,比较了单个核苷酸位置的熵p-value和折叠变化如图所示。1C. CHB和HCC患者很多部位的熵值有明显的差异。
模型开发和性能
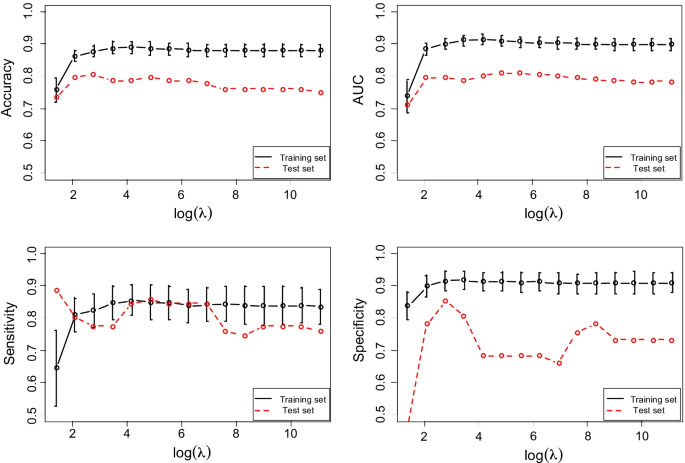
由于CHB和HCC患者之间存在太多具有不同熵的核苷酸位置,因此应采用更复杂的方法来研究核苷酸熵与HCC发展之间的关系。因此,我们利用SLR技术研究了具有准种数据的HCC/CHB组的分类[33].将该模型与训练数据集(46 HCC/63 CHB,上海数据集)进行拟合,并应用于测试集(71 HCC/41 CHB,上海数据集)进行预测。为了调整控制变量(核苷酸位置)选择的参数λ,我们在训练集中运行5倍CV 50次。λ的值从0.5开始2以0.5的比例减小,λ的长度设为15。数字2给出了所有λ的预测结果。在训练数据中,当λ小于0.53.(第2点),4个评价标准均稳定,敏感性SD最大。在测试集中,准确度和AUC从λ = 0.5开始稳定3..这里λ被选为0.53.、表2显示分类结果。训练集的准确率和AUC均值分别为0.861 (SD = 0.032)和0.883 (SD = 0.043),测试集的准确率和AUC均值分别为0.794和0.795。表中SLR模型比经典逻辑回归模型表现更优2.这表明HBV准种与HCC发展之间存在高度相关性。
利用单反得到的最终预测模型为:
其中,每个变量的下标表示所选的点突变位置。对于每个样本,在计算出每个位置的熵后,通过减去平均熵对样本进行集中。然后应用上述公式计算发生CHB或HCC的概率,发生CHB的概率较小。
其他机器学习方法也进行了研究。我们将上述结果与支持向量机(SVM)的结果进行比较[35,36]和稀疏支持向量机(SSVM) [37].支持向量机是机器学习中常用的分类方法,它使用所有考虑的变量对样本进行分类。与单反相似,SSVM被表述为一个铰链损失函数l1进行分类时选择相关变量的惩罚项[37].我们使用R包' e1071 '实现支持向量机,使用R包' sparseSVM '实现SSVM [37),分别。使用与SLR类似的方法,我们使用训练集训练模型,并将其应用于测试集。测试数据集的预测结果如表所示2.对于SSVM,我们还使用R包“platt”[38],实现普氏校准。普氏校准是通过拟合逻辑回归模型到分类器的分数,将分类输出转换为类的概率分布。通过校准,可提高SSVM的性能。由于SLR输出每个样本是HCC患者的概率,我们直接给出了其校准图。将单反雷达和校准后的SSVM的预测结果和可靠性图放入附加文件1:图S1和附加文件6:表S2,性能相似。支持向量机的AUC虽然比单反的AUC高,但不能识别相关变量。SSVM和校准SSVM的性能都比单反差得多。因此,我们接下来对CHB/HCC分类的分析是基于SLR。
在香港数据集进行独立验证
我们首先将SLR应用于香港数据集的熵数据作为训练集。由于样本量小,我们使用10倍CV 50次在该数据集中进行实验并记录结果。最后选取λ的值为0.56.表格3.“训练结果”(香港数据集)显示结果。在香港数据中,平均精度和AUC分别达到0.822 (SD = 0.031)和0.724 (SD = 0.054)。然后,我们使用上海数据集训练的模型来预测香港数据集的HCC/CHB患者作为测试集。测试结果见表中的“测试结果”3..预测精度为0.694,AUC为0.607。独立测序实验进一步证实HCC的发展与HBV准种相关。
与HCC发展相关的核苷酸位置准种
我们检查了当λ = 0.5时,用我们的整个训练数据训练的模型中的系数3..选择了10个与HCC相关的位点:10,45,2858,2861,2992,3046,3090,3093,3098和3207。当λ小于0.5时3.,除位置45外,其余9个位置均保留在模型中。这表明HCC与9个核苷酸位置高度相关。在无花果。3.,这些核苷酸点的熵在HCC和CHB患者之间有显著差异。我们进行了t-test这10个位置的熵数据。所有这些指标在HCC和CHB患者之间均有显著差异,且差异最小p-value在位置45和最大值p-位置3207的值。附加文件中也有这10个部位在HCC和CHB患者中的基频2:图S2,可以看出HCC和CHB样本的碱基分布也不同。
训练样本量对HCC/CHB患者预测准确性的影响
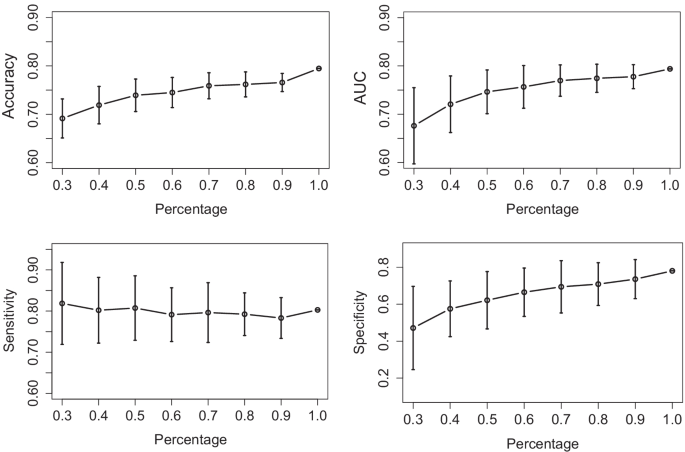
由于样本有限,当应用于新患者时,所开发的模型可能会表现得更差,我们进一步做了实验来评估预测模型,并了解SLR的性能如何取决于样本量[39].我们从λ = 0.5的原始训练集中随机选择给定百分比的样本3.训练模型,然后将其应用于测试数据。百分比从30%到100%不等,100%对应于所有的训练样本。我们为每个百分比重复采样50次。所有四个评价标准的平均值和SD绘制在图中。4.随着样本量的增加,准确性、AUC和特异性均相应增加,而SD降低。灵敏度相对稳定,值在0.80左右。即使只有30%的数据,即只有33个训练样本,平均精度和AUC也在0.70和0.67左右,这比使用普通LR得到的结果要高,与使用SSVM的结果相当。当使用70%的训练样本(数量小于80个)时,结果明显优于LR和SSVM,且趋于稳定。这些显示了单反模型在我们的实验设置中的效率。
所有患者基因型分析
为了观察不同基因型样本之间的差异,我们分别研究了基因型C和基因型B的样本。用同样的模型训练方法,我们选择λ = 0.58λ = 0.53.基因型B和基因型C的患者。结果对不同的λ ' s显示在附加文件3.:图S3,所选λ的结果列在Table中4.与使用所有患者的结果相比,b基因型患者的特异性增加,敏感性降低。C基因型患者的特异性降低,敏感性增加数倍。
我们还进行了交叉预测,以检查预测性能。我们用训练集中的C或B基因型患者训练模型,并预测测试集中的其他基因型患者。结果添加在Table中4.准确性和AUC均与同一基因型内患者相当,但敏感性和特异性变化较大。因此,出于预测的目的,这一发现意味着我们可以将所有个体组合在一起,以产生更大的样本量,正如我们的实验所证明的那样。
HCC患者HBV准种与临床参数的相关性
对于HCC患者,我们调查了HBV准种与临床参数之间的关系。对于分类临床参数和非正态分布的定量参数,我们应用了SLR。对于服从正态分布的参数,我们应用SPLS [39].由于样本量小,我们运行10倍CV 50次以选择参数λ、η和κ。当我们应用SPLS模型时,η设置在0.1 ~ 0.9之间,步长设置为0.1,κ设置在2 ~ 10之间。如果独立检测的AUC大于0.60,我们认为该临床参数与HBV准种相关。最后,我们发现血清指标:乙型肝炎e抗原(HBeAg)、HBVDNA和碱性磷酸酶(ALP)与HBV准种相关。在附加文件中给出了不同λ和η值下的分类结果4:图S4表格5给出了所选λ和η的分类结果。
在训练集中对hbeag阳性和阴性患者进行分类时,准确率和AUC均在0.9左右。而测试集的准确性和AUC分别为0.672和0.607。对于参数HBVDNA,训练集中所有η′s的准确性和AUC都在0.7左右。在测试集中,准确性和AUC分别下降到0.676和0.675。同样,对于ALP,精度和AUC都是稳定的,所有η在训练集中都在0.8和0.7左右。而在测试集中,准确率和AUC分别为0.634和0.648。
讨论
在这项工作中,我们用NGS方法研究了CHB和HCC患者的HBV前s区准种。CHB与HCC患者前s区核苷酸熵水平无明显差异,这与既往研究不一致。16].即便如此,本研究中HCC患者前s区核苷酸点熵较高,与既往研究呈现相同的趋势[16].此外,两组患者preS1和preS2的核苷酸熵水平分别存在明显的熵散。患者组的组成可能是前一项研究与我们的研究结果不同的原因之一。另一方面,基因组中的不同片段可能表现出独特的核苷酸熵,并表现出特殊的功能。
然后我们利用单反技术对前s区深度测序数据进行了研究,研究了HCC与HBV准种之间的相关性。利用核苷酸频率熵对HCC和CHB患者进行分类,在独立试验集中预测准确率为0.794,AUC为0.795,优于经典的HCC标志物:AFP [40,41].在另一个来自香港的独立数据集中,预测精度和AUC分别为0.695和0.607。这些结果证明了HCC与HBV准种之间的高度相关性。香港数据集的准确性和AUC下降可能是由于不同的测序方案,特别是preS区域的测序起点和终点不同。由于目标序列采用大规模并行测序分析,即使是微小的差异也会被放大数千倍。其他原因可能包括不同的测序过程,不同的患者体质,不同的患者干预等。其原因值得进一步研究。
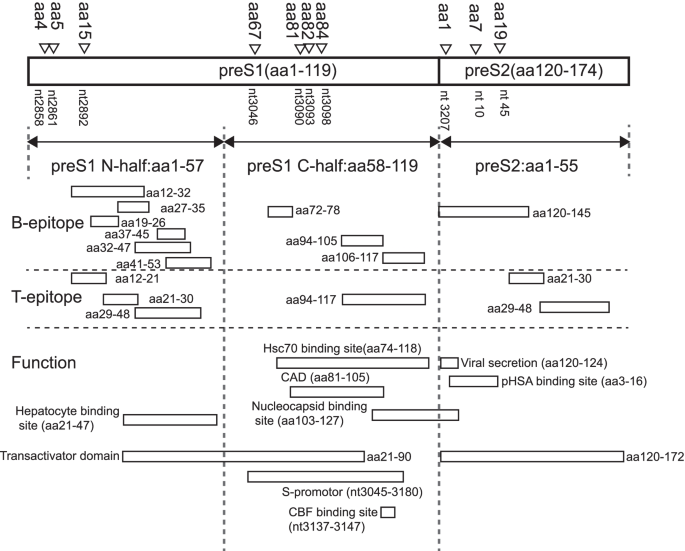
单反法的优点之一是在预测模型拟合的过程中可以选择显著变量。SLR选择的10个与HCC相关的位置在熵值和核苷酸频率数据上均有显著差异。我们将所选位置的核苷酸映射到氨基酸上,并检查这些位置的功能域[6].数字5显示了核苷酸的位置,对应的氨基酸和功能。除了2858和2861,其他职位都有相关的职能。位置3090、3093、3098、3207、10、45属于B细胞表位。位置2892属于T细胞表位。这些表位的突变可能有助于免疫逃逸并影响病毒-宿主免疫相互作用。位置3046、3090、3093、3098、3207、10、45属于转激活域。位置3046、3090、3093和3098位于s启动子区域。这些突变可能在病毒基因的转录和表达过程中起作用。位置10属于聚合人血清白蛋白(pHSA)结合位点,参与病毒结合和进入肝细胞的过程,该位点的突变可能会影响病毒与细胞的相互作用[42].位置3090、3093和3098都属于热休克同源蛋白70 (Hsc70)结合位点。
在这10个部位中,文献报道有3个部位与HCC相关,分别为10、2858和3098 [16,40,41,42].与无HCC hbsag阳性患者相比,C10A突变被报道为HCC患者的危险因素[43].我们的研究显示10位突变在HCC组和CHB组之间有显著差异p- 6.61 × 10的值8.除了在[中报道的' A '高度突变外43],我们还发现了' T '的高突变。“A”突变在HCC组中更高,而“T”突变在CHB组中更高。位置2858最近被发现与HCC发展相关[16].在这项研究中,3个密码子,包括4、27和167,被发现与HCC的发展相关。位置2858属于密码子4。如附加文件所示,与CHB相比,HCC中该位置的' C '突变要高得多2:图S2T3098C的突变也与HCC的进展有关[43,44’T’到‘C’的突变在HCC患者中比在CHB患者中要高得多。这与我们的研究是一致的,在附加文件中清楚地显示2:图S2除了' C '突变外,我们还发现在3098位点,HCC中的' G '突变高于CHB。
在分别对B和C基因型患者的实验中,预测性能略差于所有患者,但在独立测试集中仍达到了准确性,AUC大于0.7。预测的AUC下降可能是由于两个原因。首先,每种基因型病毒都可能有其特殊的疾病状态分类特征,这些特征可能在单基因型感染或混合基因型感染中起作用。病毒基因型混合感染患者按主感染基因型进行分离时,轻感染基因型特征在疾病状态分类中不起作用。先前的研究表明,不同HBV基因型的合并感染并不罕见。45,46因此,当患者首次按感染基因型分组时,分类效率下降。其次,HCC进展与C型HBV感染相关[46,47在我们的研究中,与CHB患者相比,大多数HCC患者感染了基因型C病毒(卡方检验,p-value = 0.02)。所以一些决定感染基因型的特征也可能决定疾病的状态。当按感染基因型进行人群分离时,这些特征无法进一步用于疾病分类,这也导致预测效果下降。
研究了HCC患者的临床检查参数与准种之间的关系。发现HBeAg、HBVDNA和ALP 3个参数与HBV准种相关。既往研究表明HBeAg和HBVDNA与HBV准种相关[9,49].我们的研究进一步显示了它们在前s区的关联。目前ALP尚无文献报道,值得进一步研究。与训练集相比,这3个参数在测试集中的AUC和准确性都有所下降。这可以归因于训练集和测试集的数据大小不平衡。我们应用置换检验来检验这三个参数与HBV准种之间的关联是否人为的。我们对响应标签进行了排列,并使用相同的方法来查看预测AUC。则由排列数据得到的AUC明显小于由真实数据得到的AUC (p-value < 0.05)。这证实了这些关联的存在。与这三个参数相关联的所选位置列在附加文件中7:表S3。
在我们的研究中,主要的统计方法是SLR和SPLS。利用稀疏性约束,我们可以同时找到相关变量并拟合预测模型。这些方法已应用于疾病相关生物标志物和分类的研究[34,50,51].虽然我们的团队也使用了其他机器学习方法,并基于HBV逆转录酶准种取得了更好的预测效率[41], SLR仍有优势,特别是在变量选择方面,可以促进对预测模型和HCC进展机制的理解。
在本研究中,我们只关注点突变,尽管HCC和HBV准种之间存在高度相关性。HBV基因组中也存在其他变异,如缺失、插入等,这些核苷酸变化可能与准种变异存在一定联系。我们在目前的研究中没有考虑到这些问题。如果能考虑到这些因素,应该会有更好的结果。这是我们未来的作品之一。此外,有限的样本量是本研究的一个弱点。最好是根据以前研究中描述的标准提前估计样本量[52,53],并使用足够的样本进行预测模型开发和验证。最后,外部验证的性能下降也表明,基于NGS的预测模型的应用应严格限制,包括相同的测序策略和生物信息学分析过程的预设条件。
结论
在本文中,我们利用SLR证明了NGS探测的前s区HBV准种与HCC之间的相关性。我们还发现前s区10个点突变与HCC的发展相关。利用这些点突变,HCC/CHB患者的预测显示出有希望的结果。这些结果有助于理解推动HBV发展为HCC的分子机制。
数据和材料的可用性
在当前研究中生成和分析的数据集可根据合理要求从通讯作者处获得。
缩写
- 乙肝病毒:
-
乙型肝炎病毒
- 慢性乙肝:
-
慢性乙型肝炎
- 肝细胞癌:
-
肝细胞癌
- LR:
-
逻辑回归
- 门店:
-
下一代测序
- ROC曲线:
-
受试者工作特征曲线
- 单反:
-
稀疏逻辑回归
- 期限:
-
稀疏偏最小二乘
- 支持向量机:
-
支持向量机
- SSVM:
-
稀疏支持向量机
- 法新社:
-
甲胎蛋白
- 治疗组:
-
总胆红素
- DBIL:
-
直接胆红素
- TP:
-
血清总蛋白
- 铝青铜:
-
白蛋白
- 高山:
-
碱性磷酸酶
- ALT:
-
丙氨酸转氨酶
- AST:
-
天冬氨酸转氨酶
- GGT:
-
γ谷氨酰转移酶
- e抗原:
-
乙型肝炎e抗原
- HBsAb:
-
乙型肝炎抗体
- HBeAb:
-
乙型肝炎e抗体
- PVTT:
-
门静脉血栓
参考文献
Sung H, Ferlay J, Siegel RL, Laversanne M, Soerjomataram I, Jemal A, Bray F. 2020年全球癌症统计:全球185个国家36种癌症的发病率和死亡率。中华癌症杂志。2021;71(3):209-49。
A.肝细胞癌。中华实用医学杂志,2019;29(3):344 - 344。
杨俊杰,Hainaut P, Gores GJ, Amadou A, Plymoth A, Roberts LR。肝细胞癌的全球观点:趋势、风险、预防和管理。中华胃肠病杂志,2019;16(10):589-604。
准种及其对病毒性肝炎的影响。病毒决议2007;127(2):131-50。
朱艳,金艳,郭霞,白霞,陈涛,王洁,钱刚,Groopman JD,顾娟,李娟,涂华。乙型肝炎病毒全序列的比较研究发现了与肝细胞癌相关的核心基因新突变。癌症流行病学生物标志物,2010;19(10):2623-30。
陈宝芳,刘佳杰,乔国明,陈普军,高建华,陈德生。前体s缺失在进行性肝病乙型肝炎病毒携带者中的高患病率和定位胃肠病学。2006;130(4):1153 - 68。
方志林,Sabin CA,董伯强,魏少生,陈启勇,方kx,杨建勇,黄娟,王晓霞,Harrison TJ。乙型肝炎病毒s前缺失突变是肝细胞癌的危险因素:一项匹配的嵌套病例-对照研究中华病毒学杂志,2008;39 (Pt 11): 2882-90。
黄娟,邓强,王强,李凯凯,戴建辉,李楠,朱志东,周波,刘晓霞,刘rf,费清良,陈辉,蔡波,周波,肖海生,秦丽霞,韩志刚。乙型肝炎病毒相关肝细胞癌的外显子组测序中国科学,2012;44(10):1117-21。
李峰,张东,李艳,姜东,罗松,杜宁,陈伟,邓玲,曾超。乙型肝炎病毒准种全基因组大规模并行焦磷酸测序。临床微生物感染。2015;21(3):280-7。
刘珊珊,张慧,顾超,尹娟,何颖,谢娟,曹刚。乙型肝炎病毒突变与肝细胞癌发病风险的meta分析。中华肿瘤学杂志,2009;30(4):344 - 344。
沈春峰,苏爱军,吴慧慧,谢永华,姚文军,杨kc,张天涛,谢慧娟,蔡慧恩,黄伟。一种pre-S基因芯片检测乙型肝炎病毒大表面抗原pre-S缺失作为慢性乙型肝炎病毒携带者肝癌风险的预测标志物。中国生物医学工程学报。2009;16:84。
宋伟坤、郑浩、李胜、陈荣、刘旭、李勇、李乃鹏、李伟辉、Ariyaratne PN、Tennakoon C、Mulawadi FH、王凯夫、刘亚敏、潘rt、范st、陈kl、龚震、胡勇、林志、王刚、张强、Barber TD、周炜、Aggarwal A、郝凯、周伟、张灿、Hardwick J、Buser C、徐俊、阚铮、戴宏、毛敏、Reinhard C、王杰、陆建明。肝细胞癌复发性HBV整合的全基因组调查。中国科学,2012;44(7):765-9。
叶婷婷,苏敏,吴俊,杨华伟,张明明,赖文伟,陈天涛,林春春,叶涛,李卫国。肝组织中乙型肝炎病毒dna水平和基础核心启动子A1762T/G1764A突变独立预测肝细胞癌术后生存。肝脏病学。2010;52(6):1922 - 33所示。
尹娟,谢娟,张慧,沈强,韩玲,陆伟,韩艳,李超,倪伟,王辉,曹刚。不同preS基因突变与乙型肝炎肝硬化或肝细胞癌的相关性研究。中华胃肠病杂志,2010;45(10):1063-71。
张东,董鹏,张凯,邓龙,巴赫C,陈伟,李峰,Protzer U,丁宏,曾晨。抗病毒治疗过程中HBV全基因组缺失谱及preS缺失突变体的积累。微生物学杂志。2012;12:307。
张哦,赖CL,黄财政年度,濑户工作,Fung J,黄DK,袁MF。HBV前s区准种的深度测序分析及其与肝细胞癌的关系。中华胃肠病杂志,2017;52(9):1064-74。
Pollicino T, Cacciola I, Saffioti F, Raimondo G.乙型肝炎病毒PreS/S基因变异:病理生物学和临床意义中华肝病杂志,2014;61(2):408-17。
谢瓦列兹,帕洛茨基,JM。在资源有限的环境中筛查、诊断和监测乙型和丙型肝炎的新病毒学工具。中华肝病杂志,2018;69(4):916-26。
高思思,吴海波,朴志强,李华昌,李智杰。超深焦磷酸测序分析乙型肝炎病毒耐药突变单倍型。临床微生物感染杂志,2012;18(10):E404-11。
罗德里格斯C,谢瓦列斯S, Bensadoun P, Pawlotsky JM。用超深焦磷酸测序技术研究乙型肝炎病毒对阿德福韦的耐药性动态。肝脏病学。58 2013;(3):890 - 901。
龚玲,韩艳,陈琳,刘峰,郝萍,盛杰,李小红,于德明,龚庆明,田芳,郭晓科,张xx。下一代测序与克隆测序在乙型肝炎病毒逆转录酶准种异质性分析中的比较临床微生物学杂志,2013;51(12):4087-94。
韩艳,龚亮,盛杰,刘峰,李小红,陈琳,于德明,龚庆明,郝萍,张xx。通过前处理乙肝病毒逆转录酶准种异质性预测病毒学反应:使用下一代测序的优势。临床微生物感染。2015;21(8):791-7。
刘文伟、吴志刚、李玉春、林正大、郑俊华、林玉杰、甄杰杰、郑培恩、李普芬、郑奕奕、郑培文、孙KT、阎少林、林俊杰、杨建昌、张建昌、何超、曾伟、张建斌、吴建昌、张婷婷。利用乙型肝炎病毒全基因组高通量分析确定的肝细胞癌相关单核苷酸变异和缺失中华病毒学杂志,2017;43(2):176-92。
白霞,贾娟,方梅,陈松,梁霞,朱松,张松,冯军,孙峰,高成。HBV前S区深度测序显示HBV基因型高度异质性及词型频率与HCC的相关性。Plos Genet, 2018;14(2):e1007206。
贾佳、梁霞、陈松、王红、李红、方梅、白霞、王铮、王敏、朱松、孙峰、高成。新一代测序结果显示HBV基因组preS区缺失在不同HBV相关肝病中存在差异。中国生物医学工程学报,2017;29(4):344 - 344。
滕芳芳,李廷涛,黄海燕,林剑辉,陈文伟,舒玉伟,吴兴华,彭春春,苏爱军,郑兰彬。基于新一代测序技术定量检测血浆中乙型肝炎病毒Pre-S突变体预测肝癌复发。病毒。2020;12(8):796。
滕芳芳,黄海燕,李廷涛,舒玉伟,吴慧霞,林春春,苏爱军,郑兰彬。基于新一代测序技术的肝癌患者血浆中乙肝病毒Pre-S突变体定量检测平台。科学通报2018;8(1):14816。
张哦,赖CL,黄财政年度,濑户工作,Fung J,黄DK,袁MF。乙型肝炎病毒Pre-S突变在肝细胞癌发展前的进化变化PLoS ONE。2015; 10 (9): e139478。
张安,赖晨,潘涛,黄峰,Seto W,冯军,黄德奎,袁敏。乙型肝炎病毒在肝细胞癌中的全基因组突变和准种。中华胃肠肝病杂志,2016;31(9):1638-45。
Collins GS, Reitsma JB, Altman DG, Moons KG。透明报告个人预后或诊断的多变量预测模型(TRIPOD): TRIPOD声明。中华外科杂志,2015;32(3):344 - 344。
Magoc T, Salzberg SL. FLASH:快速调整短读长以改善基因组组装。生物信息学。2011;27(21):2957 - 63。
Myers R, Clark C, Khan A, Kellam P, Tedder R. HBV STAR中使用位置特异性评分矩阵从整个和亚基因组片段中提取乙型肝炎病毒基因分型。中华病毒学杂志,2006;87(Pt 6): 1459-64。
Tibshirani R, Bien J, Friedman J, Hastie T, Simon N, Taylor J, Tibshirani RJ。在套索型问题中丢弃预测器的强规则。中国生物医学工程学报。2012;32(2):366 - 366。
杨春华,李志强。基于稀疏偏最小二乘回归的同时降维和变量选择。中国生物医学工程学报。2010;32(1):1 - 2。
支持向量机分类器-支持向量机分类微阵列数据的一个综合java接口。BMC Bioinform. 2006;7(增刊4):S25。
高贵的WS。什么是支持向量机?生物技术学报。2006;24(12):1565-7。
黄CYJ。弹性网惩罚huber损失回归和分位数回归的半光滑牛顿坐标下降算法。中国计算机工程学报,2017;26(3):547-57。
普拉特JC。支持向量机的概率输出和正则似然方法的比较。剑桥:麻省理工学院出版社;1999.p . 61 - 74。
Steyerberg EW, Bleeker SE, Moll HA, Grobbee DE, moonkgm。预测模型的内部和外部验证:小样本中偏差和精度的模拟研究。临床流行病学杂志,2003;56(5):441-7。
董霞,何红,张伟,于东,王霞,陈艳。血清RASSF1A甲基化与AFP联合检测是HCC合并慢性HBV感染的一种有前景的非侵入性生物标志物。诊断病理学。2015;10:133。
陈松,张震,王颖,方梅,周娟,李颖,戴娥,冯志,王辉,杨震,李颖,黄霞,贾佳,李松,黄超,佟玲,肖欣,何颖,段勇,朱松,高晨。利用乙肝病毒准种模式深度测序和机器学习预测肝癌。中华流行病学杂志,2011;23(11):1887-96。
Dash S, Panda SK, Nayak NC。不同肝脏疾病中聚合白蛋白与血清结合的意义及其与乙型肝炎病毒感染的关系。中华胃肠病杂志。1990;5(1):16-24。
张强,尹杰,张艳,邓艳,季霞,杜艳,蒲蓉,韩艳,赵军,韩霞,张宏,曹刚。HLA-DP多态性可能通过与病毒突变相互作用影响慢性乙型肝炎病毒感染的结局。中国病毒学杂志,2013;87(22):12176-86。
乙型肝炎病毒转染的Hep G2细胞在复制HBV的细胞中表现出遗传改变和从头病毒整合。穆特决议2000;452(2):163-78。
蒋淑珍,高志勇,李涛,李玉娟,陈小梅,王玲,陆丰发,庄华。HBV基因型C的T3098C和T53C突变与HBV感染进展相关。生物环境科学,2009;22(6):511-7。
Jose-Abrego A, Roman S, rebelo PJ, de Castro V, Panduro A.乙型肝炎病毒(HBV)基因型混合、病毒载量和HBV合并人类免疫缺陷病毒患者的肝损伤前沿微生物。2021;12:640889。
汉农C, Krogsgaard K, Horal P, Lindh M.干扰素治疗患者乙型肝炎病毒基因型混合。中华流行病学杂志,2002;26(6):529 - 529。
谢艳,乙型肝炎病毒相关肝细胞癌。Adv Exp医学生物学2017;1018:11-21。
黄德坤,Kopaniszen M, Omagari K, Tanaka Y,方迪元,濑东威K,冯杰,黄飞,张艾,洪宇夫,黎春林,袁文明。乙型肝炎病毒逆转录酶变异对恩替卡韦治疗反应的影响。中华感染杂志,2014;21(5):701-7。
梁勇,刘超,栾晓泽,梁克勤,陈天明,徐志斌,张慧。基于L1/2惩罚的稀疏logistic回归在癌症分类中的应用。BMC bioinfo . 2013;14:198。
刘震,姜峰,田刚,王松,Sato F, Meltzer SJ,谭明。基于Lp惩罚的稀疏logistic回归生物标志物识别。中华生物医学杂志2007;6:e6。
Riley RD, Snell KI, Ensor J, Burke DL, Harrell FE Jr, Moons KG, Collins GS。开发多变量预测模型的最小样本量:第二部分-二进制和事件时间结果。中华医学杂志2019;38(7):1276-96。
Riley RD, Debray TPA, Collins GS, Archer L, Ensor J, Smeden M, Snell KIE。具有二元结果的临床预测模型的外部验证的最小样本量。中华医学杂志2021;40(19):4230-51。
确认
不适用。
资金
本研究由上海市卫生健康委创新群项目(2019CXJQ03)、国家自然科学基金项目(no . 81572072、81171664、11471082、61572139)、上海市科委项目(16JC1402600)和美国NIHR01GM120624资助。
作者信息
作者及隶属关系
贡献
FS, CG和JF构思了这项研究。JJ负责数据生成和手稿撰写。SQZ使用计算模型分析数据并撰写手稿。SC和MF对数据生成有贡献。XB, XL和SFZ对数据分析有贡献。DKW和AZ提供了香港数据集。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
中国上海东方肝胆外科医院伦理委员会批准了这项研究(批准号# EHBHKY2015-01-004)。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1。图S1:
单反相机无标定可靠性图(一个),并使用普氏校准(B).纵轴表示观测到的HCC患者在数据集中的比例,横轴表示预测的HCC患者比例。由于SLR输出每个样本是HCC患者的概率,我们直接给出校准图。我们使用“platt”进一步校准SSVM,并绘制了图。校准后的SLR结果与SSVM结果相似。
附加文件2。图S2:
通过稀疏logistic回归选择十个位点突变的HCC和CHB个体的核苷酸碱基频率。
附加文件3。图S3:
不同HBV基因型HCC/CHB患者使用SLR的分类结果一个不同λ基因型的HCC/CHB患者的分类结果。B.不同参数λ C基因型HCC/CHB患者的分类结果。
附加文件4。图S4:
3个临床变量与不同参数的SLR显示的HBV准种之间的关系。一个.不同λ区HBeAg与HBV准种的关系。B.当K = 4时,不同ηs的HBVDNA与HBV准种之间的相关性。C.当K = 2时,不同ηs下ALP与HBV准种间的相关性。
附加文件5。表S1:
三脚架清单。
附加文件6。表S2:
采用LR、SLR、SVM、SSVM和校准的SSVM对HCC/CHB进行分类。
附加文件7。表S3:
所选位置与三个临床参数相关:HBeAg, HBVDNA和ALP。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
贾,农协。,Zhang, S., Bai, X.et al。稀疏logistic回归揭示了HBV PreS准种与肝细胞癌之间的相关性。性研究J19114(2022)。https://doi.org/10.1186/s12985-022-01836-9
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12985-022-01836-9
关键字
- 肝细胞癌
- HBV准物种
- 下一代测序(NGS)
- 稀疏逻辑回归
- 总统地区