摘要
背景
最近的基因组序列技术揭示了一种新型的遗传重排,称为复杂结构变异(SVs)。先前的研究已经阐明了人类乙型肝炎病毒(HBVs)中的复杂SVs。在本研究中,我们研究了非人类灵长类动物hbv (NHPs)中复杂SVs的存在。
方法
使用PubMed搜索NHP HBV的核苷酸序列,并从数据库中检索基因序列。利用CLUSTALW程序和MAFFT对含有复杂sv的候选基因序列进行分析。进行了额外的生物信息学分析,以确定具有复杂SVs的菌株,并阐明NHP HBV菌株的特征。
结果
从数据库中鉴定出154株来自NHPs的HBV株。11株(7.1%)存在SVs和复杂SVs。3株长臂猿HBV (GiHBV)在前s1区出现了插入和缺失的复杂sv。一株GiHBV株具有6-nt的插入,这通常是针对人类HBV基因型a (HBV/ a)的核心区域,进一步的分析阐明了6-nt的插入不是由重组引起的,而是由简单的插入引起的。另一种黑猩猩HBV株在前s1区显示了复杂的SV,由人类HBV/E、g特异性多态性SV和额外的6-nt插入组成。
结论
在本研究中,如既往研究所示,除了人类HBV株外,还在NHPs的HBV株中观察到复杂的SVs。这些数据表明,复杂的sv也可以在肝病毒的其他成员中发现,并可能在它们的遗传多样性中发挥作用。
背景
全世界有超过2.57亿人感染了慢性乙型肝炎病毒(HBV), 20%至30%的慢性乙型肝炎病毒感染未经治疗的患者发展为肝硬化,这可能导致肝衰竭和肝细胞癌(HCC) [1].每年共有50万至120万人死于乙肝病毒感染,主要是由于肝硬化和HCC,后者每年造成32万人死亡[2].目前,HBV感染与疟疾、人类免疫缺陷病毒一样,是全球范围内严重的公共卫生问题之一。
HBV基因改变在病毒学和临床中都很重要。在HBV基因组中观察到的基因改变中,有报道的点突变,如前核突变或核心启动子突变,这些突变影响HBV相关肝病的发病机制[3.,4,5,6].此外,据报道,重组是重要的基因变化[7,8,9].
人类HBV是家族的原型Hepadnaviradae,又分为两属:Orthohepadnavirus会感染哺乳动物Avihepadnavirus会感染雀鸟[10].此外,最近的研究在两栖动物中发现了新的肝病毒[11],和鱼[12].对这些非人类肝病毒的研究非常重要。如乙肝病毒感染的初始步骤实验,最初是用鸭乙型肝炎病毒进行的,后来发现了在HBV感染中非常重要的因素——钠/牛磺胆酸共转运多肽(NTCP)受体[13,14].
最近的基因组序列技术揭示了一种新型的遗传重排,被称为复杂结构变异(SVs)。复杂sv是由由多个断点组成的基因序列定义的,这些断点的起源不能用单个末端连接或DNA交换事件来解释[15],实际上是由2个或2个以上的sv共同出现在同一个位点[16].最初使用未报告重排的独特HBV株[17],我们在人类HBV中发现了一种新的非典型形式的遗传变化,称为复杂SVs [18,19].此外,我们在人类HBV基因型A - H和非人类灵长类动物(NHPs)的HBV中发现了s1前区域的多态SVs [18].此外,最近的研究表明,在各种正肝病毒中观察到多态SVs [20.].本研究在前人报道人类HBV中存在各种类型的复杂SVs的基础上,旨在从NHPs中阐明HBV中存在复杂SVs。
方法
HBV基因序列搜索
使用PubMed检索NHP HBV的核苷酸序列,从DDBJ/EMBL/Genbank检索基因序列。此外,在PubMed数据库中发现了分析NHP HBV核苷酸序列的研究文章。
参考序列
以V00866 (HBV/A)为参考序列。此外,采用CLUSTALW程序确定人HBV/A、HBV/B、HBV/C、HBV/D、HBV/E、HBV/F、HBV/G、HBV/H、HBV/I的一致序列[21]分别分析150、31、168、78、37、38、13、30和3个全基因组序列。此外,使用CLUSTALW还确定了每个非人灵长类动物HBV的一致序列。
简单和复杂SVs的分析
通过对HBV/A参考序列(V00866)的多次比对来确定简单的典型sv。复杂sv是由存在多个断点的sv定义的,由删除、插入和重复的复杂混合组成[15,16].利用CLUSTALW程序和MAFFT对含有复杂SVs的候选基因序列进行分析[22],并与参考序列(HBV/A, V00866)进行比对。当序列中包含部分与参考序列相似度较低(有间隙和无间隙)时,采用NCBI BLAST 2.2.31 [23].然后结合NCBI BLAST分析的结果进行额外的人工检查,并根据以前发表的文章中复杂SVs的模式确定复杂SVs,作为参考[18,19].
系统发育分析和重组分析
使用MEGA软件version 6进行邻居连接方法的系统发育分析[24].进行1000个重复的自举、再采样和重建。利用集成在MEGA软件中的Kimura双参数模型进行遗传距离计算和成对距离比较。SIMPLOT程序3.5.1版本[25]用于分析HBV株的基因型间重组。
百分比同一性分析
采用MAFFT分析HBV株从人基因型A - I和NHPs的同源性百分比[22].
结果
来自NHPs的HBV株
从数据库和发表的文章中检索来自NHPs的HBV基因序列。从NHPs中获得154条HBV基因序列(109条全基因组序列和45条部分基因组序列)。其中,长臂猿HBV (GiHBV) 91例,黑猩猩HBV (ChHBV) 16例,猩猩HBV (OuHBV) 6例,大猩猩HBV (GoHBV) 6例。超过一半的基因序列来自GiHBV。关于NHP HBV基因序列的详细信息见附加文件1.使用63个来自人类和新型乙型肝炎病毒株的全基因组序列进行的系统发育分析表明,人类和新型乙型肝炎病毒株表现出不同的群体(附加文件)1:图S1)。采用MAFFT进行多对准分析[22],并将所有NHP HBV株与参考序列(V00866, human HBV/A,或AF046996, Woolly monkey HBV (WMHBV))和人类HBV基因型A ~ H一致序列,以及NHPs中的HBV进行比较。对于复杂SVs,既往报道中描述的70株具有复杂SVs的HBV株[18,19]被认为是搜索复杂sv的原型。
来自NHPs的HBV株中的sv
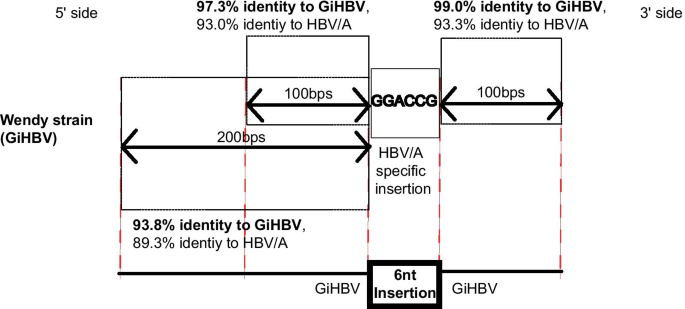
从NHPs中提取的HBV株中,有11株(7.1%)存在SVs1).从NHPs中检出7株(4.5%)典型简单sv。其中,4个(2.6%)是缺失,3个(1.9%)是插入。有趣的是,1株GiHBV (Wendy, AY330914)在核心区域出现了6-nt插入,这通常只在人类HBV/A中观察到(图3)。1).来自NHPs的所有其他HBV株均不具有6-nt插入(表6-nt插入)1).MAFFT鉴定百分比分析显示Wendy株6-nt插入的5个ʹ(100和200 bps)和3个ʹ(100 bps)位点均与HBV/A株没有高基因序列一致性,说明Wendy株6-nt插入不是由GiHBV与HBV/A重组引起的(图)。2,附加文件1:表S1-S3)。因此,Wendy菌株在6-nt插入的5ʹ和3ʹ端均显示出与GiHBV较高的基因同源性(图2)。2,附加文件1:表S1-S3),提示在Wendy菌株中观察到的HBV/A特异性6-nt核苷酸片段是插入。
Wendy株核心区6-nt插入5ʹ侧和3ʹ侧百分比同一性分析百分比身份分析的详细结果显示在附加文件中1:表S1-S3。GiHBV,长臂猿乙型肝炎病毒;HBV/A, HBV基因型A
分析了所有用于确定一致序列(150 HBV/A、31 HBV/B、168 HBV/C、78 HBV/D、37 HBV/E、38 HBV/F、13 HBV/G、30 HBV/H和3 HBV/I)的人HBV株的核心区是否存在6-nt插入。1株HBV/A株缺乏6-nt插入。进一步分析HBV株。在缺失的6-nt区5个ʹ侧和3个ʹ侧显示HBV/A序列。推测该菌株中6-nt插入缺失。因此,在151/152(99.3%)的人HBV/A株中,核心区域的6-nt插入是保守的。在HBV/B - I和NHPs的HBV中,只有Wendy株(1/445(0.2%))在核心区域有6-nt插入。
来自NHPs的HBV株中的复杂sv
3株GiHBV在前s1区与参考序列存在序列间隙。利用NCBI-BLAST分析gihbv中存在间隙的序列片段[23].BLAST搜索的结果显示,这些碎片来自该区域的5ʹ侧。因此,重排分为删除和插入两部分,如图所示。3..
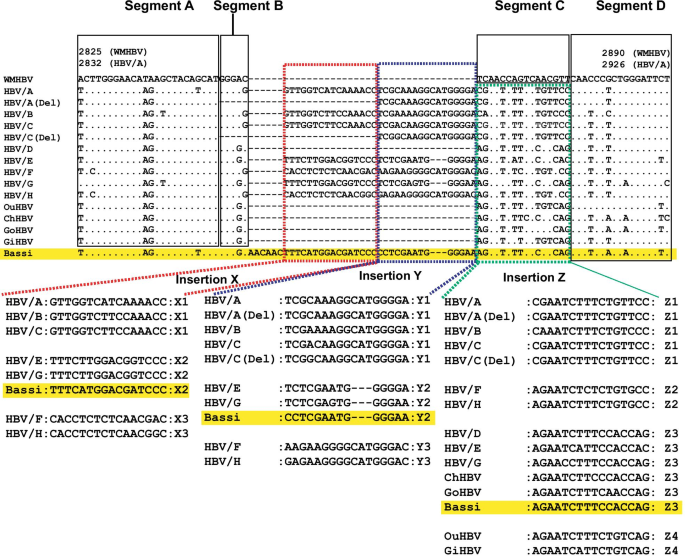
在之前的研究中[18],在人类HBV基因型A - H和NHP HBV的前s1区发现了多态sv。简单地说,重排由未知来源的插入(X, Y, Z)和删除WMHBV的C段组成,如图所示。4.因此,根据核苷酸序列一致性小于80.0%的百分比,将插入X、Y和Z分为X1 ~ X3、Y1 ~ Y3和Z1 ~ Z4。部分HBV/A、C株出现X和B段缺失,表现为HBV/A (Del)和HBV/C (Del)。人类HBV/D和非人类灵长类HBV不具有插入X和y。对于插入Z, HBV/D、E、G、ChHBV和GoHBV表现出相似的模式(Z3),而OuHBV和GiHBV表现出不同的模式(Z4)。综合来看,HBV/A→C、HBV/E→G、HBV/F→H呈相似模式;X1 + y₁+ Z1, X2 + Y2 + Z3,分别和X3 + Y3 + Z2 [18].这些变异在所有人类hbv和NHP hbv中基本保守。而1株ChHBV (Bassi, AB046525)在前s1区表现出复杂的sv。如图所示。4人类HBV/D和NHP HBV在X + Y区域具有缺失型模式。在Z区,Bassi株呈human HBV/D、E、G、ChHBV、GoHBV的共同模式。此外,Bassi菌株在插入X区域的5ʹ侧含有Bassi特异的6-nt插入“AACAAC”,如图所示。4.在其他人类或NHP hbv中未观察到这种插入。因此,该区域的Bassi序列由2个不同的插入组成:X2 + Y2,是人类HBV/E、G的特征,另一个是“AACAAC”。没有人HBV序列显示类似的模式。
人类和非人灵长类动物(NHP) hbv在s1前区域的多态性结构变异(SVs),以及Bassi菌株中观察到的复杂SVs。之前的一项研究分析了在人类和NHP hbv中观察到的独特多态SVs [18],并以毛猴HBV (WMHBV)为参考建立了多个比对。此外,在s1前区观察到Bassi应变特异性复合物sv。A段、B段、D段为保守区。插入X、Y、Z为多态性sv,分别分为X1-X3、Y1-Y3、Z1-Z4 [18].(Del)显示仅在人类HBV/A和HBV/C中观察到的缺失型小变异(B段缺失和插入X)。Bassi菌株以黄色突出显示。HBV/A,乙型肝炎病毒基因型A;德尔、删除;OuHBV,猩猩乙型肝炎病毒;ChHBV,黑猩猩HBV, GoHBV,大猩猩HBV;GiHBV,长臂猿HBV
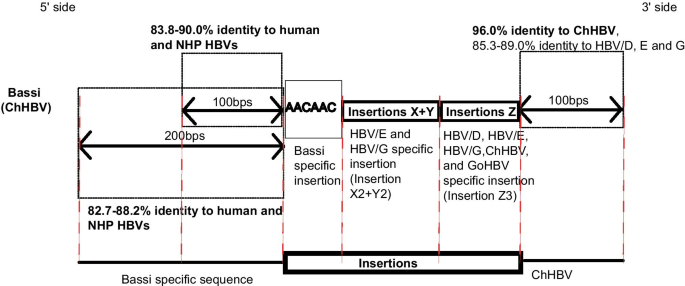
对该复杂SVs(100和200 bps)的5个ʹ侧的同源性分析显示Bassi具有Bassi特异性序列,这表明无论是人类HBV毒株还是NHP HBV毒株与Bassi毒株的同源性均不高(图2)。5,和附加文件1:表S4-S6)。在重组分析中未观察到与人HBV基因型A-I或NHP HBV重组的明确证据。此外,对该复合SVs的3ʹ侧(100 bps)进行了百分身份分析,结果显示Bassi株属于ChHBV(图2)。5,和附加文件1:表S4-S6)。在上述的前s1区,每个HBV基因型和NHP HBV都有特异性的多态SVs/复杂SVs, Bassi菌株也有特异性的复杂SVs,由2个插入组成。Bassi菌株在复杂SV位点的3个ʹ侧具有与黑猩猩相容的基因序列,在复杂SV位点的5个ʹ侧具有自己特定的基因序列。
Bassi品系复杂sv 5ʹ和3ʹ侧同源性百分比分析ChHBV,黑猩猩乙型肝炎病毒;HBV/D, HBV基因型D;NHP,非人灵长类。详细的百分比身份分析显示在附加文件1:表S4-S6
讨论
在人类和小鼠基因组分析中首次报道了复杂的SVs [15,16].HBV中的复杂SVs首次在人类HBV中报道,此外,在人类和NHP HBV的前s1区观察到独特的多态SVs [18,19,20.].在本研究中,对NHP HBV中的SVs和复杂SVs进行了搜索和澄清。3株GiHBV在前s1区(参考株V00866的nt 3093-3102)显示了由插入和删除组成的复杂sv。在之前的人类HBV分析中,超过90%的复杂SVs出现在nt 1500-2000,其中包含X和Core区域[19].复合物sv在gihbv中的位置和插入基序与在人类HBV中观察到的不相似。复杂SVs的插入和删除模式是人类HBV中观察到的最常见的复杂SVs模式。
分析表明,Wendy菌株中6-nt的插入不是由重组引起的,而是由SV引起的。Wendy中6-nt插入的5个ʹ和3个ʹ位点均未显示出与人类HBV/A的高序列同源性,而与gihbv的高序列同源性。Bassi pre-S1插入的5个ʹ和3个ʹ位点与HBV/E和HBV/G的序列一致性均不高;因此Bassi的pre-S1区域是由2个插入组成的复杂sv。温蒂和巴西出现SV和复杂SV的原因尚不清楚;然而,据推测,2个区域(HBV/A的Core区域的6-nt插入,以及HBV中观察到多态sv的前s1区域)是可能发生核苷酸断点的独特区域。关于前s1区复杂SV位点5ʹ侧的Bassi特异性序列,此前报道过Bassi株与其他人类和NHP hbv在Core区域的独特部分分化[26].本研究明确了Bassi株在部分独特的Bassi特异性基因序列的3ʹ端存在复杂的SVs。
前s1区多态SVs是非常独特的结构。既往研究报道该地区HBV/D与NHP HBV相似,HBV/E与HBV/G相似[27,28].先前的研究[18进一步阐明了pre-S1区域的遗传结构。这些多态的sv导致了人类和NHP hbv长度的差异。人HBV基因型与NHP HBV长度的差异(人HBV/ B、C、F、H、I长度3215 nt;3212 nt human HBV/E长度;人类HBV/D和NHP HBV的3182 nt长度是由s1前区域的多态sv引起的。此外,在Core区域插入6 nt和pre-S1结构(HBV/A、B、C共有)决定了人类HBV/A的长度为3221 nt,在Core区域插入36 nt和pre-S1结构(HBV/E和G共有)决定了人类HBV/G的长度为3248 nt。HBV中的SVs尚未被广泛研究;然而,我们的研究以及之前的研究[18,20.已经阐明了它们的重要性。SVs表现为多态性改变(在某些HBV基因型中观察到)和散发性改变(在3个gihbv, Bassi和Wendy中观察到)。这些sv也暗示基因组断点同时发生多态和零星。
乙型肝炎病毒和非乙型肝炎病毒之间的跨物种传播被认为是乙型肝炎病毒的起源,以及其他理论[10].s1前sv的多态性提供了一个可能的理论基础。如上所述,多态的pre-S1 sv高度保守。在NHP hbv中,Pre-S1缺失类型总体上是保守的。在人类中,仅在HBV/D中观察到pre-S1缺失类型。例如,在印度尼西亚,HBV/B和HBV/C作为人类HBV基因型分布[29];HBV/B和C均在s1前区存在插入X和Y,且印尼NHP HBV中普遍存在s1前X + Y缺失型。这表明在印度尼西亚,NHP HBV不太可能直接传播给人类HBV。此外,印度尼西亚的NHP HBV与地理位置遥远的HBV/D之间的接触不太可能。最近的一项研究报道,从新石器时代人类骨骼(7000年和5000年前)中提取的2株HBV病毒株与非洲NHP中的HBV病毒株关系最密切,从中世纪人类骨骼(3000年前)中提取的1株HBV病毒株属于HBV/D [30.].这项研究的数据与我们的结果一致。如果发生跨种传播,考虑到pre-S1区域普遍缺乏插入X和Y的多态性,第一个人类HBV应该与来自NHP的HBV相似,HBV/D应该是第二个人类HBV。此外,在Bassi株中观察到的前s1区复杂的sv可能提示了人类HBV多态性多样性的起源。在人HBV/A-C、E和G、F和H中观察到的多态SVs可能已经插入到X和Y缺失的原型中,就像在Bassi菌株中观察到的那样,并在此后一直保持。目前,这些解释可能不足以澄清乙肝病毒的起源。其余未回答的问题可能会逐步澄清的数据,基因序列的正肝病毒进一步积累。
如前一篇文章所述,复杂的sv可以重新排列基因组以创建新的蛋白质,将启动子或增强子洗牌成新的调控配置[15].对人HBV毒株中复杂SVs的实验研究表明,HBV基本核心启动子中与肝细胞核因子1 (HNF1)结合位点的复杂SVs可引起乙型肝炎核心蛋白在细胞核和核周的积累[17],另外,在感染含有复杂SVs的HBV株的患者肝脏病理中也观察到同样的现象。实验数据还表明,含有HNF1结合位点的复杂SVs结构具有更高的前基因组RNA和前S/S RNA水平[17,31].复杂的SVs可以通过影响转录和蛋白质产生来调节HBV的病理生物学。进一步的研究可能会阐明复杂SVs在HBV中的作用。
数据和材料的可用性
本研究中使用和/或分析的数据集可根据合理要求从通讯作者处获得。
缩写
- SV:
-
结构变化
- 乙肝病毒:
-
乙型肝炎病毒
- 额定马力:
-
非人灵长类动物
- GiHBV:
-
长臂猿乙肝病毒
- ChHBV:
-
黑猩猩乙肝病毒
- OuHBV:
-
猩猩乙肝病毒
- GoHBV:
-
大猩猩乙肝病毒
参考文献
https://www.who.int/news-room/fact-sheets/detail/hepatitis-b.
乙型肝炎病毒流行病学、疾病负担、治疗以及当前和新出现的预防和控制措施。中华流行病学杂志2004;11:97-107。
Carman WF, Jacyna MR, Hadziyannis S, Karayiannis P, McGarvey MJ, Makris A, Thomas HC。突变预防慢性乙型肝炎感染患者乙型肝炎e抗原形成《柳叶刀》杂志。1989;2:588 - 91。
冈本H, Tsuda F, Akahane Y, Sugai Y, Yoshiba M, Moriyama K, Tanaka T, Miyakawa Y, Mayumi M. e抗原抗体携带者e抗原阴性表型核心启动子突变的乙型肝炎病毒中国病毒学杂志,1994;68:8102-10。
Orito E, Mizokami M, Sakugawa H, Michitaka K, Ishikawa K, Ichida T, Okanoue T, Yotsuyanagi H, Iino S. B型和c型乙型肝炎病毒临床和分子生物学差异的病例对照研究肝脏病学。2001;33:218-23。https://doi.org/10.1053/jhep.2001.20532.
高俊华,陈普军,赖美民,陈德生。乙型肝炎病毒基础核心启动子突变增加乙型肝炎携带者发生肝细胞癌的风险。胃肠病学杂志。2003;124:327-34。https://doi.org/10.1053/gast.2003.50053.
Bollyky PL, Rambaut A, Harvey PH, Holmes EC。不同基因型乙型肝炎病毒序列的重组。中华药理学杂志,1996;42(2):377 - 382。
菅内F, Orito E,一田T,加藤H, Sakugawa H, Kakumu S,石田T, Chutaputti A, Lai CL, Gish RG,等。基因型B与基因型c重组的乙型肝炎病毒流行病学和病毒学特征。消化病学。2003;124:925-32。https://doi.org/10.1053/gast.2003.50140.
Araujo NM。全球乙型肝炎病毒基因间型重组:概述。感染Genet Evol 2015; 36:500-10。https://doi.org/10.1016/j.meegid.2015.08.024.
李志刚,李志刚,李志刚。乙型肝炎病毒和丁型肝炎病毒的起源和演变。冷泉港展望医学。2016;6:a021360。https://doi.org/10.1101/cshperspect.a021360a021360.
迪尔JA,加缪AC,利里JH,迪吉亚罗纳多F,霍尔姆斯EC,吴恩达TF。来自鱼类和两栖动物的独特病毒谱系揭示了肝病毒复杂的进化史。中国病毒学杂志,2016;90:7920-33。https://doi.org/10.1128/JVI.00832-16.
Hahn CM, Iwanowicz LR, Cornman RS, Conway CM, Winton JR, Blazer VS.白色吸盘中新型肝病毒的特征描述(Catostomus commersonii)产于美国五大湖地区。中国病毒学杂志,2015;89:11801-11。https://doi.org/10.1128/JVI.01278-15.
Glebe D, Urban S.参与肝病毒进入的病毒和细胞决定因素。世界胃肠杂志。2007;13:22-38。https://doi.org/10.3748/wjg.v13.i1.22.
闫红,钟刚,徐刚,何伟,景智,高志,黄勇,齐勇,彭波,王辉,等。牛磺胆酸钠共转运多肽是人乙型和丁型肝炎病毒的功能性受体。电子学报。2012;3:e00049。https://doi.org/10.7554/eLife.00049.
昆兰AR,霍尔IM。生殖系和体细胞基因组复杂结构变异的特征。《趋势》,2012;28:43-53。https://doi.org/10.1016/j.tig.2011.10.002.
王凯,王晓燕,王晓燕,王晓燕,王晓燕。小鼠基因组结构变异的精细尺度结构研究。中国生物工程学报。2012;13:R18。https://doi.org/10.1186/gb-2012-13-3-r18.
杨春梅,李志刚,李志刚,李志刚,李志刚。慢性乙型肝炎病毒基因型乙型肝炎病毒ⅰ型突变的研究进展。中华乙型肝炎病毒杂志,2005;29(4):344 - 344。https://doi.org/10.1128/JVI.79.22.14404-14410.2005.
Fujiwara K, Matsunami K, Iio E, Nojiri S, Joh T. HBV基因组的新型非典型遗传重排被称为“复杂结构变异”。病毒决议2017;238:84-93。https://doi.org/10.1016/j.virusres.2017.06.009.
藤原K,松浦K,松美K, Iio E, Nojiri S.乙型肝炎病毒复杂结构变异的特征。BMC微生物学。2018;18:202。https://doi.org/10.1186/s12866-018-1350-1.
藤原K,松浦k,松美K, Iio E,长仓Y,野尻S,等。被称为“结构变异多态性”的新型遗传重排有助于矫正肝病毒的遗传多样性。病毒。2019;11(9):871。
汤普森JD,希金斯DG,吉布森TJ。CLUSTAL W:通过序列加权、位置特定的间隙惩罚和权重矩阵选择,提高渐进式多序列对齐的灵敏度。核酸研究,1994;22:4673-80。
Katoh K, Standley DM. MAFFT多序列比对软件版本7:性能和可用性的改进。中国生物医学杂志,2013;30:772-80。https://doi.org/10.1093/molbev/mst010.
Altschul SF, Madden TL, Schaffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ。gapping BLAST和PSI-BLAST:新一代蛋白质数据库搜索程序。核酸研究,1997;25:3389-402。
Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:分子进化遗传学分析版本6.0。中国生物医学杂志,2013;30:2725-9。https://doi.org/10.1093/molbev/mst197.
Lole KS, Bollinger RC, Paranjape RS, Gadkari D, Kulkarni SS, Novak NG, Ingersoll R, Sheppard HW, Ray SC.来自印度c亚型感染血清转换者的人类免疫缺陷病毒1型全长基因组,有亚型间重组的证据。中国病毒学杂志,1999;73:152-60。
Starkman SE, MacDonald DM, Lewis JC, Holmes EC, Simmonds P.非人类灵长类动物乙型肝炎病毒基因型的地理和物种关联。病毒学。2003;314:381 - 93。
高桥K,三次郎S,王子AM。来自中非黑猩猩的新型乙型肝炎病毒株(Pan troglodytes troglodytes)具有不同寻常的核心蛋白抗原性。Intervirology。2001;44:321-6。https://doi.org/10.1159/000050065.
Utsumi T, Wahyuni RM, Lusida MI, Yano Y, Priambada NP, Amin M, Purwono PB, Istimagfiroh A, Soetjipto, Brule A,等。印度尼西亚加里曼丹中部长臂猿和看护人乙型肝炎病毒的全基因组特征和系统发育分析。中国生物医学工程学报。2015;https://doi.org/10.1007/s00705-014-2323-9.
Yano Y, Utsumi T, Lusida MI, Hayashi Y.印度尼西亚乙型肝炎病毒感染情况。世界胃肠杂志。2015;21:10 14 - 20。https://doi.org/10.3748/wjg.v21.i38.10714.
Krause-Kyora B, Susat J, Key FM, Kuhnert D, Bosse E, Immel A, Rinne C, Kornell SC, Yepes D, Franzenburg S,等。新石器时代和中世纪病毒基因组揭示了乙型肝炎的复杂进化。Elife, 2018;7: e36666。https://doi.org/10.7554/eLife.36666.
Gunther S, Piwon N, Iwanska A, Schilling R, Meisel H, Will H.免疫抑制严重肝病患者乙型肝炎病毒核心启动子/增强子II突变的类型、患病率和意义中国病毒学杂志1996;70:8318-31。
确认
不适用。
资金
本研究由JSPS资助(资助号19K08427)。
作者信息
作者及隶属关系
贡献
K.F.概念化;k.f.和Y.N.方法论;K.F, y.n.k.m, and e.i.,形式分析,数据管理;K.F.和Y.N.写作-初稿准备;K.F., y.n.y.t., h.k.,写作,评审和编辑,K.F.项目管理,资金获取。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们没有竞争利益。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1。
人类HBV序列列表。非人灵长类hbv名单。表S1。Wendy、人类HBV/A- i和非人灵长类HBV在核心区域HBV/A特异性6nt插入5 '侧100 bp处的一致性百分比。表S2。Wendy、人类HBV/A- i和非人灵长类HBV在核心区域6nt插入5 '侧200 bp HBV/A特异性位点的一致性。表S3。Wendy、人类HBV/A- i和非人灵长类HBV在核心区域6nt插入3 '侧100 bp内HBV/A特异性的一致性百分比。表S4。Bassi、人类HBV/A-I和非人灵长类HBV在多态preS1区域5 '侧100 bp的同源性百分比表S5。Bassi、人类HBV/A-I和非人灵长类HBV在多态preS1区域5 '侧200 bp的同源性。表S6。Bassi、人类HBV/A-I和非人灵长类HBV在多态preS1区域3 '侧100 bp的同源性百分比图S1。采用邻居连接法对人类和非人灵长类(NHP)乙型肝炎病毒(HBV)进行了系统发育分析。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
长仓,Y,藤原,K,松浦,K。et al。非人灵长类乙型肝炎病毒的复杂结构变异。性研究J18, 200(2021)。https://doi.org/10.1186/s12985-021-01667-0
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12985-021-01667-0
关键字
- 乙型肝炎病毒
- 非人灵长类动物
- 复杂的结构变异