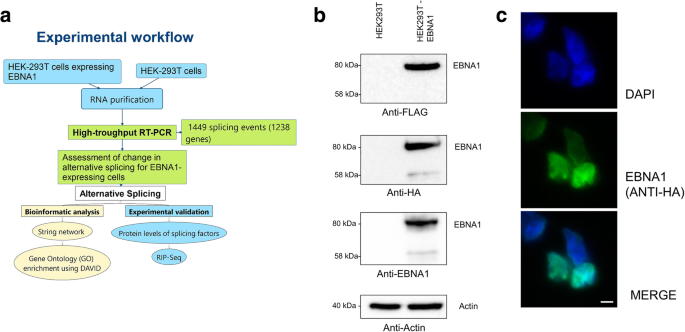
生成稳定的EBNA1 HEK293T细胞
利用MSCV-N EBNA1质粒制备表达EBNA1- flag - ha蛋白的稳定HEK293T细胞。MSCV-N EBNA1是来自Karl Munger的礼物(附加基因质粒# 37954)[29]。转染后,用5 μg/mL嘌呤霉素孵育20天。为了保持细胞的选择性,将细胞保存在3 μg/ml嘌呤霉素- dmme -10%胎牛血清中。
Western blot验证EBNA1表达
HEK293T细胞和表达EBNA1-FLAG-HA的HEK293T稳定细胞在T-75烧瓶中融合生长,在1500 rpm, 5 min下胰蛋白酶化并成粒。将细胞重悬于RIPA Buffer (1% Triton X-100, 1%脱氧胆酸钠,0.1% SDS, 1 mM EDTA, 50 mM Tris-HCl pH 7.5和完全蛋白酶抑制剂(ROCHE))中,用13%振幅,5 s的冰上超声裂解2次。细胞碎片在13000 RPM, 4°C, 10 min下成球。如果染色体DNA在离心后仍然漂浮,则进行第二次超声和离心处理。采用标准Bradford法(Thermo Scientific comasassie protein assay)对裂解液进行三份的总蛋白剂量测定。取适量的蛋白质在水中稀释至10微升,用1倍(终浓度)的Laemmli缓冲液补全,在95°C下加热5分钟。样品上载10% sds -聚丙烯酰胺凝胶,在150 V下进行电泳。将凝胶转移到聚偏二氟乙烯(PVDF)膜上,温度为4℃,温度为1 h15,温度为100 V。将膜在5%的TBS-T (10 mM Tris-HCl pH 8.0, 220 mM NaCl, 0.1% Tween 20)中阻断,室温下1小时。将小鼠抗Flag抗体(Sigma Aldrich)、抗HA或抗EBNA1抗体(Santa Cruz Biotechnology)在2.5%的牛奶/PBS中稀释为1:1000 (Flag, HA)或1:100 (EBNA1),用TBS-T洗涤后,与膜一起在4°C的潮湿室中孵育过夜。 The membrane was washed 3 times in TBS-T and incubated with a sheep anti mouse-HRP secondary antibody 1:5000 (Amersham Biosciences) during 1 h in a humid chamber at room temperature. Membrane was washed 3 times with TBS-T and one time with PBS. Bound antibodies were revealed using an enhanced chemiluminescence (ECL) kit (Perkin Elmer) and scanned on ImageQuant LAS4000 (GE Healthcare Life Science). Mouse anti-β-actin loading control (Sigma) was done on the same membrane after stripping the membrane by boiling it in PBS for 1 min. The procedure was then the same from the blocking upon revelation using the anti-β-actin antibody diluted 1:2000 (in 2.5% milk/PBS) and the secondary anti-mouse (1:5000) antibody from Amersham.
稳定表达EBNA1 HEK293T的免疫荧光研究
前一天,将稳定表达EBNA1-FLAG-HA蛋白的HEK293T细胞以5 × 104个/孔的密度接种于24孔板。用PBS洗涤细胞两次,在室温下用4%多聚甲醛和4%蔗糖在PBS中固定20分钟。在室温下,用0.15% triton X-100在PBS中渗透5分钟,然后用10%正常山羊血清(Wisent)阻断细胞。抗ha抗体(Santa Cruz Biotechnologies)室温孵育4小时,检测EBNA1-FLAG-HA蛋白。细胞洗涤后,用DyLight 488标记的山羊抗小鼠二抗(ThermoFisher Scientific)在黑暗中孵育1小时。1 μg/ml Hoechst染色,室温下染色15 min。用SlowFadeGold上载介质(Life Technologies)将盖玻片安装在载玻片上,然后使用Nikon Eclipse TE2000-E可见光/表观荧光倒置显微镜,使用Hoechst和DyLight 488带通滤光片进行荧光显微镜观察。
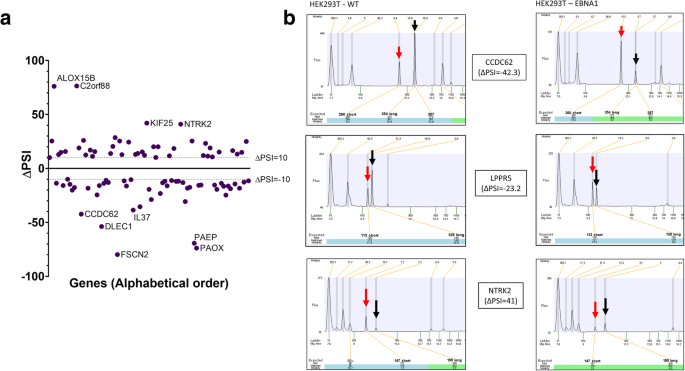
AS事件的高通量RT-PCR筛选
表达EBNA1-FLAG-HA的HEK293T和HEK293T细胞在融合和成粒后生长。按照制造商的方案,使用TRIzol (Invitrogen)和氯仿对细胞颗粒进行总RNA提取。回收水层,与一体积70%乙醇混合,直接应用于RNeasy Mini Kit色谱柱(Qiagen)。柱上的dna酶处理和总RNA回收按照制造商的方案进行。使用Agilent 2100生物分析仪(Agilent Technologies)评估RNA完整性。用转录逆转录酶(Transcriptor)、随机六聚体、dNTPs (Roche Diagnostics)和10单位RNAse OUT (Invitrogen)按照生产商的方案对2.2 μg总RNA进行反转录,总体积为20 μl。所有正向和反向引物分别重悬于20-100 μM的Tris-EDTA缓冲液(IDT)中,并在RNase无dna水(IDT)中稀释为一对引物至1.2 μM。在10 μL终体积中对10 ng cDNA进行终点PCR反应,每个dNTP为0.2 mmol/L, MgCl2为1.5 mmol/L,引物为0.6 μmol/L,铂Taq DNA聚合酶(Invitrogen)为0.2单位。95°C初始孵育2分钟,随后在94°C 30秒、55°C 30秒和72°C 60秒进行35次循环。在72°C下孵育2分钟完成扩增。 PCR reactions were carried on thermocyclers GeneAmp PCR System 9700 (ABI), and the amplified products were analyzed by automated chip-based microcapillary electrophoresis on Caliper LC-90 instruments (Caliper LifeSciences). Amplicon sizing and relative quantitation was performed by the manufacturer’s software, before being uploaded to the LIMS database.
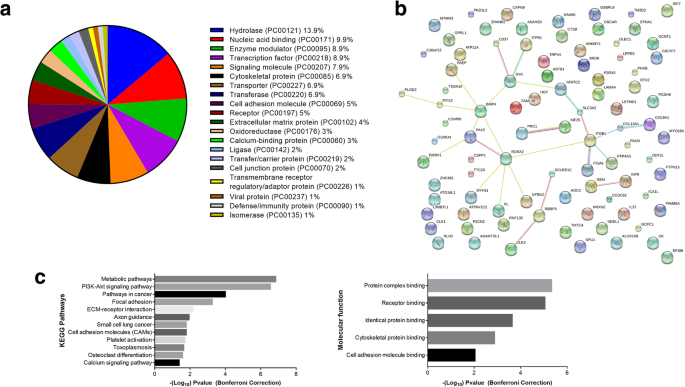
线网络
使用STRING数据库[30.[10.0]版本,基因提交生成蛋白-蛋白相互作用网络智人interactome。P蛋白-蛋白相互作用富集的-值直接从STRING分析中恢复。
基因本体分析
注释、可视化和集成发现数据库(DAVID) [31使用Bonferroni修正的6.8版本。参考背景由高通量RT-PCR分析的所有基因组成,以考虑癌症相关基因的偏倚。
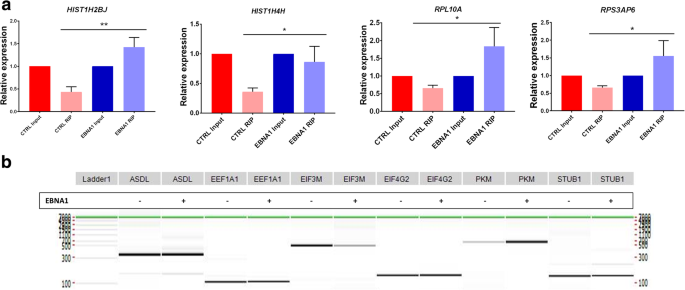
剪接因子的蛋白水平
如前所述,HEK293T细胞和表达EBNA1-FLAG-HA的HEK293T稳定细胞在RIPA缓冲液中经融合、胰蛋白酶化和裂解培养。裂解液加量并装入10%丙烯酰胺凝胶。抗体以1:1000稀释为SF1 (Abgent), Rbm23 (Abgent);hnRNPA1(兔抗ASASSSQRGR肽多克隆,见[32])、Fox-2 (Abcam)和1:100的SRSF3 (Santa Cruz Biotechnologies)。前4个抗体采用1:5000稀释的兔抗二抗(Cell Signaling Technology), SRSF3抗体采用1:5000稀释的小鼠抗(Cell Signaling Technology)。本文给出的结果是两到三个独立实验的代表性结果。
RNA immunoprecipation
HEK293T细胞和表达EBNA1-FLAG-HA的HEK293T稳定细胞在P150培养皿中汇合生长,收获并成粒。然后在冷PBS中洗涤两次,并以相同体积的多体裂解缓冲液(10 mM Hepes pH 7.0, 100 mM KCl, 5 mM MgCl2, 0.5% NP-40, 1 mM DTT, 100 u/mL RNase抑制剂和完全蛋白酶抑制剂(罗氏))重悬。在冰上孵育5分钟后,在- 80°C冷冻以完成裂解。快速解冻后,试管在4°C, 13000 g下离心20分钟。上清液采用Bradford法(Thermo Scientific comasmase Protein assay)加药,一式三份。每个IP取50 μL Anti-Ha matrix (Roche), 13400 rpm离心1 min,丢弃上清。用1 mL NT2缓冲液(50 mM Tris-HCl, pH 7.4, 150 mM NaCl, 1 mM MgCl2, 0.05% NP-40)洗涤微珠,13400 rpm离心1 min,离心5次。然后用900 μL NET-2缓冲液(NT2缓冲液中添加20 mM EDTA、pH 8.0、1 mM DTT和100 U/mL RNase抑制剂)和100 μL (NT-2缓冲液调节至总蛋白量为2.5 mg)分别加入对照或表达EBNA1细胞的裂解液中。试管倒置几次,在13400 rpm下离心1分钟,4°C,去除100 μL作为输入,以评估RNA降解。所有试管在4°C的转轮上孵育过夜。 Beads were precipitated at 5000 g for 5 min, 4 °C and washed five times with ice-cold NT2 buffer. Upon the last washing, beads were resuspended in 90 μL of NT-2 buffer and 10 μL of RQ1 DNase and incubated at 37 °C for 30 min. Input tubes were directly added 10 μL of RQ1 DNase. Dnase-treated IP were diluted with 1 mL NT-2 buffer, centrifuged and supernatant was discarded. Beads were resuspended in 150 μL of proteinase K buffer (1% SDS, 1.2 mg/mL proteinase K from Boehringer Mannheim), and input tubes were directly added SDS and proteinase K to the same volume. Tubes were incubated at 55 °C during 30 min with inversion at every 10 min. RNA was then extracted with phenol-chloroform, followed by a second chloroform extraction step. Precipitation of RNA using 272 mM ammonium acetate, 122 mM LiCl and 27 μg/mL glycogen in ice-cold ethanol was carried out during 2 h at − 80 °C and followed by high-speed centrifugation. Pellets were washed in 75% ethanol, ethanol was thoroughly removed, and RNA was resuspended in water.
RIP-Seq库准备
在Agilent Nano芯片(目录号5067-1511)上进行质量(输入)和数量(输入和IP)评估。来自对照和EBNA1表达细胞的输入均显示出良好的RNA完整性(RIN分别为8.9和9.2),因此由于实验程序,底层RNA未被降解。按照制造商的协议,使用Illumina Ribo-Zero rRNA去除试剂盒对RNA进行核糖体去除。利用Illumina SSV21106试剂盒从9 μl的去核RNA中构建RNA-seq文库。使用Agilent DNA HS芯片(目录号5067-4626)评估文库质量。按照Illumina Kappa文库定量方案,采用qPCR进行文库定量。HEK293T和HEK293T- ebna1文库进行多路转换,测序使用Illumina HiSeq 4000在McGill大学和gsamome quacimbec创新中心测序服务进行100 bp配对端读取。
RIP-Seq分析
测序后,对照和EBNA1 RIP文库分别获得22,887,816和21,858,499个reads,平均质量评分分别为39和38。首先使用Trimmomatic (Galaxy Tool Version 0.32.2)修剪与这两种条件相对应的读取并删除适配器。使用STAR(版本2.5.1b)将reads与来自ENSEMBL数据库的注释版本89的hg38人类基因组进行比对。通过samtools(版本1.3.2)按名称对Reads进行排序,并使用rmdup函数去除PCR和测序重复。然后使用samtools(版本1.3.2)和自定义的awk命令删除未映射或映射到scaffold及其在头中的相应ID,因为它们不适合使用RIPSeeker进行分析:
Samtools视图-h文件。b一个m | awk ‘((NR < =197 && length($2) < 10) || (NR > =198 && length($3) < 5 && $3! ~ /[*]/)){print $0}’ > file.out.sam.
然后使用samtools将输出文件转换为bam格式,并使用R包RIPSeeker(版本1.10.0)进行分析。之所以选择RIPSeeker包,是因为它是专门设计用于在RIP实验中分配峰值的唯一程序之一[33]。在HEK293T- ebna1和HEK293T上使用RIPSeeker,以HEK293T作为对照,以确定分配峰的可靠性。
数据可用性
本文中讨论的数据已发表在NCBI的基因表达综合数据库[34],可通过GEO系列注册号GSE107808 (https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE107808)。
qPCR
在1.7 μg RNA(剪接因子上的qPCR)、600 ng和300 ng (RIP-qPCR,分别复制1和2-3个)上用转录逆转录酶、随机六聚体、dNTPs(罗氏诊断公司)和10单位RNAse OUT (Invitrogen公司)按照制造商的方案进行反转录,总体积为10 μl。所有正向和反向引物分别重悬于20-100 μM的Tris-EDTA缓冲液(IDT)中,并在RNase无dna水(IDT)中稀释为一对引物至1 μM。在CFX-96型热循环仪(BioRad)上,用5 μl 2X iTaq Universal SYBR Green Supermix (BioRad)、10 ng (3 μl) cDNA(剪接因子qPCR)或5 ng (3 μl) cDNA (RIP-qPCR)和200 nM (2 μl)最终引物对溶液,在10 μl孔板上进行定量PCR (qPCR)反应。使用以下循环条件:95℃下3 min;50个循环:在95℃下15 s,在60℃下30 s,在72℃下30 s。使用qBASE框架计算相对表达水平。对于每次PCR运行,在没有模板的情况下对每个引物对进行对照反应,这些反应一致为阴性。
As-PCR
使用Qiazol (Qiagen)按照制造商的方案从细胞中提取总RNA。用1 μg RNA和4 μl iScript Reverse transcription Supermix (BIO-RAD)进行反转录,最终体积为20 μl, PCR程序如下:25°C, 5 min;46℃下20min;在95°C下1分钟。AS特异性引物(IDT)设计为只扩增一个ASE,并在1 μM下重悬在一起。使用的引物和预测的扩增子可在附加文件中找到1表S2。每个反应由ThermoPol缓冲液(NEB)、dNTPs、引物、cDNA和TAQ (NEB)组成,按照以下程序孵育:在94℃下孵育2 min;34次循环:94°C 30 s;55℃下30 s;72℃下1 min;72°C下2分钟(最终伸长率)。PCR产物采用Caliper LC-90毛细管电泳(Caliper LifeSciences)进行分离。