- 研究
- 开放获取
- 发表:
证据表明洛克恩病毒(家族Peribunyaviridae)是一种重组病毒,它的大、小基因组片段来自主干病毒,中基因组片段来自一种未被发现的病毒
病毒学杂志体积15,文章号:122(2018)
摘要
背景
Lokern病毒(LOKV)是一种节肢动物传播的病毒,属于该属Orthobunyavirus(家庭Peribunyaviridae).该属病毒均为三段式单链负意义RNA基因组,三个RNA片段分别为小、(S)、中(M)和大(L)。LOKV病毒M RNA片段的559 nt.区域已被测序,尚未获得其S或L RNA片段的序列数据。本研究的目的是对LOKV的基因组进行测序。
方法
采用无偏高通量测序、cDNA末端5 '和3 '快速扩增、逆转录-聚合酶链反应和Sanger测序对LOKV基因组进行全测序。
结果
LOKV的S和L RNA片段分别由952和6864 nt组成,与主漏病毒(MDV)的相应区域具有99.0%的核苷酸同源性。相比之下,4450-nt。M RNA片段与MDV相应区域的核苷酸同源性仅为59.0%,与Genbank数据库中所有其他M RNA片段序列的核苷酸同源性不超过72.7%。系统发育数据支持这些发现。
结论
这项研究提供了证据,证明LOKV是一种天然重组病毒,其S和L RNA片段来自MDV,其M RNA片段来自一种未被发现的,可能已经灭绝的病毒。全基因组序列数据的可用性有助于对病毒和病毒感染的准确检测、识别和诊断,对于具有分段基因组的病毒尤其如此,因为在不完全序列数据可用时,很难甚至不可能区分重组体及其前体。
背景
Lokern病毒(LOKV)是该属中一种特征较差的病毒Orthobunyavirus(家庭Peribunyaviridae).该属以前曾被分为血清组,LOKV属于Bunyamwera (BUN)血清组[1].该血清群中的其他病毒包括巴泰病毒(BATV)、布尼亚姆韦拉病毒(BUNV)、缓存谷病毒(CVV)、马瓜里病毒(MAGV)、阿里病毒(NRIV)、西北病毒(NORV)和天锯病毒(TENV)。LOKV最初是从库蚊tarsalis1962年加州的蚊子[2].此外,还从美国西部的各种节肢动物以及兔子和野兔中分离出了更多的分离物,大多数分离物是从这些动物中提取的库蠓属摇蚊[2,3.,4,5].血清学资料显示,LOKV的地理范围还包括美国的东半部[6].LOKV不是一种公认的人类或脊椎动物病原体,但其致病能力尚未得到广泛研究。
属中的所有病毒Orthobunyavirus具有三段式单链负意义RNA基因组[7].这三个RNA片段分别为小,(S),中(M)和大(L),长度分别约为1.0,4.5和6.9 kb。互补和高度保守的核苷酸序列位于每个RNA片段的5 '和3 '端。这些序列的碱基配对导致了长柄结构的形成,这被认为是基因组转录和复制启动所必需的[8,9].S RNA片段编码核衣壳(N)蛋白和非结构蛋白NS年代在重叠的阅读框中[10].M RNA片段编码一个多蛋白,该多蛋白经过翻译后加工,可生成N端和c端糖蛋白Gn和Gc,以及一个非结构蛋白NS米[11].L RNA片段编码作为RNA依赖性RNA聚合酶的L蛋白[12].
当载体或宿主同时感染两种或多种病毒时,亲缘关系密切的正布尼亚病毒之间可发生RNA片段交换(基因组重组)[13,14].重组被认为是这些病毒进化的主要手段,因为由此产生的子代可以具有新的和有利的遗传和表型特征。重组有时产生的病毒比它们的供体病毒更具致病性。一个例子是NRIV,它从BUNV获得S和L RNA片段,从BATV获得M RNA片段[15,16].NRIV与东非人类出血热的大规模暴发有关,而与其前体病毒相关的最严重疾病表现为低热和肌痛。
LOKV的基因组尚未完全测序。M RNA片段的559 nt.区域横跨部分5 '非翻译区(UTR)和Gn编码区已被测序(Genbank Accession no. 1)。AY593736.1),没有S或L RNA片段的序列数据。本研究的目的是对LOKV的基因组进行完全测序,并确定其与其他BUN血清组病毒的遗传和系统发育关系。
方法
病毒
LOKV(分离的FMS 4332)从德克萨斯州加尔维斯顿的德克萨斯大学医学分部的新兴病毒和虫体病毒世界参考中心获得。在收到病毒之前,该病毒已在乳鼠大脑中传代了13次,并在爱荷华州立大学的非洲绿猴肾(Vero)细胞中进行了一次额外的传代。
高通量测序
使用Trizol试剂(ThermoFisher, Carlsbad, CA)从lodv感染的Vero细胞的次融合单分子层中提取总RNA,使用RNase III (New England BioLabs, Ipswich, MA)进行碎片化,并使用Agilent 2100生物分析仪(Agilent, Santa Clara, CA)评估质量。使用离子总RNA-Seq Kit v2 (ThermoFisher)构建文库,并在爱荷华州立大学基因组技术设施使用离子质子测序仪(ThermoFisher)进行质量评估和分析。离子激流读取到的基因组Chlorocebus sabaeus使用Bowtie 2来识别宿主细胞序列[17].使用sortMeRNA程序分析未映射的reads以去除rrna相关的reads [18].所有剩余的Phred值≥33的读数都进行de novo SPAdes (ver 3.5.0)组装[19].Contigs被映射到正布尼亚病毒基因组(MDV为S和L RNA片段序列;CVV for M RNA片段序列)使用LASTZ [20.].在Tablet上手动验证对齐文件[21].
逆转录聚合酶链反应和sanger测序
通过逆转录-聚合酶链反应(RT-PCR)和Sanger测序验证高通量测序生成的LOKV序列,并缩小contigs之间的间隙。使用Superscript III逆转录酶(ThermoFisher)生成互补dna,并使用高保真度进行pcrTaq聚合酶(ThermoFisher)根据制造商的说明。引物是根据高通量测序生成的序列设计的,可根据要求提供。RT-PCR产物使用purelink凝胶提取试剂盒(ThermoFisher)纯化,并使用3730 × 1 DNA测序仪(Applied Biosystems, Foster City, CA)测序。
5 '和3 ' cDNA末端快速扩增
每个RNA片段的5 '和3 '端分别用cDNA端5 '和3 '快速扩增得到。简单地说,用200 U的T4 RNA连接酶2 (New England BioLabs)将20 pmol的预腺苷化DNA适配器(5 ' -rApp/TGGAATTCTCGGGTGCCAAGGT/ddC-3 ')连接到1 μg病毒基因组RNA和抗基因组RNA上,并在25°C下孵育1小时。通过乙醇沉淀回收连接产物,使用Superscript III逆转录酶和适配器特异性引物创建互补cdna。使用适配器和基因特异性引物进行pcr, RT-PCR产物纯化并进行Sanger测序。
核苷酸和氨基酸序列比对
利用BLASTn和BLASTp分别将LOKV的核苷酸序列和预测氨基酸序列与Genbank数据库中的其他序列进行比对。校准也使用Clustal Omega(可在http://www.ebi.ac.uk/Tools/msa/clustalo/),以计算所选序列之间核苷酸和氨基酸相同的百分比。
系统发育分析
利用快速傅里叶变换对核苷酸序列进行多重比对。基于马尔可夫链蒙特卡罗的贝叶斯分析由MrBayes使用一般时间可逆+不变位点+伽玛分布4模型[22].为了达到一致性,运行了1000万次迭代,每1000次迭代采样一次。最大分支可信度树是使用TreeAnnotator(版本1.8.2)创建的,带有10%的老化[23并使用FigTree(版本1.4.0)进行可视化。
结果
测定LOKV基因S、M和L RNA片段的完整核苷酸序列(Genbank Accession no . MG696865、MG820264和MG828823)。S RNA片段由952个nt.组成,与MDV的对应区域关系最密切,同源性99.0%,其次是NRIV和NORV,同源性分别为85.0和84.6%(表2)1).LOKV的S RNA片段包含两个开放阅读框(orf)。最长的5 ' AUG启动最长的ORF,包含核苷酸位置77到778,并编码N蛋白。预测翻译产物由233个氨基酸组成,与MDV N蛋白的同源性最高,同源性为99.6%。第二个ORF包含核苷酸位置96到401,并编码NS年代蛋白质。预测的翻译产物由101个氨基酸组成,与NS相同年代MDV蛋白。
LOKV的M RNA片段由4450 nt.组成,与CVV及其变体Fort Sherman病毒、Playas病毒和Tlacotalpan病毒的对应区域关系最密切,具有72.4 ~ 72.7%的nt.同源性,其次是MAGV和NORV,分别具有72.2和71.4%的nt.同源性(表4)1).先前在Genbank数据库中获得的LOKV的部分M RNA片段序列与我们序列的相应区域具有99.6%的nt.同源性。这些序列在两个核苷酸位置上不同;在5 ' UTR上有一个核苷酸取代,在Gn编码区有一个同义取代。对Birao病毒、Bozo病毒、Mboke病毒、Shokwe病毒和xinu病毒的部分M RNA片段序列进行了成对核苷酸序列比对,结果显示与LOKV相应区域的nt.同源性为60.2 ~ 68.8%。LOKV的M RNA片段包含一个单一的ORF,编码1434个氨基酸的多蛋白,预计可产生Gn, Gc和NS米分别含有286、866和173个氨基酸的蛋白质。推导出的多聚蛋白的氨基酸序列与CVV及其变体的相应区域具有最大的一致性(77.9 ~ 78.4%)1).利用推导出的单个蛋白质的氨基酸序列进行比对,揭示了Gn, Gc和NS米LOKV蛋白与CVV及其变体蛋白的同源性分别为87.0、79.1和67.0%。
LOKV的L RNA片段由6864 nt.组成,与MDV对应区域的同源性最密切,同源性99.0%,其次是NORV和TENV,同源性分别为86.7和86.1%1).LOKV的L RNA片段包含一个单一的ORF,包含核苷酸位置49到6765,并编码L蛋白。预测的翻译产物包含1434个残基,与MDV L蛋白的同源性为99.0%。
在LOKV所有三个RNA片段的5 '和3 '端鉴定出互补序列(图2)。1).S RNA片段5 '端和3 '端的21个末端核苷酸中有17个是互补的,对M和L RNA片段序列的检查显示,20个和27个nt中的19个和24个分别是互补的。在这三个片段的5 '末端的前11个nt.由序列5 ' -AGUAGUGUACU-3 '所表征。除了位置9和- 9的不匹配外,当处于反向互补方向时,所有三个片段的3 '端都存在相同的序列。
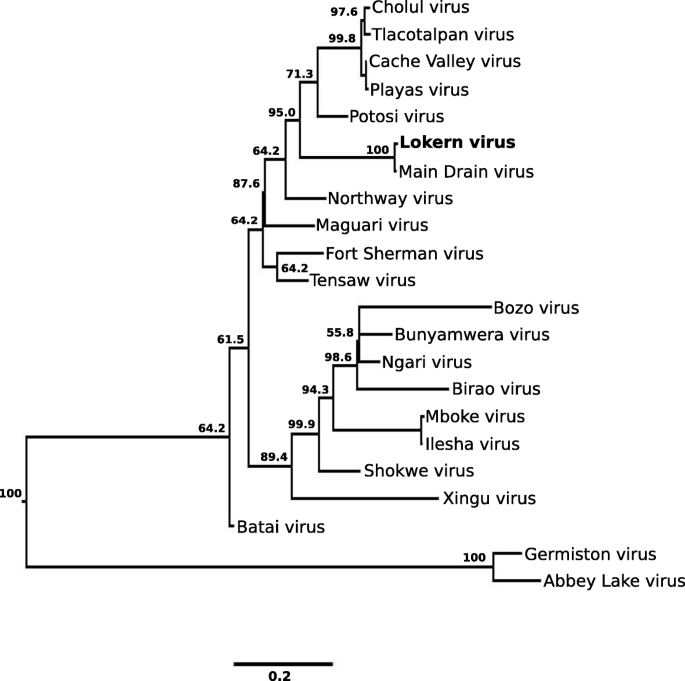
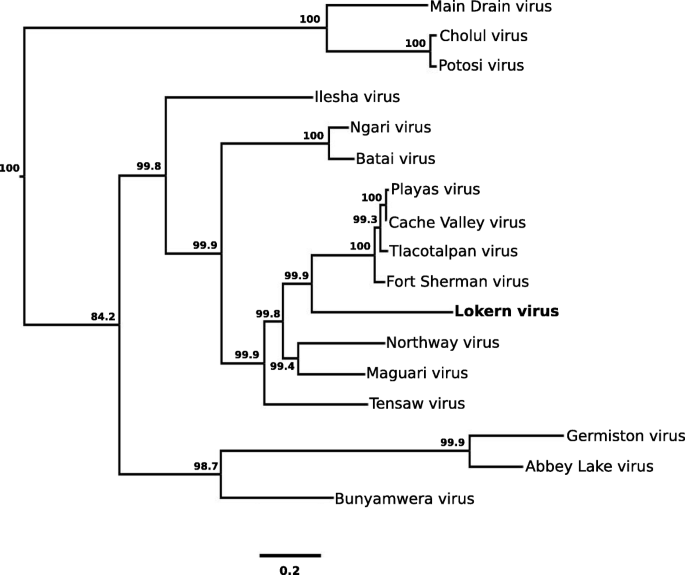
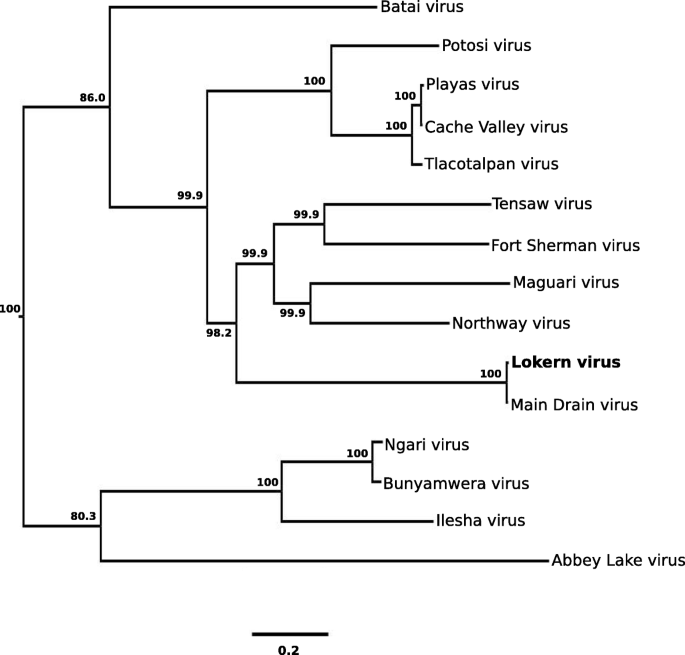
利用LOKV病毒S、M和L RNA片段的核苷酸序列和其他正bunyya病毒,用贝叶斯方法构建系统发育树(图2)。2,3.,4).LOKV的S RNA片段序列与MDV的相应片段在系统发育上关系最为密切。这种拓扑结构的贝叶斯后验支持度为100%。同样,在用L RNA片段序列构建的贝叶斯树中,LOKV与MDV的亲缘关系最为密切,并具有较强的后验支持(100%)。相比之下,LOKV的M RNA片段序列在系统发育上与CVV及其变体的相应区域最密切相关。这种拓扑结构的后验支持度为99.9%。这些病毒,连同NORV, MAGV和TENV,组成了一个独特的分支,有强大的后支持(99.9%)。
Lokern病毒小RNA片段的系统发育分析。该分析基于702 nt。Lokern病毒小RNA片段的区域和所有其他Bunyamwera血清组病毒的相应区域,这些序列数据是可用的。选择后验概率表示为节点下的百分比。分支长度与核苷酸差异的数量成正比。分析中使用的Genbank接入号列在表的脚注中1
Lokern病毒培养基RNA片段的系统发育分析。该分析基于4345-nt。Lokern病毒中RNA片段的区域,以及所有其他有这些序列数据的Bunyamwera血清组病毒的相应区域。选择后验概率表示为节点下的百分比。分支长度与核苷酸差异的数量成正比。Genbank的加入编号列在表的脚注中1
Lokern病毒大RNA片段的系统发育分析。该分析基于6722-nt。Lokern病毒大RNA片段的区域和所有其他Bunyamwera血清组病毒的相应区域,这些序列数据是可用的。选择后验概率表示为节点下的百分比。分支长度与核苷酸差异的数量成正比。Genbank的加入编号列在表的脚注中1
讨论
这项研究提供了证据,证明LOKV是一种天然重组病毒,其S和L RNA片段来自MDV,其M RNA片段来自一种未被发现的,可能已经灭绝的病毒。MDV被认为是供体病毒之一,因为它的S和L RNA片段与LOKV的几乎相同。这两种病毒具有相似的地理分布,以及媒介和宿主之间的关联,从而为发生重组提供了机会。LOKV和MDV都发生在美国西部,大多数节肢动物和脊椎动物衍生的病毒分离都来自于库蠓属摇蚊及家兔,分别[3.].另一种不太可能的解释是,LOKV和MDV都是从同一个祖先那里获得S和L RNA片段的重组体。有人认为MDV是一种重组病毒,其S RNA片段来自MAGV或NORV, M RNA片段来自Potosi或Kairi病毒,L RNA片段来自CVV或MAGV [24].
LOKV的M RNA片段被认为是从一种未发现的病毒中获得的,因为它与所有其他已知正bunyya病毒的M RNA片段的同源性不超过72.7%。另一种可能性较小的解释是,M段供体是一种已知病毒,但重组事件不是最近发生的,因此有足够的时间通过遗传漂变发生相当数量的序列分歧。所有病毒的基本定位到LOKV图中。3., MAGV和NORV是它的近亲。MAGV不太可能是M RNA片段供体,因为其已知的地理分布与LOKV不重叠。MAGV发生在拉丁美洲和加勒比地区,但从未在美国被发现(疾病控制和预防中心虫媒病毒目录,https://wwwn.cdc.gov/arbocat/VirusDetails.aspx?ID=276&SID=12, 2018年4月2日访问)。然而,NORV和LOKV的地理分布、媒介和宿主范围重叠。NORV发生在美国西部和加拿大,并已从兔子和其他啮齿动物以及蚊子中分离出来[3.,25].
我们的数据支持早期的研究,即在BUN血清组病毒中最常见的重组事件是M RNA片段交换。在一项经典研究中,在实验室中使用BUNV/BATV和BUNV/MAGV进行异源杂交,所有由此产生的重组都从同一亲本病毒获得其S和L RNA片段[26].此外,MDV、NRIV和波托西病毒都是由M RNA片段交换引起的自然重组病毒[15,16,24,27].Cholul病毒是个例外;它是由S RNA片段交换产生的自然重组[28].对于通常具有同源M- l和S-M关联的加利福尼亚血清组病毒和似乎有利于同源M- l关联的C组病毒来说,M RNA片段交换不是首选的重组模式[29,30.].
病毒属中所有病毒基因组的特征Orthobunyavirus是在每个RNA片段的5 '和3 '端存在互补核苷酸序列[7,31,32,33].病毒的每个RNA片段之间以及属内每个病毒之间的序列高度保守。一致序列由11 nt.组成,定义为5 ' -AGUAGUGUACU-3 ',如我们用LOKV观察到的那样,在两端的位置9和- 9处不匹配。末端11 nt后面是3到4个核苷酸,它们是互补的,在特定片段的基础上高度保守,后面是几个核苷酸,它们是互补的,通常是单个病毒和片段独有的[29,30.].对LOKV基因组序列的检查显示,在每个片段的末端存在21到27个nt.的末端互补序列,并有1到4个错配,这与先前的报道一致,互补区域超出了高度保守的11 nt.共识序列。
结论
总之,本研究中提供的序列和系统发育数据提供了证据,证明LOKV是由M RNA片段交换引起的重组子。我们认为LOKV的S和L RNA片段很可能来自MDV,其M RNA片段来自一种未被识别的病毒,但也提供了其他解释。完整的基因组序列数据是精确检测和识别病毒所必需的,特别是当有关病毒具有分段基因组时,因为否则重组体及其前体可能被误认为彼此。
缩写
- BATV:
-
Batai病毒
- 包子:
-
Bunyamwera
- BUNV:
-
Bunyamwera病毒
- CVV:
-
缓存谷病毒
- 李:
-
大
- LOKV:
-
Lokern病毒
- M:
-
媒介
- MAGV:
-
Maguari病毒
- 护士:
-
核衣壳
- NORV:
-
Northway病毒
- NRIV:
-
阿里病毒
- 子:
-
开式阅读架
- rt - pcr:
-
逆转录聚合酶链反应
- 史:
-
小
- TENV:
-
Tensaw病毒
- UTR:
-
翻译区
参考文献
Blitvich BJ, Beaty BJ, Blair CD, Brault AC, Dobler G, Drebot MA, Haddow AD, Kramer LD, LaBeaud AD, Monath TP, Mossel EC, Plante K, Powers AM, Tesh RB, Turell MJ, Vasilakis N, Weaver SC.布尼亚病毒分类学:与当前用于物种划分的ICTV标准相关的局限性和误解。中华热带医学杂志,2018;39(1):1 - 6。在新闻
Scrivani RP。Lokern (LOK)株。FMS 4332。中华热带医学杂志,1990;19(6):1111-2。
Calisher CH, Francy DB, Smith GC, Muth DJ, Lazuick JS, Karabatsos N, Jakob WL, McLean RG。1956-1984年北美Bunyamwera血清组病毒的分布。中华热带医学杂志1986;35:429-43。
鹤GT, Elbel RE, Francy DB, Calisher CH.虫媒病毒从美国犹他州西部,1967-1976。中华医学昆虫杂志1983;20:294-300。
李丽娟,李丽娟,李丽娟。植物虫媒病毒的分离库蠓属在科罗拉多州发生水疱性口炎的动物流行病期间,人们发现了蠓(双翅目:蠓科)。中华医学昆虫学杂志,1990;27:487-93。
Meyers MT, Bahnson CS, Hanlon M, Kopral C, Srisinlapaudom S, Cochrane ZN, Sabas CE, Saiyasombat R, Burrough ER, Plummer PJ, O 'Connor AM, Marshall KL, Blitvich BJ。美国绵羊中Cache Valley病毒和其他Bunyamwera血清群病毒抗体流行率与操作水平相关的管理因素媒介传播人畜共患疾病,2015;15:683-93。
Schmaljohn CS, Nichol ST.布尼亚病毒科。在:Knipe DM,编辑。菲尔兹病毒学。第五版。费城:利平科特·威廉姆斯和威尔金斯;2007.p . 1741 - 89。
Barr JN, Wertz GW。布尼亚病毒RNA的合成需要3 ' -和5 ' -末端序列的配合。中国病毒学杂志,2004;78:1129-38。
Pardigon N, Vialat P, Girard M, Bouloy M.杰米斯顿病毒rna(布尼亚病毒)末端的长柄和发夹结构。病毒学。1982;122:191-7。
富勒F,鲍恩AS,主教DH。布尼亚病毒核蛋白N和非结构蛋白NSs由S RNA中重叠的阅读框编码。中国病毒学杂志,1983;64:1705-14。
Fazakerley JK, gonzez - scarano F, Strickler J, Dietzschold B, Karush F, Nathanson N.雪鞋兔布尼亚病毒中RNA片段的组织。病毒学。1988;167:422-32。
Endres MJ, Jacoby DR, Janssen RS, Gonzalez-Scarano F, Nathanson N.加州血清组布尼亚病毒的大病毒RNA片段编码大病毒蛋白。中华病毒学杂志,1989;70:223-8。
布氏T型、卡利舍CH型、希格斯s型布尼亚病毒科病毒:所有分离株都是重组株吗?病毒学。2013;446:207-16。
Beaty BJ, Sundin DR, Chandler LJ,主教DH。双感染蚊布尼亚病毒基因组重组进化(伊蚊triseriatus).科学。1985;230:548-50。
布里斯T,伯德B,卡普尔V,尼科尔ST,利普金WI。巴泰病毒和阿里病毒:M段重组和与东非严重发热疾病暴发的关联。中国病毒学杂志,2006;
Gerrard SR, Li L, Barrett AD, Nichol ST. Ngari病毒是一种布尼亚姆韦拉病毒重组体,可与非洲出血热的大规模暴发有关。中国病毒学杂志,2004;78:8922-6。
朗米德B,萨尔茨堡SL.快速间隙阅读对齐领结2。Nat Methods. 2012; 9:357-9。
Kopylova E, Noe L, Touzet H. SortMeRNA:在meta转录组数据中快速准确地过滤核糖体rna。生物信息学。2012;28:3211-7。
班克维奇A,努尔克S,安提波夫D,古列维奇AA,德沃金M,库利科夫AS,列辛VM, Nikolenko SI, Pham S, Prjibelski AD, Pyshkin AV, Sirotkin AV, Vyahhi N, Tesler G, Alekseyev MA, Pevzner PA。SPAdes:一种新的基因组组装算法及其在单细胞测序中的应用。中国生物工程学报。2012;19:455-77。
改进的基因组DNA成对排列:宾夕法尼亚州立大学;2007.可以在:http://www.bx.psu.edu/~rsharris/rsharris_phd_thesis_2007.pdf
Milne I, Stephen G, Bayer M, Cock PJ, Pritchard L, Cardle L, Shaw PD, Marshall D.使用平板电脑进行第二代测序数据的可视化探索。生物信息学报。2013;14:193-202。
Ronquist F, Huelsenbeck JP。MrBayes 3:混合模型下的贝叶斯系统发育推断。生物信息学。2003;19:1572-4。
Drummond AJ, Rambaut A. BEAST:抽样树的贝叶斯进化分析。BMC进化生物学。2007;7:214-21。
布里斯T,卡普尔V,利普金WI。波托西病毒和主漏病毒的m段自然重组:正布尼亚病毒进化的意义。《寄生虫学》,2007;152:2237-47。
坎贝尔GL,哈迪JL,埃尔德里奇BF,里维斯WC。1969-1985年加利福尼亚州和俄勒冈州蚊子中Northway血清型和其他Bunyamwera血清型布尼亚病毒的分离中华热带医学杂志。1991;44:581-8。
Pringle CR, Lees JF, Clark W, Elliott RM。利用分子克隆互补DNA探针进行点杂交分析布尼亚病毒基因组亚基重组。病毒学。1984;135:244-56。
Yanase T, Kato T, Yamakawa M, Takayoshi K, Nakamura K, Kokuba T, Tsuda T. Batai病毒的遗传特征表明自然界正bunyvirus之间存在基因组重组。《病毒学》,2006;151:2253-60。
Blitvich BJ, Saiyasombat R, Dorman KS, Garcia-Rejon JE, Farfan-Ale JA, lorino - pino MA。序列和系统发育数据表明,最近在墨西哥尤卡坦半岛发现的正布尼亚病毒是波托西病毒和卡什谷病毒的一种新型重组病毒。《寄生虫学》2012;157:1199-204
乌尔基迪五世主教拉克罗斯病毒和雪鞋兔病毒三方RNA基因组之间的非随机重组。中华病毒学杂志。1992;73:2255-65。
Nunes MR, da Rosa AP, Weaver SC, Tesh RB, Vasconcelos PF.美洲C组病毒(布尼亚病毒科,正布尼亚病毒)的分子流行病学。中国病毒学杂志,2005;49:10561 - 70。
艾略特RM。布尼亚病毒科分子生物学。中华病毒学杂志,1990;71:501-22。
Elliott RM, Schmaljohn CS, Collett MS.布雅病毒科基因组结构与基因表达。中华微生物学杂志,2001;39(4):344 - 344。
葛拉赫P, Malet H, Cusack S, Reguera J.布尼亚病毒复制及其vRNA启动子调控的结构研究。细胞。2015;161:1267 - 79。
致谢
作者感谢Robert Tesh提供LOKV隔离。
资金
这项研究得到了爱荷华州立大学的校内资助。
数据和材料的可用性
当前研究的所有数据均可根据要求从通讯作者处获得。
作者信息
作者及隶属关系
贡献
CT和JC进行了高通量测序实验。CT还进行了rt - pcr、Sanger测序、cDNA末端5’和3’快速扩增和系统发育分析。BJB构思了这个想法,并准备了初稿。所有作者审阅并批准了手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
所有作者均同意发表。
相互竞争的利益
作者声明没有利益竞争。
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
坦古杜,c.s.,查尔斯,J.和Blitvich, B.J.证据表明洛克恩病毒(家族Peribunyaviridae)是一种重组病毒,它的大、小基因组片段来自主干病毒,中基因组片段来自一种未被发现的病毒。性研究J15, 122(2018)。https://doi.org/10.1186/s12985-018-1031-6
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12985-018-1031-6
关键字
- Lokern病毒
- 水管病毒
- Orthobunyavirus
- Peribunyaviridae
- 重组
- 基因组序列
- 高通量测序