- 研究文章gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
评估全基因组测序在牛呼吸道合胞病毒实验感染和自然暴发中追踪传播途径的潜力gydF4y2Ba
兽医研究gydF4y2Ba体积gydF4y2Ba53gydF4y2Ba、物品编号:gydF4y2Ba107gydF4y2Ba(gydF4y2Ba2022gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
牛呼吸道合胞病毒(BRSV)是牛呼吸道疾病的主要病因。基因组测序可以解决病毒种群之间的系统发育关系,可用于推断传播途径,并可能为生物安全措施的设计提供信息。对15000 -nt BRSV基因组短片段(<2000 nt)的测序揭示了BRSV种群的地理和时间聚类,但变异不足以区分从空间和时间上紧密聚集的感染群体收集的病毒。本研究调查了全基因组测序的潜力,以揭示足够的基因组变异,推断畜群之间的传播途径。下一代测序(NGS)数据来自于瑞典Jämtland和乌普萨拉县的实验感染和自然暴发。分别从47到20个样本中获得了足够的覆盖深度来分析一致性和次一致性序列多样性。在实验传输中观察到的共识级多态性很少(范围:6个实验中的0-6个多态性)。在疫情样本的一致序列中发现了更高水平的多样性(146个多态性位点)。大多数多态性(144/146)发生在县与县之间,而不是县与县之间,这表明共识全基因组序列在推断直接传播途径方面存在空间分辨率不足的问题,但可能允许在区域范围内确定暴发源。相比之下,实验样本内的多样性普遍高于爆发样本。 Analyses to infer known (experimental) and suspected (outbreak) transmission links from within-sample diversity data were uninformative. In conclusion, analysis of the whole-genome sequence of BRSV from experimental samples discriminated between circulating isolates from distant areas, but insufficient diversity was observed between closely related isolates to aid local transmission route inference.
介绍gydF4y2Ba
牛呼吸道合胞病毒(BRSV)是牛呼吸道疾病的主要病因。由于BRSV感染的福利和经济影响,以及需要减少抗生素的使用以防止细菌重复感染,因此控制BRSV具有高度优先性[gydF4y2Ba1gydF4y2Ba]。为了阻止BRSV的传播,需要接种疫苗和/或采取卫生措施,这两项措施都不是直截了当地的,特别是考虑到关于BRSV传播途径的大量知识差距。如果能更好地了解BRSV的流行病学,将有助于设计更有效的控制措施。目标是改进北欧国家目前正在制定的控制方案的设计,以改善牛的健康[gydF4y2Ba2gydF4y2Ba],现在的主要目标是确定BRSV的传播途径。gydF4y2Ba
牛是BRSV的天然宿主,但其他反刍动物物种也可能在传播中发挥作用。其他动物物种(例如雀鸟和狗)可作为被动传病媒介,但在某些情况下是有限的[gydF4y2Ba1gydF4y2Ba]。一些数据表明,BRSV可能在受感染的牛体内持续存在[gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba],但证明BRSV从先前感染的动物身上再排泄的尝试失败了[gydF4y2Ba6gydF4y2Ba]。与人类RSV (HRSV)一样,BRSV通过与受感染个体的直接接触,通过受感染动物的大呼吸道飞沫或鼻分泌物传播。即使可能在同一建筑物内或由通风系统连接的马厩内经空气传播[gydF4y2Ba7gydF4y2Ba],不太可能发生在距离较远的象群之间[gydF4y2Ba8gydF4y2Ba]。人类作为被动载体传播BRSV,这在HRSV中已被观察到[gydF4y2Ba9gydF4y2Ba,不能排除。人类访客有时与动物没有直接接触,但被怀疑会引起一些BRSV疫情。然而,这一点从未得到证实,需要进行更多的工作来支持这一假设。gydF4y2Ba
BRSV是一种包膜、无节段、负链RNA病毒,属于gydF4y2BaOrthopneumovirusgydF4y2Ba属内属gydF4y2BaPneumoviridaegydF4y2Ba家庭(gydF4y2Ba10gydF4y2Ba]。基因组包含约15000个核苷酸,编码11种蛋白质,包括病毒RNA聚合酶。与其他RNA病毒一样,这种聚合酶也会产生错误,而这种错误无法通过校对机制得到缓解。对于BRSV G序列,估计单个野外样本中每个位点的错误结合频率可能高达5.2 × 10gydF4y2Ba4gydF4y2Ba-10.1 × 10gydF4y2Ba4gydF4y2Ba核苷酸/网站[gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba]。因此,核苷酸变化应该是频繁的,因为病毒基因组相对较长,并且通过正链中间体进行复制。观察到的核苷酸替换频率根据基因位点而变化,优先发生在耐受变化的基因组部分。BRSV的G糖蛋白和SH基因已被证明是最易变的[gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba],但这些片段很短(分别为840 nt和420 nt),因此,部分基因组测序可能无法区分在空间和时间上紧密聚集在一起的病毒的系统发育关系[gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba]。通过研究全基因组,我们希望能提高菌株之间的基因区分水平。gydF4y2Ba
事实上,通过全基因组测序,结合流行病学调查,有可能重建几种RNA病毒的传播树[gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba]包括口蹄疫病毒[gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba]。序列数据显示,在单次暴发期间发生了大量进化[gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba],并利用遗传变异在高时空分辨率下重建传播树[gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba]。gydF4y2Ba
对BRSV毒株全基因组的系统发育分析可能同样有助于追踪该病毒在农场之间的传播,并确定引入源。然而,为了实现这一目标,首先在个体和群体水平上描述感染期间BRSV全基因组的遗传变异水平是有用的。大多数追踪传播途径的方法使用共识病毒基因组序列结合流行病学数据(如感染时间、空间接近度、接触者追踪数据)来衡量潜在传播链接的相对可能性[gydF4y2Ba24gydF4y2Ba]。最近,开发了利用样本内(亚共识)遗传多样性的方法[gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba,gydF4y2Ba27gydF4y2Ba,gydF4y2Ba28gydF4y2Ba]。为了使基因组序列多样性有助于传播树的重建,在密切相关的感染场所之间必须存在多样性,无论是在共识水平还是在次共识水平上,并且样本之间共享多样性的数量必须能够提供关于它们在传播树上的接近程度的信息。为了评估全基因组序列在帮助推断畜群之间传播联系方面的效用,我们评估了沿着实验传播链和瑞典最近爆发的BRSV疫情样本之间的多样性水平,从个体牛之间的直接传播到距离最远达450公里的牛群样本。该研究的总体目标是建立一种BRSV全基因组测序的技术方法,并确定是否可以通过对病毒全基因组测序数据的分析来重建暴发期间BRSV的传播。目标是:(1)建立BRSV的NGS方法并评估其可靠性;(2)衡量样本之间(在共识序列水平上)和样本内部(在次共识水平上)的总体多样性水平;(3)研究利用共识和次共识多样性推断传输链路的可行性;(4)比较样本之间以及预期可变区域(SH + G)和较少可变区域(基因F,基因组的其余部分)之间的样本内多样性水平。gydF4y2Ba
材料与方法gydF4y2Ba
样品收集gydF4y2Ba
本研究分析的BRSV样本既来自实验性感染,也来自自然暴发。在通过气溶胶吸入或自然感染BRSV的小牛的实验研究中收集了支气管肺泡灌洗液、鼻拭子和攻击病毒(表2)gydF4y2Ba1gydF4y2Ba)。总共对19只实验感染4种不同菌株之一的动物的36份样本进行了测序。此外,对通过细胞培养或小牛传代获得的由四种不同毒株组成的六种攻毒病毒样本进行了测序(表1)gydF4y2Ba1gydF4y2Ba) [gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba]。活体动物鼻孔采集鼻拭子,死后用400 mL磷酸盐缓冲盐水(PBS)经气管冲洗左肺叶,进行支气管肺泡灌洗(BAL)。用无菌纱布过滤BAL,取10 mL室温、200 ×离心gydF4y2BaggydF4y2Ba去除上清液,用750µL TRIzol™Reagent (Thermo Fisher, Life Technologies Corporation, USA)重悬微球,- 75°C冷冻至RNA提取。此外,从2020年5个猪群的BRSV暴发中收集了7份鼻拭子样本。gydF4y2Ba
实验样品gydF4y2Ba
实验A至F是先前发表的小牛BRSV感染实验,目的是评估不同疫苗的效果[gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba]。在这些研究中,如上所述,在死后收集BAL,并/或在BRSV攻击前后每天从小牛的一个鼻孔收集鼻拭子。挑战包括在细胞培养物或小牛中传代的病毒,如表所述gydF4y2Ba1gydF4y2Ba。gydF4y2Ba
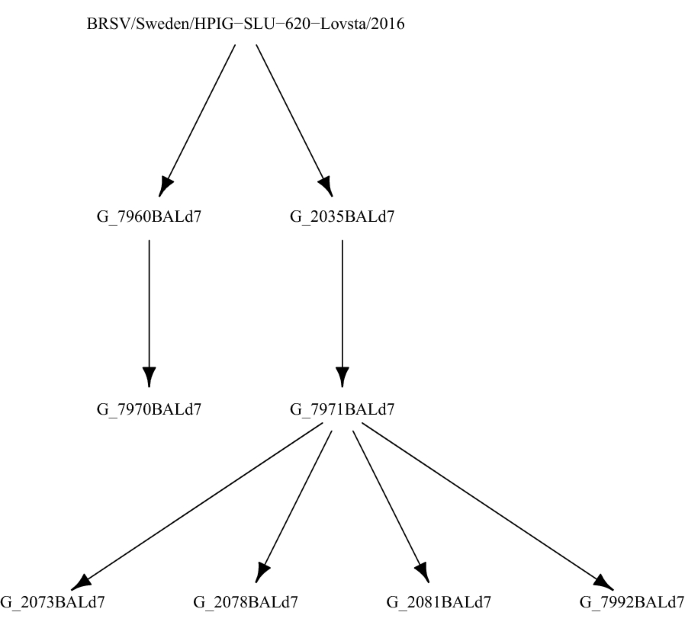
实验G包括病毒在犊牛之间传代,旨在研究BRSV基因组沿序列传代的进化(图5)gydF4y2Ba1gydF4y2Ba;附加文件gydF4y2Ba1gydF4y2BaA, D)。10头小牛来自瑞典畜牧研究中心(SLU, Uppsala)。他们被常规饲养3-7周龄的瑞典荷斯坦和瑞典红白品种的雄性和雌性小牛,并根据没有呼吸道疾病史或接种疫苗而选择。犊牛成对饲养在稻草床围栏内,每天两次喂代乳和犊牛颗粒,以及干草和水gydF4y2Ba没有限制地gydF4y2Ba。适应1周后,7960和2035分别饲喂1.84 ~ 2.35 × 10gydF4y2Ba5gydF4y2Ba相当于TCIDgydF4y2Ba50gydF4y2Ba采用实时荧光定量PCR方法检测一株BRSV病毒(BRSV/Sweden/HPIG-SLU-620-Lovsta/2016)的气溶胶接种结果[gydF4y2Ba31gydF4y2Ba]。从感染后第0天(PID)至第7天或第10天收集每日鼻拭子(病毒拭子UTM™,Copan,意大利)。挑战前后进行日常临床检查,并对呼吸系统疾病的临床症状进行评分,如前所述[gydF4y2Ba31gydF4y2Ba]。临床和病毒学数据见附加文件gydF4y2Ba1gydF4y2Baeg。攻毒后第7天,对7960和2035犊牛进行过量全身麻醉(氯胺酮5 mg/kg,戊巴比妥钠40 mg/kg,放血)安乐死,切除肺,通过观察和触诊对宏观病变进行观察和量化,并收集BAL。这一过程在接下来的两轮中重复了两次。用7960和2035犊牛获得的BAL分别接种7970和7971犊牛。然后将7971小牛在PID 7上获得的BAL用于接种2073、2078、2081和7992小牛,并在PID 7上从这4头小牛获得BAL(附加文件)gydF4y2Ba1gydF4y2BaA, D).对这八头小牛进行了全基因组测序。剩下的两头小牛(7965和7962)作为伴侣动物。来自这些动物的病毒未用于进一步接种或基因组测序。gydF4y2Ba
爆发样本gydF4y2Ba
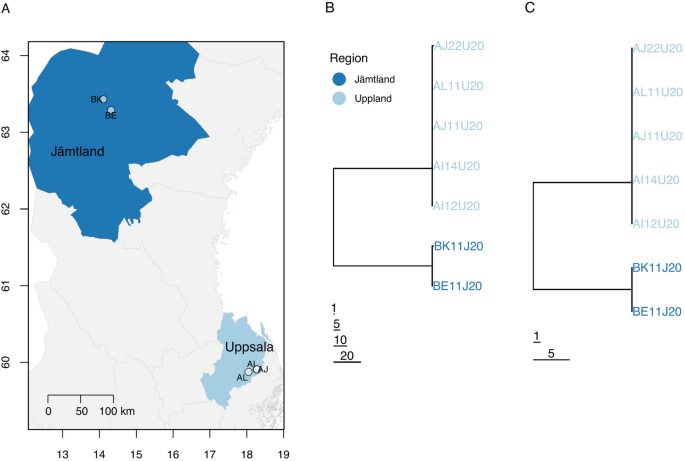
现场暴发样本(研究O)是从乌普萨拉县三个农场(分别于2020年2月18日、19日和21日对AI、AJ和AL农场进行采样)和Jämtland县两个农场(分别于2020年4月3日和13日对BE和BK农场进行采样)中患有呼吸道疾病的牛的鼻拭子采集的(图3)gydF4y2Ba2gydF4y2BaA).农场AI和AJ相距500-1000米,与AL相距13公里,而BE和BK相距19公里。奥普兰县和Jämtland相隔约450公里。这两项先前未发表的研究(G和O)都得到了瑞典乌普萨拉大学伦理委员会的批准。5.8.18-16188/2017)。BRSV菌株直接从鼻分泌物中测序,无需细胞培养传代。gydF4y2Ba
样品命名法gydF4y2Ba
这里列出的所有样本名称都以实验字母(A、B、C、D、F或G)或爆发样本的字母O为前缀。实验动物样本命名如下:[gydF4y2Ba实验的信gydF4y2Ba) _ (gydF4y2Ba4位动物IDgydF4y2Ba] [gydF4y2Ba取样方法:BAL(支气管肺泡灌洗)或NS(鼻拭子)gydF4y2Ba感染后]]d[天。例如,在感染后6天通过鼻拭子从动物8863中取样B_8863NSd6作为实验B的一部分。爆发样本的命名如下:O_[gydF4y2Ba两个字母的匿名农场代码gydF4y2Ba] [gydF4y2Ba两位数动物代码gydF4y2Ba] [gydF4y2Ba县代码:gydF4y2BaUgydF4y2Ba乌普萨拉;gydF4y2BaJgydF4y2Ba对于JamtlandgydF4y2Ba] [gydF4y2Ba采样年份为两位数gydF4y2Ba]。例如O_AJ22U20是2020年在乌普萨拉的AJ农场从22号动物身上采集的暴发样本(这意味着在第二次访问时,这是第二只采样的动物)。gydF4y2Ba
测序gydF4y2Ba
使用TRIzol (Invitrogen, Carlsbad, CA, USA)和RNeasy色谱柱(Qiagen, Hilden, Germany)联合从临床样品中提取总RNA。利用Superscript IV第一链合成试剂盒(Invitrogen)将RNA转化为cDNA。每个样品的浓度采用高灵敏度试剂和Qubit 2.0荧光计(Invitrogen)测定。为了减少偏倚,没有PCR扩增步骤。然而,通过RT-qPCR平行分析样品,仅包括CT值低于25的样品。使用NexteraXT试剂盒(Illumina, San Diego, CA, USA)根据制造商说明制备DNA文库。每个DNA样本最初由生物分析仪(Agilent Technologies,使用DNA1000芯片)进行分析,以确定文库质量和平均大小分布。使用MiSeq 600 cycle v3试剂盒(Illumina)在Illumina MiSeq上以配对端模式对DNA文库进行测序。使用Genomics软件包workbench 10 (CLC Bio, Aarhus, Denmark)分析和绘制原始序列数据。对原始读取进行裁剪,并重新组装每个裁剪数据集的子集,以生成每个数据集的参考序列。 Basic trimming was done by CLC-bio software to generate FastQ files. The FastQ files were deposited in the NCBI Sequence Read Archive database (accession numbers are provided in Additional file1gydF4y2BaB),在生物计划PRJNA893434下收集。gydF4y2Ba
生物信息学管道gydF4y2Ba
对FastQ文件进行适配器修剪和对端读取的质量控制gydF4y2Ba减少大量的gydF4y2Ba版本0.6.6 [gydF4y2Ba34gydF4y2Ba]。短于50 bp的Reads被丢弃,Phred质量评分低于25的低质量末端被修剪。每个实验中来自攻毒病毒样本的修剪reads与一个密切相关的参考序列对齐[gydF4y2Ba32gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba)使用gydF4y2BaBWAgydF4y2Ba版本0.7.17 [gydF4y2Ba37gydF4y2Ba],使用MEM算法和默认值gydF4y2BaBWAgydF4y2Ba选项。然后将所有实验样本与该实验特有的攻毒病毒的共识序列对齐,将BWA缺口打开惩罚从默认的6增加到100,以防止插入。这确保了管道生成的主机内多样性数据适用于不允许插入或删除的下游多样性分析gydF4y2BaBetabinomial瓶颈gydF4y2Ba和gydF4y2BaBadTrIPgydF4y2Ba)。现场暴发样本与实验G BRSV/Sweden/HPIG-SLU-620-Lovsta/2016的攻毒菌株一致,该攻毒菌株是从2016年Uppland自然感染样本中分离出来的。初步分析表明,前500个基地的覆盖率往往异常低。因此,我们从参考序列中排除了基因NS1(碱基1-572)和NS1与NS2之间的序列(573-578),并从NS2(碱基579)开始进行比对。因此,我们分析了96%的BRSV基因组;然而,为简洁起见,我们将这些序列称为全基因组序列。转换为bam对齐格式后使用gydF4y2BasamtoolsgydF4y2Ba1.9版[gydF4y2Ba38gydF4y2Ba共识序列和每基因组位置碱基计数使用gydF4y2BaVSensusgydF4y2Ba[gydF4y2Ba39gydF4y2Ba]。gydF4y2Ba
统计分析gydF4y2Ba
序列质量分析gydF4y2Ba
我们进行了三次初步分析,目的是预测和潜在地增加覆盖率。首先,我们使用了线性混合效应回归模型gydF4y2Balme4gydF4y2Ba包gydF4y2BaRgydF4y2Ba4.1.1版考察抽样方法的效果。我们询问是否可以使用鼻拭子样本获得高覆盖率的基因组序列,或者是否有必要使用更具侵入性和劳动密集型的BAL方法。结果变量是覆盖率,loggydF4y2Ba10gydF4y2Ba-变换以满足正态分布均方差残差的假设。我们使用匹配的样本(使用一种方法的样本只有在同一天以另一种方法从同一动物身上收集的另一个样本才被纳入),并纳入抽样事件(动物日)的随机效应。对4只动物进行支气管肺泡灌洗和鼻拭子取样,共获得8个支气管肺泡灌洗样本和8个鼻拭子样本。其次,我们使用线性混合效应模型来评估实时PCR (RT-PCR) Ct值在预测平均覆盖率方面的效用,目的是利用Ct值将测序资源集中在高覆盖率的样本上。该模型根据采样方法(支气管肺泡灌洗或鼻拭子)的影响进行了调整,并纳入了样本ID的随机效应,以解释相同样本的重复测序。方差在对数中的比例gydF4y2Ba10gydF4y2BaCt值解释的覆盖率估计为边际gydF4y2BaRgydF4y2Ba2gydF4y2Ba。该分析包括40个样本的71个序列。第三,我们对log进行了配对t检验gydF4y2Ba10gydF4y2Ba50个样本的平均覆盖率,以询问是否可以通过关注含有基因SH和G的1344个核苷酸序列(包括其39个核苷酸的基因间序列)从低覆盖率序列中恢复出具有样本内多样性信息的高覆盖率序列,该序列占基因组的9%,预计是其最可变的区域[gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba]。我们称之为SH + G区域。gydF4y2Ba
病毒多样性数据的可靠性gydF4y2Ba
为了提供足够的分辨率来分析低频多态性,我们从样本内多样性分析中排除了平均覆盖率低于500 ×或第10百分位覆盖率低于125 ×的序列。第二个标准的目的是确保覆盖率的一致性,但在实践中,所有符合第一个标准的样本也符合第二个标准。我们将其称为500 ×覆盖数据集。通过这两个阈值的序列包括10个超过一次测序的样本(6个两次,4个三次)。我们使用了一个二项广义线性混合效应模型gydF4y2Balme4gydF4y2Ba软件包来评估测序方法的可靠性,并选择1%的错误频率阈值,低于该阈值,多态性可能是测序过程的人工产物。在这个分析中,我们将多态性定义为少数变体。即将所有非多数碱基汇总计算为错配,并计算基因组位点的错配比例gydF4y2Ba我gydF4y2Ba从样本gydF4y2BajgydF4y2Ba(gydF4y2BajgydF4y2Ba= 1,…,gydF4y2BakgydF4y2Ba)被定义为gydF4y2BapgydF4y2BaijgydF4y2Ba据估计gydF4y2BaxgydF4y2BaijgydF4y2Ba/gydF4y2BangydF4y2BaijgydF4y2Ba在哪里gydF4y2BaxgydF4y2BaijgydF4y2Ba不匹配的总数是和gydF4y2BangydF4y2BaijgydF4y2Ba是覆盖率。为了找到最优的误差阈值,我们对现场的不匹配进行了建模gydF4y2Ba我gydF4y2Ba从样本gydF4y2BajgydF4y2Ba由于是由二项分布产生的,gydF4y2BaxgydF4y2BaijgydF4y2Ba~ Binom (gydF4y2BangydF4y2BaijgydF4y2Ba,gydF4y2BapgydF4y2BaijgydF4y2Ba)。我们将不匹配频率和误差变化的站点间变化建模为logit尺度上的正态分布偏差:logit(gydF4y2BapgydF4y2BaijgydF4y2Ba) =gydF4y2BaμgydF4y2BajgydF4y2Ba+γgydF4y2Ba我gydF4y2Ba+εgydF4y2BaijgydF4y2Ba,在那里gydF4y2BaμgydF4y2BajgydF4y2Ba样本中所有站点的logit不匹配频率平均值是多少gydF4y2BajgydF4y2Ba,γgydF4y2Ba我gydF4y2Ba~ n (0, σgydF4y2BaγgydF4y2Ba2gydF4y2Ba)表示位点间真正的失配变化,εgydF4y2BaijgydF4y2Ba~ n (0, σgydF4y2BaεgydF4y2Ba2gydF4y2Ba)表示重复测序样本之间不共享的非二项误差变异。我们将同一样本重复序列之间的相关性定义为潜在尺度的类内相关系数ICC = σgydF4y2BaγgydF4y2Ba2gydF4y2Ba/(σgydF4y2BaγgydF4y2Ba2gydF4y2Ba+σgydF4y2BaεgydF4y2Ba2gydF4y2Ba)。在相同样本的重复序列之间,真正多态性的频率应该是高度相关的,而误差变异应该是不相关的。对于每一组重复测序的样本,我们估计ICC从gydF4y2BaxgydF4y2BaijgydF4y2Ba和gydF4y2BangydF4y2BaijgydF4y2Ba从20个具有更严格误差阈值的数据集。在第一个数据集中,去除平均错配频率低于0.1%的位点,并以0.1%的步骤将该错误阈值提高到2.0%。来自可比数据的误差阈值通常在0.5-1.0%之间[gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba]。我们推断,ICC应该随着错误阈值的增加而增加,一旦超过正确的错误阈值,ICC就会趋于平稳。为了检测ICC在更高的误差阈值下趋于平稳的人为趋势,我们从三个数据集估计ICC,其中站点身份被随机排列以消除两个样本之间的相关性(期望ICC = 0)。使用此分析选择的低于1%阈值的变异频率被假定为错误并设置为零。gydF4y2Ba
Consensus-level多样性gydF4y2Ba
在排除平均覆盖率低于20倍或第10百分位覆盖率低于5倍的序列后,称为共识序列(尽管,与多样性分析一样,所有符合第一个标准的样本也符合第二个标准)。我们将其称为20倍覆盖数据集。通过分离位点的数量来衡量每个实验中共识序列之间和现场样本之间的多样性。为了说明野外暴发样本的遗传结构,利用每对一致序列之间不同位点的遗传距离来测量遗传距离,构建了一个无根邻居连接(NJ)系统发育树。我们仅使用SH + G区域重新构建了NJ树,以测试是否可以通过部分测序获得同等信息的系统发育树。gydF4y2Ba
Within-sample多样性gydF4y2Ba
每个位点的样本内多样性被量化为少数变异的频率(仅包括频率高于1%误差阈值的变异)。也就是说,所有非多数碱基被汇总并计算为错配,并在现场计算错配频率gydF4y2Ba我gydF4y2Ba(gydF4y2Ba我gydF4y2Ba= 1,…,gydF4y2BaNgydF4y2Ba)被定义为gydF4y2BapgydF4y2Ba我gydF4y2Ba估计为gydF4y2BaxgydF4y2Ba我gydF4y2Ba/gydF4y2BangydF4y2Ba我gydF4y2Ba在哪里gydF4y2BaxgydF4y2Ba我gydF4y2Ba不匹配的总数是和gydF4y2BangydF4y2Ba我gydF4y2Ba覆盖范围在现场吗gydF4y2Ba我gydF4y2Ba。利用香农多样性估算样本内的多样性,gydF4y2Ba\ (H = - \ sum_ {i = 1} ^ {N} {p} _{我}\ mathrm {ln} ({p} _{我})\)gydF4y2Ba,在那里gydF4y2Ba\ (0 \ * \ mathrm {ln} (0) \)gydF4y2Ba等于0。通过绘制每个位点的香农多样性图(定义为单个位点对香农多样性的贡献)来考察多样性沿基因组的分布。我们还将多样性估计为错配频率高于1%误差阈值的位点的比例。为了研究SH + G区域在共识水平上的更大多样性是否扩展到亚共识多样性,将每个样本中的两种多样性测量方法在SH + G和基因F之间进行了比较,根据以往的研究,预计F比SH + G变化更小[gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba在SH + G和整个BRSV基因组之间。gydF4y2Ba
瓶颈大小和传输树距离gydF4y2Ba
我们假设样本内多样性可以帮助推断传输链路。我们使用两种方法来验证这一假设,这两种方法在容忍测序错误的同时利用了样本内的多样性:一种基于群体遗传模型的方法,用于估计传播瓶颈的大小[gydF4y2Ba42gydF4y2Ba];和gydF4y2BaBadTrIPgydF4y2Ba,一种贝叶斯传输树重建方法[gydF4y2Ba26gydF4y2Ba]。gydF4y2Ba
我们将瓶颈方法应用于实验G个样本,其中传输树已知(图1)gydF4y2Ba1gydF4y2Ba),仅使用超过500 ×覆盖度阈值的样本,通过估算两两传播树距离与两两共享遗传多样性之间的相关性。如果样本内多样性是传递树的信息,我们会期望每对样本之间的传递步骤数量与它们共享的遗传多样性数量之间呈负相关。使用精确的β -二项方法来估计瓶颈大小及其95%置信区间(CI),该方法在Sobel Leonard等人的R脚本中实现,经过修改以减少计算时间[gydF4y2Ba43gydF4y2Ba]。瓶颈大小可以解释为来自感染供体的单个病毒粒子在受体中建立新种群的数量。它是共享遗传多样性的一种度量,因为通过传播瓶颈转移的遗传多样性的数量与其大小成正比。如果在被多个传输步骤分开的供方和受方之间估计瓶颈大小,则预计类似大小的重复瓶颈会累积起作用,从而导致预计传输树距离与感知瓶颈大小之间存在负相关关系。我们还估计了超过500倍覆盖率阈值的所有对现场爆发样本之间的瓶颈大小。对于实验G,我们只估计已知传播方向的瓶颈大小,对于现场暴发样本,传播方向未知或不适用,因此我们估计两个方向的瓶颈大小。在没有已知传播树的情况下,我们使用时空接近度和接触者追踪数据来分配顺序的“流行病学距离”类别:(1)同一农场;(2)有明显接触证据的不同养殖场;(3)有中度接触证据的不同养殖场;(4)不同养殖场无接触证据。gydF4y2Ba
的gydF4y2BaBadTrIPgydF4y2Ba方法利用蒙特卡罗马尔可夫链(Monte Carlo Markov chain, MCMC)从基因组多样性数据中推断出传播树的后验分布,以每个位点的4个观察到的碱基频率的形式。gydF4y2BaBadTrIPgydF4y2Ba作为一个包实现(gydF4y2BaBADTRIPgydF4y2Bav1.0.1)gydF4y2Ba野兽gydF4y2Bav2.5.2 [gydF4y2Ba44gydF4y2Ba],并应用于实验G的样本和现场爆发样本。为了使蒙特卡洛马尔可夫链(MCMC)在几天而不是几周的时间尺度上收敛,有必要只分析两到五个样本的子集。出于同样的计算可行性原因,覆盖率随机下采样至最大50倍。通过这些调整,gydF4y2BaBadTrIPgydF4y2Ba平均在4天(范围1-20)内收敛,其中收敛被定义为超过有效样本量(ESS),从后验模型概率至少超过200,从所有参数至少超过100。为了分离遗传数据对传播树重建的贡献,提供了相同的非信息流行病学数据gydF4y2BaBadTrIPgydF4y2Ba对于每个样品,所有采样时间设置为第0天,所有曝光窗口设置为±100天。将PoMo模型的虚拟种群大小设为25,对测序错误率进行估计而非固定,基频固定为样本均值频率,F81替代模型[gydF4y2Ba45gydF4y2Ba是假定的。瓶颈大小的对数正态先验分布均值为0,标准差为2。gydF4y2Ba
结果gydF4y2Ba
序列质量分析gydF4y2Ba
国家统计局样本的平均覆盖率(gydF4y2BangydF4y2Ba= 8)高出10.7倍(95% CI: 3.9, 35.3;χgydF4y2Ba2gydF4y2Badf = 1gydF4y2Ba= 14.41,gydF4y2BaPgydF4y2Ba= 0.0001;边际gydF4y2BaRgydF4y2Ba2gydF4y2Ba= 54%)比BAL样本(gydF4y2BangydF4y2Ba= 8)取自相同的四只动物(附加文件gydF4y2Ba1gydF4y2BaH),因此BAL不能传递覆盖优势来抵消更大的采样成本。NS样本的覆盖率与Ct值呈负相关(gydF4y2BangydF4y2Ba= 37个序列;χgydF4y2Ba2gydF4y2Badf = 1gydF4y2Ba= 4.71,gydF4y2BaPgydF4y2Ba= 0.030),但在BAL样本中没有关联的证据(gydF4y2BangydF4y2Ba= 34个序列;χgydF4y2Ba2gydF4y2Badf = 1gydF4y2Ba= 0.29, p = 0.59;交互χgydF4y2Ba2gydF4y2Badf = 1gydF4y2Ba= 4.78,gydF4y2BaPgydF4y2Ba= 0.029)。然而,在两种样本类型中,由Ct变化解释的对数转换覆盖率的方差比例都很低(BAL为0.5%,NS为11.3%),这表明无论采用何种抽样方法,Ct值都不是覆盖率的有力预测因子。SH + G区域的平均盖度比全基因组的平均盖度高13%(几何平均值分别为349 ×和310 ×);对数的配对t检验gydF4y2Ba10gydF4y2Ba意味着覆盖:gydF4y2BatgydF4y2Badf = 49gydF4y2Ba= 2.09,gydF4y2BaPgydF4y2Ba= 0.042),而在低覆盖度样本(覆盖度< 500 ×)中,几何平均值分别为92 ×和83 ×;gydF4y2BatgydF4y2Badf = 29gydF4y2Ba= 1.46,gydF4y2BaPgydF4y2Ba= 0.156)。这些结果表明,总体覆盖度较低的样本在SH + G区域内的覆盖度也较低,因此不太可能通过集中在该区域进行拯救。gydF4y2Ba
病毒多样性数据的可靠性gydF4y2Ba
对相同样本重复序列的分析表明,该测序方法对频率在大约1%以上的多态性给出了可靠的结果。在超过一次测序的10个样本中的9个样本中,ICC和错误阈值之间的关系大致符合预期模式,在最低的错误阈值处从0.4上升到0.8,在9个样本中有7个样本高于0.9,另外两个样本约为0.7(附加文件)gydF4y2Ba1gydF4y2Ba这种模式表明,正如预期的那样,较低频率的多态性不太可靠,因此更有可能是错误的,而测序方法是可靠的。因此,合并来自重复序列的FastQ文件,得到最终的多样性分析数据集,该数据集由16个实验样本和4个野外暴发样本的序列组成,覆盖率至少为500倍,多态性频率低于1%的误差阈值设置为零(表1)gydF4y2Ba2gydF4y2Ba)。gydF4y2Ba
Consensus-level多样性gydF4y2Ba
从27只动物和6种攻毒病毒收集的50个测序样本中,来自26只动物和6种攻毒病毒的47个序列通过了共识序列的覆盖标准(20 ×覆盖数据集)。47个一致序列被存入NCBI核苷酸数据库(登录号见附加文件)gydF4y2Ba1gydF4y2BaB).每个样本沿基因组的覆盖分布以及与参考序列对应的reads比例(平均值:3.8%;范围:0.1-24.0%)在附加文件中报告gydF4y2Ba2gydF4y2Ba。在产生多个测序样本的五个实验(B、C、D、F和G)中,以分离位点数量衡量的共识水平多样性水平非常低(表1)gydF4y2Ba2gydF4y2Ba;额外的文件gydF4y2Ba3.gydF4y2Ba)。在实验B中,攻击病毒与感染后4-6天从两头小牛身上采集的5个样本之间没有观察到共识水平的变化;或在实验G中,攻击病毒与8个样本之间在初次感染后长达21天和三个传播步骤。在实验C中,有一个共识水平的多态性与挑战病毒有关,在6个样本中的3个样本中发现,两只小牛(表1)gydF4y2Ba3.gydF4y2Ba)。这种多态性在同一天用不同方法(BAL和NS)从同一动物身上采集的两个样本之间也存在差异。然而,在6个实验C样本中,该位点的少数变异频率接近50%的共识阈值(范围为39.6-49.6%),因此这些样本之间的共识序列差异反映了该位点病毒群体多样性差异的窄谱。在实验D中,在来自一只动物的两个样本中观察到与攻毒病毒有关的一个共识水平的变化,但在来自另一只动物的单个样本中没有观察到。实验D中的两只动物都直接被攻毒病毒感染。在实验C中,在基因L的第305个氨基酸上也观察到相同的共识水平的变化,但在实验F中也观察到相反的方向(表1)gydF4y2Ba3.gydF4y2Ba)。这是在感染后4-7天从五个宿主收集的13个实验F样本之间的一致序列中唯一不同的位点,存在于来自两头小牛的三个样本的一致序列中。其他6个位点在宿主样本和攻击病毒之间存在差异,但在宿主样本之间没有差异。在实验样本中观察到的共识水平变化的次共识多样性揭示了一个共同的模式。在实验C、D和F中观察到的小牛的所有共识水平变异在攻毒病毒中以低于共识的频率出现,频率从15%到40%不等,这表明共识水平的多样性来自攻毒病毒中预先存在的遗传变异,而不是来自新的突变。gydF4y2Ba
与40个实验样本相比,7个实地样本之间的共识水平多样性相对较高,观察到146个隔离点。2个Jämtland牧群(BE和BK)和3个Uppland牧群(AI、AJ和AL)之间存在明显的多样性分化,144/146个多态性具有区域特异性(图2)gydF4y2Ba2gydF4y2BaB;额外的文件gydF4y2Ba3.gydF4y2Ba)。每个地区的畜群之间只有一个共识水平的差异。gydF4y2Ba
正如预期的那样,SH + G区域的共识水平多样性高于整个基因组,在7个野外暴发序列之间有27/1344(2.0%)个位点存在差异,而在整个基因组中只有146/ 14562(1.0%)个位点存在差异。因此,SH + G区域仅在9%的基因组中占据了18%的共识序列多样性。此外,全基因组树和SH + gnj树高度相似(图2)gydF4y2Ba2gydF4y2BaB和C)。gydF4y2Ba
Within-sample多样性gydF4y2Ba
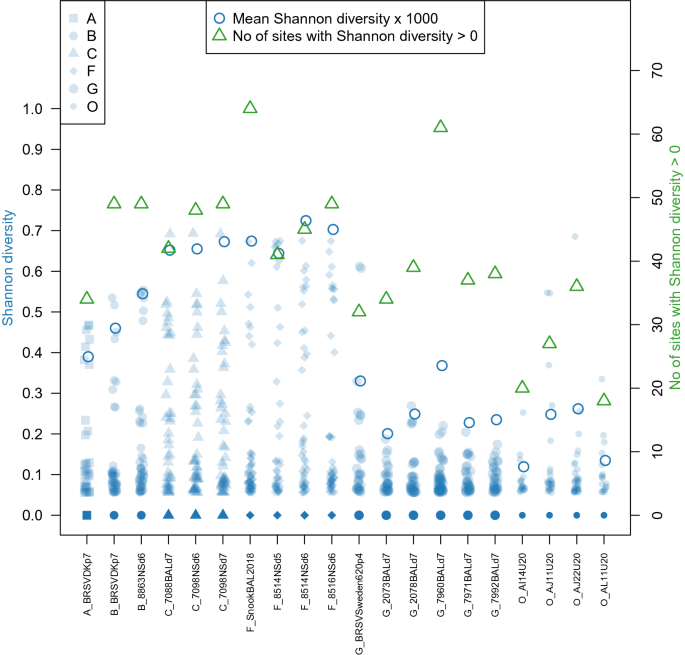
利用Shannon多样性和多样性位点数对500 ×覆盖度数据集中20个序列的多样性进行量化(图2)gydF4y2Ba3.gydF4y2Ba)。与共识水平的多样性相反,实验样本的样本内多样性往往高于暴发样本,尽管实验G的多样性低于其他实验,接近实地样本的水平。在实验样本中,超过1%误差阈值的亚共识多样性位点的数量为32至64个(基因组的0.2-0.4%),在野外暴发样本中为18至36个(基因组的0.1-0.2%)(图2)gydF4y2Ba3.gydF4y2Ba(绿色三角形)。在500倍覆盖率数据集中的5个实验G样本中,更高的频率多样性尤其稀缺,任何样本中都没有位点的错配频率超过10%,尽管在攻毒病毒样本中有3个这样的位点。在实验A、B、C和F中发现了较高水平的样本内共同变异,每个样本有6至12个位点高于10%错配频率阈值,在4个实地暴发样本中处于中间水平(范围为0-4个位点,错配频率为10%)。gydF4y2Ba
500 ×覆盖序列集的20个样本的样本内多样性。gydF4y2Ba图中绘制了三种多样性指标:每个地点的香农多样性(蓝色阴影点,左轴);整个基因组的平均香农多样性(开放的蓝色圆圈,左轴)乘以1000,以便与特定地点的香农多样性在相同的尺度上绘制;和Shannon多样性站点数量> 0(绿色三角形,右轴)。假设小于1%的基本比例是由于NGS误差造成的,并设置为零,因此对多样性估计没有贡献。每个样本的平均点位数为14 562个(范围为14 561-14 563)。实验G的挑战病毒BRSV/Sweden/HPIG-SLU-620-Lovsta/2016,缩写为G_BRSVSweden620p4。gydF4y2Ba
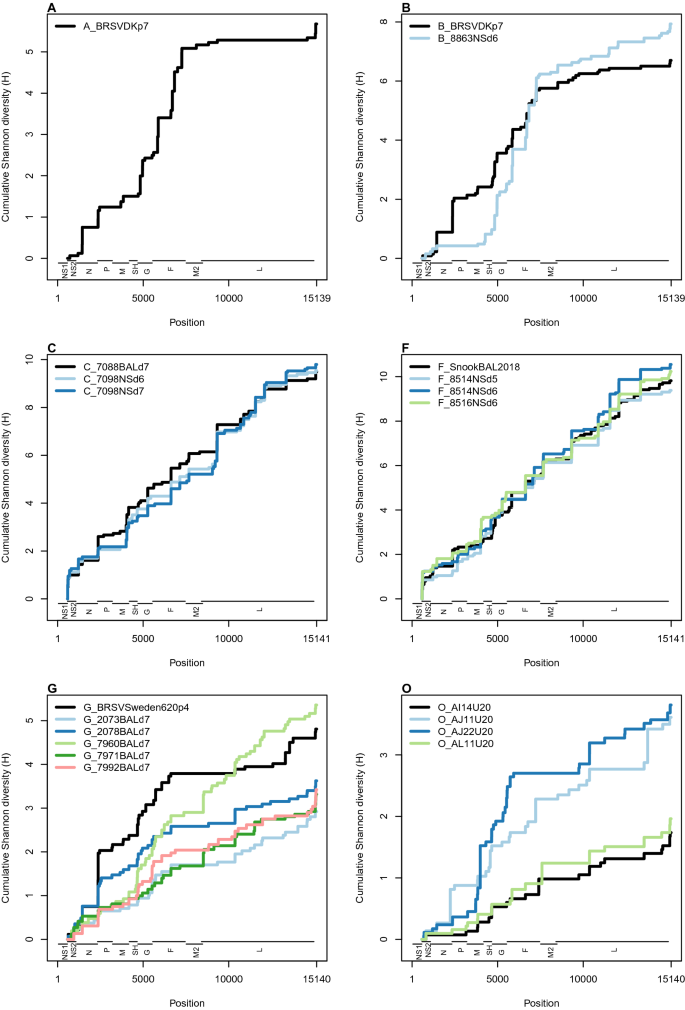
Shannon多样性的累积分布如图所示gydF4y2Ba4gydF4y2Ba。在实验A和B中,多样性最高的位置在5000 ~ 7500之间,这个区域大致跨越了G、F和M2基因。在实验C和F中,多样性沿基因组均匀分布。实验G多样性最高的区域约为2000-6000个,包括P、M、SH和G基因。4个野外暴发样本中3个(AI14U20、AJ11U20、AL11U20)的多样性沿基因组分布近似均匀。其余大田样品AJ22U20多样性最高的区域位于4000 ~ 6000位点之间,覆盖SH和G基因。实验A、B和G的几个序列以及大田暴发的几个序列在3 '未翻译的尾部区域的15050 ~ 15100位点附近的多样性急剧增加。gydF4y2Ba
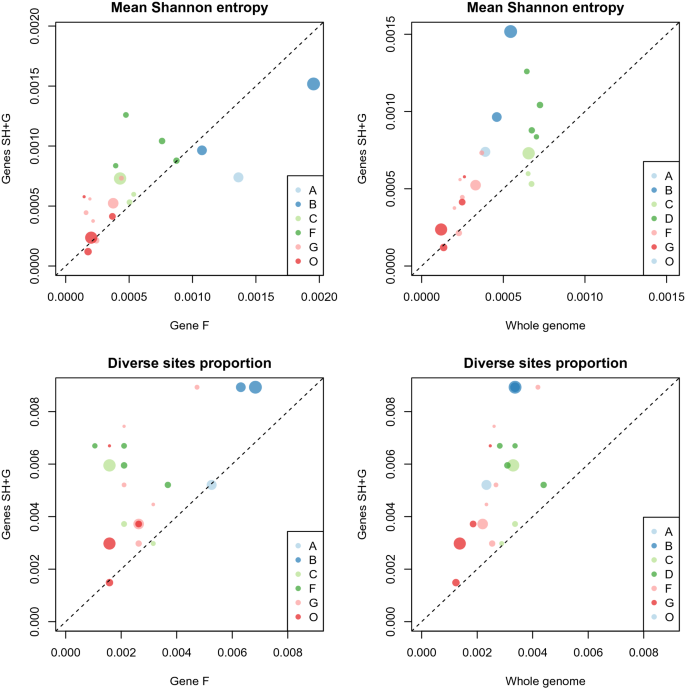
基因SH和基因G之间的多样性比较,其中多样性被假设集中,基因F表明,对于大多数实验和现场暴发样本,意味着Shannon多样性更高,基因SH和基因G比基因F有更多的多样性位点(图1)gydF4y2Ba5gydF4y2Ba)。这种模式的主要例外是实验A和B,其中基因f的多样性较高。与整个BRSV基因组的平均多样性相比,基因SH和G的多样性趋势更加明显。gydF4y2Ba
瓶颈大小和传输树距离gydF4y2Ba
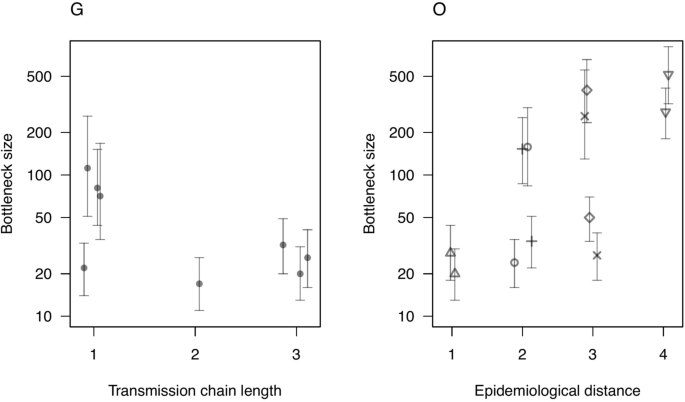
来自实验G的6个样本和来自现场暴发的4个样本都是在4天内在Uppland地区采集的,它们都通过了500 ×覆盖阈值,因此适合用于估计传播瓶颈大小(图2)gydF4y2Ba6gydF4y2Ba;额外的文件gydF4y2Ba1gydF4y2BaC).实验G的6只动物之间的瓶颈大小为17至112个病毒粒子,而4个实地暴发样本之间每个方向的6个瓶颈大小估计为20至516个。由于通过500 ×覆盖率阈值的样本数量较少,因此无法推断传输树距离与瓶颈大小之间的关系。gydF4y2Ba
在实验G的样本和现场暴发的样本之间没有推断出高可信度的传播联系gydF4y2BaBadTrIPgydF4y2Ba从BE农场到BK农场的传播方向推断的置信度最高,概率为68%,这与基于接触者追踪的疑似传播方向一致;也就是说,据估计,疑似传播方向的可能性大约是相反方向的两倍(表1)gydF4y2Ba4gydF4y2Ba)。对于其余的野外爆发样本和实验G,发现所有可能的树的概率大致相等。表格gydF4y2Ba4gydF4y2Ba总结的结果gydF4y2BaBadTrIPgydF4y2Ba与来自共识序列的证据一起进行分析,并对现场暴发样本进行接触者追踪,同时详细分析gydF4y2BaBadTrIPgydF4y2Ba结果显示在附加文件中gydF4y2Ba1gydF4y2BaJ和K。gydF4y2Ba
讨论gydF4y2Ba
BRSV是牛呼吸道疾病的主要原因,在大多数生产系统中,预防BRSV感染至关重要。当疫情激增时,许多农民依靠生物安全措施而不是疫苗接种来阻止该病毒进入其畜群。为提高效率,生物安全措施需要根据病毒的生命周期和所有病毒传播方式进行调整。为了了解BRSV是如何在农场中引入或维持的,我们评估了全病毒基因组序列分析是否比部分测序能提供更好的分辨率,以更好地追踪病毒。为此,在首次BRSV全基因组测序研究中,我们使用Illumina MiSeq平台建立了可靠的深度测序方法(目标1),我们使用该方法在共识和亚共识水平上量化瑞典牛群的实验传播和现场爆发的基因组多样性(目标2)。我们评估了使用共识水平和亚共识水平基因组多样性来推断牛个体和牛群之间传播联系的可行性(目标3)。最后,我们研究了亚共识多样性与样本类型、Ct值和基因组位置之间的关系(目标4)。我们通过分析来自实验和现场暴发BRSV样本的47个全病毒基因组测序来实现这些目标。gydF4y2Ba
对来自Uppland三个农场和Jämtland两个农场的一致性序列的差异分析表明,在两个区域对应的两个主要进化支之间存在强烈的基因组差异,这两个区域相距约500公里,在区域内的局部(< 20公里)尺度上差异很小。基因组变异的强烈区域聚类表明,这两种病毒种群在独立进化的时间尺度远远大于可能分隔区域内样本的几天或几周,受感染动物在区域之间的移动非常有限。这提出了一种令人兴奋的可能性,即共识序列可用于确定新的BRSV暴发的起源区域。然而,需要对来自瑞典各地的更大的序列样本进行分析,以确定共识序列变异的地理结构模式及其在追踪区域间疫情方面的潜在效用。这样的分析,如果包括小于20公里规模的农场,也可以揭示我们所观察到的非常低水平的区域内农场之间的共识序列差异-并且对追踪当地农场之间传播的前景没有希望-是否是整个瑞典的典型。值得注意的是,当地农场间规模(0.5-20公里)的一致性序列差异与沿着短实验传播链的动物之间观察到的差异相似。例如,实验G的共识序列在21天内和三个传播步骤之间没有差异。gydF4y2Ba
根据使用G和SH基因部分序列的有限研究,似乎具有相似基因序列的病毒在时间和空间上聚集[gydF4y2Ba12gydF4y2Ba,gydF4y2Ba46gydF4y2Ba]。在其他几个实地研究中观察到的BRSV的遗传多样性可能是从其他地区引入新谱系的结果,而不是反映BRSV在局部传播后的快速进化[gydF4y2Ba14gydF4y2Ba,gydF4y2Ba47gydF4y2Ba,gydF4y2Ba48gydF4y2Ba,gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba,gydF4y2Ba51gydF4y2Ba,gydF4y2Ba52gydF4y2Ba,gydF4y2Ba53gydF4y2Ba,gydF4y2Ba54gydF4y2Ba,gydF4y2Ba55gydF4y2Ba,gydF4y2Ba56gydF4y2Ba,gydF4y2Ba57gydF4y2Ba]。的确,正如Larsen等人先前所表明的那样。[gydF4y2Ba13gydF4y2Ba]和Deplanche等gydF4y2Ba。gydF4y2Ba[gydF4y2Ba58gydF4y2BaG基因的一致序列在犊牛传代和细胞培养过程中是非常稳定的。实验G中BRSV连续在犊牛间传代3次后,临床体征逐渐增加(数据未显示)。因此,我们预计遗传变异可能出现在基因组的其他部分,而不是g基因。然而,尽管在小牛连续传代之间临床表达增加(数据未显示),但在三次传代中没有出现共识水平的变异,而在每次传代测序的病毒中检测到的亚共识多样性程度并不大于在挑战病毒中引入的低水平(图1)gydF4y2Ba3.gydF4y2Ba)。gydF4y2Ba
从深度测序数据推断的一致性序列的分析丢弃了样本内基因组多样性的信息,这可以提供有关传播链接的信息。因此,我们采用了两种利用样本内多样性来推断瓶颈大小的方法[gydF4y2Ba42gydF4y2Ba]及传播途径(gydF4y2BaBadTrIPgydF4y2Ba) [gydF4y2Ba26gydF4y2Ba在已知传播树的实验样本与可获得流行病学距离信息的现场暴发样本之间进行比较。既没有使用瓶颈大小估计方法,也没有使用传输路由推断方法gydF4y2BaBadTrIPgydF4y2Ba是关于传播链接的信息,这表明样本内多样性在推断本地传播路径时可能没有价值。然而,从这些结果中得出悲观的结论是值得注意的。在瓶颈分析中,覆盖率足够高的样本太少,无法得出瓶颈大小与传输树距离之间关系的结论,因此使用更大的样本集仍有可能发现两者之间的关系。同样,严重的下降采样覆盖率要求gydF4y2BaBadTrIPgydF4y2Ba就统计能力而言,计算上可行的方法可能代价高昂,特别是在实验G中,因为它相对于其他实验的多样性水平较低。因此,尽管这些结果并不令人乐观,但从样本内序列多样性推断传播途径的其他方法(例如:gydF4y2Ba25gydF4y2Ba])应用于更大的BRSV病例样本将在推断传播途径方面取得更大的成功,特别是在结合流行病学数据时。gydF4y2Ba
值得注意的是,尽管假定现场暴发样本之间存在较大的流行病学差异,但实验G(17-112)和现场暴发样本(20-516)之间的瓶颈大小估计大致重叠,这将预测较小的瓶颈大小。以往,无论是通过实验还是在自然暴发中,都没有估计过BRSV的传播瓶颈大小。使用不同的统计方法,Lau等。[gydF4y2Ba59gydF4y2Ba]估计成人HRSV实验感染的下限瓶颈大小为25,这在我们研究中估计的瓶颈大小范围内。Orton等人在我们的研究中采用了类似的测序和统计方法。[gydF4y2Ba60gydF4y2Ba估计在暴发样本(范围2-9)和实验传播(范围4-39)中FMDV传播瓶颈狭窄。这里估计的中度至狭窄传播瓶颈对病毒进化的影响(例如,对疫苗接种的反应)是难以预测的[gydF4y2Ba61gydF4y2Ba]。理论上,狭窄的瓶颈可能会阻碍适应,例如,通过增加有害突变的频率或减少总体遗传变异,或通过快速建立宿主间病毒多样性来促进适应。gydF4y2Ba
BRSV体外链结样本中G基因序列的样本间和样本内多样性水平(gydF4y2BangydF4y2Ba= 4),体内(gydF4y2BangydF4y2Ba= 3) Deplanche等对实验进行评价。[gydF4y2Ba58gydF4y2Ba]。与本研究一致,在7个样本之间没有观察到共识水平的变化,而通过对每个样本的26-29个克隆进行测序,显示出低但显著的亚共识多样性水平,估计为0.7-1.0 × 10gydF4y2Ba−3gydF4y2Ba每个核苷酸的取代。我们使用二项累积分布函数估计替代率为10gydF4y2Ba3gydF4y2Ba如果我们将Deplanche等人的研究(基因G中约27个序列,约660 nt)扩大到我们的研究(整个基因组的平均覆盖率为1413 × 14 562 nt),那么在整个基因组中均匀操作的每个核苷酸将不会产生超过1%的误差阈值的多样性。虽然我们在比较使用不同测序技术获得的全基因组序列和部分基因组序列的多样性时应该谨慎,但比较表明,Deplanche等人观察到的显著差异水平并不一定高于本研究中观察到的全基因组差异水平,其中多样性最少的样本中有18个位点的多样性高于1%的误差阈值。对于口蹄疫等其他病毒,以前估计样本内的遗传多样性程度为0.31 × 10gydF4y2Ba−3gydF4y2Ba每个核苷酸的取代数[gydF4y2Ba21gydF4y2Ba, 0.21 × 10gydF4y2Ba−3gydF4y2Ba每个核苷酸的取代数[gydF4y2Ba62gydF4y2Ba], 0.07 ~ 0.77 × 10gydF4y2Ba−3gydF4y2Ba每个核苷酸的取代数[gydF4y2Ba63gydF4y2Ba表明类似的短期替代率与当前的研究基本一致。BRSV样本中缺乏遗传多样性和一致性序列的强稳定性可以用病毒复制后产生的绝大多数突变体更强的负选择压力来解释。gydF4y2Ba
G和SH被认为是基因组中最易变的部分,因此被用于研究RSV的多样性和进化[gydF4y2Ba12gydF4y2Ba,gydF4y2Ba46gydF4y2Ba]。因此,本研究的第二个目的是比较部分基因组序列,特别是基因组中更多样化的SH + G区域,与全基因组在追踪传播途径方面的可用信息。我们确认样本内多样性集中在SH + G区域,从多样性分布的峰值可以看出(图1)gydF4y2Ba4gydF4y2Ba),并通过比较SH + G与基因F和整个基因组之间的多样性估计值(图2)gydF4y2Ba5gydF4y2Ba)。在共识水平上,我们表明SH + G区域的多样性大约是整个基因组的两倍,并且与整个基因组相比产生了几乎相同的系统发育树。这一结果提出了一种可能性,即SH + G测序可能比全基因组测序更具成本效益,例如在瑞典建立BRSV地理结构的大规模分析中。然而,低覆盖率序列在SH + G区域的覆盖率并不高于整个基因组的覆盖率,这表明专注于SH + G不太可能导致低覆盖率样本的恢复。然而,使用特定的巢式RT-PCR对该区域进行扩增和测序可能有助于在病毒RNA水平较低的样品中鉴定BRSV分离物。gydF4y2Ba
全基因组测序方法的一个局限性是,序列必须具有足够的覆盖率才能使用,而平均覆盖率变化很大(本研究的范围为1 ×至8614 ×)。50个测序样本中有3个未进行分析,其平均覆盖率低于我们为共识序列设定的20 ×阈值,30个序列未达到样本内多样性分析的500 ×阈值。因此,能够预测高覆盖率样本,并利用这些信息来优化覆盖率,并在浪费资源进行测序之前识别低覆盖率样本将是有价值的。在本研究中,样本采集方法强烈预测了平均覆盖率,相对方便且无侵入性的鼻拭子法比支气管肺泡灌洗法的覆盖率更高,这表明鼻拭子应用于未来的BRSV病例采样。然而,Ct值并不能预测平均覆盖率,因此似乎没有潜力优先考虑高覆盖率的样本。然而,具有非常高的Ct值的样品已经被过滤掉了,所以这个结果表明,额外的过滤将是无用的。gydF4y2Ba
分析BRSV的全基因组序列可以区分遥远地区的流行分离株。然而,在对有限数量的实地样本进行体内传播研究和分析后,在具有密切流行病学联系的分离株中观察到非常低水平的突变和多样性。因此可以得出结论,尽管全基因组测序可以提高分辨正在传播的分离株的分辨率,但该病毒没有显示出足够程度的快速进化和多样性,无法在没有流行病学信息支持的情况下在群体水平上进行高度可靠的追踪。gydF4y2Ba
数据和材料的可用性gydF4y2Ba
本研究生成的核苷酸序列数据可在NCBI SRA和nucleotide数据库中获得,并在BioProject PRJNA893434下收集。NCBI加入号在附加文件中提供gydF4y2Ba1gydF4y2BaB.所有其他生物信息学和数据分析脚本可在格拉斯哥大学的启发研究数据库(gydF4y2Bahttps://doi.org/10.5525/gla.researchdata.1287gydF4y2Ba)。gydF4y2Ba
参考文献gydF4y2Ba
Valarcher JF, Taylor G .(2007)牛呼吸道合胞病毒感染。箴言38:153-180gydF4y2Ba
Stokstad M, Klem TB, Myrmel M, Oma VS, Toftaker I, Osteras O, Nodtvedt A(2020)利用生物安全措施防治牛呼吸道疾病:挪威牛呼吸道合胞病毒和牛冠状病毒控制规划。《前沿兽医科学》7:7 7gydF4y2Ba
De Jong MC, van der Poel WH, Kramps JA, Brand A, van Oirschot JT, Mourits MC, nienen M, Frankena K, Schukken YH(1996)奶牛呼吸道合胞体病毒种群持续性和复发性暴发的定量调查。Am J Vet Res 57:628-633gydF4y2Ba
Thomas LH, Stott EJ, Jones PW, Jebbett NJ, Collins AP(1980)呼吸道合胞病毒的可能作用gydF4y2Ba巴斯德菌gydF4y2Ba小牛呼吸道疾病中的SPP。Rec 107:304-307gydF4y2Ba
张建军,张建军,张建军,张建军,张建军,张建军。(2001)牛呼吸道合胞病毒对B淋巴细胞持续感染的影响。病毒学291:55 - 67gydF4y2Ba
Van der Poel WH, Langedijk JP, Kramps JA, Middel WG, Brand A, Van Oirschot JT(1997)牛中持续存在牛呼吸道合胞病毒的血清学指征和检测该病毒的尝试。Arch Virol 142:1681-1696gydF4y2Ba
Mars MH, Bruschke CJ, van Oirschot JT(1999)在实验条件下,BHV1, BRSV和BVDV在牛之间的空气传播是可能的。兽医微生物,66:197-207gydF4y2Ba
Ohlson A, Emanuelson U, Traven M, Alenius S(2010)牛冠状病毒和牛呼吸道合胞病毒抗体水平与奶牛群体疾病发病率、繁殖和群体特征的关系。兽医学报52:37gydF4y2Ba
张国荣,张国荣,张国荣,等(1980)呼吸道合胞病毒的传播途径。[J] .中国传染病杂志(英文版)gydF4y2Ba
李丽娟,李建军,李建军,李建军,王立军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军。[J] .中国生物医学工程学报,1998,19 (4):391 - 391gydF4y2Ba
(2000)牛呼吸道合胞病毒的进化。[J]中国生物医学工程学报(英文版)gydF4y2Ba
Larsen LE, Tjornehoj K, Viuff B(2000)在封闭牛群中反复爆发分离的牛呼吸道合胞病毒的广泛序列差异。[J]中华微生物学杂志,38 (4):422 - 422gydF4y2Ba
Larsen LE, Uttenthal A, Arctander P, Tjornehoj K, Viuff B, Rontved C, Ronsholt L, Alexandersen S, Blixenkrone-Moller M(1998)牛呼吸道合胞病毒(BRSV)的血清学和遗传学特征表明丹麦分离株属于中间亚群:没有证据表明先前的细胞培养适应和在细胞培养或小牛中传代对G蛋白核苷酸序列的变动性有选择性影响。兽医微生物杂志62:265-279gydF4y2Ba
biokhti MR, Traven M, Ohlson A, Zarnegar B, Baule C, Belak S, Alenius S, Liu L(2012)近期饲养场和奶牛群暴发的牛呼吸道合胞体病毒的系统发育分析。Arch Virol 157:601-607gydF4y2Ba
Spada E, Sagliocca L, soudis J, Garbuglia AR, Poggi V, De Fusco C, Mele A(2004)基于最小生成树模型的丙型肝炎病毒感染医院暴发的分子流行病学调查。[J]中国临床微生物学杂志42 (2):442 - 436gydF4y2Ba
张丽,Diaz RS, Ho DD, Mosley JW, Busch MP, Mayer A(1997)人类免疫缺陷病毒1型体内进化的宿主特异性驱动因子。[J]中国生物医学工程学报gydF4y2Ba
Wong CW, Albert TJ, Vega VB, Norton JE, Cutler DJ, Richmond TA, Stanton LW, Liu ET, Miller LD(2004)利用高通量、高密度重测序阵列追踪SARS冠状病毒的进化。基因组杂志14:398-405gydF4y2Ba
Savolainen C, Mulders MN, Hovi T(2002)连续流行季节收集的鼻病毒分离株的系统发育分析。病毒Res 85:41-46gydF4y2Ba
刘健,林士林,阮勇,凌爱玲,吴丽凤,Drosten C, Liu ET, Stanton LW, Hibberd ML (2005) SARS在新加坡传播模式的病毒序列变异分析。PLoS医学2:e43gydF4y2Ba
Cottam EM, Haydon DT, Paton DJ, Gloster J, wilessmith JW, Ferris NP, Hutchings GH, King DP(2006) 2001年英国口蹄疫病毒爆发的分子流行病学。[J]中国生物防治杂志80:11274-11282gydF4y2Ba
Cottam EM, King DP, Wilson A, Paton DJ, Haydon DT(2009)自然感染上皮内口蹄疫病毒核苷酸序列变异分析。病毒Res 140:199-204gydF4y2Ba
Cottam EM, Thebaud G, Wadsworth J, Gloster J, Mansley L, Paton DJ, King DP, Haydon DT(2008)利用遗传和流行病学数据确定口蹄疫病毒传播途径。生物学报(英文版)275:887-895gydF4y2Ba
Cottam EM, Wadsworth J, Shaw AE, Rowlands RJ, Goatley L, Maan S, Maan NS, Mertens PP, Ebert K, Li Y, Ryan ED, Juleff N, Ferris NP, wilessmith JW, Haydon DT, King DP, Paton DJ, Knowles NJ(2008) 2007年英国口蹄疫病毒传播途径。PLoS Pathog 4:e1000050gydF4y2Ba
张建军,张建军,张建军,张建军(2019)口蹄疫疫情传播网络模型的研究进展。科学报告9:4809gydF4y2Ba
李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军(2018)昆廷:基于病毒准种基因组数据的疾病传播重建。生物信息学34:163 - 170gydF4y2Ba
De Maio N, Worby CJ, Wilson DJ, Stoesser N(2018)基于基因组变异的疫情传播贝叶斯重建。计算机工程学报(英文版)14:e1006117gydF4y2Ba
李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军。生物进化35:719-733gydF4y2Ba
Alamil M, Hughes J, Berthier K, Desbiez C, Thebaud G, Soubeyrand S(2019)从深度测序数据推断流行病学联系:人类,动物和植物疾病的统计学习方法。[j] .中国生物医学工程学报,2016,34 (4):557 - 557gydF4y2Ba
贺隆德,胡克峰,李建军,张建军,张建军,张建军,张建军。(2004)牛呼吸道合胞病毒iscoms抗体的研究进展。疫苗23:646 - 655gydF4y2Ba
张晓明,张晓明,张晓明,张晓明,张晓明,张晓明,张晓明,张晓明,张晓明。(2011)牛呼吸道合胞体病毒iscoms的免疫和免疫保护。疫苗29:8719 - 8730gydF4y2Ba
刘建军,刘建军,刘建军,刘建军,刘建军,刘建军,刘建军,刘建军,刘建军,刘建军,刘建军,刘建军。(2014)重组牛呼吸道合胞病毒(BRSV)重组人呼吸道合胞病毒(RSV)亚单位疫苗的安全性和有效性评价。PLoS One 9:e100392gydF4y2Ba
Riffault S, Meyer G, Deplanche M, Dubuquoy C, Durand G, Soulestin M, Castagne N, Bernard J, Bernardet P, Dubosclard V, Bernex F, Petit-Camurdan A, Deville S, schwarz - cornil I, Eleouet JF(2010)基于核蛋白纳米颗粒的亚单位疫苗对小牛呼吸道合胞病毒的部分临床和病毒学保护。疫苗28:3722 - 3734gydF4y2Ba
Valarcher JF, Hagglund S, Naslund K, Jouneau L, Malmstrom E, Boulesteix O, Pinard A, Leguere A, Gauthier D, Dubuquoy C, Pietralunga V, Remot A, Falk A, Shevchenko G, Lind SB, Von Bromssen C, Vargmar K, Zhang BS, Kwong PD, Rodriguez MJ, Duran MG, schwarz - cornil I, Taylor G, Riffault S(2021)牛呼吸道合胞病毒(BRSV)特异性母源抗体免疫后长期保护的比较评价。疫苗9:236gydF4y2Ba
减少大量的gydF4y2Bahttps://www.bioinformatics.babraham.ac.uk/projects/trim_galore/gydF4y2Ba访问日期:2022年12月1日gydF4y2Ba
Viuff B, Uttenthal A, Tegtmeier C, Alexandersen S(1996)用原位杂交确定自然感染小牛的牛呼吸道合胞病毒复制位点。兽医病理学33:383-390gydF4y2Ba
Thomas LH, Gourlay RN, Stott EJ, Howard CJ, Bridger JC(1982)犊牛肺炎接种新微生物的研究。兽医科学33:170-182gydF4y2Ba
Heng L .(2013)利用BWA-MEM比对序列reads、克隆序列和装配序列。出来了。gydF4y2Bahttps://doi.org/10.48550/arXiv.1303.3997gydF4y2Ba
李辉,韩志强,李志强,李志强,李志强,李志强,李志强,阮杰,Homer N, Marth G, Abecasis G, Durbin R,基因组计划数据处理[J]。生物信息学25:2078 - 2079gydF4y2Ba
VSensusgydF4y2Bahttps://github.com/rjorton/VSensusgydF4y2Ba访问日期:2022年12月1日gydF4y2Ba
Orton RJ, Wright CF, Morelli MJ, King DJ, Paton DJ, King DP, Haydon DT(2015)从RT-PCR中识别低频突变和病毒深度测序数据的序列错误。BMC Genomics 16:229gydF4y2Ba
Stoler N, Nekrutenko A (2021) Illumina测序仪器的测序误差分析。NAR genombioinform 3:19gydF4y2Ba
Sobel Leonard A, Weissman DB, Greenbaum B, Ghedin E, Koelle K(2017)基于病原体深度测序数据的传播瓶颈大小估算及其在甲型流感病毒中的应用。[J]中国生物医学工程学报(英文版)gydF4y2Ba
瓶颈估计器gydF4y2Bahttps://github.com/pcdjohnson/BB_bottleneckgydF4y2Ba访问日期:2022年12月1日gydF4y2Ba
Bouckaert R, Heled J, Kuhnert D, Vaughan T, Wu CH, Xie D, Suchard MA, Rambaut A, Drummond AJ (2014) BEAST 2:贝叶斯进化分析的软件平台。计算机工程学报,10(4):393 - 393gydF4y2Ba
Felsenstein J (1981) DNA序列的进化树:最大似然方法。[J] vol . 17:368-376gydF4y2Ba
Valarcher JF, Bourhy H, Gelfi J, Schelcher F(1999)基于核蛋白基因的巢式逆转录- pcr诊断自发性和实验性牛呼吸道合胞病毒感染的评价。[J]中华微生物学杂志37 (7):1858 - 1869gydF4y2Ba
贾帅,姚翔,杨艳,牛超,赵艳,张翔,潘锐,姜鑫,肖波生,乔鑫,管鑫,徐艳(2021)东北牛呼吸道合胞病毒III亚群分离鉴定及系统发育分析。毒性12:404 - 414gydF4y2Ba
Giammarioli M, Mangili P, Nanni A, Pierini I, Petrini S, Pirani S, Gobbi P, De Mia GM(2020)意大利奶牛群的高致病性牛呼吸道合胞病毒变异。兽医科学6:740-745gydF4y2Ba
李春华,李春华,李春华,等(2018)牛呼吸道合胞病毒在意大利分离株的遗传特征:新分支循环的证据。[J]中国兽医学报(英文版)gydF4y2Ba
Klem TB, Rimstad E, Stokstad M(2014)挪威呼吸道疾病暴发中牛呼吸道合胞病毒的发生和系统发育分析。BMC兽医Res 10:15gydF4y2Ba
Nettleton PF, Gilray JA, Caldow G, Gidlow JR, Durkovic B, Vilcek S, Elvander M, Baule C, Uttenthal A, Ballagi-Pordany A, Belak S(2003)最近从英国分离的牛呼吸道合胞病毒与美国分离的菌株比早期的英国和目前的欧洲大陆分离的菌株更接近。[J] .兽医学[J] .中国兽医学报(自然科学版)gydF4y2Ba
熊谷一,川内K,安多h K, Hatama S .(2021)日本牛呼吸道合胞病毒G基因序列及其独特系统发育。[J]中国医学杂志,33 (3):162 - 166gydF4y2Ba
(2009)爱琴海地区牛呼吸道合胞病毒的遗传特征及危险因素分析。火鸡Trop动物健康项目54:4gydF4y2Ba
zyazici Z, Ozan E, Tamer C, Muftuoglu B, Barry G, Kurucay HN, Elhag AE, Cagirgan AA, Gumusova S, Albayrak H(2020)土牛呼吸道合胞病毒在土耳其牛中的传播:首次分离和分子鉴定。动物10:1700gydF4y2Ba
Kresic N, Bedekovic T, Brnic D, Simic I, Lojkic I, Turk N(2018)克罗地亚牛呼吸道合胞病毒的遗传分析。Comp Immunol microol infection Dis 58:52-57gydF4y2Ba
Timurkan MO, Aydin H, Sait A(2019)牛副流感病毒-3和牛呼吸道合胞病毒的鉴定和分子特征——来自土耳其的首次报告。[J]兽医学杂志63:167-173gydF4y2Ba
Leme RA, Dall Agnol AM, Balbo LC, Pereira FL, Possatti F, Alfieri AF, Alfieri AA(2020)巴西牛呼吸道合胞病毒野生型株的分子特征揭示了该病毒的遗传多样性和一个假定的新亚群。兽医Q 40:83-96gydF4y2Ba
Deplanche M, Lemaire M, Mirandette C, Bonnet M, Schelcher F, Meyer G(2007)牛呼吸道合胞病毒基因组准种分布的体内证据。[J]中国生物防治杂志(英文版)gydF4y2Ba
刘建伟,Kim YI, Murphy R, Newman R, Yang X, Zody M, DeVincenzo J, Grad YH(2017)来自成人感染研究和自然感染婴儿的RSV深度测序揭示异质多样化动态。病毒学510:289 - 296gydF4y2Ba
Orton RJ, Wright CF, King DP, Haydon DT (2020) FMDV在宿主内部和宿主之间传播的病毒瓶颈大小及其对病毒进化速度的影响。接口焦点10:20190066gydF4y2Ba
Zwart MP, Elena SF(2015)大小问题:病毒感染的遗传瓶颈及其对进化的潜在影响。《维罗尔年鉴》2:161-179gydF4y2Ba
顾俊杰,郑云云,张强,石丽玲,李勇,曲顺生(2006)利巴韦林作为RNA病毒诱变剂对口蹄疫病毒的抗病毒机制研究。[J]生物化学学报,39:9-15gydF4y2Ba
Airaksinen A, Pariente N, Menendez-Arias L, Domingo E(2003)用利巴韦林治疗持续感染细胞中的口蹄疫病毒涉及强化诱变。病毒学311:339 - 349gydF4y2Ba
致谢gydF4y2Ba
我们感谢Sabine Riffault提供的实验样品,感谢瑞典农业科学大学(SLU,瑞典乌普萨拉)、瑞典国家兽医研究所(SVA,瑞典乌普萨拉)和Val de Loire INRAE (PFIE,法国Nouzilly)的技术人员对实验动物的照顾。我们感谢兽医和农民参与处理现场疫情,感谢SLU的kv实验室提供技术援助。生命细胞平台部分由SLU基础设施委员会创立,感谢其提供的设施和设备。我们感谢Nicola di Maio(英国剑桥欧洲生物信息学研究所)协助实施gydF4y2BaBadTrIPgydF4y2Ba李超(英国格拉斯哥大学)协助向NCBI提交NGS数据。最后,我们感谢两位匿名审稿人的宝贵意见和建议。gydF4y2Ba
资金gydF4y2Ba
本项目由瑞典研究委员会(Formas, Sweden)资助,批准号:2016 - 00463。部分样本是通过资助协议n633184(项目名称sapir)下的欧盟地平线2020研究、技术开发和示范计划,或瑞典研究委员会(瑞典Formas)、生物技术和生物科学研究委员会(英国BBSRC)和法国国家研究机构(法国ANR)收集的。通过欧洲研究区网(ERA-NET)的牲畜新发传染病和主要传染病(EMIDA)项目,FP # 87。gydF4y2Ba
作者信息gydF4y2Ba
作者及单位gydF4y2Ba
贡献gydF4y2Ba
JFV, SH, SZ和DTH在PCDJ和KN的贡献下构思和设计了这项研究。JFV、SH、KN、GM和GT进行了传动试验。JFV、SH、KN和SZ提供了来自现场暴发样本的数据。SZ和KN生成基因组数据并进行基因组分析。PCDJ分析了RJO, DTH, SH, SZ和JFV的贡献数据。PCDJ, DTH, JFV, SH, SZ和RJO解释了结果。PCDJ, JFV, SH和SZ撰写了手稿的初稿。所有作者都对稿件进行了批判性的审查和编辑。所有作者都阅读并批准了最终的手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
伦理批准并同意参与gydF4y2Ba
在这项工作中进行的实地研究和实验研究得到了瑞典乌普萨拉大学伦理委员会的批准。5.8.18-16188/2017)。在先前发表的研究中收集的样本由瓦尔德卢瓦尔伦理委员会(apafis# 9903-2017051513402904v6和apafis# 27192015110908345685v3)批准,或由瑞典乌普萨拉地区法院伦理委员会(Ref. no. 5)批准。C330/11、C68/10和C247/2)。gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者宣称他们没有竞争利益。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba
伟德体育在线b施普林格《自然》杂志对已出版的地图和机构的管辖权要求保持中立。gydF4y2Ba
补充信息gydF4y2Ba
13567 _2022_1127_moesm1_esm.docgydF4y2Ba
附加文件1。gydF4y2Ba表和图给出了实验G的设计和结果的额外细节,NCBI加入号,以及覆盖、可靠性、错误阈值选择、瓶颈大小估计和概率传输路由推断的分析的额外细节。gydF4y2Ba
13567 _2022_1127_moesm2_esm.pdfgydF4y2Ba
附加文件2。gydF4y2Ba绘制了来自实验A、B、C、D、F和G的47个全病毒基因组序列的覆盖深度的基因组分布(gydF4y2BangydF4y2Ba= 40),从现场爆发样本(gydF4y2BangydF4y2Ba= 7)。gydF4y2Ba
13567 _2022_1127_moesm3_esm.pdfgydF4y2Ba
附加文件3。gydF4y2Ba从观察到共识水平变异的三个实验(C、D和F)和疫情样本(O)的共识序列中对可变位点进行比对。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接获得版权所有者的许可。如欲查阅本许可证副本,请浏览gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba。创作共用公共领域免责声明(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本文中提供的数据,除非在数据的信用额度中另有说明。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
约翰逊,p.c.d., Hägglund, S., Näslund, K.。gydF4y2Baet al。gydF4y2Ba评估全基因组测序在牛呼吸道合胞病毒实验感染和自然暴发中追踪传播途径的潜力。gydF4y2Ba兽医ResgydF4y2Ba53gydF4y2Ba, 107(2022)。https://doi.org/10.1186/s13567-022-01127-9gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s13567-022-01127-9gydF4y2Ba
关键字gydF4y2Ba
- BRSVgydF4y2Ba
- 全基因组测序gydF4y2Ba
- 分子流行病学gydF4y2Ba
- 牛gydF4y2Ba
- 传播树gydF4y2Ba