摘要
非洲猪瘟病毒(ASFV)是一种感染家猪的大型DNA病毒,发病率和死亡率高。重复序列是基因组中重复两次以上的DNA序列元件,在ASFV基因组中起着重要作用。然而,大多数重复序列尚未被系统地识别和表征。本研究在非洲猪瘟病毒基因组中鉴定出微卫星、小卫星和短穿插核元件(short interspersed nuclear element, SINEs)三种重复序列,并对其分布、结构、功能和进化历史进行了研究。大多数重复序列位于非编码区和基因组的5 '端。非编码重复序列倾向于形成增强子,而编码重复序列的α -螺旋和β -片比较低,环结构和表面氨基酸比非重复序列高。此外,重复序列倾向于编码穿透性和抗菌肽。进一步分析重复序列的进化,发现泛重复序列呈开放状态,显示了重复序列的多样性。最后,CpG岛与重复序列的发生呈负相关,表明它们可能影响重复序列的产生。总的来说,这项研究强调了重复序列在asfv中的重要性,这些结果有助于理解病毒的功能和进化。
介绍
非洲猪瘟(ASF)是一种由非洲猪瘟病毒(ASFV)引起的猪的出血性毁灭性传染病,死亡率高达100% [1,2]。非洲猪瘟对许多国家的养猪业造成重大影响[3.]。非洲、欧洲和亚洲有50多个国家爆发过非洲猪瘟[3.,4]。非洲猪瘟于2018年8月首次在中国出现,并迅速蔓延到全国几乎所有省份[5]。病毒基因组是双链DNA,大小从170到194 kb不等。6]。ASFV基因组编码超过150个开放阅读框(orf),它们形成成熟的ASFV病毒粒子,具有大的、包膜的和复杂的结构,由于对该病毒的不完全了解,使得开发有效的疫苗和药物变得困难[6,7,8]。
重复序列是在基因组中反复出现的DNA序列,对病毒感染和变异至关重要[9,10,11]。根据目前的发现,重复序列在ASFV基因组中广泛分布,并可能促进同源重组的发生[12]。一些重复序列已被发现在宿主细胞中ASFV感染中起重要作用[11,13]。例如,重复肽([KPCPPP]3.)作为细胞穿透肽,潜在地帮助病毒进入细胞[11]。此外,基因内或基因间区域重复序列数量的变化有助于非洲猪瘟的遗传多样性[12,14,15]。虽然已经确定了病毒中少数重复序列的功能,但绝大多数重复序列的结构、功能和进化尚未得到研究[13,15]。需要对非洲猪瘟重复序列进行系统的研究来弥补这一空白,这将有助于病毒的预防和控制。
串联重复序列和正弦序列是最常见的重复序列类型[16,17]。它们不仅对进化、遗传和变异有影响,而且在基因表达、转录和调控中也起着重要作用[18,19]。例如,人类染色体末端的串联重复序列在细胞生长、增殖和凋亡过程中发挥着重要的调节作用,如维持染色体的完整性和稳定性,防止染色体被核酸酶降解,避免染色体末端融合和重组,提供端粒酶底物,调节细胞生命周期等。20.,21]。sine存在于许多脊椎动物和无脊椎动物物种中,具有谱系特异性,在基因表达的调控和RNA基因的产生中起着特别重要的作用[22,23]。例如,含有CTCF结合位点的SINEs可作为影响染色质结构和转录的边界元件[22,24]。本研究对ASFV重复序列进行了系统的鉴定和分类,并对其结构、功能和进化特征进行了进一步的研究。研究结果表明,重复序列对ASFV的表达和进化至关重要。这项工作将有助于更好地了解非洲猪瘟病毒的功能和进化,并为今后研究其他病毒的重复序列铺平道路。
材料与方法
非洲猪瘟病毒基因组重复序列的鉴定
从NCBI GenBank中共获得86条ASFV基因组序列[25]数据库于2021年8月15日生效。从ASFV基因组中去除间隙和非标准碱基。副本(版本1)[26]及信托基金(第一版)[27]用默认参数鉴定ASFV基因组中的重复序列。
非洲猪瘟病毒基因组的注释
使用GeneMarkS(4.28版)预测ASFV基因组编码的基因及其相关蛋白[28],使用默认参数。为了推断ASFV蛋白的功能,将它们与从NCBI RefSeq和ASFVdb下载的所有ASFV蛋白进行比对[29]数据库使用NCBI BLAST程序(版本2.9.1)[30.]。
非洲猪瘟病毒基因组重复序列的结构功能分析
如表所示1,重复序列的结构和功能使用各种公开可用的工具进行了研究。
系统发育树的推断和基因型的确定
先前发表的研究基于p72基因的c端确定了24种基因型[37]。为了确定所有分析的ASFV毒株的基因型,我们使用了MAFFT (version 7) [38]对p72基因c端415bp区域的基因组序列进行比对;然后,我们使用MEGAX中的最大似然算法推断系统发育[39用100个bootstrap重复。各非洲猪瘟病毒的基因型是根据以往的研究确定的[37,40]。
非洲猪瘟重复序列的全基因组分析
将相同的重复序列作为一个簇来聚类。采用PanGP和DG采样算法绘制ASFV泛重复序列和核心重复序列的特征曲线[41]。花图描绘了ASFV的核心重复序列簇和必要重复序列簇的数量,包括独特的重复序列plotrix ()function in R (version 4.0.3) [42]。
CpG岛的鉴定
使用Cpgplot (version 1)检测ASFV基因组中的CpG岛[43]。
统计分析
所有统计分析均在R(4.0.3版)中进行[42]和Python(3.6版本)[44]。采用Wilcoxon秩和检验wilcox.test ()函数计算相关系数cor.test ()R中的函数。
结果
非洲猪瘟病毒基因组重复序列的鉴定、分类和分布
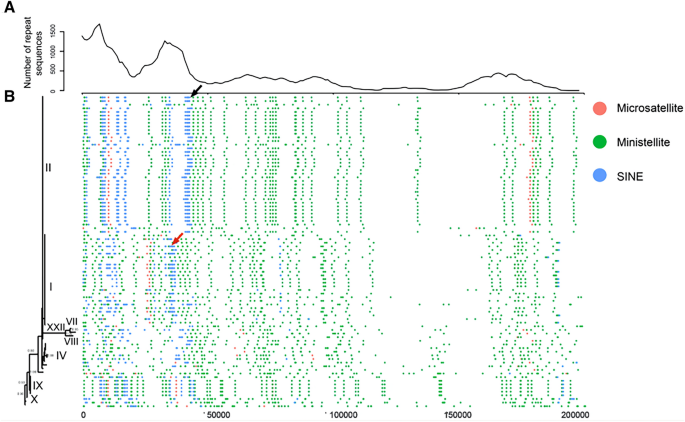
从NCBI GenBank数据库中共获得86个asfv基因组序列;这些在附加文件中列出1.发现asfv含有丰富的重复序列(图2)1)。这些重复序列分为三种类型:(1)微卫星(microsatellite),定义为重复单元小于10 bp的小串联重复序列;(2)小卫星(miniatellites),定义为重复单元10-300 bp的小串联重复序列;(3)短穿插核元件(short interspersed nuclear element, SINEs),定义为重复单元100-500 bp的不连续分布。重复序列在非洲猪瘟病毒基因组中的位置分析显示,大部分重复序列位于基因组的5 '或3 '端(图2)1A).具体来说,90%以上的微卫星和sine位于ASFV基因组的5 '端,而小卫星则不规则地分布在整个基因组中。
绝大多数重复序列被发现是基因型特异性的。检索到的基因组包含9个基因型。大多数非洲猪瘟病毒基因组,包括最近在欧洲和中国发现的基因组,都是基因型I或基因型II。基因型1包括26个ASFV基因组,其中超过30%的重复序列具有基因型特异性(见图中红色箭头标记的例子)1B).基因型II包含38个ASFV基因组,其中超过一半的重复序列是基因型特异性的(见图中黑色箭头标记的例子)1B)。
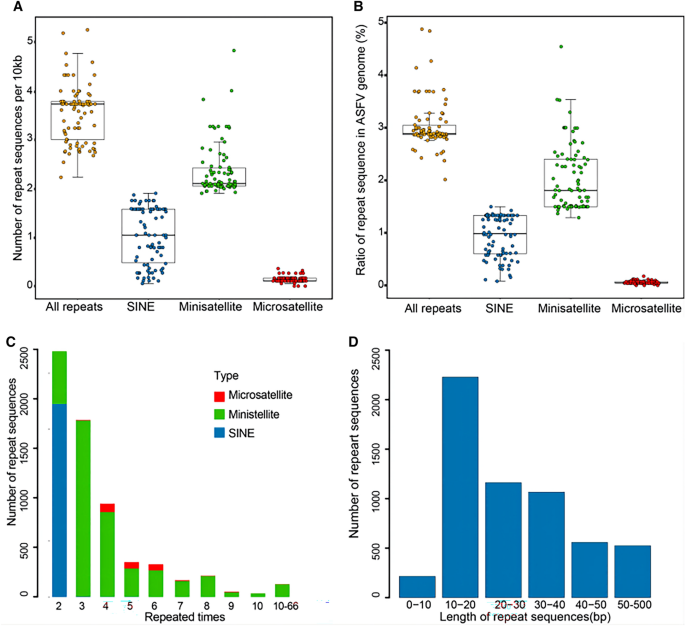
对86个非洲猪瘟病毒基因组的重复序列数量和重复序列比例进行了分析,发现每10kb基因组序列中有2.2 ~ 5.3个重复序列,中位数为3.7个(图2)2A).这些重复序列中有60%以上是小卫星,每10 kb基因组序列中有1.9 ~ 4.9个重复序列。这些小卫星中位数覆盖了1.8%的非洲猪瘟病毒基因组(图2)2B).大约30%的重复序列由sin组成,每10 kb基因组序列的数量在0.05到1.9之间,中位数为0.9%的ASFV基因组。其余的重复序列属于微卫星序列,每10 kb基因组序列的重复序列少于0.5个。
由于重复单位是重复序列的基本单位,因此我们分析了非洲猪瘟病毒基因组中重复单位的数量(图2)2C).重复单元的数量从2到66不等,中位数为3。在约80%的重复序列中,重复单元数小于4次;只有2%的重复序列重复单元数超过10次。对于几乎所有的正弦卫星,重复单元只重复两次,对于超过40%的微型卫星,重复单元只重复三次。
然后分析重复序列中重复单元的长度(图2)2D).重复单位长度从1到281 bp不等,中位数为24 bp。其中,超过一半的重复单位长度不超过30 bp,约9%的重复单位长度超过50 bp。有趣的是,最常见的重复单位长度为10-20 bp。
重复序列在非编码区发生率高
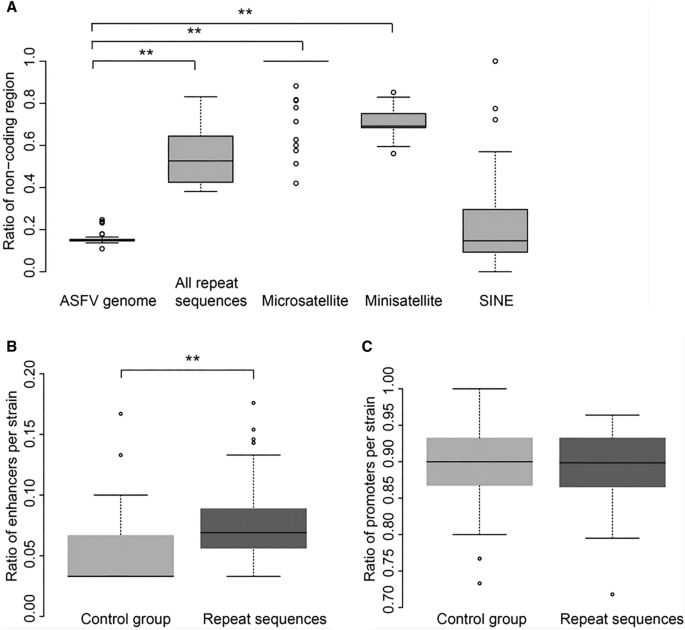
我们评估了非编码区和编码区重复序列的比例,发现大约50%的重复序列位于非编码区(图2)3.A).具体来说,大约70%的迷你卫星和95%的微型卫星位于非编码区,而不到20%的正弦信号位于非编码区。编码区的SINEs分析显示,70%的SINEs位于多基因家族(MGF)蛋白中,其余的SINEs位于其他蛋白和未知蛋白中(附加文件)2)。这表明,SINEs可能导致氨基酸插入或缺失,从而影响ASFV蛋白的功能。
非编码区重复序列的结构和功能表征
由于大多数重复序列位于非编码区,因此我们研究了重复序列在转录调控中的作用(图2)3.A).在位于非编码区的重复序列中,鉴定出两种功能元件:增强子和启动子。为了进行比较,从每个ASFV基因组的非编码区随机选择与重复序列大小相同且数量相同的非重复序列(对照组)。重复序列中增强子的比例显著高于非重复序列(0.08比0.04);p值< 0.01)3.令人惊讶的是,两个重复序列中启动子与对照组的中位数比值均大于0.9(图2)3.C),两者之间无显著差异。
编码区重复序列的序列、结构和功能表征
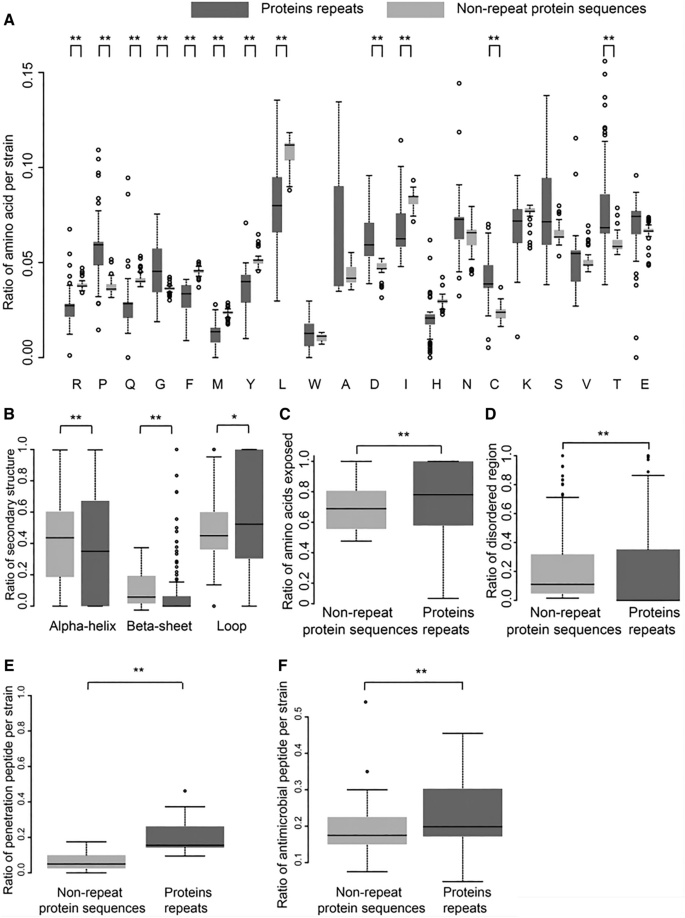
然后,对编码区的重复序列进行分析。对于每个重复序列,使用该重复序列编码的蛋白序列(为清晰起见定义为重复蛋白序列)和同一蛋白中的其他蛋白序列(定义为非重复蛋白序列)进行分析。计算重复蛋白序列与非重复蛋白序列的氨基酸比值。有趣的是,在两种类型的蛋白质序列中观察到氨基酸比例的巨大差异(图2)4A)。在重复蛋白质序列中,苯丙氨酸(F)、蛋氨酸(M)、亮氨酸(L)和异亮氨酸(I)等几种疏水氨基酸的比例显著低于非重复蛋白质序列,而在重复蛋白质序列中,半胱氨酸(C)和苏氨酸(T)等三种亲水性氨基酸的比例显著高于非重复蛋白质序列(p值< 0.01)。
我们进一步研究了重复蛋白序列的结构特征。计算了三种二级结构单元(α -螺旋、β -片和环)的比值。重复序列α -螺旋比值中位数为0.35,显著低于非重复序列(0.35 vs. 0.43);p值< 0.01)4B),重复蛋白序列中β -sheet的中位数比值仅为0.0,显著低于非重复蛋白序列(0.0 vs. 0.06;p值< 0.01)。有趣的是,重复蛋白序列中环的中位数比率为0.52,显著高于非重复蛋白序列(0.52 vs. 0.45)。p值< 0.05)。然后,计算暴露的氨基酸比例,观察到重复蛋白序列的氨基酸暴露比例高于非重复蛋白序列(图2)4C)。此外,我们计算了无序区(定义为不可能形成确定的蛋白质三维结构的区域)的氨基酸比例,观察到重复的蛋白质序列在无序区的氨基酸比例低于非重复的蛋白质序列(图2)4D)。
进一步分析了重复蛋白序列的潜在功能。先前的研究表明,重复蛋白序列在单纯疱疹病毒和人呼吸道合胞病毒中可作为穿透肽或抗菌肽[11,45]。因此,穿透肽和抗菌肽在重复蛋白序列中被鉴定出来。中位数分别为18%和21%的重复蛋白序列具有穿透肽和抗菌肽的功能(图2)4E和F),显著高于非重复蛋白序列。总的来说,超过30%的重复蛋白序列具有抗菌肽或穿透肽的功能(附加文件)3.),提示该重复蛋白序列可能在ASFV感染中起重要作用。
非洲猪瘟基因组重复序列的泛基因组分析
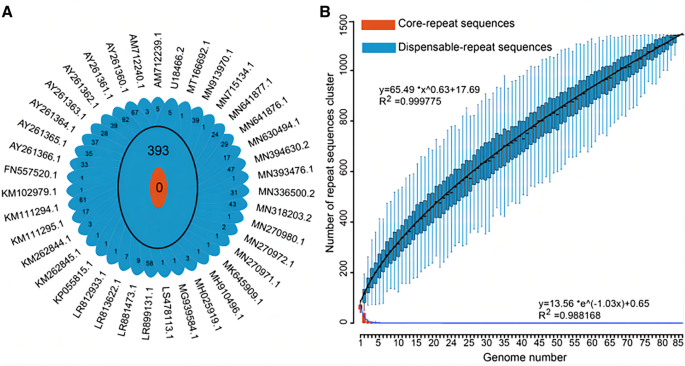
对ASFV基因组重复序列进行泛基因组分析。所有在ASFV基因组中鉴定到的重复序列均被定义为泛重复序列。基于100%的序列相似性,将它们分类为1141个聚类。得到了核心重复序列和出现在一个或多个ASFV基因组中的可有可无重复序列。在可有可无的重复序列中,那些只出现在一个基因组中的重复序列被称为唯一重复序列。pan-repeat序列分析显示,尽管存在1141个必要重复序列,包括748个独特重复序列,但未发现核心重复序列(图2)5一个)。
随着基因组数量的增加,用幂律回归模型(y)拟合可缺重复簇数锅=一个潘*xBpan+ C锅),核心重复簇数采用指数模型(y核心=一个核心*eBcore * x+ C核心),根据赵的研究[41]。两种模型的数学函数在图上进行了描述。B锅的幂律回归模型中,均大于0.5,说明asfv泛重复序列处于开放状态(见图中黑色曲线)5B).这一趋势反映了asfv具有灵活的重复序列,并且泛重复序列的大小可能随着每个基因组的增加而扩大,这有助于新的重复序列的产生。随着分析基因组数量的增加,核心重复序列簇的数量明显减少,趋于零(见图中蓝色曲线)5B).这一趋势反映了非洲猪瘟基因组重复序列的多样性。
考虑到重复序列的基因型特异性,我们进一步分析了基因型I和II的泛重复序列。共检测到核心重复序列5个、32个,非必需重复序列251个、73个,其中独特重复序列138个、独特重复序列22个,构成ASFV基因型I、II泛重复序列(附加文件)4)。B锅在幂律回归模型中基因型I和基因型II均大于0.5(附加文件)4),表明基因型内的泛重复序列也处于开放状态。随着分析基因组数量的增加,核心重复序列聚类曲线呈现收敛趋势。这一趋势反映了基因型内的重复序列在进化过程中是保守的。
非洲猪瘟病毒基因组中CpG岛与重复序列的关系
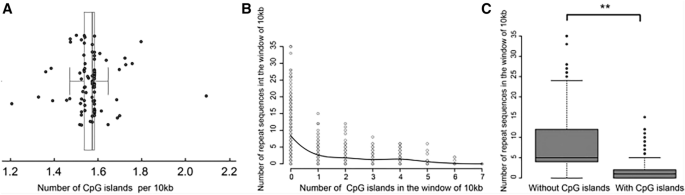
先前的研究表明,CpG岛有助于稳定基因组,并可能阻止重复序列的形成。因此,我们研究了CpG岛对重复序列的影响。在ASFV基因组中鉴定到的CpG岛数量为每10kb基因组序列1.2 ~ 2.1个(图2)6A),中位数为1.57。有趣的是,观察到ASFV基因组每10 kb窗口鉴定的CpG岛数量与同一区域鉴定的重复序列数量呈负相关(Pearson相关系数= - 0.48)6具体而言,具有CpG岛的窗口只有一个重复序列的中位数,而没有CpG岛的窗口有五个重复序列的中位数(图2)6C)。
讨论
研究了非洲猪瘟病毒基因组重复序列的分类、分布、结构、功能和进化。本研究首次揭示了ASFV重复序列的四个特征。首先,在ASFV基因组的5 '端富集重复序列。这与Campbell等人在鸡痘病毒基因组末端区域发现大量简单重复序列(SSRs)的研究一致[46]。重复序列往往位于非编码区,因为在编码区插入和删除重复序列可能会破坏ORF,从而可能对病毒造成严重损伤甚至死亡[47,48]。因此,在登革热病毒(DEN)和黄热病等病毒基因组的编码区只观察到少数重复序列[49,50]。然而,非编码区的重复序列在病毒生命周期中也起着重要的作用[51,52,53]。一些重复序列可以影响病毒的复制和转录[51,53]。例如,单纯疱疹病毒的重复序列包含一个可调节病毒基因转录的基因启动子[51]。因此,我们的研究结果提供了证据,证明重复序列是ASFV基因组的结构要素,支撑其独特的功能。
其次,与非重复序列相比,重复序列具有更高的环状结构比例和更低的无序区域比例。与以往研究结果相似,重复蛋白序列具有较大的环状结构比例,可以提供更多的蛋白结合位点[54,55]。重复序列由于其灵活的性质,可能不太可能形成无序区域。由于重复序列含有低比例的极性氨基酸(如酪氨酸(Y)和精氨酸(R)),因此很难形成聚电解质,而聚电解质是无序区的重要组成部分[56]。基于这些结果,我们得出结论,重复序列在非洲猪瘟特异性结构肽的构象中很重要。
第三,我们的研究结果表明,ASFV重复序列倾向于编码穿透肽和抗菌肽。少量的重复序列已被鉴定为编码穿透肽和抗菌肽[45,57]。编码穿透肽的重复序列在ASFV通过受体介导的内吞作用进入宿主细胞中发挥重要作用[j]。11]。此外,编码抗菌肽的重复序列可以帮助开发新的抗菌肽药物[58,59],因为源自病毒蛋白的抗菌肽是创新给药系统发展的关键因素[45,57]。这些发现表明ASFV重复序列在功能肽中具有特定的作用。
结果表明,所有ASFV的泛重复序列呈开放状态,核心重复序列不存在。与细菌类似,开放泛重复序列表明非洲猪瘟病毒的遗传物质可能不断发生突变[60]。在ASFV中未观察到核心重复序列,说明ASFV重复序列具有多样性。此外,有报道称重复序列与同源重组有关[12]。因此,重复序列的多样性可以进一步促进ASFV的多样性。总之,我们的发现强调了重复序列在非洲猪瘟基因组进化和变异中的作用,强调了进一步研究其他病毒重复序列的重要性。
这项研究有两个需要承认的局限性。首先,在ASFV基因组中观察到的重复序列的位置、大小和数量可能受到重复序列识别算法的影响。为了全面检测重复序列,采用TRF和repter两种互补方法对ASFV基因组重复序列进行鉴定。其次,预测的重复序列的结构和功能特征还没有经过实验检验。需要更多的努力来验证这些预测,并澄清非洲猪瘟基因组重复序列的作用。
总之,本研究首次阐明了asfv重复序列的分布、结构、功能和进化特性,从而促进了我们对该病毒的了解,并促进了有效抗asfv药物的开发。本研究建立的方法框架可用于指导今后对其他病毒重复序列的研究。
参考文献
Parker J, Plowright W, Pierce M(1969)非洲猪瘟的流行病学。rev 85:668-674
冯志强,王志强,等(2020)猪感染非洲猪瘟的比较病理学和发病机制。前沿兽医科学7:282
Gaudreault NN, Madden DW, Wilson WC, Trujillo JD, right JA(2020)非洲猪瘟病毒:一种新兴的DNA虫媒病毒。《前沿兽医科学》7:15 5
孙娥,张忠,王忠,何翔,张翔,王丽,王伟,黄丽,席芳,黄福华,蔡国光,霍华,孙洁,田忠,夏伟,于鑫,李飞,刘锐,关勇,赵东,布忠(2021)2020年中国家猪自然发生的低毒力非洲猪瘟病毒的发生和流行情况。中国科学:生命科学64:752-765
葛松,李军,范鑫,刘峰,李丽,王强,任伟,鲍健,刘超,王宏,刘毅,张勇,徐涛,吴翔,王铮(2018)非洲猪瘟病毒分子特征分析,中国,2018。新兴感染病24:21 1 - 2133
Dixon LK, Chapman DA, Netherton CL, Upton C(2013)非洲猪瘟病毒复制和基因组学。病毒Res 173:3-14
Cackett G, Matelska D, Sýkora M, Portugal R, Malecki M, Bähler J, Dixon L, Werner F(2020)非洲猪瘟病毒转录组。[J]中国生物医学工程学报(英文版)
李建军,李建军,李建军,等(2018)非洲猪瘟病毒的蛋白质组学分析。[J]中国生物医学工程学报
Versalovic J, Koeuth T, Lupski R(1991)真细菌中重复DNA序列的分布及其在细菌基因组转录中的应用。核酸学报,19:6823-6831
姚强,Compans RW(1996)人副流感病毒融合蛋白七肽重复序列对应的肽是病毒感染的有效抑制剂。病毒学223:103 - 112
杨松,张翔,曹勇,李松,邵军,孙松,郭华,尹生(2021)非洲猪瘟病毒CD2v蛋白新细胞穿透肽的鉴定。药物传递:28:957-962
朱忠,肖春春,范宇,蔡忠,陆超,张刚,姜涛,谭宇,彭宇(2019)非洲猪瘟病毒同源重组形成遗传多样性。兽医微生物学报236:108380
张建军,张建军,张建军,等(2018)高致病性非洲猪瘟病毒(ASFV)流行内基因组变异分析。病毒J 15:190
Goller KV, Malogolovkin AS, Katorkin S, Kolbasov D, Titov I, Höper D, Beer M, Keil GM, Portugal R, Blome S(2015)非洲猪瘟病毒串联重复插入,俄罗斯,2012。急诊感染病21:731
Kim shwe, Lee SI, Jeong HG, Yoo J, Jeong H, Choi Y, Son K, Jheong WH(2021)韩国非洲猪瘟病毒不同串联重复序列数变异的快速出现。跨界急诊68:1726-1730
尚文华,Hori T, Toyoda A, Kato J, Popendorf K, Sakakibara Y, Fujiyama A, Fukagawa T(2010)鸡具有延长串联重复序列和短非串联重复序列的着丝粒。Genome Res 20:1219-1228
马文华(2011)重复序列、基因组不稳定性与巴雷特食管腺癌。移动通信技术,1:208-212
Boeva V(2016)真核细胞中转录因子结合和转录调控的基因组序列基序分析。前日内瓦7:24
Lynch VJ, Leclerc RD, May G, Wagner GP(2011)转座子介导的基因调控网络重组对哺乳动物妊娠进化的影响。生物学报43:1154-1159
Gymrek M, Willems T, Guilmatre A, Zeng H, Markus B, Georgiev S, Daly MJ, Price AL, Pritchard JK, Sharp AJ, Erlich Y(2016)短串联重复序列对人类基因表达变异的丰富贡献。Nat Genet 48:22-29
Gymrek M, Willems T, Reich D, Erlich Y(2017)利用突变约束解释人类短串联重复序列变异。[au:] Nat Genet 49:14 . 95 - 1501
Lunyak VV, Prefontaine GG, Núñez E, Cramer T, Ju BG, Ohgi KA, Hutt K, Roy R, García-Díaz A,朱霞,Yung Y, Montoliu L, Glass CK, Rosenfeld MG(2007)器官发生中sinb2重复序列的发育调控激活。科学317:248 - 251
卢建勇,邵伟,常丽,尹勇,李涛,张宏,洪毅,Percharde M,郭玲,吴忠,刘玲,刘伟,严鹏,Ramalho-Santos M,孙勇,沈翔(2020)基因组重复序列对不同功能基因的协同调控。Cell Rep 30:3296-3311.e5
Schmidt D, Schwalie PC, Wilson MD, Ballester B, gonalves Â, Kutter C, Brown GD, Marshall A, Flicek P, Odom DT(2012)反转录转座子扩增重组基因组组织和CTCF结合的波浪。细胞148:335 - 348
Sayers EW, Cavanaugh M, Clark K, Ostell J, Pruitt KD, Karsch-Mizrachi I(2019)基因银行。核酸学报47:D94-D99
李建军,李建军,李建军,等(2001)基因组重复序列分析的研究进展。核酸学报29:4633-4642
Benson G(1999)串联重复查找器:分析DNA序列的程序。核酸:573 - 580
李建军,李建军,李建军,等(2001)微生物基因组基因序列的自训练预测方法。在调控区域寻找序列基序的意义。核酸学报29:2607-2618
朱忠,孟刚(2020)非洲猪瘟病毒基因组和蛋白质组学分析的整合资源。https://asfvdb.popgenetics.net/.2021年3月20日
Altschul SF, Madden TL, Schäffer AA,张军,张志,Miller W, Lipman DJ (1997) gap BLAST和PSI-BLAST:新一代蛋白质数据库搜索程序。核酸学报25:3389-3402
刘波,李凯,黄德生,周克成(2018).基于集成学习方法的增强器- el识别及其强度。生物信息学34:3835 - 3842
刘波,杨峰,黄德生,周克成(2018).基于多窗口的启动子- 2l双层预测因子及其类型识别。生物信息学34:33-40
张晓东,张晓东,张晓东,等(2012)中国生物医学工程学报。生物信息学28:503 - 509
Klausen MS, Jespersen MC, Nielsen H, Jensen KK, Jurtz VI, Soenderby CK, Sommer MOA, Winther O, Nielsen M, Petersen B, Marcatili P (2019) NetSurfP-2.0:基于集成深度学习的蛋白质结构特征预测。蛋白质87:520 - 527
王晓明,王晓明,王晓明,等(2016)抗微生物肽的基因序列、结构和特征分析。核酸学报44:D1094-D1097
Manavalan B, Subramaniyam S, Shin TH, Kim MO, Lee G(2018)基于机器学习的细胞穿透肽预测及其摄取效率提高的准确性。[J] .中国生物医学工程学报(英文版),17 (2):444 - 444
Bastos AD, Penrith ML, crucere C, Edrich J, Hutchings G, Roger F, Couacy-Hymann E, Thomson R(2003)非洲猪瘟病毒p72部分基因的分型。Arch Virol 148:693-706
Katoh K, Standley DM (2013) MAFFT多序列比对软件版本7:性能和可用性的改进。摩尔生物进化(30:772-780)
李建军,李建军,李建军,李建军(2018)基于遗传算法的分子进化遗传分析。生物进化35:1547-1549
Muangkram Y, Sukmak M, Wajjwalku W(2015)基于p72基因序列的非洲猪瘟病毒系统地理分析。热学杂志14:45 - 66
赵燕,贾翔,杨健,凌云,张震,于健,吴健,肖健(2014)PanGP:一种快速分析细菌泛基因组谱的工具。生物信息学30:1297 - 1299
R核心团队。R(2019)统计计算的语言和环境。维也纳,奥地利:R基金会统计计算。https://www.R-project.org/.2020年7月12日访问
Rice P, Longden I, Bleasby A (2000) EMBOSS:欧洲分子生物学开放软件套件。趋势,基因,16:276-277
Sanner, M. F (1999) Python:用于软件集成和开发的编程语言。[J]摩尔图模型17:57-61。https://www.python.org/.2020年6月10日
陈建军,陈建军,陈建军,陈建军,等。(2015)基于病毒蛋白的抗微生物和细胞穿透性药物递送肽。生物信息学31:2252 - 2256
Campbell JI, Binns MM, Tomley FM, Boursnell ME(1989)鸡痘病毒基因组末端区的串联重复序列。[J]中华微生物学杂志,70:145-154
Singh M, Kishore A, Maity D, Sunanda P, Krishnarjuna B, Vappala S, Raghothama S, Kenyon LC, Pal D, Sarma JD(2019)小鼠肝炎病毒刺突糖蛋白融合肽脯氨酸插入缺失对神经病理学的影响。[J] .中国生物医学工程学报(英文版)
王铮,潘琪,Gendron P,朱伟,郭峰,岑森,Wainberg MA,梁超(2016)CRISPR/ cas9衍生突变抑制HIV-1复制并加速病毒逃逸。电话代表15:481-489
Shurtleff AC, Beasley DW,陈建军,倪浩,Suderman MT,王宏,徐瑞,王娥,Weaver SC, Watts DM, Russell KL, Barrett AD(2001)登革热病毒3 '非编码区遗传变异。病毒学281:75 - 87
王娥,王伟,王志强,王志强,王志强(1996)非洲地区黄热病病毒3′非编码区遗传变异的研究。病毒学225:274 - 281
Chou J, Roizman B(1986)单纯疱疹病毒基因组末端a序列包含位于L组分重复序列中的一个基因启动子。[J]中国医学杂志(英文版)
Madhry D, Pandey KK, Kaur J, Rawat Y, Sapra L, Kumar R, Srivastava RK, Bhattacharyya S, Verma B(2021)非编码rna在登革热病毒与宿主相互作用中的作用。前沿生物科学(学校版)13:44-55
Vychodil T, Conradie AM, Trimpert J, Aswad A, Bertzbach LD, Kaufer BB(2021)马立克病病毒需要两个反向重复区域的拷贝才能有效地在体内复制和发病。[J]中国生物医学工程学报
Weng Y, Weiss CD(1998)人类免疫缺陷病毒1型跨膜蛋白gp41螺旋结构域残基的突变分析。[J]中国生物医学工程学报(英文版)
祁东,DeYoung BJ, Innes RW (2012) RPS5抗病蛋白卷曲和富含亮氨酸重复结构域的结构-功能分析。植物生理学158:1819-1832
Das RK, Ruff KM, Pappu RV(2015)内在无序蛋白形成和功能的相关序列编码信息。当前结构生物学32:102-112
Fox JL(2013)抗菌肽卷土重来:更好地理解抗菌肽的作用机制、修饰和合成正在重新点燃商业开发。生物科技学报31:37 79 - 383
黄燕,江燕,王辉,王军,申明明,卞勇,何宏,梁勇,杨文辉(2013)抑制“特洛伊木马”方法的挑战:实现有效而安全的细胞穿透肽基药物递送的智能策略。Adv Drug delivery Rev 65:1299-1315
Moretta A, Scieuzo C, Petrone AM, Salvia R, Manniello MD, Franco A, Lucchetti D, Vassallo A, Vogel H, Sgambato A, Falabella B(2021)抗菌肽:生物医学和制药领域的新希望。前细胞感染微生物11:668632
王忠,贾磊,李军,刘宏,刘东(2020)非洲猪瘟病毒泛基因组分析。Virol Sin 35:662-665
致谢
国家自然科学基金项目(32170651)和湖南省自然科学基金项目(2020JJ3006)资助。
作者信息
作者及单位
贡献
ZZ, SG和YP进行了实验并起草了手稿。YP, ZZ, XW和LM批判性地修改了稿件和实验设计。ZZ、SG、RJ、ZC、YW、CL、PF、XW和YP协助实验。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
相互竞争的利益
作者宣称他们没有竞争利益。
额外的信息
出版商的注意
伟德体育在线施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。
补充信息
13567 _2022_1119_moesm1_esm.doc
附加文件1。研究中使用的病毒。
13567 _2022_1119_moesm2_esm.tif
附加文件2。ASFV短穿插重复序列(sin)蛋白的功能分类。
13567 _2022_1119_moesm3_esm.tif
附加文件3。穿透肽和抗菌肽在重复蛋白序列中的交叉。
附加文件4。ASFV基因型I和II的泛重复序列分析。
(A和C) ASFV基因型I和II的核心重复序列和可省去重复序列簇的花图。该图分别描绘了ASFV基因型I和基因型II的核心重复序列簇数(橙色)和必要重复序列簇数(蓝色)。在ASFV基因组的加入号旁边显示了每个菌株中唯一重复序列的数量。(B和D) ASFV基因型I和II的基因组数量与可省去重复序列和核心重复序列谱的关系。其中,黑色曲线和蓝色曲线分别为可忽略重复簇和核心重复簇的平均数目相对于基因组数目的最小二乘拟合。两种模型的数学函数和r平方值在图上被描绘出来。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接获得版权所有者的许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非在数据的信用额度中另有说明。
关于本文
引用本文
朱忠,葛,S,蔡,Z。et al。非洲猪瘟病毒基因组重复序列的系统鉴定和表征。兽医Res53, 101(2022)。https://doi.org/10.1186/s13567-022-01119-9
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s13567-022-01119-9
关键字
- ASFV
- 重复序列
- 进化
- 遗传多样性