摘要
背景
肝星状细胞(HSC)向肌成纤维细胞的转化是肝成纤维过程中的关键事件。然而,hsc向肌成纤维细胞转变的分化轨迹以及这一过程中的开关基因仍不清楚。
方法
我们利用单细胞测序数据重建了体外造血干细胞转分化的单谱系伪时间轨迹,并分析了沿该轨迹的基因表达模式。geneswitch用于识别HSC激活过程中关键基因表达和功能事件的顺序。
结果
发现了一种新的细胞状态,属于这种状态的HSC可能是癌症相关成纤维细胞(CAFs)的重要来源。结合单细胞转录组学和geneswitch分析,我们鉴定了一些不同的开关基因,以及这些开关分别发生在新状态的HSC和经典培养激活的HSC上的顺序。基于顶级切换基因,我们建立了一个四基因组合,在预测非酒精性脂肪性肝病(NAFLD)或乙型肝炎(HBV)患者的晚期肝纤维化方面表现出高度的诊断准确性。
结论
我们的研究揭示了一种可能与CAFs相关的HSC激活过程中的新细胞状态,并确定了可能在HSC转分化中发挥关键作用的开关基因,并可作为慢性肝病患者晚期纤维化的预测标志物。
简介
肝纤维化是多种慢性肝病常见的伤口愈合反应。肝纤维化的发病机制主要表现为肝内细胞外基质(ECM)的过度积聚[1],主要来源于活化的肝星状细胞(HSC)。在不同类型的肝损伤中,静止的造血干细胞被激活并转化为增生性、成纤维性和收缩性肌成纤维细胞,其特点是表达高水平的Acta2(平滑肌肌动蛋白α 2)。HSC激活被认为是肝纤维化发生过程中的关键事件[2,3.].
单细胞转录组分析广泛应用于基础科学和临床研究,重塑了我们对许多复杂生物学过程的认识。与传统测序方法相比,单细胞RNA测序(scRNA-seq)可以前所未有地深入了解HSC的异质性,并在单细胞分辨率下识别与HSC激活高度相关的基因。最近,HSC分区被认为是肝纤维化反应的决定因素[4].通过单细胞转录组分析,肝纤维化期间的HSC和肌成纤维细胞亚群已被鉴定[5].然而,hsc向肌成纤维细胞转变的分化轨迹以及这一过程中的开关基因仍不清楚。
在本研究中,我们基于scRNA-seq数据在体外重建了HSC激活的单谱系伪时间轨迹。有趣的是,我们在HSC转分化过程中发现了一种新的细胞状态,这可能与癌症相关的成纤维细胞(CAFs)有关。此外,我们分析了有助于HSC激活的关键开关基因,然后,我们建立了一个基于顶级开关基因的四基因组合,并评估了其在预测人类晚期肝纤维化中的诊断准确性。
材料与方法
scRNA-seq数据分析
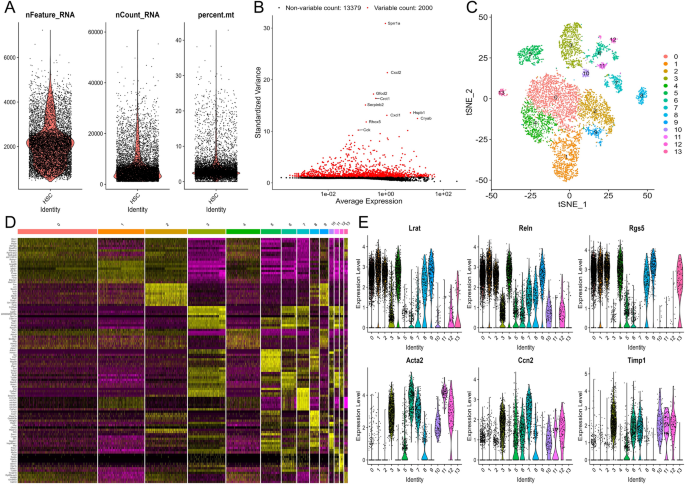
已发表的scRNA-seq数据检索自基因表达Omnibus (GEO)数据集GSE132662 [5].其中包括原代造血干细胞的单细胞转录组数据,这些原代造血干细胞是从健康小鼠肝脏中分离出来的,体外培养,并在第0、1、3、7和9天收获。scRNA-seq下游分析使用R包Seurat 3.0 [6].低质量细胞(< 200个基因/细胞,> 6000个基因/细胞,< 3个细胞/基因和> 20%的线粒体基因)从数据集中过滤掉。然后,用线性回归模型将剩余细胞中的基因表达归一化。鉴定出高变量基因,并选择主成分分析(PCA)以确定显著可用的维度[7].然后采用t分布随机邻居嵌入(t-SNE)算法进行降维和聚类分析[8].
为了在单细胞分辨率下解剖体外静止HSC的激活过程,我们使用了Monocle2 [9]进行HSC转分化的硅伪时间轨迹分析。简单地说,使用“DDRTree”约简方法,将单个细胞排列成具有分支点的轨迹。不同分支的细胞被认为处于不同的细胞分化状态。分支间表达水平存在差异的基因被定义为状态特异性基因。
GeneSwitches分析
为了发现HSC激活过程中关键基因表达和功能事件的顺序,我们应用geneswitch [10],该工具可以处理scRNA-seq数据以及伪时间轨迹,以识别在细胞状态之间起开关作用的基因(称为开关基因),以及重要的是这些开关发生的顺序。GeneSwitches首先将输入基因二进制化为“开启”或“关闭”基因表达状态,以方便识别开关事件。对于每个细胞中的每个基因,用二值化的基因表达作为因变量进行逻辑回归,每个细胞的伪时间值作为自变量。利用这一点,GeneSwitches计算整个伪时间内基因表达的概率,并使用麦克法登的伪估计拟合质量R2[11].激活开关基因与假时间呈正相关(R2> 0)被定义为上调基因,沉默开关基因与伪时间负相关(R2< 0)定义为下调基因。伪时间相关性越高,说明开关基因与轨迹过程的关系越密切。
此外,GeneSwitches还包括由分子特征数据库(MSigDB)提供的通路[12], C2京都基因与基因组百科全书[13]和C5基因本体论基因集集合,可用于排序通路或基因集(如功能本体论)。geneswitch还可以从两个相关的伪时间轨迹比较开关基因的顺序。两条轨迹之间的共同开关基因可以被绘制出来。此外,geneswitch可以识别和绘制每个轨迹特定的不同开关基因[10].
人和老鼠表达数组
分别从GEO数据库GSE49541检索了两项非酒精性脂肪性肝病(NAFLD)患者研究和一项乙型肝炎(HBV)患者肝纤维化分期研究的表达数据[14], gse89632 [15],及GSE84044 [16].
批量RNA测序数据来自数据集GSE154170 [17],在4种类型的小鼠样本中进行了实验:静态HSC分离株、胆管结扎(BDL)小鼠胆道纤维化模型的HSC分离株、3,5-二氧基羰基-1,4-二氢collidine (DDC)饮食处理的小鼠胆道纤维化模型的HSC分离株以及YAP/AKT或KRAS/sg-p19诱导的肝内胆管癌(CCA)中HSC来源的CAF分离株。
细胞培养和免疫荧光
人肝星状LX2细胞在Dulbecco 's改良Eagle 's培养基(DMEM) (Invitrogen, USA)中培养,并添加10%胎牛血清(Gibco, USA)。重组人TGF-β1从美国R&D Systems公司获得。抗体TAGLN购自Cell Signaling Technology (USA)。整合素αv (ITGAV)抗体购自BD生物科学公司(美国)。荧光二抗与Alexa Fluor 594(红色)或Alexa Fluor 488(绿色)(Invitrogen,美国)偶联。所有样品均使用荧光显微镜(Nikon,日本)进行检查。
统计分析
组间比较采用R版4.1.0 Kruskal-Wallis检验。Cytoscape中的ClueGO(版本2.5.8)和CluePedia(版本1.5.8)插件(https://cytoscape.org/),对鉴定的基因进行功能富集分析。受试者工作特征曲线(AUROC)下面积采用pROC [18].人类配对样本的比较在R中进行,并使用ggplot2进行可视化[19].p值< 0.05为有统计学意义。预测模型的开发和验证在附加文件中有详细说明1.
结果
健康小鼠造血干细胞单细胞分析
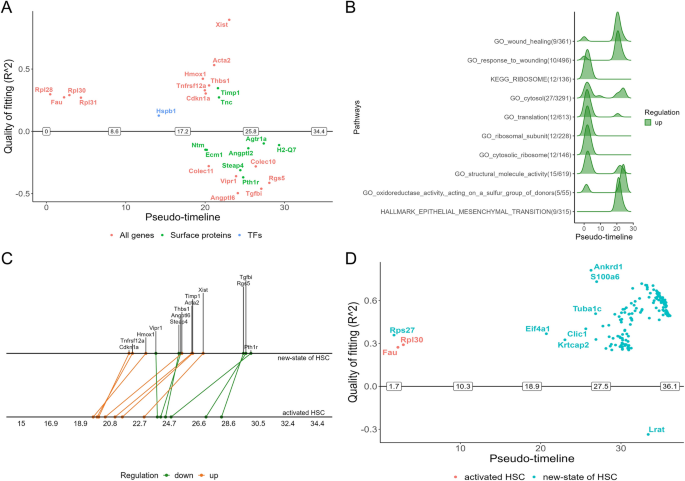
经过scRNA-seq数据的质量控制和归一化(GSE132662) [5], 8500个细胞和15,379个基因被纳入我们的分析(图。1A),并鉴定出2000个高变异基因用于后续分析(图。1B).然后,在体外培养的第0、1、3、7、9天收获的造血干细胞通过t-SNE算法成功划分为14个单独的簇(图。1C),所有14个聚类的顶级标记基因如图所示。1D.根据标记基因的表达模式,聚类0、1、2、4、8、9和13的细胞被标记为静态HSC,表达高水平的静态标记,如Lrat,Reln,Rgs5[20.],而簇3、5、6、7、10、11和12的细胞被标记为激活的HSC,表达高激活标记Acta2,Ccn2,Timp1(无花果。1E)。
造血干细胞分化轨迹分析
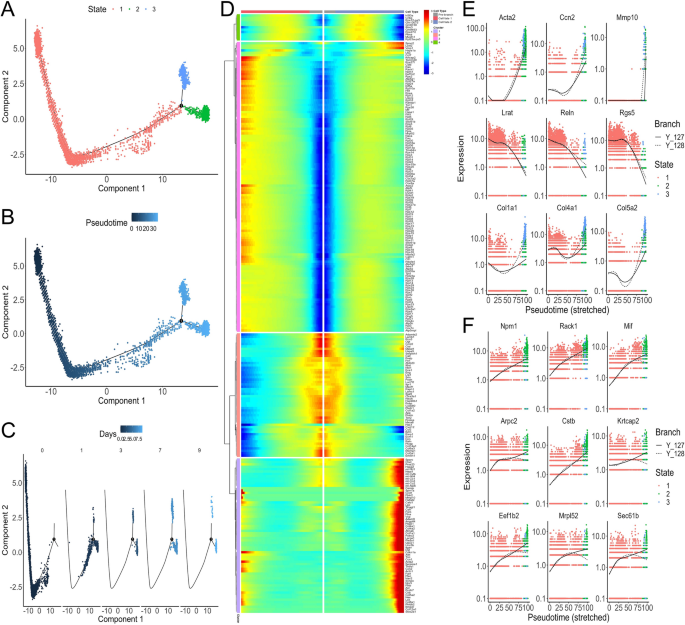
此外,我们可视化了造血干细胞的转录谱,并沿假时间轨迹绘制了它们。我们的数据显示了从静止的HSC到激活的HSC的分化轨迹,从而为HSC分配了3种伪时间依赖性分化状态(图2)。2A)。此外,体外造血细胞转分化的伪时间与造血细胞培养活化过程恰好一致(图1)。2值得注意的是,造血干细胞分叉为两个不同的分支(状态2和3)(图2)。2A),代表两种细胞命运(细胞命运1和2)(图。2分别D)。从细胞来源来看,状态1(分支前)主要由第0天收获的造血干细胞填充,部分在第1天收获。与状态2(细胞命运1)相似,状态3(细胞命运2)的细胞组成也来自于第3、7、9天和部分第1天收获的造血干细胞(图2)。2A, C, D)。因此,状态2和3可能对应hsc向肌成纤维细胞转变的状态。为了深入了解HSC转分化的过程,我们根据前250个差异表达基因的动态,进行了一个分支热图来显示这两个细胞命运分支的基因表达模式。这些基因被分成4个簇(图2)。2D)根据其特征模式,并在附加文件中列出2:表S1。
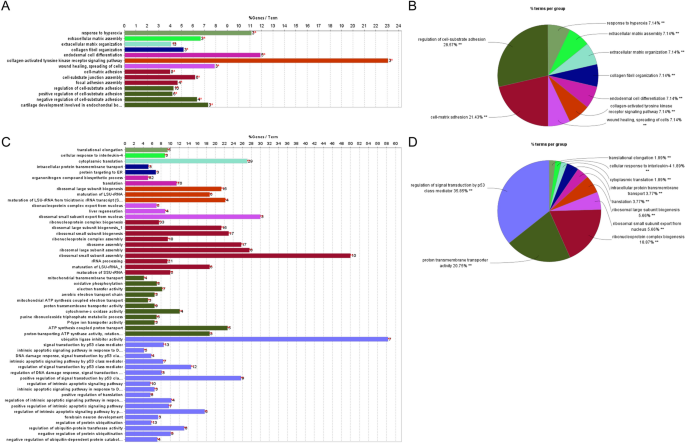
如图所示。2D,聚类1的顶层成员基因从分支前开始(状态1)逐渐上调,并在细胞命运2的最后阶段(状态3)达到较高水平。使用ClueGO和CluePedia进行功能富集分析,结果表明这些基因在“调节细胞-基质粘附”、“细胞-基质粘附”、“细胞外基质组装”等生物过程中富集较多(图3)。3.A, B).同时高表达HSC激活标志物(如:Acta2,Ccn2,Mmp10,Col1a1,Col4a1,Col5a2)、静息标记(Lrat,Reln,Rgs5分支2(包括状态1和状态3)的速度从状态1开始迅速下降(图1)。2E).因此,状态3的细胞代表典型的体外培养激活的hsc(肌成纤维细胞)。同时,伴随着分支1(状态1和状态2)中沉默标记的下调,表达水平降低Acta2,Ccn2,Mmp10在状态2中,激活的HSC标记物也较高(图2)。2E),提示属于状态2的造血干细胞也可能被激活。但状态2中部分胶原蛋白基因的表达量相对低于状态3,如Col1a1,Col4a1,Col5a2(无花果。2E)。
有趣的是,大多数核糖体蛋白基因在细胞命运1(状态2)中表达高于细胞命运2(状态3)(图3)。2D),这表明状态2的细胞可能比状态3的细胞有更高的翻译能力来适应HSC的转分化和增殖。除核糖体亚单位基因外,簇3中的其他基因在状态1中表达逐渐上调,但在状态3中表达不再上调,甚至下调,并且沿着分支1(状态1和状态2)不断上调(如:Npm1,Rack1,Mif,Cstb,Arpc2,Krtcap2等)(图;2F).然后选择聚类3中的基因进行功能富集分析,发现对应基因比例最高的功能群为“p53类中介因子调控信号转导”,包括“DNA损伤反应调控、p53类中介因子信号转导”、“p53类中介因子凋亡内在信号通路调控”、“蛋白质泛素化调控”(图。3.C, D;额外的文件3.:表S2),均与肿瘤密切相关。特别是一些基因如Npm1,Rack1,Mif,Cstb,Arpc2参与肝癌的发生[21,22,23,24,25].根据HSC的分化轨迹分析,我们提出状态2可能是HSC的特定状态,可能与肿瘤发生的调控有关。
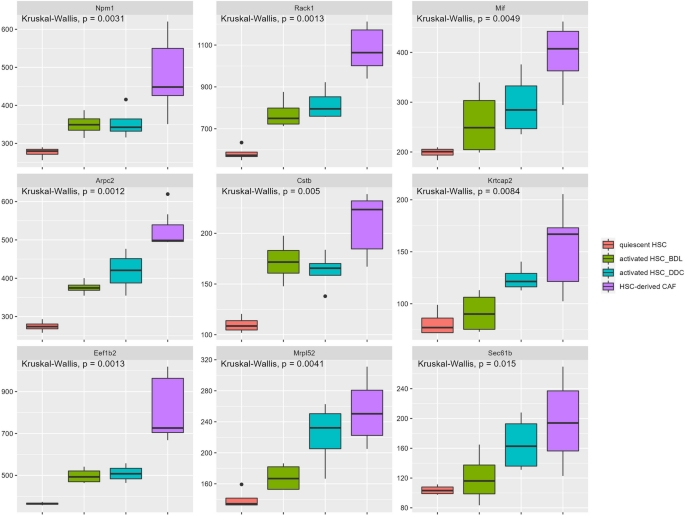
研究表明,来自活化的造血干细胞的CAFs构成了肝细胞癌(HCC)基质中的主要群体,并对HCC的进展有积极影响[26].然后,我们分析了数据集GSE154170的大量rna测序数据,包括静态造血干细胞、来自BDL或DDC饮食胆纤维化模型的激活造血干细胞以及来自CCA模型的造血干细胞来源的CAFs的基因表达谱。结果表明,基因如Npm1,Rack1,Mif,Cstb,Arpc2,Krtcap2在造血干细胞来源的CAFs中表达明显高于BDL或DDC活化的造血干细胞(图2)。4).在属于状态2的造血干细胞中表达的这些癌症相关基因似乎与造血干细胞来源的CAFs中表达的基因具有相似的表达特征(图2)。2F).因此,我们推测状态2的造血干细胞可能是肝脏肿瘤微环境(TME)中CAFs的重要来源。
此外,为了深入了解体内造血干细胞的激活,我们分析了scRNA-seq数据集GSE137720 [4]从健康和CCl中分离的造血干细胞4-处理过的老鼠肝脏。如附加文件所示4:图S1,造血干细胞的分化轨迹也显示出两个分支。状态1的细胞组成主要来自静止的HSC,状态2(细胞命运1)对应HSC向肌成纤维细胞转变的状态,状态3(细胞命运2)可能是HSC的新状态。功能富集分析显示,在“泛素连接酶抑制剂活性”、“核糖体大亚基生物发生”、“p53类中介信号转导”等生物过程中,hsc中表达最多的基因属于状态3(附加文件)5:图S2),也与肿瘤相关。一些基因,比如Npm1,Dynll1,跑,Cfl1,Eif5a,Mif,Arpc2与HCC相关[21,23,25,27,28,29,30.].
HSC激活过程中开关基因的鉴定
在无花果。2, geneswitch首次应用于单轨迹分支2(状态1和3),在该分支中,细胞从静止的造血干细胞(状态1)分化为经典的培养激活的造血干细胞(状态3)。如图所示。5A,核糖体亚单位基因的诱导是沿着伪时间轨迹的早期开关事件,随后是的上调Hspb1,Hmox1,Tnfrsf12a,Cdkn1a,Thbs1,Acta2,Xist的下调特种加工,Ecm1,Colec11,Vipr1,Angptl6等。其中,Xist(x不活跃的特定转录本)和Angptl6(血管生成素样6)显示最高的拟合质量由麦克法登的伪R2[10],可能是调控HSC激活的关键开关基因(图。5A)。来自GO和沿着假时间轨迹分支2差异表达基因的HALLMARK富集分析的顶级生物通路显示,伤口愈合相关基因在早期上调,随后是核糖体通路、氧化还原酶相关本体论和EMT(上皮间充质转化)在假时间后期上调(图2)。5B),模拟一种体外基因表达模式,该模式与肝成纤维过程中静止的HSC转变为肌成纤维细胞的模式相似。
根据Monocle2进行的伪时间分析,hsc向肌成纤维细胞转变的轨迹从状态1开始,状态1是静止的hsc。经典的培养激活的HSC处于状态3,而新的HSC状态为状态2(图2)。2A).通过GeneSwitches,我们绘制了两个分支之间常见的开关基因,以比较它们的顺序。结果表明,顶部拟合常见的开关基因,如Cdkn1a,Tnfrsf12a,Hmox1,Thbs1,Acta2,Timp1,Xist相继上调,而Vipr1,Steap4,Angptl6,Rgs5,Tgfbi,Pth1r下调(图;5C). geneswitch还发现了一些不同的开关基因,用于HSC的新状态,例如Rps27,Eif4a1,Krtcap2,Clic1,Ankrd1等,而状态3只获得能力而且Rpl30在早期(图;5D)。
高预测性纤维化标志物的鉴定
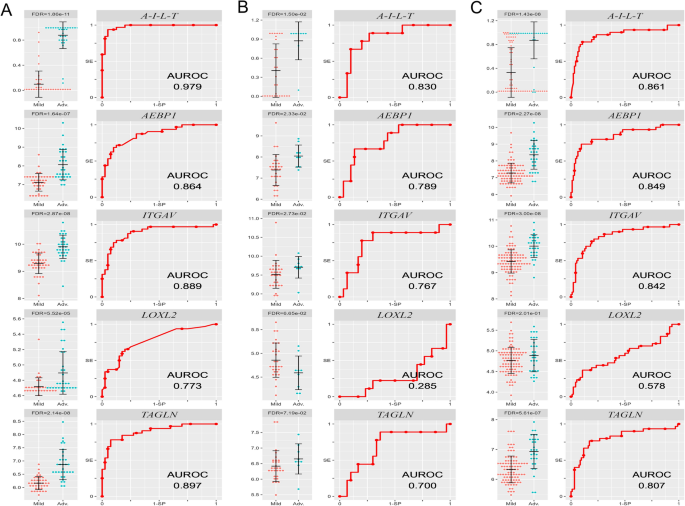
如图所示。2分支2(状态1和状态3)遵循经典的HSC培养激活轨迹,状态3的顶级开关基因是目前临床指标普遍表现不佳的晚期或重度纤维化的潜在诊断标志物。为了确定哪些基因能够区分不同阶段的纤维化,我们分析了NAFLD患者肝活检的公开微阵列数据[14].
如图所示。6,表示AEBP1,ITGAV,LOXL2,TAGLN轻度NAFLD患者(F0-F1纤维化期,n = 40)与晚期NAFLD患者(F3-F4, n = 32)之间存在显著差异(虚假发现率[FDR] < 0.05)。6A).然后我们评估了单个基因和四个基因组合的诊断准确性,AEBP1,ITGAV,LOXL2,TAGLN(A-I-L-T),通过logistic回归从顶部开关基因中鉴定。模型公式为:\ (\ mathrm {P} (\ mathrm{概率})= \压裂{{e} ^ {\ mathrm {R}}} {1 + {e} ^ {\ mathrm {R}}} \), R =−86.283 +(4.733 *表达水平的AEBP1) +(5.405 *表达水平的ITGAV) +(−7.488 * expression level ofLOXL2) +(5.354 *表达水平的TAGLN).值得注意的是组合基因A-I-L-TAUROC为0.979(95%可信区间0.953-1.000),可有效地识别严重纤维化。6A),敏感性为0.938,特异性为0.950,95%的患者为晚期纤维化。
慢性肝病患者高预测性纤维化标志物的鉴定一个来自HSC分化轨迹分析的开关基因在轻度(F0-F1, n = 40)或晚期(F3-F4, n = 32) NAFLD患者活检中的差异表达。平均表达式,标准误差,和fdr值从差分表达式分析显示。ROC曲线显示了各转录本及其组合的鉴别能力AEBP1,ITGAV,LOXL2,TAGLN(A-I-L-T).为了验证这四基因组合,使用来自另一个NAFLD队列的轻度(F0-F2, n = 31)或显著(F3-F4, n = 8)纤维化的芯片数据绘制ROC曲线(B),以及轻度(F0-F2, n = 96)或重度(F3-F4, n = 28)纤维化的HBV患者(C).SE敏感性,SP特异性,AEBP1脂肪细胞增强子结合蛋白1,ITGAV整合素亚基V,LOXL2赖氨酸氧化酶2,TAGLNtransgelin
此外,这四个基因组合在另一个NAFLD队列中的验证[15对于晚期纤维化(F3-4期)的AUROC为0.830(0.689-0.971)。6B).此外,在HBV队列中[16),A-I-L-T基因组合对显著纤维化(F3-F4期)也表现出良好的鉴别能力,AUROC为0.861(0.772-0.951)(图。6C)。这些结果表明,我们从HSC激活轨迹衍生的4个基因组合转化为人类肝纤维化,并准确预测了不同病因引起的肝纤维化的严重程度。
大多数hcc由严重的肝纤维化和肝硬化发展而来。因此,我们从公共数据集中研究了这四种基因在HCC和正常组织中的mRNA表达。结果显示ITGAV而且LOXL2在肿瘤组织中表达高于癌旁组织(TCGA-LIHC,https://portal.gdc.cancer.gov/projects/TCGA-LIHC) (p< 0.001,附加文件6:图S3A)。在GEO数据库的另一个HCC和癌旁样本中获得了类似的结果(GSE41804) [31) (p< 0.05,附加文件6:图S3B)。
人源性星状细胞表达基因的验证
为了验证上述在人类HSC中的发现,基于数据集GSE68000,研究了从三个人类肝脏分离的配对(静止和培养激活)HSC之间与HSC激活、HSC新状态和预测纤维化相关的基因表达[32].如附加文件所示7:图S4A,激活标记物的表达ACTA2,MMP10,COL1A1,COL4A1,COL5A2在活化的造血干细胞中高于在静止的造血干细胞中(p< 0.05)。而静息标记的表达LRAT,RELN,RGS5激活的造血干细胞没有减少。两个基因(RACK1而且ARPC2)在活化的HSC中表达高于静止的HSC (p< 0.05,附加文件7:图S4B)。其他新状态相关基因的表达,如NPM1,MIF,KRTCAP2,EEF1B2,MRPL52,SEC61B激活的造血干细胞也增加,但两组间差异无统计学意义。纤维化标志物ITGAV在活化的造血干细胞中表达高于静止的造血干细胞(p< 0.001,附加文件7:图S4C)。的表达式AEBP1,LOXL2,TAGLN激活的hsc也升高,但两组间差异无统计学意义。
重组人TGF-β1可明显增强LX2细胞(人HSC细胞系)的活化[33].然后,我们验证了的归纳ITGAV而且TAGLN在LX2细胞中进行免疫荧光分析,结果显示TGF-β1处理的LX2与未处理的LX2相比,两种基因的表达均显著增加(附加文件)8:图S5)。
讨论
造血干细胞异常激活、增殖和迁移导致肝纤维化和肝硬化[34].hsc向肌成纤维细胞转变是多种慢性肝损伤诱导肝成纤维过程中的关键事件。本研究分析了体外培养的造血干细胞在0、1、3、7和9天收获的scRNA-seq数据(GSE132662)。我们的结果揭示了在HSC激活过程中出现的一种新的细胞状态,这可能与CAFs有关。此外,我们发现了一些有助于HSC激活的开关基因,并建立了一个四基因组合来预测NAFLD或HBV患者的肝纤维化进展。
从健康小鼠中分离出的原代造血干细胞在无涂层塑料培养皿中培养长达7天是造血干细胞向肌成纤维细胞转变的经典模型[35].在本研究中,我们基于体外培养激活的星状细胞的单细胞转录组数据,利用硅片重建了星状细胞转分化的伪时间轨迹,并确定了星状细胞的一种新状态。与经典激活的HSC相比,属于新状态的细胞表达了较高的HSC激活标记,胶原蛋白基因表达相对较低,但表达了较高水平的p53类中介调控信号转导的基因,其中一些参与了与癌症相关的生物学途径。例如,已经证明Rack1在活化的HSC中高表达,并在HCC中上调[36此外,)Rack1能够促进肝癌患者的癌症干细胞自我更新[22].Npm1在不同的细胞过程中被发现,如核糖体生物发生、细胞增殖和肿瘤抑制因子p53/TP53的调节[37].它与肝癌有关[21].的表达式Mif而且Cstb在肝组织中也被证实参与肝癌的发生[23,24].
肝纤维化是肝损伤后的伤口愈合和疤痕修复反应。肿瘤被认为是“永远无法愈合的伤口”,80%以上的HCC是由病毒性肝炎、NAFLD等慢性肝病引起的肝硬化发展而来[38,39].在HCC的间质中发现活化的造血干细胞[40],而激活的瘤周造血干细胞被证明与肿瘤复发和死亡率相关[41].CAFs是肝癌发生的关键分子,可能主要来源于HSC,可能在HCC和CCA的发病机制中发挥重要作用[17,42,43].TME定义为周围基质的复杂混合物中的肿瘤细胞群,包括CAFs、内皮细胞、免疫细胞和ECM [44].最近,有人提出肝癌的癌前微环境(PME)应与TME区分。PME以慢性肝损伤、炎症和纤维化为特征,先于肿瘤发展[43].我们推测属于新状态的hsc可能是PME的重要组成部分,并有可能在TME中转化为CAFs。进一步研究HSC这种特定状态的命运追踪和HSC向caf转变的调控机制,可能有助于实现针对PME或TME中的肌成纤维细胞进行肿瘤预防或治疗的目标。
通过geneswitch,我们在HSC转分化的伪时间轨迹上发现了许多开关基因。Xist,一种长链非编码RNA,可通过增强乙醇诱导的HSC自噬来调节HSC的激活[45].有趣的是,其他开关基因Hmox1,Hspb1,Tnfrsf12a,Cdkn1a,Thbs1也被认为是自噬的调节剂[46,47,48,49,50].我们认为,自噬在早期阶段可能强烈地促进了星状细胞的激活。另一方面,的表达Angptl6,是内皮细胞趋化活性的调节剂和新生血管的诱导剂[51]在HSC激活期间下调。我们推断Angptl6可能与健康肝脏中的星状细胞功能和肝损伤中的星状细胞向肌成纤维细胞转变有关。此外,geneswitch分别为新状态的星状细胞和经典培养激活的星状细胞识别了一些不同的开关基因。这些特定于每个轨迹(状态)的开关基因及其时间的识别可能有助于进一步的实验,以揭示这些分叉的决定因素。
利用Monocle2和GeneSwitches方法,我们在伪时间内确定了与HSC激活相关的顶级开关基因。我们的结果表明,从NAFLD或HBV患者的活检基因表达数据来看,反映HSC转分化轨迹的基因在预测晚期肝纤维化方面具有潜在的诊断价值。特别是四基因组合,AEBP1,ITGAV,LOXL2,TAGLN,在预测纤维化严重程度方面表现出较高的准确性。AEBP1编码蛋白在脂肪形成和平滑肌细胞分化中起作用。重要的是,在非酒精性脂肪性肝炎患者的肝活检中,其表达随着纤维化的恶化而上调[52].LOXL2对结缔组织的生物发生和介导胶原交联至关重要。该蛋白在小鼠纤维化肝中表达较强,并能抑制LOXL2减轻硫代乙醯胺诱导的肝纤维化[53].整合素αV,由ITGAV基因,与多个器官的纤维化密切相关[54].TAGLN是平滑肌分化的早期标记物,并被认为是抗hbv阳性HCC的重要靶点[55].根据COMPARTMENTS亚细胞定位数据库(http://compartments.jensenlab.org) [56,我们发现AEBP1而且LOXL2编码分泌的蛋白质ITGAV而且TAGLN位于细胞质或细胞外。我们的研究结果表明ITGAV而且TAGLN主要在活化的人HSC细胞质中表达。这四个基因有潜力作为晚期纤维化患者的预测标志物。
这项研究有几个局限性。首先,从人肝脏中分离的造血干细胞大量RNA-seq数据分析的结果与从小鼠肝脏中分离的造血干细胞scRNA-seq数据分析的结果存在一定程度的差异。除物种差异外,从人体内分离的造血干细胞样本量过小可能是导致结果差异的原因。在进一步的研究中,我们计划获得从人肝脏分离的造血干细胞(包括静止的和培养激活的造血干细胞)的scRNA-seq数据,并进行分析以验证我们从小鼠造血干细胞中获得的结果。其次,在使用四基因组合之前(A-I-L-T为了在慢性肝病患者中识别晚期肝纤维化,需要在健康和患者中评估这四种基因的转录本或蛋白质的血清水平。
结论
综上所述,我们揭示了HSC激活过程中的一种新的细胞状态,并推断该状态下的HSC可能是TME中CAFs的重要来源。此外,我们发现了一些与HSC激活相关的关键开关基因,并建立了一个四基因组合,可以作为慢性肝病患者晚期纤维化的预测标志物。
数据和材料的可用性
本文包含了本次研究中分析的所有数据。
缩写
- ECM:
-
细胞外基质
- HSC:
-
肝星状细胞
- Acta2:
-
平滑肌肌动蛋白2
- scRNA-seq:
-
单细胞RNA测序
- 战乱国家:
-
癌症相关成纤维细胞
- 地理:
-
基因表达综合
- 主成分分析:
-
主成分分析
- t-SNE:
-
t分布随机邻居嵌入
- MSigDB:
-
分子特征数据库
- KEGG:
-
京都基因与基因组百科全书
- 非酒精性脂肪肝:
-
非酒精性脂肪肝
- 乙肝病毒:
-
乙型肝炎
- BDL:
-
胆汁duct-ligated
- 监护系统:
-
3 5-Diethoxycarbonyl-1 4-dihydrocollidine
- CCA:
-
胆管癌
- AUROC:
-
接收机工作特性曲线下面积
- 肝细胞癌:
-
肝细胞癌
- Xist:
-
X非活性特异性转录本
- Angptl6:
-
血管生成素样6
- 走:
-
基因本体论
- 时差:
-
肿瘤微环境
- EMT:
-
上皮间质转化
- 罗斯福:
-
错误发现率
- 中外职业:
-
癌变前的微环境
- AEBP1:
-
脂肪细胞增强子结合蛋白1
- ITGAV:
-
整合素亚基V
- LOXL2:
-
赖氨酸氧化酶样2
- TAGLN:
-
Transgelin
参考文献
肝纤维化:病理生理学,发病靶点和临床问题。《医学杂志》2019;65:37-55。
李雅,华莱士MC,弗里德曼SL.肝纤维化的病理生物学:一个翻译成功的故事。肠道。2015;64:830-41。
王鹏,小山勇,刘旭,徐健,马友友,梁松,等。有希望的治疗肝纤维化的候选疗法。前沿物理。2016;7:47。
多比R,威尔逊-卡纳莫里JR,亨德森BEP,史密斯JR,马切特KP,波特曼JR,等。单细胞转录组学揭示肝纤维化期间间质功能分区。Cell rep 2019;29(7):1832-1847.e8。
Krenkel O, Hundertmark J, Ritz TP, Weiskirchen R, Tacke F.单细胞RNA测序鉴定肝纤维化中肝星状细胞和肌成纤维细胞亚群。细胞。2019;8(5):503。https://doi.org/10.3390/cells8050503.
Butler A, Hoffman P, Smibert P, Papalexi E, Satija R.整合不同条件、技术和物种的单细胞转录组数据。中国生物工程学报。2018;36(5):411-20。https://doi.org/10.1038/nbt.4096.
李志强,李志强,李志强。基于结构感知的单细胞rna序列数据的主成分分析。计算生物学杂志,2018。https://doi.org/10.1089/cmb.2018.0027.
张志刚,张志刚,张志刚。单细胞基因表达数据的空间重构。中国生物工程学报。2015;33(5):495-502。https://doi.org/10.1038/nbt.3192.
邱霞,毛青,唐勇,王玲,Chawla R, Pliner HA,等。反向图嵌入解决复杂的单细胞轨迹。光子学报。2017;14(10):979-82。https://doi.org/10.1038/nmeth.4402.
曹永毅,欧阳景峰,Rackham OJL。基因开关:单细胞实验中的基因表达和功能事件排序。生物信息学。2020;36(10):3273 - 5。https://doi.org/10.1093/bioinformatics/btaa099.
定性选择行为的条件logit分析。《前沿经济学家》1974;104-142。
Liberzon A, Birger C, Thorvaldsdóttir H, Ghandi M, Mesirov JP, Tamayo P.分子特征数据库(MSigDB)标志基因集收集。科学通报。2015;1(6):417-25。https://doi.org/10.1016/j.cels.2015.12.004.
后藤凯格:京都基因基因组百科全书。核酸研究,2000;28(1):27-30。https://doi.org/10.1093/nar/28.1.27.
Moylan CA, Pang H, Dellinger A, Suzuki A, Garrett ME, Guy CD,等。肝脏基因表达谱区分症状前患者轻度和重度非酒精性脂肪性肝病。肝脏病学。2014;59(2):471 - 82。
Arendt BM, Comelli EM, Ma DW, Lou W, Teterina A, Kim T,等。非酒精性脂肪肝患者肝脏基因表达改变与肝脏n-3和n-6多不饱和脂肪酸降低有关。肝脏病学。2015;61(5):1565 - 78。
王敏,龚强,张娟,陈琳,张震,卢玲,等。hbv相关肝纤维化患者基因表达谱的特征和ITGBL1作为纤维化发生的关键调节因子的鉴定。科学代表2017;7:43446。https://doi.org/10.1038/srep43446.
阿福S, Nair A, Brundu F, Ravichandra A, Bhattacharjee S, Matsuda M,等。不同癌相关成纤维细胞亚群促进胆管癌生长。中国生物医学工程学报,2017;39(6):866-882.e11。https://doi.org/10.1016/j.ccell.2021.03.012.
Robin X, Turck N, Hainard A, Tiberti N, Lisacek F, Sanchez JC等。pROC:用于分析和比较ROC曲线的R和S+的开源包。BMC生物信息学。2011;12:77。https://doi.org/10.1186/1471-2105-12-77.
Wickham H. ggplot2:用于数据分析的优雅图形。柏林:施普林格;2009.
Terkelsen MK, Bendixen SM, Hansen D, Scott EAH, Moeller AF, Nielsen R,等。肝损伤后肝窦相关细胞的转录动态。肝脏病学。2020;72(6):2119 - 33所示。https://doi.org/10.1002/hep.31215.
刘旭,刘东,钱东,戴杰,安勇,姜松,等。核色素(NPM1/B23)与活化转录因子5 (ATF5)蛋白相互作用,促进肝细胞癌细胞中蛋白酶体和caspase依赖性的ATF5降解。中国生物医学工程学报,2012;https://doi.org/10.1074/jbc.M112.363622.
曹娟,赵敏,刘娟,张鑫,裴勇,王娟,等。RACK1在人肝细胞癌中通过稳定nanog促进癌症干细胞的自我更新和化疗耐药性。开展。2019;9(3):811 - 28。https://doi.org/10.7150/thno.29271.
李艳,陆超,邢刚,朱艳,何峰。巨噬细胞迁移抑制因子直接与肝生成素相互作用并调控肝癌细胞增殖。科学通报,2004;30(2):379-87。https://doi.org/10.1016/j.yexcr.2004.07.019.
周旭,王旭,黄凯,廖旭,杨超,于涛,等。乙型肝炎病毒相关肝细胞癌患者胱抑素基因的临床意义及潜在分子机制研究中国农业科学,2019;42(1):189-201。https://doi.org/10.3892/or.2019.7154.
张超,吴松,杨晓东,徐辉,马涛,朱庆霞。丁型肝炎病毒相关肝细胞癌关键基因的生物信息学分析。中华胃肠病杂志,2021;32(2):169-77。https://doi.org/10.5152/tjg.2020.191003.
尹震,董超,姜凯,徐震,李锐,郭凯,等。癌相关成纤维细胞的异质性及其在肝细胞癌的进展、预后和治疗中的作用中华血液病杂志2019;12(1):101。https://doi.org/10.1186/s13045-019-0782-x.
Berkel C, Cacan E. DYNLL1以肿瘤分期和分级依赖的方式低甲基化和上调,与肝细胞癌死亡率增加有关。Exp Mol Pathol. 2020;117: 104567。https://doi.org/10.1016/j.yexmp.2020.104567.
姚玲,周勇,隋智,张勇,刘勇,谢辉,等。hbv编码的miR-2在肝癌细胞中通过下调TRIM35而上调RAN发挥癌基因作用。EBioMedicine。2019; 48:117-29。https://doi.org/10.1016/j.ebiom.2019.09.012.
姚波,李勇,陈涛,牛勇,王勇,杨勇,等。低氧诱导的cofilin 1通过调节PLD1/AKT通路促进肝细胞癌的进展。中华医学杂志2021;11(3):e366。https://doi.org/10.1002/ctm2.366.
李玲,李霞,张强,叶涛,邹松,闫娟。EIF5A在肝细胞癌中的表达及其诊断意义。中华癌症杂志,2021;12(16):4774-9。https://doi.org/10.7150/jca.58168.
虎藤Y,本田M,田中A,野村Y,新井K,山下T,等。白介素- 28b基因型与慢性丙型肝炎患者肝细胞癌复发的关系。中国临床癌症杂志,2013;19(7):1827-37。https://doi.org/10.1158/1078-0432.CCR-12-1641.
El Taghdouini A, Sørensen AL, Reiner AH, Coll M, Verhulst S, Mannaerts I,等。DNA甲基化和基因表达模式的全基因组分析纯化,未培养的人肝细胞和活化的肝星状细胞。Oncotarget。2015;6(29):26729 - 45。https://doi.org/10.18632/oncotarget.4925.
陈伟,吴霞,闫霞,徐安,杨安,尤慧。多转录组分析揭示了与肝纤维化进展相关的优先级基因。中国医学杂志。2019;316(6):G744-54。https://doi.org/10.1152/ajpgi.00339.2018.
田建平,李建平。肝星状细胞活化机制的研究。中华胃肠病杂志,2017;14(7):397-411。
陈志强,李志强,李志强,等。肝星状细胞在肝纤维化中的作用及其分离技术。中华胃肠病杂志,2012;6(1):67-80。
Bourd-Boittin K, LePabic H, Bonnier D, L 'Helgoualc 'h A, Théret N. RACK1,一种新的ADAM12相互作用蛋白。对肝纤维化的贡献。中国生物医学工程学报,2008;29(3):344 - 344。https://doi.org/10.1074/jbc.M709829200.
任喆,Aerts JL, Vandenplas H,王佳,Gorbenko O,陈建平,等。磷酸化的STAT5通过BRCA1/BARD1-NPM1和MDM2调控p53的表达。细胞死亡,2016;7(12):e2560。https://doi.org/10.1038/cddis.2016.430.
O 'Rourke JM, Sagar VM, Shah T, Shetty S.肝纤维化背景下的癌变:对肝细胞癌管理的意义。世界胃肠杂志,2018;24(39):4436-47。https://doi.org/10.3748/wjg.v24.i39.4436.
德沃夏克高频。肿瘤:无法愈合的伤口。肿瘤基质生成与伤口愈合的相似性。中华实用医学杂志,2000;26(2):357 - 357。https://doi.org/10.1056/NEJM198612253152606.
Amann T, Bataille F, Spruss T, Mühlbauer M, Gäbele E, Schölmerich J,等。活化的肝星状细胞促进肝细胞癌的成瘤性。癌症杂志2009;100(4):646-53。https://doi.org/10.1111/j.1349-7006.2009.01087.x.
鞠敏梅,邱世杰,范军,肖玉生,高强,周杰,等。肝细胞癌根治性切除术后,瘤周活化的肝星状细胞预示着较差的临床预后。中华临床病理学杂志,2009;31(4):498-510。https://doi.org/10.1309/AJCP86PPBNGOHNNL.
张志刚,张志刚,张志刚,等。肝纤维化和肝相关成纤维细胞在肝细胞癌发病机制中的作用。国际分子化学杂志,2019;20(7):1723。https://doi.org/10.3390/ijms20071723.
阿福,于丽霞,施华贝RF。癌症相关成纤维细胞和纤维化在肝癌中的作用。《末日启示》2017;12:153-86。https://doi.org/10.1146/annurev-pathol-052016-100322.
Tahmasebi Birgani M, Carloni V.肿瘤微环境,肝细胞癌进展和治疗的范例。中华分子化学杂志,2017;18(2):405。https://doi.org/10.3390/ijms18020405.
谢志勇,王法芬,肖志华,刘顺丰,赖丽丽,唐立林。长链非编码RNA XIST通过miR-29b/HMGB1轴增强乙醇诱导的肝星状细胞自噬和激活。IUBMB生命科学。2019;71(12):1962-72。https://doi.org/10.1002/iub.2140.
觉得西南。血红素加氧酶-1/一氧化碳作为自噬和炎症的调节剂。生物化学学报。2019;678:108186。https://doi.org/10.1016/j.abb.2019.108186.
孙鹏,张松,秦霞,常霞,崔霞,李华,等。口蹄疫病毒衣壳蛋白VP2激活细胞EIF2S1-ATF4通路,通过HSPB1诱导细胞自噬。自噬。2018;14(2):336 - 46。https://doi.org/10.1080/15548627.2017.1405187.
Winer H, Fraiberg M, Abada A, Dadosh T, tamim - yeecheskel BC, Elazar Z.自噬对TNF受体Fn14的差异调控地理学报。2018;9(1):3744。https://doi.org/10.1038/s41467-018-06275-1.
王晓明,王晓明,王晓明,袁军。CDKN1A基因在细胞分化、迁移和肿瘤治疗中的作用。癌症(巴塞尔)。2019; 11(9): 1220。https://doi.org/10.3390/cancers11091220.
Kalas W, Swiderek E, Switalska M, Wietrzyk J, Rak J, Strzadala L. Thrombospondin-1受体介导ras表达癌细胞的自噬并触发肿瘤生长抑制。中国癌症杂志,2013;33(4):1429-38。
冈崎H,平川S,树斗M,中冈Y,白方Y,宫田K,等。血管生成素相关生长因子(Angptl6/angiopoietin相关生长因子)在皮肤中的靶向过表达促进血管生成和淋巴血管扩张。中华皮肤医学杂志,2012;39(4):366-74。https://doi.org/10.1111/j.1346-8138.2011.01396.x.
Gerhard GS, Hanson A, Wilhelmsen D, Piras IS, Still CD, Chu X,等。AEBP1的表达随着NASH纤维化的严重程度而增加,并受葡萄糖、棕榈酸盐和miR-372-3p的调控。PLoS ONE。2019; 14 (7): e0219764。https://doi.org/10.1371/journal.pone.0219764.
Ikenaga N, Peng ZW, Vaid KA, Liu SB, Yoshida S, Sverdlov DY,等。选择性靶向赖氨酸氧化酶样2 (LOXL2)抑制肝纤维化的进展并加速其逆转。肠道。2017;66(9):1697 - 708。https://doi.org/10.1136/gutjnl-2016-312473.
Henderson NC, Arnold TD, Katamura Y, Giacomini MM, Rodriguez JD, McCarty JH,等。以αv整合素为靶点,发现了调控多个器官纤维化的核心分子通路。中华外科杂志,2013;19(12):1617-24。https://doi.org/10.1038/nm.3282.
刘震,徐勇,张伟,高旭,罗刚,宋华,等。利用iTRAQ蛋白质组学鉴定JS-K抗hbv阳性人肝细胞癌HepG2.2.15细胞的靶点。科学通报2021;11(1):10381。https://doi.org/10.1038/s41598-021-90001-3.
宾德JX, Pletscher-Frankild S, Tsafou K, Stolte C, O 'Donoghue SI, Schneider R,等。隔间:统一和可视化的蛋白质亚细胞定位证据。数据库(牛津大学)。2014; 2014:012。https://doi.org/10.1093/database/bau012.
确认
不适用。
资金
没有资金。
作者信息
作者及隶属关系
贡献
ZW和SZ构思并设计了这个研究。HW和HJ进行了scRNA-seq数据分析。ZW, SZ, XW和FZ起草了手稿。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称不存在利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
王浩,郑寿,姜浩,王浩。et al。单细胞转录组分析揭示了一种新的细胞状态和肝星状细胞激活过程中的开关基因。翻译医学杂志20., 53(2022)。https://doi.org/10.1186/s12967-022-03263-4
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12967-022-03263-4
关键字
- 肝纤维化
- 肝星状细胞
- 单细胞RNA测序