摘要
背景
为了推动转化医学,现代生物库需要与其他数据来源(临床、基因组学)整合,以支持新的数据密集型研究。目前,大量的研究和临床数据仍然处于竖井中,由个别研究人员持有和管理,在不同的标准和治理结构下运行;一个阻碍数据共享和有效使用的框架。在本文中,我们描述了不列颠哥伦比亚省妇科癌症研究项目(OVCARE)将传统的肿瘤生物库、结果单元和数据仓库集合转移到一个集成的数据公共空间,以支持数据标准化和协作治理下的资源共享,作为一种手段,为不列颠哥伦比亚省的妇科癌症研究社区提供来自数千名患者的组织样本和相关临床和分子数据。
结果
通过与我们研究社区内不同研究机构的利益相关者的几次接触,我们确定了优先事项,并评估了优化和支持数据收集、存储和共享所需的基础设施需求,主要研究领域包括:(1)生物标本收集,(2)分子和基因组学数据,以及(3)临床数据。我们进一步构建了治理模型和资源门户,以实现无缝收集、管理和治理互操作数据的协议和标准操作程序,使基因组和临床数据可供更广泛的研究界使用。
结论
适当的数据收集、共享和治理基础设施是转化研究的必要条件。我们已经将我们的数据储备整合到一个数据共享空间中,并制定了标准化的操作程序,以满足不列颠哥伦比亚省妇科癌症社区的研究和道德要求。开发的基础设施汇集了不同的数据、计算框架以及用于管理、分析和共享数据的工具和应用程序。通过提供数据密集型科学所需的大型数据集,我们的数据公域弥合了精准医疗和妇科癌症诊断、治疗和预防方法的数据访问差距和障碍。
背景
用于病理诊断的人类生物标本的收集、储存、管理和分发[1,2,3.]可以追溯到20世纪。[3.].为满足后基因组时代的研究需要,现代生物储存库[4]支持科学家获得针对特定疾病的见解[5]通过协助研究遗传基础[6,7,8],阐明病因,评估疾病进展和治疗反应;他们是精准医疗的中坚力量。9,10]、生物医学和转化研究[1,2,11].
过去十年见证了生物技术的进步,如下一代测序(NGS),以及用于精准医疗的“组学”技术的出现(例如基因组学、转录组学、蛋白质组学、代谢组学和表观基因组学)。这些创新与计算、人工智能(AI)和分析技术的突破相吻合,使疾病之间的区分更加精确。12].这就产生了对高质量生物标本和相关数据的前所未有的需求,包括临床、分子、成像和研究过程中产生的其他类型的数据[11].支持数据密集型科学的数据库云存储和计算基础设施方面的创新进一步促进了可用于满足现代研究需求的资源革命[13,14].由于用于聚合数据和生物材料的联合模型已成为识别足够多具有特定临床或分子特征的患者的有利方法,生物库和相关数据库之间互操作性的重要性已得到强调[4,7,15].标本收集已变成虚拟的[13],灵活和互操作,托管在国际协调的基础设施上[7]并优化用于二次研究[7,13].目前的研究环境和需求导致了发展和实施数据共享[16,17],在一个研究社区内,将不同的数据、计算基础设施以及用于管理、分析和共享互操作数据的工具和应用程序汇集在一起。这为最大化合作和扩大原始数据收集所产生的价值创造了机会[18].
2016年,作为BC省多学科妇科癌症研究团队(OVCARE)的一部分,我们对本地研究环境中可用的数据资产进行了全面审查,并评估了支持我们研究社区内数据存储和共享所需的基础设施需求。在此,我们描述了创建数据共享的路线图,将传统的肿瘤生物库和数据筒仓集合转化为集成和全面的基础设施,以支持不断扩大的团队当前和未来的研究需求。
结果
将技术解决方案与研究需求相匹配
OVCARE始于2000年,是不列颠哥伦比亚省癌症机构、不列颠哥伦比亚大学和温哥华海岸健康研究所共同发起的一项倡议,旨在加快研究发现和转化为临床环境,并改善患有卵巢癌或有风险的女性的生活。今天,OVCARE是一个由医生和科学家组成的国际公认的多学科团队,他们在改善所有妇科癌症的诊断、预防和治疗方面开辟了新的领域[19,20.,21,22,23,24,25,26,27].
OVCARE的研究由妇科肿瘤库和谢丽尔·布朗妇科癌症预后部门提供支持。在研究过程中,大量的分子和基因组数据一直由生成它们的研究人员持有。同样,从图表回顾中获得的临床数据用于支持临床研究,并与临床医生一起进行。这些数据的格式不兼容,需要大量的手工操作和管理才能集成。此外,每次收集都受到不同的道德协议的约束,这些协议限制了数据的使用,并将数据保存在竖井中。这正在成为需要整合多个数据源的新型数据密集型研究的障碍;进行这样的项目是具有挑战性的,耗时的,并且容易出错;OVCARE领导层认识到,目前的研究需求无法通过现有的基础设施来满足。
2016年开展了广泛的利益相关者参与工作,目标是与各机构的研究人员、临床医生、科学家和技术人员合作,制定集体未来愿景,确定研究需求,并重新思考现有的基础设施。与主要利益相关者的接触确定了研究优先事项,并将其扩展为基本需求列表(表2)1)与生物标本、临床和分子/基因组学数据的收集和优化有关,以及由此产生的基础设施的治理模型。除了提高效率、限制错误和尊重患者的同意外,基本的研究要求还包括最大限度地利用数据的二次利用,使为一个目的收集的数据能够用于完全不同的环境。例如,在我们药房分发的化疗药物被收集用于管理目的(计费),但也可以用于与患者的表型、基因型和结果联系起来,以调查哪些患者比其他患者更受益于这些治疗。另一个重要的需求是通过同时考虑以前无法同时考虑的各种数据来产生新的研究假设。以前可能不明显的模式可能会出现,从而推动未来的创新研究。另一个重要的需求是使用转化研究来帮助为患者护理提供信息,以及使用患者护理产生的数据来提出新的研究问题,以不断尝试更好地填补对疾病病因学和进展的理解方面的空白。在接下来的章节中,我们将更详细地描述这些需求。
Biospecimen集合
OVCARE采用两种模式进行生物标本招募:第一种是一般的银行模式,具有广泛的科学目标,从同意的参与者那里获得标本并保存到需要时。第二种是基于研究的银行模式,参与者被招募来解决特定的研究目标,有预先定义的方案和预先计划的样本收集。为了适应这两种方法,在两种招募模式下,生物储存库基础设施需要以患者为中心的方法管理标本的积累,保留患者临床病史的背景,并支持在不同地点的多个研究中进行基本的生物标本收集、存储和分配。这包括库存控制,跟踪样本可用性和位置的能力,以及跟踪生成的衍生品(例如,异种移植和类器官)。基础设施需要适应研究、项目之间的需求变化,以及随着时间的推移,能够保存数据的自然历史。不同用户组的访问控制有所不同,这是确保遵守监管要求和卫生研究最佳做法的一个关键特征。出于同样的原因,除了需要在最少的机构和研究IT支持下操作和管理生物储存库外,数据安全、标本的去识别和同意跟踪也很重要。
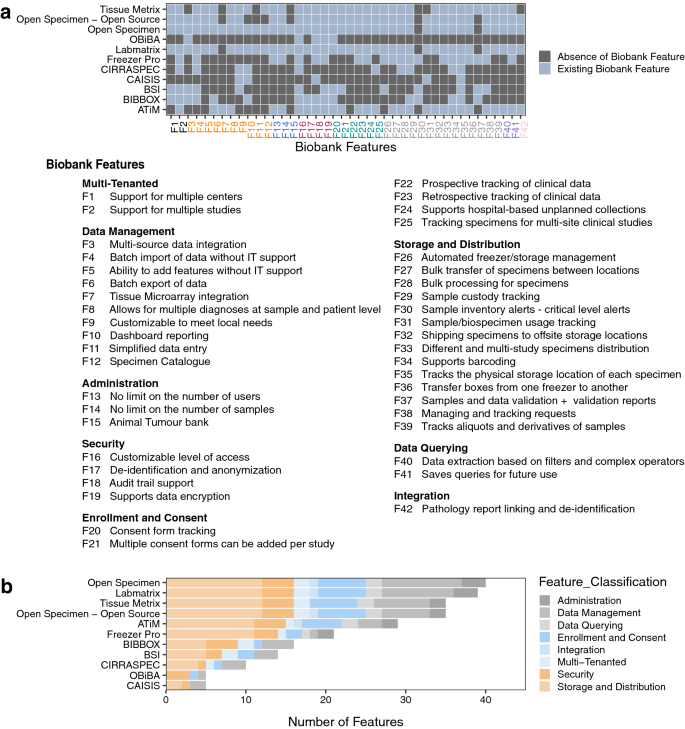
我们编译了一个全面的需求列表(附加文件2:表S1)来自我们的利益相关者会议,我们使用它来指导我们扫描现有实验室信息管理系统(LIMS)的景观(附加文件)2表S2-S11、图1).这导致了opensample [28],基于caTissue的LIMS [29],这是一个成熟的系统,已被研究界使用超过15年。与我们考虑的其他选项相比,opensample满足了我们列表中的更多需求。它是一个有商业支持的开源软件,被20个国家的70多个生物库使用。商业支持确保了持续的软件测试、更新和持续改进。除此之外,还提供技术支持,并通过活跃的论坛访问有经验的用户社区。
在这个LIMS中,生物标本可以单独或批量处理,基于条形码的快速扫描可一次输入多个患者样本的信息。这使得我们能够从遗留的LIMS进行高吞吐量处理和高效迁移。数据注释和存储管理选项使我们能够优化样本存储,这是我们研究社区的一种昂贵资源(例如,−80个冰柜)[29].
opensample LIMS允许通过图形用户界面(web界面)定制数据输入表单,以匹配特定的研究需求,而不需要软件开发。该平台满足了我们大部分的IT需求,因为它支持基于角色的访问控制,并提供了每个用户操作的审计跟踪[30.].该系统还易于使用基于图形的查询,可以搜索关于参与者、生物标本或项目的存储数据,而不需要任何编程,使大多数用户都可以访问适度复杂的查询。查询也可以通过REST API(具象状态传输应用程序编程接口)使用类似SQL(结构化查询语言)的查询语言来执行。通过合并查询脚本,这促进了分析管道数据下载的自动化。
该系统通过软件开发工具包支持独立插件。这些插件可以公开提供给社区。例如,组织微阵列(TMA)插件可以通过链接到供体块并描述在TMA块的不同切片上所做的实验细节来管理opensample上的TMA。最后,与其他系统的互操作性对于扩大数据共享内的联系非常重要。该供应商提供与电子数据捕获应用程序(REDCap, Open Clinica),电子病历系统(EPIC, Velos),病理系统(CoPath, Cerner, Aperio)以及健康级别7 (HL7)消息的集成;一种能够进一步支持包含来自分布式系统的参与者和生物标本信息的能力。
分子和基因组学数据
在研究过程中产生了各种分子和基因组学数据。其中包括下一代测序、蛋白质组学、基因表达、靶向测序以及免疫组化数据。这些数据的产生主要是为了回答特定的研究假设,并得到了公共、政府和慈善基金的支持,隐含的义务是尽量减少重复工作,并优化它们在后续研究中的二次使用。同时考虑所有这些数据的能力可以揭示新的模式、趋势和未知的相关性。这可能会引发新的假设,并激发对新的研究方向的新见解。为了实现这种级别的集成,我们需要跟踪对哪些样品进行了哪些分析分析,并链接回这些数据。为了方便对这些复杂数据的查询,需要一个探索工具来可视化生成的多维数据集,同时研究分子特征和临床属性。
我们采用了癌症基因组学的cBioPortal [31],是最受推荐及广泛使用的[32,33,34,35,36泛癌症分析web工具,以促进从各种癌症研究中收集的肿瘤样本衍生的多维数据集的交互式探索、挖掘、分析和可视化[31,37].该平台由纪念斯隆·凯特琳癌症中心(MSK)开发,用于大型癌症基因组研究(TCGA [38],目标[39]),而公众可在我们的资料集旁下载及查询。
cBioPortal能够收集每个肿瘤样本的各种基因组数据,包括非同义突变、拷贝数改变(cna)、mRNA和microRNA表达数据、DNA甲基化数据、蛋白质和磷蛋白水平数据[31].这些数据类型中的每一种都被集成并存储在基因水平上,以允许研究人员探测特定生物事件的存在(例如,每个样本中的基因突变、缺失、扩增和表达水平)[37],并在样本和多个集成数据类型之间比较离散的基因组事件和模式[31].存储的基因水平数据与去识别的临床数据相结合,以探测患者的临床结果,以支持对特定癌症中频繁改变的基因的假设的发展或测试[31,37].此外,它还有助于研究某些基因在妇科和其他癌症中的预后作用[34]、突变、表达谱、临床病理特征和某些癌症类型的潜在诊断和治疗靶点之间的相关性。
临床数据
OVCARE获得和收集临床数据的目的是评估结果,提高患者护理质量,以及进行研究。其中一些数据由Cheryl Brown Outcomes Unit历史管理,用于对BC癌症(省三级癌症中心)转诊的卵巢癌患者进行结果研究。BC省癌症登记处定期向Cheryl Brown Outcomes Unit提供数据更新,如癌症患者的识别及其生命统计数据,并辅以详尽的图表审查。除了Cheryl Brown Outcomes Unit,临床医生还经常对其他临床研究进行图表审查;所得数据单独保存。2016年,Cheryl Brown Outcomes Unit的数据收集范围仅限于卵巢癌,没有充分利用其他可用数据。收集临床数据是资源密集型的,从长远来看,所需要的努力是不可持续的。此外,谢丽尔·布朗结果小组的任务扩大,使OVCARE的研究人员能够研究BC省的所有妇科癌症,特别是那些不需要转诊到癌症中心的癌症(例如,在BC省,高达50%的子宫内膜癌患者由他们所在社区的妇科医生治疗)。因此,该团队的一个重要优先事项是提高临床数据收集的效率,并将来自各种来源的所有妇科癌症临床数据标准化、整合和链接,并将临床数据整合到单个数据库中。这将允许研究人员了解哪些临床数据已经可用,从而简化他们自己的数据收集策略,反过来,这将直接有助于主数据库。 To maximize the re-use of clinical data, standardization of ontologies across projects was needed, as well as the creation of infrastructure to serve as permanent storage with an easy-to-use data collection interface adaptable to fit the needs of various research projects. This would allow standardization of data collection, to the extent possible, and minimization of errors. Consequently, this would improve the overall quality of data, maximize interoperability and reusability, and optimize data analysis. Management of sensitive clinical data requires security, privacy and the use of tools and technology with institutional approval. We also needed rigorous security and privacy measures, and comprehensive audit trails for tracking data manipulation, exports, and downloads for both single and multi-centered research studies, including tracking data access.
为了支持OVCARE的临床数据需求,我们采用了研究电子数据捕获(REDCap),这是一种广泛使用、免费和灵活的基于web的应用程序[40,41]由范德比尔特大学开发,用于临床和转化研究。它是最受欢迎的研究电子数据系统之一,在141个国家实施,超过1,000,000 [42研究,包括我们的机构。REDCap灵活的设计支持永久数据库集合,可以通过以患者/研究为中心的调查或数据收集形式进行扩充,并包括一组丰富的模块,支持当今多样化和多规模的生物医学研究操作[41].
治理结构
为了管理各种集成数据集(生物标本、分子、基因组和临床数据),我们需要确保适当的治理、协议和标准操作程序,以支持数据共享,简化数据请求和查询,进行科学审查或请求,并确保伦理批准的可用性。我们设想了一个用于所有请求和查询的单一门户应用程序,并使用后端数据库跟踪请求研究人员的详细信息、项目描述、所需资源以及与之相关的伦理应用程序和批准证书。这个基础设施将有助于遵守道德规范,并维护所有活动的日志。
我们采用了Oracle Application Express (APEX) [43],甲骨文公司,开发此门户应用程序。我们的机构APEX已经提供了一个低代码、数据驱动的平台,用于快速开发和部署可扩展和安全的web应用程序。应用程序在预配置的环境中实现;所有的开发都是通过一个主要基于GUI(图形用户界面)的web界面完成的。web应用软件堆栈的中间层功能,例如解析超文本传输协议(HTTP)请求和会话管理,都是完全自动化的,系统的所有操作方面(数据备份、软件补丁和更新)都由机构IT管理。
实现
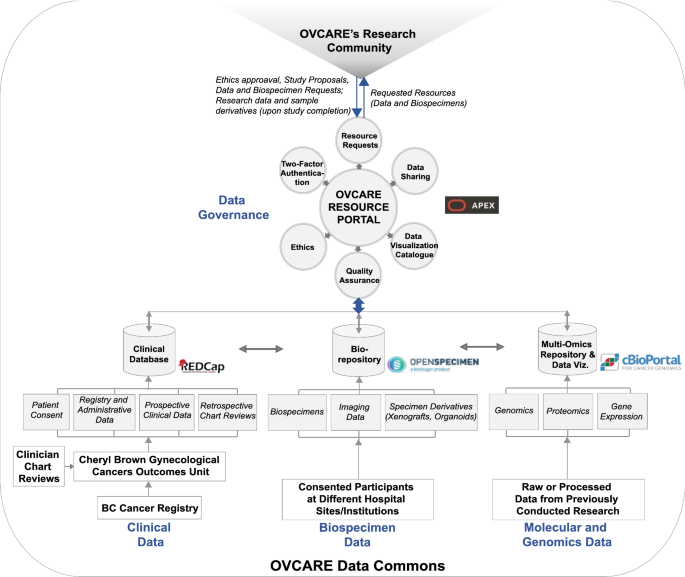
为满足前一节中描述的领域特定需求而确定的数据公共基础设施和软件的各种组件如图所示。2).这个基础设施是在机构防火墙后面实现的,只有资源门户可以通过万维网访问。实现这一基础设施的路径不是线性的,并且还在继续发展,尽管图中给出了线性的时间轴。3.).
OVCARE的数据公共基础设施和软件堆栈。整个数据共享基础设施由五个主要组件组成:(2)图书馆信息管理系统(opensample),该系统存储和管理从不同医院(即温哥华总医院、英属哥伦比亚大学医院、温哥华BC癌症中心和现在在BC省的其他几个中心)的同意参与者收集的生物标本。(3)支持对患者肿瘤样本的临床属性和分子谱进行探索、分析和可视化的cbiopportal,(4)基于规定的协议、标准操作程序和研究伦理管理数据和资源共享的OVCARE资源门户(ORP),以及(5)研究社区(包括OVCARE内部研究和信息学团队,以及OVCARE服务的更广泛的研究社区)。为满足我们的研究需求而确定的每个组件(REDCap、opensample、cBioPortal、ORP)分别托管在我们医院的计算环境中,并通过API调用以编程方式相互连接。来自不同领域的数据使用系统范围内的唯一标识符相互链接,将患者与其生物标本收集和分子/基因组学数据联系起来。为了访问积累的临床和生物标本收集,OVCARE研究社区中经过认证的研究人员将数据和样本采集请求发送到ORP,如果满足所有规定的要求,包括伦理批准,信息学人员将通过ORP满足这些请求。在成功获取数据和样本后,研究人员进行各自的研究,从他们的研究中生成的数据(原始的或加工的,和/生物标本衍生物)将返回OVCARE,使其可用于重新利用/二次使用。此外,返回到数据公域的分子数据被链接回可用和存储的患者生物标本。与临床结果一起,使用cBioPortal进一步探索、分析和可视化这些分子谱
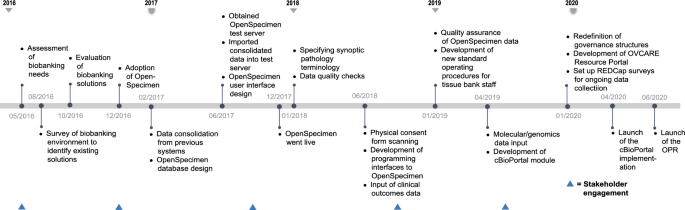
2017年初,我们完成了对现有生物银行解决方案的调查,以选择最适合我们当时需求的解决方案。2017年6月,获得了一个测试服务器,用于运行所选LIMS opensample的本地实例,以执行该软件所有组件的功能、集成和单元测试。这使我们能够直接评估opensample的特性,并确定在当前计算和研究环境中以最佳性能运行基础设施所需的资源。我们测试了性能,并评估了不同类型的用户(包括技术和非技术用户)执行日常生物银行活动的操作工作流。我们在2017年12月完全采用了opensample。在这次迁移之后,我们与研究人员一起收集了可用的基因组数据集,并将它们的可用性与opensample中各自的生物标本联系起来,并指出数据保存的位置。随着我们继续扩大这一资源,我们将增加与每个肿瘤块相关的病理幻灯片图像的可用性,并链接到它们。为了建立cBioportal集成的原型,我们收集了一种卵巢癌亚型的分子数据,这些数据来自之前的研究,这些研究与cBioportal的样本可用性和关键临床结果相结合,使用样本ID。我们最近推出了这个原型,目前正在评估中。
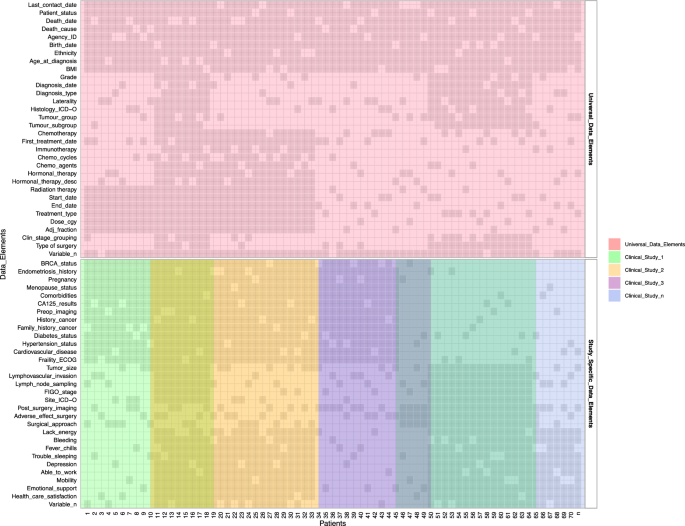
对于临床数据,我们扩大了Cheryl Brown Outcomes Unit的授权,将不列颠哥伦比亚省诊断的所有妇科癌症患者的临床和结局数据包括在内。我们还获得了伦理批准,可以永久保留本组所有临床研究的临床和结果数据。我们最大限度地利用了我们可以从行政来源(如BC癌症登记处)获得的数据,因为这为所有患者提供了临床数据的访问,并最大限度地减少了对广泛图表审查的需求(图2)。4).我们纳入了诊断日期、最后一次临床预约日期、生命统计数据、国际疾病分类(ICD -10)形态学代码、肿瘤分期和分级等要素。我们目前正在调查其他数据,如全身治疗(接受化疗和放疗)。临床数据整合的第二步包括将临床研究与图表回顾相结合。为了实现这一点,我们需要将不同的数据元素映射到唯一的概念。这进一步促进了我们小组研究人员最感兴趣的变量的识别。然后,我们为每个数据元素开发了一致的数据定义、标准和语义,以确保所有数据都可以集成到数据公有空间中。未来的数据收集将参考这些数据标准,以确保前瞻性的协调临床数据。
最后,为了管理所有数据资产和资源,我们开发了OVCARE资源门户(ORP)。该解决方案根据OVCARE用户的需求进行设计和定制,在APEX软件中实现,并于2020年6月推出。该门户网站有助于整合工作流程以及所有数据和资源请求,有助于确保适当的治理和符合协议、标准操作程序和研究伦理委员会的要求。
这些实现(REDCap、opensample和cBioPortal)分别托管在医院的研究IT网络上,仅供信息学人员访问。研究人员只能访问资源门户来提出请求。数据通过唯一的标识符进行集成,这些标识符在患者级别或标本级别上连接每个数据库的各个表。数据链接通过API调用以编程方式完成,以满足各种研究需求。
为了请求数据,研究人员在ORP上创建用户帐户,如果需要,将主要研究人员配置文件与他们的帐户关联。经过认证的研究人员可以提交有关需要资源的研究的信息(研究计划、伦理批准和研究要求)。然后将为进度跟踪创建的项目参考号发给研究人员,并将orp生成的电子邮件发送给信息学人员,通知他们新的研究计划。收到的建议随后被处理,并发送给从OVCARE社区中选择的审查人员委员会进行审查和批准,之后资源请求得到满足。研究人员将其研究结果的任何原始和处理过的数据,以及其研究产生的任何衍生品(细胞系、DNA提取物、类器官)返回到数据公域。
讨论
我们描述了实施数据共享以造福不列颠哥伦比亚省妇科癌症社区的过程。这一基础设施民主化了整个社区共享资源的获取,并将整个卑斯省妇科癌症界聚集在一起,为实现一个共同目标而努力:减少患有妇科恶性肿瘤的妇女的死亡和痛苦。为了保护我们的数据资产并使其效用最大化,我们创建了一个统一的基础设施,以及标准化的操作程序,以满足研究和道德规范的需求。在这一过程中开发的数据管理和信息学核心专业知识提高了数据收集的效率,以最大限度地提高数据的价值,并通过优化数据的二次使用来扩大研究资金。拟议的治理结构简化了要求,确保项目的科学完整性,并遵守患者特定数据的隐私、安全和道德披露。
通过调查,我们发现没有单一的解决方案可以满足所有不同的数据需求。相反,多个解决方案的集成可以帮助我们实现预期的结果。虽然用于实现当前基础设施的软件和技术堆栈将在不久的将来(5年)为我们服务,但数据存储和管理领域正在以非常快的速度发展,我们可能很快就需要重新评估我们的需求。在选择我们的软件堆栈时,我们需要平衡与开源和开放获取相关的风险,后者提供了负担得起的解决方案和更多的控制,但可用的支持很少,软件代码可能会停止维护,而与之相对的是使用提供更多技术支持和责任,但设置和维护可能非常昂贵的企业软件。为了缓解这一问题,我们尽可能地使用混合模型,并选择拥有活跃用户社区的软件,并支持一定程度的定制。
我们作为主要研究的一部分或出于管理目的收集的数据需要进行协调以进行整合。例如,一些数据源将“肿瘤等级”报告为“高或低”,而另一些数据源则报告数值等级:1,2,3,4;性别也可以用“M和F”、“1和0”或“1和2”来表示[44].这些数据的整合提出了“独特的技术、语义和伦理挑战”[45],也可能导致大量无法使用的数据,由于翻译损失。预先制定标准可以简化语义和本体,避免数据浪费,提高数据质量,并支持有效的数据集成、共享和可重用性,同时还节省了池化、处理和共享数据所需的大量时间和成本[44,46].未来与其他生物储存库和来自其他中心的类似数据库连接的努力依赖于采用标准化的本体来促进数据共享。还确定了确保数据质量和安全的政策,包括建立团队和用户角色以及数据访问级别;确保从数据获取到分发的所有过程都符合规定的政策和研究道德。
数据共享由三名主要研究人员监督,其中包括一名信息学家、一名医学肿瘤学家和一名妇科肿瘤学家。运营这个基础设施的团队包括一个兼职的数据库管理人员和一个从事各种数据集成的数据科学家。实验室技术人员和临床协调员在各种合作学生的帮助下促进标本采集、存储以及数据收集。偶尔需要与病理学和肿瘤学人员会诊。
我们的团队继续整理和协调可用数据,以最大化其效用。例如,在明年,我们将添加数字病理图像,并能够将我们收集的数据上传到数据飞地,在那里它可以与其他管理数据链接,包括卫生服务使用和处方药。这将导致一个非常丰富的数据生态系统,这将为新的科学发现提供成熟的条件,并可以实现前所未有的研究。
在不久的将来,我们将扩大我们的数据公域,使其更加以患者为中心。我们正在启动一个在线同意程序,以便我们能够接触到更广泛的患者群体,邀请他们参与研究。我们还将患者报告的结果(PRO)添加到数据共享中。
结论
与传统的生物储存库相比,将来自各种分布式系统、临床研究和研究机构的异构数据集和生物标本整合为数据公有物,为推动转化医学提供了重要机会。通过共享政策和技术,以及保护隐私的开放计算机架构和存储平台,可以实现临床和研究数据的无缝数据环境。
数据公地的成功和可持续性首先取决于培养一个能够使用开放和互联数据环境的科学社区。其次,需要有适合每种数据类型的适当技术解决方案;没有单一的解决方案可以适用于所有的数据收集,但多个解决方案应该集成在一起。最后,需要适当的治理结构,以应对跨机构和多学科研究、资源整合、数据共享和数据协调方面的独特挑战,以提高互操作性。
在本文中,我们介绍了开发和应用的方法,成功地建立了一个联合的、可扩展的基础设施,将OVCARE的传统肿瘤生物库、结果单元和数据筒仓集合扩展为一个集成的数据公有物。为此,我们收集并分析了参与机构在三个主要领域下的所有研究需求:(1)生物标本收集,(2)分子和基因组学数据,(3)临床数据,并确定、开发和实施了满足这些需求的解决方案。我们进一步建立了治理模式和资源门户,以实施协议和标准操作程序,支持所有参与机构的数据和生物材料聚合、共享、协调和治理。我们相信,这样的基础设施将有助于打破获取大型数据集的障碍,这些数据集是阐明和提高我们对复杂和罕见疾病的理解所必需的,为知识发现和转化为改善患者护理提供了强大的机会。
方法
需求评估
为了确定研究需要和收集基础设施需求,所有参与机构的利益相关者都参与了研究。与个别研究人员进行了讨论和一对一会议,并举行了头脑风暴会议,以确定未来5-10年的总体研究方向和要求。与机构研究及资讯科技人员进行进一步讨论,以了解保安、数据管理及可持续发展的需求。确定的方向和优先级扩展为需求列表(附加文件2:表S1)与生物标本、临床和分子/基因组学数据的收集和优化,以及由此产生的基础设施的治理模型有关。
技术解决方案
对于每个领域特定的需求(治理、生物标本、临床和分子/基因组学数据),都确定了技术解决方案来满足该领域下建立的需求。管理临床和分子/基因组学数据所需的解决方案(分别为REDCap和cBioPortal)之前已经得到了良好的建立、测试、实施和验证,以满足我们研究环境中这两个数据领域的需求。
为了确定满足所有/大部分已确定生物标本要求的LIMS解决方案,我们调查了生物库和LIMS环境(附加文件)1),并确定了我们比较评估的9个突出的软件解决方案。基于出版物和在线文档,我们收集并分析了所有已确定的生物银行软件的数据,并根据我们的要求检查了每个软件的特性和功能(附加文件)2:表S12)。我们还与各种软件供应商进行了会议、访谈和现场互动演示。每个已识别平台的功能列表(附加文件2表S2-S11)生成,我们的每个需求都被考虑,以确定最能满足我们需求的解决方案(附加文件2:表S12)。在第二次涉众会议上,我们讨论了确定的lim的适用性和实用性,并决定进一步评估opensample。
基于收集到的生物标本数据,我们定义了数据库概念(实体、属性、关系和约束),并定制了后端opensample数据库(运行MySQL)。我们获得了一个测试服务器(用Java和Apache Tomcat实现),并在我们的计算环境中安装了一个基于linux的opensample本地实例。在这些试验运行期间,经常向软件供应商询问特性、组件、集成和互操作性功能,包括识别缺失的需求。在成功测试之后,利用opensample的批处理上传实用程序将来自遗留系统的数据整合到服务器中。在将opensample投入生产之前,我们进一步设计和开发了用户界面,并根据我们独特的需求对其进行了配置和定制。
数据标准化和集成
现代转化医学的愿景在很大程度上取决于对患者的大规模临床和分子特征的整合,以得出假设和对患者疾病的新见解[45,47,48].OVCARE的数据来自多个不同的来源。为了整合来自多个数据库的数据,我们开始了严格的数据验证和质量控制检查。我们广泛审查了所有生物标本数据,包括:(1)检查、定位和上传所有物理同意书,以确保我们的数据库中有数字记录,(2)上传所有物理生物标本申请表格,(3)审查所有病理诊断(由具有妇科亚专科的病理学家进行),以及(4)定位和确认所有标本的可用性。将分子和基因组学数据集集成到opensample的过程需要与具有这些数据解释专业知识的研究人员密切合作。2019年初,我们从所有OVCARE研究人员那里获得并整合了之前收集的“组学”数据集。作为第一步,我们将组学数据映射回样本,并创建标记,表明它们在opensample患者档案中的可用性。该过程的第二步始于2020年4月,实施了用于数据可视化和分析的cBioPortal。
为了巩固临床数据,我们采用了两步方法,即使用所有患者的最小数据元素集,并辅以来自不同亚群患者的其他研究数据。我们评估了所有可从管理来源(如BC癌症登记处)获得的可用数据元素的准确性、一致性和完整性。我们选择了一组符合质量标准的数据元素。我们部署了一个管道,根据一组规则定期对数据元素执行质量检查,这些规则可以通过编程方式应用,以验证各个元素之间的完整性、一致性和逻辑,然后再进行集成。只有通过质量检查的数据才会被合并到永久的临床数据库中;未通过质量检查的数据将由数据管理员进一步调查,以确定错误的来源。来自BC省癌症登记处的临床结果数据在与REDCap托管的永久数据库合并之前被去标识,并每季度更新一次。
为了补充从注册中心获得的数据,我们流程的第二步涉及整合通过临床研究获得并保存在竖井中的临床数据。为了确保数据可以在研究中聚合、比较、分析、共享和重用,定义了数据标准以解决标准化差异[44].从七个临床研究中收集独特的数据变量,以了解我们临床数据库中数据的广度。我们创建了一个标准化的数据字典,目标是将数据元素映射到BC省所有临床数据集合中的相同数据概念,这些概念反过来可以与公共数据模型omp - cdm匹配[49]以最大限度地提高与外部数据集的互操作性。
数据治理、道德规范和标准操作程序
在对所有数据源进行标准化和聚合之后,我们开发了一个集中治理模型,并定义了协议、标准操作程序(sop)和政策,以管理OVCARE研究社区的数据访问、存储、保护、共享和允许使用。为了实现治理框架,我们设计、开发、测试和部署了OVCARE资源门户(ORP)。该门户网站使用Oracle APEX开发,为所有内部研究和合作团队提供在线界面,以请求包括生物标本、临床、分子、成像数据以及信息学和数据分析支持在内的资源。
缩写
- 人工智能:
-
人工智能
- 顶:
-
Oracle Application Express
- API:
-
应用编程接口
- CBGOU:
-
谢丽尔·布朗妇科癌症预后部门
- 必须:
-
副本编号更改
- GUI:
-
图形用户界面
- HL7:
-
健康等级7
- HTTP:
-
超文本传输协议
- ICD:
-
《国际疾病分类》
- LIMS:
-
实验室信息管理系统
- 信使rna:
-
信使核糖核酸
- MSK的:
-
纪念斯隆凯特琳癌症中心
- MySQL:
-
我的结构化查询语言
- 门店:
-
下一代测序
- OMOP-CDM:
-
观察性医疗结果伙伴关系-通用数据模型
- ORP:
-
OVCARE资源门户
- OVCARE:
-
卵巢癌研究计划
- 正方观点:
-
患者报告的结果
- 搬运工:
-
研究电子数据采集
- 其他:
-
具象状态转移
- 标准作业程式:
-
标准操作程序
- SQL:
-
结构化查询语言
- 目标:
-
肿瘤改变与基因组学驱动的治疗相关
- TCGA:
-
癌症基因组图谱
- TMA,
-
组织微阵列
参考文献
生物银行成熟:向生物标本科学的过渡。中国药理学杂志,2016;56(1):211-28。
沃特J,凯利A,休伊特R.回顾国际生物银行和网络:成功因素和关键基准。生物储备生物库。2009;7(3):143-50。
人体组织来源手册:人体组织样本的国家资源。圣莫尼卡:兰德;1999.p。251。
科波拉L,希安弗洛内A,格里马尔迪AM,因科罗纳托M,贝维拉夸P,梅西纳F,等。卫生保健中的生物银行:演变和未来方向。中华医学杂志2019;17(1):172。
格林伯格B,克里斯蒂安J,亨利LM,利维M,摩尔H.生物储存库。评估患者结果的注册表附录:用户指南,第三版。罗克维尔(马里兰州):医疗保健研究和质量机构(美国);2018.(AHRQ有效保健方法)。http://www.ncbi.nlm.nih.gov/books/NBK493632/.2021年6月22日访问。
Cortes A, Albers PK, Dendrou CA, Fugger L, McVean G.在英国生物银行的医院数据中识别遗传风险的跨疾病成分。植物学报。2020;52(1):126-34。
Harris JR, Burton P, Knoppers BM, Lindpaintner K, Bledsoe M, Brookes AJ,等。朝着全球健康生物银行路线图迈进。《植物学报》,2012;20(11):1105-11。
Cole JB, Florez JC, Hirschhorn JN。英国生物银行对饮食习惯的全面基因组分析确定了数百种遗传关联。自然科学进展。2020;11(1):1467。
Collins FS, Varmus H.精准医疗的新举措。中华实用医学杂志,2015;29(9):795 - 95。
刘a, Pollard K.生物银行用于个性化医疗。入:Karimi-Busheri F,编辑。21世纪的生物银行。湛:施普林格国际出版;2015.55 - 68页。
德·索萨YG,格林斯潘JS。生物银行的过去、现在和未来:责任和利益。艾滋病。2013; 27(3): 303 - 12所示。
王勇,王志强,王志强。人工智能在神经发育障碍中的应用。中华数字医学杂志2019;2(1):112。
生物银行正在改变世界。2019.https://www.forbes.com/sites/cognitiveworld/2019/08/12/biobanking-is-changing-the-world/?sh=6cf563943792.进入2020年8月16日。
李J-E。未来生物库中的人工智能:生物库中的当前问题和人工智能未来的可能性。生物医学学报,2018;7:3。
kihntopf M., Krawczak M.生物银行与国际互操作性:样本。胡文杰,2011;130(3):369-76。
格罗斯曼RL,希斯A,墨菲M,帕特森M,威尔斯W.数据公共案例:数据科学作为一种服务。计算科学与工程,2016;18(5):10-20。
Jensen MA, Ferretti V, Grossman RL, Staudt LM。NCI基因组数据共享作为精准医疗的引擎。血。2017;130(4):453 - 9。
Hinkson IV, Davidsen TM, Klemm JD, Chandramouliswaran I, Kerlavage AR, Kibbe WA。癌症研究大数据的综合基础设施:加速癌症研究和精准医疗。前沿细胞发展生物学。2017;5:83。
Köbel M, Rahimi K, Rambau PF, Naugler C, Le Page C, Meunier L,等。卵巢癌的免疫组织化学分型算法。中华妇产科病理学杂志,2016;35(5):430-41。
Shah SP, Köbel M, Senz J, Morin RD, Clarke BA, Wiegand KC,等。突变的FOXL2卵巢颗粒细胞瘤。中华实用医学杂志,2009;26(2):319 - 319。
Wiegand KC,沙SP, Al-Agha OM,赵Y,谢霆锋K,曾T, et al。ARID1A子宫内膜异位症相关卵巢癌的突变。中华实用医学杂志,2010;29(4):344 - 344。
Errico A. SMARCA4在SCCOHT中发生突变。中华儿科杂志。2014;11(6):302-302。
王玉凯,Bashashati A, Anglesio MS, Cochrane DR, Grewal DS, Ha G,等。异常DNA修复机制的基因组后果使卵巢癌组织类型分层。植物学报,2017;49(6):856-65。
杨伟,杨文杰,杨文杰,杨文杰,等。ProMisE的确认:一个简单的,基于基因组学的子宫内膜癌临床分类器:EC的分子分类。癌症。2017;123(5):802 - 13所示。
杨伟,周晨,杨伟强,等。分子分类背景下子宫内膜癌预后免疫组化标记物的评估:新后tcga时代子宫内膜癌的额外IHC生物标记物。中华流行病学杂志,2017;3(4):279-93。
李志强,李志强,李志强,等。诊断标本上子宫内膜癌的分子分类与最终子宫切除术高度一致:早期预后信息指导治疗。妇科肿瘤学杂志2016;143(1):46-53。
张志刚,张志刚,张志刚,张志刚,等。人乳头瘤病毒(HPV)非依赖性外阴鳞状细胞癌比HPV相关疾病预后更差:一项回顾性队列研究组织病理学。2017;71(2):238 - 46所示。
Krishagni解决方案。OpenSpecimen。2012.https://www.openspecimen.org.2016年8月17日访问。
McIntosh LD, Sharma MK, Mulvihill D, Gupta S, Juehne A, George B等。caTissue suite to opensample:开发一个可扩展的,开源的,基于web的生物银行管理系统。中国生物医学杂志,2015;
Krishagni解决方案。opensample生物银行LIMS功能。2012.https://www.openspecimen.org/biobanking-lims-features/.2016年8月17日访问。
张志强,张志强,张志强,张志强,等。cBio癌症基因组学门户:探索多维癌症基因组学数据的开放平台。癌症杂志,2012;2(5):401-4。
癌症基因组图谱研究网络。卵巢癌的综合基因组分析。大自然。2011;474(7353):609 - 15所示。
Jonckheere N, Van Seuningen I.癌症基因组图谱和癌细胞系百科全书大规模基因组数据库的综合分析:MUC4/MUC16/MUC20特征与人类癌症的低生存率有关。中华医学杂志,2018;16(1):259。
崔旭,景霞,易强,龙成,谭波,李旭,等。STAT3在癌症中的基因表达改变和临床结果的系统分析。Oncotarget。2018;9(3):3198 - 213。
癌症基因组图谱网络。人类乳腺肿瘤的综合分子图谱。自然。2012;490(7418):61 - 70。
长泽S,池田K,堀-井上K,佐藤S,武田S,长谷川K,等。从日本上皮性卵巢癌患者的rna测序数据鉴定卵巢癌相关基因的新突变。中国生物医学工程学报(英文版);2011;
高娟,王志强,王志强,王志强,等。使用cBioPortal对复杂癌症基因组学和临床资料进行综合分析。科学通报。2013;6(269):pl1。
2005年癌症基因组图谱计划。https://www.cancer.gov/about-nci/organization/ccg/research/structural-genomics/tcga.2021年4月12日访问。
国家癌症研究所,癌症基因组学办公室(OCG)。目标:产生有效治疗的治疗应用研究。2006。https://ocg.cancer.gov/programs/target.2021年4月15日访问。
哈里斯PA,泰勒R,希尔克R,佩恩J,冈萨雷斯N,康德JG。研究电子数据捕获(REDCap) -提供转化研究信息学支持的元数据驱动方法和工作流过程。中华生物医学杂志,2009;42(2):377-81。
哈里斯PA,泰勒R,小BL,埃利奥特V,费尔南德斯M,奥尼尔L,等。REDCap联盟:打造软件平台合作伙伴的国际社区。中国生物医学杂志。2019;95:103208。
搬运工。研究电子数据捕获(REDCap)。2004.https://www.project-redcap.org.2021年4月15日访问。
Oracle。甲骨文Apex 2019https://apex.oracle.com/en/.访问14 april 2021。
华盛顿(特区)。共享临床研究数据:研讨会总结。华盛顿(DC):美国国家科学院出版社;2013.https://www.ncbi.nlm.nih.gov/books/NBK137818/.2021年6月26日访问。
Seneviratne MG, Kahn MG, Hernandez-Boussard T.合并异质临床数据以实现知识发现。生物计算。2019;24:439-43。
Huser V, sstry C, Breymaier M, Idriss A,西米诺JJ。临床研究方案和病例报告表的标准化数据交换:临床数据交换标准联盟(CDISC)操作数据模型(ODM)适用性的评估。中国生物医学工程学报,2015;
De Maria MR, Di Sante G, Piro G, Carbone C, Tortora G, Boldrini L,等。精准医疗时代的转化研究:我们在哪里,我们将去哪里。中华儿科杂志2021;11(3):216。
田强,Price ND, Hood L.系统癌症医学:实现预测、预防、个性化和参与性医学(P4):系统癌症医学中华临床医学杂志(英文版),2012;
观察健康数据科学与信息学(OHDSI)。公共数据模型。https://www.ohdsi.org/data-standardization/the-common-data-model/.2021年7月2日访问。
确认
作者对所有捐赠样本用于研究的女性表示深深的感谢。没有他们的慷慨,妇科癌症研究和护理的进步是不可能的。作者要特别感谢Jane & Maurice Wong和Gray Family的远见卓瞩,他们为数据公地的工作提供了资金,这对研究人员来说已经并将继续是一个巨大的资源。作者还感谢来自BC癌症基金会、VGH和UBC医院基金会、英属哥伦比亚大学和加拿大卵巢癌(OVCARE, BC妇科癌症研究团队)的资助。
资金
这项工作由Jane & Maurice Wong和Gray Family的捐赠资助。此外,还获得了BC癌症基金会、VGH和UBC医院基金会、英属哥伦比亚大学和加拿大卵巢癌(给OVCARE, BC省妇科癌症研究团队)的资助。
作者信息
作者及隶属关系
贡献
AT与MW和SL合作,将OVCARE转变为一个概念数据共享与卫生署、JNM及AT协商.RA与SL和AT合作进行了LIMS调查、分析和结果解释。数据标准化和集成由SL、SW、SL和RW在AT的监督下进行。SL、SL、RW、MW和AT在与DH、JNM和AT协商后,建立了数据共享治理模型、政策和标准操作程序。数据解决方案的设计、测试和实施由SL、RA和SL在AT的监督下进行。稿件组成和起草由RA完成,AT与SL, SL, RW, SW共同完成。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
阿思威、林绍生、梁绍生。et al。从生物库和数据仓库到数据共享:聚合以支持转化医学。翻译医学杂志19, 493(2021)。https://doi.org/10.1186/s12967-021-03147-z
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12967-021-03147-z
关键字
- 起生物
- Biospecimens
- Biobank-technologies
- 精密医学
- 数据共享
- 实验室信息管理系统(LIMS)
- 联邦系统
- 数据治理