摘要
背景
复杂激酶重排是一种涉及一条或两条染色体的突变过程,具有聚集重排断点,干扰基于dna的下一代测序(NGS)对激酶融合的准确检测。我们研究了络合物的特性碱性多分子检测在非小细胞肺癌中的重排
方法
非小细胞肺癌患者样本通过靶向捕获dna - NGS分析,探针定位融合伙伴基因的内含子区,rna - NGS, RT-PCR,免疫组化(IHC)和荧光原位杂交(FISH)。
结果
在6576名非小细胞肺癌患者的大队列中,343例(5.2%)病例为隐性肺癌碱性重新安排已确定。14例复杂碱性基于dna的NGS对重排进行了鉴定,并结合多种基因组特征将重排分为基因间重排(n = 3)、基因内重排(n = 5)和“桥关节”重排(n = 6)三种类型。所有13例有足够样本的病例均表现为典型针对有eml4 - alk基于rna的NGS证实融合转录本。13例中13例ALK IHC阳性,11例中9例FISH阳性。复杂的患者碱性重新安排谁收到碱性抑制剂治疗(n = 6),与常规治疗患者相比,无进展生存期(PFS)无差异碱性融合n = 36,P= 0.9291)。
结论
本研究首次揭示了复合物的分子特征和临床疗效碱性NSCLC中的重排,对ALK抑制剂治疗敏感,并强调了利用探针在基于dna的NGS中选择融合伙伴基因的内含子区域进行精确融合检测的重要性。RNA和蛋白质水平检测可能是验证复合物功能的关键碱性临床实践中的重新安排以获得最佳治疗决策。
背景
间变性淋巴瘤激酶的重排(碱性基因在大约3-7%的非小细胞肺癌(NSCLC)患者中被鉴定,棘皮类微管相关蛋白样4 (EML4),代表最常见的融合伙伴[1,2].碱性-重排NSCLC定义了一个独特的分子亚群,对碱性酪氨酸激酶抑制剂(TKIs)克唑替尼,耐受良好的第一代药物碱性抑制剂(3.,4],已获美国食品和药物管理局批准用于治疗碱性-在2011年对NSCLC进行了重新安排。第二代碱性抑制剂,如艾乐替尼和塞瑞替尼,不仅对克唑替尼初治患者有效[5],但也适用于获得性对克唑替尼耐药的患者[6,7,8,9].识别碱性重新安排并批准了多项碱性TKIs已经彻底改变了患者隐藏的治疗方法碱性融合。因此,准确检测为碱性重新安排至关重要。
精确肿瘤学面临的一个挑战是识别新颖或复杂的易位。传统的方法,包括荧光原位杂交(FISH)和免疫组化(IHC),有局限性,如FISH不允许鉴定碱性伴侣基因或非典型断点,ALK IHC原则上可能因过度表达而混淆碱性而不是真正的融合蛋白10].而新一代测序(NGS)技术可以有效、准确地检测已知和新型的致癌融合物,并已广泛应用于临床诊断。
复杂激酶重排,在这里指的是一个突变过程涉及一个或两个染色体聚集重排断点。最近的研究表明,在非吸烟者的肺腺癌中,复杂的基因组重排产生了74%的已知融合癌基因,包括针对有eml4 - alk,CD74-ROS1,KIF5B-RET[11].然而,在基于dna的NGS检测中,复杂的基因组重排常常阻碍正确的捕获[12].越来越多的证据表明,由于基因组的复杂性,DNA测序确定的基因组断点是转录水平断点的不可靠预测器[13,14].复杂激酶重排的鉴定和临床功能验证仍然难以捉摸,这使得肿瘤学家在选择合适的治疗方法时感到困惑。基于dna的NGS技术和基于rna的NGS技术的组合方法学为探索癌症基因组的突变过程提供了独特的机会。尽管已经有了具有里程碑意义的研究来描述复杂的基因间断点融合[14],在很大程度上是基于外显子组和选择的内含子碱性基因。目前还缺乏使用基于dna的NGS的研究,这些NGS设计用于来自已知可能包含基因组断点的融合伙伴基因的内含子区域。
在本文中,我们利用专门设计的基于dna的NGS面板,使用多个探针来研究融合伙伴基因的内含子区域,并鉴定出三种类型的复合物碱性14例NSCLC患者的重排。通过RNA或蛋白质检测进行的进一步功能验证阐明了DNA和RNA为基础的NGS对激酶融合的全面检测和指导最佳治疗决策的重要性。
方法
患者和样本
收集了2018年1月至2020年7月6576例NSCLC患者的样本进行分子检测。病理和临床资料来源于临床记录。该研究得到了郑州大学第一附属医院机构审查委员会的批准。所有患者均为这些基因组分析提供了知情的书面同意。
DNA / RNA提取
常规苏木精、伊红染色玻片确认病理诊断,将相应的肿瘤细胞含量不低于20%的最优块转送进行DNA/RNA提取。根据制造商说明,使用AllPrep DNA/RNA FFPE试剂盒(Qiagen, USA)从福尔马林固定石蜡包埋(FFPE)肿瘤组织样本中提取基因组DNA (gDNA)和总RNA。作为对照,使用MagPure blood DNA DA Kit (Magen, China)根据制造商的说明从白细胞样本中提取gDNA。纯化的DNA/RNA质量用凝胶电泳检测,用Qubit®4.0荧光仪(Life Technologies, USA)定量。提取的DNA量超过30 ng被认为足以进行分析。在提取的FFPE RNA样品中,28S和18S rRNA条带被降解,≥200 ng的RNA具有较高的分析灵敏度。
基于dna的门店
将纯化后的gDNA先用酶法(5X WGS Fragmentation Mix, Qiagen, USA)裂解成约300 bp的DNA片段,进行末端修复、T-adaptors结扎、PCR扩增,得到预文库。一个内部设计的面板,针对86个癌症相关基因中的大多数外显子和选定的内含子,用于捕获DNA片段,以检测SNV/Indel,拷贝数变异和基因融合(附加文件)1:表S1)。特别是,混合捕获探针耕耘的内含子区域碱性(基因内区18日至19日),EML4(内含子6,13,20)和KIF5B(内含子15 - 16,24)设计用于检测碱性重组事件。PCR扩增后生成测序文库,在NovaSeq 6000平台(Illumina, San Diego, USA)上以150PE模式测序。
使用BWA对人类参考基因组hg19进行初始读图和比对处理[15].SAMtools [16]和基因组分析工具包GATK 3.8 [17]被用来调用snv和小的indel变体。大的插入和染色体重排(包括碱性重新排列)使用Fusionmap进行分析[18].保留从患者数据库中收集的癌症热点区VAF > 0.5%或VAF > 0.1%的非同义snv作进一步分析。确定并报告覆盖≥300和支持突变reads数≥3的融合。对于基因间区域的断点,每个方向上最近的基因被报告为预测的融合伙伴。
RNA-based挥动
基于混合捕获测序(Berry Oncology Corporation)的内部设计的RNA融合面板用于检测基因融合,该面板查找癌症中常见融合基因的所有编码外显子,并允许检测已知和新的融合,而不受融合伙伴或断点的限制。简单地说,纯化的总RNA首先通过逆转录反应转化为互补DNA(互补DNA, cDNA)。预文库构建包括末端修复、接头结扎和PCR扩增,其中总数量优化为≥600 ng。后续的杂交捕获文库在NovaSeq 6000平台(Illumina, San Diego, USA)上测序,成对端150 bp。基于Fusionmap软件,称为基因融合[18].通过人工检查断点来验证生物信息识别的融合。
鱼
简而言之,FFPE肿瘤组织样本使用Vysis LSI ALK双色、分离重排探针(Abbott/Vysis, Abbott Park, IL, USA)进行FISH分析。在每个样本的50个被评分的肿瘤细胞中,如果超过15%的被评分的肿瘤细胞分裂了一个或两个ALK 5 '和3 '探针信号或分离了3 '信号,则该样本被认为是FISH阳性。每一张FISH切片都由两位病理学家独立评估。
包含IHC
ALK蛋白的免疫组化在全自动Ventana Benchmark XT染色机上进行(Ventana医疗系统,罗氏集团,亚利桑那州图森)。使用预稀释的Ventana抗alk (D5F3)兔单克隆一抗和匹配的兔单克隆阴性对照Ig抗体,以及Optiview DAB IHC检测试剂盒和Optiview Amplification试剂盒对FFPE肿瘤样本进行染色。每一张免疫组化切片由两位病理学家独立评估。用ALK免疫组化法标记的肿瘤细胞根据先前描述的方法评估DAB信号的存在与否[19].任何肿瘤细胞以任何百分比呈强颗粒状细胞质染色均为ALK阳性,而肿瘤细胞无强颗粒状细胞质染色则为ALK阴性。
临床反应评价及统计分析
对于接受靶向治疗的一小部分病人碱性根据实体肿瘤反应评价标准(RECIST) 1.1版,基于CT成像评估临床反应。采用卡方检验研究患者特征与临床病理因素的相关性。采用Kaplan-Meier法计算无进展生存期(PFS),采用log-rank检验计算变量差异。一个双边P< 0.05为差异有统计学意义。使用GraphPad Prism(版本7.04)进行统计分析。
结果
患者特点及碱性重组
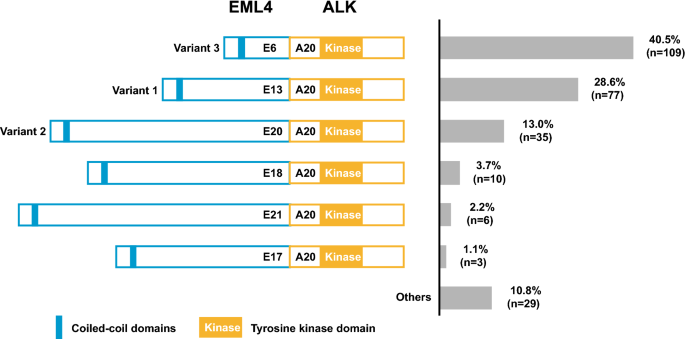
2018年1月至2020年7月期间,所有6576份来自NSCLC患者的样本都使用基于dna的NGS进行了分析。患者的临床特征见表1.碱性343例(5.2%)患者发生融合,女性、< 60岁或腺癌患者发生率较高。规范针对有eml4 - alk融合最常见,占78.4%(269/343)。大多数的基因组断点碱性在内含子19中检测到基因,而在内含子19中检测到基因EML4潜在的断点不同,可能在基因组水平上产生各种融合蛋白变体。如图所示。1,针对有eml4 - alk变异3 (E6:A20, 109/269, 40.5%)是最主要的类型,其次是变异1 (E13:A20, 77/269, 28.6%)和变异2 (E20:A20, 35/269, 13.0%)。
复合物的鉴定和验证碱性重组
343人中碱性融合案例,复杂碱性使用基于dna的NGS在86个癌症相关基因面板上靶向鉴定了14个病例的重排,并使用多个探针定位融合伙伴基因的内含子区域(表2)2).这些病例通过整合多种基因组特征可分为三种类型,包括基因间(n = 3)、基因内(n = 5)和“桥关节”重排(n = 6)。保留了足够样本的13个病例的子集被验证为额外的基于rna的NGS,用于所有常见融合基因的编码外显子。令人惊讶的是,我们发现融合基因和断点位置在DNA测序和RNA测序之间存在显著差异。所有13个病例实际上都表达了规范针对有eml4 - alk融合转录本。13例中13例ALK IHC阳性,11例中9例FISH阳性。
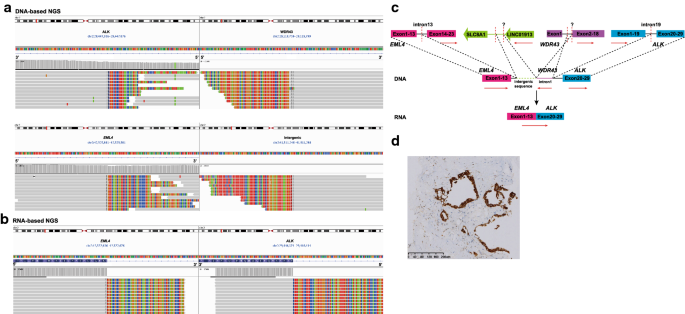
案例1为典型的基因间复合体重排案例WDR43-ALK(3 ' intron1: 3 ' intron19)和EML4-intergenic基于dna的NGS识别的融合(图;2A), ALK IHC法检测阳性结果(图。2D),但基于rna的NGS检测到规范针对有eml4 - alk融合转录本加入EML4外显子13到碱性外显子20(图;2B).测序数据表明,基因间复合体重排涉及多个融合连接,包括EML4,LINC01913上游基因间区,WDR43而且碱性(无花果。2C).基于dna的NGS也在病例2和病例3中检测到这种基因间复合体重排的证据,其中包含一个典型针对有eml4 - alk经RNA测序鉴定为变体3 (E6:A20), IHC和FISH检测均为阳性(表2)2).
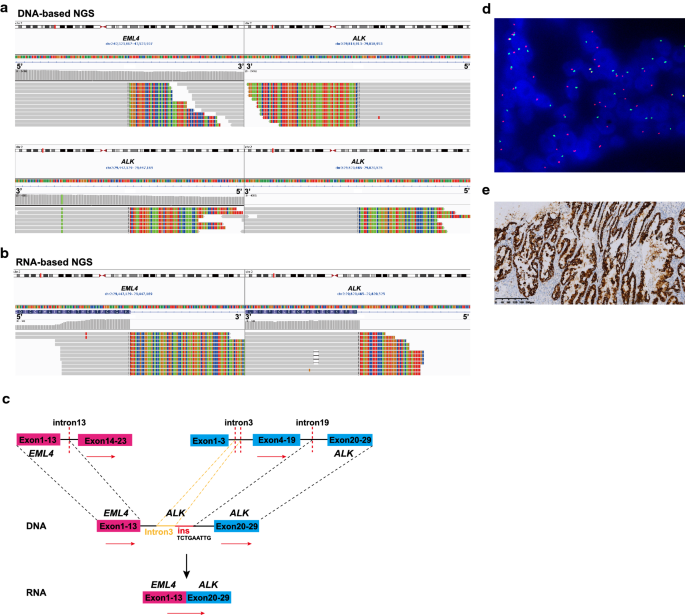
病例4-8中更为显著的观察是罕见而复杂的基因内重排碱性或EML4在DNA水平上鉴定的基因。案例4通常包含多个不同的重排,涉及碱性轨迹,由5 '组成EML4(内含子13)和3 '碱性(内含子3)融合,ALK-ALK内含子3的融合碱性与内含子19相连碱性通过9-bp插入,GALM -3 'EML4融合和5 'ALK-intergenic融合(图。3.A).只有前两个连接的融合-癌基因相关重排似乎能够产生功能性的致病融合转录本连接EML4外显子13到碱性基于rna的NGS数据检测外显子20(图;3.B和C)。另外两个没有转录产物的融合可能是互反融合。同时,清晰的分裂信号碱性使用分离探针试剂盒进行FISH检测(图;3.D),手术切除样本的IHC检测结果为阳性(图。3.E).同理,case 6 harbor 5 'EML4(内含子6)和3 '碱性(内含子4)融合39bp插入和ALK-ALK内含子4的融合碱性与内含子19相连碱性带有57-bp的插入,表示产品规范针对有eml4 - alk变体3 (E6:A20)转录本没有足够的标本进行验证试验。在情形5中,一个特殊的反转碱性检测到内含子18到内含子19的基因,其中3 '内含子18碱性与3 '内含子19相连碱性19的5 '内含子碱性连接5 '内含子6的EML4基于rna的NGS检测到规范针对有eml4 - alk变体3 (E6:A20)转录本。类似地,的逆序EML4在病例7和病例8中鉴定出内含子6,它们也藏匿着正典针对有eml4 - alk变体3 (E6:A20)转录,IHC和FISH阳性2).
基因内复合体重排产生的典型例子针对有eml4 - alk病例4的融合记录。一个DNA测序显示EML4(intron13) -碱性(intron3)融合和碱性(intron19)碱性(intron3)融合区通过IGV软件进行可视化。BRNA测序结果表明针对有eml4 - alk融合区由IGV软件可视化。C涉及DNA和RNA水平融合断点的基因组重排的可能示意图。基于dna的NGS检测到内含子13EML4与内含子3融合碱性的内含子3碱性与内含子19相连碱性插入9-bp。基于rna的NGS检测到规范融合转录本连接EML4外显子13到碱性外显子20。红色虚线表示融合断点,红色箭头表示转录方向。D积极的碱性FISH模式,红色和绿色信号分开。E免疫组化法检测ALK阳性
在病例9-13中,发现了多个基因融合,在此定义为“桥关节”重排,因为两者都是EML4而且碱性在基因组水平上分别与一个相同基因连接。以案例9为例,DNA-based NGS检测到的内含子13EML4的内含子1的下游区域融合LCLAT1的内含子1的上游区域LCLAT1的内含子19相连碱性(无花果。4由于内含子剪接,基于rna的NGS识别规范是合理的针对有eml4 - alk的变体1 (E13:A20)转录本LCLAT1(无花果。4B和C)。免疫组化检测显示ALK蛋白表达明显阳性,但FISH显示阴性结果,可能是由于分离探针设计或技术方面产生假阴性结果的风险(图。4D和E) [20.].类似地,在病例10-13中,基于dna的NGS揭示了EML4融合伙伴基因首先连接到一个新的“桥”基因的内含子区,然后连接到的内含子碱性激酶基因,如“桥关节”复合体重排。大多数新“桥”基因的内含子区域被剪接去除,导致正则针对有eml4 - alk记录。特别是“桥”基因的外显子6 (RUNX1)参与了复杂的重新排列和RUNX1-ALK(exon6: exon20)转录本在病例12中检测到,这可能是该病毒的一部分EML4-RUNX1-ALK(exon5: exon6: exon20)转录本难以识别。此外,EML4 -碱性(exon5: exon20)转录本在病例12中也被检测到,可能是由于替代剪接。有趣的是,案例14,窝藏EML4-RPIA而且MAP4K3-ALK融合,被认为是典型的针对有eml4 - alk变体1 (E13:A20)的转录本,表明RPIA而且MAP4K3都是“桥”基因和它们的内含子区域连接在一起(表2).
靶向治疗与复杂的临床结局碱性重组
14例复杂病例中碱性重排,仅有8例患者接受靶向治疗碱性抑制剂(克唑替尼或艾乐替尼)治疗,包括2个基因间复合体重排,3个基因内复合体重排和3个“桥接头”重排。表中概述了RECIST v1.1定义的治疗和治疗反应3.结果显示,6例患者(75%)达到临床客观缓解,其中5例部分缓解(PR), 1例完全缓解(CR)。
基因间复合体重排的病例1和病例3均对克唑替尼有阳性反应,但仍未达到无进展生存(PFS)终点,分别持续至少8和7个月(表2)3.).微分碱性在基因内重排变异中观察到抑制剂反应碱性-阳性肺腺癌(病例5、病例6、病例8)针对有eml4 - alk融合5号和8号病例,都有转录连接EML4外显子6到碱性外显子20和FISH和IHC阳性结果,对克唑替尼、CR(病例5)和进展性疾病(病例8)治疗5个月后的临床结果有很大差异。我们推测另一种变体发生在情况8中,TP53p.R273C突变,增强癌细胞增殖、侵袭及耐药[21].至于“桥关节”重排,三例中的一例,病例14,在克唑替尼治疗4周后表现出稳定的疾病(SD)(表3.),与其他2例患者的PR状态不同,提示临床效果较差碱性某些患者的抑制剂可能是由对靶向治疗的原发性药物耐药引起的[22].
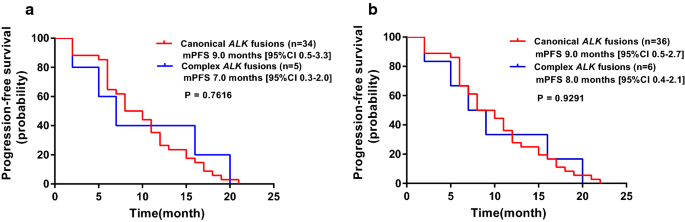
复杂的患者碱性接受克唑替尼治疗的融合患者(n = 5)表现出相当的中位无进展生存期(mPFS)碱性融合(n = 34),如图。5A的值为7.0个月(95% CI 0.3-2.0),而9.0个月(95% CI 0.5-3.3), P值为0.7616。同样,在复合体和正典之间,mPFS没有显著差异碱性同时分析阿莱克替尼和克唑替尼治疗患者的融合情况(8.0个月[95% CI 0.4-2.1]和9.0个月[95% CI 0.5-2.7], P = 0.9291,图2。5B)。
讨论
在这项研究中,我们确定了三种类型的复合物碱性重排、基因间复合体重排、基因内复合体重排和“桥接头”重排。复杂的碱性重排可归因于一种被称为嗜铬症的独特机制,在所有癌症中至少发生2%至3%,并经常促进多种肿瘤的肿瘤发生[23].数据2,3.而且4显示了一种可能由反转和嗜铬作用介导的突变过程。它引起了一次性染色体断裂和随后染色体片段的随机重组,从而导致碱性而且EML4分别连接到基因间/基因内/“桥连接”区域,这些区域在转录过程中被移除并产生正则针对有eml4 - alk致癌融合。碱性分离FISH分析显示,肿瘤细胞中有更多的异常染色体2片段和分散,可能是由于嗜色症导致染色体结构严重破坏(图2)。2D)除嗜色菌病外,易位被报道为一种新的基因内机制碱性2014年成神经细胞瘤肿瘤重排情况[24].最近的另一项研究表明,基因内复合体重排与RB1失活的表皮生长因子受体-突变肺癌细胞[25].我们在肺腺癌中检出5例基因内复合体重排,其中3例为基因内复合体重排碱性重排和2个基因内EML4重排,所有这些都产生了规范针对有eml4 - alk融合转录本中除一份因样本不足未进行RNA-based NGS检测外。
NGS技术已广泛应用于基于dna的重排检测,成为美国等高收入国家的一线病理方法。基于dna的NGS的采样要求和质量指标不像基于rna的方法那么严格。在我们的研究中,有一个病例没有成功进行基于RNA的NGS,因为样本不能满足更严格的RNA质量标准。基于dna的NGS可以识别基因组重排,不限于融合,如扩增碱性基因座,揭示了一种新的截断形式并激活驱动程序,但不导致融合转录物和蛋白质[26].基于靶向捕获dna的NGS面板通常用于靶向激酶基因的外显子和精选的内含子区域,这些区域有很高的概率包含基因组断点,可以有效地识别激酶融合。然而,当靶向捕获内含子过长、包含重复元素或涉及复杂的基因组事件时,基于dna的NGS存在一些固有的局限性[27].当肿瘤基因融合是由一个或多个复杂的DNA重排引起时,基于DNA的NGS板不能完全捕获基因组重排。相比之下,基于RNA的NGS提供了一种更直接的方法来检测临床可行的融合,因为RNA测序专注于剪接后的外显子,这可能会绕过基因组的复杂性[27].基于rna的NGS检测是目前最全面、最有效的精确融合转录本检测策略,广泛应用于基因融合的分子诊断[28].在我们的同龄人中,很复杂碱性重排表达规范针对有eml4 - alk基于DNA和rna的NGS检测到13例融合转录本。的内含子区域内的基因组断点EML4参与了这个综合体吗碱性在没有探针捕获的情况下,普通NGS面板很难检测到重排EML4内含子。使用优化的探针耕耘所选择的内含子区域EML4的内含子区域内的基因组断点EML4被基于dna的NGS清晰地检测到,并阐明了整个复合物的可能结构碱性重组。我们的发现表明,利用基于dna的NGS和优化的探针挖掘融合伙伴的内含子区域,然后利用基于rna的NGS可能是至关重要的,这可以有效地识别准确的致癌重排,并全面指导最佳治疗决策,不仅在肺癌中,而且在不同类型的肿瘤中。此外,有2例患者样本(病例9和病例12)在FISH和其他检测之间的结果不一致。多种分子检测之间的差异可以被认为是肿瘤学家的一个“警钟”,通过识别和功能验证临床相关的复杂基因组重排来确保更准确的分子诊断。
克唑替尼,口服小分子酪氨酸激酶抑制剂(TKI)靶向碱性,见过,ROS1酪氨酸激酶,已经被批准碱性-在美国、欧盟、中国和其他国家的NSCLC重排,客观缓解率(ORR)约为60.8%,中位无进展生存期(mPFS)为9.7个月[29].除克唑替尼外,还开发了多种第二代ALK-TKIs(如alectinib和ceritinib)用于糖尿病患者碱性-阳性NSCLC,均比克唑替尼效力高[30.,31,32,33].尽管ALK-TKI已经极大地扩展了碱性在非小细胞肺癌中,存在复杂基因组重排的患者是否能从这种靶向治疗中获益的实质性问题尚未完全确定。Kodama等人证实,艾勒替尼和克唑替尼均有效针对有eml4 - alk在患者来源的细胞系中,通过嗜铬症介导的-阳性肿瘤,并且艾乐替尼的效力大约比克唑替尼高13倍[23].在我们随访的临床资料中,有8例患者同时存在复合体碱性重新排列显示最佳反应为1个CR(阿莱克替尼治疗)、5个PR(1个阿莱克替尼治疗和4个克唑替尼治疗)、1个SD(克唑替尼治疗)和1个PD(克唑替尼治疗)。看来阿莱克替尼对complex的反应更为显著碱性重排比克唑替尼在这个“真实世界”的数据集中。然而,未来还需要进行更多的研究,以更大的队列验证结果。
有趣的是,病例8与病例5相比,克唑替尼的临床结果有很大差异,病例8在治疗5个月后出现PD,病例5出现CR。两例患者均有相同的罕见和复杂的基因内病变针对有eml4 - alk基于DNA和rna的NGS检测到重排,FISH和IHC阳性。结果不佳的可能原因可能是TP53基于dna的NGS中检测到p.R273C突变,有报道称NGS可增强癌细胞增殖、侵袭和耐药性[21].这是合理的,在mPFS之间没有发现明显的差异携带复杂和规范碱性融合,无论他们的一线治疗,克唑替尼或阿勒替尼,因为他们都产生典型针对有eml4 - alkRNA水平的转录。本研究生存分析的局限性包括合并并发症的病例数碱性接受靶向治疗的融合相对较小(附加文件1).
结论
本研究首次揭示了复合物的分子特征和临床疗效碱性NSCLC中的重排,对ALK抑制剂治疗敏感,并强调了优化NGS面板探针设计对筛选融合伙伴基因内含子区域的重要性。复合物的不和谐结果碱性基于DNA和rna的NGS之间的重排表明,基于DNA和rna的NGS检测在融合检测中都应该得到保证。RNA和蛋白质水平检测可能是验证复合物功能的关键碱性临床实践中的重新安排以获得最佳治疗决策。
数据和材料的可用性
本研究中使用和/或分析的数据集可根据合理要求从通讯作者处获得。
缩写
- 筛选:
-
间变性淋巴瘤激酶
- EML4:
-
棘皮动物微管相关蛋白like-4
- FFPE:
-
福尔马林固定蜡包埋
- 鱼:
-
荧光原位杂交
- 包含IHC:
-
免疫组织化学
- 门店:
-
新一代测序
- 非小细胞肺癌:
-
非小细胞肺癌
- PFS:
-
无进展生存
- TKI:
-
酪氨酸激酶抑制剂
参考文献
林俊杰,赖利GJ,邵志强。针对ALK:精准医疗出现耐药性。癌症发现,2017;7:137-55。
林俊杰,朱大众,Yoda S, Yeap BY, Schrock AB, dagoggo - jack I, Jessop NA,蒋光宇,Le LP, Gowen K,等。EML4-ALK变异对alk阳性肺癌耐药机制和临床结局的影响中华临床杂志。2018;36:1199-206。
Kwak EL, Bang YJ, Camidge DR, Shaw AT, Solomon B, Maki RG, Ou SH, Dezube BJ, Janne PA, Costa DB,等。间变性淋巴瘤激酶在非小细胞肺癌中的抑制作用。中华外科杂志,2010;
肖at, Kim DW, Nakagawa K, Seto T, Crino L, Ahn MJ, De Pas T, Besse B, Solomon BJ, Blackhall F,等。克唑替尼与化疗在晚期alk阳性肺癌中的比较。中华外科杂志,2013;
Seto T, Kiura K, Nishio M, Nakagawa K, Maemondo M, Inoue A, Hida T, Yamamoto N, Yoshioka H, Harada M,等。CH5424802 (RO5424802)用于alk重排晚期非小细胞肺癌患者(AF-001JP研究):一项单臂、开放标签、1-2期研究。《柳叶刀》2013;14:590-8。
Ou SH, Ahn JS, De Petris L, Govindan R, Yang JC, Hughes B, Lena H, Moro-Sibilot D, Bearz A, Ramirez SV,等。艾乐替尼治疗克唑替尼难治性alk重排非小细胞肺癌:全球II期研究中华临床医学杂志。2016;34:661-8。
Shaw AT, Gandhi L, Gadgeel S, Riely GJ, Cetnar J, West H, Camidge DR, Socinski MA, Chiappori A, Mekhail T,等。alk阳性、克唑替尼耐药的非小细胞肺癌的Alectinib:一项单组、多中心、2期试验《柳叶刀》2016;17:234-42。
肖AT, Kim DW, Mehra R, Tan DS, Felip E, Chow LQ, Camidge DR, Vansteenkiste J, Sharma S, De Pas T,等。Ceritinib在alk重排非小细胞肺癌中的应用。中华外科杂志,2014;
Kim D-W, Mehra R, Tan DSW, Felip E, Chow LQM, Camidge DR, Vansteenkiste J, Sharma S, De Pas T, Riely GJ,等。ceritinib在ALK重排非小细胞肺癌患者中的活性和安全性(ASCEND-1):来自多中心、开放标签、一期试验的最新结果。《柳叶刀》2016;17:452-63。
Wiesner T, Lee W, Obenauf AC, Ran L, Murali R, Zhang QF, Wong EW, Hu W, Scott SN, Shah RH,等。替代性转录起始导致一种新的ALK亚型在癌症中的表达。大自然。2015;526:453-7。
李JJ,公园,公园H,金,李·J·李J,尤区J, K, Y,公园本土知识,et al。肺腺癌突变史中癌基因重排的追踪。细胞。2019;177:1842-57。
戴维斯KD,艾斯纳DL。醒醒吧,闻闻这种融合:单模态分子检测忽略了驱动因素。临床癌症Res. 2019; 25:4586-8。
Davies KD, Le AT, Sheren J, Nijmeh, Gowan K, Jones KL, Varella-Garcia M, Aisner DL, Doebele RC。一组阳性患者样本中ROS1重排检测的分子检测方法比较中华外科杂志,2018;13:1474-82。
李伟,刘颖,李伟,陈玲,应俊。非小细胞肺癌基因间断点的检测。中华外科杂志,2020;15:1223-31。
李华,杜斌。基于Burrows-Wheeler变换的快速精确短读对齐。生物信息学。2009;25:1754-60。
Li H, Handsaker B, Wysoker A, Fennell T, Ruan J, Homer N, Marth G, Abecasis G, Durbin R.基因组计划数据处理S:序列比对/图谱格式和SAMtools。生物信息学。2009;25:2078-9。
McKenna A, Hanna M, Banks E, Sivachenko A, Cibulskis K, Kernytsky A, Garimella K, Altshuler D, Gabriel S, Daly M, DePristo MA。基因组分析工具包:用于分析下一代DNA测序数据的mapReduce框架。基因组Res. 2010; 20:1297-303。
葛红,刘凯,李娟,方峰,王晓明,何伟。基于碱基对分辨率的fusion map融合基因检测。生物信息学。2011;27:1922-8。
应健,郭玲,邱涛,单玲,凌云,刘霞,鲁宁。新型全自动免疫化学检测ALK重排在原发性肺腺癌中的诊断价值。杨晓明。2013;24(4):369 - 369。
McLeer-Florin A, Lantuejoul S.为什么技术方面而不是生物学解释alk阳性非小细胞肺癌的细胞异质性。中华外科杂志2012;4:240-1。
李娟,杨玲,高胜,张凯,吴霞,袁玉春,李红,胡松,翁勇,颜艳。突变体TP53 p.R273H和p.R273C增强癌细胞恶性,p、R273G不增强。胡姆穆塔特,2014;35:575-84。
杜霞,邵勇,高红,张霞,张红,班勇,秦红,太勇。ALK抑制剂克唑替尼无反应患者的ctr1 -ALK融合。癌症生物学杂志2018;19:962-6。
Kodama T, Motoi N, Ninomiya H,坂本H,北田K,冢口T,佐藤Y,野村K,长野H,石井N,等。患者来源细胞系中嗜色症介导的EML4-ALK重排的新机制。中华外科杂志,2014;9:1638-46。
Fransson S, Hansson M, Ruuth K, Djos A, Berbegall A, Javanmardi N, Abrahamsson J, Palmer RH, Noguera R, Hallberg B,等。基因内间变淋巴瘤激酶(ALK)重排:易位作为成神经细胞瘤肿瘤中ALK激活的新机制。基因染色体癌症。2015;54:99-109。
Pros E, Saigi M, Alameda D, Gomez-Mariano G, Martinez-Delgado B, Alburquerque-Bejar JJ, Carretero J, Tonda R, estev - codina A, Catala I,等。非吸烟相关肺癌细胞的全基因组分析显示,在egfr突变肿瘤中,常见的RB1重排与组织病理学转化相关。安·昂科尔,2020;31:274-82。
Cazes A, Louis-Brennetot C, Mazot P, Dingli F, Lombard B, Boeva V, Daveau R, Cappo J, Combaret V, Schleiermacher G,等。涉及ALK基因重排的特征揭示了成神经细胞瘤中与肿瘤侵袭性相关的一种新的截断形式。癌症决议2013;73:195-204。
Benayed R, Offin M, Mullaney K, Sukhadia P, Rios K, Desmeules P, Ptashkin R, Won H, Chang J, Halpenny D,等。肺腺癌可靶向激酶融合的高RNA测序率,DNA测序未检测到有丝分裂驱动改变,肿瘤突变负担低。临床癌症Res. 2019;25:47 - 22。
舒艳,李红,尚红,陈娟,苏霞,乐伟,雷勇,陶玲,邹超,吴伟。新型MPRIP-ROS1融合蛋白的鉴定及克唑替尼治疗晚期肺腺癌的临床疗效:1例报告。Onco Targets Ther. 2020; 13:10387-91。
Camidge DR, Bang Y-J, Kwak EL, Iafrate AJ, Varella-Garcia M, Fox SB, Riely GJ, Solomon B, Ou S-HI, Kim D-W,等。克唑替尼在alk阳性非小细胞肺癌患者中的活性和安全性:一项一期研究的最新结果《柳叶刀》2012;13:1011-9。
Peters S, Camidge DR, Shaw AT, Gadgeel S, Ahn JS, Kim DW, Ou SI, Perol M, Dziadziuszko R, Rosell R,等。艾乐替尼与克唑替尼在未治疗alk阳性非小细胞肺癌中的比较。中华医学杂志。2017;37:829 - 38。
中川K, Hida T, Nokihara H, Morise M, Azuma K, Kim YH, Seto T, Takiguchi Y, Nishio M,吉冈H,等。J-ALEX对alk阳性非小细胞肺癌患者艾乐替尼和克唑替尼的最终无进展生存期研究结果。肺癌。2020;139:195-9。
Camidge DR, Dziadziuszko R, Peters S, Mok T, Noe J, Nowicka M, Gadgeel SM, Cheema P, Pavlakis N, de Marinis F,等。在全球III期ALEX研究中,更新的疗效和安全性数据以及EML4-ALK融合变体对alectinib治疗未治疗alk阳性晚期非小细胞肺癌疗效的影响。中华外科杂志,2019;14:433 - 443。
谭ds, Araujo A,张娟,Signorovitch J,周志勇,蔡霞,刘刚。ceritinib和crizotinib作为alk靶向治疗早期NSCLC的疗效比较:与外部对照的调整比较。中华外科杂志2016;11:1550-7。
确认
我们感谢病理学系的所有作者对这个项目的贡献,以及贝里肿瘤公司,他们进行了有针对性的下一代测序。
资金
国家自然科学基金项目(No. 81272371)、河南省科技发展计划项目(No. 212102310134)、国家科技重大专项(No. 2018ZX10302205)、郑州市协同创新重大项目(郑州大学,No. 18XTZX12007)资助,夏培毅博士和李攀博士独立获得郑州大学第一附属医院青年创新基金资助。中国
作者信息
作者及隶属关系
贡献
PX和GJ设计了研究;PX、LZ、PL、EL、WL、JZ进行实验;PX、LZ、PL收集临床资料;XS进行数据生物信息学分析;PX、HL、GJ撰写稿件。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
该研究得到了郑州大学第一附属医院机构审查委员会的批准。所有患者均为这些基因组分析提供了知情的书面同意。
发表同意书
研究人员获得了所有参与研究的个体的知情同意,授权他们为研究目的访问他们的临床信息和肿瘤样本。
相互竞争的利益
李慧和苏晓星是贝里肿瘤公司的员工。所有其他作者声明没有利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:表S1。
基于dna的NGS面板中的基因列表。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
夏鹏,张磊,李鹏,李鹏。et al。复合物的分子特征和临床结果碱性通过下一代测序在非小细胞肺癌中发现重排。翻译医学杂志19, 308(2021)。https://doi.org/10.1186/s12967-021-02982-4
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12967-021-02982-4
关键字
- 碱性融合
- 复杂的重组
- 非小细胞肺癌
- 新一代测序
- 靶向治疗