- 研究gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
mybl2诱导的PITPNA-AS1通过miR-520d-5p和DDX54上调SIK2在三阴性乳腺癌中发挥致癌功能gydF4y2Ba
转化医学杂志gydF4y2Ba体积gydF4y2Ba19gydF4y2Ba,文章号:gydF4y2Ba333gydF4y2Ba(gydF4y2Ba2021gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
近年来,长链非编码rna (long non-coding RNAs, lncRNAs)因其在包括三阴性乳腺癌(triple-negative breast cancer, TNBC)在内的肿瘤发生和发展中的调控作用而备受关注。LncRNA PITPNA反义RNA 1 (PITPNA- as1)在一些癌症中已被探索,但其在TNBC中的功能和分子机制尚不清楚。gydF4y2Ba
方法gydF4y2Ba
RT-qPCR检测PITPNA-AS1在TNBC组织和细胞中的表达。采用CCK-8检测TNBC细胞活力、增殖、迁移、侵袭、集落形成、创面愈合、transwell检测。流式细胞仪检测细胞凋亡情况。western blot检测emt相关标记的表达。通过RNA pull down、荧光素酶报告、RIP和ChIP等方法探讨了PITPNA-AS1的分子机制。gydF4y2Ba
结果gydF4y2Ba
PITPNA-AS1在TNBC组织和细胞中表达量较高。敲低PITPNA-AS1可抑制TNBC细胞的活力、增殖、迁移、侵袭,抑制小鼠异种移植瘤生长。在机制上,PITPNA-AS1通过海绵miR-520d-5p和招募DDX54蛋白上调SIK2的表达。拯救试验结果表明,沉默的PITPNA-AS1对TNBC细胞过程的抑制作用部分通过过表达SIK2或miR-520d-5p抑制和DDX54过表达联合被拯救。更重要的是,我们发现TNBC细胞中PITPNA-AS1的上调与转录因子MYBL2有关。gydF4y2Ba
结论gydF4y2Ba
MYBL2激活的PITPNA-AS1通过上调SIK2在TNBC中发挥致癌作用。gydF4y2Ba
简介gydF4y2Ba
乳癌的发病率呈快速上升趋势,每年新确诊病例超过100万例[gydF4y2Ba1gydF4y2Ba].根据HER-2、雌激素受体(ER)、ki-67、孕酮受体(PR)的分子特征,可将BC分为HER-2过表达、Lumina A、Lumina B、“正常样”乳腺肿瘤和基底样肿瘤等不同亚型。根据定义,三阴性乳腺癌(TNBC)是一种HER-2、ER和PR阴性的肿瘤[gydF4y2Ba2gydF4y2Ba].目前,由于缺乏有效的生物标志物,手术切除和化疗是TNBC患者的主要全身治疗方法[gydF4y2Ba3.gydF4y2Ba].与其他BC亚型患者相比,TNBC患者的转移率和复发率明显更高[gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba].因此,探索更有效的生物标志物对TNBC的早期诊断和深入治疗至关重要。gydF4y2Ba
人们普遍认为非编码rna (non-coding RNAs, ncRNAs)是调控基因表达的关键因素,从而对肿瘤或非肿瘤细胞的表型发挥作用[gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba].长非编码rna (Long non-coding RNAs, lncrna)通常被认为是长度超过200个核苷酸的基因组转录物[gydF4y2Ba8gydF4y2Ba].lncrna缺乏不可缺少长度的开放阅读结构,在蛋白质编码中受到限制[gydF4y2Ba9gydF4y2Ba].多种研究强调lncrna与癌症发展的分子机制有关,可作为有前景的生物标志物和癌症治疗靶点[gydF4y2Ba10gydF4y2Ba].此前,许多研究强调了lncrna在调节细胞生长、凋亡、迁移和侵袭等多种细胞生物学行为中的重要作用[gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba].增加lncRNAs,如NEAT1 [gydF4y2Ba13gydF4y2Ba], borg [gydF4y2Ba14gydF4y2Ba]及LINC01638 [gydF4y2Ba15gydF4y2Ba],已被确定为TNBC中的癌基因。gydF4y2Ba
lncrna通过多种机制参与肿瘤发生和癌症进展[gydF4y2Ba16gydF4y2Ba].重要的是,lncRNA可以作为竞争的内源性RNA (ceRNA)通过海绵microRNA (miRNA)上调mRNA的表达。例如,lncRNA EPB41L4A-AS2通过海绵miR-301a-5p上调FOXL1表达抑制肝细胞癌进展[gydF4y2Ba17gydF4y2Ba].LncRNA NORAD通过靶向miR-590-3p/SIP1轴促进宫颈癌细胞增殖和侵袭[gydF4y2Ba18gydF4y2Ba].此外,lncRNAs可以通过与rna结合蛋白(RBPs)结合来调节肿瘤起始,以维持mRNA的稳定。如LINC00324在胃癌中通过与HuR蛋白结合,正调控FAM83B mRNA的稳定性,促进细胞增殖[gydF4y2Ba19gydF4y2Ba].LBX2- as1通过招募FUS蛋白稳定LBX2 mRNA表达,促进胃癌进展[gydF4y2Ba20.gydF4y2Ba].LncRNA PITPNA反义RNA 1 (PITPNA- as1)先前被报道作为肝细胞癌的癌基因,其作用是ceRNA [gydF4y2Ba21gydF4y2Ba].但其在TNBC中的功能和分子机制尚不清楚。gydF4y2Ba
在本研究中,我们研究了PITPNA-AS1在TNBC细胞过程中的功能和调控机制。我们的研究结果显示,mybl2激活的PITPNA-AS1海绵miR-520d-5p并招募DDX54蛋白来增加SIK2的表达,从而促进TNBC中的细胞活性。这一发现提示PITPNA-AS1是TNBC治疗的有前途的生物标志物。gydF4y2Ba
材料与方法gydF4y2Ba
组织样本gydF4y2Ba
从山西医科大学第二医院(山西,中国)诊断为TNBC的患者中收集56对TNBC组织及其邻近的非肿瘤组织。所有患者术前均签署知情同意书,均未接受任何抗癌治疗。从患者身上采集的标本在液氮中快速冷冻,随后在- 80°C保存。采用56对TNBC组织和相应的非肿瘤组织进行基因表达分析,经山西医科大学第二医院机构伦理委员会批准。gydF4y2Ba
细胞系gydF4y2Ba
从ATCC (Manassas, VA)购买1个正常乳腺上皮细胞MCF10A和4个人TNBC细胞系(HCC1937, MDA-MB-468, MDA-MB-231, MDA-MB-436)。细胞在含有10%胎牛血清(FBS)和1%青霉素/链霉素的DMEM (Invitrogen, Carlsbad, CA)中在37°C和5% CO的湿空气中孵育gydF4y2Ba2gydF4y2Ba.gydF4y2Ba
细胞转染gydF4y2Ba
HCC1937和MDA-MB-468细胞转染靶向PITPNA-AS1 (sh-PITPNA-AS1#1/2)、DDX54 (sh-DDX54#1/2)、MYBL2 (sh-MYBL2)和相应阴性对照(sh-NC)的sh- rna(质粒骨架:pGPU6/Hygro),最终浓度为100 nM。通过对潮霉素B抗性的检测,筛选出稳定转染的细胞。为了过表达SIK2、E2F6、PAX5、FOXP2、ELK5、MYBL2和YY1,将靶向相应基因的pcDNA3.1载体和空pcDNA3.1载体(对照)转染到HCC1937和MDA-MB-468细胞中。对于过表达或沉默miR-520d-5p, miR-520d-5p模拟物或抑制剂分别转染到HCC1937和MDA-MB-468细胞中,NC模拟物或抑制剂作为阴性对照。以上所有质粒或寡核苷酸均购自GenePharma,用Lipofectamine 2000 (Invitrogen)转染HCC1937和MDA-MB-468细胞2天。gydF4y2Ba
RT-qPCR分析gydF4y2Ba
采用TRIzol Reagent (Invitrogen)从MCF10A、HCC1937、MDA-MB-468、MDA-MB-231、MDA-MB-436细胞中提取总RNA,用反转录试剂盒(Takara)反转录成cDNA。A一步结核绿gydF4y2Ba®gydF4y2Ba利用PrimeScript™RT-PCR Kit (Takara)在Bio-Rad CFX96 Real-Time PCR系统上进行RT-qPCR。2gydF4y2Ba−∆∆CtgydF4y2Ba方法(gydF4y2Ba22gydF4y2Ba]用于计算相对表达倍数变化。GAPDH作为PITPNA-AS1和mrna的内参,U6作为miRNAs的内参。热循环条件设置为:95°C, 5min, 95°C, 10s, 55°C, 30s,循环45次,从55°C到95°C, 2 min,每0.2°C得到一次熔融曲线分析。相关引物序列见表gydF4y2Ba1gydF4y2Ba,用GETprime软件设计,由RiboBio公司合成。gydF4y2Ba
亚细胞分离试验gydF4y2Ba
采用亚细胞分割法评估PITPNA-AS1的亚细胞定位。根据制造商说明,使用细胞质和核RNA纯化试剂盒(Norgen)分离从TNBC细胞中提取的核和细胞质组分。RT-qPCR检测PITPNA-AS1、GAPDH或U6的表达。GAPDH为细胞质对照,U6为核对照。gydF4y2Ba
荧光原位杂交(FISH)法gydF4y2Ba
采用FISH法检测PITPNA-AS1的亚细胞位置。HCC1937和MDA-MB-468细胞(5 × 10gydF4y2Ba3.gydF4y2Ba每孔培养细胞),24 h后弃上清。PBS洗净,4%多聚甲醛固定后,用含0.5% Triton X-100的PBS渗透细胞。然后,用预杂交溶液在37℃下阻断细胞4 h,然后用PITPNA-AS1特异性探针(RiboBio)在37℃下杂交过夜,然后在42℃下用杂交溶液在黑暗中洗涤。然后,用4 ' -6-二氨基氨基-2-苯基吲哚(DAPI)染色细胞核10min,用荧光显微镜(Olympus)捕捉细胞图像。gydF4y2Ba
细胞计数试剂盒-8 (CCK-8)测定gydF4y2Ba
根据制造商的要求,细胞活力通过CCK-8试剂盒(Boster生物技术公司,CA,美国)进行验证。简单地说,转染的HCC1937和MDA-MB-468细胞以2 × 10的密度被镀到96孔板上gydF4y2Ba3.gydF4y2Ba细胞/孔,孵育0、24、48和72 h。然后,每孔中加入10 μL CCK-8溶液,5% CO中继续孵育gydF4y2Ba2gydF4y2Ba在37°C下1 h后细胞粘附。然后取出培养液,用PBS清洗两次。最后,用微孔板阅读器(EL340;Bio-Tek Instruments, Hopkinton, MA, USA)用于检测细胞活力。gydF4y2Ba
菌落形成试验gydF4y2Ba
采用菌落形成试验观察细胞增殖情况。简而言之,1 × 10gydF4y2Ba3.gydF4y2BaHCC1937和MDA-MB-468细胞分别接种于6孔板。培养2周后,菌落用甲醇固定15 min,用结晶紫(Sigma-Aldrich)在PBS中染色20 min,然后用流动水慢慢洗去结晶紫。平板反向风干,人工计数染色菌落数量。gydF4y2Ba
流式细胞术分析gydF4y2Ba
转染HCC1937和MDA-MB-468细胞后,采用碘化丙啶(PI)和FITC-Annexin V染色检测细胞凋亡率。HCC1937和MDA-MB-468细胞以1 × 10的浓度接种于6孔板gydF4y2Ba5gydF4y2Ba细胞在5 μL Annexin V-FITC中孵育10 min, 10 μL PI在4℃黑暗中孵育15 min。随后,使用CellQuest软件(BD Biosciences, San Jose, CA),流式细胞仪分析细胞(FACScan;BD Biosciences),并评估TNBC细胞的凋亡率。早期凋亡细胞分布在第二象限,晚期凋亡和坏死细胞分布在第三和第四象限。凋亡率(%)定义为第三象限的细胞百分比。gydF4y2Ba
伤口愈合试验gydF4y2Ba
采用创面愈合试验检测细胞迁移。TNBC细胞(1.5 × 10gydF4y2Ba6gydF4y2Ba)被镀进6孔板。然后,在细胞达到80%合流后,用移液管尖端水平划伤。细胞洗涤两次后,细胞在无血清培养基中孵育24小时。在低倍率相衬显微镜下拍摄细胞之间的间隙(Olympus MK, Tokyo, Japan)。gydF4y2Ba
Transwell化验gydF4y2Ba
Transwell实验检测细胞在体外的迁移情况。基质凝胶涂层的上腔(8.0 μm孔径,BD Biosciences)填充TNBC细胞(5 × 10gydF4y2Ba4gydF4y2Ba/well)悬浮在DMEM中。将DMEM添加10% FBS后置于底室。48小时后,上腔的细胞被棉签刮掉。下腔细胞用甲醇固定,用0.5%结晶紫染色。最后,使用倒置显微镜(Olympus)在随机选择的5个视野下,用Image J软件计算平均染色细胞数。gydF4y2Ba
Western blot分析gydF4y2Ba
采用含蛋白酶抑制剂的RIPA裂解缓冲液提取HCC1937和MDA-MB-468细胞总蛋白。在转移到聚偏氟乙烯膜(Millipore)之前,用10%十二烷基硫酸钠聚丙烯酰胺凝胶电泳分离等效蛋白质。用5%脱脂奶阻断1 h后,用抗E-cadherin (ab40772, 1/20000)、N-cadherin (ab76011, 1/5000)、Vimentin (ab92547, 1/200)、Slug (ab27568, 1/500)、Twist (ab175430, 1/1000)、SIK2 (ab53423, 1/1000)、DDX54 (ab76947, 1/2000)、GAPDH (ab9485, 1/2500)的一抗在4℃孵育过夜。加入与HRP结合的二抗(ab6721, 1/10000),室温孵育1 h。所有抗体均购自Abcam (Cambridge, USA)。应用化学发光检测系统对蛋白质进行可视化。利用ImageJ软件分析各蛋白条带的灰度值,计算目标蛋白的灰度值与GAPDH的比值。gydF4y2Ba
体内实验gydF4y2Ba
动物实验:20只6周龄BALB/C雌性裸鼠;重20-30克),购自石莱克公司。动物实验得到了山西医科大学第二医院(山西,中国)的伦理批准。裸鼠皮下注射HCC1937细胞悬液0.2 mL (3 × 10gydF4y2Ba6gydF4y2Ba)稳定转染sh-PITPNA-AS1或sh-NC。每4天测量一次肿瘤体积。gydF4y2Ba\({\文本{卷}}\,({\文本{mm}} ^{3}) = 1/2 \,文本{L}}{\ \ *{文本\ D{}} ^{2} \)。gydF4y2BaL为肿瘤最大直径,D为肿瘤最短直径。28天后,对小鼠实施安乐死,切除肿瘤进行称重。gydF4y2Ba
免疫组化(IHC)gydF4y2Ba
肿瘤组织用4%的多聚甲醛固定,然后在乙醇溶液中脱水。接下来,将组织包埋在石蜡中,并切成4 μm的切片。切片常规去亲和,然后进行抗原提取。PBS洗涤15分钟后,用山羊血清阻塞组织1小时,然后用Ki67 (ab92742, 1/500)一抗在4℃下孵育一夜。随后用酶标二抗(ab6721, 1/1000)培养。然后用苏木精反染1 min,然后用中性香脂裱片。使用Histostain™SP-9000免疫组化染色试剂盒(Zymed Laboratories, South San Francisco, CA, USA)检测Ki67阳性。使用OLYMPUSBX-41显微镜(Olympus)捕捉图像。gydF4y2Ba
RNA拉下试验gydF4y2Ba
用50 nM生物素标记的PITPNA-AS1转染HCC1937和MDA-MB-468细胞48 h,然后在RIPA裂解缓冲液中孵育10 min, 14,000×离心gydF4y2BaggydF4y2Ba获得上清液。蛋白裂解液用M-280链霉亲和素珠孵育,链霉亲和素珠预先包被无rnase的牛血清白蛋白和酵母tRNA。接下来,珠粒在4℃孵育3小时。用TRIzol纯化结合miR-520d-5p,并用RT-qPCR检测其表达。根据制造商的指南,使用Pierce Magnetic RNA-Protein Pull-Down Kit(赛默飞世尔科学公司)检测与PITPNA-AS1结合的蛋白质。从HCC1937或MDA-MB-468细胞中提取的蛋白质在4℃条件下被生物素化的PITPNA-AS1 (50 pmol)和M-280链霉素磁珠(Invitrogen)浸泡1小时。最后用RIPA缓冲液洗涤细胞5次,用5×负载缓冲液在95℃孵育5 min。western blotting检测洗脱后的DDX54、FMR1、IGF2BP1、IGF2BP2蛋白。gydF4y2Ba
荧光素酶报告试验gydF4y2Ba
对于荧光素酶报告检测,通过将与miR-520d-5p或SIK2 3'UTR互补的PITPNA-AS1的野生型(WT)或突变型(Mut)序列亚克隆到pmirGLO双荧光素酶载体中构建PITPNA-AS1-WT/Mut或SIK2-WT/Mut报告。然后,这些构建的报告细胞与miR-520d-5p模拟物或NC模拟物共转染到HCC1937或MDA-MB-468细胞中。对于PITPNA-AS1启动子-荧光素酶分析,将野生型或突变的PITPNA-AS1启动子序列(位点1和位点2)亚克隆到pGL3-Basis荧光素酶载体(Promega, Madison, WI, USA),并将这些结构与pcDNA3.1/MYBL2或pcDNA3.1共转染到HCC1937和MDA-MB-468细胞中。2天后,使用双荧光素酶报告测定系统(Promega)测定荧光素酶活性。相对荧光素酶活性定义为萤火虫荧光素酶相对光单位与荧光素酶相对光单位的比值gydF4y2BaRenillagydF4y2Ba荧光素酶。gydF4y2Ba
RNA免疫沉淀(RIP)试验gydF4y2Ba
基于制造商的仪器,应用了EZ-Magna RIP工具包(Millipore)。总之,HCC1937和MDA-MB-468细胞用预冷的PBS洗涤,然后在RIPA裂解缓冲液中冰浴裂解5min, 12000 ×离心gydF4y2BaggydF4y2Ba在4℃下放置10分钟。然后,将细胞提取物与磁珠包被的人抗ago2 (Millipore)、抗ddx54 (Millipore)或对照抗igg (Millipore)在4℃孵育过夜。最后,经蛋白酶K纯化后,采用RT-qPCR方法分析Ago2免疫沉淀的PITPNA-AS1、miR-520d-5p、SIK2的表达和DDX52免疫沉淀的PITPNA-AS1、SIK2的表达。gydF4y2Ba
放线菌素D测定gydF4y2Ba
放线菌素D可抑制mrna的合成。sh-NC、sh-PITPNA-AS1#1、sh-DDX54#1转染48 h后,用放线菌素D (5 μmol/L)处理HCC1937和MDA-MB-468细胞0、4、8、12 h, RT-qPCR检测SIK2的相对表达水平。gydF4y2Ba
染色质免疫沉淀(ChIP)试验gydF4y2Ba
利用Magna ChIP Kit (Millipore)进行ChIP,以探索MYBL2与PITPNA-AS1启动子之间的结合位点。简单地说,通过超声将交联染色质DNA分离成200-2000 bp的片段。接下来,将裂解物与抗mybl2或抗igg(内控)在4℃下免疫沉淀过夜。随后,将混合物离心,沉淀物分别用低盐缓冲液、高盐缓冲液、LiCl溶液和微量元素溶液洗涤。蛋白- dna复合物用250 μL ChIP Wash Buffer洗脱,用20 μL 5 M NaCl去交联。采用RT-qPCR检测免疫沉淀DNA数量。gydF4y2Ba
统计分析gydF4y2Ba
数据以均数±标准差表示,并使用SPSS 22.0 (SPSS,芝加哥,美国)对来自三个生物和技术复制的数据进行分析。用Student 's进行方差显著性评价gydF4y2BatgydF4y2Ba两组间的差异进行方差分析或三组间的差异进行方差分析。以P < 0.05为有统计学意义的阈值。gydF4y2Ba
结果gydF4y2Ba
PITPNA-AS1在TNBC中表达上调,并在细胞质中定位gydF4y2Ba
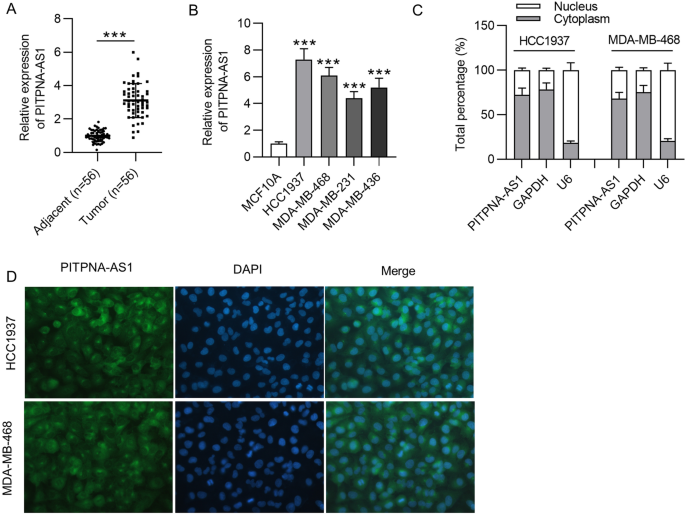
首先,采用RT-qPCR检测PITPNA-AS1在TNBC中的表达谱。与邻近非肿瘤组织相比,TNBC组织中PITPNA-AS1表达显著上调(图2)。gydF4y2Ba1gydF4y2Ba此外,PITPNA-AS1在TNBC细胞系(HCC1937、MDA-MB-468、MDA-MB-231和MDA-MB-436)中的表达水平高于MCF10A细胞系(图2)。gydF4y2Ba1gydF4y2BaB).由于HCC1937和MDA-MB-468细胞含有较高的PITPNA-AS1表达,因此使用它们进行以下检测。亚细胞分离实验结果显示,PITPNA-AS1主要分布在HCC1937和MDA-MB-468细胞的细胞质中(图3)。gydF4y2Ba1gydF4y2BaC)。此外,FISH实验结果证实PITPNA-AS1是TNBC中的细胞质RNA(图。gydF4y2Ba1gydF4y2BaD),提示PITPNA-AS1可能在转录后水平调控基因表达。gydF4y2Ba
PITPNA-AS1的敲除抑制了TNBC细胞过程gydF4y2Ba
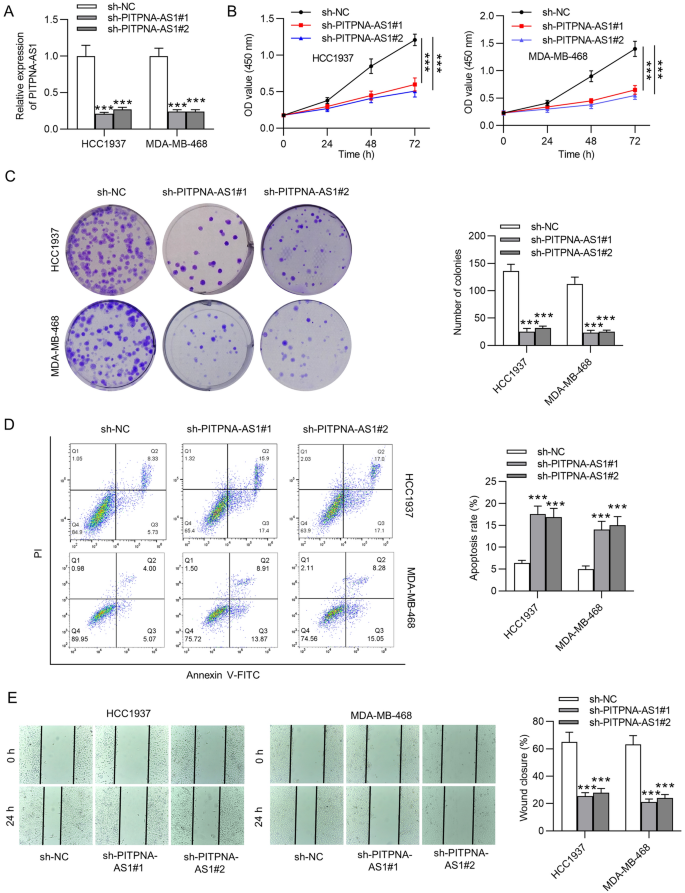
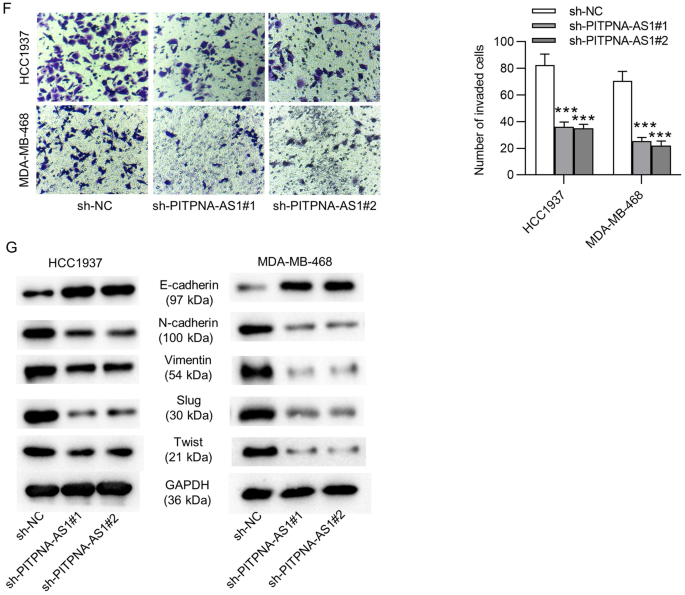
考虑到PITPNA-AS1在TNBC中的表达上调,我们通过功能缺失检测来探讨PITPNA-AS1在TNBC中的功能作用。通过sh-PITPNA-AS1#1和sh-PITPNA-AS1#2转染,HCC1937和MDA-MB-468细胞中PITPNA-AS1的表达稳定沉默。gydF4y2Ba2gydF4y2BaA).如图所示。gydF4y2Ba2gydF4y2Ba沉默PITPNA-AS1可抑制B、HCC1937和MDA-MB-468细胞的活力。此外,集落形成实验结果表明,PITPNA-AS1抑制HCC1937和MDA-MB-468细胞增殖(图3)。gydF4y2Ba2gydF4y2BaC)。此外,PITPNA-AS1表达降低促进了HCC1937和MDA-MB-468细胞的凋亡率(图。gydF4y2Ba2gydF4y2BaD).通过伤口愈合实验的结果,我们发现PITPNA-AS1的下调抑制了HCC1937和MDA-MB-468细胞的迁移能力(图。gydF4y2Ba2gydF4y2BaE)。此外,下调的PITPNA-AS1抑制了HCC1937和MDA-MB-468细胞的侵袭能力(图。gydF4y2Ba2gydF4y2BaF).类似地,PITPNA-AS1缺失导致E-cadherin蛋白水平升高,N-cadherin、Vimentin、Slug和Twist蛋白水平下降,揭示了沉默的PITPNA-AS1抑制了HCC1937和MDA-MB-468细胞的上皮-间充质转化(EMT)过程(图)。gydF4y2Ba2gydF4y2BaG)。gydF4y2Ba
沉默PITPNA-AS1的体外生物学功能。gydF4y2Ba一个gydF4y2BaRT-qPCR分析验证PITPNA-AS1在HCC1937和MDA-MB-468细胞中的敲除效率。gydF4y2BaBgydF4y2Ba,gydF4y2BaCgydF4y2BaCCK-8和集落形成检测评估了sh-PITPNA-AS1#1/2或sh-NC转染HCC1937和MDA-MB-468细胞的增殖能力。gydF4y2BaDgydF4y2Ba流式细胞仪检测敲低PITPNA-AS1对HCC1937和MDA-MB-468细胞凋亡的影响。gydF4y2BaEgydF4y2Ba,gydF4y2BaFgydF4y2Ba通过伤口愈合和Transwell试验分析pitpna - as1沉默细胞的细胞迁移和侵袭。gydF4y2BaGgydF4y2Bawestern blot检测sh-PITPNA-AS1#1/2和sh-NC组EMT过程相关蛋白水平。***p < 0.001gydF4y2Ba
PITPNA-AS1沉默抑制体内肿瘤生长gydF4y2Ba
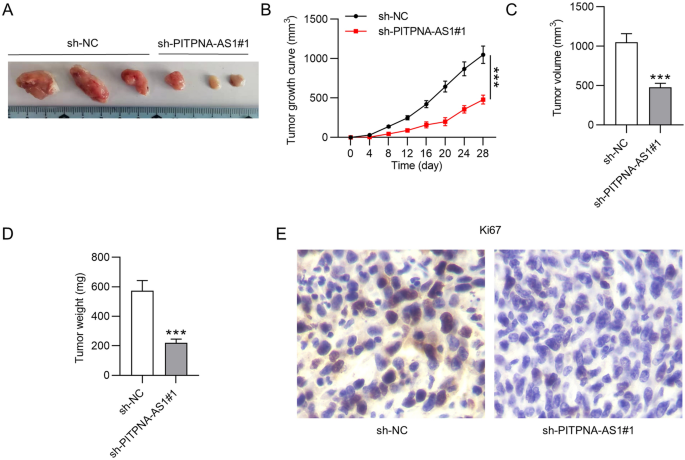
随后,通过动物实验探索PITPNA-AS1在体内对肿瘤生长的生物学作用。首先将稳定转染sh-PITPNA-AS1#1或sh-NC的HCC1937细胞皮下注射到裸鼠体内。我们发现sh-PITPNA-AS1#1组肿瘤大小小于sh-NC组(图。gydF4y2Ba3.gydF4y2BaA). sh-PITPNA-AS1#1组肿瘤生长较sh-NC组慢(图。gydF4y2Ba3.gydF4y2BaB)。此外,通过敲除PITPNA-AS1,肿瘤体积和肿瘤重量均降低(图。gydF4y2Ba3.gydF4y2BaC, D)。更重要的是,异种移植瘤的IHC检测结果证实,PITPNA-AS1缺失降低了Ki67的表达,提示PITPNA-AS1下调抑制了肿瘤细胞增殖(图。gydF4y2Ba3.gydF4y2BaE)。gydF4y2Ba
PITPNA-AS1在TNBC细胞中隔离miR-520d-5pgydF4y2Ba
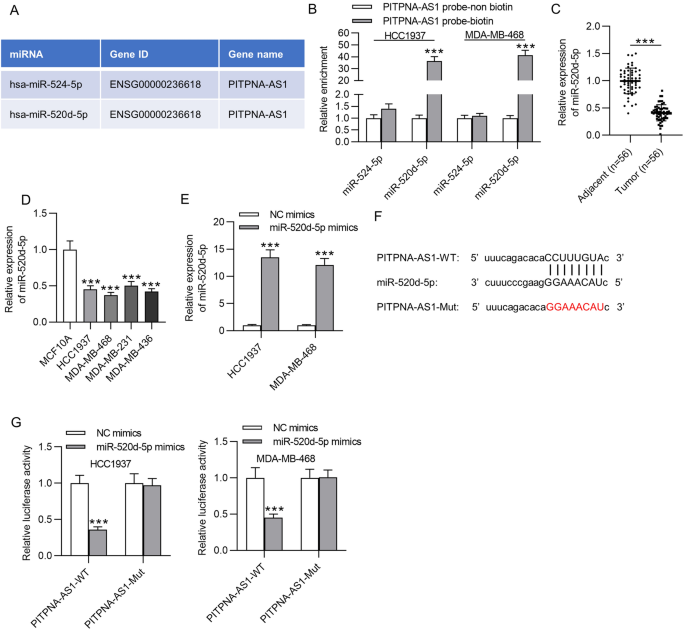
基于PITPNA-AS1在TNBC细胞中的转录后调节作用,我们推测PITPNA-AS1可能通过ceRNA网络海绵特异性miRNA来调节TNBC的进展。我们使用starBase (gydF4y2Bahttp://starbase.sysu.edu.cn/gydF4y2Ba).在CLIP数据≥3、Degradome数据≥3的条件下,发现了两个假定的miRNAs (miR-524-5p和miR-520d-5p)(图5)。gydF4y2Ba4gydF4y2Ba此外,RNA下拉实验显示,PITPNA-AS1生物素探针组miR-520d-5p高度富集,而PITPNA-AS1生物素探针组miR-524-5p没有显著富集(图5)。gydF4y2Ba4gydF4y2BaB),表明PITPNA-AS1可能与miR-520d-5p结合,而不与miR-524-5p结合。随后,通过RT-qPCR检测到TNBC组织中miR-520d-5p显著下调(图。gydF4y2Ba4gydF4y2BaC)。此外,在TNBC细胞系中也观察到miR-520d-5p的低表达(图。gydF4y2Ba4gydF4y2BaD)。随后,miR-520d-5p模拟物转染到HCC1937和MDA-MB-468细胞中,过表达miR-520d-5p进行进一步分析(图。gydF4y2Ba4gydF4y2BaE)。此外,starBase揭示了PITPNA-AS1和miR-520d-5p之间的互补结合序列(图。gydF4y2Ba4gydF4y2BaF).为了确认PITPNA-AS1与miR-520d-5p的相互作用,进行荧光素酶报告实验。结果显示miR-520d-5p上调诱导PITPNA-AS1-WT荧光素酶活性减弱。转染miR-520d-5p模拟物后,与转染NC模拟物后相比,HCC1937和MDA-MB-468细胞中PITPNA-AS1-Mut荧光素酶活性无显著差异(图5)。gydF4y2Ba4gydF4y2BaG)。gydF4y2Ba
PITPNA-AS1作为miR-520d-5p的海绵。gydF4y2Ba一个gydF4y2BaPITPNA-AS1的潜在mirna从starBase获得。gydF4y2BaBgydF4y2Ba通过RNA下拉实验评估PITPNA-AS1与预测mirna的结合能力。gydF4y2BaCgydF4y2Ba通过RT-qPCR检测MiR-520d-5p在TNBC及邻近非肿瘤标本中的表达。gydF4y2BaDgydF4y2BaRT-qPCR检测miR-520d-5p在TNBC细胞系和MCF10A细胞系中的表达。gydF4y2BaEgydF4y2BaRT-qPCR检测MiR-520d-5p模拟物转染HCC1937和MDA-MB-468细胞中MiR-520d-5p的表达gydF4y2BaFgydF4y2BaPITPNA-AS1和miR-520d-5p之间存在一个结合位点。gydF4y2BaGgydF4y2Ba荧光素酶报告实验验证了PITPNA-AS1与miR-520d-5p的结合。***p < 0.001gydF4y2Ba
SIK2是miR-520d-5p在TNBC细胞中的靶基因gydF4y2Ba
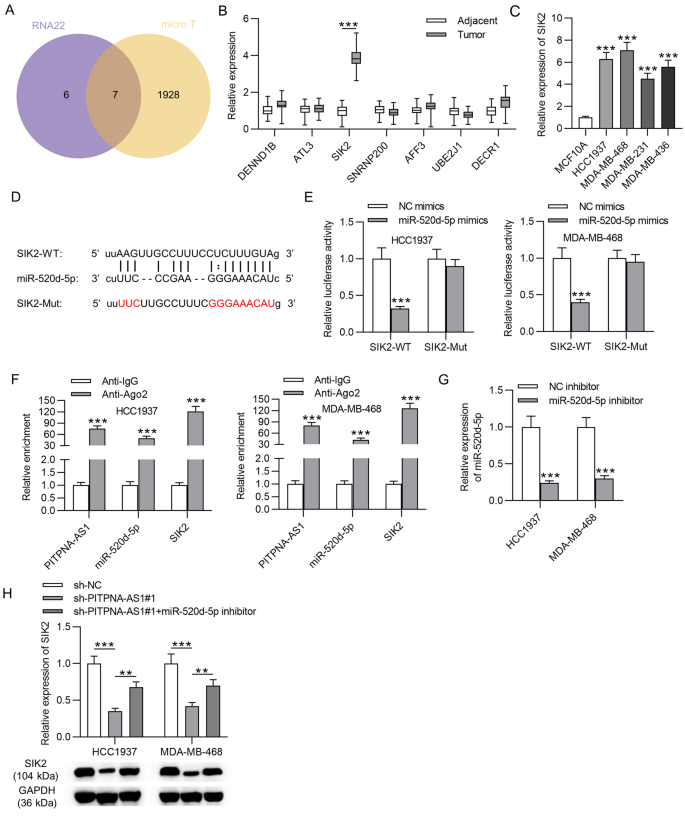
为了进一步支持PITPNA-AS1介导的ceRNA机制假设,我们探索了miR-520d-5p在TNBC中的靶基因。结合两种在线工具(RNA22和microT)的结果,预测了7种mrna作为miR-520d-5p的下游基因(图5)。gydF4y2Ba5gydF4y2BaA). RT-qPCR分析证实了这些mrna在TNBC组织中的表达,仅发现SIK2在TNBC组织中显著上调(图。gydF4y2Ba5gydF4y2BaB)。随后,与MCF10A细胞系相比,TNBC细胞系中SIK2的表达更高(图。gydF4y2Ba5gydF4y2BaC)。随后,starBase预测了miR-520d-5p与SIK2之间的结合位点(图。gydF4y2Ba5gydF4y2BaD)。荧光素酶报告实验表明,miR-520d-5p主要抑制SIK2-WT的荧光素酶活性,而不是SIK2-Mut(图2)。gydF4y2Ba5gydF4y2Ba此外,RIP实验证实PITPNA-AS1、miR-520d-5p和SIK2在rna诱导的沉默复合物(RISCs)中共存(图5)。gydF4y2Ba5gydF4y2BaF)。此外,转染miR-520d-5p抑制剂后,miR-520d-5p在HCC1937和MDA-MB-468细胞中的表达被抑制(图。gydF4y2Ba5gydF4y2Ba我们发现,下调miR-520d-5p可以部分逆转下调PITPNA-AS1导致的SIK2 mRNA和蛋白表达下降,这表明,除了miR-520d-5p之外,PITPNA-AS1还可以通过其他分子调控SIK2的表达(图520d-5p)。gydF4y2Ba5gydF4y2BaH)。gydF4y2Ba
MiR-520d-5p在TNBC中靶向SIK2。gydF4y2Ba一个gydF4y2Ba基于DIANA和microT数据库预测miR-520d-5p的靶向mrna。gydF4y2BaBgydF4y2Ba通过RT-qPCR检测预测mrna在TNBC组织和邻近非肿瘤组织中的表达水平。gydF4y2BaCgydF4y2BaRT-qPCR检测SIK2在TNBC细胞和MCF10A细胞中的表达。gydF4y2BaDgydF4y2Ba通过starBase预测miR-520d-5p在SIK2 3'UTR上的结合序列。gydF4y2BaEgydF4y2Ba荧光素酶报告实验证实了miR-520d-5p与SIK2的结合。gydF4y2BaFgydF4y2BaRIP实验显示PITPNA-AS1, miR-520d-5p和SIK2在与抗igg或抗ago2偶联的珠子中富集。gydF4y2BaGgydF4y2BaRT-qPCR检测miR-520d-5p对HCC1937和MDA-MB-468细胞的抑制效果。gydF4y2BaHgydF4y2BaRT-qPCR和western blotting检测各组HCC1937和MDA-MB-468细胞中SIK2的表达。**p < 0.01, ***p < 0.001gydF4y2Ba
PITPNA-AS1募集DDX54蛋白稳定SIK2 mRNAgydF4y2Ba
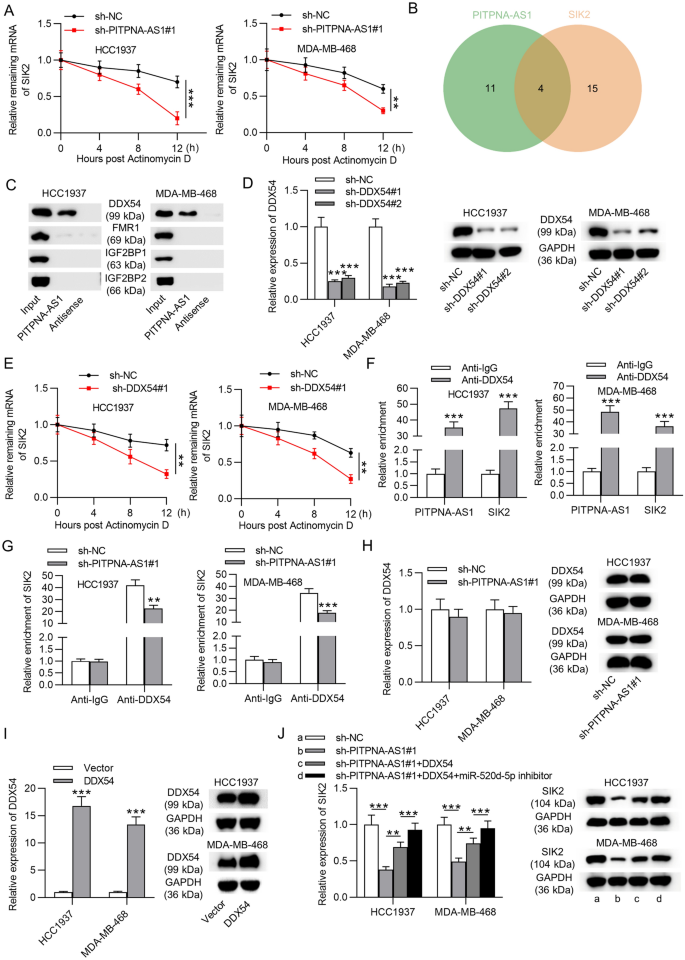
lncRNA通过招募RBPs来维持mRNA的稳定性,存在另一种转录后调控[gydF4y2Ba19gydF4y2Ba].基于上述发现,我们假设PITPNA-AS1可能不仅作为ceRNA调节SIK2的表达,还可能与rbp相互作用。采用RT-qPCR检测放线菌素D处理的TNBC细胞中SIK2 mRNA的表达。结果表明,与sh-NC组相比,PITPNA-AS1敲除抑制了SIK2 mRNA的稳定性(图2)。gydF4y2Ba6gydF4y2BaA).随后,我们探索了可能与PITPNA-AS1和SIK2相互作用的潜在rbp。通过starBase,发现了4个RBPs (DDX54、FMR1、IGF2BP1和IGF2BP2)(图。gydF4y2Ba6gydF4y2BaB).为了缩小选择范围,我们进行了RNA下拉实验,结果表明只有DDX54可以在HCC1937和MDA-MB-468细胞中与PITPNA-AS1结合(图。gydF4y2Ba6gydF4y2BaC)。随后,为了验证DDX54是否对SIK2 mRNA稳定性有作用,我们在HCC1937和MDA-MB-468细胞中转染sh-DDX54#1/2敲除了DDX54,表明sh-DDX54#1具有更好的敲除效率(图。gydF4y2Ba6gydF4y2BaD)。在放线菌素D处理的HCC1937和MDA-MB-468细胞中,DDX54的下调显著降低了SIK2 mRNA的稳定性(图。gydF4y2Ba6gydF4y2BaE)。随后,RIP实验结果进一步证实DDX54蛋白与PITPNA-AS1和SIK2结合(图。gydF4y2Ba6gydF4y2BaF).此外,PITPNA-AS1的沉默削弱了DDX54蛋白与SIK2 mRNA之间的相互作用(图。gydF4y2Ba6gydF4y2Ba在sh-PITPNA-AS1#1转染的TNBC细胞中,DDX54 mRNA和蛋白水平的结果保持不变,这表明PITPNA-AS1招募DDX54来维持SIK2 mRNA的表达,而不是调节DDX54的表达(图2)。gydF4y2Ba6gydF4y2BaH).通过RT-qPCR和western blotting验证DDX54过表达效率(图;gydF4y2Ba6gydF4y2BaI).如图所示。gydF4y2Ba6gydF4y2BaJ,沉默的PITPNA-AS1抑制的SIK2 mRNA和蛋白表达被过表达的DDX54部分抵消,而通过上调DDX54和抑制miR-520d-5p完全恢复。gydF4y2Ba
PITPNA-AS1通过招募DDX54提高SIK2 mRNA的稳定性。gydF4y2Ba一个gydF4y2Ba采用RT-qPCR检测经放线菌素D处理的sh-PITPNA-AS1#1或sh-NC转染的HCC1937和MDA-MB-468细胞SIK2 mRNA的稳定性。gydF4y2BaBgydF4y2Ba利用starBase预测了PITPNA-AS1 (CLIP Data≥2)和SIK2 (CLIP Data≥5)的4种常见rbp。gydF4y2BaCgydF4y2Ba采用RNA拉下法检测PITPNA-AS1与DDX54、FMR1、IGF2BP1、IGF2BP2的结合能力。gydF4y2BaDgydF4y2Ba通过RT-qPCR和western blot检测DDX54对HCC1937和MDA-MB-468细胞的沉默效果。gydF4y2BaEgydF4y2Ba采用RT-qPCR方法评价放线菌素D处理后0、4、8、12 h转染细胞SIK2 mRNA的稳定性。gydF4y2BaFgydF4y2Ba通过RIP实验验证DDX54蛋白与PITPNA-AS1或SIK2 mRNA的相互作用。gydF4y2BaGgydF4y2BaRIP实验显示,PITPNA-AS1敲除后,DDX54蛋白与SIK2 mRNA的结合发生了变化。gydF4y2BaHgydF4y2BaRT-qPCR和western blot检测PITPNA-AS1沉默后DDX54在mRNA和蛋白水平上的表达。I RT-qPCR和western blot验证了DDX54的过表达效率。gydF4y2BaJgydF4y2BaRT-qPCR和western blot检测转染相应质粒的HCC1937和MDA-MB-468细胞中SIK2的表达。**p < 0.01, ***p < 0.001gydF4y2Ba
PITPNA-AS1通过miR-520d-5p和DDX54上调SIK2在TNBC细胞中发挥致癌作用gydF4y2Ba
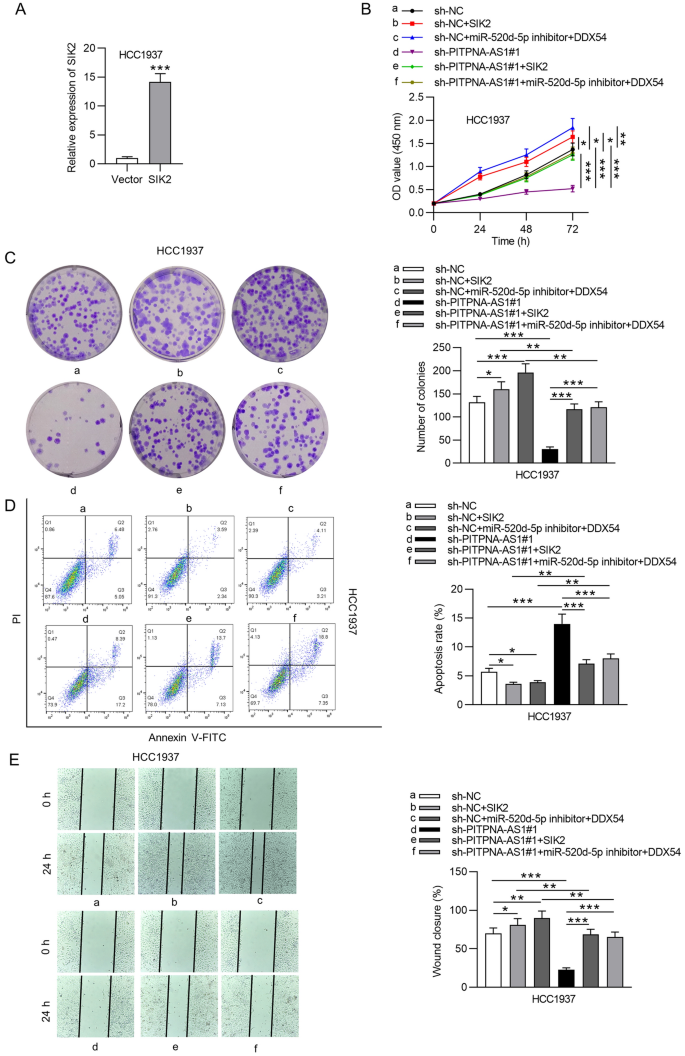
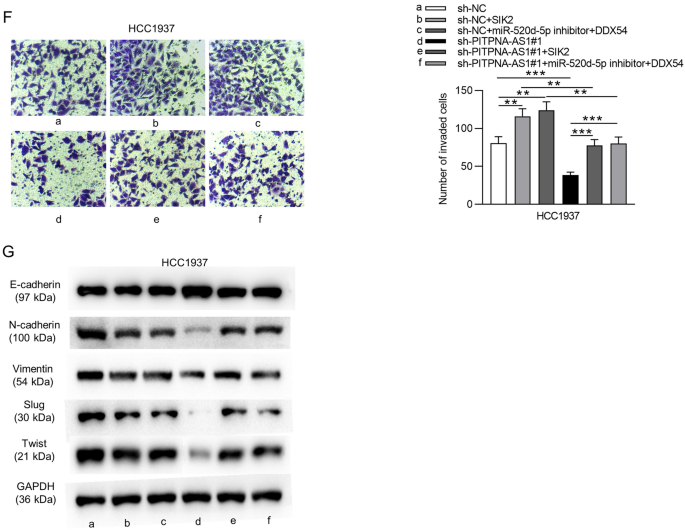
最后,我们进行抢救试验,进一步验证上述PITPNA-AS1的机制能够推动TNBC的发生发展。因此,转染pcDNA3.1/SIK2可上调HCC1937细胞中SIK2的表达。gydF4y2Ba7gydF4y2BaA).通过CCK-8和集落形成检测结果,我们发现PITPNA-AS1缺失抑制了细胞活力和增殖,转染pcDNA3.1-SIK2或共转染miR-520d-5p inhibitor + pcDNA3.1-DDX54促进了细胞活力和增殖。PITPNA-AS1缺失对hcc1937细胞活力和增殖的抑制作用通过SIK2过表达或抑制miR-520d-5p后上调的DDX54得以挽救(图5)。gydF4y2Ba7gydF4y2BaB, C).转染pcDNA3.1-SIK2或共转染miR-520d-5p inhibitor + pcDNA3.1-DDX54可抑制HCC1937细胞凋亡率。上调SIK2可抑制PITPNA-AS1沉默诱导的细胞凋亡;抑制miR-520d-5p和过表达DDX54的共同作用也逆转了这一效应(图5)。gydF4y2Ba7gydF4y2BaD)。此外,通过过表达SIK2或抑制miR-520d-5p表达同时上调DDX54, PITPNA-AS1下调抑制的细胞迁移和侵袭被抵消(图。gydF4y2Ba7gydF4y2Ba此外,SIK2过表达或miR-520d-5p抑制以及DDX54上调挽救了沉默的PITPNA-AS1对EMT过程的影响(图5)。gydF4y2Ba7gydF4y2BaG)。gydF4y2Ba
PITPNA-AS1通过靶向miR-520d-5p/DDX54/SIK2轴促进TNBC细胞过程。gydF4y2Ba一个gydF4y2Ba通过RT-qPCR检测转染pcDNA3.1 (Vector)或pcDNA3.1/SIK2 (SIK2)的HCC1937细胞系中SIK2的表达。gydF4y2BaBgydF4y2Ba,gydF4y2BaCgydF4y2Ba用CCK-8和集落形成试验检测转染细胞的活力和增殖。gydF4y2BaDgydF4y2Ba流式细胞仪检测显示各组均有细胞凋亡。gydF4y2BaEgydF4y2Ba,gydF4y2BaFgydF4y2Ba用伤口愈合试验和Transwell试验测定转染后细胞的迁移和侵袭情况。gydF4y2BaGgydF4y2Bawestern blot检测各组HCC1937细胞系中emt相关蛋白。*p < 0.05, **p < 0.01, ***p < 0.001gydF4y2Ba
MYBL2在TNBC中转录激活PITPNA-AS1的表达gydF4y2Ba
转录激活是导致基因异常表达的主要原因。因此,我们探讨了PITPNA-AS1的上调是否与此有关。通过UCSC (gydF4y2Bahttp://genome.ucsc.edu/gydF4y2Ba),发现了一些可能与PITPNA-AS1启动子结合的转录因子。RT-qPCR分析显示,TNBC细胞中E2F6、PAX5、FOXP2、ELK5、MYBL2、YY1的表达水平高于MCF10A细胞gydF4y2Ba1gydF4y2Ba:图S1A)。随后,通过转染pcDNA3.1过表达质粒,在HCC1937和MDA-MB-468细胞中上调上述转录因子进行进一步分析(附加文件gydF4y2Ba1gydF4y2Ba:图S1B)。从附加文件观察gydF4y2Ba1gydF4y2Ba:图S1C,我们发现PITPNA-AS1的表达主要在mybl2过表达的细胞中升高。进一步,我们敲除MYBL2,发现MYBL2沉默导致PITPNA-AS1表达水平下调(附加文件)gydF4y2Ba1gydF4y2Ba:图S1D)。因此,我们假设在TNBC细胞中PITPNA-AS1可以被MYBL2转录激活。基于Jaspar在线数据库,预测了MYBL2 DNA基体和四个推定的MYBL2在PITPNA-AS1启动子上的结合位点(附加文件)gydF4y2Ba1gydF4y2Ba:图S1E)。ChIP检测显示MYBL2与PITPNA-AS1启动子结合在P1区段,该区段包含位点1和位点2(附加文件)gydF4y2Ba1gydF4y2Ba:图S1F)。接下来,荧光素酶报告子实验进一步证实MYBL2与PITPNA-AS1启动子的相互作用。我们发现含有PITPNA-AS1启动子WT和Mut 1序列的载体在过表达MYBL2后荧光素酶活性增强,而含有PITPNA-AS1启动子Mut 2和Mut 1/2序列的载体在过表达MYBL2后荧光素酶活性保持不变,这表明MYBL2与PITPNA-AS1启动子在位点2发生相互作用(附加文件)gydF4y2Ba1gydF4y2Ba:图S1G)。gydF4y2Ba
讨论gydF4y2Ba
越来越多的文献报道lncRNAs通过海绵miRNA或招募RBPs参与各种人类癌症的发生发展[gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba].尽管异常表达的lncrna在TNBC进展中的重要作用已被强调[gydF4y2Ba14gydF4y2Ba,gydF4y2Ba25gydF4y2Ba], lncRNA PITPNA-AS1在TNBC中的潜在作用和机制尚不明确,值得进一步探讨。我们的研究表明,PITPNA-AS1在TNBC组织和细胞中表达高于对照组织和细胞,且主要分布在TNBC细胞的细胞质中。降低PITPNA-AS1抑制TNBC细胞增殖,促进细胞凋亡,抑制细胞迁移和侵袭。此外,PITPNA-AS1的沉默抑制了体内异种移植瘤的生长。总的来说,PITPNA-AS1在TNBC中表现出致癌特性。gydF4y2Ba
mirna是另一类长度约为22-24个核苷酸的ncrna,在癌症进展中发挥重要作用[gydF4y2Ba26gydF4y2Ba,gydF4y2Ba27gydF4y2Ba].已有证据表明lncrna可与特异性miRNA结合促进或抑制肿瘤的发生或发展[gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba].本研究通过生物信息学分析和一系列分子机制实验鉴定miR-520d-5p,进行进一步探索。此前,研究发现miR-520d-5p在胶质瘤中通过靶向PTTG1抑制细胞增殖和细胞周期[gydF4y2Ba30.gydF4y2Ba].在胃癌中,miR-520d-5p是细胞增殖和存活的重要调节因子[gydF4y2Ba31gydF4y2Ba].此外,miR-520d-5p在结直肠癌中发挥抗癌基因作用,通过调控CTHRC1抑制肿瘤生长和转移[gydF4y2Ba32gydF4y2Ba].在本研究中,我们发现miR-520d-5p在TNBC中与PITPNA-AS1具有结合能力。此外,miR-520d-5p在TNBC组织和细胞系中表达水平较低。这些发现表明PITPNA-AS1在TNBC中隔离miR-520d-5p。gydF4y2Ba
盐诱导激酶2 (SIK2)已被证实在多种癌症中发挥促肿瘤功能,包括TNBC [gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba].然而,在TNBC细胞中SIK2和miR-520d-5p(或PITPNA-AS1)之间的关系需要进一步研究。在我们的研究中,证实了SIK2在TNBC中被miR-520d-5p直接靶向。更重要的是,我们的研究结果表明,PITPNA-AS1不仅通过海绵miR-520d-5p正向调节SIK2的表达。gydF4y2Ba
DEAD-box解旋酶54 (DDX54)被认为是rbp的成员,并被报道为一些癌症的致癌基因[gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba].在本研究中,PITPNA-AS1的下调抑制了SIK2 mRNA的稳定性。DDX54在TNBC细胞中与PITPNA-AS1(或SIK2)相互作用。此外,恢复实验表明,被沉默的PITPNA-AS1抑制的TNBC细胞过程被SIK2过表达或miR-520d-5p抑制和DDX54上调的共同作用所挽救。gydF4y2Ba
新近的研究表明,转录因子介导的转录调控是lncRNAs异常表达的主要原因[gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba].MYB原癌基因样样2 (MYBL2)是肺腺癌的转录因子[gydF4y2Ba39gydF4y2Ba].我们的研究发现,MYBL2正向调控PITPNA-AS1的表达,并与PITPNA-AS1启动子结合,这表明TNBC中PITPNA-AS1的上调是由MYBL2转录诱导的。gydF4y2Ba
结论gydF4y2Ba
总之,我们的研究表明,mybl2诱导的PITPNA-AS1通过miR-520d-5p和DDX54上调SIK2的表达,从而驱动TNBC细胞过程。这为进一步探索TNBC治疗策略提供了有意义的理论依据。gydF4y2Ba
数据和材料的可用性gydF4y2Ba
这项研究的所有数据都可以在这篇发表的文章中找到。gydF4y2Ba
缩写gydF4y2Ba
- lncRNAs:gydF4y2Ba
-
长非编码rnagydF4y2Ba
- PITPNA-AS1:gydF4y2Ba
-
LncRNA PITPNA反义RNAgydF4y2Ba
- 公元前:gydF4y2Ba
-
乳腺癌gydF4y2Ba
- 呃:gydF4y2Ba
-
雌激素受体gydF4y2Ba
- 公关:gydF4y2Ba
-
孕激素受体gydF4y2Ba
- TNBC:gydF4y2Ba
-
三阴性乳腺癌gydF4y2Ba
- ncRNAs:gydF4y2Ba
-
非编码rnagydF4y2Ba
- 龙头:gydF4y2Ba
-
竞争内源性RNAgydF4y2Ba
- microrna的:gydF4y2Ba
-
微gydF4y2Ba
- RBPs:gydF4y2Ba
-
rna结合蛋白gydF4y2Ba
参考文献gydF4y2Ba
卡多so F, Harbeck N, Barrios CH, Bergh J, Cortés J, El Saghir N,等。乳腺癌的研究需要。中国科学院学报,2017;28(2):208-17。gydF4y2Ba
翟倩,李宏,孙玲,袁勇,王霞。应用生物信息学分析鉴别三阴性乳腺癌与非三阴性乳腺癌差异表达基因。中华癌症杂志,2019;26(6):784-91。gydF4y2Ba
张晓明,张晓明,张晓明。三阴性乳腺癌的研究进展。中华癌症杂志2016;17(4):1595-608。gydF4y2Ba
雷迪总经理,苏雷什PK,派RR。三阴性乳腺癌的临床病理特征。中国临床诊断杂志,2017;11(1):Ec05-8。gydF4y2Ba
塔库尔V,库蒂RV。纳米otheranotics治疗三阴性乳腺癌的最新进展。中国临床肿瘤学杂志,2019;38(1):429。gydF4y2Ba
Perez DS, Hoage TR, Pritchett JR, Ducharme-Smith AL, Halling ML, Ganapathiraju SC,等。长且大量表达的非编码转录本在癌症中被改变。胡文杰,2008;17(5):642-55。gydF4y2Ba
Guttman M, Donaghey J, Carey BW, Garber M, Grenier JK, Munson G,等。lincRNAs在控制多能性和分化的电路中起作用。自然。2011;477(7364):295 - 300。gydF4y2Ba
彭婷,李志强,李志强。长链非编码rna的进化与功能。细胞。2009;136(4):629 - 41。gydF4y2Ba
项俊,郭松,姜松,徐勇,李娟,李林,等。沉默长链非编码RNA MALAT1促进胶质瘤细胞凋亡。中华医学杂志,2016;31(5):688-94。gydF4y2Ba
埃文斯JR,冯飞,覃乃燕AM。暗物质的光明面:癌症中的lncrna。中华临床医学杂志,2016;26(8):379 - 379。gydF4y2Ba
Gibb EA, Vucic EA, Enfield KS, Stewart GL, Lonergan KM, Kennett JY,等。人类癌症长非编码RNA转录组。PloS ONE。2011; 6 (10): e25915。gydF4y2Ba
Moran VA, Perera RJ, Khalil AM。哺乳动物长链非编码rna的新功能和机制范式。中国生物医学工程学报,2012;40(14):6391-400。gydF4y2Ba
申伟,陈杰,卓伟文,肖明山,何长泉,王霞,等。长链非编码RNA NEAT1通过调节化疗耐药和癌干性在三阴性乳腺癌中发挥致癌作用。细胞死亡,2019;10(4):270。gydF4y2Ba
古丁AJ,张B, Gunawardane L, Beard A, Valadkhan S, Schiemann WP。lncRNA BORG促进三阴性乳腺癌的生存和化疗耐药。致癌基因。2019;38(12):2020 - 41。gydF4y2Ba
罗玲,唐辉,凌玲,李楠,贾旭,张震,等。lncRNA在三阴性乳腺癌中通过阻止spop介导的c-Myc降解激活MTDH-Twist1信号。致癌基因。2018;37(47):6166 - 79。gydF4y2Ba
科普F,门德尔JT。长链非编码rna的功能分类与实验解剖。细胞。2018;172(3):393 - 407。gydF4y2Ba
王一刚,王涛,石敏,翟斌。Long非编码RNA EPB41L4A-AS2通过海绵miR-301a-5p和靶向FOXL1抑制肝细胞癌的发展。中国临床肿瘤学杂志,2019;38(1):529。gydF4y2Ba
霍华,田娟,王锐,李艳,曲春,王楠。龙非编码RNA NORAD上调SIP1表达,促进宫颈癌细胞增殖和侵袭。Biomed Pharmacother, 2018; 106:1454-60。gydF4y2Ba
邹智,马涛,何霞,周健,马辉,谢敏,等。长基因间非编码RNA 00324通过与HuR结合稳定FAM83B表达促进胃癌细胞增殖。细胞死亡,2018;9(7):717。gydF4y2Ba
杨震,董旭,蒲敏,杨华,常伟,纪峰,等。LBX2- as1 /miR-219a-2-3p/FUS/LBX2正反馈环路有助于胃癌的增殖。胃癌。2020; 23(3): 449 - 63。gydF4y2Ba
孙杰,张勇,李波,董勇,孙超,张峰,等。PITPNA-AS1取消了miR-876-5p对WNT5A的抑制,促进了肝细胞癌的进展。细胞死亡,2019;10(11):844。gydF4y2Ba
Livak KJ, Schmittgen TD。利用实时定量PCR和2(-Delta Delta C(T))方法分析相关基因表达数据。方法。2001;25(4):402 - 8。gydF4y2Ba
冯红,魏波,张颖。龙非编码RNA HULC通过下调microRNA-15a促进胰腺癌细胞增殖、迁移和侵袭。中国生物医学杂志,2019;26(6):891 - 891。gydF4y2Ba
文东,黄忠,李忠,唐旭,文新,刘杰,等。LINC02535与PCBP2共同作用,通过稳定RRM1 mRNA调节宫颈癌DNA损伤修复。中国生物医学工程学报,2015;29(3):344 - 344。gydF4y2Ba
唐健,钟刚,张宏,于波,魏峰,罗林,等。LncRNA DANCR通过激活RXRA的丝氨酸磷酸化上调PI3K/AKT信号。细胞死亡,2018;9(12):1167。gydF4y2Ba
王斌,陆永飞,史荣荣,冯永德,赵小东,陆志平,等。MiR-26b通过下调Pgp调控人结直肠癌5- fu耐药。中国癌症杂志,2018;8(12):2518-27。gydF4y2Ba
周勇,任宏,戴斌,李娟,尚林,黄娟,等。肝细胞癌来源的外泌体miRNA-21通过将肝细胞星状细胞转化为癌症相关成纤维细胞来促进肿瘤进展。中国临床癌症杂志,2018;37(1):324。gydF4y2Ba
张艳,杨光,罗玉龙非编码RNA PVT1通过靶向miR-200a促进胶质瘤细胞增殖和侵袭。中华实验医学杂志2019;17(2):1337-45。gydF4y2Ba
李高J,刘L, G, Cai M,谭C,汉族人X, et al。LncRNA GAS5通过调控miR-106b/IER3轴赋予宫颈癌细胞无线电敏感性。中国生物医学杂志,2019;26(2):994 - 1001。gydF4y2Ba
支涛,姜坤,徐旭,于涛,吴伟,聂娥,等。MicroRNA-520d-5p通过直接靶向PTTG1抑制人胶质瘤细胞增殖并诱导细胞周期阻滞。中国地质大学学报(自然科学版),2017;9(11):4872-87。gydF4y2Ba
李涛,郭辉,赵霞,金健,张玲,李辉,等。胃癌细胞的增殖和存活是由亲环素B/STAT3/miR-520d-5p信号反馈环实现的。罐头决议2017;77(5):1227-40。gydF4y2Ba
闫玲,于俊,谭峰,叶桂婷,沈志智,刘华,等。sp1介导的microRNA-520d-5p通过靶向CTHRC1抑制结直肠癌肿瘤生长和转移中华癌症杂志,2015;5(4):457 - 457。gydF4y2Ba
赵娟,张鑫,高涛,王松,侯勇,袁鹏,等。SIK2通过PI3K/Akt信号通路促进卵巢癌细胞脂肪酸和胆固醇的合成,促进肿瘤生长。细胞死亡,2020;11(1):25。gydF4y2Ba
Maxfield KE, Macion J, Vankayalapati H, Whitehurst w。SIK2限制自噬通量支持三阴性乳腺癌生存。分子生物学杂志,2016;36(24):3048-57。gydF4y2Ba
张艳,郭慧,张慧。SNHG10/DDX54/PBX3反馈环对胃癌细胞生长的影响《挖掘疾病科学》2020。gydF4y2Bahttps://doi.org/10.1007/s10620-020-06488-9gydF4y2Ba.gydF4y2Ba
米列克,伊玛米,穆克吉,博尔托利,尤兹纳尔,哈扎比斯,等。DDX54在DNA损伤应答过程中调控转录组动态。中国生物工程学报,2017;27(8):1344-59。gydF4y2Ba
乔凯,宁松,万林,吴华,王强,张旭,等。LINC00673被YY1激活,通过miR-515-5p/MARK4/Hippo信号通路促进乳腺癌细胞增殖。临床癌症研究进展。2019; 38(1): 418。gydF4y2Ba
Tran DDH, Kessler C, Niehus SE, Mahnkopf M, Koch A, Tamura T.肝癌Myc靶基因,长基因间非编码RNA, Linc00176通过检测抑癌microRNAs调节细胞周期和细胞存活致癌基因。2018;37(1):75 - 85。gydF4y2Ba
刘超,张玉华,黄涛,蔡颖。肺腺癌重组转录因子的鉴定。中国医学杂志,2017;83:52-7。gydF4y2Ba
确认gydF4y2Ba
不适用。gydF4y2Ba
资金gydF4y2Ba
本工作得到山西省奖学金资助委员会科研项目(No. 2020-188)、2018年湖南省科学卫生联合基金(No. 2018JJ6122)、中国国际医学基金(No. 2018JJ6122)的资助。z - 2017 - 26 - 1902 - 2)。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
FX和JJG参与了文献检索、数据分析和解读,并撰写了稿件。BLL、PBY、LHW、YY提供项目指导,修改稿件,提供实验场地和资金。所有作者都阅读并批准了最终的手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
伦理批准并同意参与gydF4y2Ba
所有参与者均获得书面知情同意。该研究符合《赫尔辛基宣言》,并获得了山西医科大学第二医院伦理委员会的批准。gydF4y2Ba
发表同意书gydF4y2Ba
不适用。gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者宣称他们之间没有利益冲突。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
补充信息gydF4y2Ba
附加文件1:图S1。gydF4y2Ba
MYBL2通过转录诱导PITPNA-AS1的上调。(A) TNBC细胞中PITPNA-AS1水平较高的6种转录因子。(B)上述转录因子在HCC1937和MDA-MB-468细胞中的过表达效率。(C)通过RT-qPCR检测6个预测转录因子过表达后PITPNA-AS1的表达情况。(D)通过RT-qPCR检测sh-MYBL2或sh-NC转染的TNBC细胞中MYBL2和PITPNA-AS1的表达水平。(E) MYBL2 DNA基序和MYBL2与PITPNA-AS1启动子的结合位点。(F)采用ChIP法检测MYBL2与PITPNA-AS1启动子的结合。(G)荧光素酶报告试验证实了MYBL2与PITPNA-AS1启动子之间的结合位点。**p < 0.01, ***p < 0.001。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.创作共用公共领域奉献弃权书(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本条所提供的资料,除非在资料的信用额度中另有说明。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
刘,B,姚,P,肖,F。gydF4y2Baet al。gydF4y2Bamybl2诱导的PITPNA-AS1通过miR-520d-5p和DDX54上调SIK2在三阴性乳腺癌中发挥致癌功能。gydF4y2Ba翻译医学杂志gydF4y2Ba19gydF4y2Ba, 333(2021)。https://doi.org/10.1186/s12967-021-02956-6gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12967-021-02956-6gydF4y2Ba
关键字gydF4y2Ba
- PITPNA-AS1gydF4y2Ba
- mir - 520 d - 5 - pgydF4y2Ba
- DDX54gydF4y2Ba
- SIK2gydF4y2Ba
- TNBCgydF4y2Ba