摘要
背景
在我们的遗传性癌症区域项目的背景下,满足标准的个体进行生殖系突变检测,随后建立临床管理。我们的标准诊断方法侧重于对几个典型的高风险基因进行测序,这种方法通常会产生信息不足的遗传结果。本研究旨在检验按需面板提供的提高产量。
方法
我们设计了一个按需面板,用于分析总共128例遗传性乳腺癌和卵巢癌(HBOC)和遗传性非息肉病性结直肠癌(HNPCC)中35个与遗传癌症易感性相关的基因。
结果
在两种常规(Brca2 mlh1 msh2 pms2)及非常规性(Atm, blm, brip1, chek2, mutyth)测试的基因。筛选扩展到35个基因,这些基因由携带几种未知意义变异(VUS)的患者提供,最多可达6种。此外,我们在RNA水平上证实了先前未报道的剪接中断BRIP1剪接变异。使用按需面板,我们确定了18个致病突变携带者,其中7个是传统分析无法注意到的。
结论
我们的研究结果加强了NGS基因面板在诊断常规中的效用,以提高基因检测的性能,特别是在具有重叠癌症表型的家庭个体中。
背景
癌症易感基因的种系突变携带者在所有癌症病例中占很小但相关的比例。这是因为当遗传易感性被确定后,可以在家庭中实施有效的临床管理[1].传统的遗传筛查是基于对经典的高外显率基因的分析,这些基因只能解释少数家族的遗传易感性[2].此外,这一过程既耗时又昂贵,强调了不仅需要增加分析基因的数量,而且需要一种可扩展的方法。在这方面,基因癌症小组的发展,大规模平行测序技术的应用,正在取代顺序的单基因检测。使用基因面板,可以同时探索几个基因,增加了发现因果突变的机会。此外,基因面板代表了一种可负担的下一代测序(NGS)应用于临床实践。关于基因面板实施的一个关键决定是寻找特定遗传综合征的正确基因。为了满足这一需求,定制面板已经开始提供。
这些定制的面板正在大量诊断实验室中使用,提供了讨论实施过程和增加突变产量的机会[3.,4,5].一般来说,由于表型的重叠,癌症病例的队列可以满足不同的证候的标准。在实际水平上,该小组应包括候选基因,与样品的表型相匹配。为了评估这些自订面板的实施情况和优化其用途,对结果进行彻底的分析是必不可少的[6].
我们的目标是将与HBOC(遗传性乳腺癌和卵巢癌)和HNPCC(遗传性非息肉性结肠癌)相关的按需基因面板的研究纳入Castile & Leon遗传社区癌症项目的一个参考实验室的临床常规。
材料与方法
病人
本研究共纳入128例指标病例。该组包括72例HBOC(58例乳腺癌和14例卵巢癌)和57例HNPCC,阿姆斯特丹标准阳性。他们是由卡斯蒂利亚和莱昂(西班牙)地区遗传性癌症预防计划选定的。收集伦理委员会批准、知情同意、家族史和临床特征。
DNA和RNA提取
罗氏MagNaPure自动提取外周血基因组DNA®按照制造商的说明,使用“MagNA纯紧凑型核酸分离试剂盒I-Large Volume”(Roche Diagnostics, Penzberg, Germany)。使用GeneMATRIX人血液RNA纯化试剂盒(EURx, Gdánsk,波兰)从外周血淋巴细胞中提取RNA。
基因突变筛查
使用Ion S5系统(ThermoFisher Scientific, Waltham, MA, USA)上的按需研究分析(on - demand Research Assay)对所有DNA样本进行种系突变筛选。使用Qubit 3.0荧光仪(Thermo Fisher Scientific)检查每个DNA样本的浓度。然后将输入DNA的浓度调整为50 pmol。
文库和模板制备使用自动化Ion Chef系统进行,然后根据制造商的说明,使用Ion 520芯片(全部为Thermo Fisher Scientific)在Ion S5中测序。
测序结果与hg19人类参考基因组比对,并使用Ion Reporter Software Version 5.10 (Thermo Fisher Scientific)进行分析。
基因种系突变检测的变异频率截止值定义为20%,这是先前研究推荐的[7].
按需研究法检测单核苷酸变异(snv)和小缺失的灵敏度为99.85%,特异性为100%,假阳性率为0%,准确率为99.99%。
桑格测序
采用直接自动Sanger测序对大量平行测序检测结果进行确认。为此,我们在ABI 3100 DNA测序仪上使用了BigDye终结者测序试剂盒v3.1 (Applied Biosystems)。应用生物系统公司)。在可能的情况下进行了共分离研究。
RT PCR应承担的
当Human Splicing Finder (HSF)软件(http://www.umd.be/HSF3/)预测由突变引起的剪接中断,我们进行了基于dna的分析。为此,根据制造商的协议,使用转录第一链cDNA合成试剂盒(罗氏)将从淋巴细胞中分离的RNA逆转录成cDNA。然后,我们通过进行PCR来评估可能对转录的影响,随后在低熔点2%琼脂糖凝胶中分离产物,并用Red Safe™染色。然后分离条带,用NucleoSpin提取DNA®凝胶和PCR Clean‐up (Macherey‐Nagel, d
In-silico分析
根据ExAC数据,蛋白质注释和次要等位基因频率(MAF) < 0.01的突变使用Cancer Genome Interpreter (https://www.cancergenomeinterpreter.org)、Human Splicing Finder TM 3.0 (HSF)和(http://www.umd.be/HSF3)。为了评估变异对蛋白质功能的潜在影响,我们使用了联合注释依赖耗尽(CADD)方法,该方法释放了与排名最高的致病性相关的分级c分数:CADD- score -10意味着您处于破坏性突变的前10%,CADD- score -20,前1%,CADD- score -25,前0.5%和CADD- score -30, 0.1%。CADD通过对比在自然选择中幸存下来的变异和模拟突变,将多种注释整合到一个分数中[8].
变体的分类
如果变异起源于一个过早终止密码子,它们位于规范的剪接位点,或者有文献证据,那么它们被归类为有害变异。剩余变异的潜在危害性使用联合注释依赖损耗(CADD)方法进行评估。我们认为cad得分为bbbb20的变体是值得考虑的(破坏性变体的前1%)。
基因型-表型相关性及统计分析
检测了个人/家族数据与样本突变谱之间的基因型-表型相关性。使用卡方检验来研究分类变量之间的关系,使用Spearman 's Rank相关系数来评估两个变量之间的关联强度。使用R统计编程语言(v3.5.1)进行统计测试。
结果
我们将一个包含35个与癌症易感综合征相关基因的按需小组,特别是HBOC和HNPCC,整合到我们的常规基因检测中(表1)1)。所有这些基因之前都被确定为易患癌症的基因。随需应变面板的分析简化和标准化了实验室工作流程的单一程序,以检测遗传性综合征。本研究纳入的128个样本在连续5次实验中测序。
所有样品的覆盖均匀度均大于90%。总对齐reads的平均值为1,040,207 (89%),50 ×时目标覆盖率的平均值为88.6%,区域覆盖深度的中位数为206×(范围:29-549)。
然后在生物信息学分析中对测序结果进行筛选,只分析符合质量标准的选定变体。设法充分覆盖,以确保roi内的所有基地至少覆盖30倍。调整了Ion报告器管道参数,以确保更好地控制变量调用质量。在第一种方法中,使用高外显率基因的不同突变的训练集来评估面板的性能。所有的变异都被正确排序和注释(数据未显示)。
在18例癌症病例(14%)中共鉴定出18种致病性或可能致病性变异(PV/LPV),影响9种不同的基因,每种遗传性癌症病症目前具有临床效用(表1)2)。
这些变异包括13个单核苷酸变异(snv), 4个缺失和1个插入,均发生在杂合中,导致7个错义变异,5个移码变异,3个无义变异(导致过早终止密码子)和3个剪接变异(1个尚未在参考数据库中报道)。该PV组包括6名HBOC和12名HNPCC,符合阿姆斯特丹标准。
关注受影响的基因,最常发生突变的基因是MUTYH有5个变异,其中4个是相同的单等位基因突变p.Gly396Asp。有趣的是,与我们的相比,ExAc中这种突变的MAF非常低,这表明在我们的人群中频率很高。在受影响的携带者中,乳腺癌、卵巢癌、结肠癌和胃癌是癌症类型。另一个MUTYH1例胃癌患者PV为p.Ala385fs。为自动取款机,Brca2, brip1, chek2和MSH2,在每个基因中发现两个pv。最后,在以下基因中鉴定出一个PV:BLM,一种和PMS2。在卵巢癌病例中发现了ATM PV (p.Ser1993Argfs和p.Arg447Ter)和BRIP1突变之一(C .1140 + 1G > C);BRCA2 (p.Val220fs和p.Ala938fs)、CHEK2 (c.593-1G > T和p.Thr476Met)、BLM (p.Gln548Ter)和其他BRIP1 PV (c.206-2A > G)在乳腺癌患者中被鉴定出来。有趣的是,在一名乳腺癌患者中检测到MLH1中的p.Pro747fs PV,在一名被诊断为皮肤癌的女性中检测到MSH2中的p.Gly71Arg。相反,正如预期的那样,另一个MSH2 PV (p.a g711ter)和PMS2突变(p.Ser46Ile)出现在结肠癌病例中。值得注意的是,携带ATM型PV的卵巢癌患者,以及携带CHEK2型PV的乳腺癌患者,其家族史均符合HNPCC和HBOC标准。
提出的一个问题是,与专门筛选BRCA和MMR基因相比,通过应用按需面板,我们是否提高了检测潜力。对于这个问题,我们估计了面板设计的生产力,比较了传统筛选基因和其他基因中发现的PV的诊断产量。通过这种比较分析,我们的目的是确定PV的增加是由于纳入了与传统分析不同的其他基因(BRCA基因和MMR基因)。此外,为了根据遗传性癌症综合征的类型评估该小组的适用性,我们对每种综合征、HBOC和HNPCC进行了分析。结果如图所示。1。
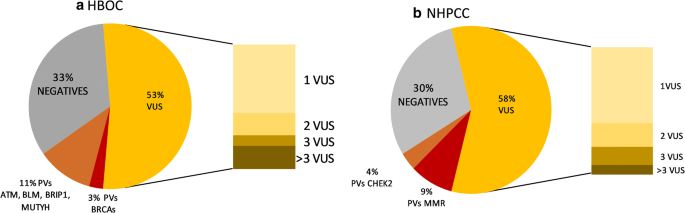
在HBOC的病例中,除BRCA1和BRCA2外,在其他基因中发现的PV占11%,而BRCA基因对应的PV占3%(特别是,所有PV均仅在BRCA2中发现)。相反,对于HNPCC,大多数PV仍然驻留在传统筛选的基因中,占9%,而其他基因占4%,特别是,CHEK2。如果我们只关注具有临床适用性的pv,设计的按需面板在HBOC的情况下比HNPCC更有效。这将提出一个前提,即不为两种不同的综合征使用相同的定制面板,因此需要考虑不同的设计。
正如预期的那样,35个基因面板意味着检测到大范围的VUS,占两种综合征遗传结果的50%以上。在HBOC病例中,29%的病例携带1个VUS;10%的病例携带2型;4%的病例为3,10%的病例超过3vus。对于HNPCC, 35%的病例携带一个VUS;11%的病例2;9%的病例为3,4%的病例超过3 VUS。
在临床中,VUS并不代表一个附加价值,然而,尽管如此,这些变异可以为基于多基因模型的癌症遗传易感性提供另一种解释。此外,VUS所代表的大百分比间接减少了HBOC和HNPCC两种综合征的阴性病例总数。
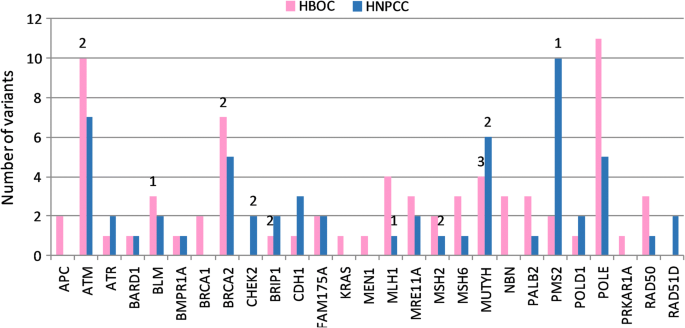
我们描述了PV和VUS沿不同基因和每种综合征的分布。在HBOC病例中,共鉴定出71个相关变异;对于HNPCC,有60个变种。在无花果。2,这是值得赞赏的Atm, brca2, mutyh, pole和FAM175A突变频繁,在HBOC和HNPCC中分别积累了58%和45%的变异;对于其他基因,变异分布是发散的。特别是,在HNPCC的一些基因中,发现了一个不相关的变体,损害了它们在我们的病例队列中的效用。有趣的是,几个VUS和PV在MUTYH在HBOC中,一个通常只在HNPCC背景下研究的基因。
为了在我们的诊断常规中利用NGS实施的结果,我们进一步研究了MAF < 0.01的VUS,使用CADD进行了计算机分析。CADD分数bbb20表示可能由变体(附加文件)引起的功能影响1:表1)。
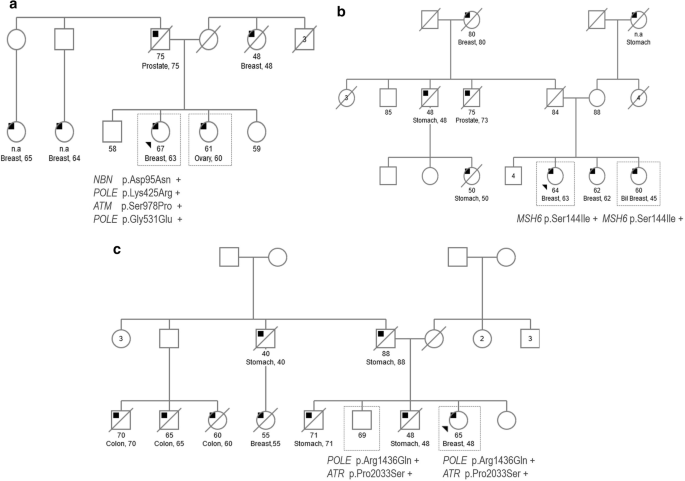
我们能够在来自3个家族的有限数量的个体中分析VUS的分离,通过累积效应增加更多关于它们在表型中可能作用的信息(图2)。3.)。在A家族的病例中,在一个62岁被诊断患有乳腺癌的妇女中,检测到四种对致病性有相互矛盾解释的变异。四个变体中的两个(NBN公司禁止p.Asp95Asn和极p.Lys425Arg)在其患卵巢癌的姐妹中也检测到。在B族中,VUS在MSH6p.Ser144Ile与两个被分析的乳腺癌病例分离,其中一个是双侧的,这是遗传性癌症的标志。在C家族中,由于不同的癌症病例都已去世,我们只能对指示病例的一个健康的兄弟进行VUS检测(p.a g1436gln)极p.Pro2033Ser inATR);他们两人携带两种型号。
有趣的是,一些患者积累了几个高cadd评分的变体:11例2 VUS, 8例3 VUS, 3例4 VUS, 5例5 VUS, 1例共携带6 VUS。根据多基因风险模型,这些具有预测功能影响的罕见错义变异将具有累积效应。值得注意的是,通过检查家族史,我们发现携带多个VUS的指标病例属于肿瘤类型多样性高的家族。数字4汇编研究队列的家族史和有关各自VUS的信息,包括CADD评分和所发现的基因。
高CADD评分变异的癌症病史与遗传结果的相关性代表携带两个或更多高CADD分数VUS的样本的个人和家族史。对于每个变异,cadd评分和受影响的基因都被显示出来。索引病例及家族成员的不同肿瘤类型编码如下:Br,乳腺;机汇,卵巢;上校,结肠;锅,胰腺;气,胃;你的甲状腺;公关,porstatic; End, endometrial; H&N, head and neck; Liv, liver; Kid, kidney; CNS, central nervous system; Skin, skin; Lun, lung and CUP, cancer of unknown primary origin. When multiple cancer cases in the family or several variants in the same gene existed, we used the “x” symbol to represent the multiplicative condition: 1x (1), 2x (2), 3x (3), etc
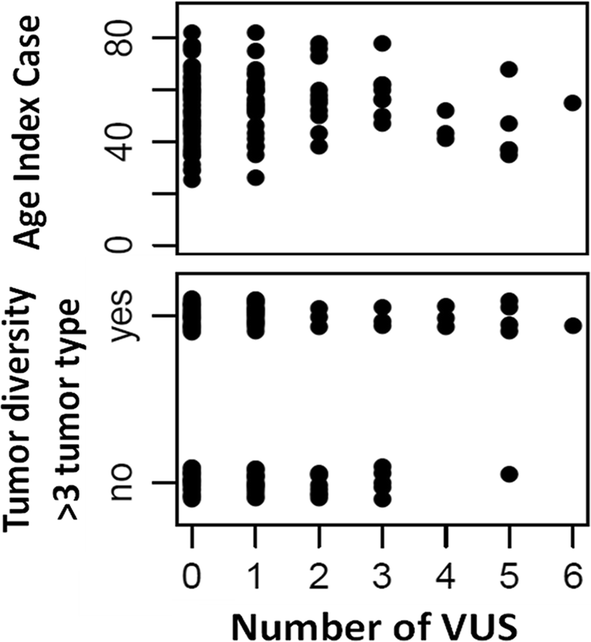
我们探讨了携带VUS的遗传状况(范围从1到6)与指标病例的诊断年龄之间的可能相关性。携带VUS的遗传状况(1 ~ 6)与指标病例的诊断年龄呈弱负相关(Spearman ρ =−0.376;无花果。5)。接下来,我们测试了家族中携带VUS的状况与肿瘤多样性之间是否存在关联。考虑“携带两个或两个以上VUS”和“家族中有三个或三个以上不同肿瘤类型”这两个二元变量,相关性无统计学意义(卡方检验,p = 0.348)。然而,当第一个变量被重新定义为“携带三个或更多VUS”时,相关性是显著的(卡方检验;p = 0.045)。
当数据可视化时,虽然没有统计学意义,但我们确实观察到一个趋势:携带较多VUS的病例(从4个VUS开始),并且属于含有3种以上不同癌症类型的家庭(更高的肿瘤多样性),呈现出更早的癌症发病(图2)。5)。
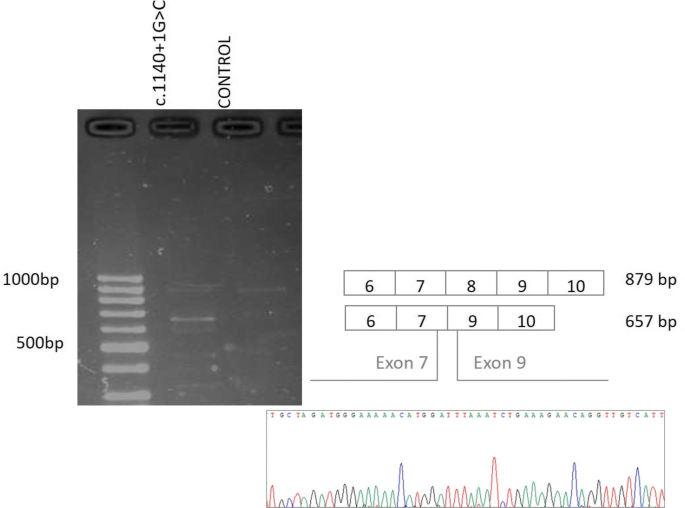
由于剪接中断可导致功能失调的蛋白质产物,我们在RNA水平上研究了那些影响规范或调节剪接位点的突变。对于位于典型剪接位点的突变,我们证实了先前未报道的C .1140 + 1G > C引起的剪接中断BRIP1突变。在无花果。6,我们可以观察到C .1140 + 1G > C变异载体的异常转录本的存在BRIP1;对照cDNA只出现一条与野生型转录物对应的条带,载体cDNA在琼脂糖凝胶中出现一条额外的条带。对异常转录本的测序随后证实了C .1140 + 1G > C的携带者外显子8的跳跃BRIP1变体。
此外,HSF表明c.4076 + 4T >g变异在BLM,和c.4473C >t自动取款机可能会改变剪接。此外,文献对这两种变异的致病性提出了相互矛盾的解释。具体来说,c.4076 + 4T >g变种BLM可以激活一个内含子隐体供体关键位点,一个新的供体位点。相反,c. 447c . b> . T自动取款机可能导致外显子增强子位点断裂。我们没有检测到c.4076 + 4T >g变异的任何异常转录BLM或为c.4473C >t in自动取款机。
为了有助于临床可操作性,特别是治疗选择,我们查阅了OncoKB数据库。我们正在寻找与患者突变谱相关的特定靶向药物的注册反应。可以认为自动取款机,BRCA2和CHEK2病原突变可能决定PARP抑制剂的敏感性,正如已经记录的那样。此外,在……的情况下自动取款机因此,DNA-PKc和ATR抑制剂预计是有效的。根据这一信息,共有6名患者可以从靶向治疗方案中受益。
讨论
为了在遗传性癌症的背景下应用成功的临床管理,确定遗传易感性的原因至关重要。传统筛选的高外显率基因的突变解释了少数病例[9];然而,需要新的策略来解释尚未解决的易感性。
新一代测序,特别是多基因面板的应用,已被证明在遗传性癌症诊断方面是成功的[4,10,11].NGS在我们实验室的实施证实了该技术的成本效益:按需面板使我们能够在4个连续实验中分析总共128个样品(每个实验5天,包括生物信息学分析),证实了可扩展性并大大缩短了周转时间[12].此外,使用靶向测序,我们确保了在感兴趣的区域有足够的覆盖,保证了健壮的变体调用[13].
在我们的研究中,超过50%的样本携带一个或多个VUS,其次是非信息性病例超过30%,PV为14%,与使用NGS多基因面板的其他组相似[4,14].
针对PV,基因筛查对患者有直接影响;一旦确定了遗传风险,就可以提供预防措施和管理[15].尽管临床可操作性受到指南数量减少的限制[16,17],对于大多数已确定的pv,可以进行遗传咨询[18,19].
提出的一个问题是,通过应用按需面板,我们是否增强了检测潜力,不仅检测BRCA和MMR基因的致病性突变,而且还检测其他传统上未筛查的基因。在这方面,按需小组使我们能够在传统的高外显率BRCAs和MMR基因筛选中未被注意到的10例中检测到PV。特别是,与HNPCC扩展分析的产量适度增加(3%)相比,HBOC中由于按需面板而增加的PV比率(11%)更为明显。这提出了一个前提,即需要根据病例的临床表型进行特定的设计。在这方面,对于表型重叠是否需要提供泛癌症小组还是癌症特定小组,实验室的意见不同[20.].一种保守但方便的策略似乎是使用表型驱动面板,对传统的高外显率BRCA和MMR基因进行机会性测试[6].尽管需要进行优化,但按需面板应用导致了中等外显率基因突变的报告,否则这些突变将无法被检测到[16].
基因面板检测提示VUS的鉴定[16,21].而VUS的分类在临床应用上是一个特殊的限制和挑战[22];在研究背景下,可以进一步研究这些VUS,以探索基因型-表型相关性,并使用多基因模型解释遗传易感性。一方面,限制性过滤器和硅工具可用于将注意力集中在减少的突变列表上。另一方面,在许多患者中同时存在多个VUS,这将使我们有机会使用多基因模型来解释遗传因果关系;几个VUS的累积功能影响可能会带来重大风险。其他作者将DNA修复基因中共同发生的变异与乳腺癌的早期发病联系起来,提出了一种累积效应[23].在我们的研究中,全球分析揭示了一个复杂的变异景观,其中每个个体可能产生功能影响的变异数量从1到6不等。与其他分析类似[24],我们的数据揭示了在不同基因中检测到的变异数量的多样性,但35个基因中只有5个积累了50%的VUS和pv。
由于计算机分析不能解决变异的临床意义[25,我们对有限数量的家庭进行了隔离研究。p.Asp95Asn突变NBN公司禁止和p.Lys425Arg in极与乳腺癌和卵巢癌病例分开(图2)。3.一个);VUS在MSH6p.Ser144Ile在一个HNPCC家族的两例乳腺癌中被鉴定出来,其中一例是双侧的。3.b)。虽然先前已在结肠癌病例中发现该变异[26],我们的分析提出了可能对乳腺癌表型的贡献。在一个有结肠、胃和乳腺肿瘤的家族中,p.a g1436gln In极p.Pro2033Ser inATR在一个乳腺癌病例和她的健康兄弟的索引病例中被确定。影响ATR功能的变异与结肠癌的高风险有关[27]及乳癌[28].关于突变极,它们对结直肠癌易感性的影响已经被描述[29].此外,p.Arg1436Gln在极cadd得分非常高,35分。因此,考虑到家族史,对未受影响的个体携带者进行程序控制内窥镜检查是值得的。另一方面,在该家族中发现的VUS,以及结肠癌、胃癌和乳腺癌病例的共存,突出了不同癌症表型之间的重叠表型。这使得单个基因的靶向分子检测不太有利。沿着这些思路,通过小组测试扩大研究基因的范围可能会带来意想不到的基因型-癌症表型相关性。
如前所述,尽管家庭分离研究可以产生强有力的数据来重新分类VUS,但家庭规模小,招募的probans数量有限,限制了其潜力[30.].然而,在临床常规中应该考虑家庭共分离研究,以增加更多的遗传发现信息。
虽然没有发现明显的基因型-表型关联,但可以指出一些值得注意的观察结果:携带多个VUS(特别是超过3个)的情况可能决定了病例中较年轻的诊断年龄和各自家庭中较高的肿瘤多样性。对大群体的分析可以让我们建立这些假定的相关性。
我们发现了一个新的剪接BRIP1C. 1140 + 1G >在clivar中,在同一位置有G >a的变化,这将其归类为可能致病。众所周知,影响剪接供体或受体位点的变异意味着蛋白质功能的丧失[31而BRIP1功能的丧失具有致病作用[32].为了深入了解这种变异的潜在影响,我们在RNA水平上进行了表征,证实了位于剪接供体位点的核苷酸变化导致了8号外显子的丢失。
剪接改变通常是外显子内含子侧翼区域突变的结果,但一些外显子变异可以影响mRNA的加工,引发功能改变[33].其机制可能是基于位于外显子区域的增强位点(ESE)或剪接沉默位点(ESS)的改变[34].因此,我们探索了HSF预测的两个可能的剪接干扰因子外显子变异对mRNA加工的可能影响:c.4076 + 4T > G变异BLM和c.4473C > T自动取款机。无论是BLM突变也不是自动取款机变异导致患者样本中的RNA加工异常。因此,尽管HSF算法已被证明是预测剪接变化的有效计算工具[35],只要有可能,就应该努力在RNA患者水平上证实这一预测。
如前所述,创建综合基因面板是为了增强pv的检测。有趣的是,根据家族史,一些pv聚集在与最初建立的表型没有直接关系的基因上。鉴于此,必须强调两点主要观察结果。首先,单等位基因PV inMUTYHc.1187G > A (p.Gly396Asp)在4个不相关的患者中被鉴定出来,与ExAC中登记的数据相比,频率显著高(0.002)。考虑到MUTYH是隐性基因[36],MUTYH-单等位基因- pv不能解释肿瘤表型;相反,最近的几项研究支持单等位基因突变在糖尿病中的可能作用MUTYH赋予风险[37用于结直肠癌、乳腺癌、胃癌和子宫内膜癌。特别是c.1187G . >a变异的患者MUTYH分别被诊断为卵巢癌、子宫内膜癌、结直肠癌和胃癌,强调PV在MUTYH对这些癌症类型来说,甚至是杂合的。其次,我们发现了两个有害的变化CHEK2, c.593-1克>T和p.Thr476Met,在两名患乳腺癌的患者中,一种与该基因中的PV相关的癌症类型[38].有趣的是,两名患者的个体表型,乳腺癌(与家族表型,HNPCC相反),将决定不同的筛选基因列表的偏好。然而,在这种情况下,癌症类型的重叠可能会促使我们考虑与家族综合征不完全相关的基因突变。相反,携带p.Pro747fs变异的患者一种47岁时得了乳腺癌。原则上,一种不会成为乳腺癌筛查的候选基因,但在本例中,她的家庭符合HNPCC标准的事实使我们能够确定患者的PV。
因此,筛选与其他癌症表型相对应的基因可以让我们有机会定义测试基因谱,特别是当家族史聚集了几种癌症类型,匹配不止一种遗传性癌症综合征时。
在肿瘤靶向治疗的新时代,药物反应记录可作为治疗选择的参考。事实上,许多拟议的治疗方法都是基于合成致命性原则,并结合突变概况,这将导致提高有效性和减少副作用。了解到基因检测结果的超越性取决于其对临床实践的转移,我们希望利用遗传信息进行治疗选择,使个性化医疗概念的应用成为可能[39,40].根据其他研究,PV在自动取款机,BRCA2和CHEK2可以决定PARP抑制剂的敏感性[41].此外,对于自动取款机PV与DNA-PKc和ATR抑制剂有合成致死性协同作用[42].这一信息可用于对我们的6例患者设计靶向治疗方案。
结论
在上述讨论的基础上,基因面板的实施可以快速、经济地改善受影响家庭的临床管理。此外,将分析扩展到其他基因,有机会发现罕见的变化,并提供不同癌症的可靠分子画像。
数据和材料的可用性
所有数据可根据要求提供。
缩写
- CADD:
-
结合注释依赖耗尽
- HBOC:
-
遗传性乳腺癌和卵巢癌
- HNPCC:
-
遗传性非息肉病性结直肠癌
- HSF:
-
人类剪接查找器
- 加:
-
次要等位基因频率
- 门店:
-
下一代测序
- 光伏/ LPV:
-
致病的或可能致病的变种
- SNVs:
-
单核苷酸变异
- VUS开头:
-
意义不明的变体
参考文献
贾伯杰,欧菲特。遗传性癌症易感性综合征。中华临床医学杂志。2016。https://doi.org/10.1200/JCO.2005.10.042。
李建平,李建平。高渗透遗传性癌症综合征研究进展。致癌基因。2004;23:6445 - 70。
Feliubadaló L, Tonda R, Gausachs M, Trotta j R, Castellanos E, López-Doriga A等。全外显子组测序和特别的设计了遗传性癌症基因检测面板。科学代表2017;7:37 . 984。
Ramírez-Calvo M, García-Casado Z, Fernández-Serra A, de Juan I, Palanca S, Oltra S,等。在遗传性癌症综合征的遗传诊断中实施大规模测序:瓦伦西亚社区遗传性癌症方案(FamCan-NGS)的诊断表现。遗传肿瘤临床实践。2019;17:3。
Soto JL, Blanco I, Díez O, García Planells J, Lorda I, Matthijs G,等。关于下一代测序在遗传性癌症基因诊断中的应用的共识文件。中华医学杂志,2018;51(1):80 - 90。
Feliubadaló L, López-Fernández A, Pineda M, Díez O, Del Valle J, Gutiérrez-Enríquez S等。BRCA1、BRCA2和错配修复基因的机会性检测提高了表型驱动的遗传性癌症基因面板的产量。中华医学杂志,2019;45(5):391 - 391。
申顺,金勇,哲欧赫,刘宁,李世涛,罗崔杰,等。用于BRCA1和BRCA2基因检测的离子流s5xl测序仪和在线工作流程的验证和优化。Oncotarget。2017;8:34858 - 66。
李建民,李建民,李建民,李建民,李建民,李建民,李建民。中国生物医学工程学报,2019;47(4):886 - 894。
张建军,张建军,张建军,等。乳腺癌易感基因的研究进展。癌症治疗杂志,2015;41:1-8。
李建军,李建军,李建军,等。多基因面板的临床应用:下一代咨询和癌症风险管理的挑战。《前沿杂志》,2015。https://doi.org/10.3389/fonc.2015.00208。
王晓明,王晓明,王晓明,等。家族性乳腺癌/卵巢癌患者的基因面板测序也发现了BRCA1/2以外的基因中的多种新突变。国际癌症杂志,2017;39(4):391 - 391。
Soukupova J, Zemankova P, Lhotova K, Janatova M, Borecka M, Stolarova L,等。CZECANCA(捷克癌症临床应用小组)用于遗传癌症综合征的靶向ngs分析的验证。PLoS ONE。2018; 13: e0195761。
刘建军,刘建军,刘建军,刘建军。应用新一代测序技术改善癌症管理:临床效果和成本效益综述。中华医学杂志,2018;33(3):533 - 544。
董宁,林奴,Kidd J, Allen BA, Singh N, Wenstrup RJ,等。乳腺癌患者中25种癌症易感基因的种系突变频率。JCO。2016; 34:1460-8。
李建平,李建平,李建平。BRCA1/2基因突变携带者的生存分析。中华临床医学杂志,2010;28(2):391 - 391。
Yorczyk A, Robinson LS, Ross TS.在癌症遗传学诊所中使用小组检测代替单基因检测。中华临床医学杂志,2015;88:278-82。
奥库五世,钟伟。遗传性癌症基因组对临床护理的影响及经验教训。冷泉Harb Mol Case螺栓。2017。https://doi.org/10.1101/mcs.a002154。
刘建军,刘建军,刘建军,等。遗传性乳腺癌和卵巢癌的多基因面板检测向医疗保健的转移:对患者和家庭管理的影响是什么?Oncotarget。2017;8:1957 - 71。
张建军,张建军,张建军,等。BRCA突变携带者和其他乳腺癌/卵巢癌遗传性综合征的预防和筛查:eSMO癌症预防和筛查临床实践指南。人工智能学报,2016;27(增刊5):v103-10。
刘建军,刘建军,刘建军,等。遗传性癌症小组的实验室观点评估。巨蟹座。2016;15:689-96。
Kurian AW, Hare EE, Mills MA, Kingham KE, McPherson L, Whittemore AS,等。用于遗传性癌症风险评估的多基因测序面板的临床评价。中华临床医学杂志,2014;32(2):2001 - 9。
Colas C, Golmard L, de Pauw A, Caputo SM, Stoppa-Lyonnet D.“解码遗传性乳腺癌”的多基因面板检测的益处和问题。乳房。2019;45:29-35。
Sepahi I, Faust U, Sturm, Bosse K, Kehrer M, Heinrich T,等。在brca1阳性女性中,研究dna修复基因中额外的截断变异对乳腺癌风险的影响。中国癌症杂志,2019;19:787。
Castellanos E, Gel B, Rosas I, Tornero E, Santín S, Pluvinet R,等。一个全面的定制面板设计常规遗传性癌症检测:保持控制,提高诊断和揭示复杂的变异景观。科学代表2017。https://doi.org/10.1038/srep39348。
Schulz WL, Tormey CA, Torres R.计算方法在临床下一代测序中注释未知意义的变异。实验室医学。2015;46:285-9。
王晓明,林德杰,王晓明,等。免疫组织化学检测发现,在pMSH6表达正常的遗传性非息肉病性结直肠癌家族中,MSH6突变是常见的。中华医学杂志,2012;20:47 - 47。
王松,张勇,陈敏,王勇,冯勇,徐铮,等。ATR-CHEK1和ATM-CHEK2通路基因变异与中国人群结直肠癌风险的关系Oncotarget。2018;9:26616-24。
林文勇,Brock IW, Connley D, Cramp H, Tucker R, Slate J,等。ATR和CHEK1单核苷酸多态性与乳腺癌的关系PLoS ONE。2013.https://doi.org/10.1371/journal.pone.0068578。
张建军,张建军,张建军,等。影响POLE和POLD1校对结构域的种系突变易导致结直肠腺瘤和癌。生物医学工程学报。2013;45(5):136 - 44。
加勒特LT, Hickman N, Jacobson A, Bennett RL, Amendola LM, Rosenthal EA,等。不确定分类变体分类的家族研究:当前实验室临床实践和一种新的基于网络的教育工具。中华病毒学杂志,2016;25(1):1 - 6。
张建军,张建军。基因剪接在基因序列变化中的应用。中华医学杂志,2005;42(2):737 - 748。
肖尔S, Thompson D, Renwick A, Elliott A, Kelly P, Barfoot R,等。范可尼贫血J基因BRIP1的截断突变是低外显率的乳腺癌易感等位基因。生物医学学报。2006;38:1239 - 1241。
李建军,李建军,张建军,等。bapp1基因突变的研究进展[j]一个b> T (p.E685V)通过在人类间皮瘤细胞系中产生一个新的5 '剪接位点,完全破坏了正常的剪接。PLoS ONE。2015; 10: e0119224。
张建军,张建军,张建军,等。外显子突变对基因剪接的影响。中国生物医学工程学报,2002;22(3):571 - 571。
Moles-Fernández A, Duran-Lozano L, Montalban G, Bonache S, López-Perolio I, mensamendez M,等。用于预测乳腺癌/卵巢癌基因剪接缺陷的计算工具:它们在预测RNA改变方面的效率如何?Front Genet, 2018。https://doi.org/10.3389/fgene.2018.00366。
李建军,刘建军,刘建军。临床应用基因卡:mutyh相关息肉病(MAP),常染色体隐性结直肠腺瘤性息肉病,多发性结直肠腺瘤,多发性腺瘤性息肉(MAP) - 2012年更新。[J] .中国生物医学工程学报,2013;
Win AK, Reece JC, Dowty JG, Buchanan DD, Clendenning M, Rosty C,等。患有MUTYH双等位基因和单等位基因突变的人患结肠癌的风险。国际癌症杂志,2016;39(4):557 - 563。
李建平,李建平。乳腺癌中CHEK2基因突变的研究进展。乳腺癌。2017;9:31 - 5。
Hintzsche J, Kim J, Yadav V, Amato C, Robinson SE, Seelenfreund E,等。影响:全外显子组测序分析管道,用于整合临床样品中的分子谱和可操作的治疗方法。中华医学杂志,2016;23:721 - 730。
Schuh A, Dreau H, Knight SJL, Ridout K, Mizani T, Vavoulis D,等。通过全基因组测序鉴定癌症患者的临床可操作突变谱。冷泉保温箱螺栓,2018;4:a002279。
李建军,李建军。聚(adp -核糖)聚合酶抑制剂的研究现状及发展趋势。Onco目标,2017;10:5195-208。
康提泽OL, Velichko AK, Luzhin AV, Petrova NV, Razin SV。ATM、ATR和DNA-PKcs的合成致死性相互作用。癌症,2018;4:755-68。
致谢
这项工作得到了巴利亚多利德大学,巴利亚多利德(西班牙)和卡斯蒂利亚地区政府(León)的支持。我们也要感谢Alan Hynds对这份手稿的书面英语的批判性评论。
资金
这项工作得到了西班牙巴利亚多利德巴利亚多利德大学卡斯蒂利亚和莱昂地区政府的支持。卡斯蒂利亚和莱昂地区卫生管理:GRS1547/A/17和GRS175/A/18。Carolina Velázquez项目得到了Castile & Leon地区政府的博士前奖学金(ORDEN EDU/1083/2013)的支持。
作者信息
作者及单位
贡献
这项工作的所有作者都参与了这项工作的实现,无论是在医院对患者进行分析,还是在实验室进行遗传研究。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
所有作者都同意这一点。
相互竞争的利益
作者宣称不存在竞争利益。
额外的信息
出版商的注意
伟德体育在线b施普林格《自然》杂志对已出版的地图和机构的管辖权要求保持中立。
补充信息
附加文件1。
表S1。遗传分析中鉴定的不同VUS的CADD评分。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接获得版权所有者的许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非在数据的信用额度中另有说明。
关于本文
引用本文
Velázquez, C, lasta, E, Avila Cobos, F。et al。用于常规遗传性癌症检测的综合定制小组评估:提高种系突变检测的产量。J翻译医学18, 232(2020)。https://doi.org/10.1186/s12967-020-02391-z
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12967-020-02391-z
关键字
- 生殖系基因突变
- 遗传咨询
- 遗传性癌症综合征
- 按需基因面板
- 下一代测序