摘要
背景
辐射性口腔黏膜炎(OM)是头颈部癌症最常见的急性并发症之一。严重的OM与放射治疗中断有关,这不利于肿瘤的成功管理。放射基因组学研究表明,基因变异与放射治疗的不良反应有关。
方法
对1467例鼻咽癌患者进行了大规模全基因组扫描,其中753例接受2D-CRT治疗,来自放疗毒性和预后遗传结构(GARTP)队列,714例接受IMRT治疗(192例来自GARTP, 522例新招募)。进一步用放射治疗技术进行亚组分析。我们还对风险位点进行了物理和调控定位,并对候选靶基因进行了基因集富集分析。
结果
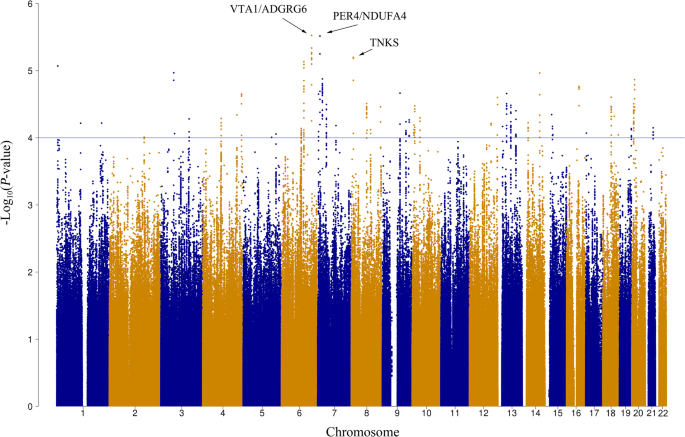
通过位置作图、表达数量性状位点(eQTL)作图、染色质相互作用作图和基于基因的分析,我们确定了50个相关基因组位点和64个基因,其中36个位点在亚群分析中得到重复。有趣的是,位于TNKS(一个与辐射毒性相关的基因)的顶级位点之一与OM风险增加相关,其先导SNP rs117157809的OR = 3.72 (95% CI 2.10-6.57;P= 6.33 × 10−6).基因集分析表明,64个候选靶基因在调控端粒封盖和维持以及端粒酶活性的生物学过程中富集(TopP= 7.73 × 10−7).
结论
这些结果增强了对放射治疗毒性的生物学认识。在端粒功能调节中富集的相关信号暗示了潜在的潜在机制,并值得进一步的功能研究和潜在的个体放射治疗应用。
背景
放射治疗是头颈部癌症的主要治疗方案。口腔黏膜炎(OM)是头颈部癌症最常见的急性辐射毒性反应之一,可导致饮食困难、体重减轻、疲劳、疼痛、睡眠不足和功能障碍[1].无法忍受的口腔黏膜炎可导致计划外的治疗中断,对治疗方案的疗效和治疗结果产生负面影响[2].由于放射治疗对鼻咽癌的肿瘤控制效果较好,放射治疗的不良反应,特别是放射诱发的鼻咽癌越来越受到人们的关注。3.].
在很大一部分患者中,使用阿片类镇痛药并不能充分缓解症状。因此,粘膜炎的对症治疗不足以避免不良临床结果,显然需要降低粘膜炎发生率的药物[1].放射基因组学研究表明,常见的遗传变异与放疗不良反应有关,基于单核苷酸多态性的预测分析以及临床因素可用于估计癌症患者因放疗产生不良反应的风险。这种检测方法可用于个性化治疗和预防严重的不良反应,从而提高患者的生活质量[4].
对于放疗的不良反应,考虑根据遗传风险调整治疗剂量,以实现个体化治疗。它提出了一种假设,即生殖系遗传学有助于辐射损伤的发展。到目前为止,辐射诱导的正常组织毒性机制是复杂的,尚不完全清楚。然而,据报道,至少有14种典型途径参与放化疗患者发生OM的过程[5].我们的目标是通过全基因组关联方法在中国南方人群中鉴定与辐射诱导OM发展相关的新位点和途径。
材料与方法
研究对象
参与者是从两个部分招募的。2005年至2007年间,从GARTP研究中筛选了960名受试者(放射治疗毒性和预后的遗传结构,注册于http://www.chictr.org.cn/(ChiCTR-ROC-17012658),根据以下标准:病理证实的鼻咽癌,以前未经治疗,以前没有放疗和/或化疗,接受全疗程的根治性放疗,成人(年龄大于18岁)。采用2D-CRT治疗的患者,对原发肿瘤的累积辐射剂量为68 ~ 76gy,每个分数为2gy。对于IMRT治疗的患者,规定的治疗方案为原发肿瘤总体积(GTV-P)规划靶体积(PTV)的30-33分68-70Gy,淋巴结总体积(GTV-N)规划靶体积(PTV)的30-33分64-68Gy。我们在2006-2014年从中国中山大学癌症中心(SYSUCC)招募了553名鼻咽癌患者。有一位患者因为没有完成整个放疗疗程而被排除在外。记录患者年龄、性别、TNM分期(采用2009年第7期UICC/AJCC分期系统)、放疗技术及治疗方案等特征。
粘膜炎评价
观察并记录放疗引起的口腔黏膜炎。根据放射治疗肿瘤小组或欧洲癌症研究与治疗组织(RTOG/EORTC)的急性辐射毒性分级标准,对其进行评估并划分为0-5级[6].根据分级结果,我们将患者分为两组:重度OM(≥3级)和轻度OM(≤2级)。
基因分型、质量控制和归责
使用商业DNA提取试剂盒(Qiagen)从整个外周血样本中提取基因组DNA,并使用PicoGreen试剂(Invitrogen)进行量化。我们在Human610-Quad芯片和Infinium Global Screening Array-24 BeadChip上对GARTP研究样本进行了基因分型。Human610-Quad芯片的基因分型和质量控制可在我们以前的出版物中找到[7].对于Infinium全球筛选阵列-24,我们使用我们的内部数据生成了一个集群文件,其中包括来自我们癌症中心的大约2000个样本,并根据制造商的协议称为基因型[8,9].对调用率低、聚类指标差或杂合度极高的变异进行人工重新聚类或去除。然后,我们根据以下标准在样本水平和SNP水平进行质量控制:(1)个体水平:呼叫率< 95%,性别差异,杂合度率异常值(> 6 sd.),基于血统的配对身份的意外重复或可能的亲属(PI_HAT > 0.5),群体分层异常值(> 6 sd.);(2) SNPs水平:非常染色体,呼叫率< 95%,小等位基因频率(MAF) < 0.001,偏离Hardy-Weinberg平衡(HWE) (P< 10−12).所有过滤后的样本都采用两阶段imputation方法,使用SHAPEIT2 [10]用于相位和IMPUTE2 [11]以作归咎。imputation在5mb非重叠间隔内进行。频度为> 1%且可归咎为INFO > 0.8的snp被纳入下游分析。
然后我们合并重叠的snp,并对snp进行进一步的质量控制。我们排除了调用率< 95%,偏离Hardy-Weinberg均衡(P< 10−12),或MAF < 0.01。我们使用PLINK 1.09进行质量控制过滤[12].最后,GWAS共分析了1467例患者(945例来自GARTP研究,522例来自2006-2014年招募的患者)和3968928例遗传变异。
全基因组关联分析
将临床因素与OM进行单因素logistic回归分析。考虑到临床变量之间存在共线性,对过滤后的变量进行多元回归分析。在全基因组关联分析中对多变量回归中显著相关的临床因素进行了调整。在加性遗传效应假设下,以主成分的前5个特征向量为协变量,采用logistic回归模型调整治疗方案、辐射技术,进行全基因组关联分析。我们还使用R包“qqman”创建了分位数-分位数图和曼哈顿图。使用分位数-分位数图来评估GWAS的总体显著性,以及观察值与预期分布的偏差P数值由膨胀因子(λGC).考虑到不同放射技术亚组(2D-CRT和IMRT)的不同发病率,我们使用logistic模型进行了进一步的关联分析,仅对两个亚组的治疗方案分别进行了调整,以检查顶部变量。
基因组风险位点和功能注释
使用fua进行功能注释[13,这是一个用于基因变异功能映射的在线平台。我们首先将“独立的重要snp”定义为超过预定义阈值的snpP值(1 × 10−4),并表现出中低连锁不平衡(r2< 0.6)。我们进一步将“先导snp”定义为独立snp的子集(r2< 0.1)。此外,我们通过合并具有相近物理位置(< 250 kb)的独立显著snp的LD块来定义基因组风险位点。在1000个基因组数据中,所有已知的snp都具有(r2> 0.6)和任何独立显著SNP被纳入注释,包含所有这些“候选SNP”的区域被认为是单个独立的基因组位点。所有LD信息均计算自1000G阶段3东亚人口[14].
通过进行ANNOVAR得到snp的功能结果[15]基于基因的注释,使用Ensembl基因。根据染色体、位置、参照和替代等位基因进行SNP匹配,并通过CADD评分进行标注(评分> 12.37表示有害SNP [16]), RegulomeDB分数[17](得分越低表明调控功能的潜力越高),以及路线图表观基因组学项目中基于127个表观基因组5个染色质标记的隐马尔可夫模型预测的染色质状态(得分越低≤7表示基因组区域的可达性越高)。CADD评分将不同的注释集成到与致病性、疾病严重程度、实验测量的调节作用和复杂性状关联相关的单一测量中。
基因图谱
利用三种策略将基因组风险位点中的snp定位到fua中的基因。
首先,位置定位是基于人类参考组合(GRCh37或hg19)中已知蛋白质编码基因的物理距离(在10 kb窗口内)。第二种策略,eQTL映射,使用了来自三个数据存储库(GTEx [18], Blood eQTL浏览器[19]和BIOS QTL浏览器[20.]),并基于显著的eQTL关联(即基因的表达与SNP的等位变异相关)将SNP映射到基因上。eQTL映射基于顺式eQTL (1 Mb内的局部调控效应)。应用0.05的错误发现率(FDR)来定义显著的eQTL关联。第三种策略,染色质相互作用映射,基于显著的染色质相互作用,将snp映射到基因的启动子区域。这种类型的映射是SNP区域和基因区域之间的三维DNA-DNA相互作用,没有距离边界。fua目前包含来自研究的21种组织/细胞类型的Hi-C数据[21].由于染色质相互作用通常在一定的分辨率(40kb)下定义,一个相互作用区域可能跨越多个基因。因此,该方法将这些区域内的所有snp映射到相应相互作用区域的基因上。为了优先考虑候选基因,我们整合了路线图表观基因组学项目中某些组织和细胞类型的预测增强子和启动子[22],包括血液、胃肠组织和皮肤。利用这些信息,fua选择了染色质相互作用,其中一个参与相互作用的区域与预测的增强子重叠,另一个与预测的启动子重叠,分别位于基因TSS上游250 bp和下游500 bp。我们使用了1 × 10的FDR−6定义重要的相互作用。
基因集分析
在fua中使用GENE2FUNC程序进一步研究了GWAS SNPs映射涉及的基因,该程序提供了MSigDB基因集中映射基因列表富集的超几何测试[23],包括BioCarta, KEGG, Reactome和Gene Oncology (GO)。的调整P采用Benjamini-Hochberg方法提供基因集富集分析的FDR值。调整阈值P-value为0.05。输入基因与被测基因集重叠的最小数量为2个。
结果
人口特征
研究人群由1467名NPC组成。其中349例患者(23.79%)放疗后出现严重OM(≥3级),未见5级粘膜炎。采用单因素logistic回归分析临床特征。据报道,肿瘤分期、临床分期、放疗技术和治疗方案与严重骨髓瘤相关(表2)1).采用不同放射治疗的患者严重中耳炎的发生率存在显著差异,2D-CRT和IMRT患者的严重中耳炎发生率分别为14.9%和33.2%。不同亚组(2D-CRT和IMRT)的临床特征见附加文件1:表S1。与单独放疗相比,接受诱导化疗和/或辅助化疗的患者的OM风险相似,or为1.07 (95% CI 0.55-2.09)。然而,与仅接受放射治疗的患者相比,同时接受放化疗的患者发生严重OM的风险更高,OR为6.96 (95% CI 4.50-10.77)。多因素logistic回归结果显示,不同的放射技术和治疗方案是放射性骨髓炎发生的重要临床因素(附加文件)2:表S2),并在GWAS中被视为协变量。
与严重口腔粘膜炎相关的snp
采用logistic回归模型,调整放疗技术和治疗方案,在加性假设下,共纳入3,968,928个snp进行全基因组关联分析。观察到的与预期的分布P数值显示在具有λ的分位数-分位数图中GC= 1.01(附加文件3.:图S1)。3个最高引物snp位于6号染色体的基因间区rs9484606 (OR = 1.70, 95% CI 1.36-2.13,P= 2.98 × 10−6, VTA1/ADGRG6), rs16876733 (OR = 1.95, 95% CI 1.47 ~ 2.59,P= 3.05 × 10−6, PER4/NDUFA4)和rs117157809 (OR = 3.72, 95% CI 2.10-6.57,P= 6.33 × 10−6),分别表示。1).我们选择了独立的snpP-value < 1×10−4对所有患者进行GWAS分析,并在阈值以下的两个治疗亚组(2D-CRT和IMRT)中进一步检查他们P= 0.05。产生的重要snp如表所示2.
基因图谱
利用FUMA中的三种基因作图策略(位置作图、eQTL作图和染色质相互作用作图),我们进一步绘制了与基因相关的显著变异,并确定了50个基因组风险位点和64个与辐射诱导的口腔黏膜炎相关的基因(附加文件)4,5:表S3, S4)。亚群分析中重叠的SNPs和基因的结果见表2.
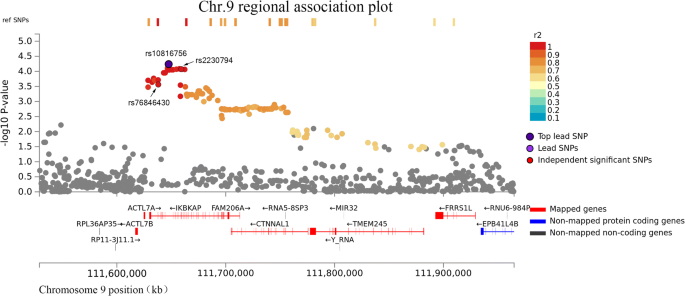
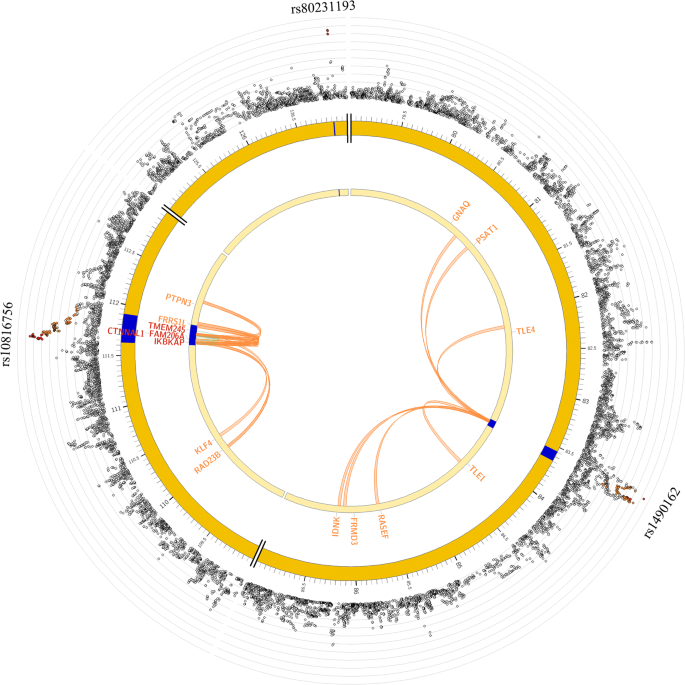
IKBKAP和DHTKD1两个基因均被三种策略定位。IKBKAP位于9号染色体位点,其前导SNP rs10816756位于该基因内含子,小等位基因OR为1.87 (95% CI为1.38 ~ 2.53)。P= 5.77 × 10−5).位于IKBKAP外显子的两个snp rs2230794和rs76846430均与rs10816756 (r2= 1,图。2).rs2230794是误义变量(OR = 1.83, 95% CI 1.35-2.49,P= 9.85 × 10−5), rs76846430是一个拼接位点变体(OR = 1.78, 95% CI 1.30-2.42,P= 2.7 × 10−4).我们进一步进行了表达数量性状位点(eQTL)分析,发现随着rs10816756风险等位基因数量的增加,整个外周血中IKBKAP的mRNA水平升高(附加文件6:表S5)。我们进一步分析了危险位点上染色质的功能相互作用,在该位点上鉴定出与染色质相互作用的8个基因,如IKBKAP、KLF4、RAD23B(图2)。3.和附加文件7:表S6)。另一个基因DHTKD1位于10号染色体位点,其前导SNP rs7068532位于内含子区,小等位基因OR为1.60 (95% CI 1.28-2.00,P= 3.33 × 10−5).eQTL分析表明,当危险等位基因增加时,外周血全血DHTKD1 mRNA水平降低(附加文件6:表S5)。风险位点与基因DHTKD1有染色质相互作用(附加文件)8:图S2和附加文件5,6:表S4, S5)。
此外,我们在GeneRIF数据集中搜索了64个映射基因,以检查它们与辐射诱导的OM的相关性。其中有两个低频变异rs117157809和rs6814005,分别位于TNKS和MAPK10的内含子中。携带rs117157809小等位基因的患者发生严重OM的风险更高,每个等位基因OR为3.72 (95% CI 2.10-6.57,P= 6.33 × 10−6).相关性OR为3.34 (95% CI 2.31-4.84,P= 1.10 × 10−3), OR为5.06 (95% CI 3.13-8.18,P= 6.99 × 10−4)在2D-CRT治疗的患者中。TNKS公司3 ' -UTR位点的SNP rs79488099与先导SNP rs117157809 (rs79488099: r2= 0.63,P= 1.48 × 10−4), rs79488099的CADD评分为16.17,为有害突变。表中显示了rs6814005的类似结果2.另一个前导SNP rs13227327位于SDK1的内含子中,小等位基因OR为1.62 (95% CI为1.30-2.03,P= 2.24 × 10−5).在该风险位点中,两个snp rs601424和rs671694均位于SDK1的外显子中,且与rs13227327 (rs601424: r2= 0.69,P= 4.54 × 10−4;rs671694: r2= 0.69,P= 3.31 × 10−4;额外的文件9:图S3)。
基于基因集的分析
fua工具涉及64个基因,为相关遗传变异的可能后果提供了更广泛的信息。使用这些基因进行基于基因集的分析,以进一步评估负责遗传信号的潜在疾病机制。附加文件中列出了20个重要的氧化石墨烯生物过程10:表S7。在这些基因集中,有8个GO基因集参与端粒或端粒酶活性的调控,包括端粒盖帽的调控(P= 7.73 × 10−7),端粒酶活性的正向调节(P= 2.13 × 10−6)和端粒维持的正向调节(P= 1.77 × 10−5).4个基因(TNKS, NEK2, NBN和KLF4)在这些GO集中复发。此外,20个重要基因集中有4个参与DNA代谢、生物合成和复制。其中一种途径是Wnt信号通路,据报道它可以调节辐射抵抗[24].
讨论
在本研究中,我们发现一些临床因素与放射诱导的OM有关,特别是化疗与放疗同步,在癌症控制中得到推广,并广泛应用于癌症治疗[25].然而,它大大增加了正常组织的辐射敏感性和辐射诱发OM的发生。调整这些临床因素,我们的全基因组关联研究通过使用近1500个总样本量确定了50个风险位点和64个定位基因。许多om相关基因参与端粒生物学过程,包括端粒封盖、维持和端粒酶活性,而其他一些基因参与DNA生物学过程,包括DNA代谢、生物合成和复制。
据我们所知,这项研究是目前最大的针对鼻咽癌放疗患者口腔黏膜炎的GWAS研究。已发表的研究调查了头颈癌和鼻咽癌中snp与辐射诱导的OM之间的关系。他们大多采用候选基因的策略来评估关联。这些候选基因和途径包括涉及双链断裂修复基因的DNA损伤和修复[26]和基底切除修复通路[27].其他重要的细胞信号通路包括Wnt/β-catenin通路[28],细胞周期调控基因,NF-κB信号通路[29]、血管生成相关基因[30.],以及GAS5 lncRNAs [31].这些研究基于100-500的小样本量,评估了候选基因中有限数量的snp,并且在未校正的水平上发现了显著的snpP值为0.05。只有一项研究显示了24名鼻咽癌患者的全基因组水平分析[32].对于辐射诱发的其他副作用的研究,许多已确定的易感基因与DNA损伤反应和修复途径、氧化应激和细胞凋亡有关[33].我们基于基因集的分析确定了Wnt信号通路,该通路已被报道与辐射诱导的OM有关[28].更重要的是,我们发现了其他潜在的途径,包括端粒和DNA生物过程。特别是端粒生物过程的基因集对患者的放射敏感性有显著影响。
已经证明端粒功能障碍与延迟DNA断裂修复动力学和对电离辐射的敏感性相关。例如,端粒酶缺陷小鼠模型表明,端粒短决定了对电离辐射过敏的情况,因此生存率下降[34].体外实验表明,未转化的人上皮细胞的辐照敏感性随着端粒功能障碍而增强,因为功能障碍的短端粒通过连接辐射诱导的DNA断端而干扰了DNA的有效修复,同时也降低了DNA断端修复的保真度[35].此外,该研究还阐明了端粒在预测癌症患者个体放射敏感性中的重要性[36].
在本研究中,与端粒功能相关的基因集如端粒封盖、维持和端粒酶活性主要涉及TNKS、NEK2、KLF4和NBN。据报道,这些基因与辐射引起的损伤有关。例如,TNKS的缺失与降解的蛋白酶体介导的DNA-PKcs观察到的缺陷性损伤反应有关,包括对电离辐射诱导的诱变、染色体畸变(末端缺失)、端粒融合和细胞杀伤的敏感性增加[37].据报道,NEK2的活性被电离辐射抑制,这种反应依赖于ATM和PP1与NEK2的结合[38].NEK2的缺失促进了细胞凋亡,减少了细胞数量[39].其他证据表明,NEK2通过调节EZH2促进胶质瘤干细胞的辐射抵抗[40].此外,据报道,KLF4可防止中心体扩增,并在γ辐射诱导的DNA损伤后表现出抗凋亡活性[41,42].在动物实验中进一步证实,KLF4是γ辐射诱导小鼠肠道损伤后肠道的辐射保护因子[43].据报道,NBN与辐射诱发的口腔黏膜炎有关[44],在放射敏感相关综合征和奈梅亨断裂综合征中均发现其突变[45].体外研究表明,NBN对辐射超敏反应的影响伴随着γ辐射诱导的人淋巴母细胞凋亡的增强[46].我们的发现进一步阐明了辐射损伤的潜在机制。
一些在两个治疗亚组中都显著的基因是进一步感兴趣的,包括IKBKAP, SDK1和MAPK10。IKBKAP位于9号染色体26号危险位点。rs10816756是该位点的领先SNP,有适度的迹象P= 5.77 × 10−5.IKBKAP被认为是一种通过c-Jun n -末端激酶(JNK)信号通路调控哺乳动物应激反应激活的支架蛋白[47], JNK信号通路被认为参与辐射诱导的OM病理[48].SDK1是一种粘附分子,在细胞应激下被激活,特别是在活性氧条件下[49].此外,MAPK信号通路成员MAPK10基因能够调控p38-MAPK、PI3K-MAPK等级联,参与电离辐射响应[50].对三名辐射暴露个体的淋巴细胞进行的初步分析表明,MAPK10是一种与细胞对电离辐射反应相关的诱导基因[51].
结论
总之,我们的全基因组关联研究确定了辐射诱发口腔黏膜炎的50个基因组风险位点和64个候选基因。使用生物数据资源的功能注释和基因定位的组合策略为相关遗传变异的可能后果提供了广泛的信息。我们强调了通过多种途径涉及的几个基因,并提出了一套丰富的基因靶点和功能随访的生物学机制。基因集分析对潜在的端粒途径提供了新的见解,证实了端粒功能在发展辐射诱导的不良反应中的重要性。需要更大的样品和功能实验。目前的研究为辐射诱导的口腔黏膜炎提供了新的线索和功能假设,对预测个体放射敏感性和促进个性化放疗策略具有重要意义。
数据和材料的可用性
本研究中使用和分析的数据集可根据合理要求从通讯作者处获得。
缩写
- OM:
-
口腔粘膜炎
- GARTP队列:
-
放射治疗毒性和预后队列的遗传结构
- eQTL:
-
表达数量性状位点
- 全国人大:
-
鼻咽癌
- RTOG / EORTC:
-
放射治疗肿瘤小组或欧洲癌症研究和治疗组织
- PTV:
-
规划目标卷
- SYSUCC:
-
中山大学癌症中心
- 加:
-
小等位基因频率
- GWAS:
-
全基因组关联研究
参考文献
Elting LS, Keefe DM, Sonis ST, Garden AS, Spijkervet FK, Barasch A,等。患者报告的头颈部癌症患者在接受放疗和不接受化疗的情况下口腔粘膜炎的测量结果:显示增加的频率、严重程度、对姑息治疗的耐药性和对生活质量的影响。癌症。2008;113(10):2704 - 13所示。
Russo G, Haddad R, Posner M, Machtay M.头颈部癌症放射治疗中断与溃疡性粘膜炎。肿瘤学家。2008;13(8):886 - 98。
蔡mlk, Wee JTS, Hui EP, Chan ATC。鼻咽癌。柳叶刀》。2016;387(10022):1012 - 24。
克恩斯,H,罗森斯坦,b。放射基因组学:利用遗传学来确定在放射治疗后发生不良反应的风险的癌症患者。中国癌症杂志,2014;4(2):155-65。
Sonis S, Haddad R, Posner M, Watkins B, Fey E, Morgan TV,等。外周血细胞的基因表达变化为头部和颈部癌症患者治疗方案相关毒性的生物学机制提供了深入了解。口腔医学杂志,2007;43(3):289-300。
Cox JD, Stetz J, Pajak TF。放射治疗肿瘤小组(RTOG)和欧洲癌症研究与治疗组织(EORTC)的毒性标准。中华放射医学杂志,1995;31(5):1341-6。
戴杰,沈伟,文伟,常杰,王涛,陈辉,等。利用中国人群全基因组关联研究数据估计九种常见癌症的遗传力。中华癌症杂志,2017;40(2):329-36。
王志刚,王志刚,王志刚,张志刚,张志刚,等。肿瘤阵列联盟:理解常见癌症遗传结构的网络。中国癌症流行病学杂志,2017;26(1):126-35。
Michailidou K, Lindstrom S, Dennis J, Beesley J, Hui S, Kar S,等。关联分析确定了65个新的乳腺癌风险位点。大自然。2017;551(7678):92 - 4。
李志强,李志强,李志强,等。在亲缘关系的全谱单倍型分期的一般方法。公共科学图书馆。2014;10(4):e1004234。
王志强,王志强,王志强,等。基于全基因组关联研究的基因型植入方法。公共科学学报,2009;5(6):e1000529。
Purcell S, Neale B, Todd-Brown K, Thomas L, Ferreira MA, Bender D,等。PLINK:全基因组关联和基于人群的连锁分析工具集。王志强,王志强。2007;21(3):357 - 357。
渡边K, Taskesen E, van Bochoven A, Posthuma D.与fua基因关联的功能定位和注释。地理学报。2017;8(1):1826。
基因组计划C, Auton A, Brooks LD, Durbin RM, Garrison EP, Kang HM,等。人类遗传变异的全球参考。自然。2015;526(7571):68 - 74。
王凯,李敏,Hakonarson H. ANNOVAR:基于高通量测序数据的基因变异功能注释。中国生物医学工程学报,2010;38(16):e164。
李文杰,李志强,李志强,李志强。人类遗传变异相对致病性的研究进展。植物学报。2014;46(3):310-5。
马晓波,李志强,李志强,等。使用RegulomeDB注释个人基因组的功能变异。基因组学报,2012;22(9):1790-7。
联盟GT,实验室DA,协调中心-分析工作G,统计方法组-分析工作G,增强Gg,基金NIHC。人类组织中基因表达的遗传效应。大自然。2017;550(7675):204 - 13所示。
韦斯特拉,张志强,张志强,等。系统鉴定反式eqtl作为已知疾病关联的假定驱动因素。植物学报。2013;45(10):1238-43。
Zhernakova DV, Deelen P, Vermaat M, van Iterson M, van Galen M, Arindrarto W,等。全血环境依赖性表达数量性状位点的鉴定。中国机械工程,2017;49(1):139-45。
Schmitt AD,胡敏,荣毅,徐震,邱勇,谭春林,等。染色质接触图的纲要揭示了人类基因组中的空间活跃区域。Cell rep 2016;17(8): 2042-59。
Kundaje A, Meuleman W, Ernst J, Bilenky M, Yen A,等。111个参考人类表观基因组的综合分析。大自然。2015;518(7539):317 - 30。
Liberzon A, Subramanian A, Pinchback R, Thorvaldsdottir H, Tamayo P, Mesirov JP。分子特征库(MSigDB) 3.0。生物信息学。2011;27(12):1739 - 40。
赵颖,陶玲,易军,宋华,陈玲。Wnt经典信号在调节辐射抗性中的作用。中国生物医学工程学报。2018;48(2):419-32。
林景昌,杨建军,徐春春,梁文敏,姜瑞,王文文。晚期鼻咽癌同期放化疗与单独放疗的III期研究:对总生存期和无进展生存期的积极影响。中华临床杂志,2003;21(4):631-7。
任建辉,戴晓峰,闫桂兰,金敏,刘长武,杨宇凯,等。鼻咽癌放疗患者急性口腔黏膜炎与DNA DSB修复基因多态性的关系。中华放射生物学杂志,2014;30(3):344 - 344。
王杰,郭超,龚霞,敖峰,黄燕,黄玲,等。碱基切除修复通路基因多态性对鼻咽癌患者化疗放疗疗效及急性毒性的影响Oncotarget。2017;8(45):78633 - 41。
于洁,黄勇,刘玲,王洁,殷杰,黄玲,等。Wnt/ -catenin通路基因的遗传多态性与鼻咽癌患者放射治疗的疗效和毒性相关。Oncotarget。2016;7(50):82528 - 37。
郭超,黄勇,余娟,刘玲,龚霞,黄敏,等。细胞周期基因和NF-kB通路单核苷酸多态性对鼻咽癌患者放疗疗效和急性毒性的影响Oncotarget。2017;8(15):25334 - 44。
马伟林,刘睿,黄汉林,邹超,黄娟,王杰,等。血管生成相关基因多态性对鼻咽癌患者放射治疗临床结局的影响。中国临床医学杂志,2017;44(5):539-48。
金赵王郭Z, Y, Y, Y, L,吴B, et al。长链非编码RNA GAS5基因多态性预测鼻咽癌患者铂基同步放化疗反应Oncotarget。2017;8(37):62286 - 97。
乐志,牛鑫,陈勇,欧旭,赵刚,刘强,等。鼻咽癌放疗患者急性口腔黏膜炎的预测单核苷酸多态性标记。Oncotarget。2017;8(38):63026 - 37。
Pavlopoulou A, Bagos PG, Koutsandrea V, Georgakilas AG。正常和肿瘤组织放射敏感性的分子决定因素:生物信息学方法。癌症杂志,2017;403:37-47。
王佳奎,张珊珊,魏乐SR, Ganesan S, Chaudhuri J,朱晨,等。端粒功能障碍损害DNA修复,增强对电离辐射的敏感性。中国科学,2000;26(1):85-8。
杨晓明,张晓明,张晓明,等。非转化人上皮细胞增殖相关端粒功能障碍对辐射敏感性的影响。衰老细胞。2009;8(4):414-25。
Mirjolet C, Boidot R, Saliques S, Ghiringhelli F, Maingon P, Crehange G.个体化放疗时代端粒在预测癌症患者个体放射敏感性中的作用。中华癌症杂志2015;41(4):354-60。
周娟,周建平,李丽娟,李文杰,李志刚。坦基酶1在端粒和DNA修复中的调节作用:抑制T-SCE和稳定DNA- pkcs。老龄化(奥尔巴尼纽约)。2010; 2(10): 691 - 708。
米俊,郭超,陈国强,陈国强。蛋白磷酸酶-1通过Nek2调节中心体分裂。癌症杂志2007;67(3):1082-9。
弗莱彻L,塞尔尼格利亚GJ,尼格EA,伊德TJ,穆谢尔RJ。DNA损伤后中心体分离的抑制:Nek2的作用。辐射科学,2004;62(2):128-35。
王洁,程鹏,Pavlyukov MS,余华,张震,金盛华,等。靶向NEK2通过破坏组蛋白甲基转移酶EZH2的稳定来减弱胶质母细胞瘤的生长和辐射抵抗。中国临床医学杂志,2017;29(8):369 - 369。
Yoon HS, Ghaleb AM, Nandan MO, Hisamuddin IM, Dalton WB, Yang VW。克虏伯样因子4可防止γ辐照诱导DNA损伤后中心体扩增。致癌基因,2005;24(25):4017 - 25所示。
Ghaleb AM, Katz JP, Kaestner KH, Du JX,杨大众。克虏伯样因子4在γ辐射诱导的DNA损伤后表现出抗凋亡活性。致癌基因。2007;26(16):2365 - 73。
郑新军,郑志军,郑志强,张志强,等。克虏伯样因子4是γ辐射诱导小鼠肠道损伤后肠的辐射保护因子。中华实用医学杂志,2015;30(2):344 - 344。
Venkatesh GH, Manjunath VB, Mumbrekar KD, Negi H, Fernandes DJ, Sharan K,等。头颈部癌症患者放射反应基因的多态性及其与急性毒性的关系。PLoS ONE。2014; 9 (3): e89079。
Tauchi H, Matsuura S, Kobayashi J, Sakamoto S, Komatsu K.奈梅根断裂综合征基因,NBS1及其与基因组稳定性因素的分子联系。致癌基因。2002;21(58):8967 - 80。
Sagan D, Mortl S, Muller I, Eckardt-Schupp F, Eichholtz-Wirth H. cd95介导的凋亡增强与NBS淋巴母细胞辐射超敏反应有关。细胞凋亡。2007;12(4):753 - 67。
Holmberg C, Katz S, Lerdrup M, Herdegen T, Jaattela M, Aronheim A,等。I κ B激酶复合物相关蛋白在细胞质应激信号传导中的新特异性作用。中国生物医学工程学报,2002;27(3):339 - 344。
张丽娟,李丽娟,张丽娟,等。放射性口腔黏膜炎。Front Oncol. 2017;7:89。
马基-内瓦拉,Sarhadi VK, Knuuttila, Scheinin I, Ellonen P, Lagstrom S,等。外显子组测序检测石棉暴露肺腺癌和恶性间皮瘤的驱动基因和新突变。肺。2016;194(1):125 - 35。
邓特P, Yacoub A, Contessa J, Caron R, Amorino G, Valerie K,等。应激和辐射诱导的多种细胞内信号通路的激活。辐射科学,2003;39(3):366 - 366。
Sakamoto-Hojo ET, Mello SS, Pereira E, Fachin AL, Cardoso RS,军政府CM,等。基因毒性胁迫下人类细胞的基因表达谱。Mutat res 2003;544(2-3): 403-13。
确认
不适用。
分析
FUMA,http://fuma.ctglab.nl/.
千人基因组计划第三阶段,http://www.internationalgenome.org/.
ANNOVAR,http://annovar.openbioinformatics.org/en/latest/.
CADD v1.3,http://cadd.gs.washington.edu/.
RegulomeDB v1.1,http://regulomedb.org/index.
15核染色质状态,http://egg2.wustl.edu/roadmap/web_portal/chr_state_learning.html.
GTEx v6 / v7中,http://www.gtexportal.org/home/.
Blood eQTL浏览器,http://genenetwork.nl/bloodeqtlbrowser/.
BIOS QTL浏览器,http://genenetwork.nl/biosqtlbrowser/.
GSE87112(高c),https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE87112.
表观基因组学项目路线图,http://egg2.wustl.edu/roadmap/web_portal/DNase_reg.html.
MsigDB v6.1中,http://software.broadinstitute.org/gsea/msigdb.
R v3.4.0,http://www.r-project.org/.
资金
本研究由中瑞联合研究计划(81861138006)资助;国家自然科学基金(81973131,81803319,81903395,81502056);广州市科技计划项目(201804020094);国家重点研发计划项目(2016YFC1302704);国家杰出青年科学基金(81325018);国家自然科学基金国际合作与交流重点项目(81220108022);广州市健康医疗协同创新项目(201604020003)。
作者信息
作者及隶属关系
贡献
由DWY进行数据收集、数据分析和解读,并起草稿件。TMW:数据和算法的质量控制,数据分析和解读,稿件编辑。JBZ参与了研究设计和稿件评审。XZL负责样品采集、质量控制和数据采集。YQH参与稿件编辑。RX、WQX、XHZ、PFZ、SDZ、YZH、GPS进行表型收集和数据采集。MC和YS参与了稿件评审。WHJ构思并设计了这项研究,编辑并审阅了手稿。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
这项研究得到了中山大学癌症中心伦理委员会的批准。在入组时获得每位参与者的书面知情同意。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:表S1。
放射治疗两个亚组的临床特点。
附加文件2:表S2。
临床因素与急性口腔黏膜炎的多因素logistic回归分析。
附加文件3:图S1。
全基因组分位数-分位数图P关联的价值。
附加文件4:表S3。
协会P-重度口腔黏膜炎的所有先导snp值。
附加文件5:表S4。
所有基因定位为基于snp的重度口腔黏膜炎(FUMA)。
附加文件6:表S5。
eQTL将严重口腔粘膜炎的GWAS风险snp与已定位的基因联系起来。
附加文件7:表S6。
染色质相互作用区域连接严重口腔粘膜炎GWAS风险位点与已定位基因。
附加文件8:图S2。
与辐射诱导的口腔黏膜炎相关的10号染色体基因组区域的交叉位点相互作用。
附加文件9:图S3。
rs13227327的关联区域图。
附加文件10:表S7。
辐射诱发口腔黏膜炎的前20个显著基因集分析。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
杨,DW。,Wang, TM., Zhang, JB.et al。全基因组关联研究确定辐射诱导的急性口腔黏膜炎的遗传易感位点和途径。翻译医学杂志18, 224(2020)。https://doi.org/10.1186/s12967-020-02390-0
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12967-020-02390-0
关键字
- 辐射伤害
- 口腔粘膜炎
- Radiogenomics
- 全基因组关联研究
- 功能性映射