抽象性
后台
越来越多的证据表明,免疫相关基因在肝细胞癌的开发与演进中发挥着关键作用。免疫相关基因评价HCC病人预测作用仍然不足。研究旨在探索HCC内免疫相关基因的基因特征和预测值
方法论
综合基因表达数据 从374HCC和50正常组织获取 癌症基因组地图集偏差表达基因分析与单词反射分析,以识别关乎总体生存的DEG使用Lasso和多变量Cox回归分析构建了免疫预测模型并应用Cox回归分析识别高专委独立预测因子免疫签名和免疫细胞渗透之间的关联分析也得到了调查。最后,签名在一个外部独立数据集中验证
结果
共检测出329个不同表达式免疫基因64个免疫相关基因与HCC病人使用单向回归分析总体生存有显著关系并搭建TF网络探索这些基因的规范机制Lasso和多变量Cox回归分析应用构建基于免疫预测模型,该模型由九种与免疫有关的基因组成进一步分析显示,这种免疫预测模型经调整以适应其他临床因素后可成为独立的预测指标风险评分模型和免疫细胞渗透之间的关系显示九元签名可反映肿瘤免疫微环境状态九元预测模型预知值在一个独立数据库中进一步成功验证
结论
联合筛选潜在的预测性免疫相关基因并创建新颖的基于免疫预想模型HCC,该模型不仅提供新的潜在预测性生物标志和治疗目标,还加深我们对肿瘤免疫微环境状态的理解,为免疫法打下理论基础
后台
肝癌全球名列第六大癌症,死亡数居第二位一号..全世界每年60多万人肝癌死亡,近85万新例一号,2..肝细胞癌占所有肝癌的85-90%并受到公众关注尽管新测试和处理快速提高,HCC5年生存率仍然不到五分之一3..外科目前仍然是肝癌早期主要治法4..相当大比例的病人会复发或远程改发5..最近Sorafenib和Regorafenib等药证明对高级HCC有效6,7..值得一提的是,病理类型和临床阶段相同的病人经相同处理后往往产生不同结果,这主要是由于病人遗传异性[8..
免疫系统被认为是癌症开发的决定性因素九九,10包括HCCImune细胞是肿瘤微环境的主要构件,并在许多关键步骤HCC开发中发挥作用,从肿瘤生长到异位化开发11,12..此外,发现大量煽动性调停者与高管开发相关联IL-22属于Citokine家族,HCC微环境表达过度并导致肿瘤生长13..Chemokines公司CXCL12,CCL20,作为免疫系统免疫分子成员,也在HCC生长、入侵和转移中发挥关键作用14,15..证明免疫性使HCC免疫法前景大有希望16..研究进度发现编程细胞死一路径HCC免疫法新目标17..单克隆抗体Nivolumab阻塞PD-1并恢复机体抗癌免疫响应,干扰信号路径,从而预防T细胞激活18号..高电委Nivolumab显示在客观响应率和总体生存方面大有裨益19号..ivolumab可能为HCC提供安全、有效和有希望的处理方法20码..越来越多的研究表明,HCC中与免疫有关的基因与HCC的肿瘤生成和开发密切相关[HCC21号..然而,目前尚没有基于免疫相关基因的预测模型系统评价肿瘤免疫环境并预测HCC病人总体预测构建基于免疫预测模型可可靠预测高频CC预测具有重大临床意义
第一步研究中,我们通过大规模排序数据库生物信息分析筛选与HCC密切相关的不同表达式免疫相关基因下一步检测出与预测有重大关联的免疫相关基因之后我们搭建了免疫相关预测模型 整合免疫相关基因独立国际癌症基因组集团数据库进一步验证了我们免疫相关预测模型的预测值提供新生物标志有效预测并监控HCC病人肿瘤免疫微环境
方法论
资料收集
Gene表达资料和HCC样本临床资料取自TCGA数据门户https://portal.gdc.cancer.gov/cart脱机至2019年9月16日RNA-Seq FPKM编程数据374HCC和50相邻HCC正常组织下载供进一步分析仔细搜验224名HCC病人并发乙型肝炎病毒其中包括81HBsAg阳性病人、60HBsAg和HBsAb均呈阳性病人和83个历史风险因素为乙型肝炎的病人国际癌症基因组联合会https://dcc.icgc.org/search?filters=%7B%22donor%22:%7B%22projectId%22:%7B%22is%22:%5B%22LIRI-JP%22%5D%7D,%22availableDataTypes%22:%7B%22is%22:%5B%22exp_seq%22%5D%7D%7D%7D网络门户提供50种不同肿瘤类型综合分子遗传剖面GIC为分析基因组在基因组学和运算学上的宝贵数据库验证组别从ICCC数据库检索基因表达数据及231名HCC病人的相应生存信息1811免疫基因通过免疫数据库分析门户下载https://www.immport.org/shared/genelists数据库中17类免疫基于各种分子函数22号..Cistrone癌症http://cistrome.org/CistromeCancer/CancerTarget/数据库对生物医学和遗传学研究有用,并完全包括318分解因子23号..为了调查免疫相关基因管理机制,我们提取这些TF供后续研究使用因为我们的数据直接从公共数据库下载,我们严格遵守TCGA和ICCC提供的发布指南,因此不需要道德认证
差分表达式分析
差分表达的HCC相关基因和TF使用Welcoxon测试法检测正常组织log2foldchange++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++热图包生成热图,火山块用R软件操作评估偏差表达的免疫相关基因生物函数GeneOntology24码和京都基因和基因百科分析25码由集群剖析器包执行26中功能类别调整P值 < 0.05被视为重要路径
生存分析
生存分析仅使用后续时间小于2000天的病人求生存包执行单词剖析仅这些P值 < 0.01基因被视为预测性免疫相关基因GO和KEGG分析进一步分析这些预测性免疫相关基因评估TF如何调节这些免疫相关基因时,我们先使用单向Cox分析 < 0.01步用Cor.test函数执行R,核心方法为Pearson测试相关系数和P值由cer.test计算截取标准设定为相关系数 > 0.5和P < 0.05为了让图片清晰化,我们只选择调控九大免疫相关基因的TFCytoscape用于构建并直观管理网27号..
HCC构建免疫签名
开发预测模型时使用Lasso和多变量Cox回归分析评估预测性免疫相关基因表达式与总体生存关系为了避免过配删除高度关联基因,LassoCox使用生存和gmnet包件回归通过Lasso算法检测到的基因通过阶梯智能多变化Cox回归分析评价风险分数基于基因表达式乘法从多变量Cox回归中获取回归系数的线性组合病人按中位风险评分分配到高风险和低风险组卡普兰-Meier分析通过sessions接收器操作特征曲线由R软件包生存矩形此外,使用单动多变分析评估风险分数对总体生存和数个临床特征的影响
免疫签名和免疫细胞渗透相关分析
探索预测模型和免疫细胞渗透之间的联系时,我们使用TumorImmune估计资源28码有效资源综合分析肿瘤渗透性免疫细胞TIMER算法允许用户估计六类肿瘤渗透性免疫细胞子集的构成(B细胞、CD4+T细胞、CD8+T细胞、宏数组、中子机和嵌入式细胞)。HCC病人的免疫渗透水平取自TimeR网站,预测模型和6个肿瘤渗透免疫细胞的关联性在R
遗传改变免疫相关签名
CBio癌症基因组门户网站是一个重要在线平台,可视化分析各种癌症基因组数据29,30码..CBioPortal分析HCC病人遗传变换反癌症药 目标这些基因
外部验证免疫相关签名
验证免疫相关签名风险评分模型预测值时,我们使用ICCC数据库验证组使用同一种公式计算风险分数并基于最优截点划分为高风险和低风险组卡普兰-Meier和ROC曲线分析如上所述
统计分析
所有分析均使用Rversion3.5.1除另行注明外,P < 0.05被认为意义重大。
结果
差异表达免染基因和HCCTF
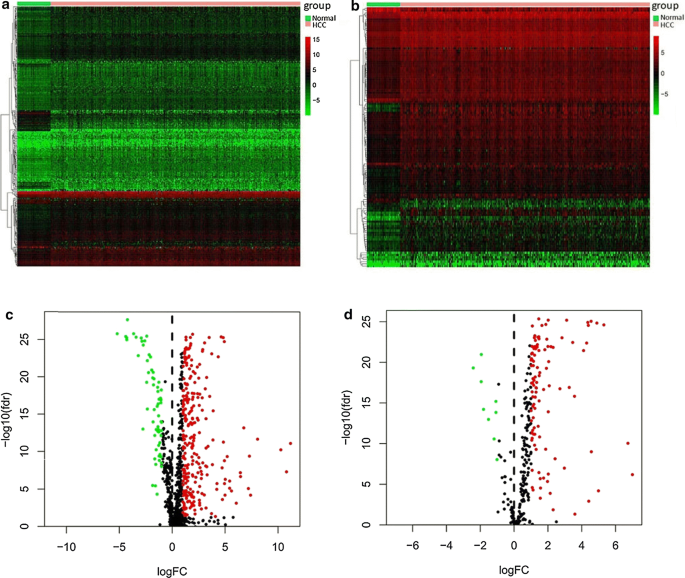
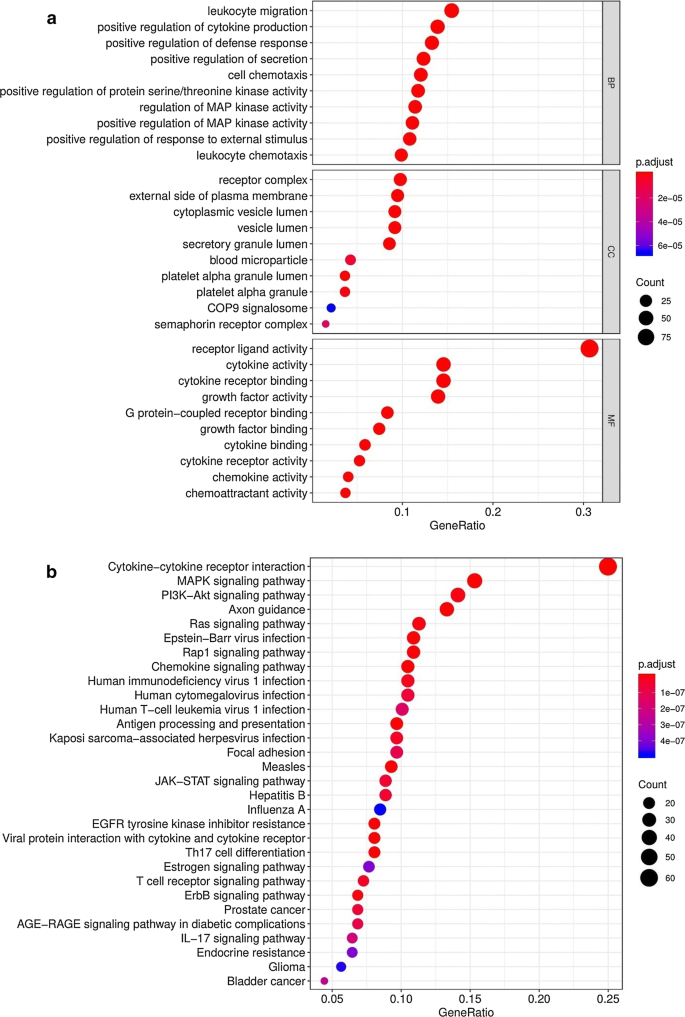
总计329个免疫相关基因(267调高和62调低)和117TFs(108调高和9调低)被识别为HCC组织与正常组织相比有差分表达法热映射显示,HCC样本可明显与正常样本区别开来,按微分表示免疫相关基因和TFs一号ab)Volcano图显示高频控制器和正常控制器间微值免责基因和TF分布一号Cd.GO和KEGG分析进一步分析329差分表示免疫相关基因GO分析显示生物过程初级功能类为leukocyte迁移、对细胞基生产的积极调控和对防御响应的积极调控细胞组件大浓缩GO条件为等离子膜受体复杂外侧最富手机组件为受体Ligand活动、cytokine活动以及cytokine受体绑定2a))KEGG路径显示,微分表示免疫相关基因主要与Cytokine-cytokine受体交互作用、MAPK信号路径和PI3K-Akt信号路径相关2b)
筛选与免疫相关基因并预知值
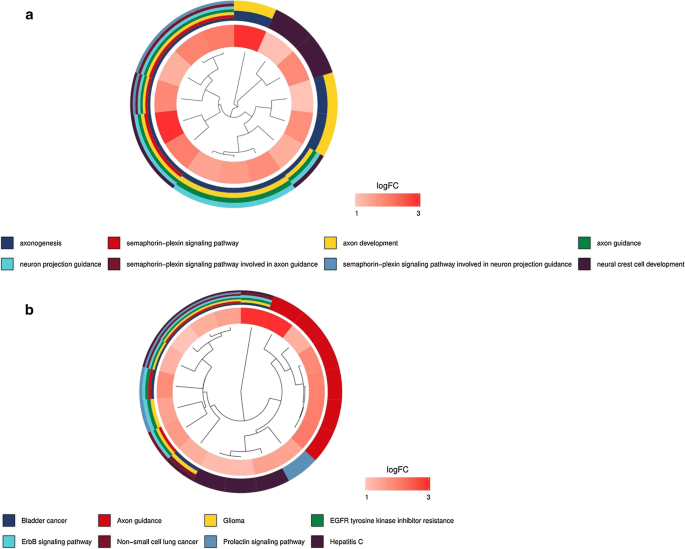
337HCC样本中329个基因表达方式通过单词剖析Cox来评价,以确定有预测特征的不同表达式免疫相关基因完全64个免疫基因被发现与OS相关预测性免疫相关基因显示于表一号.GO和KEGG分析显示,这些预测性免疫相关基因主要参加Semaphorin-Listen信号路径ErbB信号路径HepatitisC显示这些路径与癌症开发关系极大(图二图二图二图二图二图二图二图二图二图二图二图二图二图二图二图二图二图二图二图二图二图二图二图二图二图二图二图二图二图二3ab)
TF监管网络
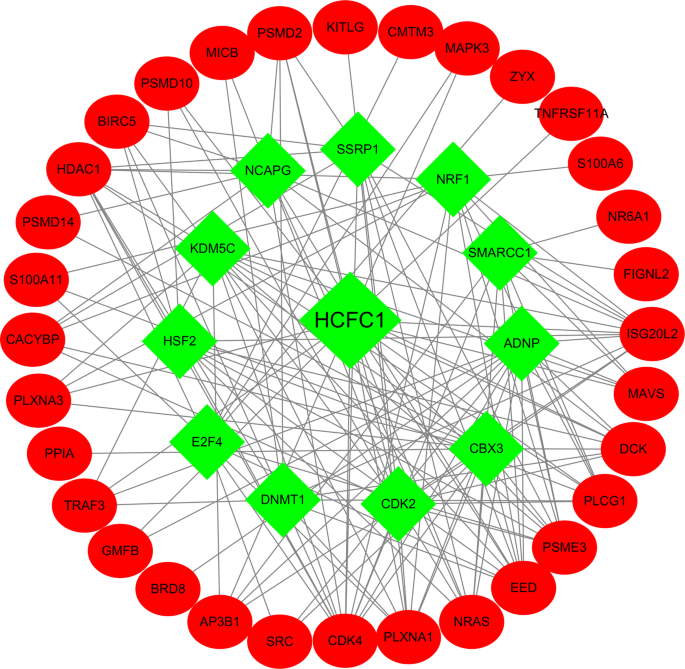
探索预测性免疫相关基因管理机制时,我们选择HCC病人中预测相关TF单变量Cox回归分析检测到54TF与病人总体生存相关2)依据54TFs和64免疫相关基因建立监管网络TF调控网络分三步建设:(1)54个预测相关TF和64个预测免疫相关基因(2) 每种TF和每种免疫基因间的关联测试使用cr.test函数进行,核心方法为Pearson测试基于截取标准,确定12种预测相关TF和32种预测性免疫相关基因建立网络3) 我们使用子形构建并视觉化主调网fig显示4HCFC1管理大部分与免疫有关的基因并占据支配地位转录调控网络揭示了这些与免疫相关基因之间的调控关系
构建HCC免疫预测模型
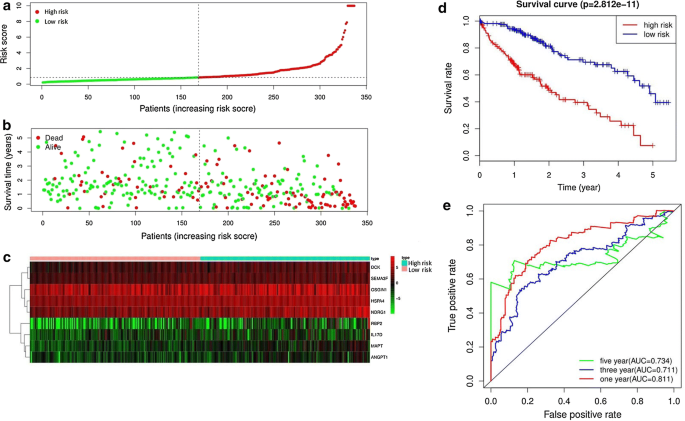
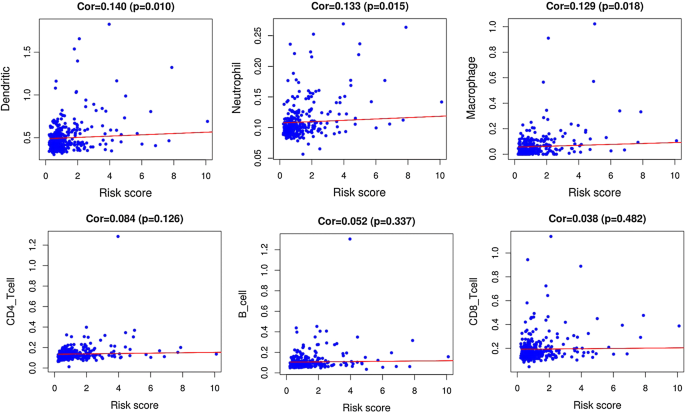
64个预测性免疫相关基因接受LassoCox分析并过滤21个基因并进行了多变量Cox分析并最后选择9个基因建立预测模型公式显示为:风险评分=0.2940*表达级别ANGP1+0.1753*表达级别MAPT+0.1066*表达级别DK+0706*表达级别SEMA3F+0703*表达级别IL17D+0703*表达级别HSPA4+0.0204*表达级别RBP2+DRG1+0.0084*表达级别OSGIN1所有九大基因均风险预测基因,危险比大于1风险分数基于基因表达度乘法回归系数计算多变量Cox回归风险评分不单与这些基因表达层次相关,而且还与相关系数相关337 HCC样本根据中位风险评分划分为高风险组(n=168)和低风险组(n=169)5a))生存概述和基因表达热图用Fig显示5b-c.生存分析显示,高风险组病人总体生存明显比低风险组病人差(P < 0.0001!微博5公元前ROC曲线下区域1年3年5年OS为0.811、0.711和0.734,表示预测模型敏感度和特性良好(图二图二图二图二)。5e)风险评分模型和免疫细胞渗透之间的关系得到了调查fig显示6嵌入式细胞、中子元和宏与风险评分呈正相关B单元、CD4+T单元、CD8+T单元和风险评分之间没有观察到重大关联
免疫相关预测签名从其他临床因素独立
207名HCC病人的临床信息包含性别、年龄、组织学等级、病理阶段和TNM阶段供进一步分析单变多变Cox回归分析评估免疫相关预测签名的独立预测力单变量分析显示,病态级(P < 0.0001)、T分类(P < 0.0001)和免疫预测模型(P < 0.0001)与总体生存有明显的关联性。多变量分析后,只有免疫预测模型仍作为OS相关独立预测因子(P < 0.0001!表23)
九大免疫相关预测基因基因基因基因变换
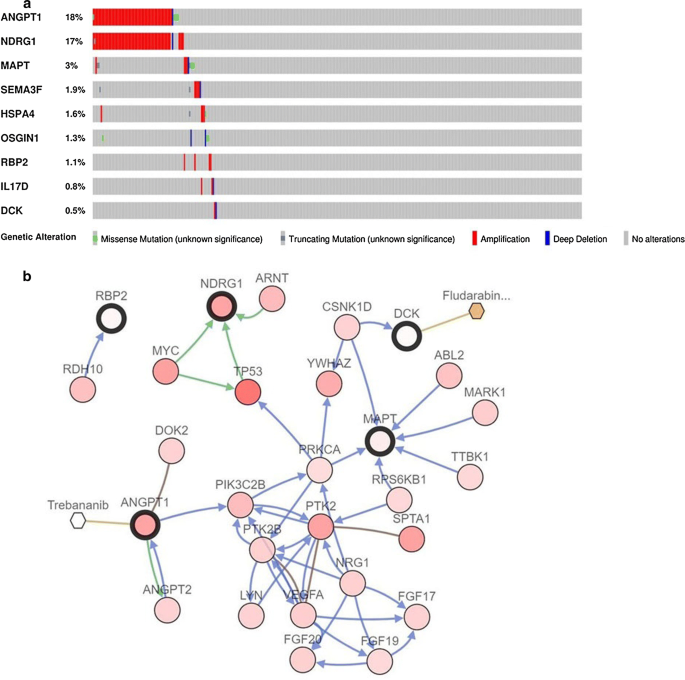
CBioPortal工具用于分析九种免疫相关预测基因的基因变换和潜在药fig显示7a,ANGPT1和NDRG1最常变换基因放大是主要的频繁遗传变换和九种免疫相关预测基因在373例中的94例中修改(25.2%)图7显示网络由九种免疫相关预测基因和50种最常变异近邻基因所构建以这些基因为对象的抗癌药展示其中有两种基因(DCK和ANGPT1)目前被视为毒品目标我们认为其他基因 可能作用为 潜在新治疗目标
GIC数据库验证免疫预测签名
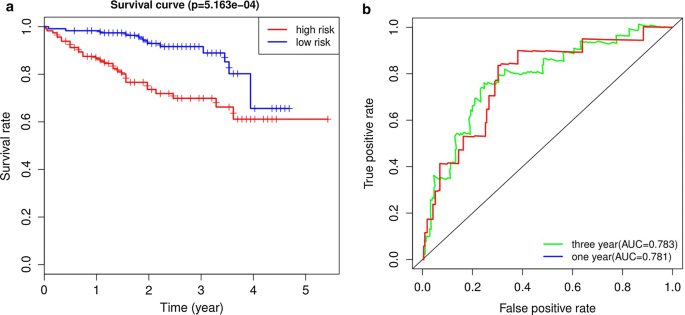
GICC数据库包括231个HCC样本用于验证免疫相关签名中位风险评分显示,我们把病人划分为高风险组(n=115)和低风险组(n=116)。kaplan-Meier曲线表示高危险组病人总体生存比低危险组病人明显差(P < 0.001!微博8a))AUC系统1年3年OS数为0.781和0.783,显示预测OS中与免疫有关的签名性能良好8b)因为只有一个病人 5年后续周期, 我们没有绘制5年ROC曲线
讨论
肝细胞癌仍是一种致命恶性肿瘤,极不讨好全球预测精确预测HCC总体生存对选择治疗方法和改善预测意义重大迄今尚没有可靠有效生物标志精确预测HCC病人生存关键需求识别强生物标志和预测模型预测高管结果
在当前研究中,基于对TCGA数据集分析,从374例HCC和50例正常组织中筛选出329个差分表达的免疫相关基因GO浓缩结果显示,所述基因主要与免疫响应相关KEGG路径大都集中在几条癌症相关路径上(例如MAPK信号路径和PI3K-Akt信号路径)。单向回归分析有微分表达的免疫相关基因中发现64个基因显示与OS关系重大深入预测性免疫相关基因管理机制,搭建TF介质网络披露关键TF,这些关键TF能够调节这些免疫相关基因主网络表示HCFC1是网络关键密钥调节器HCFC1(主机手机因子C1)属于主机手机因子组值得注意的是,最近的一项研究称HCFC1为临床中心基因,与HCC患者生存时间、等级和TNM阶段有显著关联31号..至今为止,HCFC1对HCC开发的贡献尚不清楚。需要进一步实验证据探索HCFC1分子机制
最近据异常mRNA显示基因签名引起广泛关注并揭示HCC预测的巨大潜力举例说,Long等搭建四元预测模型显示高频CC预测性能良好32码..另一项研究还搭建六元预知签名,用于HCC基于TCGA基因表达数据总体生存预测三十三..最近研究TP53相关免疫基因预测值时发现并验证二元模型(TREM1和EXO1预测模型[34号..但这些研究没有使用大量样本全面探索免疫基因与高频委预测之间的关系与前几类研究相比,本项研究有几大长处:(1) 我们使用专用免疫学数据库,使我们能够分析尽可能多的免疫基因据我们所知,这是首项探索大量免疫相关基因与HCC病人预测关系的研究(2) 我们获取数种预测性免疫相关基因并创建新颖的免疫相关预测模型预测模型展示OS预测显赫性能深入分析显示,免疫预测模型经调整以适应其他临床因素后证明为独立预测指标由九种免疫相关基因构成的模型随后成功验证为国际遗传学委员会独立数据集预知性因子所有上述结果显示,免疫相关预测模型可有效标记高频CC预测
肿瘤免疫微环境状态特征调查与免疫相关预测模型和免疫细胞渗透之间的关系数据显示,高风险病人中可观察到更高渗透度的二叉状细胞、中子元子和宏图endritic细胞、中微分量和宏数显示与免疫相关预测模型有正相关关系,显示模型可预测增强免疫细胞渗透最近的一项研究报告,进化细胞内渗透与HCC病人预测差有密切关系35码和我们发现一致现有研究报告,高合体内神经元渗透可能显示与不良临床结果相联36号..神经元化促进免疫细胞的激活、调控和效果函数37号万事通并能够通过隐蔽多类细胞素[38号从而证明他们在HCC发源地中的关键作用数项研究报告增宏与高管预测差相关三十九..宏图渗透肿瘤微环境可促进肿瘤生长、血管生成、入侵和转移40码..目标宏法被认为是HCC病人有希望的ajivant免疫法41号,42号..HCC内免疫细胞作用尚未完全说明扩大免疫细胞与肿瘤进化之间的关系,也许是有希望的解毒法
九大免疫相关基因组成预知模型,HCC确认为潜在生物标志在九大基因中,RBP2、NDRG1和HSPA在HCC与其他免疫相关基因比较后得到了很好的研究RBP2(retinol绑定蛋白2)归FABP家族所有43号..先前证明RBP2在高频委中表达过量,并关联到高频委不讨好预测44号..高清晰度RBP2与AFP和TNM阶段明显相关RBP2对HCC成形和进化可能至关紧要高度NDRG1表达式HCC45码..NDRG1建议在HCC的转移、复发和预测中发挥重要作用46号..高表达NDRG1显示与 hepatocarci47..目标NDRG1可能为HCC提供有吸引力的治疗策略HSPA4,又称hsp70,证明增强各种癌症的扩散、入侵和转移48号..HSPA4高表示式与HCC总体生存差大相联slo独立预测参数OS49号sad4显示作为HCC处理中治疗目标的巨大潜力HCC组织检测到高调ANGPT1表示法与正常肝组织对比50码,而预测 HCC隐含性没有研究先前的一项研究表明IL17D诊断值51号..华协IL17D的具体作用鲜为人知只有一个研究报告OSGIN1可能是HCC下调肿瘤抑制器52..确切作用HCC尚不清楚迄今没有相关研究报告HCCMAPT、DCC和SEMA3F需要进一步研究来说明HCC中这些潜在的免疫相关基因的功能
研究中的一些缺陷应解决第一,这项研究完全以公共数据库为基础,结果应当通过进一步的实验外部验证第二,应在使用实验方法的大量HCC样本中确定免疫相关预测模型的效率第三,HCC九大免疫相关基因的生物功能需要由一系列实验进一步检验
结论
最后,首次检测到许许多多与免疫相关基因与CC综合分析预测大相联此外,我们搭建了一个新的免疫预测模型,作为HCC独立预测器外部GCC数据库验证进一步确认该模型预测值预测模型也可以预测增强免疫细胞渗透,证明其在肿瘤免疫微环境中的关键作用当前研究加深了我们对HCC中免疫相关基因的理解,并提供了新的潜在预测和治疗生物标志
提供数据和资料
不适用
缩写
- HCC:
-
肝细胞癌
- TCGA:
-
癌症基因组地图集
- DEGs:
-
偏差表达式基因
- TFs:
-
纹理因子
- PD-1:
-
程序化手机死一
- GIC:
-
国际癌症基因组联合会
- Timers:
-
肿瘤免疫估计资源
- GO:
-
Gene文理数据库
- KEGG:
-
京都基因百科全书
- ROC:
-
接收者操作特征
- AUC:
-
ROC曲线下区域
- OS:
-
整体生存
引用
LlovetJM、Zucman-RossiJ、PikarskyE、SangroB、SchwartzM、ShermanM和GoresG肝细胞癌Nat RevDS素数2016年2:16018
RaoCVASY频繁变异基因/路径和基因组不稳定性作为肝癌预防目标卡西诺生成2017;38:2-11
AllemaniC公司、WeirHK公司、Carrela H公司、HarewoodR公司、SpikaD公司、Wang XS公司、BannonF公司、AhnJV公司、JohnsonCJ公司、BonaventryA公司等癌症生存全球监视1995-2009年:个人数据分析,来自67个国家的279个人口寄存器寄存器(COONCORD-2)的25 676 887名病人柳叶刀2015;385:977–1010.
Julit GR/AllemannP/LabgaaI/DemartinesN/Halkic循环肝细胞癌的处理和结果Langenbecks大桥Surg2017;402:737–44.
布鲁克斯JGJMZAZACHERROV肝细胞癌:临床前沿和透视口语2014;63:844-55
LlovetJM、RicciS、MazzaferroV、HilgardP、GaneE、BlancJF、de OliveraAC、SontoroA、RawelJL、FORNERA等Sorafenib高级肝细胞癌NEnglJMD2008;359:378–90.
Bruix J、QinS、MerleP、GranitoA、黄YH、BodokyG、PrachtM、YokosukaO、RosmorderO、BrederV等Regorafenib面向接受sorafenib处理的肝细胞癌患者:随机测试双盲安眠药控制阶段3柳叶刀2017;389:56-66
LinDC、Mayakondaa、Dinh HQ、HuangP、LinL、Liu X、DingLW、WangJ、BermanBP、SongEW等基因组异同异性肝细胞癌癌症解析2017;77:2255–65.
安卓J从免疫上下文到Imunoscore:预知性和预测性免疫标志作用Curr OpinImunol2013年;25:261-7
Gentles AJ、NewmanAM、LiuCL、BratmanSV、Feng W、KimD、NairVS、徐Y、KhuongA、HoangCD等预测基因景观 渗透性免疫细胞 遍历人类癌症纳特美德2015年;21:938-45
格里文尼科夫SI/GretenFR/KarinM豁免性、炎症和癌症手机内2010;140:883–99.
施密特R免疫作用产生并处理肝细胞癌狄格迪斯2016年;34:429-37
江RTANZLTENYY XAY高YWANXSInterleukin-22激活STAT3推广人体肝细胞癌赫帕托科2011/54:900-9
苏顿A号、FriandV号、Brue-DonnegerS号、ChaigneauT号、ZiolM号、Sainte-CatherineO号、PoireA号、SaffarL号、KraemerM号、VassyJ号等stromal细胞衍生因子-1/chemokine(C-X-Cmotif)ligand12刺激人体肝细胞生长、迁移和入侵Mole癌症解析2007年5:21-33
HouKZ、FuQQChemokineligand20增强肝细胞肿瘤的进化世界J气管2015年;21:475-83
Kabilova to、Kovtonyuk LV、Zonov EV、RyabchikovaEI、Popova NA、NikolinVP、KaledinVI、ZenkevaMA、VlassovVV和ChernolovskayaELImmno处理小双串RNA染色体BMC癌症201414338
福纳A ReigMBruixJ肝细胞癌柳叶刀2018;391:1301–14.
树井K新模S佐罗高桥Z无痛甲状腺炎和卷积型1型糖尿病患者Nivolumab东北JExped2018;244:33-40
El-KhooiryAB、SangroB、YauT、CrocenziTS、KudoM、HsuC、KimTY、ChooSP、TrojanJ、WellingTHR等高级肝细胞肿瘤患者Nivolumab柳叶刀2017;389:2492–502.
Deleon BA公司、Vasmatzis G公司、Truty MJ公司、Oklu R公司等小说定向治癌和肝细胞癌策略FutOncol2018;14:553-66
Budhu A、ForgesM、YeQH、JiaHL、HeP、ZanettiKA、KammulaUSS、Chen Y、QinLX、TangZY、Wang XW预测肝细胞癌的静态转移、复发和预测癌症细胞2006年;10:99-111
巴塔查里亚S公司、安道夫S公司、戈梅斯L公司、邓普公司、SchaeferH公司、庞地乌斯J公司、BergerP公司、DesboroughV公司、SmithT公司、CampbellJ公司等ImmPort:向公众传播未来免疫学数据imunolRes2014年;58:234-9
Mei S、Meyer CA、Zheng R、QinQ、WuQ、JangP、LiB、Shi X、WangB、FanJ等Cistrone癌症网络资源 综合基因规范建模癌症解析2017;77:e19-22
本体学CG基因本体学项目2006年核酸Res2006年;34:D322-6
KanehisaM,FurumichiM,TanabeM,SatoY,森岛KKEGG:基因组新视角、路径、疾病和药核酸Res2017;45:D353–d361.
余G王龙汉YHEQY集群Profiler:R包比较基因集群的生物主题OMICS2012年:16:284-7
香农P、MarkielA、OzierO、BaligaNS、WangJT、RamageD、AminN、SchwikowskiB和IdekerT子形环境生物分子交互网络综合模型软件环境基因组Res2003;13:2498–504.
Li、FanJ、WangB、TraughN、ChenQ、LiJS、LiB和LiXSETERR:网络服务器综合分析肿瘤渗透性免疫细胞癌症解析2017;77:e108–10.
塞拉米EGOJGARGRUSOZUGLSERSOOMERSOBAGOGERSYBAJOGSECBio癌症基因组门户:开放平台探索多维癌症基因组数据癌症磁盘2012年2:401至4.
Gao J、AksoyBA、Dogrusoz U、DresdnerG、Gross B、SumerSO、Sun Y、JacobsenA、SinhaR、LarssonE等综合分析复杂癌症基因组学和使用cBioPortal的临床剖面Sci信号2013年6:pl1
秀大大大大队、丰大队、赖大队、秦Y队、刘LM队、FUHX队、HERQ队、WUHYLG队、DangYW队、ChengG队、LuoDZ队高通量cRNA排序分析揭示出对硝化物机制新洞察力细胞死亡磁盘2019106.58
龙JWLLWANXLJLIJBIYWWWUJJJJZHOH四元预测模型预测患肝细胞癌病人总体生存J细胞MolMed2018;22:5928–38.
WangZ,TengD,LiY,HUZ,LiuLZH六元预知特征 肝细胞癌总体生存预测生活科学2018;203:83-91
LongJ、WangA、BaiY、LinJ、YangX、WangD、YangX、JiangY和Zha开发并验证TP53相关肝细胞癌免疫模型EBioMedicine公司2019;42:363-74
周治JshinHYLLJHLLLL内等离子细胞硬化细胞 算作治疗剖析后肝细胞癌的微弱预知因子癌症imunolImunun2019;68:1223–33.
广大大区 赵大区 大区 王大区 徐大区 宜新大区 郑大区周期性神经元子分解通过培养肝细胞癌动脉生成对疾病递增的炎症响应赫帕托尔2011/54:948-55
曼托瓦尼A/Cassatellama/CostiniC/JaillonS内生自适应豁免激活调控中的神经元Nat RevImunol201111519-31
LuC、RongD、ZhangB、ZhengW、WangX、ChenZ和TangW当前透视肝细胞肿瘤微环境:挑战与机遇Mole癌症201918130
天津湖湖湖湖湖湖湖湖湖湖湖宏字和肝细胞癌细胞Biosci2019979
吴SD、MaYS、FangY、LiuLL、FuD和ShenXZ微环境作用肝细胞癌癌症处理Rev2012年:38218-25
曼托瓦尼A/马切西F/马累西A/拉吉L/AllavenaP肿瘤相关宏图作为肿瘤学处理目标Nat RevClinOncol2017;14:399–416.
德格罗特H公司、范德伦东克A公司、GeertsA公司、Van ViererbergheH公司、DevischerL术前和临床治疗策略影响肝细胞癌中的肿瘤相关宏JimunolRes2018;2018:7819520.
梁三军、增军、WangL、FangM大队、WangQ大队、ZhaoM大队、徐三队、LiuZ队、LiW队、LiOS队等histonedemothase视网膜2蛋白过量表达出肝细胞癌并受hsa-miR-212负控PLOS一号20138:e69784
WangZY、YangJ、LiuCK和SQ高视波绑定蛋白2(RBP2)表达式医学科学Monit2017;23:2736–44.
丘亚MSSSSS过分表达NDRG1表示肝细胞癌预测不良模范Pathol2007年20:76-83
程J、秀X、WuJ、WeiX、SuR、ZhangW、LvZ、ZhengS和ZhouLNDRG1生物标识器处理肝细胞癌的转移、复发和不良预测癌症Lett2011年310:35-45
LuWJ,ChuaMS SoSK抑制N-Myc下游受控基因1恢复隐性信号并抑制脑细胞肿瘤生长卡西诺生成2014年;35:915-22
崇J江三M毛党卫士YuXN黄文热休克蛋白70抑制肝细胞扩散、迁移和肿瘤农科LETT2017;14:2703-8
MaC、XuT、SunX、ZhangS、LiOS、FanS、LeiC、TangF、ZhaiC、LiC等网络药理学和生物信息学方法揭示Baicalein在肝细胞肿瘤作用的治疗机制Evid基础补代Med2019;2019:7518374.
TorimuraT公司、UenoT公司、KinM公司、原田R公司、谷口E公司、中村T公司、SakataR公司、Hashmoto O公司、SakamotoM公司、KumashiroR公司等agiopoietin-1和angioietin-2赫帕托尔2004;40:799–807.
孙大元大元大元大元大元大元大元大元大元大元大元大元大综合分析全基因组脱氧和基因表达数据FEBS开放生物2018;8:1093–103.
刘MLLLLLTHLLLULLTTTETTETDDAYYDLLYYHLIYETAL单量偏移氧化应激抑制器Gastroenterology.2014;146:1084–96.
Acknowledgements
作者们衷心感谢分享癌症基因组地图集数据库和国际癌症基因组联合会数据库中的数据
供资问题
资助机构:山东省医院中国自然科学基金会创业基金
赠款数:QYPY2019NSFC0803
作者信息
作者和附属关系
交文
ZW构思研究并进行生物信息分析ZW和JZ下载并整理临床和基因表达数据ZW、YL和WW进行了统计分析ZWJZ和CL写手稿FC和LM严格修改文章基本知识内容和行政支助所有作者阅读并批准最终手稿
对应作者
道德申报
道德核准并同意参赛
不适用
协议发布
不适用
竞技兴趣
作者声明他们没有竞技兴趣
附加信息
发布器注解
伟德体育在线Springer自然对发布地图和机构附属关系中的司法请求保持中立
权限和权限
开放存取文章依据创用CC4.0国际许可使用,允许使用、分享、改编、分发和复制任何介质或格式,只要你适当信任原创作者和源码提供链接并显示是否修改图片或其他第三方素材均载入文章Creative公共许可中,除非素材信用栏中另有表示素材未载入文章Creative公共许可中, 法定规则不允许使用或超出许可使用范围时, 需直接从版权持有者处获取许可 。查看许可副本访问http://creativecommons.org/licenses/by/4.0/.创用公共域免责http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非信用线对数据另有说明。
关于此文章
点上此文章
王珠珠珠et al.开发并验证肝细胞癌新免疫预测模型J Transl me18号67(2020)https://doi.org/10.1186/s12967-020-02255-6
接收:
接受:
发布:
多尔市:https://doi.org/10.1186/s12967-020-02255-6
关键字
- 肝细胞癌
- 免疫相关基因
- 预测
- 预测式签名
- 生物信息学