摘要
背景
免疫检查点抑制剂在某些肺腺癌(LUAD)病例中是有效的。全外显子组测序显示,肿瘤突变负担(TMB)与免疫检查点抑制剂患者的临床获益相关。已经开发了几种商业突变面板来评估TMB,而不考虑癌症类型。然而,不同的癌症类型有不同的突变景观;因此,本研究旨在开发一个小型癌症类型特异性突变组,用于高精度估计LUAD患者的TMB。
方法
我们为LUAD患者开发了一种基于编码序列(CDSs)而非基因的小型癌症类型特异性突变面板。利用肿瘤基因组图谱(TCGA)数据库中486例LUAD患者的体细胞CDSs突变数据,我们预先选择了一组突变状态与TMB显著相关的CDSs,并使用遗传算法从中选择了与TMB最显著相关的CDS突变组。
结果
研究人员开发了一个包含106个CDSs的100个基因的突变组,其长度仅为0.34 Mb,远短于目前商业突变组的0.80-0.92 Mb。该突变组与TMB的相关性在两个独立的LUAD数据集中得到了验证,这些数据分别包含nivolumab + ipilimumab和pembrolizumab免疫疗法患者的无进展生存数据。在两个测试数据集中,生存分析显示,通过106-CDS突变面板(切点为每兆酶6.20个突变)预测的高TMB患者,训练数据集中的中位数面板评分,比预测TMB低的患者有明显更长的无进展生存(log-rank)p= 0.0018, hr = 3.35, 95% ci 1.51 ~ 7.42;log-rankp= 0.0020, hr = 5.06, 95% ci 1.63 ~ 15.69)。这个小小组比目前的商业突变小组更好地预测免疫治疗的疗效。
结论
仅0.34 Mb的small-CDS突变板优于现有的商业突变板,能更好地预测LUAD患者的免疫治疗效果,且成本低、时间密集,更适合临床应用。
背景
肺腺癌是最常见的非小细胞肺癌,约占肺癌病例的80-90% [1].目前,根据国家肿瘤综合网络临床实践指南,大约35-75%的LUAD患者在接受常规治疗后5年内复发或死亡[2].最近,通过激活免疫系统来消除肿瘤的免疫疗法[3.],显示出对NSCLC的巨大希望[4,5].例如,免疫检查点抑制剂nivolumab靶向程序性细胞死亡蛋白-1 (PD-1),可以通过阻断PD-1与其配体程序性死亡配体1 (PD-L1)之间的相互作用,并允许细胞毒性T淋巴细胞作用于肿瘤细胞,从而显著提高晚期NSCLC的生存率[6].此外,抑制免疫反应的细胞毒性T淋巴细胞相关抗原4 (CTLA-4)抑制剂(ipilimumab)已被批准用于治疗非小细胞肺癌[5]和其他一些癌症[7].然而,免疫检查点抑制剂反应的异质性极大地混淆了NSCLC的治疗[3.].因此,确定可能受益于这些免疫检查点抑制剂的患者是很重要的。
此前,非小细胞肺癌患者中PD-L1蛋白表达已被批准作为某些PD-1/PD-L1抑制剂(包括派姆单抗)的辅助预测标志物[8].然而,PD-L1蛋白表达本身并不能完全解释免疫检查点抑制剂治疗患者的生存获益[8,9,10,11].此外,由于病理学家使用不同的标准或解释对免疫染色结果进行主观诊断,因此通过免疫组化(IHC)分析PD-L1蛋白表达具有挑战性[12].
先前的几项研究报告了通过全外显子组测序(WES)确定的高肿瘤突变负担(TMB),表明患者更有可能携带新抗原,可以预测对免疫治疗的敏感性[13,14].例如,高tmb患者与nivolumab (PD-1抑制剂)+ ipilimumab (CTLA-4抑制剂)免疫治疗的增强反应相关[15].此外,高TMB与对PD-1/PD-L1抑制剂的反应比与通过免疫组化检测的PD-L1蛋白表达更显著相关[16].然而,测定TMB所必需的WES在临床实践中并不是常规的,因为它昂贵、耗时和劳动密集,并且需要大量的序列[3.,17,18].先前的研究已经报道,使用包含数百个基因的更小的基因组,如324基因突变组(FoundationOne CDxTM检测),可以准确地估计TMB [6,19,20.,21]和341基因突变组(MSK-IMPACT) [22,23],已被临床应用。这些突变面板的成本效益比WES更有利于测序深度,因此具有更高的检测突变的能力,即使是某些肿瘤细胞中的突变基因[24].然而,这些商业突变面板是从与癌症相关的基因中选择的,而不管癌症类型,而不是通过特征选择方法开发的;因此,突变面板仍然可以改进。特别是,由于不同癌症类型有不同的突变景观,有必要建立一个癌症类型特异性突变面板来估计LUAD样本的TMB [25].最近,Lyu等人。[3.]构建了LUAD特异性的24基因模型,用于预测LUAD样本的TMB。然而,该面板也是基于面板基因的完整外显子,由面板基因中的数千个外显子组成,其中大多数外显子未发生突变,这只会增加不必要的测序成本和时间。
在本研究中,我们基于LUAD中突变频率较高的编码序列(CDSs),建立了CDS突变面板来估计LUAD样本的TMB。随后,我们在两个独立的数据集中确定了突变组中CDSs与TMB的相关性。从两个数据集(Matthew和Rizvi)中,我们使用免疫检查点抑制剂治疗的晚期LUAD患者的无进展生存(PFS)数据来估计CDS突变组用于预测免疫治疗疗效的性能。此外,CDS突变组与两个商业突变组(324基因和341基因组)和一个luad特异性突变组(24基因组)进行了比较。
方法
数据来源和预处理
3个LUAD体细胞突变数据集(表1)构建并验证用于估计TMB的突变面板。训练突变数据从The Cancer Genome Atlas (TCGA)数据库(https://portal.gdc.cancer.gov/),包括486个LUAD样本,并配对mRNA表达数据。为了验证,我们获得了两个独立的体细胞突变数据集,其中包括Matthew等人报道的59个LUAD样本。5]和Rizvi等人报道的29个LUAD样本[4].Matthew数据集中的患者接受nivolumab (PD-1抑制剂)+ ipilimumab (CTLA-4抑制剂)治疗,Rizvi数据集中的患者接受pembrolizumab (PD-1抑制剂)免疫治疗。
之前对这些TCGA数据与肿瘤组织和匹配的正常组织或血液进行了全外显子组测序,用于过滤种系突变和筛选体细胞突变[26];详细方案见原始文献[27].简单地说,从每个样品中提取0.5-3µg DNA用于文库制备,并使用Illumina HiSeq平台进行测序。肿瘤和种系DNA靶向碱基的平均覆盖率分别为97.63和95.83。排除肿瘤组织中等位基因变异分数< 0.05的突变。只包括非同义突变,包括错义突变、无义突变、不间断突变、帧移突变和帧内突变,生成包含16,961个基因的82,574个cds的离散突变谱。对于两个测试突变数据,对肿瘤组织和匹配的正常组织或血液进行全外显子组测序。详细方案在原始文献中有进一步描述[5,28].最后,生成包括9400个基因的18793个CDSs和5504个基因的8711个CDSs的离散突变谱,其中CDSs突变矩阵使用来自GENCODE (https://www.gencodegenes.org/human/releases.html).
用于估计TMB的CDS突变面板的开发
首先,我们从TCGA LUAD体细胞突变数据中,利用人类参考基因组注释文件(GRCh38)提取CDSs中的突变,并选择非同义突变构建CDSs米*ncds突变矩阵,其中米为基因中cds的数量n表示样本数量。TMB估计为(CDSs总突变数/ CDSs总碱基)* 106.
然后进行Spearman秩相关分析,估计CDSs突变状态与TMB的相关性。在此,我们将分析限制在超过5%的癌症样本中突变的CDSs [29,30.过滤掉低频突变的“乘客”基因,因为它可能受到随机突变的影响,而不是具有致瘤优势。使用Benjamini-Hochberg程序调整p值[31]用于多次测试以控制错误发现率(FDR)。选择与TMB显著相关的cds作为候选。
最后,使用遗传算法(GA包)从候选CDS中生成最终CDS面板,其面板得分与TMB相关性最大。遗传算法在种群规模为5000,交叉分数为0.9的情况下实现;如果在100代中最佳子集的优化目标没有得到改善,则终止。关于遗传算法的细节在附加文件中显示1.相关性(R)2)通过线性回归分析估计[32].在这里,面板得分计算如下(公式1):
在哪里n为面板中cds的数量,l面板的长度,和\ (k_{我}\)突变的数量是多少我th cd;β\ (\ \)而且\ (C \)通过线性回归分析得到,β\ (\ \)为平衡panel-score和TMB的系数,\ (C \)是常数。
由于没有关于TCGA患者免疫治疗的临床数据,我们无法确定用于预测免疫治疗疗效的CDS面板的最佳切点。因此,我们将CDS面板的切点设置为TCGA中的中位数面板分数。
生存分析
PFS被定义为在疾病治疗期间和之后的一段时间,其中患者患有疾病,但病情没有加重。使用Kaplan-Meier方法估计生存曲线,并使用log-rank检验进行比较(生存包:' survdiff ') [33].使用单变量Cox比例风险回归模型(生存包:' coxph ')来评估突变组的预测性能。此外,在调整了包括年龄、性别和吸烟在内的临床因素后,使用多变量Cox模型(生存包:' coxph ')来评估我们的CDS突变组的独立预后价值。使用Cox比例风险模型(生存包:' Cox ')生成风险比(HRs)和95%置信区间(ci)。
功能富集分析
富集分析的功能通路于2018年11月从基因本体论(GO)下载。首先,我们使用5% FDR对照进行Student 's t检验,以在CDS小组分类的高tmb组和低tmb组之间选择差异表达基因(DE基因)。在这里,17680个基因被用于差异表达分析。然后,利用超几何分布模型,通过随机概率来判断在一个功能项中观察到的DE基因数量是否显著大于预期。
所有统计分析均采用R软件包3.4.2版(http://www.r-project.org/).重要性定义为p多重试验< 0.05或FDR < 0.05。
结果
CDS突变面板的构建
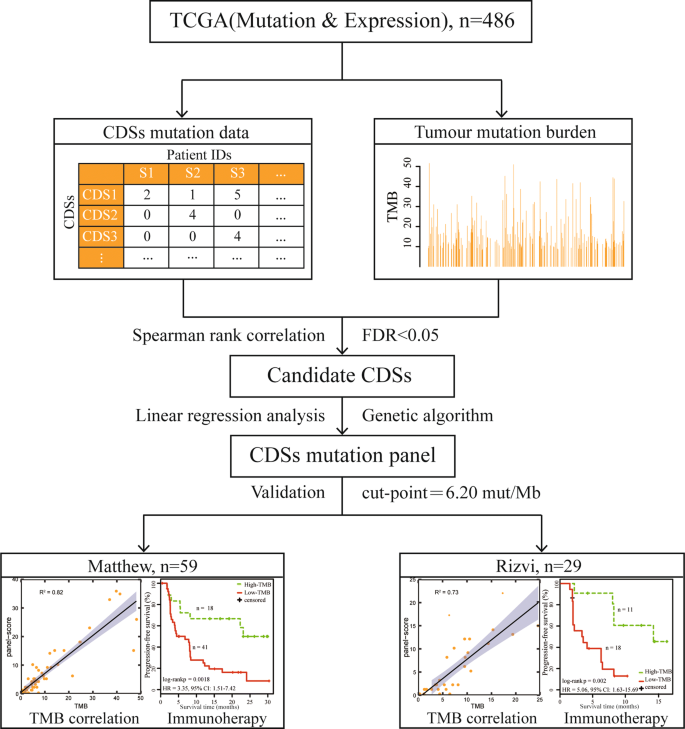
数字1提供研究方案的示意图。
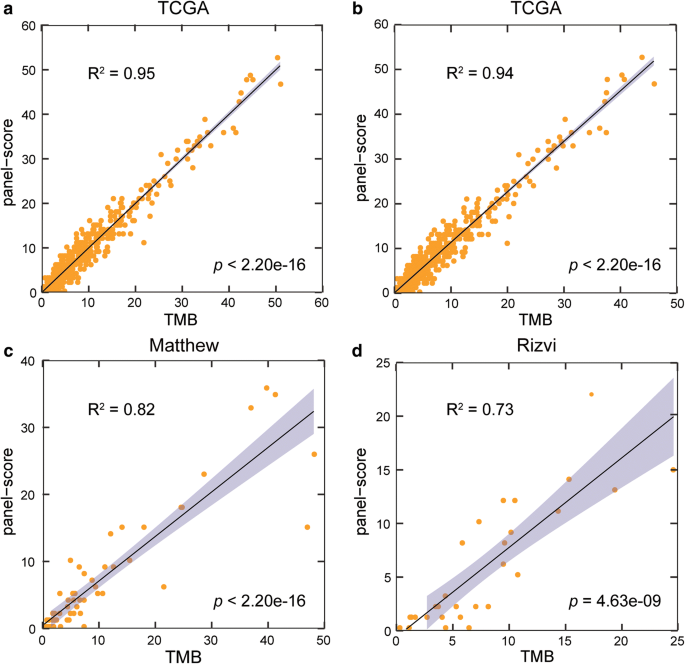
从TCGA的LUAD突变数据中,我们使用人类参考基因组注释文件(GRCh38)从CDSs中提取突变数据。在选择非同义突变后,生成了包含82574个CDSs和486名患者的突变矩阵。随后,采用Spearman秩相关分析,在5% FDR对照下,219个cds与TCGA数据衍生的LUAD样本的TMB显著相关。使用遗传算法("方法),我们提取了100个基因中106个CDS组成的CDS面板,长度为0.34 Mb(附加文件2:表S1),其面板评分与LUAD样本的TMB相关性最显著(R2= 0.95,图2a).该突变组被称为106-CDS组。在小组得分公式中,\(\beta = 0.33\),\({\text{C = 0}}。2 7 \)均由线性回归分析[32].附加文件中描述了用于TMB评估的106-CDS面板的技术细节3.:表S2。
此外,我们利用Spearman秩相关估计了临床因素和肿瘤细胞比例与TMB的相关性,发现只有年龄和性别与TMB显著相关(年龄:p= 0.0055;性:p= 0.0442,附加文件4:表S3)。因此,我们对106-CDS组、年龄和性别一起使用多元线性回归模型来估计它们与TMB的相关性。因此,106-CDS组与TMB仍显著相关,而年龄和性别不相关,表明106-CDS组是TMB的独立预测因子。此外,为了防止高估TMB,由于基因组通常大量针对复发突变的基因组区域,我们在排除癌症相关基因突变和超过5%的样本中发生复发性CDSs后重新确定了TMB,并发现相关性(R2重新计算TMB的106-CDS面板的)也接近0.94(图。2b)。
106-CDS面板验证
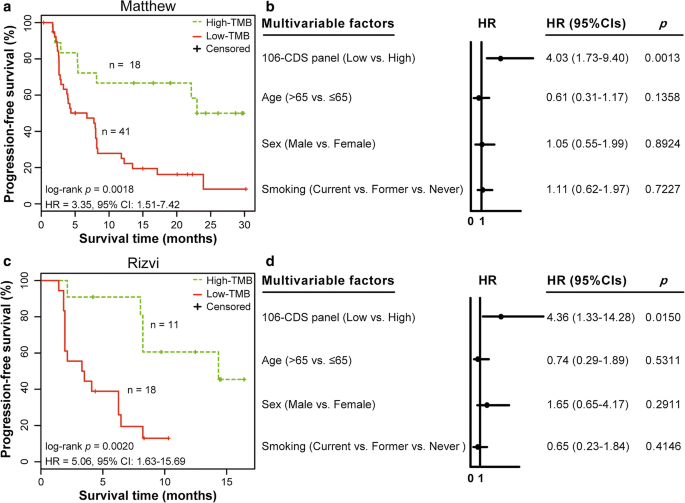
首先,我们将106-CDS面板应用于Matthew数据集中的59个样本,其中包括接受nivolumab + ipilimumab免疫治疗的患者的PFS数据。相关性(R)2106-CDS的面板评分与TMB之间的差异为0.82(线性回归分析,p< 0.0001,图;2c).当小组评分按照6.20个突变/兆酶(mut/Mb)进行二分类时,我们的106-CDS小组将18名和41名患者分别分为高危组和低危组。单因素生存分析显示,预测的高tmb患者的PFS明显长于预测的低tmb患者(log-rankp= 0.0018, HR = 3.35, 95% CI 1.51 ~ 7.42,图;3.a).预测高tmb患者的1年PFS率为0.67,显著高于预测低tmb患者的1年PFS率(0.25)。多变量Cox分析显示,切点为6.20 mut/Mb的106-CDS组与PFS显著相关(p= 0.0013, HR = 4.03, 95% CI 1.73 ~ 9.40,图;3.b)在调整年龄(> 65 vs.≤65岁)、性别(男性vs.女性)和吸烟状况(目前vs.以前vs.从不)后。
106-CDS面板在测试数据集中预测免疫治疗疗效的性能。一个Matthew数据集中59例晚期肺腺癌(LUAD)患者无进展生存(PFS)的Kaplan-Meier曲线。的p数值采用log-rank检验确定。使用单变量Cox回归模型确定风险比(HR)和95%可信区间(CI)。b在Matthew数据集中对106-CDS面板、年龄、性别和吸烟状况进行多元Cox分析。实圈代表死亡风险的HR,开放式水平线代表95% CI。的p使用多元Cox回归模型确定值、HR和CI。cRizvi数据集中29例晚期LUAD患者PFS的Kaplan-Meier曲线。的p数值采用log-rank检验确定。HR和95% CI采用单变量Cox回归模型确定。d在Rizvi数据集中对106-CDS组、年龄、性别和吸烟状况进行多元Cox分析。的p使用多元Cox回归模型确定值、HR和CI
Rizvi数据集也得到了类似的结果,其中相关性(R2)与TMB之间为0.73(线性回归分析,p< 0.0001,图;2d).使用切点为6.20 mut/Mb的106-CDS面板预测的高tmb患者的PFS明显长于预测的低tmb患者(log-rankp= 0.0020, HR = 5.06, 95% CI 1.63 ~ 15.69,图;3.c).预测的高tmb患者的1年PFS率为0.61,显著高于预测的低tmb患者的1年PFS率(0.13)。多变量Cox分析显示,切割点为6.20 mut/Mb的CDS组与PFS显著相关(p= 0.0150, HR = 4.36, 95% CI 1.33-14.28,图3.D)在调整年龄(> 65 vs.≤65岁)、性别(男性vs.女性)和吸烟状况(目前vs.以前vs.从不)后。
106-CDS组与3个突变组的比较
我们将106-CDS面板与两个商业突变面板(324基因[6,20.,21]和341基因[23由食品和药物管理局(FDA)批准的),以及一个LUAD特异性突变小组(24个基因小组),用于预测LUAD免疫治疗的疗效。单因素生存分析结果汇总于表中2.
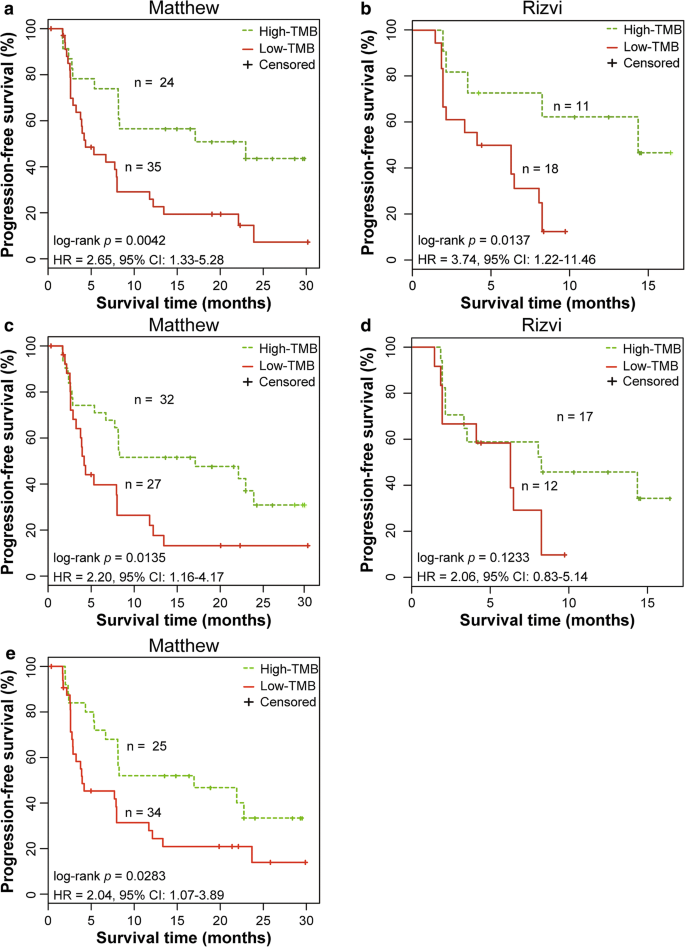
剪切点为10 mut/Mb的324基因突变面板[19],包含6130个跨度为0.80 Mb的CDSs,单因素生存分析显示,在Matthew数据集中,使用该小组分类的两组患者在接受免疫治疗后的PFS有显著差异(log-rank p = 0.0042, HR = 2.65, 95% CI 1.33-5.28,图。4a),在Rizvi数据集中(log-rank p = 0.0137, HR = 3.74, 95% CI 1.22-11.46,图。4b).然而,单因素生存结果显示,在两个数据集中,其在预测免疫治疗疗效方面的表现(HR)比我们的106-CDS小组差(表2)2).
在测试数据集中预测免疫治疗疗效的其他突变组的表现。一个Matthew数据集中59例晚期肺腺癌(LUAD)患者的无进展生存(PFS) Kaplan-Meier曲线,使用324基因组。bRizvi数据集中29例晚期LUAD患者PFS的Kaplan-Meier曲线,使用324基因组。cMatthew数据集中59例患者PFS的Kaplan-Meier曲线,使用341个基因组。dRizvi数据集中29例患者PFS的Kaplan-Meier曲线,使用341基因组。eMatthew数据集中59例患者的PFS Kaplan-Meier曲线,使用luad特异性24基因组。的p数值采用log-rank检验确定。使用单变量Cox回归模型确定风险比(HR)和95%可信区间(CI)
剪切点为7.40 mut/Mb的341基因突变组[22],包含6773个CDSs,跨度为0.92 Mb,其在Matthew数据集(log-rankp= 0.0135, HR = 2.20, 95% CI 1.16-4.17,图4c)和Rizvi数据集(log-rank . c)p= 0.1233, HR = 2.06, 95% CI 0.83-5.14,图4d)低于我们的106-CDS面板(表2).
对于切割点为141的luad特异性突变组(24基因组)[3.],包含833个跨度为0.18 Mb的CDSs,单因素生存分析显示,在Matthew数据集(log-rank .)中,由小组分类的两组患者在免疫治疗后的PFS有显著差异p= 0.0283, HR = 2.04, 95% CI 1.07-3.89,图;4e).然而,它的性能(HR)比我们的106-CDS面板差得多(表2).由于Rizvi数据集是用于确定24基因组在预测派姆单抗免疫治疗益处时的切点(141)的训练集,我们没有将我们的106-CDS组与数据集中的24基因组进行比较,因为它不是24基因组的独立测试数据。
106-CDS面板的功能特性
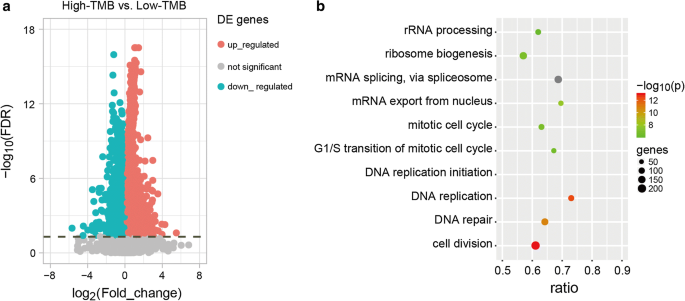
在TCGA数据集中,采用切点为6.20 mut/Mb的106-CDS面板,将220个样本分为高tmb组,266个样本分为低tmb组。我们发现两组间有7181个基因差异表达(Student’s t检验,FDR < 0.05,图。5a,附加文件5:表S4),在22个功能通路中均显著富集(超几何分布模型,FDR < 0.05,图4。5b、附加文件6:表S5),包括与基因组不稳定性相关的基因,如DNA修复[34], DNA复制[35]和染色体分离[36].这些结果表明,与使用106-CDS组预测的低tmb患者相比,预测的高tmb患者可能具有更高的基因组不稳定性,因此可能受益于免疫治疗,因为他们更有可能拥有新抗原。
讨论
本研究描述了由100个基因的106个CDSs组成的突变面板的生成,跨度为0.34 Mb。先前的研究报道了由300多个癌症相关基因组成的测序面板可以帮助预测TMB;然而,当面板中的基因数量小于150时,其性能明显较低[37].然而,这些商业突变面板(如324基因和341基因面板)并没有通过任何特征选择方法选择;因此,它们与TMB的高相关性主要是由于面板中包含大量的基因。相比之下,我们使用遗传算法开发的106-CDS突变面板,包含更多与TMB相关的主要变量,预计在估计TMB时是可靠的,其性能在两个独立的测试数据集中得到了验证。在两个测试数据集中,我们的106-CDS面板和TMB的相关性存在某些差异,因此可能解释了它们不同的样本量或样本集合;这些相关性需要在大规模临床试验中进一步验证。
目前的结果表明,106-CDS的切点为6.20 mut/Mb,较好地预测了晚期LUAD患者的免疫治疗效果。对于通过106-CDS(切点为6.20 mut/Mb)组预测的高tmb患者,nivolumab + ipilimumab免疫治疗将1年PFS率提高到0.67,显著高于预测的低tmb患者的1年PFS率(0.25)。同样,预测的高tmb患者的1年PFS率为0.61,明显高于预测的低tmb患者在派姆单抗治疗后的1年PFS率(0.13)。然而,我们认为,106-CDS面板的切点设置在训练数据集中的中位数面板评分,可能不是预测各种免疫治疗药物疗效的最佳阈值。为了评估特定切点对预测免疫治疗疗效的影响,我们另外将CDS面板的切点分别设置在训练数据集面板评分的上百分位数(9.17 mut/Mb)和四分位数(12.13 mut/Mb),并在两个测试数据集中进行估计。单因素生存分析显示,具有上四分位数切点(12.13 mut/Mb)的106-CDS组具有最佳预测性能(log-rank)p= 0.0079, HR = 3.81, 95% CI 1.33-10.93,附加文件7:图S1A)比中位数(log-rank .p= 0.0018, HR = 3.35, 95% CI 1.51 ~ 7.42,图;3.A)和上百分位(log-rankp= 0.0298, HR = 2.59, 95% CI 1.07-6.27,附加文件7:图S1B)作为Matthew数据集中nivolumab + ipilimumab治疗患者的切割点。然而,它的性能最差(log-rankp= 0.1258, HR = 2.58, 95% CI 0.72-9.21,附加文件7:图S1C)比中位数(log-rank .p= 0.0020, HR = 5.06, 95% CI 1.63 ~ 15.69,图;3.C)和上百分位(log-rankp= 0.0081, HR = 5.82, 95% CI 1.33-25.51,附加文件7:图S1D)用于Rizvi数据集中接受pembrolizumab治疗的患者。这些结果表明,以6.20 mut/Mb为切点的106-CDS面板可以有效预测可能受益于免疫治疗的患者,但特定免疫治疗药物的最佳切点需要在大规模临床试验中进一步探索。
突变组中包含的基因数量越多,与TMB的预期相关性越高。我们的研究结果表明,尽管106-CDS组的基因数量比324- cds组的基因数量少两倍[19]和341基因[22]面板,我们的106-CDS面板在预测免疫治疗的疗效方面表现更好。虽然106-CDS组(0.34 Mb)的长度比24-基因组(0.18 Mb)长,但其在预测免疫治疗疗效方面的表现明显更好。这些结果表明106-CDS突变组可能具有更高的抗原性,有待进一步证实。
功能注释显示包括TP53在内的多个基因[38], amer1 [39]和TEX15 [40]在106-CDS面板中参与DNA修复和细胞周期阻滞,在基因组不稳定性中发挥关键作用。使用切点为6.20 mut/Mb的106-CDS面板分类的两组DE基因在与基因组不稳定性相关的几个途径中显著富集,如DNA修复[34], DNA复制[35],以及染色体分离[36].这些功能分析表明,与使用106-CDS组预测的低tmb患者相比,高tmb患者可能具有更高的基因组不稳定性,更有可能含有新抗原。
结论
CDS突变面板仅为0.34 Mb,可通过准确估计TMB,有效预测LUAD患者的免疫治疗效果。这种小面板由于成本低、耗时短,是临床样品的首选。
缩写
- LUAD:
-
肺腺癌
- 非小细胞肺癌:
-
非小细胞肺癌
- PD-1:
-
程序性死亡1
- PD-L1:
-
程序性死亡配体1
- CTLA-4:
-
细胞毒性T淋巴细胞相关抗原
- 包含IHC:
-
免疫组织化学
- 三甲:
-
肿瘤突变负担
- 韦斯:
-
whole-exome测序
- 信用违约互换:
-
编码序列
- PFS:
-
无进展生存
- TCGA:
-
癌症基因组图谱
- 罗斯福:
-
错误发现率
- 小时:
-
风险的比率
- 顺式:
-
置信区间
- 走:
-
基因本体论
- 德基因:
-
差异表达基因
- 食品药品监督管理局:
-
食品和药物管理局
参考文献
Chansky K, Sculier J-P, Crowley JJ, Giroux D, Van Meerbeeck J, Goldstraw P,等。国际肺癌分期研究协会:手术治疗非小细胞肺癌的预后因素和病理TNM分期中华外科杂志。2009;4:792-801。
Siegel RL, Miller KD, Jemal A.癌症统计。中华癌症杂志2015;
吕桂英,叶永华,叶永昌,王永昌。突变负荷估计模型作为癌症免疫治疗反应的预测因子。NPJ基因组医学2018;3:12。
Rizvi NA, Hellmann MD, Snyder A, Kvistborg P, Makarov V, Havel JJ,等。癌症免疫学。突变格局决定非小细胞肺癌中PD-1阻断的敏感性。科学。2015;348:124-8。
helmann MD, Nathanson T, Rizvi H, Creelan BC, Sanchez-Vega F, Ahuja A,等。晚期非小细胞肺癌患者联合免疫治疗反应的基因组特征癌症杂志,2018;33(843-852):e4。
Chalmers ZR, Connelly CF, Fabrizio D, Gay L, Ali SM, Ennis R,等。对10万个人类癌症基因组的分析揭示了肿瘤突变负担的格局。基因组医学。2017;9:34。
张文杰,李志强,李志强,等。联合尼伏单抗和伊匹单抗治疗脑转移性黑素瘤。中华医学杂志。2018;39:722 - 30。
李志强,李志强,刘志强,孙志强,等,Rodríguez-Abreu D。Pembrolizumab vs化疗治疗pd - l1阳性非小细胞肺癌中华医学杂志(英文版),2016;
Hui R, Garon EB, Goldman JW, Leighl NB, Hellmann MD, Patnaik A,等。派姆单抗作为pd - l1阳性晚期非小细胞肺癌患者的一线治疗:一项一期试验安·昂科尔,2017;28:874-81。
Carbone DP, Reck M, Paz-Ares L, Creelan B, Horn L, Steins M,等。nivolumab用于IV期或复发性非小细胞肺癌的一线治疗。中华外科杂志,2017;
Garon EB。癌症免疫治疗试验不能避免不精确的患者选择。中华医学杂志。2017;37:2483 - 5。
Brunnström H, Johansson A, westbomh - fremer S, Backman M, Djureinovic D, Patthey A,等。PD-L1免疫组化在肺癌临床诊断中的应用:病理学家间变异性高于检测变异性。Mod Pathol. 2017; 30:1411-21。
Koeppel F, Blanchard S, Jovelet C, Genin B, Marcaillou C, Martin E,等。全外显子组测序测定晚期癌症患者液体活检中肿瘤突变负荷。PLoS ONE。2017; 12: e0188174-e0188174。
李志强,李志强,李志强,等。免疫治疗非小细胞肺癌患者肿瘤突变负担的临床应用。翻译肺癌Res. 2018; 7:647-60。
Ready N, Hellmann MD, Awad MM, Otterson GA, Gutierrez M, Gainor JF,等。nivolumab + ipilimumab治疗晚期非小细胞肺癌(CheckMate 568):通过程序性死亡配体1和肿瘤突变负担作为生物标志物的结局中华临床医学杂志2019;37:992-1000。
Teixidó C, Vilariño N, Reyes R, Reguart N. PD-L1在非小细胞肺癌中的表达检测。中华医学杂志。2018;10:1758835918763493。
Meléndez B, Van Campenhout C, Rorive S, Remmelink M, Salmon I, D’haene N.肿瘤组织中肿瘤突变负荷的测量方法。翻译肺癌Res. 2018; 7:661-7。
Allgäuer M, Budczies J, Christopoulos P, Endris V, Lier A, Rempel E,等。在常规诊断中实施肿瘤突变负担(TMB)分析-分子病理学家和临床医生的引物。翻译肺癌Res. 2018; 7:703-15。
helmann MD, Ciuleanu T-E, Pluzanski A, Lee JS, Otterson GA, Audigier-Valette C,等。Nivolumab联合ipilimumab治疗肿瘤突变负担高的肺癌。中华外科杂志,2018;
王凯,王宁,何杰,等。基于大规模并行DNA测序的临床癌症基因组分析测试的开发和验证。中国生物工程学报,2013;
张志刚,张志刚,张志刚。PD-1抑制对肿瘤突变负担和应答率的影响。中华外科杂志。2017;37:2500 - 1。
李丽娟,李丽娟,李丽娟,等。非小细胞肺癌患者抗程序性细胞死亡(PD)-1和抗程序性死亡配体1 (PD- l1)阻断反应的分子决定因素中华临床医学杂志,2018;36:633-41。
程DT, Mitchell TN, Zehir A, Shah RH, Benayed R, Syed A,等。纪念斯隆凯特林-可操作癌症靶点的综合突变分析(MSK-IMPACT):实体肿瘤分子肿瘤学的基于杂交捕获的下一代测序临床分析。中国生物医学工程杂志,2015;17(2):326 - 326。
Heemskerk B, Kvistborg P, Schumacher TNM。癌症抗基因组。Embo j . 2013; 32:194-203。
Lawrence MS, Stojanov P, Polak P, Kryukov GV, Cibulskis K, Sivachenko A,等。癌症的突变异质性和寻找新的癌症相关基因。大自然。2013;499:214-8。
王晓燕,王晓燕,王晓燕,等。肿瘤分析方法和基因组数据策略对癌症精准医疗的影响。基因组医学。2016;8:79。
网络CGAR。肺腺癌的综合分子谱分析。大自然。2014;511:543-50。
Havel JJ, Bruggeman C, Wong P, Moreira AL, Merghoub T, Garon EB,等。突变格局决定非小细胞肺癌中PD-1阻断的敏感性。科学(80 -)。2015; 348:124-8。
于俊,吴文坤,李旭,何俊,李晓霞,吴森明,等。结直肠癌中新的复发突变基因和预后突变特征。肠道。2015;64:636-45。
齐琳,李涛,石刚,王杰,李霞,张松,等。预测肺腺癌转移的个体化基因表达特征。Mol Oncol, 2017; 11:1630-45。
benjamin Y, Hochberg Y.控制错误发现率:多重测试的一个实用而强大的方法。中国社会科学院学报(自然科学版),2001;
Schneider A, Hommel G, Blettner M.线性回归分析:科学出版物评价系列的第14部分。Dtsch Arztebl Int. 2010; 107:776-82。
布兰德JM,阿尔特曼DG。洛格兰克检验。BMJ。2004; 328:1073。
Kastan MB, Bartek J.细胞周期检查点与癌症。大自然。2004;432:316-23。
马文杰,李文杰,李文杰,李文杰,李文杰,李文杰,李文杰,李文杰。大自然。2018;559:279 - 84。
钟安,陈培林,陈勇,常明,高锐,赖利,等。高糖条件下异常DNA损伤反应和DNA修复途径。J Can Res Updates. 2018; 7:64-74。
李志强,李志强,李志强,等。在临床实践中,综合癌症基因面板可用于估计PD-1阻断的突变负荷和预测临床获益。Oncotarget。2015;6:34221-7。
Vogelstein B, Kinzler KW. p53功能和功能障碍。细胞。1992;70:523-6。
Kim MKH, Min DJ, Rabin M, Licht JD。Wilms肿瘤抑制因子WTX和肿瘤相关突变体的功能特征。致癌基因。2010;30:832。
林霞,陈震,高鹏,高震,陈宏,齐杰,等。TEX15:与汉族前列腺癌风险相关的DNA修复基因。前列腺癌。2017;77:1271-8。
确认
一个也没有。
资金
国家自然科学基金(批准号:61701143、81872396、61673143)、福建省联合科技创新基金(批准号:2016Y9044)资助。
作者信息
作者及隶属关系
贡献
LSQ构思并设计了实验,并撰写了手稿。LA设计了分析方法并撰写了手稿。ZG想出了这个主意。YL和WBJ设计并进行了实验。MYL对数据进行了处理和分析。XL和ZYZ进行实验。SNZ和YXL收集和处理数据。WYZ和YYG解释数据。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1。
用于CDS面板搜索的遗传算法。
附加文件2:表S1。
106-CDS面板描述。
附加文件3:表S2。
TMB评估的技术细节。
附加文件4:表S3。
临床因素及肿瘤细胞比例与肿瘤突变负荷的相关性。
附加文件5:表S4。
通过106-CDS面板预测高tmb组和低tmb组之间不同表达的基因列表。
附加文件6:表S5。
不同表达基因富集的功能通路。
附加文件7:图S1。
106-CDS组的表现与其他预测免疫治疗疗效的切点。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
李,Y.,蒋,W.,李,T。et al。鉴定编码序列的小突变组以预测肺腺癌免疫治疗的疗效。翻译医学杂志18, 25(2020)。https://doi.org/10.1186/s12967-019-02199-6
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12967-019-02199-6
关键字
- 免疫疗法
- 肿瘤突变负担
- CDS突变组
- 肺腺癌
- 临床应用