摘要
背景
脓毒症仍然是重症监护病房面临的一大挑战,由于缺乏具有足够敏感性的快速诊断工具,脓毒症的死亡率高得令人无法接受。因此,迫切需要用一种新的方法取代费时的血液培养。理想情况下,这种方法还提供了病原菌的全面分析,以促进治疗决策。
方法
我们开发了一种具有平衡子采样的随机森林,基于少量血液样本中的无细胞DNA (cfDNA)测序数据筛查致病菌并诊断败血症。此外,我们基于一组正常和败血症样本构建了细菌共生网络,以推断未观察到的细菌。
结果
仅基于三个独立的脓毒症数据集的cfDNA测序信息,我们以令人满意的表现将脓毒症与健康样本区分开来。该策略还提供了全面的细菌分析,允许医生为败血症病例选择最佳的治疗策略。
结论
败血症鉴定和细菌推断策略的结合是基于cfdna的无创诊断的成功,这有可能极大地提高疾病检测的效率,并提供对病原体的全面了解。相比之下,基于培养的病原体分析需要长达5天,仅对三分之一到一半的患者有效,而cfDNA测序可以在1天内完成,我们的方法可以识别所有患者中的大多数病原体。
背景
脓毒症是由皮肤、肺部、腹部和尿道的各种感染引起的一种危及生命的紧急情况,是医院的一大挑战,并导致重症监护医学中高得令人无法接受的死亡率[1,2].近几十年来,人们对败血症的研究付出了巨大的努力,并开发了针对致病菌的新疗法。为了保证有效的治疗策略,快速准确地检测导致败血症的细菌或其他病原体至关重要。根据最近的一项指南,尽早(最好在1小时内)采用适当的抗生素治疗对脓毒症患者至关重要[3.].例如,在伴有低血压的感染性休克患者中,有效抗生素治疗每延迟1小时,死亡风险就会增加7.6% [4].然而,脓毒症患者病原体检测的标准程序是以培养为基础的(例如,在确认试验后进行血液培养)。由于这种方法依赖于细菌生长,因此需要相当长的时间(最多5天)[3.,5].此外,它有时无法识别脓毒症患者的特定病原体。只有三分之一到一半的败血症患者在血液培养中产生阳性结果[6].因此,迫切需要一种更快速的方法来诊断败血症样本和全面的细菌谱。
无细胞DNA (cfDNA)是指在几乎所有体液(包括血浆和血清)中都能检测到的自由循环DNA的小片段。这些DNA片段大多是人类的,在细胞凋亡过程中脱落到血液中[7]和细胞坏死[8].然而,cfDNA也包括来自其他生命形式的片段,如细菌、病毒、真菌[9,10,11],甚至透过食物摄取植物[12].随着新一代测序(NGS)技术的发展,cfDNA是一种有前途的、非侵入性的工具,可用于几种人类疾病的早期检测。它已被用于寻找癌症的预测性生物标志物[8,13,14,15],作为损伤的诊断工具[16]以及作为一种实时监测器官移植排斥反应的方法[10].最近,血液中高浓度的cfDNA被观察到是越来越多传染病的副作用[17,18].血浆中cfDNA的这些和其他用途代表了生物医学中一个快速发展的领域。
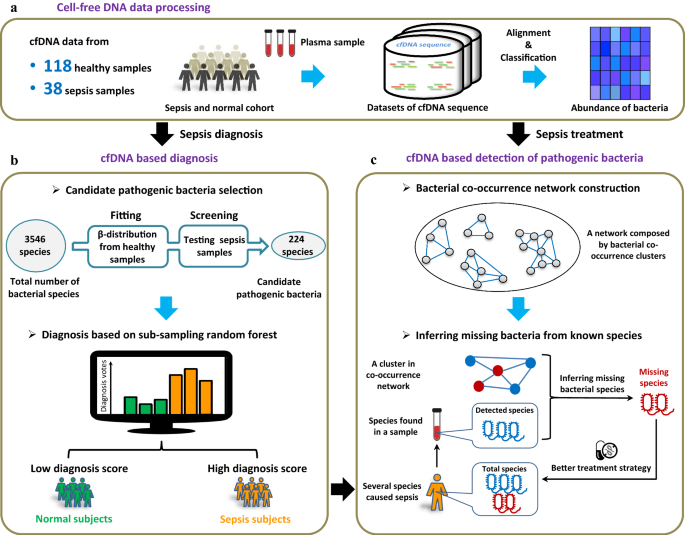
在这项研究中,我们实现了两个目标:(1)我们开发了一种基于cfdna的策略,可以快速诊断败血症患者并准确地描绘出负责的细菌;(2)构建了脓毒症特异性细菌共现网络,从cfDNA测序数据推断未观察到的细菌种类。为了实现第一个目标,cfDNA从血液样本中分离出来并进行测序(图2)。1A)健康和败血症队列。在此基础上,通过统计模型对候选致病菌进行鉴定和排序。我们的快速脓毒症诊断方法实现了93%的ROC曲线下面积(AUC)。我们的第二个目标是识别缺失的细菌,这具有实际意义,因为由于血液样本的体积有限,并不是所有引起感染的细菌都可以在cfDNA中检测到。不完整的细菌图谱可能对治疗决策产生偏差。我们通过模拟实验验证了该方法的有效性和鲁棒性。特别是,当一些细菌物种从模拟样本中随机去除时,我们的方法可以以很高的速度回忆起这些物种。事实上,即使样本中80%的细菌被随机去除,所有存在的细菌的回收率仍然是60%。因此,这种方法可以全面了解引起脓毒症和感染相关的细菌种类,极大地促进脓毒症治疗的治疗决策。
材料与方法
数据收集和处理
本研究使用的cfDNA测序数据来自38个败血症和118个健康样本。原始测序读数来自三个先前发表的数据源:38个败血症和15个健康样本,来自欧洲核苷酸档案(ENA,研究1,No. 1)。PRJEB13247 [19]), 103个健康样本来自欧洲基因组-表型档案(EGA,研究2,No. 2)。EGAS00001001754 [20.]), 165个无症状样本和187个有症状的样本来自欧洲核苷酸档案(ENA,研究3,No. 3)。PRJNA507824) [21].从血浆中提取上述研究的样本,然后进行全基因组和单端测序。来自ENA(PRJEB13247)和ENA(PRJNA507824)的原始读取被清除了类似人类的读取和低复杂度延伸的读取。对于EGA数据,使用快速比对程序Bowtie2对原始测序reads进行预处理,以去除人类和类人类的reads [22].
读取比对和量化
使用离心机将非人类测序读数与微生物基因组序列数据库进行比对[23],这是一个开源微生物分类引擎,能够快速准确地标记读数和量化物种。具体地说,该图谱是基于由Centrifuge (https://ccb.jhu.edu/software/centrifuge/manual.shtml).
沿着分类学树向上遍历,离心机映射到分类单元节点,并为每个分类类别分配“物种丰度”。丰度是估计的分数\ \(α= \离开({\ alpha_ {1}, \ alpha_ {2}, \ ldots \ alpha_{年代}}\)\)最大化似然函数;也就是说,
有了可能性\ (L \)给出的
R是读取的次数,年代是物种的数量,\ (\ alpha_ {j} \)是物种的丰富吗j(\(总和\ mathop \ \ nolimits_ {j = 1} ^{年代}\ alpha_ {j} = 1, 0 < \ alpha_ {j} < 1 \)),\ (l_ {j} \)物种基因组的平均长度是多少j.的系数\ (C_ {ij} \)如果读取,则为1我按物种分类j,否则为0。丰度向量α是通过期望最大化(EM)程序获得的。
通过该程序,从败血症和健康样本中获得两种细菌丰度矩阵。对于每个矩阵,一行代表一个细菌种类,一列代表一个样本。
候选致病菌鉴定
为了检测cfDNA样本中异常的细菌丰度,我们需要首先建立健康条件下细菌丰度的背景分布。我们用Beta分布拟合健康样本中每个物种的预期丰度。(这是定义在区间[0,1]上的连续概率分布族,并由两个正参数参数化。)具体来说,针对每种细菌j,其在健康样本训练集上观察到的丰度值用于拟合由参数定义的物种特异性Beta分布一个j而且bj.
以确定细菌种类j是否为候选病原体,我们比较丰度值αj从一个新的样本(健康或败血症)到Beta分布。具体来说,我们计算概率P观察比…高得多αj假设样本是健康的:
如果P是非常小的,那么我们可以拒绝该样本中观察到的该细菌种类的丰度是由健康条件下确定的Beta分布产生的假设,因此得出该物种的丰度异常高,是败血症的候选病原体。在我们的研究中,如果一个细菌物种至少满足一个败血症样本的这一条件,那么它就被归类为候选病原体。
随机森林平衡子抽样
随机森林是一种有效的分类方法,可以生成许多二叉决策树[24并汇总他们的结果。每个决策树都在原始训练数据的自举子样本上进行训练,并搜索决策阈值,将样本有效地划分为随机选择的输入特征子集中的类别(在我们的例子中,所有细菌物种都是病原体候选)。随机森林的最终决策是通过将每棵树的决策以多数票的方式聚合而成。随机森林及其扩展在近年来的疾病诊断研究中得到了广泛的应用。随机森林变量Ada用于癌症类型的cfDNA鉴别[25].基于稀疏回归的随机森林被设计用于预测阿尔茨海默病[26].
由于健康和败血症样本的大小不平衡,传统的随机森林可能会产生有偏差的预测。因此,我们采用重复平衡子抽样的方法来构建我们的脓毒症诊断模型。该技术将训练数据分成多个随机子样本,同时确保每个子样本中的类大小相等。在我们的案例中,我们生成了大小为30的子样本,其中15个来自健康患者,15个来自败血症患者。对于子样本组的训练集,拟合决策树。我们利用平衡子抽样策略构建了一个由500棵二叉决策树组成的森林,从而从聚合决策中生成无偏诊断模型。
共现网络推理
人类血液中的细菌DNA片段可能来自许多物种[27].这些细菌自然存在于整个人体,从皮肤到内脏,甚至在以前认为无菌的环境中,如循环中的血液中[28].了解来自不同栖息地的不同物种的DNA片段是如何聚集在一起的是非常重要的。数据中强烈的分类间关联可能表明一个群落(甚至包括不同的生命领域,如细菌和古生菌)起源于一个共同的生态位空间,或者群落成员之间可能存在直接的共生关系。这些信息在许多微生物类群的基本生态学和生活史策略仍然未知的环境中特别有价值。此外,探索不同微生物之间的共生模式可以帮助确定潜在的生物相互作用、栖息地亲和性或共同的生理学,从而指导更集中的研究或实验设置[29].特别是,我们能从血液样本中出现的其他种类的细菌中推断出某种细菌的存在吗?
共现网络是通常一起出现的实体之间关系的可视化。例如,它可以用来研究生物种群的分布[30.],以预测癌症风险[31]或分析文本集合[32].我们构建了一个基于cfDNA的细菌共生网络,其中两个物种被认为是共生的,如果它们的丰度由cfDNA估计是强相关的。网络中的每个节点代表一种细菌,而每条边代表一种共生关系。
为了构建细菌共生网络,我们首先生成两个矩阵:(1)观察到的丰度矩阵O(与n物种,米样本);(2)期望丰度矩阵N(也有n物种,米样本)。根据区域物种分布模型的预测,后者被填充在每个局部样本中,该模型被称为留白黄土模型[29].一个\(n \乘n\)协方差矩阵Σ由任意一个计算O或N通过比较行(即所有样本中2个物种的丰度)。从这个协方差矩阵的逆,偏相关\ (C_ {ij} \)对细菌种间的计算如下:
在哪里米是一个\(n \乘以m\)输入矩阵(O或N).
这两个C(O),C(N)根据式(4).然后是标准效应之间的相关关系O而且N是通过缩放计算的吗C(O)C(N).最后,通过计算p每对物种的相关系数值我而且j零假设,即观测值不相关。最后,我们的共现网络通过在每对细菌物种之间放置具有显著链接的边来生成。网络构建的详细算法描述在[33].
结果
基于cfDNA的脓毒症快速诊断新策略
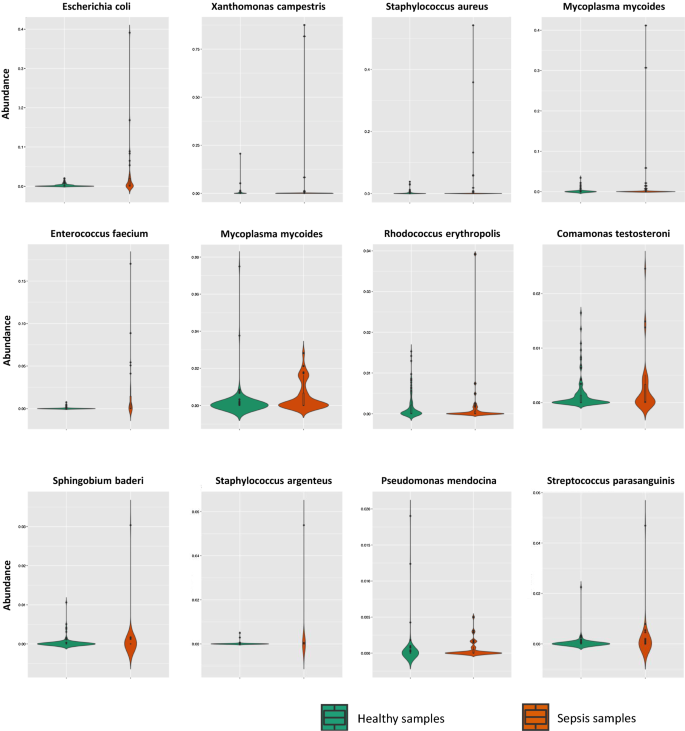
按照图中所示的程序。1A, b,我们开发了一种快速诊断脓毒症的两步方法,该方法已通过交叉验证和独立数据集验证。对于交叉验证,首先,我们通过对118个健康和38个败血症样本的cfDNA测序读数进行比对和分类,鉴定出3546种细菌。对应的列表P值的T-test,用于测量研究1 (No. 1)中败血症与健康样本之间的差异。PRJEB13247)和研究2 (No。EGAS00001001754),作为附加文件提供1:表S1。所有样本随机分为两组:2/3(78个健康样本和25个脓毒症样本)用于训练,1/3(40个健康样本和13个脓毒症样本)用于测试。对于每个物种,我们根据其细菌丰度载体与来自健康训练样本的78个元素拟合Beta分布。然后逐个测试来自脓毒症训练样本的25个丰度与Beta分布,以生成25P值。在这里,如果一个物种至少有一个满足条件,就可以认为是一个候选病原体P-value < 0.01。通过这样的筛选程序,筛选出约220个候选致病菌。数字2显示了这些候选病原体的一些例子,它们在健康和败血症样本的细菌丰度之间具有显著不同的分布。
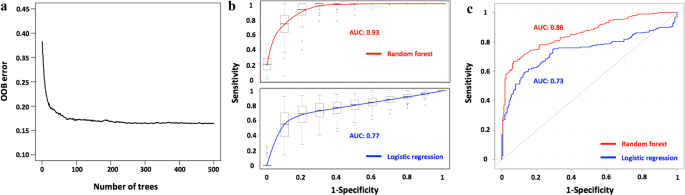
其次,仅根据观察到的候选致病菌丰度,我们使用平衡子采样训练随机森林以生成准确的分类器。最后,我们使用该分类器测试为此目的保留的其他三分之一的正常和败血症样本。通过引导,上述管道重复了1000次。如图所示。3.a,当决策树数量足够大(> 100)时,平均出袋误差(OOB误差)为0.16。诊断策略的表现令人满意,平均AUC为0.926,敏感性为0.91,特异性为0.83。作为一种替代方法,我们也尝试了逻辑回归方法作为比较(平均AUC为0.77,敏感性为0.71,特异性为0.80)(图。3.b).候选菌种在随机森林模型中按重要性排序的列表见附加文件2:表S2。
为了验证独立数据集的有效性,研究1 (No. 1)的118个健康和38个脓毒症样本分别来自研究1。PRJEB13247)和研究2 (No。以EGAS00001001754)为训练集;PRJNA507824)设置为独立验证。AUC表明,该方法在独立数据集上也表现良好(图2)。3.c)。
基于cfDNA的细菌共生网络
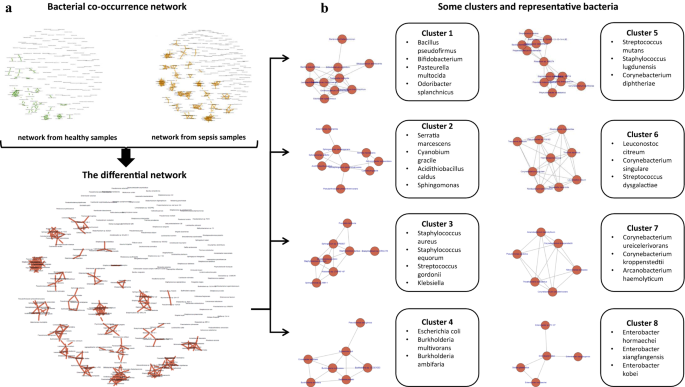
使用来自78个健康和25个败血症样本的细菌丰度矩阵进行训练,我们构建了两个细菌共存网络(图2)。4a).每个网络包含224个节点,代表在健康和败血症样本中具有显著不同丰度分布的224个候选致病菌。如上所述,血液中可能含有cfDNA片段,这些片段由居住在人体所有部位的细菌释放。因此,我们期望健康和败血症样本的共现网络包括通常不涉及败血症的“无害”物种之间的一些关联。为了专注于脓毒症特异性的关联,我们从脓毒症共现网络中排除了在健康共现网络中发现的所有关联模式,从而生成了一个差异网络。4a).我们发现了19个簇(图。4b)微分网络中的物种,即图中可见的强连通分量。4a.在25个败血症样本中,聚类中的所有物种在丰度水平方面均强相关。详细的集群信息在附加文件中提供3.:表S3。
为了分析群落的生物学特征,我们从呼吸模式、代谢栖息地和生长速率三个方面对每个群落的物种进行了描述。
首先,在所有候选病原体中,35.52%、3.66%和52.12%分别为厌氧、好氧和兼性(其余8.7%未知)。大多数簇在呼吸模式方面表现出相似性:9簇表现出对兼性物种的偏好(簇3、5、6、10、14、15、16、17和19),7簇表现出对厌氧物种的偏好(簇1、2、7、11、12、13和18)。样本中的少数厌氧物种不占任何集群的主导地位。
其次,在引起血液感染之前,这些细菌通常起源于特定的代谢环境。细菌代谢生境分为4种类型:宿主相关、陆生、水生和多样化。类群3、4、5、9、14、15、17、18、19主要为寄主伴生物种,类群10主要为陆生物种,类群3主要为水生物种,类群1、6、7、10、12、13、16包含不同代谢环境的物种。
第三,细菌生长与代谢变异和同居水平显著相关。双倍时间的数据导致了一项重要的发现,即参与翻译和转录的基因表达水平的变化会影响生长速率[34,35].我们根据其成员物种的加倍时间将簇分为两组:“快速”和“缓慢”生长的簇是那些中位数复制时间比所有物种的平均值短或长至少一个标准偏差[36].聚类6、7、11和13中的物种翻倍时间中位数大于1(快速生长簇),而聚类1、3、4、5、15和16中的物种翻倍时间中位数小于0.6(缓慢生长簇)。请注意,快速生长速度是表现出生态多样性的物种的典型特征,因此“快速”集群的识别符合前一段中分析的代谢栖息地。
针对每个集群的病原体,可提供特定的抗生素治疗方法[37].附加文件中显示了可能用于每个集群的抗生素列表3.:表S3。
从已确定的物种中推断缺失的细菌
脓毒症患者可携带多种病原体[38].因此,如果我们要提供快速有效的抗生素治疗,了解所有存在的细菌是至关重要的。同时,致病种的生长策略和环境要求(如好氧或厌氧、酸性等)范围广泛,这使得在单一培养物中很难检测到所有种。此外,由于血液样本的体积有限,并不是所有的致病物种都能从cfDNA中鉴定出来。简而言之,未观察到的细菌种类是有效治疗的主要障碍。
基于细菌共现网络,可以从已识别的细菌种类中推断出缺失的细菌种类。具体来说,通过构建细菌共现网络,我们知道某些物种通常在败血症样本中具有一致的丰度水平。因此,当在败血症样本中识别出群集中的某些物种时,从统计学上讲,群集的所有成员都很可能存在。如果缺失的细菌属于一个集群,我们可以通过这种方式推断“缺失”细菌的存在。
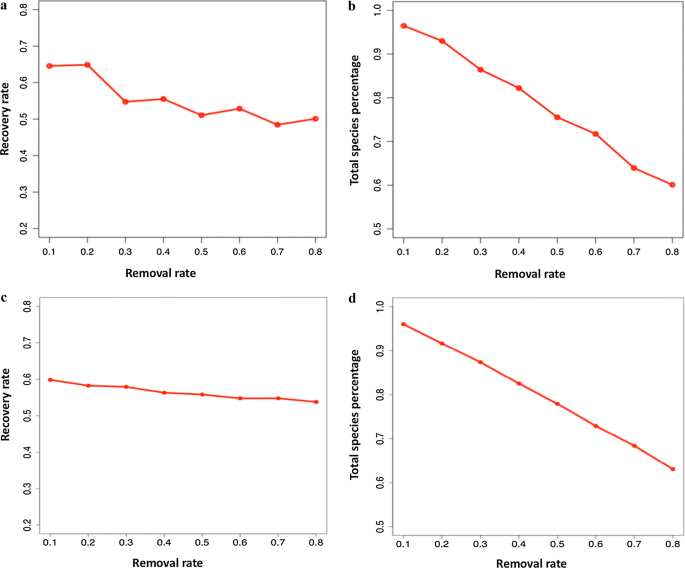
为了测试这种细菌推断方案的有效性和稳健性,每个样本从已识别的物种池中随机提取一定比例的物种,用于交叉验证和独立数据集的验证。我们试图根据细菌共现网络,从剩下的物种中推断出缺失细菌的存在。数字5A, c表明回收率在50-60%左右,随着去除率的提高,回收率逐渐降低。总体效果比较满意,如图所示。5b, d.即使80%的观察到的物种被随机移除,恢复的物种总数(包括那些没有被随机移除的物种)仍然是60%。这些结果证明了细菌共现网络的有效性,以推断来自已识别物种的未观察到的细菌的存在。这种方法有很大的潜力,特别是在基于cfDNA的分析中,因为在10毫升的血液样本中,cfDNA的数量非常有限,其中只有一小部分是微生物的cfDNA。
讨论
败血症或菌血症是一种常见的严重疾病,需要快速准确地诊断和鉴定病原体,以便选择适当的抗生素治疗。标准程序包括确认试验(例如,识别临床体征和症状,降钙素原试验,败血症细胞试验)和基于培养的病原体鉴定。根据最近的研究报道,基于培养的诊断是耗时的,并且需要严格的厌氧条件来促进细菌生长。此外,只有三分之一到一半的败血症患者血培养呈阳性[6].在这项工作中,我们开发了一种非侵入性的方法来诊断败血症和病原体鉴定使用cfDNA测序数据映射到细菌基因组。这种方法不需要培养,大大提高了诊断效率。我们的方法达到了93%(交叉验证)和88%(独立验证)的AUC,这远远优于血液培养方法。用我们的方法推断出的细菌与血培养的细菌之间的比较在附加文件中得到了证明4:表S4。血培养检测出的致病菌与本方法检测出的致病菌的符合率为84.69%。
我们的方法估计周转时间约为一天,这是目前cfDNA测序所需的时间。由于技术的进步和更快的测序速度,这个时间在未来将进一步缩短。因此,我们的方法可以提供准确和快速的鉴定脓毒症样本。
此外,差异细菌共生网络支持一个推理方案,以寻找“失踪”的细菌基于观察和识别的物种。这种方法可以对参与感染过程的所有细菌进行全面分析。它特别适用于只有少量血液样本(例如10毫升),而且许多细菌无法观察到的情况。这种快速脓毒症诊断和病原体推断的组合特别适合基于cfdna的诊断,现在已被认为是一种有前途的、无创的疾病检测工具。
结论
在这项工作中,我们从有限的败血症样本中鉴定出了引起败血症的细菌。随着越来越多的全基因组深度测序数据从健康和败血症队列中获得,可以识别更多的脓毒症引起物种,并且可以生成更准确的共现网络。因此,我们期望这种方法在不久的将来能够达到更高的精度。此外,我们期望从患者身上采集血液样本的时间序列可以进一步提高脓毒症的预后和诊断。这项研究仅仅是使用cfDNA诊断败血症的第一步,因为它展示了一种利用cfDNA测序数据和网络方法实现快速疾病诊断的新方法。
数据和材料的可用性
原始测序读数来自两个已发表的数据源:European Nucleotide Archive (https://www.ebi.ac.uk/ena/data/view/PRJEB13247,https://www.ncbi.nlm.nih.gov/Traces/study/?acc=SRP172792)及欧洲基因组表型档案(https://www.ebi.ac.uk/ega/studies/EGAS00001001754).
参考文献
Mayr FB, Yende S, Angus DC。严重脓毒症的流行病学。毒性。2014;5(1):4。
Walkey AJ, Wiener RS, Lindenauer PK.感染性休克中中心静脉导管的使用模式和结果:一项基于人群的研究。重症监护医学。2013;41(6):1450-7。
Dellinger RP, Levy MM, Rhodes A, Annane D, Gerlach H, Opal SM, Sevransky JE, Sprung CL, Douglas IS, Jaeschke R,等。幸存的败血症运动:严重败血症和败血症休克的国际管理指南,2012。重症监护医学。2013;41(2):580-637。
何王,倪夏皮罗,Angus DC, Yealy DM.美国急诊科严重败血症的国家评估。危重护理医学。2007;35:1928-36。
Vincent JL, Brealey D, Libert N, Abidi NE, O 'Dwyer M, Zacharowski K, Mikaszewska-Sokolewicz M, Schrenzel J, Simon F, Wilks M,等。危重患者感染的快速诊断,血流感染、肺炎和无菌部位感染分子检测的多中心研究。重症监护医学。2015;43(11):2283-91。
脓毒症检测:脓毒症联盟;2018.https://www.sepsis.org/sepsis/testing-for-sepsis/.2018年9月13日访问。
李志强,李志强,李志强,等。通过血浆DNA全基因组测序推断表达基因。植物学报。2016;48(10):1273。
萧晓波,胡恩,潘涛。无细胞核酸作为癌症患者的生物标志物。中华癌症杂志,2011;11(6):426。
龙勇,张勇,龚勇,等。新一代无细胞DNA测序技术在ICU患者败血症诊断中的应用中国医学杂志,2016;47(5):365-71。
De Vlaminck I等人。循环无细胞DNA使心脏移植排斥反应的无创诊断成为可能。科学翻译,2014;6:241ra77。
De Vlaminck I等人。肺移植术后感染和排斥反应的无创监测。美国国家科学研究院。2015; 112:13336-41。
Spisák S, Solymosi N, Ittzés P,等。完整的基因可以从食物传递到人体血液。PLoS ONE。2013; 8 (7): e69805。
康硕,李强,陈强,等。CancerLocator:使用无细胞DNA甲基化谱进行非侵入性癌症诊断和组织起源预测。中国生物医学工程学报。2017;18(1):53。
李伟,李强,康松,等。CancerDetector:使用无细胞DNA甲基化测序数据,在个人读取分辨率下进行超灵敏和非侵入性癌症检测。中国生物医学工程学报,2018;46(15):e89。
蒋平、陈长泉、陈嘉嘉、郑绍生、黄俊、黄文文、黄国良、陈绍林、莫志涛、陈海林、黎宝。肝细胞癌患者血浆DNA的延长和缩短。中国生物医学工程学报,2015;29(4):344 - 344。
王志强,王志强,等。中性粒细胞来源的循环游离DNA (cf-DNA/NETs)是严重烧伤患者死亡的潜在预后标志物。中华急诊外科杂志,2010;36(6):551-7。
Eshoo MW, Crowder CD, Li H, Matthews HE,孟S, Sefers SE, Sampath R, Stratton CW, Blyn LB, Ecker DJ,等。应用PCR和电喷雾电离质谱法检测和鉴定血液中的埃利希菌。临床微生物学杂志,2010;48(2):472-8。
Kaleta EJ, Clark AE, Cherkaoui A, Wysocki VH, Ingram EL, Schrenzel J, Wolk DM. pcr -电喷雾电离/质谱(MS)与MALDI-TOF/MS在阳性血培养瓶中细菌和酵母鉴定中的比较分析。临床化学杂志,2011;57(7):1057-67。
格鲁兹S,史蒂文斯P,格鲁兹C,等。脓毒症患者菌血症的新一代测序诊断。基因组医学,2016;8(1):1 - 13。
张志刚,张志刚,张志刚。MYC基因扩增与TP53基因突变在人类癌症中的共同发生。中国科学,2016;48(2):104。
Blauwkamp TA, Thair S, Rosen MJ, Blair L, Lindner MS, Vilfan ID, Kawli T, christian FC, Venkatasubrahmanyam S, Wall GD,张a .传染病无微生物细胞DNA测序检测的分析和临床验证。中国微生物学杂志,2019;4(4):663。
朗米德B,萨尔茨堡SL。快速间隙读取对齐领结2。光子学报。2012;9(4):357-9。
金D,宋林,Breitwieser FP,等。离心机:快速、灵敏的宏基因组序列分类。基因组学报,2016;26(12):1721-9。
李志强,李志强,李志强,等。随机森林:用于复合分类和QSAR建模的分类和回归工具。化学与计算科学,2003;43(6):1947。
邹杰,王娥。eTumorType,一种利用血液中循环肿瘤细胞或无细胞dna鉴别癌症类型的算法。中国生物工程学报。2017;15(2):130-40。
黄玲,金勇,高勇,唐克汉,沈东,阿尔茨海默病神经影像学研究中心。基于随机森林的软分裂稀疏回归预测阿尔茨海默病的纵向临床评分神经生物学老化。2016;46:180-91。
张晓东,张晓东,张晓东,等。通过循环的无细胞DNA,可以发现在人类中定居的许多未鉴定的和高度分化的微生物。美国国家科学研究院。2017; 114(36): 9623。
Proal AD, Albert PJ, Marshall TG。炎症疾病和人体微生物群《医学杂志》2014;17(95):257。
Barberán A, Bates ST, Casamayor EO,等。利用网络分析探讨土壤微生物群落共生模式。中华医学杂志。2012;6(2):343。
王志刚,王志刚,等。河流网络组织印迹微生物共生网络。美国国家科学研究院。2014; 111(35): 12799。
邹杰,王娥。eTumorRisk,一种基于个体种系基因组共突变基因网络预测癌症风险的算法。bioRxiv。2018.https://doi.org/10.1101/393090.
Gegov E, Gegov A, Gobet F,等。基于复杂网络的语言习得认知模型[M]//。Hauppauge: Nova Science出版社;2012.
Morueta-Holme N, Blonder B, Sandel B,等。从共现数据推断物种关联的网络方法。描述生态学。2016;39(12):1139 - 50。
磐石EPC。从tRNA的角度来看密码子使用偏差:冗余、专门化和用于翻译优化的有效解码。基因组学报,2004;14(11):2279-86。
时装设计师E, Rocha EPC。复制相关的基因剂量效应塑造了快速生长的细菌的基因组,但仅适用于转录和翻译基因。中国生物医学工程学报。2010;39(5):1506-18。
傅立奇,李志强,李志强,等。代谢网络驱动的细菌生态策略分析。中国生物医学工程学报。2009;10(6):R61。
Gyssens I C, Bax H I, Schippers E F,等。成人脓毒症患者的抗菌治疗。2010。
子宫动脉栓塞辅助完全性前置胎盘终止妊娠后脓毒症1例报告。2018年国际医学杂志。https://doi.org/10.1177/0300060517723257.
确认
不适用。
资金
这项工作得到了加州大学洛杉矶分校为XJZ提供的启动一揽子计划的支持;国家自然科学基金项目(no . 11771152, 11901203, 11971176);广东省基础与应用基础研究基金(2019B151502062);美国国立卫生研究院资助项目(no . R01CA246329, U01CA237711);中国博士后科学基金资助项目(2019M662895);中央高校基本科研业务费专项资金(2019MS111)。
作者信息
作者及隶属关系
贡献
XJZ构思了该项目;XJZ和RL监督了该项目。PC和RL进行计算和分析。SL, JR, WL和FS提供了分析工具和数据来源。所有的作者都写了手稿。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:表S1。
所有具有p值的物种。
附加文件2:表S2。
随机森林对候选菌种的重要性。
附加文件3:表S3。
细菌共生网络的聚类信息。
附加文件4:表S4。
细菌共生网络和血液培养的细菌推断。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
陈P,李S,李W。et al。基于游离DNA的脓毒症快速诊断和综合细菌谱分析。翻译医学杂志18, 5(2020)。https://doi.org/10.1186/s12967-019-02186-x
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12967-019-02186-x
关键字
- 脓毒症
- 菌血症
- 快速诊断
- 无细胞DNA序列
- 细菌共生网络
- 细菌分析