摘要
背景
大约30%的类风湿关节炎(RA)患者使用雷公藤多苷(TG)片治疗未能实现临床改善,这意味着预测生物标志物和工具的重要性。在此,我们的目的是识别可能的生物标志物预测TG片治疗RA的疗效。
方法
使用Affymetrix EG1.0阵列检测从TG片处理的发现队列中获得的外周血单个核细胞的基因表达谱。结合差异表达数据分析和基因信号转导网络分析,筛选出TG片反应的候选基因生物标志物。在此之后,基于候选基因生物标志物在RA患者中的表达水平构建偏最小二乘(PLS)模型,并使用验证队列进行评估。
结果
六个候选基因生物标记(Mx1, oasl, spink1, crk, grapl而且RNF2)被认为是TG治疗的预测因子。在构建基于pls的外周血表达水平模型后,5倍交叉验证和独立数据集验证都显示了该模型的高预测效率,并表明基于6个候选基因生物标志物表达的pls模型在预测RA患者对TG片的反应方面比常用的临床和炎症参数以及单独的基因生物标志物有显著改善。
结论
这项产生假设的研究确定了Mx1, oasl, spink1, crk, grapl而且RNF2作为这是RA治疗干预的新靶点,基于这些候选生物标志物表达水平的PLS模型可能在TG片治疗的RA患者中具有潜在的预后价值。
背景
雷公藤虎钩F (TwHF)是一种传统中药,在中国用于治疗类风湿关节炎(RA)已有数百年的历史[1].黄芪被认为是开发治疗类风湿性关节炎新药的潜在来源,因为它大大改善了类风湿性关节炎患者的前景[2].雷公藤多苷(TG)片是雷公藤多苷的主要有效成分,是雷公藤多苷治疗中最常用的一种,根据近期临床观察,雷公藤多苷片的治疗效果优于几种一线减病抗风湿药物[3.].然而,大约30%的使用TG片治疗的RA患者未能实现临床改善,表现出由表观遗传、生理、环境,特别是遗传因素联合或单独引起的显著个体间差异[4,5].RA患者在病理表现、疾病进展和治疗反应上的异质性,提示在分子水平上存在多种疾病亚型[6].研究人员正试图找出风湿疾病治疗反应的各种生物标志物,以进一步促进个体化治疗和疗效,包括microRNAs、基因、蛋白质等。[7].然而,现有的反映骨和软骨转换的生物标志物不足以替代预测某些药物的治疗结果[8].因此,识别和开发预测RA患者对TG片反应的生物标志物和工具具有重要的临床意义。
近年来,高容量基因组学和转录组学技术,如同时和全面检测众多基因表达谱的基因微阵列,在识别疾病行为的生物标志物和治疗反应预测方面发挥着重要作用[9,10].虽然基因表达芯片具有高灵敏度和高通量的特点,但尚不足以全面阐明RA的整个生物调控过程。此外,样本大小和质量的变化可能经常导致微阵列识别的众多差异表达基因之间的不一致性[11].为了解决这些问题,我们设计了一项结合基因表达谱的高通量优点和分子网络分析药物-疾病相互作用的综合说明的假设生成研究,评估对TG片有反应性和无反应性RA患者之间的差异表达基因,并根据差异表达模式和网络拓扑特征识别候选基因生物标志物。并根据候选基因生物标志物在外周血中的表达水平构建偏最小二乘(PLS)模型,用于RA患者对TG片的分层和预测(图2)。1).
方法
道德声明
本研究根据《赫尔辛基宣言》的人类指南进行,并得到了广安门医院研究伦理委员会的批准。所有患者均获得知情同意。
病人
两组RA患者队列分别为发现队列(n = 12,6名有反应者和6名无反应者,收集于2015年1月至2015年12月,广安门医院风湿病科)和验证队列(n = 31,15名有反应者和16名无反应者,收集于2016年1月至2017年6月,广安门医院风湿病科)。该发现队列用于检测外周血单个核细胞(pmcs)的基因表达谱,并训练PLS模型预测TG片的反应。验证队列通过qPCR法验证TG片反应候选基因生物标志物的表达水平,并在独立数据集测试中评估基于pls模型的预测效率。两个队列的纳入标准包括:(1)根据美国风湿病学会(ACR) 1987年RA标准或2010年ACR/欧洲风湿病联盟(EULAR)标准诊断为活动性RA [12];(2)症状持续时间小于1年;(3)以前没有使用dmard;(4) TG片(购自浙江德德股份有限公司,Z33020422,浙江新昌)开始服用12周后临床和实验室参数的可获得性,以及外周血样本的可获得性。患者口服TG片(20mg.tid.po) 12周。对TG片有反应者定义为服用TG片12周后ACR达到20的患者,无反应者定义为服用TG片12周后未达到ACR 20的患者[13].两组间临床和实验室参数无显著差异(表2)1和附加文件1).
基因表达谱
在TG片治疗前,在EDTA血浆管中采集RA患者外周血标本(1.5 mL)。采用标准Ficoll密度梯度离心分离外周血单个核细胞(pmcs),并在无菌磷酸盐缓冲盐水(PBS)中洗涤两次。使用QIAGEN RNeasy Mini Kit从PBMC中分离细胞总RNA,按照制造商的方案(Cat.No.;217004, Qiagen, Hilden,德国)。使用RNase-Free DNase Set (Cat.No. 1)去除残留的DNA污染。79254, Qiagen, Hilden,德国)。使用NanoDrop分光光度计(NanoDrop technologies, Montchanin, DE, USA)测量总RNA浓度。总RNA用15 μl无rnase水洗脱,- 80℃保存。
采用上海GMINIX生物科技有限公司的Affymetrix EG1.0阵列分别检测TG片有反应者和无反应者外周血单核细胞的mRNA表达谱。GSE106893的mRNA表达微阵列数据由国家生物技术信息中心基因表达Omnibus (https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE106893).
差异表达mrna筛选
应答者组和无应答者组之间表达有显著差异的mrna通过RVM以|log2 fold change (FC)| > 0.5和P值< 0.05为标准进行鉴定t以及。采用R (version 1.0.2, R Core Team, Vienna, Austria)中的热图包进行分层聚类分析。
基因信号转导网络分析
根据从公共数据库STRING (Search Tool for Known and Predicted Protein-Protein interaction, version 10.0, Search Tool for Known and Predicted Protein-Protein interaction, version 10.0,http://string-db.org/) [14].选择高可靠的基因-基因相互作用数据,综合证据评分高于所有评分的中位数。在网络中,节点指响应组和非响应组之间表达差异的基因,边指节点之间的相互作用。
为了确定对TG片反应的候选基因生物标志物,我们通过计算以下四个拓扑特征来评估每个节点的拓扑重要性:(1)节点度:节点i与其他基因连接强度的总和,它衡量一个基因与网络中所有其他基因的相关性;(2)节点间性:网络中一个节点相对于其他节点的重要性;(3)节点紧密度:测量将信息从节点I依次传播到所有其他节点所需要的时间。节点度/间度/紧密度越大,说明该节点在网络中越重要。
途径富集分析
为了了解通过差异表达数据分析和网络计算筛选的候选生物标志物的功能,使用数据库可视化和集成发现软件(DAVID,http://david.abcc.ncifcrf.gov/home.jsp基于从京都基因和基因组百科全书数据库(KEGG,http://www.genome.jp/kegg/,于2016年11月18日更新)[15,16].仅选取Bonferroni和Benjamini算法校正了富集P值(P < 0.05)的功能注释进行进一步分析。
基于基因表达特征的pls模型构建及5重交叉验证模型性能评价
采用PLS算法,根据候选基因生物标志物在外周血中的表达水平,构建预测RA患者对TG片反应的模型。正如我们之前的描述[17],在PLS中构造分量的客观准则是依次最大化响应变量和预测因子的线性组合之间的协方差。让\({x} \)是\(n{\times}p \)矩阵的\(n \)例和\(p \)候选基因生物标志物。同时,让\(y \)表示\(n{\times}1 \)响应值的向量,如响应者或无响应者的指标。在样本协方差的基础上,构造了最大化客观准则的分量\(y \)而且\(x_{c} \).这样,我们就找到了权向量\(w \)满足以下客观标准。
然后,利用训练数据集计算PLS模型中不同基因生物标记物的权重系数。PLS模型中6个候选基因生物标志物分别为:
每个样本的PLS模型得分定义为:
在哪里\({L}_{{P}_{{i}}}} \)指候选基因生物标志物的表达水平\({p}_{{i}} \)在每个RA患者中。
然后,利用训练数据集输入PLS模型,计算阈值\(t \)(2)受试者工作特征(ROC)曲线下面积(\(auc \))是最大的。最后,PLS分类器决定:if\(Score\,\text{>}\,T \),该样品可预测为TG片的反应剂。
为了进行5倍交叉验证,将发现队列中6个候选基因生物标记物的表达水平分为训练数据集和测试数据集两部分。由于发现队列的样本量较小,因此进行了5次交叉验证。平均\(准确度\),\({灵敏度}\)而且\({专用性}\),以及平均曲线下面积(\(auc \))根据受试者工作特征(ROC)曲线计算,计算公式如下:
在哪里\(tp \),\(tn \),\(fp \),\(fn \)分别是指一次试验中真阳性、真阴性、假阳性和假阴性结果成分的个数,\(n \)表示预测样本总数。
定量PCR分析
为了使用独立数据集测试评估我们基于基因表达特征的pls模型的预测性能,我们使用从验证队列中获得的外周血样本对模型中的6个候选基因进行了定量PCR分析[18,19].GAPDH和RPS18作为候选基因表达归一化和量化的两个内控。使用附加文件中列出的引物对在ABI 7900HT qPCR体系上进行定量PCR分析和数据收集2.采用比较周期阈值法(CT)评价基因表达的相对定量。将原始定量分别归一化为每个样品的GAPDH和RPS18值,在每三个重复的三个独立实验中,fold变化以mean±SD表示。
用独立数据集测试评估模型性能
候选基因生物标志物的差异表达模式和我们基于基因表达特征的pls模型的性能都通过使用验证队列的独立数据集测试进行了验证,这些模型预测了TG片剂的反应。准确度和ROC曲线下面积(area under ROC curves, AUC)按公式4-6计算。
统计分析
使用SPSS软件进行统计分析(版本13.0,社会科学统计程序,公司:芝加哥,伊利诺伊州,美国)。通过单因素方差分析比较应答组和非应答组之间候选基因生物标志物的表达水平。P-值< 0.05为显著性。
结果
与TG片反应相关的差异表达基因
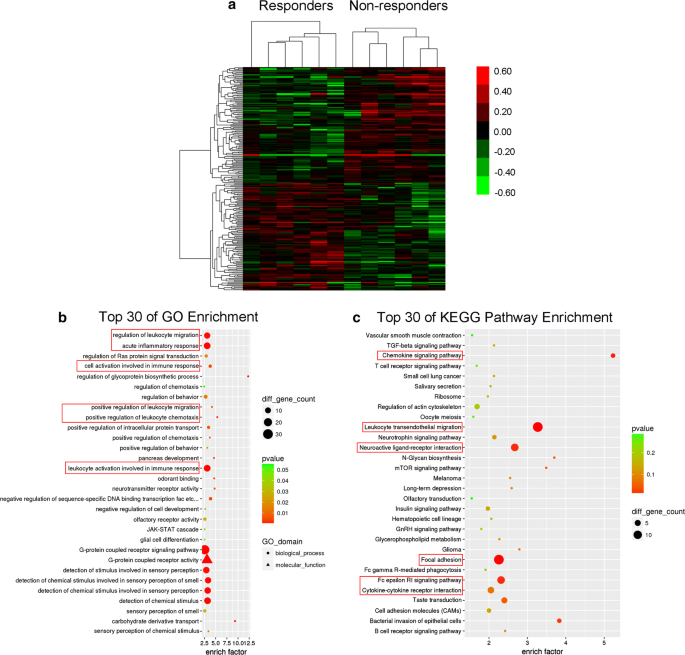
通过比较应答组和非应答组之间的基因表达谱来评估与TG片应答相关的差异表达基因。两组间共有212个基因(上调102个,下调110个)表达水平存在显著差异(所有折叠变化> 1.2,P < 0.05)。差异表达基因在附加文件中列出3..
此外,热图(图;2a)和上述差异表达基因谱的无监督层次聚类揭示了对TG片有反应者和无反应者的不同模式。途径富集分析显示,TG片反应相关的差异表达基因在炎症反应和免疫反应的生物学过程和途径中显著富集,包括急性炎症反应、参与免疫反应的白细胞活化、参与免疫反应的细胞活化、趋化因子信号通路、白细胞跨内皮迁移、正向调控白细胞迁移、白细胞趋化性、白细胞激活参与免疫应答、白细胞迁移、Fc epsilon RI信号通路及细胞因子-细胞因子受体相互作用的正向调控(均P < 0.05,图。2b, c).越来越多的证据表明,RA的发病机制可能与免疫调节缺陷和许多炎症机制有关[20.,21,22],因此,差异表达基因,如CRK,GHR,RNF2,RNF8,VAV2这些生物学过程可能会影响TG片在RA进展过程中控制炎症反应和调节免疫的治疗效果。此外,一些差异表达基因,如CRK,VAV2,IGF1,与局灶粘连显著相关(图;2c),已被证实可促进滑膜细胞的增殖、迁移和侵袭,促进RA病理改变的发生和发展[23].
基于发现队列,识别预测TG片剂反应的候选基因生物标志物
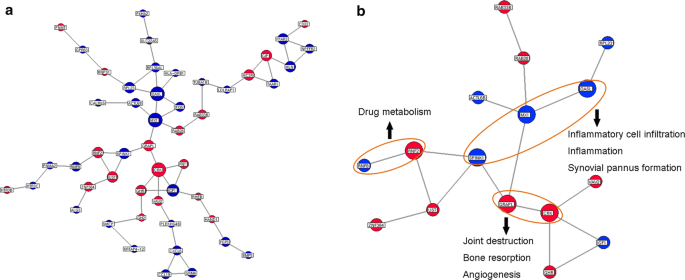
为鉴定预测TG片反应的候选基因生物标志物,从STRING数据库中获取212个差异表达基因的基因-基因互作数据,构建基因信号转导网络如图所示。3.a.通过对度值、亲近度值和中介中心值的计算,同时筛选出3个特征值高于相应中值的16个基因,这些基因是与TG片反应相关的基因-基因相互作用网络中具有较大拓扑重要性的主要基因(表2)2).此外,16个主要基因的子模块(图;3.B)是利用它们之间的直接相互作用构建的,这意味着这些主要基因之间在网络中具有紧密的联系。根据应答组与非应答组比较中16个主要基因的表达水平差异,mx₁,OASL,SPINK1,CRK,GRAPL而且RNF2表现出更明显的失调[\(Fold - change_{(responder/non responder)} \, > \, 1。5 \)而且P< 0.05]高于其他基因(表2).此外,根据GeneCards数据库(http://www.genecards.org/, Version 4.5.1), 6个候选基因生物标志物功能参与了RA进展过程中与炎症细胞浸润、炎症、滑膜泛膜形成、血管生成、关节破坏和骨吸收以及药物代谢等主要病理事件相关的几个信号通路(表3.和无花果。3.b).考虑到它们表达模式的显著差异、网络拓扑重要性以及与RA的功能相关性,我们选择了它们mx₁,OASL,SPINK1,CRK,GRAPL而且RNF2作为候选基因生物标志物及其在外周血中的表达水平,将用于构建基于pls的预测TG片反应的模型。
基于验证队列预测TG片剂反应的候选基因生物标志物的验证
在确定了六个最常见的基因(mx₁,OASL,SPINK1,CRK,GRAPL和RNF2)作为TG片反应的候选生物标志物,我们尝试使用31例患者的验证队列,通过定量PCR分析验证微阵列数据。的表达水平一致mx₁,OASL而且SPINK1根据两种内部对照,从有反应者获得的外周血中明显低于无反应者RPS18而且GAPDH(P均< 0.001,图;4a - c)。此外,表达水平CRK,GRAPL而且RNF2与无反应者相比,有反应者显著升高,这也与微阵列数据一致(均P < 0.001,图;4d, e)。
基于pls的模型有效预测TG片的反应
基于pls模型的六种基因生物标志物的表达水平(mx₁,OASL,SPINK1,CRK,GRAPL而且RNF2)。利用发现队列确定各基因生物标志物的权重值和模型的阈值。因此,的权重值mx₁,OASL,SPINK1,CRK,GRAPL而且RNF2分别为−0.4694、−0.2494、−0.5592、0.3429、0.4054、0.3504,阈值为−0.03。
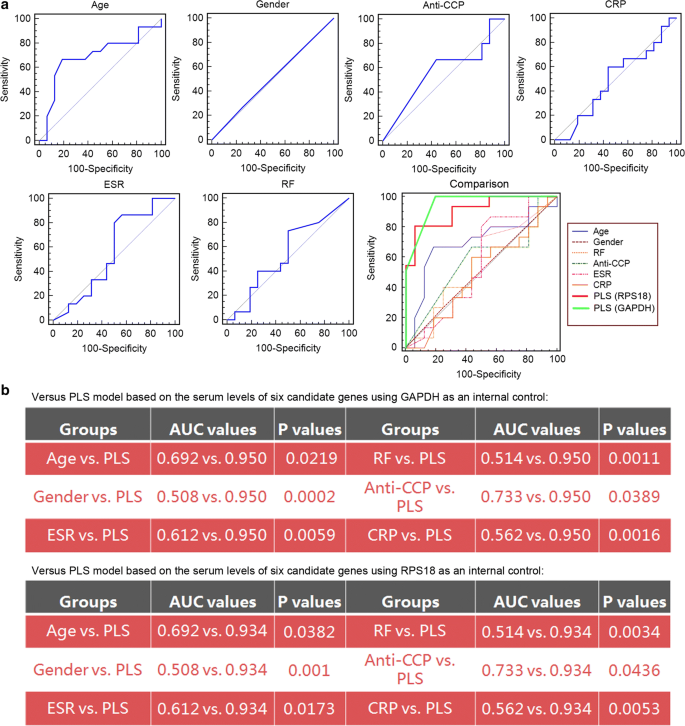
此外,5倍交叉验证证实,模型在5次试验中的准确率分别为83.33、100.00、83.33、83.33、100.00%,AUC值分别为1.000、0.889、1.000、1.0000、0.875。在独立试验验证中,的表达水平Mx1, oasl, spink1, crk, grapl而且RNF2在31个RA外周血样本(分别用GAPDH和RPS18归一化)中使用,以验证我们的模型的性能。结果表明,以GAPDH为内控的基于6种基因生物标志物表达水平的pls - s模型的准确性和AUC值分别为90.32%和0.950,与以RPS18为内控的基于6种基因生物标志物表达水平的pls - s模型的准确性和AUC值一致(准确度为87.10%,AUC值为0.934)。5倍交叉验证和独立试验验证均表明,利用不同的试验数据集筛选RA患者对TG片的反应者具有较高的可靠性和有效性。
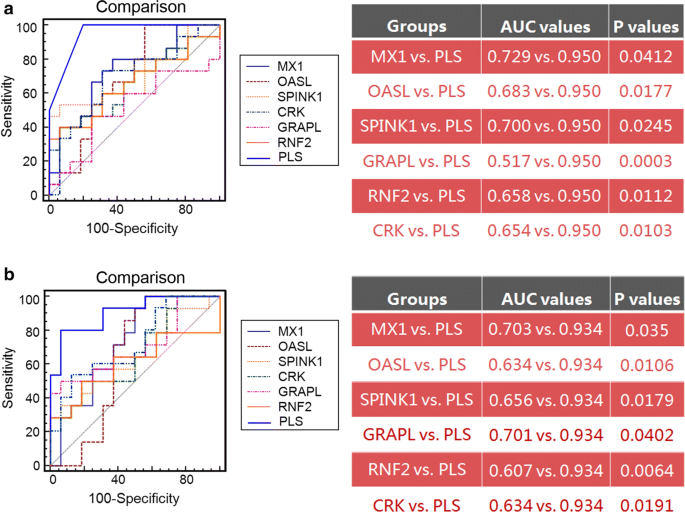
由于六个候选基因生物标记物在与TG片反应相关的基因信号转导网络中的拓扑重要性和相关性,我们结合它们的表达水平构建了基于pls的模型。为了验证该设计的合理性,我们首先基于验证队列,将基于pls的模型与6个候选基因生物标志物单独进行性能比较。如图所示。5,单基因生物标志物的表达水平和其平均水平在预测TG片反应方面均优于以GAPDH和RPS18为内对照,基于所有6个候选基因生物标志物表达水平构建的pls模型。
为了确定我们基于pls的模型在预测TG片反应优于各种常用的RA临床和炎症参数方面的优势,我们将其预测效果与患者的年龄、性别、红细胞沉降率(ESR)以及c反应蛋白(CRP)、类风湿因子(RF)和抗环桂氨酸肽(CCP)抗体水平进行了比较。ROC比较分析显示,基于6个候选基因生物标志物表达水平的PLS模型的性能明显优于临床和炎症参数(AUC值比较,均P < 0.05,图。6).
讨论
由于临床观察表明,并非所有RA患者都能从TG片的治疗中获得相同程度的受益,因此,药物反应生物标志物的识别和基于生物标志物的预测工具的开发可能对确定TG片反应概率低的患者具有重要意义,允许临床医生在疾病的早期阶段选择替代药物,而不会延迟有效的治疗。在目前的研究中,我们整合了与药物反应相关的基因表达谱和基因-基因相互作用,以确定6个预测TG片反应的候选基因生物标志物。然后,我们应用PLS算法,基于这些候选基因生物标志物的表达水平,构建治疗结果的预测模型。5倍交叉验证和独立数据集验证均表明,该模型具有较高的预测准确率(87.50-100.00%)和ROC曲线下面积(0.875-1.000)。更重要的是,我们的数据表明,在预测RA患者对TG片的反应时,基于六种候选基因生物标记物的表达水平的pls模型比常用的临床和炎症参数以及单独的基因生物标记物的表达水平有显著改善。据我们所知,这是第一个确定RA患者对TG片反应的基因生物标志物,并评估其在临床治疗结果预测模型中的应用的研究。
为了促进个体化医疗的实施和改善RA患者的治疗结果,最近的研究一直在试图识别各种治疗反应的预测因子,包括单核苷酸多态性、基因、蛋白质和microRNAs,分别基于基因组学、转录组学、蛋白质组学和miRNomics [2,24,25,26].然而,仅基于“组学”数据分析的研究可能无法完全解释RA进展过程中发生的所有变化。为了更深入地了解分子调节网络如何在治疗中发挥作用,可能会增加同时对多个“组学”维度建模的努力。在此背景下,我们将微阵列数据的差异表达分析与基因信号转导网络的拓扑特征进行了系统的整合,主要有两个优势:一是可以充分利用微阵列数据提供的基因共表达信息,这些信息被认为比单个基因的表达变化更有利于目标基因的识别。其次,网络分析是了解疾病发生和发展,以及治疗和药物反应的强大工具。通过整合生物网络的拓扑特征,可以将一些差异表达分析中丢失的信息添加到预测TG片反应的候选基因生物标志物的鉴定中。函数,如表所示3.这些候选基因生物标志物与RA的发病机制和药物代谢相关。特别是MX1和OASL都是干扰素(IFN-I)诱导基因,主要参与免疫系统的信号通路。最近的研究表明,RA中的IFN-I特征可能显示出与疾病发作和治疗反应相关的临床相关性[27,28].Sanayama等。[29]发现MX2(与MX1属于同一家族成员)和OASL作为预测RA患者对tocilizumab治疗反应的生物标志物。RNF2(环结构域包含E3泛素蛋白连接酶环指蛋白2)参与组蛋白H2A水平的维持并影响转录活性[30.].RING E3连接酶已被发现参与多种细胞过程的控制,并被认为是许多人类疾病的候选治疗靶点,包括RA [31].泛素/蛋白酶体蛋白降解途径被发现有助于延长RA组织中滑膜成纤维细胞的存活[32].有趣的是,托瑞等人。33]发现E3泛素连接酶可能正向调节I型干扰素反应并促进神经炎症的发病,这意味着RNF2与MX1和OASL之间存在几种可能的联系。据报道,Crk家族适配器在多种细胞外刺激下广泛表达并介导信号转导蛋白复合物的及时形成。久川等人。[34他们发现RA患者滑膜液T细胞中crk相关底物淋巴细胞型(Cas-L)的酪氨酸磷酸化显著增强。Miyake-Nishijima等。[35]在RA小鼠模型中发现了Cas-L蛋白的过表达及其酪氨酸磷酸化升高,并观察到大量Cas-L阳性淋巴细胞浸润到RA患者的炎性病变,提示了Cas-L在RA病理生理中的重要作用。上述文献报道支持了本研究中确定的候选基因生物标志物可能与RA的疾病进展和治疗结果相关的证据。
为了确定候选基因生物标志物的临床应用价值,我们利用六种候选基因生物标志物在RA患者中的表达水平,建立了基于pls的TG片治疗预测模型。PLS是一种通过潜在空间对两组多元数据之间的关系建模,并在该空间中执行最小二乘回归的方法。它将输入和输出变量映射到低维空间,从而使潜在空间中数据的协方差最大化。该算法能从大量特征中提取有效信息,有效区分两个数据集[36,37,38].值得注意的是,交叉验证和独立临床队列验证均表明,该模型可能能够预测TG片治疗RA患者的疗效,并通过比较常用临床和炎症因子、单个基因生物标志物和基因组合模型的表现,证实了模型构建的必要性。
结论
本假设研究确定MX1、OASL、SPINK1、CRK、GRAPL和RNF2为RA治疗干预的新靶点,基于其表达水平的PLS模型可能对TG片治疗的RA患者具有潜在的预后价值。我们的PLS模型可能不仅值得在前瞻性研究中进行评估,而且可能有利于RA在日常临床环境中的个体化治疗。但是,本研究使用的临床队列相对较少,用于预测模型的生成和验证,可能会导致一些模型过拟合。因此,这是一项产生假设的研究,未来需要基于大型临床队列的研究来验证六种基因生物标志物和PLS模型在预测和监测基于twf的治疗结果方面的效用。
缩写
- 雷公藤:
-
雷公藤钩F
- 类风湿性关节炎:
-
类风湿性关节炎
- DMARD:
-
治疗疾病的抗风湿药物
- PBMCs:
-
外周血单个核细胞
- TG:
-
雷公藤多苷片
- 请:
-
偏最小二乘回归
参考文献
Tao X, Lipsky PE。中草药抗炎和免疫抑制雷公藤华北大黄病临床杂志2000;26:29-50。
蒋敏,查强,张超,卢超,闫鑫,朱伟,刘伟,涂胜,侯磊,王超,张伟,梁强,范斌,余杰,张伟,刘鑫,杨健,何欣,李磊,牛欣,刘颖,郭辉,何斌,张刚,边忠,卢安雷公藤基于Hook f的类风湿关节炎治疗:从开放到双盲随机试验。科学通报2015;5:9700。
类风湿性关节炎和骨关节炎的补充和替代医学:系统综述综述。Curr Pain头痛代表2011;15:431-7。
吕庆文,张伟,史强,郑文军,李旭,陈辉,吴庆军,蒋伟力,李海波,龚磊,魏伟,刘宏,刘建军,金浩晖,王建新,刘小明,李志斌,刘斌,沈敏,王强,吴小楠,梁东,殷艳芳,费艳芳,苏建明,赵立德,蒋艳,李娟,唐福林,张春辉,Lipsky PE,张旭雷公藤钩f与甲氨蝶呤治疗活动性类风湿关节炎(TRIFRA):一项随机对照临床试验。安·莱姆,2015;74:1078-86。
王娟,崔敏,焦华,童燕,徐娟,赵颖,韩敏,刘娟。中医药疗效系统评价内容分析。中华传统医学杂志2013;33:156-63。
McInnes IB, Schett G.类风湿关节炎治疗的病理见解。《柳叶刀》杂志。2017;389:2328-37。
Burmester GR,教皇JE。类风湿性关节炎的新治疗策略。《柳叶刀》杂志。2017;389:2338-48。
Cooper C, Bardin T, Brandi ML, Cacoub P, Caminis J, Civitelli R, Cutolo M, Dere W, Devogelaer JP, Diez-Perez A, Einhorn TA, Emonts P, Ethgen O, Kanis JA, Kaufman JM, Kvien TK, Lems WF, McCloskey E, Miossec P, Reiter S, Ringe J, Rizzoli R, Saag K, Reginster JY.平衡糖皮质激素在风湿病和其他炎症性关节疾病中的益处和风险:来自新兴数据的新见解。来自欧洲骨质疏松症和骨关节炎临床和经济方面协会(ESCEO)的专家共识论文。Aging clinex res 2016; 28:1-16。
Márquez A, Martín J, Carmona FD。类风湿性关节炎诊断、预后和治疗反应的分子生物标志物的新兴方面。中国生物医学杂志,2016;16:663-75。
刘志刚,刘志刚,刘志刚。表达谱分析在类风湿关节炎治疗中的应用。Ann Rheum, 2013; 72:1118-24。
施neckener, Arden NS, Schuppert A.通过微阵列衍生临床生物标志物的基因列表排序的量化稳定性。BMC医学基因组学,2011;4:73。
杨森R,格林鲍姆D, Gerstein M.全基因组表达数据与蛋白质相互作用的关系。基因组Res. 2002; 12:37-46。
李超,李华。基因组数据分析的网络约束正则化和变量选择。生物信息学。2008;24:1175 - 82。
Szklarczyk D, Morris JH, Cook H, Kuhn M, Wyder S, Simonovic M, Santos A, Doncheva NT, Roth A, Bork P, Jensen LJ, von Mering C. 2017年的STRING数据库:质量控制的蛋白质-蛋白质关联网络,广泛可访问。核酸研究,2017;45:D362-8。
Dennis G Jr, Sherman BT, Hosack DA, Yang J, Gao W, Lane HC, Lempicki RA。DAVID:用于注释、可视化和集成发现的数据库。中国生物工程学报。2003;4:P3。
KEGG:京都基因和基因组百科全书。核酸Res. 2000; 28:27-30。
张勇,王松,李东,张军,顾东,朱勇,何峰。一种基于生物分类器的肝细胞癌诊断方法。PLoS ONE。2011; 6: e22426。
张艳,郭旭,熊玲,余玲,李铮,郭强,李铮,李波,林娜。综合分析microrna调控蛋白相互作用网络,揭示microRNA-149通过靶向AKT-mTOR通路在人肝细胞癌中的抑瘤作用。Mol Cancer, 2014;13:253。
林志智,黄永青,张永青,韩志东,何兴辉,凌晓辉,付旭,戴启启,蔡超,陈建辉。MicroRNA-224通过下调TRIB1抑制前列腺癌的进展。国际癌症杂志2014;135:541-50。
了解动力学:类风湿关节炎发病机制的相关途径。风湿病学(牛津大学)。51 2012; (5): v3-11。
Rabquer BJ, Pakozdi A, Michel JE, Gujar BS, Haines GK 3rd, Imhof BA, Koch AE。关节黏附分子C介导类风湿性关节炎滑膜白细胞黏附。风湿性关节炎。2008;58:3020-9。
Szekanecz, Koch AE。趋化因子通路靶向类风湿性关节炎的成功与失败。Nat Rev Rheumatol. 2016; 12:5-13。
Shelef MA, Bennin DA, Yasmin N, Warner TF, Ludwig T, Beggs HE, Huttenlocher A.黏附激酶是滑膜成纤维细胞入侵所必需的,但不是小鼠炎性关节炎。关节炎。2014;16:464。
Castro-Villegas C, Pérez-Sánchez C, Escudero A, Filipescu I, Verdu M, Ruiz-Limón P, Aguirre MA, Jiménez-Gomez Y, Font P, Rodriguez-Ariza, Peinado JR, Collantes-Estévez E, González-Conejero R, Martinez C, Barbarroja N, López-Pedrera C.循环mirna作为抗tnf α治疗类风湿性关节炎患者治疗有效性的潜在生物标志物。关节炎。2015;17:49。
Fransen J, Kooloos WM, Wessels JA, Huizinga TW, Guchelaar HJ, van Riel PL, Barrera P.预测MTX单药治疗在DMARD失败后类风湿关节炎患者反应的临床药物遗传学模型药物基因组学,2012;13:1087 - 94。
Krintel SB, Dehlendorff C, Hetland ML, Hørslev-Petersen K, Andersen KK, Junker P, Pødenphant J, Ellingsen T, Ahlquist P, Lindegaard HM, Linauskas a .阿达木单抗治疗应答的预测:早期类风湿性关节炎患者循环microRNA的双盲安慰剂对照研究药物基因组学杂志,2016;16(2):141-6。
de Jong TD, Lübbers J, Turk S, Vosslamber S, Mantel E, Bontkes HJ, van der Laken CJ, Bijlsma JW, van Schaardenburg D, Verweij CL。早期关节炎患者外周血白细胞亚群中的I型干扰素特征:主要由粒细胞贡献。关节炎,2016;18:165。
李春华,李春华,李春华。系统性红斑狼疮患者血浆I型干扰素的功能测定及其与抗rna结合蛋白自身抗体的关系。风湿性关节炎。2006;54:1906-16。
Sanayama Y, Ikeda K, Saito Y, Kagami S, Yamagata M, Furuta S, Kashiwakuma D, Iwamoto I, Umibe T, Nawata Y, Matsumura R, Sugiyama T, Sueishi M, Hiraguri M, Nonaka K, Ohara O, Nakajima H.类风湿性关节炎患者对tocilizumab治疗反应的预测:通过全基因组DNA微阵列分析外周血单个核细胞的基因表达确定的生物标志物。风湿性关节炎。2014;66:1421-31。
王华,王琳,Erdjument-Bromage H, Vidal M, Tempst P,等。组蛋白H2A泛素化在多梳沉默中的作用。大自然。2004;431:873-8。
Yagishita N, Aratani S, Leach C, Amano T, Yamano Y, Nakatani K, Nishioka K, Nakajima T无名指型泛素连接酶抑制剂是治疗类风湿关节炎的新候选药物。中华药理学杂志,2012;30:381 - 381。
李峰,李霞,寇利,李勇,孟F,马峰。sumo偶联酶UBC9促进类风湿关节炎成纤维细胞样滑膜细胞增殖和迁移。炎症。2014;37:1134-41。
Torre S, Polyak MJ, Langlais D, Fodil N, Kennedy JM, Radovanovic I, Berghout J, leva - torres GA, Krawczyk CM, Ilangumaran S, Mossman K, Liang C, Knobeloch KP, Healy LM, Antel J, Arbour N, Prat A, Majewski J, Lathrop M, Vidal SM, Gros p。中华免疫杂志,2017;18:54-63。
Hisakawa N, Tanaka H, Hosono O, Nishijima R, Ohashi Y, Saito S, Nishiya K, Hashimoto K, Morimoto C.类风湿性关节炎患者滑膜液T细胞对RANTES的异常反应。中华风湿病杂志2002;29:1124-34。
Miyake-Nishijima R, Iwata S, Saijo S, Kobayashi H, Kobayashi S, Souta-Kuribara, Hosono, Kawasaki H, Tanaka H, Ikeda E, Okada Y, Iwakura Y, Morimoto C. crk相关底物淋巴细胞类型在tax转基因小鼠和人类类风湿关节炎病理生理学中的作用。风湿性关节炎。2003;48:19 90 - 900。
Yoshida K, Shimizu Y, Yoshimoto J, Takamura M, Okada G, Okamoto Y, Yamawaki S, Doya K.利用偏最小二乘回归预测临床抑郁评分和检测全脑变化。PLoS ONE。2017; 12: e0179638。
陈志伟,陈志伟,陈志伟。基于偏最小二乘的脑功能图像空间模式分析。科学杂志。1996; 3:143-57。
麦金托什AR,洛博新泽西州。神经影像数据的偏最小二乘分析:应用与进展。科学杂志。2004; 23: s250 - 63。
作者的贡献
LN构思了这项研究,参与了设计并审阅了手稿。ZY设计了研究,进行了数据分析,并起草了手稿。WH进行了临床样本的收集,并起草了部分稿件。其他作者参与临床样本采集并进行统计学分析。所有作者都阅读并批准了最终的手稿。
确认
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
数据和材料的可用性
不适用。
发表同意书
不适用。
伦理批准并同意参与
本研究根据《赫尔辛基宣言》的人类指南进行,并得到了广安门医院研究伦理委员会的批准。所有患者均获得知情同意。
资助声明
本研究得到北京市新星计划(Z1511000003150126)、北京市自然科学基金(7152107)、国家自然科学基金(81673804,81673834)、中华人民共和国人力资源和社会保障部归国留学人员科研基金(31470962)、中央公益性科研院所基本科研业务费(Z2017082、ZZ0708079、L2017018、GH2017-06)资助。
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
作者信息
作者及隶属关系
相应的作者
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
张勇,王宏,毛X。et al。类风湿关节炎患者对雷公藤多苷片反应的一种新的基于基因表达特征的预测模型。翻译医学杂志16, 187 (2018)https://doi.org/10.1186/s12967-018-1549-9
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12967-018-1549-9
关键字
- 个性化医疗
- 类风湿性关节炎
- 雷公藤多苷片
- 基因表达谱
- 基因信号转导网络
- 偏最小二乘模型