摘要
背景
长期以来,乳腺癌家族史一直被认为表明存在易患这种疾病的遗传遗传事件。在北非,与西方人群相比,在乳腺癌家族中观察到许多特定的流行遗传学特征。尽管有这些特异性,在北非进行的大多数乳腺癌遗传学研究仍然局限于对乳腺癌的研究乳腺癌易感基因1而且BRCA2基因。因此,缺乏来自当地患者的全外显子组或全基因组水平的全面数据。
方法
使用基于家庭的方法对7个突尼斯乳腺癌家庭进行了全外显子组测序(WES)。我们重点分析了BC-TN-F001家族,其中包括两个使用WES测序的受影响成员。在BC-TN-F001中发现的相关变异已通过Sanger测序得到确认。然后,我们将我们的结果与其他WES研究的结果进行了整合分析,以找出新鉴定的基因的遗传传播模式。生物网络构建和蛋白质-蛋白质相互作用分析已经完成,以破译可能解释这些基因在乳腺癌风险中的作用的分子机制。
结果
测序、过滤策略和验证分析已经实现。对于BC-TN-F001,在已知的乳腺癌基因上没有发现有害突变。然而,在其他候选基因上已鉴定出373个杂合、外显子和罕见变异。经过多次筛选,筛选出了12个相关的高危变异。我们的研究结果表明,这些变异似乎是在家族特定模型中遗传的。这一假设在对报道的WES研究进行彻底分析后得到了证实。丰富的生物过程和蛋白质-蛋白质相互作用网络导致了四个新的乳腺癌候选基因的鉴定MMS19, DNAH3,波尔克而且KATB6。
结论
在突尼斯乳腺癌患者的首次WES应用中,我们强调了下一代测序技术在识别新型乳腺癌候选基因方面的影响,这可能会为乳腺癌发生的生物学机制带来新的见解。我们的研究结果表明,乳腺癌易感性在非BRCA家庭可能是种族和/或家庭特定的。
背景
一系列遗传和非遗传风险因素导致乳腺癌的发展[1].到目前为止,通过家族连锁、DNA重测序和全基因组关联分析,已经分别确定了几种高、中、低外显率的遗传变异对乳腺癌风险的影响[2].额外的乳腺癌相关基因的鉴定对于解释缺失的乳腺癌遗传性至关重要。最近的研究表明,乳腺癌易感性可能是由遗传的多基因风险模型来解释的,在该模型中,大量的共同snp对风险有增殖作用[3.].随着新一代测序技术的引入[4,5]许多研究表明,其余乳腺癌遗传率的很大一部分可归因于新的罕见风险等位基因,这些等位基因在常染色体显性遗传模式中分离。
迄今为止,使用了两种不同的全外显子组测序研究设计:病例/对照关联研究和基于家族的方法。病例/对照设计被认为是检测遗传变异与乳腺癌疾病之间显著关联的主要有前途的工具[6].然而,由于某些变异的极度罕见,这种方法需要大规模的队列来确认这些变异与乳腺癌风险之间的关联。第二个WES设计是基于家庭的方法[7],其中乳腺癌家族成员进行外显子测序,受影响个体之间的共享变异可能包括家族乳腺癌风险等位基因。因此,关注相关变异的家族分离有望比筛选不相关病例和对照更好地发现新的易感变异。
已经对遗传性乳腺癌进行了几项WES研究[7,8].几乎有108个乳腺癌家族使用基于家族的方法进行了全外显子组测序,并报告了许多相关变异存在于相关的受影响个体中,而在未受影响的个体中不存在。到目前为止,WES已经确定了5个与乳腺癌风险相关的新基因,其中4个基因是使用基于家庭的方法确定的,即:XRCC2[9),MAPKAP1[10),FANCM[11),RINT1[12]而只有一个基因REQCL,是采用病例/对照方法确定的[13].已知乳腺癌易感基因突变仅在四个家族中报告[10,11,12,13,14].
在突尼斯,乳腺癌是女性中最常见和最致命的癌症。15].一些流行病学、遗传学和临床乳腺癌特征已被观察到是独特的突尼斯和北非人口。的确,与西方人群相比,乳腺癌的发病率较低,但发病年龄较低,炎症性乳腺癌和三阴性乳腺癌等侵袭性乳腺癌形式的发病率相对较高[16].因此,特定于这个种族群体的遗传倾向是合理的,[8,17,18].此外,有可能乳腺癌风险变异非常罕见,以至于它们是“家族特异性的”,这意味着在易发疾病的家庭中可以检测到遗传易感性,但不一定与患有相同疾病的其他基因不相关的家庭共享[19,20.,21].
到目前为止,对突尼斯乳腺癌患者进行的基因研究主要集中在BRCA基因使用传统的桑格技术。因此,在这些未开发人群的遗传调查中使用下一代测序技术可能有助于识别新的乳腺癌风险等位基因,并解释尚未解决的乳腺癌遗传问题。
在本研究中,我们对7个具有较强家族史的突尼斯BRCAx乳腺癌家族进行了全外显子组测序,以确定可能与乳腺癌风险相关的遗传变异。使用基于家庭的方法,我们通过对三个受影响姐妹中的两个进行测序,将我们的分析集中在一个非BRCA家族上。在将我们的结果与之前WES研究中确定的结果进行比较并进行生物网络分析后,我们确定了一组新的乳腺癌候选基因,这些基因似乎是以家族特定的方式遗传的。
方法
病人
根据以下标准选择了7个突尼斯乳腺癌家庭进行WES:(1)存在至少3个相关的一级或二级乳腺癌病例;(2)年龄小于35岁的年轻乳腺癌患者,(3)至少有2例乳腺癌或卵巢癌,与年龄无关,相关的一级或二级患者中至少有1例胰腺癌或前列腺癌。已经从受影响的家庭成员那里采集了血液样本,并在突尼斯阿利亚纳Abderrahman Mami医院肿瘤科进行了采样。所有参与者均获得书面知情同意。根据《赫尔辛基原则宣言》获得了突尼斯巴斯德研究所生物医学伦理委员会的伦理批准(2017/16/E/Hôpital a-m/V1)。
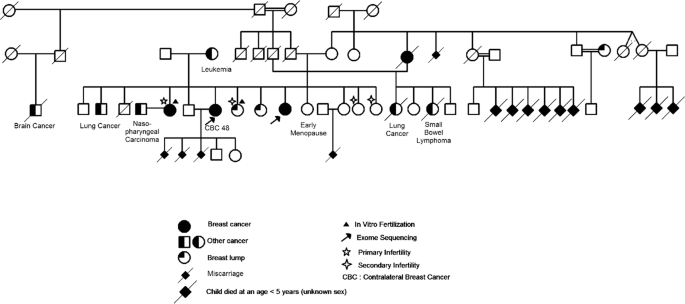
BC-TN-F001中3个患病姐妹中有2个已进行了全外显子组测序。先证者43岁时被诊断为原发性乳腺癌,48岁时被诊断为对侧浸润性导管乳腺癌。参与这项研究的第二个家庭成员在56岁时被诊断出患有侵袭性乳腺癌。表中描述了受影响家庭成员的表型特征1.
全外显子组测序和数据分析
根据制造商的说明,使用盐析法或Qiagen公司的DNeasy血液试剂盒从每个参与者的外周血中分离出总基因组DNA。DNA纯度和浓度使用NanoDrop™分光光度计测量。
样品根据安捷伦SureSelect协议1.2版本进行制备,并根据安捷伦SureSelect协议进行富集。富集后的样品在Illumina HiSeq 2000平台上使用TruSeq v3化学对端测序(2 × 100pb)。
使用Burrows-Wheeler Aligner (BWA)包将外显子组DNA序列映射到其在人类基因组构建中的位置(hg19/b37)。后续的SAM文件使用Samtools转换为BAM文件。使用Picard删除重复读取。然后使用GATK重新校准基础质量评分以及SNP和短INDEL呼叫。使用VarAFT(变体注释和过滤工具)对潜在致病变体进行注释和优先级排序(http://varaft.eu).为了注释变量,VarAFT使用ANNOVAR,这是一个命令行工具。根据以下几个标准对注释的INDELs和snp进行筛选:(1)将乳腺癌视为常染色体显性疾病,去除纯合子状态的变异;(2)剔除内含子型、基因间型、无编码或同义型变异;(3)假设因果变异很罕见,我们去除Exac中所有等位基因频率为> 1%的变异。22], 1000个基因组[23]或ESP6500 (http://evs.gs.washington.edu/EVS/),(4)良性或耐受变异,根据不同的硅预测工具也被去除。最后,通过筛选其表型相关性,获得显著的候选变异。
桑格测序
桑格测序技术首次用于检测BRCA受影响家庭成员的状态,然后验证从整个外显子组测序中鉴定的变异。按照标准方案对基因组DNA (gDNA)进行PCR反应,然后使用自动测序仪进行Sanger测序(ABI 3500;应用生物系统公司,福斯特城,CA)使用循环测序反应试剂盒(大染料终结者试剂盒,应用生物系统公司)。数据分析使用BioEdit Sequence Alignment Editor Version 7.2.5。
在硅预测工具
我们选择了四种硅预测工具来评估候选变异的功能效果:从耐受性到不耐受性的排序(SIFT) (http://sift.jcvi.org/)研究不同物种氨基酸残基的保存程度,以及蛋白质结构和功能的变化;PolyPhen-2 (http://genetics.bwh.harvard.edu/pph2/)及变异品尝师(http://www.mutationtaster.org/)以评估突变对蛋白质功能的影响,并观察对剪接或mRNA表达的影响和对齐GVGD (http://agvgd.iarc.fr),将查询序列中的错义变体分为七个等级,从危害最大的C65到危害最小的C0,中间等级为C15、C25、C35、C45和C55 [24].该程序基于Grantham计算,Grantham变异(GV)测量在对齐的特定位置观察到的生化进化变异量,Grantham偏差(GD)测量错义残基之间的生化差异和在对齐的该位置观察到的变异范围。
功能注释与生物网络构建
为了识别候选乳腺癌基因的含义,一些生物信息学工具被用于探索它们的生物学途径和可能的蛋白质-蛋白质相互作用。
我们首先使用浓缩铀平台进行功能分析[25],这是一种基于生物信息学的工具,包括60多个基因集库,如基因本体[26], KEGG, Wikipathways,以及Jensen-diseases。显著丰富路径和本体术语的选择标准为ap值小于0.05(附加文件1:表S1)。
为了更好地可视化和解释与所选乳腺癌候选基因及其上游调控因子相关的生物学过程,我们使用了ClueGO [27],一个用户友好的Cytoscape插件,用于分析生物网络中术语和功能组的相互关系[28].简而言之,我们采用富集(右侧)超几何分布检验,p值显著性水平≤0.05,然后对术语和Kappa-statistics得分阈值设置为0.5的组进行Bonferroni调整,并根据最高显著性选择领先术语组。
蛋白质-蛋白质相互作用网络,包括我们基因组的物理和功能关联,使用字符串db 10.0 [29],自信心得分0.4。
结果
使用全外显子组测序分析了来自7个BRCAx突尼斯乳腺癌高危家庭的8名患者。全文总结了整个外显子组测序患者的reads数、样本覆盖率和测序深度等结果1:表S2。
我们目前的分析主要集中在第一个问题上BRCA消极的家庭;BC-TN-F001(无花果。1).三分之二的受影响家庭成员已被选中进行全外显子组测序。
已知乳腺癌易感基因变异分析
在应用过滤器之前,方法部分描述的步骤,我们首先调查了以下29个已知与遗传性乳腺癌和卵巢癌相关的基因: atm, bard1, brca1, brca2, blm, brip1, cdh1, chek2, fam175a, fancc, fancm, mapkap1, mlh1, mre11a, msh2, nbn, nf1, palb2, pms2, pten, rad50, rad51b, rad51c, rad51d, recql, rint1, stk11, tp53而且XRCC2(表2).这些基因共鉴定出59个共享杂合变异,其中51个(86.4%)为常见非编码变异,5个外显子变异,3个剪接SNPs。外声子变体包括aBRCA2罕见变异(rs4987047, MAF = 0.0089), 3个常见的外显子多态性BARD1(rs2070094, rs2229571和rs1048108),以及一个变体MAPKAP1(rs1201689)。没有发现任何杂合变异Brca1, blm, fam175a, fancm, pten, rad50, rint1, stk11, tp53而且XRCC2在两个测序的家族成员之间共享。
基于乳腺癌信息核心(BIC)和ClinVar数据库,在这些经典乳腺癌和卵巢癌基因上鉴定的59个变异中,没有一个被归类为致病性。因此,我们认为这个家族的乳腺癌遗传易感性可能是由于新的乳腺癌候选基因的新变异。
新候选变异的鉴定
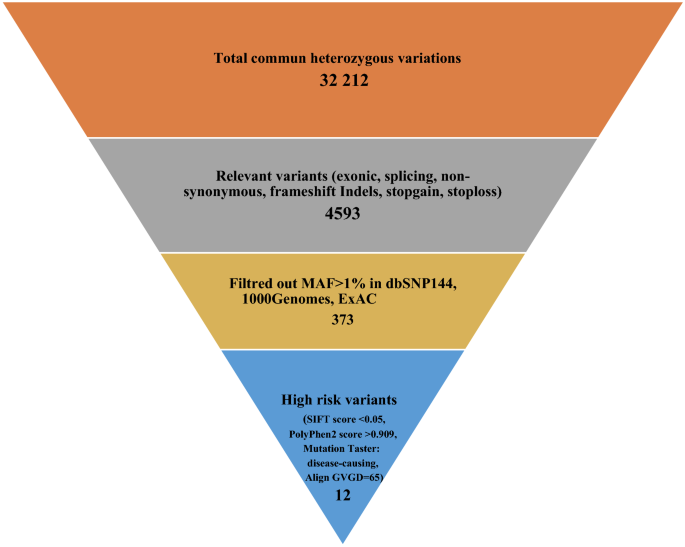
两种病例共鉴定出32,212个杂合变异(图2)。2).其中被称为杂合、外显子、剪接和非同义snp的有4593个。MAF > 1%的变体已被排除。因此,我们选择了373个罕见变异进行进一步研究,其中包括39个以前没有报道过的变异。事实上,由于突尼斯人口在公共数据库中没有被代表,报告的变体并没有被排除。
为了选择最相关的SNPs,我们使用SIFT(得分< 0.05)、PolyPhen(得分> 0.909)、Mutation Taster(得分> C55)和Align GVGD(得分> C55)作为计算机预测工具来评估373个变异的功能效果。
基于对硅预测的兴趣,选择了12个高风险变异的列表(表2)3.),其中有七个非同义变体HSD3B1,PBK, ITIH2, MMS19, PPL, DNAH3和RASSF2, 1剪接变化上雌性生殖道, 2个停止增益变量CALCOCO2而且LRRC29, 1移码删除开启PABPC3和1移码插入ZNF677.ClinVar数据库中没有列出这些变异,除了CFTR-rs1057516216变异似乎“可能致病”。
家庭特定假说
我们首先针对另外6个BRCAx外显子组测序的乳腺癌家族(BC-TN-F002_BC-TN-F007)筛选了这个候选基因和变体列表。所有已鉴定的变异只在BC-TN-F001中发现,除了PABPC3在其他突尼斯BRCAx家族中发现了这种变异。
然后,我们将该家族中确定的变异列表与其他关于BRCAx家族的WES研究结果进行了比较。同样,本研究中发现的变异只在BC-TN-F001中发现,这表明家族特异性乳腺癌易感性。在其他4项WES研究中,这种家族特异性假说已被用于解释乳腺癌易感性[8,19,20.,21].
因此,我们根据4个家庭特异性WES研究结果和当前的研究结果进行了文献整理,以探索这个家庭特异性乳腺癌易感性。额外的文件1表S3总结了通过这些研究确定的54个基因列表,这些基因是在家族特定模型中遗传的新的潜在乳腺癌候选基因。我们观察到,每个外显子组测序家族都表现出特定的遗传模式,具有不同的候选基因集。只有KAT6B在两项独立研究的两个不同家庭中均有报道[19,20.].
在最近对五个BRCAx埃及家庭进行的WES研究中[8],即四个基因LOC100129697,NPIPB1, NBPF10而且PABPC3在不止一个家庭中被发现。PABPC3在目前的研究中,也发现了三个埃及家庭和四个突尼斯家庭共享的基因序列。
基因集富集分析
由于大多数通过家族特异性易感性研究确定的乳腺癌候选基因缺乏参与乳腺癌发生的功能证据,我们汇集了单独WES研究中确定的54个候选基因(附加文件)1:表S3),我们进行了功能注释分析,以探索这些基因之间是否存在任何生物相互作用,从而加强其与乳腺癌的关联(附加文件)1:表S1;额外的文件2:图S1)。
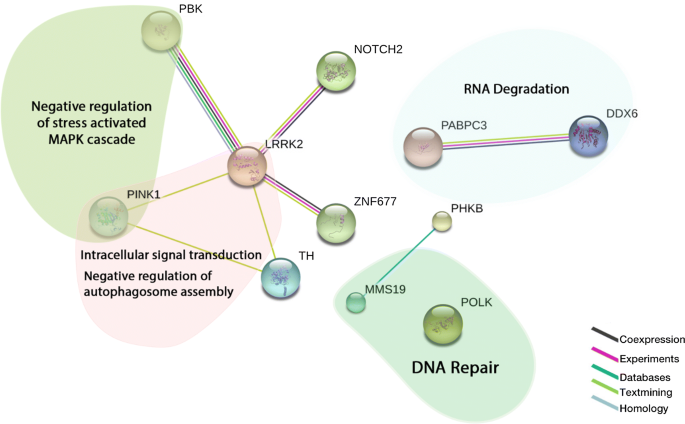
此外,使用enrichment r和Stringdb webtools进行了综合基因集富集和蛋白质-蛋白质相互作用分析。结果显示MMS19而且波尔克基因参与了DNA修复途径(图;3.).其余基因是参与癌症病因学的几种途径的一部分,如:应激激活MAPK级联的负调控(PBK而且PINK1)、细胞内信号转导及自噬体组装的调控(LRRK2而且PINK1)和RNA降解(PABC3而且DDX6).NOTCH2而且ZNF677高度预测与?共表达PBK而且LRRK2(无花果。3.).
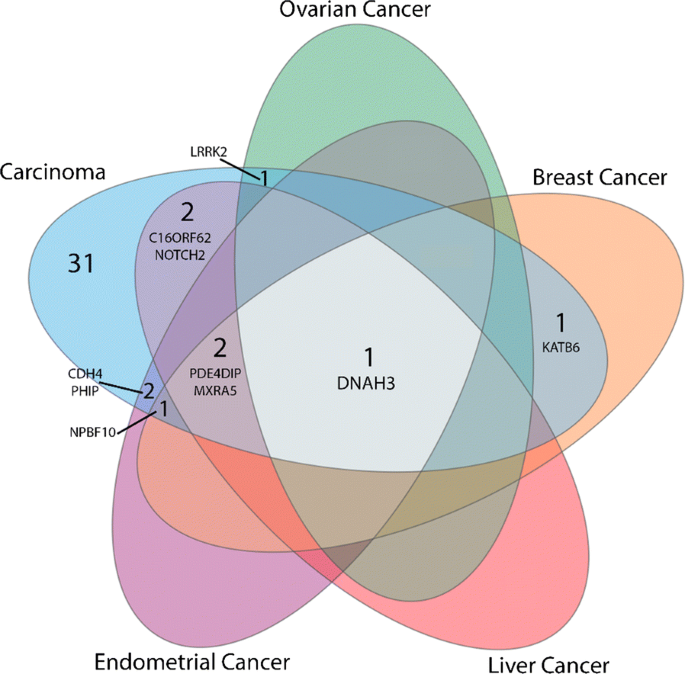
最后,我们使用Jensen疾病数据库(PMID: 25484339)进行了疾病基因关联分析,将候选基因聚类为涉及同一疾病的亚群。因此,我们检查了这些亚簇与不同癌症之间的重叠,即乳腺癌、卵巢癌、肝癌和子宫内膜癌(图2)。4).获得的结果显示了与乳腺癌相关的五个最重要的基因Dnha3, katb6, pde4dip, mxra5而且NBPF10.值得注意的是,NBPF10也与子宫内膜癌有关DNHA3是所有这些癌症中唯一可能出现的。
讨论
大多数患有家族性乳腺癌的BRCAx患者缺乏遗传易感性的证据。人们提出了多种模型来解释缺失的遗传力。首先,已提出隐性和多基因传播模型,以解决乳腺癌的一部分剩余遗传[30.].导致家族性乳腺癌风险的另一类遗传变异包括大量缺失和拷贝数变异[31].遗传变异和环境危险因素之间的相互作用仍然是解释多家庭乳腺癌易感性的有趣模型。然而,该模型在很大程度上尚未被探索,因为大多数可以解决该模型的关联研究都不足[32].最后,使用基于家族的方法的NGS应用代表了一种合适的方式来识别具有常染色体显性遗传机制的额外基因,从而解释了乳腺癌家族成分的额外部分[7].
在目前的研究中,两个受影响的姐妹从一个非BRCA突尼斯乳腺癌家族已经使用全外显子组测序进行了探索。我们在测序的个体中排除了未受影响的成员,因为他们可能是非渗透携带者。
这两个已测序的家族成员之间共有数千个杂合变异。然而,在已知的乳腺癌基因中还没有发现有害的变异。BRCA2-rs4987047是已知乳腺癌易感基因上唯一罕见的外显子变异。尽管它具有潜在的功能作用[33], ClinVar预测将这种变异归为良性。
值得注意的是,在此前10项乳腺癌WES研究中报道的108个外显子组测序家族中,已知乳腺癌基因突变仅在4个家族中报道,因为通常在使用全外显子组测序方法之前进行BRCA检测[10,11,12,13,14].此外,突尼斯人口的高血亲率可能通过降低高穿透突变的频率来降低乳腺癌的患病率[34].
然而,在BC-TN-F001中发现了几个位于已知乳腺癌易感基因上的常见变异(表2)2).这些变异中的一些已经被报道为与不同的癌症相关的低渗透多态性。事实上,两种常见的外显子变体在BARD1基因(rs2229571和rs1048108)在中国人群中被鉴定为低穿透性乳腺癌基因座[35].此外,PALB2-rs249954已被报道与乳腺癌风险相关[36),CHEK2-rs2236142可能与中国人群食管癌和淋巴结转移风险降低有关[37),RAD51C-rs12946397已知与头颈癌风险相关[38),自动取款机-rs664143已被报道与肺癌有关[39].考虑到多个家庭成员都受到其他癌症的影响,如肺癌和小肠淋巴瘤(图。1),这些变异可能与该家族的癌症易感性有关。因此,我们不能抛弃突尼斯乳腺癌家族中乳腺癌易感性的多基因模式。
尽管这些变异在白种人中被报道为常见的低渗透变异,但我们无法估计其在突尼斯人群中的外显率。事实上,由于不同的遗传结构和等位基因频率在群体之间的差异,变异外显率可能在一个群体与另一个群体之间存在差异,一个群体中的低外显率变体可能在另一个群体中具有高外显率。需要在突尼斯人群中进行进一步的关联研究,以评估这些变异在突尼斯人群中的外显率。
在研究了已知的乳腺癌基因后,我们探索了其他尚未报道的与乳腺癌相关的基因。在以下基因上选择了12个高风险变异,由四种不同的硅预测工具预测为有害的,并显示出表型相关性:Hsd3b1, cftr, pbk, itih2, mms19, pabpc3, ppl, dnah3, lrrc29, calcoco2, znf677而且RASSF2。
ClinVar数据库中没有列出在这些基因中发现的变异,除了雌性生殖道-rs1057516216变体,似乎“可能致病”。雌性生殖道(囊性纤维化跨膜电导调节基因)是一种编码atp结合盒(ABC)转运体超家族成员的基因[40].该基因的突变会导致囊性纤维化,这是北欧血统人群中最常见的致命遗传疾病[41].然而,雌性生殖道由于其体积大,可能偶然反复突变,其参与乳腺癌发生存在争议,因此不能视为潜在的乳腺癌候选基因。事实上,有人提议a雌性生殖道基因突变可预防乳腺癌[42],然而,在另一项CFTR表达水平与乳腺癌组织学分级相关的研究中,表明血清中CFTR的高水平与高分级、低分化的肿瘤相关[43].
当将确定的基因集与其他乳腺癌WES研究中报道的其他基因进行比较时,我们发现每个外显子组测序家族都具有特定的遗传模式,具有不同的候选基因集。除了PABPC3在突尼斯乳腺癌家族中发现的基因尚未在其他乳腺癌外显子组测序家族中报道,这表明家族对该疾病有特定的遗传易感性。PABPC3四个突尼斯家庭和三个埃及全外显子组测序家庭共享。此外,LOC100129697,NPIPB1, NBPF10已在三个完整外显子组测序的埃及家族中发现[8].这些来自特定族群(突尼斯人和埃及人)的家族共享的基因表明,在高血缘和内婚制率的人群中,种族特异性乳腺癌易感性模型也是合理的。PABPC3在mRNA代谢的细胞质调节过程中起作用[44].的参与PABPC3我们在本研究中对生物过程和蛋白质-蛋白质网络的分析已经证实了RNA降解途径中的这种作用(附加文件)2:图S1、图3.).
我们还表明,其余基因也与有趣的新途径有关,如:应激激活的MAPK级联的负调控和细胞内信号转导和自噬体组装的调节。只有两个基因(MMS19而且波尔克)参与DNA修复途径,被认为是乳腺癌基因参与的传统途径[45].
MMS19作为早期作用的细胞质铁硫组装成分和细胞靶铁硫蛋白子集(如ERCC2/XPD, FANCJ和RTEL1)之间的适配器,从而在核苷酸切除修复(NER)和RNA聚合酶II (POL II)转录中发挥关键作用[46].值得注意的是,人类MMS19也以不依赖配体的方式与雌激素受体相互作用[47].波尔克是Y家族DNA聚合酶的一员,通过修复穿过DNA损伤的复制叉发挥作用[48].最近,波尔克已被报道为新的卵巢癌易感基因[49].
使用Jensen疾病文库进行的额外功能注释分析显示,与乳腺癌相关的最重要基因是Katb6 pde4dip mxra5 dnha3而且NBPF10.kat6b -一种参与DNA复制、基因表达和调控以及染色体结构表观遗传修饰的组蛋白乙酰转移酶[50]在两项独立的WES研究中被报道与乳腺癌有关[19,20.].
与我们的结果一致的是,有报道说DNHA3与不同的癌症有关,包括乳腺癌[51,52,53].DNHA3(Dynein Axonemal Heavy Chain 3)基因属于动力蛋白家族,该家族成员编码微管相关运动蛋白复合物的组成部分大蛋白[54].在其相关途径中,我们指出了呼吸电子传递,通过化学渗透偶联合成ATP,以及通过解偶联蛋白质产生热量。然而,很少有证据存在的作用PDE4DIP,MXRA5,NBPF10在乳腺癌发生过程中。
综上所述,这些WES研究结果和本研究进行的功能注释,共同表明Mms19, dnha3, polk而且KATB6是有趣的乳腺癌候选基因。位于这些基因上的变异似乎是在家族特定模式中遗传的。PABPC3似乎是另一个有趣的乳腺癌候选基因,它可能与乳腺癌以特定种族的方式相关,因为在另一个北非人群中也有报道[8].
虽然NGS代表了一种前所未有的方法来破译不同遗传疾病的遗传易感性,但它也带来了许多挑战。事实上,不同的乳腺癌WES研究产生的不同的基因列表可能部分是由用于分析这些数据的不同管道和生物信息学工具所解释的。此外,NGS数据用户应用不同的过滤器来帮助确定变量的优先级,例如可能对某些变量错误分类的内嵌预测工具,从而导致错误地包含或排除某些变量。
因此,为了评估家族特异性假设在多大程度上是可信的,我们建议收集所有乳腺癌全外显子组测序家族的原始数据,并使用共同和共识的策略重新分析结果数据。COMPLEXO小组在通过下一代合作确定缺失的乳腺癌遗传性方面所做的努力代表了克服这些NGS数据分析挑战的绝佳举措[55].
结论
在本研究中,我们报告了一份新的乳腺癌候选基因列表,这些基因似乎是在家族特异性和种族特异性模型中遗传的。需要对BRCAx突尼斯家族进行进一步的WES研究和进一步的体外或体内功能分析,以了解它们的影响并确认它们与乳腺癌风险的关系。为了更好地解释NGS数据,科学界应该首先克服NGS数据分析的挑战,以生成更有意义的NGS数据和更多临床可操作的变体。
缩写
- 美国广播公司(ABC):
-
磷酸腺苷磁带
- BAM:
-
二进制对齐图
- BIC:
-
乳癌资讯核心
- BRCAx:
-
非BRCA
- BWA:
-
burrows - wheeler对准器
- 背景:
-
脱氧核糖核酸
- GD:
-
格兰瑟姆偏差
- gDNA:
-
基因组DNA
- 问:
-
格兰瑟姆的变化
- INDEL:
-
insertion-deletion
- 加:
-
小等位基因频率
- 信使rna:
-
信使核糖核酸
- 尼珥:
-
核苷酸切除修复
- 门店:
-
下一代测序
- 聚合酶链反应:
-
聚合酶链反应
- 波尔II:
-
RNA聚合酶II
- RNA:
-
核糖核酸
- 山姆:
-
序列比对图
- 筛选:
-
区分不宽容和宽容
- SNP:
-
单核苷酸多态性
- VarAFT:
-
变量注释和过滤工具
- 韦斯:
-
全外显子组测序
参考文献
乳腺癌流行病学与危险因素研究。中华妇产科杂志,2016;59(4):651-72。
Maxwell KN, Nathanson KL.后cogs时代常见乳腺癌风险变异:一项综合综述。乳腺癌,2013;15(6):212。
李俊杰,等。多基因风险、个性维度与青少年酒精使用问题:一项纵向研究中国药理学杂志,2017;28(3):442-51。
Shendure J,等。先进的测序技术:方法和目标。中国科学院学报。2004;5(5):335。
沈忍,季华。下一代DNA测序。生物技术学报。2008;26(10):1135。
Sokolenko AP,等。全外显子组测序鉴定新型遗传性癌症基因。中华癌症杂志,2015;36(2):344 - 344。
Chandler MR, Bilgili EP, Merner ND。遗传性乳腺癌易感基因发现的全外显子组测序研究综述。胡木,2016;37(9):835-46。
Kim YC,等。外显子组测序揭示了五个埃及家族乳腺癌家族种系变异的独特特征。PLoS ONE。2017; 12 (1): e0167581。
Park D,等。XRCC2的罕见突变会增加患乳腺癌的风险。王志强,王志强。2012;30(4):344 - 344。
Gracia-Aznarez FJ,等。全外显子组测序表明,大部分非brca1 /BRCA2家族性乳腺癌是由于中外显率和低外显率易感等位基因。PLoS ONE。2013; 8 (2): e55681。
Kiiski JI,等。外显子组测序确定FANCM是三阴性乳腺癌的易感基因。自然科学进展,2014;29(4):344 - 344。
Park DJ等。RINT1的罕见突变易使携带者患乳腺癌和Lynch综合征谱癌。癌症杂志,2014;4(7):804-15。
Cybulski C,等。生殖系RECQL突变与乳腺癌易感性相关。中国科学,2015;47(6):643。
汤普森ER,等人。外显子组测序发现DNA修复基因FANCC和BLM中罕见的有害突变为潜在的乳腺癌易感性等位基因。公共科学学报,2012;8(9):e1002894。
Dimassi K,等。突尼斯育龄妇女的癌症死亡率。突尼斯医学,2016;94(1):16-22。
Corbex M, Bouzbid S, Boffetta P.发展中国家乳腺癌的特征,来自北非的例子。中华癌症杂志,2014;30(10):1808-18。
Al-Eitan LN, Jamous RI, Khasawneh RH。约旦阿拉伯后裔乳腺癌的候选基因分析:一项病例对照研究。癌症投资杂志,2017;35(4):256-70。
Bayraktar S,等。BRCA突变携带者的基因型-表型与种族和突变位置的相关性年代.乳房杂志,2015;21(3):260-7。
Wen H,等。家族特异性的,新颖的,有害的种系变异为确定BRCAx家族性乳腺癌的遗传易感性提供了丰富的资源。BMC癌症。2014; 14(1): 470。
Lynch H,等。家族性乳腺癌的未知易感性是家族特异性的吗?乳房杂志,2013;19(5):520-8。
Noh JM,等。一个无BRCA突变的乳腺癌家族的外显子组测序中国生物医学工程学报,2015;33(2):349。
Lek M,等。60706人蛋白质编码遗传变异分析。大自然。2016;536(7616):285。
G.P.人类遗传变异的全球参考。大自然。2015;526(7571):68。
Tavtigian SV,等。在用基于序列比对的方法进行误义替换的计算机分析中。胡木,2008;29(11):1327-36。
陈永毅,等。enrichment:交互式协作HTML5基因列表丰富分析工具。中国生物医学工程学报。2013;14(1):128。
Ashburner M,等。基因本体论:生物学统一的工具。中国科学。2000;25(1):25。
Szklarczyk D,等。STRING v10:蛋白质-蛋白质相互作用网络,整合在生命树上。中国农业科学,2014;43(D1): D447-52。
陈永毅,等。Expression2Kinases:与多个上游调控层相连的mRNA分析。生物信息学,2011,28(1):105 - 11所示。
Shannon P,等。细胞景观:生物分子相互作用网络集成模型的软件环境。基因组学报,2003;13(11):2498-504。
法老PD,等。乳腺癌的多基因、风险预测和靶向预防。中华实用医学杂志,2008;26(4):366 - 366。
Enyedi MZ,等。在生殖系DNA和FFPE肿瘤样本中同时检测BRCA突变和大规模基因组重排。Oncotarget。2016;7(38):61845。
Nickels S,等。常见乳腺癌易感位点与既定环境危险因素之间基因-环境相互作用的证据。公共科学学报,2013;9(3):e1003284。
Johnson N,等。计数BRCA1、BRCA2和ATM中的潜在功能变异可以预测乳腺癌易感性。胡文杰,2007;16(9):1051-7。
Denic S, Bener A.血缘关系降低乳腺癌风险,宫颈癌不受影响。中华癌症杂志2001;85(11):1675。
刘浩,等。BARD1基因非同义突变与汉族女性乳腺癌之间的横断面研究《亚太公共卫生杂志》,2013;25(4_增刊):8S-14S。
陈平,等。常见PALB2多态性与乳腺癌风险的关联:一项病例对照研究临床癌症杂志,2008;14(18):5931-7。
顾华,等。CHEK2变异等位基因与中国人群食管癌淋巴结转移风险降低相关Mol生物学杂志2012;39(5):5977-84。
格雷斯纳P,等。Rad51C:一种新的抑制基因调节头颈部癌症的风险。生物化学学报。2014;762:47-54。
沈璐,等。ATM多态性与癌症风险的关联:一项荟萃分析。Mol生物学杂志2012;39(5):5719-25。
Hyde SC,等。atp结合保护与囊性纤维化、多药耐药和细菌转运相关的结构模型。大自然。1990;346(6282):362。
削减GR等。囊性纤维化导通调节蛋白的第一个核苷酸结合折叠中的一组囊性纤维化突变。大自然。1990;346(6282):366。
李毅,等。囊性纤维化跨膜电导调节基因突变与肺癌风险。中华癌症杂志。2010;70(1):14-21。
Southey MC,等。CFTR ΔF508在一项以人群为基础的病例对照研究中,乳腺癌携带者状况、40岁前乳腺癌风险和组织学分级。中华肿瘤学杂志,1998;39(5):489 - 497。
Ozturk S,等。聚(A)结合蛋白基因EPAB, PABPC1和PABPC3在非阻塞性无精子症不育男性中表达差异。辅助生殖学报,2016;33(3):335-48。
Katsuki Y, Takata M.人类疾病背后的同源重组修复缺陷:FA和HBOC。中国癌症杂志,2016;23(10):T19-37。
Hatfield MD等人。在NER和转录中具有不同功能的MMS19结构域的鉴定。DNA修复。2006;5(8):914-24。
吴霞,李辉,陈景德。酵母DNA修复和TFIIH调节剂MMS19的人类同源物是af -1特异性雌激素受体的辅激活剂。中国生物医学工程学报,2001;26(2):339 - 339。
Lone S,等。人DNA聚合酶κ环DNA:错配延伸和病变绕过的意义。生物化学学报。2007;25(4):601-14。
Stafford JL,等。BRCA1/2阴性高危卵巢癌患者的再分析揭示了新的种系风险位点和对缺失遗传性的见解。PLoS ONE。2017; 12 (6): e0178450。
香槟N等。与单核细胞白血病锌指蛋白相关的人组蛋白乙酰转移酶的鉴定。中国生物医学工程学报,1999;29(4):339 - 339。
Ichikawa T,等。肺癌模拟组织性肺炎的免疫组化和遗传特征。肺癌。2017;113:134-9。
McIver LJ,等。微卫星基因分型揭示了乳腺癌外显子体的一个特征。乳腺癌治疗与治疗。2014;34(3):379 - 379。
Suo C,等。体细胞突变、表达和功能数据的整合揭示了预测乳腺癌生存的潜在驱动基因。生物信息学,2015,31(16):2607 - 13所示。
威斯戴德B,海鸥K.真核生物的动力蛋白:比较基因组分析。交通。2007;8(12):1708 - 21所示。
Southey MC,等。COMPLEXO:通过下一代合作确定乳腺癌缺失的遗传性。乳腺癌,2013;15(3):402。
作者的贡献
研究构想与设计:YH和SA。数据采集:YH、MB、CN。资料分析与解释:YH、MB、NM对患者临床病理资料进行分析与解释。生物信息分析和网络:CBH, KG和YH。对结果解释的贡献HB, SL, NMJ, HE和ND技术实验:MCH, MB, NM。全文编校:YH。参与稿件起草:SA, MB, NM文章的重要修订:SA, MSB, RM和OM。提交程序:MBR、NM、OM。所有作者都阅读并批准了最终的手稿。
确认
作者非常感谢患者的参与使这项工作成为可能。
相互竞争的利益
作者宣称他们之间没有利益冲突。
数据和材料的可用性
在这项研究中产生或分析的所有数据都包含在这篇发表的文章及其附加文件中。
发表同意书
不适用。
伦理批准并同意参与
所有参与者均获得书面知情同意。根据《赫尔辛基原则宣言》获得了突尼斯巴斯德研究所生物医学伦理委员会的伦理批准(2017/16/E/hôpital a-m/V1)。
资金
这项工作得到了突尼斯公共卫生部(PEC-4-TUN)、突尼斯高等教育和科学研究部(LR11IPT05和LR16IPT05)以及E.C. FP7项目GM-NCD-Inco No 295097赠款协议的支持。
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
作者信息
作者及隶属关系
财团
相应的作者
附加文件
附加文件1:表S1。
基因集富集分析。表S2。7个突尼斯乳腺癌BRCAx测序家族中snp和Indels的总结。表S3。假定易感性家庭特定基因在几个WES研究中使用基于家庭的方法。
附加文件2:图S1。
功能注释分析确定的生物网络和丰富的基因本体通路。基于生物过程的共享候选疾病基因及其上游调控因子富集网络,使用ClueGO Cytoscape插件。超几何(右手)富集分布检验,p值显著性水平≤0.05,然后根据显著性最高选择术语和领先术语组进行Bonferroni调整。节点的大小和颜色越深,说明富集的意义越大。
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
哈姆迪,Y.,布杰马,M.,本·瑞卡亚,M.。et al。家族特异性乳腺癌遗传易感性:突尼斯全外显子组乳腺癌病例测序结果。翻译医学杂志16, 158(2018)。https://doi.org/10.1186/s12967-018-1504-9
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12967-018-1504-9
关键字
- 乳腺癌
- 外显子组测序
- 家庭特定易感性
- 非BRCA突尼斯家庭