摘要
背景
骨骼肌纤维类型分布对人体健康、肌肉功能和表现有影响。这些知识是通过劳动密集型和昂贵的方法收集的,这限制了这些研究。在这里,我们提出了一种基于肌肉组织RNA测序数据(totRNAseq)的方法,以估计冷冻人体样本中骨骼肌纤维类型的分布,从而允许更多数量的个体进行测试。
方法
以单核RNA测序(snRNAseq)数据为参考,将聚类基因标记的基因表达平均,然后将其应用于totRNAseq数据,并通过线性矩阵分解推断肌纤维核类型,得到聚类表达签名。然后将这一估计值与两个独立队列中62个肌肉样本的atp酶染色测量的纤维类型分布或肌球蛋白重链蛋白异构体分布进行比较(n= 39和22)。
结果
基于测序的方法与其他两种方法的相关性为ratpa= 0.44 [0.13-0.67], [95% CI], r肌凝蛋白= 0.83[0.61-0.93],与p= 5.70 × 103和2.00 × 106,分别。纤维类型组成的反褶积推理即使在非常低的totRNAseq测序深度下也是准确的,即平均可低至约10,000对末端reads。
结论
这种新方法(https://github.com/OlaHanssonLab/PredictFiberType)因此,可以使用totRNAseq以一种成本和劳动效率高的方式测量大量样品的纤维类型分布。在大型有力的研究中,研究纤维类型分布与健康结果之间的关系现在是可行的。
简介
我们的身体有大约30-40%的骨骼肌,它是三种肌肉中数量最多的,其他两种是平滑肌和心脏肌。骨骼肌由不同类型的纤维(即肌肉细胞类型)组成,这些类型的相对比例因肌肉、肌肉内的位置、个体和个体的性别而异[1,2,3.,4].不同类型纤维的氧化和糖酵解潜能以及收缩特性差异很大,富含线粒体的慢缩纤维(I型)具有更高的氧化能力,而快缩纤维(IIa型和IIx型)具有更高的糖酵解能力[1].随着年龄的增长,纤维的比例也会发生变化,II型纤维最容易受到肌肉减少症的影响。5].锻炼骨骼肌是血糖和血脂分解代谢的主要部位,该组织的代谢特征影响着优秀运动员的成绩[6,7]和个人的疾病倾向,例如葡萄糖和脂质运输到肌细胞的障碍可促进糖尿病和动脉粥样硬化性血管疾病[8,9,10].虽然环境因素,如训练或久坐行为和衰老,导致骨骼肌中毛细血管密度和纤维组成的适应性改变(IIa型与IIx型的增加),但这些特征部分受遗传控制[11,12].然而,I型纤维向II型纤维以及II型纤维向I型纤维转变的程度仍不确定[6].肌肉组织通常根据主要的肌球蛋白重链(MyHC)异构体进行分类,但这是一种简化的分类,忽略了骨骼肌中表达的大量其他蛋白质[2,13].因此,估计和比较人类肌肉纤维类型并不简单,例如,由于存在很大的异质性和方法的局限性,在冷冻的死后样本中甚至更具挑战性。
除了来自不同类型肌肉纤维的细胞外,活检样本还包含成纤维细胞、内皮细胞、脂肪细胞、平滑肌细胞和神经元相关雪旺细胞等细胞。骨骼肌的基因表达分析由于其组织的复杂性而变得困难。尽管来自肌肉活检的转录组测序仍然可以提供个体之间功能差异的视角,但来自分离细胞的靶向RNA测序提供了揭示不同细胞群特异性差异的机会[14].先前研究骨骼肌的研究已将单细胞RNA测序(scRNAseq)应用于提取的单核细胞[15,16,17,18,19,20.].例如,这些研究描述了不同细胞类型的复杂图景,例如,两种不同的肌肉祖细胞群[15],提供有关肌肉再生的详细知识[19]和肌肉疾病的病因[20.].肌肉和其他固体组织中单细胞基因组研究的一个挑战是捕获难以在悬浮液中分离的细胞类型[21],例如,在上述scRNAseq研究中,肌肉纤维尚未测序。然而,一些研究分离并测序了单个核,包括小鼠多核初级肌纤维的核[22,23,24],例如,发现肌纤维类型主要表达MyHC蛋白的慢速异构体或快速异构体之一,而只有一小部分杂交纤维可以表达一种以上的MyHC [22].然而,目前还没有关于人类多核原代肌纤维的研究。
结果
在这里,我们提出了一种仅使用肌肉组织RNA测序(totRNAseq)数据估计骨骼肌纤维类型比例的方法,这些数据可用于冷冻样本。该方法基于一个人类个体的snRNAseq信息,然后在人类骨骼肌的两个独立的更大的totRNAseq数据集(肌肉卫星细胞研究,MSAT,n= 39,以及JMS的Juntendo肌肉研究,n= 23)。
一种基于单核RNA测序的新型肌纤维类型预测模型
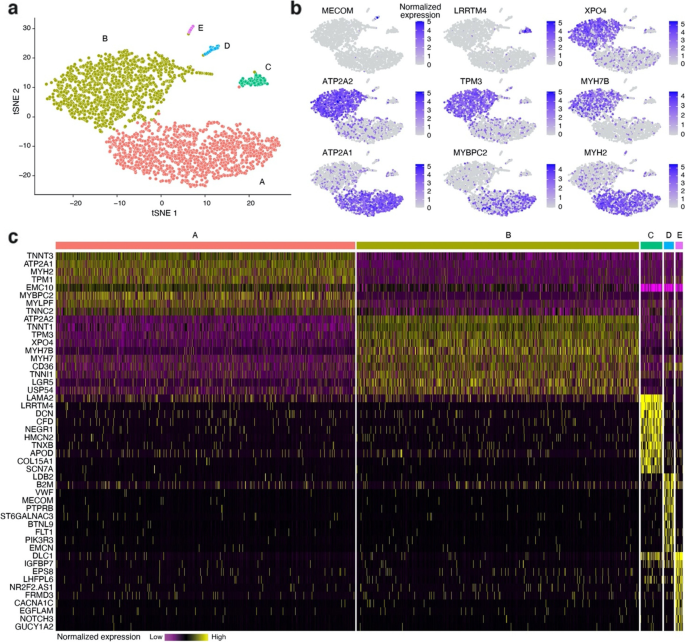
过滤及品质控制后(请参阅“材料与方法), snRNAseq数据集包含2699个核,有22084个表达基因。平均每个细胞核发现约500个基因(补充图。1).利用基于鲁汶模块化优化的图聚类方法,确定了5个主要的核簇[25].为了使核群可视化,应用了t分布随机邻居嵌入(tSNE)非线性降维(图2)。1a).可以清晰地观察到I型(B簇)和II型(a簇)纤维核的分离,即I型和II型纤维的基因标记(如:ATP2A2,TPM3,MYH7B与ATP2A1,MYBPC2,MYH2)在不同聚类中表现出不同的表达模式(图;1b).簇D可能代表内皮细胞,即富集了基因标记,如LDB2,VWF,BTNL9,FLT1.C和E簇的身份尚不清楚,但可能分别代表成纤维细胞周细胞。5个聚类的48个基因标记的完整列表如图所示。1c。
利用冷冻样本的总RNA测序进行肌纤维分型
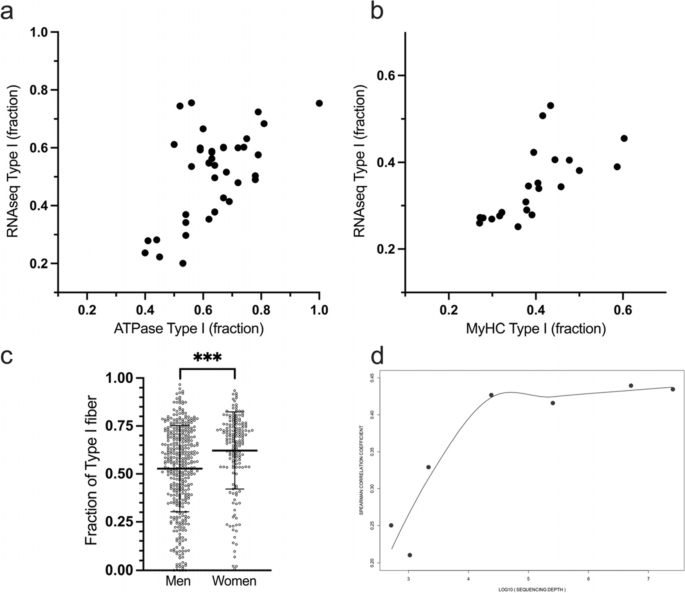
为了评估从totRNAseq数据中估计肌肉样本中I型与II型纤维核比例的有效性,进行了反褶积分析[26]在由39名人类受试者组成的数据集中进行,即MSAT研究(表2)1).简而言之,以snRNAseq数据为参考,通过将聚类基因标记的基因表达量平均,然后通过线性矩阵分解推断肌纤维核类型,将其应用于totRNA MSAT数据集,从而产生聚类表达签名[26],然后最后将这一估计值与同一肌肉样本的atp酶染色测量的纤维类型比例进行比较。来自totRNAseq数据的肌纤维核类型估计比例与来自atp酶染色的肌纤维类型之间的相关性为r= 0.44 [0.13-0.67], [95% CI], p斯皮尔曼= 5.70 × 103,n= 39(图2a).同样的反褶积分析在第二个数据集上进行,该数据集由来自日本的23个人类受试者组成,即JMS(表2)2).从totRNAseq数据估计的肌纤维核类型比例与在JMS中通过MyHC蛋白异构体分布测量的肌纤维类型之间的相关性是r= 0.83 [0.61-0.93], [95% CI], p斯皮尔曼= 2.00 × 106,n= 22(图2b). 1例因测序质量低被排除。为了进一步验证该方法,骨骼肌totRNAseq数据从基因型组织表达项目(n= 569),并测试了男性和女性之间纤维类型分布的差异。正如预期的那样,我们观察到与男性相比,女性中I型纤维的比例更大(68%[54-76%]比56%[39-71%],中位数[95% CI],pMann-Whitney= 3.1 × 107,n女性= 171,和n男人= 398,图2c)。
一个totRNAseq数据估计的肌纤维核类型比例与MSAT研究中atp酶染色的肌纤维类型之间的相关性r= 0.44 [0.13-0.67], [95% CI]p斯皮尔曼= 5.70 × 103,n= 39。b来自totRNAseq数据的肌纤维核类型估计比例与JMS研究中来自肌球蛋白重链分布的肌纤维类型之间的相关性,r= 0.83 [0.61-0.93], [95% CI]p斯皮尔曼= 2.00 × 106,n= 22。c从基因型-组织表达项目的totRNAseq数据估计肌纤维核类型的比例(n合计= 569)。女性I型纤维核的比例高于男性,分别为68%[54-76%]和56%[39-71%],中位数[95% CI],pMann-Whitney= 3.1 × 107,n女性= 171,和n男人= 398。d不同测序深度下,totRNAseq数据中估计的肌纤维核类型比例与MSAT研究中atp酶染色的肌纤维类型之间的相关性
为了测试将该方法应用于大量样本的纤维分型的可能性,我们估计了totRNAseq数据所需的最小测序深度,以准确推断骨骼肌纤维类型组成。在MSAT研究的39个样本的totRNAseq数据中,越来越多的随机选择的reads被移除。通过该方法,对3500万对端(成对端,PE) reads的初始平均测序深度进行了“下采样”。2d)和均方误差(补充图。2),计算I型纤维的ATPase和totRNAseq之间的预测分数。即使在非常低的测序深度,即平均~ 10,000个PE reads,纤维类型组成反褶褶推断的准确性也与3500万PE reads水平相似(图2)。2d和补充图。2).
讨论
在这里,我们提出了一种从冷冻样本中估计骨骼肌纤维类型比例的方法,允许以标准化、成本和劳动效率的方式测量更多的样本。骨骼肌纤维类型的分布是体能表现的决定因素[27,28,29,30.]和整体健康状况[31,32,33,34,35]在人类中具有高度的遗传性[11,12].例如,氧化慢缩I型纤维比例的降低与糖尿病肌肉中胰岛素敏感性的降低有关[31,32,33,34]和肌肉萎缩,例如,与年龄相关的肌肉减少症以特定纤维类型的方式进展[35].最近也有研究表明,运动训练后的骨骼肌反应和恢复取决于纤维类型组成,因此在制定个性化训练建议时,骨骼肌反应和恢复是一个需要考虑的重要因素[36,37,38].
然而,上述许多与纤维类型分布的关联都是基于低功率的研究。限制因素之一是确定纤维类型分布的方法是劳动密集型的,因此相对昂贵。这里提出的方法允许使用totRNAseq以标准化和相对快速的方式分析大量的样本。用这种新方法,可以估计I型和II型纤维的分布,但不能区分IIa型和IIx型纤维。然而,这只需要少量的肌肉组织(~ 5-10毫克)就可以实现,例如,通过微创显微活检技术取样[39].纤维类型组成的反褶积推理即使在非常低的测序深度下也很准确,即平均可达~ 10,000个PE读数。这意味着一个浅覆盖的trnaseq实验(或靶向RNAseq)将足以以低成本(< 1美元/样本)准确估计骨骼肌纤维类型组成。因此,这种新方法可以测量更多样本的纤维类型分布,并使研究纤维类型分布与健康之间的联系成为可能。它还可以用于估计totRNAseq数据公共存储库中的纤维类型分布(例如,基因型-组织表达项目),并在纤维类型关联的硅晶片分析中执行。综上所述,totRNAseq可以有效地估计冷冻样品的骨骼肌纤维类型分布。
材料与方法
snRNAseq数据生成
为了从冷冻组织中分离核,所有以下步骤都在带有预冷缓冲液的冰上进行,离心步骤在4°C下进行。在0.1% NP40存在的冰冷均质缓冲液(0.32 M蔗糖,3 mM CaCl2, 2 mM醋酸镁,0.1 mM EDTA, 1 mM DTT, 10 mg/ml BSA, 10 mM Tris-HCL,蛋白酶抑制剂(Sigma-Aldrich))中进行均匀均质,破坏组织并释放细胞核。核悬液依次通过40 μm、30 μm和20 μm细胞过滤器(Miltenyi Biotec),并在1000 g下离心10分钟。颗粒在1% RNase-free BSA的PBS中重悬,并使用DAPI (1:1000, BD Pharmingen)染色。使用dapi阳性事件群对完整的单个核进行批量排序,以单细胞排序精度,并使用100微米喷嘴(BD FACS AriaIII)将其放入PBS/1% BSA中。计数核数并装载在10 ×铬微流控芯片上,目的是通过分选获得尽可能多的目标核数。使用10 ×基因组学单细胞3 ' kit v.2进行单核实验,将细胞核与条形码标记的珠子一起封装,生成并扩增cDNA并生成测序文库。每个池库使用10 × Genomics提供的i7条形码进行条形码编码。cDNA和测序文库的质量和数量采用安捷伦的高灵敏度DNA检测。在Illumina的HiSeq平台上以150-bp的配对端模式对文库进行池化和测序。
snRNAseq数据处理与分析
后处理管道Cell Ranger (https://support.10xgenomics.com/single-cell-gene-expression/software/)用于解复用、比对、过滤、条形码和唯一分子标识符(UMI)计数。管道生成FASTQ和BAM格式的文件,以及UMI计数矩阵。我们使用Seurat工作流对snRNAseq基因表达数据进行进一步的质量控制和下游分析。初始数据集包含2937个核和22336个表达基因。来自线粒体和核糖体基因的细胞核比例高的细胞核被去除。接下来,在至少一个细胞核中选择至少一个UMI计数的基因。在这些步骤之后,2699个细胞核和22084个基因被纳入下游分析。我们使用Seurat的LogNormalize方法对基因表达数据进行归一化,并在进行主成分分析之前对计数值进行标准化(补充图)。3.).JackStraw程序[40]作为去噪步骤,以选择最优的主成分个数(PCs),即保留3个主成分供进一步下游分析,可视为识别snRNAseq数据的真实内在维数。此外,使用在细胞周期中被注释为功能的基因列表来研究预测具有增殖能力的细胞数量。41].绝大多数细胞未被检测到增殖,循环细胞没有形成任何单独的簇(补充图。4a - b)。基于Louvain模块化优化的图聚类[25],分辨率参数为0.1,用于检测不同核居群之间的边界,采用tSNE非线性降维,根据k-nearest neighbors rule of thumb,取困惑度50为核数的平方根,用于核居群的可视化(图。1a). snRNAseq数据上传到Gene Expression Omnibus (GEO),登录号为GSE190489。
反褶积分析
反褶积分析[26以snRNAseq数据为参考,执行。为此目的,snRNAseq数据被用于产生聚类签名,这是每个聚类细胞间平均的标记基因表达。在此之后,通过线性矩阵分解推断大量RNAseq样本中各类型骨骼肌纤维的基因表达[42].采用DeconRNASeq R/Bioconductor包进行反褶积分析,结果如图所示。2a、b。
研究对象
MSAT队列:39名瑞典男性受试者(表2)1)通过社交媒体广告和当地的自行车俱乐部参与了这项研究。纳入标准如下:(1)男性,(2)健康,未服用任何药物,(3)年龄在20至55岁之间。在受试者给出书面知情同意之前,他们会得到关于实验过程的口头和书面信息。每个参与者在不同的时间点进行了三次访问。所有受试者均完成了三次随访。第一次访问包括定期的医生检查血液样本和测量人体特征。第二次访问包括,一夜禁食后,温盖特测试,随后肌肉活检和VO2马克斯是在第三次和最后一次来访时测量的。该研究得到了隆德大学当地伦理委员会的批准(Dnr 2015/593)。用于测定峰值无氧功率(Wingate)和VO2马克斯,受试者被要求在每次测试前48小时只进行简单的训练。为确定峰值无氧功率,需进行30秒全力温盖特试验[43]在循环功率计(Monark Peak power)上进行。在测试前,进行5分钟~ 150w的低强度热身,并指示每分钟进行5秒的高节奏演练。测试开始时,受试者尽可能快地踩踏板。当节奏达到120 rpm时,制动阻力相当于0.7 N × m × kg−1应用于飞轮,并在30秒内保持不变。在整个测试过程中,受试者都被要求坐着。全程给予强烈的口头鼓励,以确保提供最大的努力。对衰竭进行增量测试以确定VO2马克斯.试验以3 W × kg的循环3 min开始−1(四舍五入到最接近10w),然后每2分钟增加35w,直到自愿耗尽或无法保持≥60rpm。自始至终都给予强烈的口头鼓励。签证官2使用带有混合室的Oxycon Pro (Jaeger GmbH,德国)进行测量,采样时间为30秒。气体传感器在每次测试前都根据供应商的说明进行校准。最大摄氧量被确定为在任何30秒期间的运动中获得的两个最高值的平均值。
Juntendo队列:23名日本受试者(表2)被招募来检测日本人群中RNA表达谱和肌肉纤维组成之间的关系。所有受试者在纳入研究前均签署知情同意书。研究方案由俊藤多大学伦理委员会批准,并按照《赫尔辛基宣言》执行。在该队列中未进行额外的试验。
肌肉活组织检查和组织学检查
在MSAT队列(表1),肌肉活组织检查取自股外侧肌肌肉在无菌条件下进行局部麻醉(1%利多卡因),使用5毫米Bergström针并冷冻在液氮中。活检在温盖特试验后5分钟内进行。连环教派。(10 μm)用低温恒温器在−20℃下切割。肌原纤维atp酶组织化学在pH值4.4、4.6和10.3的预孵育下进行,以确定纤维类型[44];纤维类型的比例(即I型、IIa型或IIx型)计算为每种类型的纤维数量除以截面中的纤维总数。计算机图像分析使用图像分析设备(BioPix IQ 2.0.16软件,BioPix AB,瑞典)。
在Juntendo队列(表2),肌肉活组织检查取自股外侧肌在无菌条件下,局部麻醉(1%利多卡因),使用处置针活检仪(Max Core;C. R. Bard, Covington, GA)。在超声成像下,从每个受试者双腿髌骨上方约15厘米处采集活检(Noblus;Aloka, Tokyo, Japan),并尽可能避免皮下脂肪和筋膜下和肌腱部分的包涵。此外,从活检样本中去除任何可见的非肌肉组织(例如脂肪组织)。样品立即在液氮中冷冻,并在- 80℃保存,直到进一步分析。肌球蛋白重链(MyHC)蛋白异构体被评估为肌肉纤维组成的标记物。冷冻肌肉样品在冰冷的裂解缓冲液[50 mM HEPES (pH 7.4), 10 mM EDTA, 4 mM EGTA, 50 mM β-甘油磷酸,25 mM NaF, 5 mM Na]中均质3.签证官4],含有磷酸酶抑制剂(PhosSTOP片;Roche Diagnostics, Indianapolis, IN)和蛋白酶抑制剂(完全片剂;罗氏诊断)。得到的裂解物在4℃下,以10000 g离心10分钟。将离心后得到的不溶性颗粒悬浮在足够体积的SDS样品缓冲液[30%甘油,5% β-巯基乙醇,2.3% SDS, 0.05%溴酚蓝和62.5 mM Tris-HCl (pH 6.8)]中,在95℃下煮沸5分钟。根据Kakigi等人的研究,MyHC组成由甘油SDS- page测定。[45].简单地说,蛋白质样品通过甘油SDS-PAGE[堆叠凝胶:4%丙烯酰胺,34.7%甘油,125 mM Tris-HCl (pH 6.8);分离凝胶:8%丙烯酰胺,33.3%甘油,375 mM Tris-HCl (pH 8.3)]。电泳在60 V下开始,8℃下堆叠凝胶。当跟踪染料完全进入分离凝胶后,在8℃下运行18 h,电压设置为150 V。分离后,用考马斯亮蓝染色(Biosafe G250;Bio-Rad实验室,赫拉克勒斯,加州),并反复用水冲洗。使用校准密度计扫描每个凝胶(ChemiDoc触摸成像系统;Bio-Rad Laboratories), MyHC-I、MyHC-IIa和MyHC-IIx的相对比例使用校准密度计(ChemiDoc Touch Imaging System)和分析软件(Image Laboratory软件版本5.2.1;Bio-Rad实验室)。
RNA提取和totRNAseq分析
在MSAT队列中,使用TissueLyser II (Qiagen)和miRneasy Mini Kit (Qiagen)从25 ~ 30 mg肌肉活检中提取RNA。RNA浓度使用NanoDrop ND-1000分光光度计(A260/A280 > 1.8和A260/A230 > 1.0 (NanoDrop Technologies, Wilmington, DE, USA)测定。RNA完整性使用2200 TapeStation仪(Agilent Technologies, CA, USA)进行验证,所有样品的平均RNA完整性数(RIN)均高于8。在Juntendo队列中,使用Micro Smash MS-100R (Tomy Seiko, Japan)以3000转/分两次,在2°C下,用5.0 mm氧化锆珠粉碎冷冻肌肉样本,持续15秒。根据制造商的协议,使用TRIzol®试剂(Thermo Fisher Scientific, Waltham, MA, USA)从肌肉样本中提取总RNA。RNA浓度和纯度使用NanoDrop 8000 UV-Vis分光光度计(赛默飞世尔科学公司,威尔明顿,DE,美国)检查。RNA完整性使用2200 TapeStation仪(Agilent Technologies, CA, US)进行验证,所有样品的平均RNA完整性数(RIN)均高于8。
所有来自MSAT和Juntendo队列的样本均在隆德大学使用800 ng输入RNA进行测序。文库制备使用TruSeq总RNA文库准备试剂盒和riboo - zero Human/Mouse/Rat Set A (Illumina), 75 bp的配对端测序使用NextSeq®500/550 High Output Kit v2(150个周期)(Illumina)在NextSeq仪器上进行。测序质量采用fastQC v0.11.9 (http://www.bioinformatics.babraham.ac.uk/projects/fastqc)及multiQC [46v1.9。基因表达用Salmon [47中的例子)。是processing v1.2.1外显子表达量通过STAR [48[v2.7.6 and counting with featurets]49v2.0.1(选项:-p -f -C -O)。我们使用了GRCh38 ensemble v77 [50作为参考基因组。我们使用Seqtk工具(https://github.com/lh3/seqtk)对MSAT数据进行逐步随机下采样,并应用Salmon对下采样的RNAseq数据进行基因表达量化。每次下采样迭代的定量基因表达用TMM归一化[51],并使用上述snRNAseq数据中为慢和快抽搐人类聚类识别的基因标记进行反褶积分析。我们计算了每次下采样迭代的斯皮尔曼相关系数和预测与真实纤维类型组成之间的均方差。
数据和材料的可用性
使用这种新方法所需的所有脚本都可以在https://github.com/OlaHanssonLab/PredictFiberType.支持本文结论的数据集可在Gene Expression Omnibus存储库(GSE190489)中获得https://www.ncbi.nlm.nih.gov/geo.totRNAseq数据可在欧洲基因组-表型组档案中获得,EGAS00001006362 - Juntendo肌肉研究(JMS), EGAS00001006363 -肌肉卫星细胞研究(MSAT)https://ega-archive.org.
缩写
- totRNAseq:
-
肌肉组织RNA测序数据
- scRNAseq:
-
单细胞RNA测序
- snRNAseq:
-
单核RNA测序
- I型:
-
Slow-twitch纤维
- II型:
-
增大纤维
- MyHC:
-
肌球蛋白重链
- MSAT:
-
肌肉卫星细胞研究
- JMS:
-
Juntendo肌肉研究
- tSNE:
-
t分布随机邻居嵌入
- ATP2A2:
-
atp酶肌浆/内质网Ca2 +转运2
- TPM3:
-
原肌球蛋白3
- MYH7B:
-
肌球蛋白重链7B
- ATP2A1:
-
atp酶肌浆/内质网Ca2 +转运1
- TNNT3:
-
肌钙蛋白T3,快速骨骼型
- MYH2:
-
肌球蛋白重链2
- TTN:
-
肌
- MEG3:
-
母性表达3
- 体育:
-
Paired-end
- UMI:
-
唯一的分子标识符
- 电脑:
-
主成分
- 主成分分析:
-
主成分分析
- 地理:
-
基因表达综合
- RIN:
-
RNA完整性数
- EGA:
-
欧洲基因组-表型档案
参考文献
哺乳动物骨骼肌中的肌球蛋白异构体。应用物理学报。1994;1985(3):344 - 344。
哺乳动物骨骼肌中的纤维类型。物理学Rev. 2011; 91:1447-531。https://doi.org/10.1152/physrev.00031.2010.
Saltin B, Henriksson J, Nygaard E, Andersen P, Jansson E.久坐男性和耐力跑者骨骼肌纤维类型和代谢潜能。南京大学学报(自然科学版),2001;
Simoneau JA, Bouchard C.人类骨骼肌纤维类型比例和酶活性的变化。中华医学杂志。1989;257:E567-572。
Snijders T, Verdijk LB, van Loon LJ。肌肉减少症和运动训练对骨骼肌卫星细胞的影响。老化决议Rev. 2009; 8:328-38。https://doi.org/10.1016/j.arr.2009.05.003.
威尔逊JM,等。耐力、力量和力量训练对肌纤维类型转换的影响。J Strength Cond res 2012; 26:1724-9。https://doi.org/10.1519/JSC.0b013e318234eb6f.
Harridge SD,等。人类的全肌肉和单纤维收缩特性和肌球蛋白重链异构体。Pflugers Arch. 1996; 432:913-20。
Tanner CJ,等。肌肉纤维类型与肥胖和体重减轻有关。中华内分泌杂志,2002;28(2):397 - 397。https://doi.org/10.1152/ajpendo.00416.2001.
Mogensen M,等。2型糖尿病患者骨骼肌线粒体呼吸降低。糖尿病。2007;56:1592-9。https://doi.org/10.2337/db06-0981.
Simoneau JA, Colberg SR, Thaete FL, Kelley DE.骨骼肌糖酵解和氧化酶能力是肥胖女性胰岛素敏感性和肌肉成分的决定因素。王志强,1995;9:379 - 379。
Komi PV,等。男女同卵和异卵双胞胎的骨骼肌纤维和肌肉酶活性。物理学报。1977;100:385-92。
张志刚,张志刚。人类骨骼肌纤维类型比例的遗传决定。法斯伯j . 1995; 9:1091-5。
Schiaffino S, Reggiani C, Murgia M.基于质谱的骨骼肌纤维类型多样性的研究。《组织病理学杂志》2020;35:39 - 46。https://doi.org/10.14670/HH-18-170.
邓强。单细胞RNA测序:技术进步和生物学应用。Mol Aspects Med. 2018; 59:36-46。https://doi.org/10.1016/j.mam.2017.07.003.
De Micheli AJ, Spector JA, Elemento O, Cosgrove BD.人类骨骼肌组织的参考单细胞转录组图谱揭示了分叉的肌肉干细胞群。骨骼肌,2020;10:19。https://doi.org/10.1186/s13395-020-00236-3.
Jensen JB,等。从人骨骼肌活检中分离和鉴定肌肉干细胞、纤维成脂肪祖细胞和巨噬细胞。Am J Physiol Cell Physiol. 2021。https://doi.org/10.1152/ajpcell.00127.2021.
van den Heuvel A等。单细胞RNA测序在面肩肱肌营养不良病病因学及发展中的应用。Hum Mol Genet 2019; 28:1064-75。https://doi.org/10.1093/hmg/ddy400.
Barruet E,等人。通过单细胞RNA测序鉴定功能异质的人类卫星细胞。Elife。2020;9。https://doi.org/10.7554/eLife.51576.
人类骨骼肌图谱确定了干细胞和祖细胞的发展轨迹,并从人类多能干细胞。细胞干细胞。2020;27:58 - 176.e110。https://doi.org/10.1016/j.stem.2020.04.017.
坎普斯J等。间质细胞重塑促进营养不良肌肉的异常脂肪生成。Cell rep 2020;31:107597。https://doi.org/10.1016/j.celrep.2020.107597.
吴海涛,吴海涛,王晓明,王晓明。成人肾脏单核RNA测序的优势:肾脏纤维化中罕见的细胞类型和新细胞状态。中国生物医学杂志。2019;30:23-32。https://doi.org/10.1681/ASN.2018090912.
Dos Santos M,等。单核RNA-seq和FISH在哺乳动物肌纤维中鉴定协同转录活性。Nat Commun. 2020;11:5102。https://doi.org/10.1038/s41467-020-18789-8.
Kim M,等。单核转录组揭示合胞骨骼肌细胞的功能区隔化。Nat Commun. 2020;11:6375。https://doi.org/10.1038/s41467-020-20064-9.
Petrany MJ,等。单核RNA-seq鉴定多核骨骼肌纤维的转录异质性。Nat Commun. 2020;11:6374。https://doi.org/10.1038/s41467-020-20063-w.
Blondel VD, Guillaume JL, Hendrickx JM, de Kerchove C, Lambiotte R.随机网络的局部领导者。物理学报。2008;47:036114。https://doi.org/10.1103/PhysRevE.77.036114.
龚T, Szustakowski JD。DeconRNASeq:基于mRNA-Seq数据的异构组织样本反褶积的统计框架。生物信息学。2013;29:1083-5。https://doi.org/10.1093/bioinformatics/btt090.
高志平,王志强,王志强。优秀长跑运动员肌肉纤维组成与酶活性的关系。医学体育。1976;8:96-100。
年轻竞技长跑运动员的单肌纤维收缩特性。应用物理学报。2008;1985(10):629 - 636。https://doi.org/10.1152/japplphysiol.00995.2007.
维德里克JJ,特拉普SW,科斯蒂尔DL,菲茨RH。来自精英跑步大师和久坐男性的单个肌肉纤维的力-速度和力-功率特性。中华医学杂志。1996;21:C676-683。https://doi.org/10.1152/ajpcell.1996.271.2.C676.
Bellinger P,等。1500米配速和极限计时赛最后一圈速度的决定因素。欧洲J应用物理。2020。https://doi.org/10.1007/s00421-020-04543-x.
Oberbach A等人。2型糖尿病患者骨骼肌中纤维分布及纤维特异性糖酵解和氧化酶活性的改变。糖尿病护理。2006;29:895-900。
Lillioja S,等。骨骼肌毛细血管密度和纤维类型可能是人体内胰岛素抵抗的决定因素。《金融与投资》,1987;30(4):344 - 344。https://doi.org/10.1172/JCI113088.
Henriksen EJ,等。大鼠骨骼肌葡萄糖转运蛋白含量及葡萄糖转运能力。中华医学杂志。1990;259:E593-598。https://doi.org/10.1152/ajpendo.1990.259.4.E593.
Daugaard JR等人。人体骨骼肌中GLUT4纤维类型特异性表达:运动训练的影响。糖尿病。2000;49:1092-5。https://doi.org/10.2337/diabetes.49.7.1092.
Ciciliot S, Rossi AC, Dyar KA, Blaauw B, Schiaffino S.肌肉消瘦的肌肉类型和纤维类型特异性。中国生物化学杂志,2013;https://doi.org/10.1016/j.biocel.2013.05.016.
Deshmukh AS,等。冻干人体肌肉活检的深层肌肉蛋白质组学分析揭示了纤维类型对运动训练的适应性。Nat Commun. 2021;12:304。https://doi.org/10.1038/s41467-020-20556-8.
Bellinger P,等。肌肉纤维类型与过度训练反应中过度伸展的发生率相关。应用物理学报。2020;1985(12):823-36。https://doi.org/10.1152/japplphysiol.00314.2020.
Lievens E, Klass M, Bex T, Derave W.肌肉纤维类型实质上影响从高强度运动恢复的时间。应用物理学报。2020;1985(12):648-59。https://doi.org/10.1152/japplphysiol.00636.2019.
Hayot M,等。骨骼肌显微活检:一项微创技术的验证研究。中国科学院学报,2005;25:431 - 432。https://doi.org/10.1183/09031936.05.00053404.
钟国忠,Storey JD。在高维数据中驱动系统变异的变量的统计意义。生物信息学。2015;31:545-54。https://doi.org/10.1093/bioinformatics/btu674.
Kowalczyk MS,等。单细胞RNA-seq揭示了造血干细胞衰老过程中细胞周期和分化程序的变化。Genome Res. 2015; 25:19 60 - 72。https://doi.org/10.1101/gr.192237.115.
装饰蛋白在癌症中的多重作用。Sofeu Feugaing, d.d., Götte, M. & Viola, M. More than matrix。中国生物医学工程学报(英文版);2013;https://doi.org/10.1016/j.ejcb.2012.08.004.
Bar-Or O.温盖特厌氧试验。研究方法、信度和效度的更新。体育医学1987;4:381-94。https://doi.org/10.2165/00007256-198704060-00001.
布鲁克MH,凯撒KK。三、“肌球蛋白腺苷三磷酸酶”系统:其pH不稳定性和巯基依赖性的性质。组织化学杂志。1970;18:670-2。
Kakigi R,等。热应激增强人体骨骼肌抗阻运动后mTOR信号通路。中国生物医学工程学报(自然科学版),2011;https://doi.org/10.1007/s12576-010-0130-y.
Ewels P, Magnusson M, Lundin S, Käller M. MultiQC:在一份报告中总结多种工具和样品的分析结果。生物信息学。2016;32:3047-8。https://doi.org/10.1093/bioinformatics/btw354.
Patro R, Duggal G, Love MI, Irizarry RA, Kingsford C. Salmon提供了快速和偏见意识的转录表达量化。2017; 14:417-9。https://doi.org/10.1038/nmeth.4197.
Dobin A,等。STAR:超快通用RNA-seq对准器。生物信息学。2013;29:15-21。https://doi.org/10.1093/bioinformatics/bts635.
廖勇,史伟。featurets:一种高效的通用程序,用于分配序列读取基因组特征。生物信息学。2014;30:923-30。https://doi.org/10.1093/bioinformatics/btt656.
耶茨AD,等。运用2020年。Nucleic Acids res 2020;48: D682-8。https://doi.org/10.1093/nar/gkz966.
Robinson MD, Oshlack A. RNA-seq数据差异表达分析的标度归一化方法。中国生物工程学报。2010;11:R25。https://doi.org/10.1186/gb-2010-11-3-r25.
确认
作者要感谢生命科学实验室、国家基因组学基础设施、NGI和瑞典国家计算基础设施(SNIC)在UPPMAX的支持,部分资金由瑞典研究委员会通过no。2018-05973为大规模并行测序和计算基础设施提供帮助。
资金
由隆德大学提供的开放获取资金。这项工作得到以下资助:克努特和爱丽丝·瓦伦堡基金会的设备,瑞典研究委员会项目拨款2018-02635,Crafoord基金会,诺和诺德基金会,Påhlsson基金会,糖尿病健康,瑞典糖尿病基金会,Hjelt基金会,JSPS kakeni, Dnr 16KK0188,以及Juntendo大学健康和运动科学与医学研究所。LUDC-IRC:瑞典战略研究基金会,Dnr IRC15-0067; EXODIAB:瑞典研究委员会,战略研究领域,Dnr 2009-1039。NO和BN由克努特和爱丽丝·瓦伦堡基金会资助,作为瑞典国家生物信息基础设施的一部分。资助机构在研究设计、数据收集、分析和解释以及撰写手稿方面没有影响力,也没有以任何其他方式参与。
作者信息
作者及隶属关系
贡献
NO、SP、BN、OH撰写主要手稿文本,NO、GC、BT、DK、OH准备图表。OE, EMM, NF和OH组成了表格。MS, KS和BM进行实验。NO、GC、HP、DK、BT、OH进行生物信息学分析。OE、EMM、ML、HK、RK、HN、KFE、NF、SP和OH进行人体研究和/或收集样本。作者审阅并批准了手稿。
相应的作者
道德声明
伦理批准并同意参与
MSAT研究得到了隆德大学当地伦理委员会的批准(Dnr 2015/593), JMS研究得到了Juntendo大学当地伦理委员会的批准。所有参与者在参与研究前均知情同意。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:图S1。
Seurat snRNAseq工作流质量控制统计。从左到右,第一个图报告了每个细胞表达的基因的数量,第二个图报告了每个细胞UMIs(文库大小)的数量,第三个图显示了每个细胞中映射到线粒体基因的reads的比例。图S2。对3500万对端(PE) reads的初始平均测序深度进行了“下采样”,并计算了I型纤维的ATPase和totRNAseq预测片段之间的均方误差。图S3。主成分分析(PCA)图中来自人类骨骼肌snRNAseq的基因表达由聚类注释着色,见图1A,由对显著主成分数量的Louvain聚类获得(根据Seurat工作流)。图S4。(a)主成分分析(PCA)图,(b)人类骨骼肌snRNAseq基因表达的tSNE图,由Seurat工作流中的CellCycleScoring函数获得的细胞周期注释着色。从PCA和tSNE图中均未观察到基于细胞周期的明显聚类形成。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
Oskolkov, N., Santel, M., Parikh, H.M.et al。来自RNA测序数据的高通量肌纤维分型。骨骼肌12, 16(2022)。https://doi.org/10.1186/s13395-022-00299-4
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s13395-022-00299-4