摘要
在过去的10年里,高密度SNP阵列和DNA重测序已经阐明了许多生物的大部分基因型空间,包括人类、玉米、水稻和拟南芥.对于任何愿意在许多个体中定义和评分表型的研究人员来说,全基因组关联研究(GWAS)提供了一个强大的工具,可以将这一特征重新连接到其潜在的遗传学。在这篇综述中,我们讨论了支撑成功分析或其他方面的生物学和统计学考虑因素。生物学因素的相关性,包括效应大小,样本量,遗传异质性,基因组混淆,连锁不平衡和虚假关联,以及统计工具来解释这些被提出。GWAS可以为后续验证提供对性状结构或候选位点的有价值的首次洞察。
一个物种内的遗传多态性和个体间观察到的表型差异之间的因果关系是基本的生物学兴趣。要预测人类疾病的遗传风险因素和植物的生长速度和产量等农学上的重要性状,需要了解构成表型的特定位点和性状的遗传结构。至少自孟德尔假设存在遗传给下一代的“内部因素”以来,表现型和基因型之间的这种关系一直备受关注。
正向遗传学是对许多基因型不同的个体进行筛选,以寻找感兴趣的表型,这是解决此类问题的一个非常强大的工具。一般来说,筛选的原始遗传差异要么通过诱变获得,要么从自然群体中取样。通过各种映射方法,包括数量性状位点(QTL)映射,任何被识别的表型差异都与潜在的致病位点相关联。从这个角度来看,我们考虑连接基因型-表型图的补充和强大的工具,全基因组关联研究(GWAS)。
在F2群体或重组自交系(RIL)家族中,QTL作图已经证明,并且仍然是识别与给定性状共分离的基因组区域的有力方法。开花时间通路的关键成分拟南芥都被这样解剖过[1- - - - - -3.];的自然变异和QTL作图综述拟南芥参见[4].尽管取得了这样的成功,QTL作图仍然存在两个基本的局限性;只有特定F2杂交亲本之间或RIL群体内部分离的等位基因多样性才能被检测[5],其次,在RIL群体创建过程中发生的重组数量限制了映射分辨率。在建立RIL群体时,通过几代交交可以显著提高分辨率,例如,高级交交RIL [6].与此同时,在建立RILs之前,通过多亲本高级代互交(MAGIC)和多亲本高级代互交(MAGIC),可以在一定程度上增加映射群体内的等位基因多样性拟南芥多家母公司信实集团(AMPRIL) [7,8].
然而,在任何这样的实验室群体中出现的等位基因频率和组合将不同于自然群体[9].对于许多应用来说,这并不构成问题,但它确实混淆了例如上位性的分析,并且仅提供了自然种群中存在的功能多样性的有限观点。
GWAS克服了上面提到的QTL分析的两个主要局限性,但也引入了其他几个缺点(下面讨论)。一般来说,在确定感兴趣的表型后,GWAS可以作为一个基础实验,通过提供对性状遗传结构的深入了解,允许对QTL分析的亲本进行知情选择,并建议突变和转基因的候选者。因此,GWAS通常是QTL定位的补充,当一起进行时,它们可以减轻彼此的局限性[10,11].
GWAS的基本方法是评估每个基因分型标记和在大量个体中得分的感兴趣的表型之间的关联。这种方法是近十年前在人类遗传学领域率先采用的[12],迄今已有近1500例已发表的人类GWAS [13].GWAS现在常规应用于一系列模式生物,包括拟南芥[14]和老鼠[15],以及包括农作物在内的非模式系统[16- - - - - -18]和牛[19].
在这篇综述中,我们将讨论运行GWAS的优点和局限性拟南芥,这些问题通常与其他生物有关。我们考虑样本量和映射面板组成,统计方法来克服遗传混淆和方法来识别和解释复杂的遗传结构。
Self-fertilisation使拟南芥特别适合GWAS
拟南芥thaliana已被证明是进行GWAS的几乎理想的生物体,因为它可以通过持续的自我受精维持为自交系,因此有可能重复显型基因相同的个体。因为超过1300个不同的接入已经对250000个snp进行了基因分型[20.研究者所需要的只是感兴趣性状的几百个系的表型。除了具有里程碑意义的107种表型的概念验证GWAS研究[14],许多其他特征,包括硫代葡萄糖苷水平[21],避影[22]、重金属[23]和耐盐性[24],开花时间[25],以及其他生活史特征[26]已被成功分析。
重要的是,统计方法最近出现了重大改进,包括使用考虑到遗传背景混淆效应的混合模型。这已经通过各种R和Python包实现,或者作为第一个调用点,可以使用在线工具:http://gwas.gmi.oeaw.ac.at[27].这个web应用程序预装了所有常用种质的基因型数据,提供了几种统计选项,并促进了跨已发表性状的元分析。几年前,对几百个个体的全基因组扫描可能需要一天的时间,而在PC或基于web的应用程序上对数十万个snp进行简单的单标记扫描(称为边缘测试,忽略上位性和其他相互作用)只需要几分钟。
遗传结构;大效应的罕见变体,还是小效应的常见变体?
进行GWAS的动机可以是识别给定性状的致病/预测因素,也可以是确定性状遗传结构的各个方面(即贡献基因座的数量以及它们各自对表型的贡献)。一些性状是由少数具有大效应量的位点所支撑的(一个简单的遗传结构),并且高度服从GWAS。这种情况可能在生物选择下的性状中很常见[28].其他特征可能具有更复杂的体系结构,这给GWAS带来了困难。有两种可能,一种是一个性状由许多罕见的变异控制,每个变异对表型都有很大的影响,另一种是许多常见的变异对表型的影响很小。在这两种情况下,致病变异可能聚集在一个或少数基因中,或跨越许多基因(多遗传)。
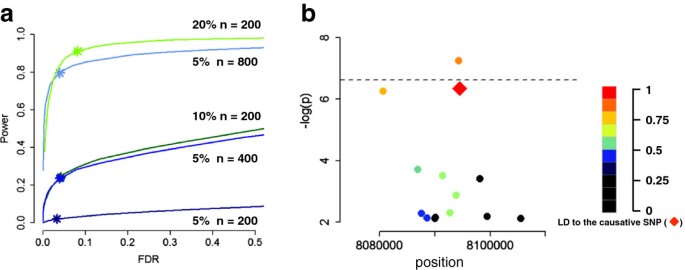
GWAS识别SNP和性状之间真正关联的能力取决于由SNP解释的种群内表型方差(图1a).表型方差由两个等位变异在表型效应(效应量)上的差异程度以及它们在样本中的频率决定。因此,罕见的变异和较小的效应量都是GWAS的问题[29,30.].
此外,罕见变异体与基因组内许多其他非致病罕见变异体存在强烈或完全关联,而不考虑LD衰退,因此单个致病位点可能会导致许多合成关联[31].如果考虑一个人体内的多个私有snp,这一点就能清楚地说明:无论它们的基因组位置如何,它们都是完全相连的。
当变异频率低或效应量小时,如何提高检测有意义关联的能力?以下将讨论几个重要的考虑因素,包括样本量、不完全基因分型、遗传异质性和混杂遗传背景的解释。然而,我们注意到,稀有变异对于特定性状的重要性也可以通过QTL分析来解开,因为稀有变异通过交叉方案被提升到中频。
样本量和遗传异质性:如何选择你的映射面板?
到目前为止,大多数分析是用拟南芥只使用了几百个个体,但对于某些性状,用少于100个的样本就可以获得有意义的结果[14].这表明,所考虑的性状仅由解释大部分表型方差的少数位点所支持。人类的情况看起来不同,通常会发现大量的小效应位点,大多数分析需要数千个个体来检测这些位点。32,33].在其他动物中观察到具有许多小影响的遗传结构[34]或maize [35].异交和自交物种之间是否存在不同遗传结构的总体趋势还有待观察。另一方面,人类疾病状态实际上可能是由许多小效应有害突变驱动的一类特殊性状,而具有中等效应大小的位点已被证明是人眼和肤色等性状的基础[36,37].
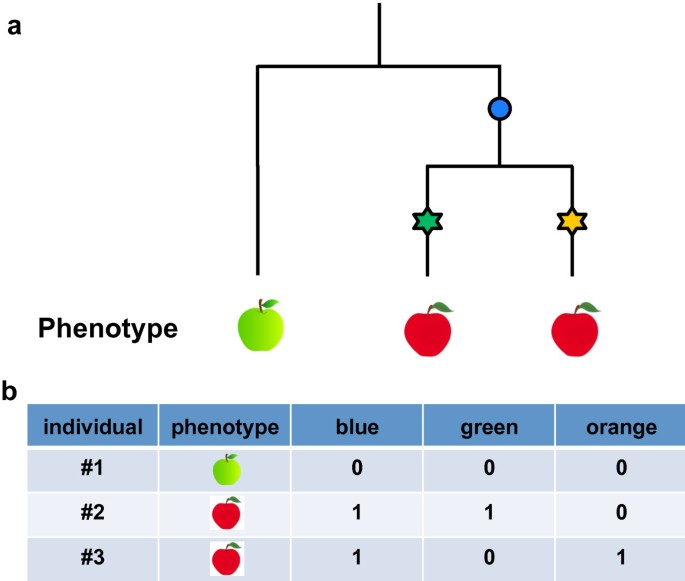
尽管GWAS在拟南芥,许多性状将是多基因的,但效应量较小;因此,增加样本量将提高恢复有意义关联的能力(图1a).鉴于此,如何选择映射面板?一种方法是使用星形设计,包括地理位置遥远的站点。这将最大化样本内的遗传方差[25],但有可能引入遗传异质性。由于包括局部适应在内的原因,从不同地点收集的样本中,不同的变异可能是一种性状的基础[26].这种遗传异质性将降低恢复任一变体的能力,因为它削弱了表型和任何特定变体之间的相关性(图2).
有趣的是,遗传异质性可以导致非致病标记比致病标记更能描述表型[38].考虑两种影响相同表型的近期罕见突变的情况:任何与两种等位基因相关的标记,尽管是非致病的,但与表型的关联比单独的两个单个标记更强(图2).这种合成关联,虽然在某种意义上是假阳性,因为它们不会导致表型,但仍然被证明是有价值的,因为它们是预测表型所需要的全部。
通过在混合模型设置中包括“竞争性”变异作为辅助因子,可以解开遗传异质性的贡献[39].如果事实上,最显著的SNP是唯一的致病标记,将其作为辅助因子应解释该基因组区域贡献的所有表型方差。通过将多个snp拟合到混合模型中,人们可以潜在地解开隐藏在不同GWAS峰值下的最小snp数量。然而,在这样的模型中识别要包含的snp集并不是简单的,并且可能导致过度拟合。
第二种增加样本量的方法是密集抽样显示表型多样性的当地人口。这具有最小化遗传异质性的潜在优势,但缺点是与全球表型多样性相关的变异可能保持在低等位基因频率或完全不存在。
然而,增加样本量可能并不总是能解决罕见变体的体系结构。一种建议的解决方案是将一个区域的几个snp分解成一个单一的指示变量,并将其用作复合基因型[40].人们可以想象这是一种将高度复杂的变异模式简化为仅两个单倍型的方法。不幸的是,如何瓦解snp的理性并非平凡[41- - - - - -43].
不完美的基因型
值得注意的是,在目前可用的基于阵列的SNP数据集中,大多数表型的因果变异不太可能存在。其中有250k个snp拟南芥只代表种群中分离的少数snp。最近的全基因组测序揭示了更高的snp密度拟南芥[44- - - - - -46],在全球样本中约有700万个snp。尽管如此,还是可以检测到显著的关联,因为致病变异(无论是snp还是结构变异)通常与基因分型标记存在足够的连锁不平衡(LD)。在拟南芥, LD一般在5 Kb内衰减50% [45];因此,250k SNP(平均每600bp一个SNP)标记几乎所有的非重复基因组,从而使GWAS [14,47].
在不久的将来,超过1000个基因的“完整”基因组序列将可获得(http://www.1001genomes.org).这组“全部”snp、结构变异、拷贝数和转座因子变异可能包括大多数致病变异。值得注意的是,原则上这些基因型中的任何一种都可以用于GWAS。
包含这样完整的序列真的有帮助吗?无论包含多少变异,数据的LD结构和在内部(有时在之间)snp之间观察到的不寻常的长范围LD拟南芥染色体(46]总是会使从相关的中性标记中分离致病变异变得困难。然而,在随后的假设检验和后续研究中,通过了解所有变体所获得的好处将大大超过这种LD结构带来的任何缺点。
缺失或低质量的数据是SNP芯片和重测序数据集的主要问题。仅从一部分个体中排除基因分型差的变异,会导致不同位点的样本量不相等,使下游统计更加复杂。通常,这是通过对缺失数据进行归因来克服的[48],其中一个非基因分型标记的状态是从其他个体的单倍型推断出来的。当由于技术原因(低覆盖率测序或与基因分型阵列的差杂交)导致数据缺失时,这种方法可能是有效的;然而,如果一个位点上存在两个以上的等位基因,则很可能会误判正确的状态,当SVs和CNVs重叠一个SNP时就会发生这种情况。另一种可能是基因型的不确定性[49通过计算每个SNP的概率分数,然后用于加权回归。
亲缘关系引起的混淆
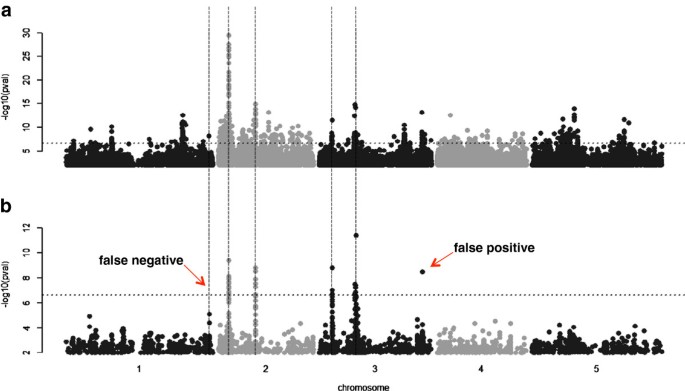
上面讨论的两个主要问题:相关个体共享因果和非因果等位基因,以及这些位点之间的LD可以导致合成关联,实际上是一个单一的问题,即由于遗传背景造成的混淆[50].动物育种领域首先开发了一种强大的方法来解释这种人工现象:混合模型,通过解释由于遗传相关性(即在模型中包括关系或亲属关系作为随机术语)造成的表型协方差的数量来处理种群结构。此后,混合模型被应用于GWAS [11,51- - - - - -53],并可显著减少假阳性关联的数量(图3.).
不幸的是,任何用来校正种群结构的关系矩阵都只能作为真正潜在遗传背景的代表。50].直观地说,人们只希望纠正与感兴趣的特征相关的混淆标记。一种方法是在关系矩阵中只包含与性状线性相关性最强的snp [54].
判断结果
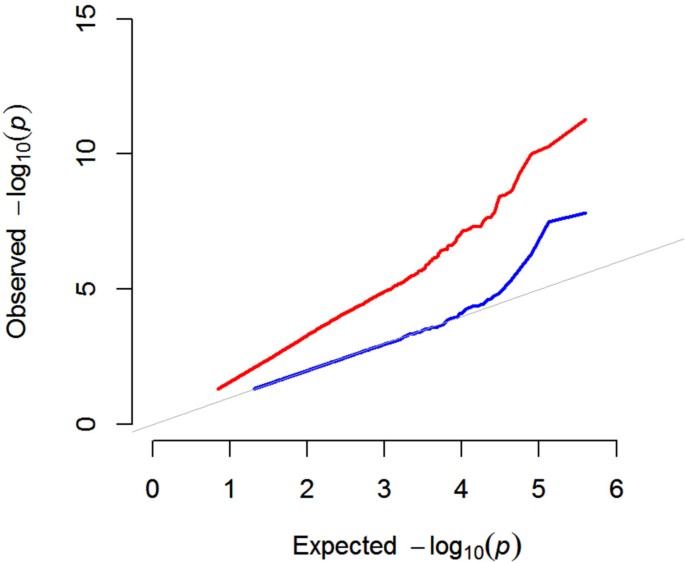
根据什么标准可以判断一个特定性状最合适的GWAS方法?最基本且通常信息丰富的方法是对多次测试进行校正(通常使用5% Bonferroni阈值),并检查Q-Q图和Manhattan图以寻找证据P价值膨胀(数字)3.而且4).这两种方法都给出了数据的总体印象,即相对于先前的预期,重要的snp是太多还是太少?这些修正的主要限制是假设每个测试的SNP都是独立的。结构拟南芥人口显然违背了这一假设,因此由于数据中的LD,许多虚假关联在多次测试校正中幸存下来。
最具信息量的性能标准是模拟数据集中(保持真实总体的LD特征)的假阳性和假阴性关联的比例,通常表示为错误发现率(FDR)和功率(图2)1).考虑到研究的目的,一些项目可以考虑高FDR(例如研究性状的遗传结构),而另一些项目则考虑低FDR(例如确定后续研究的候选位点)。
综上所述:遗传性
狭义遗传力是衡量加性遗传变异对观察到的表型方差的贡献;这可以被认为是表现型与基因型的联系有多强。用于运行GWAS的混合模型将观察到的表型方差划分为可加的遗传成分和非遗传成分。这些估计值可以(在无穷小模型的假设下)用于计算遗传力,通常称为伪遗传力。
对于某些性状(开花时间在拟南芥例如)伪遗传力实际上可能超过复制估计的遗传力(A. Korte个人观察)。然而,对于许多性状(尤其是人类),伪遗传力要低得多。这可以被认为是遗传性缺失的一个特例。缺失遗传力通常是指无法用所有显著snp解释的遗传方差部分[32].这种差异的部分原因可能是致病变异和基因型之间的不完全联系,或者是由于罕见的变异[31](然而,请参阅[55])。那么,问题是GWAS在识别小效应的变体方面的一个基本限制,或者从另一个角度来看,在一个小而异构的样本中运行GWAS的限制。包含全序列数据和增加样本量理论上可以克服这个问题。
此外,有人认为表观遗传状态也可能有助于遗传拟南芥[56,57].鉴于可变的表观遗传状态可能调节SNP和性状之间的联系,考虑这种中间生物学信息是合适的。这可以通过将遗传、表观遗传、基因表达和表型信息整合到一个联合模型中来实现。估计这样一个模型的参数需要大量的样本量。
解释了基因组和环境之间的相互作用
边际GWAS不考虑位点间的遗传互作(上位性),也不考虑位点与环境的互作。基因间的上位性相互作用对关联作图提出了一个问题,但有可能对关联作图做出重要贡献拟南芥表型(58].
虽然已经提出了在GWAS中识别上位性相互作用的策略[59- - - - - -61],全基因组相互作用扫描(如果不是计算上的,至少在统计上)受到需要执行的大量测试的影响。使用图形处理器(gpu)可解决计算问题[62].
从数万亿的成对测试中识别有意义的关联是一个严峻的挑战。减少测试数量的各种方法只考虑先前在边缘GWAS中显示的重要位点,或使用降维方法[63,64].结合了拟南芥交互组数据[65是另一种可能。例如,通过考虑已知的网络拓扑并逐个测试特定的模型,最近的一项研究使用了特征良好的硫代葡萄糖苷通路,并将其与GWAS结合起来,以确定对环境波动敏感的新位点[21].
一个基因对一个性状的贡献可能因环境条件而异,已经有人提出了鉴定这种基因-环境(GxE)相互作用的方法[66].在这种情况下,重复显型同一基因型的能力(由于自交)允许一个人在几个不同的环境中测试相关性(例如在两种温度下的开花时间或在不同位置的适应度)。有人提出了分析相关性状的统计模型(可以将在两个环境中测量的性状视为两个相关性状),同时还可以校正种群结构[67].这允许检测以前未检测到的关联,并将影响分解为遗传和环境成分,从而揭示性状结构[67,68].有趣的是,这种方法在检测GxE交叉效应方面更强大,在这种效应中,一个等位基因在不同环境中的作用是相反的,而在识别缩放效应方面则不那么强大,在这种效应中,只有效应的大小发生了变化[67,69].
表型相同拟南芥在受控制的环境条件下,多次行增加了性状均值的精度,但也允许人们估计表型方差。模拟表明,选择对方差控制位点的影响更为强烈[70].为了使这种方法起作用,表型缓冲的程度必须因人而异,这是一种可能的情况。迄今为止,大多数GWAS只考虑了性状的平均值,旨在了解遗传对特定表型的贡献本身.然而,通过考虑表型方差,有可能揭示鲁棒性和可塑性的遗传基础。
有趣的是,等位基因可能经历的最戏剧性的环境变化是转向不同的遗传背景。虽然从技术上讲,任何导致的差异都是上位性(基因-基因相互作用)的结果,但人们基本上可以将其建模为GxE效应。
期待
GWAS方法已经取得了很大的进展,它现在是一个强大的工具,用于分析加性遗传场景下的简单性状,以及解剖更复杂的遗传结构。人类和植物中许多感兴趣的表型都是高度定量的,因此GWAS可能无法揭示我们所寻找的致病位点。一种可能的解决方案是通过对更接近潜在遗传学的性状进行评分来改进感兴趣的表型[71].这有可能减少对性状有贡献的基因座的数量,从而增加检测它们的能力。
这是一个重要的考虑(或限制),即使在图中所示的具有高遗传力的单个致病位点的简单模拟场景下1b,最显著的SNP并不一定是真正的致病位点。这种综合关联是数据的联系和误差结构的自然结果,因此尽管样本量增加,这种关联仍可能持续存在。
现在的文献包含了许多揭示潜在遗传学的GWAS的例子。然而,缺失的基因型、遗传异质性、意外的LD、小效应量、低等位基因频率或复杂的遗传结构仍然是一个挑战。考虑到这些因素的GWAS方法的收集将继续增长。然而,成功的最佳预测因素仍然是一个明确的特征,一个适当的统计模型,最后是对候选人的验证。
术语表
影响的大小一个位点上两个等位基因的平均表型差异。
基因结构遗传变异网络:构成某一特定性状的遗传变异网络,包括致病等位基因的数量、效应大小和等位基因频率,以及它们之间的所有加性和上位性相互作用
遗传背景所有在特定环境中对特定性状没有贡献的基因座。群体结构等因素可导致遗传背景与性状的部分相关。
遗传异质性当不同的位点,无论是在单个基因内(等位基因异质性)还是在不同的基因内(基因/位点异质性),在不同的个体中产生相同的表型效应。
基因-环境(GxE)相互作用当一个位点的表型效应在不同的环境中不同时(见[72])。
基因-基因(GxG)相互作用或上位两个或多个位点的非加性相互作用(见[73])。位点间的等位基因组合可能导致比单个位点效应量预期的更高(正上位性)或更低(负上位性)的表型。
遗传在特定环境中归因于基因型方差(广义遗传力)的表型方差比例(见[74])。加性遗传变异(即不包括显性和上位性)的贡献是狭义遗传力(或育种值),可以通过后代表型值对亲本表型的回归来估计。
连杆不平衡(LD)两个或多个等位基因的非随机共存。LD自然发生在距离较近的位点之间,并通过重组分解。通过选择或群体结构,即使在不同的染色体上,也可以保持高于预期的LD。
混合模型一种同时包含固定效应和随机效应的统计模型,用于估计表型和基因型之间的相关性,同时考虑到个体之间的相关性。
表型方差一个群体中特征值传播的度量。表型变异源于遗传(见遗传力)和环境因素。由单个位点解释的表型方差的比例是其效应大小和等位基因频率的乘积。
Pseudo-heritability从混合模型估计狭义遗传力。这是可以通过个体之间的遗传相关性来解释的表型方差的比例(由SNP数据的全基因组亲缘矩阵估计)。
合成协会由一个或多个致病标记和/或一个不可测量的误差源的关联驱动的非致病标记与给定性状的关联。
参考文献
Alonso-Blanco C, El-Assal SE, Coupland G, Koornneef M:拟南芥Landsberg erecta和Cape Verde Islands生态型开花时间基因座的自然等位基因变异分析。中国生物医学工程学报,1998,14(2):749-764。
刘志刚,刘志刚,刘志刚,刘志刚:拟南芥开花时间的QTL分析。中国生物医学工程学报,2004,26(3):379 - 379。10.1007 / BF02191594。
Kowalski SP, Lan TH, Feldmann KA, Paterson AH:拟南芥开花时间自然变异的QTL定位。生物化学学报,2004,26(5):548-555。
胡晓明,李文杰,王晓明,等。拟南芥遗传变异的研究进展。植物学报,2004,29(3):344 - 344。10.1146 / annurev.arplant.55.031903.141605。
Borevitz JO, Nordborg M:基因组学对拟南芥自然变异研究的影响。植物学报,2003,32(2):718-725。10.1104 / pp.103.023549。
Balasubramanian S, Schwartz C, Singh A, Warthmann N, Kim MC, Maloof JN, Loudet O, Trainer GT, Dabi T, Borevitz JO:拟南芥高级杂交重组自交系QTL定位。公共科学学报,2009,4 (2):e4318-10.1371/journal. peer .0004318。
Kover PX, Valdar W, Trakalo J, Scarcelli N, Ehrenreich IM, Purugganan MD, Durrant C, Mott R:拟南芥多亲本高级代数量性状精细图谱研究。公共科学学报,2009,5 (7):e1000551-10.1371/journal.pgen.1000551。
黄X, Paulo MJ, Boer M, Effgen S, Keizer P, Koornneef M, van Eeuwijk FA:拟南芥多亲本重组自交系群体自然等位基因变异分析。美国国家科学研究院。2011, 38(4): 489 - 497。10.1073 / pnas.1100465108。
拟南芥的自然变异:从分子遗传学到生态基因组学。中国生物医学工程学报,2012,29(1):1 -2。10.1104 / pp.111.189845。
Brachi B, Faure N, Horton M, Flahauw E, Vazquez A, Nordborg M, Bergelson J, Cuguen J, Roux F:拟南芥自然开花时间的连锁和关联映射。公共科学学报,2010,6 (5):e1000940-10.1371/journal.pgen.1000940。
赵k, Aranzana MJ, Kim S, Lister C, Shindo C, Tang C, Toomajian C, Zheng H, Dean C, Marjoram P:拟南芥结构样本中关联映射的一个例子。公共科学学报,2007,3 (1):e4-10.1371/journal.pgen.0030004。
Hirschhorn JN, Daly MJ:常见疾病和复杂性状的全基因组关联研究。植物学报,2005,6(2):95-108。
Hindorff LA, Sethupathy P, Junkins HA, Ramos EM, Mehta JP, Collins FS, Manolio TA:人类疾病和性状的全基因组关联位点的潜在病因学和功能意义。美国国家科学研究院。2009, 106(23): 9362-9367。10.1073 / pnas.0903103106。
Atwell S, Huang YS, Vilhjalmsson BJ, Willems G, Horton M, Li Y,孟D, Platt A, Tarone AM, Hu TT:拟南芥自交系107个表型的全基因组关联研究。自然科学进展,2010,38(4):344 - 344。10.1038 / nature08800。
弗林特J,埃斯金E:小鼠全基因组关联研究。植物学报,2012,13(11):807-817。10.1038 / nrg3335。
黄霞,赵颖,魏霞,李超,王安,赵强,李伟,郭勇,邓龙,朱春:世界水稻种质开花时间与产量性状的全基因组关联研究。植物学报,2012,44(1):32-39。
Ranc N, Munos S, Xu J, Le Paslier MC, Chauveau A, Bounon R, Rolland S, Bouchet JP, Brunel D, Causse M:番茄(Solanum lycopersicum var. cerasiforme)的全基因组关联作图是可能的。G3(贝塞斯达)。2012, 2(8): 853-864。2012.
王敏,姜宁,贾涛,Leach L, Cockram J, Comadran J, Shaw P, Waugh R,罗忠:大麦高结构群体农艺性状和形态性状的全基因组关联定位。应用理论与实践,2012,29(2):344 - 344。10.1007 / s00122 - 011 - 1697 - 2。
Olsen HG, Hayes BJ, Kent MP, Nome T, Svendsen M, Larsgard AG, Lien S:挪威红牛全基因组关联作图确定了BTA12上生育和产奶的数量性状位点。植物学报,2011,42(5):466-474。10.1111 / j.1365-2052.2011.02179.x。
Horton MW, Hancock AM, Huang YS, Toomajian C, Atwell S, Auton A, Muliyati NW, Platt A, Sperone FG, Vilhjalmsson BJ:基于RegMap的拟南芥全基因组遗传变异模式。植物学报,2012,44(2):212-216。10.1038 / ng.1042。
陈ek, Rowe HC, Corwin JA, Joseph B, Kliebenstein DJ:结合全基因组关联作图和转录网络鉴定拟南芥硫代葡萄糖苷控制基因。公共科学图书馆,2011,9 (8):e1001125-10.1371/journal.pbio.1001125。
Filiault DL, Maloof JN:一项全基因组关联研究确定了拟南芥避荫反应的变异。公共科学学报,2012,8 (3):e1002589-10.1371/journal.pgen.1002589。
Chao DY, Silva A, Baxter I, Huang YS, Nordborg M, Danku J, Lahner B, Yakubova E, Salt DE:重金属ATPase3是拟南芥叶片镉自然变异的主要决定因素。公共科学图书馆,2012,8 (9):e1002923-10.1371/journal.pgen.1002923。
Baxter I, Brazelton JN, Yu D, Huang YS, Lahner B, Yakubova E, Li Y, Bergelson J, Borevitz JO, Nordborg M:钠转运体AtHKT1的自然变异驱动拟南芥钠积累的海岸渐变;公共科学学报,2010,6 (11):e1001193-10.1371/journal.pgen.1001193。
李艳,黄艳,Bergelson J, Nordborg M, Borevitz JO:拟南芥局部气候敏感数量性状位点的关联定位。美国国家科学研究院。中国农业科学,2010,29(4):379 - 379。10.1073 / pnas.1007431107。
傅尼尔- level A, Korte A, Cooper MD, Nordborg M, Schmitt J, Wilczek AM:拟南芥局部适应图谱。科学通报,2011,34(6):344 - 344。10.1126 / science.1209271。
Seren U, Vilhjalmsson BJ, Horton MW,孟D, Forai P, Huang YS, Long Q, Segura V, Nordborg M:拟南芥全基因组关联作图的网络应用。植物细胞学报,2012,24(12):4793-4805。10.1105 / tpc.112.108068。
Louthan AM, Kay KM:跨性状类型的适应性景观比较:生物选择下性状的更大QTL效应量。中国生物医学工程学报,2011,29(4):344 - 344。
李志强,李志强,李志强,等。复杂性状的稀有变异关联分析方法。植物学报,2010,44:293-308。10.1146 / annurev -麝猫- 102209 - 163421。
Gibson G:罕见和常见的变体:20个参数。植物学报,2011,13(2):135-145。
Dickson SP, Wang K, Krantz I, Hakonarson H, Goldstein DB:罕见的变异产生合成的全基因组关联。公共科学图书馆,2010,8 (1):e1000294-10.1371/journal.pbio.1000294。
Manolio TA, Collins FS, Cox NJ, Goldstein DB, Hindorff LA, Hunter DJ, McCarthy MI, Ramos EM, Cardon LR, Chakravarti A:寻找复杂疾病的缺失遗传性。自然科学进展,2009,37(4):344 - 344。10.1038 / nature08494。
博德默W,博尼拉C:常见疾病多因素易感性的常见和罕见变异。植物学报,2008,40(6):695-701。10.1038 / ng.f.136。
Flint J, Mackay TF:小鼠,苍蝇和人类数量性状的遗传结构。基因组学报,2009,19(5):723-733。10.1101 / gr.086660.108。
田飞,Bradbury PJ, Brown PJ, Hung H, Sun Q, Flint-Garcia S, Rocheford TR, McMullen MD, Holland JB, Buckler ES:玉米巢状关联定位群体叶片结构的全基因组关联研究。植物学报,2011,43(2):159-162。10.1038 / ng.746。
Sulem P, Gudbjartsson DF, Stacey SN, Helgason A, Rafnar T, Magnusson KP, Manolescu A, Karason A, Palsson A, Thorleifsson G:欧洲人头发、眼睛和皮肤色素沉着的遗传决定因素。植物学报,2007,39(12):1443-1452。10.1038 / ng.2007.13。
Beleza S, Johnson NA, Candille SI, Absher DM, Coram MA, Lopes J, Campos J, Araujo II, Anderson TM, Vilhjalmsson BJ:非洲-欧洲混合人群皮肤和眼睛颜色的遗传结构。公共科学学报,2013,9 (3):e1003372-10.1371/journal.pgen.1003372。
Platt A, Vilhjalmsson BJ, Nordborg M:在这种条件下,全基因组关联研究将具有积极的误导性。遗传学报,2010,186(3):1045-1052。10.1534 / genetics.110.121665。
Segura V, Vilhjalmsson BJ, Platt A, Korte A, Seren U, Long Q, Nordborg M:结构化群体中全基因组关联研究的高效多位点混合模型方法。植物学报,2012,44(7):825-830。10.1038 / ng.2314。
冯涛,朱旭:检测罕见变异。方法分子生物学杂志,2012,85:453-464。10.1007 / 978 - 1 - 61779 - 555 - 8 -大于。
戴勇,郭磊,董娟,蒋锐:基于数据自适应前向选择策略的罕见和常见变异折叠改进功率。BMC Proc 2011, 5 Suppl 9: S114-
Bhatia G, Bansal V, Harismendy O, Schork NJ, Topol EJ, Frazer K, Bafna V:一种检测罕见变异和常见表型之间遗传关联的覆盖方法。中国生物医学工程学报,2010,26 (10):10954 - 101371 /journal.pcbi.1000954。
李波,Leal SM:常见疾病罕见变异相关性检测方法:在序列数据分析中的应用。中国生物医学工程学报,2008,29(3):344 - 344。10.1016 / j.ajhg.2008.06.024。
曹j, Schneeberger K, Ossowski S, Gunther T, Bender S, Fitz J, Koenig D, Lanz C, Stegle O, Lippert C:拟南芥多居群全基因组测序。植物学报。2011,43(10):956-963。10.1038 / ng.911。
Gan X, Stegle O, Behr J, Steffen JG, Drewe P, Hildebrand KL, Lyngsoe R, Schultheiss SJ, Osborne EJ, Sreedharan VT:拟南芥的多参考基因组和转录组。自然科学学报,2011,37(6):419-423。10.1038 / nature10414。
Long R,孟,Huber, Farlow, Platzer, Zhang, Vilhjalmsson, Korte, Nizhynska, Voronin, Korte, Sedman, Mandakova, Lysak, Seren, Hellmann, Nordborg:瑞典拟南芥的大规模基因组变异和强选择。Nat麝猫。在新闻
Kim S, Plagnol V, Hu TT, Toomajian C, Clark RM, Ossowski S, Ecker JR, Weigel D, Nordborg M:拟南芥重组与连锁不平衡。植物学报,2007,39(9):1151-1155。10.1038 / ng2115。
焦山,徐玲,Hutter CM, Peters U:估算值在全基因组关联研究meta分析中的应用。中华流行病学杂志,2011,35(7):597-605。10.1002 / gepi.20608。
Taub MA, Schwender H, Beaty TH, Louis TA, Ruczinski I:将基因型不确定性纳入基因型TDT的主要效应和基因-环境相互作用。中华流行病学杂志,2012,36(3):225-234。10.1002 / gepi.21615。
Vilhjalmsson BJ, Nordborg M:全基因组关联研究中混杂的性质。植物学报,2012,14(1):1-2。
姜HM, NA Zaitlen, Wade CM, Kirby A, Heckerman D, Daly MJ, Eskin E:模型生物关联映射中群体结构的有效控制。中国生物医学工程学报,2008,39(3):379 - 379。10.1534 / genetics.107.080101。
Yu J, Pressoir G, Briggs WH, Vroh Bi I, Yamasaki M, Doebley JF, McMullen MD, Gaut BS, Nielsen DM, Holland JB:一种统一的混合模型关联映射方法,该方法考虑了多个层次的相关性。植物学报,2006,38(2):203-208。10.1038 / ng1702。
张震,Ersoz E,赖cq, Todhunter RJ, Tiwari HK, Gore MA, Bradbury PJ, Yu J, Arnett DK, as JM:用于全基因组关联研究的混合线性模型方法。植物学报,2010,42(4):355-360。10.1038 / ng.546。
李士强,李柏德,李秀珍,李秀珍,李秀珍。基于线性混合模型的全基因组关联研究。Nat方法,2012,9(6):525-526。10.1038 / nmeth.2037。
Wray NR, Purcell SM, Visscher PM:由罕见变异产生的合成关联不能解释大多数GWAS结果。公共科学图书馆,2011,9 (1):e1000579-10.1371/journal.pbio.1000579。
Johannes F, Porcher E, Teixeira FK, Saliba-Colombani V, Simon M, Agier N, Bulski A, Albuisson J, Heredia F, Audigier P:评估跨代表观遗传变异对复杂性状的影响。公共科学学报,2009,5 (6):e1000530-10.1371/journal.pgen.1000530。
杨晓明,王晓明,王晓明,等。拟南芥生态相关性状的基因鉴定。植物学报,2010,11(12):867-879。10.1038 / nrg2896。
Ehrenreich IM, Stafford PA, Purugganan MD:拟南芥芽分枝的遗传结构:候选基因关联与数量性状位点定位的比较评估。中国生物医学工程学报,2007,26(2):339 - 339。
Cordell HJ:检测人类疾病背后的基因-基因相互作用。植物学报,2009,10(6):392-404。
马奇尼,唐奈利,李志刚:基于基因多态性的复杂疾病诊断方法。植物学报,2005,37(4):413-417。10.1038 / ng1537。
Kam-Thong T, Putz B, Karbalai N, Muller-Myhsok B, Borgwardt K:基于gpu穷尽枚举的定量表型上位性检测。生物信息学,2011,27 (13):i214-221。10.1093 /生物信息学/ btr218。
Hemani G, Theocharidis A, Wei W, Haley C: EpiGPU:在消费级显卡上并行的穷举成对上位扫描。生物信息学,2011,27(11):1462-1465。10.1093 /生物信息学/ btr172。
Turner SD, Berg RL, Linneman JG, Peissig PL, Crawford DC, Denny JC, Roden DM, McCarty CA, Ritchie MD, Wilke RA:知识驱动的多位点分析揭示了两个独立emr链接生物库中影响HDL胆固醇水平的基因-基因相互作用。公共科学学报,2011,6 (5):e19586-10.1371/journal.pone.0019586。
吴s,李j,权MS, Weir B,河K, Park T:一种基于基因MDR的全基因组关联研究中高阶基因-基因相互作用的新方法。13补充9。2012, 13增刊:S5-
联盟目的:拟南芥相互作用组图谱中网络进化的证据。科学通报,2011,32(3):344 - 344。
Thomas D:基因环境关联研究:新兴方法。植物学报,2010,11(4):259-272。10.1038 / nrg2764。
Korte A, Vilhjalmsson BJ, Segura V, Platt A, Long Q, Nordborg M:结构化群体中相关性状全基因组关联研究的混合模型方法。植物学报,2012,44(9):1066-1071。10.1038 / ng.2376。
戴瑞,杨j,戴维斯,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚。自然科学进展,2012,29(3):344 - 344。
拉卡泽X,海斯PM, Korol A:大麦表型可塑性的遗传:QTL分析。遗传(Edinb)。2009, 102(2): 163-173。10.1038 / hdy.2008.76。
陈晓明,陈晓明,陈晓明:变异控制基因的选择:适应性还是稳定性。自然科学进展,2012,39(12):379 - 379。10.1111 / j.1558-5646.2012.01753.x。
Benjamin DJ, Cesarini D, Chabris CF, Glaeser EL, Laibson DI, Guethnason V, Harris TB, Launer LJ, Purcell S, Smith AV:基因经济学的承诺和陷阱*。经济季刊,2012,4:627-662。10.1146 / annurev -经济- 080511 - 110939。
Hunter DJ:人类疾病中的基因-环境相互作用。植物学报,2005,6(4):287-298。
上位性-基因相互作用在遗传系统的结构和进化中的重要作用。植物学报,2008,9(11):855-867。10.1038 / nrg2452。
Visscher PM, Hill WG, Wray NR:基因组学时代的遗传性——概念和误解。植物学报,2008,9(4):255-266。
致谢
我们感谢JackiHeraud-Farlow, Matt Horton和Magnus Nordborg对手稿的评论和讨论。AK得到了德国农业协会(Deutsche Forschungsgemeinschaft, DFG) (KO4184/1-1)的资助。
作者信息
作者及隶属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
作者的贡献
AK和AF共同撰写并编辑了手稿。两位作者都阅读并批准了最终的手稿。
亚瑟·科特和阿什利·法洛对这项工作做出了同样的贡献。
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用属性许可协议(http://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
Korte, A., Farlow, A.用GWAS进行性状分析的优点和局限性:综述。工厂方法9, 29(2013)。https://doi.org/10.1186/1746-4811-9-29
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1746-4811-9-29
关键字
- GWAS
- 拟南芥
- 混合模型
- 影响的大小
- 遗传异质性