简介
果实大小是水果作物的重要性状,包括番茄(茄属植物lycopersicum).它影响产量,这是植物育种和改良计划的首要任务。研究表明,抗病性的渗透,通常是成功培育品种的必要条件,对产量有负面影响(Ning等,2017).因此,除了感兴趣的新性状外,不损害现有性状的遗传资源总是需求量很大,因为这类资源可以非常有利于将新性状迅速纳入育种背景。鉴于此,在DNA序列水平上利用这些负面影响的知识一直是(应用)植物科学社会的兴趣所在。
为了提供丰富的序列资源,发现与果实尺寸缩小相关的候选基因,我们重点研究了枯萎病抗性我渗入(两者我基因和它的侧翼区域通常覆盖多兆酶),它是从野生番茄(美国pennellii;(斯科特和琼斯,1989)变成驯化番茄(美国lycopersicum),在历史上被认为可以减少驯化番茄的果实大小(重量)(Scott1999;Chitwood-Brown等人,2021年,一个).有趣的是,最近,缩短了我通过杂交获得的基因渗入证明,短的基因渗入并没有减少果实大小,这意味着1)限制减少果实大小的连锁阻力被破坏,2)停留在野生番茄基因组区域的基因与多兆酶杂交可能是果实大小减少的主要原因。果实尺寸缩小导致基因的鉴定依赖于基因组区域上的基因发现,该区域已被交叉,目前基于参考基因组与> 236-kbp歧义核苷酸存在缺口。三种番茄有相同的基因背景,除了我选用耐腐蚀的Fla. 8814我渐渗(估计4.2-Mbp),这表明减少的果实大小(此后,佛罗里达州8814长),耐腐蚀的Fla. 8814我通过交叉(s),以较短的间隔(估计140 kbp)渗入,这并不显示减少果实大小(Fla. 8814短)和易感的佛罗里达8814我等位基因,也不显示减少的果实大小(佛罗里达州8814没有一个).
研究证明,基因组组装,特别是通过长读,可以增强序列变异的检测,尤其是结构变异(SVs)(即长度≥50 bp) (Wang et al.,2021).此外,在本研究中使用的Fla. 8814和作为完全测序参考的驯化番茄的Heinz 1706之间的遗传变异(番茄基因组联盟,2012)如果仅将序列片段与驯化番茄基因组进行比对,可能会导致对变异的误解和/或无法发现最初源自野生番茄的现有变异。因此,我们使用Oxford Nanopore和Illumina NovaSeq技术对三个番茄基因组进行了测序,并在从头组装的基础上构建了contigs。
结果
我们对三种番茄进行了超过100 ×基因组覆盖的长读基因组序列数据(表S1).结合短读误差校正的从头组装使每个番茄的装配长度为2.7 ~ 5.3 mbp(表S2).基于短读对程序集的比对,高映射率(> 98%)和覆盖率(> 94%)表明程序集和读之间具有较高的一致性(表S .)3.).此外,每个装配的BUSCO得分至少为96.9%(表S4),表示装配的完整性很高。最后,将组装的contigs与两个参考基因组进行比对,结果表明参考基因组与contigs在宏观水平上存在高度共线性(图2)。1,图S1).
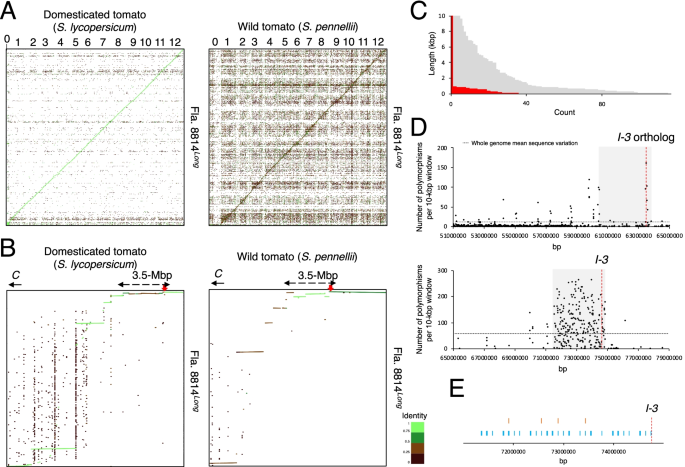
枯萎病抗性的序列鉴定我侧翼果实大小减小区。一个参考基因组(水平)与Fla. 8814的contigs之间的点图比较长(垂直)(一个而且B).数字代表番茄染色体。序列相似度用从0到1的颜色编码(一个而且B;身份摘要放在旁边B).B放大图(14mbp间隔)捕捉我7号染色体的渐渗。推断出我用破折号箭头表示渐渗(约3.5-Mbp)。的位置我基因。C着丝粒。C在野生番茄基因组4-Mbp区间内,发现115个缺口长度分布我渐渗(灰色)和36个由contigs(红色)重叠的间隙。D枯萎病抗性常见序列变异密度图我渐渗现象。顶部和底部面板显示了基于参照驯化番茄(即,在佛罗里达州8814中发现的序列变体)比对的常见序列变体长佛罗里达州8814号短但不是在佛罗里达州8814没有一个)和参考野生番茄(即,在佛罗里达州8814短佛罗里达州8814号没有一个但不是在佛罗里达州8814长),分别。推断出我渐渗(约3.5-Mbp)用灰色表示。的大致位置我- - - - - -3正交(Solyc07g05564063,514,724 - 63,521,342 bp;顶部面板D),我(Sopen07g029010介于74,627,845至74,630,497 bp之间;底部面板D而且E)用红线表示。图S3为每个Fla. 8814的序列变异密度图长佛罗里达州,8814短佛罗里达州8814号没有一个.物理位置是基于个体的参考基因组。E在Fla. 8814中发现了结构变异(SVs)(长度≥50 bp)的位置长仅与同源DNA序列(即,在佛罗里达州8814中均未发现)进行比较短也没有佛罗里达州8814号没有一个)(橙色竖条),在佛罗里达州8814短佛罗里达州8814号没有一个但不是在佛罗里达州8814长(蓝色竖条)。物理位置是基于野生番茄的参考基因组
序列对齐将几个contig放置在一个14 mbp的间隔中,该间隔携带到我渐渗现象(图1 b,图S2).很明显,中心体-近端我侧翼区域与驯化番茄基因组的相似性较小。相比之下,这个着丝粒-近端侧翼区域与野生番茄基因组具有很高的相似性。这一观察结果与先前的一项研究一致,该研究报告称,在驯化番茄背景中,大多数剩余的野生番茄序列都是着丝粒-近端。当前版本的野生番茄基因组在7号染色体上有115个缺口(即每个缺口≥50 bp),这些缺口充满了不明确的核苷酸(即Ns),间隔为71至75-Mbp。在佛罗里达州8814号长然而,比对结果显示,其中36个间隙与来自大型contigs的明确序列(> 1.0-Mbp)重叠(图2)。1 c、表。年代5),从而使无间隙基因组组装更容易实现。
高序列变异频率明显靠近我从基于contigs比对的变体发现到参考基因组(图2)。1 d,图S3.表S6).与之前间接推断序列变异的方法(Chitwood-Brown et al.,2021 b),很明显,的大小我渗入量接近3.5-Mbp(驯化番茄在60.4 ~ 63.7-Mbp之间,野生番茄在71.4 ~ 74.6-Mbp之间)。同时,在3.5-Mbp范围内唯一鉴定出72个sv我佛罗里达州8814的侧翼间隔长与Fla. 8814相同大小的间隔进行了比较短佛罗里达州8814号没有一个与佛罗里达州8814的3.5-Mbp有相似之处长(无花果。1 e表S7).有趣的是,一个SV(从野生番茄染色体7号位置72,195,816 bp开始)在佛罗里达州8814短佛罗里达州8814号没有一个但不是在佛罗里达州8814长,包含野生番茄基因的部分外显子区Sopen07g026470(显示与kruppel-like因子的相似性Solyc07g052913).对于一个简略的我观察到一个连续高序列变异的区间(在驯化番茄的63.43和63.58-Mbp之间约150-kbp),与先前估计的结果相似(表S .)8).
讨论
在当前的研究中,我们报告了两个主要贡献。首先,许多缺失的DNA序列在我侧翼区域通过组装长读数据进行序列解析。目前LA716的参考基因组是使用Illumina短对端/配偶对和bac端测序组装的(Bolger et al.,2014).我们的contigs和在野生番茄渐进中鉴定的sv一起,现在提供了对先前未知的番茄遗传变异区域的访问。第二,我佛罗里达州8814号的侵入长极有可能接近3.5 mbp。用目前的基因组技术,确定精确的基因渐渗边界是一项挑战。它进一步受到另一种存在的野生番茄渗入的阻碍(图s的左面板)3.).根据美国新鲜市场大果(圆)番茄的计算重组率(1.0至2.6 cM/Mbp),驯化番茄和野生番茄之间的重组水平通常低于两个驯化番茄之间的杂交(Bhandari和Lee,2021;班达里等人,2022),则在我轨迹。
绵延不断的缺口我侧翼区域表示在该抗病位点附近有复杂的遗传变异区域。其他长读和超长读测序平台与完全测序的大插入克隆(如细菌人工染色体/Fosmid)相结合,可以用于识别近基因组的完整遗传变异谱我,从而减少了这种模型水果作物的果实大小。组装和生物信息学技术的进步也可能揭示先前未组装的基因组序列并纠正错误序列。
数据和材料的可用性
基因组集合已存入GenBank,登录代码PRJNA841967。
参考文献
Bhandari P, Lee TG。大果鲜食番茄的遗传图谱和连锁面板。地球科学学报,2001;26(2):344 - 344。https://doi.org/10.21273/JASHS04999-20.
Bhandari P, Shekasteband R, Lee TG。鲜食番茄共识遗传图谱及连锁面板。地球科学学报,2002,26(3):344 - 344。
Bolger A, Scossa F, Bolger ME, Lanz C, Maumus F, Tohge T,等。耐胁迫野生番茄物种的基因组茄属植物pennellii.Nat Genet, 2014; 46:1034-8。https://doi.org/10.1038/ng.3046.
奇伍德-布朗J,瓦拉德GE,李TG,赫顿SF。番茄抗枯萎病育种研究进展。基因。2021;12:1673。https://doi.org/10.3390/genes12111673.
李建平,李志刚,等。枯萎病3种抗性基因连锁阻力的鉴定与消除。理论应用,2021b; 134:2129-40。https://doi.org/10.1007/s00122-021-03810-5.
宁云,刘伟,王桂兰。作物免疫与产量的平衡。植物科学进展,2017;22:1069-79。https://doi.org/10.1016/j.tplants.2017.09.010.
斯科特JW,琼斯JP。番茄单基因抗性尖孢镰刀菌f.sp。黄瓜比赛3。Euphytica。1989;40:49-53。
番茄基因组联盟。番茄基因组序列为肉质水果的进化提供了见解。大自然。2012;485:635-41。https://doi.org/10.1038/nature11119.
王艳,赵艳,波拉斯,等。纳米孔测序技术、生物信息学及应用。清华大学学报(自然科学版)。https://doi.org/10.1038/s41587-021-01108-x.
抗枯萎病3种基因杂合的番茄植株比纯合的抗枯萎病植株果实大。佛罗里达州法典1999:305-7。
确认
作者感谢Samuel F. Hutton分享番茄材料。
资金
不适用。
作者信息
作者及隶属关系
贡献
TGL进行了这项研究。作者阅读并批准最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1。
材料与方法。
附加文件2:图S1。
参考基因组(水平)与Fla. 8814的contigs之间的点图比较短佛罗里达州8814号没有一个(垂直)。顶部和底部分别以驯化番茄和野生番茄基因组为目标序列。序列相似度用从0到1的颜色编码。
附加文件3:图S2。
放大图(14mbp间隔)捕捉我7号染色体的渐渗。序列相似度用从0到1的颜色编码。C着丝粒。
附加文件4:图S3。
序列变异密度图的我渐渗现象。左右面板分别显示了参照驯化番茄和参照野生番茄排列基础上的序列变异。在Fla. 8814中59.5 - 61.0-Mbp的高序列变异频率(左图)表明存在另一种野生番茄渗透(Chitwood-Brown et al.,2021 b;S.F. Hutton,个人沟通)。推断出我渐渗(约3.5-Mbp)用灰色表示。在佛罗里达州8814号短,序列变异频率在63.52-Mbp附近达到峰值我直接同源Solyc07g05540(63,514,724至63,521,342 bp)位于(左面板)。物理位置是基于个体的参考基因组。
附加文件5:表S1。
序列数据统计。
附加文件6:表S2。
contigs的统计。
附加文件7:表S3。
短读映射统计。
附加文件8:表S4。
对标通用单副本骨科(BUSCO)评估。
附加文件9:表S5。
野生番茄第7号染色体上的间隔为71 ~ 75 mbp,间隔为4-Mbp。
附加文件10:表S6。
在参照驯化番茄比对的基础上,序列变异频率在14-Mbp区间内。
附加文件11:表S7。
结构变异在3.5-Mbp范围内我佛罗里达州8814的侧翼间隔长与佛罗里达州8814号相比短佛罗里达州8814号没有一个
附加文件12:表S8。
基因居住在一个缩短我渐渗现象。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
Lee, T.G. Long-read DNA测序可以更完整地描述枯萎病抗性基因两侧的果实尺寸缩小区域的序列特征。摩尔园艺2, 16(2022)。https://doi.org/10.1186/s43897-022-00037-w
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s43897-022-00037-w