摘要
背景
蝙蝠是人畜共患病毒的主要宿主,人们对描述蝙蝠固有免疫和炎症的特异性特征越来越感兴趣。最近的研究揭示了影响干扰素(IFN)信号和IFN刺激基因(ISGs)的蝙蝠特异性适应,但我们对塑造蝙蝠免疫进化的遗传机制的了解仍然有限。在这里,我们研究了小棕蝙蝠I型IFN反应期间转座元件(TEs)的转录和表观遗传动力学。鼠耳蝠lucifugus)原代胚胎成纤维细胞,使用RNA-seq和CUT&RUN。
结果
我们发现了多种蝙蝠特异性te,它们对IFN的响应经历了位点特异性和家族水平的转录诱导。我们的转录组重组鉴定了多个从蝙蝠特异性TEs获得新外显子的isg,包括NLRC5,SLNF5和之前未标注的异构体IFITM2基因。我们还发现了te衍生的调控元件的例子,但没有发现强有力的证据支持te响应IFN的全基因组表观遗传激活。
结论
总的来说,我们的研究发现了大量由干扰素诱导的te衍生转录本、蛋白质和替代异构体鼠耳蝠lucifugus细胞,突出可能有助于蝙蝠特异性免疫功能的候选位点。
背景
蝙蝠日益被认为是人畜共患病毒的重要储存库,包括狂犬病毒、登革病毒、埃博拉病毒和冠状病毒。[1,2].值得注意的是,蝙蝠的病毒感染与最低的致命性和减少的炎症表型相关,这导致了旨在揭示蝙蝠特异性免疫特征的广泛研究[3.,4,5,6].
最近对蝙蝠的基因组和功能研究已经开始揭示影响先天免疫反应的物种特异性适应。例如,干扰素(IFN)基因在不同的蝙蝠物种中经历了进化的扩张和收缩,一些物种在低水平上表现出IFN的组成性表达[1,7].蝙蝠也表现出独特的isg子集[8].比较I型干扰素反应的时间过程分析狐阿勒克图和人类揭示了ifn刺激基因(ISG)调控的独特动力学,其中与人类相比,蝙蝠表现出更快的ISG下调[9].除了影响IFN信号的适应外,其他促炎基因也经常突变或丢失,包括TLR基因[10]、炎性小体的成分[11,12], cGAS/STING通路[13],以及OAS/RNASEL通路[9,14].这些研究已经开始揭示蝙蝠免疫特异性的遗传基础,这可以帮助我们理解它们作为病毒储存库的倾向。
虽然很明显,蝙蝠已经进化出了许多影响先天免疫途径的独特适应能力,但我们对导致这些变化的遗传机制仍然知之甚少。我们的研究重点是TEs作为一个潜在的重要但尚未得到充分研究的突变来源,形成蝙蝠的免疫力。
TEs被广泛认为是蝙蝠进化的重要贡献者[15,16,17,18,19],包括蝙蝠特有的免疫功能[20.,21].虽然所有哺乳动物物种的基因组都包含大量的谱系特异性转座子,但蝙蝠的基因组以最近活跃的DNA转座子为特征,这种转座子在大多数其他哺乳动物谱系中已经灭绝[15,16,17,18].除了DNA转座子,蝙蝠基因组还被其他哺乳动物中常见的te广泛塑造,包括LTR逆转录转座子,如内源性逆转录病毒(ERVs)、LINEs和SINEs。对其他哺乳动物谱系的研究一再表明,谱系特异性TEs通过多种机制促进先天免疫功能。例如,TE和病毒源性蛋白已多次被选为免疫蛋白,通常通过显性负活性限制病毒[22]反刍动物[23]、啮齿动物[24],以及灵长类[25].te衍生的非编码转录物容易形成免疫刺激双链rna或dna [26,27,28].最后,TEs可作为调控元件调节干扰素诱导基因的表达[29,30.,31,32,33].值得注意的是,最近的一份报告发现了一种Rhinolophus -的外显子内的特定LTR插入OAS1,它破坏了马蹄蝠对SARS-CoV2的抗病毒活性,对OAS1有重大影响-人类对SARS-CoV2的介导反应[14].这个例子进一步支持TEs对免疫功能的反复独立增选可能反映了它们通过增加基因组变异来促进适应的能力,特别是在宿主-病原体共同进化军备竞赛的背景下[27,28].
然而,除了这个例子之外,由于缺乏研究蝙蝠免疫学的实验和功能基因组资源,TEs对蝙蝠免疫力的潜在影响在很大程度上仍未得到研究。
为了对蝙蝠先天免疫中的TEs进行全面的全基因组研究,我们对蝙蝠的I型IFN反应进行了转录组和表观基因组分析鼠耳蝠lucifugus原代成纤维细胞。我们使用RNA-seq和CUT&RUN来表征ifn诱导的转录组和调控元件,这使我们能够系统地检查TEs对定义蝙蝠ifn诱导反应的位点的贡献。
结果
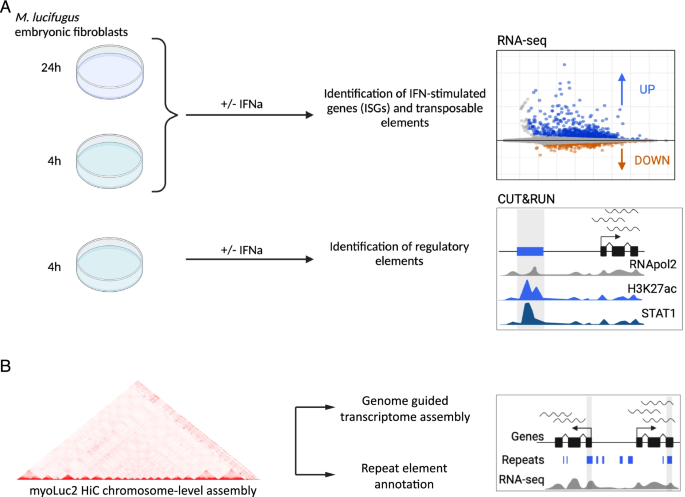
为了表征TEs对蝙蝠IFN反应的贡献,我们对蝙蝠I型IFN反应进行了转录组学和表观基因组分析m . lucifugus原代胚胎成纤维细胞(图;1).我们使用重组通用IFN α (IFNa)刺激细胞,并使用RNA-seq在0、4和24 h时间点对转录组进行分析。我们使用qPCR检测典型ISGs的表达,证实了通用IFN处理的细胞反应(图S1),如上文所示m . lucifugus皮肤成纤维细胞[8].我们还利用CUT&RUN对0和4小时的时间点进行了分析,以绘制H3K27ac、POLR2A和STAT1的全基因组定位图。我们将这些数据与小棕色蝙蝠基因组的染色体尺度HiC组装(myoLuc2.0_HiC) [34,35],这是可用的最连续的组件(支架N50约95.5 Mb)。
实验设计。一个鼠耳蝠lucifugus用1000u /ml的通用IFNa (+IFNa)或匹配容量的DPBS(−IFNa)处理胚胎成纤维细胞原代细胞24小时(24小时)和4小时(4小时)。提取两个时间点的总RNA,并作为RNA文库制备和测序的输入,以鉴定差异表达基因和转座元件(TEs)。为了表征IFN处理后染色质可达性的变化,细胞同样处理4小时,并对H3K27ac、POLR2A和STAT1进行CUT&RUN协议。B染色体水平的基因组组装鼠耳蝠lucifugus作为参考,执行从头重复元素识别和注释。结合我们RNA-seq数据集的基因组引导转录组组装,自定义重复元件注释用于识别te衍生和病毒衍生的异构体和转录起始位点(TSS)。
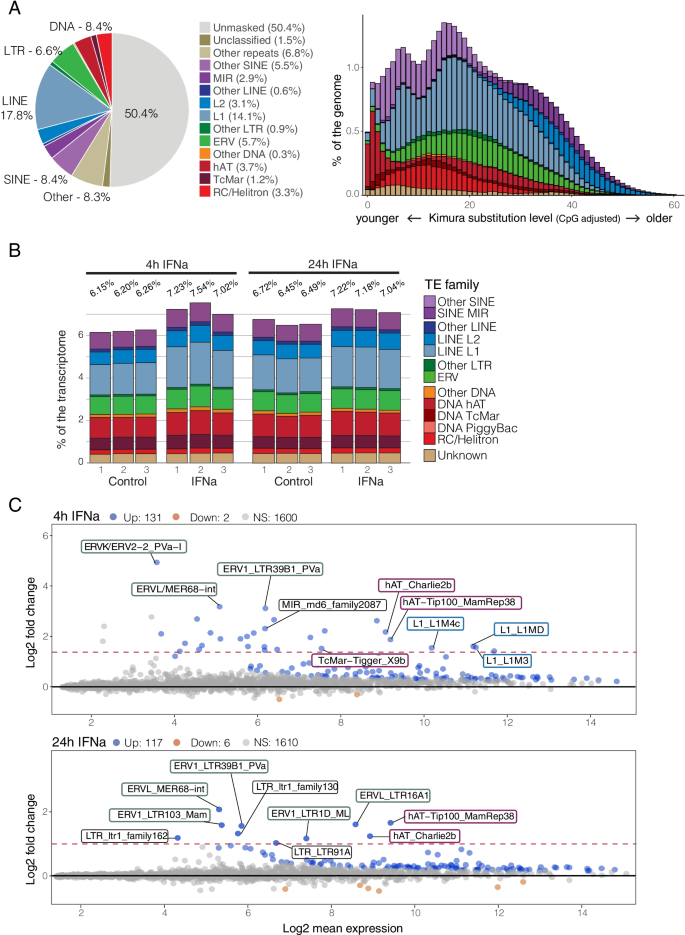
在分析我们的功能基因组数据之前,我们使用RepeatModeler2对myoLuc2.0_HiC组件进行了从头重复识别[36]及HelitronScanner [37],然后使用RepeatMasker重复注释[38,39].我们注释了来自TEs的42.7%的基因组,相比之下,在myoLuc2非hic组装中注释了35.5% (https://www.repeatmasker.org/species/myoLuc.html;无花果。2).L1系代表最丰富的TE组(14.1%),其次是病毒衍生元素(ERVs;5.6%)和DNA hAT和Helitron元件(分别占基因组的3.7和3.2%)(图。2A).如前所述[15],我们的分析显示了DNA转座子的谱系特异性扩增,首先由Helitron元件主导,最近由可能通过水平转移引入的hAT元件的多个亚家族主导(图2)。2) (40].与之前的注释相比,我们的注释包含了更多的种特异性Helitron和LINE1 TEs(分别多出2.3和1.2%),并将额外5.3%的基因组分配给“其他重复序列”,这是一种将卫星重复序列和低复杂度重复序列组合在一起的分类。这种被掩盖重复序列的增加与我们研究中使用的更好质量的基因组组装是一致的。
重复元素组成,进化动态和转录谱。通过结合从头识别和基于同源的搜索对重复元素进行标注。一个饼图显示了TE主要家族的相对丰度(总数为42.7%)占基因组的百分比;直方图显示了主要TE超家族的组成占基因组的百分比,作为与每个TE的参考共识序列的散度(Kimura2Distance)的函数。考虑到与共识的背离与换位时间之间的相关性,K2D(左)越低,TE越年轻。正如先前在Myotis和其他蝙蝠物种中发现的那样,我们恢复了多种DNA元素的近期扩张和活性,特别是Helitrons和最近的hAT元素。B直方图显示了主要TE家族的表达水平,作为处理后4小时和24小时ifna刺激和未刺激细胞的RNA测序数据中归一化读取计数的一部分(%)。Capeglm的地图[41]转换后的数据显示IFN处理后4小时(上)和24小时(下)te表达差异。对于4 h时间点,仅标记Log2倍变化最高的前3个家庭te。对于两个时间点,只有满足调整阈值的tep标记为-value < 0.05和log2 fold change > 1.5(接受fdr = 0.05)。上调(蓝点)和下调(橙点)的te计数基于调整后的p值(< 0.05;仅接受fdr = 0.05)。在4小时的诱导TEs中(根据我们的截止时间共45个),我们恢复了大部分ERV逆转录转座子(绿色轮廓),DNA hAT转座子(红色轮廓)和L1线(浅蓝色轮廓)。在处理后24小时,我们只发现了10个符合过滤标准的家族,大多数ERV与4小时数据集共享
IFN刺激下的基因和TE表达谱
为了分析TE家族水平的转录活性,我们使用TETranscripts将RNA-seq reads映射到基因和TE家族[42(图。2B,图S3.).平均而言,在未受刺激的细胞中,6.38%的RNA-seq读命中了TEs,而在IFN处理4小时后,7.26%的读命中了TEs,在IFN处理24小时后,7.15%的读命中了TEs。这些百分比略高于在其他具有相似TE基因组含量的哺乳动物中观察到的百分比(即小鼠和人类,[43])。然而,TE的表达在同一生物的不同组织和细胞类型之间有很大的差异,我们在原代培养的成纤维细胞中观察到的可能并不能反映TE的生理转录谱,因为TE的转录水平受到染色质可及性和TE调节机制的影响[43].我们鉴定的最丰富的te衍生转录本包括L1 LINEs、DNA/hAT元素、ERVs、sin和L2 LINEs。45例TEs在4 h表现出显著的家族水平转录诱导(adj。假定值< 0.05;log2FC > 1.5),根据相同的截止阈值在24 h诱导8个家族(图;2C)。这些包括治疗后4小时的多个ERV家族(21),L1 LINEs(6)和DNA转座子(6),以及IFN治疗后24小时的ERV(5)。这些发现表明,多种蝙蝠特异性te在IFN处理后表现出家族水平的转录诱导,在诱导后4小时达到峰值,在诱导后24小时逐渐减弱,但仍然存在。
如前所述[43], TE转录图谱概括了基因组TE含量。当只分析支系特异性te时(图S3.,请参阅方法),我们发现了相似的趋势,转录组组成大致概括了相似年龄TEs的基因组格局,Helitrons、其他sin和L1 LINEs是转录最多的TEs。值得注意的是,最近的TEs(定义为与共识序列差异小于或等于20的TEs;看到方法)平均占转录te的50%,这一关系适用于ERVs、L1 LINEs和大多数DNA元件。因为在蝙蝠辐射开始时,Helitrons在蝙蝠基因组中扩展[18],我们仅在分析最近的te时发现Helitrons的转录水平没有变化。类似地,sin在最近的转录组中占主导地位,最近在基因组中扩展。一个例外是MIR sin,它主要是古代TE,因此在TE转录组中有很高的代表性。令人信服的是,“其他sin”几乎占最近TE转录组的30%(图S3.),但它们只构成基因组的一个更边缘的部分(图。2).这可能是最近在强净化选择下的扩张(即[44]),或支持基因组随机转录模型的假设[43,45].此外,我们发现在干扰素治疗后,近期te的诱导作用很小。这一结果与分析整个TE转录组时观察到的结果形成了鲜明的对比。这里检测到的模式可能是不同的限制因素的基础,在高度调控的生理过程中,这些限制因素专门抑制了最近的和潜在的转位能力TEs的过度转录。
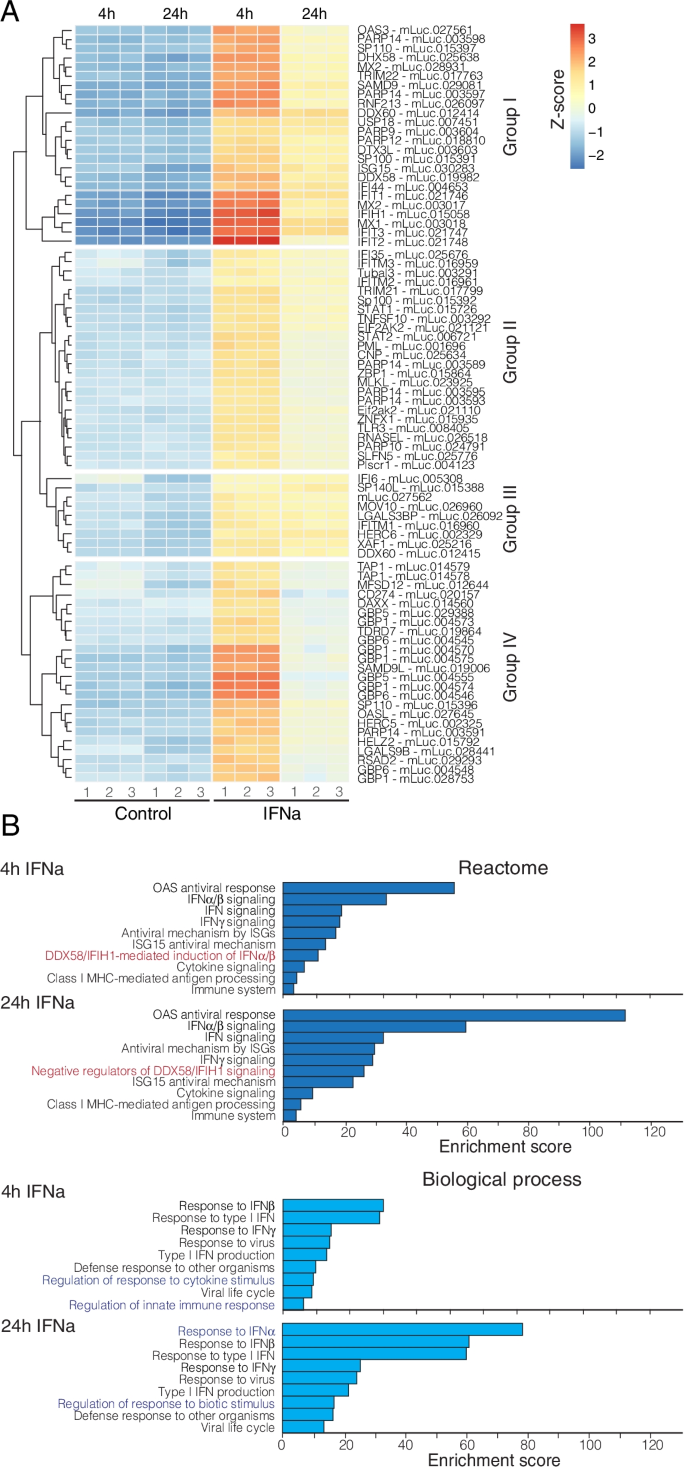
接下来,我们分析了RNA-seq数据集,以识别ifn刺激基因(ISGs),由DESeq2在处理后4小时和24小时成对比较处理后和未处理样品的基因计数确定(表S1和S2).我们首先使用DNAzoo提供的基于同源的注释作为参考转录组。使用adj的截断值。假定值< 0.05和log2FC > 1.5,我们在IFN刺激后4小时鉴定出213个上调转录本(对应138个独特基因)和1个下调基因,在24小时鉴定出91个上调转录本(对应66个独特基因)。根据它们的表达动态,鉴定出4个主要的转录本簇(图。3.A): I)转录本在4小时显示对IFN的强烈反应,在24小时下降;II)转录本在4 h时显示轻度诱导,在24 h时下降;III)显示轻度、稳定诱导的转录物;和IV)转录本在4 h时表现出强烈的诱导,在24 h时迅速下降到与未刺激细胞相似的水平。诱导后4小时和24小时,ISGs均富集了典型ISGs和其他参与免疫信号传导的基因(图2)。3.值得注意的是,我们观察到DDX58/IFIH1 (RIG-I/MDA5)参与的基因在4小时诱导IFNa/ B,随后在24小时诱导该途径的负调控因子(图2)。3.B).同样地,在治疗后4小时检测到与细胞因子刺激反应相关的基因富集,但在治疗后24小时没有检测到。这些观察到强烈的早期反应,随后在IFN刺激后24小时下降,这与黑狐的观察结果一致(狐阿勒克图) [9].
基因表达谱和富集分析。一个热图显示了vsd转换数据中样本间表达方差最高的140个基因(聚类方法:“欧几里德”)。胚胎成纤维细胞随着时间的推移呈现出四种不同的基因诱导动态:i) 24 h快速诱导和下降的基因(i组);ii) 4 h诱导轻度、24 h诱导下降幅度较小或诱导稳定的基因(ii组和III组);iii) 4 h快速诱导、快速下降的基因(IV组)。B柱状图显示了IFNa处理后4小时和24小时差异表达基因的功能过度表达分析(ORA)结果。尽管两个时间点之间的大部分表达是相同的,但我们发现DDX58/IFIH1通路在IFNa刺激后4小时是活跃的,而在24小时是负调控的。同样,对细胞因子刺激的反应出现在治疗后4小时,而不是24小时
TE对ISG转录本结构的贡献
为了提高对可能未注释的ifn诱导的te衍生转录物和异构体的检测,我们使用StringTie2在我们的RNA-seq联合数据集上进行了基因组引导的转录组重组[46](补充资料1),并使用SwissProt数据库根据同源性注释组装的基因。这产生了一个扩展的转录组,包含68,110个stringtie组装的转录本,其中3,2137个转录本异构体与Swissprot中可能的蛋白质编码基因相匹配,其余很可能对应于长非编码rna和蝙蝠特异性基因。在进行成对差分表达式分析后(表S3.)在4 h时,我们鉴定出1243个ifn诱导的转录物,对应836个StringTie基因(740个转录物,对应449个Swissprot中同源匹配的基因);在24 h时,717个转录物,对应500个StringTie基因(385个转录物,对应239个Swissprot中同源匹配的基因)。其中606个转录本对应于392个基因(358个转录本,214个基因匹配Swissprot)在4小时至24小时处理时间内共享。
我们使用我们改进的转录组重组来研究TEs对本构性和ifn诱导的转录结构的贡献。首先,我们根据外显子的重叠(> 50%序列长度)和标注的TE特征识别包含外显子TE的转录本。考虑到所有表达的(TPM≥0.5)多外显子转录本,我们发现1039个转录本对应于749个StringTie基因(648个转录本和470个匹配Swissprot基因),这些转录本包含至少一个te衍生的外显子(表S .)4).
首先关注从组成性表达基因重建的转录本,我们确定EEF1A1例如,271 bp(代表第三和第四外显子的一部分;编码外显子2和3)被注释为源自Zisupton DNA转座子。Zisupton DNA转座子在小鼠中并不丰富m . lucifugus基因组(我们的掩蔽只恢复了一个有174个拷贝的家族)。它们有可能水平转移到蝙蝠基因组中,就像在多种鱼类中发生的那样[47].对Zisupton基因组拷贝的年龄分析表明,它们可能是最近引入的(Kimura 2距离共识序列~ 18),但只能扩展很短的一段时间(与共识序列的分歧峰值在4-9),当时其他DNA元件已经活跃。我们通过多次BLAST搜索(补充数据)进一步验证了这一发现2).首先,我们对齐EEF1A1将StringTie转录本(STRG.21546.2, Scaffold 10: 12654609-12,761,039)添加到NCBI数据库(XM_006089332.3)上的转录本序列。在确认两个转录本之间的匹配后,我们使用沉积的mRNA序列作为对Repbase转座元素数据库的查询[48以及对抗蝙蝠和哺乳动物mRNA的收集。所有小蝙蝠亚目,蝙蝠科特别是共享高度同源的编码外显子3和4(补充数据3.),表明该事件发生在事件发生之前或期间小蝙蝠亚目而且Pteropodinae辐射。尽管有共同的同源性,但在人类或其他真肢动物身上没有发现Zisupton匹配特征EEF1A1同系物。因此,我们的分析成功识别了蝙蝠EEF1A1作为一个基因可能被蝙蝠特有的TE改变的例子。
接下来,我们筛选含有te衍生外显子的ifn诱导转录本,发现在4小时诱导的34个基因(其中16个用SwissProt同源性注释)中有44个转录本,在24小时诱导的24个基因(11个注释)中有31个转录本(表s .)4).这些基因包括在免疫反应中具有既定作用的基因,比如PARP9,SLFN5(无花果。4A)和一个候选的新异构体IFITM2之前未被发现(图S4B).该分析揭示了许多组成性表达和ifn诱导基因在编码区或utr中从蝙蝠特异性te中获得了外显子(表1).
我们接下来的目标是确定增选te衍生蛋白的潜在例子(如合胞素[49];)可能不会被RepeatMasker屏蔽,因为他们的年龄或低拷贝数。我们使用tblastx根据反转录转座子和DNA元件特异性的参考TE蛋白序列文库查询表达的StringTie转录本(见方法).根据串联的StringTie转录组组装,我们发现总共有7个StringTie基因具有已知的注释同源谱,156个StringTie基因基于完整的ORF预测(表S .)5;方法)。通过使用完整ORF预测方法,我们确定了9个isg(即:1英镑,DDX58而且PARP14),至少有一个逆转录转座子衍生的特征,主要来自ERVK和L1 LINEs。这些te衍生序列大多位于最后一个外显子,在那里它们提供了停止密码子和3'UTR,或新的编码和/或调控序列.最后,我们采用同样的方法来识别与已知病毒蛋白匹配的新的蛋白编码序列,这些蛋白编码序列可以代表驯化的病毒蛋白[50].通过利用gEVE和自定义合胞素蛋白数据库,我们发现总共有3个组成型表达的StringTie基因与合胞素蛋白同源,9个与pol、gag、逆转录转座酶或AP病毒蛋白同源(表S .)6).只有一个pol (RVT1)衍生基因,UBP18,在IFN处理后表达差异。
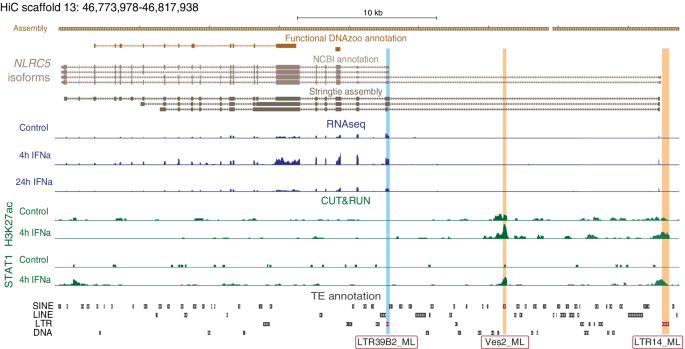
除了研究TE对蛋白质编码序列的贡献外,我们还搜索了TE衍生启动子的例子。我们将StringTie转录本坐标折叠到它们的转录起始位点(TSS),并将转录本TSS与生成的TE注释相交。我们共鉴定出11个TSS衍生自TE的转录本,这些转录本在4小时内可被ifn诱导,其中9个与24小时子集共享(表S7).其中大部分属于已知参与免疫功能和调节的基因,比如NLRC5(无花果。4),EIF2AK2,1英镑,mx₁,(,PHF11,SAMD9而且XAF1,而其他人则喜欢PARP14而且CS012都是非规范的isg。值得注意的是,我们还观察到te衍生的启动子在支架13和20的组织相容性位点上的反复使用,其中4小时时78个转录本(对应21个注释基因),24小时时7个转录本(对应3个注释基因)具有te衍生的tss或编码外显子。虽然组织相容性位点可能容易出现与其高度多态和复杂结构相关的序列组装伪像,但我们的分析表明,TEs可能影响了蝙蝠组织相容性位点的进化,这被认为是蝙蝠特异性免疫功能的基础[51,52].
干扰素刺激的表观遗传谱
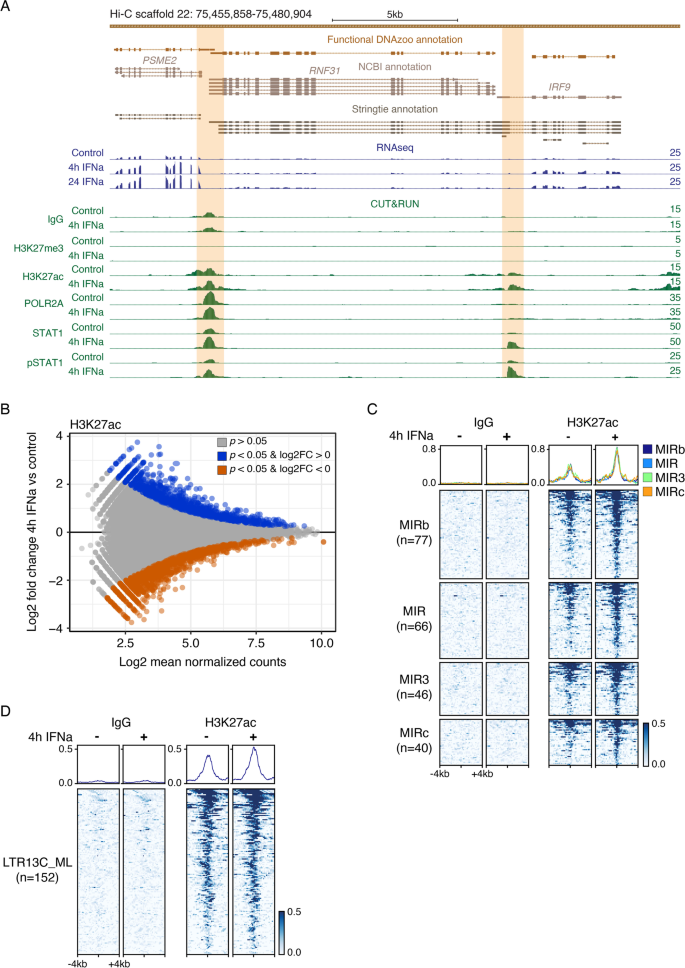
在分析了TEs对isg的贡献后,我们接下来询问TEs如何对由H3K27ac、POLR2A或STAT1活性定义的可诱导调控元件做出贡献,以响应IFN。我们在预测的启动子处观察到STAT1信号的增加IRF9而且PSME2对I型IFN的反应(图;5A).使用0和4小时的峰值归一化CUT&RUN数据,我们使用DESeq2定义ifn诱导的调控元件,如前所述[29].出乎意料的是,我们没有观察到来自其他物种的ifn刺激细胞具有的强大的ifn诱导的染色质变化[29,30.].我们没有发现任何虚假发现率(FDR) < 0.05的H3K27ac诱导元素。5B;表的年代8),定义了一组1113个元素,显示H3K27ac信号增加,显著性阈值放宽为未调整假定值< 0.1(图S5A).该组ifn诱导元件富集干扰素刺激反应元件(ISRE)基序(e值1.43 × 10)−10)(表S9),与IFN刺激的激活相一致。因此,虽然我们的CUT&RUN分析成功地识别出了一些显示ifn诱导活性的元素,但我们的分析显示了令人惊讶的适度的染色质水平变化,尽管根据RT-qPCR从取自相同部分细胞的RNA中获得了强有力的ISG诱导(图S .)1).
未处理和ifna刺激的胚胎成纤维细胞原代细胞的表观基因组分析。一个基因组浏览器的视图IRF9而且PSME2位点。RNA-seq和CUT&RUN轨迹每百万次读取被标准化。信号轨迹最大值显示在每条轨迹的右侧。IFNa-inducible (假定值< 0.10, log2FC > 0) STAT1峰值以橙色突出显示。Bifna诱导的MA图(未调整假定值< 0.10, log2FC > 0,蓝色)和ifna - suppressed(未调整假定值< 0.10, log2FC < 0,橙色)H3K27ac区域从H3K27ac CUT&RUN。地区与未调整假定值> 0.10用灰色表示。使用apeglm函数v1.8.0对Log2次折叠变化值进行收缩[41].C热图显示正常化的CUT&RUN信号(每百万次读取的信号)超过ifna诱导(未调整)p-val < 0.10), h3k27ac标记的MIR家族。D热图显示h3k27ac标记的LTR13C_ML族的标准化CUT&RUN信号(每百万次读取的信号)
在这些区域中,我们发现1113个区域中有466个完全重叠至少一个TE(表S10).此外,我们确定了766个可诱导的stat1结合TEs,它们位于ISG的100 kb范围内(表S11).这包括一个LTR14_ML元素,它可以作为的启动子NLRC5除了内含子Ves2_ML的sin元件外,还有一个基因座。4).然而,与之前对其他物种的研究相比,[29,30.,31,32,33],在这个集合中我们没有观察到任何TE家族的过高代表(图S5B;表的年代12).唯一重叠超过10个ifn诱导的H3K27ac区域的亚家族对应于蝙蝠进化之前的古代哺乳动物MIR和L2家族(图2)。5C;无花果。5C)。独立查询未处理或ifn刺激条件下的H3K27ac区域,我们观察到MIR3、AmnSINE1和LTR13C_ML家族的富集程度非常低(图2)。5D;无花果。6).综上所述,我们的分析表明TE贡献了数百个参与IFN信号传导的调控元件,但与其他物种的研究相比,我们没有在IFN诱导的调控元件中发现谱系特异性TE家族的全基因组富集。然而,鉴于我们的CUT&RUN分析揭示了全基因组水平上的诱导调控元件相对较少,我们识别富集TE家族的能力受到限制。
讨论
我们的研究描述了蝙蝠TEs中IFN反应的转录和表观遗传动力学m . lucifugus细胞。为了促进我们在功能基因组数据中绘制TEs的能力,我们使用150 bp的配对端读进行了RNA-seq和CUT&RUN,并使用染色体规模的组装生成了改进的重复注释。我们的分析显示,TEs塑造了ifn诱导的转录组,但我们没有发现强有力的证据证明TEs在塑造相关表观遗传变化方面的全球作用。功能性研究将是必要的,以验证本研究中确定的任何元素是否对蝙蝠免疫具有意义,但考虑到在人类和小鼠中验证的例子越来越多,一些TEs很可能已被用于蝙蝠的先天免疫功能。
在我们的研究中,我们生成了匹配的转录组和表观基因组数据集,分析了I型干扰素的反应m . lucifugus主细胞。我们的IFN反应的转录组分析m . lucifugus胚胎成纤维细胞证实了先前报道的蝙蝠先天免疫的特征。我们发现这些细胞在转录水平上对IFN刺激有反应,在较早的时间点(治疗后4小时)对ISGs有更强和更广泛的诱导。我们还发现,只有一小部分在4小时过表达的基因在24小时保持高表达水平,而大多数基因的表达降低到较低水平或与未刺激细胞中记录的水平相似。这与基因表达谱是一致的狐阿勒克图[9].与基因表达平行,我们描述了te衍生转录本的表达谱,并发现了类似的趋势。在IFN处理后4小时,TE的总表达量更高,与未受刺激和24小时的细胞相比,TE家族的差异表达越来越多。虽然这些转录本的表达并不能直接指示功能,但ifn诱导的蝙蝠特异性TE家族的表达可能作为非编码转录本的来源,可以进一步激活先天免疫途径,类似于人类癌症中特征的“病毒拟态”途径[26,27].间接支持TEs可能参与先天免疫的假设,来自于观察,尽管TE的总转录组在组成上遵循TE的基因组内容,但对于差异表达的TE家族来说,这是不正确的。由于我们的分析不能区分TE在单个位点水平上的表达,有可能一个特定TE家族中只有少数成员过表达,而整个家族则不是。总之,很可能存在ifn响应序列特异性特征(即ifn响应启动子)鼠耳蝠属.
我们还通过筛选编码区和转录起始位点(TSS)中与转座元件或病毒蛋白具有同源性的基因转录本,探讨了特异性TEs是否可能影响宿主基因的转录本结构。我们的基因组引导转录组组装确定了多个te衍生和病毒蛋白衍生的exonization事件实例,两者都是可选的(IFITM2)或保守(PARP9)外显子和TSS (NLRC5).其中一些转录本代表规范的(PARP9,DDX60)或非规范(PLAAT3,SP140Lisg。这些分析提供了强有力的证据,证明TEs已被吸收到蝙蝠isg的外显子中,其中一些外显子化事件可能具有重要的功能后果。例如,我们的分析确定了多个鼠耳蝠属特定的TE插入和exonization事件NLRC5基因。NLRC5已被确定为MHC i类依赖免疫反应的关键调节因子[53],并可能参与炎性小体激活和I型IFN反应的调节[54].需要进一步的研究来验证这些te衍生序列的潜在影响,但有可能鼠耳蝠属特异性TEs改变NLRC5功能和/或调节。
我们还鉴定了te衍生的组成性表达基因的实例。我们通过多次BLAST和序列比对验证了EEF1A1蛋白的~ 100个氨基酸小蝙蝠亚目而且很可能Pteropodinae从一个Zisupton DNA转座子中获得的蝙蝠。我们分析发现的这个例子强调了TEs除了参与免疫功能的基因外,还塑造了蝙蝠生物学的其他方面的可能性。
虽然RNA-seq分析已被越来越多的研究应用于转录组水平的蝙蝠免疫分析,但迄今为止还没有研究在表观基因组水平上描述蝙蝠免疫。出乎意料的是,我们的rna测序分析m . lucifugus细胞与IFN处理的强烈转录反应相吻合,我们根据CUT&RUN表观基因组分析观察到相对温和的染色质变化。这一观察结果与其他哺乳动物ifn处理细胞中通常观察到的稳健染色质变化形成对比[29,55,56].因此,我们没有观察到强有力的证据支持家族水平的TE调节诱导,如之前在其他物种中观察到的,如人类[30.]和cow [29],部分原因是在全基因组水平上缺乏明确诱导的元素。
对于我们在蝙蝠细胞中观察到的相对温和的ifn诱导的表观遗传反应,有多种可能的解释。首先,我们的研究是最早在蝙蝠细胞上进行CUT&RUN的研究之一,本研究中使用的抗体还没有完全得到验证m . lucifugus.然而,虽然用于STAT1的抗体可能对蝙蝠ortholog的识别能力较差,但组蛋白和POLR2A高度保守,有望被标准抗体有效靶向。其次,我们的研究涉及用重组通用型I IFN刺激衍生的胚胎成纤维细胞培养。尽管这些条件仍然显示出强烈的ISGs转录诱导,但在体内IFN反应的内源性激活过程中,染色质动力学可能是不同的.
最后,我们的观察可能反映了蝙蝠免疫的一个独特属性,与一些蝙蝠物种表现出构成性IFN表达的想法一致[7,57].虽然I型IFN基因座m . lucifugus虽然特征不明显,但我们能够注释至少11个未特征的基因,这些基因可能反映了IFNw簇的基因重复扩展(图S7) [58],而没有发现IFNa基因。在11个IFNw序列中,5个在未受刺激的细胞中显示出明显的低表达,并在处理后4小时诱导(图S7).这些观察结果表明鼠耳蝠属由于IFN的组成性表达,表观基因组被“启动”,并且可能能够驱动强大的转录激活,而不表现出通常与诱导染色质活性相关的表观遗传变化,例如H3K27ac或POLR2A水平的增加。
结论
我们的研究首次系统地研究了TEs对蝙蝠I型IFN反应的贡献。我们发现了大量te衍生转录本、替代外显子和调控元件的例子,它们塑造了基因组对IFN的反应m . lucifugus.我们的研究表明,TEs在其他蝙蝠谱系中,如狐蝠而且Rhinolophus也可能形成ifn诱导的转录组,这可能会激发功能研究,以确定它们在蝙蝠免疫方面的生物学意义。我们的发现为TE增效在形成物种特异性免疫反应进化中的广泛作用提供了额外的支持。
方法
转座因子鉴定与分析
鼠耳蝠lucifugus根据同源性和从头鉴定方法对基因组重复元件进行了注释。我们使用RepeatModeler2(包含在dfam-tetools-1.1中)执行从头TE识别[36]和HelitronScanner v1.1 [37]以匹配新发布的高度连续的小棕色蝙蝠基因组染色体水平组装(myoLuc2_HiC) [34,35,59,60].基因组组装依赖于长读组装策略(即HiC [34,61])更适合在连续的染色体水平支架上捕获全长元素。
简单地说,我们使用RepeatModeler和HelitronScanner执行从头TE识别,然后将这两个库合并为一个小棕色蝙蝠从头库,使用RepeatMasker v4.1.0用于基于同源的TE注释[39].为了最大化元素识别,我们遵循了自定义的多步映射策略[62使用多个库作为屏蔽过程的参考,顺序如下:(i)包含在RepeatMasker提供的Repbase库中的bat特定重复;(ii)科斯比博士提供的蝙蝠专用库[19];(iii)我们新建的小棕蝙蝠图书馆;(iv)由RepeatMasker提供的整个四足动物Repbase库[48].
细胞系和治疗
鼠耳蝠lucifugus原代胚胎成纤维细胞是Mario Capecchi(犹他大学)的礼物。细胞在37°C和5% CO下生长2并在DMEM (ThermoFisher #10566016)中传代,添加10%胎牛血清、5% MEM非必需氨基酸、100 U/mL青霉素-链霉素和1 mM丙酮酸钠。将细胞以2 × 10的优化密度播种到6孔板中5每孔细胞在2毫升培养基中(或2 × 106每15厘米培养皿细胞切割和运行)。第二天(或CUT&RUN 48小时)细胞用1000u /ml的通用型I IFNa重悬在DPBS (PBL Assay Science #11200)中的2 ml培养基中;对照组细胞用等量DPBS在2 ml培养基中处理。在处理后4和24小时收集细胞进行RNA提取(4小时用于CUT&RUN)。
RNA测序的RNA分离和文库制备
去除培养基后,用1ml DPBS清洗细胞,每孔加入400ul 0.25%胰蛋白酶分离细胞。在37°C孵育10分钟后,用1.6 ml培养基中和胰蛋白酶。细胞悬液转移到1.7 ml试管中,在300×g离心5分钟制成颗粒。然后按照制造商的说明,在300ul RNA裂解缓冲液(zyymo Research #R1060-1-50)中裂解细胞,并在−80°C下存储,直到使用Quick-RNA MiniPrep试剂盒(Zymo Research #R1054)进行RNA提取。
如上所述,在三个生物重复中制备每个时间点和条件的总RNA样本。NanoDrop One分光光度计(赛默飞世尔科学公司)用于测定RNA浓度和质量;所有样品均通过质量评估。根据制造商的协议,使用KAPA mRNA HyperPrep Kit (KAPA Biosystems #8098115702)进行PolyA富集和文库制备。简单地说,使用500 ng RNA作为输入,并以最终浓度为10 nM添加单指标适配器(Kapa Biosystems #08005699001)。纯化的,适配器连接的文库按照制造商的方案被扩增了总共11个周期。最终的文库在Illumina NovaSeq 6000 (University of Colorado Genomics Core)上进行汇总和测序,成对末端reads为150 bp。
CUT&RUN样本和库的准备
CUT&RUN下拉使用[]协议生成63,64].所有的缓冲液都是根据“高钙2+/低盐”部分的协议使用0.04%洋地黄苷(EMD Millipore #300410)。5 × 105每一次下拉都使用活细胞。使用以下抗体:兔抗小鼠IgG (1:100, Abcam #ab46540),兔抗h3k27me3 (1:100, Cell Signaling #9733),兔抗h3k27ac (1:100, Millipore #MABE647),兔抗prpb1 - ser5 (1:50, Cell Signaling #135235S),兔抗stat1 (1:100, Cohesion #3322),兔抗pstat1 - ser727 (1:100, Active Motif #39634)。pagg - mnase(制备方法为[63,64]),以最终浓度为700 ng/mL的一抗孵育后添加到每个样品中。染色质消化、释放和提取根据[63,64].将酵母尖刺DNA (Steven Henikoff赠送)添加到淬灭(“1× STOP”)缓冲液中,最终浓度为100 pg/mL。在进行文库准备之前,通过Qubit dsDNA High Sensitivity和TapeStation 4200 HSD5000确定下拉成功。
库是使用KAPA HyperPrep Kit使用的修改协议生成的。简单地说,每次下拉的全部容量(50 uL)用于根据制造商的协议生成库,并进行了以下修改。新稀释的0.200 uM单指标适配器(Kapa Biosystems #08005699001)以低浓度(9 nM)添加到每个库中,以最大限度地减少适配器二聚体的形成。适配器连接库使用KAPA Pure Beads (KAPA Biosystems #07983280001)进行双面0.8X/1.0X清理。使用以下PCR循环条件扩增纯化的适配器连接文库:98°C 45 s, 15 ×(98°C 15 s, 60°C 10 s), 72°C 60 s。放大库进行了双面0.8X/1.0X清理。最终的文库使用Qubit dsDNA High Sensitivity和TapeStation 4200 HSD5000进行量化。文库在Illumina NovaSeq 6000 (Novogene)上合并并测序,成对端reads为150 bp。
成对RT-qPCR
5 × 105从相同的CUT&RUN群体(未处理和4小时IFN)中提取活细胞RNA进行RT-qPCR分析,以确认在CUT&RUN文库制备之前IFN诱导基因的诱导。细胞在300ul RNA裂解缓冲液中裂解(Zymo Research, #R1060-1-50)。按照制造商的说明,将制备好的裂解物存储在−80°C,直到使用Quick-RNA MiniPrep试剂盒(Zymo Research #R1054)进行RNA提取。
如上所述,在三个生物重复中制备每个时间点和条件的总RNA样本。NanoDrop One分光光度计(赛默飞世尔科学公司)用于测定RNA浓度和质量;所有样品均通过质量评估。RNA表达水平CTCF,STAT1,IFIH1根据制造商说明书,使用Luna Universal One-Step RT-qPCR试剂盒(New England Biolabs #E3005L)进行定量。简而言之,对于每个反应,25ng RNA与5ul 2× Luna Universal One-Step reaction Mix、0.5ul 20× Luna WarmStart RT Enzyme Mix、0.4ul 10uM正向引物和0.4ul 10uM反向引物结合。使用CFX384触摸式实时PCR检测系统(Bio-Rad)扩增反应,PCR循环条件如下:55℃10分钟,95℃1分钟,40次(95℃10秒,60℃30秒)。通过熔体曲线分析评估靶上扩增。每种处理条件设两个生物重复,每个生物重复进行技术重复。使用阈值为的双尾配对Student 's t检验评估统计学意义假定值< 0.05。
转录组分析
使用BBDuk v.38.05对端150 bp读长度的FASTQ文件进行质量和适配器修剪[65];使用FastQC v0.11.8进行质量检查[66]并通过MultiQC v1.7检验[67].然后在STAR v2.7.3a中使用2-pass方法将过滤后的FASTQ文件映射到myoLuc2_HiC基因组[68].STAR按照默认参数运行,并允许多映射读取(带有选项-winAnchorMultimapNmax 100 -outFilterMultimapNmax 100’),这是在输出文件中包含TE-mapping读取的必要条件。DNAzoo上可用的注释文件,这里称为“功能注释”(www.dropbox.com/sh/xt300ht42mihjov/AADoENW7RTvR3jTh1a8qUOmRa)作为映射过程的参考。在第二轮绘图中,我们过滤出了映射到线粒体基因组的新连接hic_scaffold d_93,该连接基于使用LASTZ v1.02.00[对参考线粒体基因组进行的最有可能的对齐命中。69].结果对齐文件已排序。bam然后提供TETranscripts v2.1.4中TE和基因表达量化的输入[42]使用相同的基因注释和来自RepeatMasker的自定义TE注释。为了分析分支特定的TE表达式,我们过滤了自定义TE注释文件,只包含Kimura2D < = 20的TE。在IFN处理后4小时和24小时,对DESeq2 v1.32进行成对差异表达分析[70].差异表达基因的功能富集分析。假定值< 0.05, log2FC > 1.5)使用WebGestalt web工具[71].
基因组引导转录组组装和分析
运行STAR生成的短读RNA-seq比对文件(见前段)使用SAMtools v1.10进行合并、排序和索引[72],结果。bamStringTie v1.3. 3.3b中基因组引导转录组组装的输入文件[46].StringTie是按照默认参数运行的,除了在连接上对齐所需的最小拼接读取数量从1增加到5使用选项'- j 5”。生成的StringTie gtf输出(补充数据1)转换为FASTA格式gffread使用选项的实用程序-M, -F和-Z袖扣v.2.2.1 [73].我们遵循基于同源的方法来注释组装的StringTie基因和异构体。通过SwissProt数据库查询StringTie基因序列[74]通过blastxBLAST 2.12.0+ [75使用选项”-max_target_seqs 1 - value 10”。然后筛选匹配的共享序列标识等于或大于50%。
为了找到与TEs相交的编码区域的转录本,我们应用了BEDTools v2.28.0 [76一个外显子至少50%的序列来源于TE的事件- f 0.5”。简单地说,从StringTie注释中提取外显子坐标,并与我们的自定义TE注释相交鼠耳蝠lucifugus.为了缩小候选StringTie转录本列表并限制假阳性匹配的冗余,输出过滤了百万分之转录本(TPM)值等于或高于0.5的多外显子转录本。采用RSEM v1.3.0在异构体水平定量TPM值[77].筛选后的StringTie候选转录本与DESeq2 v1.32中差异表达分析的结果进行交叉引用[70](表S3.)以识别isg中的TE免除事件。候选isg通过与DNAzoo和NCBI注释进行视觉检查来验证鼠耳蝠lucifugus以及RNA-seq覆盖轨迹。为了识别可能有助于替代转录起始位点(TSS)的TE,我们使用一个自定义的python脚本从StringTie注释中提取TSS坐标,并如前所述将这个折叠文件与TE注释文件交叉。
最后,我们通过从TE参考序列的Repbase库中提取的DNA转座子和反转录转座子数据库查询stringtie衍生的组装转录本[48].tblastx根据先前指定的参数使用,并进一步筛选比对大于或等于300 bp且序列恒等式大于或等于90%的结果。为了识别任何带有完整的病毒来源蛋白质编码序列的转录本,我们运行blastxi) gEVE逆转录病毒蛋白库[78]和ii)从ncbi资料库收集的合胞体蛋白自订资料库[79].对gEVE冲击波的输出进行筛选,寻找大于或等于200 bp且共享序列单位大于或等于50%的比对。对syncytin blast的输出进行筛选,筛选出大于100 bp且共享序列同一性为50%及以上的比对。对TE序列和蛋白质数据库进行了相同的blast分析,并对运行该函数发现的大于50aa的开放阅读帧(orf)进行了分析usearch[80) (Usearch -fastx_findorfs -orfstyle在StringTie汇编文本文件上。
急忙逃走的分析
适配器和低质量读取使用BBDuk v38.05进行修剪[65使用选项' ktrim=r k=34貂=11 hdist=1 tpe tbo qtrim=r trimq=10”。使用Bowtie 2 v2.2.9对myoLuc2_HiC程序集进行对齐[81有选择的——局部-非常敏感-局部-不统一-不混合-不不协调-I 10 -X 700,并且只保留MAPQ最小为10的唯一映射读取。对准线粒体基因组的片段被移除。对象独立对齐的已修剪读取酿酒酵母组件(GCF_000146045.2)使用Bowtie 2 v2.2.9 [81有选项的——局部-非常敏感-局部-不统一-不混合-不不协调-不重叠-不吻合”。myoLuc2_HiC读深度根据对齐到的片段数进行归一化酿酒酵母使用deepTools v3.0.1生成对应于每100万个规范化读的读覆盖率的规范化bigWigs [82,83]以显示热图。
使用MACS2 v2.1.1的完整和大小的子对齐文件执行峰值调用[84]在两步过程中,使用单端选项分别调用不同的峰值组。——format BAM——shift=-75——extsize=1502)配对端选项- - -格式BAMPE”。对于两种模式只有峰值与未调整假定值< 0.01保留。每个模式的峰值随后被合并。从每个下拉峰值集中减去IgG峰值以最小化背景。仅保留MACS2峰值评分下降的前2万个峰值进行进一步分析。
为了识别ifn诱导的cut和run峰,在特定的下拉(所有重复,未经处理和ifn刺激)中,所有样本的前20,000个峰被连接到一个单一的列表中,并使用BEDTools v2.28.0 [76].使用DESeq2 v1.26.0称为ifn诱导峰[70],但我们无法确定FDR < 0.10的IFN响应显著上调的峰值。因此,我们采取了一种更轻松的方法,保留了所有未调整的峰值假定值< 0.10和log2FC > 0。使用apeglm函数v1.8.0收缩log2FC值[41]的可视化。Motif分析使用XSTREME v5.4.1 [85有选项的’——minw 6——maxw 20——streme-motifs 20——对齐中心’查询JASPAR CORE 2018脊椎动物数据库[86].
为了评估TEs在调节IFN反应中的作用,我们将IFN诱导的H3K27ac峰值与所有注释的te交叉,要求所有报告的te完全包含在峰值内。我们使用BEDTools v2.28.0[通过将stat1标记的TEs识别为到最近的ISG (FDR < 0.05, log2FC > 1.5)转录起始位点距离的函数,进一步描述了调控景观[76].为了评估家庭水平的富集,GIGGLE v0.6.3 [87]使用上述自定义TE数据库创建myoLuc2_HiC基因组中的TE数据库。然后在TE数据库中查询ifn诱导的H3K27ac峰。结果按Giggle富集评分递减进行排序,富集TE家族根据优势比(Fisher 's two-tailed)确定假定值,和重叠数。TE热图是通过在不同家族中选择重叠IFN诱导的H3K27ac区域或未处理或IFN条件下的任何H3K27ac区域的元素来制备的。信号从酿酒酵母使用deepTools v3.0.1绘制峰值、CPM标准化大腕的热图[82,83].
数据和材料的可用性
新生成的RNAseq和CUT&RUN原始文件已保存在GEO SuperSeries接入GSE200833下。经过处理的数据,包括TE注释,可以在这里可视化为UCSC基因组浏览器自定义跟踪:https://genome.ucsc.edu/s/GiuliaPasquesi/myoLuc2_HiC.
参考文献
Gorbunova V, Seluanov A, Kennedy BK.世界像蝙蝠一样:更长寿,更能忍受病毒。Cell Metab, 2020; 32:31-43。
Luis AD, Hayman DTS, O 'Shea TJ, Cryan PM, Gilbert AT, Pulliam JRC,等。蝙蝠和啮齿动物作为人畜共患病毒宿主的比较:蝙蝠是特殊的吗?中国生物工程学报,2013;280:20122753。
Schuh AJ, Amman BR, Sealy TK, Spengler JR, Nichol ST, Towner JS。埃及花环蝙蝠对马尔堡病毒感染保持长期的保护性免疫,尽管抗体水平下降。科学代表2017;7:8763。
李文杰,李志强,李志强,等。翼类蝙蝠被确认为亨尼帕病毒的宿主库:病毒传播的综合实验研究。中华热带医学杂志,2011;85:946-51。
王志强,王志强,王志强,等。用伊波拉病毒实验接种动植物。《新兴感染杂志》1996;2:21 1 - 5。
Munster VJ, Adney DR, van Doremalen N, Brown VR, Miazgowicz KL, Milne-Price S,等。中东呼吸综合征冠状病毒在牙买加果蝠体内的复制和脱落(Artibeus jamaicensis).科学代表2016;6:21878。
周鹏,Tachedjian M, Wynne JW, Boyd V,崔杰,Smith I,等。蝙蝠I型IFN位点的收缩和IFN-α的异常本构表达。中国科学院学报(自然科学版),2016;
肖爱华,张志刚,顾志刚,张志刚,等。通过I型干扰素反应的多物种比较揭示了哺乳动物先天免疫系统的基本特性。科学通报,2017;15:e2004086。
De La Cruz-Rivera PC, Kanchwala M, Liang H, Kumar A,王丽芳,邢晨,等。蝙蝠的IFN反应显示出独特的IFN刺激的基因表达动力学与非典型性RNASEL诱导。中华免疫杂志,2018;30(4):344 - 344。
Escalera-Zamudio M, Zepeda-Mendoza ML, Loza-Rubio E, Rojas-Anaya E, Méndez-Ojeda ML, Arias CF,等。蝙蝠核酸感应toll样受体的进化。分子生物学学报。2015;24:5899-909。
安敏,崔杰,王丽峰。哺乳动物中蝙蝠PYHIN基因家族的独特损失:对炎性小体感测的影响。科学代表2016;6:21722。
安敏,安德森·德,张强,谭长波,林伯林,卢科,等。在蝙蝠中抑制nlrp3介导的炎症及其对特殊病毒库宿主的影响。《微生物学报》2019;4:789-99。
谢娟,李勇,沈鑫,高光,朱勇,崔健,等。蝙蝠sting依赖性干扰素激活的抑制。细胞宿主微生物,2018;23:297-301.e4。
柳根哈根A, Sugrue E, Lytras S, Kuchi S, Noerenberg M, Turnbull ML,等。烯丙化dsRNA传感器可预防严重的COVID-19。科学。2021;374:eabj3624。
Ray DA, Feschotte C, Pagan HJT, Smith JD, Pritham EJ, Arensburger P,等。最近蝙蝠DNA转座子活动的多波,鼠耳蝠lucifugus.基因组决议2008;18:717-28。
grzija I, Messing SA, Thomas J, Cosby RL, Bilic I, Miskey C,等。从蝙蝠中重建的Helitron转座子揭示了真核生物基因组改组的新机制。Nat Commun. 2016;7:10716。
Mitra R, Li X, Kapusta A, Mayhew D, Mitra RD, Feschotte C,等。piggyBat的功能描述鼠耳蝠lucifugus揭示了一个活跃的哺乳动物DNA转座子。中国科学院学报(自然科学版),2013;
Pritham EJ, Feschotte C.蝙蝠谱系中滚动圈转座子的大量扩增鼠耳蝠lucifugus.中国科学院学报(自然科学版),2007;
张锐,张晓东,张晓东,张晓东,等。通过转座酶捕获脊椎动物转录因子的反复进化。科学。2021;371年。https://doi.org/10.1126/science.abc6405.
Skirmuntt EC, Escalera-Zamudio M, Teeling EC, Smith A, Katzourakis A.内源性病毒元件在蝙蝠进化中作为人畜共患病毒宿主的潜在作用。2020年修订版;可以从:https://www.annualreviews.org/doi/abs/10.1146/annurev-virology-092818-015613.
skskirmuntt EC, Katzourakis A.蝙蝠内源性逆转录病毒包膜基因的进化及其对宿主生物学的潜在贡献。病毒决议2019;270:197645。
冯志刚,冯志刚。内源性病毒序列对宿主细胞功能的增选。《中国医学杂志》2017;25:81-9。
Mura M, Murcia P, Caporale M, Spencer TE, Nagashima K, Rein A,等。内源性逆转录病毒转显性gag诱导的晚期病毒干扰。中国科学院学报(自然科学版)2004;101:11117 - 1122。
Young GR, Yap MW, Michaux JR, Steppan SJ, Stoye JP。逆转录病毒限制基因Fv1的进化之旅。中国科学院学报(自然科学版)2018;115:10130-5
布兰科-梅洛D,吉福德RJ, Bieniasz PD。原始人类祖先中用于宿主防御的内源性逆转录病毒包膜的增选。Elife。2017;6。https://doi.org/10.7554/eLife.22519.
Chiappinelli KB, Strissel PL, Desrichard A, Li H, Henke C, Akman B,等。抑制DNA甲基化通过dsRNA(包括内源性逆转录病毒)在癌症中引起干扰素反应。细胞。2015;162:974 - 86。
王勇,王志强,王志强,王志强,等。dna去甲基化剂通过内源性转录物诱导病毒拟态靶向结直肠癌细胞。细胞。2015;162:961 - 73。
Schmidt N, Domingues P, Golebiowski F, Patzina C, Tatham MH, Hay RT,等。流感病毒触发的SUMO开关协调增选的内源性逆转录病毒刺激宿主抗病毒免疫。中国科学院学报(自然科学版)2019;116:17399-408。
陈丽丽,陈丽娟,陈丽娟。反刍动物特异性反转录转座子形成牛免疫的调节进化。bioRxiv。2021:2021.10.01.462810可从:https://www.biorxiv.org/content/10.1101/2021.10.01.462810.引用2022年2月25日。
张志勇,张志勇,张志勇,等。内源性逆转录病毒对先天性免疫调节进化的影响。科学,2016;351:1083-7美国科学促进会。
Srinivasachar Badarinarayan S, Shcherbakova I, Langer S, Koepke L, Preising A, Hotter D,等。HIV-1感染激活内源性逆转录病毒启动子,调节抗病毒基因表达。核酸res 2020; 48:10890-908。
van de Lagemaat LN, Landry J-R, Mager DL, Medstrand P.哺乳动物转座因子促进具有专门功能的基因的调节变异和多样化。趋势,2003;19:530-6。
Bogdan L, Barreiro L, Bourque G.转座因子在细菌感染时被激活。中国生物医学工程学报(英文版);
Dudchenko O, Batra SS, Omer AD, Nyquist SK, Hoeger M, Durand NC,等。从头组装的埃及伊蚊使用Hi-C的基因组产生染色体长度支架。科学。2017;356:92-5。
Myotis_lucifugus。DNA动物园。可以从:https://www.dnazoo.org/assemblies/Myotis_lucifugus.引用2022年6月7日。
费林JM, Hubley R, Goubert C, Rosen J, Clark AG, Feschotte C,等。RepeatModeler2用于自动发现转座因子家族的基因组。中国科学院学报(自然科学版)2020;
熊伟,何磊,赖军,杜春华,杜春华。HelitronScanner在许多植物基因组中发现了一个被忽视的Helitron转座子。中国科学院学报(自然科学版),2014;
使用和理解RepeatMasker。方法Mol biology 2012; 859:29-51。
RepeatMasker主页。可以从:http://www.repeatmasker.org.引用2022年4月12日。
Ray DA, Pagan HJT, Thompson ML, Stevens RD.带有hATs的蝙蝠:Myotis属近期DNA转座子活性的证据。分子生物学杂志,2007;24:632-9。
朱A,易卜拉欣JG,爱米。序列计数数据的重尾先验分布:去除噪声和保留大差异。生物信息学。2019;35:2084 - 92。
金勇,吴婷,潘亚agua E, Hammell M. TEtranscripts:一种在RNA-seq数据集差异表达分析中包含转座因子的包。生物信息学。2015;31:3593-9。
Pasquesi GIM, Perry BW, Vandewege MW, Ruggiero RP, Schield DR, Castoe TA。脊椎动物谱系在不同的组织中表现出不同的转座因子调节和表达模式。中国生物科学。2020;12:506-21
薛亚平,薛晓燕,王晓燕,王晓燕。基于全基因组数据和人口学模型的脊椎动物抗LINE逆转录转座子选择的差异效应。中国生物工程学报。2018;10:1265-81。
ENCODE项目联盟。人类基因组中DNA元素的综合百科全书。大自然。2012;489:57 - 74。
Kovaka S, Zimin AV, Pertea GM, Razaghi R, Salzberg SL, Pertea M.长读RNA-seq与StringTie2对齐的转录组组装。《基因组生物学》2019;20:278。
Böhne A,周Q, Darras A, Schmidt C, Schartl M, Galiana-Arnoux D,等。Zisupton -最近活跃于鱼类DNA转座元件的新超家族。《生物化学学报》2011;29:631-45
鲍文华,郭志刚,李志刚。Repbase序列的序列分析。暴徒的DNA。2015; 6:11。
Mi S, Lee X, Li X, Veldman GM, Finnerty H, Racie L,等。合胞素是一种参与人类胎盘形态发生的逆转录病毒包膜蛋白。大自然。2000;403:785-9。
堀江文M,小林Y,本田T,藤野K,赤坂T,科尔C,等。蝙蝠基因组中的一种依赖RNA聚合酶基因,来源于一种古老的负链RNA病毒。科学通报2016;6:25873。
张刚,Cowled C,史震,黄震,Bishop-Lilly KA,方X,等。蝙蝠基因组的比较分析提供了深入了解飞行和免疫的进化。科学。2013;339:456-60。
潘文伟,李志强,李志强,等。埃及Rousette基因组揭示了蝙蝠抗病毒免疫的意想不到的特征。细胞。2018;173:1098 e18——110.。
小林KS,范登埃尔森PJ。NLRC5: MHC i类依赖免疫反应的关键调节因子。中华免疫杂志2012;12:813-20。
本克托斯,Kovács EG,赫兹尔F,库弗TA。NLRC5的功能超出了MHC I的调节-到目前为止我们知道什么?前沿免疫,2017;8:150。
Ostuni R, Piccolo V, Barozzi I, Polletti S, Termanini A, Bonifacio S,等。在分化细胞中被刺激激活的潜在增强子。《科学》2013;152:157-71
乔勇,Giannopoulou EG,陈超,Park S- h,龚松,陈杰,等。干扰素-γ诱导的染色质重塑和toll样受体信号通路协同激活炎症细胞因子基因。免疫力。2013;39:454 - 69。
Bondet V, Le Baut M, Le Poder S, Lécu A, Petit T, Wedlarski R,等。蝙蝠IFNα蛋白的构成。Front Immunol. 2021;12:735866。
Kepler TB, Sample C, Hudak K, Roach J, Haines A, Walsh A,等。手翼龙I型和II型干扰素基因由统计基因家族组装器从基因组测序痕迹推断。BMC基因组学。2010;11:444。
林伯韬,李志强,李志强,等。用29种哺乳动物绘制的人类进化限制的高分辨率地图。大自然。2011;478:476 - 82。
杜琴科O,沙米姆MS,巴特拉SS,杜兰德NC,穆西亚NT,莫斯托法R,等。Juicebox组装工具模块可以使用染色体长度的支架进行哺乳动物基因组的从头组装,价格低于1000美元。bioRxiv。2018:254797可从:https://www.biorxiv.org/content/10.1101/254797v1.引用2022年4月18日。
张晓东,张晓东,张晓东,张晓东,张晓东。一种用于基因组结构捕获的综合技术。方法。2012;58:268 - 76。
Pasquesi GIM, Adams RH, Card DC, Schield DR, Corbin AB, Perry BW,等。有鳞爬行动物挑战鸟类和哺乳动物设定的基因组重复元素进化范式。Nat Commun. 2018;9:2774。
Janssens D, Henikoff S. CUT&RUN:针对低细胞数量v3的高效原位全基因组分析。协议。io: ZappyLab, Inc.;2019.可以从:https://www.protocols.io/view/cut-amp-run-targeted-in-situ-genome-wide-profiling-zcpf2vn
Meers MP, Bryson TD, Henikoff JG, Henikoff S.改进的CUT&RUN染色质分析工具。Elife。2019;8。https://doi.org/10.7554/eLife.46314.
BBMap。SourceForge。可以从:https://www.sourceforge.net/projects/bbmap/.2019年2月
Babraham bioinformatics - FastQC高通量序列数据的质量控制工具https://www.bioinformatics.babraham.ac.uk/projects/fastqc/.版本0.11.08 - 2018/04/10
Ewels P, Magnusson M, Lundin S, Käller M. MultiQC:在一份报告中总结多种工具和样品的分析结果。生物信息学。2016;32:3047-8。
杜宾A, Davis CA, Schlesinger F, Drenkow J, Zaleski C, Jha S,等。STAR:超快通用RNA-seq对准器。生物信息学。2013;29:15-21。
CCGB:米勒实验室,LASTZ。可以从:https://www.bx.psu.edu/~rsharris/lastz/.2021年2月进入。
Love MI, Huber W, Anders S.用DESeq2调节RNA-seq数据的折叠变化和分散估计。基因组生物学,2014;15:550。
廖颖,王杰,Jaehnig EJ,史志,张波。WebGestalt 2019:改进ui和api的基因集分析工具包。核酸研究,2019;47:W199-205。
李华,Handsaker B, Wysoker A, Fennell T, Ruan J, Homer N,等。序列对齐/映射格式和SAMtools。生物信息学。2009;25:2078-9。
GFF实用程序:GffRead和GffCompare。F1000Res。2020; 9。https://doi.org/10.12688/f1000research.23297.2.
bairroch A, Apweiler R. SWISS-PROT蛋白质序列数据库及其补充TrEMBL 2000。核酸Res. 2000; 28:45-8。
卡马乔C, Coulouris G, Avagyan V, Ma N, Papadopoulos J, Bealer K,等。BLAST+:架构和应用程序。生物信息学。2009;10:421。
昆兰AR,霍尔IM。BEDTools:用于比较基因组特征的一套灵活的实用程序。生物信息学。2010;26:841-2。
李B,杜威CN。RSEM:从RNA-Seq数据中精确的转录物定量,有或没有参考基因组。生物信息学。2011;12:323。
中川S,高桥沐。gEVE:一个基于基因组的内源性病毒元素数据库,提供了哺乳动物基因组中全面的病毒蛋白编码序列。数据库。2016;2016年。https://doi.org/10.1093/database/baw087.
Sayers EW, Bolton EE, Brister JR, Canese K, Chan J, Comeau DC,等。国家生物技术信息中心数据库资源。核酸Res. 2022;50: D20-6。
埃德加钢筋混凝土。搜索和聚类速度比BLAST快几个数量级。生物信息学。2010;26:2460-1。
朗米德B,萨尔茨堡SL.快速间隙阅读对齐领结2。Nat Methods. 2012; 9:357-9。
Ramírez F, Dündar F, Diehl S, Grüning BA, Manke T. deepTools:探索深度测序数据的灵活平台。中国生物工程学报,2014;42:W187-91。
Ramírez F, Ryan DP, Grüning B, Bhardwaj V, Kilpert F, Richter AS,等。deepTools2:用于深度测序数据分析的下一代web服务器。核酸研究,2016;44:W160-5。
利用基于模型的ChIP-Seq (MACS)分析胚胎干细胞中蛋白质- dna相互作用测序产生的短读。入职:基德·BL,编辑。干细胞转录网络:方法和协议。纽约:施普林格纽约;2014.p . 81 - 95。
葛瑞敏,李志强。生物序列数据集的综合基序分析。bioRxiv。2021;可以从:https://www.biorxiv.org/content/10.1101/2021.09.02.458722v1.
Fornes O, Castro-Mondragon JA, Khan A, van der Lee R, Zhang X, Richmond PA,等。JASPAR 2020:更新转录因子结合谱开放获取数据库。Nucleic Acids res 2020;48: D87-92。
Layer RM, Pedersen BS, DiSera T, Marth GT, Gertz J, Quinlan AR. GIGGLE:用于大规模集成基因组分析的搜索引擎。Nat Methods. 2018; 15:123-6。
确认
未经公布的基因组集合在DNA动物园联盟(dnazoo.org)的许可下使用。鼠耳蝠lucifugus原代胚胎成纤维细胞是Mario Capecchi(犹他大学)赠送的礼物。我们感谢bifrontiercomputing核心在本研究期间提供的技术支持。我们感谢科罗拉多大学基因组学共享资源和生物前沿计算核心在这项研究中的技术支持。
资金
这项研究得到了NIH (1R35GM128822)、Alfred P. Sloan基金会、David和Lucile Packard基金会以及Boettcher基金会的支持。
作者信息
作者及隶属关系
贡献
EC和GP设计了这项研究。GP和CK进行实验。GP, CK, AO和EC分析数据并解释结果。GP和CK创建并编辑图形和表格。EC、GP和CK根据所有合著者的意见撰写了手稿。所有作者最终同意出版。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:图S1。
鼠耳蝠lucifugus胚胎成纤维细胞对IFNa刺激的反应。图S2。鼠耳蝠lucifugus基因组重复元素组成。图S3。鼠耳蝠lucifugus总的和最近的TE转录组组成。图S4。TE免责事件鼠耳蝠属isg。图S5。A)热图显示IFNainducible H3K27ac(未调整)上的标准化CUT&RUN信号(每百万次读取的信号)p-val < 0.10, log2FC > 0,n= 1113)。B)火山图显示ifna诱导的家族水平TE富集(未调整)p-val < 0.10, log2FC > 0) H3K27ac区域。TE家庭与费舍尔的双尾p-val < 0.05根据报道的优势比定义为富集(蓝色)或贫化(绿色)。不重要的家庭用灰色表示。C)热图显示正常化的CUT&RUN信号(每百万次读取的信号)超过ifna诱导(未调整)p-val < 0.10, log2FC > 0) h3k27ac标记的L2家族。图S6。热图显示在h3k27ac标记的A) MIR3和B) amnine1家族上正常化的CUT&RUN信号(每百万次读取的峰值信号)。图S7。I型IFN位点的重建。
附加文件2:表S1。
RNAseq 4hrs对比IFNvsUN。
附加文件3:表S2。
RNAseq 24hrs对比IFNvsUN。
附加文件4:补充数据
自定义StringTie转录组汇编的gtf格式注释文件。
附加文件5:表S3。
IFNvsUN。
附加文件6:表S4。
StringTie组装te -外显子相交。
附加文件7:补充数据
Zisupton-EEF1A1对齐。
附加文件8:补充数据
蝙蝠物种EEF1A1序列比对。
附加文件9:表S5。
StringTie转录组与TE参考序列tblastx分析。
附加文件10:表S6。
StringTie转录组与病毒蛋白tblastx分析。
附加文件11:表S7。
te转录起始位点重叠。
附加文件12:表S8。
M. lucifugus胚胎成纤维细胞H3K27ac CUT&RUN DESeq2结果。
附加文件13:表S9。
M. lucifugus胚胎成纤维细胞H3K27ac CUT&RUN motif分析。
附加文件14:表S10。
te重叠ifna诱导的H3K27ac峰按家族划分。
附加文件15:表S11。
te重叠STAT1峰到最近的ISG (log2FC > 1.5) TSS的距离。
附加文件16:表S12. 5
家庭级TE丰富使用GIGGLE。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
帕斯奎,g.i.m.,凯利,c.j.,奥多涅斯,A.D.et al。在I型IFN反应中转座因子的转录动力学鼠耳蝠lucifugus细胞。移动DNA13, 22(2022)。https://doi.org/10.1186/s13100-022-00277-z
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s13100-022-00277-z
关键字
- 蝙蝠的免疫力
- 转座的元素
- 表观基因组学