摘要
背景
ice是整合在细菌染色体中的可移动遗传元素,可以切除并转移到新细胞中。它们在水平基因传递中起着重要作用,并携带辅助基因,可能为细菌提供有趣的表型。在这里,我们试图研究冰碛体在300种具有最大科学和经济影响的植物致病细菌基因组中的存在和作用。
结果
共鉴定出78个ice(45个不同元素)根癌土壤杆菌,Dickeya dadantii,和d .以上,Pectobacterium carotovorum和p . atrosepticum,两,Ralstonia solanacearum种类复杂,黄定.有趣的是,在一些样品中观察到四种ice同时出现p .两菌株。此外,我们还发现了31个新元素,携带396个可能影响毒力和适应度的辅助基因,如编码T3SS、细胞壁降解和耐重金属等相关功能的基因。我们也提出了分析先前报道的数据有关的货物基因的表达与毒力p . atrosepticum这证明了这些基因在烟草植物感染过程中的作用。
结论
综上所述,本文强调了ICEs在影响这些植物病原的致病性和生活方式方面的潜力,并指导了重要的推定毒力基因在植物病原细菌中的传播。
背景
整合和结合元件(ICEs)是自传播的移动元件,在细菌适应过程中发挥核心作用;因此,它们可以直接影响宿主的进化[1,2].这些广泛分布的元件目前被发现整合到细菌染色体中,因为它们能够通过重组元件侧面的直接重复序列(dr)来进行切割,并通过共轭转移,以模块化结构携带其机制[3.,4,5].在ice中发现的主要遗传模块包括编码功能的基因,这些功能与它们与宿主染色体的整合和切除、偶联和调节有关。这些模块也有可变的内容,这导致这些功能由来自不同基因的不同机制执行[5,6].ice通常携带货物基因,因此赋予宿主细胞显著的表型,如毒性、对抗生素和重金属的耐药性,这些对细菌的适应度很重要。例如,冰Tn43716061年发现的铜绿假单胞菌,对碳青霉烯类抗生素和ICE产生耐药性Pm1的变形杆菌,Providencia stuartii和摩根氏菌属morgani,它们携带编码黏附蛋白酶和铁获取系统的基因,有助于这些细菌的毒性[4,7,8,9].
ice广泛分布在细菌染色体中,一些研究表明,它们存在于植物致病菌中,这是一种微生物,利用生物膜形成和毒素等毒力因子,通过宿主组织入侵,参与了重大作物损失[3.,10].例如,在菌株中发现了不同的ice两pv。actinidiae对重金属的抗性,ICE HAI2Pectobacterium atrosepticum,它传播编码一种重要毒力因子生物合成簇的基因[11,12].在这些植物病原细菌中,Mansfield等人在2012年根据它们的科学相关性和经济影响对它们进行了分类,其排名由两,青枯农杆菌,农杆菌,黄oryzae pv。oryzae,黄定,黄axonopodis pv。manihotis,欧文氏菌amylovora,Xylella fastidiosa,Dickeya(dadantii和以上),Pectobacterium(carotovorum和atrosepticum)[13].
大部分涉及ice的先驱研究仅基于货物基因赋予的表型,没有提供关于这些元素的更广泛的知识[5].然而,全基因组测序(WGS)的发展导致了完整基因组序列的大量可用性,使研究得以进行,以阐明ICEs在细菌进化中的作用[14,15,16].因此,我们在这里寻找整合到300个主要植物病原细菌完整基因组中的ice,并分析假定的负载基因及其在毒性或适应性中的潜在作用。
结果
发现的最全面的集成植物致病菌基因组的ICEs数据集
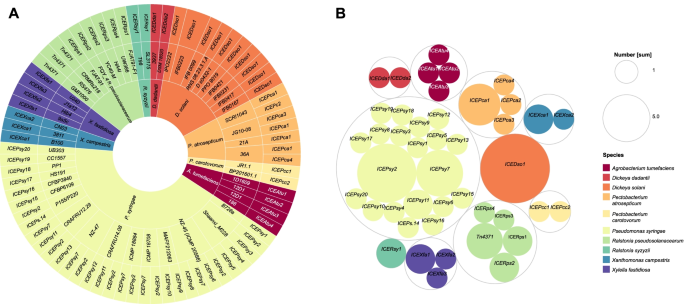
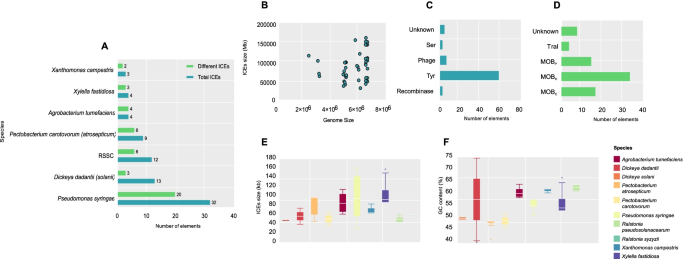
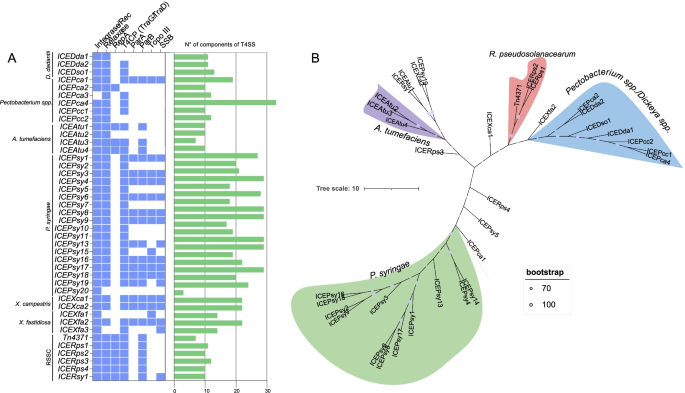
我们首先试图在分子植物病理学中识别和分析重要植物致病菌基因组中的ICEs。在9种植物病原菌中共发现78种推测元素,其中包括丹参、茄属、茄属、茄属、瘤属、紫丁香属、油菜属、苛养属和r . solanacearum物种复合体(RSSC)(图。1和表年代11).在这些元素中,发现了45个不同的ice,并且在基因组中发现了更多的icep .两(33个,其中20个是不同的元素)(图。2A).在染色体序列中未发现icex oryzae,x axonopodis,或大肠amylovora.
ice在菌株间的分布。太阳爆炸图表显示了所有菌株中发现的元素。细菌种类按元素数最多到元素数最少的顺序排列,并按颜色进行区分:两页;深蓝色的:d .以上;浅蓝色:r . pseudosolanacearum;绿色(分别从最暗到最亮):atrosepticum, X. fastidiosa, A. tumfaciens, X. campestris;黄色:r . syzigii;光橙色:p . carotovorum;暗橙色:d . dadantii.从内到外的图表:细菌的种类,菌株的名称,以及每个菌株中存在的已识别的元素。b细菌种周围ice分布的分层组织,如图例所示,用颜色对物种进行编码。图旁的刻度显示了每种物种的ice数量
各元素序列大小差异较大。最大的元素ICEPsy10被发现在p .两有161 KB,是最小的ICEDda2例发现于d . dadantii40 kb。总的来说,元素的平均大小是80 kb。GC含量在40 ~ 66%之间ICEPat2,ICEXfa2,分别。平均含量为55%。2F和表S12).正如已经预期的那样,在宿主基因组的增大和这些元素的存在之间没有发现任何关系(图1)。2B)。
大多数(88%,n = 68)插入到tRNA基因中,9个插入到其他基因中。附件网站(丙氨酸)在49例ice中被识别;但是,我们的搜索方法没有找到丙氨酸整合到RSSC基因组的任何元素的位点(表S13).58个ice编码Tyr重组酶家族,34个ice编码MOBHrelaxase基因(图。2C, D;表的年代14).
有趣的是,我们在其他物种中发现了具有核心模块基因的ice。的ICEPca2从p . carotovorum是在?的基因组中发现的沙雷氏菌属plymuthicaC-1菌株,比较分析表明,元素具有99%的核苷酸相似性(数据未显示)。同时,ICEPca1与一种新的ICE有82%的核苷酸同一性p . aroidearum哈佛应变;ICEXca1个与一个新的ICE有88%的序列一致性黄arboricolapv。juglandis应变Xaj 417。我们数据集中的两个ice,ICEDda1d . dadantii和Tn4371的r . pseudosolanacearum之前被列为家族成员吗Tn4371[15,17].然而,这两种元素之间的比较分析表明相似性很低(核苷酸识别率为45%)。
ice携带对植物致病菌的致病性和生活方式有潜在影响的基因
元素所携带的基因也被调查和分类为他们假定的作用。正如预期的那样,更多的假设蛋白质和功能未知的基因被识别出来,其次是结合基因和货物基因(图4)。3.A).具体而言,在货物基因中,氧化还原过程和抗性编码功能在元素中orf(开放阅读框)中出现的数量较多,毒力因子次之(图1)。3.B).通过在特定蛋白质数据库中的注释,验证了大多数ICEs的推定毒性和适应性作用。编码毒力功能的基因在28个元素中被追踪到,而在假定的适应性下,在12个ICEs中发现了作用,以及其他重要作用,如金属和抗生素耐药性(表S15).在毒力方面,基因也根据其假定的功能进行了组织,最常见的一类毒力基因是编码由III型分泌系统(T3SS)及其组成部分易位的蛋白质的基因,其次是编码参与宿主细胞壁降解的水解酶的基因(图)。3.C).综上所述,这两部分揭示了高影响植物致病细菌基因组中存在的ICEs及其对其致病性和生活方式的可能影响。现在,我们将进一步介绍个别细菌种类的具体结果。
冰在根癌土壤杆菌
首先,我们研究了NCBI数据库中17个可用菌株的染色体序列,每个菌株都有两条染色体。然后,我们在32个序列中搜索ice农染色体(表3.).我们的方法允许在三个菌株的序列中识别四种新元素:ICEAtu1在菌株1D1609的染色体上被发现,ICEAtu2,ICEAtu3,分别在12D1株的圆染色体和线性染色体上,ICEAtu4在菌株186的圆形染色体上被发现(图1)。1).这些元素的平均大小为86 kb;ICEAtu序列最大的元素为3 (114 kb),其次为ICEAtu4(101 kb)ICEAtu2(68 kb)ICEAtu1是最小的元素,长度为61 kb(表S12).这些元素的平均GC含量为60%,范围为58% (ICEAtu3)至63% (ICEAtu1).唯一的元素ICEAtu2和ICEAtu312D1菌株的GC含量分别低于基因组的58%和58%(表S12).的丙氨酸网站的ICEAtu3和ICEAtu4已确定,关于整合场地,元素和ICEAtu2插入到tRNA序列中,而ICEAtu1插入到guaA基因(表13和14).ice的序列比对农透露,ICEAtu2和ICEAtu4具有53%的核苷酸同一性,主要是由于基因簇代表了基于同位分析的共轭和集成模块(图S1,年代2,年代3.,年代4,年代5,年代6,年代7,年代8,年代9和S10一个)。
农ICEs编码的蛋白质可能具有重要的致病性功能,如半胱氨酸水解酶和糖苷酶ICEAtu1和endo1,4 - β -木聚糖酶ICEAtu4.同样,烯烃还原酶和谷胱甘肽s -转移酶上ICEAtu1和通用应激蛋白ICEAtu3可能对这些细菌的适应有假定的作用。我们还鉴定了一种L.D -转肽酶的编码基因ICEAtu3和MBL折叠金属水解酶ICEAtu4,提供了对抗生素的假定耐药性(表S15).
冰在迪克亚(达达蒂和索拉尼)
我们在13个染色体序列中寻找iceDickeya基因组,其中两个来自d . dadantii, 11个基因组属于d .以上物种(表9).在这些序列中,我们发现了三种不同的元素,总共有13个ice:ICEDda1在3937株和ICEDda2,在菌株DSM 18020的染色体中。有趣的是,ICEDso1都存在于D.以上因此,所有被研究的菌株都存在ice(图5)。1).在这些元素中,只有ICEDda1被引用在文献中,作为元素Tn4371家庭(表16)[17].
关于这些元素的大小,ICEDda1最大的元素是74 kb,其次是ICEDso1(赠),而ICEDda2最小,为40kb(表S12).各元素GC含量均低于基因组含量,平均值为51%,为52%ICEDso152%,ICEDda2,和49%ICEDso1(表12).这两个ICEDda1和ICEDso1是否确定了它们的附着位点,并插入到tRNA序列中ICEDda2是插入bamE基因(表13和14).这些元素之间的比较显示出彼此之间的相似性很低(< 50%的核苷酸同源性)。4B).然而,在所有三个元素之间都观察到同源基因簇,这些基因与重组酶基因、一些转录调节基因和与VI型分泌系统(T6SS)相关的装载基因有关。此外,ICEDda1和ICEDso1可能也共享它们的接合和调控模块,因为共质基因簇包括IV型分泌系统(T4SS)和毒素-抗毒素系统的基因(图S2).在这些ICEs的主要装载基因中,我们确定了一个推定的耐碲基因(terB)和条目排除(eexN)ICEDda1.此外,T6SS的组成基因,hcp感受器,tssI-vgrG对所有三种不同元素的毒力作用进行了验证(表S15).
冰在Pectobacteriumcarotovorum(和atrosepticum)
八个基因组Pectobacterium被调查,四从p . carotovorum(Pcc)和四个来自p . atrosepticum(Pca)。共识别出9种元素,其中6种是不同的ice(图1)。1,表10).在Pcc中,我们发现ICEPcc1和ICEPcc2在菌株JR1.1和BP201601.1的基因组中分别发现。我们观察到在基因组中出现了几次Pectobacterium,包括两名行政主任(ICEPca1和ICEPca2),在SCRI1043染色体上的ICEPca1和ICEPca3)和元素ICEPca1和ICEPca4在36A菌株的基因组中。在这里,我们确定了该属的五个新元素,而且只有ICEPca1曾有过报道[12,18,19(图。1,表16).
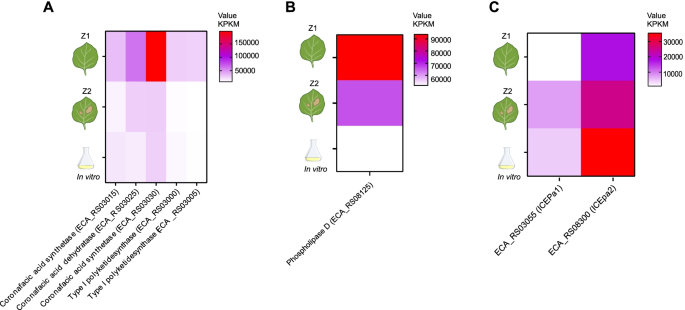
的Pectobacteriumice与ICEPca2和ICEPcc1有69.5%的核苷酸同源性,而在ICEPca2和ICEPca4,为689%(图S10C).共时分析显示,大多数元素似乎有相似的共轭模(高同一性),除了ICEPca1,是由接合基因和T4SS基因组成的高度共效的基因簇,以及接合基因之间的相似性ICEPcc1,ICEPca2,ICEPcc2和ICEPca4(无花果。3.).就这些元素的主要载货基因而言,冠藻酸的生物合成簇ICEPca1以SCRI1043分离物的毒性而闻名[12].基因编码蛋白质推定的毒性作用也发现ICEPca2(磷脂酶D),ICEPca4(精氨酸酶家族蛋白)和ICEPcc2(谷胱甘肽过氧化物酶)。此外,我们鉴定了假定的进入排斥基因ICEPca2 ICEPca4,和ICEPcc1.最后,我们观察到编码蛋白质的基因可能带来抗生素耐药性:MBL折叠金属水解酶ICEPca3和ICEPcc2,mipA/ompV家庭的蛋白质ICEPca4(表15).
我们的差异基因表达分析表明,高表达的基因簇冠酸生物合成携带ICEPca1在无症状阶段p . atrosepticum与症状期和体外培养相比,烟草SCRI1043感染的RPKM值更高(图1)。4),由[20.].并验证了其推定的毒力基因磷脂酶D的表达,其表达模式与该基因的表达模式相同ICEPca1且在无症状感染阶段RPKM值较高(图1)。4).最后,我们分析了其携带的松弛酶基因的表达ICEPca1(基因座标签ECA_RS03055)和ICEPca2(基因座标记ECA_RS08300),以指示这些元素可能发生动员的感染阶段。一个不同的结果模式被验证,和放松ICEPca1在感染有症状期表达量较大,体外培养次之,无症状期表达量较低(图1)。4).的relaxaseICEPca2在体外培养中表达上调,其次是症状期的表达(图。4).
冰在两pathovars
发现的ice数量最多的是假单胞菌pathovars这可能是由于该物种有多个可用的基因组。对28条染色体进行了研究,发现了33个元素,其中20个不同元素(图4)。1,表1).关于这些元素在细菌基因组中的分布,ICEPsy2是最常见的元素,存在于9个品系中,其次是ICEPsy7,在7株菌株中检测到(图。1).我们找到了7个p .两ice已经在文献中引用了。因此,这项工作带来了13个新的假定要素(表S16).
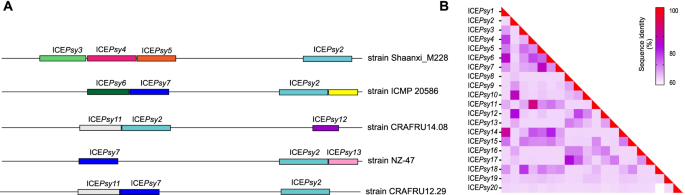
ICE共现的有趣事件已在p .两染色体序列以shaanx_m228和ICMP 20,586菌株为主,各有4个单片段缺失。此外,我们在CRAFRU14.08、NZ-47和CRAFRU12.29的基因组中发现了三个单块ice;MAFF212063中的两个元素:ICMP 18708、ICMP 18884和P155。1).其中一些元素可以被观察到分组在串联上的染色体序列与上的相同丙氨酸其排列如图所示。5答:这可能是文献中第一次在一个基因组中追踪到三种以上的元素。
的大小p .两元素的范围从30 KBICEPsy20至166kbICEPsy10,平均值为99 kb(表S12), GC含量平均值为55%,范围为50 ~ 57%(表S12).确定了15个元素的附加位点,所有32个元素都被整合到一个tRNA序列旁边(表S13和14).在元素之间,观察到更大的相似性ICEPsy4和ICEPsy11,85%核苷酸的同源性ICEPsy1和ICEPsy14,核苷酸同源性为82%(图。5B).基因簇的比较分析表明在p .两这使我们能够根据同纤基因簇,将这些元素分成三组。组1组成ICEPsy3,ICEPsy15,ICEPsy16,ICEPsy18和ICEPsy19.这些元素大多共享共轭和积分模块,以及一些IS序列(图S4).第二组是最大的,由ICEPsy1,ICEPsy4,ICEPsy6,ICEPsy9,ICEPsy11,ICEPsy13,ICEPsy14,ICEPsy17和ICEPsy20;该组不仅具有第1组的偶联和整合模块,还具有编码相关功能的负载基因,如其他负载基因之间的抗铜和抗砷基因簇ICEPsy20,是最独特的元素p .两只有一个整合酶和一个维持基因(ardR)(图。5).最后,第三组由ICEPsy2,ICEPsy5,ICEPsy7,ICEPsy10和ICEPsy12;所有元素还共享主模块(集成和接合)。此外,ICEPsy2和ICEPsy10也有一个共效基因簇,包含T3SS效应子、一个纤维素酶、一个转运基因簇和趋化性基因簇,这些基因簇可能与其他货物基因一样参与元素调控。这种比较分析允许我们验证序列之间的差异ICEPsy2在ICMP20586和shaanx_m228菌株中,由于IS插入元素(图S6).
大量编码的货物基因被确认为重要的作用p .两ice,主要是铜和砷抗性基因簇ICEPsy8和ICEPsy13,这些在文献中已经有描述(Colombi等人,2017b)。然而,我们在这里报告这些基因簇ICEPsy1和ICEPsy14(耐铜和砷),ICEPsy4和ICEPsy11(只有砷阻力)。在一些菌株中发现了大量编码具有毒力作用的蛋白质的基因p .两元素,如T3SS效应基因ICEPsy2,ICEPsy5,ICEPsy7,ICEPsy9,ICEPsy10,ICEPsy12和ICEPsy18.也鉴定了假定的抗生素耐药的货物基因,如crpP家族蛋白,由十二种元素组成,还有mupB的ICEPsy18.此外,还发现了一个编码碲抗性的基因簇ICEPsy2(表15).
冰在Ralstonia solanacearum种类复杂
染色体序列为100r . solanacearum对菌株进行了检测,发现了7种独特的ice(6种发现于r . pseudosolanacearum基因组和一个发现于r . syzygii)(图。1,表2).其中大部分元素已经在我们研究小组的另一项工作中描述过了。15)(表16),这里我们报告一个已知元素的存在(ICERps1)在另外两个品系FJAT91-F1和FJAT91-F8中,ICERps4,在UW386检测到。ICERps4大小为56.3 kb,占GC内容的64.4%,并插入到tRNA序列中(表S12,年代13和S14).对序列的比对显示了两者之间的相似性ICERps4和ICERps1,ICERps2和ICERps3,核苷酸识别率分别为79%、81%和78%(图S3.D)。用这些元素之间的同源基因簇(包括共轭模组和其他货物基因)加以说明(图S7).这些元素携带的货物基因编码在细菌适应中的假定作用,如作为元素Tn的谷胱甘肽s -转移酶4371和ICERps3和γ -谷氨酰环转移酶携带ICERps1.同样,我们能够确定对毒力有推定影响的基因,如来自ICERsy1和SDR家族氧化还原酶,存在于Tn4371和ICERps3.最后,ICERps1也携带超氧化物歧化酶基因,据推测具有抗氧化作用(表S15).
冰在黄定pathovars
我们从18个染色体序列开始研究x定菌株中,我们追踪到两种不同的ice,这是两种新元素(ICEXca1和ICEXca2).ICEXca1在B100和3811菌株的染色体中发现ICEXca2在CN03染色体上的表达(图。1,表5).元素的大小ICEXca1不同品系间差异较大:B100株为64.1 kb, 3811株为64.5 kb。而且,大小ICEXca2元素为83.7 kb;GC含量分别为61%和60%(表S12).描述了这些元素的附着位点,并将它们插入tRNA序列中(表S13和S14).这些元素的相似性很低,核苷酸识别率为47%(图S10E),然而,它们有高度共音的基因簇(图S8).在x定携带编码推定毒性作用的货物基因。在冰Xca1在ICE中,我们发现了编码氨基转移酶和裂解慕麟转糖基酶的基因Xca2.该成分还携带两个推定的致病效应基因和货物基因,如肌醇六磷酸,这可能与这些细菌的适应性有关(表S15).
冰在Xylella fastidiosa
21条染色体的分析x fastidiosa结果发现了四种元素中三种不同的ice(图1)。1,表8).元素的大小从88 kb到ICEXfa1至158kbICEXfa3,平均GC含量为56,介于54至66之间(表S12).元素的插入ICEXfa1由于该元素被发现与ABC转运子基因结合在一起,因此与其他基因分离;ICEXfa2和ICEXfa3被发现整合在tRNA序列旁边。而且,只有依恋位点的ICEXfa2(表S13和S14).到目前为止,只有ICEXfa2在Hib4菌株中已发现[21].因此,我们的工作在x fastidiosa.比对分析表明核苷酸同源率小于或等于42%的元素之间的相似性很低(图S10F).基因聚类比较分析能够显示由ICEXfa3和ICEXfa1,包括它们的偶联和维持模块,以T4SS基因和毒素抗毒素基因簇为代表,以及其他装载基因;ICEXfa2与该分析中的其他元素相似度也很低(图S9).在这三种基因中都发现了可能在毒性中起作用的货物基因x fastidiosa元素,包括可以在ICEXfa1和ICEXfa3.此外,在两种元素(MBL折叠金属水解酶-ICEXfa1)和(3-羟基豆蔻酰氨基氨基酰基转移酶-ICEXfa3).我们鉴定了假定的抗氧化性基因(超氧化物歧化酶)和假定的适应作用,如协比例phyrinogen III氧化酶ICEXfa2.此外,我们还发现了一个假定的入口排斥基因ICEXfa3(表15).
植物病原ICEs核心基因的保存和进化历史
除了ice携带的货物基因外,我们还展示了ice核心基因的保护和进化历史,这些基因可能在元素移动中发挥作用。整合酶/重组酶、松弛酶、iv型偶联蛋白(T4CPs)、ParA、ParB、拓扑异构酶III和单链dna结合蛋白(SSB)是我们数据集中最保守的核心基因(图1)。6A).此外,我们发现来自同一物种的ice具有共同的核心基因,包括其存在/缺失和T4SS成分的平均值,而缺乏大多数核心基因的可能是缺陷元素,如ICEPsy20从p .两(无花果。6一个)。p .两ICEs的T4SS成分平均最高(22个基因集)。接下来,我们将这些核心基因从假定的完整元素中分组,并基于8个骨干基因对齐连接构建了一个树。在系统发育树上,与物种相关的ice被划分为4个类群。p .两ice是最保守的集群,农,r . pseudosolanacearum,和包含来自的ice的集群Pectobacterium spp.和Dickeya spp.(无花果。6B)这些发现可能意味着这些元素有相同的共同起源。然而,我们发现了不同物种之间的聚类,这表明ice基因本身是保守的(图1)。6B)。
讨论
近年来,由于全基因组序列的可用性增加,人们已经克服了对ice的有限理解[4,5,22].一些研究已经讨论了植物病原细菌中存在的ice,但没有深入研究。然而,有必要充分了解ICEs与经济上重要的植物致病菌之间的关系[3.,10].通过一个in-silico利用NCBI提供的300个基因组对ICEs进行了研究,我们已经确定并表征了78个假定的集成到十大植物致病细菌基因组中的ICEs。正如预期的那样,大多数ice被发现整合到tRNA序列中[4].这些元素编码了核心模块基因,包括允许整合/切除模块、偶联、维护和调节模块的基因[4].总体而言,GC含量等ICEs特征的证据表明宿主基因组中的值略低于预期,这表明这些元素可能是最近获得的,很可能是在选择压力下适应密码子宿主的密码子使用[23,24].一些元素还携带进入排斥基因(exn和traG),可以避免ICEs的冗余转移,从而节省宿主能量[25,26]
我们没有在x oryzaepv。oryzae, x axonopodis或e . amylovora可能是由于所使用的软件系统存在偏差,因为我们正在处理高度多样化的元素和这些物种的完整基因组的数量。然而,我们在83个完整的基因组中寻找了icex oryzaepv。Oryzae和,尽管它有大量的插入序列27,28],没有发现ICE。同样,我们研究了100个RSSC完整基因组序列,比[15,以及一部小说ICE (ICERps4)为RSSC报告。我们假设,目前不同种类细菌基因组中ice的变化可能与一种控制这些元素进入细胞的机制有关,该机制或多或少严格,从而允许或不允许宿主细胞获取ice。
大多数被鉴定的元素被发现携带具有推定的适应性和毒性功能的基因。推测的抗生素耐药基因也被鉴定出来,它们可能与ICEs维持模块有关,尽管它们没有以相同的方式进行分类。在发现的毒力因素方面,观察到在ICEs中T3SS orf数量增加。这种分泌系统是植物细菌的一个重要毒力特征,它可以通过改变植物细胞的代谢或抑制防御,使效应蛋白转位到植物细胞中[29,30.].此外,我们还发现编码参与宿主细胞壁降解的水解酶的基因是我们的ice中第二常见的毒力因子。因此,我们的工作强调了ICEs在寄生进化中可能发挥作用的重要性。在ICEs中发现的保守核心基因可能有助于元素的迁移,并促进重要的推定毒力基因在植物病原细菌中的传播。
综上所述,我们的研究结果表明,冰激菌与植物病原菌的适应度之间可能存在关联。我们的硅研究为进一步的实验研究打开了大门,这些研究应该进行,以提高对植物病原细菌中ICEs及其装载基因作用的认识。
材料和方法
数据
300个具有经济和科学影响的植物病原细菌完整基因组[13从国家生物技术信息中心(NCBI -)下载http://ftp.ncbi.nih.gov/assembly), 2020年7月。使用GenBank格式的染色体序列搜索ICEs(表S1,年代2,年代3.,年代4,年代5,年代6,年代7,年代8,年代9和S10).
综合元素和共轭元素的识别和定界
为了揭示细菌染色体中的ice序列,我们采用了一种类似于Gonçalves和Santana [16].因此,我们使用BLASTn [31]与储存在“冰山”数据库内的已知ice [32,33,只对e值小于或等于10的序列进行检测5且覆盖率大于或等于50%被选择。我们将ice的核苷酸序列提交给ICEfinder软件系统(https://db-mml.sjtu.edu.cn/ICEfinder/ICEfinder.html),这是一个由ICEberg 2.0提供的在线工具,可以识别细菌基因组中整合酶基因、T4SS和定向重复序列(DRs)等整合和结合元素的特征特征[32,33], OriTfinder [34) (https://tool-mml.sjtu.edu.cn/oriTfinder/oriTfinder.html)识别细菌染色体中的转移起源序列,这表明ICE的存在,MOBscan (https://castillo.dicom.unican.es/mobscan/)识别松弛酶MOB家族,MacSyFinder的concscan模块(https://galaxy.pasteur.fr)识别细菌基因组中寻找IV型分泌系统的结合系统[35].我们还对完整的和注释的染色体序列进行手动搜索,以寻找ICEs签名基因:参与接合转移的T4SS的一部分基因,如交易,vir0,或民国,和整合酶(int)[5].附件区域(丙氨酸提供元素上下边界的站点)在ICEfinder中被识别出来,但当丙氨酸区域没有被检测到,我们使用BLASTn手动识别它们。我们首先通过寻找接近trna的整合酶基因来划分元素的上界,然后,这些基因之间的区域被选择,使BLAST在基因组的另一个位置寻找重复序列,该序列接近ICEfinder给出的元素可能的最终部分的坐标。ice的命名遵循已经描述过的模式[36].
携带基因的特征
蛋白质序列的注释使用Uniprot (https://www.uniprot.org/)[37]和Pfam (http://pfam.xfam.org/)[38蛋白质数据库。通过对病原菌-宿主相互作用数据库(http://www.phi-base.org/)[39]、毒力因子数据库(http://www.mgc.ac.cn/VFs/)[32,33]及III型分泌系统效应器资料库(http://effectors.bic.nus.edu.sg/blast.php)[40].用于识别序列编码蛋白的参数为:e值小于或等于105氨基酸同一性大于30% [15].随后,将所识别元素的核苷酸序列下载到GenBank格式,并使用genous®软件系统(Biomatters Ltd.)进行分析,以表征其切割和整合、偶联、调控和维护模块。
差异表达分析
采用RNAseq数据进行差异表达分析p . atrosepticum从烟草植株中分离出对应于两个感染阶段(无症状和有症状)的SCRI1043,并进行体外培养。数据可在NCBI生物项目中获取(登录号PRJNA403794) [20.].使用genous软件系统进行分析,跟随带有默认参数的Expression analysis教程。为了研究基因组中存在的ICEs的基因表达,我们以元素序列为参考绘制reads,并测量差异表达。在GraphPad Prism 8.4.3版本中绘制每千基每百万(RPKM)的读数,以生成热图。
比较分析
以fasta格式的ice核苷酸序列提交给ClustalW [41]生成用于创建热图的成对恒等矩阵,使用GraphPad Prism 8.4.3版本的Windows。序列也提交给Mauve [42和clker clustermap.js [43来进行基因簇比较和共时性分析。核心基因在ClustalW中对齐[44],并在Mesquite软件版本3.70 (http://www.mesquiteproject.org).对最大似然树进行对齐并使用MEGA X [45,基于8个骨干基因的比对。一般时间可逆模型和自举置信度值1000应用于树。
数据和材料的可用性
本研究分析的数据集可以在文章/补充材料中找到。这里描述的ICEs序列保存在ICEberg数据库中。
参考文献
整合和共轭元素的稳定机制。《微生物学杂志》2017;38:44-50。https://doi.org/10.1016/j.mib.2017.03.014.
综合元素与共轭元素的二元论性质。生物基因元素。2015;5(6):98-102。https://doi.org/10.1080/2159256x.2015.1102796.
Guglielmini, J., Quintais, L., Garcillán-Barcia, m.p, de la Cruz, F., & Rocha, e.p.c.。原核生物中ICE的序列强调了共轭作用的统一性、多样性和普遍性。公共科学图书馆遗传学。2011;7(8)。https://doi.org/10.1371/journal.pgen.1002222.78(1), 138 - 157。https://doi.org/10.1111/j.1365-2958.2010.07317.x
约翰逊CM,格罗斯曼广告公司。整合和结合元素:它们的功能和工作原理。2015;49:57 - 601。https://doi.org/10.1146/annurev-genet-112414-055018.
整合和接合元件:马赛克移动遗传元件,使动态横向基因流动。微生物学报。2010;8(8):552-63。https://doi.org/10.1038/nrmicro2382.
杜桑A、梅林C.移动元件作为功能模块的组合。质粒。2002;47(1):26 - 35周不等。https://doi.org/10.1006/plas.2001.1552.
Flannery EL, Antczak SM, Mobley HLT。整合和结合元件ICEPm1在临床分离株之间的自我传递需要功能整合酶、放松酶和IV型分泌系统。J Bacteriol。2011;193(16):4104 - 12所示。https://doi.org/10.1128/JB.05119-11.
Fonseca EL, Marin MA, Encinas F, Vicente ACP。大流行中发现的携带金属-β-内酰胺酶blaSPM-1和双环霉素bcr1耐药基因的整合和结合元件的完整特征铜绿假单胞菌克隆SP / ST277。中国抗菌化学杂志,2015;70(9):2547-50。https://doi.org/10.1093/jac/dkv152.
Flannery EL, Mody L, Mobley HLT。在产脲酶尿路病原体中广泛存在的模组化致病性岛的鉴定,该致病性岛与一组不同的移动元素具有相同的特征。感染与免疫。2009;77(11):4887-4894。https://doi.org/10.1128/IAI.00705-09.
Mole BM, Baltrus DA, Dangl JL, Grant SR.植物病原细菌的全球毒力调节网络。趋势Microbiol。2007;15(8):363 - 71。https://doi.org/10.1016/j.tim.2007.06.005.
科伦比E,斯特劳布C, Künzel S,邓普顿MD,麦肯HC,雷尼PB。猕猴桃致病菌丁香假单胞菌(Pseudomonas syringae pv actinidiae)对铜抗性的进化与整合结合元素和质粒的获取。环境Microbiol。2017;19(2):819 - 32。https://doi.org/10.1111/1462-2920.13662.
潘达P,万加BR,卢A,菲里斯M,费纳兰PC,巴特勒R,阿姆斯特朗K,荣森CW,皮特曼AR。Pectobacteriumatrosepticum和Pectobacteriumcarotovorum含有独特的、独立获得的编码冠状淀粉酸的结合和结合元素,增强对马铃薯茎的毒性。Microbiol前面。2016;7:1-13。https://doi.org/10.3389/fmicb.2016.00397.
曼斯菲尔德J, Genin S, Magori S, Citovsky V, Sriariyanum M, Ronald P, Dow M, Verdier V, Beer SV, Machado MA, Toth I, Salmond G, Foster GD。分子植物病理学十大植物致病菌。植物病理学杂志,2012;13(6):614-29。https://doi.org/10.1111/j.1364-3703.2012.00804.x.
da Silva GC, Gonçalves OS, Rosa JN, França KC, Bossé JT, Santana MF, Langford PR, Bazzolli DMS。移动遗传元件驱动耐药基因在巴氏杆菌科物种中的传播。Microbiol前面。2022;12:773284。https://doi.org/10.3389/fmicb.2021.773284.
Gonçalves OS, de Queiroz MV, Santana MF。整合和结合元素(ICEs)和基因组岛的潜在进化影响Ralstonia solanacearum物种复杂。Sci众议员2020;10(1):1-13。https://doi.org/10.1038/s41598-020-69490-1.
Gonçalves OS,桑塔纳MF。酸杆菌基因组中单片段整合和结合元素的共存。基因。2021;777:145476。https://doi.org/10.1016/j.gene.2021.145476.
Toussaint A, Merlin C, Monchy S, Benotmane MA, Leplae R, Mergeay M, Springael D.联苯和4-氯联苯分解代谢转座子Tn4371,一个与IncP和Ti质粒相关的新基因组岛家族成员。应用环境微生物学,2003;69(8):4837-45。https://doi.org/10.1128/AEM.69.8.4837-4845.2003.
贝尔KS、塞拜伊亚M、普里查德L、霍尔顿MTG、海曼LJ、霍列娃MC、汤姆逊NR、宾利SD、邱彻LJC、蒙格尔K、阿特金R、巴森N、布鲁克斯K、齐灵渥斯T、克拉克K、道格特J、弗雷泽A、汉斯Z、豪泽H、托特IK。肠杆菌植物病原艾winia carotovora subsp atroseptica基因组序列及其毒力因子的鉴定。中国环境科学与工程学报2004;29(6):891 - 891。https://doi.org/10.1073/pnas.0402424101.
万加BR, Ramakrishnan P, Butler RC, Toth IK, Ronson CW, Jacobs JME, Pitman AR.植物病原体诱导植物水平获得岛2的动员Pectobacterium atrosepticum其中包括假定的松弛酶ECA0613和群体感应。环境Microbiol。2015;17(11):4730 - 44。https://doi.org/10.1111/1462-2920.13024.
Gorshkov V, Gubaev R, Petrova O, Daminova A, Gogoleva N, Ageeva M, Parfirova O, Prokchorchik M, Nikolaichik Y, Gogolev Y.转录组分析有助于识别隐藏到暴力行为的潜在和真实的分子开关Pectobacterium atrosepticum在烟草植物的系统定植过程中。植物病理学杂志,2018;32(4):359 - 359。https://doi.org/10.1007/s10658-018-1496-6.
Denancé N, Briand M, Gaborieau R, Gaillard S, Jacques MA。植物遗传关系和亚种特征的鉴定Xylellafastidiosa.BMC基因组学,2019;20(1):21。https://doi.org/10.1186/s12864-019-5565-9.
王晓东,王晓东,王晓东,等。抗生素耐药进化过程中整合和结合元素的作用。趋势Microbiol。2021;29(1):仅。https://doi.org/10.1016/j.tim.2020.05.011.
劳伦斯JG, Ochman H.大肠杆菌基因组的分子考古学。中国生物医学工程学报1998;
Marri PR, Golding GB。基因改良的证明:细菌中新生基因的旅程。基因组。2008;51:164-8。
ICE元素中的一种新的入口排除方式为元素及其宿主提供了选择优势。微评论。2019;112(7):1061 - 5。https://doi.org/10.1111/mmi.14342.
Eex和TraG的进入排斥的决定因素是细胞质。J Bacteriol。2007;189(17):6469 - 73。https://doi.org/10.1128/JB.00522-07.
Cesbron S, Briand M, Essakhi S, Gironde S, Boureau T, Manceau C, fisher - le Saux M, Jacques MA。致病性和非致病性菌株的比较基因组学、黄arboricola揭示与病理适应相关的分子和进化事件。植物科学进展2015;6:1126。https://doi.org/10.3389/fpls.2015.01126.
萨尔茨伯格SL,索默DD,沙茨MC,菲利普AM,拉比诺维奇PD, Tsuge S, Furutani A, Ochiai H, Delcher AL, Kelley D, Madupu R, Puiu D, Radune D, Shumway M, Trapnell C, Aparna G, Jha G, Pandey A, Patil PB, Bogdanove AJ。水稻致病菌水稻黄单胞菌PXO99A的基因组序列与快速进化BMC基因组学。2008;9(5月):1 - 16。https://doi.org/10.1186/1471-2164-9-204.
III型分泌系统的组装和功能。微生物学报2000;54:735-74。
III型分泌系统效应蛋白:细菌疾病和植物防御的双因子。植物病理学报2004;42(1):385-414。https://doi.org/10.1146/annurev.phyto.42.040103.110731.
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ。基本的本地对齐搜索工具。中华分子生物学杂志1990;215(3):403-10。https://doi.org/10.1016/s0022 - 2836 (05) 80360 - 2.
刘斌,郑东,金强,陈磊,杨杰。VFDB 2019:一个具有交互式web界面的比较病理基因组平台。核酸修正案2019;47(D1): D687-92。https://doi.org/10.1093/nar/gky1080.
刘敏,李鑫,谢煜,毕东,孙娟,李娟,太超,邓卓,欧海燕。冰山2.0:细菌整合和结合元素数据库的更新。核酸修正案2019;47(D1): D660-5。https://doi.org/10.1093/nar/gky1123.
李鑫,谢颖,刘敏,太春,孙杰,邓卓,欧海燕。基于网络的细菌移动遗传元件DNA序列转移起源识别工具。核酸学报2018;46(W1): W229-34。https://doi.org/10.1093/nar/gky352.
Cury J, Touchon M, Rocha EPC。综合和结合元素及其宿主:组成、分布和组织。核酸学报2017;45(15):8943-8956。https://doi.org/10.1093/nar/gkx607.
Burrus V, Pavlovic G, Decaris B, Guédon G.共轭转座子:冰山一角。摩尔Microbiol。2002;46(3):601 - 10。https://doi.org/10.1046/j.1365-2958.2002.03191.x.
Bateman a . UniProt:蛋白质知识的全球中心。核酸修正案2019;47(D1): D506-15。https://doi.org/10.1093/nar/gky1049.
Bateman A, Coin L, Durbin R, Finn RD, Hollich V, griffith - jones S, Khanna A, Marshall M, Moxon S, Sonnhammer ELL, Studholme DJ, Yeats C, Eddy SR. Pfam蛋白家族数据库。核酸学报,2004;32:38 - 41。https://doi.org/10.1093/nar/gkh121.
厄本M, Cuzick A, Rutherford K, Irvine A, Pedro H, Pant R, Sadanadan V, Khamari L, Billal S, Mohanty S, hammon - kosack KE。PHI-base:多物种病原体-宿主相互作用数据库的新接口和进一步添加。核酸AcidsResearch。2017; 45 (D1): D604-10。https://doi.org/10.1093/nar/gkw1089.
Tay, d.m., Govindarajan, K. R., Khan, a.m., Ong, t.y.r ., Samad, h.m., Soh, W. W., Tong, M., Zhang, F., & Tan, T. W. T3SEdb:由细菌III型分泌系统分泌的毒力效应物的数据仓库。BMC生物信息学。2010;11(增刊。7).https://doi.org/10.1186/1471-2105-11-S7-S4
Larkin MA, Blackshields G, Brown NP, Chenna R, Mcgettigan PA, McWilliam H, Valentin F, Wallace IM, Wilm A, Lopez R, Thompson JD, Gibson TJ, Higgins DG。Clustal W和Clustal X 2.0版本。生物信息学。2007;23(21):2947 - 8。https://doi.org/10.1093/bioinformatics/btm404.
Darling ACE, Mau B, Blattner FR, Perna NT. Mauve:保守基因组序列多重对齐与重排。基因组研究》2004;14(7):1394 - 403。https://doi.org/10.1101/gr.2289704.
吉尔克里斯特,C. L. M. & choi, Y. H. clinker & clustermap.js:基因簇比较图的自动生成。BioRxiv。2020https://doi.org/10.1101/2020.11.08.370650
胡拉舍克K, Borowiak M, Tausch SH, Malorny B, Käsbohrer A, Otani S,等。CLUSTAL W:通过序列加权、位置特定的间隙惩罚和权重矩阵选择来提高渐进式多序列对齐的灵敏度。核酸决议2019;5:102。https://doi.org/10.3390/microorganisms9030598.
Kumar S, Stecher G, Li M, Knyaz C, Tamura K. MEGA X:跨计算平台的分子进化遗传学分析。Mol Biol Evol 2018; 35:1547-9。https://doi.org/10.1093/molbev/msy096.
资金
这项工作得到了国家Desenvolvimento议会(Conselho de Desenvolvimento Científico e Tecnológico-CNPq和Coordenação de Aperfeiçoamento de Pessoal de Nível Superior -巴西(cape) -财政代码001的部分支持。
作者信息
作者和联系
贡献
MFS和JCSA构思并设计了该研究;JCSA, OSG和ASF。收集和分析数据;JCSA起草了初稿;OSG, ASF, MVQ, DMSB和MFS起草了后续版本的手稿。所有作者都对文章做出了贡献,并批准了提交的版本。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
作者声明,该研究是在不存在任何可能被解释为潜在利益冲突的商业或金融关系的情况下进行的。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
补充信息
补充文件1:补充表1。
本工作分析了丁香假单胞菌的基因组。补充表2。本文分析了茄兰氏菌种复合体的基因组。补充表3。本工作分析了农杆菌的基因组。补充表4。米黄单胞菌的基因组。Oryzae在这项工作中分析。补充表5。本文对油菜黄单胞菌的基因组进行了分析。补充表6。本研究对axonopoodis的基因组进行了分析。补充表7。本文分析了扁豆豆属植物的基因组。补充表8。本文对苛养木杆菌的基因组进行了分析。补充表9。本文分析了迪克亚属(dadantii和solani)的基因组。补充表10。本文分析了胡萝卜Pectobacterium carotovorum(和P. atrosepticum)的基因组。补充表11。在主要植物致病细菌的基因组中发现了ice。补充表12。已识别元素的大小和GC含量。补充表13。附件网站。补充表14。插入位点,整合酶和松弛酶的类型。补充表15。在这项工作中,研究人员确定了由ICEs携带的显著的货物基因。补充表16所示。文献中描述了这一工作的要素。
附加文件2:补充图1。
农杆菌ICEs的基因簇比较。补充图2。Dickeya ice的基因簇比较。补充图3。颈胡萝卜Pectobacterium carotovorum(和atrosepticum) ICEs的基因簇比较。补充图4。丁香假单胞菌第1组的基因簇比较。补充图5。丁香假单胞菌第2组的基因簇比较。补充图6。丁香假单胞菌第3组的基因簇比较。补充图7。RSSC ice的基因簇比较。补充图9。苛养木杆菌的基因簇比较。补充图10。ICEs单位矩阵热图。
权利和权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。欲查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
德·阿西斯,j.c.s., Gonçalves, o.s.,费尔南德斯,A.S.et al。基因组分析揭示了整合和结合元件在植物致病菌中的作用。移动DNA13, 19(2022)。https://doi.org/10.1186/s13100-022-00275-1
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s13100-022-00275-1
关键字
- 基因组进化
- 水平基因转移
- 可移动遗传元件(MGE)
- 植物病理学