摘要
背景
粉虱是一种农业害虫,在全球范围内对作物产量造成负面影响,有时会造成严重的经济损失和粮食不安全。的烟就其广泛的作物寄主范围和作为400多种植物病毒载体的能力而言,白蝇种类复合物的破坏性最大。属于该物种复合体的粉虱基因组提供了有价值的基因组数据;然而,这些基因组中的转座因子(TEs)仍未被探索。这项研究首次提供了TE含量的精确表征烟粉虱物种复杂。
结果
本研究发现,三种粉虱(MEAM1、MEDQ和SSA-ECA)的基因组中平均有40.61%由TEs组成。经鉴定的TEs大多为DNA转座子(平均22.85%),而SINEs(平均0.14%)最少。本研究还将三种粉虱基因组的TE含量与其他三种半翅目基因组进行了比较,发现粉虱基因组中DNA转座子明显增多,LINEs明显减少。在三种粉虱物种中共鉴定出63个TE超家族(39个DNA转座子,6个LTR, 16个LINE和2个sin)。将识别的TE序列聚类,共生成5766个TE簇。总共有2707个簇被鉴定为在粉虱基因组中唯一发现的簇,而生成的簇中没有一个来自粉虱和非粉虱的TE序列。
这项研究首次描述了不同类型的te烟粉虱他创建了一个标准化的注释工作流程,可用于分析未来的白蝇基因组。
结论
这项研究是第一个描述te景观的烟粉虱白蝇种类复杂。三种粉虱基因组中这些元素的特征表明TEs占据了重要的部分烟粉虱其中DNA转座子占绝大多数。本研究还确定了TE超家族和潜在感兴趣的TE序列簇,提供了必要的信息,并为该物种复合体中TE的未来研究提供了框架。
简介
粉虱是一种农业害虫,可造成数十亿美元的作物损失[1,2,3.].超过1500种粉虱已被确认,其中,成员烟就其广泛的作物寄主范围(例如豆类、木薯、棉花、马铃薯、番茄)和作为> 400植物病毒载体的能力而言,粉虱种类复合物是最具集体破坏性的[4,5,6].
农业集约化和气候变化导致了高度繁殖力的种群烟粉虱通过受感染植物的国际贸易在各大洲和全球蔓延[1,7,8].几十年来,这种有害物种的严重程度形成了几个国家和国际合作项目[3.],在过去十年中,基因组和转录组资源急剧增加。这些研究有助于探索这种害虫复杂物种多样化的机制,如不同的寄主特异性和解毒机制,以及植物病毒相互作用[9,10,11,12,13].在过去几年中,基因组组装草案(MEAM1、MED/Q和SSA-ECA)已发表,同时注释了与杀虫剂抗性、解毒和病毒传播相关的基因组特征[14,15,16].然而,转座因子(TEs)在所有这些研究中都被忽视了,迄今为止还没有在这个白蝇物种复合体中发现TEs的详细描述。
TEs的鉴定在基因组组装分析中是不可或缺的,因为TEs在真核生物基因组中丰富,可以繁殖,移动,影响基因调控,并扩展宿主的基因组[17,18,19,20.,21].TEs根据其转座子的方法可分为两大类:DNA转座子和反转录转座子[22,23,24,25].DNA转座子借助DNA中间体进行转座子,可以是自主的,也可以是非自主的[23,26,27].自主元素可以自行转位,而非自主元素则需要其他te来促进其移动[27,28].大多数DNA转座子使用“剪切-粘贴”转座子的方法;其中转座子从其位置被“切割”,然后“粘贴”(插入)到一个新的目标位点[18,28,29].
逆转录转座子是一种能在RNA中间体的帮助下进行转座子[30.,31,32].DNA转座子编码转座子,而逆转录转座子产生RNA转录物,它们在逆转录酶的帮助下从RNA转录成DNA,然后将序列整合到基因组中的新位点[30.,31].它们在基因组中的动员不需要切除,因此它们的运动被称为“复制-粘贴”[31,33].逆转录因子可根据其结构进一步分为两类:长末端重复逆转录转座子(LTR)和非LTR逆转录转座子[30.,31,32].
TEs可以进一步划分为超科,它们在不同节肢动物物种中的存在差异很大,目前低至2.6%贝尔基卡号南极洲以高达72.8%在Sitophilus oryzae[34,35].这些元素的功能通常是未知的,但它们在基因组中的存在与诱导宿主生物的各种变化有关。大多数昆虫TE研究都是在果蝇中进行的,其中最具特征的TE之一是P元素[36,37].P元素是在黑腹果蝇并被证明会导致杂交发育不良[38],当雌性的d .腹缺乏磷元素的雄性品系与具有自主磷元素的雄性品系交配[36,39].由此产生的组合导致后代患有不育疾病,突变率升高,染色体重排和重组增加[36,39,40].不同类型的TEs具有不同的影响,这些元素在其他昆虫物种中的特征为我们提高对这些元素潜在影响的理解奠定了基础。
TEs在基因调控和表达中的作用已经被描述[28,41,42,43,44,45,46不同白蝇基因组中TEs的丰度和类型可能决定了物种复合体的进化。TEs也与基因复制有关,其中TE的插入位置会影响正常的复制过程[17,47].过程改变的确切机制取决于TE的类型,其影响程度也相应不同[44,47,48,49].
TEs占很大比例烟粉虱基因组,约占已发表的两个基因组草案的40-44%烟粉虱称为小中东亚1 (MEAM1)和地中海Q (MED/Q)的种[14,16].最新发布烟粉虱撒哈拉以南非洲人群(SSA-ECA)的基因组草案报告了略低的TE含量(38.5%),但注意到513 Mb基因组组装丢失了大约四分之一的基因组数据[15].因此,SSA-ECA基因组的重复序列含量不能被认为是准确的。
除了经颅电损伤的比例之外烟粉虱基因组中发现的TEs,我们所知甚少烟粉虱物种复杂。此外,在上述两个完整的TE订单的报告估计中存在显著差异烟粉虱基因组草案。虽然所有的研究都报告了大约40%的基因组由TEs组成,但据报道MEAM1和SSA-ECA白蝇基因组具有丰富的DNA转座子[14,15]特别是螨虫(小型倒置重复转座元件),而ltr被报道[16在MED/Q基因组中是最丰富的。委员会成员烟粉虱物种复合体表现出非常不同的生物学和表型特性,因此这些对比的结果被认为是潜在的重要的。
研究报告了不同的TE课程比例烟粉虱粉虱基因组[14,15,16]采用了不同的TE注释工作流。在MEAM1和SSA-ECA注释中[14,15],在MED/Q重复注释工作流中加入了一种mite特异性识别工具(MITE-Hunter),而ltr特异性识别工具(LTR-Finder) [16].陈等人。[14,15]使用RepeatModeler (RECON和RepeatScout)创建了他们的特定物种重复库,并包括用于鉴定螨虫的MITE-Hunter。谢等。[16]使用Piler-DF和RepeatScout来创建他们的重复库,并包括LTR-FINDER来识别ltr。
三种粉虱的基因组组装采用了不同的基因组测序技术和组装方法。MEAM1粉虱DNA测序采用Illumina HiSeq 2500系统,Illumina配对端reads由Platanus v1.2.1组装,间隙随后使用PacBio长reads和PBJelly填充[14].MED/Q基因组组装也由Illumina配对末端reads构建,但组装使用SOAPdenovo [16]然后使用细菌人工染色体(BAC)库来改进组装。对于最近发布的SSA-ECA草图基因组组装,HiSeq 2500的配对末端和配对配对文库与Platanus [15].皮隆是用来填补空白的。SSA-ECA出版物[16]指出,尽管约25%的基因组缺失,但大多数基因空间被认为已经正确组装。
使用不同的组装方法和工作流阻碍了之前报告的三个TE类的准确比较烟粉虱基因组。可靠的推论基于在TE组成中发现的显著差异在已发表的基因组烟粉虱因此无法形成物种复合体。此外,研究人员尝试复制已发表数据中报告的鉴定工作流程,结果与使用相同基因组组合的已发表估计不一致。为了解决使用不同TE注释工作流的程序集的问题,本研究开发了一个可重复的工作流,用于识别和分类其中发现的TE烟粉虱基因组。应用相同的工作流跨所有发布烟粉虱基因组提供了标准化的TE注释过程,并强调了目前发表的一些TE组成的错误分类和高估烟粉虱基因组。本研究首次对英语教学中的TE类进行了准确的探索烟粉虱物种复杂。
结果
使用repeatmasker repBase库识别te
3个草案基因组(MEAM1, MED/Q,和SSA-ECA)烟粉虱迄今为止所发表的隐种复合体是分析的重点。这些基因组中的te最初是通过RepeatMasker使用RepBase库(version RepBase_RepeatMasker-edition20180826 library)识别出来的。使用RepeatMasker RepBase库的TE识别结果明显低于各自出版物中报道的结果(表1);MEAM1 (18.92% vs 43.82%)、MED/Q (17.28% vs 40.29%)和SSA-ECA (13.41% vs 38.52%)。
搜索RepBase库烟粉虱特异性TE和282个不同TE一致序列被鉴定出来。鉴定结果表明,只有部分鉴定的TE一致序列被提交到RepBase,在这些提交的TE一致序列中,只有不到一半的TE被鉴定出来。试图在公开可用的存储库中找到其余的共识序列是不成功的。
因此,RepBase库测试了其识别te的能力黑腹果蝇基因组(第6版[50]),以确定在这项研究中测试的半翅人基因组的异常是否是由于用户错误造成的。RepBase文库能够识别17.44%的TE基因组比例,而已发表的结果表明,在不同的果蝇研究中,< 20%的基因组被识别为TE [51,52,53,54].因此,鉴定结果与报告中在该物种中发现的结果一致,证实了文库的搜索是正确的。
使用RepeatMasker RepBase文库进行TE鉴定的结果表明,该文库不能用于粉虱基因组中TE的表征和比较。为了解决这个问题,开发了一个注释工作流,以标准化跨白蝇基因组的TEs识别。这在使用不同TE识别工具的已发表研究中有所不同;MEAM1,和SSA-ECA [14,15]使用DNA转座子特异性工具,而MED/Q [16]使用LTR特定的识别工具。注释工作流程的标准化是精确比较三个基因组的te所必需的。使用相同的工具为每个基因组创建了一个特定物种的定制重复文库,以识别和分类每个基因组中的te。工作流中TEs的识别结合了识别元素的几种方法:基于结构的和从头的;而识别的元素的分类使用序列相似性,结构和机器学习(详细信息见方法部分)。
开发的注释工作流的性能通过一个特征良好的基因组进行了验证,以确定其是否适合于在特征不太明显的昆虫基因组中注释TEs。的d .腹基因组(第6版[50])被选中进行验证,因为它被认为是最准确的TE注释之一,其参考基因组发布了几次迭代,并同时发布了TE信息[50,55].开发的注释工作流与RepeatMasker RepBase库进行了比较,因为后者使用了包含来自多个TE研究的更新的数据库和包含TE注释的库d .腹基因组释放[24,56].
在该基因中发现了穿插重复序列的基因组比例为17.44%d .腹使用RepeatMasker文库与使用特定物种定制文库发现的16.88%的穿插重复序列基因组比例相比(表2).发现的大多数重复序列是ltr,在RepeatMasker和定制库之间,这一类别的差异为0.46%。sin类元素是最不常见的;RepeatMasker库识别了81 bp的SINEs,而定制库没有发现(0 bp)。对于DNA转座子,两个文库之间的差异为0.58%,而LINEs的检测差异为0.42%。差异< 1%的总TEs中确定d .腹基因组和低于< 1%的每个顺序支持识别基因组内发现的TEs的工作流程的能力。
节肢动物基因组中的TEs
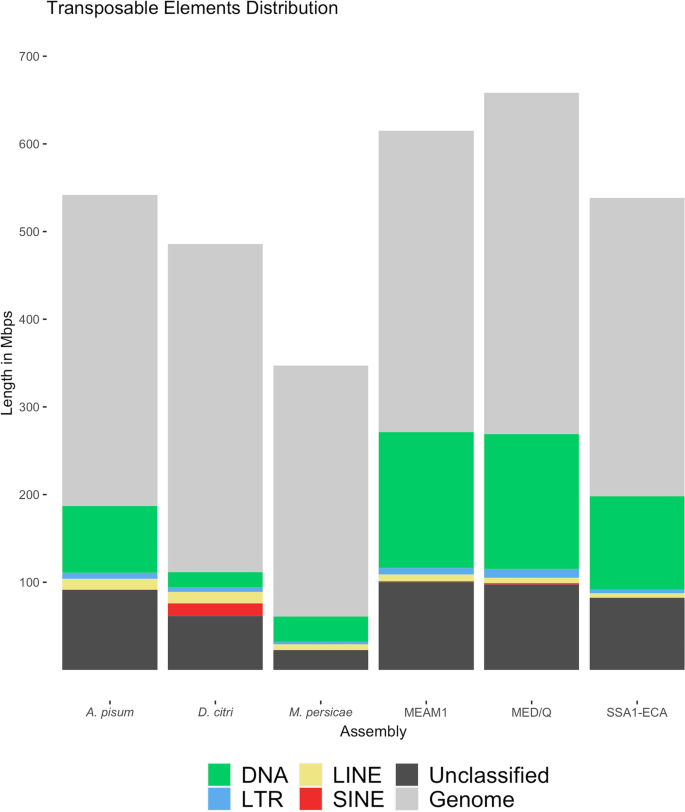
经过验证的开发工作流用于识别每个目标基因组的TE含量(图2)。1),从而为每个被研究的基因组建立了一个定制的物种特定文库。除了三个白蝇基因组(MEAM1、MED/Q和SSA-ECA)外,还包括三个半翅目基因组作为一般比较,即Acyrthosiphon pisum,Diaphorina citri,而且Myzus persicae.3个粉虱基因组TE含量均高于3个非粉虱基因组(TE基因组占比平均为25.01%),TE含量平均为40.61%。MEAM1在6个基因组中TE含量最高,为44.14%答:pisum非粉虱基因组中TE含量最高,为34.54%。SSA-ECA草案基因组(已知缺失约25%的基因组数据)的TE含量在白蝇基因组中最低,为36.80%,比白蝇基因组的TE含量高2%以上答:pisum基因组组装。的桃蚜基因组组装TE含量在6个基因组中最低,为17.52%。
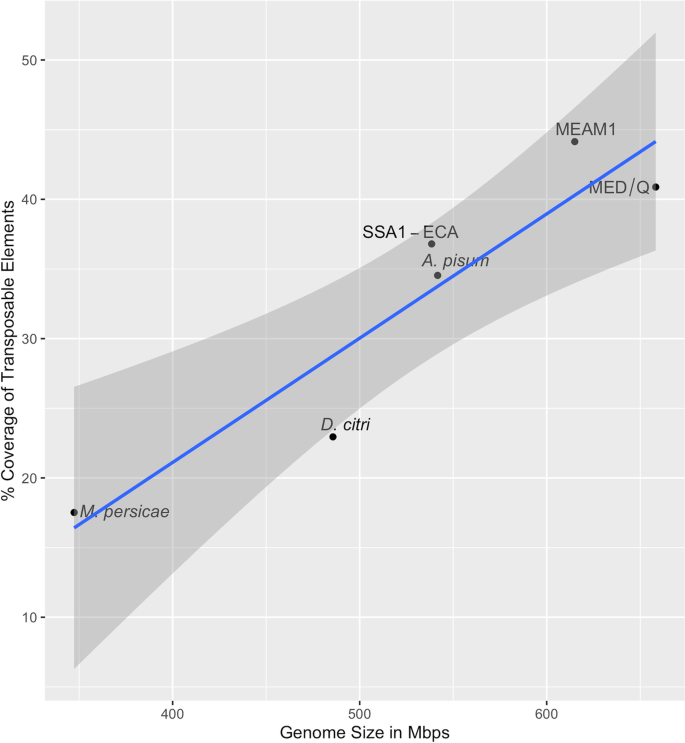
6个基因组的组装大小与其TE含量之间的关系采用Spearman秩相关检验(图。2).TE比例与装配尺寸呈正相关(r= 0.93,p= 0.006)。6个基因组中TE含量最高的是MEAM1基因组(615 Mbp),达到44.14%m . persicae基因组组装(347 Mbp) TE含量最低,为17.52%。在粉虱基因组中,SSA-ECA的组装尺寸最小(538.48 Mbp), TE基因组比例最低(36.80%)。
TE含量在基因组间分布的差异
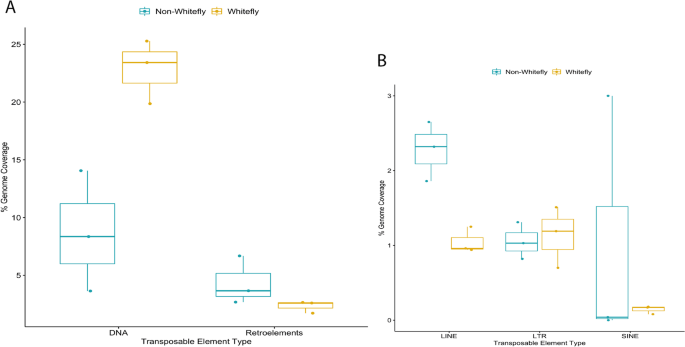
差异无统计学意义(p白蝇基因组组装大小(平均603.92 Mbp)与非白蝇基因组组装大小(平均458.24 Mbp)的差异为0.09)。这使我们能够比较两组,而不会因为基因组组装大小的变化而对我们的结果产生显著的偏差。比较了6个基因组中TEs占基因组百分比的分布情况。白蛉基因组内的分类元件以DNA转座子为主,平均占22.85%。MEAM1在三种粉虱基因组中的分布最高,为25.28%,SSA-ECA最低,为19.86%。反转录转座子在白蝇基因组中所占的比例要低得多,平均为2.32%,LTRs是三个序列中最丰富的,平均为1.13%,其次是LINEs,平均为1.05%。
在三种非白蝇基因组中,DNA转座子的含量最高答:pisum(14.06%)和m . persicae(8.35%)基因组,而反转录转座子在基因组中含量最高d . citri基因组(6.68%)。在非白蝇基因组中,反转录转座子的平均比例为4.34%。其中,LINEs的逆转录转座子序列最多答:pisum基因组(2.32%)和m . persicae基因组(1.86%),而SINEs在d . citri基因组(3%)。
在四目TEs中,SINEs最小,平均为0.58%(粉虱基因组为0.14%,非粉虱基因组为1.01%)。在所有六个基因组中d . citriSINEs序列在全基因组中所占比例最高,为3%,而TE序列在全基因组中未检测到m . persicae基因组组装。
通过比较两组基因组之间的分布,进一步探索了TEs在基因组之间的分布,以确定是否有任何特定的特征似乎是针对所研究的白蝇基因组的。使用双样本t检验(DNA转座子、LTR和LINE)和Wilcoxon秩和检验(sin)比较了粉虱和非粉虱基因组TEs顺序的分布(图2)。3.).对于方差相同的序列(DNA转座子、lts和LINEs)采用标准t检验,而对于SINEs采用Wilcoxon秩和检验作为两组基因组比例分布,因为它们具有非正态分布。DNA转座子的平均TE含量(p= 0.01)和LINEs (p= 0.008),而LTRs中无显著差异(p= 0.7856)和sin (p= 0.6625)。与研究的三种非白蝇半翅目基因组相比,在白蝇基因组中发现了明显更多的DNA转座子,而LINEs明显更少。
未分类的元素仍然可以在已识别的TEs中找到。在6个基因组中,平均13.70%的基因组比例仍未分类(白蝇基因组为15.43%,非白蝇基因组为11.98%)。利用NCBI非冗余蛋白数据库和UniProtKB/Swiss-Prot节肢动物蛋白质序列对白蝇种特异性TE文库中的未知一致序列进行检索。计划将命中的重复序列从最终的TE库中排除;然而,没有找到匹配。
最后,应当指出,当未分类的元素被分类时,这些元素的相对比例将会发生变化;然而,在白蝇基因组中鉴定的DNA转座子的比例非常高,这意味着这类将仍然是在所有三种分析的白蝇基因组中鉴定的最大顺序的元素(补充表2).
TE超家族横跨基因组
根据不同目TE的单系起源和基序的同源性,可以进一步划分为超科[27,56,57].在每个基因组中都鉴定出超家族(表3.).在粉虱基因组中共鉴定出98个TE超家族,在非粉虱基因组中鉴定出89个TE超家族。在两组基因组中共鉴定出69个TE超家族(39个DNA转座子,8个LTR, 19个LINE和3个sin)。大多数被鉴定的超家族被归类为DNA转座子,共有66个不同的超家族,其中19个是粉虱基因组所独有的,8个是非粉虱基因组所独有的。sin超家族最少,共有11个超家族,其中4个是白蝇基因组所独有的,另外4个是非白蝇基因组所独有的。LINE超家族是最确定的逆转录转座子,有29个独特的超家族,其中3个是粉虱基因组所独有的,7个是非粉虱基因组所独有的。
MEAM1的超家族数量最多,为82个m . persicae基因组最少,有61个超家族。在所有基因组中,DNA转座子超家族是最常见的,在白蝇基因组中平均有47个,在非白蝇基因组中平均有36个。MED/Q和MEAM1的转座子超家族数量最多,分别为49个和48个d . citri基因组至少有30个超家族。sin超家族最少,平均有4个超家族。的d . citri基因组中发现的sin超家族数量最多,共7个,未发现任何sin超家族m . persicae基因组组装。
通过聚类来自6个物种特异性文库的TE共识,进一步分析了基因组组装中发现的超家族。聚类基于TEs长度和80%序列相似度。总共创建了5766个集群;来自非粉虱TE一致序列的1131个簇,来自粉虱TE一致序列的2707个簇,以及来自同一基因组组装的TE一致序列的1928个簇(补充表)3.).在同一基因组组装中发现的TE一致序列的1928个簇是预期的。这些簇是从一个基因组的物种特异性文库中发现的相同超家族中创建的(即来自MEAM1的吉普赛人元素被鉴定为与另一个MEAM1吉普赛人元素80%相似)。这些预期的重叠被忽略,因为在创建特定于物种的TE库期间已经执行了聚类(参见方法).尽管基于它们的分类顺序,在基因组组装中发现了相似的重复超家族,但根据序列相似性和长度,每个组(粉虱与非粉虱)的TE一致序列都没有被确定为共享的。
粉虱TEs中2707个集群的分解(表4MEAM1和MED/Q基因组共享的簇数最多,为987个,而MED/Q和SSA-ECA共享的簇数最少,为441个。三家公司共确定了733个共享集群烟粉虱基因组。有216个已知TE簇被鉴定为DNA转座子,其中37个簇来自Helitrons, 31个簇来自不同的hAT家族,31个簇来自不同的TcMar家族。共有174个集群被确定为ltr,其中三个超家族占大多数:61个Copia集群,56个Gypsy集群和54个Pao集群。共有120个簇被确定为LINEs,其中三个超家族占了一半以上的簇:25个Jockey簇,19个L2簇和17个R1簇。分类为sin的集群被识别得最少,在733个集群中只有2个sin集群。
最后,有相当数量(1004)的粉虱TE聚类来自未分类的TE一致序列。来自MEAM1和MED/Q的未分类TE一致性序列创建了最多的聚类(318个簇),其次是来自MEAM和SSA-ECA的TE一致性序列(283个簇),三个白蝇TE一致性序列共创建了220个簇。
重复的风景
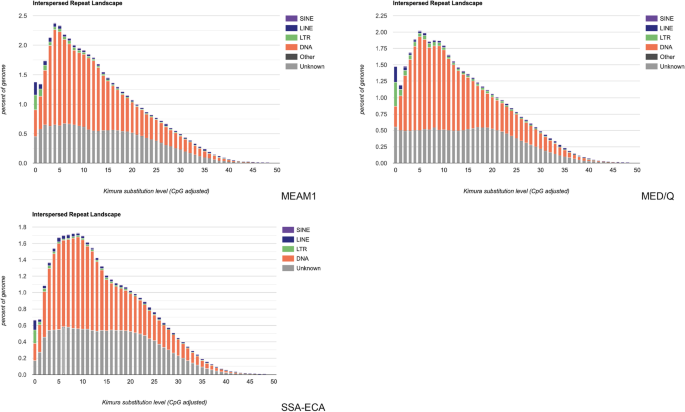
在RepeatMasker中包含的脚本的帮助下,生成了几个重复的景观。这些重复的景观显示了每个基因组内由木村距离测量的序列分歧。下图展示了每种转座子(DNA转座子、LTR、LINE、SINEs、Unknown等)拷贝的基因组覆盖分布及其与共识序列的差异。一个副本的发散度可以推断出它的活动和插入的年龄。较低的分化度分数意味着较近期的转位因子活动,而较大的分化度分数代表较早转位事件的副本。在这些图中也可以观察到活动的峰值,它们代表了特定转座因子进化史中的转座爆发[58].
数字4显示的重复景观烟粉虱基因组装配。SSA-ECA显示,在Kimura评分5 ~ 10分左右,基因组TE活性有一次延长的增加。在9 Kimura评分处,DNA转座子的基因组比例为1.12%,lts的基因组比例为0.02%,LINEs的基因组比例为0.03%,SINEs的基因组比例< 0.01,未分类TE的基因组比例为0.56%。MEAM1在4 Kimura评分时活性峰值,其DNA转座子基因组比例为1.62%,lts为0.05%,LINEs为0.06%,SINEs为0.01,未分类TE序列为0.65%。MED/Q在5 Kimura评分处活性峰值,DNA转座子的基因组比例为1.43%,lts为0.06%,LINEs为0.03%,SINEs为0.01%,未分类TE序列为0.50%。
对超家族活动的进一步分析表明,大多数DNA转座子超家族在每个白蝇基因组中达到峰值的时间轴与它们的活动峰值大致相同,而反转录转座子的活动峰值在所有三种环境中都可以在0 Kimura得分处发现(补充表)4).
讨论
这项研究是第一个描述经颅电损伤的烟粉虱物种复杂,并创建一个标准化的注释工作流,可用于分析未来白蝇基因组的发布。该物种复合体的前三个公开基因组是本次分析的重点(MEAM1、MED/Q和SSA-ECA)。我们的研究结果强调了先前发表的数据表明物种之间存在显著的TE类差异[14,15,16]是由于MED/Q草案基因组中TEs的错误识别。改进和标准化的TE注释工作流将允许在未来的研究中更准确地分析TE在白蝇物种复合体中的分布。
在基因组中鉴定TEs
与已发表的白蝇TEs鉴定结果相比,使用RepBase库的鉴定结果显著降低(表2)1)基因组,而RepBase库准确地识别了TEsd .腹基因组(表2).在所有三种白蝇基因组中,使用RepBase文库识别的TEs都不到它们各自出版物中报道的一半[14,15,16].这些结果表明RepBase库并没有包含在先前研究中识别和发表的所有白蝇TE一致序列[14,15,16].此外,从2019年4月12日起,RepBase不再公开提供,需要订阅才能访问最新版本。这些问题阻碍了TE在物种复合体中的进一步探索,并促进了TE注释工作流的开发,该工作流将标准化多个白蝇基因组的注释。
所开发的工作流程被证明可以准确地描述在基因组中发现的ted .腹基因组(表2).重复识别在不同d .腹研究报告,< 20%的基因组由TEs组成[51,52,53,54],本研究中开发的注释工作流的结果与这些发现一致。TE订单分布比例的相似性表明了所开发工作流的准确性。研究d .腹TEs最早可追溯到1980年[59], TE注释能够准确识别这些元素。
本研究试图运行Chen等人描述的TE注释工作流。[14,15]和谢等人。[16]研究以比较结果;然而,这些尝试并没有产生类似的结果,而且使用的一些工具未能与研究中包括的其他半人基因组一起运行。白蝇基因组研究公布了它们的基因组组装、TE分布内容和TE副本的GTF文件;然而,TE共识库不可用。这表明TE库是Chen等人开发的。[14,15]和谢等人。[16]研究没有提交到RepBase图书馆(或任何其他TE数据库)。
在白蝇基因组中,开发的工作流程能够在MEAM1和SSA-ECA基因组中识别相似比例的TE顺序。陈等人。[14,15]报告MEAM1(29.25%)和SSA-ECA(25.94%)中DNA转座子的丰度(表1).相比之下,据报道,在MED/Q基因组中,lts是最丰富的元素,占18.5%,只有15.66%的基因组被DNA转座子占据[16].这项研究的结果表明,在这些范围内,TEs的比例有显著的变化烟粉虱基因组是前人使用不同TE注释方法研究的产物。在MEAM1和SSA-ECA中,使用了DNA转座子特异性鉴定工具,而在MED/Q注释工作流中包含了LTR鉴定工具。这项研究强调了实施标准化工作流程以准确识别基因组间TEs差异的必要性。
TE含量与基因组组装大小
在节肢动物基因组中,TE含量与基因组大小呈正相关[34,60,61]以及其他基因组[18,44,62].彼得森等人进行的节肢动物广泛研究[34]的分布最为广泛,表明节肢动物基因组中TE比例与基因组组装大小有关。彼得森等人研究的最大基因组[34]研究(状花序migratoria5759.8 Mbp) TE比例最大(63.55%基因组比例),而研究的最小基因组(贝尔基卡号南极洲89.54 Mbp) TE比例最低(2.58%基因组比例)。
在这项研究中包括的六个基因组中也发现了同样的正相关性。的烟粉虱平均而言,白蝇基因组比非白蝇基因组更大,包含更多的te(图2)。2).的m . persicae该研究中最小的非白蝇基因组(347.31 Mbp)的TE含量最低(17.52%)。尽管基因组中的TE含量一直被证明与基因组大小相关[34,60,63],目前尚不清楚TEs是如何直接促成这一现象的,因为不同的节肢动物基因组有不同的TEs景观。在鳞翅目动物中,TE的长度和活性与基因组扩张有关;然而,工商业扩张的确切顺序仍不清楚[61].一个特定的TE顺序(DNA转座子)和基因组组装大小的关联被确定在Clitarchus hookeri基因组(60];然而,这种关系的程度尚未得到充分探讨。
TE的分类
这项研究确定了最丰富的TE在烟粉虱基因组是DNA转座子,与研究中包括的其他半翅目基因组相比,白蝇物种的基因组明显更高。平均而言,与彼得森等人研究中分析的不同节肢动物进化支相比,三种白蝇基因组的DNA转座子也更高(22.85%)[34];半翅目(平均3.24%),鳞翅目(平均1.40%),膜翅目(平均2.83%),果蝇(1.67%)。
DNA转座子在植物基因组中大量存在,并被观察到具有不同的作用;基因表达、基因组扩增、基因调控与基因组进化[42,60,61].DNA转座子可以作为顺式调控元件,增加附近基因的表达,也可以由于其产生的小rna而减少和沉默基因的表达[41,42].
在节肢动物中,DNA转座子已被观察到在基因组扩张中发挥作用[34,60].DNA转座子被鉴定为最丰富的TEc . hookeri基因组和与其他三种多翅目基因组的比较显示了DNA转座子和基因组组装大小的关联[60].多翅目动物基因组中特定DNA TE超家族的存在和缺失揭示了这种关联;然而,TEs引起的膨胀机制还有待进一步研究。在DNA转座子中发现的显著差异烟粉虱类群可能是为什么在物种复合体中发现的基因组比研究中包括的其他半翅类基因组更大的原因之一。
物种复合体中DNA转座子的丰度已在MEAM1中报道过[14]及SSA-ECA [15]基因组,但没有进一步探索。在粉虱基因组中存在常见和独特的DNA转座子超家族,突出了这种TE顺序在该物种中的重要性。为了进一步了解这些元素在物种复合体中存在的背景,需要在特征描述之外进行更详尽的探索。
与研究的三种非白蝇基因组相比,白蝇中的LINEs明显更少。平均而言,与彼得森等人研究中分析的不同节肢动物进化支相比,三种粉虱基因组中鉴定出的LINEs也更少(1.05%)[34];半翅目(平均5.14%)、鳞翅目(平均5.17%)和果蝇目(4.34%)。大多数昆虫的LINE研究都是在果蝇上进行的。在d .腹,携带特定的非ltr逆转录转座子的菌株表现出杂交发育不良[28,64].这些昆虫的后代变得不育,突变和染色体重排的频率增加[28,64].已经观察到line在宿主生物的进化生命周期中成功地维持自己[65,66,67].R1和R2 LINE超家族在28S核糖体RNA基因附近的位点特异性插入确保了其繁殖,同时也有证据表明另一个LINE超家族通过非位点特异性插入成功地维持了自身[65,66,67,68].在不同的昆虫基因组中可以发现不同的LINEs超家族,每个超家族都可能导致不同的影响,这取决于插入的类型和区域[28,34,61,69].在粉虱物种复合体中LINEs低分布的后果是未知的,在昆虫进化的更广泛背景下探索这些元素是有必要的。
SINEs是白蝇基因组中最低的TEs。正弦的转置需要直线[28,70],物种复合体中SINEs的低分布可能与LINEs的低分布有关。然而,应该注意的是,即使使用RepBase库识别了已知的sin,工作流也难以识别sin1;补充表1).工作流也无法识别在m . persicae而且d .腹基因组。
在不同节肢动物TE研究中,SINEs的识别困难一直是一个挑战。在Petersen等人进行的节肢动物范围TE识别中[34],在研究中包括的73个基因组中的7个中没有发现sin序列。有可能在这些基因组中真的没有发现sin;然而,在同一生物体中发现的已识别的SINEs的比例有多个不一致的报告。彼得森等人[34]报告了2.07%的SINEs基因组比例邮差墨尔波墨基因组和9.41%b .森基因组。在h·墨尔波墨TE分析,Lavoie等[69]在基因组比例为8.22%时识别更多;当在b .森由osanai - futaashi等人完成的TE分析[71], 12.8%的基因组被鉴定为SINEs。反转录转座子的大小增加了用自动化TE注释工具识别sin的难度[72,73].sin是TEs中最短的,在识别这些元素时受到的影响最大。
相当大比例的te在白蝇物种复合体基因组中已识别的元素中仍未分类。这些未知元素被筛选为潜在的蛋白质序列和基因片段;然而,筛查没有得到阳性结果。其中一些未知元素也被发现在三种白蝇基因组中共享。这些未知元素的意义值得进一步研究和验证,以改进对这些元素的分类和理解。
最后,三分之一的元素仍然未知,应该注意的是,TE类中的类的分布可能会发生变化;然而,DNA转座子在烟粉虱物种复合体仍然是最丰富的,因为在物种复合体中超过一半的已识别元素是DNA转座子。
TEs和超家族的景观烟粉虱基因组
重复景观突出了TEs及其活动的差异烟粉虱基因组。三种白蝇基因组中最丰富的DNA转座子超家族是hAT超家族。hAT超家族是最具代表性的转座子元件之一,它还包括第一个被发现的可移动DNA元件,即激活玉米转座子[45,74].hAT超家族的一般结构为2.5-5 k bp,末端倒置重复序列跨度可达50 bp,产生长达8 bp的目标位点重复(TSD),编码包含转座酶结构域的单个蛋白[27].在这项研究中还出现了13个代表不同谱系的hAT超家族。hAT家族中的一些特定元素已被探索,以确定它们的功能、结构和演化[27,74].hat相关序列存在于不同的生物中,包括人类、线虫、苍蝇、真菌和植物[74].
对于逆转录转座子,在这三个基因中发现了三个活跃的重复家族烟粉虱基因组。结果表明,Gypsy和Pao是所有基因组中LTR超家族中最活跃的两个。吉普赛人元素首次出现在d .腹基因组及其序列与脊椎动物逆转录病毒具有高度相似性[28].Gypsy元件有很高的转位率,并被证明插入内含子并通过破坏正常的转录控制来影响基因表达[75,76].Pao元素是LTR元素,与吉普赛元素有关,据说起源于家蚕基因组(28,77].Pao元件编码GAG和pol蛋白,并在插入基因组后产生4-6个TSD [25,77].在LINEs中,RTE-BovB是最活跃的超家族烟粉虱基因组。在RepBase分类中,BovB被归为RTE组,该组中的重复序列具有编码蛋白的两个功能域的能力;ap -核酸内切酶(无嘌呤酶)和逆转录酶[25,27].Bov-B(牛b)元素已在牛在其他真核生物基因组中观察到有水平转移事件[78,79,80,81].
MEAM1和MED/Q基因组中重复序列的分布形状相似。MEAM1和MED/Q共享的集群数量也最多。这两个白蝇种是相对密切相关的系统发育分析烟粉虱物种复合体[8,82].除了分布的形状外,两个基因组之间的扩展趋势也相似,因为它们都有目前最活跃的超家族,即CACTA, hAT, RTE-BovB, Copia, Pao和Gypsy。科皮亚的活动在烟粉虱在非白蝇组中基因组覆盖率较低除了答:pisum基因组组装。Copia元件是自主的LTR反转录转座子,其定义特征是整合酶结构域的位置[27,83].Copia元素可以追溯到更远的植物中,而在昆虫中则更活跃[84,85].他们最近一直活跃在果蝇基因组,假设它们可能是水平传播的[85].
DNA转座子活性扩增降低,LINE和LTR活性增加烟粉虱基因组装配。目前还不完全清楚这些趋势如何影响它们各自的基因组。采用木村替代模型确定元素的相对年龄;然而,为了在更客观的时间尺度内确定元素的年龄,需要确定粉虱的进化速度。
粉虱物种复合体TE研究的未来
随着标准化工作流程的可用性和粉虱物种复合体的特征性TEs,现在可以对这些元素的活性进行进一步研究。TEs对生物特性(如寄主植物定植、多食、解毒、病毒传播)和粉虱物种复合体成员多样化的影响将是进一步研究的优先领域。
结论
TEs占据了白蝇基因组的重要部分,但迄今为止,还没有研究准确地描述了在白蝇基因组中发现的TEs的分布烟粉虱物种复杂。这项研究首次探索了TE在物种复合体中的分布,并创建了一个工作流来标准化多个白蝇基因组中元素的表征。TE注释工作流的标准化已经在物种复合体中确定了大量的DNA转座子,并在所有发表的文章中证明了这一点烟粉虱与先前发表的结果相矛盾的基因组[16].其他值得注意的TE超家族也被鉴定出来,其中一些超家族被证明是特定于白蝇基因组的。未分类的元素仍然重要,已知元素的生物学意义也仍然未知。这些问题强调了进一步探索这种粉虱物种复合体不同基因组中的这些元素的必要性。这项研究为未来在该物种内的TE研究提供了基础,并希望这些元素的初步表征将增加人们对在该物种内发现的TE的兴趣烟粉虱物种复杂。
方法
基因组数据集
这项研究包括了六种不同的节肢动物基因组。其中三个基因组来自烟粉虱研究包括隐种复合体;MEAM1 [14], med / q [16],以及SSA-ECA [15].MEAM1 (烟粉虱1)基因组组装从GenBank获得,登录号为GCA_001854935.1。MED/Q (烟粉虱地中海)的基因组组装www.gigadb.org/dataset/100286.SSA1-ECA(撒哈拉以南1中东部非洲)基因组组装从ftp://www.whiteflygenomics.org/pub/whitefly/SSA-ECA/v1.0/.
其他三个节肢动物基因组是非粉虱基因组,被纳入工作流程的性能评估,并与粉虱基因组鉴定的TEs结果进行比较;Acyrthosiphon pisum(项目加入ABLF01000000)[86),Diaphorina citri(项目加入AWGM01000000)[87),而Myzus persicae(项目代码LXJY01000000)[88].所有三个基因组组合都是从NCBI使用他们的项目登录代码获得的。
重复鉴定
本研究中为创建特定物种重复库而执行的工作流。要研究的基因组组装首先提交给MITETracker (https://github.com/INTABiotechMJ/MITE-Tracker) [89]和转座子psi_08222010 (http://transposonpsi.sourceforge.net/)作为识别的第一步。然后将基因组组装提交给genometools v1.5.9 (LTRHarvest和LTRDigest)。末端重复长度为100 ~ 6000bps且相似度≥85%的元素被识别为lts。由MITETracker和基因组工具生成的TEs代表序列被组合起来创建一个文库,并提交给RepeatMasker以屏蔽在基因组组装中发现的TEs副本。然后将被掩盖的基因组组装提交给RepeatModeler v1.0.11 [90]用于从头TE鉴定。已经识别的TE副本的副本的屏蔽阻止了RepeatModeler识别和建模已经识别的重复序列。来自MAKER-P管道的实用脚本也被用于帮助genometools v1.5.9 (LTRHarvest和LTRDigest), RepeatModeler v1.0.11和RepeatMasker v4.1.1结果的解析[91].
每个程序都有候选序列,它们被识别为重复元素,四个输出随后合并到一个库中,然后提交给USEARCH v11.0.667 [92,93].USEARCH的聚类算法采用了一种称为“贪婪算法”的算法,它基于当前选项实现“最佳”解决方案。这意味着序列输入顺序在识别候选共识序列时很重要,因为每个聚类的选项是基于库中序列的顺序的。使用USEARCH的“-sortbylength”命令进行排序,并根据≥80%的序列相似度创建聚类。然后从每个聚类中产生一个共识序列,以获得一个具有代表性的序列。代表性序列与成员序列相似度≥80%。所有具有代表性的序列之间相似度< 80%。该过程减少了冗余,并有助于识别退化重复元素。
重复识别工作流生成的重复库经过一系列步骤对每个一致性序列进行分类。用于分类的第一种方法是基于同源的方法。重复库被提交给RepeatClassifier(https://github.com/rmhubley/RepeatModeler/blob/master/RepeatClassifier),然后将未分类的序列提交至网页浏览器版本的Censor [94].在继续进行下一步分类之前,删除< 70 bp的序列,并将该方法分类的序列进行分类。该库随后提交给TEClass v2.1.3 [95]和PASTEClassifier v1.0 [96].人工策展分析两种工具的结果。排序是基于序列的相似度和序列对齐的长度。当两种工具的结果相似且跨度≥元素长度的80%时,该分类被接受。当分类结果在类别级别上(即DNA转座子和retro转座子)存在差异时,该元素仍然未知。当分类导致顺序差异(即LINEs vs sin, LTR vs NonLTR)并且识别出≥80%的序列长度时,则使用其更一般的分类级别对元素进行分类。任何序列长度小于80%的结果均被忽略。
利用2019年7月10日获得的NCBI nr蛋白数据库(version 2019.08.05)和UniProtKB/Swiss-Prot Arthropoda蛋白序列对未知序列进行Blast检索。该计划是识别命中的未知序列并解析结果,并从特定物种库中删除命中超过50 bps的序列。所有未知序列均未产生爆炸结果,未知序列被接受为未分类TEs。
将基于同源性的分类结果、TEClass和PASTEClassifier的共识分类结果以及未知序列进行组合,生成最终的文库。对本研究中包括的基因组产生的每个重复文库重复该过程。
基因组组装大小和TE分布跨物种分析
每个基因组中发现的te比例从RepeatMasker v4.1.1输出表中获得。6个基因组的基因组组装大小与TE含量之间的关系采用斯皮尔曼秩rho相关检验。斯皮尔曼排名相关性测试两个排名变量或一个排名变量和一个测量变量之间的关联。关系确定变量是否共变(当另一个变量的值变化时,变量增加/减少)。
使用标准t检验和Wilcoxon秩和检验进一步比较各组基因组中各阶TEs的比例。这两个测试都比较测量变量的平均值,并确定平均值是否有显著差异。在本研究中,测试确定了粉虱与非粉虱基因组中各目TE比例是否存在显著差异。方差相近的TE阶数采用标准t检验,非正态分布的TE阶数采用Wilcoxon秩和检验。
数据和材料的可用性
MEAM1,答:pisum,d . citri,m . persicae基因组组装可在NCBI获得,登录号为GCA_001854935.1,项目代码为ABLF01000000,项目代码为AWGM01000000,项目代码为LXJY01000000。MED/Q基因组组装可在www.gigadb.org/dataset/100286.SSA1-ECA基因组组装可在ftp://www.whiteflygenomics.org/pub/whitefly/SSA-ECA/v1.0/.特定物种重复库已提交给DFAM,目前正在审查中。物种特定的重复库和相关的下游分析脚本也可从相应的作者请求。
改变历史
2022年6月8日
缩写
- BAC:
-
细菌人工染色体
- GTF:
-
基因转移格式
- 线:
-
长时间穿插的核元素
- LTR:
-
长终端重复
- 螨:
-
微型反向重复转座元件
- NonLTR:
-
非长终端重复
- sin:
-
短而穿插的核元素
- TE:
-
转座因子
参考文献
Seal SE, VandenBosch F, Jeger MJ。影响海棠病毒进化的因素及其日益增长的全球意义:对可持续控制的影响。植物科学2006;25:23-46。https://doi.org/10.1080/07352680500365257.
Naranjo SE, Chu CC, Henneberry TJ。棉花烟粉虱(同翅目:烟粉虱科)经济伤害水平:作物价格、防治成本和防治效果的影响作物,1996;15:779-88。https://doi.org/10.1016/s0261 - 2194 (96) 00061 - 0.
Oliveira MRV, Henneberry TJ, Anderson P.烟粉虱的历史、现状和合作研究项目。作物学报2001;20:709-23。
Martin JH, Mound LA。世界上白蝇的注释清单。木兰出版社;2007.www.mapress.com/zootaxa/.
Abd-Rabou S, Simmons AM。埃及烟粉虱生殖寄主植物调查(半翅目:烟粉虱科)及新寄主记录。昆虫学报。2010;121:456-65。https://doi.org/10.3157/021.121.0507.
Navas-Castillo J, Fiallo-Olivé E, Sánchez-Campos s。植物病理学杂志,2011;49:219-48。
MacFadyen S, Paull C, Boykin LM, De Barro P, Maruthi MN, Otim M,等。东非农业景观中的木薯粉虱、烟粉虱(半翅目:烟粉虱科):数量决定因素综述。Bull Entomol res 2018; 108:565-82。https://doi.org/10.1017/S0007485318000032.
马志伟,李志强,李志强,等。乌干达农作物和未栽培植物上白蝇(Bemisia spp.)的遗传多样性:对控制非洲这种毁灭性害虫物种的影响。中国生物医学工程学报(英文版);https://doi.org/10.1007/s10340-021-01355-6.
Malka O, Santos-Garcia D, Feldmesser E, Sharon E, Krause-Sakate R, Delatte H,等。烟粉虱的物种复杂多样化和寄主-植物关联:通过RNA-Seq分析揭示的植物防御和解毒视角。分子生物学。2018;27:42 - 56。https://doi.org/10.1111/mec.14865.
马尔卡O, Feldmesser E, van Brunschot S, Santos-Garcia D, Han WH, Seal S,等。烟粉虱物种复合体中决定不同程度多食的分子机制。人类进化,2021;14:807-20。https://doi.org/10.1111/eva.13162.
艾德林·哈拉里O,桑托斯·加西亚D,穆塞里M,莫什茨基P,帕特尔M,维森迪P,等。烟粉虱物种复合体中谷胱甘肽s -转移酶家族的分子进化。基因组生物学进化2020;12:3857-72。
迟勇,潘丽丽,Bouvaine S,范YY,刘永强,刘思生,等。斯里兰卡木薯花叶病毒由三种隐种烟粉虱复合体传播的差异。病毒学。2020;540:141-9。https://doi.org/10.1016/j.virol.2019.11.013.
范友友,钟玉文,赵娟,迟勇,Bouvaine S,刘ss,等。烟粉虱囊泡相关膜蛋白2与begomovirus相互作用并在病毒获取中发挥作用。细胞。2021;10(7):1700。
陈伟,长谷川DK, Kaur N, Kliot A, Pinheiro PV, Luan J,等。全球作物害虫烟粉虱MEAM1的基因组草案为病毒传播、宿主适应和杀虫剂抗性提供了新的见解。BMC生物学2016;14:110。https://doi.org/10.1186/s12915-016-0321-y.
陈伟,吴秀娜,长谷川DK, Casinga C,史瑞玛RR, Fiaboe KKM,等。非洲木薯粉虱基因组及非洲木薯定殖粉虱的分布和遗传多样性。生物化学学报,2019;https://doi.org/10.1016/j.ibmb.2019.05.003.
谢伟,陈超,杨震,郭林,杨霞,王东,等。甘薯粉虱MED/Q的基因组测序。Gigascience。2017;6:1-7。https://doi.org/10.1093/gigascience/gix018.
Correa M, Lerat E, Birmelé E, Samson F, Bouillon B, Normand K,等。人类基因的转座因子环境因其复制状态和本质不同而不同。中国生物医学工程学报。2021;13:eva062。https://doi.org/10.1093/gbe/evab062.
Kidwell毫克。真核生物中转座因子与基因组大小的进化。遗传。2002;115:49 - 63。https://doi.org/10.1023/A:1016072014259.
Smith CD, Edgar RC, Yandell MD, Smith DR, Celniker SE, Myers EW,等。双翅目昆虫重复识别和掩蔽的改进。基因。2007;389:1-9。https://doi.org/10.1016/j.gene.2006.09.011.
Holt C, Yandell M. MAKER2:用于第二代基因组项目的注释管道和基因组数据库管理工具。生物信息学。2011;12:491。https://doi.org/10.1186/1471-2105-12-491.
张志刚,张志刚,张志刚,等。单分子转录本测序在真核基因预测中的应用。中国生物工程学报。2015;16:184。https://doi.org/10.1186/s13059-015-0729-7.
芬尼根DJ。真核转座因子与基因组进化。《趋势》,1989;5 C: 103-7。
芬尼根DJ。转座的元素。《生物学报》1992;2:861-7。
Jurka J, Kapitonov VV, Pavlicek A, Klonowski P, Kohany O, Walichiewicz J. Repbase update,一个真核生物重复元素数据库。细胞遗传学杂志,2005;
刘志强,李志强,李志强,等。真核生物转座因子的统一分类体系。Nat Rev Genet. 2007; 8:973-82。
Piégu B, Bire S, Arensburger P, Bigot Y.转座元素分类系统的调查-呼吁基本更新,以满足其多样性和复杂性的挑战。生物信息学学报,2015;
小岛KK。真核生物转座因子的结构和序列多样性。基因工程学报。2019;94:233-52。
Galun E.转座元素。多德雷赫特:施普林格荷兰;2003.https://doi.org/10.1007/978-94-017-3582-7.
费肖特C,普里瑟姆EJ。DNA转座子与真核生物基因组的进化。《热内年鉴》2007;41:31 31 - 68。https://doi.org/10.1146/annurev.genet.40.110405.090448.
Eickbush TH。反转位子活动。在:布伦纳S,米勒jhht - e的G,编辑。遗传学百科全书。纽约:学术出版社;2001.p . 1699 - 701。https://doi.org/10.1006/rwgn.2001.1111.
艾克布什TH,马利克HS。逆转录转座子的起源和进化。在:移动DNA 2。美国微生物学会;2014.1111 - 44页。
芬尼根DJ。反转位子活动。中国生物医学杂志,2012;22:R432-7。https://doi.org/10.1016/j.cub.2012.04.025.
Kazazian HH, Scott AF.“复制和粘贴”人类基因组中的转座因子。中国科学(d辑),2001;https://doi.org/10.1172/JCI116400.
Petersen M, Armisén D, Gibbs RA, Hering L, Khila A, Mayer G,等。节肢动物中转座因子的多样性和进化,特别是昆虫。BMC进化生物学2019;19:11。https://doi.org/10.1186/s12862-018-1324-9.
Parisot N, Vargas-Chávez C, Goubert C, Baa-Puyoulet P, Balmand S, Beranger L,等。谷物害虫米象富含转座因子的基因组。BMC生物学。2021;19:2021.03.03.408021。https://doi.org/10.1186/s12915-021-01158-2.
葛瑞菲斯a. JF,格巴特WM,列万廷RC,米勒JH。现代遗传分析。第二。纽约:W.H. Freeman & Co. Ltd;2002.2020-03-23。
吉尔伯特C, Peccoud J, Cordaux R.转座因子与昆虫进化。昆虫学报。2021;66:355-72。https://doi.org/10.1146/annurev-ento-070720-074650.
黑腹果蝇雄性的自发重组。自然科学学报(英文版),1971;https://doi.org/10.1073/pnas.68.2.268.
马宗达* S,里约热内卢DC。P果蝇和其他真核生物的转座因子。微生物学杂志2015;3:MDNA3-2014。https://doi.org/10.1128/microbiolspec.mdna3-0004-2014.
凯莱赫。通过piRNA沉默镜头重新审视黑腹果蝇p元素的入侵。遗传学。2016;203:1513-31。https://doi.org/10.1534/genetics.115.184119.
内藤K,张峰,Tsukiyama T,齐藤H, Hancock CN, Richardson AO,等。突然大量转座子扩增对水稻基因表达的意外后果。大自然。2009;461:1130-4。
韩明梅,周庆泽,张海辉,童霞,陆超,张震,等。昆虫中微型倒转重复序列转座元件的全基因组图谱。数据库。2016;2016:baw48。https://doi.org/10.1093/database/baw148.
Kim J, Martignetti JA, Shen MR, Brosius J, Deininger P.鼠类BC1 RNA基因作为ID元素扩增的主基因。中国科学院学报(自然科学版)1994;https://doi.org/10.1073/pnas.91.9.3607.
Bourque G, Burns KH, Gehring M, Gorbunova V, Seluanov A, hamell M,等。关于转座因子你应该知道的十件事06生物科学0604遗传学基因组生物学,2018;19:199。https://doi.org/10.1186/s13059-018-1577-z.
玉米变异位点的起源和行为。自然科学学报。1950;36:344-55。https://doi.org/10.1073/pnas.36.6.344.
Biémont C.转座因子地位简史:从垃圾DNA到进化中的主要参与者。遗传学。2010;186:1085 - 93。https://doi.org/10.1534/genetics.110.124180.
王晓明,蒋晓明。转座元件对宿主基因复制的影响。Curr Opin Genet Dev. 2018; 49:63-9。https://doi.org/10.1016/j.gde.2018.03.005.
Morgante M, Brunner S, Pea G, Fengler K, Zuccolo A, Rafalski A.利用helitron样转座子进行基因复制和外显子改组产生玉米种内多样性。Nat Genet, 2005; 37:997-1002。
王华。转座因子在植物基因组结构、功能和进化中的作用。植物学报。2014;65:505-30。
Hoskins RA, Carlson JW, Wan KH, Park S, Mendez I, Galle SE,等。黑腹果蝇基因组的Release 6参考序列。Genome Res. 2015; 25:45 - 58。https://doi.org/10.1101/gr.185579.114.
Goubert C, Modolo L, Vieira C, Moro CV, Mavingui P, Boulesteix M. De novo用dnaPipeTE对亚洲虎蚊(Aedesalbopictus)重复组进行组装和注释,并与黄热病蚊(埃及伊蚊)进行比较分析。中国生物工程学报。2015;7:1192-205。https://doi.org/10.1093/gbe/evv050.
汤姆·希尔。转座元素动力学在果蝇系统发育中是一致的,尽管其含量有很大差异。bioRxiv。2019; 2:1-29。https://doi.org/10.1101/651059.
Mérel V, Boulesteix M, Fablet M, Vieira C.果蝇转座因子。暴徒。DNA。2020; 11:23。https://doi.org/10.1186/s13100-020-00213-z.
Repeatmasker.org。D. melanogaster[果蝇]基因组数据集。http://www.repeatmasker.org/species/dm.html.2020年1月12日访问。
Kaminker JS, Bergman CM, Kronmiller B, Carlson J, Svirskas R, Patel S,等。果蝇黑腹常染色质的转座因子:基因组学的观点。中国生物医学工程学报。2002;3:research0084 - research0084。https://doi.org/10.1186/gb-2002-3-12-research0084.
鲍文华,郭志刚,张志刚,张志刚,等。生物基因组重复元素数据库的研究进展。暴徒的DNA。2015; 6:11。
袁耀文,魏斯勒。真核生物剪切粘贴转座酶超家族的催化结构域。中国科学院学报(自然科学版),2011;https://doi.org/10.1073/pnas.1104208108.
通过核苷酸序列的比较研究估计碱基取代的进化速率的一种简单方法。中华药理学杂志,1980;16(4):344 - 344。
李志刚,李志刚,李志刚,李志刚。果蝇杂交发育异常的研究进展。科学(80-)。1980; 207:606-11。https://doi.org/10.1126/science.6766221.
吴c, Twort VG, Crowhurst RN, Newcomb RD, Buckley TR.组装大基因组:对竹节虫(Clitarchus hookeri)基因组的分析揭示了与繁殖相关的高重复含量和性别偏向基因。BMC Genomics, 2017;18:884。https://doi.org/10.1186/s12864-017-4245-x.
张晓东,李志强,李志强,等。在木白(瘦蝶)蝴蝶中,由于转座因子过度活跃,基因组大小迅速增加。中国生物医学杂志,2017;9:2491-505。https://doi.org/10.1093/gbe/evx163.
Naville M, Henriet S, Warren I, Sumic S, Reeve M, Volff JN,等。由非自主转座元件扩展驱动的基因组大小的巨大变化。中国生物医学杂志2019;29:1161-1168.e6。
张志刚,张志刚,张志刚。26种蝇类基因组大小和转座因子含量的系统发育惯性。生物学杂志2016;12:20160407。https://doi.org/10.1098/rsbl.2016.0407.
福西特DH,李斯特CK,凯勒特E,芬尼根DJ。控制D. melanogaster I-R杂交发育不良的转座因子与哺乳动物系相似。细胞。1986;47:1007-15。
车床WC,伯克WD, Eickbush DG, Eickbush TH。果蝇属R1逆转录转座因子的进化稳定性。中国生物医学杂志,1995;12:1094-105。
车床WC,艾克布什TH。R2逆转录转座因子的单一谱系是果蝇rDNA位点的一个活跃的、进化稳定的组成部分。中国生物医学杂志,1997;https://doi.org/10.1093/oxfordjournals.molbev.a025732.
蚊虫中的Juan非ltr逆转录转座子:基因组影响、垂直传播和近期广泛活动的迹象。BMC进化生物学。2007;7:12 2。https://doi.org/10.1186/1471-2148-7-112.
Jakubczak JL, Burke WD, Eickbush TH。逆转录转座元件R1和R2可以阻断大多数昆虫的rRNA基因。中国科学院学报(自然科学版),2001;https://doi.org/10.1073/pnas.88.8.3295.
Lavoie CA, Platt RN, Novick PA, Counterman BA, Ray DA。鞘翅目转座因子的进化提示鳞翅目基因组的多样性。暴徒的DNA。2013; 21。https://doi.org/10.1186/1759-8753-4-21.
大岛K,冈田N.正弦和线:真核生物基因组的共生体与一个共同的尾巴。细胞遗传学杂志,2005;https://doi.org/10.1159/000084981.
杨晓东,杨晓东,杨晓东,陈晓明。家蚕转座因子在家蚕中的分布与筛选。家蚕昆虫生物化学。2008;38:1046-57。https://doi.org/10.1016/j.ibmb.2008.05.012.
王志伟,Rodriguez-Tomé P,史佩柏,王志伟,王志伟,等。人类内源性逆转录病毒花叶型的分类和特征是常见的。Retrovirology。2016;第13章第7节。https://doi.org/10.1186/s12977-015-0232-y.
费林JM, Hubley R, Goubert C, Rosen J, Clark AG, Feschotte C,等。RepeatModeler2用于自动发现转座因子家族的基因组。中国科学院学报(自然科学版)2020;https://doi.org/10.1073/pnas.1921046117.
鲁宾E,利斯威克G,利维AA。hAT转座子超家族的结构与演化。遗传学。2001;158:949-57。https://doi.org/10.1093/genetics/158.3.949.
Herédia F,洛雷托ELS,瓦伦蒂VLS。吉普赛人在果蝇物种中的复杂进化。Mol Biol evolol 2004; 21:1831-42。https://doi.org/10.1093/molbev/msh183.
Kim A, Terzian C, Santamaria P, Pélisson A, Prud’homme N, Bucheton A.无脊椎动物中的逆转录病毒:吉普赛逆转录转座子显然是一种具有传染性的黑腹果蝇逆转录病毒。中国科学院学报(自然科学版),1994;https://doi.org/10.1073/pnas.91.4.1285.
熊勇,Burke WD, Eickbush TH。Pao,一种来自家蚕的高度发散的逆转录转座因子,包含长末端重复序列和推定的R区串联拷贝。核酸决议1993;21:2117-23。https://doi.org/10.1093/nar/21.9.2117.
裴康J,卢瓦索V, Cordaux R,吉尔伯特C.昆虫转座因子的大规模水平转移。中国科学院学报(自然科学版),2017;https://doi.org/10.1073/pnas.1621178114.
Walsh AM, Kortschak RD, Gardner MG, Bertozzi T, Adelson DL。广泛的逆转录转座子水平转移。中国环境科学学报,2013;26(2):366 - 366。https://doi.org/10.1073/pnas.1205856110.
Elsik CG, Tellam RL, Worley KC, Gibbs RA, Muzny DM, Weinstock GM,等。牛磺酸牛的基因组序列:反刍动物生物学和进化的窗口。科学(80-)。2009; 324:522-8。https://doi.org/10.1126/science.1169588.
伊万切维奇AM,科尔沙克RD,贝尔托齐T,阿德尔森DL。BovB和L1反转录转座子在真核生物中的水平转移。基因组生物学,2018;19:85。https://doi.org/10.1186/s13059-018-1456-7.
De Barro PJ, Liu SS, Boykin LM, Dinsdale AB.烟粉虱的物种地位。昆虫学报,2011;56:1-19。
邱飞,王志刚,王志刚。三种野生向日葵长端重复逆转录转座子的基因组丰度和转录活性。BMC植物生物学,2018;18:6。https://doi.org/10.1186/s12870-017-1223-z.
Sabot F, Schulman AH。植物的寄生和逆转录转座子生命周期:基因组的搭便车指南。遗传(Edinb)。2006; 97:381-8。https://doi.org/10.1038/sj.hdy.6800903.
White SE, Habera LF, Wessler SR.正常植物基因侧翼区域的反转录转座子:复制样元件在基因结构和表达进化中的作用。中国科学院学报(自然科学版)1994;https://doi.org/10.1073/pnas.91.25.11792.
Richards S, Gibbs RA, Gerardo NM, Moran N, Nakabachi A, Stern D,等。豌豆蚜虫Acyrthosiphon pisum的基因组序列。公共科学图书馆。2010;8(2):e10000313。
Saha S, Hosmani PS, Villalobos-Ayala K, Miller S, Shippy T, Flores M,等。柑橘黄龙病昆虫载体的改进注释:由不同基因组学群落的生物发生。数据库。2017;2017:bax032。https://doi.org/10.1093/database/bax032.
马泽涛,陈勇,李志强,李志强,等。复制基因簇的快速转录可塑性使无性繁殖蚜虫能够在不同的植物物种中繁殖。《基因组生物学》2017;18:27。https://doi.org/10.1186/s13059-016-1145-3.
Crescente JM, Zavallo D, Helguera M, Vanzetti LS。MITE追踪器:一种在大基因组中识别微型倒位重复转座元件的准确方法。生物信息学杂志,2018;19:348。https://doi.org/10.1186/s12859-018-2376-y.
Smit A, Hubley R. RepeatModeler Open-1.0。2008.http://www.repeatmasker.org.
Campbell MS, Law MY, Holt C, Stein JC, Moghe GD, Hufnagel DE,等。MAKER-P:用于快速创建、管理和质量控制植物基因组注释的工具包。植物物理学报。2014;164:513-24https://doi.org/10.1104/pp.113.230144.
埃德加钢筋混凝土。搜索和聚类速度比BLAST快几个数量级。生物信息学。2010;26:2460-1。https://doi.org/10.1093/bioinformatics/btq461.
埃德加钢筋混凝土。usparse:从微生物扩增子读取的高精度OTU序列。南京大学学报(自然科学版),2013;https://doi.org/10.1038/nmeth.2604.
Kohany O, Gentles AJ, Hankus L, Jurka J. Repbase中重复元素的注释、提交和筛选:RepbaseSubmitter和Censor。BMC生物信息学,2006;7:474。https://doi.org/10.1186/1471-2105-7-474.
Abrusán G, Grundmann N, Demester L, Makalowski W. TEclass -用于未知真核生物转座因子自动分类的工具。生物信息学。2009;25:1329-30。
Hoede C, Arnoux S, Moisset M, Chaumier T, Inizan O, Jamilloux V,等。一个自动转位元素分类工具。PLoS ONE。2014; 9: e91929。https://doi.org/10.1371/journal.pone.0091929.
确认
作者要感谢格林尼治大学为JPS提供副校长奖学金。这项工作也得到了比尔和梅林达·盖茨基金会的部分支持[资助号OPP1149777]。根据基金会的授权条件,创作共用署名4.0通用许可已经被分配给作者接受的手稿版本,该版本可能由本次提交。
资金
JPS由格林尼治大学的副校长奖学金资助。PV, SOS, SB和SS的捐款部分来自格林威治大学,部分来自比尔和梅林达·盖茨基金会[资助号OPP1149777]。
作者信息
作者及隶属关系
贡献
JPAS收集、分析和解释了本研究中使用的数据。JPAS和SOS开发并测试了TE识别工作流。PV验证了研究中使用的方法。SES、PV和SB对JPAS进行监督。JPAS起草了手稿,所有作者都参与了最终手稿的编辑。作者阅读并批准最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
所有作者都没有竞争利益需要声明。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
这篇文章的原始版本被修改了:作者要求更改鸣谢和资助部分的措辞。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
西卡特,j.p.a.,维森迪,Pet al。的转座元件的特征烟物种复杂。移动DNA13, 12(2022)。https://doi.org/10.1186/s13100-022-00270-6
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s13100-022-00270-6
关键字
- 转座的元素
- 粉虱
- 生物信息学
- 烟
- DNA转座子
- TE注释