摘要
背景
我们通过讨论当前的争议,对高等灵长类动物中驯化基因SETMAR的历史和生物学活性进行了回顾。我们的目的是打开一个新的前景,将作为未来关于SETMAR工作的框架,可能在认知发展领域。
主体
关于SETMAR的最新进展可以总结为:(1)整个蛋白序列处于强大的纯化压力下;(2)它的作用是加强现有的生物功能,而不是提供新的功能;(3)它表现出一种组织特异性的表达模式,至少对于它所经历的选择性剪接是这样的。
本文报道的研究表明,SETMAR蛋白可能参与调控复制、转录和翻译的基本网络。此外,在胚胎发生过程中,SETMAR似乎有助于大脑发育。
简短的结论
我们的综述首次强调了SETMAR直接与与发声和声乐学习相关的脑功能相关的基因相互作用。这些发现为未来关于SETMAR和高等灵长类认知能力发展的工作铺平了道路。
背景
在人类中,最显著的进化特征是在数百万年的智人进化过程中认知能力的增强。更好地理解这种进化的基础是一个真正的挑战。高达20%的遗传智力已被解释为补充方法,如行为和遗传,候选基因,全基因组关联,连锁分析和最近的大规模遗传研究[1,2].
与此同时,驯化转座因子(TEs)已被证明是快速出现适应性新事物的有效驱动因素,如增强的认知能力和最近的物种形成[3.].“分子驯化”的概念是由Miller及其合作者在1997年提出的,其中有几个案例说明了转座元件如何提供te衍生的蛋白质,这些蛋白质已被宿主重新利用[4].自那以来,在包括人类在内的高等真核生物中,已有多达150例病例被充分记录[5,6].TEs提供了数万个TEs衍生的DNA结合位点,为参与DNA或RNA代谢的转录因子和调控蛋白服务[7],大量te衍生的结合事件维持细胞类型特异性[8].
在这里,我们通过讨论当前的争议,对这种驯化基因之一SETMAR的历史和生物学活性进行了回顾。我们给一个概述Hsmar1它们仍然存在于人类基因组中,以及它们作为SETMAR网络成员的可能作用。然后,我们将利用ChIP-seq和RNA-seq现有数据,建立SETMAR及其网络、参与中枢神经系统(CNS)发育的基因及其控制之间的联系。我们的目的是在认知发展领域为SETMAR的未来工作开辟一个新的框架。
SETMAR历史和生化活动
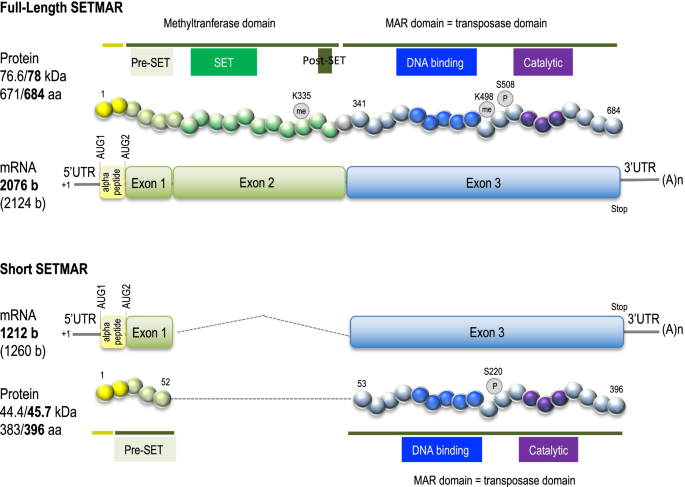
研究人员正在研究转座因子水手是第一个在人类基因组中寻找这些元素并发现嵌合基因的[9[后来命名SETMAR或METNASE.生于4500万年前SETMAR只存在于高等灵长类动物中[10].SETMAR由三个外显子组成,第一个外显子来自集基因和编码对于组蛋白甲基转移酶的功能,第三个来自于Hsmar1转座酶基因及重组酶功能编码[11(图。1).首先归因于SETMAR的活动在逻辑上是那些现在形成新基因的祖先蛋白质:组蛋白甲基转移酶(用于SET结构域)和DNA链转移(用于MAR结构域)[11].30篇关于SETMAR生化特性的文章汇总在图中。2以后再讲。
正如后面将要展示的那样,全长的SETMAR蛋白被描述为基因组守护者,在主要组织中表达,但表达水平不同[11(图。3.a).在癌细胞中SETMAR基因过表达,全长的SETMAR维持致癌过程。在某些情况下,SETMARpre-mRNA经历选择性剪接,导致更短的蛋白质的产生,在癌症干细胞中富集[12,14].SETMAR和细胞状态之间的关系已被记录[14,15,16].下面介绍的工作涉及全长蛋白(78kDa),称为SETMAR,除非另有说明。
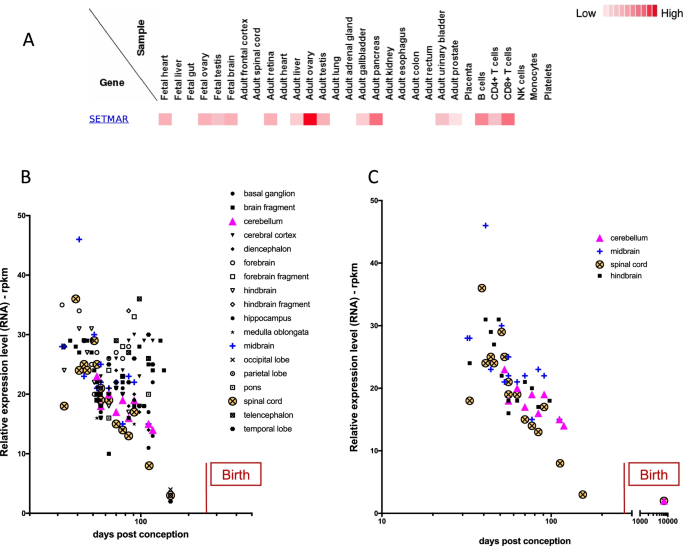
胚胎发生过程中SETMAR mRNA在人脑中的表达。一个数据来自Kim等人[13],使用“SETMAR”作为查询。所示为SETMAR蛋白被搜索的组织(测试样本)。颜色代码在右边给出。B胎儿大脑中SETMAR mRNA的相对水平。根据颜色代码报告18个地区或地区。注明出生时间(怀孕后270天)。C细节来自A,计算小脑的Spearman相关性(p= 0.027,r=-0.75),中脑(p= 0.0048,r=-0.72),后脑(p= 0.0049,r=-0.74)和脊髓(p= 0.0003,r= -0.8)。注明出生时间(怀孕后270天)
SETMAR在DNA结合、修复和外源DNA整合中的作用
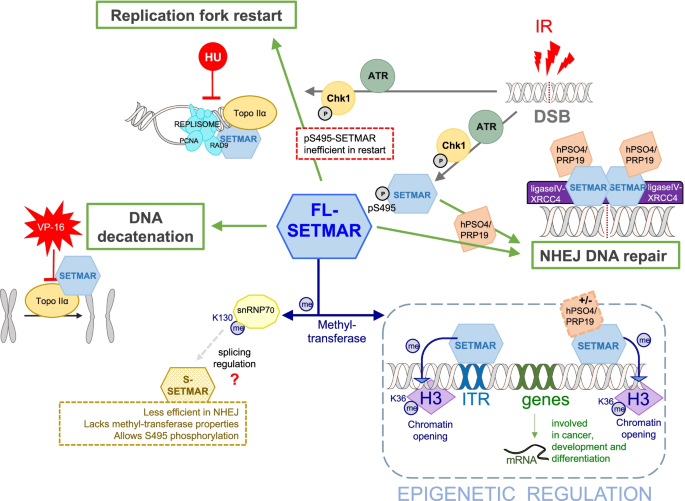
就像Hsmar1MAR结构域由一个DNA结合结构域和一个重组酶结构域组成。SETMAR绑定Hsmar1被称为TIRs(末端倒置重复)的四肢首次被研究。正如预期的那样,SETMAR能够与30 bp的TIRs序列相互作用,类似于重建的祖先转座酶(HSMAR-RA) [10,17,18,19].一些研究表明SETMAR与Hsmar1而它不能以同样的效率识别其他序列。SETMAR的这一特征至关重要,因为在当前的人类基因组中存在数千个TIRs,并代表着尽可能多的特定固定位点[10].贝克和合作者[20.,21]也证明了hPSO4/PRPF19是一个SETMAR伙伴,可以修改其结合属性,并提高对其他序列的识别和结合Hsmar1行动。
与SETMAR重组酶结构域相关的第二个特征是它能够整合外源DNA,利用转座酶共享的DNA磷脂酰转移酶活性来完成转座的最后一步,即.集成。李等[11]测量了线性化向量的自由端被SETMAR连接的能力在体外.他们的结果首次表明,SETMAR可能构成非同源末端连接(NHEJ)修复系统的一个组成部分。从那时起,一些研究已经证实SETMAR是NHEJ通路的有效伙伴,有助于产生未受损的3 '端,适合修补和结扎,旁路和移除阻塞的末端[22,23,24].它显示[25,26SETMAR定位于双链断裂,在那里它提高了修复效率,并促进磷酸化H2Ax焦点的解决,这是DNA双链断裂在塌陷分叉的标志。此外,多种NHEJ因子被证明可以直接与SETMAR相互作用,如hPSO4/PRPF19 [21]和连接酶IV-XRCC4复合物[27].尽管这些活动往往自发地与MAR结构域相关,但SET结构域的贡献正式地通过导致NHEJ效率降低的突变来维持,特别是对于精确修复[11]及仅使用SET域[28].这些后来和最近的工作表明,当SET和MAR结构域没有连接在一起时,它们都可以在DNA修复和非法整合中发挥作用(需要澄清它们的确切贡献),然而整个SETMAR蛋白只是微弱地负责这些活动。虽然与前面提到的研究不一致,但这些结果很好地说明了许多意想不到的参数会影响细胞中的SETMAR活性。他们还表明,使用过表达的重组蛋白是一种简化的方法/愿景,难以解决复杂的问题。
细胞周期和复制
在磷脂酰转移酶活性的上游,转座酶需要在转座开始时进行DNA双链断裂以切除转座子。这些特性(一致裂解和重新结扎)都是染色体十废化的主要特征,十废化是指在S期合成的姐妹染色单体被解开,以确保有丝分裂中染色单体的正确分离,以防止染色体在有丝分裂后期断裂和不适当的修复[29].因此,研究证明SETMAR与拓扑异构酶IIα (Topo IIα)发生物理相互作用并共同定位,拓扑异构酶IIα是一种关键的染色体清除酶[30.,31].此外,它增强了Topo IIα的癸烯化,增加了细胞对Topo IIα抑制剂的抗性[32],常用作化疗药物。在复制过程中也需要Topo IIα,在复制分叉之前展开DNA,允许分叉在DNA合成过程中进行。细胞在复制过程中特别容易受到DNA损伤,因为多个损伤会导致复制分叉暂停,甚至暂停,直到DNA断裂被修复。复制叉重启完成复制是细胞周期的一个重要过程。关于SETMAR在复制应激反应中的作用的信息,通过帮助复制叉重新启动,是通过羟基脲(HU)处理解决的,羟基脲是一种阻止复制的化学物质。研究表明,敲除SETMAR可提高细胞对HU处理的敏感性,延迟反应并重新启动DNA合成[26].SETMAR在S期的参与得到了证实,因为它与PCNA和RAD9共免疫沉淀,PCNA-like RAD9- hus1 - rad1内S检查点复合物的成员[26].综上所述,这些工作表明SETMAR在NHEJ的DNA修复、染色体失十位和复制应激反应中有很强的参与。
因为细胞周期是由DNA损伤感知启动的磷酸化事件级联调控的,并且因为转座酶的DNA损伤水手家族被证明是由磷酸化调节的[33],研究了SETMAR和CHK1之间的磷酸化事件。研究表明,电离辐射造成的DNA损伤可诱导CHK1对Ser-495 (pS495)的SETMAR磷酸化[34].pS495-SETMAR在重新启动崩溃的复制叉时效率较低,但它可以更有效地招募到DNA双链断裂以加强修复。因此,SETMAR是CHK1在复制应激中的效应因子之一,保护细胞并修复DNA断裂。此外,pS495-SETMAR增强CHK1的稳定性[35].最近的一项研究提出,SETMAR可能存在于复制起点,但这一点仍有待于使用同步培养细胞来证实[14].
SET域呢?
SETMAR的组蛋白甲基转移酶活性也是先导性工作中最先研究的之一。使用在体外方法,李等[11]已经证明SETMAR能够二甲基化H3K4和H3K36,这是开放和可接近染色质的两个表观遗传标记。尽管这一数据一直受到质疑[36],最近通过综合表观遗传学方法比较过表达SETMAR在SET域突变或未突变的重组SETMAR,证实了H3在K36位点的SETMAR二甲基化[37].的在活的有机体内SETMAR甲基化H3K36me2的能力也通过下调SETMAR的表达来证实,SETMAR的表达显著降低了胶质母细胞瘤细胞系中H3K36me2的水平[24].SETMAR促进H3K36me2的能力不仅与其在NHEJ中的作用相关,还通过增强需要修复的染色质位点的开放。谢等[15]还提到SETMAR可能参与了膀胱癌细胞中检测到的H3K27me3标记。值得注意的是,这些发现涉及非典型SETMAR抄本(变式5,见表1),而不是编码FL-SETMAR的那个。变体5编码了一种从未被检测到的蛋白质在活的有机体内(SETMAR-L),并且不包含完整的SET域。即使翻译过来,SETMAR-LmRNA允许产生接近S-SETMAR的SETMAR蛋白,这不能直接解释膀胱癌细胞中检测到的H3K27me3标记[15].
除了组蛋白H3的甲基化,SETMAR被证明可以促进自甲基化(在K335)和剪接因子snRNP70在K130的甲基化[36].在这些情况下,SETMAR是一种单甲基转移酶。卡尔森的命题等[36关于SETMAR在K335上的自甲基化的一个重要发现是,它只涉及细胞SETMAR的一小部分在活的有机体内,而且它可能不会改变酶的催化活性,而是改变了与它的伙伴蛋白质的相互作用。在过表达SETMAR的细胞中,K130-me1 snRNP70的数量增加,这一事实证明了SETMAR参与剪接调控,但这一点仍有待探索。最后,SETMAR也已被证明对其残基K485进行自动甲基化,该残基调节染色体的癸烯化[31].
在过去的十年中,SET域的作用似乎是有争议的。然而,最近基于高通量分析技术的工作使我们相信,SET结构域确实参与了不同靶蛋白的甲基化:组蛋白H3 (K36)、snRNP70和SETMAR本身。最后,研究表明结直肠细胞不会产生包含SET结构域的SETMAR变体[14].这一观察提醒我们Western blot方法(检测内源性蛋白质)的重要性,作为验证RNA方法的必要补充。
SETMAR表达式
数据库(Ensembl和NCBI Gene)描述了SETMAR转录本的各种亚型。其中一些在不同的细胞类型中被发现[12,38而其他的则从未被发现在活的有机体内(表1).
根据数据库信息,其中5个转录本可以编码蛋白质。两篇论文实际上展示了检测内源性SETMAR蛋白的实验[12,14].只有一种商业抗体确实是特异性的。它直接针对蛋白质的n端部分,因此允许检测所有假定的SETMAR蛋白(至少是数据库预测的那些)。目前,已检测到四种内源性SETMAR蛋白(表1):分别有一个表观分子质量约为77 kDa [12]和三个分别在48-52 kDa左右的表观分子质量[12,14].请注意,仅根据西方印迹,似乎很难排除杜索索瓦首先描述的短变体S-SETMAR等[12不是安托万-洛昆最近描述的两种短变体之一等[14].
SETMAR翻译
直到2014年,通常被用作SETMAR第一个氨基酸(aa)的蛋氨酸位于ATG2(图2)。1).Jeyaratnam等[38]首次提出SETMAR开放阅读框可以从ATG1开始,导致一个684个氨基酸的蛋白质,即.包括后来被称为“α肽”的13aa序列[14].所有使用重组SETMAR进行的实验均使用全长为671 aa的SETMAR变体,不含“肽alpha”(见图)。1).这导致了两种不同的SETMAR氨基酸编号系统。例如,文献中描述了MAR结构域的磷酸化位置S495 [35),即.在全长为671的SETMAR中,而SET结构域的甲基化是在K335位置[36),即.在684全长SETMAR中。我们现在建议,社区应该采用考虑ATG1翻译蛋白的编号,如图所示。1.
Hsmar1残余和相关的SETMAR网络
转座酶是自然界中数量最多、最普遍的基因[39].它们是进化过程中各种TEs驯化产生的数百个“新基因”的起源[40],一如既往Hsmar1.第一个Hsmar1近4500万年前,拷贝被插入到灵长类祖先的基因组中[10].经过数百万年的转换和放大,数百份Hsmar1被创建。这种放大的爆发之后是Hsmar1失活,由于转座酶基因和/或TIRs序列的累积突变和缺失。今天,人类基因组包含大约250个缺陷基因Hsmar1以及近12,500个TIR,这些TIR组织在单个TIR或缺乏转座酶编码基因的微型元件中,被称为螨虫[41],两者都来自于全长元素的部分切除或重组。
hsa - mir - 548
由于mite具有近乎完美的回文结构,其中TIRs在发夹结构中被几个碱基对分开,因此有人提出它们可能位于内源性小非编码rna (siRNA和/或miRNA)的起源上,这些rna在转录后水平的基因调控中起着重要作用。在2007年,Hsmar1衍生的螨虫被证明是一种名为hsa-miR-548的活性miRNA的起源[42].因为,在人类基因组中发现了超过75个hsa-miR-548基因,导致大约80个miRNA的表达,靶向估计1000到6000个基因,这取决于所使用的数据库(miRBase, miRWalk, miRanda和TargetScan)。已发表的80多篇论文证明了hsa-miR-548在多种疾病基因调控中的作用,如癌症(乳腺癌、膀胱、肝癌、……)、缺血或冠状动脉疾病[43,44,45,46,47].此外,SETMAR mRNA被预测为(至少)11 hsa-miR-548的靶标(miRWalk数据库)。
Hsmar1行动
在将近12500人中Hsmar1TIRs仍然存在于现代人类基因组中,约4000个显示了允许SETMAR结合的序列[41,48],促进组织这里所说的“SETMAR网络”。与该网络潜在相关的遗传控制可能以多种方式发生:(1)通过SETMAR的SET域,(2)通过与伙伴因子竞争或相互作用,或(3)通过抑制TEs活性的调节机制。最后这一假设已在人类和非灵长类动物细胞中得到证实(即.海拉与CHO细胞),其中活跃的(重建的)Hsmar1拷贝被稳定地插入到结构中,允许对重组体进行跟踪Hsmar1切除。有趣的是,初代CHO细胞的遗传背景是允许的Hsmar1转位,而HeLa细胞不是[41].作者证明了重组Hsmar1复制被DNA甲基化和H3K9me3沉积沉默。相反,内生性Hsmar1, TIRs或SETMAR并不是特别沉默的。它们显示出与整个人类基因组相似的表观遗传特征。这种整体的方法不排除一些的可能性Hsmar1序列可能是表观遗传调控的目标,可能是细胞特异性的,但这仍有待详细分析。事实是Hsmar1tir的可访问性加强了SETMAR是实现(epi)基因网络的良好候选的想法。Tellier和Chalmers最近对这一假设进行了调查。他们表明,在培养的骨肉瘤细胞(U2OS)中,SETMAR过表达诱导了近1500个基因mRNA表达的2倍以上的转移[37].表达的移位与SET结构域活性高度相关。使用ChIP-seq方法,他们证明了带有SETMAR结合TIR的基因(157个基因)在374个免疫沉淀基因中明显过度代表。然而,47个免疫沉淀基因包含一个不被SETMAR占据的TIR。在227个基因中,SETMAR识别并结合除Hsmar1行动。尽管最近在一个相当人工的模型(单倍体细胞系与内源性KOSETMAR基因及重组基因的过表达)[48],这一观察结果有力地支持了Beck和合作者的结果[21],表明hPSO4/PRPF19是修改其绑定属性的SETMAR伙伴(见下文)。含有hPSO4/PRPF19的SETMAR复合物显示出较低的特异性结合特性,扩大了SETMAR网络的领域和可能的靶基因的数量,如最近的两项工作所示[14,37].最后,Tellier和Chalmers研究中SETMAR控制的基因[37]被描述为参与器官发育和/或分化细胞事件,这些事件在癌细胞中被错误调节。令人惊讶的是,与SETMAR结合的基因表达率发生了显著变化(2倍),显示出与突触发生或某些认知技能(如语言学习)有关的基因出人意料地丰富。因此,在提出与SETMAR相关的网络假设12年后[10],经实验证实。未来的工作将不得不处理特殊的具有挑战性的问题,如确定真正使用的SETMAR结合位点或SETMAR结合靶基因的生物学后果。另一个挑战涉及SETMAR变体的作用和出现。事实上,Antoine-Lorquin等[14]的研究结果表明,结直肠细胞(无论是否癌变)都不表达全长形式的SETMAR(见下文),这提出了一个由Dussaussois等人勉强提出的假设[12: SETMAR选择性剪接可以是组织或细胞特异性的。这个新的假设应该让我们重新考虑我们认为理所当然的东西:SETMAR可能不是一种普遍存在的蛋白质。
SETMAR和疾病
SETMAR和癌症
一些研究表明,SETMAR量与细胞增殖呈正相关[26,30.,36],而另一种则不这么认为[28].如果细胞增殖与SETMAR正相关,那么癌细胞预计会过度表达它。因此,SETMAR蛋白已在患者的肿瘤中被发现,如血液肿瘤[38]、神经胶质瘤及胶质母细胞瘤[12].在这些组织中,与健康组织相比,SETMAR mRNA增加了70倍(取决于分析的样本)。SETMAR mRNA是多种选择性剪接的结果。其中,编码完整SETMAR蛋白(FL-SETMAR,全称全长SETMAR)的mRNA始终是最丰富的。在血液肿瘤(急性髓系白血病(AML)、套细胞淋巴瘤和慢性髓系白血病)中发现了10种不同的mRNA,在胶质瘤和胶质母细胞瘤中发现了4种不同的mRNA,这与在血液肿瘤中发现的4种相同。根据它们的序列,6个mRNA中有3个编码功能蛋白:全长蛋白和缺乏部分SET结构域的蛋白质,或缺乏整个SET结构域和部分pre-SET结构域的蛋白质。然而,它们都包含了整个MAR域[12,38].在AML中,全长mRNA表达的增加与低水平的染色体易位相关,这表明高SETMAR表达具有保护作用[38].直到最近,SETMAR蛋白在患者肿瘤组织中的存在仅限于胶质母细胞瘤(GBM) [12].已经检测到两种变体,全长的SETMAR和缺少部分pre-SET域和SET域的截断形式(S-SETMAR简称SETMAR),均由外显子2编码。编码这种短变异的mRNA先前已在血液肿瘤中检测到。最近在结直肠癌中也发现了非常相似的蛋白质[14].在GBM中,每种SETMAR变体的相对丰度与相关mRNA的相对丰度不匹配,因为两种SETMAR蛋白都在GBM中富集,而只有全长mRNA被过度表达。杜索索伊斯等[12]证明了S-SETMAR仍然能够执行NHEJ,但效率低于FL-SETMAR,从而证实了SET域在SETMAR函数中的重要性。为了理解S-SETMAR被过度表达这一有趣的事实(与其mRNA水平有关),一个由13个氨基酸组成的短n端序列的作用得到了解决,并提供了证据,证明该肽(称为α -肽)将两种SETMAR蛋白的半年期从几小时增加到几天[12].当时,由于缺乏抗-肽抗体,无法检查内源性SETMAR中该肽的存在。然而,最近的数据证实,大多数内源性SETMAR含有一种α -肽[49].每个SETMAR蛋白的作用都需要明确,但第一个证据表明,S-SETMAR在癌症干细胞中优先表达,而FL-SETMAR在分化细胞中优先表达。SETMAR被证明与GBM的复发有关,因为其在治疗耐药细胞中的表达上调[24].由此产生的高水平H3K36me2有助于激活NHEJ修复通路。预期,治疗耐药细胞与sh-SETMAR降低H3K36me2的整体水平,导致细胞不可逆衰老[24].从GBM和GBM干细胞中恢复的数据被结肠癌干细胞数据所加强[16].在这里,siRNA敲除SETMAR的外显子3(编码MAR结构域,存在于所有SETMAR蛋白中,见图。1)与NANOG、OCT3/4、SOX2等干性因子下调直接相关。即使这些发现不能区分S-和FL-SETMAR,它们也清楚地建立了SETMAR与茎干和/或分化之间的关系。最近的研究结果支持了这一观点,研究表明NONO(一种在细胞分化过程中基因激活和mRNA处理中起作用的因子)50])才能在膀胱癌细胞中保留SETMAR外显子2 [15].这加强了SETMAR异构体和细胞分化之间的联系。Xie等人发现的唯一不足之处[15他们的作品涉及一个非典型的SETMAR抄本(变式5,见表1),而不是编码FL-SETMAR的那个。
根据这些结果,可以提出第一个工作假设。FL-SETMAR可能是基因组守护者,在几乎所有成人健康组织中都有表达(在mRNA水平上)[11],但在蛋白质水平上的量是可变的[14].它的作用可能是提高除高等灵长类动物以外的其他物种中存在的生物机制的效力。在癌细胞中SETMAR基因过表达,FL-SETMAR随后维持致癌过程和癌细胞或癌症干细胞的发育。在某些情况下,SETMAR pre-mRNA经历选择性剪接,导致更短的SETMAR异构体,包含或不包含α -肽。不能排除的是,选择性剪接发生在不同的细胞类型上,导致在组织特异性之后产生不同的短变体。这些更短且可能更稳定的蛋白质可以破坏FL-SETMAR的活性(通过与伴侣蛋白竞争,与DNA结合位点竞争,通过S-和FL-SETMAR之间的异二聚体组装……)。事实上,高水平的S-SETMAR (GBM中发现的短变异)是GBM患者预后良好的一个因素[49可能是这种主导的消极活动的结果。
SETMAR作为治疗靶点
如前所述,SETMAR被视为涉及放射治疗和化疗的经典癌症治疗的假定障碍(图。2).因此,开发SETMAR抑制剂来改善癌症治疗显得很有趣。在MAR结构域链转移活性的基础上,已经筛选了各种SETMAR抑制剂[51].特别有趣的是,环丙沙星会损害SETMAR的裂解和修复DNA的能力。这增加了癌细胞和异种移植瘤模型对已经临床可用的化疗的敏感性,通过阻断化疗诱导的DNA损伤的修复。另一方面,一些hsa-miR-548被描述为直接参与肿瘤发生;例如,miR-548a-3p抑制SIX1,这是一种控制糖酵解基因的转录因子。在乳腺癌中,SIX1随着miR-548a-3p水平的降低而过表达,该水平是患者预后的良好预测指标[52].另一种hsa-miR-548, miR-548b,被证明通过抑制MTA2,转移瘤相关蛋白-2,抑制胶质瘤细胞(来自脑肿瘤)的增殖[53].在这两种情况下,治疗方法可以考虑恢复增加的hsa-miR-548水平。
SETMAR和整合病毒
因为SETMAR被认为是重要的外源DNA整合到宿主细胞基因组,Williamson等人[54]研究了SETMAR表达水平是否会影响慢病毒基因组整合(更准确地说,是VSV-G伪型hiv -骨干慢病毒)。他们表明,SETMAR的表达水平与活慢病毒整合相关,这种活性依赖于MAR结构域的效率。相比之下,SETMAR对细胞病毒RNA、cDNA或2-LTR环的总量没有影响。
其他疾病吗?
为了确定SETMAR在遗传性疾病中的潜在作用,已经使用了两种互补的方法。
首先,大规模的基因改变涉及SETMAR已使用ClinVar数据收集(表2).没有删除或复制的情况SETMAR只有基因被报道过。在9个案例中,SETMAR而且SUMF1(仅)共删除,但无临床意义相关。由于SETMAR属于SUMF1内含子,因此这种共缺失是意料之中的。
第二,准时或小突变内SETMAR基因被检查,使用基因组聚合数据库(gnomAD)的布罗德研究所。同义和错义SETMAR功能丧失蛋白变体根据其预期进行评分与所观察到的事件,给出的比值约为1(分别为0.98和0.96),这表明SETMAR基因处于低(或无)选择状态。pLI(功能丧失不耐受的概率)为0证实了这一点,这意味着SETMAR可以耐受突变而不显示疾病表型。事实上,在一般人群中,许多个体在他们的基因组的这一部分显示杂合缺失(基因组变异数据库),杂合状态意味着缺失或准时突变是无害的。相比之下,Cordaux等[10]的研究结果表明,MAR结构域的n端(即。DNA结合域)正处于强烈的净化选择之下,这一点最近得到了Tellier的证实等[37].这一点最近由安托万-洛昆完成等[14],他们认为整个SETMAR序列处于强净化选择状态。如果我们认为gnomAD中鉴定的SETMAR变体仅作为杂合子存在,那么这个明显的矛盾就可以解决。不能排除纯合突变会对表型产生影响。
综上所述,基于gnomAD和pLI, SETMAR似乎不是一个必需基因,即能够耐受突变而不表现出疾病表型,至少是杂合的,但与癌症相关或与选择性剪接相关的表达水平变化有关。未来的研究应该考虑一个新的假设:SETMAR作为一个基因,即。包括所有的转录本,可以被认为是无处不在的表达,而它显示组织和细胞类型的特异性,就每一个转录本。
SETMAR和认知
在最近的一篇综述中,罗斯和迪克[2调查人类特有的高级认知能力是否为非人类动物所共有,即使是在初步形态中。他们检查了9个行为项目或能力(工具使用或制造、解决问题、凝视跟随、镜像自我识别、模仿、元认知、心理理论、有意识注意、亲社会行为)。他们的研究结果表明,这些能力都不能被认为是人类独有的。它们都有非人类灵长类动物的前体。此外,幼儿(三岁以前)的能力与猿类和旧世界的猴子并没有真正的区别。信息处理能力与皮层神经元数量、堆积密度和轴突速度有良好的相关性。对于灵长类动物来说,人类的大脑构成了这些特征的优化,依赖于大脑皮层的发育及其在进化过程中的重组。为了解决社会、生态、现实和心理领域的问题,这些增强的信息处理使人类和非人类动物之间的差异变得最为显著,即。铰接式,句法式和语法式语言的出现[2].
综上所述,我们可以将灵长类动物的智力进化分为三个阶段,这与通常对这些灵长类动物所接受的系统发育分类相一致:第一个阶段以原猴和新大陆猴为代表,第二个阶段由旧世界的猴子和类人猿以及人类的幼儿代表,第三个阶段由老年人代表。因为SETMAR与类人猿谱系(旧世界的猴子、类人猿、人类)相伴随,人们很容易考虑它是否可能是皮层发育和高级大脑组织的参与者之一。
2018年,弗洛里奥及其合作者发表了一项前沿研究[55],该研究提出通过从五个不同的转录组中搜索早期特异性表达在胎儿新皮层祖细胞(cCNP)中的基因来识别参与人类新皮层扩张的基因。他们确定了35个灵长类特异性基因(在非灵长类基因组中没有直系同源),由15个人类特异性基因完成,所有50个优先表达在cCNPs中。其中,有17个基因是类人猿的灵长目动物共有的。所有这些基因都是从祖先基因的复制(部分或全部)或逆转进化而来的。其中10个编码锌指(ZNF)家族的DNA结合蛋白,另外7个参与各种细胞过程(tRNA成熟、免疫、脂质运输和金属离子稳态)。它们共同代表了类人猿灵长类动物皮层扩张的第一组候选基因。另一组候选基因可以由类人猿灵长类动物从TEs中吸收的基因提供[56].其中,SETMAR的特征使其成为一个迷人的候选者,可能有助于包括人类在内的高等灵长类动物的认知发展。虽然从未从这个角度考虑过,但我们在这里提出了一个关于SETMAR进化功能的原始假设。这一假设考虑了SETMAR的一些生物学和生物化学特性,它的表达谱(在蛋白质和mRNA水平上),它所承受的高选择压力,它与转座衍生DNA结合位点网络的联系,以及SETMAR结合基因的基因本体。SETMAR确切的生物学功能尚不清楚,一些方面仍在争论中。其他SETMAR属性从未被完全解释,例如它在各种癌症中的表达增加及其相关网络,由Hsmar1结合位点。
人类胚胎发生过程中SETMAR的表达
质谱数据来自Kim等人的LC-MS分析[13]的研究表明,在人类胎儿中,SETMAR蛋白在整个大脑、性腺和心脏中都有适度的检测到。有趣的是,在成人中的表达谱完全不同,SETMAR蛋白在B细胞、胰腺、膀胱、胆囊、性腺和视网膜中被检测到,但在整个大脑中没有(图2)。3.a).这些数据不允许区分SETMAR变体,因为检测到用于分析的肽(AIFLFEFK)来自外显子3的开始。这个外显子编码MAR结构域,所有SETMAR蛋白共享的结构域。
更精确的分析已经在mRNA水平上完成,在早期胚胎发生期间大脑的各个特定区域内。来自Ensembl网站的数据收集自有关胎儿大脑的项目,并进行分析增刊的数据详情)。他们明显地表明,SETMAR mRNA的整体表达水平在胚胎发生时下降,在怀孕结束时达到一个微弱的水平(图2)。3.b).在小脑、中脑、后脑和脊髓四个区域,SETMAR mRNA表达的降低与时间有统计学相关性,Spearman检验证实(图。3.c).尽管仅凭这些相关性不足以构成或支持我们的假设,但它们允许考虑SETMAR在神经发生过程中的作用。
大脑中的SETMAR网络
BrainSpan数据库中的RNA表达数据已被用于识别人类大脑发育期间(0-38 mpc)的SETMAR共表达基因,假设基因共表达网络捕获生物学上重要的模式[57)(见增刊的数据详情)。因此,我们寻找(i)在脑胚胎发生过程中与SETMAR共同调节的基因,以及(ii)在这些基因中可能是SETMAR靶基因的基因,因为它们含有Hsmar1TIR的序列。500个最强烈的共调控基因(正的[r在0.648到0.993之间]或负的[r在-0.557到-0.834之间])被保留用于分析(表1和表2,表S1).基因本体[58]显示,大部分正共调控基因参与翻译和转录调控,而负共调控基因参与阳离子跨膜转运(表S2).这些发现与Florio等人基因列表的基因本体论显示很匹配[55] (ZNF蛋白、tRNA成熟、脂质转运和金属离子稳态)。这些初步的发现还远远不足以得出结论,SETMAR在神经发生中发挥作用,更不用说在高等灵长类动物的认知能力中了。因此,我们通过比较几组基因来进行分析:至少包含一个TIR的基因[41(列表#3),来自Florio等人研究的基因[55](列表#5),SETMAR正向和负向共同调节基因(列表#1和#2),以及参与智力障碍(ID)的基因(列表#4),可在IDGenetics网站上获得。完整的基因列表见表S1.我们的主要发现见表格3.而且4.
在大脑发育过程中,有三个基因既参与ID,又在SETMAR中共同调控,在它们的序列中显示出一个SETMAR结合位点(LRPPRC和PDE4D(正共同调控);CYP27A1(负共调节))。总的来说,不同群体的基因本体并没有比前面提到的类别给出更多或更好的东西。值得注意的例外是参与ID并具有SETMAR结合位点(TIR)的60个基因,其中基因本体显示参与发声学习(fold enrichment > 100)和发声行为(fold enrichment = 51.5)的基因高度富集。这些基因是NRXN1、CNTNAP2和GLI3。声乐学习和发声是认知发展的基本特征。因此,这些发现支持了我们的假设,这仍有待实验证实。事实上,这里给出的结果仅仅基于来自不同数据库的数据之间的相关性。
其他SETMAR网络
泰利尔及查尔默斯[37]使用ChIP-seq分析首次描述了人类细胞中被SETMAR结合的基因。虽然这项工作还没有在CNS细胞上完成,但它是唯一描述SETMAR和靶基因之间生物学相互作用的工作。已经发现279个基因被SETMAR物理靶向(列表#6,表S1).其中上调2倍以上的有28例(表7,表S1).基因本体论显示,在八个最具代表性的类别中,七个涉及大脑特定的生物过程([37]参见补充数据):突触组装与功能(5项),学习与记忆(1项),发声(1项)。此外,最富集的基因是那些参与发声行为的基因(fold enrichment >100),在60个参与ID的基因中发现它们的序列中有一个SETMAR结合位点。总的来说,这些分析显示了涉及同一途径的两个简短的基因列表,即.其中,SETMAR可能是一个重要的调节因子。泰利尔及查尔默斯[37我们鉴定的基因是NRXN1、NRXN3和SHAK2,我们鉴定的基因是CNTNAP2、GLI3和NRXN1。
这些发现非常令人惊讶,因为它们是通过不同的方法获得的,但它们非常具有挑战性。他们有力地支持了我们的假设,即SETMAR网络参与了高等灵长类动物的认知技能发展,并确定NRXN1是该网络的重要组成部分。
结论
上一次对SETMAR函数的回顾始于2010年[60此后,许多研究都强调了它在某些癌症中的重要性及其网络功能。在这里,我们对其已知的功能进行了最新的综述,同时对这种多效蛋白的假定作用提出了一个前沿的观点。在撰写这篇综述时,我们注意到在理解SETMAR活动时缺少了一个重要的元素。已知SETMAR以二聚体的形式与DNA相互作用[10].自从短变异体被发现以来,人们从未探索过在不同变异体之间形成异二聚体的可能性。如果这一假设是正确的,它将通过所有变体之间的可能组合,为监管提供更多的可能性。
关于SETMAR的最新进展可以总结为:(1)整个蛋白序列处于强大的纯化压力下;(2)它的作用是加强现有的生物功能,而不是提供新的功能;(3)它表现出一种组织特异性的表达模式,至少对于它所经历的选择性剪接是这样的。
在总体水平上,本文报道的研究表明,SETMAR蛋白可能参与调控复制、转录和翻译的基本网络。在胚胎发生过程中,SETMAR似乎有助于大脑发育。有趣的是,SETMAR已被描述为与发声和声乐学习相关的基因相互作用。发声在包括人类在内的许多脊椎动物中都是必不可少的,因为它们会影响母婴依恋、社会和行为发展。对人类来说,发声和咿呀学语是学习声音和单词的先决条件,换句话说,是学习口语的先决条件[61].这些发现为未来关于SETMAR和高等灵长类认知能力发展的工作铺平了道路。
数据和材料的可用性
在当前研究中分析的数据集可在以下存储库中获得:
•ClinVar数据(https://www.ncbi.nlm.nih.gov/clinvar)
•博德研究所基因组聚合数据库(gnomAD)https://gnomad.broadinstitute.org)
•基因组变异数据库(http://dgv.tcag.ca/dgv/app/home)
•数据来自Kim等人[13]http://www.humanproteomemap.org/query.php
•Kim等人的质谱数据[13]可在Expression Atlas网站(https://www.ebi.ac.uk/gxa/experiments/E-PROT-1/Results?geneQuery=%5B%7B%22value%22%3A%22ENSG00000170364%22%7D%5D&filterFactors=%7B%22DEVELOPMENTAL_STAGE%22%3A%5B%22fetus%22%5D%7D)
•Ensembl网站(关于胎儿大脑的项目)(http://www.ensembl.org/Homo_sapiens/Gene/ExpressionAtlas?db=core;g=ENSG00000170364;r=3:4303304-4317567)
•来自BrainSpan网站的转录组数据。0- 38pcw在人的整个大脑。(https://www.brainspan.org/rnaseq/searches?domain=10173,10185,10278,10194,10163,10291,10208,10209,10225,10236,10235,10243,10252,10268,10269,10294,10361,10551,10550,10552,10333,10391,10398,10665,10656,10657&stages=IIA,IIB,IIIA,IIIB,IV,V&search_type=correlation&search_term=&seed=1099763)
•IDGenetics网站(已知snv和indel列表)。
缩写
- AML:
-
急性髓系白血病
- cCNP:
-
胎儿新皮层的祖细胞
- 中枢神经系统:
-
中枢神经系统
- “绿带运动”:
-
胶质母细胞瘤
- ID:
-
智力障碍
- KEGG:
-
京都基因与基因组百科全书
- 质:
-
液相色谱-质谱法
- LTR:
-
长末端重复
- 螨虫:
-
微型反向末端重复
- 货币政策委员会:
-
个月post-conception
- NHEJ:
-
非同源端连接
- 铝:
-
翻译后修饰
- TE:
-
转座因子
- 行动:
-
末端反向重复
- ZNF:
-
活力的手指
参考文献
王晓明,王晓明,王晓明。认知能力的分子遗传学研究。《生物学报》1998;70:281-96。
人类认知的起源与演化。Prog Brain res 2019; 250:285-316。
夏皮罗农协。21世纪的移动DNA与进化。暴徒的DNA。2010; 1:4。
王文杰,王志强,王志强,等。分子驯化研究进展。遗传。1997;100:261 - 70。
Jangam D, Feschotte C, Betrán E.转座因子驯化对进化冲突的适应。《趋势》,2017;33:817-31。
Etchegaray E, Naville M, Volff J-N, Haftek-Terreau Z.转座因子衍生的脊椎动物发育序列。暴徒的DNA。2021; 12:1。
张勇,张志刚。转座因子的调节作用:从冲突到利益。Nat Rev Genet, 2017; 18:71-86。
孙达兰,程燕,马震,李东,邢晓霞,Edge P,等。转座因子对基因调控网络创新的广泛贡献。Genome res 2014; 24:63 - 76。
罗伯逊HM, Zumpano KL.人类基因组中古水手转座子Hsmar1的分子进化。基因。1997;205:203-17。
Cordaux R, Udit S, Batzer MA, Feschotte C.通过从移动元件中捕获转座酶基因来诞生嵌合灵长类基因。美国国家科学研究院。2006; 103:8101-6。
Lee S-H, Oshige M, Durant ST, Rasila KK, Williamson EA, Ramsey H,等。SET结构域蛋白Metnase介导外源DNA整合,并将整合与非同源末端连接修复联系起来。美国国家科学研究院。2005; 102:18075 - 80。
杜索索伊-蒙塔涅A, Jaillet J, Babin L, Verrelle P, Karayan-Tapon L, Renault S,等。胶质母细胞瘤中的SETMAR亚型:蛋白质稳定性问题。Oncotarget。2017;8:9835-48。
金M-S, Pinto SM, Getnet D, Nirujogi RS, Manda SS, Chaerkady R,等。人类蛋白质组图的草图。大自然。2014;509:575 - 81。
Antoine-Lorquin A, Arensburger P, Arnaoty A, Asgari S, Batailler M, Beauclair L,等。在人类结肠细胞中,在一些增强子和复制起点中富集的两个重复基元与SETMAR亚型结合。基因组学。2021;113:1589 - 604。
谢锐,陈鑫,程琳,黄敏,周强,张娟,等。NONO通过SETMAR选择性剪接抑制膀胱癌淋巴转移。Mol Ther, 2020。https://doi.org/10.1016/j.ymthe.2020.08.018.
Apostolou P, Toloudi M, Kourtidou E, Mimikakou G, Vlachou I, Chatziioannou M,等。Metnase转座酶融合基因通过调控关键基因在结肠癌中的潜在作用。PLoS ONE。2014; 9: e109741。
刘d, Bischerour J, Siddique A, Buisine N, Bigot Y, Chalmers R.人类SETMAR蛋白保留了祖先Hsmar1转座酶的大部分活性。中国生物医学工程学报,2007;27:1125-32。
Miskey C, Papp B, Mátés L, Sinzelle L, Keller H, Izsvák Z,等。古老的水手再次航行:人类Hsmar1元件的转座子重构转座子和SETMAR蛋白在转座子末端的活性。中国生物医学工程学报,2007;
李永杰,李永杰,李永杰,李永杰,等。SET和转座酶融合蛋白Metnase的生化特性:其DNA结合和DNA切割活性。生物化学,2007;46:11369 - 76。
Beck BD, Lee SS, Hromas R, Lee S-H。结合伙伴Pso4对Metnase TIR结合活性的调控。生物化学学报,2010;
Beck BD, Park S-J, Lee Y- j, Roman Y, Hromas RA, Lee S-H。人Pso4是一种metnase (SETMAR)结合伙伴,调节metnase在DNA修复中的功能。中国生物医学工程学报。2008;
李世华,李志强,李志强,李志强,等。通过Metnase和Artemis内切酶修剪受损的DNA双链断裂3 '悬垂。DNA修复(Amst)。2013; 12:422-32。
Beck BD, Lee S-S, Williamson E, Hromas RA, Lee S-H。metnase内切酶活性的生化表征及其在NHEJ修复中的作用。生物化学,2011;50:4360 - 70。
Kaur E, Nair J, Ghorai A, Mishra SV, Achareker A, Ketkar M,等。在残留病变细胞中抑制SETMAR-H3K36me2-NHEJ修复轴可预防胶质母细胞瘤复发。神经肿瘤防治杂志。https://doi.org/10.1093/neuonc/noaa128.
Fnu S, Williamson EA, De Haro LP, Brenneman M, Wray J, Shaheen M,等。组蛋白H3赖氨酸36的甲基化通过非同源端连接增强DNA修复。美国国家科学研究院。2011; 108:540-5。
De Haro LP, Wray J, Williamson EA, Durant ST, Corwin L, Gentry AC,等。Metnase促进停止和崩溃的复制分叉的重新启动和修复。核酸研究,2010;38:5681-91。
Hromas R, Wray J, Lee S-H, Martinez L, Farrington J, Corwin LK,等。人组和转座酶结构域蛋白Metnase与DNA连接酶IV相互作用,提高了非同源末端连接的效率和准确性。DNA修复(Amst)。2008; 7:1927-37。
Tellier M, Chalmers R.人类SETMAR (Metnase)蛋白在非法DNA重组和非同源端连接修复中的作用。DNA修复(Amst)。2019; 80:26-35。
阿什沃斯a .哦,它编织了一张多么复杂的网:BRCA1和DNA decatenation。中华癌症杂志2005;8:95-7。
Wray J, Williamson EA, Sheema S, Lee S- h, Libby E, Willman CL,等。Metnase介导急性白血病细胞染色体失十位。血。2009;114:1852-8。
Williamson EA, Rasila KK, Corwin LK, Wray J, Beck BD, Severns V,等。SET和转座酶结构域蛋白Metnase增强染色体去十位:通过自甲基化调节。核酸决议2008;36:5822-31。
Wray J, Williamson EA, Royce M, Shaheen M, Beck BD, Lee S-H,等。Metnase介导乳腺癌细胞对拓扑异构酶II抑制剂的耐药性。PLoS ONE。2009; 4: e5323。
Bouchet N, Jaillet J, Gabant G, Brillet B, Briseño-Roa L, Cadene M,等。cAMP蛋白激酶磷酸化Mos1转座酶并调节其活性:来自质谱和生化分析的证据。Nucleic Acids res 2014; 42:1117-28。
赫罗玛斯R, Williamson EA, Fnu S, Lee Y-J, Park S- j, Beck BD,等。Chk1磷酸化的Metnase增强DNA修复,但抑制复制叉重启。致癌基因。2012;31:4245-54。
王伟,吴勇,李世华,等。DNA修复成分Metnase调节Chk1的稳定性。Cell Div. 2014;9:1。
Carlson SM, Moore KE, Sankaran SM, Elias JE, Gozani O.一种蛋白质组学策略通过SETMAR识别剪接因子snRNP70的赖氨酸甲基化。生物化学杂志2015。https://doi.org/10.1074/jbc.M115.641530.
Tellier M, Chalmers R.人类SETMAR是一种DNA序列特异性组蛋白甲基化酶,对转录组有广泛的影响。核酸res 2019; 47:122-33。
Jeyaratnam DC, Baduin BS, Hansen MC, Hansen M, Jørgensen JM, Aggerholm A,等。已知和新的SETMAR (Metnase)基因转录变异的描述和在血液肿瘤中的表达谱。Exp内科杂志。https://doi.org/10.1016/j.exphem.2014.02.005.
阿齐兹RK,布莱巴特M,爱德华兹RA。转座酶是自然界中最丰富、最普遍的基因。核酸研究,2010;38:4207-17。
转座因子与调控网络的进化。Nat Rev Genet, 2008; 9:397-405。
雷诺S,让蒂M,加博里A,布瓦诺C,埃诺C, Dugé德伯农维尔T,等。人类DNA转座子HsMar1的表观遗传调控。BMC Genet 2019;20:17。
Piriyapongsa J, Jordan IK。来自微型倒置重复转座元件的人类microRNA基因家族。PLoS ONE。2007; 2: e203。
Sánchez-Chaparro MM, Garza-Veloz I, Zayas-Villanueva OA, Martinez-Fierro ML, Delgado-Enciso I, Gomez-Govea MA,等。BRCA1和BRCA2基因3'UTR的遗传变异及其对遗传性乳腺癌和卵巢癌microRNA机制的推测影响诊断(巴塞尔)。2020; 10。
蔡文春,蒋文华,吴春华,李永春,Campbell M,黄培华,等。miR-548aq-3p是一种新型远红外辐射靶点,可预测冠状动脉疾病内皮集束形成细胞的反应性。科学通报2020;10:6805。
杨超,李强,陈旭,张震,牟忠,叶峰,等。环状RNA circRGNEF通过miR-548/KIF2C轴调控促进膀胱癌进展。老龄化(奥尔巴尼纽约)。2020; 12:6865 - 79。
金敏,卢松,吴勇,杨超,石超,王勇,等。Hsa_circ_0001944通过作为miR-548的竞争性内源性RNA促进膀胱癌细胞的生长和转移。临床癌症杂志2020;39:186。
Elgharably H, Okamoto T, Ayyat KS, Niikawa H, Meade S, Farver C,等。人肺气道上皮上调MicroRNA-17和MicroRNA-548b对冷缺血和体外再灌注的反应。移植。2020;104:1842-52。
Miskei M, Horváth A, Viola L, Varga L, Nagy É, Feró O,等。人类基因组中转座酶衍生SETMAR蛋白结合位点的全基因组图谱。中国生物工程学报,2017;19(4):344 - 344。
Lié O, Virolle T, Gabut M, Pasquier C, Zemmoura I, Augé-Gouillou C. SETMAR短亚型:胶质母细胞瘤的一个新的预后因素。Front Oncol. 2021;11:638397。
马C, Karwacki-Neisius V,唐华,李伟,史志,胡华,等。Nono,二价结构域因子,调控Erk信号通路和小鼠胚胎干细胞多能性。细胞报告,2016;17:997-1007。
Williamson EA, Damiani L, Leitao A, Hu C, Hathaway H, Oprea T,等。靶向DNA修复成分Metnase的转座酶域增强化疗。癌症法案2012;72:6200-8。
李林,梁勇,康林,刘勇,高松,陈松,等。SIX1对癌症Warburg效应的转录调控。《癌症杂志》2018;33:368-385.e7。
潘艳,梁伟,赵霞,刘玲,青云,李艳。miR-548b通过靶向转移瘤相关蛋白-2抑制恶性胶质瘤的增殖和侵袭。Neuroreport。2016;27:1266 - 73。
Williamson EA, Farrington J, Martinez L, Ness S, O 'Rourke J, Lee S- h,等。人DNA修复蛋白metnase的表达水平影响慢病毒基因组整合。Biochimie。2008;90:1422-6。
傅立文,张文杰,张文杰,等。进化和细胞类型特异性的人类特异性基因优先表达在胎儿新皮层的祖细胞。Elife。2018;7。
Oliver KR, Greene WK。移动DNA和te -推力假说:来自灵长类动物的证据。移动的DNA。2011; 2:8。
张志刚,张志刚,张志刚,张志刚,等。共表达网络揭示了转录和剪接的组织特异性调控。Genome Res. 2017; 27:1843-58。
米H, Ebert D, Muruganujan A, Mills C, Albou L-P, Mushayamaha T,等。PANTHER版本16:修订的族分类,基于树的分类工具,增强器区域和广泛的API。核酸res 2020;49: D394-403。
Heberle H, Meirelles GV, da Silva FR, Telles GP, Minghim R. InteractiVenn:一个基于web的工具,用于通过Venn图分析集。生物信息学杂志。2015;16:169。
Shaheen M, Williamson E, Nickoloff J, Lee S-H, Hromas R. Metnase/SETMAR:一种驯化的灵长类转座酶,增强DNA修复,复制和decatenation。遗传。2010;138:559 - 66。
学习单词和学习声音:语言发展的进步。中华精神病学杂志,2017;
确认
不适用。
资金
这项工作得到了国家癌症研究所(INCA_11693 PLBIO 2017)和图尔大学的支持。没有一个资助机构参与研究的设计、数据的收集、分析或解释,也没有参与手稿的撰写。
作者信息
作者及隶属关系
贡献
SR分析和解释组学数据。OL和CAG是撰写手稿的主要贡献者。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
Lié, O.,雷诺,S. & Augé-Gouillou, C. SETMAR,灵长类动物增选基因的一个案例:走向新视角。移动DNA13, 9(2022)。https://doi.org/10.1186/s13100-022-00267-1
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s13100-022-00267-1
关键字
- SETMAR
- 网络
- 认知
- 新第三纪
- 监管
- 灵长类动物