摘要
自从首次在细菌中发现逆转录酶,后来又在古生菌中发现,细菌和古生菌的逆转录因子被定义为它们共同的酶,协调不同的功能。然而,进化的细化在微生物生命树中产生了不同的回溯元素,也许最好的描述是它们的程序化rna——一种为复杂机制保留核心信息的紧凑序列。从这个角度来看,逆转录酶被选为在各种RNA模板中执行自然指令的模块化工具。有益的回溯元素——那些可以为宿主提供适应度优势的元素——在广泛的微生物及其病毒中进化成现有的形式,几乎跨越了所有的栖息地。在每个专门的retroelement类中,不同类群之间似乎有一些共同的特征,而具体的功能和机制见解仅基于临床分离物的少数模型retroelement系统。目前,人们对不同生物群落中逆转录元素的细胞功能多样性和生态意义知之甚少。随着分离、宏基因组组装和单扩增基因组的可用性的增加,原核逆转录元件的分类和功能宽度正在变得更加清晰。这篇综述探讨了细菌和古生菌的有益的,但辅助的逆转录元素的最新特征。我们描述了这些专门的机制是如何利用一种固定的移动性的形式,从而逆转录因子不会在整个基因组中自私地增殖。此外,我们还讨论了从大量序列库中系统识别逆转录元素的计算方法,并重点介绍了它们在自然界中的明显分布和生态意义方面的最新发现。 Lastly, we present a new perspective on the eco-evolutionary significance of these genetic elements in marine bacteria and demonstrate approaches that enable the characterization of their environmental diversity through metagenomics.
模板中的遗传记忆
RNA在微生物细胞中有许多不同的形式,可以将信息从DNA传递到蛋白质,作为核酶进行生化反应,或形成执行各种细胞功能的核糖核蛋白复合体。在回溯元素中,非编码rna具有保存和传递遗传信息的能力,作为一种动态形式的记忆,在生物冲突中被回忆起来。在细菌和古生菌中发现了几类基因组元件,它们具有保存和传递序列信息的独特作用,这些信息与细胞之间的对抗性相互作用耦合,或对环境压力做出反应。为了减轻生物冲突,逆转录元件可以将这种记忆存储在编码短RNA的基因组区域,我们称之为模板。尽管这些模板本身并不直接编码蛋白质,但它们的序列代表了蛋白质-蛋白质或蛋白质-配体相互作用的潜在状态。因此,有益的逆转录因子由其基本的RNA模板来定义,这些模板保存了紧凑的序列信息,从而在解决冲突中发挥关键作用。
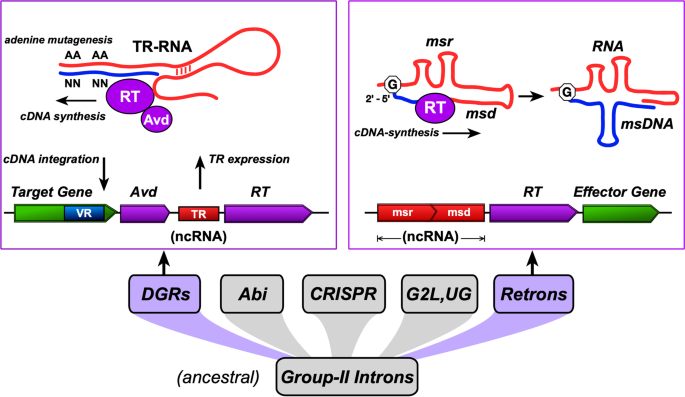
通过RNA模板存储和转移遗传记忆的能力是为细菌、古生菌及其病毒提供特定选择优势的两种机制的共同定义特征:多样性生成逆转录元件(DGRs)和逆转录子。DGRs具有序列变异的模板,可传递到蛋白质中,以实现与大量配体的相互作用[1,2,3.].Retrons由一个小DNA分子的保守模板组成,它与免疫和效应蛋白相互作用,最近发现这些效应蛋白作为噬菌体感染的反应触发生长停滞[4,5].
通过对自然系统的直接测序,我们可以看到正在进行的微生物进化的快照,包括可能经历了几个增殖和细化到当前基因组环境的动态阶段的专门元素的作用。对于任何给定的回溯元素,过去移动的时间或未来扩散的可能性都很难重建。Retrons和DGRs被认为具有复杂的进化历史,从祖先的RTs驯化到与提供独立细胞和病毒特性的蛋白质的功能偶联。在这里,我们回顾了这两类驯化的回溯元素——逆激子和产生多样性的回溯元素——它们已经被改进,以提供特定的好处,因为它们的“固定”移动性。
Diversity-generating retroelements
微生物基因通过进化优化来适应,这可能涉及多个协同突变的组合。如果一个基因存在多个协同突变,单靠随机突变和选择可能无法达到最优适应度;进化到这些最佳状态可能只有通过一种专门的机制来快速探索同时发生的突变。超突变可以自发地影响微生物基因组,其中的全球现象包括不完美的DNA损伤修复和复制错误[6,7].虽然随机超突变在环境压力下更受青睐,但在适应良好的种群中通常会减少[8].或者,局部变异可以由定向重组、动态启动子反转或密码子重写引起[9,10].多样性生成逆转录元件(DGRs)是细菌、古生菌和温带病毒目标基因高突变的驱动因素[11,12].通过在模板序列中精确定位腺嘌呤,DGRs能够以单核苷酸分辨率靶向突变。每当DGR重新激活时,突变就会被覆盖,这表明在稳定的细胞条件下,高突变的表型可能会被抑制,以减轻适应度的潜在损失。
DGRs的分子机制(图;1)涉及模板RNA的逆转录和模板腺嘌呤的选择性重写到随机碱基,这反过来推动整合后靶基因中的氨基酸取代[13,14].除了必需的逆转录酶(RT)和不变的模板RNA序列外,DGRs可能还需要一个辅助基因来协调cDNA的合成和整合(即逆转录)。此外,几独联体-作用序列特征增强或引导寻巢,包括目标区域下游的诱变寻巢(IMH)起始位点,以及IMH侧翼的一个或多个茎环特征[14].
在模型DGR系统中博代氏杆菌属在噬菌体中,模板重复(TR) RNA分子编码一段短序列(~ 150 bp),对应于目标基因的可变重复(VR),而其尾部RNA序列(~ 200 bp)预计具有与逆转录酶(RT)相互作用的保守结构,与辅助蛋白[15,16].最近的实验工作与体外系统包括博代氏杆菌属DGR-RT和Avd为dgr合成cDNA过程中腺嘌呤突变的作用提供了新的见解[17].Handa等人证明了RNA模板腺嘌呤的误掺和与DGR-RT的低催化效率有关,同时也发现了突变依赖于模板嘌呤碱基分别在腺嘌呤和鸟嘌呤的C6位置具有胺或羰基的证据。虽然DGR系统突变机制的原型正在出现,但靶标整合的分子决定因素仍然难以捉摸。
DGR分布与进化史
早期对宏基因组数据集和现有细菌基因组的系统调查导致预测DGRs广泛存在于细菌及其噬菌体中[1,18,19].最近的环境研究在古生菌中发现了DGRs [20.],也是地下水生生态系统中未培育的候选门成员的显著特征[21].这些未培养的细菌和古菌谱系预计主要由纳米大小的生物组成,这些生物依赖于微生物宿主的分子资源,以适应其生物合成的缺陷[22].尽管DGRs被预测在这些假定的表观生物的多样化附着蛋白中发挥作用,但它们的大多数多样化靶基因仍然完全未被表征。最近,对所有公开可用的基因组和宏基因组数据库进行了检查,从而对DGR在分类学、功能和生物地理尺度上的分布有了更全面的了解[23,24].DGRs在人类微生物组的微生物组分中尤其普遍,其中它们在肠道微生物中特别丰富[23,24,25,26].通过生物信息学筛选,在来自人类肠道样本的许多病毒和细胞基因组中发现了DGRs。在感染的肠道噬菌体中也发现了DGRs拟杆菌dorei[27],在CrAss-like噬菌体中,目标基因-尾领纤维蛋白-也保守在缺乏DGRs的噬菌体基因组中[28].尽管一些DGRs似乎存在于固定的染色体位点中,但它们也可以出现在噬菌体、前噬菌体、质粒和偶联元件中,这为这些元件在微生物基因组之间具有移动的动态潜力提供了证据[23].这种跨越分类学界限的交换能力使确定这些元素的进化史的尝试复杂化。
对蓝藻基因组的综合分析发现,DGRs在该门的数十个成员中是共同的,而DGR-RT的系统发育重建似乎描绘了远亲类群之间的水平交换[29].的成员束毛藻属,念珠藻属,淡水藻类的一种,眉藻属在其他属中,具有多个不同的DGR-RT基因,这些基因似乎是独立获得的,可能是通过门内的HGT获得的。非蓝藻基因组中最接近RT的亲属属于Chlorobi和Chloroflexi [29],这可能是蓝藻中DGR的早期来源。DGR-RT在特定进化支中的缺失,如原,球藻Gloeobacter,或质体的代表,进一步表明DGRs要么是最近在蓝藻亚群中获得的,要么是它们在门中的分布是特定谱系中损失的结果。
DGR功能多样性
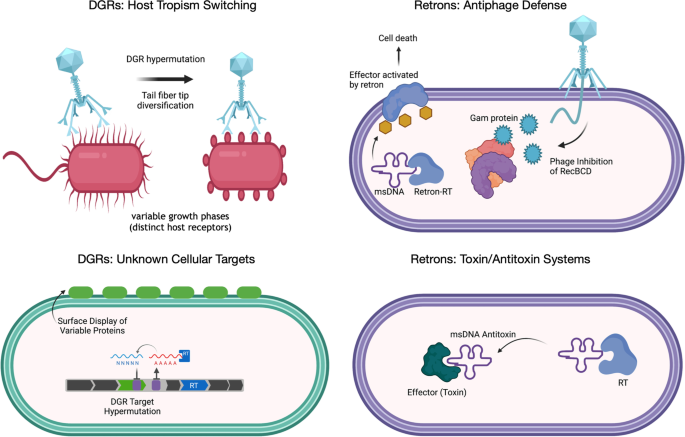
虽然在噬菌体中发现了DGRs,它们使宿主附着的基因多样化[1],这些逆转录元件似乎提供了广泛的用途,作为多种细胞和病毒靶点超突变的模块化工具(图。2).迄今为止,很少有分子靶点被实验描述,然而根据丰富的微生物基因组序列数据的预测表明,大约一半的dgr可变蛋白似乎具有细胞(即非病毒)功能[23,24].高变蛋白在一些临床菌株嗜肺性军团菌,它们似乎积极地使显示在细胞外膜上的脂蛋白的基因多样化[30.].的DGR靶蛋白梅毒denticola(贩运受害者保护法》确定),据预测,脂蛋白信号序列参与向外膜输出,可能附着于其他细菌或上皮细胞[3.].在肠道微生物组中,各种不同的DGRs似乎在靶向噬菌体和细胞基因方面发挥作用[24,25,26],但这些可变蛋白质仍然在功能上未被表征。
由于大量已知细菌和古菌DGRs的可用实验数据很少,我们在理解大多数超突变靶点的功能意义方面受到限制。许多属于未培养的寄生虫或形成候选门谱系的表观生物的DGRs被预测参与调节宿主相互作用[21].对于蓝藻DGR靶标,跨DGR靶标的蛋白质结构域预测表明可变clc样结构域的融合n端成分可能具有不明确的调节功能[29].据推测,应激反应蛋白的一种灵活的配体结合模块可能使这些生物能够快速适应不断变化的环境[29].超变异的另一个潜在优势可能是减轻或绕过流产途径效应蛋白的功能,这样DGR变异可能使个体避免程序性细胞死亡。
Retrons
事实上,跨越生命之树的所有生物在其基因组中都编码了各种非编码rna,包括大多数功能仍未确定的元素。Retrons是第一个从细菌中发现的逆转录元素[31,32](后来,古生菌[33]),多年来,它们在细菌细胞中的功能作用仍然难以捉摸[34,35].这些小DNA分子最初在几种模式细菌物种中具有特征,通过ncRNA模板的逆转录产生,ncRNA模板成熟为RNA-DNA杂交体,通常在细菌细胞中产生许多副本(图2)。1).尽管重激子及其多拷贝单链DNA (msDNA)的功能自发现以来多年来一直未知,但最近的证据表明,重激子在流产防御系统中发挥了特定作用,以减轻噬菌体感染(图2)。2) [4,5].此外,新的初步发现还揭示了以前未知的基团(UG)和Abi-like逆转录因子在防御噬菌体感染方面的类似生物学作用[36].
从基因组的角度来看,逆转录子是由逆转录酶(RT)基因与逆转录子RNA和DNA区域一起组成的msr而且默沙东公司,分别(图;1).从msr-msd-RT序列产生一个单一的转录本,并形成一个RNA二级结构,在鸟苷残基处进行2 ' -OH引物后,作为cDNA合成的模板。msd RNA序列逆转录为msDNA, msDNA在5 '和3 '端与模板msr-RNA结合(图2)。1).的成员内的Retrons已被实验表征Deltaproteobacteria,Gammaproteobacteria,Alphaproteobacteria,拟杆菌[37],其中rt介导的msDNA合成被证明是这些元素的普遍属性。
重子的RNA分子是形成RNA - dna杂交分子的必要前体,直到最近,它的细胞功能还不清楚。细菌的逆激子通常与效应基因一起编码在防御岛中,包括dna结合蛋白(HTH、锌指等)、冷休克蛋白、内切酶和蛋白酶[38].这些效应子可能被“保护”或被retron RNA-DNA杂化分子激活,如该研究所证明的大肠杆菌recon, Ec48,可防御多种噬菌体[4,5,39].Retrons及其效应蛋白被假设为一种毒素-抗毒素系统(图。2),通过噬菌体感染导致流产细胞死亡,确保个体细胞牺牲以造福种群[5].逆激子的抗噬菌体活性似乎依赖于msDNA的结构和功能域,如拓扑异构酶引物酶(TOPRIM)、toll-白细胞介素样受体(TIR)、atp酶和HNH核酸酶域[38,40].鉴于在几种细菌谱系中发现了一系列不同的基因操纵子,而其他的逆激子似乎与近端效应基因缺乏明确的联系[38],除了失败的抗噬菌体反应外,retrons可能还被用于不同的细胞功能。
Retron分布和进化史
基因组调查已经估计了逆转录子在细菌门中的分布,最全面的努力探索了来自大约20万个逆转录酶序列的9141个非冗余代表的数据库,这些序列包括每个逆转录元素类,以及未知的RT谱系[41,42].在微生物基因组中发现的逆转录子根据其RT系统发育进行分组:已描述了11个分支,它们与各种ncRNA结构和各种效应基因或RT融合域相关[38].
细菌重激子在不同的基因组中显示出垂直和水平交换的证据。例如,不同黏菌菌株中密切相关的逆子与其他保守黏菌基因具有相似的密码子使用特征[43].相比之下,广泛的大肠杆菌逆转录子与核心基因的密码子使用并不密切匹配,这表明它们是在这些肠杆菌的进化史上最近获得并水平交换的[43].有趣的是,重激子通常存在于细菌基因组的前噬菌体序列中,其中噬菌体转导可能是它们迁移到新受体的驱动因素[34,44].而实验验证的前噬菌体重激子来自大肠杆菌基因组,前噬菌体编码的重子很可能在其他细菌基因组中发现。
回溯元素复杂的进化史之前曾被描述为ii族内含子的早期祖先,其他类别的内含子都是从它们进化而来的[38,45,46].Retrons和DGRs可能有一个最近的共同祖先,因为它们有一些共同的机制特征,与大多数ii族内含子相比,它们的关系更密切。重转录子和DGRs具有共同的关键特征:i)生成一个单一的小RNA转录本,从中形成DNA-RNA异双工;ii)通过RT合成cDNA是模板引物。然而,拒通过不同的机制,重传子似乎缺乏DGRs和ii族内含子都表现出的整合回巢能力[13,47].
驯化逆转录因子的基因组学与生态学
过去十年见证了基因组数量的巨大增长,为微生物生命的基因组成提供了途径。除了改进传统微生物分离方法的培养策略的进展之外[48,49,50],单扩增基因组和宏基因组组装基因组为获取在实验室环境中难以培养的生命分支的基因组提供了额外的手段[51,52,53,54].越来越多的长读测序技术[55]预测微生物基因组的数量、广度和质量将继续提高。
迄今为止,对DGRs和retrons的比较基因组研究强调使用基于同源的工具进行特征检测的识别和分类[19,21,23,24,38,46,56].这些方法最近已经系统地鉴定了数千个定义分类和生物地理分布的新回溯元件,以及相关蛋白质家族的功能多样性[24,38].然而,基因组的用途并没有扩展到捕获逆转录元素的生态和进化意义及其在自然微生物种群中的动态性质。基因组通常代表种群中的单个成员,不能立即访问动态区域,其中专门机制的活动、多样性或流动性在环境种群的所有成员中可能不一致。然而,个体基因组在理解密切相关环境种群的基因组动态方面的不足至少可以通过宏基因组读取招募部分解决。事实上,宏基因组的阅读招募策略不仅被用于了解跨环境的单个类群的生物地理[57,58,59],还可以阐明个别环境种群内的遗传变异[60,61],使我们能够更深入地了解环境变化在时间和空间尺度上的人口动态。
基因组、宏基因组和计算策略的日益可用性为研究由于驯化逆转录因子的活性而在自然微生物种群中出现的遗传变异的动态但未被充分研究的景观提供了一个新的前沿[21,24,62].在这里,我们通过调查海洋种群来一瞥这些机会束毛藻属erythraeum使用一个孤立的基因组和海洋宏基因组。
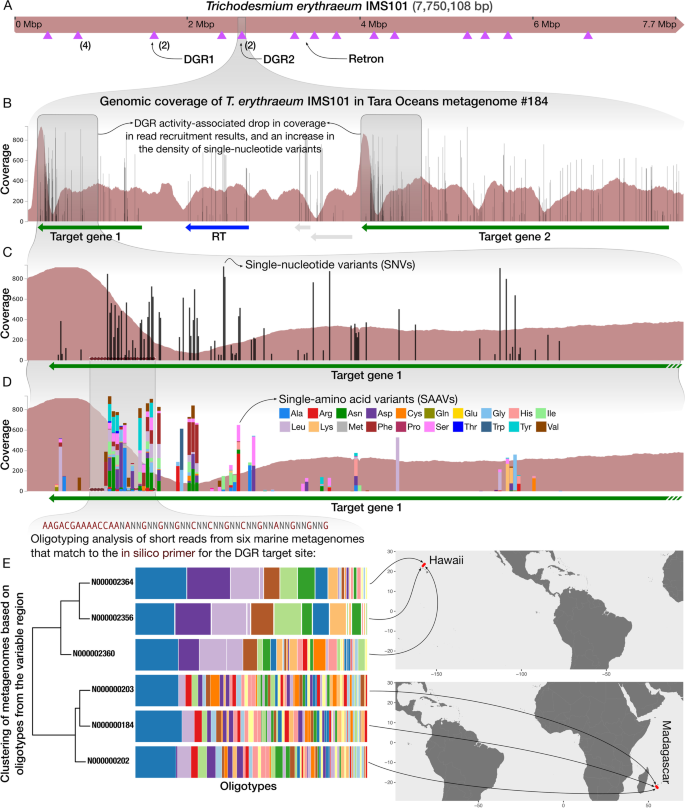
对参考基因组中的逆转录因子的调查表明,蓝藻门是最富含推测的DGRs和逆转录子的物种[23].束毛藻属,是海洋蓝藻的一个属,包括分布广泛和丰富的贫营养热带和亚热带海洋的谱系[63],并有助于海洋生境中氮的生物地球化学循环[64,65],例如t . erythraeum.Pfreundt等人证明DGRs的模板rna在t . erythraeumIMS101 [66],然而,在实验室培养中未检测到靶向诱变[66].探讨环境回溯元素的生态学t . erythraeum种群,我们使用分离的基因组束毛藻属erythraeumIMS101,包含至少10个不同的逆转录酶基因,包括两个DGR-RTs和一个retron(表S1,无花果。3.),以及来自塔拉海洋计划的六个宏基因组[67],其中t . erythraeum含量丰富(表S2).
生物信息学工作流调查宏基因组中的DGR活性和生态。一个基因组图谱束毛藻属erythraeum菌株IMS101,显示两个DGRs,一个retron和几个DGR靶基因的位置(紫色三角形)。Banvi 'o [68的一个部分的覆盖的可视化t . erythraeum从塔拉海洋计划宏基因组N000000184中招募的短读基因组,以及单核苷酸变异(SNVs)的分布。C靶基因1中SNV密度的增加和覆盖率的减少的详细表示,这表明DGR在环境种群中存在活性。D单氨基酸变异(saav)在基因同一区域的分布。saav的计算基于在宏基因组短读中发现的密码子的频率,这些短读完全映射到基因中给定的密码子位置[61],因此,它们可以更有效地量化环境种群的非同义变异程度束毛藻属积累了。E所有与引物序列完全匹配的宏基因组短reads的寡分型分析,其位置由面板上的横杆指示C而且D.序列模式考虑了读取募集结果中snv揭示的保守和可变的核苷酸位置。对每个样本中与模式匹配的宏基因组短读进行寡核苷酸分型分析[69],在此期间,高熵核苷酸位置将序列划分为不同的、生态相关的组。条形图显示了每个样本的寡型分布及其多样性,左侧的树状图显示了基于Jaccard相似性的样本之间的关系。最后,箭头指向夏威夷和马达加斯加附近的采样点,这些采样点与本分析中使用的宏基因组相对应
基因组由几个分散的逆转录酶基因组成(表2)S1),包括两个dgr -RT (Tery_1035和Tery_1728)和一个retron RT (Tery_2145),如前所述[29,38,66].每个DGR都有两个近端DGR靶基因(图2)。3.A),除了之前描述的远程靶基因外[29,66].在我们发现DGR-TR/VR同源性的18个位点中,至少有11个位点显示了宏基因组read招募结果显示的局部超变异模式(表2)S3).在宏基因组read募集结果中,局部的超变异性通常表现为覆盖范围的急剧下降和单核苷酸变异(SNVs)的增加,这是由于环境序列与参考基因组的一致性降低,这是DGR活动的可能结果。无花果。3.B在DGR2靶基因的背景下证明了这一现象。覆盖率和snv的变化是群体内阅读招募结果高变异性的有效指标(图2)。3.C),然而,snv并不是影响氨基酸组成的变化的充分指标[61].然而,这些信息可以通过分析单氨基酸变体(saav)来恢复,saav表示基于完全覆盖密码子的短读数的单密码子位置上氨基酸的等位基因频率。尽管密码子位置中的绝大多数非同义多样性通常由两个氨基酸表示[61]中相同DGR2目标区域对应的saavt . erythraeum揭示了氨基酸的大量多样性(图。3.D),证实了在宏基因组read招募结果中已知的dgr介导的序列进化机制。另一个重要的问题是观察到的变异是否与特定种群的生物地理有关。这个问题也可以通过宏基因组读取招募结果来解决。要深入描述高变区,需要使用原始的短读,因为即使来自基因组的同源区域,读取招募结果也会排除许多短读,因为它们的低同一性。一种选择是通过分析发现的读招募结果的snv来确定保守和可变核苷酸的模式,并在高质量过滤的配对端短读中搜索与这个“硅引物”匹配的读。识别这样的阅读t . erythraeum并对其进行寡核苷酸分型分析,根据假定的DGR活性,从夏威夷和马达加斯加收集的宏基因组之间存在明显的区别(图2)。3.E).该分析展示了一个生物信息学工作流程,可以通过使用宏基因组学(URL)来揭示逆向元素的动态生态和进化模式https://merenlab.org/dgrs-in-metagenomes详细介绍了类似应用程序的数据生成和可视化步骤)。结合短间隔纵向采样,这种方法可以帮助识别和表征任何栖息地自然发生的微生物种群的快速进化模式。
结论
DGRs和retrons都是神秘的遗传元素,因为它们在自然生态系统中的特定细胞功能、分子机制和活动方面仍有许多未被发现的地方。此外,这些有益的逆转录元素的进化史还远远没有解决,尽管理解这一复杂的问题将导致关于微生物适应生物冲突和环境压力的令人兴奋的启示。功能基因组实验可能会揭示DGRs或retrons的新的生物学作用,超越现有的宿主附着或动态抗噬菌体防御的预期变异范式。新的计算工具正在应用于多组学数据集,以研究不同生物群落中DGRs和retrons的生态和进化动态。为此,对个体基因组或泛基因组进行集中的时间检查,可能会导致关于逆转录因子对特定微生物的重要性的关键见解。
数据和材料的可用性
数据共享不适用于本文,因为在当前研究期间没有生成或分析新的数据集。
参考文献
杜拉托夫,刘敏,刘德华,等。bortela噬菌体向性转换定义了一个多样性生成逆转录因子家族。大自然。2004;431:476 - 81。可以从:https://doi.org/10.1038/nature02833.
McMahon SA, Miller JL, Lawton JA, Kerkow DE, Hodes A, Marti-Renom MA,等。c型凝集素折叠是大量序列变异的进化解决方案。分子生物学杂志,2005;12:886-92。
黎柯q,高希,等。密螺旋体多样性生成逆序元素中c型凝集素折叠的守恒。中国科学院学报(自然科学版)2011;
Millman A, Bernheim A, Stokar-Avihail A, Fedorenko T, Voichek M, Leavitt A,等。细菌逆转录子在抗噬菌体防御中起作用。细胞。2020;183:1551 - 61. e12汽油。
Bobonis J, Mitosch K, Mateus A, Kritikos G, Elfenbein JR, Savitski MM,等。噬菌体蛋白阻断和触发逆激毒素/抗毒素系统。可以从:https://doi.org/10.1101/2020.06.22.160242.
编码错误:由重复DNA序列错位介导的突变和重排。Mol Microbiol, 2004; 52:1243-53。
丁平B,范P,古德曼MF。容易出错的复制,无论好坏。微生物学。2004;12:288-95。可以从:https://doi.org/10.1016/j.tim.2004.04.004.
王志强,王志强,王志强,等。细菌种群的突变率动态反映了适应和遗传负荷之间的紧张关系。自然科学进展,2013;可以从:https://doi.org/10.1073/pnas.1219574110.
Martincorena I, Luscombe NM。非随机突变:有目标的超突变和低突变的进化。BioEssays。2013; 35:123-30。
Darmon E, Leach DRF报道。细菌基因组不稳定性。Microbiol Mol Biol rev 2014; 78:1-39。可以从:https://doi.org/10.1128/mmbr.00035-13.
梅达卡,米勒,j.f。Diversity-generating retroelements。《微生物学》,2007;10:388-95。
郭海华,郭伟林,郭志强,李志强。噬菌体和细菌基因组中产生多样性的逆转录因子。2.微生物学研究。2014;可以从:https://doi.org/10.1128/microbiolspec.MDNA3-0029-2014.
郭华,谢璐,Barbalat R, Sivaamnuaiphorn S,徐明,Doulatov S,等。产生多样性的逆序元素寻的再生目标序列用于重复的密码子重写和蛋白质多样化。Mol Cell, 2008; 31:13 - 23。
郭华,谢璐,聂卫华,Czornyj E, Williams S, Oukil S,等。目标站点识别的多样性生成回溯元素。PLoS Genet, 2011;7:e1002414。
Alayyoubi M, Guo H, Dey S, Golnazarian T, Brooks GA, Rong A,等。基本多样性生成逆序元素蛋白bAvd的结构及其与逆转录酶的重要功能相互作用。结构。2013;21:266 - 76。
韩达,姜杨,陶s, Foreman R, Schinazi RF, Miller JF,等。模板辅助合成腺嘌呤诱变cDNA的逆转录蛋白复合物。核酸res 2018; 46:9711-25。
王晓燕,王晓燕,王晓燕,等。腺嘌呤基因突变的遗传机制研究。核酸res 2021; 49:1033-45。
Simon DM, Zimmerly S.细菌中未鉴定的逆转录酶的多样性。核酸Res. 2008; 36:7219-29。
薛林杰,李志刚,陈志刚,陈志刚。基于DiGReF的生物多样性生成回溯元素综合数据集分析。BMC基因组学,2012;13:430。
Paul BG, Bagby SC, Czornyj E, Arambula D, Handa S, Sczyrba A,等。通过地内古菌和古菌病毒产生目标多样性。Nat community . 2015;6:6585。
Paul BG, Burstein D, Castelle CJ, Handa S, Arambula D, Czornyj E,等。回溯元素引导的蛋白质多样化存在于细菌和古生菌的大量谱系中。微生物学报。2017;2:17045。可以从:https://doi.org/10.1038/nmicrobiol.2017.45.
Castelle CJ, Brown CT, Anantharaman K, Probst AJ, Huang RH, Banfield JF。CPR和DPANN辐射中的生物合成能力、代谢多样性和不寻常生物学。微生物学报。2018;16:629-45。
吴玲,马金丽,王志强,王志强,等。多样性生成回溯元素:从大规模基因组调查推断的自然变异、分类和进化。核酸决议2018;46:11-24。
Roux S, Paul BG, Bagby SC, Nayfach S, Allen MA, Attwood G,等。全球微生物群超突变的生态学和分子靶标。Nat Commun. 2021;12:3076。
Minot S, Grunberg S, Wu GD, Lewis JD, Bushman FD。人类肠道病毒群中的高变异位点。自然科学学报(英文版)2012;
叶旸。人类微生物群落中产生多样性的逆转录元素的鉴定。中国生物医学工程学报(英文版);2014;
肖晓华,杨晓华,杨晓华,等。一种由全球普遍存在的拟杆菌噬菌体编码的产生多样性的反转录元素。微生物。2018;6:191。可以从:https://doi.org/10.1186/s40168-018-0573-6.
Morozova V, Fofanov M, Tikunova N, Babkin I, Morozov VV, Tikunov A.第一个编码多样性生成逆序元素(DGR)的cras - like噬菌体基因组。病毒。2020;12:573。可以从:https://doi.org/10.3390/v12050573.
Vallota-Eastman A, Arrington EC, Meeken S, Roux S, Dasari K, Rosen S,等。蓝藻中多样性生成逆转录因子调节途径的作用。BMC基因组学,2020;21:664。
王伟,王志强,王志强,王志强,等。由军团菌多样性生成回溯元件大量可变脂蛋白的表面显示。自然科学进展,2013;可以从:https://doi.org/10.1073/pnas.1301366110.
Lampson BC, Inouye M, Inouye S.黄粘球菌分枝rna链msDNA无细胞合成中的逆转录酶伴核糖核酸酶H活性。细胞。1989;56:701-7。
林D, Maas WK。大肠杆菌b细胞中一种共价连接的分枝DNA-RNA化合物的逆转录依赖合成。1989;50:891-904。可以从:https://doi.org/10.1016/0092 - 8674 (89) 90693 - 4.
Rest JS, Mindell DP。古生菌中的Retroids:系统发育和横向起源。《生物学杂志》2003;20:1134-42。
Lampson BC, Inouye M, Inouye S. Retrons, msDNA和细菌基因组。细胞遗传学杂志,2005;
崔维萨诺,井上。逆转录因子:未知功能的逆转录因子。微生物学,1995;3:209-11讨论211-2。
马志强,王志强,王志强,López-Beltrán A, González-Delgado A, Martínez-Abarca F等。UG/Abi:与防御功能相关的高度多样化的原核逆转录酶家族。bioRxiv。2021.2021.12.02.470933页。[引自2021年12月30日]。可以从:https://doi.org/10.1101/2021.12.02.470933v1.abstract.
西蒙AJ,艾灵顿AD,芬克尔斯坦IJ。逆转录子及其在基因组工程中的应用。核酸res 2019; 47:11007-19。
Mestre MR, González-Delgado A, Gutiérrez-Rus LI, Martínez-Abarca F, Toro N.细菌逆激子功能相关基因的系统预测和编码三方系统的分类。核酸res 2020; 48:12632-47。
Maxwell KL. Retrons:在噬菌体防御中补充CRISPR。Crispr j . 2020:226-7。可以从:https://doi.org/10.1089/crispr.2020.29100.kma.
高林,althai - tran H, Böhning F, Makarova KS, Segel M, schmiddleburgk JL,等。不同的酶活性介导原核生物的抗病毒免疫。科学。2020;369:1077 - 84。
Toro N, Martínez-Abarca F, Mestre MR, González-Delgado A.与CRISPR-Cas系统相关的多重逆转录酶来源。RNA生物学。2019;16:1486-93。可以从:https://doi.org/10.1080/15476286.2019.1639310.
González-Delgado A, Mestre MR, Martínez-Abarca F, Toro N.原核逆转录酶:从逆转录元件到专门的防御系统。45. FEMS微生物学Rev. 2021;可以从:https://doi.org/10.1093/femsre/fuab025.
Rice SA, Lampson BC。粘杆菌中retron元件的系统发育比较:垂直遗传的证据。中华微生物学杂志,1995;
Dodd IB, Barry Egan J.大肠埃希氏菌逆转录子Ec67和Ec86在186相关前噬菌体的cosite和转录终止子之间替换DNA。病毒学。1996;219:115-24。可以从:https://doi.org/10.1006/viro.1996.0228.
吴林。细菌逆转录酶的多样性研究。微生物学杂志2015;3:33-MDNA3-0058-2014。
Toro N, Nisa-Martínez R.细菌逆转录酶的综合系统发育分析。公共科学图书馆,2014;9:e114083。
Lambowitz AM, Belfort M.内含子作为移动遗传元件。生物化学,1993;62:587-622。
Cross KL, Campbell JH, Balachandran M, Campbell AG, Cooper CJ, Griffen A,等。逆向基因组学对未培养细菌的定向分离和培养。自然出版集团。2019;37:1314-21。
朱可夫DV, Khorosheva EM, Khazaei T, Du W, Selck DA, Shishkin AA,等。微流控滑片装置,用于纳米升规模的多步复合生物化学。芯片实验室。化学学报。2019;19:3200-11。
王文杰,张文杰,张文杰,等。基于液滴的高通量培养用于精确筛选抗生素耐药肠道微生物。生命科学出版有限公司;2020.[引自2021年12月31日]。可以从:https://elifesciences.org/articles/56998.
Anantharaman K, Brown CT, Hug LA, Sharon I, Castelle CJ, Probst AJ,等。数千个微生物基因组揭示了含水层系统中相互关联的生物地球化学过程。7. Nat Commun. 2016;[引自2021年12月31日]。可以从:https://pubmed.ncbi.nlm.nih.gov/27774985/.
帕克斯DH, Rinke C, Chuvochina M, Chaumeil PA, Woodcroft BJ, Evans PN,等。近8000个宏基因组组装基因组的恢复大大扩展了生命之树。2.生物工程学报。2017;[引自2021年12月31日]。可以从:https://pubmed.ncbi.nlm.nih.gov/28894102/.
杜德蒙,Quince C, Shaiber A, Esen ÖC, Lee ST, Rappé MS,等。表面海洋宏基因组中有丰富的Planctomycetes和Proteobacteria固氮种群。2 .生物工程学报,2018;[引自2021年12月31日]。可以从:https://pubmed.ncbi.nlm.nih.gov/29891866/.
帕索里E, Asnicar F, Manara S, Zolfo M, Karcher N, Armanini F,等。跨越年龄、地理和生活方式的超过15万个宏基因组揭示了广泛的未探索的人类微生物组多样性。细胞。2019;176年。[引自2021年12月31日]。可以从:https://pubmed.ncbi.nlm.nih.gov/30661755/.
Amarasinghe SL, Su S, Dong X, Zappia L, Ritchie ME, Gouil Q.长读测序数据分析的机遇与挑战。Genome Biol BioMed Central, 2020; 21:1-16。
叶艳。MyDGR:用于多样性生成回溯元素识别和表征的服务器。核酸研究,2019;47:W289-94。可以从:https://doi.org/10.1093/nar/gkz329.
王志刚,王志刚,王志刚。用于菌株分析的集成宏基因组学管道揭示了细菌传播和生物地理学的新模式。Genome Res冷泉港实验室出版社,2016;26:1612-25。
Edwards RA, Vega AA, Norman HM, Ohaeri M, Levi K, Dinsdale EA,等。广泛传播的人类肠道病毒裂解的全球系统地理学和古代进化。《自然微生物》自然出版集团2019;4:1727-36。
陈志强,陈志强,陈志强,陈志强,等。来自人类肠道考古组的1167个基因组目录。微生物学性质。自然出版集团。2022;7:48-61。
Simmons SL, DiBartolo G, Denef VJ, AliagaGoltsman DS, Thelen MP, Banfield JF。参与酸性矿井排水形成的钩端螺旋体II组菌株变异的群体基因组分析。公共科学图书馆,2008;6:e177。
杜丽娟,陈建平,陈建平,陈建平,Rappé MS,等。单氨基酸变异揭示了形成全球SAR11亚分支生物地理的进化过程。生命科学出版有限公司;2019.[引自2021年12月31日]。可以从:https://elifesciences.org/articles/46497.
李志强,李志强,叶勇,等。密螺旋体的基因组和宏基因组分析。前沿微生物。2016;7:852。[引自2021年12月31日]。可以从:https://doi.org/10.3389/fmicb.2016.00852.
卡彭DG。Trichodesmium,一种全球重要的海洋蓝藻。科学。1997;276:1221-9。可以从:https://doi.org/10.1126/science.276.5316.1221.
柏格曼B,桑德G,林S,拉尔森J,木匠EJ。Trichodesmium——一种广泛分布的海洋蓝藻,具有不同寻常的固氮特性。FEMS微生物学杂志2013;37:286-302。[引自2021年12月31日]。可以从:https://pubmed.ncbi.nlm.nih.gov/22928644/.
铁锹戳。发现非重氮养Trichodesmium物种丰富和广泛在公海。118.美国国立自然科学研究院,2021;[引自2021年12月31日]。可以从:https://www.pnas.org/content/118/46/e2112355118.abstract.
普弗朗特,科普夫,贝尔金,伯曼-弗兰克,赫斯。海洋重氮营养菌红毛霉IMS101的初级转录组。科学通报2014;4:6187。
Sunagawa S, Coelho LP, Chaffron S, Kultima JR, Labadie K, Salazar G,等。海洋浮游生物。全球海洋微生物群的结构和功能。科学。2015;348年。[引自2021年12月31日]。可以从:https://pubmed.ncbi.nlm.nih.gov/25999513/.
艾伦,谢伯尔,李志强,等。社区主导,集成,可重复的多组学与anvi 'o。Nat Microbiol. 2021; 6:3-6。可以从:https://doi.org/10.1038/s41564-020-00834-3.
Eren AM, Maignien L, Sul WJ, Murphy LG, Grim SL,等。寡分型:利用16S rRNA基因数据区分密切相关的微生物类群。方法生态学杂志,2013;4:1111-9。可以从:https://doi.org/10.1111/2041-210x.12114.
确认
BGP得到了Gordon and Betty Moore基金会和G. Unger Vetlesen基金会的支持。AME由西蒙斯基金会和阿尔弗雷德·p·斯隆基金会资助。
资金
BGP得到了Gordon and Betty Moore基金会和G. Unger Vetlesen基金会的支持。AME由西蒙斯基金会和阿尔弗雷德·p·斯隆基金会资助。
作者信息
作者及隶属关系
贡献
B.G.P.和a.m.e写了手稿。B.G.P.准备了图1。& 2和A.M.E.准备了图3。两位作者都审阅了手稿。作者们阅读并批准了最终的手稿。
相应的作者
道德声明
相互竞争的利益
作者声明没有利益竞争。
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商说明
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:
表S1。红毛霉IMS101中的逆转录酶基因及其相关逆转录元素特征的坐标(如适用)。表S2。假定远端DGR目标基因的坐标,以及基于同源性的相应模板重复序列(TR)。表S3。TARA全球海洋数据集的宏基因组细节用于毛孢泛基因组分析。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
保罗,b.g.,艾伦,A.M.微生物基因组中驯化逆转录因子的生态进化意义。移动DNA13, 6(2022)。https://doi.org/10.1186/s13100-022-00262-6
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s13100-022-00262-6