摘要
背景
自主逆转录元素长穿插元素-1 (LINE-1)使用RNA中间体(逆转录转位)通过复制和粘贴机制进行动员。在人类进化过程中,大约有50万个LINE-1序列在基因组中积累。这些序列大多属于祖先的LINE-1亚家族,包括L1PA2-L1PA7,并且不能再被调动。只有一小部分的LINE-1序列,大约80到100份属于L1Hs亚家族,是完整的,仍然能够进行逆转录转位。虽然在大多数细胞中沉默,但关于癌细胞中LINE-1失调的许多问题仍然存在。
结果
在这里,我们优化了CRISPR Cas9 gRNAs,以特异性靶向L1Hs 5’utr启动子的调控序列。我们发现了三种对L1Hs更特异性的gRNAs,与较老的LINE-1序列(L1PA2-L1PA7)结合有限。我们还采用C-BERST方法(限制性空间标记在基因组元件上的dCas9-APEX2生物素化)来识别癌细胞中的LINE-1转录调控因子。我们的LINE-1 C-BERST筛选显示了已知的和新的LINE-1转录调控因子,包括CTCF、YY1和DUSP1。
结论
我们对gRNA特异性的优化和评估以及C-BERST方法的应用为研究LINE-1在癌症中的调控机制创造了一个工具。此外,我们还鉴定了双重特异性蛋白磷酸酶DUSP1作为一种新的LINE-1转录调节因子。
简介
Long Interspersed element -1 (LINE-1)是人类基因组中唯一的自主移动元件。在数百万年的时间里,LINE-1序列通过逆转录转位的过程积累在我们的DNA中,这需要复制和粘贴RNA中间体[1,2].估计在人类基因组中存在50万个LINE-1拷贝,使LINE-1及其逆转录转位的研究复杂化[3.].然而,由于5 '截断、突变和反转,大多数LINE-1序列是无功能的。这些无功能序列主要是祖先的LINE-1序列,包括L1PA2-L1PA7亚家族,这些亚家族不再能调动[1,4,5].剩下的80-100个全长LINE-1序列属于人类特异性的LINE-1亚家族(L1Hs),能够逆转录转位,具有重塑我们基因组、改变基因表达和破坏基因组完整性的潜力[6,7,8].
LINE-1包含一个5 ' UTR,两个编码ORF1p和ORF2p蛋白的开放阅读帧,以及一个带有polyA尾巴的3'UTR [9,10].一个完全下游的意义启动子位于5'UTR内,控制LINE-1 mRNA的表达[11].此外,在5'UTR中发现了一个反义启动子,该启动子已被证明可以控制反义链上第三个开放阅读框ORF0的表达,以及其他反义转录本的表达[12,13,14].在LINE-1序列的进化过程中,5'UTR获得了新的调控序列,表现出宿主因子结合的快速进化,特别是KRAB锌指结合蛋白[15].LINE-1编码的蛋白质ORF1p和ORF2相对保守,两者都在逆转录转座中起作用[16,17].ORF1p是一种核酸伴侣,形成同源三聚体并结合LINE-1 mRNA [18,19].ORF1p也被证明可以结合ssDNA以及非line -1 mrna [20.,21].ORF2p的内切酶和逆转录酶结构域为逆转录转位提供了必要的酶活性[22,23].一旦表达,ORF1p和ORF2p结合LINE-1 mRNA形成核糖核蛋白(RNP)。在有丝分裂过程中,当核破裂时,RNP进入细胞核,在细胞核中,ORF2p内切酶切割DNA,并通过靶启动反转录(TPRT)产生一个新的LINE-1副本[24,25,26,27].LINE-1也被证明在非分裂细胞中具有逆转录转座作用,这表明有一种额外的进入细胞核的方式[28].
许多机制已经发展到在体细胞中沉默LINE-1的表达,限制了其调动的潜力。DNA甲基化、组蛋白修饰、RNA干扰和转录因子结合均被证明在限制LINE-1表达和限制逆转录转座中发挥作用[29,30.,31,32,33,34,35,36].其中许多机制在癌细胞中被破坏,允许LINE-1的重新表达和动员[37,38,39].在大约47%的肿瘤中观察到LINE-1蛋白ORF1p,并被认为是某些癌症侵袭性疾病的指标[37,40,41].在大约53%的研究肿瘤中也发现了新的LINE-1插入。在前列腺癌中,60%的肿瘤包含至少一个新的LINE-1插入,并且在转移性疾病中逆转录转位的速度加快[38].然而,这在不同的癌症中有所不同,因为在透明细胞肾细胞癌(ccRCC)中,在评估的肿瘤样本中没有检测到新的插入[42].这种在不同类型癌症之间LINE-1表达和逆转录转位的变化提示了LINE-1可能的细胞类型特异性调控。虽然LINE-1插入经常在内含子和非编码区被发现,但在肿瘤抑制基因中也发现了新的外显子插入,包括APC结直肠癌[33,43].除了直接破坏基因外,新的插入还可以通过诱导新的甲基化模式来改变基因组的调控格局[44,45,46].
LINE-1改变基因表达和驱动基因组不稳定性的潜力表明它可能促进癌症。更好地理解LINE-1的转录调节将有助于深入了解其在癌细胞中的异常调节和活性。迄今为止,Suv39h H3K9me3、HUSH复合物、SETDB1和KAP1都被证明在胚胎细胞中调控LINE-1元素[47,48,49,50].更多的研究通过motif分析在LINE-1 5'UTR上鉴定了Myc、CTCF、YY1和RUNX3的结合位点[32,51,52,53].对这些转录因子的进一步功能分析揭示了它们在转录调控和转录启动中的作用[32,52].在这里,我们优化了一个独特的CRISPR C-BERST模型,以进行一项无偏倚的研究,确定癌细胞中活性LINE-1 (L1Hs)的转录调控因子。虽然这项技术可以识别传统的转录因子,但它也可以识别不直接与DNA结合但也在转录调节中发挥作用的蛋白质。
结果
用dCas9 C-BERST靶向line - 15 ' utr启动子
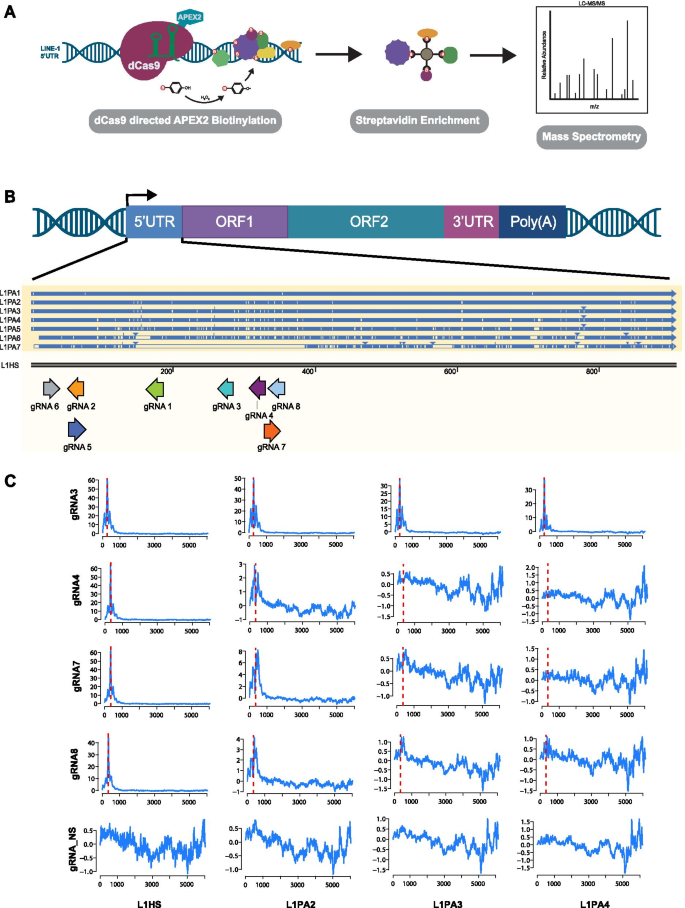
为了更好地理解LINE-1在癌细胞中的转录调控,我们利用dCas9 C-BERST (dCas9-APEX2B基因组上的IotinylationE克莱门特。由Restricted年代的可Tagging)方法映射与LINE-1启动子结合的调控蛋白[54].C-BERST利用核酸酶缺陷的Cas9 (dCas9)与抗坏血酸过氧化物酶APEX2融合。当表达时,dCas9-APEX2使用引导rna (gRNA)定向到特定的DNA位点。在用生物素-苯酚和过氧化氢处理后,APEX2生成生物素-phenoxyl自由基,这些自由基在约20nm半径内与生物素标记的蛋白质共价(图2)。1) (54].LINE-1启动子位于其5'UTR内,并被转录为LINE-1 mRNA的一部分,以在逆转录转位期间保存它[11].为了绘制癌症细胞中指导LINE-1转录的调控蛋白,我们将gRNAs定向到LINE-1 5'UTR启动子区域。
利用C-BERST方法定位line - 15 ' utr。一个用L1Hs特异性gRNAs将标记了dCas9的APEX2招募到line - 15 5'UTR启动子上。细胞与生物素-苯酚孵育30分钟,用双氧水处理1分钟,触发位于LINE-1 5'UTR启动子的蛋白质的生物素化。生物素化蛋白通过链霉亲和素免疫沉淀富集,并通过质谱鉴定。图及基于Gao等的方法[54].BL1PA2-L1PA7与L1Hs的比对。蓝线表示对l1h的正确对齐,奶油色线表示对l1h的不匹配对齐,箭头标记l1h中缺少的其他未对齐序列。设计了8个引导rna (gRNA)靶向LINE-1的5'UTR。优先选择与l1h排列良好的导线,但缺乏与旧的LINE-1序列(L1PA2-L1PA7)的对齐。C在表达8种LINE-1 5'UTR gRNAs的细胞中进行dCas9染色质免疫沉淀(ChIP),以及非靶向对照(NS)。使用MapRRCon分析软件将ChIP reads与LINE-1 L1Hs以及旧的L1PA2-L1PA7进行比对[32].红线表示每个gRNA的目标位置。gRNA 3、4、7和NS的代表性数据。所有grna的完整数据集可以在补充图中找到1
为了选择性地将dCas9引导到活跃的、逆转录转位有能力的LINE-1序列上,我们设计了8个专门针对L1Hs 5’utr的gRNAs。gRNA设计了5'UTR的目标区域,这些区域与旧的LINE-1序列L1PA2-L1PA7排列不佳(图2)。1B).接下来,我们在每个5'UTR gRNA和一个非靶向gRNA对照(NS)存在的情况下,对dCas9进行染色质免疫沉淀(ChIP)。使用LINE-1专用软件MapRRCon [32],我们将Cas9 ChIP reads对齐到L1Hs,以及较老的LINE-1元素L1PA2-L1PA7。正如预期的那样,所有8个grna都对准了L1Hs 5'UTR的目标区域(补充图)S1).当我们将ChIP读取序列对准较老的、无功能的LINE-1序列(L1PA2-L1PA7)时,gRNA 4、gRNA 7和gRNA 8富集了L1Hs结合,并开始失去对准以L1PA7开始的较老序列的质量。导路4、7和8几乎失去了所有与L1PA3-7的对齐,使它们最不可能靶向祖先的LINE-1(图1)。1C)。所有其他导联均显示与L1PA2和L1PA3对齐,如gRNA 3所示,但当它们与较旧的LINE-1序列对齐时,在5'UTR中失去了显著的峰。1C、补充图S1).选择gRNA 4和gRNA 7进行C-BERST检测,是因为它们对较年轻的、活跃的L1Hs序列具有高度特异性,对较老的L1PA2-L1PA7序列的募集最少。
通过dCAS9-APEX2生物素化鉴定line - 15 ' utr定位蛋白
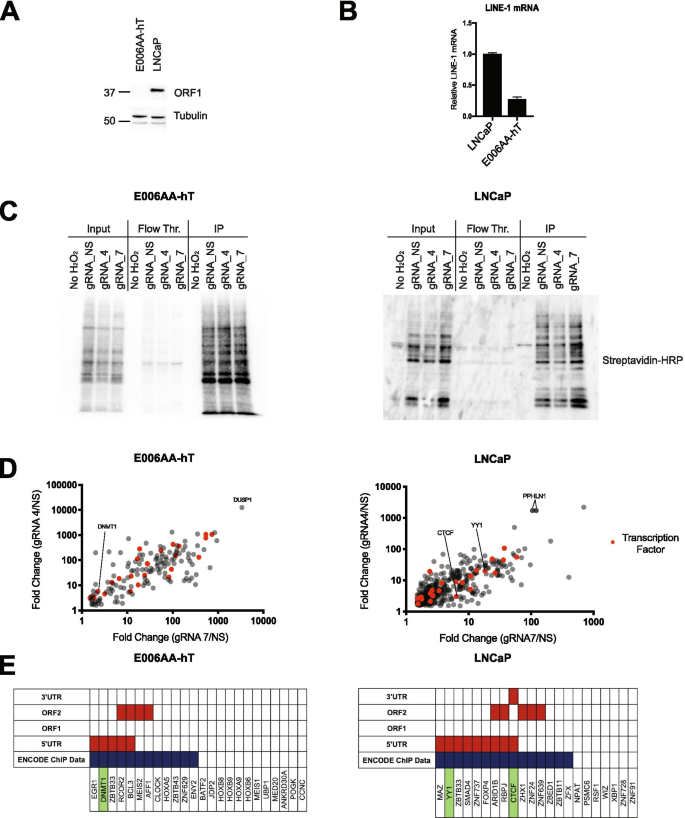
我们用来鉴定line - 15 ' utr结合蛋白的两种细胞系是LNCaP和E006AA-hT。LNCaP细胞是一种雄激素依赖性前列腺癌细胞系,已被证明表达line -1 ORF1蛋白和mRNA(图。2A和B). E006AA-hT细胞不表达可检测到的LINE-1 ORF1蛋白[55],并且LINE-1 mRNA的水平很低(图。2虽然E006AA-hT细胞最初被认为是前列腺癌细胞系,但后来发现它是肾细胞癌细胞系786‐O的克隆[56].这些细胞系中LINE-1表达的变化为更好地理解癌细胞中LINE-1转录激活和抑制提供了一个引人注目的模型。例如,E006AA-hT细胞中与LINE-1 5'UTR结合的蛋白质可能具有抑制活性,因为细胞不表达LINE-1。
5'UTR生物素化转录因子的C-BERST鉴定。一个Western blot检测E006AA-hT和LNCaP细胞全细胞裂解液中LINE-1 ORF1p的表达。(微管蛋白加载控制)。BqPCR检测LINE-1 mRNA的相对水平。CWestern blot检测C-BERST链霉亲和素免疫沉淀。收集表达gRNA-4、gRNA-7或gRNA-NS的细胞,并通过链霉亲和素免疫沉淀收集生物素化蛋白。A无过氧化氢对照(无H2O2)被包括在收获中,以消除内源性生物素化蛋白。用链霉亲和素HRP标记输入、通过和免疫沉淀(IP)。D与非靶向对照(gRNA NS)相比,C-BERST鉴定的蛋白在两种LINE-1靶向gRNA (gRNA-4, gRNA-7)中富集至少1.5倍。转录因子用红色标出。E与非靶向对照(gRNA NS)相比,在两种LINE-1靶向gRNA (gRNA-4, gRNA-7)中富集至少1.5倍的转录因子的完整列表。先前鉴定的调节LINE-1的蛋白质用绿色突出显示。蓝色方块表示ENCODE芯片数据可用性。红色方框为MapRRCon分析中LINE-1指定位置上的峰值
每个细胞系稳定转染dCas9-APEX2和gRNA (gRNA 4, gRNA 7或gRNA NS)。与之前的优化一样,细胞被排序以选择低dCas9 (mCherry)表达,以减少背景[54].多西环素诱导dCas9-APEX2 21 h,生物素-苯酚孵育30 min,双氧水处理1 min。双氧水淬灭并分离细胞核后,通过链霉亲和素免疫沉淀收集生物素化蛋白,质谱鉴定蛋白(图2)。2我们的筛选显示LNCaP细胞中有22个转录因子(356个总富集蛋白),E006AA-hT细胞中有24个转录因子(149个总富集蛋白),在两种LINE-1特异性指南中都比gRNA NS富集至少1.5倍(gRNA 4, gRNA 7)(图4)。2在这两个筛选中,我们都鉴定出了先前显示的调节LINE-1表达的蛋白,包括LNCaP细胞中的YY1、PPHLN1和CTCF,以及E006AA-hT细胞中的DNMT1(图2)。2E) (32,48,52,53,57].
MapRRCon验证转录因子
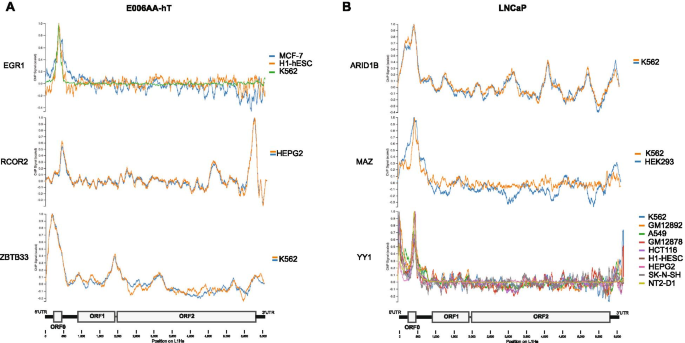
为了评估LINE-1 5'UTR上富集转录因子的存在,我们使用MapRRCon软件分析了现有的ENCODE ChIP数据。对于LNCaP细胞,我们分析了从C-BERST筛选中获得的15个转录因子,这些转录因子中有12个(80%)在LINE-1上显示峰值,9个(60%)映射到5'UTR上(图2)。2E和3.B)。同样,我们还分析了E006AA-hT C-BERST实验中富集的12个转录因子,发现7个(58.3%)在LINE-1上有峰,5个(41.6%)映射到5 ' utr上(图2)。2E和3.A).在两种细胞系中,都有转录因子在全长LINE-1上显示出不止一个峰值,包括RCOR2(图2)。3.).有趣的是,ENCODE ChIP数据是从多种细胞类型中收集的,并通过MapRRCon分析LINE-1上的峰值。许多鉴定的转录因子在多种细胞类型中都有峰值,这表明在调节LINE-1转录中具有广泛的作用(图。3.),而BCL3等则表现出明显的细胞类型差异(补充图)S2).
C-BERST蛋白的MapRRCon ChIP图谱。C-BERST富集蛋白的MapRRCon分析一个E006AA-hT和BLNCaP细胞。每个图都是对多个细胞系中可用的ENCODE ChIP数据的分析(列在每个图的右侧)。观察到的峰的位置由沿图底部的全长L1Hs图表示。其他MapRRCon图可在补充图中找到2
C-BERST揭示了LINE-1 mRNA表达的新调控因子
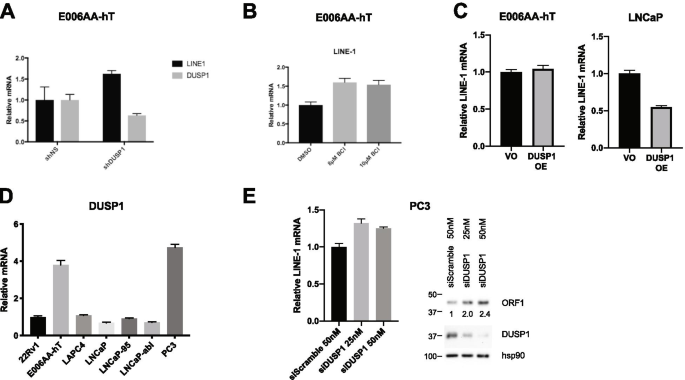
在我们的E006AA-hT C-BERST筛选中富集最多的蛋白是DUSP1,这是一种已知抑制MAPK的双特异性蛋白磷酸酶[58].为了测试DUSP1对LINE-1表达的影响,我们在E0006AA-hT细胞中用shRNA敲低DUSP1。我们用qPCR方法评估了LINE-1 mRNA水平,发现敲除DUSP1后LINE-1转录水平增加了1.6倍(图1)。4A).接下来,我们使用DUSP1抑制剂BCI,并通过qPCR评估其对LINE-1转录水平的影响。治疗后,我们再次看到LINE-1转录水平增加了1.5-1.6倍(图2)。4B). DUSP1仅在E006AA-hT C-BERST筛选中发现,说明DUSP1抑制LINE-1表达;为了验证这一假设,我们检测了其对LNCaP细胞中LINE-1转录水平的影响。在LNCaP细胞中过表达DUSP1后,我们观察到LINE-1转录水平下降了45%(图2)。4C)支持我们的假设。DUSP1在E006AA-hT细胞中的过表达导致LINE-1水平没有变化,可能是由于DUSP1在这些细胞中的饱和。我们还在PC3细胞中观察到高DUSP1 mRNA水平,PC3是一种前列腺癌细胞系,line -1表达水平较低(图2)。4D) (55].为了研究DUSP1是否在PC3细胞中发挥调节LINE-1的作用,我们用siRNA敲除DUSP1,并评估LINE-1 mRNA和ORF1蛋白水平(图2)。4E).敲低后,我们观察到LINE-1 mRNA和ORF1p蛋白水平均有升高。与siScramble相比,ORF1p蛋白水平增加了2.0倍和2.4倍。总之,我们的研究结果表明DUSP1在抑制癌细胞中LINE-1转录中发挥作用。
DUSP1调节LINE-1 mRNA的表达。一个表达shRNA到DUSP1或非靶向对照(NS)的E006AA-hT细胞中LINE-1 mRNA水平的qPCR。B载体(DMSO)或DUSP1抑制剂BCI处理的E006AA-hT细胞中相对LINE-1水平的qPCR。C仅过表达载体(VO)或DUSP1细胞E006AA-hT或LNCaP细胞中相对LINE-1 mRNA水平的qPCR。D前列腺癌细胞系(包括E006AA-hT肾细胞癌系)中DUSP1 mRNA水平的qPCR检测E用siScramble或siDUSP1 siRNA处理的PC3细胞中LINE-1 mRNA相对水平的qPCR(左)和LINE-1 ORF1p、DUSP1和hsp90(加载对照)蛋白水平的western blot(右)。定量ORF1蛋白相对于hsp90的水平,siScramble设为1
讨论
LINE-1在癌症中的异常调控及其扰乱基因组完整性的潜力强调了理解LINE-1转录调控的重要性。然而,人类基因组中丰富的祖先LINE-1序列增加了研究这一重复元素的挑战。在这里,我们利用染色质免疫沉淀和LINE-1特异性分析软件MapRRCon来缓解这一挑战,并评估8种CRISPR Cas9 gRNAs的L1Hs特异性。我们的分析显示,3个gRNAs横跨5'UTR 47 bp的区域,显示出对L1Hs的高特异性,而对L1PA2-L1PA7的低募集。这三种gRNAs与L1PA2一致序列差异1个(gRNA4)或2个(gRNA7, gRNA8)碱基对,与L1PA3差异2个(gRNA4, gRNA8)或3个(gRNA7)碱基对。虽然我们的gRNA与旧的LINE-1序列之间的这些差异很微妙,但我们的ChIP分析表明,它们破坏了gRNA向L1PA2-L1PA7的募集。在这三种gRNA中,我们选择使用gRNA 4和gRNA7进行C-BERST检测。虽然gRNA 8对L1Hs也表现出高度特异性,但它与我们之前确定的LNCaP细胞中表达的活性L1Hs序列并不完全一致(Xp22.2(2)) [21].由于Xp22.2(2)是LNCaP细胞中表达最高的LINE-1位点之一,我们决定将其排除作为参考。然而,gRNA 8可以作为可行的LINE-1 gRNA用于替代细胞类型。对这些grna的分析不仅为C-BERST靶向L1Hs建立了平台,还为用于靶向LINE-1 L1Hs的其他CRISPR应用提供了见解。
我们在两种不同的细胞类型LNCaP和E006AA-hT中制备了C-BERST,以评估LINE-1调控的不同机制。在LNCaPs中,我们之前已经发现许多不同的LINE-1位点被积极表达,观察到显著的ORF1p水平[21,55].虽然LNCaP细胞表达大量的LINE-1位点,但通过ORF1p RNA免疫沉淀分析,它们包含从高表达的LINE-1位点到不可检测的位点的表达范围[21].另一方面,E006AA-hT细胞有非常低的LINE-1 mRNA表达,没有检测到ORF1p的表达(图。2在LNCaP细胞中,我们在两种gRNAs的5'UTR位点上都发现了356个富集蛋白,其中22个是转录因子。在E006AA-hT细胞中,我们只观察到149个富集蛋白,包括24个转录因子。每个细胞系中富集蛋白总数的差异可能是由于LNCaP细胞中line -1表达的差异。不同的LINE-1位点可能有多种用于LINE-1激活或抑制的蛋白复合物,从而产生更多的富集蛋白。这种差异也可能是特定于细胞类型的,或者可能是由于LNCaP细胞的更高背景。在鉴定的转录因子中,只有ZBTB33在LNCaP和E006AA-hT细胞系中都被富集。这些结果表明在细胞和/或癌症类型之间LINE-1调控存在显著差异。
C-BERST方法被开发用于识别结合或定位于特定位点的蛋白质。我们的筛选显示了许多先前已被证明可调节LINE-1表达的蛋白质,并确定了新的假定的LINE-1调节因子。转录因子YY1和CTCF都在LNCaP细胞的5'UTR位点富集,之前已被证明与LINE-1 5'UTR结合并调节转录[32,52,53].PPHLN1是HUSH复合体的一个组成部分,在我们的LNCaP筛选中高度富集,并且也被证明可以抑制LINE-1的表达[48,59].HUSH复合物通过招募SETDB1来维持H3K9me3, SETDB1是另一种被证明可以调节LINE-1表达的蛋白质,在我们的实验中发现富集[60,61].然而,除了PPHLN1之外,HUSH复合体中没有其他成分被鉴定为富集,这可能是因为LNCaP细胞中许多LINE-1位点上的LINE-1没有被抑制。此外,在E006AA-hT细胞中,DNMT1(一种DNA甲基转移酶,先前已被证明可抑制年轻的LINE-1元件)在5'UTR位点富集[57].除了这些先前发现的蛋白,我们的MapRRCon分析在E006AA-hT细胞中发现了5个蛋白,在LNCaP细胞中发现了9个蛋白,这些蛋白在LINE-1 5'UTR中证实了ChIP峰。在我们的MapRRCon分析中,其他蛋白质在ORF2和3'UTR中显示出峰值。这些蛋白可能是由于LINE-1位点的次级染色质结构而富集的,然而,需要进一步分析来确定它们在LINE-1调控中的潜在作用。
双特异性磷酸酶1 (DUSP1)是E005AA-hT细胞中富集程度最高的蛋白。DUSP1通过去磷酸化苏氨酸/酪氨酸使MAPK失活,包括p38 MAPK, JNKs和ERKs [58,62].我们推测DUSP1可能会去磷酸化MAPKs,从而改变L1HS 5’utr蛋白复合物中下游转录因子的活性。在早期前列腺癌和膀胱癌中,DUSP1表达水平较高,但随着组织学分级的进展,DUSP1水平下降。我们的研究结果表明,DUSP1在E006AA-hT细胞和PC3细胞中始终有助于LINE-1的抑制。有趣的是,在LINE-1表达较低的细胞系中观察到DUSP1的高表达(图2)。4D) (55].虽然我们的C-BERST结果强烈表明它位于5'UTR附近,但还需要进一步的分析来探索DUSP1底物在LINE-1调控中的作用。由于我们要求蛋白质在两种引导物中都富集,因此我们的严格性可能消除了重要的辅因子和DUSP1底物。C-BERST的应用使我们能够识别直接结合在LINE-1 5'UTR上的蛋白质(ZBTB33, ERG1, GATAD1等),以及在传统序列和ChIP分析中可能被遗漏的瞬态调控蛋白(DUSP1)。总的来说,我们的LINE-1优化C-BERST实验能够识别细胞类型特异性的LINE-1转录调控因子。
结论
基因组中丰富的祖先LINE-1序列对研究主动L1Hs调控提出了重大挑战。在我们的研究中,我们发现了三种专门针对活性L1Hs的CRISPR Cas9 gRNAs,与旧的LINE-1序列结合最少。我们还利用限制性空间标记方法C-BERST来识别定位于line - 15 ' utr启动子的蛋白质。我们应用C-BERST方法在癌细胞中鉴定了已知的和新的LINE-1转录调控因子,包括双特异性磷酸酶DUSP1。我们优化了C-BERST方法,专门针对L1Hs启动子,创造了一个工具,可以用来更好地理解癌细胞中LINE-1表达的调控。
材料与方法
细胞培养
E006AA-hT (CRL-3277)和LNCaP (CRL-1740)细胞株购自ATCC。E006AA-hT细胞维持在添加10%胎牛血清的DMEM中。LNCaP细胞维持在添加10%胎牛血清的RPMI 1640中。定期评估细胞支原体污染情况。
siRNA可拆卸的
Human DUSP1 siRNA SMARTpool (# L-003484-02-005)和非靶向控制pool (# D-001810-10-05)购自Dharmacon。PC3细胞(2.5 × 105每孔细胞)分别接种于6个孔板上。根据制造商说明书,使用Lipofectamine RNAiMAX试剂(Life Technologies)转染25和50 nM sirna。连续两天进行转染。第一次转染72 h后收集RNA和蛋白。整个细胞裂解物在RIPA缓冲液(50 mM Tris pH 8, 150 mM NaCl, 1% NP-40, 0.1% SDS, 10 mM EDTA, 10 μg/mL抑质子素和leuptin, 1 mM PMSF, 1 mM Na)中收集3.签证官4),蛋白浓度用Bradford法定量。
shRNA击倒
将DUSP1和scramble shRNA克隆到pTRIPZ主干中。用Lipofectamine Reagent (Thermo Fisher 18324012) (2 μg pMD2G, 3 μg psPAX2, 5 μg pTRIPZ)将pTRIPZ质粒和病毒包装质粒转染HEK 293 T细胞,48 h后收集病毒并过滤。病毒上清液(4 mL)加入新鲜培养基(2 mL)和聚brene (8 μg/mL),与E006AA-hT细胞孵育4 h, 48 h后用嘌呤霉素(1 μg/mL)处理细胞。选中shRNA后,用1 μg/mL强力霉素诱导shRNA表达48 h。
sgRNA创建和设计
sgrna使用MIT指导RNA设计工具(CRISPR.MIT.edu)进行设计。至少有一个与旧的LINE-1 (L1PA2-L1PA7)序列不匹配的sgrna被优先考虑。一旦选择了序列,通过将sgNS序列替换为LINE-1靶向引导序列,将sgRNAs结构克隆到pejs614_pttet - p2a - bfpnls /sgNS中。引导序列可在补充表中找到2.
DUSP1过度
从DUSP1 GenScript质粒(OHu10841D)中扩增出DUSP1序列,并克隆到pCW57-MCS1-2A-MCS2主干(Addgene #71782)中。用Lipofectamine Reagent (Thermo Fisher 18324012) (2 μg svv - g, 3 μg gag-pol, 5 μg Overexpression质粒)将过表达质粒和病毒包装质粒转染HEK 293 T细胞,48 h后收集病毒并过滤。病毒上清液(4 mL)加入新鲜培养基(2 mL)和聚brene (8 μg/mL),与E006AA-hT或LNCaP细胞孵育4 h, 48 h后用嘌呤霉素(1 μg/mL)处理细胞。选中后,用1 μg/mL强力霉素诱导DUSP1表达48 h。
BCI治疗
E006AA-hT细胞接种于6孔板,37℃孵育过夜。然后用DMSO或BCI (Axon Medchem #2178)在37°C下处理细胞3小时。用Qiagen RNeasy Plus Mini Kit(74134)收集RNA,方法如下所示,并进行qPCR检测。
RNA分离和qPCR
使用Qiagen RNeasy Plus Mini Kit(74134)从细胞中分离RNA,并根据制造商的协议使用Turbo无DNA脱氧核糖核酸酶消化(赛默飞世尔科学AM1907)消化污染DNA。cDNA使用Verso cDNA试剂盒(Thermo Scientific- AB1453A)进行制备。qPCR使用SYBR Green Master Mix (Life Technologies 4344463)进行,相关mRNA水平使用ΔΔCT计算。RPL19被用作标准化的内部控制。引物序列见补充表2.qPCR LINE-1引物已发表[63].
染色质免疫沉淀反应
E006AA-hT和LNCaP细胞(~ 20 × 106)稳定表达dSpyCas9-mCherry-APEX2与gRNA融合度达到80%。用250 nM Sheild1 (Clontech)和2 μg/mL强力霉素处理细胞21 h,诱导dCas9表达。细胞在室温下与甲醛(PBS中含1%甲醛)交联10分钟,用1 mL 2.5 M甘氨酸淬灭,轻轻摇动5分钟。细胞用PBS洗涤,在4℃下以425xg成粒5分钟,再悬浮在法纳姆裂解缓冲液中(5 mM PIPES pH 8.0, 85 mM KCL, 0.5% NP-40, Halt蛋白酶抑制剂(Thermo fish -87786))。将悬浮液重新制成颗粒状,并在液氮中快速冷冻。将球丸重悬于含有Halt蛋白酶抑制剂的1ml法纳姆裂解缓冲液中,通过25刻度注射器15次,在4℃下以425xg旋转5分钟。将球丸在RIPA缓冲液(1 × PBS, 1%NP-40, 0.5% na -脱氧胆酸盐,0.1% SDS, Halt蛋白酶抑制剂)中重悬,并通过25号注射器20次。裂解物在Diagenode Bioruptor中超声30分钟,开30秒,关30秒,以20800xg旋转10分钟。剪切的DNA用4 μg mCherry抗体(Thermo PA5-34,974)在4℃孵育过夜,用50μL蛋白a /G珠在4℃孵育3小时。用LiCl洗涤缓冲液(100 mM Tris pH7.5, 500 mM LiCl, 1% NP-40, 1% Na-脱氧胆酸盐)洗涤5次,用TE缓冲液(10 mM Tris - hcl pH7.5, 0.1 mM Na)洗涤3分钟2珠子在蛋白酶K/SDS溶液(0.5% SDS, 0.2 mg/mL蛋白酶K, 1X TE)中重悬,在55°C下孵育3小时,65°C过夜以逆转交联。样品置于磁条上收集上清。在样品中加入600μL PB和4μL RNaseA (17500u/mL),使用Qiagen PCR纯化试剂盒(28104)对样品进行纯化。用35μL、10 mM Tris pH 8洗脱样品2次。Illumina文库使用NEB Next DNA文库Prep Ultra II试剂盒(E7645S)根据制造商的协议生成。文库在Illumina NextSeq 500上测序。读取用Illumina bcl2fastq v2.20解复用,需要与索引BC序列完美匹配。
细胞分类
LNCaP和E006AA-hT细胞经强力霉素(2 μg/mL)和Sheild1 (250 nM)处理21 h后进行分选。细胞使用SONY SY3200并行分拣机(SONY Biotechnology, San Jose, CA)进行分类,使用100µm孔口喷嘴和大约25 psi的系统压力。mCherry和BFP双阳性细胞如前所述纯化[54].
C-BERST化验
生物素化:7个15厘米厚的E006AA-hT或LNCaP细胞板(~ 6 × 107用2 μg/mL强力霉素和250 nM shield 1处理细胞21 h。生物素-苯酚(500 μM)在37℃、1 mM h下孵育30 min2O2在室温下加入细胞1分钟。为了停止生物素化反应,加入猝灭剂溶液(5 mM trolox, 10 mM抗坏血酸钠和10 mM叠氮化钠),并将细胞放置在冰上。用淬冷剂溶液进行三次额外洗涤,然后用PBS洗涤两次。
核分离:从平板上刮取细胞,在4℃下300 × g离心5分钟。颗粒在7.5核分离缓冲液(10 mM PIPES pH 7.4, 0.1% NP-40, 10 mM KCl, 2 mM MgCl)中重悬2, 1 mM DTT和Halt蛋白酶抑制剂)。细胞在冰上孵育10分钟,用Dounce均质器(~ 20倍)使细胞破裂。细胞进一步在冰上孵育20分钟,重复均质。将裂解液轻轻添加到含有20 mL 30%蔗糖和3.5 mL 10%蔗糖(10 mM PIPES pH 7.4, 10 mM KCl, 2 mM MgCl)的蔗糖垫中2, 30%或10%蔗糖,1mm DTT)。蔗糖垫和细胞裂解液在1000 × g下旋转15 min,去除上清,用800μL PBS重悬核(球)。悬浮液在4℃下以1500 × g纺丝5 min。加入500μL RIPA裂解缓冲液(50 mM Tris-HCl pH 7.5, 150 mM NaCl, 0.125% SDS, 0.125%脱氧胆酸钠,1% Triton X-100), 4℃孵育。裂解物在Diagenode Bioruptor中超声15分钟(开30秒,关30秒),然后在15800 × g, 4℃下离心10分钟。用Bradford法测定蛋白质浓度,并将样品归一化。
免疫沉淀:每个样品加入MyOne Streptavidin T1 Dynabeads (Thermo Fisher 65,601) (400μL), 4℃孵育过夜。用RIPA(两次)、1 M KCl、0.1 M Na清洗珠子2有限公司3., 2 M尿素在10 mM Tris-HCl pH 8.0,再与RIPA(两次)。水洗后,对珠子进行质谱分析链霉亲和素结合蛋白的珠上消化).协议基于之前描述的C-BERST技术[54].
Western blot/streptavidin blot
细胞裂解物在SDS加载缓冲液中在98°C下煮沸5分钟。样品在聚丙烯酰胺凝胶上通过SDS-PAGE分离,并使用BioRad Trans-Blot Turbo Transfer System转移到PVDF上。在TBS中5% BSA中阻断印迹,并用链霉菌亲和素- hrp (Thermo Fisher SA10001)、ORF1p (Millipore MABC1152)、DUSP1 (Cell Signaling 48625)或HSP90 (BD Biosciences 610419)进行检测。Western blots使用BioRad Clarity Western ECL Substrate (1705060S)进行显影,并在iBrightCL1000成像仪上进行可视化。蛋白质条带用ImageJ定量[64].
链霉亲和素结合蛋白的珠上消化
链霉亲和素珠用1 mL 50 mM NH冲洗两次4HCO3.交换缓冲区。用50 μl 50 mM NH重悬4HCO3.含有20 ng/μl trypsin/Lys-C (Promega),然后在37°C下孵育一夜,并在恒温器中剧烈混合(Eppendorf)。孵育后将珠粒制粒,上清液转移到新管中。加入20%七氟丁酸5 μl酸化,室温孵卵5 min, 16000 g离心5 min澄清。根据制造商的说明,使用C18旋转尖端(Thermo Scientific)对澄清样品中的肽进行脱盐。脱盐的多肽在真空下干燥,并在LC-MS分析之前在0.1%甲酸中重新溶解。在Nanodrop One (Thermo Scientific)上测量205 nm处的肽浓度。
质分析
在Orbitrap Fusion Lumos质谱仪上结合Dionex Ultimate 3000 UHPLC对多肽进行LC-MS分析。每次运行时,从单个样品中提取0.5-2 μg多肽,在50cm长的EASY-Spray柱(Thermo Scientific)上以0.25 μl/min的流速,用4-40%乙腈在0.1%甲酸溶液中线性梯度分解90 min。依赖数据的采集方法基于已发布的协议[65]除了每个周期被设置为持续2秒而不是3秒。
多肽鉴定和无标签定量在Proteome Discoverer 2.1中完成。Sequest HT搜索引擎的蛋白质数据库包括从UniProt下载的人类蛋白质组(www.uniprot.org)和链霉菌亲和素的氨基酸序列。设置参数以搜索长度至少为5个氨基酸的多肽,其中包含最多2个缺失的胰蛋白酶裂解。动态修饰包括:丝氨酸、苏氨酸或酪氨酸的磷酸化,蛋白n端乙酰化,赖氨酸和精氨酸的单甲基化和二甲基化。使用Proteome Discoverer中的“前体离子区域检测器”模块进行基于ms1的无标签定量。首先用检测到的链霉亲和素的强度归一化样品。将面积值为0的样品替换为检测到的最低MS1强度。接下来,H2O2仅(内源性生物素化)值从+ H中减去2O2样本(gRNA NS, gRNA 4和gRNA7)。从重复中计算平均值,目标导向(gRNA 4和gRNA 7)除以每个检测到的蛋白质的gRNA NS值。大于gRNA NS对照至少1.5倍的蛋白被认为富集,并纳入进一步分析。
数据和材料的可用性
试剂可根据要求提供。
缩写
- 1号线:
-
长穿插元素-1
- C-BERST:
-
限制性空间标记法在DCas9-APEX2基因组元素上的生物素化
- DUSP1:
-
双特异性蛋白磷酸酶
参考文献
Konkel MK, Walker JA, Batzer MA。灵长类动物进化的正弦线。人类进化。2010;19(6):236-49。
Boeke JD,等人。Ty元素通过RNA中间体转置。细胞。1985;40(3):491 - 500。
Lander ES,等。人类基因组的初步测序和分析。自然。2001;409(6822):860 - 921。
Szak ST,等。人类基因组L1插入的分子考古学。中国生物医学工程学报,2002;3(10):research0052。
格里马尔迪G, Skowronski J,辛格MF。定义KpnI族段的开始和结束。中国科学d辑,2004;3(8):1753-9。
Brouha B,等。热l1基因占人类逆转录转位的大部分。中国科学(d辑:自然科学版),2003;21(3):344 - 344。
黄cr, Burns KH, Boeke JD。基因组中的主动转位。《热内年鉴》2012;46:651-75。
Beck CR,等。人类基因组中LINE-1逆转录转位的活性。细胞。2010;141(7):1159 - 70。
Scott AF,等人。人类L1元件的起源:从一致的DNA序列推断出的先祖基因。基因组学。1987;1(2):113 - 25所示。
Dombroski BA,等。分离出一个活跃的人类转座因子。科学。1991;254(5039):1805 - 8。
Swergold GD。人LINE-1启动子的鉴定、鉴定和细胞特异性。分子生物学杂志,1990;10(12):6718-29。
Criscione SW,等。人类L1反义启动子驱动转录本的全基因组特征。BMC Genomics. 2016;17:463。
邓利AM,等。灵长类动物特异性ORF0有助于逆转录转座子介导的多样性。细胞。2015;163(3):583 - 93。
人类L1逆转录转座子的反义启动子驱动相邻细胞基因的转录。分子生物学杂志2001;21(6):1973-85。
Jacobs FM,等。KRAB锌指基因ZNF91/93和SVA/L1逆转录转座子之间的进化军备竞赛。大自然。2014;516(7530):242 - 5。
王晓明,王晓明,王晓明,等。人类LINE-1逆转录转座子的分子进化及其扩增速度。基因组学报,2006;16(1):78-87。
莫兰JV,等。培养哺乳动物细胞的高频逆转录转位。细胞。1996;87(5):917 - 27所示。
Martin SL,等。L1逆转录转位中一种必需蛋白的三聚体结构。中国科学(d辑:自然科学),2003;29(3):344 - 344。
马丁SL,布西曼FD。小鼠LINE-1逆转录转座子ORF1蛋白的核酸伴侣活性。分子生物学杂志,2001;21(2):467-75。
Kolosha VO, Martin SL.来自小鼠LINE-1的第一个ORF蛋白的体外性质支持其在逆转录转位过程中核糖核蛋白颗粒形成中的作用。中国科学院学报(自然科学版),2001;29(2):379 - 379。
Briggs EM等人。RIP-seq揭示LINE-1 ORF1p与p体富集mrna的关联。暴徒的DNA。2021; 12(1): 5。
冯Q,等。人L1逆转录转座子编码逆转录转座子所需的保守内切酶。细胞。1996;87(5):905 - 16。
Mathias SL,等。由人类转座因子编码的逆转录酶。科学。1991;254(5039):1808 - 10。
Mita P,等。LINE-1蛋白在细胞周期中的定位和功能动态。Elife。2018;7:e30058。
成本GJ,等。人L1元素靶启动的体外逆转录。中国生物医学工程学报。2002;21(21):5899-910。
栾丹丹,等。R2Bm RNA的逆转录是由染色体靶位点上的一个缺口启动的:非ltr逆转录转位的机制。细胞。1993;72(4):595 - 605。
Idica A,等。MicroRNA miR-128通过下调核输入因子TNPO1抑制LINE-1 (L1)逆转录转位。中国生物医学工程学报,2017;
Kubo S,等。非分裂和原代人体细胞L1逆转录转位。中国科学(d辑),2006,26(3):344 - 344。
陈琳,等。自然发生的内源性sirna通过DNA甲基化沉默人类细胞中的LINE-1逆转录转座子。表观遗传学。2012;7(7):758 - 71。
Goodier JL,张LE, Kazazian HH Jr. MOV10 RNA解旋酶是细胞逆转录转位的有效抑制剂。公共科学学报,2012;8(10):e1002941。
尤德JA,沃尔什CP,贝斯特TH。胞嘧啶甲基化与基因组内寄生虫的生态学。植物学报,1997;13(8):335-40。
孙X,等。转录因子分析揭示了人类逆转录转座子表达的分子编排和关键调控因子。中国科学(d辑),2018;29(3):366 - 366。
Scott EC,等人。热L1逆转录转座子逃避躯体抑制并引发人类结直肠癌。基因组学报,2016;26(6):745-55。
Nguyen THM,等。L1逆转录转座子在卵巢肿瘤细胞进化中的异质性。Cell Rep. 2018;23(13): 3730-40。
尤因AD,等。纳米孔测序可实现全面的转座元件表观基因组分析。中国生物医学工程学报。2020;40 (5):915-928.e5。
Grundy EE, Diab N, Chiappinelli KB。转座因子在癌症中的调控和表达。2021年2月。https://doi.org/10.1111/febs.15722
Rodic N,等。长时间穿插的元素1蛋白表达是许多人类癌症的标志。中华流行病学杂志。2014;34(5):366 - 366。
Tubio JM,等。癌症中的移动DNA。L1逆转录转位介导的非重复DNA在癌症基因组中的广泛转导。科学。2014;345(6196):1251343。
KH燃烧。癌症中的转座因子。中华癌症杂志,2017;17(7):415-24。
哈里斯CR,等。长分布的核元素-1蛋白在乳腺肿瘤中的核定位与不良预后的相关性。中华癌症杂志。2010;1(2):115-24。
Ting DT,等。胰腺和其他上皮性癌症中卫星重复序列的异常过表达。科学。2011;331(6017):593 - 6。
Helman E,等人。全基因组和外显子组测序显示人类癌症中的体细胞反转录转位。基因组学报,2014;24(7):1053-63。
Miki Y,等人。结肠癌中L1序列反转座子插入对APC基因的破坏。癌症杂志1992;52(3):643-5。
Beck CR,等。结构变异与疾病中的LINE-1元素。中国生物工程学报。2011;12:187-215。
格兰迪FC等。反向转位产生倾斜的海岸:低甲基化CpG岛对侧翼CpG位点的分级影响。中国生物工程学报,2015;25(8):1135-46。
Garcia-Perez JL等。人胚胎癌细胞中L1逆转录转位事件的表观遗传沉默。自然。2010;466(7307):769 - 73。
Bulut-Karslioglu A,等。suv39h依赖的H3K9me3标记完整的逆转录转座子并沉默小鼠胚胎干细胞中的LINE元素。中国生物医学工程学报。2014;36(2):379 - 379。
Robbez-Masson L等人。HUSH复合物与TRIM28合作抑制年轻的反转录转座子和新基因。基因组学报,2018;28(6):836-45。
卡斯特罗-迪亚兹等人。胚胎干细胞中L1的进化动态调控。基因开发,2014;28(13):1397-409。
麦克法兰TS,等。内源性逆转录病毒和邻近基因被LSD1/KDM1A协调抑制。基因开发,2011;25(6):594-607。
杨n,等。RUNX3在人类L1转录和逆转录转位中的重要作用。中国生物医学工程学报,2003;31(16):4929-40。
Athanikar JN, Badge RM, Moran JV。精确的人类LINE-1转录起始需要yy1结合位点。中国生物医学工程学报,2004;32(13):3846-55。
Sanchez-Luque FJ,等。人类表观遗传抑制的LINE-1逃避。中国生物医学工程学报,2019;29 (3):374 - 374 .e12。
高晓东,等。C-BERST:用dCas9-APEX2定义基因组元素的亚核蛋白质组学景观。光子学报。2018;15(6):433 - 36。
Briggs EM等人。前列腺癌细胞中长时间穿插的核元素-1表达和反转录转位。暴徒的DNA。2018; 9:1。
Koochekpour S,等。更正:原发性雄激素反应性非裔美国人前列腺癌细胞系E006AA的建立和鉴定。前列腺60 2004;(2):145 - 152。前列腺癌。2019;79(7):815。
Jonsson ME,等。在人类神经祖细胞中,通过LINE-1元件对全局DNA去甲基化的神经元基因激活。地理学报。2019;10(1):3182。
富兰克林CC,卡夫AS。U937细胞中丝裂原活化蛋白激酶(MAPK)磷酸酶MKP-1的条件表达优先抑制p38 MAPK和应激活化蛋白激酶。中国生物医学工程学报,2001;29(3):344 - 344。
Tunbak H,等。HUSH复合体是I型干扰素的看门人,通过LINE-1s的表观遗传调控。网络学报。2020;11(1):5387。
奎利亚尔TL等。SETDB1沉默逆转录转座子抑制急性髓系白血病的干扰素反应。中国生物医学工程学报,2017;26(4):457 - 457。
Tchasovnikarova IA,等。基因沉默。HUSH复合物的表观遗传沉默介导人类细胞中的位置效应杂色。科学。2015;348(6242):1481 - 5。
朱毅,等。有丝裂原激活的蛋白激酶磷酸酶PAC1、MKP-1和MKP-2具有独特的底物特异性,并且在体内对ERK2 7 - maker突变的活性降低。中国生物医学工程学报。1996;21(11):457 - 457。
戈林W,利巴斯卡T,舒尔茨W。人类前列腺癌逆转录因子表达的选择性改变。致癌作用。2011;32(10):1484 - 92。
Schneider CA, Rasband WS, Eliceiri KW. NIH图像到ImageJ: 25年的图像分析。自然科学进展。2012;9(7):675 - 5。
戴维斯S等。利用电荷有序平行离子分析(CHOPIN)结合广泛特异性蛋白水解扩大蛋白质组的覆盖范围。中国生物医学工程学报,2017;16(3):1288-99。
确认
Erik Sontheimer慷慨地提供了Cas9-Apex2和sgRNA质粒用于这些实验。(Addgene质粒# 108649,# 108570)。所有测序均由纽约大学朗格尼系统遗传学研究所完成。我们还要感谢Raven Luther和Megan Hogan的专业技术。
利益冲突
Jef Boeke是CDI Labs, Inc.的创始人和董事,Neochromosome, Inc.的创始人,重开诊断公司的创始人和SAB成员,并担任或曾担任以下公司的科学顾问委员会:Sangamo, Inc., Modern Meadow, Inc., Sample6, Inc.和Wyss研究所。
资金
这项工作得到了美国国立卫生研究院拨款R01CA112226(给s.k.l.), F31CA225053-01A1(给E.M.B.), P01AG051449(分包给J.D.B.), R21CA235521给J.D.B., R01GM127267(给E.N.),布拉瓦尼克家庭基金会(E.N.)和霍华德休斯医学研究所(E.N.)的支持。
作者信息
作者及隶属关系
贡献
E.M.B, p.m., s.k.l.和J.D.B.设计了这项研究。E.M.B, p.m., S.H.和Z.R.L.进行了实验并分析了数据。X.S, P.M.和J.D.B.用MapRRCon软件分析了ENCODE芯片数据。N.V.进行了质谱分析。S.K.L, J.D.B.和E.N.提供监督监督。emb写了手稿。所有作者都提供了输入并批准了手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:补充图1。
CRISPR Cas9 gRNA定位到LINE-1的完整ChIP评估gRNA1-8和gRNA- ns在细胞中用dCas9表达。在每个指导下进行ChIP,并用MapRRCon评估ChIP数据,以量化每个gRNA的LINE-1 (L1Hs, L1PA2-L1PA7)定位。沿x轴在全长LINE-1上的位置。沿着y轴折叠输入以上的富集量。红色虚线标记gRNA目标站点。
附加文件2:补充图2。
C-BERST富集转录因子的MapRRCon ChIP附加图。具有确定的LINE-1峰的富集转录因子的ChIP图。
附加文件3:补充表1。
LNCaP和E006AA-hT细胞中C-BERST富集蛋白。与非靶向对照(gRNA NS)相比,gRNA 4和gRNA 7中富集至少1.5倍的蛋白质的完整列表。
附加文件4:补充表2。
gRNA和qPCR引物序列
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
布里格斯,e。m。米塔,P。et al。利用CRISPR Cas9对LINE-1启动子进行无偏置蛋白质组学定位。移动DNA12, 21(2021)。https://doi.org/10.1186/s13100-021-00249-9
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s13100-021-00249-9
关键字
- 1号线
- 转录调控
- 癌症
- C-BERST
- CRISPR Cas9限制性空间标记