摘要
背景
正弦序列构成了动物基因组的重要组成部分,并被用于研究不同分类群的进化。尽管在脊椎动物和高等真核生物的研究中取得了重大进展,但人们对它们自身的进化知之甚少。
结果
我们发现并详细描述了一种新的Squam3 SINE,专门用于有鳞爬行动物(Squamata)。该基因的亚科在鳞片动物基因组中表现出不同的分布,并结合蜥蜴中类似基因的数据,使我们能够在爬行动物进化的背景下提出它们的进化方案。
结论
在大多数基因组中保存少量的祖先正弦可以产生特定分类群的正弦家族。分析这方面的正弦可以揭示爬行动物基因组中正弦变异的历史和机制。
背景
基因组受到各种重复元件的入侵,其中最丰富的(至少在高等真核生物中)是长穿插元件和短穿插元件(分别为LINEs和sin)。这些逆转录转座子的扩增周期包括其基因组拷贝的转录、逆转录和整合到基因组中。细胞系依赖于细胞RNA聚合酶II的转录,而逆转录和整合则由它们自己的酶完成。SINEs不编码任何酶,并利用RNA聚合酶III (pol III)转录的细胞机制和它们的伙伴LINE的机制进行逆转录和整合到染色体中。因此,SINEs具有用于转录的pol III启动子和被其伙伴LINE酶识别的序列,用于逆转录/整合。
一个典型的SINE包括来自细胞RNA种类之一的头部(tRNA, 7SL RNA或5S RNA);身体,其末端部分被伴侣逆转录酶(RT)识别;尾巴是一段简单的重复。有各种各样的变化;某些正弦函数没有主体,或者其主体包含未知起源和功能的序列(其中一些称为中心结构域),这些序列在其他不相关的正弦函数家族之间共享,等等。[1]。
系存在于所有高等真核生物的基因组中。很明显,正弦不能没有线而存在,反之亦然;有一些罕见的基因组有LINEs但没有sin(例如,酿酒或果蝇)。在进化过程中,LINE(亚)家族可能变得不活跃,它们的伙伴sin也停止扩增。如果另一个LINE家族在特定基因组中变得活跃,替换其RT识别的序列可以重新激活SINE [2]。通常,一个基因组包含一个或几个SINE家族;其中一些可能是无活性的,并在祖先中扩增。对不同分类群中SINE变化的分析使我们能够将其作为可靠的系统发育标记[3.,4]。
爬行动物-鸟类分支的主要分支是有鳞爬行动物(Squamata)、鳄蜥(Rhynchocephalia)、海龟(Testudines)、鳄鱼(Crocodilia)和鸟类(Aves)。鳞目是爬行动物中最大的一目,包括以下主要的分支:蛇类、鬣蜥类(包括鬣蜥类、龙蜥类、变色龙类)、Anguimorpha、Scincomorpha、Lacertoidea、Gekkota和Amphisbaenia。由于分子、形态学和古生物学数据提供的相互矛盾的信号,鳞片类爬行动物之间的系统发育关系备受争议。它们与舌头动物中唯一现存的代表物种鳄蜥一起,构成了单系超目鳞翅目,是古龙目的姐妹类群,古龙目包括古龙(鳄鱼和鸟类)和海龟[5]。
1990年,人们在中华塘龟身上发现了首个SINE爬行动物[6];目前,我们知道爬行动物中大约有10个SINE家族[1]具有不同的分类分布,例如,Cry仅限于海龟和羊膜动物祖先中活跃的amnsin的退化副本[7],可以在爬行动物之外找到。另一个例子是Ther1,最初被描述为哺乳动物的sin (MIR),但后来更名为[8,9]。几个已知的Ther1/MIR亚家族(MIRb, MIRc和MIR_Testu)与Ther1有轻微的差异,除了在基因上的差异鳄鱼mississippiensis的带有扩展删除的MIR1_AMi。此外,在非鸟类爬行动物中也发现了活跃的Ther1/MIR sin,因此在它们的基因组中可以预期有丰富多样的衍生sin [10]。爬行动物L2的积极多样化进一步证实了这一点[11]。
尽管对各种蜥蜴和蛇的基因组进行了积极的测序,但没有在不同分类群中对SINE家族在目水平上的详细比较基因组研究。我们在…的基因组中发现了一个名为Squam3的新SINEDarevskia和安乐蜥蜴。进一步分析表明它们分布在整个鳞片中;在鳄蜥中也发现了类似的SINE [12但在其他爬行动物或鸟类中没有。然而,Squam3在近40个鳞片基因组中未被发现。在此,我们分析了Squam3及其近亲的结构、分布和进化。
结果
Squam3识别
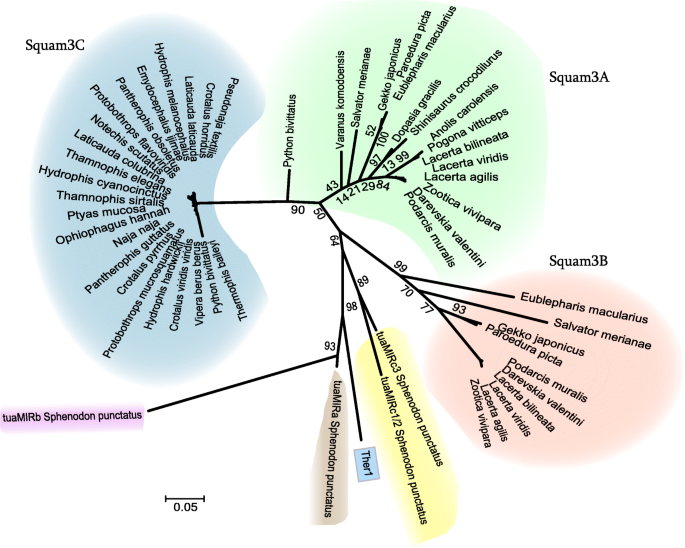
的共识序列DarevskiaSquam3用于搜索有鳞爬行动物的基因组。它存在于所有已测序的基因组中(以及各种未测序的鳞动物物种的GenBank序列中);表S1)。除Squamata外,未发现Squam3(见下文)。对其一致序列的分析揭示了三个主要亚家族,我们称之为Squam3A, Squam3B和Squam3C。
Squam3结构
Squam3是典型的正弦函数[13由trna衍生的头,身体具有中心结构域和与伙伴LINE相匹配的3 '端,以及尾巴是由几个简单的重复组成的一段。一致序列范围为218 ~ 239 nt(不含尾)。对特定tRNA物种没有明确的偏好(这在sine中并不罕见)。
的身体与CORE中心域的片段相似;明显的相似性跨度超过28 nt(图中双下划线)。1)。也有一个相似之处,非常3 '端线的L2分支确定在Darevskia valentini(数据未显示),并且与的L2线的相似性不太明显安乐有害无益(Repbase中的L2-26_ACar和L2-24_ACar)。
Squam3的尾部主要由(TAAA)n或(总部)n;然而,某些物种有(GTT)n(ATT)n,或poly(A)(表)1)。Squam3的目标位点复制率非常低。这在sin中是不寻常的,但并非例外,并且可以指出伴侣LINE内切酶在不同DNA链中的另一种切割模式[13]。
Squam3亚科
sin的基因组拷贝受到随机突变的影响;因此,单核苷酸突变只能用于鉴定高度保守的SINEs亚家族。我们使用扩展插入/删除来区分三个主要的Squam3亚家族,即Squam3A, Squam3B和Squam3C。1)。Squam3B具有特征性的11-nt插入(图中粉红色标记)。1), Squam3C具有特征性的7-nt插入(图中蓝色标记)。1)。Squam3亚家族之间也有细微的差异。此外,还存在亚亚科;其中之一(Squam3B3)已成为两个壁虎科物种的主要变体。
对鳄蜥蜴基因组中squam3相关序列的进一步分析发现了一个类似的sin (tuaMIRa),其插入32 nt(图中苋苋标记)。1)。这一插入恢复了CORE中心域,并使该元素类似于Ther1 (MIR)。值得注意的是,Squam3和tuamirmir相对于Ther1的缺失不同于MIR1_AMi的缺失(图S2A)。TuaMIR sine在LINE-derived区域也有一个8-13-nt的缺失(图中以紫色标记)。1)。此外,另一个具有类似插入的元件(tuaMIRb)缺乏在其他Squam3-和ther1相关的sin中保守的CORE和LINE-derived区域之间的~ 40-nt区域,但由于77-nt的插入(在图中的芒果中标记),它具有更长的L2 LINE-derived区域。1)。最近报道了这些鳄蜥SINE家族的序列[12],但只提到了与MIR (Ther1的前身)的关系以及所有Ther1相关序列的平均散度。
除此之外,Squam3亚家族的不同之处在于尾巴,这在很大程度上是(TAAA)n在3a /C或(CTT)n在Squam3B。平均序列相似度在不同亚家族之间也存在差异,在Squam3B中最高(高达94%),而在Squam3C(~ 63%)和Squam3A中较低(54-63%)。数字2可视化蜥蜴、蛇和鳄蜥基因组中Squam3的多样性。大多数蛇类的Squam3C在种间差异不大;这与Squam3A和Squam3B亚家族的多样性形成对比。tuatara sin显然是一个独立于Ther1的星团。
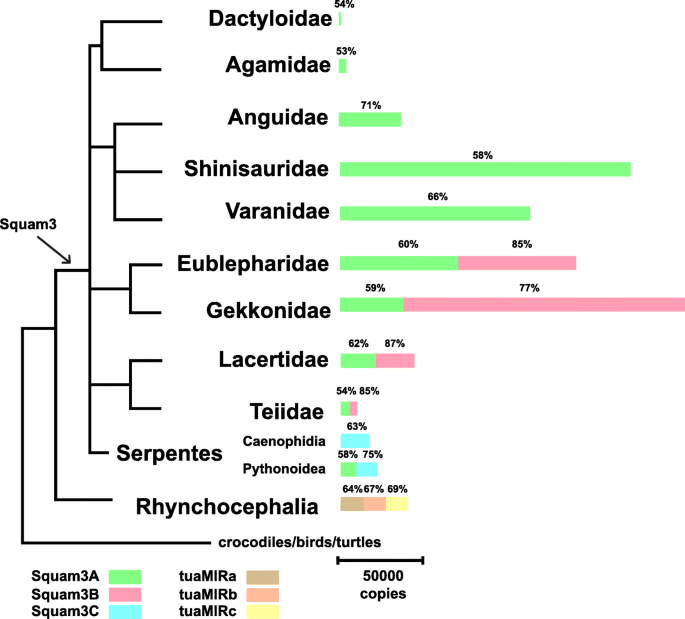
Squam3全长副本的数量变化范围很广:从~ 500英寸安乐有害无益到~ 26万Gekko粳稻(分别占基因组长度的0.005%和2.55%)(图2)3.)。大多数物种Squam3亚科的平均相似性为60-65%,但伊瓜尼亚的Squam3B和Squam3A亚科的相似性为53%。
Squam3在爬行动物基因组中的分布
接下来,我们在鳞片及其邻近分类群的基因组中寻找Squam3亚家族的一致序列。总体而言,38种鳞片动物、鳄蜥、海龟(Trachemys scripta elegans)、鳄鱼(Crocodylus porosus)和鸟(背带吊裤带)进行分析。Squam3在所有有鳞动物中均有发现,但在其他爬行动物和鸟类中均未发现1)。在鳄蜥(斑点楔齿蜥)。当这项工作进行时,Gemmel等人。[12报道了这些正弦函数,所以我们使用他们对蜥蜴正弦函数的命名法。
Gekkota和Lacertoidea (Gekkonidae, Eublepharidae, Lacertidae和Teiidae科)的基因组中Squam3A和Squam3B亚科的比例相似(尽管Squam3A的比例偶尔会低至12%)。除了蟒蛇有43%的Squam3A外,其他蛇类都有Squam3C亚家族。其余鳞片动物(家蝇科、鳗鲡科、蠓科、蠓科和鹬科)均为Squam3A亚科(表2)1)。表中未列出的鳞目动物的NCBI序列分析1在很大程度上证实了这一模式,除了在另外三个蛇科(Elapidae, Lamprophiidae和Viperidae)中发现了一些高度不同的Squam3A序列(表S1)。我们专门在一种高级蛇中搜索了Squam3A (Vipera贝鲁),发现了约330份拷贝。
鳄蜥(Sphenodontidae)有一组与Squam3和Ther1相关的tuaMIR家族。因此,我们专门在Squamata的基因组中寻找这些序列。没有发现tuaMIRb或tuaMIRc,而在所有被分析的鳞状动物基因组中都存在少量的tuaMIRa,从单个全长拷贝到~ 500 (inShinisaurus crocodilurus(表S2)。所有的蛇在相同的基因组位点上都有一个单一的tuaMIRa拷贝(通过非常相似的侧翼区域来判断)。
Squam3和其他类似的CORE sin
我们将Squam3与tuaMIR和其他脊椎动物的含core - sin进行了比较。虽然所有核心的5 ' -序列相似,但图中苋菜标记的特征缺失。1)将所有Squam3和tuaMIRc与其他正弦函数区分开来(图S2C)。
讨论
正弦函数最有趣的一个方面是它们是如何出现和发展的。这项研究为我们提供了一个独特的机会,在非常广泛的分类群中追踪单个SINE家族。Squam3 SINE在有鳞爬行动物(Squamata)中被发现,但在tuatara (Rhynchocephalia)和其他包括鳄鱼,鸟类和海龟在内的谱系中没有发现。我们发现了三个主要的亚家族,以相对较长的插入/删除(Squam3A, Squam3B和Squam3C)为特征。它们在拷贝数和平均序列相似性方面也有所不同,这表明了SINE亚家族的年龄(准确地说,是其扩增的时间),因为SINE基因组拷贝不受选择压力的影响,并且随着时间的推移逐渐积累突变。
Squam3的进化
总的来说,在Squamata祖先的基因组中可能有一小部分(几百个?)不太活跃的Squam3A。在一些谱系中(Shinisauridae和Varanidae), Squam3A扩增非常活跃,没有明显的序列修改(在2008年达到165,000个拷贝)Shinisaurus crocodilurus;Squam3拷贝数仅在Gekko对虾基因组大两倍)。Squam3A扩增在鳗鲡中也很活跃(约35000拷贝)Dopasia股薄肌),但考虑到这种无腿蜥蜴的SINE序列的高平均相似性(71%),它开始得相对较晚。相反,Squam3A在Agamidae中逐渐减少(~ 4500个拷贝,平均相似度为53%)Pogona vitticeps)。最后,Squam3A在Dactyloidae(< 500拷贝)中停止繁殖(和进化)安乐有害无益)。
当Squam3亚家族在鳞科动物谱系中出现时,Squam3A在Gekkota和Lacertoidea中继续扩增(从~ 5000个拷贝到~ 65000个拷贝),但在蛇中没有扩增(除了原始蛇,在蛇类中约9000个拷贝)Python bivittatus)。我们在那里只能找到大约300份拷贝Vipera贝鲁;在其他四个蛇科的非基因组序列中也发现了单个拷贝(表S2)。
在Gekkota和Lacertoidea中,Squam3A减少后,它们的基因组产生了Squam3B亚家族。它可以说是最年轻的Squam3亚家族。令人惊讶的是,Squam3B的平均相似度非常高蝎虎星座agilis(92%)和l .冬青(94%),但只有75%l . bilineata.这表明Squam3B可能在l .冬青和l . agilis但不是l . bilineata属的代表同一属的在壁虎科中,更多产的Squam3B3亚家族在2009年出现了约18万个拷贝Gekko对虾这是所有Squam3亚家族中数量最多的)。由于某些原因,Squam3A和Squam3B在Teiidae (出来merianae),但Squam3B的扩增时间仍晚于Squam3A。
Squam3C亚家族仅限于蛇;此外,它是大多数蛇中唯一的主要亚科。Squam3A数量可能存在于所有鳞片中,但在大多数蛇中不繁殖。相反,在高级蛇类(Caenophidia)中,Squam3C的活跃时间稍晚或在同一时期(Squam3C的平均相似性为61-65%,而在Squam3A中为51-71%)。这种模式对Python bivittatus其中,Squam3A的扩增量紧随其后,Squam3C的扩增量平均相似度分别为58%和75%。
Squam3的起源
我们非常兴奋地发现了鳄蜥进化中所谓的“缺失环节”。的基因组斑点楔齿蜥除了Squam3相对于其中两个(tuaMIRa和tuaMIRb)缺失32 nt外,在最左边~ 120 nt处有3个与Squam3相似的SINE家族。因此,在两个tuaMIR SINEs中删除了一个较大的CORE片段。另一种蜥蜴sin (tuamirmir)有这种缺失,并且在该区域内与Squam3相似(但在头部和线派生区域不同)。在哺乳动物、爬行动物、鸟类甚至腔棘鱼的共同祖先中活跃的Ther1的祖先似乎是合理的[9,51]在鳞翅目祖先的CORE区域内获得了32-nt的缺失,并且在相关的sin中也存在相同的区域(图2)。S2B和S2C)。这个前体sin在tuatara中产生tuamirr,在Squamata中产生Squam3。
结论
我们在所有(38个)有鳞爬行动物(Squamata)的基因组测序中发现了一个新的SINE Squam3。尽管蜥蜴和蛇的基因组数据不断增加,但这种相当多产的SINE以前没有报道过。sin家族和亚家族的进化动力学是模糊的,与基因组的分化有关。这项研究在理解正弦函数如何出现和衰退方面迈出了一步。我们鉴定并描述了Squam3亚科,并直接比较了各种主要鳞类动物类群中Squam3亚科的结构特征和拷贝数,并与相关的蜥蜴sin家族进行了比较。这项研究深入了解了SINE家族是如何出现和发展的。
方法
大多数基因组数据是从NCBI基因组(https://www.ncbi.nlm.nih.gov/genome)除了安乐有害无益,虽然murali(运用,https://www.ensembl.org),Dopasia股薄肌,Shinisaurus crocodilurus(diArkhttps://www.diark.org/diark),Darevskia valentini(17]。我们使用了基因组序列蝎虎星座agilis和Thamnophis线虫获得脊椎动物基因组计划的许可。表中未列出的鳞目动物的个体序列1也从NCBI (https://www.ncbi.nlm.nih.gov/taxonomy/advanced)。如果出版物或动物基因组大小数据库中没有关于基因组大小的数据[52],它被计算为最接近物种的平均值。
我们使用基于Smith-Waterman搜索的自定义Perl脚本来查找具有至少65%同一性和90%长度与共识重叠的正弦序列的基因组副本。在确定所有Squam3家族后,依次使用其共识序列耗尽基因组库,并将所有命中组合进行进一步分析。
生成多个序列比对MAFFT(53],由GeneDoc(54]。亚家族是手动识别的,必要时在更大的样本中进行分析。我们只考虑了充足的亚家族(≥1%的全长拷贝总数)。在爬行动物/鸟类基因组中搜索tuaMIRa序列,首先对所有与共识序列至少65%相似的拷贝进行初步鉴定,然后对可能包含将其与tuaMIRa序列分离的特定突变的候选拷贝进行人工亚采样和重新排列。平均相似性是确定100个随机选择的序列(或所有可用的,如果更少)使用alistat艾迪·S.,剑桥,[55])。利用MEGA软件构建了一个带有1000次自举复制和“部分删除”选项的邻居连接树。
数据和材料的可用性
生成的数据可在手稿支持文件中找到。Squam3正弦序列库,以及随机正弦序列的多个比对,可根据要求为每个物种提供。
参考文献
Vassetzky NS, Kramerov DA。sinbase:一个用于正弦分析的数据库和工具。核酸学报,2013;41:D83-9[引自2014年6月4日]。可以从:http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=3531059&tool=pmcentrez&rendertype=abstract.
克拉默洛夫DA,瓦塞茨基NS。真核生物基因组中正弦序列的起源和演化。遗传(Edinb)。2011; 107:487-95来源:http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=21673742.
谢洛安,高桥K,冈田N.物种形成的正弦:用反转录子追踪谱系。生态学报。2004;19:545-53http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=16701320.
李建军,李建军,李建军,等。雀形目鸟类早期进化过程中sin逆转录子的从头出现。移动DNA。2017; 8:1-8。
Crawford NG, Parham JF, Sellas AB, Faircloth BC, Glenn TC, Papenfuss TJ,等。龟类的系统发育分析。生物化学学报,2015;33(3):557 - 557。可以从:https://doi.org/10.1016/j.ympev.2014.10.021.
Endoh, Nagahashi S, Okada N.乌龟基因组中一个高度重复和可转录的序列可能是一个逆转录子。生物化学杂志。1990;189:25-31http://www.ncbi.nlm.nih.gov/pubmed/1691979.
nishhara H, Smit AF, Okada N.功能性非编码序列在哺乳动物基因组中的应用。Genome Res. 2006; 16:864-74可从:http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=16717141.
Smit AF, Riggs AD。MIRs是经典的trna衍生的正弦,在哺乳动物辐射之前被放大。核酸研究。1995;23:98-102来源:http://www.ncbi.nlm.nih.gov/pubmed/7870595.
李建军,李建军,李建军,等。中华生物医学杂志。2000;28 (3):357 - 357http://www.ncbi.nlm.nih.gov:80/entrez/query.fcgi?cmd=Retrieve&db=PubMed&list_uids=10772856&dopt=Abstract.
Shedlock AM, Botka CW, Zhao S, Shetty J, Zhang T, Liu JS,等。非鸟类爬行动物的系统基因组学和祖先羊膜动物基因组的结构。科学通报,2007;24(4):397 - 397。
夏德罗克。爬行动物cr1line多样性的系统基因组学研究。系统生物学。2006;55:902-11。
germell NJ, Rutherford K, Prost S, Tollis M, Winter D, Macey JR,等。蜥蜴基因组揭示了羊膜动物进化的古代特征。大自然。2020;584:403-9。
克拉默洛夫DA,瓦塞茨基NS。正弦。Wiley交叉Rev RNA。2011; 2:772-86[引自2014年6月4日]。可以从:http://www.ncbi.nlm.nih.gov/pubmed/21976282.
刘毅,周强,王毅,罗磊,杨军,杨玲,等。Gekko对虾基因组揭示了粘附趾垫和尾巴再生的进化。6自然出版集团。2015。[引自2020年11月29日]。可以从:https://pubmed.ncbi.nlm.nih.gov/26598231/.
原阳,竹内M, Kageyama Y, Tatsumi K, Hibi M, Kiyonari H,等。马达加斯加地壁虎基因组分析的特征是重复基因的不对称命运。生物医学工程学报,2018;16(1):1 - 19。
熊志,李峰,李强,周磊,甘布尔特,郑杰,等。绘制豹纹壁虎的基因组,Eublepharis macularius.GigaScience。5可从:https://doi.org/10.1186/s13742-016-0151-4.
Darevskia (ID 327916) - BioProject - NCBI [Internet]。[引自2020年12月9日]。可以从:https://www.ncbi.nlm.nih.gov/bioproject/327916
GenomeArk -蝎虎星座agilis(互联网)。[引自2020年12月2日]。可以从:https://vgp.github.io/genomeark/Lacerta_agilis/
Kolora SRR, Weigert A, Saffari A, Kehr S, Walter Costa MB, Spröer C,等。近缘哺乳动物基因组的分化进化蝎虎星座冬青和l . bilineata,以及对物种形成的影响。科学学报。2019;8:22 NLM (Medline)。[引自2020年11月29日]。可以从:http://orcid.org/0000-0001-7839-735X.
刘建军,刘建军,刘建军,等。翼雀素和类胡萝卜素基因的调控变化是壁虎平衡颜色多态性的基础。科学通报,2019;16(6):563 - 542[引自2020年12月2日]。美国国家科学院。可以从:https://www.pnas.org/content/116/12/5633.
Yurchenko AA, Recknagel H, Elmer KR.普通蜥蜴的染色体水平组装(一只)基因组。生物学报。2020;12(1):53 - 60。
Roscito JG, Sameith K, Pippel M, Francoijs KJ, Winkler S, Dahl A,等。泰古蜥蜴的基因组出来merianae结合Illumina, PacBio和光学制图数据,生成高度连续的组件。牛津大学出版社。2018;7:1-13。
Ullate-Agote A, Milinkovitch MC, Tzika AC.玉米蛇的基因组序列(Pantherophis guttatus),是EvoDevo研究鳞片的宝贵资源。中国生物医学工程学报,2014;38(1):481 - 481。
Pantherophis obsoletus(ID 88953) -基因组- NCBI[互联网]。[引自2020年12月2日]。可以从:https://www.ncbi.nlm.nih.gov/genome/88953?genome_assembly_id=889057
Ptyas粘膜(ID 44753) -基因组- NCBI[互联网]。[引自2020年12月2日]。可以从:https://www.ncbi.nlm.nih.gov/genome/44753?genome_assembly_id=884075
GenomeArk -Thamnophis线虫(互联网)。[引自2020年12月2日]。可以从:https://vgp.github.io/genomeark/Thamnophis_elegans/
Thamnophis sirtalis(ID 16688) -基因组- NCBI[互联网]。[引自2020年12月2日]。可以从:https://www.ncbi.nlm.nih.gov/genome/16688?genome_assembly_id=245767
李建军,高永远,谢磊,邓超,石平,关丽丽,等。异温蛇高海拔适应的比较基因组研究。科学通报,2018;33(5):444 - 444。
Kishida T, Go Y, Tatsumoto S, Tatsumi K, Kuraku S, Toda M.海蛇嗅觉丧失为羊膜动物的水生适应提供了新的视角。生物工程学报(英文版);2019;28。
Hydrophis cyanocinctus(ID 75161) -基因组- NCBI[互联网]。[引自2020年12月2日]。可以从:https://www.ncbi.nlm.nih.gov/genome/75161?genome_assembly_id=437861
Hydrophis hardwickii(ID 75162) -基因组- NCBI[互联网]。[引自2020年12月2日]。可以从:https://www.ncbi.nlm.nih.gov/genome/75162?genome_assembly_id=437862
Suryamohan K, Krishnankutty SP, Guillory J, Jevit M, Schröder MS, Wu M,等。印度眼镜蛇参考基因组和转录组可以全面识别毒液毒素。Nat Genet. 2020; 52:106-17 Springer US。可以从:https://doi.org/10.1038/s41588-019-0559-8.
Notechis scutatus(ID 14408) -基因组- NCBI[互联网]。[引自2020年12月2日]。可以从:https://www.ncbi.nlm.nih.gov/genome/14408?genome_assembly_id=408294
Vonk FJ, Casewell NR, Henkel CV, Heimberg AM, Jansen HJ, McCleary RJR,等。眼镜王蛇基因组揭示了蛇毒系统中动态的基因进化和适应。科学通报,2013;33(4):551 - 556。
Pseudonaja textilis(ID 72610) -基因组- NCBI[互联网]。[引自2020年12月2日]。可以从:https://www.ncbi.nlm.nih.gov/genome/72610?genome_assembly_id=408420
Castoe TA, De Koning APJ, Hall KT, Card DC, Schield DR, Fujita MK等。勘误:缅甸蟒蛇基因组揭示了蛇极端适应的分子基础(美国国家科学院院刊(2013)110,51,(20645-20650)DOI: 10.1073/pnas。1314475110)。中国科学d辑,2014;33(1):1 - 4。
Crotalus澳洲(ID 16679) -基因组- NCBI[互联网]。[引自2020年12月2日]。可以从:https://www.ncbi.nlm.nih.gov/genome/16679?genome_assembly_id=274149
张建军,张建军,张建军,张建军,张建军,等。蛇类内源性肝炎病毒、病毒性肝炎病毒和圆环病毒的研究进展。中国生物医学工程学报(英文版);2014;01 - 01。
绿豆,绿豆(id71654) -基因组- NCBI[互联网]。[引自2020年12月2日]。可以从:https://www.ncbi.nlm.nih.gov/genome/71654?genome_assembly_id=434976
Shibata H, Chijiwa T, Oda-Ueda N, Nakamura H, Yamaguchi K, hatori S,等。哈布基因组揭示了毒液蛋白基因的加速进化。科学通报,2018;8:1-11。
张建军,张建军,张建军,张建军。pitviper的种群基因组分析揭示了毒液化学背后的微观进化力量。中国生物医学工程学报,2017;9(2):444 - 444。
Vipera + us + us(ID 14467) -基因组- NCBI[互联网]。[引自2020年12月2日]。可以从:https://www.ncbi.nlm.nih.gov/genome/14467?genome_assembly_id=214193
高军,李强,王忠,周勇,Martelli P,李峰,等。濒危中华鳄蜥蜴基因组的测序、重新组装和注释Shinisaurus crocodilurus.Gigascience。2017;6:1-6。
宋斌,程生,孙勇,钟旭,金杰,关睿,等。一份无腿蜥蜴的基因组草图,Ophisaurus股薄肌.Gigascience。2015;4:15-7。
Lind AL, Lai YYY, Mostovoy Y, Holloway AK, Iannucci A, Mak ACY,等。科莫多龙的基因组揭示了巨蜥心血管和化学感觉系统的适应性。中华生态学报。2019;3:1241-52。可以从:https://doi.org/10.1038/s41559-019-0945-8.
乔治A,李强,连军,O 'Meally D, Deakin J,王忠,等。澳大利亚龙蜥蜴基因组的高覆盖率测序和注释组装Pogona vitticeps.GigaScience。2015;4可从:https://doi.org/10.1186/s13742-015-0085-2.
Alföldi J, Di Palma F, Grabherr M, Williams C, Kong L, Mauceli E,等。绿色变色蜥蜴的基因组以及与鸟类和哺乳动物的比较分析。大自然。2011;477:587 - 91。
Brian Simison W, Parham JF, Papenfuss TJ, Lam AW, Henderson JB。红耳滑龟的染色体水平参考基因组注释(Trachemys scripta elegans)。生物学报。2020;12:456-62。
王晓明,王晓明,王晓明,等。高质量的咸水鳄参考基因组组合Crocodylus porosus揭示鳄鱼科的选择模式。基因组生物进化。2019;12:36 . 35 - 46牛津大学出版社。[引自2020年12月2日]。可以从:https://pubmed.ncbi.nlm.nih.gov/31821505/.
Hillier LW, Miller W, Birney E, Warren W, Hardison RC, Ponting CP,等。鸡基因组的序列和比较分析为研究脊椎动物进化提供了独特的视角。大自然。2004;432:695 - 716。
Nikaido M, Noguchi H, Nishihara H,丰田A, Suzuki Y, Kajitani R,等。腔棘鱼基因组揭示了从水到陆地进化过渡的特征。基因工程学报,2013;23:1740-8。
动物基因组大小数据库。2020。可以从:http://www.genomesize.com
Yamada KD, Tomii K, Katoh K. MAFFT序列比对程序在大数据中的应用——链式导向树有用性的再检验。生物信息学。2016;32:3246-51。
Nicholas KB, Nicholas HBJ。GeneDoc:遗传变异的分析和可视化1997。可以从:http://www.nrbsc.org/gfx/genedoc/index.html
SQUID - C序列分析函数库;2005.
致谢
我们感谢Dmitri Kramerov博士对手稿的批判性阅读。
资金
本研究由俄罗斯科学基金会(RSF)项目No. 19-14-00083资助。
作者信息
作者及单位
贡献
NSV和APR,概念化;所有,基因组数据分析;NSV和SAK,研究设计和稿件准备;APR和VIK,监督;APR,项目管理和资金获取。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们没有竞争利益。
额外的信息
出版商的注意
伟德体育在线施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。
补充信息
附加文件1:图S1。
物种特异性Squam3序列的比对。绿色,Squam3A;红色,Squam3B;蓝色- Suam3C。物种名称为:Squam3EmA;Eublepharis macularius;Squam3GjA,Gekko对虾;Squam3PpA,Paroedura picta;Squam3Vk,Varanus komodoensis;Squam3Ch,Crotalus澳洲;Squam3Pt,Pseudonaja textilis;Squam3Cp,Crotalus皮拉斯;Squam3Pg,Pantherophis guttatus;Squam3Nn,眼镜蛇眼镜蛇;Squam3Oh,眼镜;Squam3Pmc,Protobothrops mucrosquamatus;Squam3Ts,Thamnophis sirtalis;Squam3Hc,Hydrophis cyanocinctus;Squam3Te,Thamnophis线虫;Squam3Pf,Protobothrops flavoviridis;Squam3Po,Pantherophis obsoletus;Squam3Cv,Crotalus冬青;Squam3Hh,Hydrophis hardwickii;Squam3Tb,Thermophis baileyi;Squam3Vb,Vipera贝鲁;Squam3EjEmydocephalus ijimae;Squam3Hm,Hydrophis melanocephalus;Squam3Ll,Laticauda laticaudata;Squam3Lc,Laticauda colubrina;Squam3Ns,Notechis scutatus;Squam3Pr,Protobothrops mucrosquamatus;Squam3PbC,Python bivittatus;Squam3DvB,Darevskia valentini;Squam3LbB,蝎虎星座bilineata;Squam3LaB,蝎虎星座agilis;Squam3LvB,蝎虎星座冬青;Squam3PmB,虽然murali;Squam3ZvB,Zootica只;Squam3GjB,Gekko对虾;Squam3PpB和Squam3PpB3,Paroedura picta;Squam3GjB3,Gekko对虾;Squam3EmB,Eublepharis macularius;Squam3SmB,出来merianae.
附加文件2:图S2。一个。
Ther1/MIR亚家族的比对。B。Squam3、tuaMIR和其他CORE sine全长一致序列与tRNA-和l2衍生区域的比较相应的区域标示在序列上方。C。脊椎动物CORE sine的CORE结构域。在苋菜中标记出特有的Squam3缺失(如图所示)。1)。
附加文件3:图S3。
若干L2 line的tuaMIRb和Ther1及3′端序列的line衍生区域比对。圆括号中给出了原点和总长度。
附加文件4:表S1。
在表中未列出的有鳞动物的NCBI序列中发现的Squam3拷贝1.
附加文件5:表S2。
研究动物基因组中tuaMIR亚家族的分布。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接获得版权所有者的许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非在数据的信用额度中另有说明。
关于本文
引用本文
Vassetzky, n.s., Kosushkin, s.a., Korchagin, V.I.et al。新的ther1衍生的有鳞爬行动物的SINE Squam3。移动DNA12, 10(2021)。https://doi.org/10.1186/s13100-021-00238-y
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s13100-021-00238-y
关键字
- 正弦
- 反转位子活动
- 有鳞目
- 爬虫类
- 进化