摘要
背景
II类内含子是可移动的逆转录元件,能够侵入DNA中的新位点。它们是自我剪接的核酶,与内含子编码的蛋白质结合形成剪接后靶向DNA的核糖核蛋白。这些分子可以入侵DNA位点,具体来说,通过一个被称为逆转录的过程,或者可以通过逆转录转位入侵异位位点。特别是,逆转录转位可以受到环境和细胞因素的强烈影响。
结果
为了研究影响逆转录转位的宿主因素,我们使用IS进行了随机插入突变S1转座子来生成超过1000个突变体的库Lactococcus lactis它是Ll的原生宿主。LtrB族II内含子。通过筛选这个文库,我们鉴定出了92个逆转录转位频率增加的突变体(RTP-ups)。我们发现氨基酸转运和代谢的突变倾向于增加逆转录转位频率。我们进一步探索了这些RTP-up突变体的一个子集,其中最引人注目的是核糖体RNA甲基转移酶的突变体rlmH,显示出逆转录转位频率可重复增加20倍。体外和体内实验结果表明,核糖体在体外和体内均有一定的作用rlmH突变体在m3Ψ修饰中存在缺陷,与内含子RNA的结合减少。
结论
综上所述,我们的结果加强了原生宿主生物在调节II组内含子逆转录转位中的重要性。特别是,证据来自rlmH突变提示核糖体修饰在限制逆转录转位猖獗方面的作用。
背景
II组内含子是在古细菌、细菌和细胞器基因组中发现的自我剪接的可移动遗传元件,被认为是真核生物剪接染色体内含子和反转录转座子的祖先[1,2,3.,4].具有催化活性的内含子RNA与其内含子编码蛋白(IEP)一起形成核糖核蛋白(RNP)复合体,其中IEP协助内含子的剪接和迁移[2,5].剪接通过两个可逆的酯交换反应发生,并导致II族内含子套索和连接外显子的形成[2].剪接后,内含子可以在IEP的帮助下反向剪接到目标DNA中,要么通过一个称为逆转录的过程,即内含子专门入侵目标DNA的归位位点,要么通过一个效率较低的称为逆转录转位(RTP)的过程,即它入侵异位位点,通常在染色体的复制叉处[1,2,3.,6,7,8].
RNPs中的II族内含子是高度结构化的RNA分子。RNA结构对活性至关重要,由一个高度保守的折叠组成,包含六个结构域(DI-DVI) [2,9].域I (DI)包含外显子结合位点(EBS),与外显子内含子结合位点(IBS)相互作用,实现靶剪接和反剪接[2].域IV (DIV)包含编码IEP的开放阅读框(ORF),以及IEP翻译所需的核糖体结合位点(RBS),以及剪接和反向剪接所需的IEP结合位点。翻译后,IEP与DIV结合并阻断RBS,从而抑制IEP的进一步翻译[10].然后,拼接并完全形成的RNP可以作为一种能够入侵DNA的逆转录因子。
逆转录回巢和逆转录转位都已使用Ll仔细解剖。LtrB II族内含子Lactococcus lactis在模式生物中,大肠杆菌,揭示了各种宿主因素在抑制和促进逆行性方面的重要性[6,11,12,13,14,15,16].例如,一项研究揭示RNase E通过降解内含子RNA来下调II组内含子的逆转录性[15],而进一步的研究表明核糖体如何与内含子结合并阻断这种效应[16].然而,这些影响可以根据宿主背景而改变,甚至可以在不同的宿主生物中通过不同的机制途径进行逆转录转位[13].在它的原生宿主中,l . lactis,即Ll。LtrB内含子主要通过复制叉(Okazaki片段作为反转录引物)进行逆转录转位到单链DNA,主要通过内切酶无关途径[6,7].然而,在大肠杆菌内含子主要通过内切酶依赖通路进行逆转录转位,这与位点特异性逆转录有许多相似之处[13].综上所述,这些研究表明逆转录转位对宿主环境很敏感,宿主基因在调节逆转录转位中起着重要作用。
由于不同宿主之间的逆转录转位差异,人们已经开始努力阐明Ll。LtrB内含子及其原生宿主,Lactococcus lactis.噢。的LtrB内含子l . lactis在偶联质粒pRS01上发现,并能够通过偶联[17,18,19,20.].由于逆转录转位是一种低频事件,因此通常使用遗传报告内含子供体质粒pLNRK-RIG进行研究。1a和Table1).该质粒含有一个逆转录转位指示基因(RIG),可以根据卡那霉素耐药的获得来量化逆转录和逆转录转位频率[6,21(图。1b).使用该报告器,已表明Ll。LtrB内含子本身存在,不仅能切割自身的pRS01质粒DNA启动偶联,还能切割脱靶DNA位点,促进逆转录转位[21].
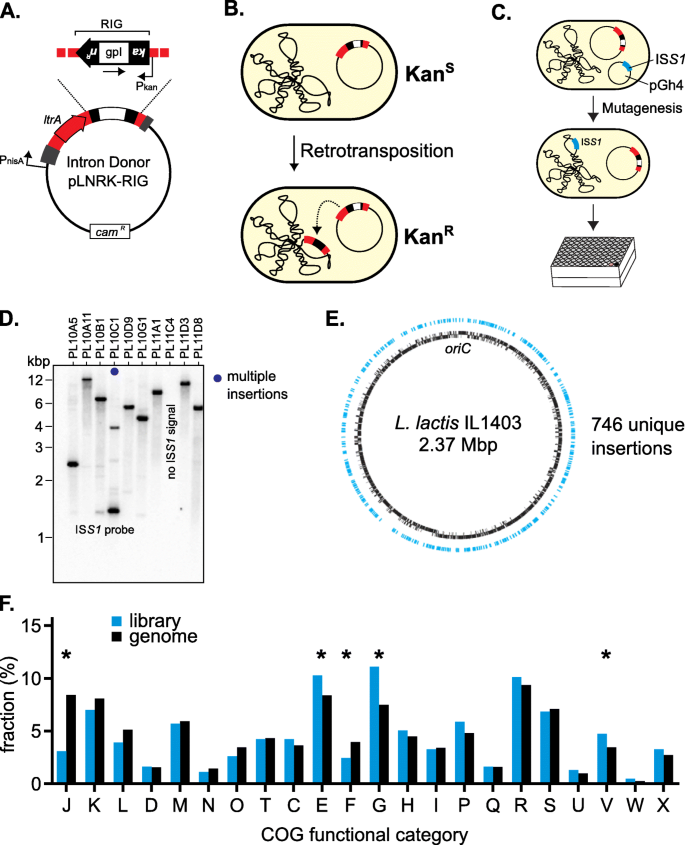
突变库的生成和描述。一个。内含子供体质粒pLNRK-RIG。逆转录转位指示基因(RIG)位于Ll。LtrB II族内含子(红色),受nisin启动子(P原子力安全保安院).RIG含有一个卡那霉素抗性基因(黑色框箭头)和它自己的启动子(P菅直人与转录方向相反),被I族内含子(gpI,白框)中断。噢。LtrB内含子两侧有部分原生外显子,内含子编码蛋白LtrA。pLNRK主链含有一个氯霉素抗性基因(凸轮R) [21].b。逆转录转位试验通过获得卡那霉素耐药来识别逆转录转位事件(KanR)在I组内含子被剪接后。c。是S1-介导突变示意图。含有内含子供体质粒pLNRK-RIG的细胞被pGh4:IS转化S1并对红霉素耐药进行了筛选。培养包含两种质粒的培养物以允许IS的转位S1转座子,然后进行温度移位去除温度敏感的pGh4:ISS1筛选红霉素耐药性的丧失,并与各种对照一起存放在96孔板中。详情请参见附加文件1:图S1d。IS库的Southern blot分析S1拷贝数。50个IS的随机子集S1变种人被用来研究ISS1使用Southern blotting插入。深蓝色圆点表示该菌株有多次插入。有关所有数据,请参阅附加文件1:图S2。e。IS的分布S1周围的突变库插入(蓝色)l . lactisIL1403染色体(黑色)。对文库进行测序和分析,鉴定出746种独特的ISS1插入。f。IS功能覆盖的比较S1突变库(蓝色)对抗l . lactis基因组(黑色),基于COG类别的相对丰度。星号表示有显著差异(p< 0.05)为超几何检验结果。所有COG类别的键都在附加文件中1表S3
为了进一步研究Ll。LtrB内含子和宿主功能,我们在内含子的原生宿主中创建并筛选突变库l . lactis.在相互作用网络中进一步表征了相对于野生型(RTP-ups)显示逆转录转位频率上调的突变体。然后,从不同功能类别中选择12个突变体进行进一步分析。从这些突变体中rlmH::S1突变体,核糖体RNA (rRNA)甲基化缺陷,始终表现出非常高的逆转录转位频率,尽管对位点特异性逆转录没有影响。核糖体来自rlmH::S1在体外和体内,突变体结合内含子RNA的能力都存在缺陷,这表明正常的核糖体-内含子相互作用降低了逆转录转位的频率,这使我们假设核糖体的功能是保护基因组免受原生宿主中II族内含子逆转录转位的侵袭。
结果
突变库的构建和初始描述
鉴定影响Ll的宿主基因。LtrB组II型内含子逆转录转座效率高,在转座插入中产生突变体l . lactis携带内含子供体质粒的IL1403株(pLNRK-RIG,图;1a)后卫+携带插入元件IS的宿主质粒S1(pGh4:S1) [23(图。1c和表1;额外的文件1:图S1)。共有1006人l . lactisIL1403是S1获得突变株,排列到11个96孔板,PL1-PL11 (PL -板)。Southern blot分析50 ISS1从库中随机选择的菌株证实了IS的存在S1它们在细菌染色体中的随机分布,其中20%(45个菌株中有9个)含有一个以上的ISS1插入(图。1d;额外的文件1:图S2A和表S1),这与最初报道的多拷贝转位的发生情况一致[23].我们还建立了IS的稳定性和垂直传输S1通过追踪它们的12代细菌(附加文件1:图S2B)。总的来说,这些结果表明我们建立了一个稳定的突变体库,主要由单个插入组成。
的库的进一步特征l . lactisIL1403是S1我们使用靶向高通量测序鉴定IS突变株S1插入站点(附加文件1:图S3)。为了尽量减少测序反应的数量,我们利用启发式直三策略进行正交池化和映射[27](附加文件1:图S3)。经过测序、初始数据处理和筛选,我们鉴定出757个ISS1插入位点,其中746个是唯一映射在l . lactisIL1403基因组图1e;额外的文件2).IS的比较S1尽管IS的上游和下游都有15和16个核苷酸碱基,但侧翼区域的序列之间没有显著的一致性S1插入位点发现腺嘌呤和胸腺嘧啶的频率比预期的要高一些(附加文件)1:图S4)。然而,伊斯兰国在很大程度上是随机的S1在l . lactisIL1403证实先前的报告[23,28].
接下来,我们想要确定突变库中是否存在任何功能偏差。为了在更大范围内比较基因功能,我们进行了同源组簇(COG)分析,该分析根据蛋白质功能将基因聚类为组[29].我们首先比较了IS的COG分布S1的突变库l . lactisIL1403(无花果。1f;额外的文件1:表S2),并使用超几何检验进行统计比较。图书馆中有几个类别得到了极大的丰富(p< 0.05),包括E, G和V. COG类E和G分别代表氨基酸和碳水化合物的运输和代谢相关的功能(附加文件1:表S2-S3)。COG类V由参与宿主防御机制的蛋白质组成,如限制修饰系统或药物外排泵(附加文件)1:表S3)。我们观察到两个类别在文库中明显代表性不足(p < 0.05): F和J(图。1f;额外的文件1:表S3)。F类由具有与核苷酸转运和代谢相关功能的蛋白质组成,而J类中的功能与翻译、核糖体结构和核糖体生物发生有关(附加文件)1:表S3)。尽管这些统计上的显著差异,大多数功能类别在我们的突变文库和基因组背景之间有非常相似的表示l . lactisIL1403。
识别内含子反转置频率升高的突变体
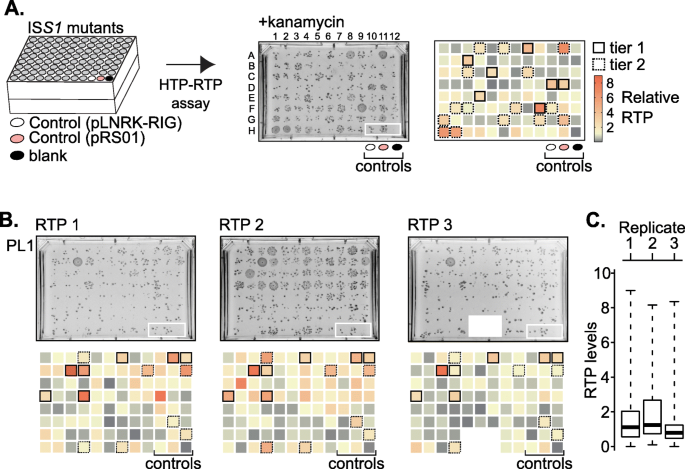
为了鉴定内含子逆转录活性改变的潜在宿主突变体,逆转录转位(RTP)检测在96孔高通量板中进行,然后在含有160 μg/mL卡那霉素的GM17矩形琼脂板上进行斑点电泳(图2)。2一个;额外的文件1:图S5)。同时对PL1到PL11板进行http - rtp测定,并使用液体处理剂独立重复三次,以确保斑点的均匀性。虽然一些在http - rtp检测板上产生相对较高数量菌落的突变体可以用眼睛识别,但我们使用sgatols进行图像分析和斑点大小量化[30.(图。2b;额外的文件1:图S5和表S4)。对于每个平板,用SGAtools计算斑点大小分数,并归一化为来自同一平板的野生型pLNRK-RIG菌株产生的斑点大小分数。因此,从反转位事件的板选择归一化菌落大小分数代表相对内含子反转位水平。
RTP-up突变体鉴定。一个。高通量RTP (http -RTP)检测。都是S1培养突变株和对照株,诱导并在卡那霉素上电镀,以鉴定具有假定的RTP-up表型的突变株。相对于野生型pLNRK-RIG对照(白色椭圆形),对选择性介质上的光斑大小进行量化,并以双梯度热图显示(min, 0;马克斯,9;基线,1).其他对照包括与pRS01相同的菌株,表型为“向上”[21](粉红色椭圆形)和一个只包含介质的空白井(黑色椭圆形)。根据每个板内逆转录转位频率的箱线图分析,将突变体分级(附加文件)1:图S5和表S4)。黑色边框表示第一层的突变体,而虚线表示第二层。b。钢板1 (PL1)的三种独立的http -RTP测定结果(RTP 1 -RTP 3)。每个板块的热图都显示出来了。RTP3板的一个污染区域被排除在分析之外(PL1 RTP3中的白色区域)。所有板的http -RTP检测结果在附加文件中1:图S5和表S4。c。PL1的箱线图分析。RTP水平报告相对于野生型。方框和须状图勾勒出四分位数,每个方框内的粗体线作为中位数。所有箱线图都在附加文件中1:图S5
接下来,我们使用箱线图分析(图。2C),这使我们能够研究数据集之间的相对内含子反转位值分布,并选择始终显示较高相对水平的突变体。我们将自己限制在逆转录转位频率增加的突变体(RTP-up突变体),因为那些产生降低频率的突变体产生不一致的表型。突变体根据其逆转录转位表型的强度和一致性被分为几层。基于箱线图分析,第一层突变体被定义为在所有三种http - rtp检测中第三个四分位数(Q3)和第四个四分位数(Q4,最大值)之间具有相对逆转录转位水平的突变体;如果突变体的逆转录转位水平在两次HTP-RTP试验中的Q3和Q4之间,但在中位数(Q2)和Q3之间一次,则突变体处于第二层(图2)。2;额外的文件1:图S5和表S4)。来自第一和第二层的突变体被指定为RTP-up突变体,并被选择用于进一步分析。
共鉴定出155株l . lactisIL1403是S1显示逆转录转位水平持续升高的突变株。这些RTP-up突变体被检查是否存在多拷贝ISS1使用如上所述的Southern blotting,以及具有多个IS的突变体S1每个染色体都被从列表中清除(附加文件1:图S6和表S4)。的是S1通过反PCR结合个体测序两阶段过程验证插入,然后在插入位点两侧进行引物验证PCR(附加文件)1:图S7-S8和表S5)。任何不能通过该过程验证的突变体都从后续调查中被省略,留下92个突变体进行进一步分析(附加文件)3.).
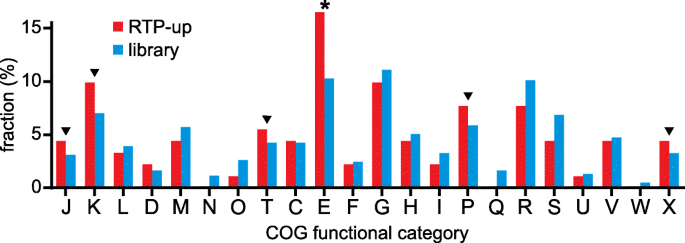
为了鉴定验证的RTP-up突变体中的功能富集,我们比较了RTP-up突变体与整个IS的COG类别分布S1突变体库。我们观察到RTP-up突变体在氨基酸转运和代谢相关基因(COG类E)中显著且具有统计学意义的富集(图5)。3.;额外的文件1:表S2-S3)。其他几个COG类别的RTP-up突变体的相对丰度升高,尽管没有统计学意义,包括与翻译(J)、转录(K)、信号转导(T)、无机离子转运和代谢(P)和移动遗传元件(X)相关的类别(图)。3.;额外的文件1:表S2-S3)。
RTP-up突变体的功能分类比较。RTP-up突变体与突变体库的COG分布比较。星号表示统计上有显著差异(p< 0.05)使用超几何检验,而三角形指向RTP-up突变体富集的类别,但没有统计学差异。在COG E类中有显著差异,对应于氨基酸的转运和代谢。RTP-ups的富集也在几个类别中观察到:J,翻译,核糖体结构和生物发生;K,转录;T,信号转导;P,无机离子运输和代谢;X是可移动遗传元件。所有COG类别的键都在附加文件中1表S3
为了探索一种可能的逆转录转位主调控因子,我们使用Cytoscape和STRING数据库生成了RTP-up突变体的蛋白质相互作用网络和突变体库背景[31,32].我们提交了IS中突变体的所有蛋白质序列S1并生成了一个由503个节点组成的网络,代表独特的蛋白质序列。在该网络中,52个节点没有交互。我们将网络按COG类别分组,并将RTP-up突变体涂成红色,使每个COG类别中RTP-up突变体的比例可视化(附加文件1:图S9)。尽管在STRING数据库中发现了许多相互作用,但没有从网络中出现连接许多RTP-up突变体的主调控因子,可以解释RTP-up表型。
选择供进一步分析的突变体
接下来,我们将重点放在92个突变体中的12个突变体上,这些突变体在http - rtp检测中显示逆转录转位升高,并具有特别有趣的基因功能(表2)2).例如,我们选择现货::S1因为之前的研究表明relA而且现货对逆转录转位有影响LtrB内含子大肠杆菌[33].我们选择hisH::S1而且rpoE::S1由于它们的基因功能与中枢代谢有关[34,35],分别代表COG类别E和K,相对于文库背景,这两类突变体在RTP-up突变体中均被过度代表(图。3.).的coiA基因,为能力蛋白编码[36],因为它在IS中出现了三次S1的不同位置有三个不同的插入点的突变体库coiA基因,RTP频率不同。我们还选择了一个插入到复制的起点,oriC,因为逆转录转位之前已被证明与DNA复制有关[6,7].类似地,我们选择矩形::S1因为它在DNA修复中的功能[37].一组特别感兴趣的突变体是那些与翻译相关的突变体(COG类J)。尽管COG类J在文库中代表性不足(图2)。1 f),该类别中的四个突变体符合http - rtp检测中持续高结果的标准:ybaK::S1,rsmB::S1,rsmE::S1,rlmH::S1.的ybaK氨酰基trna脱酰基酶的基因编码[38],而其他三种编码核糖体RNA甲基化酶[39)(表2).
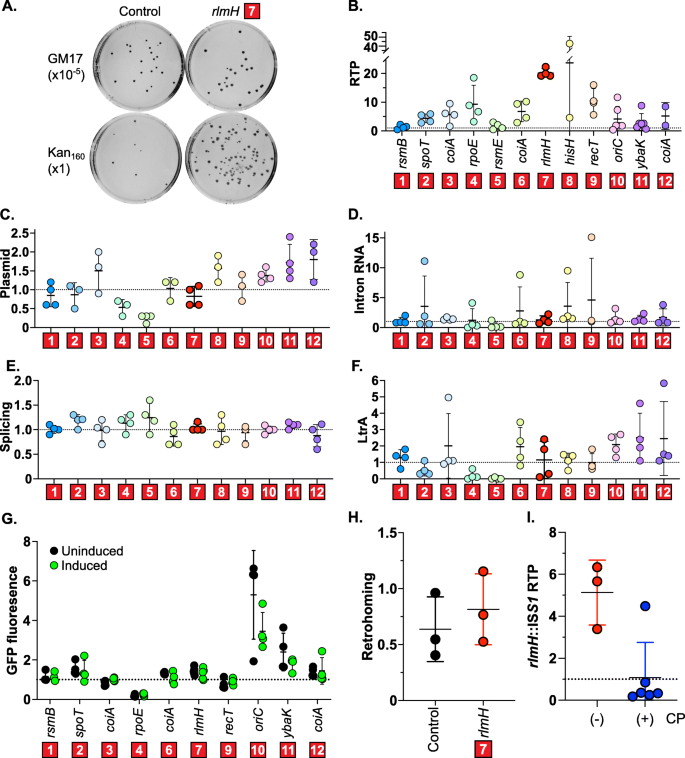
首先,我们在更大的培养物中进行了更定量的RTP试验,并在含卡那霉素和不含卡那霉素的GM17上镀上,以计算每个突变体的反转位频率,将含有卡那霉素的培养皿上反转位事件的菌落形成单位(CFU)的数量除以非选择性GM17上的总CFU(图5)。4a-b和表2;额外的文件1:图S10)。一些逆转录转位表型在http -RTP试验和个体表征之间有所不同,这可能是由于不同的生长条件或单独的RTP试验负责细胞生长,而最初的http -RTP试验严格测量卡那霉素的存活率。例如,在三个rRNA修饰突变体中,有两个,rsmB::S1而且rsmE::S1,在http - rtp试验中逆转录转位持续升高,但在个体表征中有逆转录转位的背景水平(图。4b;额外的文件1:图S10)。相比之下,rlmH::S1在两种试验中,逆转录转位均反复大幅升高。所有其他突变体的逆转录转位频率升高,尽管有差异rlmH::S1在12个选择的突变体中,作为明显的“赢家”,显示出持续20倍的反转录转位频率(图。4b和表2;额外的文件1:图S10)。
相对于对照的前12个突变体的特征。一个。Retrotransposition分析。在低通量下对每个突变体进行RTP分析,以量化逆转录转位频率。代表性的板块显示rlmH::S1突变株和等基因野生型对照株。用选择性介质上的CFU除以非选择性介质上的CFU计算逆转录转位频率。b。Retrotransposition频率。每个突变体都相对于包含pLNRK-RIG的野生型IL1403进行评估,忽略极端异常值。单个数据点显示,在平均值与标准偏差的水平条。有关所有数据点,请参阅附加文件1:图S10。c。DNA斑点。采用点印迹法测定供体质粒(pLNRK-RIG)的数量。有关数据,请参见附加文件1:图S11。d。北方的屁股。内含子RNA水平(前体和剪接内含子)针对16S rRNA归一化。有关凝胶,请参阅附加文件1:图S12。e。引物延伸分析。剪接效率计算为剪接内含子数量除以剪接内含子加上前体的总和。有关凝胶,请参阅附加文件1:图S13。f。西方的屁股。内含子编码蛋白LtrA的数量归一化为总蛋白。有关污点,请参阅附加文件1:图S14。g。由P归纳原子力安全保安院启动子。质粒pLNRK-GFP显示出GFP荧光。在诱导3小时后,每个突变体相对于野生型的归一化GFP值(绿色,诱导;黑色,uninduced)。有关所有数据,请参阅附加文件1:图S15。h。Retrohoming分析。用野生型和rlmH::S1包含内含子供体pLNRK-RIG和归巢位点pMN1343。两组间无显著差异rlmH::S1控制(p= 0.51)。我。互补的化验。用(蓝色)或不带(红色)互补质粒(CP)进行检测,并报告相对于各自的野生型对照。互补显著降低了RTP频率rlmH::S1(p< 0.05)
接下来,我们试图消除内含子过度生产可以解释反转位频率增加的可能性。因此,我们检测了每个突变体中内含子供体质粒的数量、内含子RNA水平、剪接、内含子编码蛋白(LtrA)的数量以及报告子诱导性(图2)。4c g;额外的文件1:图S11-S14)。因此,我们使用点印迹来测量质粒拷贝数(图。4c;额外的文件1:图S11), Northern blot检测内含子RNA水平(图S11)。4d;额外的文件1:图S12),引物扩展分析以测定剪接效率(图S12)。4e;额外的文件1:图S13), Western blot检测LtrA水平(图S13)。4f;额外的文件1:图S14)。最后,为了分析突变体如何更好地表达报告结构,我们用绿色荧光蛋白(GFP)基因替换II组内含子,并在nisin诱导下测量GFP荧光(图。4g和表1;额外的文件1:图S15)。尽管突变体之间的RNA剪接是一致的,但一些突变体的供体质粒、内含子RNA或LtrA水平升高(hisH::S1,oriC::S1,ybaK::S1,coiA::S1),两种突变体在所有测试中都有较低的水平(rpoE::S1而且rsmE::S1).当用GFP荧光测量诱导时,大多数突变体表现与对照相似oriC::S1而且ybaK::S1与对照组相比,无论诱导与否,突变体的报告结构表达均升高(图2)。4g;额外的文件1:图S15)。在各方面,rlmH::S1突变体的逆转录转位水平提高了20倍,质粒拷贝数、内含子RNA水平、LtrA和诱导水平与野生型对照物相当(图2)。4g)。
rRNA的甲基化影响RTP频率
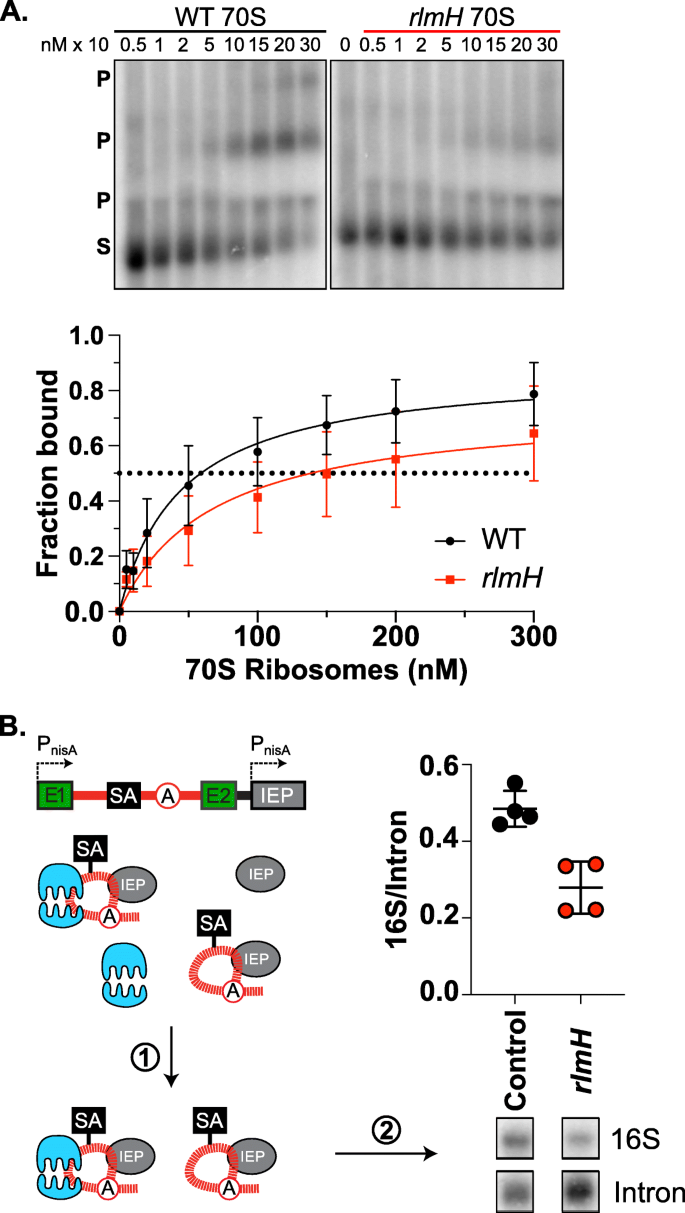
我们在http - rtp筛选试验中观察到一组逆转录转位增加的rRNA甲基化突变体:rlmH::S1, rsmB::S1,rsmE::S1。然而,在更定量的个体RTP测定中,rlmH::S1表现出相对于对照组显著增加的逆转录转位水平(20.1 +/−1.5),而rsmB::S1而且rsmE::S1均在对照组的误差范围内(分别为1.3 +/−0.7和1.7 +/−1.3)。4b和表2).因此,我们决定深入研究如何rlmH::S1突变体影响逆转录转位。首先,我们感兴趣的是rlmH::S1是否特定于逆转录转位,或者它是否也影响逆转录,因为这可能为RlmH在逆转录过程中的作用机制提供一些见解。我们改变了rlmH::S1pLNRK-RIG和对照的野生型pLNRK-RIG菌株,含有第二粒含巢位点pMN1343(表2)1),并以类似于个体RTP测定的方式测量返巢。归位位点的存在使得反向归位在数量级上成为首选过程,相比之下,反向转位的贡献可以忽略不计。的回巢频率rlmH::S1与对照组无显著性差异(图;4H),表明的影响rlmH::S1突变体是逆转录转位所特有的。
进一步验证对逆转录转位的影响是具体由于rlmH而不是IS的多效性S1插入时,我们进行了RTP分析,通过添加回rlmH基因(图。4i).为了减轻维持多个质粒的压力,我们使用了较低的抗生素浓度,这可能解释了不太戏剧性的,尽管仍然显著增加的逆转录转位频率rlmH::S1无互补质粒(5.1 +/−1.5)。在介绍rlmH互补质粒(表1),相对反转录转位频率下降到对照水平(1.1 +/−1.7)(图。4i).重新引入的反转位频率降低rlmH基因表明,在rlmH直接导致了逆转录转位的表型。
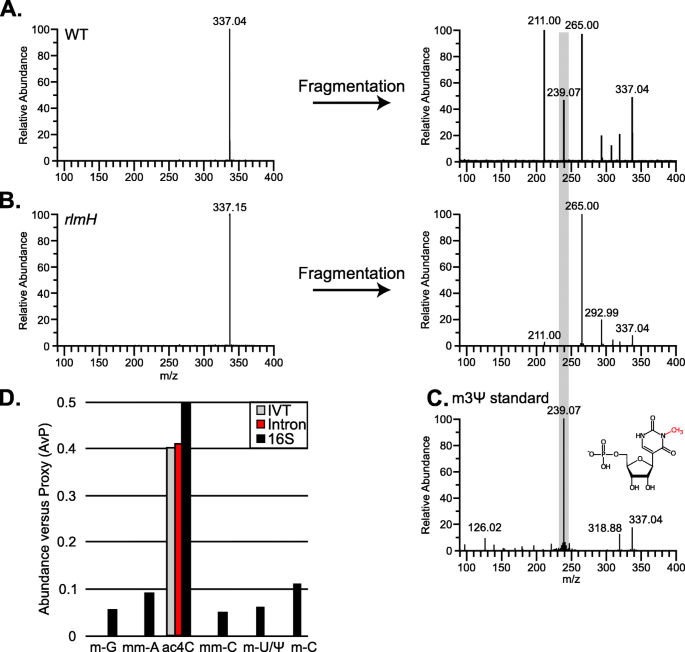
接下来,我们实验验证的功能rlmH基因l . lactisIL1403及其敲除rlmH::S1.在大肠杆菌,在完全组装的70S核糖体上,RlmH将大rRNA亚基1915位置的伪尿嘧啶转化为3-甲基伪尿嘧啶(m3Ψ) [40,41,42],质谱分析可以很容易地检测到其存在。我们的策略包括从野生型或rlmH::S1对突变细胞进行外切酶消化,将RNA还原为单个核苷酸成分,然后通过直接输注纳米流电喷雾(nanoflow electrospray, nanspray) MS分析产物[43].为了明确区分m3Ψ与一系列具有相同元素组成和质量的自然变体(即Um, m5U, m1Ψ和m3U),我们首先通过串联质谱(MS/MS)分析了相应的合成标准。数据显示,m3Ψ在239 m/z产生了一个独特的诊断片段,这是没有从任何其他异构体获得的(附加文件1:图S16)。当对70S样品进行检测时,两种消解混合物在337 m/z时都表现出丰富的信号,原则上可以对应上述异构体的任何可能组合(图2)。5左边是A和b)当提交到MS/MS时,来自野生型样品的337 m/z前体离子产生了一系列对应于任何可能的异构体组合的片段,包括m3Ψ的239 m/z信号特征(图2)。5a,右侧面板和图。5c).相比之下,这样的片段明显没有出现在337 m/z前体的MS/MS谱中rlmH::S1示例(图。5B,右边面板)。的70S核糖体中m3Ψ的缺失rlmH::S1证实该突变体完全敲除了RlmH功能,因为其70S核糖体没有显示m3Ψ,而野生型70S核糖体则显示m3Ψ。
质谱验证rlmH::S1核糖体和内含子RNA的修饰谱。一个。野生型rRNA信号在337 m/z的分离谱。分离的前体离子被提交到碰撞激活,观察到的碎片模式与标准记录的碎片模式进行比较(附加文件)1:图S16)。灰色高亮显示存在唯一的239 m/z信号,对应于m3Ψ(面板C和附加文件1:图S16)。其他突出的信号,如211和265 m/z对应于甲基化尿苷/伪尿苷等压产生的特征片段(附加文件)1:图S16)。b。从RNA中分离出的信号在337 m/z的分离和碎片谱rlmH::S1核糖体。灰色高亮显示了239 m/z信号下降的位置,如果突变rRNA中存在m3Ψ种。c。从m3Ψ标准获得的碎片光谱,显示239 m/z的突出诊断信号(灰色),这在其他等压线的光谱中是不存在的(附加文件)1:图S16)。d。内含子和/或16S rRNA中检测到的核糖核苷酸变异的相对丰度(AvP)。所示为纯化内含子RNA(红色)、16S rRNA(黑色)和体外转录内含子RNA (IVT,灰色)的修饰。缩写:m-G =甲基鸟苷,mm-A =二甲基腺苷,ac4C =乙酰胞苷,mm-C =二甲基胞苷,m-U/Ψ =甲基化尿苷或甲基化假尿苷,m-C =甲基化胞苷
在试图解释反转录转位频率增加的rlmH::S1,我们假设功能性RlmH可能混杂地修饰野生型细胞中的内含子RNA,从而限制逆转录转位。为了验证这一点,我们从野生型细胞的内含子RNPs中纯化内含子RNA,用于质谱分析(附加文件)1:图S17)。此外,我们纯化了与内含子共纯化的16S rRNA,作为阳性对照,并在体外转录的内含子RNA作为阴性对照。RNA分离后,利用天然琼脂糖凝胶电泳从16S rRNA中分离内含子RNA,提取代表内含子RNA或16S rRNA的单独条带进行分析。包括阴性对照在内的所有样品均显示乙酰化胞苷(ac4C)的存在,这可能是纯化方法的伪产物(图。5d).无论如何,与明显甲基化的16S rRNA相比,我们观察到内含子RNA上没有甲基化(图。5d).因此,内含子在野生型背景下是未修饰的,内含子本身没有甲基化并不能导致的逆转录转位频率的变化rlmH::S1.
在rlmH突变体中,核糖体与内含子RNA的结合减少
由于核糖体已被证明与II族内含子相互作用[16], RlmH作用于完全组装的核糖体[41],我们探讨了内含子RNA与70S核糖体的相互作用,包括野生型和野生型rlmH::S1突变株。为此,我们测量了体外转录的重折叠内含子RNA和纯化的70S核糖体之间的结合rlmH::S1和野生型细胞使用凝胶转移试验[16].从这些凝胶,我们观察到较弱的结合rlmH::S1突变型核糖体比野生型核糖体更明显,这表明在相同浓度的核糖体下,结合产物相对于未结合的底物更少。6一个)。
的rlmH::S1核糖体与内含子RNA的结合很差。一个。体外凝胶位移实验证明了这一点rlmH::S1突变70S核糖体与内含子RNA的结合存在缺陷。凝胶显示野生型(WT)和rlmH::S1核糖体-内含子结合,其中底物(S)在体外转录的内含子底物中被放射标记,结合产物(P)在凝胶中更高。核糖体浓度的增加显示在顶部。多重结合产物可能代表各种核糖体结合内含子RNA构象[16].下面是基于定量这些凝胶中条带强度的结合曲线,结合的平均分数与核糖体浓度的关系。b。体内内含子下拉与rlmH::S1核糖体结合内含子RNA缺陷。下拉结构在小外显子(E1和E2,绿色)中表达,内含子编码的蛋白质(IEP,灰色)在中表达反式.含有链霉亲和素适体(SA,黑色)的内含子RNA用链霉亲和素树脂(1),并对RNA进行Northern blot (2),探测内含子RNA和16S rRNA。rRNA与内含子RNA的比值被绘制出来,以单个复制为数据点,平均值为水平线,标准差为胡须。对照组rRNA与内含子RNA共纯化率显著高于对照组(p < 0.05)rlmH::S1突变株。完全印迹位于附加文件中1:图S18
为了进一步探索核糖体-内含子相互作用的作用rlmH::S1突变体,我们进行了一个下拉实验来测量体内结合。我们转化了无质粒rlmH::S1和野生型l . lactisIL1403含有内含子的质粒,内含子RNA序列中含有链霉亲和素结合适体1) [26].经过链霉亲和素琼脂糖树脂的生长、诱导和下拉,我们分离出RNA,并对内含子RNA和核糖体RNA进行Northern blot检测(图2)。6b;额外的文件1:图S18)。将16S rRNA的copurifying量与下拉内含子RNA的量归一化后,我们确定rlmH::S1与对照组(0.48)相比,突变体每个内含子的rRNA(0.28)显著减少(p< 0.05)(图6b).因此,核糖体来自rlmH::S1在体外或体内都不能结合内含子RNA以及野生型核糖体。综上所述,核糖体-内含子结合不良与反转录转位的特定增加相关,但与反归巢频率无关,这表明核糖体结合可以在复制叉处限制II组内含子的反转录转位(图2)。7一个)。
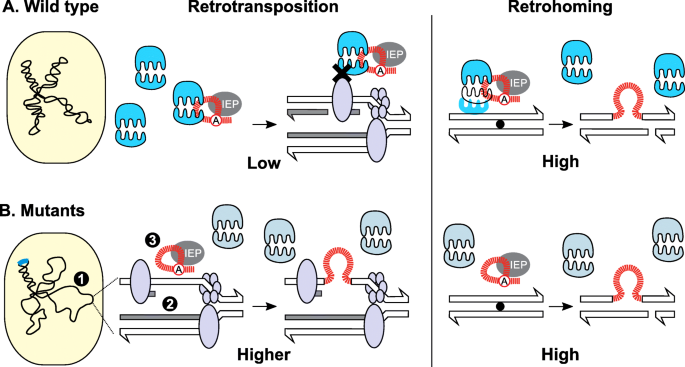
保护基因组对抗猖獗的反转录转位的模型。一个。野生型主机背景。在正常细胞条件下,细菌类核浓缩,DNA复制受到严格控制,内含子RNPs(红色RNA套索与灰色内含子编码蛋白结合)与核糖体(蓝色)结合,其功能是限制逆转录转位。核糖体结合的RNPs在拥挤的DNA复制叉处与蛋白质发生空间冲突(黑色X),抑制逆转录转位,限制了cDNA合成的引物(Low)。相反,在双链DNA中,回巢到归巢位点(黑点)不受核糖体存在的影响,回巢是有效的,与免于拥挤的复制叉(High)一致。b。突变宿主背景。突变某些宿主基因(染色体上的蓝色插入)增加逆转录转位频率。(1)氨基酸生物合成途径中断或严格反应调控不当(现货::S1,rpoE::S1,hisH::S1)导致类核重组成更有利于逆转录转位的构象。(2)由于严格反应的激活或DNA重组和修复功能的中断,导致复制分叉停滞(单链间隙增加)(coiA::S1,矩形::S1)成为反转录转位的主要目标。(3.)核糖体结合功能失调(rlmH::S1)允许内含子RNPs更自由地侵入DNA复制叉(Higher),而逆行则保持不变(High)。仅为说明目的,突变体的各种影响(编号1,2,3.)一起显示在同一幅图上
讨论
通过创建一个突变体库Lactococcus lactisIL1403, Ll的本机主机。LtrB内含子,我们强调了宿主生物遗传背景对II组内含子反转位的重要性。我们缩小了92个伊斯兰国的名单S1与对照相比,这些突变体具有更高的逆转录转位频率,并进一步研究了个体基础上的12个突变体的简短列表(图。4和表2).从这12个涉及氨基酸生物合成、严格反应、DNA重组功能和核糖体修饰的突变体中,可以发现rlmH::S1缺乏rRNA甲基化的突变体脱颖而出,具有最引人注目和一致的逆转录转座表型。此外,rlmH::是S1突变体表现出核糖体与内含子结合的缺陷,这使我们提出在体内核糖体结合在限制原生宿主内含子逆转录转位中发挥重要作用。
从L. lactis突变库带回家的信息
虽然突变体库是广泛的,但它不是全面的,因为我们没有获得完整的覆盖l . lactis基因组。然而,我们确实得到了一个随机的,有代表性的分布(图。1e).根据我们的COG分析,我们的突变体的功能分布与l . lactis尽管与翻译(J)和核苷酸代谢(F)相关的功能在文库中未被充分代表(图。1f). Since ISS1换位是随机的(图;1 e;额外的文件1:图S4) [23,28],我们怀疑突变文库中的代表性不足是基因本质的结果。在枯草芽孢杆菌,许多参与蛋白质合成的基因(J),以及参与核酸代谢的基因(F)都被认为是必不可少的[44].按照同样的逻辑,我们怀疑我们在氨基酸代谢(E)、碳水化合物代谢(G)和宿主防御机制(V)中观察到的过度表达是由于在富培养基的实验室生长条件下,其中一些基因具有非必需或冗余的性质。虽然STRING蛋白相互作用网络分析未能揭示逆转录转位的主要调控因子(附加文件1:图S9),由于我们库的不完全性,不能完全排除这样一个调节器的存在。
当我们将表现出逆转录转位增加的突变体与整个文库进行比较时,我们观察到RTP-up突变体更频繁地出现在COG类E中(图5)。3.),其中含有与氨基酸转运和代谢有关的基因[29].氨基酸代谢之间的关系,特别是饥饿和严格反应,和逆转录转位已在先前的描述大肠杆菌[33].事实上,在最感兴趣的12个突变体中,少数具有与严格反应和氨基酸代谢有关的功能,即现货::S1,hisH::S1,rpoE::S1.在大肠杆菌工作,∆relA和∆现货突变体使我们得出结论,ppgpp介导的应激反应刺激逆转录转位[33].由于ppGpp是饥饿和严格反应的关键信号分子,因此其他与氨基酸生物合成(E)功能相关的基因也影响逆转录转位就不足为奇了。在前12名突变体中,hisH是组氨酸生物合成所必需的[34].组氨酸生物合成的调节延伸到我们库中的另一个突变体,rpoE::S1,因为RpoE负责适当的监管hisH[35].考虑到这三个前12名突变体,现货::S1,hisH::S1,rpoE::S1与氨基酸饥饿或调节有关,我们怀疑它们都通过类似于研究中报道的ppgpp介导的机制来影响逆转录转位大肠杆菌[33].研究大肠杆菌假设氨基酸饥饿后的严格反应通过促进有利于反转录转位的类核结构和增加停滞复制叉的发生来刺激反转录转位[33].尽管宿主生物之间存在差异,但我们的数据表明,氨基酸代谢对逆转录转位调节的重要性在原生宿主中是保守的。7b)。
其他前12个RTP-up突变体,oriC::S1,矩形::S1,coiA::S1,破坏对DNA复制、重组和修复重要的基因或区域。的oriC::S1插入是复制的原点l . lactis染色体,这是开始DNA复制的位置。我们观察到逆转录转位频率的增加oriC::S1(无花果。4b),这可能是由于IS导致复制分叉停止S1但由于GFP试验表明,无论诱导与否,该突变体表达均显著增加(图2)。4g;额外的文件1:图S15),可能逆转录转位的增加是内含子结构表达改善的间接影响。相比之下,矩形::S1与对照组相比,逆转录转位升高,表达谱正常(图。4B和g;额外的文件1:图S10和S15)。RecT蛋白对DNA双链断裂修复很重要[37],破坏DNA修复可能为逆转录转位进入受损DNA或停滞的复制叉提供更多机会,因为II组内含子直接与DNA复制叉机制相互作用[45].虽然CoiA由三个独立的插入突变体表示,与COG类R(仅用于一般功能预测)相匹配,但进一步研究发现,CoiA蛋白通过促进重组在转化后的DNA加工中发挥作用[36].类似于矩形,这是可能的表达coiA通过刺激适当的细胞修复途径促进重组和抑制逆转录转位。总的来说,这些与DNA复制、重组和修复相关的功能与II组内含子逆转录转位途径一致,其中复制分叉停滞和修复或重组功能受损会产生单链DNA焦点,从而促进反向剪接和第二链DNA合成[6,7,46(图。7b)。
核糖体甲基化作为对抗猖獗的反转录转位的保护措施
强而一致的表型rlmH::S1需要我们验证突变体中的rRNA甲基化缺陷,并消除rlmH::S1直接作用于修饰内含子RNA(图;5d).此外,利用遗传互补,我们表明,表型是由于直接rlmH突变而不是多效效应(图;4i).因此,我们得出结论,rRNA甲基化缺陷直接抑制核糖体与内含子RNA的结合,这反过来又减轻了对逆转录转位的抑制作用(图。6).这些结果与我们之前的研究一致,即在体内和体外70S核糖体与内含子RNA有很强的特异性结合l . lactis[16].然而,在大肠杆菌核糖体结合实际上解释了反转录转位的增加,而不是保护基因组免受II组内含子入侵[15,16].这一差异是由大肠杆菌核糖体保护II组内含子免受RNase E的降解l . lactis没有RNase e的同源物。l . lactis更类似于枯草芽孢杆菌在这方面,可能使用完全不同的降解体大肠杆菌[47].因此,核糖体-内含子相互作用似乎在原生宿主中起着不同的作用。
重要的是,我们发现rlmH::S1是不与逆转录转位相反,升高与核糖体结合在DNA底物水平上的抑制作用一致,这对于这两种逆转录过程是不同的[6,7,11,12,13].逆转录转位最常发生在拥挤的复制叉上,而逆转录转位则发生在不受复杂分子机制影响的特定归巢位点上,并且似乎已经进化为高效的内切酶介导的内含子整合[6,7,11,12,13].因此,我们认为核糖体占据II族内含子会导致拥挤复制叉的空间冲突,从而阻碍原生宿主的逆转录转位(图2)。7一个)。
由于猖獗的逆转录转位对宿主生物是危险的,核糖体-内含子结合可能已经进化到限制逆转录转位,从而保护原生生物l . lactis宿主从细菌到哺乳动物的生物已经进化出了广泛的机制,以减轻逆转录转座子和其他可移动元素的负担[48,49,50,51].II组内含子的表达及其相应的逆转录转位活性对细菌的生长速度和生存都有影响[52].为了对抗这些缺陷,II组内含子逆转录转位在大肠杆菌受RNase E活性的限制[15].然而,l . lactis缺乏RNase E的任何同源物,必须应对Ll的更高逆转录转位频率。LtrB基因内区。我们的研究结果有力地证明了一种机制l . lactis处理猖獗的逆转录转位是通过核糖体-内含子结合抑制拥挤复制叉处的逆转录转位(图。7).
移动元件通常是基因组内冲突的来源,有不同的进化途径来解决[50].虽然,我们描述了核糖体和II组内含子之间的相互作用如何限制逆转录转座子以保护宿主的一个例子,但这些内含子并不是唯一的逆转录转座子,其转座子由核糖体调节:Alu元件,依赖于自主LINE逆转录转座子的蛋白质活性的非自主逆转录转座子,可以劫持和暂停翻译LINEs的核糖体,以获取Alu迁移所需的ORF2蛋白[53,54].这两个反转录转座子在核糖体水平的交叉为宿主生物的额外冲突和/或控制奠定了基础。逆转录转座子和核糖体之间的相互作用尚未被充分探索,但根据我们对II组内含子的研究结果,我们预测核糖体可以作为逆转录转座子的更普遍的调控层。
结论
正如用IS库演示的那样S1在突变体中,原生宿主的生物学是控制II组内含子逆转录转位的关键。一些相互作用似乎在生物之间是保守的,例如氨基酸转运和代谢功能突变与逆转录转位增加之间的相关性,这表明严格反应的激活可能广泛地刺激逆转录转位。相比之下,II组内含子-核糖体相互作用在原生宿主中表现明显,核糖体结合的破坏导致逆转录转位频率显著增加,而不影响逆转录归巢。因此,我们揭示了核糖体作为宿主基因组守护者的新作用,通过限制复制分叉处的反转录转座子,这可能广泛适用于其他反转录转座子。
材料与方法
细菌生长条件
本研究使用的菌株列于表中1.除非另有说明,所有菌株都在如下条件下生长。l . lactisIL1403菌株在GM17培养基(M17肉汤中添加0.5% (w/v)葡萄糖)中密闭盖管或瓶中培养,30℃无曝气。在适当的情况下,培养基中氯霉素含量为5或10 μg/mL (Cam5或凸轮10),卡那霉素浓度为160 μg/mL (Kan160),红霉素浓度为2或0.5 μg/mL (Erm2或Erm0.5)或50 μg/mL的大霉素(Spc50).大肠杆菌菌株在Luria肉汤(LB)培养基中37℃曝气培养。适当时,培养基中氨苄西林含量为100 μg/mL (AmpOne hundred.)、25 μg/mL氯霉素(Cam25)或红霉素150 μg/mL (Erm150).
质粒方法学,酶和寡核苷酸
本研究使用的质粒列于表中1.用EZNA质粒Mini试剂盒(Omega)分离纯化质粒DNA。所有寡核苷酸都列在附加文件中1:表S5;它们是通过人工检查或NEBuilder (nebuilder.neb.com),并由集成DNA技术(IDT)合成。PCR片段使用CloneAmp HiFi PCR Premix (Clontech)或Taq聚合酶2x Master Mix (Thermo Fisher Scientific)进行扩增。所有克隆都在大肠杆菌,然后转化为l . lactis菌株。
治疗l . lactisIL1403是S1以50 μL接种量和不同量的50%抗坏血酸(0、100、200 μL)继代培养1 mL,不加抗生素,培养18 h。培养物被稀释2-和103.-fold,在GM17上镀,生长18 h。GM17和GM17 Cam上的殖民地被修补10识别敏感的(Cam年代)菌落,进一步pcr验证质粒(IDT5546和IDT5547)的丢失。
突变体库的构建
IS图书馆S1突变体的生成如[23].简而言之,l . lactisIL1403携带Ll。将LtrB内含子供体质粒pLNRK-RIG新鲜转化为pGh4:ISS1质粒电穿孔。在GM17 Cam上28°C过夜生长后,选择一群共转化子10(选择pLNRK-RIG)和Erm5(选择pGh4:ISS1)琼脂。诱导IS转位S1将质粒整合到细菌染色体中,新鲜培养物在28℃下孵育2.5小时,然后转移到37.5℃下再孵育2.5小时。通过在GM17 Cam上37°C过夜生长,选择发生转位事件的突变体5和Erm2.然后将单个突变体在96孔板中37°C的1ml GM17 Cam中过夜10.治疗pGh4:IS细胞S1过夜后,培养液被稀释3.-折叠在新鲜GM17凸轮1028°C孵育18 h。然后在GM17 Cam上进行培养5琼脂(选择pLNRK-RIG)不含Erm(不选择pGh4:IS)S1),并将最终温度移至37°C 18 h。单个菌落被修补到选择性的(嗯2)和非选择性板(凸轮5), 37℃孵育。对Erm敏感表明pGh4:IS缺失S1质粒。
杂交探针制备
的是S1用高稀释pGh4:IS引物对IDT3705和IDT3706扩增探针S1质粒DNA为模板(预期产物267 bp), rig特异性探针以pLNRK-RIG为模板,IDT374和IDT375扩增(预期产物1545 bp)。PCR产物经凝胶提取纯化,并用α-[32P]-dCTP (PerkinElmer),使用随机引物DNA标记系统(Invitrogen)。
所有寡核苷酸探针均用1-2 U的T4多核苷酸激酶和25 μM γ-[32P]-ATP (PerkinElmer)在37°C下孵育1小时,然后用Illustra MicroSpin G-50色谱柱纯化(GE Healthcare)。所有实验都使用32使用Typhoon Trio磷成像仪(GE医疗)对P进行成像。
菌落印迹杂交
将Hybond XL膜(GE Healthcare)放置在具有假定突变菌落的琼脂板上5分钟,进行菌落转移。转移后,菌落在以下溶液中浸泡:两次在20 mg/mL溶菌酶中浸泡15分钟,在37℃,两次在0.5 M NaOH中浸泡3分钟,在25℃,两次在1 M Tris-HCl (pH 7.4)中浸泡5分钟,在25℃,一次在含有1.5 M NaCl和0.5 M Tris-HCl (pH 7.4)的缓冲液中浸泡5分钟。膜用Stratalinker UV 1800 (Stratagene)交联。交联膜在快速杂交缓冲液(GE Healthcare)中65°C预杂交15分钟,然后与变性的IS PCR探针孵育S1或RIG在65°C下放置2小时。印迹用6X SSC在65°C下清洗两次15分钟,然后最后用2X SSC在65°C下清洗15分钟。膜在25°C下暴露于存储荧光屏1天。
Southern blotting和杂交
所选IS基因组DNAS1使用nexttec 1-Step DNA Isolation Kit for Bacteria (nexttec biotechngie GmbH)对突变体进行分离和纯化。3 μg的基因组DNA被消化后III-HF(内)。将大约400ng消化后的DNA加载在0.7%琼脂糖凝胶上,在0.5X TBE中,在25°C下运行8小时。使用GENIE电印迹器(Idea Scientific)在12v下将凝胶电印迹30分钟到Biodyne b带电尼龙膜(Pall)上。将固定化DNA用0.4 N NaOH浸泡10 min, 2X·SSC洗涤10 min,交联。预杂交在65°C的快速杂交缓冲液中进行1小时(GE Healthcare)。(32P]标签S1引物IDT3705和IDT3706的PCR产物用0.1 vol 2 N NaOH变性8分钟,然后用0.1 vol 1 M Tris-HCl中和,pH 7.4。将预杂交缓冲液用5 mL新鲜探针杂交缓冲液代替。在65°C下杂交24 h。用2X·SSC在65℃下洗涤2次30 min,用0.1X SSC和0.1% SDS在65℃下洗涤1次30 min。然后膜成像如上所述。
利用下一代测序技术绘制ISS1插入位点
为了尽量减少测序反应的数量,我们使用直三策略进行正交池化和映射[27].简单地说,就是所有阵列的ISS1突变株在1ml GM17 Cam中培养10直到饱和。收集的11个板分为PL1-6和PL7-11两套。通过组合集合中所有板块的公共行(A到H)和列(1到12)的文化来生成池。都是S1从一个培养皿中提取突变株,形成培养皿池,PL1-6组为6个培养皿,PL7-11组为5个培养皿。
对于每个池,使用DNeasy血液和组织试剂盒(Qiagen)按照制造商的革兰氏阳性细菌方案分离基因组DNA。DNA通过琼脂糖凝胶电泳和NanoDrop(赛默飞世尔科学公司)进行评估。使用Bioruptor标准超声系统(Diagenode)对总DNA (100 μL终体积TE缓冲液中2 μg)进行碎片化,参数如下:在4℃下开/关周期30 min, 30 s。剪切的DNA在0.7% (w/v)琼脂糖凝胶上用溴化乙胺染色,用QIAquick凝胶提取试剂盒(Qiagen)从凝胶中分离出200 bp - 800 bp的片段。基因组DNA制备后,使用NEB Next DNA文库准备试剂盒(NEB)进行文库构建。必要时用QIAquick PCR纯化试剂盒(Qiagen)对片段进行纯化。样本被提交到奥尔巴尼大学功能基因组学中心进行Illumina测序,以获得高输出(75个周期,400 M reads)测序。
适配器和ISS1使用自定义生物马拉松脚本(可按要求提供)从读取中删除序列。该脚本允许精确检测IS的3 '端S1在生成的读的片段中,然后是IS的修剪S1上游序列留下了侧翼碎片。读数3 '末端的5个bp似乎质量较低,也被移除。使用Galaxy服务器(usegalaxy.org) [55].Bowtie软件用于自定义设置下的读取映射[56],其中:'种子中允许的最大错配数'设置为' 0 '(参数-n), '当存在有效对齐时是否尽可能努力寻找'设置为' try hard '(参数-y)。此外,该设置是根据地层(不匹配的数量)和不匹配位置的质量值(参数-best)报告“最佳”单例对齐。FASTQ美容师[57], SAMtools [58], deepTools [59]和BEDTools [60]也被用于分析其他在Galaxy服务器上实现的生物信息工具(usegalaxy.org) [55].
同源基因簇(COG)分析
所有注释的蛋白质l . lactisIL1403 (ASM686v1)被用作本地blastp的查询[61]根据COG数据库进行搜寻[29](最后访问时间为2018年7月10日),其中根据e值分数记录每个查询的最佳匹配。如果一个蛋白质/COG家族命中在多个类别中表示,每个类别计数一次。类别的相对丰度被用于比较。对于统计比较,我们执行了超几何检验,将我们的测试样本与背景水平进行比较(如在[62,63])。计算p-values时,我们使用统计软件R中的phyper函数,并将值调整为对应于双尾检验,以便对所做的比较进行适当的保守。同样的分析被用于比较RTP-up突变体与文库。
逆转录转位和回巢分析
逆转录转位试验l . lactis用内含子供体质粒pLNRK-RIG以高通量和更定量的低通量方式进行IL1403。在96深井板中同时对1006个突变体进行了高通量逆转录转位(http - rtp)检测。将原突变株在1ml GM17 Cam中培养过夜10然后传代培养1∶10个新鲜的GM17凸轮10直至对照菌株IL1403 pLNRK-RIG达到OD值6000.2。添加10 ng/mL nisin诱导内含子表达,培养3 h,直至在矩形GM17 Kan上点镀(5 μL)160使用机器人液体处理器JANUS G3 (PerkinElmer),配备8尖Varispan臂(PerkinElmer)。30°C孵育2天。
GM17 Kan生长(光斑大小)的稳健性160对每个菌株进行sgatols分析(sgatools.ccbr.utoronto.ca) [30.].每个培养皿的斑点大小评分归一化,与来自同一培养皿的对照IL1403 pLNRK-RIG计算的斑点大小评分相同,从而得出每个菌株的相对内含子反转置水平。为了鉴定始终显示较高相对逆转录转位的突变株,使用BoxPlotR (shiny.chemgrid.org/boxplotr),然后使用自定义Python脚本进行排序(可通过请求获得)。感兴趣的突变体被分为两层。在所有三个http - rtp试验中,在第三个四分位数(Q3)和第四个四分位数(Q4或最大值)之间表现出相对逆转录转位水平的菌株被分配到第一层。两次位于Q3和Q4之间,一次位于中位数(Q2和Q3)之间的菌株被确定为第二级。
定量低通量RTP测定在选定的菌株中进行,如前所述[6,21].简而言之,培养物在40ml GM17 Cam中培养10以1:33稀释到OD6000.2。用10 ng/mL nisin诱导内含子表达3 h,并将内含子镀在GM17和GM17 Kan上160.剩余的培养物以10ml的增量制成颗粒,并在−80°C保存,以便进一步分析(见下文)。
使用pMN1343提供归巢位点的回巢分析,执行方法如下[6],除了使用pLNRK-RIG内含子供体质粒诱导3小时外,与本研究其余部分一致。根据反转位频率计算回巢频率,用t检验评估显著性。
RTP-up突变体ISS1插入位点的反PCR定位
所选IS基因组DNAS1使用nexttec 1-Step DNA Isolation Kit for Bacteria (nexttec biotechngie GmbH)对突变体进行分离和纯化。DNA (1 μg)用分3AI和通过T4 DNA连接酶(NEB)连接循环,根据制造商的方案。用引物IDT3858和IDT3859或IDT3860和IDT3861扩增DNA片段,得到5 ' -或3 ' - IS的产物S1侧翼区域,分别。所得PCR片段在1% (w/v)琼脂糖凝胶上进行电泳,凝胶纯化并测序(EtonBio)。序列被映射到的基因组l . lactisIL1403 [22]使用BLAST (BLAST) [64]通过国家生物技术信息中心(NCBI;www.ncbi.nlm.nih.gov).参考基因组核苷酸序列的登录号为NC_002662。用基因特异性引物进行PCR鉴定(表S5)。
蛋白质相互作用网络
从NCBI检索文库中所有非冗余突变体的独特蛋白质序列(510个序列)。这些序列是根据STRING数据库(string-db.org) [32],并将提交的510个序列中的505个映射到STRING数据库。STRING数据库中未标识的其余5个序列可能是l . lactis基因组,于2020年3月22日重新注释。另外,属于转座酶的两个序列虽然名称不同,但完全相同,因此用STRING进行合并,得到503个序列。映射的STRING标识符用于生成STRING应用程序的网络(版本1.5.1)[65]在Cytoscape(3.7.2版本)[66].将COG类别添加到每个突变体中,并使用基于COG类别的分组布局重新构建网络。当一个蛋白质属于多个COG类别时,随机选择一个类别进行分组。最终网络包含503个节点,在默认参数截断置信度分数(0.4)下,其中52个节点没有相互作用。
点杂交质粒定量
为了测量质粒供体(pLNRK-RIG)拷贝数,在低通量RTP试验中产生的细胞颗粒重新悬浮在2 mL GM17中并稀释至OD6000.4。每个样品取2 μL涂抹在Hybond XL膜(GE Healthcare)上,风干。膜处理如上所述的菌落杂交。交联后,膜在快速杂交缓冲液(GE Healthcare)中42°C预杂交15分钟,然后与5 '标记的[32P]- pLNRK-RIG特异性寡核苷酸探针(IDT5059),在42°C下1小时。膜在42°C下用6X SSC洗涤三次15分钟,并使用ImageJ (imagej.nih.gov / ij) [67].
RNA定量northern blotting
通过苯酚/氯仿异戊醇(PCIA)提取、乙醇沉淀和DNase处理(RQ1 DNase, Promega)从细胞颗粒中分离总RNA,然后再进行PCIA提取和最终乙醇沉淀。如前所述,进行北方印迹[16,26].杂交与5 '标记[32P]-内含子特异性寡核苷酸探针(IDT1073)在42°C下放置3小时。为了标准化,印迹被剥离,并与5 '标记的[32P]-16S rna特异性寡核苷酸探针(IDT861),如上所述。
通过引物延伸分析测量拼接效率
引物延伸分析如前所述[5,26,68].使用SuperScript III (Invitrogen)和0.4 pmol的5 ' -标记[32P]-内含子特异性寡核苷酸(IDT1073)。反应在8%尿素-聚丙烯酰胺凝胶上分离(国家诊断),并暴露在荧光屏上。使用ImageQuant进行分析。
蛋白印迹定量
对于LtrA分析,如所述进行Western blotting [16,26作了修改。低通量RTP检测细胞颗粒的总细胞裂解物在预铸的12%聚丙烯酰胺mini-PROTEAN TGX凝胶(BioRad)上在300 V下运行30分钟,并使用BioRad的Trans-Blot涡轮转移系统转移到PVDF膜上。用10%干牛奶堵塞膜,清洗,然后用抗ltra一抗(Covance)在25℃孵育1小时。然后清洗膜,用二抗(HRP偶联,抗兔(Advansta))在25°C孵育1小时,再次清洗,并使用Advansta westerbright量子试剂检测蛋白质。膜在BioRad Chemi Doc XR系统上成像和分析。所有样品在考马斯氏染色预铸凝胶上归一化至每道总蛋白。
绿色荧光蛋白测定
pLNRK-GFP质粒通过从pLNRK-RIG中去除内含子生成太平洋标准时间我和SpeI消化,随后连接从pHGSap中扩增出的绿色荧光蛋白(GFP)基因[69使用NEBuilder (IDT6054和IDT6055)。如前所述,将单个突变体固化,用pLNRK-GFP进行电转化,然后进行PCR确认(IDT6056和IDT6057)。将携带质粒的菌株在40ml GM17 Cam中1:10传代培养10.2小时后,将培养物分成两管。其中一半培养基用10 ng/mL nisin诱导GFP 4 h,其余培养基不诱导,继续生长。每小时取1 mL转移到96深井板(Eppendorf),以3220 x g离心8分钟,清洗,并在1X PBS (137 mM NaCl, 2.7 mM KCl, 10 mM Na)中连续稀释2HPO4, 1.8 mM KH2阿宝4).将200 μL的体积转移到96孔板(透明平底,康宁)中测量OD600和GFP荧光(黑色平底,康宁)在平板阅读器(BioTek Synergy H1)上。数据使用BioTek Gen5和Microsoft Excel进行分析。
互补反转位测定
制备互补载体pGh5:P原子力安全保安院,是S1被控卫消化掉了+host5:S1 L. lactis/E。杆菌穿梭向量[23)使用后III切割部位,导致pG+host5 (pGh5)。乳清素诱导启动子P日本原子力安全保安院说,pcr从pLNRK-RIG (IDT6871和IDT 6872)中扩增,亚克隆到pGEM-T Vector System I (Promega)中,转化为大肠杆菌DH5α,随后被酶切并克隆到EcoR我和后III位点,导致pGh5“空”:P原子力安全保安院向量。的rlmH基因PCR扩增l . lactisIL1403 gDNA (IDT6924和IDT6925)克隆到pGh5:P原子力安全保安院使用NEBuilder生成pGh5:P原子力安全保安院rlmH.使用M13引物对结构物进行测序(Eton Bio),并转化为相关的结构物l . lactisIL1403菌株。野生型和ISS1含有pGh5的突变株:P原子力安全保安院或pGh5: P原子力安全保安院rlmH质粒在GM17 Cam中培养5嗯0.5和逆转录转位试验如上述所述,但在Cam中进行5.采用t检验评估统计学意义。
核糖体纯化、结合分析和70S rRNA分离
70S核糖体的分离l . lactisIL1403和rlmH::S1突变体为[16稍加修改。简单地说,裂解物被装载在40%蔗糖垫上(40%蔗糖,20 mM Tris-HCl pH 7.5, 500 mM NH4Cl, 0.5 mM EDTA, 6 mM β-巯基乙醇,20 mM MgCl2),并使用70型钛转子(Beckman Coulter)以29,500 rpm的速度超离心26小时制成颗粒。核糖体颗粒在70S重悬缓冲液(50 mM NH)中重悬4Cl, 20 mM MgCl2, 20 mM Tris-HCl pH 7.5, 0.5 mM EDTA, 6 mM β-巯基乙醇),然后在10-40%蔗糖梯度上分离。将含有70S核糖体的部分合并、浓缩,并将缓冲液交换(3X)到最终的再悬浮缓冲液(20mm Tris-HCl pH 7.4, 200mm KCl, 5mm MgCl2).纯化的核糖体与标记的体外转录内含子RNA用于核糖体结合实验,如所述[16],并使用Prism 8 (GraphPad)软件绘制数据。
从分离的70S核糖体中,PCIA提取,MS级乙酸铵乙醇沉淀,MS级水中重悬,制备用于MS分析的RNA样品。样品用外切酶消化,如下所述。
体内内含子RNA下拉
野生型和rlmH::S1将含有pLNRK-SA下拉质粒的菌株1:20传代培养至100 mL GM17 Cam中10他长大后吸毒过量600用乳酸链球菌素诱导3小时。诱导后,离心收集50 mL等份的细胞培养物,并将微球储存在−80°C。细胞微球在300 μL CB溶液中重悬500缓冲液(20 mM Tris-HCl pH 8.0, 500 mM NaCl, 0.1 mM EDTA),超声裂解。高容量链霉亲和素琼脂糖树脂(Thermo Scientific)用CB洗涤3次500然后与裂解液孵育一夜,在4°C下结壳。然后用CB洗涤树脂10次50010 mM生物素500 μL洗脱500.RNA通过两轮PCIA提取和乙醇沉淀从洗脱液中分离。如上所述,对分离的RNA进行Northern印迹(1小时杂交),用相同的凝胶并排运行,对每个探针进行一次印迹(内含子RNA为IDT1073, 16S rRNA为IDT861)。采用t检验评估统计学意义。
天然内含子纯化用于RNA修饰的质谱分析
噢。LtrB RNP诱导和纯化如前所述[5几丁质柱洗脱后,PCIA萃取分离RNA,乙醇沉淀,DNase消化。对照的体外转录内含子RNA如前所述合成[16].所有样品在0.5X TBE的1%琼脂糖凝胶上分离,分别切除内含子RNA和16S rRNA对应的条带。切除的条带在−80°C冷冻过夜,然后在4°C下以最大速度离心30分钟,取上清,浓缩RNA,用3 kDa MWCO Amicon离心柱将3X交换成10 mM乙酸铵(用10 mM乙酸铵预冲洗)。
消化和质谱分析
取1.2 μg RNA (30 μL, 10 mM乙酸铵),用1 μL Nuclease P1 (Sigma Aldrich)在37℃下孵育2 h。加入碳酸氢铵至终浓度为100 mM,然后用1 μL蛇毒磷酸二酯酶(Sigma Aldrich)在37℃处理2 h,得到MS所需的单磷酸核苷酸(NMP)混合物[70],然后将样品储存在−20°C以停止反应。在分析之前,将NMP混合物在10 mM LC-MS级乙酸铵(Sigma Aldrich)和10% 2-丙醇(fisheries scientific)中稀释至4 ng/μL。所有样品均使用Thermo Scientific LTQ-Orbitrap Velos仪器进行分析[43,71].纳米流直接输注电喷雾电离(纳米喷雾)是在负离子模式下使用自制石英发射器进行的。用凝胶加载器移液管将最多5 μL的样品加载到每个发射器中。在发射器的后端插入一根不锈钢线,以提供0.8至1.0 kV的电离电压。通过密切监测铵加合物和水团簇的发生率来调整源温度和脱溶条件。
每种修饰的相对丰度用丰度相对代理(abundance versus Proxy, AvP)表示,计算公式如下:
其中,信号强度(aix)的每一种修饰对同一光谱中四个标准基(cr我).
化学合成的甲基化尿苷和伪尿苷核苷购自Berry & Associates,用于合成相应的nmp,并进一步纯化,如[72].为了揭示每个等压NMP在气相中破碎时的独特破碎模式,如前所述,在正离子和负离子模式下进行串联质谱(MS/MS)分析[43,73].然后,将生物样品中甲基化尿苷和假尿苷分离得到的m/z的碎片模式与合成标准品的碎片模式进行比较,以确定生物样品中等压改性NMPs的组成。
数据和材料的可用性
在当前研究中使用和/或分析的数据集可根据要求从通信作者处获得。这项工作中使用的公开数据集可在COG数据库中获得,http://www.ncbi.nlm.nih.gov/COG[29和STRING数据库,http://string-db.org/[32].
参考文献
Toro N, Jiménez-Zurdo JI, García-Rodríguez FM。细菌II组内含子:不仅仅是剪接。微生物学通报,2007;31(3):342-58。
Lambowitz AM, Zimmerly S. II组内含子:侵入DNA的移动核酶。中国生物医学工程学报。2011;3(8):a003616。
Lambowitz AM, Belfort M.真核生物进化关键的可移动细菌群II内含子。在:克雷格NL,钱德勒M,盖勒特M,兰博维茨AM,赖斯PA,桑德梅尔SB,编辑。移动DNA III[互联网]。华盛顿:ASM出版社;2015.1209 - 36页。
Novikova O, Belfort M.移动组II内含子作为祖先真核元素。植物学报,2017;33(11):773-83。
曲国强,王杰,沈松松,王志刚,等。II族内含子与其逆转录酶复合物的结构。分子生物学杂志,2016;23(6):549-57。
Ichiyanagi K, Beauregard A, Lawrence S, Smith D, Cousineau B, Belfort m。LtrB II组内含子主要通过反向剪接进入DNA靶点。中华微生物学杂志,2002;46(5):1259-72。
钟杰,兰博维茨AM。利用DNA复制叉上的新生链来启动逆转录的II组内含子迁移。中国生物医学工程学报。2003;22(17):4555-65。
张晓明,张晓明,张晓明,等。ⅱ族内含子的进化研究。暴徒的DNA。2015; 6(1): 7。
贝尔福特M,兰博维茨AM。II组内含子RNPs和逆转录酶:从逆转录元件到研究工具。中国生物医学工程学报。2019;11(4):a032375。
Singh RN, Saldanha RJ, D 'Souza LM, Lambowitz AM。II组内含子编码的逆转录酶/成熟酶与其高亲和力内含子RNA结合位点的结合涉及序列特异性识别和自调节翻译。中华分子生物学杂志,2002;18(2):287-303。
Cousineau B, Smith D, Lawrence-Cavanagh S, Mueller JE, Yang J, Mills D,等。细菌II族内含子的回巢。细胞。1998;94(4):451 - 62。
Cousineau B, Lawrence S, Smith D, Belfort M.细菌II组内子的逆转录转位。大自然。2000;404(6781):1018 - 21所示。
Coros CJ, Landthaler M, Piazza CL, Beauregard A, Esposito D, Perutka J,等。逆转录转位策略Lactococcus lactis噢。LtrB II组内含子由宿主身份和细胞环境决定。中国生物医学工程学报,2005;26(2):529 - 529。
张晓明,张志刚,张志刚,张志刚,张志刚,张志刚,等。ⅱ组内含子返巢过程中宿主功能的激活与修复机制研究。基因开发,2005 Oct;19(20): 2477-87。
Coros CJ, Piazza CL, Chalamcharla VR, Belfort M.突变筛选显示RNase E作为II族内含子逆转录的沉默者大肠杆菌.RNA。2008年10月,14日(12):2634 - 44。
黄涛,陈志强,陈志强,陈志强,等。II组内含子-核糖体结合保护内含子RNA不被降解。RNA。2013; 19(11): 1497 - 509。
Belhocine K, Plante I, Cousineau B.缀合介导Ll的转移。LtrB II族内含子在不同细菌种类之间。中国微生物学杂志,2004;51(5):1459-69。
李志强,任志强,李志强,等Lactococcus lactis染色体性别因子促进Ll的传播。LtrB族II内含子。中华微生物学杂志,2005;37(3):344 - 344。
Mills DA, McKay LL, Dunny GM.乳酸球菌中涉及pRS01共轭转移的II组内含子剪接。中国生物医学工程杂志,1996;29(6):531 - 531。
LaRoche-Johnston F, Monat C, Cousineau B.细菌II组内含子的近期水平转移、功能适应和传播。中国生物医学工程杂志,2016;16(1):223。
李文杰,李志强,李志强。基因水平转移中偶联和逆转录转座因子的相互作用。公共科学图书馆。2014;10(12):e1004853。
王文杰,王文杰,王文杰,等。乳酸菌的全基因组序列乳酸乳球菌。lactisIL1403。基因组学报,2001;11(5):731-53。
Maguin E, Prévost H, Ehrlich SD, Gruss A.乳酸球菌和其他革兰氏阳性细菌的有效插入突变。中华微生物学杂志,1996;17(3):332 - 332。
Mills DA, Choi CK, Dunny GM, McKay LL。区域遗传分析乳酸乳球菌亚属。lactis质粒pRS01参与共轭转移。应用环境微生物学。1994;60(12):4413-20。
Mills DA, Manias DA, McKay LL, Dunny GM. II组内含子的归巢乳酸乳球菌亚属。lactisML3开始。中华微生物学杂志,1997;26(4):344 - 344。
Qu G, Piazza CL, Smith D, Belfort M. II组内含子通过mRNA靶向抑制细菌共轭松弛酶的表达。eLife。2018; 7: e34268。
Dale JL, Beckman KB, Willett JLE, Nilson JL, Palani NP, Baller JA,等。综合功能分析粪肠球菌在菌株OG1RF中使用一个有序的,序列定义的插入突变集合的核心基因组。mSystems。2018;3 (5): e00062-e00018 msystems / 3/5 / msys.00062 - 18.原子。
Romero DA, Klaenhammer TR.插入序列IS946的表征,一种Iso-ISS1元素,从结合乳酸球菌质粒pTR2030分离得到。中华微生物学杂志,1990;17(8):451 - 451。
Galperin MY, Makarova KS, Wolf YI, Koonin EV。扩大微生物基因组覆盖范围,改进COG数据库中的蛋白质家族注释。核酸工程学报,2015;43(D1): D261-9。
Wagih O, Usaj M, Baryshnikova A, VanderSluis B, Kuzmin E, Costanzo M,等。SGAtools:基于阵列的基因交互筛选的一站式分析和可视化。中国生物医学工程学报,2013;41(W1): W591-6。
汪培林,王培林,王晓明,王晓明。基于数据集成的网络可视化研究。生物信息学。2011;27(3):431 - 2。
肖晓明,王晓明,王晓明,等。STRING v10:蛋白质-蛋白质相互作用网络,整合在生命树上。核酸工程学报,2015;43(D1): D447-52。
Coros CJ, Piazza CL, Chalamcharla VR, Smith D, Belfort M.全球监管机构编排II组内含子逆转录。生物化学学报。2009;34(2):250-6。
Rieder G, Merrick MJ, Castorph H, Kleiner D. hisF和hisH基因产物在组氨酸生物合成中的作用。中国生物化学杂志,1994;69(20):14386-90。
薛x, Tomasch J, Sztajer H, Wagner-Döbler I. RNA聚合酶的delta亚基RpoE是一种全局调节因子变形链球菌环境适应。中国生物医学工程学报,2010;29(2):319 - 319。
德赛BV,莫里森DA。转型链球菌引起的肺炎:日食复合体的形成coiA突变提示CoiA参与基因重组。中国生物医学工程学报,2007;29(4):344 - 344。
李志强,李志强,李志强,等大肠杆菌矩形的蛋白质。中国生物医学工程学报,1998;29(4):344 - 344。
阮B, Söll D.细菌YbaK蛋白是一种Cys-tRNA箴和Cys-tRNA半胱氨酸deacylase。中国生物医学工程学报,2005;29(4):344 - 344。
Sergeeva OV, Bogdanov AA, Sergiev PV。我们对核糖体RNA甲基化有什么了解大肠杆菌?Biochimie。2015;117:110-8。
王志强,李志强,王志强,等。假尿嘧啶甲基转移酶的鉴定大肠杆菌.RNA。2008; 14(10): 2223 - 33所示。
err, Leppik M, Liiv A, Remme J. 23S rRNA修饰酶RlmH和RluD的特异性和动力学。RNA。2010; 16(11): 2075 - 84。
Purta E, Kaminska KH, Kasprzak JM, Bujnicki JM, Douthwaite S. YbeA是m3甲基转移酶RlmH,靶向23S rRNA中的核苷酸1915。RNA。2008; 14(10): 2234 - 44。
Rose RE, Quinn R, Sayre JL, Fabris D.在全转录组水平上分析核糖核酸修饰:向基于ms的外延转录组学迈出了一步。RNA。2015; 21(7): 1361 - 74。
小林K, Ehrlich SD, Albertini A, Amati G, Andersen KK, Arnaud M,等。至关重要的枯草芽孢杆菌基因。自然科学进展,2003;21(3):344 - 344。
García-Rodríguez FM, Neira JL, Marcia M, Molina-Sánchez MD, Toro N. II组内含子编码蛋白通过β-滑动钳与细胞复制机制相互作用。中国生物医学工程学报,2019;47(14):7605-17。
Ichiyanagi K, Beauregard A, Belfort M.细菌II组内含子倾向于逆转录转位进入质粒靶。自然科学进展,2003;21(3):344 - 344。
Arraiano CM, Andrade JM, Domingues S, Guinote IB, Malecki M, Matos RG,等。RNA加工和降解在基因表达控制中的关键作用。中国生物医学工程学报,2010;34(5):883-923。
包瑞德A, Curcio MJ, Belfort M.逆转录转座因子与宿主间的取与给。科学通报。2008;42(1):587-617。
古蒂杰。限制逆转录转座子:综述。暴徒的DNA。2016; 7(1): 16。
Cosby RL, Chang N-C, Feschotte C.宿主-转座子相互作用:冲突、合作与合作。基因工程学报,2019;33(17-18):1098-116。
Koonin EV, Makarova KS, Wolf YI, Krupovic M.移动遗传元素和宿主防御系统的进化纠缠:租用枪支。科学通报。2020;21(2):119-31。
李国强,李志强,李志强,等。检验原始真核细胞出现的逆转录因子入侵假说。中国生物工程学报,2018;29(4):344 - 344。
Ahl V, Keller H, Schmidt S, Weichenrieder O.核糖体失速构象中Alu RNP的反转录转位和晶体结构。中国生物医学工程学报。2015;30(5):715-27。
杜塞AJ,威鲁兹JE,三吉T,刘Y,莫兰JV。LINE-1逆转录转位需要一个3 ' poly(A)束。中国生物医学工程学报。2015;30(5):728-41。
戈克斯J, Nekrutenko A,泰勒J,银河TT。Galaxy:支持生命科学中可访问、可复制和透明的计算研究的综合方法。中国生物医学工程学报。2010;11(8):R86。
Langmead B, Trapnell C, Pop M, Salzberg SL.人类基因组短DNA序列的超快和内存高效对齐。中国生物医学工程学报。2009;10(3):R25。
公鸡PJA,田CJ,后藤N,豪雅ML,大米PM。sanger FASTQ文件格式用于具有质量分数的序列,以及Solexa/Illumina FASTQ变体。中国生物医学工程学报,2010;38(6):1767-71。
李华,Handsaker B, Wysoker A, Fennell T, Ruan J, Homer N,等。序列对齐/映射格式和SAMtools。生物信息学。2009;25(16):2078 - 9。
Diaz A, Park K, Lim DA, Song JS。ChIP-seq的归一化,偏差校正和峰值调用。中华生物医学杂志2012;11:3。
昆兰AR,霍尔IM。BEDTools:用于比较基因组特征的一套灵活的实用程序。生物信息学,2010,26(6):841 - 2。
McGinnis S, Madden TL. BLAST:一套强大而多样的序列分析工具的核心。核酸决议2004;32 (Web服务器):W20-W25。
Toft C, Williams TA, Fares MA。变形菌与昆虫共生后的全基因组功能分歧通过一种新的计算方法解开。计算机科学进展。2009;5(4):e1000344。
Waldern J, Schiraldi NJ, Belfort M, Novikova O.细菌II组内含子基因组邻居反映生存策略:隐藏和劫持。中国生物医学杂志,2020;37(7):1942-8。
阿特舒尔SF,吉什W,米勒W,迈尔斯EW,李普曼DJ。基本的本地对齐搜索工具。中华分子生物学杂志,1990;215(3):403-10。
唐彻瓦,李志强,李志强。Cytoscape StringApp:蛋白质组学数据的网络分析和可视化。中国生物医学工程学报,2019;18(2):623-32。
细胞景观:生物分子相互作用网络集成模型的软件环境。基因组学报,2003;13(11):2498-504。
Schneider CA, Rasband WS, Eliceiri KW. NIH图像到ImageJ: 25年的图像分析。自然科学进展。2012;9(7):675 - 5。
董晓霞,李志强,李志强,李志强,等。RNP基因ⅱ组内含子的结构修饰。中国生物医学工程学报,2018;46(16):8542-56。
甘戈帕德哈伊JP,姜松,保卢斯在体外以绿色荧光蛋白为指示剂的蛋白剪接抑制剂筛选系统。中国医药科学,2003;29(10):344 - 344。
DNA和RNA的制备和酶解用于质谱分析。见:酶学方法。爱思唯尔。1990:782 - 90。
Rose RE, Pazos MA, Curcio MJ, Fabris D. RNA转录后修饰的整体外延组学分析是研究应激反应外延组学的有效工具。Mol细胞蛋白质组学。2016;15(3):932-44。
陈志伟,陈志伟,陈志伟,等。高分辨率离子迁移谱-同分异构体/等压核糖核苷酸变体的质谱。质谱仪。2020;55(2):e4465。
McIntyre W, Netzband R, Bonenfant G, Biegel JM, Miller C, Fuchs G,等。阳性RNA病毒揭示了感染过程中细胞和病毒外延转录组的复杂性和动态。中国生物医学工程学报,2018;46(11):5776-91。
确认
作者感谢马特·斯坦格(Matt Stanger)展示了各种各样的数字。此外,他们还要感谢为该项目做出贡献的众多研究人员,包括Dhanushki Samaranayake博士和本科生研究人员:Jackie Carey、Maggie Huang、Anya Joynt、Arianna Lechsinska、Jonathan Lee、Mary Njie、Seymour Suncin、Celeste Verdolivo和Hanifah Yasin
资金
这项工作得到了美国国立卫生研究院(NIH) GM039422和GM044844对MB的资助。
作者信息
作者及隶属关系
贡献
JW, MB和ON构思了这项研究。JW、CLP、EJB、MB和ON起草了手稿。JW, DS, CLP, EJB和ON进行了实验。JW, NS和ON对计算分析做出了贡献。JW, DS, CLP, EJB, RN, DF, MB, ON对数据进行分析。RN和DF贡献了重要的思想和反馈。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:补充数字和表格
.此文件包含补充数据。S1-S18而且补充表S1-S5.
附加文件2:。
是S1插入映射。该文件包含IS的坐标和基因信息S1本研究产生的突变体。
附加文件3:。
RTP-up突变体列表和汇总数据。该文件列出了所有已验证的单插入RTP-up突变体,以及它们的插入位置、突变层排名和COG分类的数据。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
沃尔德恩,j.m.,史密斯,D.,皮亚扎,C.L.et al。甲基化rRNA作为宿主防御猖獗的II组内含子反转置。移动DNA12, 9(2021)。https://doi.org/10.1186/s13100-021-00237-z
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s13100-021-00237-z
关键字
- 可移动遗传元素
- 反转位子活动
- 核糖体
- RNA拼接