摘要
背景
本体匹配应该有助于FAIR数据的互操作性(可查找、可访问、可互操作和可重用)。多个数据源可以使用不同的本体来注释其数据,从而产生对动态本体匹配服务的需求。在这个实验研究中,我们评估了本体匹配系统在罕见疾病领域的真实应用环境中的性能。此外,我们提出了一种分析顶级类的方法,以提高精度。
结果
我们包括三个本体(NCIt、SNOMED CT、ORDO)和三个匹配系统(AgreementMakerLight 2.0、FCA-Map、LogMap 2.0)。我们根据生物门户和统一医学语言系统元辞典(UMLS)的参考校准来评估匹配系统的性能。然后,我们分析了匹配类的顶级祖先,以便在不参考引用对齐的情况下检测不正确的映射。为了检测这种不正确的映射,我们手动匹配本体对的语义等价的顶级类。AgreementMakerLight 2.0、FCA-Map和LogMap 2.0在biopportal的f1得分分别为0.55、0.46、0.55,UMLS的f1得分分别为0.66、0.53、0.58。使用基于投票的共识调整可以全面提高性能。对手动创建的顶级层次结构映射的评估显示,平均90%的映射类属于匹配的顶级类。
结论
我们的研究结果表明,根据我们的评估,包含的本体匹配系统自动生成了适度准确的映射。当没有参考对齐可用时,映射的层次分析似乎很有前途。总之,该系统显示出作为查询FAIR数据的本体匹配服务的一部分实现的潜力。未来的研究应侧重于开发用于评估此类映射服务的映射的方法,从而在FAIR数据生态系统中实现它们。
背景
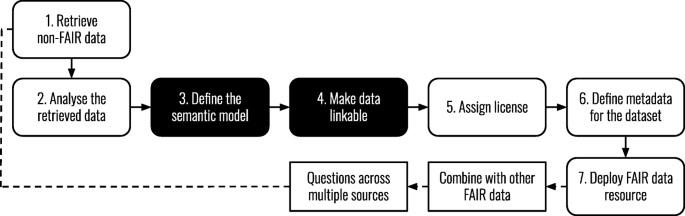
数据的生成、收集和使用对科学研究至关重要。因此,研究数据的共享和重用变得越来越重要,导致了诸如欧盟地平线2020计划的指导方针,要求开放获取科学出版物和研究数据[1].2016年,一群研究人员和其他利益相关者,对研究数据的可寻性和重用感兴趣,发布了一套原则,宣传研究数据对机器和人类的重用[2].这些原则被作为科学数据管理和管理的FAIR指导原则(可发现、可访问、可互操作和可重用)提出。使数据公平的过程通常被称为FAIRification, GO FAIR倡议将其描述为七个步骤(图1)。1) [3.].
FAIRification过程。改编自GO FAIR [3.].本研究主要关注第3步(定义语义模型)和第4步(使数据可链接)
当使数据成为FAIR时,需要定义数据的语义模型(步骤3),并且需要使数据具有可链接性(步骤4),对于这两种本体都很有用,因为它们提供了关于某些领域的某种形式的共识。使用本体对于提高数据的语义互操作性非常重要[4].不同的本体可以包含描述相同领域或概念的类,这使得它们重叠[5].如果研究人员使用不同的本体来描述同一个类,那么使数据互操作变得更加具有挑战性。例如,“囊性纤维化”类在多种生物医学本体论中都有描述。对生物医学门户中生物医学本体之间的术语重叠和术语重用的系统分析发现,本体之间类重用的重叠比例约超过25%,而重用比例不到9% [5].另一篇论文研究了生物医学本体中逻辑公理的重用,发现123个本体中有49个不应用任何类型的重用[6].因此,注释数据本身并不意味着可互操作的数据,因为需要匹配不同的本体。本体匹配的目的是通过匹配来自不同本体的语义相关的类,实现本体之间的互操作,从而实现本体之间的对齐。当数据(集)使用不同的本体进行注释时,本体匹配可以使数据具有互操作性[7].一个特定的社区可能定义整个社区应该遵守的标准,也可能不定义,包括特定的本体。然后本体匹配将用于实现社区内部和社区之间的互操作性,因为不同的社区可能使用不同的标准。本研究的背景和动机源于欧洲罕见病联合计划(EJP RD),这是一个大型的泛欧洲项目,专注于为罕见病研究和护理创建一个生态系统[8].EJP RD的目标之一是建立一个符合fair的数据发现平台,该平台描述罕见病资源,并使研究人员能够查询来自不同位置的多个资源的数据。这些源可能使用不同的本体,这就需要本体匹配来支持源之间的查询。
问题陈述
许多匹配技术和系统已经开发出来,但对现实应用的研究一直很匮乏[9].本体匹配应该通过提供使用来自不同本体的类注释的数据源之间的互操作性来促进FAIR数据。匹配本体不是一次性的任务,因为数据集和本体都会随时间变化。因此,需要动态本体匹配服务[7].尽管本体匹配背后有活跃的研究团体,但尚不清楚匹配系统和技术在FAIR数据环境下将如何执行。因此,现有本体匹配系统的有效性和性能应结合遵循FAIR指导原则的实际应用进行评估。
目标和研究问题
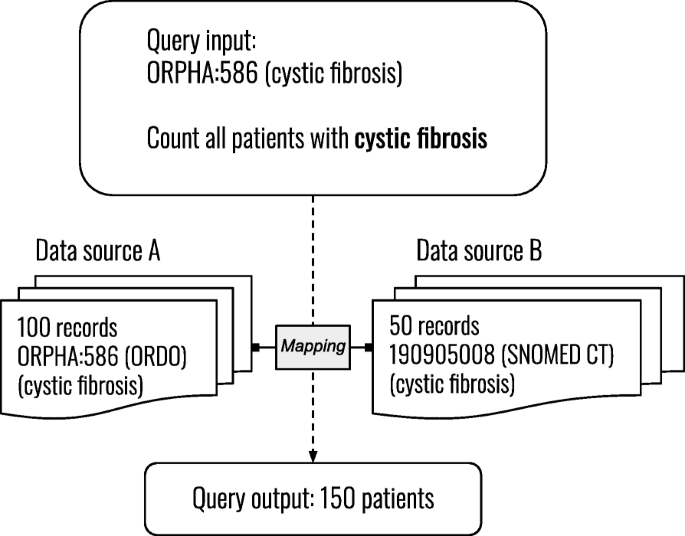
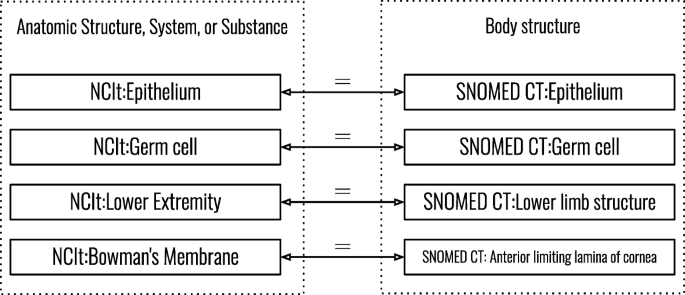
本实验研究旨在在符合fair的数据发现平台的背景下,对与罕见疾病领域相关的本体进行本体匹配系统的性能测量。通过这样做,本研究旨在为FAIR数据的本体匹配系统的使用做出贡献。数字2描述这样的数据发现平台如何从本体匹配中受益。对于这个用例,系统还应该能够确定两个匹配的类属于同一个类别,例如,通过说类“囊性纤维化”和“多发性硬化症”都是一种“疾病”。因此,我们分析匹配类的顶级层次结构,以检测其顶级祖先在语义上等价的类。将讨论以下研究问题:
- 1
在罕见病研究领域,用于暴露本体之间映射的自动化本体匹配系统的性能如何?
- 2
在关注查询分布式数据的fair相关项目中,当前可用的本体匹配系统在多大程度上是有用的?
预赛
基本的定义
我们采用的基本定义来自[10],根据本工作的需要对其进行了修改。本体匹配是寻找不同本体类之间关系的过程。关系的例子是等价(≡),包容(\ \ sqsubseteq \ ()),更通用(≥),或更具体(≤)[10].匹配过程输出对齐一个包含本体类之间的映射O而且O”.一个映射米是两个本体的不同类之间的关系(根据对齐)。有些论文把映射称为通信。形式上,映射可以由一对本体定义为三元组O而且O”和一组映射关系\(θ= \ \{\枚\ sqsubseteq \ leq \组\}\):
在哪里e∈O,e”∈O”而且r∈Θ.此外,映射可以包括元数据,如置信度值和标识符。等价映射对这项研究很有意义,例如,匹配系统应该能够检测出两类代表同一类型的疾病。
本体匹配技术的分类
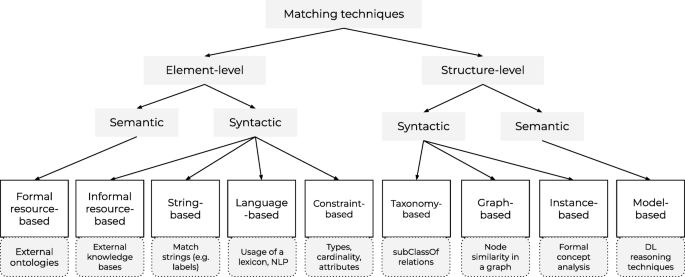
可以使用基于输入解释和粒度组织技术的模型对本体匹配技术进行分类[10].数字3.显示了这个分类模型的改编版本。匹配系统的粒度可以由两个级别定义:元素级和结构级。元素级匹配技术关注一个类而不考虑它与其他类的关系,结构级匹配技术关注本体结构中的一个类。在每个级别上,模型都对语义匹配技术和语法匹配技术进行了区分。语法匹配技术只使用类的信息而不进行解释,例如文本标签或同义词。语义匹配技术通过使用推理机或外部资源为结构信息添加意义。匹配系统的输入可以用九种技术来解释,如图所示。3.包括实现示例。本研究中使用的本体匹配系统应用了其中的一种或多种技术。
匹配技术的分类。改编自[10].对于每个类别,给出了一个可能的实现示例
方法
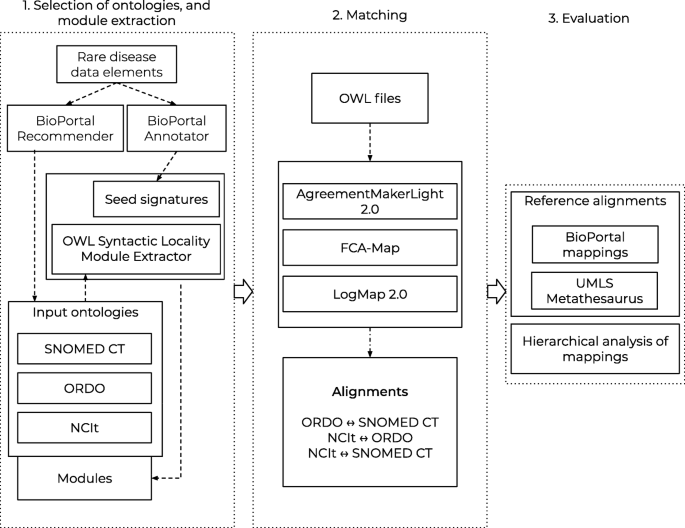
我们进行了一项实验研究,在符合fair的数据发现平台的背景下,测量与罕见疾病领域相关的本体匹配系统的性能。首先,我们使用两个参考对齐来评估由匹配系统生成的对齐。此外,我们还分析了映射中类的顶级层次结构。具体步骤如下:(1)选取相关生物医学本体,提取模块;(2)生成本体对之间的对齐;(3)对得到的排列进行两部分评估。数字4展示了所进行的实验的概述。开发使用Java版本8,数据分析使用R版本4.0.1 [11].
本体的选择
为了选择与罕见病领域相关的本体,我们使用一组与罕见病相关的关键词作为生物门户推荐器的输入[12].为了对其输入进行注释,推荐者使用生物门户注释器[13].输入关键字是从罕见病注册表的通用数据元素集中提取的(元素和编码名称列中的项目)[14],以及孤儿院罕见病的分类(所有类别,每类别随机一种疾病)[15].项,即一个或多个单词,可以有多个注释。完整的数据项目列表可在附录中找到。推荐者使用默认配置运行,并从列表中选择前两个本体,即SNOMED临床术语(SNOMED CT,国际版发布31-01-2020)[16],以及《国家癌症研究所同义词词典》(NCIt,版本20.02 2d) [17].SNOMED CT是一个主要的生物医学本体,包含超过35万个类,包括罕见疾病的内容。NCIt还涵盖了生物医学领域,有超过15万门课程。最后,我们添加了孤立罕见病本体(ORDO,版本2.9.1)作为第三个本体,因为它专门针对罕见病领域,包含近15,000个类。所有的本体论都以网络本体论语言(OWL)格式提供,该格式是由万维网联盟(W3C)维护的标准[18].
模块提取
我们从每个本体中提取一个模块,它是原始本体的一个子集。模块只允许处理本体中基于感兴趣类的相关信息[19].这些更小的子集允许我们使用更少的计算资源来运行实验,并使理解和浏览本体的结构更容易。OWL API包括一个语法位置模块提取器[20.].这个模块提取器使用所谓的种子签名作为输入,从本体中提取子集。这个种子签名是来自模块所基于的父本体的类列表。模块提取器可以提取三种类型的模块:星型、底部和顶部。顶层模块包含种子签名中的所有子类和类的(子)属性,底层模块则相反,它包含超类和(超)属性。星型模块通过包含顶部和底部模块的交集来结合这两种策略。我们从NCIt、ORDO和SNOMED CT中提取了一个星形模块。种子签名包含生物门户注释器返回的罕见病数据项的注释。为了确保模块包括原始本体的整个顶层层次结构,种子签名包括那些注释类的所有祖先。在本文的其余部分中,星型模块将被称为“模块”。
匹配系统和校准
本体匹配系统从参加2019版本体对齐评估计划(OAEI)的系统中选择[21].自2004年以来,OAEI一直是本体匹配系统性能评估的年度重复活动。事件由多个轨道组成,每个轨道处理各种本体和匹配任务。参与“大型生物医学本体论”和“疾病和表型”轨道的系统是特别感兴趣的,尽管来自所有轨道的系统都有资格被纳入。疾病和表型跟踪包括与ORDO匹配的任务[22].为了保证我们实验中系统之间的多样性,根据Euzenat等人的分类,我们选择了采用不同匹配技术的系统(图1)。4) [10].此外,我们只选择源代码在公共存储库中可用的系统(以确保我们可以使用它们)。这导致包含三个系统:协议makerlight 2.0 (AML) [23,24], fca地图[25]及LogMap 2.0 (LogMap) [26,27].表格1显示这些系统实现了图中所示的大多数匹配技术。4.
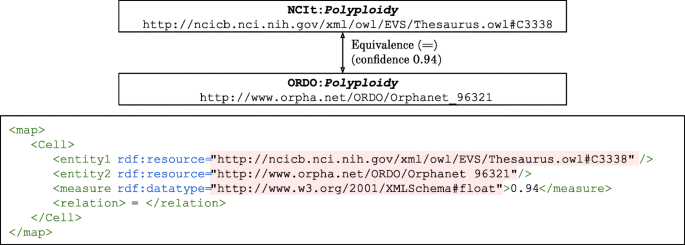
选中的匹配系统以默认配置运行,系统参数不做任何更改。匹配系统的输出,即校准,以校准API提供的一般格式保存[28].每次运行分配64GB的RAM。匹配系统在匹配过程中不需要任何用户输入,即提供自动本体匹配。使用本体对作为输入:ORDO-SNOMED CT, NCIt-ORDO, NCIt-SNOMED CT(注意匹配A到B相当于匹配B到A)。所有对齐都包含成对等价映射,并包含每个类的URI。数字5展示了NCIt和ORDO之间的映射示例。
映射的层次分析
生物医学本体的词汇标签中含有大量的信息,因此本体匹配系统通常主要使用词汇匹配技术[29].但是,包含来自不同顶级类的类的映射可能是错误的,即使这些类在词法上有相似的标签。例如,两个类分别标记为骨折第一个是这个类的后代临床发现第二个是身体结构.本体的每个顶级层次结构都包含类型相似的类,顶级类的每个后代都与其祖先共享IS-A关系。本研究的用例认为查询系统应该能够分辨来自不同本体的两个类属于同一个类别。例如,一个系统应该能够分辨两个类别都是疾病。因此,我们通过比较匹配类的顶级层次结构来分析匹配系统的映射。NCIt-ORDO、NCIt-SNOMED CT和ORDO-SNOMED CT的顶级类之间的映射是手工创建的。我们通过检查真正映射的顶级层次结构来创建这些手动映射(图。7),基于参考对准。关于引用对齐的更多细节将在下一节中给出。类描述还用于确定是否应该匹配两个顶级类。手动匹配的顶级类在语义上是等价的。数字6显示如何创建这种手动映射的示例。
绩效评估
我们使用两个参考对齐来评估由匹配系统生成的对齐。分别对每个参考比对线进行评估。第一个参考对齐包含来自biopportal的映射,第二个包含基于统一医学语言系统元辞典(UMLS)的映射[30.].选择这两者是因为它们分别被用作OAEI疾病和表型跟踪和大型BioMed跟踪的参考校准。通过删除模块中不存在的类之间的映射,本体模块的引用对齐从它们的完整对应模块派生而来。
BioPortal
bio - portal参考比对被认为是基线;在大多数情况下高度不完整的对齐[22].ORDO、NCIt和SNOMED CT的生物门映射如下skos: closeMatch基于词汇OWL本体匹配器(LOOM)的映射[31].LOOM是一种简单的字符串匹配算法,它比较两种本体中类的首选名称和同义词。使用biopportal API检索生物门户映射。
uml metathesaurus
基于umls的参考对齐被认为是银标准;不一定完全或正确的对齐[22,32].UMLS mettathesaurus使用一个代码对所有语义上等价的类进行分组:概念惟一标识符(CUI)。一个类可以有多个CUI代码。参考对齐是从UMLS mettathesaurus(版本2020AA)的一个子集中局部提取的,Jimenéz-Ruiz等人也使用了这种方法。[33].该子集是使用UMLS的MetamorphoSys工具通过检索MRCONSO获得的。RRF文件,并使用MySQL Community Server 5.6.48版本在本地安装。通过首先获取每个本体中每个类的所有可用的gui来检索成对映射。然后,包含至少一个对应CUI的本体A和本体B的所有类作为引用对齐中的映射。ORDO没有出现在UMLS中,但是在本体中包含了作为注释的CUI代码映射。因此,ORDO CUIs不是从UMLS中检索的,而是直接从本体本身中检索的。
性能指标
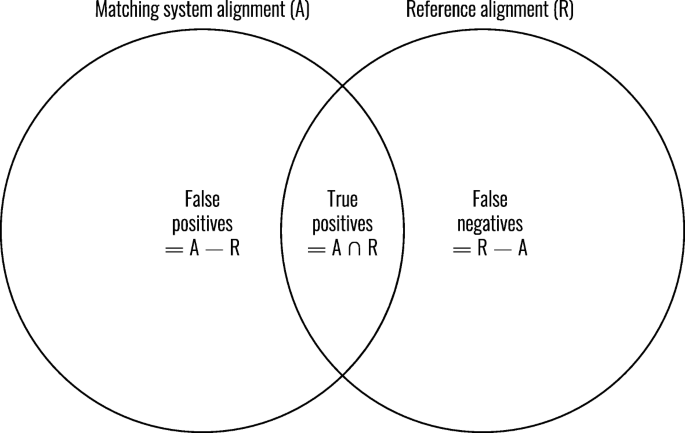
我们通过将每个映射分类为真阳性(TP,在对照比对中都存在)、假阳性(FP,只在对照比对中存在)或假阴性(FN,只在对照比对中存在)来评估比对(见图)。7).真实的底片不包括在内,因为没有黄金标准可以包含所有可能的正确映射。
使用生物门户和UMLS mettathesaurus参考比对进行评估的类别。改编自[10]
随后,我们通过计算精度(Eq。1),回忆(Eq。2)和F-measure (F1-score) (Eq。3.).精度显示在校准中被分类为真阳性的映射的比例。召回显示在引用对齐中同时存在于对齐中的映射的比例。f测量通过计算它们的谐波平均值来结合精度和召回率。对于精确度、回忆率和f测量,1分意味着完美的结果,0分是最差的结果。首先在不考虑分层分析的情况下评估映射,然后考虑分层分析。对于后者,如果它们的顶级层次结构类没有出现在手动创建的映射集中,则假阳性映射(不在UMLS或biopportal中)将被标记为不正确。我们重新计算精度和f测度后,丢弃了那些不正确的映射从校准。最后,我们根据多数投票生成共识对齐并对这些对齐进行评估。通过选择由两个或两个以上系统(投票≥2)和三个系统(投票=3)选择的映射,生成所有系统之间的一致对齐。 Additionally, we added consensus alignments of three combinations of matching system pairs (vote=2).
结果
罕见病数据元素和模块
从罕见病登记和孤儿罕见病分类的通用数据元素集中提取了117个数据项。生物门户推荐器用ORDO类注释了42%的输入,用SNOMED CT类注释了52%,用NCIt类注释了65%。种子签名包含471个SNOMED CT类、74个ORDO类和547个NCIt类。表格2显示所提取模块的详细信息。这些模块包含了整个本体类和公理总量的0.4-2%。
比对
在本体对ORDO-SNOMED CT、NCIt-ORDO和NCIt-SNOMED CT之间建立对齐。每个匹配系统总共使用6个OWL文件作为输入,每个本体(模块和整个本体)两个文件。这导致了总共18个校准,每个匹配系统6个。所有对齐都包含具有等价关系的映射。在运行时间方面,AML和LogMap最快,NCIt-SNOMED CT的整个本体对齐在几个小时内就完成了。FCA-Map较慢,NCIt-SNOMED CT需要6-8小时。表格3.显示整个本体和模块的每次对齐的映射数。
评价:UMLS mettathesaurus和BioPortal
表格4显示了从biopportal和UMLS mettathesaurus中提取的参考比对的映射数量。所有来自UMLS mettathesaurus的参考比对都比来自biopportal的包含更多的映射。NCIt-ORDO和NCIt-SNOMED CT参考对齐之间的重叠最大,具有加权重叠,占每个参考对齐映射数的差异,分别为45%和57%(整体本体)。ORDO-SNOMED CT参考对齐有最小的重叠,即14%(模块)和25%(整体本体)。
表格5显示了整个本体的平均评价结果。对于所有的本体对,通过biopportal评估的校准召回率高于通过UMLS评估的校准召回率。当使用基于umls的参考对齐时,ORDO-SNOMED CT对齐的精度(0.45精度)高于bio - portals的参考对齐(0.28精度)。ncti - ordo和ncti - snomed CT的情况相反,生物门的精度评分高于UMLS。AML对生物门户的f1分最高(0.66),所有匹配系统对生物门户的f1分总体高于UMLS。LogMap对UMLS的精确度(0.47)高于对biopportal的精确度(0.45)。AML对BioPortal的精确度(0.54)高于UMLS的精确度(0.47)。在所有本体对和系统中,NCIt-SNOMED CT的查全率和查准率最高。
表格6显示模块的平均结果。生物门户的所有本体对的召回率都高于UMLS,这与整个本体的结果相对应。对于所有本体对,UMLS精度均高于bio - portal精度。总体而言,所有匹配系统UMLS的f1得分都高于生物门户。
每个匹配系统(即所有本体对)和本体对(即所有匹配系统)的结果都取平均值,以便于解释,而且因为个体差异通常很小。参见附加文件1对于所有的个体结果。
利用映射的层次分析进行评价
表格7显示手动创建的顶层层次结构映射。我们在ORDO-SNOMED CT的顶级类之间创建了三个映射,为NCIt-ORDO创建了六个映射,为NCIt-SNOMED CT创建了13个映射。表格8显示分析结果。在对齐中,平均10%的映射(整个本体)包含的类的顶级层次结构没有出现在手动顶级映射集中。对于biopportal和/或UMLS的真正阳性映射被保留在校准中。平均而言,对齐中4.6%的映射包含的类的顶级层次结构没有出现在手动映射集中,并且为假阳性。模块对齐中平均有19%的不正确的层次结构映射,在对齐中有8.7%的映射是错误的顶层层次结构的假阳性。
去除顶级层次结构不正确的假阳性映射后,整个本体重新计算精度和F1-score的结果如表所示9.对于所有本体对和匹配系统,精确度和F1-score值在0.01到0.05之间增加。唯一的例外是FCA-Map,其bio - portal精度从0.39增加到0.45(+0.06)。表格10显示了模块的新的精度和f1得分。除NCIt-SNOMED CT精度和bio - portal的f1得分(分别为+0.29和+0.22)外,其余评分均在0 - 0.06分之间增加。
共识比对
表格11显示了基于多数投票的共识对齐的f1得分。精度和召回分数包括在附加文件1.在包含两个或更多选票的映射的所有系统之间的一致对齐导致了总体性能的提高。除了NCIt-SNOMED CT和ORDO-SNOMED CT的UMLS得分与表中的平均分相比,包含vote=3映射的对齐进一步提高了所有的m1得分5.当在匹配的系统对之间投票时(只有那些被两个系统选择的映射,投票=2),配对AML 2.0和FCA-Map的性能提高了0.03分,相比于在所有系统中使用3票的一致对齐。与Table相比,FCA-Map和LogMap 2.0一致对齐时,基于UMLS的NCIt-SNOMED CT和ORDO-SNOMED CT的f1得分增加5.从共识对齐中移除具有不正确顶级层次结构的假阳性映射并不会增加f1得分超过0.01分。
讨论
我们使用基于UMLS和生物门户的参考对齐来评估三个现有本体匹配系统的性能。此外,我们使用本体对的顶级类之间手动创建的映射分析了映射的顶级层次结构。这些实验应该有助于在FAIR数据上下文中查询分布式数据源的用例。
主要研究结果
在罕见病研究领域,用于暴露本体之间映射的自动化本体匹配系统的性能如何?
这些系统平均暴露了ORDO-SNOMED CT之间的5.726个映射,NCIt-ORDO之间的3.295个映射,NCIt-SNOMED CT之间的23.134个映射。获得的f1评分为0.55/0.66 (AML, UMLS/ bio - portal), 0.46/0.53 (FCA-Map)和0.55/0.58 (LogMap)。模块的结果与整个本体的结果相当。由于没有可用的金标准,系统的总体低精度(在0.39-0.54之间)和高召回(在0.64-0.96之间)表明(自动)评估映射的正确性确实具有挑战性。该系统检索了参考校准中的大部分映射,但也暴露了许多额外的映射,因此精度较低。已知两种参考对准都是不完整的(银标准和基线),需要进一步的研究来评估系统返回的其他映射是否正确。OAEI 2019大型生物医学跟踪(SNOMED CT-NCIt大片段任务[34])是最接近于用作解释性能的参考,因为OAEI的其他轨道和任务使用其他本体或参考对齐。使用基于umls的参考对齐(不一致的映射被标记为被忽略),AML获得了0.76的f1得分,FCA-Map 0.65, LogMap 0.71。这些OAEI结果更好,尽管由于参考对齐和本体版本不同,它不是一对一的比较。此外,OAEI报告使用SNOMED CT的一个大片段,导致比使用整个本体时更少的映射(18887 vs. AML的14200)。
使用一致对齐(即,由多个系统选择的映射)全面提高了性能(表11及附加文件1).正如预期的那样,选择更多的选票(vote=3,由所有系统选择的映射)会导致更高的精度和更低的召回率。在实践中,对于特定的应用程序,可以优先考虑精度而不是召回,反之亦然,选择符合这些需求的一致对齐的能力是很有用的。
在关注查询分布式数据的fair相关项目中,当前可用的本体匹配系统在多大程度上是有用的?
所有系统都能够在不需要用户干预的情况下生成对齐,这对数据查询非常重要。运行时间从几分钟到几小时不等,这取决于输入本体的大小。匹配系统暴露了本体对类之间的等价关系。图中描述的用例。2需要等价映射和自动匹配。这意味着在用例的上下文中应用AML、FCA-Map和/或LogMap将是一个明智的决定。然而,在用于查询数据的匹配服务的情况下,高精度比高召回率更重要,因此需要在验证映射的正确性方面进行额外的工作。所有系统都支持OWL本体作为机器可读的rdf文件的输入和导出对齐。这允许连接来自多个匹配系统的对齐。此外,分析匹配类的顶级层次结构可以有效地揭示来自相同层次结构的类之间的映射。表格8显示平均10%的映射具有不正确的顶层层次结构;90%是使用手动创建的映射匹配顶级层次结构的映射。例如,考虑一个查询:“计数所有患有罕见疾病的患者”,那么分层分析可以显示“囊性纤维化”是一种罕见疾病,它的记录应该被计数。层次分析可用于没有参考对齐可用的情况。最后,与使用整个本体相比,使用模块有助于更快地开发和测试匹配技术。如果速度或资源是重要因素,可以考虑使用模块而不是整个本体。此外,通过从本体中删除与应用程序不相关的内容,模块化可以用作一种结构级匹配[10].由于模块化从本体中删除了内容,应该注意到这可能会改善或恶化使用结构匹配技术的匹配系统的结果,尽管我们没有测试这个假设。
优势与局限
可以确定它的几个优点和局限性。本研究的一个优势是它使用FAIR数据用例进行本体匹配的实用方法,使用与罕见疾病领域相关的本体。一个局限性是AML、FCA-Map和LogMap并不是唯一可用的匹配系统,尽管它们涵盖了Euzenat等人定义的分类模型所指定的大多数匹配技术。[10].特别是,利用机器学习技术的系统不包括在内。同样,在罕见病领域也存在其他本体,特别是考虑到生物门户等存储库中存在大量本体。生物门户注释器和推荐器用于选择本体和创建种子签名,但也可以使用其他类似的工具。
另一个优点是使用基于罕见病数据元素的种子特征的模块。较小的模块使得在执行实验时手动评估映射更容易。此外,它还显示了在匹配服务中实现的潜力,当需要使用大型本体的较小块时,例如为了更快的运行时间。然而,由于罕见病数据元素列表没有经过验证,因此不可能从模块与整个本体结果之间得出任何额外的结论。例如,我们不知道模块中包含的类是否与罕见疾病领域的使用最相关。
使用生物门和UMLS参考校准进行评估
精确度、回忆率和f1得分被用作绩效衡量指标,因为它们被OAEI广泛知晓并使用。然而,在本体匹配的背景下,精确度和查全率都引入了一个必须提及的问题。如[35],精度和回忆率都是集合论的测量,它们不区分语义上相等但不相同的映射。因此,当一个映射没有出现在引用对齐中,根据定义它被认为是不正确的(假阳性)。语义精度和召回可以通过考虑在语义上接近引用对齐中的映射来解决这个问题。例如,当映射的类是引用对齐中对应类的超类或子类时。
据我们所知,UMLS和基于生物门户的参考校准是唯一提供多种本体之间映射的方法,包括SNOMED CT、ORDO和NCIt。我们考虑使用两种参考对齐来评估强度,因为它们都包含不同的映射,尽管它们有28 - 57%的重叠(表4).正如OAEI之前提到的,生物门户映射被认为是基线对齐[22].对于AML, bio - portal的精密度和召回率均为最高(全本体分别为0.54和0.96)。这对应的事实是,AML和生物门户(LOOM)的映射都只基于词汇技术。另一方面,从UMLS派生的映射被认为是银标准,因为Metathesaurus是由领域专家维护的。本研究中使用的UMLS参考比对的一个限制是来自ORDO的CUI代码,因为ORDO不包括在UMLS中,那些CUIs是从ORDO本身提取的。模块的biopportal f1得分平均比整个本体低15%,这可能是由于参考校准中的映射数量较少。最后,早期的研究提到UMLS参考对齐包含非相干映射[33],即包含从输入本体和映射集的联合而来的逻辑错误的映射[36].此外,在生物门映射中也发现了逻辑不连贯[37].在本研究的评估过程中,这些映射没有被移除和/或检查。
映射的层次分析
丢弃假阳性映射,其顶级层次结构类不是手动匹配的,并没有导致更高的精度得分(高达0.06分)。尽管如此,在对FAIR数据应用本体匹配时,分析匹配类的顶级层次结构是有价值的。首先,层次结构可以利用关于类起源的信息。例如,如果“肺炎”和“哮喘”都是“疾病”层次结构的一部分,它们可以被归类为“疾病”,即使由于缺乏参考对齐,这些类别本身是否可以互换使用仍然是未知的。这在查询多个数据源的数据时很有用(例如图。2).此外,有些类出现在多个映射中(一个类映射到多个其他类),在这种情况下,层次结构分析能够检测到不正确的映射。
我们的顶级类之间的手动映射列表可能不完整,这是层次分析的一个限制。此外,我们的方法需要手动匹配顶级类,因此不能自动完成。然而,即使是大型本体也往往只有很少的顶级类(例如,SNOMED CT和NCIt都有19个顶级类)。表格12显示四个可能不正确的映射(由匹配系统返回)及其顶级层次结构。表中的第一个映射12是软组织而且软组织紊乱,第一个是指软组织的解剖结构,第二个是指软组织的紊乱。第二个例子是动脉瘤性骨囊肿而且动脉瘤性骨囊肿,其中标签在词法上是相同的。但是,第一个是指疾病,第二个是指身体结构。最后一个例子是细胞增殖与增生,顶级层次结构显示第一个类是生物过程第二个是身体结构.现在,这个例子放大了领域专家评估映射的重要性。增生是细胞增殖的结果[38],因此可以认为映射是正确的,这取决于应用程序。此外,人工检查对齐后,我们发现真正映射,但根据我们的手动映射,其顶层层次是不正确的。这些映射没有被标记为不正确,因为它们包含在任何一个引用对齐中。然而,这些映射可能表明在引用对齐中存在不正确的映射,这超出了本文的范围。
共识比对
我们使用共识对齐比单独对齐产生了更好的结果。OAEI的某些轨道也使用共识对齐[22,39].Harrow等人提到,共识对齐只比较匹配系统之间的执行情况。当多个系统可以找到相同的、不正确的映射时,假阳性仍然可能发生。此外,正确的映射可能只在一个系统中找到,因此不会包含在一致对齐中。
与其他工作的关系
本体论匹配系统的评价涉及早期对疾病和表型本体论以及大型生物医学本体论(large BioMed)的匹配研究,两者都是OAEI [22,40].除了biopportal基线参考校准,基于投票机制的共识校准(多个系统返回相同的映射)和手动规划的映射被用于评估匹配系统。OAEI的大型生物医学本体跟踪使用UMLS作为参考对齐,这是基于一项早期工作,该工作从UMLS mettathesaurus [33].此外,我们的工作与2020年发表的一篇论文有关,该论文提出了使数据公平的通用工作流[41].当处理多个本体时,本体匹配系统应该包括在FAIRification工作流中,并且需要某种形式的类自动匹配。
未来的研究
我们探讨了在FAIR数据上下文中使用本体匹配系统的用例,并表明现有的本体匹配系统具有在此类环境中实现的潜力。一个尚未解决的问题是,如何评估映射对其特定应用程序是否有用或正确。获得完整的参考对齐是一项具有挑战性的任务,而且这样的对齐并不容易获得。在FAIR数据环境中的动态匹配服务中使用映射的(自动)评估将变得非常重要。因此,未来的研究应该集中于开发评估映射的方法,这些映射可以被这种匹配服务使用。此外,应该考虑没有参考线可用的情况。这些开发应该由特定的用例驱动。除此之外,将顶层层次结构分析作为本体匹配系统的附加方法将是有益的。未来的研究可以集中在如何将该方法集成到现有的(模块化)系统和工作流中。例如,AgreementMaker提供了一个可扩展的体系结构,可以包含我们的方法。 We should acknowledge that additional matching methods, utilizing the structure or logic of ontologies, are not limited to top-level hierarchies. Future research could focus on discovering and analyzing other methods that have not yet been implemented by existing matching systems. Lastly, our experiment did not include matching systems based on a machine learning approach. Earlier research has demonstrated that an approach based on representation learning is effective at ontology matching [42].因此,研究基于机器学习的系统可能具有附加价值。最近添加到匹配与评估工具包(MELT)框架中的机器学习扩展可以帮助这方面的工作[43].MELT还提供了所谓的过滤器,其中一个是分类器,可以训练它将映射划分为正确或不正确。这种滤波器可以用来提高对准的精度,给定它可以用正映射和负映射训练。对于后者,需要一个黄金标准,或者需要手动创建负映射。
结论
我们探讨了用于罕见病领域的本体匹配系统的性能,并在一个FAIR数据用例的上下文中分析了映射的顶层层次结构。我们的结果表明,根据我们的评估,所有三个系统(AgreementMakerLight 2.0、FCA-Map、LogMap 2.0)都能够自动生成适度精确的映射。我们发现,评估系统的性能是具有挑战性的,因为正确和完整的参考校准并不总是可用的。不完整或不正确的引用对齐阻碍了映射的评估,从而可能限制查询分布式数据的系统的可靠性。我们提出了一种在这种情况下似乎很有前途的映射层次分析,因为它不需要引用对齐。总而言之,这项工作将激发人们对在用于查询FAIR数据的动态服务中实现已演示的本体匹配系统和顶层层次分析的兴趣。
附录
数据和材料的可用性
支持本文结论的数据集可以在GitHub存储库中找到,https://github.com/PhilipvD/2021-jbiomedsem-ontology-matching-paper.
缩写
- AML:
-
AgreementMakerLight 2.0
- 崔:
-
概念唯一标识符
- EJP采访:
-
欧洲罕见病联合方案
- 公平:
-
可查找、可访问、可互操作和可重用
- FCA:
-
形式概念分析
- NCIt:
-
国家癌症研究所同义词词典
- OAEI:
-
本体对齐评估计划
- 圣务指南:
-
孤儿罕见病本体论
- snom CT:
-
临床术语
- uml:
-
统一医学语言系统元辞典
参考文献
研究和创新总局。《地平线2020年科学出版物开放获取和研究数据开放获取规则指南》。3月:欧洲委员会;2017.
威尔金森博士Dumontier M, Aalbersberg IJ,阿普尔顿G, Axton M, Baak,显得过于N, Boiten JW, da Silva桑托斯磅,伯恩PE、Bouwman J,布鲁克斯AJ,克拉克T, Crosas M, Dillo我Dumon O,埃德蒙兹,Evelo CT, Finkers R, Gonzalez-Beltran,灰色AJG Groth P, Goble C, Grethe JS, Heringa J, T Hoen PAC, Hooft R,库恩T R角,角J,酒鬼SJ, Martone我,隆起,封隔器,佩尔松B, Rocca-Serra P,鲁斯M, van Schaik R,桑松SA舒尔特E, Sengstag T,斯莱特T - G, Swertz妈,汤普森M,Van Der Lei J, Van Mulligen E, Velterop J, Waagmeester A, Wittenburg P, Wolstencroft K, Zhao J, Mons B.评论:科学数据管理和管理的FAIR指导原则。科学数据,2016;3:1-9。https://doi.org/10.1038/sdata.2016.18.
公平主动。2020年。https://www.go-fair.org/fair-principles/fairification-process/.2020年3月访问。
本体论、本体论与公平的“我”。Data Intell. 2020;2(2): 181 - 91。
Kamdar MR, Tudorache T, Musen MA。生物医学本体术语重用和术语重叠的系统分析。语义网,2017;八(6):853 - 71。https://doi.org/10.3233/SW-160238.
Quesada-Martínez M, Fernández-Breis JT。Teije A, Popow C, Holmes JH, Sacchi L,编辑。医学中的人工智能。Cham:施普林格:2017。3-13页。
Harrow I, Balakrishnan R, Jimenez-Ruiz E, Jupp S, Lomax J, Reed J, Romacker M, Senger C, splentidiani A, Wilson J, Woollard P.语义启用应用的本体映射。《今日药物发现》2019。https://doi.org/10.1016/j.drudis.2019.05.020.
欧洲委员会。欧洲罕见病联合方案。https://www.ejprarediseases.org/.2020年2月18日访问。
李志强,刘志强,刘志强,Rodríguez-Martínez FJ, Gómez-Rodríguez A.本体匹配:文献综述。专家系统应用2015;42(2): 949 - 71。https://doi.org/10.1016/j.eswa.2014.08.032.
李志强,李志明,《本体匹配》,第二版;2007,第1-333页。https://doi.org/10.1007/978-3-540-49612-0.
R核心团队:R:统计计算语言和环境。维也纳;2020.https://www.r-project.org/.已于2020年7月访问。
Martínez-Romero M, Jonquet C, O 'Connor MJ, Graybeal J, Pazos A, Musen MA。NCBO本体推荐2.0:生物医学本体推荐的增强方法。J Biomed Semant. 2017;8(1): 22页。https://doi.org/10.1186/s13326-017-0128-y.http://arxiv.org/abs/1611.05973。
Shah NH, Bhatia N, Jonquet C, Rubin D, Chiang AP, Musen MA。构建开放式生物医学注释器的概念识别器比较。生物信息学。2009;10(5。9): 1 - 9。https://doi.org/10.1186/1471-2105-10-S9-S14.
欧洲委员会。罕见病登记通用数据元素集。2019。https://eu-rd-platform.jrc.ec.europa.eu/set-of-common-data-elements_en.2019年12月访问。
Orphanet。罕见病及其分类。2020.http://www.orphadata.org/cgi-bin/rare_free.html.2020年3月访问。
snom国际。snom CT。2020.http://www.snomed.org.2019年12月访问。
弗拉戈索。G . de Coronado S . Haber M . Hartel F . Wright L. NCI词库的概述和使用。2004。https://doi.org/10.1002/cfg.445.
万维网联盟:语义网W3C。2020.https://www.w3.org/standards/semanticweb/.2020年2月。
Cuenca Grau B, Horrocks I, Kazakov Y, Sattler U.从本体中提取模块:一种基于逻辑的方法。:选。指出第一版。科学。(包括潜艇。收。Artif指出。智能。收。生物信息学):2009。https://doi.org/10.1007/978-3-642-01907-4-8.
OWL API: OWL本体的Java API。语义网,2011;2(1):乳。https://doi.org/10.3233/SW-2011-0025.
Euzenat J, Meilicke C, Stuckenschmidt H, Shvaiko P, Trojahn C.本体对齐评估倡议:6年经验。:选。指出第一版。科学。(包括潜艇。收。Artif指出。智能。收。生物信息学):2011。https://doi.org/10.1007/978-3-642-22630-4-6.
Harrow I, Jiménez-Ruiz E, splentidiani A, Romacker M, Woollard P, Markel S, Alam-Faruque Y, Koch M, Malone J, Waaler A.本体对齐评估倡议中的疾病和表型本体匹配。J Biomed Semant. 2017;8(1): 1-13。https://doi.org/10.1186/s13326-017-0162-9.
Cruz IF, Antonelli FP, Stroe C. AgreementMaker:大型现实世界模式和本体的高效匹配。Proc VLDB捐赠。2009;2(2): 1586 - 89。https://doi.org/10.14778/1687553.1687598.
法里亚D,佩斯基塔C,桑托斯E,克鲁兹IF,库托FM。AgreementMakerLight 2.0:面向高效大规模本体匹配。CEUR工作规程2014;1272:457-60。
赵敏,张松,李伟,陈刚。基于形式概念分析的生物医学本体匹配。J Biomed Semant. 2018;9(1): 1-27。https://doi.org/10.1186/s13326-018-0178-9.
Jiménez-Ruiz E, Cuenca Grau B. LogMap:基于逻辑和可扩展的本体匹配。计算机科学课堂讲稿(包括人工智能课堂讲稿和生物信息学课堂讲稿子系列)。2011;7031 lncs(1): 273-88。
Jiménez-ruiz E, Grau BC,周宇。LogMap 2.0:面向基于逻辑、可扩展和交互式的本体匹配。Nat Precedings. 2011:2-3。https://doi.org/10.1038/npre.2011.6670.1.
万维网联盟。对齐的API。2014.https://www.w3.org/2001/sw/wiki/Alignment_API.访问2020年2月05日。
法里亚D,佩斯基塔C,莫特I,马丁斯C,库托FM,克鲁兹IF。应对生物医学本体匹配的挑战。J Biomed Semant. 2018;9(1): -。https://doi.org/10.1186/s13326-017-0170-9.
美国国家医学图书馆。超类龙,2020年。https://www.nlm.nih.gov/research/umls/index.html.2020年5月。
Ghazvinian A, Noy NF, Musen MA。为生物医学中的本体创建映射:简单的方法有效。AMIA Ann Symp 2009;2009(1): 198 - 202。
Jiménez-Ruiz E, Grau BC, Horrocks I.在本体对齐评估倡议中利用umls元辞典。: E-LKR。Castellón de la Plana: CEURWS.org: 2012。
Jiménez-Ruiz E, Grau BC, Horrocks I, Berlanga R.基于逻辑的UMLS本体源兼容性评估,第2卷。2011:2。https://doi.org/10.1186/2041-1480-2-S1-S2.
本体对齐评估倡议:OAEI 2019大型生物医学跟踪2019。https://www.cs.ox.ac.uk/isg/projects/SEALS/oaei/2019/.2020年10月访问。
本体对齐评估的语义精度和查全率。见:Proc第20届人工智能国际联合会议。海德拉巴:AAAI出版社:2007年。348 - 53页。https://hal.inria.fr/hal-00817806.
Jiménez-Ruiz E, Meilicke C, Grau BC, Horrocks I.评估映射修复系统与大型生物医学本体。CEUR工作规程2013;1014:1000-10。
法里亚D, Jiménez-Ruiz E,佩斯基塔C,桑托斯E,库托FM。在生物门图中注释潜在的不连贯。计算机科学课堂讲稿(包括人工智能课堂讲稿和生物信息学课堂讲稿子系列)。2014;8797:17-32。https://doi.org/10.1007/978-3-319-11915-1-2.
维基媒体基金会。增生。2020。https://en.wikipedia.org/wiki/Hyperplasia.2020年10月访问。
Jiménez-Ruiz E, Grau BC, Horrocks I.我的本体匹配系统和你的相似吗?波士顿:CEUR-WS.org;2007.
刘志军,刘志军,刘志军,基于语义索引的本体对齐任务分解与神经嵌入。2018。http://arxiv.org/abs/1805.12402。2020年2月。
Jacobsen A, Kaliyaperumal R, Bonino da Silva Santos L, Mons B, Schultes E, Roos M, Thompson M.数据FAIRification过程的通用工作流。Data Intell. 2020;2:56 - 65。
Kolyvakis P, Kalousis A, Smith B, Kiritsis D.生物医学本体对齐:一种基于表示学习的方法。J Biomed Semant. 2018;9(1): 1 - 20。https://doi.org/10.1186/s13326-018-0187-8.
Hertling S, Portisch J, Paulheim H.用MELT监督本体和实例匹配。cerc工作计划2020;2788:60 - 71。http://arxiv.org/abs/2009.11102。
Vasant D, Chanas L, Malone J, Hanauer M, Olry A, Jupp S, Robinson PN, Parkinson H, Rath A. ORDO:连接罕见病、流行病学和遗传数据的本体。波士顿:国际计算生物学学会;2014.
确认
不适用。
资金
这项工作由欧盟的地平线2020研究和创新计划资助,该计划隶属于EJP RD联合基金-EJP N∘825575.该研究的部分资金也来自于Economía、Industria y competition vidad、Gobierno de España和欧洲区域发展基金,资助号为TIN2017-85949-C2-1-R。
作者信息
作者和隶属关系
贡献
PD, JTFB和RC构思和设计了分析。PD进行了实验并撰写了论文。NB、JTFB、JAMG、NK和RC对稿件内容进行了修改。作者们阅读并批准了最终稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者声明他们没有竞争利益。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
补充信息
附加文件1
个别评价结果。评价的全部结果。表显示每个匹配系统、本体对、本体类型(模块或整个本体)的单个结果。此外,该文件还包括具有一致性对齐的个别精度和召回评分的表。
权利与权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。欲查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于本文
引用本文
范·云顿,P. Fernández-Breis, j.t.,贝尼斯,N。et al。FAIR数据本体匹配系统的性能评估。J生物医学语义13, 19(2022)。https://doi.org/10.1186/s13326-022-00273-5
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s13326-022-00273-5
关键字
- 本体匹配
- 合理的数据
- 语义互操作性
- 罕见疾病