摘要
背景
阐明疾病和发育障碍需要理解表型变异。单物种模式生物解剖本体(ssAOs)已被建立来代表这种变化。多物种解剖本体(msAOs;脊椎动物骨骼,脊椎动物同源,硬骨骨,两栖动物AOs)已经被开发来代表跨物种的“自然”表型变异。我们的目标是为了各种目的整合ssAOs和msAOs,包括建立表型变异和候选基因之间的联系。
结果
以前,msao包含唯一的和重叠的内容的混合。这阻碍了集成和协调,因为需要维护对ssao的交叉引用或本体间等价公理,或者执行大规模的过时和模块化导入。在这里,我们将解剖学本体论统一到Uberon中,这是一个单一的本体论资源,可以实现不同数据和研究小组之间的互操作性。因此,TAO, VSAO, AAO, vHOG的独立开发已经停止。
结论
新扩展的Uberon本体论是后生动物(动物)的一个统一的跨分类单元资源,它已被大幅扩展到包括广泛的脊椎动物解剖结构的多样性,允许在灭绝和现存分类单元的解剖变异之间进行推理。Uberon是一个核心资源,它支持单个和跨物种的候选基因查询,使用来自系统学、生物多样性、医学和模式生物群落的表型注释,同时也为细胞和基因本体论中的逻辑定义提供实体。
与本文中描述的本体合并相关的本体发布文件可在以下位置获得:http://purl.obolibrary.org/obo/uberon/releases/2013-02-21/
当前的本体发布文件总是可以在:http://purl.obolibrary.org/obo/uberon/releases/
背景
随着“组学”时代的兴起,信息、集成和检索已成为系统生物学面临的重要挑战。表型组学,或基于表型的分析,是一项关键的事业,并在系统生物学方法中发挥重要作用,以了解基因功能和功能障碍。具体来说,我们的目标是发现潜在的遗传和表观遗传对发育、进化、行为和形态变异的贡献。为了支持生物多样性和生物医学研究中表型变异的计算查询,已经开发了许多语义资源。多年来,模式生物数据库(MODs)一直在聚合来自各自生物研究社区的遗传学和基因组学信息,MOD开发人员在该领域的管理和数据标准化战略的建立中发挥了关键作用。为了注释和查询基因表达和表型数据,MODs开发了物种特异性解剖本体(ssAOs),包括发育小鼠解剖本体(EMAPA [1])、成年小鼠解剖本体(MA [2])、成人本体(FMA [3.])、发育人体解剖学本体(EHDAA2 [4]),斑马鱼解剖本体(ZFA [5]),苍蝇解剖本体(FBbt [6]),以及非洲爪蟾蜍解剖学本体论7,8])。为了在不同的ssao之间标准化上层术语(例如,“组织”和“非细胞结构”),开发了通用解剖参考本体(CARO) [9].不幸的是,这并没有解决所有这些问题,因为CARO的设计目的不是为跨物种共享的特定结构提供大量术语,而是为构建解剖学本体提供一个组织框架。
MODs的一个主要目标是查询跨解剖表型数据,以支持与人类疾病相关的相关基因的鉴定(见[10])。为了实现这一目标,我们构建了Über解剖本体(Uberon),这是一个后生动物多物种解剖本体,以提供跨MODs解剖结构的泛化[11].然而,要理解模式生物之外的生物多样性,就必须更深入地了解MODs记录之外的形态变化,以及这种变化是如何进化的。为了比较不同物种之间的“自然”表型变异,已经做出了许多努力来构建分类单元特异性的多物种aoo (msAOs)。
多个分类单元特有的脊椎动物msao被分别开发用于许多不同的目的。硬骨骨解剖本体(TAO [12])由phenoscape.org (phenoscape.org)项目开发,用于从分类学文献中注释进化表型(特征状态),并实现与遗传数据的互操作性[13,14].TAO最初是从ZFA克隆的,并扩展到涵盖硬骨鱼,重点是骨骼系统。两栖动物解剖本体(AAO [15)是为了标准化不同两栖动物群体的术语而建立的,它包括了许多解剖学系统的术语。脊椎动物骨骼解剖本体(VSAO [16])被构建为适用于所有脊椎动物的高级骨骼系统术语。脊椎动物同源器官类群本体(vHOG [17)是为了定义脊椎动物中具有共同祖先(同源性)的结构,而不是描述它们的形态多样性。作为Phenoscape项目的一部分,已经进行了大量工作,以使脊椎动物msAOs (TAO, VSAO, AAO)和ssAOs (MA, XAO,和ZFA)可互操作,以连接从特征状态注释的形态变异与候选基因[18].同时,Bgee项目[19,20.]已将ssAO术语(EHDAA、EMAPA、EVoc成人、MA、XAO和FBbt)映射到vHOG本体,以支持同源结构中基因表达模式的比较[21].
在此之前,这些脊椎动物msAOs包含独特和重叠的混合内容。这导致了重复工作,因为每个人都定义了共同的类别,如“椎体”和“神经系统”。此外,从用户的角度来看,维护交叉引用或本体间等价公理的需要阻碍了集成。与此同时,表型RCN的一些成员[22]开始形式化特定领域的专家知识,例如神经嵴本体工作组[23]以及研究哺乳动物进食肌肉的FEED小组[24].为了最大限度地减少由多个小组并行工作引起的重复工作,并促进跨物种的推断,我们因此决定将工作结合到一个单一的多物种解剖本体中。我们还希望在这个范围内促进对本体开发的贡献的归因,以及一个有效的工作流程来减少手工工作。我们选择使用Uberon作为目标本体,因为它在后生动物中具有最广泛的覆盖范围,并且因为它与现有的单物种解剖本体全面集成。
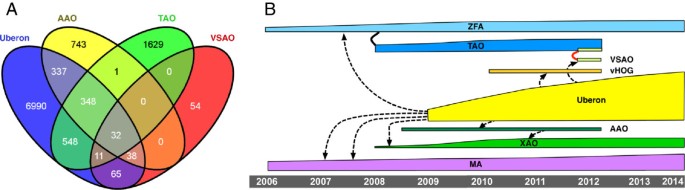
在这里,我们将四个msao (TAO, VSAO, AAO和vHOG)统一到Uberon中。对于研究人员来说,其意义在于,来自这些原本完全不同的资源(Phenoscape、Bgee、MODs)的数据现在将是可互操作的。TAO, VSAO, AAO和vHOG的开发已经停止,他们现在致力于Uberon背景下的内容开发。在我们的共同努力下,Uberon的2013-02-21版本包含了11391个类,其中2831个类是直接从TAO、AAO和VSAO中添加的。在最近的2014-03-03版本中,又增加了850个类,使类总数达到12241个。这是因为工作现在得到了协调,不再需要维护交叉引用和本体间等价公理的任务(对于尚未在MODs中表示的类)。我们详细描述了用于统一这些本体和扩展Uberon的方法,并进一步描述了Uberon的改进内容,这些改进内容是合并的一部分,并且正在作为后续集体本体开发的一部分进行。
结果
合并过程
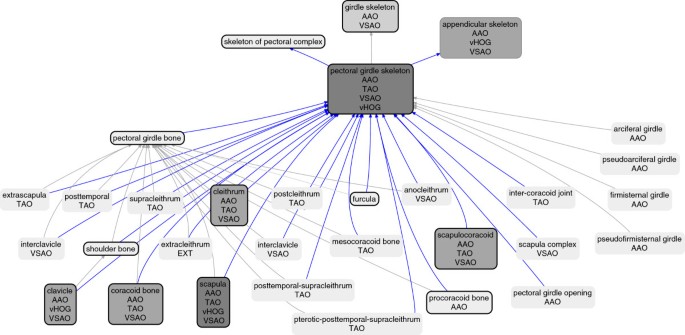
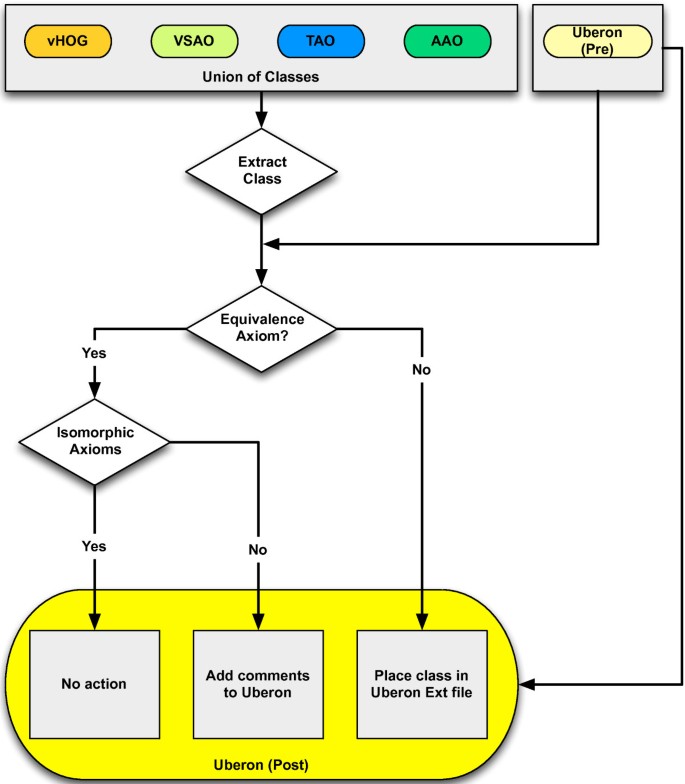
目标是维护有意义的解剖分组,以供生物学家最终用户使用,以及增强描述逻辑公理化,以支持更高级的推理,这些推理在最初的四个源本体中是有限的或不存在的。这次合并分为两个阶段。首先,手动比较来自一个或多个源本体论的类,这些类与Uberon类具有相似的文本定义、标签、同义词,或者已经与Uberon具有等价性或特定于分类单元的子类关系(参见方法)。在需要的地方,Uberon类中的包含标准被扩大,或者在某些情况下,根据来自源本体之一的更专业的定义来缩小。其次,来自Uberon中没有表示的任何源本体的类都被放置在owl格式的扩展本体中,该扩展本体导入核心Uberon并扩展核心中的类。作为第一种情况的一个例子,“胸带骨架”这个职业之前在Uberon和VSAO中都有出现[16]和其他一些源本体。因为这些定义经过了完全的专家审查,所以当这些类被导入时,Uberon中的中心表示被调整为符合VSAO。相比之下,作为第二种情况的一个例子,类' extracleithrum '以前没有在Uberon中表示,因此在Uberon名称空间中被赋予了一个新的标识符(Uberon:4200022)。1并放置在扩展本体中(参见图1).这种安排允许将更特定于分类的类的编辑权限分配给领域专家。图中突出显示了每个源本体的贡献和演化2,其中突出显示了合并前的交叉引用(Xrefs),以及这些源本体与Uberon之间的时间关系。图中显示了各个类如何相互关联的示例1.
挑战
类标签
在扩大硬骨鱼和两栖动物的范围时,有许多挑战。一个挑战是,在一个本体论范围内明确的类标签在合并到Uberon时变得模糊。例如,代表颅骨结构的AAO类“胸骨柄”(AAO:0000680)可能会与不相关的结构“胸骨柄”(UBERON:0002205)造成混淆;因此前者被重新命名为“玻璃柄”(UBERON:3000680)。
超类和关系
合并多个本体的另一个挑战是许多类缺乏超类或其他关系。在合并之后,大量的类出现在根节点或高级节点的正下方,这一点很明显。这些都是很容易纠正的目标,并且咨询领域专家以确定它们在本体中的正确位置。例如,' postminimus ' (UBERON:3010205)最初只是作为AAO中高级节点'解剖实体'的子类。这个类现在被断言为“软骨元素”的子类(UBERON:0007844)和part_of一些“跗骨骨架”(UBERON:0009879)。额外的文件1:表S1保存了对象属性和示例使用的列表。
调和不同领域的术语差异
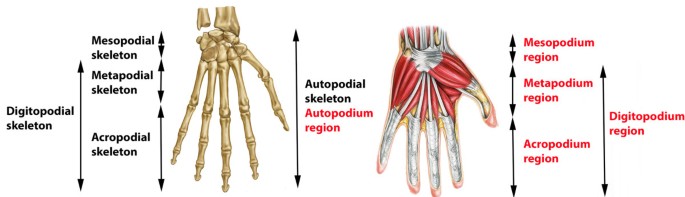
为不同目的而建造的ao的整合过程也突出了动物学和(或)医学术语之间和术语内部的不一致。这个问题的一个很好的说明来自于用于肢体不同区域的术语。在比较形态学文献中,“-podium”术语通常用于指代脊椎动物的附肢骨骼[25,26].这些术语是由海克尔提出的[27在他1895年的论文中,Systematische Phylogenie,指的是骨骼元素及其发育过程间叶原基;它们最初并不是指复合骨/肉肢体段。然而,在发展文献中,作者有时使用这些术语来指代“肢体段”(例如:[26]),通常指肢体芽[26- - - - - -30.].为了协调这些不同的用法,我们标记了一些术语,如“手动肢端骨骼”来指代骨骼,“手动肢端区域”来指代肢体部分。另一个挑战是使用一些术语(如acropodium)在不同的上下文中指代不同的结构。在某些论文中,“acropodium”仅指指骨(例如:[31]),但在其他情况下,它指的是除中足膜外的整个手掌骨骼(例如[32,33])。为了与海克尔的定义保持一致,我们决定用“肢足骨”(或“肢足骨”)来表示指骨,并引入一个新的术语“digitopodium”来表示肢足/肢足复体(图2)3.).
记录和实现设计模式
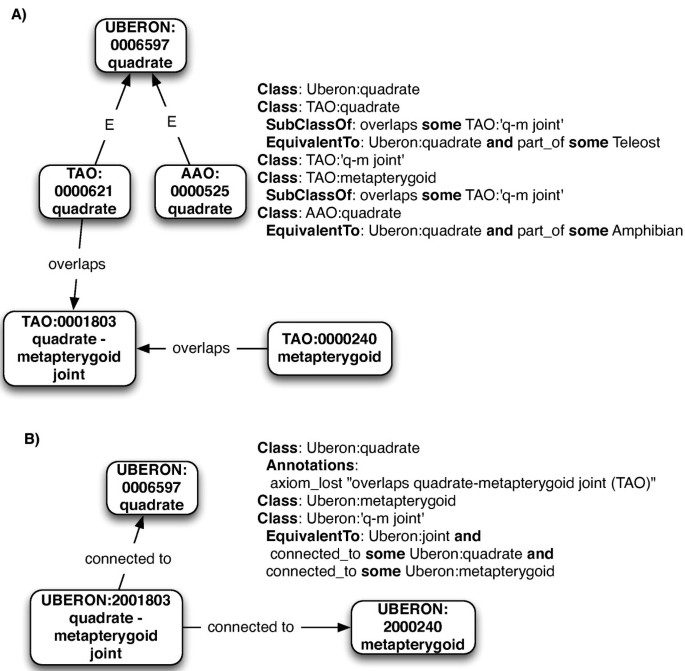
实现内聚统一本体的另一个挑战是跨源本体的建模策略、设计模式和术语约定的差异。这方面的一个例子是在合并之前在不同的本体中表示关节。在TAO中,代表不同骨骼元素的类与特定关节有关系;例如,' quadrate '和' anguloarticular '都有重叠与“方关节-角关节”的关系。在Uberon中,依赖关系是相反的——对于任何给定的骨骼元素,都没有与特定关节类的关系的假设,但关节被假设为暗示某些元素的存在。例如,在Uberon中,“方形关节”有两个connected_to与‘方形’和‘关节’(anguloarticular)的关系。在比较这两种款式时,我们决定选择后者。这是因为尽管TAO中的表示在硬骨鱼的水平上是有效的,但Uberon中表示的更广泛的分类多样性包含了脊椎动物骨骼元素连通性模式的变化。在Uberon中,关节是由它们连接的骨骼元素来定义的,而不是由各种骨骼元素的重叠来定义的,因此建模模式更稳定,因为不同物种的单个骨骼的连接可能不同。
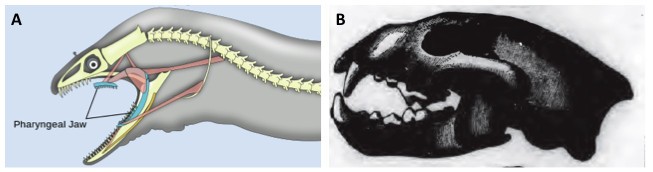
建模模式差异的另一个例子是各种本体如何表示牙齿与骨骼元素的连接。当本体论最初被结合在一起时,与不同骨骼相关联的牙齿的表示是可变的,例如,connected_to,part_of,重叠等。例如,牙齿与颌骨的关系是通过依附关系来表示的(attaches_to),相比之下part_of陶的关系。此外,许多鱼类在哺乳动物不常见的地方长有牙齿,例如在咽部(见图)4).因此,我们记录了一组核心设计和建模模式[34将应用于整个本体论,并且我们沿着这些路线继续统一组合的本体论。因此,Uberon的所有牙齿,无论是与哺乳动物的下颌骨和上颌骨相关,还是与鱼类的犁齿相关,都具有相同的关系(attaches_to)至支撑结构。对于希望查询组合本体的用户来说,这是一个主要优势,并提供了跨分类单元更稳定的建模模式。
最后一个说明需要通用设计模式的示例是肌肉及其附件的表示。例如,在脊椎动物进化的过程中,颌收肌(颌内收肌)从单一的、简单的肌肉转变为复杂的、高度分化的肌肉群,经历了好几次。TAO和ZFA只有一个类别,“下颌内收肌复合体”(TAO:0000311, ZFA:0000311),尽管大多数硬骨鱼的下颌内收肌至少有四个不同的部分(称为A1, A2, A3, Aw)。同样,在AAO中,有一个单独的类叫做“颌骨肌肉”(AAO:0000247)。两栖动物通常被描述为具有独立的内、外下颌骨内收肌(或分别为内、外提下颌骨内收肌)。这种仅基于子类和分类学的广泛表示法不足以描述复杂性。实现了一种通用的设计模式,可以对内收肌复合体中的单个肌肉进行颗粒状表示和更好的分类,该模式还基于神经支配、连通性和功能,例如“咬肌”(UBERON:0001597):
有能力参与的一些:咀嚼的
“有肌肉插入”一些下颌骨的
“有肌肉起源”一些“颧弓”
“有肌肉起源”一些上颌骨的
“部分”一些“脸”
innervated_by一些咬肌神经(三叉神经的分支部分)
正如前面提到的例子所示,在大型本体中保持一致的建模风格是很有挑战性的。在我们的本体论工作中,我们遵循记录常见“设计模式”的软件方法[35],已应用于生物本体[36].对于每个设计模式,都会创建一个文档,并在OWL中表示为本体本身的一部分[34,37].通过这种方式,文档直接耦合到与之相关的结构,从而促进应用。脚本用于为每个文档生成网页。例如,关于关节建模的文档是可用的,包括状态、贡献者、讨论和在本体中实现的问题摘要[38].
vHOG中同源组的整合
vHOG整合提出了一些具体的挑战,因为它代表了同源结构的分组,而不是正式的解剖学描述[17].这些手动审查的分组允许验证和纠正从Uberon到ssao的xref。现存类群中存在的一些高度衍生的器官合并在vHOG中,因为它们起源于共同的祖先结构。例如,鳍鱼的鱼鳔被假设与陆生脊椎动物的肺同源[39].虽然这些结构在Uberon中表示为不同的术语,但在vHOG中它们是一个单一的术语,称为“肺-鱼鳔”。在这种情况下,Uberon中保留了单独的术语,以避免创建代表假定结构或未在现有物种中描述的类。vHOG中包含的信息由Bgee组导出到一个外部注释文件,允许Uberon实体之间明确的同源关系,这些实体被认为来自一个共同的祖先结构。这也允许正式提供支持这些断言的证据。注释文件可在[40].其中一些同源断言在本体中使用OWL对象属性' homologous_to '进行复制,以表示同源实体之间的关系。但是,我们计划将它们作为派生自vHOG关联文件的独立OWL模块维护在一个地方,该关联文件可以分别导入和扩展。
基于同一器官发育状态的区别被类似地对待。“未来的大脑”(大脑的胚胎前身)和“大脑”等术语在vHOG中被合并为一个类别,从进化史的角度来看是有意义的。然而,vHOG中的这些合并需要进行分离,以适当地整合到Uberon中,其中基于发育结构的区分是分开的,分类是基于结构、功能、谱系等。然而,在vHOG中分组的发育结构之间的关系仍然可以从Uberon中检索,使用对象属性transformation_of而且immediate_transformation_of.
归因
责任的分配、编辑权利的协调和归属是我们不同社区之间合并过程的关键部分。主要贡献包括通过多个领域专家的跟踪器、设计文档、会议和研讨会对Uberon的补充。对核心的修改建议通过Uberon追踪器提交[41并由更大的社区进行审查。因为对这个社区和“科学的良好实践”来说,跟踪贡献并促进关于设计或定义的进一步讨论是非常重要的,所以需要一个更复杂的归因方法。对本体的贡献被标记为本体中的元数据,在类、公理和本体本身的级别上,使用对设计文档的引用,以及使用数据库交叉引用或ORCID id对个人的引用。设计文档也有作者,可以通过导入文件直接作为实例(见上文)包含在本体中,也可以通过Uberon.org网站进行在线链接。
讨论
有关表型的问题本质上是比较的。例如,为了了解疾病的遗传基础,临床遗传学家比较患有和没有这种疾病的人的外显子,或者生物医学研究人员比较野生型生物与转基因生物中不同基因组元素的贡献。为了了解气候变化对表型的影响,生态学家将受影响的表型与受环境控制的表型进行比较(见[42]供审阅)。为了分析生物多样性进化变化的顺序和性质,必须对世系内的基础种和衍生种进行比较。为了研究发育的进化,比较了不同物种中同源结构的表达模式。在任何这些领域中,跨表型的计算比较都需要解剖学的知识表示,该知识表示结构一致且灵活,允许添加新信息。本文描述的聚合多物种本体是代表多个研究领域观点的专家花费大量时间和精力的结果,并且已经产生了一个广泛有用的多物种解剖本体,它将为这些领域的研究人员提供实质性的好处。虽然每个分类群落都可以独立地从自己的解剖本体添加到其他物种的个体解剖本体的交叉引用,但单是时间和费用都是令人望而却步的。利用Uberon作为语义映射引擎,可以立即访问生物医学社区丰富和不断扩大的遗传和发育数据,这些数据本质上是可扩展的和可计算的。
本体集成的挑战
每一个做出贡献的团体都带来了丰富整体的新视角。硬骨鱼和两栖动物本体论给Uberon带来了一套广泛的术语和公理,其中许多是这些分类学群体独有的,这不仅增强了整体所包含的解剖学知识,而且挑战和改进了结构本身。例如,脊椎动物有各种鳞片样的成分:鱼类至少有三种,两栖动物有一种,而其他类型的骨沉积物(骨皮)在其他现存和灭绝的羊膜动物中都有发现[43].这些骨皮不怀疑与鱼类鳞片同源[44,45].类似地,vHOG和VSAO提供了更高级别的知识和分组术语,在合并后的Uberon中补充、细化和修订了鳞片和其他骨皮瘤的分类。
集成还包括达成协议,以解决备选的透视图。因此,生成的本体比任何专门为单个项目开发的本体(如Phenoscape或Bgee)都要复杂,因为它必须适应不同的建模需求,因此需要多个分类轴。对于一些用户来说,Uberon的简化子集可能更有用,例如,对于文本挖掘者来说,标签和同义词上的自动实体匹配就足够了。对于其他目的,如基因表达比较(Bgee)或识别主要进化变化的候选基因(Phenoscape),对特定解剖系统的完全公理化是有用的。我们通过提供Uberon的许多不同子集来解决这个问题,在uberon.org网站上有描述。特别是,人们可以利用Uberon的分类单元特定的slim(本体的一部分)以及利用OBO子集注释的各种本体slim。例如,实验因素本体(EFO [46])最近更新为使用Uberon的slim,这些都是使用EFO_slim注释属性记录在Uberon中的。
达成的另一项协议是在类的主标签的选择上。我们尽量使用最中立的物种术语。作为这个决定的结果,我们将Uberon中现有的类,如“手”、“脚”、“手指”、“手肌肉”等重命名为“手”、“pes”、“手手指”、“手肌肉”等。我们将其一直延续到命名的重复亚单位,例如“手指1的远端指骨”。原有的标签被保留为同义词。在某些情况下,改变标签的理由比术语偏好更深刻。例如,UBERON:0006807类先前被标记为“肱骨外侧上髁”。然而,Phenoscape团队的比较形态学家指出,“横向”位置并不适用于所有类群,因为四肢的姿势在进化中发生了变化。我们决定采用一个更独立于物种的术语,并将这个类重命名为“肱骨外髁”。之前的标签被保留为同义词,并标记为“人类首选的”同义词(即,寻找更多“同义词”的社区首选的同义词)。智人为中心”的观点)。每个类实际上都有一个唯一的主标签,尽管应用程序可以根据自己的喜好轻松使用不同的同义词类型,也可以根据需要添加更多的同义词类型。
本体的整合和对齐是生物医学中广泛使用案例的普遍挑战。正如我们在这里强调的,有许多社区对记录解剖数据有特定的需求。尽管象和鸡这样种类繁多的生物的解剖结构乍一看似乎没有太多共同之处,但任何比较形态学家的主要目标都是检查这些相似之处。因此,解剖学本体对本体对齐提出了一个特殊的问题——存在许多研究相似内容的小组,但没有协调,并且处于不同的领域。由于不同的建模策略、本体开发原则和生物学角度,相似的解剖特征最终会以不同的方式表示。理想情况下,我们应该能够使用标准工具来调和和合并这些解剖本体。在实践中,我们最终使用了我们自己的特别脚本和工具,这允许我们在任何时候对大量的本体执行操作。一旦进行了初始合并,管理员就可以使用Protégé等标准编辑环境在更细粒度的级别上进行编辑和更正。我们的自动化工具是开放和免费的(参见方法),但尚不清楚它们是否适用于其他领域,因为它们是针对我们正在使用的特定解剖本体进行大量定制的。我们建议开发解剖本体的团队从一开始就以集成的模块化方式进行,而不是事后集成,后者总是一项昂贵且耗时的任务。
调和共同的术语差异
在定义解剖实体类别的过程中,我们遇到了许多困难,通常是因为常用的类别是“模糊的”。例如,身体的大部分大肌肉都是“骨骼肌”。“骨骼肌”附着在骨骼元件上,是由躯体运动神经元支配的“随意”肌肉,来源于躯体。相比之下,鳃弓肌或咽弓肌长期以来被称为“内脏”肌肉,因为它们是鳃弓装置的一部分,源自颅间质(颅旁中胚层),而不是躯体。“内脏”一词指的是内脏或内部器官。“内脏肌肉”一词在大多数情况下是指由自主运动神经元支配的光滑不随意肌肉。然而,鳃裂肌是随意肌,它们由大的运动神经元支配,像骨骼肌一样,被认为是α运动神经元。此外,有些随意肌肉,如舌头的固有肌肉,与骨骼没有附着,但被称为骨骼肌。随意肌也是“横纹肌”,根据它们在光学显微镜下的带状外观。然而,心肌是横纹肌的一种形式,尽管它通常被认为是“不随意”的内脏肌肉,并由自主运动神经元支配。 To complicate matters further, some invertebrates, such as arthropods, have at least two types of striated muscles. Thus, the traditional terminology presents many complex, contradictory issues. Reconciling complex fuzzy nomenclature is an ongoing and future goal, and we intend to work with the respective communities (the FEED group for this example) to provide competency questions and get feedback on our modeling approaches. In this way, we hope to improve our communication, specificity, and interoperability amongst different content specialists.
超越脊椎动物
我们的长期目标是创建一个涵盖多细胞生物的联邦解剖本体集。Uberon中的许多类适用于脊椎动物之外(例如,'脊索动物'),有些广泛适用于后生动物(例如,'肌肉组织')。然而,Uberon中的大多数类都是脊椎动物特有的。一些类群将包含在Uberon中,而其他类群将作为独立的本体开发,作为多物种OWL导入链的一部分与Uberon一起工作。特别是新的多孔动物(海绵)本体(Thacker,本期)、头足类本体[47]和栉水母本体正在作为单独的本体开发,它们重用一组CARO和Uberon类。节肢动物解剖学本体论(见https://code.google.com/p/arthropod-anatomy-ontology/)进一步发展,许多来自Uberon的类(例如“蘑菇体”)将被淘汰并让与这个本体。我们欢迎领域专家提供信息,以扩展Uberon或“萌芽”新的本体,涵盖诸如刺胞动物、环节动物或棘皮动物等生物,所有这些生物目前都只有浅层覆盖。虽然这听起来很简单,但实际上相当具有挑战性。正如我们在Uberon的开发中所看到的,通常情况下,词汇表是为特定的分类单元或目的而开发的。通过表型RCN,我们为解剖学家和进化生物学家提供培训,以协助编目“生命部分”的任务。
总之,将主要脊椎动物类群的解剖系统正式整合到Uberon中,已经扩大并使我们能够努力研究疾病[48- - - - - -51]和进化[14,18]使用比较解剖学方法。
方法
本体合并
在合并之前,我们有四个源本体(VSAO, AAO, TAO, vHOG)和一个目标本体(Uberon)。在早期的工作中,我们增强了连接Uberon类与源类的等价公理集。然后,目标是通过将源类合并到Uberon本体中来消除冗余类(即等价类),这样得到的本体是内聚的,不会丢失信息,并且重复最小化。作为第一步,我们增强了连接目标类和源类的等价公理集。这是直接等价公理(例如Uberon:股骨等价to VSAO:股骨)和分类等价公理(例如Uberon:股骨和part_of一些两栖动物相当于AAO:股骨)。总体方法如图所示5详情在这里。有了等价公理之后,我们将所有源类分为Uberon中存在等价公理(直接等价或分类等价)的源类和Uberon中没有等价类的源类。例如,“方骨”的两栖类和硬骨类(TAO:0000621和AAO:0000525)属于前一组,因为它们都有等价公理将它们与UBERON:0006597联系起来(见图)6).
对于那些存在等价公理的类,我们验证了所有公理也都出现在Uberon类中;如果它们不是同构的,Uberon类就会用一个结构化的注释进行注释。例如,TAO有一个公理,说明'平方'重叠“方关节- angulo关节”。Uberon类没有添加等价关系;相反,添加了一条注释,说明TAO包含了这种更具体的关系。来自TAO的原始文本定义作为“外部定义”注释传输过来。这使我们能够保守地自动地从信息源转移信息,而无需做出在更广泛的分类学上下文中不成立的逻辑陈述。相反,如果源类中没有额外的公理约束,源类就会被废弃,直接被等价的Uberon类所取代。同义词根据需要添加到这些Uberon类中,以保留来自源类的信息。
对于每个在Uberon中没有等价公理的源类,我们创建一个过时的记录,创建一个新的Uberon类,并从源类转移公理,同时使用等价公理映射所有实体。例如,TAO类' quadrate posterodorsolateral process ' (TAO:0001611)之前没有在Uberon中表示,因此生成了一个新类(Uberon:2001611)。保留了相同的标签、定义和其他描述性信息。如果源类还包含part_of关系(例如TAO类的“四方后外侧突”part_ofTAO类' quadrate ' (TAO:0000621)),然后该关系被重写为使用等效的Uberon类(在本例中,Uberon:0006597)。以这种方式创建的所有新类都被认为是Uberon的一部分,但出于管理原因,它们被放置在一个单独的本体文件“phenoscpe -ext”中。猫头鹰或简称“Ext”。这允许本体编辑器细化翻译过程的结果,并在核心中没有出现的情况下添加新类。公理分离为核心本体文件和扩展Ext文件,允许分工和编辑权利。所有原始Uberon术语都包含在核心文件中,即那些与原始源类等价的术语。其他本体特有的术语放置在Ext文件中。
TAO的合并
TAO包含3048个类,其中许多TAO类已经被VSAO类所取代。在合并之前,Uberon有970个TAO类的等价公理,TAO中剩下的2078个类主要代表硬骨鱼特定的结构(TAO版本从2012年8月9日开始)。后一个类被淘汰,并被Uberon Ext本体中的2078个等价的新类所取代(这些类的id形式为Uberon:2nnnnnn,为方便起见保留了最后六位数字),相应的公理被保留或重写(见上文)。在这个自动化过程之后发生了额外的手动对账。因为TAO最初是通过克隆ZFA创建的,所以本体包含了大量的类(特别是细胞类型和大脑区域),这些类并没有真正泛化鲐鱼类;我们废弃了这些,因为除了已经存在于特定物种的斑马鱼解剖本体之外,它们几乎没有增加什么价值。Uberon Ext从TAO中保留了1630个课程。
AAO的合并
AAO的过程遵循了TAO的过程。AAO包含1595个类(2012年4月13日版本),在Uberon中有789个类具有分类学上的对等物。其余的类被淘汰,等价的类被添加到Ext(在ID范围UBERON:3nnnnnn)。由于创建大部分AAO的自动化过程中产生的问题,重点放在手动增强这些类上。许多类都有模棱两可的名称,如' procsus dorsalis ' (AAO: 0000655),并且缺乏定义。与这一领域的专家合作,我们产生了不那么模糊的名称和定义。
VSAO的合并
VSAO包含201个类。Uberon和VSAO的编辑在VSAO的开发过程中进行了协调,因此VSAO的大部分人在合并过程的一开始就已经出现并与Uberon保持了良好的一致。有54个类没有被表示出来(2012年7月16日的版本),这些类在UBERON:40nnnnn ID空间中成为Ext中的类。VSAO的定义和公理被保留下来,因为它们是经过专家审查和精心策划的[16].
vHOG的合并
因为Uberon和vHOG的起源都是对准现有的物种特定的解剖本体,vHOG中的大多数类都已经在Uberon中表示了。vHOG中手动验证的分组被转移到Uberon本体中的“同源注释”注释中,并用于细化和验证分类单元等价公理。
编辑和发布
Ext组件使用Protégé进行编辑。核心Uberon本体是导入的,但Ext编辑器不能直接编辑(Protégé在视觉上区分导入的类和声明的类,为编辑器提供适当的视觉提示)。核心本体文件的维护和编辑需要专门的工具来保持本体与ssao、GO和其他本体同步。但是,Ext本体包含核心类的子类,管理员可以使用Protégé进行维护,而不需要使用这种专门的工具。核心文件和Ext文件之间的区别是一个编辑工件,在发布的文件中消失了。我们使用了Protégé 4.2,加上bo- edit图形组件(Dietze, in prep)来自bo- edit [52],加上描述插件(见下文)和其他支持OBO开发风格的插件。额外的文件1:表S1保存了对象属性和示例使用的列表。与本文中描述的本体合并相关的发布文件可在以下地址获得:http://purl.obolibrary.org/obo/uberon/releases/2013-02-21/
Uberon.org网站上描述了多种形式的Uberon,例如:
http://purl.obolibrary.org/obo/uberon/ext.owl以及特定于分类的文件和简单的预先推理文件,只有三个主要关系(子类、part_of和develops_from).
所有用于生成和维护本体的工具都是开放的、免费的,并记录在这里:
https://github.com/obophenotype/uberon/wiki/Editor-tools-guide
使用Jenkins进行集中推理
如所述[11], Uberon包含了大量的公理,以确保内部一致性和与其他本体的一致性。我们要求本体总是满足这些条件。此外,我们要求检查本体是否违反某些语法条件(例如,每个类应该恰好有一个标签,并且不超过一个文本定义)。其中一些检查很难在本体开发人员的机器上本地运行,因此我们使用了OBO库Jenkins持续集成服务器[53].编辑器执行的每一次提交都会触发验证和构建管道,这也会生成一个bo-format文件,用于Phenex中的Phenoscape字符注释和CharaParser中的实体匹配[54].
与ssao的一致性检查
我们已经实现了一个过程,通过该过程,Xrefs和等价公理在单物种解剖本体和现在集成到Uberon中的本体之间进行检查。例如,如果一个斑马鱼类有一个到TAO类的Xref,这个TAO类还没有被淘汰并被Uberon类取代(使用replaced_by注释属性),这些将是不满意的,将被固定在源斑马鱼文件。
描述的整合
此更新还包括描述各种种类的本体类实例的图像。最初的描述集是通过使用现有的Uberon到Wikipedia的映射来查询DBpedia的图像来生成的(参见原始Uberon论文[11]查阅详情)。由于使用了DBpedia/Wikipedia,这个初始集主要是针对人类的,但我们一直在使用图像描述插件手动增强它[55].
尾注
一个在本文档中,我们对本体中的类使用obo风格的id。要将此转换为OWL本体中使用的URL,请在数字前加上“http://purl.obolibrary.org/obo/UBERON_,例如http://purl.obolibrary.org/obo/UBERON_4200022.
缩写
- 阳极氧化铝:
-
两栖动物解剖本体
- EMAPA:
-
小鼠大体解剖与发育,摘要
- Ext:
-
新合并的Uberon本体的一部分,主要包含来自分类单元特定内容的叶节点,以前没有出现在Uberon中
- 菲利普-马萨:
-
人体解剖学的基本模型
- 国防部:
-
模型生物数据库,如小鼠基因组信息学(MGI)或斑马鱼模型生物(ZFIN)数据库
- 马:
-
成年小鼠解剖本体
- msAO:
-
多物种解剖本体论,侧重于一个狭窄的分类群,而不是一个单一的物种,如ssAO,或广泛的分类群,如Uberon的情况
- ssAO:
-
物种特有的解剖本体
- 道:
-
硬骨骨解剖本体
- vHOG:
-
脊椎动物同源器官类群本体
- VSAO:
-
《脊椎动物骨骼解剖学本体
- XAO:
-
爪蟾解剖本体
- Xref:
-
交叉引用到另一个数据库、本体或源
- ZFA:
-
斑马鱼解剖本体。
参考文献
Bard JL, Kaufman MH, Dubreuil C, Brune RM, Burger A, Baldock RA, Davidson DR:基于系统命名法的小鼠发育解剖学的互联网访问数据库。机械工程学报,1998,29(4):344 - 344。10.1016 / s0925 - 4773(98) 00069 - 0。
Hayamizu T, Mangan M, Corradi J, Kadin J, Ringwald M:成年小鼠解剖词典:注释和整合数据的工具。中国生物工程学报,2005,6:R29-10.1186/gb-2005-6-3-r29。
生物信息学的参考本体论:解剖学的基本模型。中国生物医学工程学报,2003,36:478-500。10.1016 / j.jbi.2003.11.007。
巴德J:前7周(卡内基阶段1-20)人类发育解剖学的新本体论(结构化层次)。王志强,2012,30(4):344 - 344。10.1111 / j.1469-7580.2012.01566.x。
Van Slyke CE, Bradford YM, Westerfield M, Haendel MA:斑马鱼解剖和阶段本体:代表Danio rerio的解剖和发展。中国生物医学工程学报,2014,26(5):457 - 457。
科斯塔M, Reeve S, Grumbling G, Osumi-Sutherland D:果蝇解剖学本体。中国生物医学工程学报,2013,4:32-10.1186/2041-1480- 432。
Segerdell E, Bowes JB, Pollet N, Vize PD:非洲爪蟾解剖和发展的本体论。中国生物医学工程学报,2008,29(4):344 - 344。
Segerdell E, Ponferrada VG, James-Zorn C, Burns KA, Fortriede JD, Dahdul WM, Vize PD, Zorn AM: Enhanced XAO: Xenopus解剖和发育的本体论支持更准确的基因表达注释和Xenbase查询。中国生物医学工程学报,2013,4:31-10.1186/2041-1480- 431。
Haendel MA, Neuhaus F, Osumi-Sutherland D, Mabee PM, Mejino JLV, Mungall CJ, Smith B: CARO -通用解剖学参考本体。生物信息学的解剖学本体:原理与实践。编辑:Burger A, Davidson D, Bladock R. 2007,卷Albert Bur:施普林格
Washington NL, Haendel MA, Mungall CJ, Ashburner M, Westerfield M, Lewis SE:使用基于本体的表型注释将人类疾病与动物模型联系起来。公共科学图书馆,2009,7:e1000247-10.1371/journal.pbio.1000247。
Mungall CJ, Torniai C, Gkoutos GV, Lewis SE, Haendel MA: Uberon,一个整合的多物种解剖本体。中国生物工程学报,2012,13:R5-10.1186/gb-2012-13-1-r5。
Dahdul WM, Lundberg JG, Midford PE, Balhoff JP, Lapp H, Vision TJ, Haendel MA, Westerfield M, Mabee PM:硬骨骨解剖本体:基因组学时代的解剖学表征。中国生物医学工程学报,2010,29(4):369-383。10.1093 / sysbio / syq013。
Mabee PM, Arratia G, Coburn M, Haendel M, Hilton EJ, Lundberg JG, Mayden RL, Rios N, Westerfield M:使用本体论将进化形态学与基因组学联系起来:来自包括斑马鱼在内的Cypriniformes的案例研究。中国生物医学工程学报,2007,29(4):457 - 457。
Dahdul WM, Balhoff JP, Engeman J, Grande T, Hilton EJ, Kothari C, Lapp H, Lundberg JG, Midford PE, Vision TJ, Westerfield M, Mabee PM:进化特征,表型和本体:从系统生物学文献中整理数据。公共科学学报,2010,5:e10708-10.1371/journal.pone.0010708。
Maglia AM, Leopold JL, Pugener LAÌ, Gauch S:一种两栖动物的解剖学本体。中国生物工程学报,2007,29(3):367-378。
Dahdul WM, Balhoff JP, Blackburn DC, Diehl AD, Haendel MA, Hall BK, Lapp H, Lundberg JG, Mungall CJ, Ringwald M, Segerdell E, Van Slyke CE, Vickaryous MK, Westerfield M, Mabee PM:脊椎动物骨骼系统的统一解剖本体。公共科学学报,2012,7:e51070-10.1371/journal.pone.0051070。
Niknejad A, Comte A, Parmentier G, Roux J, Bastian F, Robinson-Rechavi M: vHOG,多物种脊椎动物同源器官类群本体。生物信息学,2012,28:1017-1020。10.1093 /生物信息学/ bts048。
Mabee PM, Balhoff JP, Dahdul WM, Lapp H, Midford PE, Vision TJ, Westerfield M: 50万条鱼表型:脊椎动物骨骼进化和发育生物学的新信息学景观。应用鱼酚学报,2012,28:300-305。10.1111 / j.1439-0426.2012.01985.x。
Bastian F, Parmentier G, Roux J, Moretti S, Laudet V, Robinson-Rechavi M: Bgee:异质转录组数据的整合与比较。数据集成,2008,124-131。
Bgee:基因表达进化数据库。http://bgee.unil.ch,
帕门提尔G,巴斯蒂安FB, Robinson-Rechavi M:同源关系:通过本体的成对对齐生成同源关系及其在脊椎动物解剖学中的应用。生物信息学,2010,26:1766-1771。10.1093 /生物信息学/ btq283。
表型研究协调网络。http://www.phenotypercn.org/,
由表型RCN赞助的神经嵴研讨会。http://wiki.phenotypercn.org/wiki/Neural_Crest_Workshop,
Wall CE, Vinyard CJ, Williams SH, Gapeyev V, Liu X, Lapp H, German RZ: FEED概述,饲喂实验最终用户数据库。中国生物医学工程学报,2011,29(5):531 - 531。10.1093 /学院/ icr047。
Shubin NH, Daeschler EB, Coates MI:四足动物肱骨的早期进化。科学通报,2004,30(4):344 - 344。10.1126 / science.1094295。
《从鳍到四肢:进化、发展和转变》,2007年,芝加哥:芝加哥大学出版社
海克尔E: Systematische Phylogenie。Entwurf eines Natürlichen在地球上的组织系统。1895,Dritter Theil: Systematische Phylogenie der wibelthiere(脊椎动物)
赵春春,韩力克M:灵长类动物肢体骨骼的进化与发育。人类学报,2002,11:94-107。10.1002 / evan.2002。
田村K, yoneii -Tamura S,矢野T,横山H, Ide H:自体:在肢体发育过程中的形成。科学通报,2008,30(增刊1):387 - 387。
陈志强,李志强,李志强,李志强,陈志强。小鼠前肢和后肢自异体的转录组分析。生物工程学报,2005,34(3):344 - 344。10.1002 / dvdy.20514。
vukievviic TT, aliic I, Slavica A, Poletto M, kujir S:克罗地亚布里朱尼岛休闲鹿(Dama Dama l.)足跖骨和肢足骨的初步骨测量分析。Veterinarski arhiv。2012, 38(4): 344 - 344。
赵春华,赵春华:四足动物肢体起源的一个假设。中国生物医学工程学报,2001,29(3):366 - 366。10.1002 / jez.1100。
Johanson Z, Joss J, Boisvert CA, Ericsson R, Sutija M, Ahlberg PE:鱼指:鱼鳍中的手指同源物。中国生物医学工程学报,2007,29(4):457 - 457。
Uberon用户手册。http://purl.obolibrary.org/obo/uberon/docs/manual,
Pree W:面向对象软件开发的设计模式。1994,Reading, Mass: Addison-Wesley
谭志强,李志强,李志强。基于细胞周期本体论的生物本体设计模式研究。中国生物医学工程学报,2008,9(增刊5):s1 -10.1186/1471-2105- s5 - s1。
Uberon文档系统。http://purl.obolibrary.org/obo/uberon/docs/About-documentation,
Uberon设计文档-建模关节。https://github.com/obophenotype/uberon/wiki/Modeling-articulations-Design-Pattern,
郑伟,王铮,Collins JE, Andrews RM, Stemple D,龚忠:斑马鱼鱼鳔与哺乳动物肺转录组的分子同源性分析。公共科学学报,2011,6:e24019-10.1371/journal.pone.0024019。
Bgee解剖相似性注释。https://sourceforge.net/p/bgee/wiki/Similarity%20annotation/,
杨晓明,李志刚,李志刚:植物适应的进化遗传学研究。植物学报,2011,27:258-266。10.1016 / j.tig.2011.04.001。
Vickaryous MK, Sire J-Y:四足动物的表皮骨骼:起源,进化和发展。王志强,2009,30(4):344 - 344。10.1111 / j.1469-7580.2008.01043.x。
朱格·格,维特·LJ,考德威尔·JP:爬虫学:两栖动物和爬行动物生物学导论。2001,伦敦:学术出版社
Zylberberg L, Wake MH:真皮虫和微盲肠虫(两栖纲:裸乳纲)鳞片的结构,以及与其他脊椎动物真皮骨化的比较。中华医学杂志,2000,26(3):344 - 344。10.1002 / jmor.1052060104。
Malone J, Holloway E, Adamusiak T, Kapushesky M, Zheng J, Kolesnikov N, Zhukova A, Brazma A, Parkinson H:基于实验因子本体的样本变量建模。生物信息学,2010,26:1112-1118。10.1093 /生物信息学/ btq099。
头足类动物本体项目。https://github.com/obophenotype/cephalopod-ontology,
Chen C- k, Mungall CJ, Gkoutos GV, Doelken SC, Köhler S, Ruef BJ, Smith C, Westerfield M, Robinson PN, Lewis SE, Schofield PN, Smedley D: MouseFinder:小鼠表型数据中的候选疾病基因。胡木,2012,33:858-866。10.1002 / humu.22051。
Doelken SC, Köhler S, Mungall CJ, Gkoutos GV, Ruef BJ, Smith C, Smedley D, Bauer S, Klopocki E, Schofield PN, Westerfield M, Robinson PN, Lewis SE:通过对人类、小鼠和斑马鱼单基因突变的跨物种计算分析揭示个体基因对CNV致病性贡献的表型重叠。机械工程学报,2013,6:358-372。10.1242 / dmm.010322。
Robinson P, Köhler S, Oellrich A, Wang K, Mungall C, Lewis SE, Washington N, Bauer S, Seelow DS, Krawitz P, Gilissen C, Haendel M, Smedley D:通过跨物种表型比较改善疾病基因的外显子组优先级。基因组决议2013,http://www.genome.org/cgi/doi/10.1101/gr.160325.113,
Smedley D, Oellrich A, Köhler S, Ruef B, Westerfield M, Robinson P, Lewis S, Mungall C:表型digm:分析将动物模型与人类疾病相关联的策策注释。数据库(牛津大学)。2013, 2013: bat025-
Day-Richter J, Harris MA, Haendel M, Lewis S, Group GOO-EW: bo - edit——生物学家的本体编辑器。生物信息学,2007,23:2198-2200。10.1093 /生物信息学/ btm112。
Mungall C, Dietze H, Carbon S, Ireland A, Bauer S, Lewis S:开放生物本体库的持续集成。Bio-Ontologies。2012年,http://bio-ontologies.knowledgeblog.org/405,
Cui H: CharaParser用于生物形态描述的细粒度语义注释。地球科学进展,2012,29(3):344 - 344。10.1002 / asi.22618。
图像描绘Protégé插件。https://github.com/balhoff/image-depictions-view,
确认
本材料部分基于由国家科学基金会资助的DBI-0641025, DBI-1062404, DBI -1062350和DBI-1062542的工作,并由国家进化合成中心在NSF EF-0423641和nsf# EF-0905606资助。本材料中表达的任何观点、发现、结论或建议都是作者的观点,并不一定反映美国国家科学基金会的观点。根据R24OD011883,对准人类和模式生物来源得到了NIH的支持。我们感谢表型本体论研究协调网络(NSF-DEB-0956049)的支持,它将参与者聚集在一起,开发本体论的关键领域。CJM感谢美国能源部基础能源科学办公室主任的支持。DE-AC02-05CH11231。Bgee得到了瑞士生物信息学研究所(SIB)、瑞士国家科学基金会(资助号31003A 133011/1)和瑞士沃州大学的支持。我们也感谢许多通过我们的追踪器为Uberon做出贡献的人,以及审稿人的建设性意见。
作者信息
作者及隶属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
作者的贡献
AD、MAH和NI是合并本体的主要本体编辑器,生成新的术语,解决合并问题,评估和修复现有的Uberon类,贡献高级设计,提供专家进化知识。CJM提供软件支持,设计合并计划,确保与现有本体的一致性,并在非骨架领域扩展本体。JPB提供了图像插件和推理质量保证。MAH执行合并计划并监督本体的开发。PM、PS和SEL监督项目,并提供所需的环境和资源。WD、YB和PM提供了专业知识,并协助TAO、VSAO和Uberon的合并。DB提供了专业知识,并协助AAO的合并。最后,JAB提供了专家知识,并协助哺乳动物结构的表示。PS、NI和AD同样有助于远古恐龙结构的表征。AN, AC, FB和MRR贡献了vHOG。 AN, YB, and AC fixed relations to species-specific ontologies. RD contributed expert knowledge about muscles from the FEED project. Authors are listed in alphabetical order with the exception of first and last authors. All authors read and approved the final manuscript.
电子辅助材料
13326 _2013_174_moesm1_esm.xlsx
附加文件1:表S1:列出了Uberon本体中可用的对象属性。属性IRI、标签、定义、定义源(definitionXrefs)、超级属性和用法示例将被显示。(xlsx21 kb)
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用属性许可协议(http://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是原创作品的名称要注明出处。
关于本文
引用本文
汉德尔,文学硕士,巴尔霍夫,j.p.,巴斯蒂安,F.B.et al。Uberon中比较生物学的多物种脊椎动物解剖学本体的统一。J生物医学语义5, 21(2014)。https://doi.org/10.1186/2041-1480-5-21
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/2041-1480-5-21
关键字
- 进化生物学
- 形态变化
- 表型
- 语义集成
- Bio-ontology