摘要
背景
近年来,药物重新定位因其在药物开发中的优势而备受关注。人工智能技术通过发现治疗性药物概况、副作用和新的靶蛋白,极大地提高了药物的复制能力。然而,随着药物数量的增加,它们的靶标和巨大的相互作用产生了不平衡的数据,这些数据可能不适合立即作为预测模型的输入。
方法
本文提出了一种基于药物化学结构和蛋白质序列预测药靶相互作用的新方案。采用药物摩根指纹图谱、药物成分描述符、蛋白质氨基酸组成、蛋白质二肽组成等方法提取药物及蛋白质特征。然后,提出了利用支持向量机一类分类器提取负样本的方法,以解决药物目标数据集的不平衡数据问题特征集。构建负样本和正样本,并将其输入不同的预测算法以识别DTIs。除了研究化学和物理特征在评估和发现药物-靶点相互作用中的有效性外,还应用了10倍CV验证测试程序来评估所提出方法的可预测性。
结果
我们的实验模型优于现有的关于接受者工作特征(AUC)、准确度、精密度、召回f分、均方误差和MCC曲线的技术。与现有方法相比,AdaBoost分类器的预测精度提高了2.74%,精度提高了1.98%,AUC提高了1.14%,F-score提高了3.53%,MCC提高了4.54%。
简介
预测潜在药物的DTIs在药物发现中起着至关重要的作用。它有助于理解生物操作,降低药物发现的成本[1,2].然而,在预测dti方面存在许多挑战。例如,药物的许多积极和消极影响是很难发现和解释的。在过去的几年里,人们在克服这些挑战和预测dti方面做出了重大努力。此外,由于人类基因组计划的完成和分子医学的不断发展,发现了更多未知的DTIs。然而,经分析验证的药物-靶点相互作用的数量仍然非常少,这促使研究科学家设计新的计算方法来克服这些挑战,以实现潜在的DTI预测[3.].
大量的DTI数据是在高性能计算技术发展之后产生的。几个流行的数据库,如KEGG [4]、药库[5], ChEMBL [6], stitch [7],及TTD [8],用于存储已确认的数据并提供相关的恢复信息,对于建立有效的计算方法以进行最佳的DTIs预测是有用的。
典型的DTI计算方案可分为三类:基于配体的方案、模拟对接方案和化学基因组方案。首先,基于配体的方案利用靶蛋白的相似性来预测药物化学结构和蛋白质序列之间的相互作用[9].
其次,基于对接的方案使用目标蛋白的动态模拟来发现新的、未知的相互作用。这种方案是一种有前景的技术,它可以加强蛋白质的3D结构,以解决预测阶段的问题[10].
化学基因组计划建立基于图论的预测模型[11,12]、网络方法[13,14],以及基于机器学习的技术[15,16].在化学基因组学方法中,机器学习方法被认为是最可靠的预测结果。机器学习方法可以分为特征方法和相似方法。
相似度技术已被开发用于计算药物化合物和靶蛋白之间的相似性[17,18].基于相似性的技术包括矩阵分解[13]、基于内核的方法和基于图的方法[11].
特征方法用带有处方载体的载体表示目标药物对。靶点-药物对的不同性质被编码为相关特征。在特征技术中,通过检测最明显的特征来预测DTIs。因此,这些技术的输入是由药物和靶标的特性组合而成的不同载体。这些载体已通过指定编码特征或生物信息学软件包计算,该软件包可以强制计算其化学和生物特征。由于这些向量通常具有多个维度,一些方法使用降维方法来减少特征的数量,从而提高性能模型和预测效率。
在药物-靶标相互作用预测中,药物和靶标都使用了许多类型的特征,例如[19],其中作者使用了构成、拓扑和几何描述符的药物特征向量。所使用的蛋白质特征是氨基酸、伪氨基酸和组成、转变和分布(CTD)描述符。此外,[20.]使用Morgan分子指纹作为药物特征载体,蛋白质特征为20个氨基酸。有许多医学库用于查找这些特性,例如RDKit库[21], RCPI库[22]和PyBioMed库[23].
一些ML技术,如XGBoost [24]、深度学习[16],支持向量机[25],使用最近邻算法可以更有效地发现可能的DTI特征。
我们正在开发一个DTI预测框架,该框架使用了最流行的药物分子指纹,摩根指纹[26],也称为ECFP4扩展传导指纹。摩根指纹以二进制形式生成。摩根指纹经常用于生物活性的预测建模,以允许有意义的化学扩散解码到化学空间。
药物的第二个特征是它的结构描述符,这是最简单的分子描述符,可以从分子结构计算出来。构成配方包括所有代表分子结构的配方,这些配方只涉及化学结构,不包含关于拓扑和一般几何结构的信息。
我们应用蛋白质最常见的特性,即由长链α-氨基酸(α-氨基酸)组成[27].AAC知道每种类型的氨基酸的数量与残基总数归一化。
蛋白质的第二个特征是二肽组成[28,29],它比简单的AAC更有用,后者提供了肽中存在的一对残基的组合。二肽组成比AAC具有更好的特征,因为它包含了氨基酸部分和局部氨基酸种类的信息。
在本文中,我们提出了一个依赖于药物化学结构和性状提取的蛋白质测序的DTI预测模型。我们提出了一种利用支持向量机一类分类器预测负样本的方法,以克服负样本和正样本之间的不平衡问题,然后从负样本和正样本药物目标数据集中构建四个特征集。最后,将这些特征集输入到预测算法中,以确定DTI。
本文的主要贡献总结如下:
- 我。
开发了一种使用支持向量机一类分类器来预测负样本的方法,以处理现有方法无法有效解决的负样本和正样本之间的不平衡问题。
- 2
从4种药物靶点特征和阴性、阳性样本中构建4个特征集。然后,将这些特征集应用于各种类型的机器学习算法来预测DTIs。
- 3
将所提出的方法与现有模型进行了比较,通过在DrugBank数据集上获得最佳性能分数,表明了所提出的模型的优越性。提出的模型的结果优于最近在DTI领域的研究。该模型的平均准确率比现有研究高2.74%,AUC、F-score和MCC分别为1.14、3.53和4.54%。
- 4
提出利用特征重要度和数据集平衡进行特征分析。
本文的结构如下。第二节介绍了现有的DTIs的相关方法。我们提出的框架,以及所使用的技术和数据集的详细描述,将在第3节中介绍。在第4节中,提供了结果和讨论。第5、6节介绍了特征分析、数据平衡以及与最新方法的比较。最后,结论在第7节中描述。
相关工作
近年来,使用机器学习算法的几种方法已经用于DTI预测活动。一般来说,首先使用一个库从输入数据中提取药物和靶标特征。然后对阳性和阴性样本进行识别,输入预测方法。最后,利用评价矩阵对模型进行评价。
表格1显示DTI-SNFRA [30.]的工作分为两个阶段:首先使用SNN,然后使用搜索空间划分组,然后从第一阶段获得的所有可能的药靶相互作用对中计算模糊原始逼近的程度,并选择合适的激发样本少计程度阈值。在[31]和[16]深度学习结构模型发现了目标的局部生存模式,成功地丰富了原蛋白序列的蛋白质优势,导致了比相关方法更好的预测结果。在[32],作者提出了一个基于多核的学习器,伴随着减少的特征和提取预测分数来表明结果,而作者在[33开发了一种FastUS算法来克服类不平衡约束。作者在[20.]提出了一种基于环路和矩阵(PSSM)的DTI预测方法。特别地,LOOP用于从PSSM中提取特征向量。相比之下,[34]使用了药物微笑的(e态)指纹和蛋白质序列的(APAAC)指纹测试的特征。在[35],作者开发了一种新的异质多分子信息网络,由蛋白质和药物之间的n个已知连接组合而成。
材料与方法
建议模型概述
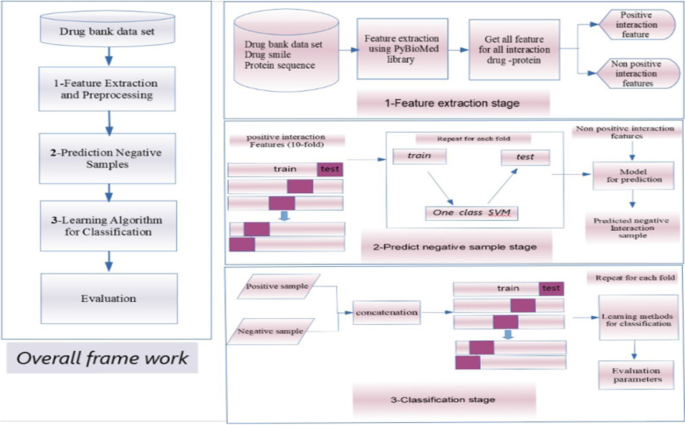
所述框架方法的原理图如图所示。1.最初,药物结构(SMILE格式)和蛋白质序列(FASTA格式)使用访问标识符从DrugBank数据库中聚合。将各种特征提取技术应用于药物和蛋白质序列,以生成不同的特征。特征使用单行支持向量机和已知的相互作用来预测负样本。最后,使用预测算法对框架进行训练,对四个特征集进行分类并对这些算法进行评估。
特征提取
药物输入用简化分子输入线输入系统表示,该系统使用短ASCII字符串描述化学物质的结构。SMILE药物包含完整的化学结构信息,根据其特定的药物ID从DrugBank数据库中汇总。
本文使用PyBioMed软件工具包[23],这是一个响应式功能丰富的python应用程序,用于操作不同文件格式的化学结构,允许它们被分析、转换和存储。PyBioMed [23可以产生18种分子指纹。
在本研究中,第一个药物特征是Morgan指纹,因为它提高了药物研究和分析的效率。为了表示药物性质,SMILE格式转换为Morgan格式,其中分子指纹模式为1024位数字序列。从每个药理化学结构中推导出1024维特征向量。
第二个药物特征是成分描述符,这是最简单和最常用的描述符,它反映了化合物的化学结构,而不涉及其分子几何结构或原子连接。从化合物的化学成分中得到30维特征向量。
对于蛋白质,从FASTA格式的蛋白质序列中提取的特征使用PyBioMed Software Toolkit从DrugBank数据库中整理[23]从蛋白质序列中获得目标特征。这些特征包括氨基酸组成(AAC)和二肽组成(DC)。AAC包含20个元素,每个元素都是蛋白质序列中20个氨基酸中的一个。二肽组成(DC)考虑的是蛋白质序列中每两个AAC残基的比例。DP的主要特点是成对捕获蛋白质序列顺序信息。DP提供400个特性。
负样本预测
在数据集部分,未知交互的数量为58,629,134。然后,构造未知交互特征集。这是存储和处理中的一个主要问题,因此我们试图在这些交互中提出一种新的建议模式来克服数据平衡。
单类SVM是一种无监督算法,用于学习新发现的决策函数:预测与训练包相同或不同的新数据。单类支持向量机算法是通过评估决定超平面上大多数数据距离的概率分布函数来构建的。决策规则将这些观测值以最显著的潜在边际分开[36].学习阶段的计算复杂度很高,因为一类SVM训练涉及四重编程问题。一旦定义了决策函数,就可以预测新测试数据的分层标记。
数字2提供了使用单类支持向量机分类器预测负样本的过程。
我们开发了一种使用单类支持向量机分类器预测负样本的方法。这个算法也可以。
- 1.
确定所有未知交互作用(等于58,629,134个交互作用)。
- 2.
使用单类支持向量机器学习算法将阳性样本分类为超平面,并在10倍交叉验证上执行。经验特征集分为训练特征集和测试特征集。此外,它还用于预测与正超平面的未知相互作用的符号距离。
- 3.
在四个特征集中应用前一步来预测有符号距离,这是所有样本到模型学习的分离超平面的距离。
- 4.
让这些特征集中的参与者构建等于32,802的预测负样本。然后,我们对这些样本进行排序,以得到预测的负样本的小符号距离。
最后,我们使用正交互和负交互(39,866个交互)从表中构建特征集。该算法的伪代码如图所示。2.
预测的方法
我们以前的工作[15]证明了基于集成学习的DTI预测算法在预测药物-靶向相互作用方面是最准确的。本文采用了这些集成学习算法,并与其他机器学习算法进行了比较。
使用了五种不同的预测算法:RF、AdaBoost、XGBoost、Light Boost和SVM。通过10倍CV验证试验,药物靶向特征集大致分为10个亚组。从十组中选取一组作为试验组,其余九组作为训练组,重复此操作(交叉验证)10次。在计算10个验证结果的平均值后,使用不同类型的预测算法从药物目标数据集中创建结果。
- 一)
支持向量机(SVM)
支持向量机是一种优秀的机器学习方法,可用于并行预测和回归问题。预测是通过识别每一类数据最具特征的平面来执行的。该方法中支持向量机的参数为{reg_p = 1.0, kn = ' rbf, ' gama = ' scale '}。
参数说明如下:
reg_p:正则化参数。
kn:指定算法中使用的内核类型。系统默认值为RBF。
gama:它是核心因子
- b)
随机森林(RF)
射频是一种用于预测的集成学习技术。RF适用于来自单个决策树的大规模数据元素。此外,即使在大量数据丢失的情况下,也可以保持精确的射频算法。该方法的参数为{max feature = 0.3, min sample split = 16, num of estimators = 115}。
参数说明如下:
最大特征是考虑分裂节点的最大随机最前特征数。
最小样本分裂是分裂内部节点所需的最小叶子数量。
估计量的数量是算法在取最大投票或取预测平均值之前构建的几棵树。
- 一)
演算法
自适应增强是将权重重新分配给每个条件,将最高权重分配给排名不正确的情况。自适应增强是一种很好的集成技术,广泛应用于并发预测和回归问题。该方法中使用的参数为{splitter = ' best ',最大深度= 6,最小样本分割= 2,算法= " SAMME ",估计器数量= 90}。
参数说明如下:
最小样本分裂是分裂内部节点所需的最小叶子数量。
估计量的数量是算法在取最大投票或取预测平均值之前构建的几棵树。
算法:采用SAMME离散增强算法。
分配器:用于在每个节点上选择分配器的策略。
最大深度:树的最大深度。
- b)
XGBoost
XGBoost根据梯度树增强来优化集成模型,这在预测任务中被广泛使用。该方法使用的参数为{max_depth = 5, learning_rate = 0.2612, n_estimators = int (75.5942), reg_alpha = 0.9925, thread =−1,objective = ' binary: logistic '}。
- 3)
光刺激
Light Boost是一种快速、高性能的统一技术,它使用了像决策树算法这样的分布技术。该方法使用的参数为学习率=[0.001,0.01,0.1,0.2,0.3],动量数=[0.0,0.2,0.4,0.6,0.8,0.9],优化器方法= SGD,目标=二进制,boosting =梯度boosting。
评价参数
用于评估和比较不同技术的药靶相互作用预测的不同措施有[15]如下:
其中TP为真阳性,TN为真阴性,FP为假阳性,FN为假阴性。
曲线下面积:
接收者工作特征(ROC)曲线显示了不同阈值下预测器的性能。
均方误差(MSE)
MSE计算误差平方的平均值。
结果与讨论
在本节中,我们强调实现这四个特征集的DTI预测模型的有效结果。每种技术都通过sci-kit-learn、集成包、Kares库、TensorFlow库和XGBoost包(3.8版)在python语言中应用。使用3.10 GHz英特尔酷睿i9处理器和64.0 GB RAM的Windows 10,算法得到了加速。
数据集
经验药物和靶向数据集来自药物库[5)数据库。药库数据库包括SMILE化学结构和FASTA序列,具有认证的、经验的、营养的、生物技术的和撤回版(组)药物和蛋白质包。我们研究的药物、靶标和实验数据集的相互作用的批准版本在最近发布的DrugBank Online(版本5.1.8,发布于2021-01-03)上。我们的数据集包括11150种药物和5260种蛋白质靶点,其中58,649,000种潜在相互作用,只有19,866种相互作用被标记为积极相互作用,如表所示2.因此,积极相互作用的数量远远低于潜在的消极相互作用。未知相互作用的数量等于58,629,134,导致数据集不平衡。为此,我们提出了一种预测负样本的方法,以控制正面和负面交互数据集之间的不平衡。DrugBank数据集统计数据显示在DrugBank数据库中。
我们将这些数据集应用于特征生成过程,并提取特征。这些特征结合了药物与蛋白质相互作用的四个特征集。表中显示了这些特征集的不同组合3..
现在,我们有五个不同数量的特性集。
负样本预测结果
支持向量机一类学习要求的选择内核用稳定系数来定义边界。即使没有确定带宽因子的精确公式或算法,通常也会选择RBF核。SVM单类学习中的第二个重要参数是ν参数,称为一阶SVM裕度,它对应于找到一个新的,但规则的,可观察到的出界nu等于0.01的可能性。
首先,在单类支持向量机中,使用正样本进行训练,在所有正样本(正超平面)中构建超平面。然后,利用该方法中的决策函数确定未知相互作用与正超平面之间的距离。接下来,在四个特性集中应用这个函数。其次,确定距离的最大负值,这表示距离正超平面的最大离群值。评价结果见表4.
预测算法结果
表中的结果5记录不同技术获得的精度、均方误差、MCC和F-score。使用特征集[1],通过AdaBoost集成学习获得精度得分最高的值为0.9999,Light Boost获得第二好的值为0.9998。
对于特征集[2],通过AdaBoost集成学习和随机森林,获得了最高的精度评分值、最佳召回值、最高的f评分值和最高的精度评分值0.9998。Light Boost获得了第二高的值0.9996。
对于特征集[3],通过AdaBoost集成学习和随机森林获得最佳精度分值、最佳召回值、最佳F-score值和最高精度分值为0.9993。XGBoost获得了第二高的值0.999。
对于特征集[4],通过AdaBoost集成学习和随机森林获得最佳精度分值、最佳召回值、最佳F-score值,最高精度分值为0.999。SVM得到最坏的值进行预测。
对于所有特征集,通过AdaBoost集成学习和随机森林获得最佳精度评分值、最佳召回值、最佳F-score值和最高精度评分值0.9993,SVM获得最差值进行预测。
从之前的结果中发现,特征集1和2的结果比其他的更好,因为它们包含了使用Morgan指纹的药物表示。这证明摩根的指纹比其他特征更能代表毒品。当使用所有特征时,我们发现结果有所下降,这意味着一些特征不能很好地描述药物和蛋白质。在发现的药物特征中,结构描述符在DTIs预测中达到最差的结果。
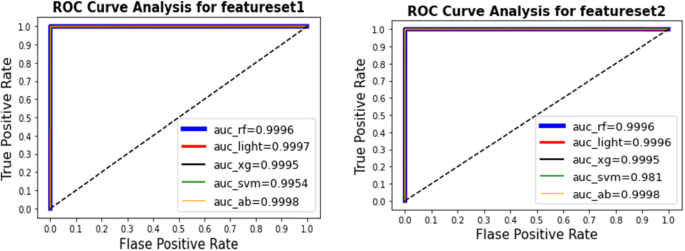
结果见Table6.记录曲线下面积(AUC)、均方误差和不同技术实现的MCC。使用特征集[1],通过AdaBoost集成学习获得AUC最高值为0.9998,Light Boost获得第二优值为0.9997。通过AdaBoost和Light Boost集成学习得到最佳MCC值为0.9996。
对于特征集[2],通过AdaBoost集成学习获得最佳AUC值为0.9998,最佳MCC值为0.9997。Random Forest和Light Boost的第二高值为0.9996。
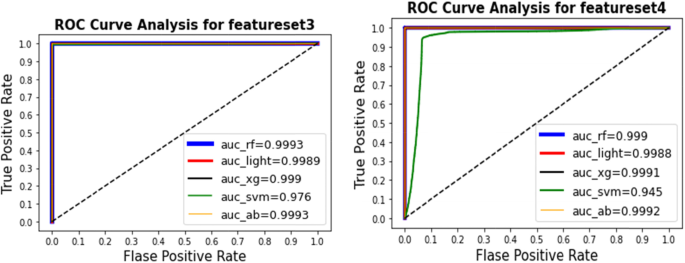
对于特征集[3],通过AdaBoost集成学习和随机森林获得最佳AUC值为0.9993,最佳MCC值为0.9986。XGBoost获得了第二高的值0.999。
对于特征集[4],通过AdaBoost集成学习、随机森林和XGBoost获得最佳AUC值为0.999,最佳MCC值为0.998。AdaBoost集成学习也获得了用于预测的最小均方误差。
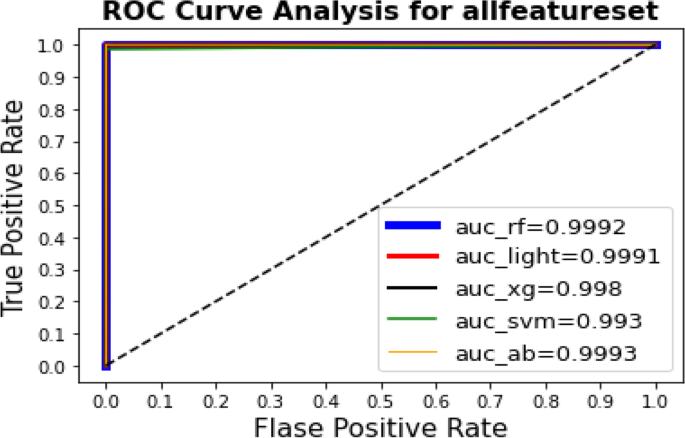
对于所有特征集,通过AdaBoost集成学习获得最佳AUC值为0.9993,最佳MCC值为0.999。此外,AdaBoost集成学习为预测提供了最小的均方误差。
AUC是根据每个模型的AUC曲线来计算的,用于描述工作质量,这为预测DTIs提供了最准确的可视化解释。
数字3.为学习技术的ROC曲线和AUC值。利用特征集(1),通过AdaBoost集成学习得到最佳AUC值为0.9998。对于特征集(2),通过AdaBoost集成学习获得最佳AUC值和最佳MCC值为0.9998。数字4为学习技术的ROC曲线和AUC值。对于特征集(3),通过AdaBoost集成学习和随机森林得到最佳AUC值为0.9993。对于特征集(4),通过AdaBoost集成学习获得最佳AUC值0.999。数字5显示了ROC曲线的结果和学习技术的AUC值。AdaBoost方法预测所有特征集的AUC = 0.9993的最大分数
由于该分类器的缺陷之一是对离群样本敏感,因此使用该分类器可以获得最好的分类结果。这表明,在使用单类SVM分类器预测负样本时,已经删除了非常大比例的离群样本,以提供最佳使用我们的方法。
特性分析
功能的重要性
在这项研究中,我们应用机器学习从使用的不同类型的特征中发现重要的特征。遗传算法[37]和XGBoost是选择的方法,因为与其他方法相比,它们获得了最高的性能。
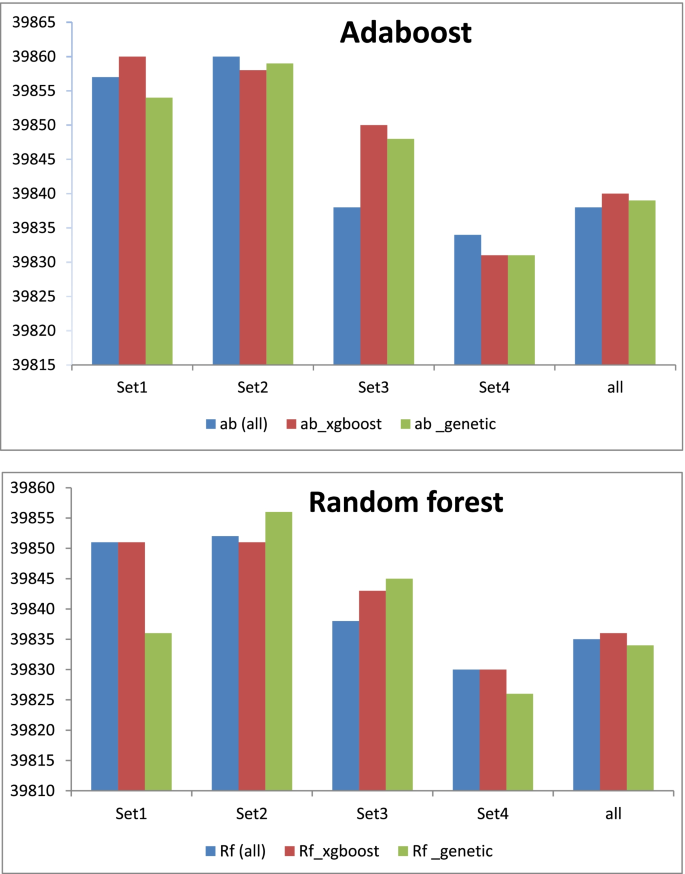
数字6显示了不同学习技术下正确分类样本的数量。利用随机森林,在特征集[2]和特征集[3]中,通过遗传方法获得正确分类样本的最佳数量。对于AdaBoost,通过XGBoost集成学习在特征集[1]、特征集[3]和所有特征集中获得正确分类样本的最佳数量。
欠采样和过采样方法
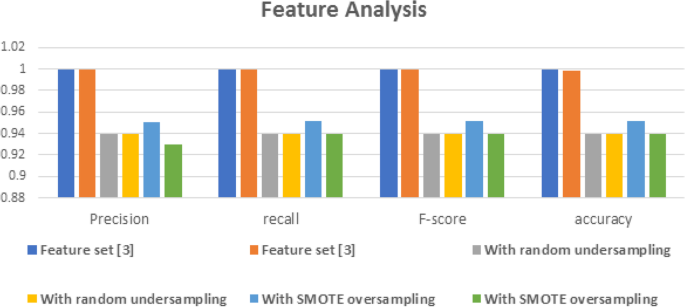
在我们的研究中,我们使用过采样和欠采样的方法来与所提出的模型进行比较,该模型使用随机欠采样技术来进行欠采样方法[38]和用于过采样方法的SMOTE技术[38].
我们的方法超过了所有其他采样不足和过采样的方法,因为我们依赖于通过评估一类支持向量机中的概率分布函数来预测负样本。
数字7表明我们的方法超过了不同学习技术的最佳表现。在功能集[3]中使用随机森林和AdaBoost。最后,我们计算道路的偏差,平均值为0.249。
与最新方法的比较
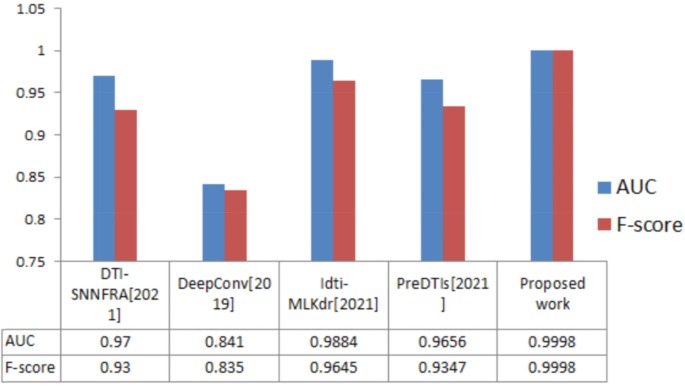
我们的框架与四种方法进行了比较[30.,31,32,33],结果如图所示。8.我们的方法在整个DrugBank中获得了最高的性能,特别是在特征集[2]中。如图所示。8时,我们的框架(最高平均精度= 0.9997)的平均精度比[中的模型高2.74%。32],平均精度比[模型提高10.98%31], AUC、F-score和MCC的均值分别比[模型高1.14、3.53和4.54%。32].
我们的模型得到了最好的结果[31,32]因为我们操作了一类SVM来确定阴性和阳性样本,这比在[中使用聚类算法得到了更好的结果。32].此外,我们在预测阶段使用了它,并且在之前的研究中我们已经证明了集成学习获得了最好的性能。
结论
我们的研究提出了一个新的计算框架,用于使用DrugBank数据集预测DTIs。这一领域存在两个关键的挑战:1)大量的药物和靶标相互作用,创造了广泛的研究领域;2)DTIs的数据集不平衡,因为迄今为止检测到的DTIs很少。因此,阴性样本的数量要比阳性样本的数量大得多。本文的贡献是确定有效预测的阴性样本,以及研究化学和物理特征在评估和发现药物-靶点相互作用中的有效性。
我们发现,使用一类支持向量机预测负样本的过程可能是选择所有样本中尚未检测到的负样本的最佳过程。此外,我们还发现特征集2中的特征,如摩根指纹和二肽组成,在表征过程中是最好的。该方法在预测阶段的性能在DTI预测中具有较高的准确性,特别是在比较各种预测时。该方法在DTI预测中具有较强的稳定性。
在检测药物-靶点相互作用时,我们面临着时间和处理能力的问题。我们已经克服了处理能力的不足,使用了特殊规格的计算机设备来完成工作,但我们仍然有时间的问题。建议采用重构方法对数据进行整体重构,以提高低质量数据的性能。
数据和材料的可用性
在这项研究中产生或分析的所有数据都包含在这篇发表的文章中。
参考文献
Núñez李文杰,李文杰。靶点-药物相互作用:基本原理及其在药物发现中的应用。今天的药物发现。2012; 17(1 - 2): 10-22。
Karine Vuignier JS, Veuthey JL, Carrupt PA, Martel S.药物-蛋白质结合:分析工具的批判性回顾。生物化学。2010;38:53 - 66。
李强,赖磊。基于简单序列性质的潜在药物靶点预测。BMC生物信息学。2007;8:353。
金永久,郭藤生,田边,田边等。大规模分子数据集的整合和解释。核酸研究,2012;40(D1): D109-14。
王志强,郭志强,王志强,等。DrugBank 5.0: 2018年对DrugBank数据库的重大更新。核酸研究,2018;46(D1): D1074-82。
Bento AP, Gaulton A, Hersey A, Bellis LJ, Chambers J, Davies M,等。ChEMBL生物活性数据库:更新。中国生物医学工程,2014;42(D1): D1083-90。
Kuhn M, Szklarczyk D, Pletscher-Frankild S, Blicher TH, Von Mering C, Jensen LJ,等。STITCH 4:整合蛋白质-化学相互作用与用户数据。中国科学通报,2014;42(D1): D401-7。
朱峰,韩波,库马尔P,刘旭,马X,魏X,等。TTD:治疗靶标数据库更新。核酸研究,2010;38(suppl_1): D787-91。
Yamanishi Y, Araki M, Gutteridge A, Honda W, Kanehisa M.从化学和基因组空间整合预测药物-靶点相互作用网络。生物信息学,2008;24 (13):i232-40。
Gönen M.使用贝叶斯矩阵分解从化学和基因组内核预测药物-靶点相互作用。生物信息学,2012,28(18):2304 - 10。
王伟,杨松,李静。基于异构图推理的药物靶点预测。《生物计算》2013。53 - 64页。
blakley K, Yamanishi Y.使用二部局部模型监督预测药物-靶点相互作用。生物信息学。2009;25(18):2397 - 403。
杨晓东,李志强,李志强。基于神经网络的药物-靶点相互作用预测方法研究。生物信息学。2013;29(16):2004 - 8。
陈旭,刘mx,闫光宇。基于异质网络随机游走的药-靶相互作用预测。生物化学学报。2012;8(7):1970-8。
El-Behery H, Attia AF, El-Fishawy N, Torkey H.预测Covid-19药物-靶点相互作用的高效机器学习模型与案例研究。中国生物工程学报。2021;49:107536。
文敏,张震,牛松,沙华,杨锐,云云,等。基于深度学习的药靶相互作用预测。中国生物医学工程学报,2017;16(4):1401-9。
肖旭,闵建林,王萍,周建昌。icdi - pseac - pseac分子指纹识别细胞网络中通道-药物相互作用。中国生物医学工程学报。2013;337:71-9。
Yamanishi Y, Araki M, Gutteridge A, Honda W, Kanehisa M.从化学和基因组空间整合预测药物-靶点相互作用网络。生物信息学,2008;24 (13):i232-40。
马苏迪-内贾德。基于进化信息的PSSM药物-靶点相互作用预测。中国药理学杂志,2016;78:42-51。
詹旭,郑敏,IEEE,蔡杰,李林,于超,潘杰AJK。基于蛋白质序列和药物指纹图谱的集成学习方法预测药靶相互作用。IEEE访问。2020; 12。
Landrum G, Kelley B, Tosco P, sriniker, gedeck, NadineSchneider等。rdkit/rdkit: 2018_03_1(2018年第一季度)发布。https://doi.org/10.5281/zenodo.1222070.
肖n,东升C,青松x。2018.
董健,姚志军,张玲,罗峰,林强,卢爱普,等。PyBioMed:一个python库,用于化学物质、蛋白质和dna的各种分子表示及其相互作用。化学通报,2018;10(1):16。
陈涛,CG。22. XGBoost:一个可扩展的树增强系统。第22届ACM SIGKDD知识发现和数据挖掘国际会议。2016;
王永春,杨志祥,王勇,邓年勇。通过支持向量机计算探测药物-蛋白质相互作用。中国医药杂志,2010;7(5):370-8。
Cereto-Massague A, Ojeda MJ, Valls C, Mulero M, garcia - vve S, Pujadas G.虚拟筛选中的分子指纹相似度搜索。方法。2015;71:58 - 63。
安德烈·毛里,v.c.和罗伯托·托德奇尼,分子描述符。出版:《计算化学手册》,2017。2065 - 2093页。
丁颖,蔡颖,张刚,徐伟。二肽组成对蛋白质热稳定性的影响。中国科学(d辑),2004;
Guruprasad K, Reddy BV, Pandit MW。蛋白质稳定性与其二肽组成之间的相关性:一种从其主要序列预测蛋白质体内稳定性的新方法。中国生物工程学报,1990;4(2):155-61。
Islam SM, Hossain SMM, Ray S. DTI-SNNFRA:基于共享最近邻和模糊粗略逼近的药物-目标相互作用预测。科学通报。2017;16(2):e0246920。
李一,Keum J, Nam H. deepconvc - dti:基于蛋白质序列卷积的深度学习预测药物-靶相互作用。公共科学图书馆,计算生物学,2019;15(6):e1007129。
马穆德,陈伟,贾汉,刘勇,哈桑。基于降维的多核框架药物-靶相互作用预测。化学智能实验室系统。2021;212:13。
陈伟,刘艳,马伟华,马伟华,刘志强。基于梯度助推的药物-靶相互作用预测方法。生物信息学报。2021;22(5):bbab046。
王超,王伟,陆凯,张军,陈萍,王波。电拓扑态指纹图谱和两亲性伪氨基酸组成预测药靶相互作用。国际分子化学杂志,2020;21(16)。
季碧,尤志华,姜海军,郭志华,郑凯。基于LINE网络表示方法的多分子网络药靶相互作用预测。中华医学杂志2020;18(1):347。
金志勇,李志强,李志强,等。基于SVM的药物-靶相互作用预测。公共科学学报,2017;12(2):e017183。
卡托奇,肖汉,库马尔。遗传算法:过去,现在和未来的回顾。中国机械工程,2016;29(5):366 - 366。
穆罕默德R, Rawashdeh J,阿卜杜拉M.机器学习与过采样和欠采样技术:概述研究和实验结果。2020年第十一届信息与通信系统国际会议(ICICS)。2020.243 - 248页。IEEE。
确认
作者要感谢匿名审稿人和编辑的深刻评论。
资金
由科学、技术和创新基金管理局(STDF)与埃及知识银行(EKB)合作提供的开放获取资金。
作者信息
作者及隶属关系
贡献
Heba El-Behery:概念化,方法学,软件,验证,形式分析,调查,资源,数据管理,写作-原创,写作-审查和编辑,以及可视化。Abdel-Fattah Attia:审查和编辑。Nawal El-Fishawy:概念化、监督、审查和编辑。Hanaa Torkey:概念化,方法学,软件,验证,形式分析,调查,资源,数据管理,写作-原创,写作-审查和编辑,和可视化。所有作者都审阅了手稿。作者阅读并批准最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
没有利益冲突需要声明。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
El-Behery, H., Attia, AF., El-Fishawy, N.et al。一种利用多特征信息和数据平衡的基于集成的药靶相互作用预测方法。J生物工程16, 21(2022)。https://doi.org/10.1186/s13036-022-00296-7
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s13036-022-00296-7
关键字
- 药物相互作用
- 数据平衡
- 支持向量机
- 机器学习