摘要
背景
DNA甲基化已被证明与成年肥胖有关。然而,类似的DNA甲基化模式是否与儿童和青少年的体重指数(BMI)有关,这在很大程度上是未知的。在更年轻的时候深入了解这种关系可能会对未来预防肥胖及其相关特征产生影响。
方法
我们使用横断面和纵向模型研究了儿童和青少年脐带血和全血中的DNA甲基化是否与2 - 18岁年龄段的BMI相关。我们对包括23项研究中的4133名儿童在内的全表观基因组关联研究进行了meta分析。我们检查了之前在儿童和成人研究中报告的结果与我们的分析和计算富集结果的重叠。
结果
三个CpGs (cg05937453、cg25212453和cg10040131)的DNA甲基化在不同年龄范围内与BMI具有Bonferroni显著性相关。P< 1.06 × 10−7甲基化每增加10%,其标准差评分(SDS)分别为0.96(标准差(SE) 0.17)、0.32 SDS (SE 0.06)和0.32 BMI SDS (SE 0.06)。横断面儿童模型中9个额外CpGs处的DNA甲基化与BMI存在错误发现率显著性相关。在我们的分析中,187个cpg的DNA甲基化的关联强度与成人BMI相关,随着儿童和青少年年龄的增长而增加。此外,这些cpg对成人和儿童和青少年的影响估计之间的相关系数也增加了。在每个年龄范围的顶级发现中,我们观察到CpGs的富集增加,这是之前在成年人(出生时)中发现的P浓缩= 1;童年P浓缩= 2.00 × 10−4;青春期P浓缩= 2.10 × 10−7).
结论
DNA甲基化与儿童和青少年BMI的相关性很小。随着儿童和青少年参与者年龄的增长,我们观察到与成年BMI相关的DNA甲基化位点改变的重叠增加。这些发现可能与DNA甲基化差异主要是肥胖的结果而不是原因的假设相吻合。
背景
越来越多的证据表明,生命早期的暴露与儿童的身体质量指数有关[1].假设DNA甲基化的变化可能是早期生活暴露与儿童肥胖之间关联的基础[2,3.,4].到目前为止,大多数关于DNA甲基化和肥胖的证据都来自成人研究[5,6,7,8,9].在超过10,000名参与者中,最大的成人表观基因组全关联研究(EWAS)确定了187个位点的DNA甲基化与BMI之间的横断面关联[5].先前关于表观基因组范围内DNA甲基化与儿童和青少年肥胖之间关系的研究规模小且不确定[10,11,12,13,14,15,16].儿童候选基因研究确定了脐带血和儿童血液中DNA甲基化与肥胖指标的关系[17,18,19,20.,21,22,23,24].样本量为40至700人的儿童和青少年表观基因组全关联研究发现,与BMI相关的胞嘧啶-磷酸鸟嘌呤位点数量有限[11,12,13,15,25].尽管一些研究结果表明,DNA甲基化的差异可能在肥胖的发展之前,但最近在成人中使用孟德尔随机化等方法的研究认为,DNA甲基化的改变主要是肥胖的结果,而不是原因[4,5,9,26,27].任何因果通路的方向还没有在儿童中得到有力的评价。在儿童时期获得更多关于DNA甲基化和肥胖之间的联系的知识,可能对未来预防肥胖及其相关特征有意义。
我们对来自23项研究的4133名参与者的BMI全表观基因组相关性研究进行了meta分析。我们评估了儿童期和青春期脐带血DNA甲基化与2-18岁儿童BMI的相关性。我们还比较了影响估计,并检查了我们的数据中是否有丰富的cpg之前确定的其与青少年和成人肥胖的关系。
方法
参与者
我们荟萃分析了脐带血或全血甲基化与儿童或青少年体重指数(BMI)的表观基因组全范围关联研究。我们使用了来自妊娠和儿童表观遗传学(PACE)联盟、生命周期项目和营养计划项目的23项研究中多达4133名参与者的数据1:表S1A-D和附加文件2:补充方法)[28,29]: ALSPAC, BAMSE, CHAMACOS, CHOP研究,CHS, DOMInO试验,GECKO Drenthe队列研究,Generation R研究,GOYA研究,Healthy Start研究,HELIX, INMA, IOW F1, IOW F2, MoBa1, MoBa2, NEST, NFBC 1986, PIAMA研究,PREDO研究,Project Viva, Raine和STOPPA(补充方法中的全称)。队列参与者主要是欧洲血统,但也有(部分)非欧洲种族(非洲人,西班牙人和土著)的队列。大多数队列是准出生队列。我们排除了多胞胎、兄弟姐妹(每个家庭最多一个孩子)、医生诊断的综合征性肥胖病例和任何类型的产妇糖尿病(包括妊娠糖尿病)。所有参与者都获得了知情同意,所有研究都获得了当地伦理委员会的批准(见附加文件)2:补充方法)。
DNA甲基化
使用Illumina Infinium®HumanMethylation450 BeadChip测定法(Illumina, San Diego, CA, USA)测定儿童和青少年脐带血和全血样本中的DNA甲基化[30.].每个队列独立地进行他们首选的质量控制和归一化方法,见附加文件2:补充方法。使用单个CpG位点的未转换归一化beta值作为暴露变量。如果在一个年龄范围内DNA甲基化和BMI的多重测量值可用,我们使用该范围内同一时间点的BMI和DNA甲基化可用的最老年龄。使用以下方法排除外围甲基化beta值:值<(第25百分位−3*四分位范围(3IQR))和值>(第75百分位+ 3IQR)被删除[31].DNA甲基化表现为DNA在特定位点被甲基化的等位基因的比例,因此取值范围从0到1。
儿童体重指数
在每项研究中,测量身高和体重的方法均在附加文件中详细描述2:补充方法。主要结果是BMI,以体重/身高计算2公斤/米2在三个年龄范围内连续测量:2-5岁(幼儿期)、5-10岁(幼儿期晚期)和14-18岁(青春期)。如果有多重BMI和DNA甲基化测量值,我们使用在同一时间点BMI和DNA甲基化可用的年龄范围内的最老年龄的测量值。然后,利用LMSGrowth将BMI值转换为性别和年龄调整标准差评分(SDS) [32,33,34].国际肥胖特别工作组(IOTF)标准用于定义体重过轻、正常体重、超重和肥胖儿童的BMI分界点,该标准是根据英国1990年的生长参考和参与者的BMI、性别和年龄信息制定的[35,36].在二次分析中,我们使用二元结果变量,以正常体重儿童为对照,超重或肥胖儿童为病例。体重过轻的儿童被排除在这些次要分析之外。如果一项研究在其中一组(病例组或对照组)中参与者≤10人,则该研究将被排除在二次分析之外。
协变量
所有模型中包含的协变量为母体协变量:母体年龄、母体教育水平(队列定义)、母体怀孕期间吸烟状况(吸烟vs不吸烟)、母体孕前或早孕BMI和胎次(多胎vs未产)以及出生胎龄。有关特定于队列的收集方法的详细信息,请参见附加文件2:补充方法。我们使用基于参考的Houseman方法和R中minfi包中的Reinius参考来估计白细胞比例(B细胞、CD8+ T细胞、CD4+ T细胞、粒细胞、NK细胞和单核细胞)[37,38,39,40].在R代和ALSPAC研究中,使用脐带血特异性Bakulski参考文献进行敏感性分析[41].使用特定于队列的方法调整了批处理效果,请参见附加文件2:补充方法。儿童期横断面分析中添加的其他协变量是出生体重和母乳喂养。青少年分析还根据青少年年龄、性别、自身吸烟状况和青春期状况进行了调整。青春期状态被分为青春期早期(如果乳房和阴毛Tanner阶段(或类似的分类)都是1、2或3,如果女孩是初潮前期或男孩还没有变声)和青春期晚期(如果乳房或阴毛Tanner阶段(或类似的分类)都是4或5,如果女孩是初潮后期或男孩已经变声)[42,43,44].进一步的细节在研究特定的附加文件中提供2:补充方法。
一些具体的分析
DNA甲基化与儿童或青少年BMI之间的关联在个体研究中进行,所有协变量数据均完整。在涉及多个种族的研究中,每一组分别进行分析。根据预先指定的分析计划,我们对BMI-SDS的连续结果使用稳健线性回归模型,对超重和肥胖与正常体重的病例/对照分析使用广义线性回归模型。EWAS分析使用DNA甲基化在三个时间点进行:出生、童年和青春期,BMI数据收集在三个时间点:早期儿童(2-5岁)、晚期儿童(5-10岁)和青春期(12-18岁)(表1).我们将儿童时期分为早期和晚期,以克服任何年龄特异性的影响以及肥胖反弹对结果的潜在影响[45].根据数据的可用性,队列参与了四项分析中的一项或多项:(分析A)脐带血DNA甲基化与儿童早期BMI的纵向关联(2-5岁;来自13项研究的3295名儿童),(分析B)脐带血DNA甲基化与儿童晚期BMI(5-10岁;来自12项研究的4133名儿童),(分析C)儿童血液DNA甲基化与儿童BMI的横断面关联(2-10岁;来自11项研究的3371名儿童)和(分析D)青少年血液DNA甲基化与青少年BMI的横断面关联(14-18岁;来自7项研究的2842名青少年1).每个分析的参与研究在附加文件中显示1:表S1A-D。
脐带血分析根据产妇年龄、教育程度、吸烟状况、孕前或早孕BMI、胎次、孕龄、批次和估计的细胞类型比例进行调整。儿童期横断面分析还对出生体重和母乳喂养等儿童协变量进行了调整;相比之下,青少年的横断面分析调整了与分析C相同的协变量加上青少年的性别、年龄、吸烟和青春期状态。
荟萃分析
在对所有研究进行质量控制后,我们使用METAL [46,47].所有随访分析均在R [39].meta分析由两个研究小组独立完成,并对结果进行比较。排除仅在一项研究中测量的探针、映射到X和Y染色体的探针以及与交替序列共杂交的探针(交叉反应探针)后,我们纳入分析A的429,959个探针,分析B的429,959个探针,分析C的429,957个探针,分析D的428,967个探针[48,49].在主要荟萃分析的结果文件中,我们标记了映射到包含单核苷酸多态性(SNP)的DNA、重复序列元素或包含INDEL的DNA的探针(附加文件)3.:表S2A-D) [48,49].我们使用Bonferroni校正对多重测试进行校正,该校正给出了显著性阈值P< 1.16 × 10−7(0.05/429,959),以及采用Benjamini和Hochberg方法的较不严格的错误发现率(FDR)阈值[50].EWAS结果总结为每个CpG每增加10%的甲基化,BMI-SDS的平均(和标准误差)差异。我们创建了火山图来可视化影响的大小和方向(甲基化减少或增加)以及统计显著性水平。我们计算了我2统计以探索研究间的异质性。的我2估计每个CpG站点的meta分析结果中由于研究之间的差异而不是随机/抽样变化而产生的变化的比例。异质性被定义为我2>值50,并在森林图中以图形形式显示。我们进行了一项遗漏分析,即我们重复进行主元分析,每次删除23项研究中的一项,以探索是否有任何研究影响了个别结果。我们使用UCSC基因组浏览器增强了Illumina提供的注释。所有注释都使用人工二月2009 (GRCh37/hg19)程序集。我们使用HUGO基因命名法手动更新了所有结果文件中的基因名称,如果在那里没有找到它们,我们于2019年11月5日使用NCBI基因网站[51,52,53].
为了探索BMI分布的极值之间的关系,我们进行了病例/对照分析(超重和肥胖与正常体重的对比)。体重过轻的儿童被排除在这些分析之外,导致样本量为N= 491例和2540例对照(分析A),N= 707例和3217例对照(分析B),N= 644例和2567例对照(分析C),和N= 507例和2188例对照(分析D)4:表S3A-D)。
为了检验我们分析中bonferroni显著性或fdr显著性cpg是否接近BMI snp,我们评估了这些cpg是否位于与儿童体重指数相关的15个遗传位点周围的4 Mb窗口(±2 Mb)内[2,54].对于因为可能受SNP影响而标记的fdr显著cpg,我们在R代研究中目视检查了密度图,以查看这些是否偏离单态(附加文件5:补充信息,图S6)。为了探索显著CpGs周围区域的DNA甲基化模式,我们评估了位于这些CpGs周围10 kb(±5 kb)窗口内的所有CpGs与相关模型中BMI的关联(附加文件)6:表S4)。
敏感性分析
为了探索种族异质性是否可能影响我们的结果,我们重复了荟萃分析,包括只对欧洲血统的参与者(N= 2902(不包括分析A的三项研究),N= 3657(不包括分析B的三项研究),N= 3026(不包括分析C的两项研究)),最大的种族亚群(附加文件7:表S5A-C)。除非在研究特定的补充方法中另有说明,种族定义是使用自我报告的问卷2).我们进行了额外的分析,排除了超重和肥胖儿童的高百分比(> 30%(排除体重过轻儿童后计算的百分比)的研究,以探索是否有任何关联可能是由更极端的BMI值(包括N= 2989(不包括分析A的两项研究),N= 3489(不包括分析B的四项研究),N= 3171(不包括C分析的一项研究)8:表S6A-C)。我们还对所有年龄组进行了第三种保守的敏感性分析,不包括非欧洲人的队列,超重或肥胖儿童比例高(30%)的研究,以及针对任何特定暴露或结果选择或丰富样本的研究(附加文件)9:表S7A-D)。
与以往研究结果的比较
我们在之前的研究中探索了CpG位点与儿童、青少年或成人BMI是否相关。对于先前的候选基因研究和较小的EWASs (N< 1000)时,我们使用bonferroni调整的方法对点击量进行了查找P每项研究的值截止值,因此每项研究的截止值为0.05/(Ncpg来自该研究)(附加文件10:表S8) [7,15,17,18,20.,21,23,24,55].如果一项研究中的特定cpg不在我们的数据集中,我们就查找所有标注到相关基因的cpg [17,24].为了确定先前报道的较大EWASs中CpG位点是否与BMI相关(N≥1000)在我们的cpg中比例过高,且cpg最小P值,我们检查了文献中的顶级cpg与我们分析中的顶级cpg的绝对重叠[5,6,9,25,26,56,57,58,59].后者的定义使用两个截止点:一个严格的截止点P取值< 1 × 10−5和一个更宽容的P值< 0.05。(附加文件11:表S9)。我们使用超几何测试来计算R Stats包中的phyper函数的富集程度。
我们检测了在最大的成人研究中发现的187个cpg (N= 10,261)到目前为止,我们的结果更详细[5].我们使用卡方检验来测试我们的分析中富集的cpg是否显著重叠。我们使用皮尔逊相关系数来检验这187个cpg对成人的影响估计与我们分析结果之间的相关性[5].采用Fisher精确检验,计算不同年龄间的相关系数是否存在显著差异。
功能分析
我们探索了最显著相关cpg的潜在功能解释(P取值< 1 × 10−4)在所有模型中使用基因本体论(GO)和京都基因与基因组百科全书(KEGG)富集分析。我们使用了missMethyl包,它使我们能够根据2020年5月5日版本的GO和2019年10月23日版本的KEGG源数据库,在450K阵列上校正每个基因的探针数量[60].为了过滤掉大型的一般途径,我们将每个基因集的基因数量分别设置在5到2000之间。我们报告的名义P值< 0.05和FDR进行富集(附加文件12、表S10)。
结果
参与者
我们纳入了2842至4133名参与者,他们来自妊娠与儿童表观遗传学(PACE)协会23个独立队列[28].我们评估了儿童早期(2-5岁)脐带血DNA甲基化与BMI的关系(N= 3295,分析A),儿童晚期(5-10岁)脐带血DNA甲基化与BMI (N= 4133,分析B),儿童DNA甲基化伴儿童BMI(2-10岁)(N= 3371,分析C),青少年DNA甲基化与青少年BMI(12-18岁)(N= 2842,分析D)。不同分析中使用的参与者和研究的详细信息见表1额外的文件1:表S1A-D和附加文件2:补充方法。
荟萃分析
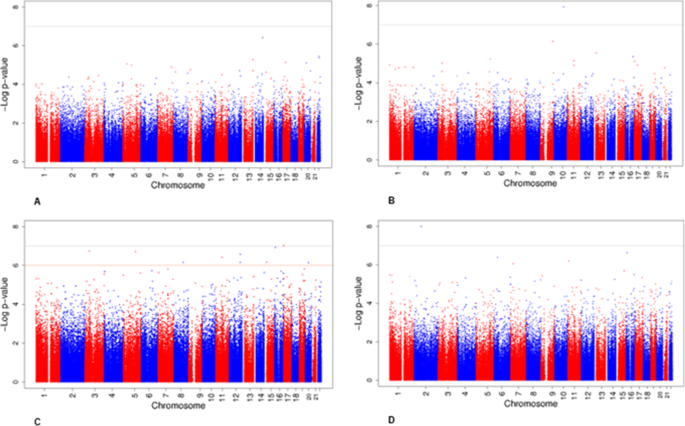
主要、次要和敏感性分析列于表中1.主要荟萃分析的基因组膨胀因子(lambdas)在0.97到1.27之间5:补充信息。1模拟)。所有队列特异性分析的基因组膨胀因子(lambdas)在附加文件中显示1:表S1A-D。主要结果见表2和无花果。1.我们没有确定脐带血DNA甲基化与儿童早期BMI在全基因组意义上的关联(分析A,图。1和附加文件3.:表S2A)。DNA甲基化在一个CpG上,cg05937453 (SFRP5),与儿童晚期BMI显著相关(分析B,图。1b,和附加文件3.:表开通)。脐带血中cg05937453位点DNA甲基化每增加10%,儿童晚期BMI增加0.96 SD(标准误差(SE) 0.17)。CpG处脐带血DNA甲基化与儿童早期BMI名义上显著相关(P值= 0.004),但在横断面分析中,儿童和青少年的DNA甲基化与BMI无关(附加文件13:表S11)。
在横断面分析(分析C)中,儿童DNA甲基化在cg25212453 (SLC43A2)与Bonferroni校正后的儿童BMI相关。cg25212453位点DNA甲基化增加10%与儿童BMI增加0.32 SD (SE 0.06)相关(图。1c和附加文件3.:表S2C)。出生和青春期CpG的DNA甲基化与BMI无关(附加文件13:表S11)。在其他基因中或附近的9个额外CpGs的儿童DNA甲基化与使用FDR的儿童BMI相关P值< 0.05。1c和附加文件3.:表S2C)。青少年的DNA甲基化在cg10040131 (SFXN5)与Bonferroni校正后的青少年BMI相关(分析D,图。1d和附加文件3.表2:d)。cg10040131 DNA甲基化增加10%与青春期BMI升高0.32 SD (SE 0.06)相关。名义上,儿童时期CpG的DNA甲基化与儿童时期BMI显著相关(P值= 0.0002)。脐带血中此CpG的DNA甲基化与儿童BMI的相关性不显著(附加文件13:表S11)。
在任何分析中,DNA甲基化与BMI的关联都没有显示出优先的影响方向(火山图,附加文件5:补充信息,图S2A-D)。我们观察到bonferroni显著相关的CpG位点之间的研究几乎没有异质性的证据我2≤50(附加文件3.:表2a-d和森林图,附加文件5:补充信息,图S3A, B和L)。我们发现了研究间异质性的证据(我2> 50)为9个与fdr显著相关的CpG站点中的3个(附加文件3.:表2c和森林图,附加文件5:补充信息,图S3C-K)。12个Bonferroni或fdr显著相关cpg的结果在一次删除一项研究后是稳定的(删除一项分析,附加文件5:补充信息,图S4A-L)。
当BMI被二分为正常和超重/肥胖时,儿童期横断面模型中只有一个CpG, cg06991974 (PRDM16-DT),显示了关联的证据。在儿童期横断面模型中,包括644名超重/肥胖儿童和2567名正常体重儿童,cg06991974位点的DNA甲基化与儿童期超重/肥胖风险增加相关(比值比(OR) 3.10, 95%可信区间(CI) 2.08, 4.63)(附加文件)4:表S3A-D)。
三个不同年龄范围的三个bonferroni显着CpGs和9个fdr显着CpGs都没有在与儿童体重指数相关的15个已知遗传位点周围的4 mb窗口内[54].
12个FDR显著CpGs中有4个含有单核苷酸多态性(SNP) [48,49].我们没有发现任何这些cpg的非单峰分布的迹象,这表明在这些位点的甲基化测量不受snp的显著影响(附加文件5:补充信息,图S6)。
三个bonferroni显著cpg中的两个(cg05937453和cg25212453)在450K数组上测量的10 kb窗口(±5 kb)内有其他附近的cpg(附加文件6:表S4)。Cg05937453(模型B)被其他24个cpg包围,其中1个名义上与BMI显著相关(P值< 0.05)。两者都位于的TSS200区域SFRP5效果估计在同一方向。Cg25212453(模型C)周围有13个cpg,其中3个名义显著(P值< 0.05)。都位于的基因体中SLC43A2效果估计在同一方向。Bonferroni和fdr显著cpg的结果显示在附加文件中6:表S4。
敏感性分析
当限制在3657个欧洲种族的参与者时,发现与主要结果一致(所有CpG站点的影响估计的Pearson相关系数为0.86-0.97,在顶级CpG站点为0.99 (P取值< 1 × 10−4)用于所有模型(附加文件7:表S5A-C))。同样,当排除了超重或肥胖儿童比例高(> 30%)的研究时,结果也与主要分析一致(所有CpG位点的效应估计的Pearson相关系数为0.89-0.98,最高CpG位点的Pearson相关系数为0.99 (P值< 1 × 10)−4)用于所有模型(附加文件8:表S6A-C))。最后,当非欧洲参与者的研究,超重或肥胖儿童的高比例,以及样本选择或丰富任何特定暴露或结果的研究都被排除在外时,结果仍然与来自主要模型的结果密切相关。所有CpG站点的Pearson相关系数分别为0.64-0.97和0.95-0.99 (P值< 1 × 10-4)的所有模型(附加文件9:表S7A-D)。
与以往研究结果的比较
在之前的候选基因研究或较小的EWASs (N< 1000)没有在我们的结果中复制(附加文件10:表S8) [7,15,17,18,20.,21,23,24,55].当比较来自最大的BMI EWASs (N> 1000)与我们在四个年龄范围内最重要的发现相一致,我们发现随着年龄的增加,重叠增加(表3.和额外的文件11:表S9) [5,6,9,25,26,56,57,58,59].我们使用两个截止点来选择我们结果中最重要的发现:aP取值< 1 × 10−5,以识别“暗示性”的结果,以及不那么严格的、象征性的结果P值< 0.05。符合这些标准的cpg数量列于表中3..首先,我们检查了成人研究和我们的研究结果之间cpg重叠的绝对数量P取值< 1 × 10−5和计算的浓缩。随着儿童期和青春期年龄的增长,我们观察到187个cpg的含量在不断增加,此前在迄今为止最大的一项研究(N= 10261) [5].对于两种脐带血模型,与成人研究结果无重叠(P浓缩= 1),对于儿童期横断面模型2/187成人命中重叠,(P浓缩= 0.0002),对于青少年的横断面模型,3/187重叠(P浓缩= 2.10 × 10−7)(表3.和额外的文件11:表S9)。使用较不严格的截止(P值< 0.05),这一趋势更加明显。成人EWAS的187个cpg与我们数据中的cpg之间的重叠P值< 0.05为8/187 CpGs (P浓缩= 0.77,分析A)脐带血DNA甲基化与儿童早期BMI和11/187 CpGs (P浓缩= 0.30,分析B)脐带血DNA甲基化与儿童晚期BMI的关系。对于儿童期的横断面模型,重叠为61/187 CpGs (P浓缩= 1.97 × 10−20,分析C),在青春期,重叠为77/187 CpGs (P浓缩= 1.68 × 10−44D)3.和额外的文件11:表S9)。在儿童和青少年模型中,有27个cpg是富集的cpg。这种重叠并不显著(P= 0.88)。
四个模型中187次命中的效果估计与cpg效果估计之间的相关系数随年龄增加而增加(分析A =−0.186 (P= 0.01),分析B =−0.013 (P= 0.86), C = 0.604 (P= 5.31 × 10−20,分析D = 0.816 (P= 7.89 × 10−46).所有比较的相关系数差异显著(P的相关系数< 0.01的比较),除模型A和模型B的比较(P= 0.09)。
在我们的分析中,这187个成人BMI cpg的关联效应大小随着我们分析中儿童年龄的增长而增加5:补充信息,图S5)。我们发现成人和青少年中其他EWASs中CpGs的富集有类似的趋势3.)[6,9,25,26,56,57,58,59].这些来自成人研究的发现,有一个命名P值(< 0.05)在我们的模型中,17-35%是由多个成人研究报告的。其中大部分是在两到三个研究中发现的,但有四个,cg06500161、cg19750657、cg12593793和cg18181703,是在之前的六到七个分析中报道的。
功能分析
使用与CpGs相关的基因进行功能富集分析P值< 1 × 10−4在每个模型中均未显示基因本体论(GO)术语或京都基因与基因组百科全书(KEGG)术语的功能富集(FDR < 0.05)12:表S10)。
讨论
在这项儿童和青少年BMI的EWASs大型元分析中,我们发现很少证据表明DNA甲基化与儿童或青少年BMI之间存在关联。三个不同cpg的DNA甲基化,每个cpg都在不同的年龄范围内,与生命早期的BMI相关。在我们的分析中,随着儿童年龄的增长,我们观察到先前确定的与青少年或成人肥胖有关的CpGs的增加。此外,对于之前最大规模的成人BMI研究中发现的187个cpg,我们发现随着年龄的增长,成人效应量和我们分析中发现的效应量之间的相关性也在增加。
对主要调查结果的解释
儿童肥胖是一个主要的公共卫生问题,与短期和长期的发病率和死亡率有关[61].尽管从先前的研究中有一些证据表明DNA甲基化可能介导妊娠相关暴露与后代肥胖的关联,但只有少数特定的CpG位点被识别出来[4,27].到目前为止,大多数关于DNA甲基化与肥胖相关的证据都来自成人研究。
在这项研究中,我们发现几乎没有证据表明DNA甲基化与儿童或青少年BMI之间存在关联。三个CpGs (cg05937453、cg25212453和cg10040131)的DNA甲基化在不同年龄范围内与BMI具有Bonferroni显著性相关。P< 1.06 × 10−7.然而,我们确实观察到,随着我们研究参与者年龄的增加,先前报道的与成人肥胖相关的CpGs的富集和点估计值的增加[5,6,9,25,26,57,58,59].此外,来自成人研究的效果估计与我们模型中的效果估计之间的相关系数随着我们研究参与者的年龄而增加。排除不变探测后(N= 114,204)以成人为参照,随着年龄的增长,与成人肥胖相关的CpGs的富集增加的趋势仍然存在。这一结果表明,据报道探针在成人中是不变的,并没有强烈影响富集分析的结果[62].这些趋势在儿童和青少年的横断面分析中最为明显,尽管两个时间点之间的丰富cpg没有显著重叠。这一趋势可能部分由研究样本量、年龄范围和模型间协变量的差异所解释。这些发现可能表明,随着时间的推移,暴露在更高“水平”的BMI中可能会导致不同的DNA甲基化。DNA甲基化已被证明对环境有反应,也可能对代谢变化和与高BMI相关的脂肪因子/细胞因子环境的改变作出反应[63,64,65].甲基化差异可能是由环境的改变引起的,也可能是在这种改变的环境中细胞选择的结果。如果差异DNA甲基化是暴露于较高BMI的结果,它可能是将肥胖与代谢和心血管疾病联系起来的途径的一部分[5,7].一些研究报告称,肥胖相关CpG位点的DNA甲基化水平与心脏代谢因素有关,如血脂、胰岛素抵抗和血压[26,64].
最近使用孟德尔随机化等方法的研究表明,DNA甲基化的改变主要是肥胖的结果,而不是原因[5,7,9,26].在这些研究中,使用孟德尔随机化研究DNA甲基化和BMI之间潜在的因果关系,独立于未测量的混杂因素,使用遗传变异作为工具变量[66,67].虽然在我们的研究中,我们无法确定这些关联是否有因果关系,但我们的结果可能与这个假设相符。对于先前在我们的数据中报道的与成人和青少年肥胖相关的CpGs的增加,另一种解释可能是不同年龄的BMI并不代表相同的生物表型。DNA甲基化谱可能只是反映了随着时间的推移,儿童BMI向不同的、更像成人的BMI表型的转变。BMI(体重(公斤)/身高(米2))可能在不同年龄有不同的生物学解释,随着年龄的增加,生物学表型与成人BMI更加相似[68].已知特定CpG位点的DNA甲基化随着年龄的变化而变化。在我们的模型中,我们没有看到任何与年龄相关的CpGs的增加,这些CpGs是在之前的儿童和青少年研究中发现的,随着年龄的增长(所有CpGs)P值> 0.19),使我们的结果不太可能代表年龄的强烈影响[69,70].
我们只观察到三个cpg,在三个不同的年龄范围内,DNA甲基化与儿童或青少年的BMI显著相关。脐带血DNA甲基化与儿童晚期BMI相关的Cg05937453被注释为分泌卷曲相关蛋白5 (SFRP5).这个基因是SFRP通过调节Wnt信号转导起作用的家族[71].Wnt家族和SFRPs在多种生物过程中发挥作用,包括胚胎发育、炎症和免疫[72].SFRP5是一种抗炎脂肪因子,可在前脂肪细胞增殖、分化和成熟过程中诱导[65,72].另外两个cpg, cg25212453和cg10040131,以及它们与肥胖的潜在关系,我们所知甚少。在儿童期横断面分析中,在溶质载体家族43个成员2的基因体中,DNA在cg25212453处甲基化(SLC43A2),与BMI相关。SLC43A2在三个队列研究的全血转录组全关联分析中,转录本被描述为与空腹胰岛素相关[73].位于Sideroflexin 5基因体cg10040131的DNA甲基化(SFXN5),与青春期的BMI有关。SFXN5尚未描述与肥胖或相关表型的关系。
根据UCSC Genome Browser提取的路线图表观基因组数据完整收集图绘制的组蛋白标记,所有3个CpG位点与血液中的一个弱转录区域重合,2个CpG位点与脂肪组织中的一个弱转录区域重合,除了cg25212453(在SLC43A2),与脂肪组织中的增强剂相一致[74].这种与关键调控元件的重叠可能表明,这些cpg上的DNA甲基化可能具有调控后果[75,76].
以前许多研究DNA甲基化和儿童BMI之间的关系的研究都不是全基因组的,样本量有限,或仅使用FDR或不太严格的截断值来衡量显著性[10,11,12,13,18,77].先前的候选基因研究报告,CpGs的甲基化注释到原黑色素皮质蛋白(POMC)、类视黄酮X受体(RXRA)和一氧化氮合酶3 (NOS3或以挪士)与儿童时期的体重指数有关[17,24].这些研究的确切cpg要么没有给出,要么没有出现在450K Illumina阵列上,因此无法在我们的数据中进行检查。然而,在我们的数据集中,没有一个注释了这些基因的cpg与我们的分析中BMI相关[17,24].此外,缺氧诱导因子3A (HIF3A),之前曾有报道称甲基化与成人和儿童的BMI存在差异,但在我们的数据中,并没有显示出与儿童或青少年的BMI有任何关联[7,20.,21,23].这一发现与最近发表的两项研究是一致的,两项研究都对大约1000名参与者进行了研究,都没有发现儿童时期的BMI和甲基化之间的联系HIF3A[21,22].
优势和局限性
这一EWAS比之前关于儿童和青少年DNA甲基化与BMI之间关系的全基因组研究要大得多。本研究的其他优势是2至18年的广泛分析,包括纵向和横断面。我们还在PACE联盟中使用了统一的分析计划和稳健的方法。然而,与成人研究相比,本荟萃分析的样本量仍然有限。所有参与的研究都使用了Infinium人类甲基化450K阵列,该阵列仅覆盖了基因组中所有CpG位点的1.7% [78].因此,我们不能排除其他未测量cpg的甲基化可能与儿童BMI有关。450K BeadChip现已被EPIC BeadChip所取代,其中包括> 850,000 CpG站点(美国加利福尼亚州圣地亚哥Illumina) [78,79].一些以前的文献包括本手稿中的一个参与研究。我们分析了儿童和青少年不同时期DNA甲基化和BMI之间的关系,但没有研究同一个体从早期到成年期间DNA甲基化与BMI之间的纵向变化。最近的一项研究对1485名成年人进行了DNA甲基化和BMI的交叉滞后分析,两者都是在两个时间点测量的[26].这些分析表明,BMI与DNA甲基化之间存在显著的单向路径,与其他横断面成人研究一致[5,7].我们用血液来测量DNA甲基化模式与BMI的关系,这可能不是最相关的组织。由于超重和肥胖与血液中的炎症表型有关,并可能影响白细胞组成,血液可能是相关的靶组织[80].然而,有许多可能与BMI相关的靶组织,包括大脑、脂肪细胞、胰腺、肝脏和许多其他组织,而DNA甲基化与BMI的关系在这些组织之间可能不同。在大规模的基于人群的研究中,从这些组织中收集样本几乎是不可能的。一项针对成年人的研究检查了血液和脂肪组织中特定CpG的DNA甲基化与BMI之间的关系是否具有可比性,并显示了组织之间的相似结果[7].我们使用成人参考数据集对儿童和青少年的细胞类型比例进行了估计,这可能不是调整这些年龄的白细胞比例的最佳方法。然而,据我们所知,没有专门针对儿童或青少年的参考小组存在[37,40].因此,我们可能无法完全解释不同年龄血液生物学的潜在差异,这可能对我们的结果产生了一些影响。特定的脐带血参考数据集只有在完成队列特异性分析后才能获得[41,81].然而,在我们最大的两个研究中,我们没有观察到结果有实质性的差异N= 789)和ALSPAC(最大值N= 669),当将我们使用成人参考文献的主要分析与使用脐带血特异性参考样本估计的细胞计数的相同分析进行比较时[37,41].所有450K CpG位点使用成人和脐带血特异性参考面板进行分析的效果估计的相关系数为r= 0.98,r分别为= 0.89。儿童体重指数受遗传、产前和产后环境因素的影响。我们对大量潜在的混杂因素进行了调整。然而,由于其他非测量因素的残留混杂可能仍然存在。参与本荟萃分析的个体研究采用了各自偏好的质量控制和甲基化归一化过程。我们之前已经证明,与使用非标准化甲基化数据相比,这对兴趣关联的影响并不大[82].对23项研究的meta分析可能会引入研究间的异质性。我们进行了多次敏感性分析,结果与主要研究结果相比较。基于我2值,大多数顶级cpg在研究之间没有表现出很大的异质性,尽管有三个fdr显著发现存在异质性。这三个CpG站点有我2取值为50.2、52.7和61.8。森林样地和留一样地没有显示出较大的异质性或一项研究的极端效应(森林样地(附加文件5:补充信息,图S3H, I和K和图S3H, I和K)。目前的分析无法确定是否有任何关联是因果关系。使用孟德尔随机化等方法的未来研究可能进一步阐明因果关系,一些成人研究已经使用了这种方法[5,9,21,83].通过EPIC BeadChip分析BMI与DNA甲基化的关系可以提供新的观点,因为与450K BeadChip相比,EPIC BeadChip查询的CpG位点数量几乎是前者的两倍,并且特别关注潜在调控区域的CpG位点[78,79].此外,亚硫酸氢盐测序法测量DNA甲基化可以提供更详细的信息。在目前的研究中,我们分析了单个CpGs的差异甲基化。未来的研究可以分析差异甲基化的区域模式(差异甲基化区域(DMRs))及其与BMI的关系,以提供进一步的生物学见解。我们主要在一般人群样本中研究BMI。如果暴露在超重环境中已经改变了儿童或青少年时期的DNA甲基化谱,那么在儿童或青少年时期具有更极端肥胖表型的人群中分析这种关联将是很有趣的。为了检验潜在干预措施的效果,研究儿童或青少年减肥前后的DNA甲基化可能是有用的。在成人中,体重减轻已被证明与显著不同的DNA甲基化模式有关[84,85,86].分析同一人群从出生到青春期不同时间点的DNA甲基化和BMI的纵向轨迹,将有助于进一步了解DNA甲基化水平变化和变化模式的生物学相关性[26,87].
结论
在这项针对儿童和青少年的大型表观基因组相关研究meta分析中,我们观察到很少有证据表明个体CpGs的DNA甲基化与儿童或青少年BMI之间存在关联。随着儿童期和青春期年龄的增长,我们观察到效应估计值的增加,成人效应大小与我们分析结果之间的相关性增加,先前确定的与成人肥胖相关的cpg的含量增加。这些发现可能与DNA甲基化差异主要是肥胖的结果而不是原因的假设相吻合,但这仍有待证实。
数据和材料的可用性
与本文主要分析相对应的全基因组DNA甲基化元分析汇总统计数据可在figshare (https://doi.org/10.6084/m9.figshare.13172873)[88].在获得所需批准后,可向相关机构申请获得个别队列水平的数据。有关参与研究的队列的信息可在附加文件中找到2:补充方法。
缩写
- 体重指数:
-
身体质量指数
- CpG:
-
Cytosine-phosphate-guanine
- 背景:
-
脱氧核糖核酸
参考文献
Mitchell EA, Stewart AW, Braithwaite I, Murphy R, Hancox RJ, Wall C,等。儿童和青少年体重指数相关因素:一项国际横断面研究。《公共科学图书馆•综合》。2018;13 (5):e0196221。
Kupers LK, Monnereau C, Sharp GC, Yousefi P, Salas LA, Ghantous A,等。对新生儿表观基因组相关研究的meta分析显示,广泛存在与出生体重相关的DNA甲基化差异。Nat Commun。2019;10(1):1893。
Demetriou CA, van Veldhoven K, Relton C, Stringhini S, Kyriacou K, Vineis P.早期生命暴露与疾病风险的生物嵌入:DNA甲基化的作用。中华临床杂志2015;45(3):303-32。
里士满RC, Timpson NJ, Sorensen TI。探索早期生活环境暴露对肥胖和肥胖发展可能的表观遗传中介作用。国际流行病学杂志2015;44(4):1191-8。
Wahl S, Drong A, Lehne B, Loh M, Scott WR, Kunze S,等。体重指数和肥胖不良后果的表观基因组全关联研究。大自然。2017;541(7635):81 - 6。
Aslibekyan S, Demerath EW, Mendelson M, zhid,关伟,梁磊,等。表观基因组范围的研究发现了与体重指数和腰围相关的新的甲基化位点。肥胖(银泉)。2015; 23(7): 1493 - 501。
Dick KJ, Nelson CP, Tsaprouni L, Sandling JK, Aissi D, Wahl S等。DNA甲基化与体重指数:全基因组分析。柳叶刀》。2014;383(9933):1990 - 8。
Demerath EW, Guan W, Grove ML, Aslibekyan S, Mendelson M,周永华,等。非裔美国成年人BMI、BMI变化和腰围的表观基因组全关联研究(EWAS)发现了多个复制位点。中华医学杂志。2015;24(15):4464-79。
Mendelson MM, Marioni RE, Joehanes R, Liu C, Hedman AK, Aslibekyan S,等。体重指数与血细胞中DNA甲基化和基因表达的关联以及与心脏代谢疾病的关系:孟德尔随机化方法《公共科学图书馆·医学。2017;14 (1):e1002215。
丁旭,郑东,范超,刘铮,董浩,陆勇,等。DNA甲基化的全基因组筛选确定了儿童肥胖的新标志物。基因。2015;566(1):74 - 83。
van Dijk SJ, Peters TJ, Buckley M, Zhou J, Jones PA, Gibson RA,等。新生儿血液中DNA甲基化与儿童早期体重指数和胰岛素敏感性的关系。中华医学杂志。2018;42(1):28-35。
Fradin D, Boelle PY, Belot MP, Lachaux F, Tost J, Besse C,等。全基因组甲基化分析确定了严重肥胖儿童的特定表观遗传标记。Sci众议员2017;7:46311。
黄景荣,Garratt ES,潘浩,吴勇,Davis EA, Barton SJ,等。全基因组甲基化分析鉴定差异甲基化CpG位点与儿童严重肥胖相关。表观遗传学。2015;10(11):995 - 1005。
Kresovich JK,郑毅,Cardenas A, Joyce BT, rifass - shiman SL, Oken E,等。儿童早期和中期脐带血DNA甲基化与肥胖的测量。表观遗传学。2017;9:86。
Rzehak P, Covic M, Saffery R, Reischl E, Wahl S, Grote V,等。学龄前儿童的dna甲基化和身体组成:欧洲儿童肥胖项目(CHOP)研究中的全表观基因组分析。Sci众议员2017;7(1):14349。
He F, Berg A, Imamura Kawasawa Y, Bixler EO, Fernandez-Mendoza J, Whitsel EA,等。肥胖相关基因DNA甲基化与青少年体重指数百分位数的关系。Sci众议员2019;9(1):2079。
戈弗雷KM, Sheppard A, Gluckman PD, Lillycrop KA, Burdge GC, McLean C,等。出生时表观基因启动子甲基化与儿童后期肥胖相关。糖尿病。2011;60(5):1528 - 34。
李志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,等。ANRIL启动子DNA甲基化:晚期肥胖的围产期标记物。EBioMedicine。2017; 19:60 - 72。
clark - harris R, Wilkin TJ, Hosking J, Pinkney J, Jeffery AN, Metcalf BS,等。pgc1α启动子在5-7岁时血液中的甲基化预示9 - 14岁的肥胖(EarlyBird 50)。糖尿病。2014;63(7):2528 - 37。
潘宏,林鑫,吴燕,陈磊,德安,苏雪,等。HIF3A与肥胖的关系:故事开始于出生前。表观基因组学,2015;7(6):937 - 50。
Richmond RC, Sharp GC, Ward ME, Fraser A, Lyttleton O, McArdle WL等。DNA甲基化和BMI:在因果框架中研究已确定的HIF3A甲基化位点。糖尿病。2016;65(5):1231 - 44。
Mansell T, Ponsonby AL, janar V, Novakovic B, Collier F, Burgner D,等。缺氧诱导因子3A基因(HIF3A)甲基化的早期生命决定因素:一项出生队列研究表观遗传学。2019;11(1):96。
王松,宋娟,杨燕,张燕,王红,马娟。HIF3A DNA甲基化与儿童肥胖和ALT的关系。公共科学图书馆-综合[电子资源]。2015; 10 (12): e0145944。
Kuehnen P, Mischke M, Wiegand S, Sers C, Horsthemke B, Lau S,等。POMC基因的一种与Alu元素相关的高甲基化变异与儿童肥胖有关。公共科学图书馆麝猫。2012;8 (3):e1002543。
王旭,潘勇,朱辉,郝刚,黄勇,Barnes V,等。一项非裔美国青年和青壮年肥胖的表观基因组研究:新发现,中性粒细胞的复制,以及与基因表达的关系。表观遗传学。2018;10:3。
孙东,张涛,苏松,郝刚,陈涛,李启智,等。体重指数驱动DNA甲基化的变化:一项纵向研究。中国保监会研究》2019;125(9):824 - 33所示。
Sharp GC, Lawlor DA, Richmond RC, Fraser A, Simpkin A, Suderman M,等。母亲孕前体重指数和妊娠体重增加,后代DNA甲基化和后代肥胖:来自雅芳父母和儿童纵向研究的发现。中华流行病学杂志2015;44(4):1288-304。
Felix JF, Joubert BR, Baccarelli AA, Sharp GC, Almqvist C, Annesi-Maesano I,等。队列简介:妊娠和儿童表观遗传学(PACE)协会。国际流行病学杂志2018;47(1):22-3u。
Jaddoe VWV, Felix JF, Andersen AN, Charles MA, Chatzi L, Corpeleijn E,等。生命周期项目-欧盟儿童队列网络:一个联邦分析基础设施和超过25万名儿童和父母的统一数据。欧洲流行病学杂志2020;35(7):709-24。
Bibikova M, Barnes B, Tsan C, Ho V, Klotzle B, Le JM,等。高密度DNA甲基化阵列,单CpG位点分辨率。基因组学。2011;98(4):288 - 95。
图基JW。探索性数据分析。阅读:addison - wesley;1977.
科尔TJ。构建标准化增长标准的LMS方法。中华儿科杂志1990;44(1):45-60。
Flegal KM, Cole TJ。疾病控制和预防中心2000年生长图表LMS参数的构建。Natl Health Stat Rep 2013(63): 1-3。
Pan H. Cole TJ。LMSGrowth,一个基于LMS方法访问增长引用的Microsoft Excel外接程序。2012年2.77版本。可以从:https://www.healthforallchildren.com/shop-base/shop/software/lmsgrowth/.
科尔TJ,格林PJ。平滑参考百分位曲线:LMS方法和惩罚似然。统计地中海。1992;11(10):1305 - 19所示。
Cole TJ, Bellizzi MC, Flegal KM, Dietz WH。建立全球儿童超重和肥胖的标准定义:国际调查。BMJ。320(7244): 1240 - 2000; 3。
Houseman EA, acacando WP, Koestler DC, Christensen BC, Marsit CJ, Nelson HH,等。DNA甲基化阵列作为细胞混合物分布的替代测量方法。BMC生物信息学。2012;13:86。
Aryee MJ, Jaffe AE, Corrada-Bravo H, Ladd-Acosta C, Feinberg AP, Hansen KD,等。Minfi:一个灵活和全面的Bioconductor软件包,用于Infinium DNA甲基化微阵列分析。生物信息学。2014;30(10):1363 - 9。
R核心团队。R:用于统计计算的语言和环境。维也纳:R统计计算基金会;2013.可以从:http://www.R-project.org/.
Reinius LE, Acevedo N, Joerink M, Pershagen G, Dahlen SE, grereco D,等。纯化的人血细胞中DNA甲基化的差异:细胞谱系和疾病易感性研究的意义。《公共科学图书馆•综合》。2012;7 (7):e41361。
Bakulski KM, Feinberg JI, Andrews SV, Yang J, Brown S, SLM,等。脐带血细胞类型的DNA甲基化:混合细胞出生研究的应用。表观遗传学。2016;11(5):354 - 62。
坦纳JM。青少年的生长与内分泌学。童年内分泌和疾病,童年,加德纳李(编)。桑德斯,费城。1975:14 - 64。
Morris NM, Udry JR.自我管理工具评估青少年发展阶段的有效性。青少年学报。1980;9(3):271-80。
多恩LD,测量青春期。青少年健康杂志。2006;39(5):625-6。
Rolland-Cachera MF, Deheeger M, Bellisle F, Sempe M, Guilloud-Bataille M, Patois E.儿童肥胖反弹:预测肥胖的一个简单指标。中华临床医学杂志。1984;39(1):129-35。
Willer CJ, Li Y, Abecasis GR. METAL:快速有效的全基因组关联扫描元分析。生物信息学,2010,26(17):2190 - 1。
Van der Most PJ, Kupers LK, Snieder H, Nolte I. QCEWAS:表观基因组全关联研究结果的自动质量控制。生物信息学。2017;33(8):1243 - 5。
陈亚,Lemire M, Choufani S, Butcher DT, Grafodatskaya D, Zanke BW,等。在Illumina Infinium HumanMethylation450芯片中发现交叉反应探针和多态CpGs。表观遗传学。2013;8(2):203 - 9。
Naeem H, Wong NC, Chatterton Z, Hong MK, Pedersen JS, Corcoran NM,等。减少错误发现的风险,通过使用HumanMethylation450阵列识别具有生物学意义的全基因组甲基化状态。BMC基因组学。2014;15:51。
控制错误发现率:多重测试的一种实用而强大的方法。中国科学(d辑)1995;
雨果基因命名委员会。genenames.org.进入2019年11月5日。可以从:https://www.genenames.org/.
国家生物技术信息中心。NCBI基因。进入2019年11月5日。可以从:https://www.ncbi.nlm.nih.gov/gene/.
Braschi B, Denny P, Gray K, Jones T, Seal R, Tweedie S,等。Genenames.org: 2019年HGNC和VGNC资源。核酸学报2019;47(D1): D786-D92。
Felix JF, Bradfield JP, Monnereau C, van der Valk RJ, Stergiakouli E, Chesi A,等。全基因组关联分析确定了三个新的儿童体重指数易感位点。中国生物医学杂志,2016;25(2):389-403。
李松,王恩恩,裴敏,阮涛,朱杰,石杰。双胞胎家庭中体重指数与血液DNA甲基化的因果关系研究。国际实用医学杂志2019;43:243-52。
Ali O, Cerjak D, Kent JW Jr, James R, Blangero J, Carless MA,等。在肥胖的全表观基因组关联研究中,SOCS3的甲基化与代谢综合征呈负相关。表观遗传学。2016;11(9):699 - 707。
Campanella G, Gunter MJ, Polidoro S, Krogh V, Palli D, Panico S,等。肥胖与肥胖相关疾病未来风险的表观基因组全关联研究国际实用医学杂志,2018;42(12):2022-35。
葛丽梅,杜格帕,朱杰杰,马卡里克E,荣昌,关伟,等。中老年人血液DNA甲基化与体重指数之间的新联系。中华医学杂志。2018;42(4):887-96。
Sayols-Baixeras S, Subirana I, Fernandez-Sanles A, Senti M, luis- ganella C, Marrugat J,等。DNA甲基化与肥胖特征:一项表观基因组范围的关联研究。2017;12(10): 909-16。
Phipson B, Maksimovic J, Oshlack A. missMethyl:用于分析Illumina公司HumanMethylation450平台数据的R包。生物信息学。2016;32(2):286 - 8。
非传染性疾病风险因素合作。1975年至2016年体重指数、体重不足、超重和肥胖的全球趋势:对1.2890亿儿童、青少年和成人的2416项基于人口的测量研究的汇总分析。柳叶刀》。2017;390(10113):2627 - 42。
Edgar RD, Jones MJ, Robinson WP, Kobor MS.一种基于人类450K甲基化阵列的经验驱动数据还原方法,用于去除组织特异性的非可变cpg。表观遗传学。2017;九11。
Rakyan VK, Down TA, Balding DJ, Beck S.常见人类疾病的表观基因组关联研究。科学通报,2011;12(8):529-41。
金铮,刘燕。人类疾病中的DNA甲基化。基因说。2018;5(1):1 - 8。
王晓燕,王晓燕。肥胖诱导的脂肪组织微环境变化及其对心血管疾病的影响。中国保监会研究》2016;118(11):1786 - 807。
瑞尔顿律师,戴维律师。两步表观遗传孟德尔随机化:建立表观遗传过程在疾病途径中的因果作用的策略。国际流行病学杂志,2012;41(1):161-76。
Richardson TG, Sanderson E, Elsworth, B, Tilling K, Davey Smith G.利用遗传变异分离早期和晚年肥胖对疾病风险的影响:孟德尔随机研究。BMJ。2020; 6 (369): m1203。
科尔TJ。体重/身高比较体重/身高2评估儿童肥胖:年龄和骨龄对青春期p的影响。中华生物学报1986;13(5):433-51。
李超,高伟,高原,于超,吕杰,吕锐,等。6-17岁儿童和青少年的年龄预测:DNA甲基化的表观基因组分析。老化(奥尔巴尼纽约)。2018; 10(5): 1015 - 26所示。
Mulder RH, Neumann A, Cecil CAM, Walton E, Houtepen LC, Simpkin AJ等。从出生到青春期晚期DNA甲基化的表观基因组范围的变化和变异。https://doi.org/10.1101/2020.06.09.142620.
Rattner A, Hsieh JC, Smallwood PM, Gilbert DJ, Copeland NG, Jenkins NA,等。分泌的蛋白质家族与卷曲受体富含半胱氨酸的配体结合域具有同源性。中国科学(d辑:自然科学版)1997;
刘立林,陈晓东,周晓燕,朱强。Wnt拮抗剂及其分泌的卷曲相关蛋白5在脂质代谢、炎症和2型糖尿病中的作用。Biosci众议员2018;(4):38 BSR20180011。
陈bh, Hivert MF, Peters MJ, Pilling LC, Hogan JD, Pham LM,等。空腹血糖和胰岛素浓度的外周血转录组特征。糖尿病。2016;65(12):3794 - 804。
Casper J, Zweig AS, Villarreal C, Tyner C, Speir ML, Rosenbloom KR,等。UCSC基因组浏览器数据库:2018年更新。核酸学报2018;46(D1): D762-D9。
杨文华,陈文华。DNA甲基化与组蛋白修饰的关系:模式与范式。中国生物技术学报2009;10(5):295-304。
组蛋白甲基化:健康、疾病和遗传的动态标志。科学通报,2012;13(5):343-57。
Samblas M, Milagro FI, Mansego ML, Marti A, Martinez JA, Members G. PTPRS和PER3甲基化水平与儿童肥胖相关:来自全基因组甲基化分析的结果Pediatr ob 2018; 13(3): 149 - 158。
所罗门O, MacIsaac J, Quach H, Tindula G, Kobor MS, Huen K,等。Illumina 450K和EPIC beadchip测定新生儿和14岁儿童血液中DNA甲基化的比较表观遗传学。2018;13(6):655 - 64。
Infinium MethylationEPIC BeadChip数据表。可以从:https://science-docs.illumina.com/documents/Microarray/infinium-methylation-epic-data-sheet-1070-2015-008/Content/Source/Microarray/Infinium/MethylationEPIC/infinium-methylation-epic-data-sheet.html.获取日期:2020年8月14日。
Timpson NJ, Nordestgaard BG, Harbord RM, Zacho J, Frayling TM, tybjeger - hansen A,等。c反应蛋白水平与体重指数:通过互惠孟德尔随机化来阐明因果关系的方向。中华医学杂志。2011;35(2):300-8。
Gervin K, Page CM, Aass HC, Jansen MA, Fjeldstad HE, Andreassen BK,等。脐带血中细胞类型特异性DNA甲基化:基于450k参考数据集和细胞计数的估计细胞类型组成验证。表观遗传学。2016;11(9):690 - 8。
Joubert BR, Felix JF, Yousefi P, Bakulski KM, Just AC, Breton C,等。新生儿DNA甲基化与孕妇吸烟:全基因组联盟meta分析。中国生物医学工程学报。2016;38(4):369 - 369。
Richmond RC, Hemani G, Tilling K, Davey Smith G, Relton CL。分子中介研究的挑战和新方法。中国生物医学杂志,2016;25(R2): R149-R56。
Nicoletti CF, Cortes-Oliviera C, Noronha NY, Pinhel MAS, Dantas WS, Jácome A,等。肥胖妇女在短期低热量饮食后DNA甲基化模式发生改变。中华流行病学杂志2020;74(9):1345-53。
Ronn T, Volkov P, Davegardh C, Dayeh T, Hall E, Olsson AH,等。6个月的运动干预会影响人类脂肪组织的全基因组DNA甲基化模式。公共科学图书馆麝猫。2013;9 (6):e1003572。
凌志,罗恩文。人类对规律运动的表观遗传适应。《今日药物发现》2014;19(7):1015-8。
史丹利JR,苏德曼M,辛普金AJ,甘特TR,赫伦J,雷尔顿CL,等。表观遗传轨迹建模的纵向分析策略。国际流行病学杂志2018;47(2):516-25。
Vehmeijer FOL, Küpers LK, Sharp GC, Salas LA, Lent S, Jima DD,等。从出生到青春期的DNA甲基化和体重指数:表观基因组相关研究的元分析。数据集。基因表达综合https://doi.org/10.6084/m9.figshare.13172873(2020)。
确认
meta分析没有具体的确认。所有参与研究的致谢可以在附加文件中找到2:补充方法。
资金
这项元分析的工作获得了欧盟地平线2020研究和创新计划(733206,LIFECYCLE;633595, DynaHEALTH)和欧洲联合规划倡议"健康饮食促进健康生活" (JPI HDHL,营养计划项目,ZonMw,荷兰,no.529051022)。所有参与研究的资助报表可在附加文件中找到2:补充方法。
作者信息
作者和联系
贡献
F.O.L.V L.K.K。,G.C.S A.B。C.L.R, h, V.W.V.J, J.F.F.构思和设计研究。一些具体分析完成L.A.S. (INMA) S.L.(项目万岁)D.D.J.(巢),G.T. (CHAMACOS),狭义相对论(MoBa1)中国区(PIAMA) O.G. (BAMSE) C.P. (MoBa2) f.i.r (F1)较低,歌星P.E.M.(雷恩),“(戈雅),通用电气公司(螺旋),公关(切),A.H. (NFBC 1986), T.G. (STOPPA) S.T.T. (PREDO),低速齿轮(CHS) J.P.R. (DOMInO) A.P.S.(健康)开始,J.W.H. (F2)较低,和P.Y. (CHAMACOS)。G.M.A, L.J.B A.B。,后代e.b., L.C,漆包线,特区,B.E。东南部,N.F。V.G, D.G, S.E.H,刘昀,K.H, R.K。I.K,让•保罗•Langhendries Johanna Lepeule, M.C.M, R.L.M, P.L.M。,.,T.A.M, E.O, K.R。S.R,镉比,砂岩,V.U,汽车,J.M.V, C.X, I.V.Y, H.Z,林文泽,W.K, d, B.S.M, C.V.B, Jari拉赫蒂,C.A。M.J, M.V, b.k. T.I.A.S, r.h.合著,S.H.A, W.N,定位,G.H.K, S.J.L,新罕布什尔州,M.B, S.K.M, M.H.导致一些具体的概念,设计,或收购。F.O.L.V.和L.K.K.对结果进行了meta分析。F.O.L.V, L.K.K和G.C.S.进行随访分析。F.O.L.V, J.F.F. L.K.K。,G.C.S A.B。C.L.R, h, V.W.V.J.解释结果。F.O.L.V在J.F.F、V.W.V.J、L.K.K、G.C.S、a.b.、C.L.R和h.s.的帮助下,完成了手稿的初稿。所有作者都阅读并严格修改了后续的草稿。所有作者都同意提交最终稿件,并同意对工作的所有方面负责,以确保有关工作的任何部分的准确性或完整性的问题得到适当的调查和解决。
相应的作者
道德声明
伦理批准和同意参与
没有为本荟萃分析的目的招募队列。所有队列在通过当地伦理委员会收集数据前获得受试者的伦理批准和知情同意;每个队列的详细信息可以在附加文件中找到2:补充方法。我们的研究符合《赫尔辛基宣言》的原则。
同意出版
不适用。
相互竞争的利益
G.T.获得了750美元的学生/新研究者旅行奖,用于参加2019年9月19日至23日在美国华盛顿特区举行的2019环境与基因组学会(EMS)会议。其余的作者声明他们没有竞争利益。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
补充信息
额外的文件1:
表S1A-D.主要荟萃分析中参与研究的特征。
额外的文件2:
补充的方法.按字母顺序排列的特定研究经费、致谢和方法,包括参考文献。
额外的文件3:
表S2A-D.在主要荟萃分析中,DNA甲基化与BMI之间的关系。cpg的结果p值< 1 x104所示。
额外的文件4:
表S3A-D.DNA甲基化水平与超重/肥胖与正常体重之间的关系。p值< 1x10的cpg结果4所示。
额外的文件5:
补充信息。图S1 - 6.
额外的文件6:
表S4。在每个模型中,距离Bonferroni和fdr显著cpg的+/- 5kb窗口内的cpg。
额外的文件7:
表S5A-C。只有欧洲血统儿童的DNA甲基化与BMI之间的关系。p值< 1x10的cpg结果4所示。
额外的文件8:
表S6A-C。DNA甲基化和儿童BMI之间的关系,不包括30%超重或肥胖的研究。p值< 1x10的cpg结果4所示。
额外的文件9:
表S7A-D。DNA甲基化与儿童和青少年BMI之间的关系,不包括非欧洲血统的研究,不包括超重/肥胖儿童比例高(30%)的研究,也不包括基于特定暴露或结果选择的人群的研究。
额外的文件10:
表S8。在先前报道的候选基因研究和较小的EWAS中查找与BMI相关的cpg (N< 1000)。
额外的文件11:
表S9。先前报道的大型EWAS中与BMI相关的CpGs富集分析结果(N≥1000)。
额外的文件12:
表S10。使用p值< 1-x10的CpGs相关基因进行功能富集分析的结果4在每个模型中使用基因本体论(GO)术语或京都基因与基因组百科(KEGG)术语(FDR <0.05)。
额外的文件13:
表S11。三个bonferroni显著cpg位点的DNA甲基化与所有年龄段的BMI之间的关系。
权利和权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。欲查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
韦梅杰,f.o.l., Küpers, L.K,夏普,G.C.et al。从出生到青春期的DNA甲基化和体重指数:表观基因组相关研究的元分析。基因组医学12105(2020)。https://doi.org/10.1186/s13073-020-00810-w
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s13073-020-00810-w
关键字
- 身体质量指数
- 儿童肥胖
- DNA甲基化
- 表观遗传学