摘要
背景
土壤是农业生产力的关键组成部分,土壤微生物群决定了许多必需植物养分的可用性。土壤的农业驯化,即将以前未开垦的土壤转化为可耕种的土壤,往往伴随着集约化的单一栽培,特别是在发展中国家。然而,对于连续栽培如何改变土壤驯化后原核土壤微生物群的结构,包括作物植物对土壤微生物群组成的影响程度,以及栽培引起的微生物群组成的变化如何影响作物的生产性能,人们的了解有限。
结果
我们在这里表明,在洪水条件下,主要粮食作物水稻的连续单一栽培(> 8个生长季节)与土壤细菌和古细菌微生物群结构向更一致的组成的显著转变有关,从而驯化了以前未耕种的微生物群。除了农业耕作方式的潜在影响外,我们提供的证据表明,水稻植物本身是驯化过程的重要驱动因素,通过在根际中选择性地富集特定分类群,包括产甲烷古菌,这些分类群与生长在相同环境中的本地植物不同。此外,我们发现水稻栽培驯化土壤中的微生物群有助于植物-土壤反馈,通过对水稻幼苗活力产生负面影响。
结论
通过水稻的连续单一栽培,土壤驯化导致土壤微生物群的组成变化,这在一定程度上是由水稻植物驱动的。其后果包括对植物性能的负面影响和温室气体排放微生物的增加。
介绍
植物的根被复杂的微生物群定植,这些微生物群主要来自周围的土壤[1,2,3.,4]。与根相关的微生物群可以通过提高养分利用率而使寄主植物受益[5],排除或防御病原体[6],并通过影响植物激素通路来促进生长[7]。与根相关的微生物群也会对植物生长产生不利影响。利用自然生态系统中的土壤和植物进行的研究发现,与在异种土壤中生长的植物相比,在同种土壤中生长的植物(即以前生长过特定植物物种的土壤)的生物量和生产力会降低[8]。这种效应被称为植物-土壤负反馈,被认为是有害微生物定植的产物[9]以及植物和微生物合成的毒素的积累[10,11]。植物-土壤负反馈主要在非农艺、陆地生态系统的背景下进行了研究,并被认为是一种通过限制排除比竞争对手更不适合的植物来增加生物多样性的机制[12,13]。
对农业环境中的植物-土壤反馈知之甚少,特别是在土壤驯化的背景下,将未开垦的土壤转化为耕种状态的过程,从而破坏自然土壤生态系统和地球化学过程[14]。作物管理措施对根系微生物群落组合的影响[3.,15],最近对花生田的一项研究表明,作物管理,即连续单一栽培与轮作,会改变土壤微生物群落并影响植物生理[16]。有氧栽培的水稻随着时间的推移产量明显下降,这种现象被称为土壤病[17,18]。然而,在淹水的水稻中,并没有这种现象的目击或报道[18,19]。最近的一项研究表明,特定的玉米基因型可以通过根分泌物成分调节栽培土壤,从而影响根相关微生物群的组成,并对茎部生物量产生负面影响[j]。20.]。此外,最近有报道称,玉米种植可以重构草原土壤微生物多样性;然而,观察到的变化归因于农业实践,而不是玉米植物[21]。从这些和早期的研究中产生了一些尚未解决但重要的问题,如源自改变的微生物群的有害影响是否是集约化农业的普遍特征,以及与农业实践相反,作物本身在多大程度上推动了微生物群的这种变化。集约化农业耕作将在满足不断增长的世界人口需求方面发挥关键作用,了解土壤生物因子如何影响作物生长和产量变得越来越重要。然而,对于密集的单一栽培作物如何影响土壤微生物群组成以及微生物群模式如何影响作物生长参数的变化,我们仍然知之甚少。在本研究中,我们研究了以下三个问题:(1)长期种植水稻是否改变了土壤微生物组的细菌和古细菌成分?(2)水稻本身是否在微生物水平上驱动土壤驯化过程?(3)与未驯化土壤微生物组相比,驯化土壤微生物组对寄主植物活力的影响是什么?研究结果揭示了水稻连续栽培对细菌和古细菌土壤微生物群(以下简称微生物群)的影响,以及农业土壤驯化对水稻植株活力的影响。
结果
土壤栽培历史影响植物根系微生物组合
为了评估水稻集约栽培对土壤-根连续体细菌和古细菌多样性的影响,我们调查了在温室水淹条件下栽培和非栽培土壤中生长的水稻植株的散装土壤、根际和内圈群落的原核分类组成(见“方法”一节)。栽培土壤(从这里开始被称为驯化土壤)是从加利福尼亚的稻田中收获的,这些稻田有超过8个季节的水稻单一栽培历史,而未栽培土壤是从稻田附近的两个未开垦的地点获得的(每个地点的位置在附加文件中标注)1:图S1A)。每种驯化和未开垦土壤的土壤化学特征表明,地理而非土壤历史在很大程度上决定了土壤的化学性质1:图S1B,C,附加文件2:表1)。每个未开垦的地点支持不同的本地植物物种(附加文件)1:图S1D)重叠最小。
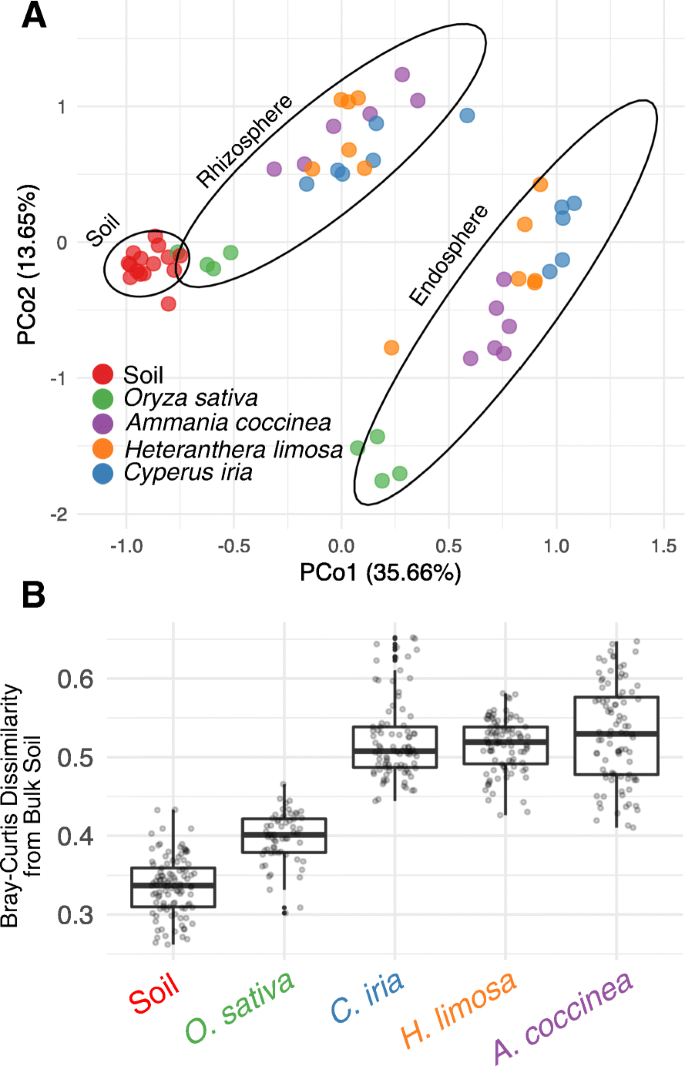
与我们之前的结果一致[3.,22,23],我们发现根室对微生物群落的显著影响(R2= 0.22,P< 0.001, perMANOVA,附加文件2表S2),当使用布雷-柯蒂斯不相似度度量时。从未开垦土壤中获得的根相关微生物群与从驯化土壤中获得的根相关微生物群有显著差异,并且聚集明显(图2)。1一个,R2= 0.18,P< 0.001, perMANOVA,附加文件2表2)。我们注意到土壤历史与根室间存在显著的交互作用项(R2= 0.05,P< 0.001, perMANOVA,附加文件2表2)。当计算其他不相似度量时,也观察到类似的模式(附加文件1:图S2,附加文件2表S3)。虽然各隔室内的原核微生物群均受土壤栽培历史的显著影响,但根际群落受土壤历史的影响大于内圈群落(R2= 0.31 vs. 0.27;P= 0.001, perMANOVA,附加文件2表2)。此外,我们观察到,与驯化土壤相比,未开垦的大块土壤、根际和内圈微生物群的变异性显著增加(附加文件)1:图S3,P< 0.05, Tukey 's诚实组内质心距离显著性差异检验,附加文件2:表S4)。因为居住在土壤中的植物区系组成可能会影响土壤微生物群落组成[24,25],在未开垦土壤之间观察到的差异可以用不同地点之间植物覆盖的差异来解释(附加文件1:图S1D)。
从散装土壤到根际群落的组成转变是根微生物组组装的第一步,涉及宿主介导的特定土壤分类群的补充和消耗。为了评估这种根际效应是否在土壤驯化状态下表现出差异趋势,我们比较了不同栽培历史根际和大块土壤群落之间的两两差异。相对于驯化的样品,未栽培的根际微生物群从各自的散装土壤对照中表现出明显更大的变化。1b,P= 7.14 × 10-26年方差分析)。这一结果表明,在单一栽培下,土壤群落可能会朝着与根际群落逐渐相似的组成状态进行重组。比较不同土壤历史类型,我们发现根际原核微生物群明显比块状土壤样品更相似(图2)。1C,“土壤类型之间”面板)。我们注意到,当比较土壤历史类型时,这不是根际群落比散装土壤群落表现出更少变化的影响。1C,“土壤类型”面板)。这种模式表明,根际微生物群的获取减少了驯化和未驯化的大块土壤群落之间的固有组成差异,因此可以反映土壤驯化在不到一个生长季节的开始。
接下来,我们确定了在驯化和未开垦土壤中生长的植物之间获得性微生物组差异的个体分类群。我们使用DESeq2来鉴定在驯化土壤和未开垦土壤中生长的水稻植株的隔室中富集或耗尽的微生物(附加文件)2表5)。由于本实验分两批进行(参见“方法”部分),我们分别对每个实验批次进行了建模,并发现批次之间驯化和未栽培土壤的每个隔室中otu的重叠显著丰富(附加文件)2表6)。我们发现在驯化土壤中生长的植物的隔室中富集了140个独特的otu(散装土95个,根际106个,内圈16个),而在未栽培土壤中生长的水稻植物的隔室中富集了256个otu(散装土163个,根际109个,内圈83个)。土壤栽培历史不成比例地影响了几个门的OTUs丰度:Euryarchaeota, Armatimonadetes, Acidobacteria, Deltaproteobacteria, Chloroflexi, Firmicutes和Crenarchaeota的OTUs在驯化土壤中生长的植物的隔室中都比预期的更丰富(额外文件)1:图S4,P< 0.05,超几何测试),而γ、β和α变形菌门、双胞菌门、plantomycetes和放线菌门成员在未开垦土壤中组装的微生物组中更不成比例地富集(附加文件)1:图S4,调整后P≤0.05,超几何检验)。产甲烷古细菌在驯化土壤中比在未开垦土壤中生长的水稻植株的隔室中富集。综上所述,这些观察结果支持了水稻种植“驯化”了土壤环境中的微生物群,使其更类似于水稻根际微生物群的假设。
与本地植物相比,水稻获得了独特的微生物群
稻田土壤驯化可能通过多种机制改变现有的土壤微生物群。洪水的遗留问题、化肥和农药的添加以及耕作造成的机械破坏都可能影响土壤的物理化学特性,从而可能重塑微生物群落。此外,寄主-微生物与水稻根系的相互作用,加上密集和连续的单一栽培,也可能在土壤原核生物群落从预栽培到驯化状态的过渡中发挥重要作用。为了解决土壤驯化可能至少部分是由于宿主-微生物与水稻根系的相互作用而导致的假设,我们比较了在阿肯色州琼斯博罗的稻田中,在相同的水淹和管理条件下生长的水稻植物与三种本地植物的根相关微生物组(见“方法”一节):Heteranthera limosa(蓝泥车前草),香iria(flatsedge),Ammania coccinea(谷redstem)。这三个物种没有密切的亲缘关系,前两个分别是百合科和草科的单子叶,第三个是独子叶。成对Bray-Curtis差异的主坐标分析(PCoA)显示,样品可以通过根室和植物种类来区分(图2)。2一个;室:R2= 0.42,P< 0.001;植物:R2= 0.14,P< 0.001, perMANOVA,附加文件2表S7)。使用替代的不相似度量也发现了类似的结果(附加文件1:图S5,附加文件2表8)。隔室与植物种类间存在显著的交互作用项(R2= 0.05,P< 0.011,附加文件2(见表S7),表明不同植物物种的微生物群差异程度取决于根隔室。我们比较了宿主物种对每个隔间之间微生物组组成的影响大小,发现内球微生物组受宿主物种的影响略大(R2= 0.42,P< 0.001,附加文件2:表S7)比根际微生物群(R2= 0.35,P< 0.001,附加文件2表S7)。在根际和内圈,水稻植株的微生物群似乎与本地植物不同,即本地植物支持的微生物群落彼此之间比与水稻更相似。我们进一步证实,在分析中排除水稻植物后,寄主植物物种解释了观察到的群落差异(根际:R2= 0.23,P< 0.001;endosphere:R2= 0.28,P< 0.001, perMANOVA,附加文件2表S7)。这些结果表明,水稻支持的与根相关的微生物群与生长在水稻田中的本地植物不同,并且每种本地植物都有与其他被调查的本地植物不同的微生物群定殖。
PCoA的另一个观察结果是,水稻根际样本比原生植物样本更接近稻田散装土壤样本(图2)。2a).根际群落与散装土的差异的直接比较表明,与原生植物相比,水稻根际微生物群与散装土的群落具有更大的相似性(图2)。2b、附加文件2表S9)。综上所述,这些观察结果表明,除了洪水和其他栽培措施外,水稻植物可能还通过根系选择性地富集和消耗微生物类群来驯化土壤微生物群。
接下来,通过使用DESeq2(附加文件)检测哪些微生物相对丰度存在显著差异,我们研究了哪些otu区分了水稻与本地植物的微生物组1:图S6A,附加文件2:表S10)。通过鉴定与本地植物相比,水稻中通常富集或减少的微生物,我们确定了一组水稻富集和耗尽的核心微生物(附加文件中的固体点)1图S6A和三路相交在附加文件1:图S6B,附加文件2(表S11)。根际稻核富集菌群不成比例地属于酸性菌门、绿柔菌门、Euryarchaeota、gemmatimonadees、Epsilonproteobacteria和CrenarchaeotaP< 0.05,超几何检验;额外的文件1:图S7)。在内球中,水稻核心富集的微生物不成比例地属于三角洲变形菌门、厚壁菌门、Euryarchaeota、Chlorobi和螺旋体(调整后)P< 0.05,超几何检验;额外的文件1:图S7)。另一方面,原生植物富集的核心微生物(即与原生植物相比,从水稻根部持续减少的微生物)主要属于根际Betaproteobacteria, Verrucomicrobia, Bacteroidetes, Planctomycetes和Gammaproteobacteria以及内圈Betaproteobacteria和Gammaproteobacteria(调整后)P< 0.05,超几何检验;额外的文件1:图S7)。
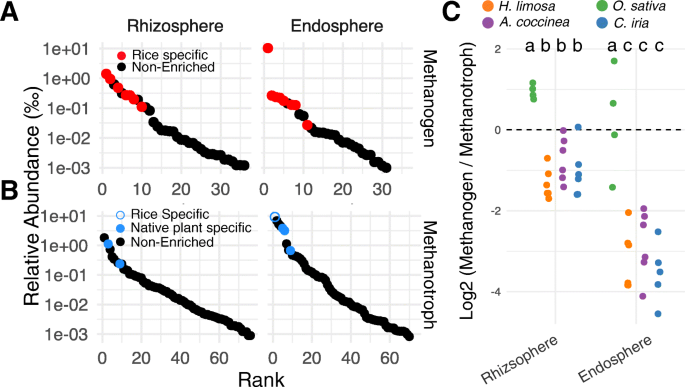
产甲烷古菌是稻田甲烷排放的重要贡献者。在差异丰度的微生物中,我们发现7个OTUs属于产甲烷分类,特别富集于水稻根际,8个OTUs富集于内球。4个otu在根际和内圈稻芯富甲烷菌之间共享。在根际检测到的36个产甲烷otu中,水稻核心富集的otu均位于最丰富的前12位(图2)。3.a).同样在内球中,在31个可检测的产甲烷otu中,水稻核心富集的都在最丰富的11个otu中(图2)。3.a).与水稻相比,我们无法鉴定出本地植物中富集的任何产甲烷OTUs。
接下来,我们分别比较了水稻和本地植物根室的产甲烷相对丰度。我们发现,总的来说,与大块土壤和内圈群落相比,水稻根际具有更大的甲烷菌相对丰度(附加文件)1:图S8),与我们和其他人之前报道的结果相似[3.,26]。然而,当我们对本地植物的根室进行类似的比较时,我们发现大块土壤比根际和内圈群落宿主的甲烷菌丰度要高得多1:图S8)。综上所述,这些结果表明,在淹水条件下,水稻植物而非本地植物在根际富集产甲烷古菌。
产甲烷的真细菌利用甲烷作为能源,从而抵消甲烷的排放。我们在根际数据集中没有发现水稻特有的甲烷营养OTU,并且在水稻核心富集的内圈微生物群中只有一种甲烷营养OTU,尽管这种特殊的OTU是我们的内圈数据集中最丰富的甲烷营养细菌(图2)。3.b).核心原生植物富集微生物含甲烷养菌较多:根际组有2个甲烷养otu,内球组有3个。在根际和内层数据中,原生植物的核心甲烷氧化菌是最丰富的。
因为总CH4通量是产甲烷微生物和产甲烷营养微生物活性的函数,我们接下来比较了每个植物物种中产甲烷古细菌和产甲烷营养细菌的相对丰度比。与内圈相比,根际通常支持更高比例的产甲烷菌和养甲烷菌(图2)。3.c).这是可以预料到的,因为根部在淹水、缺氧的环境中含有最高水平的氧气,而产甲烷菌在有氧条件下繁盛(而产甲烷菌则相反)。我们发现,与生长在相同环境下的原生植物相比,水稻根际和内圈产甲烷微生物的比例均显著高于产甲烷营养细菌。原生植物根际和内圈的平均比值均< 1,而水稻根际和内圈的平均比值均> 1。如果不知道我们数据集中产甲烷菌和氧化甲烷菌的活动水平,就不可能得出关于水稻和本地植物作为甲烷生产者或甲烷汇的效率的明确结论。然而,与本地植物相比,这些数据与水稻根系微生物组在稻田产生CH4中的主要作用是一致的。
水稻核心富集菌群在驯化土壤中表现出富集
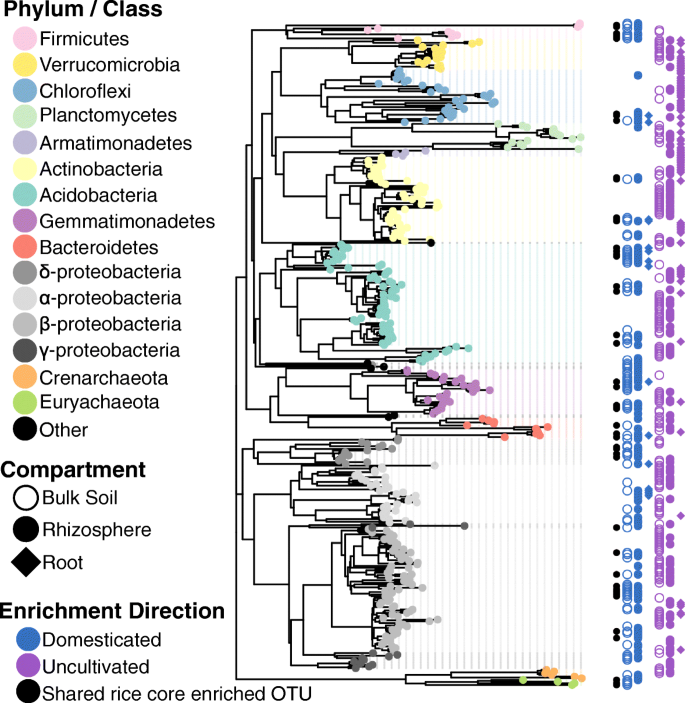
上述结果表明,与生长在相同环境下的本土植物相比,水稻获得了不同的根相关微生物群。此外,我们的研究结果表明,水稻种植与土壤微生物群从野生状态到驯化状态的相当大的转变有关。虽然洪水和养分添加可能在驯化稻田土壤中发挥作用,但我们假设水稻植物本身是通过选择性招募和排除特定微生物来驯化土壤的重要因素。为了支持这一假设,我们可能会期望驯化土壤富集的otu和水稻核心富集的otu存在重叠。我们比较了从驯化土壤中组装的微生物组中发现的显著富集的otu(附加文件)2:表S6)到稻芯富集微生物(附加文件)1:图S6,附加文件2(表S11)。在来自驯化土壤的微生物组中富集的256个独特的otu中,我们发现48个otu与水稻核心富集的分类群重叠(黑色数据点,图2)。4、附加文件2:表S12)。考虑到两个数据集之间不同的微生物群,这种重叠明显大于偶然的预期(P= 1.88 × 10−49(超几何检验)。在重叠的otu中,有两个优势产甲烷古菌类群Methanocella和甲烷八叠球菌属以及属内的四个OTUsAnaerolinea,与产甲烷菌共培养时表现出合作行为[27]。相反,只有8个水稻富集的otu与未开垦土壤富集的otu重叠(P= 0.06,超几何检验)。原生植物富集otu中,只有1个与驯化土壤富集otu重叠,12个与未开垦土壤富集otu重叠(P= 1.41 × 10−5(超几何检验)。考虑到原生植物实验是在美国阿肯色州进行的,而用于土壤驯化实验的土壤是在加利福尼亚州收集的,水稻核心富集的otu和驯化土壤otu之间的重叠程度令人惊讶。这些结果支持了一个假设,即驯化土壤中的微生物群被水稻植物显著地转向了水稻根系特有的组成。
土壤驯化改变了一组分类上多样化的otu的相对丰度。在未开垦和驯化土壤中显示不同数量OTUs的系统发育树。尖端上的彩色点表示特定OTU的门/类。树右边的彩色点表明OTU在隔区(由形状表示)的未开垦(紫色)或驯化(蓝色)群落中是否显著富集。黑点表示与extra文件中鉴定的富含水稻的otu核心集重叠的otu1图S6
与未开垦的土壤相比,驯化的土壤使水稻幼苗活力降低
在确定了土壤栽培历史对水稻根系相关微生物群组成的影响后,我们通过两个独立的实验研究了驯化和未驯化微生物群对幼苗活力性状的影响。土壤养分分析表明,土壤化学随地理位置的变化而不同1(图S1B, C)。因此,为了避免混淆土壤非生物和生物因素(例如,不同的土壤物理和化学性质,潜在的化感化合物和其他根代谢物),我们在接种了土壤来源的微生物群悬浮液的共同生长基质中种植水稻。此外,为了证实观察到的效应是由微生物群的组成差异而不是接种菌群中残留的非生物变异引起的,我们在用无菌悬浮液模拟接种的基质中种植植物(见“方法”一节)。
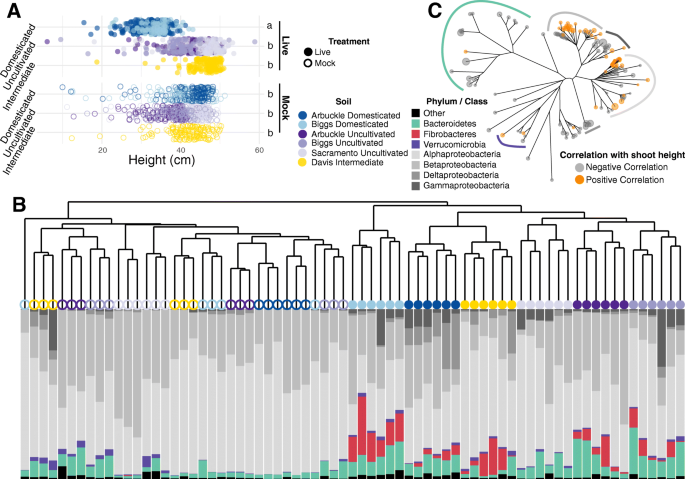
在第一个试验中,惰性煅烧粘土接种了来自两种驯化土壤和三种未开垦土壤的微生物群落。此外,第六个微生物接种物是从一个试验田收获的,该试验田在某些夏季种植水稻,而在其他夏季保持休耕,因此代表了中间土壤类型。与未驯化和中间微生物群的水稻幼苗相比,驯化土壤微生物群的水稻幼苗表现出较低的茎鲜重、干重和高。5a、附加文件1:图S9A,附加文件2(表S13)。在模拟接种的基质中生长的植株显示出均匀的茎部生物量和长度,这表明未栽培和驯化土壤接种体之间的差异本质上是生物学的(图2)。5a、附加文件1:图S9A,附加文件2(表S13)。
驯化与未栽培土壤群落组成差异与水稻植株生长差异有关。一个在煅烧粘土中生长的21日龄水稻幼苗的高度,接种了活的土壤微生物菌群悬浮液(实心圆圈)或模拟悬浮液(空圆圈)。每种颜色代表产生相应接种物的土壤的历史状态和来源。不同的字母表示处理之间的显著差异(Tukey检验;P多重比较调整后< 0.05)。参见附加文件1图S9为额外的性状测量。b基于样本间Bray-Curtis差异的内球群落分层聚类。每个点的形状和颜色分别代表接种物类型和土壤来源,方案与图中相同一个。每个点下面的条形图显示了每个群落中最丰富的门和变形菌纲的相对丰度。c在接种活菌群悬浮液的煅烧粘土中生长的植物,内圈OTUs的系统发育树与幼苗高度显著相关(Wald试验;P多重比较调整后< 0.05)。彩色弧线表示选择分支的门或变形杆菌类,并遵循与面板相同的配色方案b
在第二个试验中,将含泥炭苔藓有机质的砂质植物生长基质UC Mix III接种于两种驯化土壤和两种未开垦土壤。我们再次发现,接种活菌的植株因土壤栽培历史的不同而有显著差异:接种驯化土壤接种菌的植株比未接种土壤接种菌的植株的茎鲜重和茎高显著降低(附加文件)1:图S9B,附加文件2(表S13)。模拟接种的植物在土壤历史状态上没有显著差异,这再次表明我们在驯化和未驯化土壤微生物群的植物之间观察到的幼苗活力性状的差异本质上是生物性的(附加文件)1:图S9B,附加文件2(表S13)。
我们假设土壤类型和接种类型(活的和假的)之间植物生长性状的差异与微生物群结构有关;因此,我们接下来分析了根系相关的细菌和古细菌微生物群。在两个实验中,活体和模拟接种的幼苗所携带的根微生物群明显不同(图2)。5b、附加文件1:图S10,附加文件2表S14):与模拟接种相比,接种活疫苗的幼苗在微生物群结构上表现出更大的差异(附加文件)1:图S10),表明过滤器灭菌/高压灭菌足以破坏微生物群结构。接种了活体土壤群落的幼苗也承载了微生物群,驯化土壤和未栽培土壤之间的分离程度比接种模拟菌的植物要高(附加文件)1:图S10)。试验1中接种中间土壤类型的幼苗的微生物群与驯化土壤类型的微生物群聚集在一起(图2)。5b)尽管这些植物显示出较高的幼苗活力特征(图2)。5a).综上所述,这些结果表明,过滤器灭菌/高压灭菌降低了土壤微生物群之间的差异,表明幼苗生长参数的差异与微生物群结构有关。
接下来,我们寻找相对丰度与幼苗活力性状变异相关的细菌类群。我们从试验1接种了活土壤微生物组的植株中鉴定出151个otu与苗高呈显著的正相关或负相关(图2)。5c、附加文件2:表S15)。在模拟群落中,只有7个OTUs与株高呈显著正相关或负相关,没有一个与活体接种的活OTUs重叠。与土壤活体接种植物的相关OTUs中,62个呈正相关,89个呈负相关,分别包含4个门和9个门。与苗高呈正相关的otu主要由根瘤菌科的类群组成[22],草藻菌科[9],共聚物科[6]和嗜甲基科[4]。负相关的otu在分类上更加多样化,包括29个不同的细菌科。综上所述,这些结果表明,在重复的栽培季节中,微生物的积累会对水稻幼苗的活力产生负面影响。
讨论
土壤是一种重要的农业资源,了解土壤的生物成分如何受到作物种植的影响,以及这些变化如何反过来影响作物性能,对于维持农业生产力至关重要。这项研究表明,长期种植水稻的田地土壤中的微生物群组成与来自地理上相邻地区的未耕作的非农业土壤有很大的不同,因此可能代表了一种前驯化状态。虽然灌溉和养分添加等耕作方法可能是土壤驯化的因素,但我们的研究结果表明,水稻植株本身通过选择性地招募和减少特定微生物,也是土壤驯化过程中微生物群变化的重要驱动因素。4).在相同的田间环境中生长的本地植物在将土壤微生物群转变为驯化状态方面似乎没有明显的作用,因为这些植物获得的微生物群不同于周围土壤,不同于水稻根际和根系,也不同于彼此,并且不是稻田植物群的重要成员。土壤微生物群受植物覆盖的影响[24,25];因此,当农民采用不同的杂草控制策略时,本地植物可能在稻田土壤驯化中发挥更大的作用。
我们进一步描述了微生物群的变化对植物性能的影响。在淹水条件下连续种植水稻显著地使稻田土壤微生物群向更一致的微生物群落结构转变(图2)。1a、附加文件1:图S2),对幼苗活力产生负向影响(图S2)。5a、附加文件1:图S9)与未栽培土壤微生物接种物或灭菌接种物的比较。这种抑制效果是非常有效的,因为它可以观察到200倍稀释的土壤微生物群接种到灭菌的盆栽混合物。以前关于水稻的报告表明,需氧栽培(即不淹水)的水稻容易受到植物-土壤负反馈(也称为土壤病)的影响,并推测非生物因素是连续栽培对水稻性能有害影响的基础[19,28]。考虑到我们的接种物的稀释因素,以及通过过滤消除灭菌后的影响,我们提出生物因素,特别是微生物群的变化,是驯化土壤中植物活力下降的主要因素。我们认为幼苗活力的差异不太可能是化感作用造成的,因为水稻植株只有在暴露于浓度大于100 mg/L的根渗出液时才表现出自毒性[29我们稀释的疫苗无法达到的浓度。
驯化土壤中微生物抑制水稻生长的机制目前尚不清楚。最近对玉米的一项研究发现,生长野生型玉米的农业土壤微生物群在10倍稀释条件下可以抑制玉米的生长,但生长缺乏DIMBOA(一种对食草动物防御很重要的代谢产物)的突变玉米的农业土壤微生物群却没有抑制玉米的生长。20.]。水稻不会产生DIMBOA;因此,DIMBOA渗出不能解释稻田微生物群对生长的抑制,我们发现即使在更高的稀释度下也有效。这些观察结果表明,植物-土壤反馈是作物栽培的一般结果,其中可能涉及多种机制。从我们的研究来看,不可能确定驯化土壤所需的生长季节数量,从而对幼苗活力产生负面影响。然而,我们确实表明,在未开垦的土壤中生长的植物的根际微生物群与在驯化土壤中生长的水稻的根际微生物群比在未种植的驯化土壤和未开垦的土壤中生长的水稻的根际微生物群更相似。1b).这些数据表明,土壤驯化在发芽后6周就已经开始,并可能通过多个季节的栽培而得到加强。我们进一步发现,在稻田休耕超过一个生长季节后接种土壤疫苗的幼苗所携带的微生物群与驯化土壤更相似。5b、附加文件1:图S10)。出乎意料的是,这些幼苗表现出的活力特征等于或大于未开垦的土壤(图2)。5a、附加文件1:图S9A)。这些结果表明,如果水稻种植暂时停止,微生物群给水稻连续种植带来的负面影响可能是可逆的。
在我们的研究中观察到的生长抑制似乎不是由特定的原核生物分类群引起的。与苗高的负相关在细菌门和纲间广泛分布。但与生长的正相关在分布上较为局限,只在根瘤菌目的几个类群中存在。具体来说,我们确定了13个根瘤菌4个农杆菌OTUs, 2个农杆菌OTUsDevosiaOTUs与苗高呈正相关。根瘤菌在自然土壤中广泛分布,最近对原生草原土壤与栽培玉米地的研究也发现了这种模式,尽管没有研究根瘤菌与植物活力和植物-土壤负反馈的可能相关性[qh]21]。有趣的是,最近的一项研究发现,根瘤菌分离菌一般诱导生长促进拟南芥还有一些根瘤菌菌株干扰了mamp触发的免疫反应,可能允许根定植,而不会通过诱导免疫反应对植物生长产生负面影响[30.]。这些结果表明,水稻土壤驯化选择了对植物生长有害的微生物的富集,而牺牲了促进生长的细菌1:图S11)。我们没有研究土壤驯化可能对真菌群落的影响,因此,我们不能排除对水稻生长的反馈效应来自特定真菌分类群。然而,一项研究发现,在单一栽培的田间土壤中生长的花生植株显示出与防御细菌有关的基因表达上调,而不是真菌,这表明至少在该系统中,细菌群落对植物生长有有害影响[16]。这一推论的一个含义是,通过向在驯化土壤中生长的植物提供促进生长的微生物,对这种负面影响进行部分补救可能是可行的。有趣的是,人类和圈养的非人类灵长类动物肠道原核微生物群的主要变化已被证明与工业化社会的典型饮食有关[31,32,33,34]。尽管具体机制可能存在差异,但它们说明了一个类似的基本概念,即工业化和发展可以通过调节微生物组导致意想不到的后果。
这项研究的结果对与农业有关的温室气体的产生也有影响。水田占人为甲烷排放量的15-30% [35,36]。由于甲烷的温室效应潜能值是二氧化碳的25倍[37],预计将增加水稻种植以满足未来需求,因此了解对产甲烷菌的潜在影响非常重要。淹没的土壤,包括沼泽地,维持厌氧条件,已知有利于产甲烷的古细菌[26,38]。然而,除了洪水造成的缺氧环境外,尚不清楚驻留在根际和根组织中的甲烷菌是否表现出植物寄主特异性富集。本研究表明,与生长在相同淹水环境中的原生植物相比,特定的产甲烷古菌在水稻植物的根际和根系中独特地富集。此外,与野生土壤相比,在水稻驯化土壤中生长的水稻植物的微生物群中也富含产甲烷古细菌(图2)。4).这些数据表明,一些优势产甲烷古菌在稻田中的优势可能通过两个步骤的过程促进。淹水条件为产甲烷菌的建立提供了有利的厌氧条件,从而为水稻根际和根组织的定植奠定了基础。与其他似乎不支持产甲烷菌进入根际和内层的水生原生植物相比,水稻植物增强了特定产甲烷菌的定殖(附加文件)1:图S8)。先前的研究表明,古细菌Methanocella是水稻植物来源碳的主要利用者[39,40],其基因组编码碳同化和耐氧途径[41]。在我们的研究中,Methanocella和甲烷八叠球菌属作为水稻土壤中另一种优势产甲烷菌,与未开垦的土壤相比,驯化土壤中甲烷菌含量更高,而且这两种甲烷菌也存在于水稻核心富集微生物中(图2)。4).因此,水稻种植土壤驯化的一个重要副产品是产甲烷古细菌的积累,这可能会产生长期的气候后果。如果水稻是高度活跃的产甲烷菌的强大驱动因素,那么这种积累可能通过在田地内强制水稻种植的不连续性或通过选择甲烷排放量较低的水稻品种来改善[42这可能在甲烷菌的吸收和生长中不那么活跃。
结论
本研究描述了水稻(全球最重要的粮食作物)连续单一栽培对土壤微生物组和植物-土壤反馈产生的驯化后果。研究结果表明,土壤微生物群的组成变化似乎部分是由水稻植物驱动的,而不仅仅是耕作方式的结果。这些微生物群的变化会抑制植物生长,并可能影响农业产量,同时也会导致全球甲烷排放。在未来的研究中应该解决的问题将包括作物轮作时改变的微生物群的持续程度,或者如果耕作暂停或终止,在没有反馈回路的情况下驯化状态的衰变速度,以及与地理相关的未耕作土壤的微生物群组成的恢复程度。
方法
土壤收集与表征
用于土壤驯化和幼苗活力研究的土壤是从加利福尼亚中央山谷的多个地点收集的(附加文件)1:图S1A)。驯化土壤采自Arbuckle(39°00′42.2″N, 121°55′19.6″W)和Biggs(39°27′50.8″N, 121°44′14.4″W)稻田;在Arbuckle(39°00′44.8″N 121°53′09.4″W)、Biggs(39°27′53.0″N 121°43′49.9″W)和Sacramento(38°34′29.6″N 121°38′43.8″W)的非农业地点收获的未开垦土壤;在Davis(38°32 ' 37.9 N, 121°48 ' 44.0″W)的试验田中收获了一种中间土壤。土壤化学分析由UC Davis分析实验室进行。
土壤驯化研究
本研究分两批进行,使用四种不同的土壤(附加文件1:图S1A)。第一批包括来自萨克拉门托的未开垦土壤和来自Arbuckle的驯化土壤(均于2015年4月10日收集),而第二批包括来自Arbuckle的未开垦土壤和来自Biggs的驯化土壤(均于2016年6月3日收集)。土壤均质后放入盆中,在受控温室条件下用去碘水浸泡保存(图2)。6a).一半的花盆保留作未种植的土壤对照,另一半用于移植从表面消毒的去皮种子(70%漂白5分钟,然后在高压去离子水中洗涤三次)中萌发的7天龄的无性系水稻幼苗(品种M206)。在试验期间,植物和土壤在淹水条件下进行灌溉。每14 d向植物和土壤补充一次营养液。移植后6周,收获样品,并立即按照以下步骤处理大块土壤、根际和内圈群落。
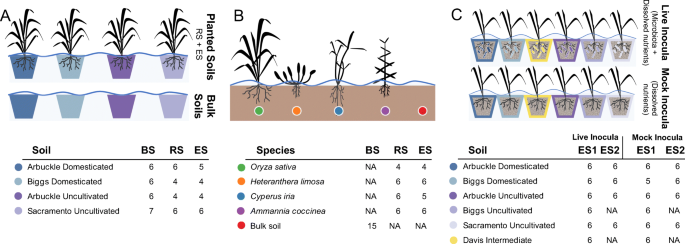
实验设计。一个土壤驯化研究:利用16S rRNA基因分析了水稻在驯化和未驯化土壤中的根际和内圈,了解土壤栽培历史对根系微生物组的影响。此外,对未种植的大块土壤进行采样,以表征土壤类型之间固有的成分差异。种植和未种植的盆栽土壤都在受控的温室环境中浸泡。b本地植物研究:水稻根际和内层(栽培稻)、泥前草(Heteranthera limosa)、莎草(Cypeus iria)和redsystem (Ammania coccinea)对16S rRNA基因进行了分析,以探索在同一水稻田中种植的单一栽培作物和本地植物物种之间的差异。此外,还收集了大量土壤样品,以了解其伴生群落与同种植物(水稻)和其他寄主获得的群落组成关系。c幼苗活力研究:为了分析具有不同栽培历史的土壤微生物组对植物生长的影响,我们将水稻生长在一个共同的基质中,并分别用驯化、未栽培或中间土壤的微生物接种剂进行处理。作为对照,植物在经过相应灭菌的接种物处理的基质中生长,以解释任何残留的非生物变异。此外,我们还对一些植物的内球进行了16S rRNA基因分析,以评估其群落结构。在所有面板中,每个图形下的表格表示每种处理/样品类型组合的重复测序数量。BS、RS和ES分别代表散装土壤、根际和内圈群落;ES1和ES2分别代表第一次和第二次幼苗活力试验收集到的内圈群落。在所有情况下,NA代表不适用
幼苗活力研究
本研究包括两个独立的实验,每个实验都有特定的生长基质、接种处理和收获时间(图2)。6b)。
实验1
该研究包括六种不同的土壤:两种来自Arbuckle和Biggs的驯化土壤;阿巴克尔、比格斯和萨克拉门托三处未开垦的土地;以及中间的戴维斯土壤(均于2018年4月5日收集)。每种土壤均质,舀入锅中,在淹没条件下保存10天。将100 g浸没土加入1 L无菌0.5×强度MS培养基中搅拌,生成土壤悬浮液。对于活菌群的处理,每种土壤悬浮液各500 mL加入10 L两次高压灭菌的煅烧粘土。将接种的底物充分混合,舀入两个72细胞的繁殖托盘中,立即用去离子水底部饱和以达到浸没状态。在模拟接种处理中,除土壤悬浮液沉淀30 min后,收集上清液并进行过滤灭菌(0.22 μm过滤膜,Millipore Sigma, SCGPU10RE),其余步骤与模拟接种处理相同。表面消毒的稻壳种子(10%漂白剂1小时,然后在高压灭菌的去离子水中洗涤三次)然后缝在接种的煅烧粘土中。在12个处理/土壤组合中,每个组合播种144粒种子。 Plants were kept under controlled greenhouse conditions and bottom-irrigated to maintain submerged conditions. Upon harvesting, the shoot height and fresh weight of 21-day-old rice seedlings were immediately registered. The collected tissue was then allowed to dry for 1 week before measuring the dry weight. Additionally, whole root systems (three per tray, six per treatment/soil combination) were collected in sterile PBS for endosphere microbiome characterization following the steps described below.
实验2
该研究包括四种不同的土壤:两种来自阿巴克尔和比格斯的驯化土壤,以及两种来自阿巴克尔和萨克拉门托的未开垦土壤(均于2016年11月收集)。每种土壤均质,舀入锅中,在淹没条件下保存14天。对于活菌群的处理,将18 g浸没土搅拌成1 L 0.5×强度的无菌MS培养基,加入1.8 kg二次蒸压的UC Mix III中。UC Mix III是加州大学校园使用的盆栽土壤混合物,主要由沙子和泥炭苔藓(https://agops.ucr.edu/soil/).对于每种土壤,将接种的基质完全均匀,舀入16个8孔聚丙烯箱中,事先穿孔以允许水流。然后将盒子均匀地分布在两个塑料托盘中,底部用去碘化水饱和。对于模拟接种处理,除了在接种UC Mix III基质前对土壤悬浮液进行高压灭菌外,其余处理均遵循相同的程序。表面消毒的稻壳种子(1%漂白2小时,然后在高压灭菌的去离子水中洗涤三次)然后缝入接种的UC混合物III中。在8个处理/土壤组合中,每个处理共播种256颗种子(聚丙烯箱每个细胞内每孔2颗种子),然后稀释到每个处理128颗幼苗。植物保持在受控的温室条件下,并进行底灌以保持淹没条件。收获后,立即登记14日龄水稻苗的苗高和鲜重。此外,在无菌PBS中收集整个根系(每个托盘三个,每个处理/土壤组合六个),按照以下步骤进行内球微生物组鉴定。
本地植物研究
大米(栽培稻,品种Sabine),河谷系统(Ammania coccinea)、蓝泥前草(Heteranthera limosa)和扁莎草(Cypeus iria)植物(n= 4-6个寄主)于2015年8月22日在阿肯色州琼斯博罗附近的一个淹水稻田收获(图2)。6c).由于植物物候影响根系微生物群组成,从植物繁殖阶段收集根系[4,23,43]。大力摇动根部,去除松散的土壤,收集到50毫升猎鹰管中,加入15毫升无菌PBS溶液。此外,未种植的大块土壤(n= 15)直接收集于50 ml Falcon管中。所有的样本都立即储存在冰上,并连夜运往加州大学戴维斯分校。收到样本后,按如下方法处理大块土壤、根际和内球区室进行DNA提取[44]。简而言之,将收获的水稻根大力摇动以去除松散的土壤,并将其收集到50毫升的猎鹰管中,并加入15毫升无菌PBS溶液。对根进行涡旋处理,收集500 μL的土壤浆液到PowerBead管中提取DNA。然后将根在新鲜PBS溶液中连续洗涤,直到所有土壤都耗尽,并在新鲜PBS中以50 Hz的频率超声三次,持续30秒,以去除所有根面微生物。然后将剩余的根放入PowerBead管中进行内球DNA提取。对于散装土壤样品,直接将~ 250 mg土壤放入PowerBead管中进行DNA提取。所有DNA提取均使用MoBio Powersoil DNA分离试剂盒进行。
16S rRNA基因扩增及测序
所有16S rRNA基因扩增均按[44]。简单地说,采用双标引PCR扩增了16S rRNA基因的V4区。对于每个PCR反应,也进行相应的阴性对照。通过在1%琼脂糖凝胶上运行PCR产物来检查所有反应是否扩增。如果一个反应的阴性对照扩增成功,那么我们放弃特定的反应并重新进行PCR。PCR反应用AMPure珠纯化,并用量子比特测量浓度。PCR产物以等摩尔浓度汇集,用AMPure珠浓缩,然后从2%琼脂糖凝胶中提取凝胶。序列文库送到加州大学DNA技术核心实验室,在Illumina Miseq平台上进行250 × 250 bp测序。
序列处理
生成的成对结束序列使用自定义Python脚本进行解复用[44],并使用PANDAseq [45]。如果包含任何不明确的碱基或长度超过275个碱基,则丢弃连续读取。然后使用NINJA-OPS基于97%的序列同一性将所有读段聚类到otu中[46]。从所有得到的OTU表中删除具有质体和线粒体分类的OTU。
统计分析
所有统计分析均采用R 3.1版本[47]。除非另有说明,我们确定的统计显著性为ɑ= 0.05,并在适当的情况下,使用Benjamini和Hochberg方法对多重假设检验进行校正[48]。对于β -多样性分析,OTU计数使用DESeq2中实现的方差稳定转换进行归一化[49,50]。使用diversity()函数计算Shannon多样性,使用capscale()函数进行PCoA和CAP分析,使用adonis()函数进行perMANOVA分析,计算到群内质心的距离(即附加文件)1(图S3)使用betadisper()函数,使用vegdist()函数计算Bray-Curtis差异,均来自Vegan包装[51]。用DESeq2包进行差异丰度分析[49,50]。线性混合效应模型用lmerTest包拟合[52]。使用betareg R包中的betareg()函数运行Beta回归[53],并使用Stats包中的aov()函数进行方差分析[47]。使用phyper()函数运行超几何测试。使用PhyloSeq包中的plot_tree()命令显示系统发育树[54]。所有其他图形和图表都是使用ggplot2包生成的[55]。
数据和材料的可用性
原始序列可在NCBI的短读档案中访问,项目编号为。PRJNA548898 [56]。所有处理过的数据集都存放在Zenodo存储库中https://doi.org/10.5281/zenodo.3372822[57]。完整分析的R笔记本在GNU通用公共许可证v3.0下可以在GitHub存储库中免费获得https://github.com/bulksoil/SoilDomestication[58]。
参考文献
王晓明,王晓明,王晓明,等。跨越动植物王国的微生物群和宿主营养。细胞宿主与微生物,2015;17(5):603-16。
Finkel m, Castrillo G, Herrera Paredes S, Salas González I, Dangl JL。了解和开发植物有益微生物。植物学报,2017;38(5):559 - 563。
王志强,王志强,Santos-Medellín C,王志强,等。水稻根系相关微生物组的结构、变异和组装。中国科学院学报,2015;112(8):911 - 920。
Lundberg DS, Lebeis SL, Paredes SH, Yourstone S, Gehring J, Malfatti S,等。定义拟南芥核心根微生物组。自然。2012;488(7409):86 - 90。
casstrillo G, Teixeira PJPL, Paredes SH, Law TF, de Lorenzo L, Feltcher ME,等。根系微生物群驱动磷酸盐胁迫和免疫的直接整合。大自然。2017;543(7646):513 - 8。
Berendsen RL, Vismans G, Yu K, Song Y, de Jonge R, Burgman WP,等。植物有益菌群的疾病诱导组合。中国生物医学工程学报,2018;12(6):1496-507。
Bulgarelli D, schlaepppi K, Spaepen S, Ver Loren van Themaat E, Schulze-Lefert P.植物微生物群的结构和功能。植物学报,2013,34(1):444 - 444。
菲茨帕特里克CR, Copeland J,王普文,Guttman DS, Kotanen PM, Johnson MTJ。被子植物根系微生物群的组合及其生态功能。中国科学:自然科学版,2018;35(6):1157 - 1165。
Klironomos约。与土壤生物群的反馈有助于植物在群落中的稀有性和入侵性。自然。2002;417(6884):67 - 70。
李建军,李建军,李建军。化感作用的生态系统和进化背景。生态学报,2011;26(12):655-62。
Lau JA, Puliafico KP, Kopshever JA, Steltzer H, Jarvis EP, Schwarzländer M,等。化感作用的推断由于活性炭对植物生长的影响而变得复杂。中国生物医学工程学报,2009;32(2):444 - 444。
李建军,李建军,李建军,等。土壤群落与植物种群动态关系的研究进展。中国生物医学工程学报,2009;30(5):591 - 591。
Mangan SA, Schnitzer SA, Herre EA, Mack KML, Valencia MC, Sanchez EI等。植物-土壤负反馈预测了热带森林中树种的相对丰度。大自然。2010;466:752。
Amundson R, Berhe AA, Hopmans JW, Olson C, Sztein AE, Sparks DL。土壤科学。21世纪的土壤与人类安全。科学。2015;348(6235):1261071。
Hartman K, van der Heijden MGA, Wittwer RA, Banerjee S, Walser J-C, Schlaeppi K.种植方式操纵根系和土壤微生物群成员的丰度模式,为智能农业铺平了道路。微生物。2018;6(1):14。
李欣,Jousset A, de Boer W, Carrión VJ,张涛,王旭,等。土地利用历史的遗留决定了土壤微生物组对植物生理的重编程。中国医学杂志,2019;13(3):738-51。
George T, Magbanua R, Garrity DP, Tubaña BS, Quiton J.水稻在好氧土壤上连续种植的快速产量损失。杨志强。2002;19(4):1 - 9。
彭s, Bouman B, Visperas RM, Castañeda A,聂璐,朴海坤。热带旱作稻与淹水稻的比较:八季试验的农艺性能。作物科学与技术,2006;32(2):391 - 391。
聂利,彭生,Bouman BAM,黄军,崔凯,Visperas RM,等。减轻好氧单作引起的土壤病害:好氧水稻对土壤烤炉加热的反应。植物学报,2007,30(1):185-95。
胡林,Robert CAM, Cadot S,张欣,叶敏,李斌,等。根分泌物代谢物通过塑造根际微生物群驱动植物-土壤对生长和防御的反馈。生态学报,2018;9(1):2738。
刘建军,刘建军,刘建军,等。美国中西部栽培和原生高草草原土壤的微生物群落结构和功能潜力。中华微生物学杂志,2018;9:1775。
Santos-Medellín C, Edwards J, Liechty Z, Nguyen B, Sundaresan V.干旱胁迫下水稻根系相关微生物群的重组。MBio。8(4): 2017; 18日e00764-17。
Edwards JA, Santos-Medellín CM, Liechty ZS, Nguyen B, Lurie E, Eason S,等。根相关细菌和古细菌微生物群的组成变化跟踪稻田植物的生命周期。生物科学进展,2018;16(2):e2003862。
schlater DC, Bakker MG, Bradeen JM, Kinkel LL。植物群落丰富度和微生物相互作用构成土壤细菌群落结构。生态。2015;96(1):134 - 42。
Prober SM, Leff JW, Bates ST, Borer ET, Firn J, Harpole WS,等。植物多样性可以预测草原土壤微生物的β多样性,但不能预测α多样性。生态学报,2015;18(1):85-95。
李海军,郑世生,金鹏军,Madsen EL, Jeon CO.淹水稻田根际土壤中细菌、古细菌、产甲烷菌和产甲烷菌的高分辨率深度分布。微生物学报,2015;6:639。
Sekiguchi Y, Yamada T, Hanada S, Ohashi A, Harada H, Kamagata Y. . Anaerolinea嗜热细菌gen. nov., sp. 11和Caldilinea嗜热细菌gen. nov., sp. nov.,新的丝状嗜热细菌代表了以前未培养的亚门水平的领域细菌。中华微生物学杂志。2003;33(6):1843 - 1851。
Kreye C, Bouman BAM, Faronilo JE, Llorca L.土壤病害对旱作水稻早期生长的影响。作物科学与技术,2009;32(2):391 - 391。
马勇,张敏,李勇,水军,周勇。水稻根系分泌物化感作用及其与稻瘟虫的关系。和小奥罗本奇Sm。发芽。植物学报,2014;9(1):722-30。
Garrido-Oter R, Nakano RT, Dombrowski N, Ma K-W, AgBiome Team, AC MH,等。根瘤菌根微生物群的模块特性及其与共生根瘤菌的进化关系。细胞宿主微生物,2018;24(1):155 - 667 - e5。
Yatsunenko T, Rey FE, Manary MJ, Trehan I, Dominguez-Bello MG, Contreras M,等。不同年龄和地域的人类肠道微生物组。大自然。2012;486(7402):222。
肠道微生物组:西化和肠道多样性的消失。当代生物学报,2015;25(14):R611-3。
Martínez I, Stegen JC, Maldonado-Gómez MX, even AM, Siba PM, Greenhill AR,等。巴布亚新几内亚农村人的肠道微生物群:组成、多样性模式和生态过程。生物医学进展,2015;11(4):527-38。
Clayton JB, Vangay P, Huang H, Ward T, Hillmann BM, al - ghalith GA,等。圈养使灵长类动物的微生物群人性化。[j] .中国科学:自然科学版,2016;13(3):10376-81。
李建军,李建军,李建军,等。氮肥对水稻根周土壤甲烷氧化的影响。大自然。2000;403(6768):421 - 4。
莱纳H-U。稻田的甲烷排放。生物科学。1993年,43(7):466 - 74。
自然来源的甲烷和一氧化二氮排放;2012.1 - 196页。
张志,Zimmermann NE, Stenke A, Li X, Hodson EL,朱刚,等。湿地甲烷排放在推动21世纪气候变化中的新作用科学通报,2017;35(6):947 - 952。
吕玉华,王晓华,王晓华,等。水稻根际产甲烷古菌的表同位素原位探测。科学。2005;309(5737):1088 - 90。
陈志强,张志强,张志强,等。一种产甲烷古菌的分离与鉴定[J] .微生物学通报,2008,31(4):929 - 936。
Erkel C, Kube M, Reinhardt R, Liesack W.水稻根际甲烷主要产生菌群I古菌基因组。科学。2006;313(5785):370 - 2。
Simmonds MB, Anders M, Adviento-Borbe MA, van Kessel C, McClung A, Linquist BA。直接播种系统中几种水稻品种甲烷和氧化亚氮的季节性排放。环境科学学报,2015;44(1):103。
张健,张宁,刘云霞,张霞,胡斌,秦勇,等。水稻根系微生物群的迁移与田间停留时间和发育阶段有关。中国科学:生命科学,2018;61(6):613-21。
王晓明,王晓明,王晓明,等。水稻根系相关微生物群的提取及16S rRNA序列分析。Bio-Protocol。2018; 8 (12): e2884。
Masella AP, Bartram AK, Truszkowski JM, Brown DG, Neufeld JD。PANDAseq: Illumina序列的成对端组装器。生物信息学,2012;13(1):31。
张建军,张建军,张建军,等。基于基因序列的标记基因序列分析。艾森JA,编辑。计算机工程学报,2016,31(1):563 - 567。
R核心团队。R:用于统计计算的语言和环境。维也纳:R统计计算基金会;2016.
刘建军,刘建军。控制错误发现率:一种实用且强大的多重测试方法。[J] .社会科学与技术。2007;32(1):1 - 4。
Love MI, Huber W, Anders S.使用DESeq2对RNA-seq数据的折叠变化和离散度进行调节估计。基因工程学报,2014;15(12):550。
不浪费,不匮乏:为什么细化微生物组数据是不可接受的。科学通报,2014;10(4):e1003531。
Dixon P. VEGAN,一个社区生态学的R函数包。植物学报,2003;14(4):927 - 930。
Kuznetsova A, Brockhoff PB, Christensen RHB。lmerTest包:测试线性混合效果模型。统计软件学报,2017;82(13):1-26。
李建军,张建军,张建军。基于多变量回归模型的实证分析[J] .计算机工程学报,2010;34(2):24 - 24。
张建军,张建军,张建军,等。微生物群落普查数据的系统分析。科学通报,2013;8(4):61217。
Wickham H. ggplo2:用于数据分析的优雅图形。纽约:斯普林格出版社;2016.
刘建军,李建军,李建军,李建军,李建军。土壤驯化对水稻根系微生物群的影响。NCBI。序列读取归档。(2019)https://www.ncbi.nlm.nih.gov/bioproject/PRJNA548898。2019年10月16日访问。
Edwards J, Santos-Medellin C, Sundaresan V.水稻栽培土壤驯化数据:通过土壤微生物群变化的植物-土壤反馈(1.0版)。数据集Zenodo. 2019。https://doi.org/10.5281/zenodo.3372822。
李建军,李建军,李建军,李建军。水稻种植对土壤微生物群的影响及其对土壤驯化的影响。源代码里。Github。2019。https://github.com/bulksoil/SoilDomestication。2019年10月16日访问。
回顾历史
评审历史记录作为附加文件提供3.。
资金
VS感谢国家科学基金奖IOS 1444974和美国农业部农业实验站项目CA-D-XXX-6973-H的支持。JE感谢Henry A. Jastro研究生研究奖和Elsie Taylor Stocking纪念研究奖学金的部分支持。C.S.M.感谢加州大学墨西哥研究所(UCMEXUS)、墨西哥国家科学研究委员会Tecnología (CONACYT)和Secretaría de Educación Pública (m
作者信息
作者及单位
贡献
JE, CSM, JK, GP和VS构思和设计研究。JE, CSM, BN, JK, ZL, EV和JN进行了研究。JE和CSM分析结果数据。JE, CSM, VS撰写论文。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
这项研究不需要伦理批准。
相互竞争的利益
作者宣称他们没有竞争利益。
额外的信息
出版商的注意
伟德体育在线施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。
补充信息
附加文件1:图S1。
驯化和未开垦土壤的收集点。图S2。加权和未加权Unifrac差异度量揭示了水稻在淹水驯化和未开垦土壤中获得的不同微生物群。图S3。未开垦的群落比驯化的群落在不同的区隔上更容易变化。图S4。不同土壤栽培历史otu的分类丰富程度差异。图S5。加权和非加权Unifrac差异度量揭示了根室和驯化历史对群落组成的影响。图S6。水稻与本地植物的组成差异源于几种otu的同时富集和耗竭。图S7。OTUs在水稻根际和内层群落的分布持续增加或减少。图S8。甲烷菌的相对丰度在大块土壤样本和水稻和本地植物的根际之间存在差异——但方向相反。图S9。驯化的土壤微生物群减少了水稻植株的生长。图S10。接种活菌悬浮液后,水稻内圈群落根据土壤来源的不同而形成。图S11。描述水稻栽培对土壤驯化及其对水稻根相关微生物群和幼苗活力影响的简化模型。
附加文件2:表S1。
驯化试验中驯化和未驯化土壤的土壤化学特征。表S2。土壤驯化研究中Bray-Curtis差异的Permanova统计。表S3。土壤驯化研究中加权和非加权单变量差异度量的持续统计。表S4。土壤驯化研究的β -色散估计。表S5。驯化与未开垦土壤各隔室丰度差异测试结果。表S6。驯化土壤与未开垦土壤中otu含量的差异。表S7。原生植物Bray-Curtis差异的Permanova统计。表S8。原生植物加权和非加权Unifrac差异度量的研究。表S9。Tukey HSD结果用于测试每个植物物种的根际微生物组与散装土壤的不相似性度量。水稻与土体的距离明显小于其他植物。表S10。水稻各室和各植物寄主菌群丰度差异试验。表S11。属于稻芯富集和稻芯枯竭微生物的分类群。表S12。在驯化土壤菌群和水稻核心菌群中富集重叠otu。表向。不同土壤接种剂对幼苗活力性状的事后比较。表S14系列。幼苗活力试验布雷-柯蒂斯差异的Permanova统计。表S15。幼苗活力试验1中OTUs与苗高呈极显著相关。
附加文件3。
回顾历史。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上不受限制地使用、分发和复制,前提是您要适当地注明原作者和来源,提供到知识共享许可协议的链接,并注明是否进行了修改。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有说明。
关于本文
引用本文
爱德华兹,J., Santos-Medellín, C.,阮,B.。et al。水稻土壤驯化通过土壤微生物群的变化导致植物-土壤反馈。基因组医学杂志20., 221(2019)。https://doi.org/10.1186/s13059-019-1825-x
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s13059-019-1825-x