摘要
背景
内聚蛋白是一种重要的基因组结构调节因子,既能调节三维基因组的组织,也能调节基因的表达。核心内聚蛋白三聚体与各种HEAT重复辅助亚基相互作用,产生具有不同组成和潜在不同功能的内聚蛋白复合物。两个相互排斥的HEAT重复序列亚基PDS5A和PDS5B的作用尚不清楚。
结果
在这里,我们确定PDS5A和PDS5B在小鼠胚胎干细胞(mESC)基因组中具有高度相似的定位模式,并且它们与其他内聚物HEAT重复附属亚基STAG1和STAG2有很强的重叠。使用CRISPR/Cas9基因组编辑来生成单个稳定的PDS5A和PDS5B敲除系,我们发现一个PDS5亚基的缺失不会改变另一个PDS5亚基的分布,也不会改变核心内聚蛋白复合体。PDS5A和PDS5B都是基因正常表达所必需的,但它们对基因靶点的作用只是部分重叠。值得注意的是,PDS5 HEAT重复蛋白双缺失后的基因表达与STAG HEAT重复蛋白双缺失引起的基因表达变化并不完全重叠,尽管这四种蛋白的基因组分布都是重叠的。此外,PDS5A和PDS5B的双重缺失降低了内聚蛋白与NIPBL和WAPL的关联,降低了SMC3乙酰化,并且不改变基因组上的内聚蛋白的总体水平。
结论
这项工作揭示了PDS5A和PDS5B对适当内聚功能的重要性。任何一个亚基的缺失对整个基因组内聚蛋白定位的影响都很小,但PDS5A和PDS5B对基因表达的要求不同。
背景
内聚蛋白复合体在整个细胞周期中动态地构建染色体,在间期介导DNA环,并在DNA复制后维持姐妹染色单体的内聚,直到有丝分裂[1,2].内聚蛋白由三个核心亚基SMC1A、SMC3和RAD21组成,并与多种HEAT重复蛋白相互作用,这些蛋白有助于不同的内聚蛋白功能。例如,当内聚蛋白与DNA结合并形成DNA环时,它与两个HEAT重复附属亚基之一STAG1或STAG2具有互斥相互作用[3.,4,5,6].早熟解离姐妹5 (PDS5)亚基,PDS5A和PDS5B,是另一对相互排斥的HEAT重复附属亚基,结合内聚- stag1 /2复合物。PDS5蛋白在姐妹染色单体内聚中具有重要作用,PDS5缺失导致姐妹染色单体排列异常,破坏发育,导致胚胎致死[7,8,9,10,11].然而,PDS5A和PDS5B亚基在内聚蛋白介导的基因组组织和基因表达中的作用尚不清楚。此外,PDS5蛋白与STAG1或STAG2的关系尚不清楚。虽然STAG1和STAG2已被证明定位于整个基因组中大部分共享的结合位点,但PDS5A和PDS5B的定位模式尚不清楚[12,13,14,15].不同组成的内聚蛋白复合物(例如,内聚蛋白- stag1 - pds5a和内聚蛋白- stag2 - pds5b)可能具有特定的作用或特性,从而对基因组结构和功能产生不同的影响。
PDS5蛋白与黏聚蛋白复合物的相互作用被认为与黏聚蛋白装载因子NIPBL相互排斥,NIPBL是另一种HEAT重复蛋白,对黏聚蛋白装载到DNA和挤压DNA环至关重要[4,5,6,16,17,18].目前的模型表明,NIPBL有助于内聚蛋白复合物加载到染色体上并挤压DNA,直到遇到远端位点,NIPBL解离和/或被PDS5A或PDS5B取代。最近假设PDS5亚基与CTCF相互作用,并参与DNA环锚点内聚蛋白复合物的捕获和稳定结合[19,20.].然而,尚不清楚内聚蛋白在缺乏CTCF的位点(如增强子和启动子)是如何稳定的[19,20.,21,22,23,24].据报道,PDS5A和PDS5B在染色质环、复制叉保护和wapl介导的间期DNA内聚蛋白卸载中起作用[20.,25,26,27,28].就基因组组织而言,HeLa细胞中PDS5A和PDS5B的同时缺失导致DNA环数量减少,并削弱了拓扑关联结构域(TADs)和区室的强度[20.].此外,PDS5A和PDS5B的缺失导致收敛定向CTCF DNA基序的环减少,而发散和/或串联定向CTCF DNA基序的环增加[20.].
内聚蛋白介导的DNA环是基因组结构的驱动力,尽管它们具有短暂性。最近的数据表明,DNA环相对较少,形成的时间小于7%,持续时间小于30分钟[29].目前尚不清楚内聚蛋白介导的DNA挤压是如何停止的,从而导致动态内聚蛋白分子在特定位置稳定。一些报道表明,在哺乳动物中,内聚蛋白亚基SMC3 (SMC3ac)的K105和K106残基(酵母中的K112和K113残基)的乙酰化可使内聚蛋白稳定结合,尽管尚不清楚SMC3乙酰化对调节基因表达的DNA环的稳定性是否重要[30.,31,32].在S期,乙酰转移酶ESCO1和ESCO2负责SMC3-K105/6乙酰化[33,34].虽然ESCO2的表达仅限于S期,但ESCO1的表达以及随后SMC3的乙酰化在整个细胞周期中普遍存在[32,35,36,37].在酵母中,由于赖氨酸残基突变而不能乙酰化的SMC3突变株是不可活的,而在哺乳动物SMC3中等效的突变株是可活的[33,38,39].此外,虽然在SMC3ac突变哺乳动物细胞中,内聚蛋白在染色质上的停留时间没有改变,但酵母SMC3ac突变细胞显示内聚蛋白结合减少,导致DNA环数量减少,并向更大的DNA环转移[33,38,39].当PDS5A、PDS5B、STAG1、STAG2或CTCF缺失时,SMC3乙酰化程度不同[11,32,37,40,41,42,43].这些不同的内聚蛋白相互作用蛋白如何共同促进SMC3的乙酰化状态的机制尚不清楚。
在这项研究中,我们利用CRISPR/Cas9基因组编辑、基因组学技术和生化分析来研究PDS5A和PDS5B在小鼠胚胎干细胞中的作用。我们发现PDS5A和PDS5B定位于整个基因组的共享结合位点,而不调节内聚蛋白与染色质结合的位置。尽管PDS5A和PDS5B在整个基因组中显示了共享的定位模式,但它们对基因表达有部分不同的影响。对所有四种HEAT重复辅助蛋白的比较分析表明,PDS5A/B和STAG1/2定位于整个基因组的共享位点,并在PDS5亚基或STAG亚基双缺失后导致SMC3乙酰化的相似降低。两种PDS5亚基的双重缺失降低了内聚蛋白与其已知调控因子NIPBL和WAPL之间的相互作用,而两种STAG蛋白的双重缺失降低了染色质结合内聚蛋白的水平。这些数据共同揭示了PDS5亚基在适当的基因表达中的需求,而不是内聚蛋白定位或基因组上的水平。
结果
PDS5A和PDS5B定位于整个基因组的共享结合位点
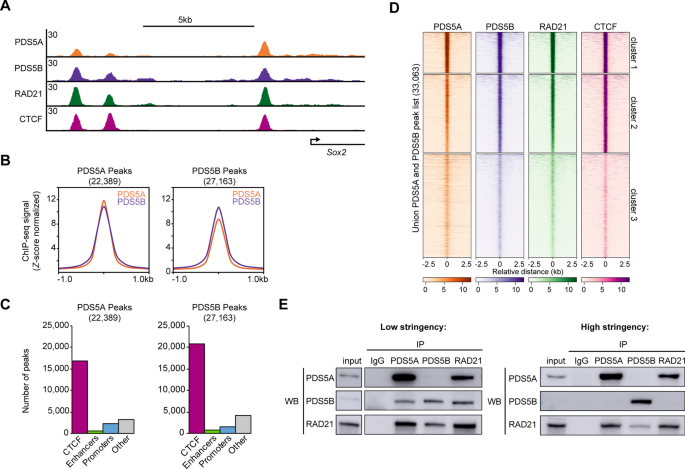
为了确定PDS5A和PDS5B的全基因组占用模式,在小鼠胚胎干细胞(mESCs)中进行染色质免疫沉淀,随后进行高通量测序(ChIP-seq),利用spike-in进行标准化(附加文件)1:表S1)。PDS5A和PDS5B在整个基因组中显示了相似的结合谱,并且具有相似数量的峰,鉴定出22,389个PDS5A峰和27,163个PDS5B峰(图5)。1A, B,附加文件2图S1A, B). PDS5A和PDS5B信号与核心内聚蛋白亚基、RAD21和CTCF信号有很强的重叠。PDS5A和PDS5B峰与CTCF位点有很强的重叠,与增强子或启动子的重叠相对较少(图5)。1C)。对所有PDS5A峰值和所有PDS5B峰值(33,063)进行k-means聚类,结果显示PDS5A信号强的位点也显示PDS5B、RAD21和CTCF的强信号(图。1D)虽然PDS5A和PDS5B定位于一个细胞群中的一组共享的基因组位点,但尚不清楚它们是否同时共同占据一个位点。共免疫沉淀(coIP)实验表明,PDS5A的免疫沉淀能够在低严格度、非交联条件下共纯化RAD21和PDS5B(图2)。1E).然而,PDS5B的免疫沉淀仅共纯化核心内聚蛋白RAD21亚基,而PDS5A未共纯化。当这些coIPs在高严格度(高盐和洗涤剂)、非交联条件下进行时,PDS5A和PDS5B之间的相互作用不再被检测到,但PDS5A和核心内聚蛋白亚基RAD21之间的相互作用保持完整。有趣的是,在高严格条件下,PDS5B与核心内聚蛋白复合物的相互作用减少,表明它与内聚蛋白环的相互作用比PDS5A更弱。这些结果表明,单个内聚蛋白复合物分子包含两个相互排斥的PDS5亚基中的一个。虽然在低严格条件下,可以检测到PDS5A和PDS5B之间的弱相互作用,但这可能反映了两个不同的内聚蛋白复合体之间可能发生的弱相互作用(内聚蛋白- PDS5A复合体与内聚蛋白- PDS5B复合体相互作用)。总之,这些结果表明,虽然PDS5A和PDS5B占据了整个基因组中的许多共享位点,但它们参与不同的内聚蛋白复杂分子,而不是多个内聚蛋白分子稳定的高阶结构的一部分。
PDS5A和PDS5B定位于mESC基因组的共享位点。一个的启动子附近的PDS5A、PDS5B、RAD21和CTCF ChIP-seq信号的基因组浏览器跟踪Sox2在WT mESCs中(Z -分数归一化)。BPDS5A和PDS5B各峰PDS5A和PDS5B各峰ChIP-seq信号平均信号图(Z -分数归一化)。CPDS5A和PDS5B峰在基因组中重叠相关功能元件的数量:CTCF位点、增强子、启动子或其他(以上均非)。DPDS5A、PDS5B、RAD21和CTCF ChIP-seq信号在PDS5A和PDS5B峰值联合列表上的聚类热图,按RAD21信号排序(k-means = 3) (Z -分数归一化)。E在低严格度和高严格度条件下,在WT核裂解液中共免疫沉淀IgG(阴性对照)、PDS5A、PDS5B和RAD21后进行Western blot分析
一个PDS5亚基的缺失不影响内聚蛋白的定位,另一个PDS5亚基也不影响内聚蛋白的定位
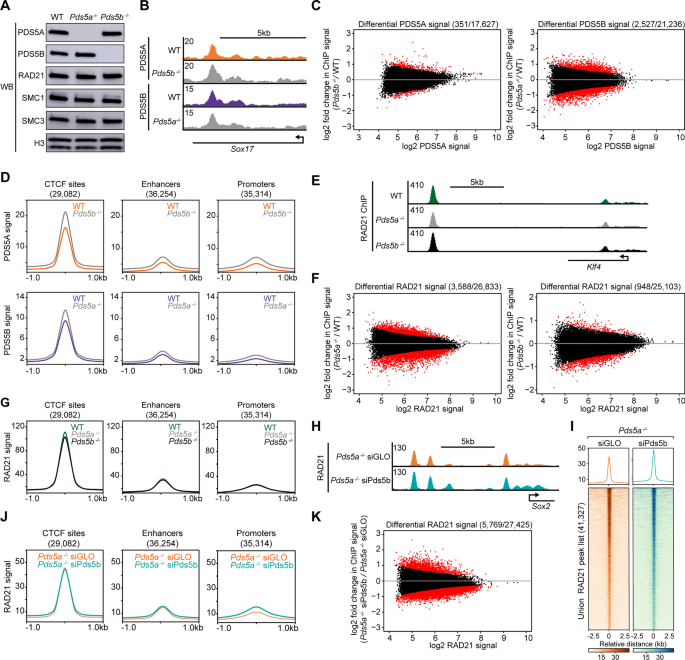
为了研究PDS5A和PDS5B在内聚蛋白生物学中的单独作用,使用CRISPR/Cas9基因组编辑为每个基因生成两个独立的敲除mESC系:Pds5a−−/代表1,Pds5a−−/代表2,Pds5b−−/代表1,和Pds5b−−/rep 2 mESCs(附加文件2:图S2A)。值得注意的是,PDS5A蛋白在Pds5a−−/mESCs和PDS5B蛋白均未检测到Pds5b−−/mESCs,和,其他内聚蛋白亚基的水平没有改变(图。2A).为了确定一个PDS5亚基是否补偿了另一个PDS5亚基的损失,进行了ChIP-seq,对野生型(WT)的PDS5A和Pds5b−−/mESCs和PDS5B在WT和Pds5a−−/制。重要的是,在WT和敲除的mESCs中,PDS5A和PDS5B的免疫沉淀效率在所有重复中是相似的2:图S2B, C)。PDS5A结合剖面在WT和Pds5b−−/分别鉴定出22,389个和22,022个峰,并且PDS5B在WT和PDS5B中的结合谱非常相似Pds5a−−/mESCs(分别鉴定出27,163和27,088个峰)2B、附加文件2:图S2D)。使用DiffBind(测量两种条件共享的结合位点的定量变化)对相对信号进行分析[44],并揭示了一小部分PDS5A的ChIP-seq信号差异位点Pds5b−−/mESCs相对WT (2.0%), PDS5B在Pds5a−−/mESCs相对于WT (11.9%)2C)。在CTCF位点、增强子和启动子上,PDS5A水平基本不因PDS5B的缺失而改变,而PDS5B水平基本不因PDS5A的缺失而改变(图5)。2D).这一结果,以及整个基因组中差异结合的小百分比,表明PDS5蛋白不相互补偿。在一个PDS5亚基的慢性缺失之后,剩余的PDS5亚基没有向新的基因组位点重新分配,也没有改变保守结合位点上剩余PDS5亚基的水平。
PDS5蛋白的缺失不会导致其他PDS5蛋白或内聚蛋白复合物的定位错误。一个Western blot分析WT中黏附蛋白复合物亚基,Pds5a−−/,Pds5b−−/mESC核裂解物。B基因组浏览器跟踪PDS5A ChIP-seq信号在WT和Pds5b−−/在WT和中mESCs和PDS5B信号Pds5a−−/mESCsSox17基因位点。CMA图显示WT和PDS5A信号的差异富集Pds5b−−/保守结合位点上的mESCs,以及WT和PDS5B信号的差异富集Pds5a−−/在保守结合位点上的mESCs。DWT和PDS5A ChIP-seq信号平均信号图Pds5b−−/mESCs,以及WT和PDS5B信号Pds5a−−/CTCF位点上的mESCs、增强子和启动子。ERAD21 ChIP-seq信号在WT中的基因组浏览器跟踪Pds5a−−/,Pds5b−−/mESCsKlf4基因位点。FMA图显示WT与RAD21信号的差异富集Pds5a−/−保守结合位点上的mESCs,以及WT和Pds5b−−/在保守结合位点上的mESCs。GWT下RAD21 ChIP-seq信号平均信号图,Pds5a−−/,Pds5b−−/CTCF位点、增强子和启动子上的mESCs。H基因组浏览器跟踪RAD21 ChIP-seq信号Pds5a−−/siGLO和Pds5a−−/siPds5b mESCs在Sox2基因位点。我RAD21 ChIP-seq信号的热图Pds5a−−/siGLO和Pds5a−−/在这两种情况下,siPds5b mESCs在所有RAD21峰的联合峰列表。JRAD21 ChIP-seq信号的平均信号图Pds5a−−/siGLO和Pds5a−−/siPds5b在CTCF位点、增强子和启动子上的mESCs。K之间显示差分RAD21 ChIP-seq信号的MA图Pds5a−−/siPds5b和Pds5a−−/保守结合位点上的siGLO mESCs
虽然PDS5A和PDS5B的定位模式在很大程度上对彼此的缺失不敏感,但尚不清楚核心内聚蛋白复合物的定位是否对PDS5亚基的缺失也不敏感。为了解决这个问题,WT中RAD21的ChIP-seq,Pds5a−−/,而且Pds5b−−/进行了mESCs,值得注意的是,所有重复都观察到类似的ChIP效率(附加文件)2:图S2E)。结果显示,WT中RAD21的结合谱和峰数相似,Pds5a−−/,而且Pds5b−−/mESCs(分别为33,665、35,981和31,595个峰)(图。2E、附加文件2:图S2F)。与这一结果一致,DiffBind分析检测到相对较少的差分RAD21信号的位点Pds5a−−/或Pds5b−−/相对于WT的mESCs(分别为13.4%和3.8%),表明整个基因组的核心内聚蛋白复合物水平在PDS5A或PDS5B缺失后显示轻微变化(图5)。2F). CTCF位点、增强子和启动子上的RAD21信号在所有三个细胞系中也惊人地相似(图。2这些结果表明,PDS5A和PDS5B亚基并不是决定内聚蛋白在基因组上定位的特异性因素,缺乏PDS5亚基的内聚蛋白复合物仍然分布在其正常的基因组位点。此外,在缺乏单个PDS5蛋白的细胞中未观察到异位PDS5结合位点,也未观察到异位RAD21结合位点,这与PDS5亚基大多与核心内聚蛋白成员复合体,不独立于核心复合体运作的模型一致。
PDS5A和PDS5B的双重缺失不会改变内聚蛋白在基因组上的定位或水平
为了研究PDS5A和PDS5B同时缺失是否会影响染色质上的内聚蛋白定位,我们对PDS5B进行了siRNA敲低Pds5a−−/制。作为对照,在WT mESCs中也进行了siPds5b和siGLO转染。经siRNA处理后Pds5a−−/mESCs, PDS5B蛋白水平相对于处理过的siGLO对照减少了95%Pds5a−−/mESCs(附加文件2:图S2G)。RAD21 ChIP-seq在四种siRNA条件下进行,采用spike-in进行归一化(图。2H、附加文件2:图S2H, I). PDS5B在WT或Pds5a−−/在任何一种条件下,mESCs都没有改变RAD21峰联合列表上的内聚蛋白定位(图2)。2一、附加文件2:图S2J)。与对照组相比,siPds5b处理细胞中CTCF位点、增强子和启动子的内聚蛋白水平没有变化。2J、附加文件2:图S2K)。PDS5B的缺失导致了研究中21%内聚蛋白结合位点RAD21水平的显著变化Pds5a−−/这与野生型mESCs中PDS5B缺失的影响相似(20.4%的位点显示差异信号),表明野生型细胞中PDS5B的急性缺失或PDS5B和PDS5A的双重缺失导致保守结合位点的内聚蛋白水平发生类似的微小变化(图5)。2K、附加文件2:图S2L)。这些结果表明,在缺乏PDS5蛋白的情况下,内聚蛋白的定位是正常的,这表明PDS5A和PDS5B并不是决定内聚蛋白在基因组上结合位点的特异性因素。
PDS5A和PDS5B对基因表达的影响既重叠又不同
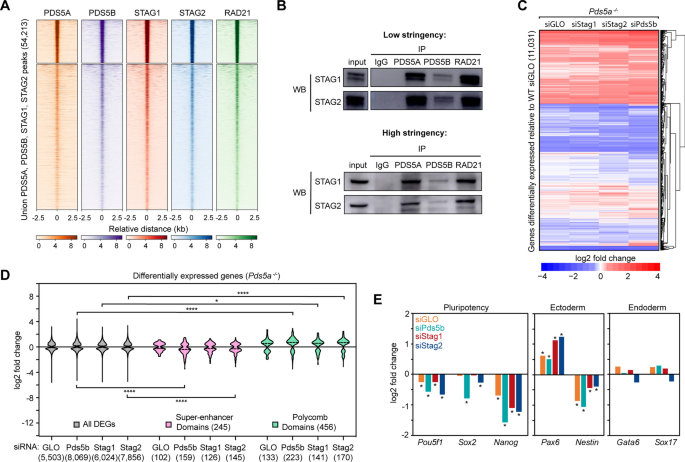
PDS5亚基是否在调节基因表达中起作用尚不清楚。为了研究这一点,在WT中进行RNA-seq,Pds5a−−/,而且Pds5b−−/利用DESeq2进行mESCs和差异基因表达分析(padj < 0.01) [45].Pds5a−−/与WT型mESCs相比,mESCs有5,503个差异表达基因(DEGs)Pds5b−−/与WT型mESCs相比,mESCs有6237个deg(附加文件)1:表S1,附加文件2:图S3A,附加文件3.:表S2)。这些DEG列表的重叠显示了2866个基因在PDS5A或PDS5B (Common)缺失时错表达,而2637个基因仅在PDS5A或PDS5B (Common)缺失时错表达Pds5a−−/mESCs和3371个基因仅在Pds5b−−/制(无花果。3.A).任何一种中所有deg基因表达的变化Pds5a−−/制或Pds5b−−/mESCs呈弱正相关(R2= 0.359);然而,在2,866个共同发展指数(R2= 0.620)(图3.B、附加文件2:图S3B)。中所有deg的组合列表Pds5a−−/而且Pds5b−−/mESCs(8,874个基因)显示了两种基因型相似的log2倍的表达变化模式。3.重要的是,PDS5A和PDS5B都占据了这些基因的启动子,与WT相比,在互反敲除细胞系中,PDS5A和PDS5B的相对ChIP-seq信号在这些启动子上没有变化(图5)。3.C).研究了超级增强子结构域(SDs)和Polycomb结构域(pd)内的基因表达,因为已知这些结构域的DNA环结构包含由超级增强子调控或被Polycomb抑制的细胞身份基因,因此这些基因通常对转录绝缘的丧失很敏感[46].PDs内的基因表达均显著增加Pds5a−−/而且Pds5b−−/制(p< 0.01),而SDs内基因的表达没有变化(图;3.D).为了检查PDS5A或PDS5B的缺失是否影响细胞特性,检查了多能性、外胚层和内胚层基因的表达。Pds5a−−/而且Pds5b−−/mESCs的多能性基因表达显著降低Pou5f1(OCT4)和Nanog,以及外胚层基因表达增加Pax6,与干细胞状态改变一致(图;3.E). PDS5A和PDS5B蛋白丰度在多种细胞系和组织中高度相似[47],虽然在WT mESCs中PDS5A转录水平高于PDS5B,但PDS5亚基中的一个亚基的缺失并没有显著改变其余PDS5亚基的转录水平。2A、附加文件2:图S3C-E)。
PDS5A和PDS5B对基因表达的影响部分重叠且不同.一个差异表达基因(DEGs)重叠Pds5a−−/而且Pds5b−−/相对于WT mESCs。所有细胞用siGLO处理作为转染对照。B中常见DEGs(2866)表达的log2倍变化的相关图Pds5a−−/而且Pds5b−−/制。坐标轴裁剪,去掉一个异常值。C热图的log2倍变化的表达,所有的deg在两者Pds5a−−/而且Pds5b−−/制。WT和PDS5A的ChIP-seq信号Pds5b−−/在WT和中mESCs和PDS5B信号Pds5a−−/mESCs显示在这些deg的启动子上。D小提琴图的log2倍变化的表达为所有的DEGs,那些在超级增强子域,和那些在Polycomb域Pds5a−−/而且Pds5b−−/制。显著性通过Kruskal-Wallis检验和Dunn多重比较检验确定。星号表示组间有显著差异(**p< 0.01, ****p< 0.0001)。E多能性基因表达的log2倍变化柱状图(Pou5f1, Sox2, Nanog)、外胚层谱系基因(Pax6而且巢蛋白),以及内胚层谱系基因(Gata6而且Sox17)Pds5a−−/而且Pds5b−−/相对于WT mESCs。星号表示与使用DESeq2测定的WT有显著差异(padj < 0.01)。F差异表达基因的重叠Pds5a−−/siGLO,Pds5b−−/siGLO,Pds5a−−/siPds5b制。常见和冗余基因类被突出显示。GWT siPds5b中DEGs组合列表表达log2倍变化的聚类热图Pds5a−−/siGLO,Pds5a−−/siPds5b mESCs都相对于WT siGLO mESCs。H基因本体论(GO)分析生物过程相关的deg是Pds5a−−/具体的,Pds5b−−/特定的,共同的,或多余的PDS5蛋白的损失。我在WT(实线)和敲除(虚线)mESCs中PDS5A、PDS5B和RAD21 ChIP-seq信号的平均信号图Pds5a−−/具体的,Pds5b−−/特定、通用或冗余DEG启动子(Z -分数归一化)。J启动子的百分比Pds5a−−/具体的,Pds5b−−/与WT内聚物重叠的特异性、共同性、冗余性或非deg基因类[46]
PDS5A和PDS5B在一组基因表达中的冗余作用
为了进一步研究PDS5A和PDS5B在调节基因表达中的潜在重叠作用,在Pds5a−−/siPds5b mESCs和三个对照条件。击倒PDS5BPds5a−−/相对于WT siGLO处理的mESCs, mESCs产生了大量的deg,而WT mESCs中PDS5B的敲除几乎没有导致基因表达的变化(分别为8069对16)(附加文件)2:图S3F)。中deg的重叠Pds5a−−/siPds5b mESCsPds5a−−/siGLO和Pds5b−−/siGLO mESCs揭示了一类基因,其中PDS5A和PDS5B起冗余作用(2165个基因)(图。3.F)。此外,该分析表明,两种PDS5亚基的双重消耗比任何一种单一扰动条件都能产生更多的deg。进一步分析了在任何条件下与WT siGLO条件相比被认为存在差异表达的基因。在双重消耗条件下,基因表达的log2倍变化总体上强于任何一种单一条件(图2)。3.G)。与两个单一敲除系相似,两个PDS5亚基的双重缺失导致PDs内基因的显著上调(p< 0.0001);然而,双重消耗也导致SDs内基因表达的显著降低,这在中未观察到Pds5a−−/或Pds5b−−/制(p< 0.0001)(附加文件2:图S3G)。多能性和外胚层基因的变化被检测到Pds5a−−/siPds5b mESCs,变化方向相似,但比in的变化更大Pds5a−−/而且Pds5b−−/mESCs(附加文件2:图S3H)。基因本体论(GO)分析揭示了与之相关的生物学过程的强烈差异Pds5a−−/具体的和Pds5b−−/特异性DEGs(图;3.H、附加文件4:表S3)。总的来说,生物过程在公共、冗余和Pds5a−−/特定的类彼此相似,而Pds5b−−/特定类别的基因显示出更明显的模式(图。3.H).总的来说,PDS5A和PDS5B发挥冗余作用的基因与神经元分化、神经元生成和RNA加工有关。尽管一些基因普遍、冗余或特异性地受到PDS5A或PDS5B亚基缺失的影响,但在这四个基因类别的启动子中,PDS5A或PDS5B的占用并没有特异性的富集(图5)。3.I).此外,PDS5A或PDS5B的富集在Pds5b−−/或Pds5a−−/细胞,分别与WT mESCs相比,表明这些基因表达的变化可能是一种间接影响(图。3.值得注意的是,在PDS5扰动后,这四个基因类别中差异表达的基因启动子比没有差异表达的基因启动子更有可能参与远程相互作用(26-42%)(18%)(图5)。3.J) [46].综上所述,这些结果揭示了PDS5A和PDS5B的冗余作用,以保持一个基因子集的正确表达,同时也对其他基因子集显示出不同的影响。
PDS5亚基和STAG亚基定位于相同的位点,但对基因表达有部分不同的影响
尽管PDS5A和PDS5B共享全基因组占用,但它们对基因表达具有不同和重叠的影响,这一发现与之前报道的另一组相互排斥的内聚蛋白亚基STAG1和STAG2的结果相似[12].然而,PDS5蛋白与STAG蛋白之间的关系尚未被直接探讨。因此,研究人员在WT mESCs中检测了PDS5A、PDS5B、STAG1和STAG2的全基因组分布,发现在包括任何单个数据集中识别的所有峰值的联合列表中,所有四个亚基的ChIP-seq信号都有惊人的重叠(54,213)(图2)。4) (12].值得注意的是,最强的PDS5峰同时也是最强的STAG峰,这表明这四个亚基的染色质结合水平都是正相关的。在低严格度和高严格度、非交联条件下进行PDS5A、PDS5B和RAD21的共免疫沉淀,以研究内聚蛋白复合物亚单位组成的潜在特异性;对STAG1和STAG2亚基的western blotting实验表明,STAG1和STAG2都能与PDS5A和PDS5B亚基共同纯化(图5)。4B).这与我们之前的报告表明STAG1和STAG2在不同的复合物中定位于共享的基因组位置,表明PDS5A或PDS5B与内聚物- STAG1或内聚物- STAG2变体复合物没有选择性结合[12].相反,由STAG1或STAG2和PDS5A或PDS5B的所有可能成分组成的内聚复合物可以存在于细胞中。虽然尚不清楚PDS5A/B和STAG1/2在基因组上的重叠分布是否依赖于彼此的存在,但在几乎不含STAG蛋白的mESCs中对PDS5A和PDS5B进行了ChIP-qPCR,在几乎不含PDS5蛋白的mESCs中对STAG1和STAG2进行了ChIP-qPCR。在WT siGLO和CTCF的各个结合位点上测定了PDS5A和PDS5B的ChIP富集Stag2−−/siStag1 mESCs,并显示PDS5蛋白占用无显著变化(附加文件2:图S4A)。在WT siGLO和中相同的CTCF结合位点测定了STAG1和STAG2的ChIP富集Pds5a−−/siPds5b mESCs,并显示STAG1水平略有下降(p< 0.01),而STAG2水平无变化(附加文件2:图S4B)。因此,PDS5和STAG蛋白的定位似乎在很大程度上是相互独立的。
PDS5蛋白定位于与STAG蛋白相同的基因组位点.一个PDS5A、PDS5B、STAG1、STAG2和RAD21 ChIP-seq信号在PDS5A、PDS5B、STAG1和STAG2峰值联合列表中的聚类热图,按RAD21信号排序(k-means = 2) (Z -分数归一化)。B在低严格度和高严格度条件下,在WT核裂解液中共免疫沉淀IgG(阴性对照)、PDS5A、PDS5B和RAD21后进行Western blot分析。本实验的对照印迹如图所示。1E。C中deg的组合列表表达的log2倍变化的聚类热图Pds5a−−/siGLO,Pds5a−−/siStag1,Pds5a−−/siStag2,Pds5a−−/siPds5b mESCs相对于WT siGLO mESCs。D小提琴图的log2倍变化的表达对所有的DEGs,那些在超级增强子域,和那些在Polycomb域Pds5a−−/siGLO,Pds5a−−/siStag1,Pds5a−−/siStag2,Pds5a−−/siPds5b mESCs相对于WT siGLO mESCs。显著性通过Kruskal-Wallis检验和Dunn多重比较检验确定。星号表示组间有显著差异(*p< 0.05, ****p< 0.0001)。E多能性基因表达的log2倍变化柱状图(Pou5f1, Sox2, Nanog)、外胚层谱系基因(Pax6和Nestin),以及内胚层谱系基因(Gata6和Sox17)Pds5a−−/siGLO,Pds5a−−/siStag1,Pds5a−−/siStag2,Pds5a−−/siPds5b制。星号表示与使用DESeq2测定的WT siGLO mESCs有显著差异(padj < 0.01)
而与基因组结合的内聚蛋白在Stag2−−/siStag1 mESCs [12], PDS5A和PDS5B在CTCF结合位点无明显降低,提示PDS5蛋白可以独立于核心内聚蛋白复合物与CTCF相互作用[19,20.].为了直接验证这一假设,我们对WT siGLO和WT siSMC3 mESCs中CTCF结合位点上的PDS5A和PDS5B进行了ChIP-qPCR检测。虽然没有观察到PDS5A的富集有显著差异,但在所有四个CTCF位点,PDS5B在内聚蛋白耗尽后显著降低(附加文件)2:图S4C)。这支持了一个模型,其中PDS5A(而不是PDS5B)能够独立于核心内聚蛋白复合体与CTCF相互作用。
虽然通过共免疫沉淀没有检测到PDS5A/B和STAG1/2亚基的优先物理相互作用,这表明所有可能的亚基组成的内聚复合物可以存在于细胞中,但尚不清楚PDS5亚基和STAG亚基是否对基因表达显示重叠效应。为了探索这种可能性,在敲除STAG1或STAG2后进行RNA-seqPds5a−−/mESCs(附加文件3.:表S2)。差异基因表达分析显示,在Pds5a−−/siStag1和Pds5a−−/中所见的siStag2 mESCsPds5a−−/siPds5b mESCs(分别为6,024、7,856和8,069)(附加文件2:图S4D-E)。值得注意的是,deg的数量Pds5a−−/siStag2和Pds5a−−/siPds5b mESCs与Pds5a−−/mESCs(5,503)和Pds5b−−/制(6237)。与WT siGLO相关的四种条件中任何一种的差异表达基因(Pds5a−−/siGLO,Pds5a−−/siStag1,Pds5a−−/siStag2,Pds5a−−/siPds5b)被鉴定并合并到一个联合列表(11031个基因)。检查此列表中deg的log2倍变化值显示细胞间的最小差异,其中STAG1, STAG2或PDS5B在细胞中被敲除Pds5a−−/相对于WT siGLO对照组处理的细胞(图。4C).此外,在中发现的deg之间有很强的重叠Pds5a−−/siPds5b,在Pds5a−−/siStag1和Pds5a−−/siStag2 mESCs(附加文件2:图S4E)。SDs内基因的表达不受影响Pds5a−−/而在siStag1 mESCs中显著降低Pds5a−−/siStag2 mESCs与之前的发现一致,即缺乏STAG2,而不是STAG1,会影响WT背景下SD基因的表达(图1)。4D) (12].STAG1或STAG2的缺失也引起多能性基因和外胚层基因表达的变化,其表达方向和程度与PDS5B的缺失相似Pds5a−−/制(无花果。4E).为了更好地检验个体敲除对基因表达的影响Pds5a−−/siGLO背景,差异表达基因相对Pds5a−−/识别了siGLO mESCs(而不是相对于WT siGLO mESCs)。该分析显示相对较少的差异表达基因(380Pds5a−−/siStag1, 2,698英寸Pds5a−−/siStag2和3485英寸Pds5a−−/siPds5b mESCs)与WT siGLO背景相比,表明在a中STAG1或STAG2的耗尽Pds5a−−/背景引起的转录变化类似于PDS5B的耗尽(附加文件2:图S4F, G)。
尽管在PDS5基因敲除背景下,单个STAG亚基的敲除类似于PDS5A和PDS5B的双重损失,但在PDS5基因敲除背景中,约有一半的deg被鉴定出Pds5a−−/siPds5b与Stag2−−/siStag1 mESCs(附加文件2:图S4H) [12].此外,PDS5亚基双缺失与STAG亚基双缺失相比,普通或冗余调控基因的重叠更少(附加文件)2:图S4I) [12].PDS5亚基的双缺失或STAG亚基的双缺失对生物学过程的影响也很少重叠(附加文件)2:图S4J,附加文件4:表S3)。有趣的是,通常由两种相反方向改变表达的双重消耗条件调节的基因不会对特定的生物过程产生任何富集。综上所述,PDS5A/B和STAG1/2亚基在基因组上的定位模式有显著的重叠,但PDS5A和PDS5B的双重缺失,或STAG1和STAG2的双重缺失,会影响不同生物学过程中相关基因的表达。
PDS5亚基和STAG亚基对SMC3乙酰化和染色质内聚蛋白水平有不同的影响
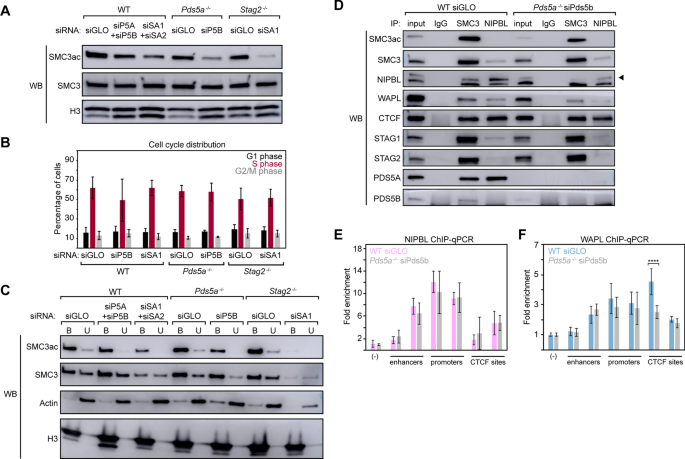
考虑到PDS5A、PDS5B、STAG1或STAG2亚基缺失后基因表达的许多变化,我们接下来研究了内聚蛋白复合体的各种性质及其与调控蛋白的相互作用。最初,我们试图测量SMC3的翻译后乙酰化,在残基K105和K106处,与间期姐妹染色单体内聚和稳定内聚蛋白结合相关[32,33,34,35,36,37].利用siRNA和敲除细胞系的组合,产生了7种急性(WT siPds5a + siPds5b和WT siStag1 + siStag2)和慢性(Pds5a−−/siPds5b和Stag2−−/siStag1)内聚蛋白亚单位耗尽(附加文件2:图S5A)。使用SMC3抗体和smc3ac特异性抗体(针对SMC3 K105ac和K106ac)检测7种细胞条件下核提取物中翻译后修饰的SMC3和总SMC3的水平。结果显示,与WT siGLO相比,在所有条件下SMC3ac水平都有所下降,在双重消耗条件下观察到的下降幅度最大:Pds5a−−/siPds5b和Stag2−−/siStag1(无花果。5A、附加文件2:图S5B)。重要的是,在7种情况下,总SMC3水平没有变化,两种抗体都显示出特异性(图2)。5A、附加文件2:图S5C, D).中SMC3ac水平的具体降低Pds5a−−/与对照的mESCs相比,在核提取物中SMC3的IP后也观察到siPds5b mESCs(附加文件)2:图S5E)。为了确定这种SMC3ac的损失是否由于细胞周期分布的变化,我们在双重耗尽条件下以及单次敲除控制下测量了G1、S和G2/M细胞的百分比。相对于WT siGLO mESCs,在任何突变条件下检测到的细胞周期分布均无显著差异(图2)。5B、附加文件2:图S5F)。值得注意的是,Pds5a−−/siPds5b mESCs的增殖率降低Stag2−−/siStag1 mESCs在每个细胞周期阶段显示出强烈的增殖缺陷和增加的时间长度(附加文件)2:图S5G, H)。这些结果表明,PDS5或STAG蛋白缺失时SMC3ac水平的降低不是由于S期或G2/M期细胞比例的改变。
PDS5蛋白和STAG蛋白对SMC3ac水平和染色质结合内聚蛋白有不同的影响。一个Western blot分析mESCs中七种siRNA条件下的核裂解物。缩写包括PDS5A (P5A), PDS5B (P5B), STAG1 (SA1)和STAG2 (SA2)。B每个细胞周期阶段的细胞百分比,n= 3。双尾未配对的t用检验来确定显著性。在细胞周期的任何阶段,突变体与WT条件之间均无显著性差异。C从七种siRNA条件下分离细胞后进行Western blot分析。每种条件都显示了染色质结合(B)和核可溶性(U)。D免疫共沉淀IgG(阴性对照)、SMC3和NIPBL后进行Western blot分析Pds5a−−/siPds5b核裂解物,在高严格度条件下。黑色三角形表示NIPBL特定波段(顶部),由NIPBL击倒确定(参见附加文件)2:图S6D)。EChIP-qPCR检测WT型siGLO和NIPBLPds5a−−/siPds5b制。描述了相对于5%输入材料的倍增富集和负控制区。数据在两个生物重复中以均值±标准差表示,每个重复中有三个技术重复。双尾未配对的t用检验来确定WT siGLO与Pds5a−−/siPds5b mESCs用于每个引物集。任何引物组均未发现显著性。FChIP-qPCR检测WAPL在WT siGLO和Pds5a−−/siPds5b制。描述了相对于5%输入材料的倍增富集和负控制区。数据在两个生物重复中以均值±标准差表示,每个重复中有三个技术重复。双尾未配对的t用检验来确定WT siGLO与Pds5a−−/siPds5b mESCs用于每个引物集。星号表示组间显著差异(****p< 0.0001),否则无统计学意义
随后,为了解决SMC3ac的减少是否影响了与基因组结合的内聚蛋白水平,我们在相同的7种条件下进行了染色质分离,以评估结合和未结合的内聚蛋白池。首先,在所有条件下,除了染色质结合和非结合部分,SMC3总量都处于相似的水平Stag2−−/与我们之前的观察一致,即在缺少STAG1和STAG2的情况下,内聚蛋白从基因组中强烈丢失(图2)。5C、附加文件2:图S6A) [12].值得注意的是,急性和慢性STAG1和STAG2亚基的双重消耗导致与染色质结合的SMC3水平下降。鉴于PDS5蛋白在促进wapl介导的内聚蛋白从基因组中卸载中的作用,令人惊讶的是,在PDS5亚单位耗尽后,结合内聚蛋白的水平没有变化,而不是增加(附加文件)2:图S6A)。其次,在急性和慢性双重消耗PDS5亚基和STAG亚基后,结合和未结合部分的SMC3ac水平均下降(图2)。5C、附加文件2:图S6B)。总的来说,丢失任何一对HEAT重复蛋白(PDS5A/B或STAG1/2)时,SMC3ac水平都降低了。
PDS5A和PDS5B在与NIPBL和WAPL调节复合物相互作用中的作用
为了研究PDS5A和PDS5B的双重缺失如何在不影响与染色质结合的内聚蛋白水平的情况下影响SMC3ac水平,在WT siGLO和Pds5a−−/评估了siPds5b mESCs和内聚蛋白相互作用的改变。PDS5亚基双缺失后SMC3的免疫沉淀显示内聚蛋白与NIPBL的相关性降低(图)。5D、附加文件2:图S6C)。值得注意的是,NIPBL抗体是特定于所示的顶部带(附加文件2:图S6D)。虽然NIPBL在WT条件下不能与SMC3ac共纯化,但它能与PDS5A强烈共纯化(图5)。5D、附加文件2:图S6C)。这是令人惊讶的,因为有报道称NIPBL和PDS5蛋白会竞争与RAD21的相互作用,并且在酵母中不占据相同的结合位点[4,16,17].NIPBL与CTCF也有很强的关联,考虑到NIPBL主要定位于增强子和启动子,而不是CTCF位点,这是令人惊讶的[48].为了确定内聚物- NIPBL关联的减少是否影响NIPBL在增强子、启动子或CTCF位点的富集,在WT siGLO和中进行了ChIP-qPCRPds5a−−/siPds5b制。尽管双PDS5缺失后NIPBL与SMC3的相关性降低,但在具有代表性的增强子、启动子和CTCF位点上,NIPBL水平没有变化(图5)。5E).然而,这些结果与报道一致,即NIPBL可以在没有内聚蛋白的情况下与染色质结合,并且内聚蛋白在染色质上的批量装载不受与NIPBL相互作用损失的影响[49,50,51].
PDS5和WAPL之间的相互作用已被证明对间期内聚物卸载很重要[25,26,52].有趣的是,PDS5亚基双缺失后SMC3的免疫沉淀仅显示SMC3与WAPL的相关性略有下降,而不是如果PDS5蛋白是WAPL与内聚蛋白结合的唯一界面所预期的全部损失(图5)。5D、附加文件2:图S6C)。这些结果支持了先前的报道,即WAPL可以与PDS5以外的内聚蛋白亚基发生物理相互作用[53].与NIPBL相似,WAPL的ChIP-qPCR在WT siGLO和Pds5a−−/siPds5b mESCs显示,不同增强子和启动子之间的染色质结合WAPL水平没有差异,但在一个有代表性的CTCF位点出现了单一的显著下降(p< 0.0001)(图5F).总之,这些结果表明,NIPBL-cohesin和WAPL-cohesin复合物可以在缺乏PDS5A和PDS5B的情况下形成。
讨论
在这里,我们首次展示了相互排斥的内聚蛋白亚基PDS5A和PDS5B定位于哺乳动物细胞基因组中的共享结合位点。PDS5A和PDS5B都在CTCF位点上富集,在增强子和启动子上富集程度较低。PDS5A和PDS5B的分布与核心内聚复合体成员RAD21以及互斥内聚亚单位STAG1和STAG2的分布高度相似[12].PDS5A/B和STAG1/2占据的强烈重叠是出乎意料的,因为STAG1/2与内聚蛋白装载到DNA和内聚蛋白介导的DNA环挤压有关,而PDS5A/B在某些情况下抑制内聚蛋白装载并促进内聚蛋白从DNA中卸载[3.,4,5,6,25,26,52].本研究确定了PDS5蛋白在哺乳动物基因组中的定位模式,并测试了PDS5蛋白在内聚蛋白占用和基因表达中的需求。
尽管单个内聚蛋白复合物被认为本质上总是包含一个STAG亚基,但PDS5与内聚蛋白复合物的相互作用被认为是更短暂的。免疫共沉淀研究表明,PDS5A亚基可与STAG1和STAG2相互作用。同样,PDS5B亚基可以与STAG1和STAG2相互作用,这表明内聚蛋白复合物中STAG和PDS5亚基的特定组合缺乏特异性。然而,PDS5B与内聚核心亚基RAD21的相互作用似乎弱于PDS5A与RAD21的相互作用。
这两种PDS5蛋白似乎并不相互补偿,因为PDS5A或PDS5B单独的损失会导致剩余PDS5亚基稳态蛋白水平的轻微但不显著的增加。此外,剩余PDS5亚基的定位模式和水平在siRNA缺失或敲除PDS5蛋白时基本不受影响,尽管这些扰动会产生可变性和不一致性。此外,核心内聚蛋白复合体成员RAD21的全基因组分布没有发生变化Pds5a−−/, Pds5b−−/,或Pds5a−−/siPds5b制。这与之前的一篇报道一致,即PDS5耗尽不会导致RAD21在HeLa细胞基因组上的重新分配[37].然而,在缺乏PDS5亚基的细胞中,相似水平的内聚蛋白在基因组上的保留是令人惊讶的,考虑到PDS5蛋白在WAPL介导的染色质内聚蛋白的脱落、WAPL或PDS5亚基耗散后基因组组织的变化以及PDS5亚基双敲除后内聚蛋白流动性的变化[20.,25,26,52,54].此外,我们的研究结果挑战了CTCF位点上PDS5亚基的占用对于停止挤压内聚蛋白复合物至关重要的观点,因为我们在缺乏PDS5A和PDS5B亚基的细胞中观察到CTCF结合位点上的内聚蛋白水平没有变化。然而,我们确实观察到,尽管内聚蛋白耗尽后,CTCF位点的PDS5B水平下降,但在WT siSMC3 mESCs和CTCF中,PDS5A水平没有变化Stag2−−/siStag1,大多数内聚蛋白复合物从基因组中丢失的情况(见此处和[12])。这些结果与PDS5A(而非PDS5B)在核心内聚复合物通过DNA挤压到达CTCF之前在绝缘体部位与CTCF相互作用的模型一致[19,20.].我们的研究结果表明,PDS5A和PDS5B并不是决定整个基因组内聚蛋白定位模式的特异性因素。结合我们之前的工作,这些结果与一个模型一致,即STAG和PDS5亚基的所有可能组合的变体内聚蛋白复合物可以存在于细胞中,并在CTCF位点、增强子和启动子上显示类似的分布,而不管其亚基组成如何。
在酵母中进行的几项研究表明,负载因子Scc2(哺乳动物中的NIPBL)和PDS5是内聚蛋白复合物的互斥亚基[4,16,17].内聚力- scc2 (NIPBL)复合物可以装载到染色体上,水解ATP并进行高效转运,而内聚力- pds5分子不水解ATP或转运,从染色体中释放。一些研究表明,当内聚蛋白挤压停止时,Scc2 (NIPBL)亚基被PDS5亚基从内聚蛋白中取代,而内聚蛋白复合物不能同时被NIPBL和PDS5结合[4,16,17].在哺乳动物细胞中,我们发现PDS5A可以与NIPBL共纯化,这表明这两种蛋白质相互作用。此外,缺乏PDS5亚基的内聚蛋白复合物没有表现出与NIPBL相互作用的增加,这与PDS5亚基和NIPBL之间缺乏竞争内聚蛋白上的单个界面一致。这一结果与先前的报道一致,并可能表明Scc2 (NIPBL)在染色质上的周转可能太快而不受PDS5丢失的影响,或太快而在这种类型的试验中检测不到[4].需要全面研究PDS5与NIPBL的相互作用,以及酵母和哺乳动物环境之间的差异,以了解这些蛋白质如何通过看似共享的RAD21 (Scc1)界面来不同地调节内聚蛋白功能。
核心内聚蛋白成员SMC3的乙酰化可以促进稳定的内聚蛋白与基因组的结合。含有SMC3ac的内聚蛋白复合物在CTCF位点和DNA环锚点富集[32].CTCF的耗竭强烈地降低了SMC3ac,支持CTCF - pds5相互作用对SMC3乙酰化和稳定内聚蛋白停留很重要的模型[32,43].我们观察到在急性情况下(WT siPds5a + siPds5b)和慢性情况下(Pds5a−−/siPds5b)导致SMC3乙酰化形式的减少。同样,急性(WT siStag1 + siStag2)和慢性(Stag2−−/STAG蛋白的缺失导致SMC3乙酰化形式的类似减少。在缺乏STAG蛋白的细胞中,SMC3ac的缺失似乎是由于内聚蛋白稳定性和与基因组结合的降低,这与报道的STAG蛋白在内聚蛋白负载和易位中的作用一致。在缺乏PDS5亚基的细胞中,SMC3ac的缺失最有可能是由于PDS5s与乙酰转移酶ESCO1/2之间的相互作用减少,因为PDS5蛋白的缺失并没有改变S期或G2/M期的细胞比例,这与先前的结果一致[37].缺乏PDS5亚基的细胞没有表现出染色质结合内聚蛋白水平的变化,证实了先前在哺乳动物SMC3ac突变体中的结果,这表明尽管PDS5亚基可以促进SMC3乙酰化,但这可能不会直接影响内聚蛋白的装载和易位[39].此外,虽然PDS5亚基已被证明通过促进内聚蛋白与WAPL的相互作用来促进内聚蛋白的卸载[25,26,52],在这里我们观察到PDS5亚基的缺失仅导致WAPL与内聚蛋白的关联略有下降,这与之前的报道一致,即WAPL与额外的内聚蛋白亚基相联系[53].总之,这些结果支持一个模型,即PDS5亚基通过促进wapl介导的内聚蛋白卸载以及通过SMC3乙酰化稳定的内聚蛋白结合来影响内聚蛋白与基因组的关联。进一步的研究应该旨在理解这种平衡的复杂性。
我们的数据表明,缺乏PDS5A或PDS5B亚基的内聚蛋白环仍然可以遍历基因组并定位到适当的目标位点;然而,它们似乎在那些正常的结合位点上显示出改变的功能。基因表达模式的分析表明,大约一半在PDS5A缺失时差异表达的基因在PDS5B缺失后也会错误表达。还有大量的基因只有在PDS5A或PDS5B缺失时才会错表达。此外,缺乏PDS5A和PDS5B的细胞比单一敲除的细胞有更多的错表达基因,并且揭示了一类只有在PDS5亚基都缺失时才会错表达的新基因。PDS5蛋白在基因表达中发挥这些部分独立作用的机制尚不清楚,这应该是未来研究的主题。重要的是,在PDS5扰动后,错表达基因的启动子比未错表达基因的启动子更有可能参与远程内聚介导的相互作用。因此,虽然在缺乏PDS5亚基的细胞中,内聚蛋白复合物在整个基因组中显示了适当的定位,但这些共同的锚定位点可能参与了不同的DNA环或具有不同的特性、动力学或稳定性。此外,尽管PDS5A/B和STAG1/2与基因组结合有很强的重叠,但PDS5亚基双缺失后的基因表达变化与STAG亚基双缺失引起的基因表达变化并不相同。虽然这项工作探讨了PDS5A和PDS5B缺失对内聚蛋白定位和基因表达的个体和组合效应,以及与STAG1和STAG2的关系,但还需要进一步的研究来确定这四种内聚蛋白附属蛋白如何不同地影响内聚蛋白动力学和基因表达的调控。
结论
这项工作利用等基因小鼠胚胎干细胞来研究HEAT重复蛋白PDS5A和PDS5B急性和慢性损失的后果。我们发现PDS5A和PDS5B定位于整个基因组的共享结合位点,这种模式与其他HEAT重复蛋白STAG1和STAG2的模式重叠。PDS5蛋白的单个和双重缺失不会引起内聚蛋白在基因组上的错位,但会导致许多基因表达的变化。尽管PDS5A、PDS5B、STAG1和STAG2亚基具有共同的定位模式,但由于PDS5亚基双缺失引起的许多基因表达变化并不与STAG亚基双缺失引起的变化重叠。然而,在PDS5亚基或STAG亚基双重缺失时,也观察到类似的SMC3ac水平下降。STAG亚基的双重缺失降低了染色质结合内聚蛋白的水平,而PDS5亚基的双重缺失降低了内聚蛋白与NIPBL和WAPL的关联。
方法
细胞培养
如前所述,在标准ESC条件下培养小鼠胚胎干细胞(mESCs v6.5,雄性)[55],使用敲除DMEM (Gibco, 10829-018),添加15%胎牛血清(VWR, 97068-085)。为了ChIP-seq spike-in标准化,在DMEM (Gibco, 11995065)中培养人胚胎肾细胞(HEK293T,女性),并添加10%牛血清(Seradigm, 2100-500)、1X GlutaMAX (Thermo Fisher, 35050-061)、100 U/ml青霉素和100 ug/ml链霉素(Thermo Fisher Scientific, 15140-122)。
基因组编辑
使用Lipofectamine 2000 (Thermo Fisher Scientific, 11-688-027)对WT mESCs转染两个质粒,目的是在感兴趣基因的单个外显子内诱导一个小的缺失。一个独特的sgRNA被克隆到包含SpCas9和两个荧光基因(eGFP或mCherry)之一的pX330主干中(AddGene, 42230)。转染后一天,12000个单细胞在CytoSort Array (Cell Microsystems, CS200S)上播种。使用CellRaft AIR系统(细胞微系统),收集、扩增、筛选和低温储存这两种基因的荧光细胞。编辑位点的序列通过感兴趣区域的PCR和Sanger测序确定。根据国际小鼠标准化遗传命名委员会的编辑摘要和官方等位基因名称如下所示。编辑站点序列在附加文件中描述2:图S2A和引导RNA寡核苷酸序列在附加文件中列出5:表S4。同基因的Stag2−−/基因敲除序列是先前生成和描述的[12].
Pds5a−−/复制1(也称为Pds5aem1Jdow的外显子2内含杂合编辑Pds5a导致纯合子敲除。等位基因1有38 bp的插入和18 bp的缺失,而等位基因2有350 bp的缺失延伸到下面的内含子。Pds5a−−/复制2(也称为Pds5aem2Jdow)在基因的第2外显子上有176 bp的纯合缺失Pds5a.Pds5b−−/复制1(也称为Pds5bem1Jdow)的第3外显子有5 bp的纯合缺失Pds5b.Pds5b−−/复制2(也称为Pds5bem2Jdow)的第3外显子有1 bp的纯合子插入Pds5b.
染色质免疫沉淀(ChIP)和测序
交联前对mESCs进行胰蛋白酶化(Gibco, 12604-013)并计数。细胞与1%甲醛(Thermo Fisher Scientific, 28906)在PBS中交联5分钟,然后用2.5 M甘氨酸淬灭。交联细胞用10 ml冷裂解缓冲液1 (50 mM Hepes-KOH pH7.5, 140 mM NaCl, 1 mM EDTA, 10%甘油,0.5% NP-40, 0.25% Triton X-100)在4℃旋转10 min裂解。在1350xg下制粒5分钟后,在5 ml室温裂解缓冲液2 (10 mM Tris-HCl pH 8, 200 mM NaCl, 1 mM EDTA和0.5 mM EGTA)中旋转10分钟,在室温下裂解核。1350 × g制粒5分钟后,去除上清液,试管用5 ml冷剪切缓冲液(10 mM Tris pH 7.5, 1 mM EDTA, 0.1% SDS)洗涤,1350 × g纺丝5分钟。染色质(颗粒)重悬在1 ml剪切缓冲液中,超声前加入5%的HEK293T染色质,使用相同的方法提取,用于之后的spike-in正切。所有缓冲液均添加1X蛋白酶抑制剂鸡尾酒(PIC) (Sigma Aldrich, 11697498001)。染色质的超声处理使用Covaris E220在milliTUBEs (Covaris, 520130)中进行,设置如下:占空比5,PIP/W 140,每脉冲200次,持续12分钟。生成200 - 1000个碱基对的染色质片段。超声处理后,将不溶性物质制成颗粒,在4℃、15,000 rpm转速下旋转样品10分钟。
使用附加文件中提到的抗体进行ChIPs1:表S1。PDS5A和PDS5B抗体用50 ul Protein G Dynabeads (Invitrogen 10004D)孵育,分别使用3.5 ug和4 ug抗体。RAD21抗体用30 ul Protein G Dynabeads和10 ug抗体孵育。抗体在IP前用小珠孵育6-8小时。在加入染色质之前,用含有1X蛋白酶抑制剂鸡尾酒(PIC) (Sigma Aldrich, 11697498001)的PBS清洗珠粒三次,以去除未结合的抗体。剪切缓冲液中的染色质加入NaCl和Triton X-100,使其处于ChIP缓冲液(20 mM Tris pH 7.5, 2 mM EDTA, 0.1% SDS, 150 mM NaCl, 1% Triton X-100)中。染色质为1 × 107将细胞加入抗体偶联珠中,在4°C下旋转孵育过夜。第二天,用ChIP缓冲液、洗涤缓冲液1 (20 mM Tris - hcl pH 8, 500 mM NaCl, 2 mM, EDTA, 0.1% SDS, 1% Triton X-100)、洗涤缓冲液2 (10 mM Tris - hcl pH 8, 250 mM LiCl, 1 mM EDTA, 1% nf -40)和洗涤缓冲液3 (10 mM Tris pH 8, 1 mM EDTA, 50 mM NaCl)洗涤珠粒,每个在4℃旋转5分钟。通过加入100 ul IP洗脱缓冲液(50 mM Tris pH 8,10 mM EDTA和1% SDS)从珠子中洗脱染色质,并在65℃下孵育1小时,每10分钟漩涡一次。上清液在65℃下孵育过夜,加入5 ul蛋白酶K (NEB, P8107S)反向交联。第二天,使用ChIP DNA清洁浓缩试剂盒(Zymo, D5205)纯化DNA。
使用Kapa HyperPrep试剂盒按照制造商的说明(Roche/Kapa, KK8502)制备文库。在收集50 bp单端reads的HiSeq 4000上进行测序(WT siGLO中的RAD21代表1和2;RAD21在Pds5a−−/而且Pds5b−−/代表1和2),或收集50 bp配对端读的NovaSeq 6000 SP (WT siPds5b中的RAD21,Pds5a−−/siGLO,Pds5a−−/siPds5b代表1和2;PDS5A在WT和Pds5b−−/练习1和2;PDS5B在WT和Pds5a−−/练习1和2)。
ChIP-seq分析
ChIP-seq分析是通过以前发布的自定义脚本执行的,该脚本可以在GitHub上找到:https://github.com/dowenlab的描述如下[56].在处理之前,任何被分割到序列器的两个通道上的样本都有原始的fastq文件连接(带有“L1”和“L2”的文件)。如果样本两端配对,则仅使用第一个读取值(“R1”)进行进一步分析。在对齐前,将生物复制进一步连接为原始fastq文件。将合并的重复使用蝴蝶结(v1.2.3)(参数−v 2−p 24−S−m1 -best -strata)对准包含小鼠基因组组装mm10和人类基因组组装hg38的合并基因组[57].Mchr被添加为小鼠染色体的前缀,以便将来与人类染色体区分开来。重复序列使用samtools (v1.11) markdup(−r−s) [58].分别使用samtools idxstats分离鼠标和人类对齐的读取,并使用awk进行计数。使用samtools视图创建了一个只包含鼠标读取的bam文件,然后使用bedtools (v2.29.0)将其转换为bed格式59].读取扩展了200 bp,扩展床文件使用MACS (v2.2.7.1)调用峰值,错误发现率为1% (macs2 callpeak−f bed−g mm−q 0.01) [60].峰顶在上游和下游扩展了50 bp,使用bedtools相交(−v)删除了与重复元素重叠的任何扩展峰(使用UCSC基因组浏览器中的重复屏蔽跟踪定义)。使用公式1/h计算每个ChIP-seq数据集的归一化因子,其中h是前面描述的以百万为单位的人类对齐读取数[61].使用bedtools genomecov(−bga−scale 1/h)将包含小鼠读取的床文件转换为床图文件,然后使用ucsctools (v320)的bedGraphToBigWig转换为bigwig文件[62].Z-评分归一化是使用Daniel McKay博士实验室的Spencer Nystrom定制的R脚本进行的。
ChIP-seq数据的信号轨迹使用IGV 2.4.10桌面浏览器可视化[63].使用deeptools (v2.4.1) computeMatrix (CTCF位点、启动子、增强子和标记峰值列表的参考点)生成平均信号图,然后使用deeptools plotProfile [64].当床具交叉进行峰重叠时,使用延长的峰。启动子列表从UCSC转录起始位点获得。增强子被定义为转录因子OCT4、SOX2和NANOG共同占据的位点(ChIP-seq峰)[65].任何由这些因子结合的转录起始位点都从增强子列表中删除。“其他”位点是在取兴趣峰值列表并删除与CTCF位点重叠的位点、增强子和启动子使用床工具相交(−v)后剩下的位点。在Microsoft Excel中生成功能站点峰值重叠的柱状图。热图是使用deeptools computeMatrix(参考点)生成的,然后是deeptools plotHeatmap。使用deeptools plotHeatmap (-kmeans n)对图例中所示的热图进行k-means聚类。通过连接各自的峰值列表并使用bedtools merge去除重复峰值(默认重叠1bp)来生成联合峰值列表。使用deeptools plotFingerprint (- skipzero)生成指纹图。相关值使用deeptools multiBigwigSummary和plotCorrelation (-removeOutliers -skipZeros -corMethod Pearson)从图中收集。在R (v2.12.0)中使用DiffBind识别差异结合位点和相应的MA图[44].
芯片- - - - - -qPCR
PDS5A ChIP (Bethyl, A300-089A)和PDS5B ChIP (Bethyl, A300-538A)分别在WT siGLO中进行两次生物重复,Stag2−−/分别在WT siGLO和sisimc3细胞中进行siStag1和WT siSMC3细胞,以及STAG1 ChIP和STAG2 ChIP进行两次生物重复Pds5a−−/如上所述的siPds5b单元。使用附加文件中列出的引物检测了一个阴性对照区域和四个CTCF位点5:表S4。NIPBL ChIP (Santa Cruz, sc-374625)和WAPL ChIP (Bethyl, A300-268A)也分别在WT siGLO和WAPL中进行了两次生物重复Pds5a−−/siPds5b细胞。NIPBL和WAPL ChIP样品在超声前用两种交联试剂处理。细胞在含有2 mM Di(n -丁二酰亚胺基)戊二酸盐(DSG) (Sigma Aldrich, 80424)的PBS中孵育,在室温下旋转45分钟,然后1%甲醛交联10分钟。WAPL ChIP使用Protein A Dynabeads (Invitrogen, 10002D)进行,而其他所有使用Protein g,使用附加文件中列出的引物检查一个阴性控制区和两个增强子、启动子和CTCF位点5:表S4。相对于阴性控制区和5%输入材料,测定ChIP富集的平均倍数变化。使用PowerUp SYBR Green Master Mix (Applied Biosciences, A25742)对每两个生物重复进行3个技术重复。数据表示为每个ChIP的6个总重复的平均平均折叠变化±标准偏差。使用2尾未配对引物组确定WT和耗尽细胞系之间的显著性t测试时用星号表示图示中的重要性。
Co-immunoprecipitation (coIP)
在PBS中刮取细胞。使用核复合物Co-IP Kit (Active Motif, 54001)提取用于共免疫沉淀的样品,并采用自制的核部分消化步骤。按照制造商的说明分离核。核在200 ul Buffer A (10 mM HEPES pH 7.9, 10 mM KCl, 1.5 mM MgCl)中裂解2, 340 mM蔗糖,10%甘油)用1X蛋白酶抑制剂鸡尾酒(PIC) (Sigma Aldrich, 11697498001),用10U Benzonase (Sigma Aldrich, E1014)在37℃下消化15分钟。用2 ul 0.5 M EDTA淬灭反应,在冰中孵育5分钟。样品在5000 × g下4℃旋转5分钟,收集含核蛋白的上清液。使用量子比特蛋白质分析定量试剂盒(Invitrogen, Q33211)定量蛋白质水平。每个IP在低严格和高严格的原生(未交联)条件下使用100 ug蛋白质,按试剂盒说明书指示。使用的IP抗体包括PDS5A (Bethyl, A300-089A)、PDS5B (Bethyl, A300-538A)、RAD21 (Abcam, ab992)、SMC3 (Abcam, ab9263)、NIPBL (Bethyl, A301-779A)和IgG (Bethyl, P120-101)。蛋白G Dynabeads (Invitrogen 10004D)在加入蛋白提取物前与抗体孵育4-6小时。按照制造商的说明对珠子进行清理。IP材料在50 ul IP洗脱缓冲液(50 mM Tris pH 8, 10 mM EDTA和1% SDS)中在65°C下洗脱1小时,每10分钟旋转一次以保持珠子悬浮。
分馏
细胞胰蛋白酶化(Gibco, 12604-013), siRNA转染48小时后计数,每种条件收集1000万个细胞。使用培养细胞亚细胞蛋白分离试剂盒(Thermo Scientific, 7884D)收集染色质结合和未结合(核可溶性)部分。按照制造商对100ul包装细胞体积的说明进行馏分的收集,在每次收集之间进行额外的PBS洗涤。
西方墨点法
用PBS清洗融合的mESCs,并通过刮痧收集。通过将细胞小球重悬于含有1X蛋白酶抑制剂鸡尾酒(PIC) (Sigma Aldrich, 11697498001)的10 ml裂解缓冲液A (10 mM HEPES pH 7.9, 10 mM KCl, 0.1 mM EDTA和0.1 mM EGTA)中,并在4℃下摇晃15分钟,收集蛋白质提取物。加入1 ml 10% NP-40,样品立即涡旋,在4℃下以1350 × g成粒5分钟。去除上清液,将颗粒悬浮在含有1X PIC的1ml冷缓冲液TEN250/0.1 (50 mM Tris-HCl pH 7.5, 250 mM NaCl, 5 mM EDTA和0.1 mM NP-40)中,并在4°C下旋转至少30分钟。样品在4°C下以最大速度旋转10分钟后,收集核部分(上清液)。使用量子比特蛋白质测定定量试剂盒(Invitrogen, Q33211)定量蛋白质水平,在4-20% Tris-Glycine凝胶(BioRad, 4568094)中运行,并转移到PVDF膜(VWR, BSP0161)。膜用5%阻断级缓冲液(BioRad, 1706404)阻断1小时,用一抗在4°C摇晃孵育过夜。使用的主要抗体有PDS5A (Bethyl, A300-089A)、PDS5B (Bethyl, A300-538A)、RAD21 (Bethyl, A300-080A)、SMC1A (Bethyl, A300-055A)、SMC3 (Abcam, ab9263)、STAG1 (Bethyl, A300-157A)、STAG2 (Bethyl, A300-158A)、CTCF (Active Motif, 61311)、组蛋白H3 (Abcam, ab1791)、Actin (Abcam, ab190476)、SMC3ac (Millipore, MABE1073)、NIPBL (Santa Cruz, sc-374625)和WAPL (Bethyl, A300-268A)。所有抗体用1X TBS-T洗涤3 × 10 min。二抗在4℃摇1 h。使用的二抗为驴抗兔(GE Healthcare, NA934)、兔抗山羊(Abcam, ab97100)和山羊抗小鼠(Invitrogen, A16072)。 Membranes were imaged using either Thermo SuperSignal West Pico PLUS (34577) or Thermo SuperSignal West Femto (34094) chemiluminescent substrates with an Amersham Imager 600 (GE Healthcare). Quantification of blots were performed using Fiji [66].在相关的图形图例中描述了量化的规范化。
RNAi
计数细胞,5 × 105每孔镀6孔板。按照制造商说明,使用DharmaFECT 1转染试剂(Dharmacon, T-2001),每孔转染50 nM siRNA或siGLO转染对照。使用的siRNA试剂包括siGLO (D-001630-01-0)、siPds5b (M-058400-01-0005)、siStag1 (M-041989-01-0005)、siStag2 (M-057033-01-0005)、siSMC3 (M-064492-01-0005)和sininipbl (M-048662-00-0005)。48小时后收集细胞进行ChIP、蛋白提取、RNA或流式细胞术。
rna测序和分析
每个基因型使用三个重复的单个CRISPR克隆。复制一用于Pds5a−−/用于mESCs和复制2Pds5b−−/制。转染siRNA 48小时后从6孔板中收集细胞。细胞在Trizol (Invitrogen, 15596018)中重悬,RNA提取和纯化使用Zymo Direct-zol RNA MiniPrep试剂盒(Zymo, R2050)。利用poly-A转录本富集制备文库,并由Novogene在NovaSeq 6000仪器上测序,配对端reads为150 bp。rna测序样本概述在附加文件1:表S1。
利用STAR (v2.7.5)将RNA测序的Reads与mm10基因组进行比对[67].差异表达基因(DEGs)用DESeq2 (v1.24.0)进行鉴定(padj < 0.01) [45].在R中使用dplyr (v1.0.8)生成DEG和所有联合DEG列表的重叠[68].使用GraphPad PRISM生成相关图,然后进行Pearson相关分析。使用pheatmap (RRID:SCR_016418)生成R基因表达log2倍变化的热图。使用Microsoft Excel绘制细胞身份基因表达的log2倍变化的柱状图,显著性由DESeq2确定。对于基因本体(GO)分析,的列表Pds5a−−/具体的,Pds5b−−/特异性、共同和冗余差异表达基因分为上调和下调基因组。使用ShinyGO软件包对这8个基因集进行GO分析(FDR < 0.05) [69].对8个deg子集也做了同样的实验Pds5a−−/siPds5b具体,Stag2−−/特定于siStag1,并且对这两种条件都通用(附加文件2:图S4I)。每个子集的前30个丰富术语在附加文件中给出4:表S3。从ProteomicsDB下载123种细胞和组织类型中PDS5A和PDS5B的相对蛋白质丰度(log10归一化iBAQ强度)[47].从癌症细胞系百科全书下载了1019个转化人类细胞系的基因表达计数。PDS5蛋白表达以Pds5a与Pds5b转录水平之比表示,并使用Microsoft Excel绘制累积分布函数。mESCs的Pds5a和Pds5b转录本的读取计数是根据我们自己在WT细胞中的RNA-seq数据确定的,使用的是DESeq2的标准化读取输出。WT和敲除基因型的转录本水平之间的显著性是用双尾不配对法确定的t测试。小提琴图是使用GraphPad Prism生成的。显著性通过Kruskal-Wallis检验和Dunn多重比较检验确定。超级增强子结构域、Polycomb结构域和ChIA-PET锚的列表从[46],使用UCSC LiftOver将坐标从mm9转换为mm10。非deg启动子对应于在每个PDS5扰动条件下没有差异表达的基因,如果在任何条件下都没有检测到转录本,则排除基因。
细胞周期分析
流式细胞术中,siRNA转染48小时后的mESCs用10um EdU (Santa Cruz, sc-284628)在37°C下孵育30分钟。按照正常胰蛋白酶化方案收集细胞。PBS洗涤后,细胞在2300 × g下旋转3分钟,在500 ul 4%多聚甲醛PBS中重悬,室温下孵育15分钟。加入1ml 1% BSA帮助细胞成粒,然后将细胞重悬于1ml 1% BSA + 0.5% Triton X-100中,在室温下再孵育15分钟。在2300 × g压球5分钟后,细胞在500 ul标记液(PBS, 1 mM CuSO)中重悬4, 1 uM AlexaFluor 647 (Life Technologies, A10277)和100 mM抗坏血酸),并在室温下避光孵育30分钟。细胞先加入1ml 1% BSA + 0.5% Triton X-100,然后以2300 × g旋转5分钟。然后将细胞重悬在500 ul DAPI染色液中(1% BSA + 0.5% Triton X-100, 100ug/ml RNAse, 1ug /ml DAPI (Life Technologies, D1306)),在37℃避光孵育1小时。细胞中加入1 ml PBS,以2300 × g旋转5 min。将细胞重新悬浮在最终容量为300ul的PBS中,通过过滤器顶部管,并进行流动分析。使用Attune NxT流式细胞仪对细胞进行分类,使用FlowJo™v10.8 Software (BD Life Sciences)进行细胞周期分析。数据以三次生物重复的平均值±标准差表示。使用双尾未配对法确定每个阶段WT和消耗条件之间的显著性t测试时用星号表示图示中的重要性。
流式细胞术中使用的相同7种条件的增殖试验,如前所述进行了3次生物重复[12].由此,计算每种条件下细胞在72到84小时之间的群体倍增时间(群体倍增时间=培养时间/ PD,其中PD = log2(收获时的#细胞/最初电镀的#细胞)。结合每个细胞周期阶段的细胞百分比,计算每个细胞周期阶段的时间长度(小时)。使用双尾未配对法确定每个阶段WT和消耗条件之间的显著性t测试时用星号表示图示中的重要性。
数据和材料的可用性
支持本文结论的数据集可在GEO存储库中获得,登录号为GSE199356。可以通过转到来访问数据https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE199356.所有个人登录号码可在附加文件1:表S1。
缩写
- 制:
-
小鼠胚胎干细胞
- WT:
-
野生型
- ChIP-seq:
-
染色质免疫沉淀,测序
- coIP:
-
Co-immunoprecipitation
- 知识产权:
-
免疫沉淀反应
- RNA-seq:
-
RNA序列
- 度:
-
差异表达基因
- SD:
-
Super-enhancer域
- 帕金森病:
-
Polycomb域
- 走:
-
基因本体论
参考文献
内聚物的作用与机制。植物学报。2009;43(1):525-58。
李志强,李志强,李志强,等。内聚蛋白复合体在染色体生物学中的作用。基因工程学报,2008;
刘伟,李丽娟,李志刚,刘志刚。Scc3内聚蛋白的染色体负载与基因序列的关系。中国生物医学工程学报。2021;6(1):3。
李志强,李志强,胡斌,等。Scc2是内聚蛋白atp酶的有效激活剂,通过不含Pds5的结合Scc1促进装载。中国生物医学工程学报,2018;40(6):1134-1148。
Davidson IF, Bauer B, Goetz D, Tang W, Wutz G, Peters J-M。DNA环被人体内聚物挤压。科学。2019;366(6471):1338 - 45。
金勇,史志,张华,于辉。人内聚物通过环挤压压缩DNA。科学。2019;366(6471):1345 - 9。
Panizza S, Tanaka T, Hochwagen A, Eisenhaber F, Nasmyth K. Pds5与内聚蛋白合作维持姐妹染色单体内聚。中国生物医学杂志,2000;10(24):1557-64。
Losada A, Yokochi T, Hirano T. Pds5在人类细胞和爪蟾卵提取物中内聚介导的内聚的功能贡献。中国生物医学工程学报,2005;29(3):344 - 344。
田中K,郝Z,凯M,冈山H.裂变酵母姐妹染色单体内聚的建立和维持机制。中国生物医学工程学报。2001;20(20):5779-90。
张斌,常杰,付敏,黄娟,Kashyap R, Salavaggione E,等。内聚蛋白调节因子PDS5对哺乳动物发育的剂量效应:内聚疾病的意义。PLoS ONE。4 (5): 2009; e5232。
Carretero M, Ruiz-Torres M, Rodríguez-Corsino M, Barthelemy I, Losada A. Pds5B是着丝粒内聚建立和极光B积累所必需的。中国生物医学工程学报。2013;32(22):2938-49。
Arruda NL, Carico ZM, Justice M,刘玉峰,周杰,Stefan HC,等。STAG1和STAG2在胚胎干细胞内聚蛋白定位和基因表达中的不同和重叠作用。表观遗传学染色质。2020;13(1):1 - 17。
卡萨V, Moronta Gines M, Gade Gusmao E, Slotman JA, Zirkel A, Josipovic N,等。内聚蛋白STAG亚基在染色质环和转录控制中的冗余和特异性作用。Genome res 2020;30(4):515-527。
Kojic A, Cuadrado A, De Koninck M, Giménez-Llorente D, Rodríguez-Corsino M, Gómez-López G,等。内聚体- sa1和内聚体- sa2在三维染色体组织中的不同作用。分子生物学杂志,2018;25(6):496-504。
Cuadrado A, Giménez-Llorente D, Kojic A, Rodríguez-Corsino M, Cuartero Y, Martín-Serrano G,等。内聚物- sa1和内聚物- sa2对胚胎干细胞TADs和多梳结构域的特异性贡献。2019;27(12):3500-3510.e4。
李志强,李志强,李志强,等。内聚蛋白卷曲线圈的折叠对于scc2 /4诱导的与染色体的结合是重要的。Elife。2021;十四10。
Kikuchi S, Borek DM, Otwinowski Z, Tomchick DR, Yu H.内聚素装载器Scc2的晶体结构及内聚病的研究。中国科学:自然科学,2016;29(4):344 - 344。
乔斯克R,白山M,舍甫琴科A,田中T,托特A,舍甫琴科A,等。内聚蛋白与染色体的结合依赖于由Scc2和Scc4蛋白质组成的单独复合体。生物化学学报,2000;5(2):243-54。
汉森。CTCF作为内聚物介导的环挤压的边界因子:多步骤机制的证据。核。2020;11(1):132 - 48。
Wutz G, Várnai C, K Nagasaka, DA Cisneros, RR Stocsits, Tang W,等。拓扑相关结构域和染色质环依赖于内聚蛋白,并由CTCF、WAPL和PDS5蛋白调控。中国生物医学工程学报。2017;36(24):3573-99。
Bauer BW, Davidson IF, Canena D, Wutz G, Tang W, Litos G,等。内聚蛋白通过“摆动和夹钳”机制介导DNA环的挤压。细胞。2021;184(21):5448 - 5464。
向勇,李志强,李志强,等。内聚蛋白结构与聚类研究。Elife。2021;1(10):1-49。
查帕德C,琼斯R,范Oepen T,谢诺斯特JC, Nasmyth K.内聚蛋白复合体中并列的smc头和kleisin之间的姐妹DNA诱捕。中国生物医学工程学报,2019;29 (2):344 - 344 .e5。
史瑞尼瓦桑,谢诺斯特,裴德华,李志强,等。内聚环利用它的铰链利用非拓扑和拓扑机制来组织DNA。细胞。2018;173 (6):1508 - 1519. - e18。
Kueng S, Hegemann B, Peters BH, Lipp JJ, Schleiffer A, Mechtler K,等。Wapl控制内聚蛋白与染色质的动态关联。细胞。2006;127(5):955 - 67。
欧阳忠,郑刚,宋杰,Borek DM, Otwinowski Z, Brautigam CA,等。人内聚蛋白抑制剂Wapl的结构。美国国家科学研究院。2013; 110(28): 11355 - 60。
Morales C, Ruiz-Torres M, Rodriguez-Acebes S, Lafarga V, Rodríguez-Corsino M, Megias D,等。PDS5蛋白是适当的内聚动力学所必需的,并参与复制叉保护。中国生物医学工程学报,2019;29(1):457 - 457。
Dauban L, Montagne R, Thierry A, Lazar-Stefanita L, Bastié N, Gadal O,等。eco1和其他伙伴对内聚蛋白介导的染色体折叠的调控。中国生物医学工程学报。2020;27(6):1279-1293。
Gabriele M, Brandão HB, gross - holz S, Jha A, Dailey GM, Cattoglio C,等。活细胞成像显示CTCF和内聚蛋白介导的染色质环的动态变化。科学。2022;376(6592):496 - 501。
Bastié N, Chapard C, Dauban L, Gadal O, Beckouët F, Koszul R. Smc3乙酰化,Pds5和Scc2控制转位酶活性,建立内聚素依赖的染色质环。分子生物学学报,2018;29(6):575-585。
Barton RE, Massari LF, Robertson D, Marston AL.依赖于eco1的内聚蛋白乙酰化锚定染色质环和内聚,定义功能性减数分裂染色体域。Elife。2022;1:11。
Wutz G, Ladurner R, St Hilaire BG, Stocsits RR, Nagasaka K, Pignard B,等。ESCO1和CTCF通过保护cohesinSTAG1不受WAPL的影响而形成长染色质环。Elife。2020;透。
Ben-Shahar TR, Heeger S, Lehane C, East P, Flynn H, Skehel M,等。姐妹染色单体内聚建立过程中eco1依赖性内聚蛋白乙酰化。科学。2008;321(5888):563 - 6。
Ünal E, Heidinger-Pauli JM, Kim W, Guacci V, Onn I, Gygi SP,等。建立姐妹染色单体内聚的分子决定因素。科学。2008;321(5888):566 - 9。
陈杰,陈志强,陈志强,陈志强,陈志强,等。Esco1和Esco2在细胞周期进程中调节不同的内聚蛋白功能。美国国家科学研究院。2017; 114(37): 9906 - 11所示。
Whelan G, Kreidl E, Wutz G, Egner A, Peters JM, Eichele G.内聚蛋白乙酰转移酶Esco2是一种细胞活力因子,是中心外异染色质内聚所必需的。中国生物医学工程学报,2012;31(1):71-82。
Minamino M, Ishibashi M, Nakato R, Akiyama K, Tanaka H, Kato Y,等。Esco1通过不同于Esco2的机制乙酰化内聚蛋白。中国生物医学杂志,2015;25(13):1694-706。
Bastié N, Chapard C, Dauban L, Gadal O, beckoulac F, Koszul R. Smc3乙酰化,Pds5和Scc2控制转位酶活性,建立内聚素依赖的染色质环。分子生物学学报,2018;29(6):575-585。
Ladurner R, Kreidl E, Ivanov MP, Ekker H, Idarraga-Amado MH, Busslinger GA,等。Sororin积极维持姐妹染色单体内聚。Embo j . 2016;35(6): 635-53。
Vaur S, Feytout A, Vazquez S, Javerzat j p。Pds5促进内聚蛋白乙酰化和稳定的内聚蛋白-染色体相互作用。EMBO报告2012;13(7):645-52。
蒙达尔G,史提夫斯M,古德B,阿什沃斯A,所罗门DA。在复制叉进展中对STAG2的需求在内聚突变型癌症中产生了可靶向的合成致死率。自然科学进展。2019;10(1):1686。
陈佳玲,李志强,李志强,李志强,等。Pds5促进和保护内聚蛋白乙酰化。美国国家科学研究院。2013; 110(32): 13020 - 5。
张志刚,张志刚,张志刚,张志刚,等。内聚蛋白通过转录CTCF和Wapl在哺乳动物基因组中定位。自然。2017;544(7651):503 - 507。
Ross-Innes CS, Stark R, Teschendorff AE, Holmes KA, Ali HR, Dunning MJ,等。差异雌激素受体结合与乳腺癌临床结局相关。自然。2012;481(7381):389 - 93。
Love MI, Huber W, Anders S.用DESeq2调节RNA-seq数据的折叠变化和分散估计。中国生物医学工程学报。2014;15(12):550。
Dowen JM,范志平,Hnisz D,任刚,Abraham BJ,张丽宁,等。细胞识别基因的控制发生在哺乳动物染色体的绝缘区。细胞。2014;159(2):374 - 87。
刘志强,刘志强,刘志强,等。ProteomicsDB:为生命科学研究提供公平的开源资源。核酸Res. 2022;50(D1): D1541-52。
唐文杰,陈志强,陈志强,等。Hübner MR。转录调控元件染色体复合体的多重结构维持。中国生物医学工程杂志,2013;1(5):371-8。
Rhodes J, Mazza D, Nasmyth K, Uphoff S. Scc2/Nipbl在加载后在染色体内聚蛋白环之间跳跃。Elife。2017;15:6。
Remeseiro S, Cuadrado A, Kawauchi S, Calof AL, Lander AD, Losada A.在Cornelia de lange综合征小鼠模型中,nipbl的减少损害局部内聚蛋白负载,影响转录,但不影响内聚蛋白依赖功能。生物化学学报。2013;1832(12):2097-102。
Zuin J, Franke V, van IJcken WFJ, van der Sloot A, Krantz ID, van der Reijden MIJA,等。NIPBL在启动子中的内聚力独立作用为CdLS提供了新的见解。公共科学图书馆。2014;10(2):1004153。
欧阳志,郑刚,罗霞,于慧。pds5依赖内聚动力学的结构基础和IP6要求。中国生物医学工程学报,2016;36(2):344 - 344。
原坤,郑刚,曲强,刘华,欧阳志,陈震,等。内聚蛋白亚复合物的结构确定了中心粒内聚中shugoshin-Wapl的直接拮抗作用。分子生物学杂志,2014;21(10):864。
Morales C, Ruiz-Torres M, Rodríguez-Acebes S, Lafarga V, Rodríguez-Corsino M, Megías D,等。PDS5蛋白是适当的内聚动力学所必需的,并参与复制叉保护。中国生物医学工程学报,2010;29(1):457 - 457。
Kagey MH, Newman JJ, Bilodeau S, Zhan Y, Orlando DA, van Berkum NL,等。中介和内聚蛋白连接基因表达和染色质结构。大自然。2010;467(7314):430 - 5。
Justice M, Carico ZM, Stefan HC, Dowen JM。WIZ/内聚蛋白/CTCF复合物锚定DNA环以定义基因表达和细胞身份。Cell rep 2020;31(2): 107503。
Langmead B, Trapnell C, Pop M, Salzberg SL.人类基因组短DNA序列的超快和内存高效对齐。中国生物工程学报。2009;10(3):1-10。
李华,Handsaker B, Wysoker A, Fennell T, Ruan J, Homer N,等。p fosequence对齐/marmat和SAMtools。生物信息学。2009;25(16):2078 - 9。
昆兰AR,霍尔IM。BEDTools:用于比较基因组特征的一套灵活的实用程序。生物信息学,2010,26(6):841 - 2。
张勇,刘涛,梅耶,Eeckhoute J, Johnson DS, Bernstein BE,等。基于模型的ChIP-Seq (MACS)分析。中国生物工程学报。2008;9(9):R137。
奥兰多·达,陈兆伟,布朗·维,索兰基·S,崔玉杰,奥尔森·ER,等。定量ChIP-Seq归一化揭示了表观基因组的整体调控。细胞科学进展,2014;9(3):1163-70。
王俊杰,苏伟杰,张志刚,张志刚,张志刚,等。UCSC的人类基因组浏览器。基因组学报,2002;12(6):996-1006。
Robinson JT, Thorvaldsdóttir H, Winckler W, Guttman M, Lander ES, Getz G,等。整合基因组学查看器。中国生物工程学报。2011;29(1):24-6。
Ramírez F, Ryan DP, Grüning B, Bhardwaj V, Kilpert F, Richter AS,等。deepTools2:用于深度测序数据分析的下一代web服务器。核酸研究,2016;44(W1): W160-5。
Whyte WA, Orlando DA, Hnisz D, Abraham BJ, Lin CY, Kagey MH,等。主转录因子和中介在关键细胞识别基因上建立超级增强子。细胞。2013;153(2):307 - 19所示。
张志刚,张志刚,张志刚,等。斐济:生物图像分析的开源平台。光子学报。2012;9(7):676-82。
杜宾A, Davis CA, Schlesinger F, Drenkow J, Zaleski C, Jha S,等。STAR:超快通用RNA-seq对准器。生物信息学。2013;29(1):15至21。
韦文杰,张文杰,张文杰,等。欢迎来到Tidyverse。[J] .软件学报。2019;4(43):1686。
葛士喜,荣德,姚荣。ShinyGO:动物和植物的图形丰富工具。生物信息学。2020;36(8):2628 - 2629。
确认
我们感谢唐恩实验室和丹尼尔·麦凯博士实验室的所有其他成员的有益讨论和评论。此外,我们感谢Robert Dowen博士对手稿的批判性阅读。我们还要感谢北卡罗来纳大学高通量测序设施的工作人员对测序的支持,以及北卡罗来纳大学流式细胞仪核心的工作人员和Jean Cook博士实验室的成员对流式细胞仪的支持。
资金
本工作得到NIGMS R35GM124764的支持。NLA部分由国家普通医学科学研究所根据5T32GM007092和国家科学基金会研究生研究奖学金计划提供资助。本材料是基于国家科学基金会研究生研究奖学金项目资助的工作。dge - 1650116。本材料中表达的任何观点、发现、结论或建议都是作者的观点,并不一定反映美国国家科学基金会的观点。
作者信息
作者及隶属关系
贡献
NLA和JMD构想了研究并设计了实验。NLA和AFB进行实验。NLA和JMD分析了数据。NLA和JMD根据空军b的输入编写了手稿。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者声明没有利益竞争。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
Arruda, N.L, Bryan, A.F. & Dowen, J.M. PDS5A和PDS5B在不改变整个基因组内聚蛋白定位的情况下差异地影响基因表达。表观遗传学与染色质15, 30(2022)。https://doi.org/10.1186/s13072-022-00463-6
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s13072-022-00463-6
关键字
- Cohesin
- 基因组
- 本地化
- 基因表达
- PDS5A
- PDS5B