摘要
背景
印迹控制区(ICRs)是在女性和男性生殖系中获得差异甲基化的富含cpg的序列,并在体细胞中以亲代起源特异性的方式保持它。尽管由于甲基化胞嘧啶的自发脱氨,icr的突变率很高,但在哺乳动物物种中,icr显示了cpg丰富度和含有cpg的转录因子结合位点的守恒。然而,对于在这些位点上维持甲基cpg高密度的机制知之甚少。
结果
为了获得维持CpG甲基化机制的功能洞察,我们试图通过确定识别小鼠ESCs中这些DNA区域甲基化六核苷酸基序的ZFP57的相互作用体来确定与ICRs的甲基化等位基因结合的蛋白质。通过使用标记方法结合LC-MS /MS分析,我们鉴定了几种蛋白质,包括参与mRNA加工/剪接、染色体组织、转录和DNA修复过程的因素。在确定的ZFP57相互作用体中存在的复制后错配修复(MMR)复合物成分MSH2和MSH6促使我们通过染色质免疫沉淀和测序来研究它们的DNA结合谱。我们证明了MSH2在未甲基化CpG岛重叠的基因启动子和重复序列中富集。我们还发现MSH2和MSH6都与ICRs的甲基化等位基因相互作用,其中它们与DNA的结合是由ZFP57/KAP1复合物介导的。
结论
我们的研究结果表明,MMR复合物集中在基因启动子上,并在小鼠ESCs中重复,这表明维持这些区域的完整性是高度增殖细胞的主要功能。此外,通过与ZFP57/KAP1的相互作用,MSH2/MSH6被募集到icr的甲基化等位基因,这表明MMR复合体在维持这些调控区域的完整性和哺乳动物基因组印迹进化中的作用。
背景
基因组印记是哺乳动物基于雌雄配子体发生过程中表观遗传标记差异获取的基因调控机制[1].这些标记在发育后期对母系遗传和父系遗传附近基因等位基因的表达产生相反的影响。印迹基因修饰的最佳特征形式是CpG甲基化,迄今为止识别的大多数印迹控制区(ICRs)重叠了富含CpG的序列(种系衍生的差异甲基化区域),其甲基化在女性和男性生殖细胞中差异建立,并在整个发育过程中忠实地维持在体细胞中。
在着床前发育期间,维持icr的差异甲基化尤为关键,这是发生表观遗传重编程的阶段。一些人类疾病(统称为印迹障碍)是由于这一阶段特定印迹位点甲基化的缺失或增加而引起的[2].包括锌指蛋白ZFP57及其辅助因子KAP1在内的多蛋白复合物特异性地识别ICRs的甲基化等位基因,并且在小鼠胚胎和胚胎干细胞中维持其DNA甲基化是必需的[3.,4,5,6].ZFP57/KAP1结合对于维持icr母系和父系等位基因上的不同组蛋白标记以及印迹基因的等位基因特异性表达也是必要的[7,8,9,10].在人类中,双等位基因功能缺失的突变ZFP57导致短暂性新生儿糖尿病1型和多基因座印记障碍[11].虽然ZFP57在ICRs的一个等位基因上识别多个含有cpg的甲基化基序,但这些调控区域的另一个非甲基化等位基因与防止重新甲基化和促进印迹基因转录所需的其他几个转录因子结合[2].
哺乳动物基因组中CpGs的突变率通常较高,因为这些二核苷酸通常是甲基化的,而甲基化的胞嘧啶是不稳定的,因为它经历了脱氨到胸腺嘧啶的过程,如果没有纠正,会产生C到T的转变[12].这一机制是哺乳动物基因组中CpG二核苷酸频率较低的主要原因。一个例外是重叠基因启动子的CpG岛,在种系中通常是非甲基化的,因此保留了它们的CpG丰富度。尽管ICRs在种系的一个等位基因上被甲基化,但它们保持了2-4 kb长的富含cpg区域的特征,并在哺乳动物中显示了含有cpg的转录因子结合位点的保存[13,14].
错配修复复合体(MMR)识别双链DNA中的错配碱基,并被要求修复由5-甲基胞嘧啶(5mC)脱氨导致的G:T错配[15].值得注意的是,未修复的5mC脱氨事件引起的突变在mmr缺陷的癌症中普遍存在,特别是由MSH2-MSH6异二聚体组成的MutSα异二聚体缺陷的癌症[15].
为了进一步了解ICRs中甲基cpg的维持机制,我们试图通过确定小鼠ESCs中的ZFP57相互作用体来确定与ICRs的甲基化等位基因相互作用的蛋白质。我们能够识别参与mRNA加工/剪接、染色体组织、转录和DNA修复过程的几个因素。我们重点研究了错配修复复合物MSH2和MSH6的组分,并通过染色质免疫沉淀和测序研究了它们的DNA结合谱,证明了它们与ICRs的相互作用。虽然发现MSH2优先富集在非甲基化的CpG岛,但MSH2和MSH6也通过ZFP57/KAP1复合物与ICRs的甲基化等位基因相互作用。
结果
LC-MS /MS鉴定zfp57相互作用蛋白
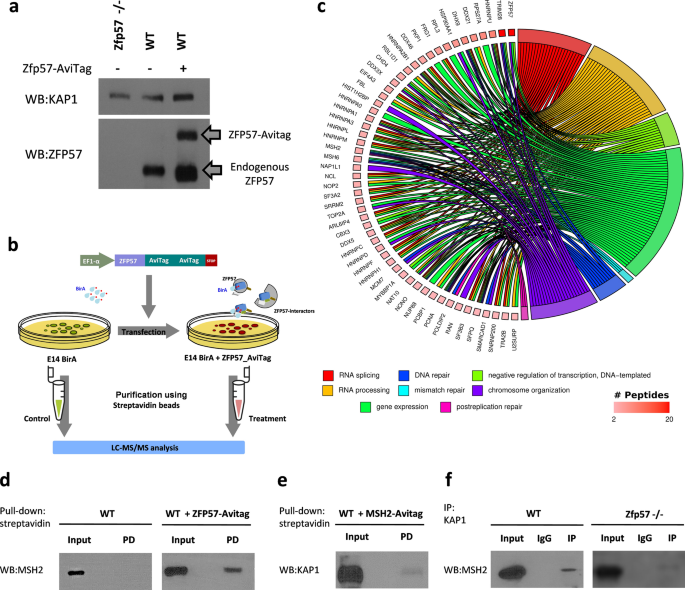
由于商业性ZFP57特异性抗体的特异性较差且检测背景较高,我们使用标记方法鉴定与ZFP57相互作用的蛋白。我们选择了可以在表达细菌生物素连接酶BirA的细胞中用生物素标记的Avi-Tag,随后用链霉亲和素拉下[16].该方法应用于小鼠胚胎干细胞(ESCs) E14,其中内源性Zfp57基因具有高表达和功能性[4].因此,表达bira的E14 ESCs被标记转染Zfp57基因(Zfp57 -AviTag)(无花果。1a)和与生物素化的ZFP57-AviTag相互作用的蛋白质用链霉亲和素包被的珠子从细胞裂解液中拉下来,纯化并用质谱分析。为了排除可能被BirA特异性生物素化的蛋白,我们还分析了模拟转染的表达BirA的E14 ESCs。只有蛋白质被检测到Zfp57 -avtagg转染细胞(附加文件1:表S1a)进行进一步分析。该过程的示意图如图所示。1b. LC-MS /MS分析使我们鉴定出60个高可信度的zfp57相互作用蛋白(附加文件1:表S1a)。该列表包括KAP1和HP1γ,它们已经被报道为ZFP57相互作用子[4,17],以及几种新蛋白质。基因本体(GO)分析允许将ZFP57相互作用子分类为几个功能类别,包括mRNA处理/剪接、染色体组织、转录和DNA修复过程(图2)。1c).使用STRING数据库[18],我们重建了zfp57相互作用蛋白的网络模型(附加文件2:图S1a)。这种方法使我们能够识别出几种可能与ZFP57相互作用的蛋白质家族或复合物,包括组蛋白H1变体、异质核核糖核蛋白和复制后DNA错配修复(MMR)复合物,包括MSH2、MSH6和PCNA。
ZFP57在小鼠ESCs中的高置信相互作用伙伴。一个Western blot分析野生型(WT) ZFP57和KAP1,Zfp57-AviTag-transfected和Zfp57- / - ESCs。请注意,avtagg -ZFP57迁移为60 kDa波段,而内源性ZFP57迁移为50 kDa波段。b标记蛋白质谱方法用于识别ZFP57相互作用体的实验工作流程。的Zfp57将cDNA克隆到表达载体pEF6-Avitag-GGGx2-Avitag中。Zfp57-AviTag转染稳定表达bira的小鼠ESCs,生物素标记的拉出蛋白使用链霉亲和素拉出。用胰蛋白酶消化沉淀蛋白复合物进行LC-MS /MS分析。共同的蛋白质Zfp57- avitag转染和mock转染bira表达的E14 ESCs被排除在进一步的分析之外。培养皿和eppendorf管的图像取自https://doi.org/10.7875/togopic.2020.104而且https://doi.org/10.7875/togopic.2022.115,分别。BirA为浅蓝色,生物素为红色,ZFP57为深蓝色,ZFP57-相互作用子为灰色、绿色和橄榄色。Zfp57- avtag转染和模拟转染bira表达的E14 ESCs分别用红色和绿色表示。c弦图显示ZFP57相互作用子的富集GO簇。根据相对富集度排序,LC-MS /MS分析鉴定的蛋白质显示在左侧,富集的氧化石墨烯簇在右侧用不同的颜色表示。LC-MS /MS分析中识别蛋白质的多肽数量以红色梯度显示(2-20个多肽)。d用链霉亲和素在WT和Zfp57- avitag转染ESCs,抗msh2抗体检测。e用链霉亲和素拉低蛋白质的Western blotMsh2- avitag转染ESCs,经抗kap1抗体检测。fWestern blot检测WT和Zfp57-/- ESCs中抗kap1抗体免疫沉淀蛋白,并检测抗msh2抗体。输入相当于1%的细胞裂解液用于免疫沉淀
我们对MMR复合物的MSH2/MSH6异源二聚体与ZFP57的相互作用特别感兴趣,因为它在icr的CpG守恒中具有潜在作用。然后,我们通过共沉淀和Western blotting验证了MSH2与ZFP57-AviTag的相互作用。1d).同样,我们证明了KAP1与avi标记的MSH2相互作用(图。1e),内源性MSH2和KAP1蛋白在野生型E14 ESCs中相互作用,并且这种相互作用在E14 ESCs中维持Zfp57-/- ESCs(图;1f).总的来说,这些结果证实了DNA错配修复复合物与ZFP57的相互作用,并表明这可能是由KAP1介导的。
MSH2的dna结合谱
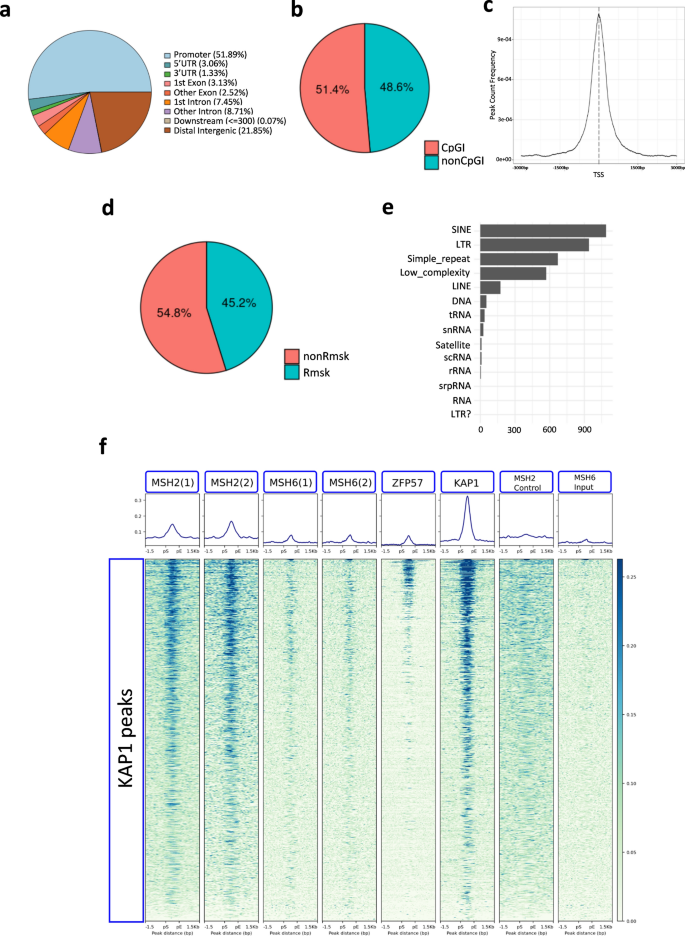
在验证了MSH2与ZFP57-KAP1复合物之间的相互作用后,我们确定了MSH2在小鼠ESCs中的DNA结合谱。我们使用了基于融合AviTag的蛋白质和链霉亲和素的下拉方法。该方法已被证明对染色质免疫沉淀和测序(Bio-ChIP-seq)有效,特别是当针对内源性蛋白的可用抗体产生高背景[19].应用Bio-ChIP-seq协议确定E14 ESCs中的MSH2结合,发现两个重复之间有4444个共享峰(附加文件)1:表S2a及S2b)。MSH2富集最多的基因组区域对应于基因启动子(52%)和CpG岛(CpGI, 51,4%),大多数峰重叠于转录起始位点(TSS,图。2a-c和附加文件2:图S2),包括几种细胞生长控制基因的启动子,如小君,Jund,”丛书而且国家管制当局方面(附加文件2:图S3)。此外,大约45%的MSH2峰与重复序列重叠,其中最丰富的类别是sin和lts(图4)。2d, e)。
Bio-ChIP-seq和ChIP-seq揭示了小鼠ESCs中MSH2和MSH6的全基因组结合谱。一个饼图显示了MSH2 Bio-ChIP-seq峰覆盖的DNA区域内基因组元素的分布。MSH2峰被定义为DNA区域富集于Msh2-AviTag细胞与模拟转染的表达bira的ESCs比较。对于特征注释,我们使用了带有annoDb = "org.Mm.eg.db"参数的plotAnnoPie函数。b饼图表示MSH2 Bio-ChIP-seq峰重叠CpG岛(CpGI)和不包括CpGI(非CpGI)的基因组区域的百分比。cMSH2峰重叠转录起始位点(TSS)的平均谱。该图显示了TSS的- 3000 bp和+ 3000 bp之间的峰值频率。d饼图表示MSH2峰重叠或不重叠掩膜重复元素的百分比分布。e显示MSH2峰覆盖的重复元素的绝对频率的条形图。f热图显示MSH2(2个重复)、MSH6(2个重复)、ZFP57和KAP1在基因组区域与KAP1 ChIP-seq峰值重叠(±1.5 kbp)。作为对照,我们添加了未转染E14 ESCs的Bio-ChIP-seq reads (MSH2对照)和MSH6 ChIP的输入DNA reads (MSH6输入)。KAP1峰值根据每个区域的平均读富集值按降序排序
为了更好地描述MMR蛋白与ZFP57/KAP1复合物之间的关系,我们分析了MSH2 ChIP-seq reads沿1343个KAP1 ChIP-seq峰的分布[7]在E14 esc中。这一分析揭示了许多MSH2峰与ZFP57和KAP1峰的共定位。2f). MSH2和KAP1的重叠主要涉及基因间区和非编码区,一般没有启动子CpGI(补充文件2:无花果。S3和S4)。然而,MSH2和KAP1在icr和非icr ZFP57结合位点的富集尤其强烈2:无花果。S5和S6)。此外,MSH2结合了许多不是ZFP57靶向的KAP1结合位点,支持了MSH2与KAP1相互作用独立于ZFP57的假设(附加文件)2:图S5)。我们还在E14 ESCs中使用针对天然蛋白的抗体进行了MSH6的ChIP-seq实验。用过氧化氧处理细胞以增加MSH6对DNA的募集[20.].虽然这种抗体能识别出很少的峰,但MSH6 ChIP-seq沿着KAP1峰的序列显示了MSH6在KAP1结合位点上的富集,证实了用MSH2获得的结果(图5)。2f和附加文件2:无花果。S4和S5)。
这些结果表明MSH2主要富集在启动子CpGIs和重复序列上。此外,我们发现在小鼠ESCs中,MSH2和MSH6结合的序列与ZFP57和KAP1结合的序列重叠。
MSH2和MSH6与icr的相互作用
然后,我们将重点放在印迹位点上,发现Bio-ChIP-seq揭示的MSH2结合在至少一个重复的10个ICRs上富集,并且MSH2的峰值与这些区域的ZFP57和KAP1的峰值重合(图5)。3.a和附加文件1表S2a, b).由于MSH2 Bio-ChIP-seq检测到的背景值相对较高,我们分别用Bio-ChIP沉淀MSH2和用常规ChIP沉淀MSH6后,通过定量PCR验证了MSH2的结合,并检测了MSH6与ICRs的结合。结果表明,MSH2和MSH6分别在16和14个icr上富集Hoxa3与CpGI不重叠的基因(图;3.b)和icr相邻序列(图;3.c)。
MSH2和MSH6在icr上结合的表征。一个UCSC基因组浏览器的屏幕截图显示,与先前确定的ZFP57和KAP1结合区域相比,在表达bira的E14 ESCs中沿8个ICRs检测到生物素标记的MSH2的Bio-ChIP-seq峰值。icr显示为水平方框,根据RRBS确定的平均DNA甲基化水平着色。Bio-ChIP-seq富集读入Msh2- avitag转染ESCs(2个重复)和模拟转染bira表达ESCs (MSH2对照)分别显示为浅蓝色和绿色;ZFP57和KAP1 ChIP-seq扩增片段分别为粉红色和紫色。b柱状图分别显示生物素标记的MSH2(蓝色)和MSH6(绿色)在WT E14 ESCs中ICRs的相对富集程度,分别由Bio-ChIP和ChIP测定。的Smg7而且Utp3以启动子CpGI为阳性对照,而基因的一个区域Hoxa3Bio-ChIP-seq检测显示不与CpGI重叠且MSH2富集较低的基因作为阴性对照。ChIP值表示为% input。误差条表示三个生物副本的SD。Bio-ChIP在ICRs中显示MSH2-AviTag显著富集Msh2- avittag -相对于Mock转染的表达bira的ESCs (Mock),并且在抗MSH6抗体相对于IgG的ICRs中显示了MSH6的显著富集。c条形图显示MSH2和MSH6在土壤中相对富集Plagl1而且Inpp5ficr和侧翼区域如b所示。来自UCSC基因组浏览器的屏幕截图显示了所分析区域的特征,报告在条形图上方。ICRs为深灰色,CpGI为绿色,ZFP57峰为粉红色。橙色条表示与ZFP57的ChIP-seq峰值重叠的icr。蓝色、橙色和浅灰色条表示用于Q-PCR的引物的位置。ChIP值表示为% input。误差条表示两个生物重复的SD,每个重复在两个技术重复上的平均值。*p< 0.05, **p< 0.01, ***p< 0.001
MSH2和MSH6以依赖zfp57的方式结合甲基化的ICR等位基因
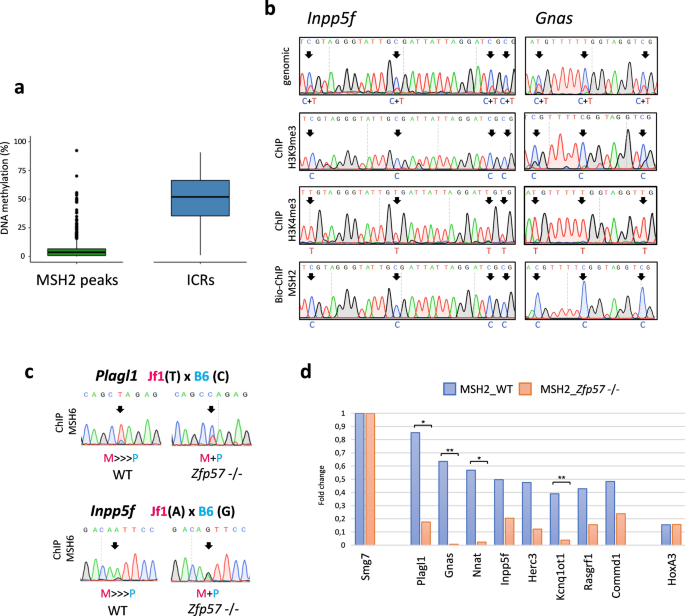
为了研究MSH2和MSH6结合序列的甲基化状态,我们用RRBS测定了E14 ESCs的全基因组甲基化谱。我们发现,大多数可以用这种方法分析的非重复msh2结合区域显示出低甲基化水平(图。4a).唯一的例外是显示平均甲基化水平为50%的icr,与它们的印记状态一致,以及其他一些非印记的zfp57结合位点,其中大多数RRBS分析显示甲基化水平更高(图2)。3.一个和4a和附加文件2:图S6)。
亲本来源特异性的MSH2和MSH6与icr结合。一个显示非重复msh2结合区域(左)和msh2结合ICRs(右)甲基化水平的箱形图。b电泳图显示DNA甲基化水平Inpp5f而且玲娜基因组DNA中的ICRs,抗h3k9me3、抗h3k4me3抗体获得的ChIP DNA和链霉亲和素拉下的Bio-ChIP DNAMsh2- avitag转染ESCs,亚硫酸氢盐测序显示。甲基化水平由甲基化(C)和未甲基化(T)胞嘧啶的比值确定。c电泳图显示杂交WT和MSH6的等位基因特异性结合Zfp57-/- ESCs在Plagl1而且Inpp5f拥有。与母系JF1或父系Black/6 (B6)等位基因相关的snv显示在顶部。母亲相对于父亲的等位基因的相对富集被报告在电泳图下面。黑色箭头表示snv。d条形图显示生物素标记的MSH2蛋白相对于WT和ICRs的富集程度Zfp57- / - ESCs。ChIP值表示为%输入,并归一化到Smg7用作阳性对照的区域
由于icr有一个甲基化的等位基因,另一个非甲基化的等位基因,我们询问MSH2和MSH6与这些基因组区域的结合是否存在任何偏倚。为了区分甲基化和未甲基化的胞嘧啶,我们用亚硫酸氢钠处理E14 ESCs中Bio-ChIP方案沉淀的msh2结合DNA,并通过Sanger测序进行分析。我们观察到甲基化的等位基因Inpp5f而且玲娜ICRs优先富集在msh2结合的DNA中(图。4b).作为对照,我们测试的甲基化状态Inpp5f而且玲娜用抗H3K9me3和抗H3K4me3抗体ChIP后恢复的ICR序列,发现H3K9me3和H3K4me3分别优先富集在甲基化和非甲基化等位基因上。这一观察结果与之前的结果一致[7].为了确定MSH6是否也以等位基因特异性的方式与DMRs相互作用,我们在来自种内小鼠杂交(JF1x C57-Black/6)的ESC系(JB1)中进行了ChIP实验。在该芯片中,icr的父、母等位基因可通过JF1-和C57-Black/6特异性snv进行识别[10].利用这种方法,我们发现MSH6优先富集在猪的JF1等位基因上Plagl1而且Inpp5fJB1 ESCs中的ICRs(图;4c).因为Plagl1而且Inpp5fICRs在母系等位基因上甲基化[10],这些结果证实了MSH2的结果。
由于MSH2和MSH6的结合位点与ZFP57的结合位点重叠,我们想知道这些蛋白在ICRs上的富集是否依赖于这种锌指蛋白。为了解决这个问题,我们首先研究了MSH2绑定到wildtype和icrZfp57-/- E14 ESCs by Bio-ChIP。结果表明,在8个icr上,MSH2富集显著降低Zfp57基因(图。4d).在JB1 ESCs中利用常规ChIP研究ZFP57对MSH6结合的作用。Sanger测序的DNA免疫沉淀与抗MSH6抗体表明,等位基因偏向在MSH6结合Plagl1而且Inpp5f在野生型JB1 ESCs中观察到的ICRs丢失Zfp57- - JB1 ESCs(图;4c)。
总之,这些结果表明MSH2和MSH6优先与ICRs的甲基化等位基因相互作用,并表明DNA结合是由ZFP57介导的。
讨论
icr是具有独特性质的DNA序列[1,2].印迹基因的亲本来源依赖甲基化和等位基因特异性表达的维持需要存在多个cpg和转录因子的结合位点,这些调控序列的两个亲本等位基因上识别甲基化或非甲基化DNA。我们使用ZFP57锌指蛋白作为诱饵,在小鼠ESCs中鉴定与ICRs的甲基化等位基因相互作用的蛋白质。通过高分辨率质谱分析,在ZFP57相互作用组中鉴定了MMR配合物的成员。我们研究了MSH2和MSH6的DNA结合谱,发现它们在非甲基化CpGI上富集,但它们也结合ICRs的甲基化等位基因。分析Zfp57MSH2/MSH6与ICRs的结合是由ZFP57介导的。
通过采用LC-MS / ms方法,我们在小鼠ESCs中鉴定了60种潜在的zfp57相互作用子。有趣的是,所有这些蛋白之前都在人类ESCs和K562细胞中被抗kap1抗体拉下,证实它们是ZFP57-KAP1复合物的真正相互作用体[21].此外,其中一些相互作用,如与h3k9me3相互作用的蛋白HP1γ和染色质重塑因子CHD4的相互作用,在以前的研究中已被描述[4,22].在本研究中发现的新型ZFP57相互作用体中,组蛋白H3.3变体此前已与icr相关,而组蛋白H1变体已被报道可将dnmt招募到小鼠ESCs的这些调控区域[23].特别有趣的是还发现了包括HNRNP-U在内的多种异质核核糖核蛋白,它可能参与塑造大规模染色质结构,控制印记位点上依赖亲本的等位基因表达[24,25].
我们的研究通过LC-MS /MS分析确定了复制后MMR复合物的三个组分(MSH2, MSH6和PCNA)作为ZFP57的相互作用剂。MMR通路对于修复5mC脱氨至关重要,因为MSH2/MSH6异源二聚体识别G:T错配并招募下游蛋白质进行校正[15,26].的确,MMR基因的缺陷增加了癌症中甲基化CpGs的全基因组突变率[15,27].同时,Msh2 / Msh6敲除小鼠表现出更高的癌症易感性、微卫星不稳定性和突变表型[28,29,30.,31].因此,MSH2/MSH6与icr的甲基化等位基因的相互作用可能降低其突变率并在快速分裂的胚胎细胞中保留印记。在MSH2靶位点中发现少量甲基化序列。它们都与ZFP57结合序列相对应,MSH2和KAP1的富集程度特别高,表明在ZFP57-KAP1复合物的介导下,MMR复合物在印迹位点上有特异性的募集。
氧化损伤后,MSH2-MSH6异源二聚体有助于人类细胞中表观遗传沉默蛋白(包括DNMT1、SIRT1和EZH2)招募到启动子CpGI [20.].当修复发生时,这种募集导致转录的短暂减少。我们发现TSS重叠CpGI上MSH2富集与上述结果一致。有趣的是,MSH2/MSH6优先结合到细胞生长控制基因的启动子上,提示了一种快速增殖细胞的保护机制。此外,与丁及其合作者的研究不同的是,[20.],在MSH2 Bio-ChIPseq实验之前,我们的细胞未用过氧化氧处理,这表明MSH2/MSH6在没有氧化损伤的情况下也能识别富含cpg的启动子。然而,应该考虑的是,基因转移后MSH2浓度的增加可能有利于它与我们细胞中DNA的相互作用。因此,不能排除MSH2/MSH6与CpG-rich启动子和ICRs的相互作用优先发生在DNA损伤后。MSH2靶向启动子CpGI似乎一般不受ZFP57/KAP1的介导,其潜在机制需要在未来的研究中阐明。我们的结果与人类细胞中显示的MSH6通过H3K36me3与基因体相互作用不一致,这表明存在物种或细胞类型特异性差异[32].
MSH2 ChIP-seq也显示MSH2在重复元素上富集,主要是sin、lts、低复杂度和简单序列重复。其中一些相互作用可能由KAP1介导,已知KAP1结合重复序列并有助于基因组稳定性[33,34].这些相互作用可能有助于MMR蛋白的抗突变和抗重组功能[35].
总之,通过确定MSH2/MSH6的基因组结合谱,我们的研究为MMR复合体的功能提供了新的视角,并揭示了通过识别ICRs的甲基化CpGs发挥的意想不到的作用,这些作用可能在维持这些调控区域的完整性和哺乳动物基因组印记的进化中起着至关重要的作用。
材料与方法
细胞系和培养条件
鼠标野生型,Zfp57- - - / - - -Msh2-/- E14 ESCs在标准无饲料条件下在糊化组织培养皿中培养,并在DMEM (Gibco, Thermo Fisher Scientific)中保存,添加100 uM 2-巯基乙醇(Sigma), 1 mM丙酮酸钠,2 mMl-谷氨酰胺、1 ×青霉素-链霉素、15%胎牛血清(HyClone)和103.U/ml白血病抑制因子(LIF)。野生型杂交种ESC系JB1,即(JF1 × C57BL/6) F1和JB1衍生系Zfp57 - / -ESC线已在前面描述[7,36].野生型和Zfp57-/- JB1 ESCs在标准无饲料条件下,使用ESGRO Complete™Plus无血清克隆级1i培养基(Merck Millipore),在3 μM Gsk3抑制剂CHIR99021的存在下,在糊化组织培养皿中培养。对H2O2曝光,0.5 mM H2O2立即用PBS稀释,然后加入培养基,30分钟后收集细胞。细胞在37℃、5% CO气氛下培养2.
克隆和转染
编码全长的cdnaZfp57而且Msh2基因克隆到含有AviTag序列的表达载体,在伸长因子-1 α (EF-1α)启动子。对于质粒转染,根据制造商的协议(赛默飞世尔科学公司),使用Lipofectamine LTX转染pEF1-BirA-V5_His和pef - avtagg - zfp57 /Msh2质粒。稳定的BirA转染细胞以0.25 mg/ml G418 (Life Technologies)选择,并维持在0.1 mg/ml G418。以模拟转染的表达bira的E14 ESCs为对照,进行MS分析和Bio-ChIP。用于克隆的引物列在附加文件中1:表S4。
蛋白质免疫沉淀分析
将两个10厘米培养皿中的细胞制成颗粒,并在NP40缓冲液(10mm Tris-HCl pH 7.4, 10mm NaCl, 3mm MgCl)中重悬20.5% NP-40)加冰,4°C孵育20分钟。分离的核在SDS裂解缓冲液(50 mM Tris-HCl pH 8.0, 10 mM EDTA和1% SDS)中在4℃下裂解10分钟。在95°C孵育10分钟后,核裂解物在冰上超声,并以最大速度离心10分钟。用30 μl蛋白A/G琼脂糖珠(SantaCruz)在4℃的旋转轮子上预先清除1 mg蛋白(KAP1、MSH6和IgG IP) 2 h。将抗kap1抗体(Abcam ab10483)、抗msh6抗体(Santa Cruz sc-137015)或小鼠IgG添加到预先清除的裂解液中,在4°C的转轮上孵育过夜。用50 μl protein A/G琼脂糖珠在4℃旋转沉淀1 h。对于生物素标记的zfp57和-MSH2免疫沉淀,1 mg蛋白与100 μl链霉霉素珠在4℃的旋转轮上孵育过夜。然后用500 μl RIPA缓冲液(10 mM Tris-HCl pH 8.0, 140 mM NaCl, 1 mM EDTA, 0.5 mM EGTA, 0.1%脱氧胆酸钠,1% Triton X-100和0.1% SDS,蛋白酶抑制剂(cOmplete, EDTA-free, Roche Life Science)洗涤5次。珠粒洗脱后进行质谱或Western Blot分析。高分辨率纳米lc串联质谱和质谱数据处理如先前报道[37].在模拟转染的表达bira的E14 ESCs中被链霉亲和素拉下并通过质谱鉴定的蛋白质(附加文件)1:表S1b)排除在进一步分析之外。LC-MS /MS识别的ZFP57交互器列表导入STRING数据库[18].聚类分析使用“kmean聚类”选项,“聚类数量”参数设置为4。集群之间的边缘设置为“虚线”。为了探索这些蛋白质参与的过程,我们使用gProfiler2 R package v 0.2.1的函数gost进行了GO:BP富集分析。38使用默认选项。我们选择了所有涉及DNA或RNA过程的术语,并进行了调整p-values < 0.05。所选术语使用GOplot包v 1.0.2绘制[39].
西方墨点法
通过95°C在Laemmli缓冲液中孵卵10分钟,用8%的丙烯酰胺凝胶从珠子中洗脱蛋白质。样品转移到PVDF膜上(Biorad Transblot)。在RT下,在5% w/v牛奶/TBST中阻塞1小时后,将膜与抗zfp57 (Abcam ab45341)、抗kap1 (Abcam ab10483)、抗msh6抗体(BD Biosciences,610,918)、抗MSH2 (Calbiochem, NA27)和抗肌动蛋白(Sigma-Aldrich, A2066)在4℃下孵育过夜。用TBST冲洗3次,用二抗孵育。使用ECL方法对信号进行可视化。
染色质免疫沉淀(ChIP)
生物素标记的msh2, MSH6, H3K9me3和H3K4me3结合的ChIP分析,在10 cm培养皿中生长的细胞分离的甲醛交联染色质上进行∼confluency 80%。简单地说,在37°C下,加入0.05%胰蛋白酶3分钟,分离细胞。加入甲醛约3 × 107在最终浓度为1%的磷酸盐缓冲盐水(PBS)中重悬,并在室温下摇晃培养10分钟。在最终浓度为0.125 m时加入甘氨酸,停止反应。细胞在冰冷的PBS中洗涤两次,离心并在裂解缓冲液1 (50 mM HEPES pH 8, 10 mM NaCl, 1 mM EDTA, 10%甘油,0.5% NP-40和0.25% Triton X-100)中重悬,在4°C下10分钟。分离的核在裂解缓冲液2 (10 mMTris-HCl pH 8.0, 200 mM NaCl, 1 mM EDTA和0.5 mM EGTA)中在4℃下裂解10分钟。使用S220聚焦超声仪(Covaris)在超声缓冲液(10 mM Tris-HCl pH 8.0, 100 mM NaCl, 1 mM EDTA, 0.5 mM EGTA, 0.1%脱氧胆酸钠和0.5% n -月桂酰肌氨酸)中剪切染色质,平均大小为100 - 400 bp。超声染色质在超声缓冲液中稀释。用30 μl蛋白A/G琼脂糖珠(SantaCruz)在4℃旋转轮上预处理MSH6、H3K9me3和H3K4me3 ChIP的染色质4 h。将链霉亲和素珠(Dynabeads MyOne Streptavidin T1, ref 65,601, Invitrogen)和抗msh6 (Santa Cruz sc-137015)、抗h3k9me3 (Abcam 8898)、抗h3k4me3 (Abcam ab8580)抗体或兔/小鼠IgG添加到预先清除的染色质中,在4°C的转轮上孵育过夜。MSH6、H3K9me3和H3K4me3 ChIP的染色质用30 μl蛋白A/G琼脂糖珠在4℃旋转沉淀4 h。珠子被洗五次500μl•瑞帕缓冲区(10毫米Tris-HCl pH值8.0,140毫米氯化钠,EDTA 1毫米,0.5毫米EGTA, 0.1%钠脱氧胆酸盐,1% Triton x - 100和0.1% SDS)一旦每个缓冲区:洗缓冲区(50 mM玫瑰,0.5%钠脱氧胆酸盐,1% Triton x - 100, 1毫米EDTA, 500毫米NaN3氯化钠和0.2%),氯化锂缓冲区(0.25 M氯化锂,NP-40 0.5%, 0.5%钠脱氧胆酸盐,1毫米EDTA和10 mMTris pH值8)和TE缓冲(10毫米三羟甲基氨基甲烷pH值8液,1毫米EDTA)。结合后的染色质用100 μl TE缓冲液洗脱。 Cross-links were reversed by incubation at 1 h at 37 °C with 1 μl RNAse cocktail (Ambion) and O/N at 60 °C after addition of 2.5 μl 20% SDS and 2.5 μl 20 mg/ml proteinase K (Sigma). The DNA was extracted by using the QIAquick Gel Extraction Kit (Qiagen). The immunoprecipitated or 1% input DNAs were analysed by real-time PCR using SBYR Green PCR Master Mix (Bio-Rad) on a CFXCONNECT Thermal Cycler (Bio-Rad). Each reaction was performed in biological duplicate with each duplicate averaged over at least two technical replicates. To evaluate significance, we applied an unpaired Student’s T-test. Asterisks indicate statistical significant differences in enrichment of DNA sequences with adjusted p-value: *p< 0.05, **p< 0.01, ***p< 0.001。引物在附加文件中列出1:表S4。
Allele-specific芯片
MSH2结合以及组蛋白H3K9me3和H3K4me3在甲基化和非甲基化等位基因上的富集Inpp5f而且玲娜在亚硫酸盐处理和pcr扩增免疫沉淀DNA直接测序后评估位点。MSH6结合在JF1和B6等位基因上Plagl1,Inpp5f是通过对存在于两个亲本基因组之间的snp进行免疫沉淀DNA分型来确定的。扩增产物测序(Eurofins Genomics),等位基因特异性dna之间的比例由电泳图确定。所有引物都列在附加文件中1:表S4。
门店分析
对于RRBS文库的制备,我们根据Illumina的说明使用了100-200 ng的基因组DNA。在IGA技术服务公司(意大利)使用NuGEN Ovation RRBS Methyl-Seq文库系统和NovaSeq6000 (Illumina, San Diego, CA)上的150 bp配对测序模式生成文库并进行测序。使用TrimGalore v. 0.6.6进行适配器修整和质量步骤后[https://github.com/FelixKrueger/TrimGalore使用RRBS模式(-rrbs),我们将读取与亩骶基因组(mm10)使用Bismark v.0.23.0默认参数[40].我们使用带有默认参数的ui -tools v.1.1.1删除了重复的读取41],然后使用Bismark甲基化萃取器得到所覆盖CpGs的甲基化水平。
对于ChIP-seq分析,使用来自免疫沉淀和输入染色质的两纳克DNA用于Illumina文库制备。在IGA技术服务公司(意大利)使用NuGen Ovation超低文库系统v2试剂盒生成文库并在Illumina NovaSeq6000平台(Illumina, San Diego, CA)上进行150 bp对端测序。在检查原始数据中没有质量差的碱基调用和适配器污染后,我们使用Bowtie2短读取校准器v2.3.5.1与默认参数[42].我们使用Picard markduplduplicate v2.22.9删除重复读取[http://broadinstitute.github.io/picard/]和多个映射读取,我们在其余的研究中只使用唯一映射的读取。
ZFP57和KAP1在E14 ESCs中的DNA结合谱来自GSE77744数据集。使用CrossMap Python脚本将坐标转换为mm10基因组[43].对于UCSC的可视化,我们使用了由BEDtools套件v2.292的GenomeCoverageBed工具生成的以每百万读取数(RPM)标准化的轨迹。44].为了定义富集区域,我们使用了带PE和-broad参数的MACS2算法[45].我们使用Bedtools相交函数进行峰值相交,并使用Bioconductor package ChIPseeker中nShuffle = 1000的richpeakoverlap函数评估其统计显著性。两个重复共享的MSH2峰分别为复制1的33%和复制2的62%。重叠(4444个峰)被认为具有统计学意义。(p= 0.0001)。我们使用ChIPseeker包注释了这些峰值[46TxDb.Mmusculus.UCSC.mm10。knownGene数据库[47].使用带有默认参数的plotAvgProf函数,我们绘制了TSS附近的峰值频率剖面。此外,对于特征注释,我们使用了带有annoDb = "org.Mm.eg.db"参数的plotAnnoPie函数。使用richpeakoverlap函数计算MSH2峰与启动子之间的重叠(TSS的−1000 + 0)的统计显著性,发现高度显著(p= 0.0001)。对于CpG岛屿和Rmsk区域的注释,我们将这些区域的坐标与从UCSC下载的这些区域的坐标相交。我们使用deepTools v3.4.3的computeMatrix和plotHeatmap功能绘制了与基因组区域相关的得分热图[48].原始和处理过的文件存放在GEO中,注册号为GSE205043。KAP1和ZFP57 ChIPseq的公开数据集来源于GEO数据库:GSE77444。
数据和材料的可用性
支持本研究结果的原始数据已以登录代码GSE205043保存在基因表达综合库中。
参考文献
Barlow DP, Bartolomei MS.哺乳动物基因组印迹。中国生物医学工程学报。2014;6:a018382。
Monk D, Mackay DJG, Eggermann T, Maher ER, Riccio A.基因组印迹障碍:基因组,表观基因组和环境相互作用的经验教训。《Nat Rev Genet》2019;20:235-48。
李霞,伊藤M,周峰,杨森N,左X, Leder P, Ferguson-Smith AC.一个母系-合子效应基因Zfp57同时保持母系和父系印记。开发细胞。2008;15:547-57。
Quenneville S, Verde G, Corsinotti A, Kapopoulou A, Jakobsson J, Offner S, Baglivo I, Pedone PV, Grimaldi G, Riccio A, Trono D.在胚胎干细胞中,ZFP57/KAP1识别甲基化的六核苷酸,影响印迹控制区的染色质和DNA甲基化。化学学报。2011;44:361-72。
Strogantsev R, Krueger F, Yamazawa K, Shi H, Gould P, Goldman-Roberts M, McEwen K, Sun B, Pedersen R, Ferguson-Smith AC. ZFP57在印迹和非印迹单等位基因表达表观遗传调控中的等位基因特异性结合。中国生物医学工程学报。2015;16:12 2。
Anvar Z, Cammisa M, Riso V, Baglivo I, Kukreja H, Sparago A, Girardot M, Lad S, De Feis I, Cerrato F, Angelini C, Feil R, Pedone PV, Grimaldi G, Riccio A. ZFP57识别多个和紧密间隔的序列基序变体,以维持小鼠胚胎干细胞的抑制表观遗传标记。核酸决议2016;44:1118-32。
Riso V, Cammisa M, Kukreja H, Anvar Z, Verde G, Sparago A, Acurzio B, Lad S, Lonardo E, Sankar A, Helin K, Feil R, Fico A, Angelini C, Grimaldi G, Riccio A. ZFP57在小鼠胚胎干细胞中维持印记基因的起源亲本特异性表达,并对非印记靶标产生差异影响。核酸决议2016;44:8165-78。
Shi H, Strogantsev R, Takahashi N, Kazachenka A, Lorincz MC, Hemberger M, Ferguson-Smith AC. ZFP57转座因子调控和基因表达的印迹域内外。表观遗传学染色质。2019;12:49。
蒋伟、史娟、赵娟、王强、丛东、陈峰、张艳、刘艳、赵娟、陈强、顾磊、周伟、王超、方志、耿生、谢伟、陈丽兰、杨艳、白艳、林红、李霞。ZFP57决定了目标印迹基因的等位基因表达开关。美国国家科学研究院。2021; 118: e2005377118。
Acurzio B, Verma A, Polito A, Giaccari C, Cecere F, Fioriniello S, Della Ragione F, Fico A, Cerrato F, Angelini C, Feil R, Riccio A. Zfp57失活说明了ICR甲基化在小鼠ESCs神经分化过程中印迹基因表达中的作用。科学通报2021;11:13802。
Mackay DJ, Callaway JL, Marks SM, White HE, Acerini CL, Boonen SE, Dayanikli P, Firth HV, Goodship JA, Haemers AP, Hahnemann JM, Kordonouri O, Masoud AF, Oestergaard E, Storr J, Ellard S, Hattersley AT, Robinson DO, Temple IK。短暂性新生儿糖尿病个体中多个印迹位点的低甲基化与ZFP57突变相关。Nat Genet, 2008; 40:949-51。
霍奇金森,埃尔-沃克,变异在哺乳动物基因组突变率。Nat Rev Genet. 2011; 12:756-66。
金,杨晓明,等。peg - 3与H19-ICR的进化模式。基因组学。2019;111:1713-9。
Takahashi N, Coluccio A, Thorball CW, Planet E, Shi H, Offner S, Turelli P, Imbeault M, Ferguson-Smith AC, Trono D. ZNF445是基因组印迹的主要调控因子。基因开发2019;33:49-54。
方辉,朱旭,杨辉,吴杰,Barbour JA,王俊华。复制无关的DNA错配修复缺陷导致癌症中5-甲基胞嘧啶脱氨突变特征。科学通报2021;7:eabg4398。
王娟,王晓明,王晓明,王晓明。小鼠胚胎干细胞中蛋白质-蛋白质和蛋白质- dna相互作用的研究。Nat protocol . 2009; 4:506-17。
左X, Sheng J, Lau HT, McDonald CM, Andrade M, Cullen DE, Bell FT, Iacovino M, Kyba M, Xu G, Li X。锌指蛋白ZFP57通过其转录抑制域招募DNA甲基转移酶并维持胚胎干细胞DNA甲基化印记。中国生物化学杂志,2012;
Jensen LJ, Kuhn M, Stark M, Chaffron S, Creevey C, Muller J, Doerks T, Julien P, Roth A, Simonovic M, Bork P, von Mering C. STRING 8 - 630种生物中蛋白质及其功能相互作用的全球观点。核酸研究,2009;37:D412-6。
Krepelova A, Neri F, Maldotti M, Rapelli S, Oliviero S. Myc和max全基因组结合位点分析将Myc调控网络与小鼠胚胎干细胞的polycomb和核心多能网络联系起来。PLoS ONE。2014; 9: e88933。
Ding N, Bonham EM, Hannon BE, Amick TR, Baylin SB, O 'Hagan HM。错配修复蛋白将DNA甲基转移酶1招募到DNA氧化损伤部位。中国生物医学杂志,2016;
张sm, Kauzlaric A, Quivy JP, Pontis J, Rauwel B, Coluccio A, Offner S, Duc J, Turelli P, Almouzni G, Trono D. KAP1促进DNA复制后异染色质的恢复。核酸res 2018; 46:8788-802。
Hoffmeister H, Fuchs A, Erdel F, Pinz S, Gröbner-Ferreira R, Bruckmann A, Deutzmann R, Schwartz U, Maldonado R, Huber C, Dendorfer AS, Rippe K, Längst g CHD3和CHD4形成不同的NuRD复合体,功能不同但重叠。核酸研究,2017;45:10534-54。
杨思敏,金宝杰,李国强,李国强。H1连接蛋白组蛋白通过调节DNA甲基化和组蛋白H3甲基化促进表观遗传沉默。美国国家科学研究院。2013; 110:1708-13。
核RNA在染色质结构和转录调控中的作用。中国生物医学杂志,2019;58:120-5。
格林I,伊什琴科AA。活性DNA去甲基化中碱基切除修复和错配修复途径的相互作用。核酸决议2016;44:3713-27。
张志刚,张志刚,张志刚,等。基因组印迹中染色质组织与基因活性的关系。Curr Opin Genet Dev. 2020; 61:17-24。
王振华,王振华。胞嘧啶甲基化与DNA复制和修复过程之间的相互作用形成了癌症基因组的突变景观。核酸研究,2017;45:7786-95。
魏凯,郭晓明,王晓明,等。人类DNA错配修复基因缺陷的小鼠模型。医学趋势,2002;8:346-53。
de Wind N, Dekker M, Berns A, Radman M, te Riele H.小鼠Msh2基因失活导致错配修复缺陷、甲基化耐受、高重组和癌症易感性。细胞。1995;82:321-30。
Reitmair AH, Schmits R, Ewel A, Bapat B, Redston M, Mitri A, Waterhouse P, Mittrücker HW, Wakeham A, Liu B,等。缺乏MSH2的小鼠能够存活并易患淋巴肿瘤。Nat Genet, 1995; 11:64-70。
李国强,李志刚。真核DNA错配修复。《现代科学》1999;9:89-96。
黄艳,顾玲,李桂梅。h3k36me3介导的错配修复优先保护活性转录基因的突变。中国生物化学杂志,2018;
Rowe HM, Jakobsson J, Mesnard D, Rougemont J, Reynard S, Aktas T, Maillard PV, Layard-Liesching H, Verp S, Marquis J, Spitz F, Constam DB, Trono D. KAP1控制胚胎干细胞中的内源性逆转录病毒。大自然。2010;463:237-40。
Ecco G, Cassano M, Kauzlaric A, Duc J, Coluccio A, Offner S, Imbeault M, Rowe HM, Turelli P, Trono D.转座元件及其KRAB-ZFP控制器调节成人组织中的基因表达。Dev Cell. 2016; 36:611-23。
Harfe BD, Jinks-Robertson S. DNA错配修复与遗传不稳定性。《热内年鉴》2000;34:359-99。
Kota SK, Llères D, Bouschet T, Hirasawa R, Marchand A, Begon-Pescia C, Sanli I, Arnaud P, Journot L, Girardot M, Feil R ICR非编码RNA表达控制Dlk1-Dio3域的印迹和DNA复制。Dev Cell. 2014; 31:19-33。
Marino MM, Rega C, Russo R, Valletta M, Gentile MT, Esposito S, Baglivo I, De Feis I, Angelini C, Xiao T, Felsenfeld G, Chambery A, Pedone PV。交互组映射将BRG1定义为SWI/SNF染色质重塑复合体的一个组分,作为转录调节因子CTCF的新伙伴。中国生物化学杂志,2019;29(4):861 - 73。
Kolberg L, Raudvere U, Kuzmin I, Vilo J, Peterson H. gprofiler2 -一个R包的基因列表功能富集分析和命名空间转换工具集g:Profiler。F1000Res。2020; 9:灵丹妙药- 709。
Walter W, Sánchez-Cabo F, Ricote M. GOplot:一个将表达式数据与功能分析可视化结合的R包。生物信息学。2015;31:2912-4。
Krueger F, Andrews SR. Bismark:用于亚硫酸氢盐- seq应用的灵活对准器和甲基化调用器。生物信息学。2011;27:1571-2。
Smith T, Heger A, Sudbery I. umi -工具:唯一分子标识符的建模测序误差以提高定量精度。基因组决议2017;27:491-9。
朗米德B,萨尔茨堡SL。快速间隙读取对齐领结2。Nat Methods. 2012; 9:357-9。
赵慧,孙志强,王娟,黄慧,Kocher JP,王磊。一种基于交叉图谱的基因组坐标转换工具。生物信息学。2014;30:1006-7。
昆兰AR,霍尔IM。BEDTools:用于比较基因组特征的一套灵活的实用程序。生物信息学。2010;26:841-2。
张勇,刘涛,Meyer CA, Eeckhoute J, Johnson DS, Bernstein BE, Nusbaum C, Myers RM, Brown M, Li W,刘xs。基于模型的ChIP-Seq (MACS)分析。基因组生物学,2008;9:1-9。
于刚,王乐林,何启勇。ChIPseeker:用于ChIP峰值注释、比较和可视化的R/Bioconductor包。生物信息学。2015;31:2382-3。
Bioconductor核心团队和Bioconductor包维护者TxDb.Mmusculus.UCSC。mm10。knownGene: TxDb对象的注释包。R包版本为3.10.0。2019.
Ramírez F, Ryan DP, Grüning B, Bhardwaj V, Kilpert F, Richter AS, Heyne S, Dündar F, Manke T. deepTools2:用于深度测序数据分析的下一代web服务器。核酸研究,2016;44:W160-5。
确认
我们感谢Salvatore Oliviero提供的AviTag载体和Bio-ChIP-seq协议。
资金
这项工作得到了以下赠款的支持:意大利Ricerca sul Cancro IG协会2020年第24405号,“坎帕尼亚地区肿瘤研究项目”赠款:I-Cure,“ateneo竞争项目V:ALERE (VAnviteLli per la Ricerca) 2019 - Università degli Studi della Campania“Luigi VAnviteLli”赠款MIRIAM。
作者信息
作者及隶属关系
贡献
概念化、AR和BA;方法:BA、FC、CG、AV、RR、MV;验证、FC、CG和BHM;形式化分析、BA和FC;数据管理、BA和FC;写作、审稿、编辑、BA、FC、AR;监督、CA、AC、AR;资助获取,AR。所有作者阅读并批准最终手稿。
相应的作者
道德声明
发表同意书
所有参与本研究的参与者均同意发表临床和分子数据。
相互竞争的利益
作者声明,他们没有潜在的竞争利益需要披露。作者声明,他们没有与本文相关的财务关系需要披露。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:
表S1。用ZFP57-AviTag提取E14 ESCs中高置信蛋白,并通过纳米lc - MS/MS鉴定。表印地。E14 BirA(对照)和E14 BirA+ZFP57_Avitag(样本)中链霉亲和素拉下的共享蛋白列表。表S2a.Bio-ChIP-seq在E14 ESCs中鉴定出MSH2峰。复制1。表开通。Bio-ChIP-seq在E14 ESCs中鉴定出MSH2峰。复制2。表S3。小鼠ESCs中印迹的gDMRs坐标*。表S4。引物序列。
附加文件2:
图S1。zfp57相互作用蛋白的STRING网络模型。60个候选zfp57相互作用蛋白中的41个被映射到通过STRING分析构建的互连网络上。该模型揭示了连接到ZFP57和KAP1(TIF1B)的关键子网络集群。基于K-means聚类,颜色代表不同的子网络。边缘厚度代表了基于数据库挖掘、实验证据和文本挖掘的交互置信度。图S2。热图显示,绝大多数与启动子重叠的MSH2 Bio-ChIP-seq峰集中在转录起始位点(TSS)上。S3无花果。.UCSC基因组浏览器的截图显示,在表达bira的E14 ESCs中,沿8个启动子重叠CpGI的细胞生长控制基因检测到生物素标记的MSH2的ChIP-seq信号。MSH2(2个重复)、ZFP57和KAP1的DNA甲基化和结合谱如图3a所示。无花果。S4.(与图2f相关)热图显示MSH2、MSH6、ZFP57和KAP1在与KAP1 ChIP-seq峰重叠(+/- 1.5 kbp)的基因组区域的reads富集,KAP1 ChIP-seq峰根据它们与各种基因组元件的重叠进行排序。图S5。(与图2f相关)热图显示MSH2、MSH6、ZFP57和KAP1在基因组区重叠(+/- 1.5 kbp), KAP1 ChIP-seq峰根据它们与ZFP57峰的重叠进行排序。无花果S6。.UCSC基因组浏览器的截图显示,在表达bira的E14 ESCs中,沿ZFP57[7]结合的5个noICR区域检测到生物素标记的MSH2的ChIP-seq信号。MSH2(2个重复)、ZFP57和KAP1的DNA甲基化和结合谱如图3a所示。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
阿库尔齐奥,B.,西塞尔,F.,嘉卡里,C.。et al。错配修复蛋白MSH2和MSH6通过ZFP57-KAP1复合物与印迹控制区相互作用。表观遗传学与染色质15, 27(2022)。https://doi.org/10.1186/s13072-022-00462-7
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s13072-022-00462-7
关键字
- 质/女士
- Chip-seq
- Allele-specific分析
- CpG岛
- 甲基CpG
- 基因组印记
- 错配修复
- 转录因子结合
- 胞嘧啶脱氨基作用