- 研究gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
白细胞特异性DNA甲基化生物标志物及其对病理表观遗传学分析的意义gydF4y2Ba
表观遗传学的通信gydF4y2Ba体积gydF4y2Ba2gydF4y2Ba,文章号:gydF4y2Ba5gydF4y2Ba(gydF4y2Ba2022gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
不同的细胞类型可以通过它们的DNA甲基化模式来识别。过去十年的许多研究都集中在癌症中的DNA甲基化变化,或者在创伤或器官移植病例中使用血浆中的无细胞循环DNA来识别受损组织。然而,关于白细胞和其他组织之间的差异甲基化模式以及它们如何在一系列情况下用作免疫活动的检测工具的研究很少。gydF4y2Ba
结果gydF4y2Ba
我们已经确定了几个在白细胞中完全甲基化的位点,但在其他一系列中胚层、外胚层和内胚层来源的组织中几乎没有甲基化。我们对唾液进行亚硫酸氢酶扩增子测序,并在体外将外周血单个核细胞和肠类器官细胞按一定比例混合,验证了这些生物标志物。有趣的是,这些甲基化生物标志物之前已被确定在各种炎症性疾病中发生改变,包括阿尔茨海默病、炎症性肠病和牛皮癣。我们假设这是由于白细胞浸润,而不是病变细胞本身的特征。此外,在六种癌症类型中,我们发现浸润的白细胞与HOXA3位点的DNA甲基化水平呈正线性关系,表明进一步的免疫细胞浸润。gydF4y2Ba
结论gydF4y2Ba
我们的数据强调了在进行DNA甲基化分析时考虑细胞组成的重要性,并证明了开发新的诊断测试来检测炎症和免疫细胞浸润的可行性。gydF4y2Ba
简介gydF4y2Ba
许多表观遗传过程被用来调节基因表达;然而,DNA甲基化是独特的,因为它可以在很长一段时间内传递生物记忆。在脊椎动物中,大多数DNA甲基化是在胞嘧啶上发现的,特别是在胞嘧啶-鸟嘌呤(CpG)二核苷酸上。CpG是一个复文序列,因此,甲基转移酶,如DNA甲基转移酶1 (DNMT1),可以在DNA复制后通过将甲基化从模板链上的胞嘧啶复制到新合成链上的互补胞嘧啶来保持DNA甲基化标记[gydF4y2Ba1gydF4y2Ba].因为体内所有细胞本质上都具有相同的DNA序列,细胞形态和功能与表达或抑制的特定基因组合有关。DNA甲基化有助于调节基因表达(例如,通过阻止转录因子结合启动子和增强子[gydF4y2Ba2gydF4y2Ba]或通过甲基结合结构域募集异染色质相关蛋白[gydF4y2Ba3.gydF4y2Ba]),因此,DNA甲基化模式有助于定义细胞身份。因此,全基因组甲基化分析的层次聚类表明,密切相关的细胞类型聚集在一起[gydF4y2Ba4gydF4y2Ba],许多不同的甲基化区域仍保留着早期发育细胞鉴定的决定[gydF4y2Ba5gydF4y2Ba].gydF4y2Ba
独特的细胞类型特定的DNA甲基化模式也可以作为生物标志物,在法医和诊断环境中识别未知的细胞类型。例如,从凋亡和坏死细胞释放到血液中的无细胞DNA (cfDNA)可以通过非侵入性血液采样收集。分析cfDNA的癌症特异性DNA甲基化模式可用于检测、监测和预测癌症[gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba].cfDNA甲基化也被用于追踪创伤性损伤或器官移植后的细胞死亡[gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba],血液样本中的甲基化分析已用于量化白细胞亚群[gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba],并应用于癌症甲基组筛选[gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba].gydF4y2Ba
虽然这些组织类型的反卷积研究本身就很强大和有用,但它们依赖于阵列技术(例如Infinium HumanMethylation450 BeadChip),从单个样本中同时分析数千个CpG位点。相比之下,相对简单的诊断测试,例如那些旨在检测炎症和免疫细胞浸润的测试,只需要将血液来源的细胞(如白细胞)与其他组织类型区分开来。因此,可能不需要从数千个位点进行复杂的反褶积[gydF4y2Ba4gydF4y2Ba].gydF4y2Ba
在这项研究中,我们首先旨在识别能够区分白细胞和所有其他细胞类型的基因组区域,并使用快速、高通量亚硫酸氢盐pcr检测来验证它们(图2)。gydF4y2Ba1gydF4y2Ba).其次,我们的目标是对照之前发表的数据集对这些位点进行基准测试,以评估甲基化疾病研究中白细胞特异性甲基化模式的报告情况。我们发现了两个区域gydF4y2BaHOXA3gydF4y2Ba和gydF4y2BaMAP4K1gydF4y2Ba在血液来源细胞中高度甲基化但在所有其他组织中未甲基化的基因座。在使用唾液和人工制造的DNA混合物进行验证后,我们发现这些位点适用于定量异质组织中白细胞的比例。gydF4y2Ba
研究方法流程图。生物标志物的发现由Moss等人完成。[gydF4y2Ba10gydF4y2Ba]和Reinius等人。[gydF4y2Ba11gydF4y2Ba)数据集。个体CpG引用有差异gydF4y2BaβgydF4y2Ba白细胞与所有组织之间的-值≥0.8被识别并称为ls-DMPs。CpG岛外的ls-DMPs被过滤掉。我们通过(1)定义的PBMC和肠道类器官DNA的体外混合以及(2)同种唾液样本验证了每个生物标志物。为了评估文献中如何报道ls-DMPs,我们收集了一系列检查炎症疾病甲基化的数据集,并确定了报道的ls-DMPs的数量。最后,为了将我们的ls-DMP与当前的反褶积方法进行比较,我们使用TCGA泛癌症图谱来评估每个样本中ls-DMP甲基化与白细胞数量之间的相关性gydF4y2Ba
结果gydF4y2Ba
生物标志物的发现gydF4y2Ba
为了充分识别候选的白细胞特异性DNA甲基化生物标志物,我们利用了Moss等人的研究。gydF4y2Ba10gydF4y2Ba甲基化图谱(GEO编号:GSE122126),其中Illumina Infinium HumanMethylation450 BeadChip阵列数据用于血细胞制备,以及另外9种不同的组织类型,这些组织类型据称不含血管和免疫种群。对于Moss数据集中的每个CpG位点(共423,213个),我们计算了组合的平均DNA甲基化水平(表示为agydF4y2BaβgydF4y2Ba- 0到1之间的值),并将其与白细胞的DNA甲基化水平进行比较。两种方法之间的差异被用于告知潜在生物标志物的选择;差异≥0.8的CpG位点被认为是不同甲基化位点(今后,我们将其称为白细胞特异性差异甲基化位点,或ls-DMPs)。共鉴定出77个ls-DMPs(表SgydF4y2Ba1gydF4y2Ba).其中19例在白细胞中高度甲基化(甲基化> 89%),58例未甲基化(甲基化< 8%)。大多数高度甲基化的ls-DMPs(19个中的15个)被发现在CpG岛(cgi)和基因体内(19个中的12个),而大多数未甲基化的位点位于公海(58个中的45个)(图S)gydF4y2Ba1gydF4y2Ba模拟)。cgi定位的候选生物标记物特别有趣,因为它们可能被额外的cpg包围,提供类似的鉴别能力。随后,我们只关注位于cgi内的CpG位点(表gydF4y2Ba1gydF4y2Ba).gydF4y2Ba
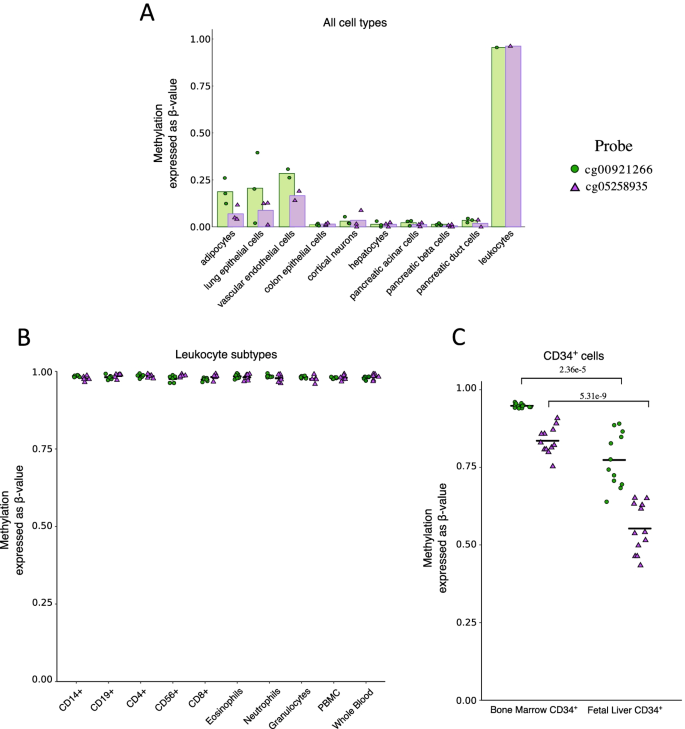
我们决定集中于仅有的两个包含多个ls- dmp的cgi。第一次是在gydF4y2BaHOXA3gydF4y2Ba基因座(chr7:27,153,187-27,153,647, hg19),含有2个ls-DMPs (cg00921266和cg08101036)。该区域也显示出强烈的进化保守性-亚硫酸氢盐测序在同源小鼠区域显示出类似的甲基化模式(数据由Hon等人发表。[gydF4y2Ba5gydF4y2Ba];图年代gydF4y2Ba2gydF4y2Ba).第二个感兴趣的区域是在CGI的26号外显子中gydF4y2BaMAP4K1gydF4y2Ba(chr19:39,086,878-39,087,304, hg19),还含有两个ls-DMPs, cg05258935和cg02798280。各基因座(cg00921266和cg05258935)的最高CpGs分布相似;白细胞DNA甲基化水平非常高;脂肪细胞、肺上皮细胞和血管内皮细胞中甲基化水平中等偏低;结肠上皮、皮层神经元、肝细胞和胰腺各种类型的细胞中几乎没有DNA甲基化(图2)。gydF4y2Ba2gydF4y2Ba一个)。gydF4y2Ba
识别标志。gydF4y2Ba一个gydF4y2Ba甲基化的柱状图gydF4y2BaβgydF4y2BaMoss等人的10个分离细胞群中cg00921266 (HOXA3,绿色)和cg0528935 (MAP4K1,紫色)的-value [gydF4y2Ba10gydF4y2Ba]数据集(GEO接入:GSE122126)。gydF4y2BabgydF4y2Ba甲基化的点图是agydF4y2BaβgydF4y2BaReinius等人在10种不同免疫细胞群中对cg00921266 (HOXA3,绿色)和cg0528935 (MAP4K1,紫色)的-value进行了研究。[gydF4y2Ba11gydF4y2Ba数据集(GEO接入:GSE35069)。gydF4y2BacgydF4y2Ba甲基化的点图是agydF4y2BaβgydF4y2BaCD34中cg00921266 (HOXA3,绿色)和cg0528935 (MAP4K1,紫色)的-value。gydF4y2Ba+gydF4y2Ba来自成人骨髓和胎儿肝脏的造血细胞gydF4y2Ba
为了进一步探索这些生物标志物的生物学基础,我们使用先前发表的基于阵列的数据集检测了白细胞亚群甲基化[gydF4y2Ba11gydF4y2Ba,gydF4y2Ba15gydF4y2Ba].这些数据表明,所有成年白细胞亚群在cg05258935和cg00921266位点都高度甲基化。gydF4y2Ba2gydF4y2Bab),尽管CD34的甲基化略有减少gydF4y2Ba+gydF4y2Ba成人骨髓干细胞(图;gydF4y2Ba2gydF4y2Bac).相比之下,胎儿肝脏CD34gydF4y2Ba+gydF4y2Ba与成人CD34相比,细胞表现出明显的去甲基化gydF4y2Ba+gydF4y2Ba(cg00921266gydF4y2BapgydF4y2Bae = 2.3gydF4y2Ba−5gydF4y2Ba;cg05258935,gydF4y2BapgydF4y2Bae = 5.3gydF4y2Ba−9gydF4y2Ba)(图。gydF4y2Ba2gydF4y2Bac).总之,这些数据表明ls-DMPs在gydF4y2BaHOXA3gydF4y2Ba和gydF4y2BaMAP4K1gydF4y2Ba在未修饰的状态下开始发育,在造血谱系的胎儿干细胞中积累甲基化,并在随后的分化、发育和衰老过程中保持这一状态。gydF4y2Ba
使用DNA甲基化准确预测细胞起源身份gydF4y2Ba
虽然识别这些生物标志物的数据集无疑是有价值的,但由于几个原因,阵列技术作为诊断平台是有限的。首先,在每个样本的基础上,数组是相对昂贵的。其次,他们依靠单个CpG探针来检测给定位点的甲基化,这可能会导致给定生物标志物区域的甲基化采样不足。此外,阵列技术无法在单分子水平上提供信息。为了解决这一问题,我们决定采用一种更具成本效益的扩增子-亚硫酸氢盐测序测试,该测试利用了密切相关的cpg通常具有相同的DNA甲基化状态(即甲基化或非甲基化)。这是因为通过增加同时分析的CpG位点的数量,我们提高了甲基化生物标志物的分辨率和鉴别能力[gydF4y2Ba9gydF4y2Ba].gydF4y2Ba
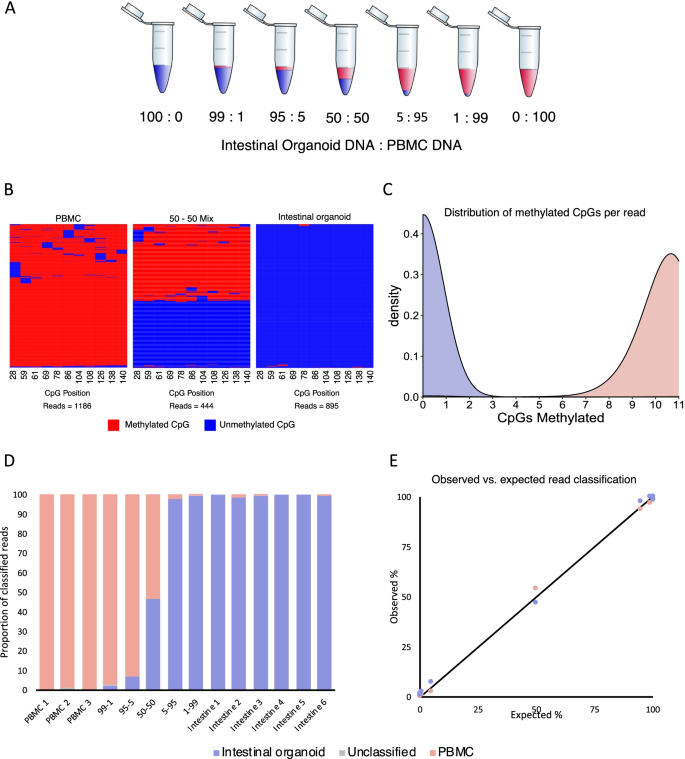
来量化甲基化gydF4y2BaHOXA3gydF4y2Ba和gydF4y2BaMAP4KgydF4y2Ba我们采用了基于双索引、四引物pcr的检测方法[gydF4y2Ba16gydF4y2Ba](请参阅“gydF4y2Ba方法gydF4y2Ba部分进行深入描述)。首先,我们的目标是验证每个感兴趣的区域,并评估改编的扩增子-亚硫酸氢盐测序试验准确测量DNA甲基化水平的能力。具体来说,我们想确定(a)是否存在对甲基化或非甲基化链的扩增偏倚以及(b)该测定方法识别少数细胞类型的能力。为了做到这一点,我们从外周血单个核细胞(pmcs)中提取纯化的DNA,因为这些细胞代表血液中有核的细胞部分,不应该含有大量来自其他组织的血清来源的cfDNA。为了从没有任何白细胞或血液来源的细胞中制备DNA,我们提取了培养的肠道类器官DNA。肠道类器官在支持Lrg5生长的精确三维培养条件下生长gydF4y2Ba+gydF4y2Ba干细胞及其衍生物[gydF4y2Ba17gydF4y2Ba],以免污染血液和血管。然后,我们将每个来源的纯化DNA以7个精确量混合(图2)。gydF4y2Ba3.gydF4y2Baa)并对每种混合物进行亚硫酸氢盐转化和PCR检测。gydF4y2Ba
基于HOXA3 DNA甲基化的体外混合PBMC和肠类器官DNA的反卷积。gydF4y2Ba一个gydF4y2Ba肠道类器官与PBMC DNA的比例用于体外混合。gydF4y2BabgydF4y2Bapbmc(左)、50:50混合(中)和肠类器官(右)HOXA3扩增子甲基化热图输出示例。每一行代表一个单独的读取,每列是扩增子内的一个CpG位点;列名表示CpG在读取中的位置。gydF4y2BacgydF4y2Ba纯肠类器官(蓝色)和纯PBMC(红色)样本中每次读取的甲基化CpG位点数量的密度图。的gydF4y2BaygydF4y2Ba-轴表示单位上的概率gydF4y2BaxgydF4y2Ba-轴,使特定区间的曲线下面积等于该区间内甲基化cpg数量的概率。带宽为0.45。gydF4y2BadgydF4y2Ba堆叠柱状图的比例分类为每个样本类型。读数分为肠类器官(蓝色)、PBMC(红色)和未分类(灰色)。gydF4y2BaegydF4y2Ba肠类器官和PBMC观察读数分类与预期读数分类的散点图gydF4y2Ba
纯肠道类器官DNA的平均总DNA甲基化为0.83%(标准差(sd) = 0.69%), 1.02% (sd = 0.65%)gydF4y2BaHOXA3gydF4y2Ba和gydF4y2BaMAP4K1gydF4y2Ba,分别。相比之下,纯PBMC DNA的平均总DNA甲基化率为94.0% (sd = 0.30%)和93.1% (sd = 0.21%)gydF4y2BaHOXA3gydF4y2Ba和gydF4y2BaMAP4K1gydF4y2Ba,分别。我们使用热图可视化了单个读取的DNA甲基化,其中读取贯穿一行,每一列表示单个CpG甲基化状态(图2)。gydF4y2Ba3.gydF4y2Bab,图SgydF4y2Ba3.gydF4y2Baa、图SgydF4y2Ba4gydF4y2Ba,图SgydF4y2Ba5gydF4y2Ba).为gydF4y2BaHOXA3gydF4y2Ba,在肠道类器官和PBMC样品中,只有一小部分reads分别完全甲基化(0.37%)或完全去甲基化(0.69%);有趣的是,最小读数包含适度的DNA甲基化。gydF4y2Ba
我们使用了二项逻辑回归模型(图SgydF4y2Ba6gydF4y2Baa、图SgydF4y2Ba6gydF4y2BaC)生成每次读取的甲基化胞嘧啶数的密度图gydF4y2BaHOXA3gydF4y2Ba和gydF4y2BaMAP4K1gydF4y2Ba位点(图gydF4y2Ba3.gydF4y2Bac、图SgydF4y2Ba3.gydF4y2Bab).两种细胞类型在两个位点上彼此分开聚集。我们试图使用混合样本的DNA甲基化模式对其进行分类。为此,我们构建了受试者工作特征(ROC)曲线,并使用R中的“pROC”包确定了最佳分类阈值(图SgydF4y2Ba6gydF4y2Bab和SgydF4y2Ba6gydF4y2Bad).该模型确定gydF4y2BaHOXA3gydF4y2Ba如果11个CpGs中有≤3个被甲基化,或者有≥4个被甲基化为PBMC衍生的扩增子(TPR = 0.993, FPR = 0.004),则应归类为肠类器官衍生扩增子。为gydF4y2BaMAP4K1gydF4y2Ba如果8个CpGs中有≤2个被甲基化,则该扩增子被分类为肠类器官来源,而≥3个被分类为PBMC来源(TPR = 0.999, FPR = 6.37e−05)。然而,由于每种细胞类型的DNA甲基化水平实际上是相反的,并且最小的reads包含中等水平的DNA甲基化,我们决定对每个扩增子使用更严格的分类系统。这是一个DNA片段gydF4y2BaHOXA3gydF4y2Ba如果扩增子上有≤3个甲基化CpGs,则被分类为肠类器官衍生扩增子;如果扩增子上有≥6个甲基化CpGs,则被分类为PBMC扩增子,两者之间的任何读数均未分类。同样,对于gydF4y2BaMAP4K1gydF4y2Ba在扩增子中,如果≤2个CpGs被甲基化,则该读数被分类为肠类器官来源,如果≥6个CpGs被甲基化,则该读数被分类为PBMC来源,任何介于两者之间的读数均未分类。gydF4y2Ba
我们将分类系统应用于每个混合样本,两个位点都显示出与每种细胞类型输入DNA的数量显著相关(gydF4y2BaHOXA3gydF4y2Ba:gydF4y2BaRgydF4y2Ba2gydF4y2Ba= 0.999,gydF4y2BaMAP4K1gydF4y2Ba:gydF4y2BaRgydF4y2Ba2gydF4y2Ba= 0.9985)。gydF4y2Ba3.gydF4y2Bac, d,图SgydF4y2Ba3.gydF4y2Bac - d)。我们的结果还表明,我们使用的亚硫酸氢盐PCR引物对高甲基化或非甲基化的reads没有任何显著的偏差,并且是高度准确的。gydF4y2Ba
唾液白细胞可与口腔上皮细胞分离gydF4y2Ba
为了进一步验证这些生物标志物与异质,未培养的组织样本,我们从人类捐赠者的唾液。我们选择唾液是因为收集唾液是非侵入性的,唾液中含有大量的口腔牙龈分泌的白细胞,以及从颊上皮脱落的非白细胞颊细胞[gydF4y2Ba18gydF4y2Ba].与口腔白细胞相比,颊部细胞大而扁平,因此使用标准组织学技术很容易识别[gydF4y2Ba18gydF4y2Ba].此外,确定唾液白细胞和颊上皮DNA甲基化的比例可能对研究人员使用唾液样本进行表观遗传学流行病学研究很有价值[gydF4y2Ba19gydF4y2Ba].gydF4y2Ba
为了确认从白细胞中提取的DNA是甲基化的,而口腔细胞是未甲基化的,我们使用细胞过滤和流式细胞术从唾液样本中纯化各自的细胞群。纯化细胞群的组织学检查显示,预测的平均纯度为97.1%的颊细胞和99.4%的白细胞(图2)。gydF4y2Ba4gydF4y2Baa, b).在双指标亚硫酸氢盐PCR检测后gydF4y2BaHOXA3gydF4y2Ba和gydF4y2BaMAP4K1gydF4y2Ba在位点上,绝大多数reads具有预期的甲基化模式;然而,在已排序的白细胞群体中,有一小部分reads亚群具有很大程度的非甲基化特征(2.7%的readsgydF4y2BaHOXA3gydF4y2Ba;2.8%gydF4y2BaMAP4K1gydF4y2Ba),反之亦然(2.3%的读入gydF4y2BaHOXA3gydF4y2Ba;2.6%gydF4y2BaMAP4K1gydF4y2Ba)(图。gydF4y2Ba4gydF4y2Bad,图SgydF4y2Ba7gydF4y2Baa、图SgydF4y2Ba8gydF4y2Ba,图SgydF4y2Ba9gydF4y2Ba).我们怀疑这些差异是由于手工细胞计数中观察到的细胞分离不完善造成的。我们使用分离的口腔和白细胞细胞群进行了相同的二项回归分析,以产生甲基化密度图(图2)。gydF4y2Ba4gydF4y2Bae,图SgydF4y2Ba7gydF4y2Bab,图SgydF4y2Ba10gydF4y2Ba).在这种情况下,两种细胞类型的分类界限不同于PBMC和肠类器官细胞。为gydF4y2BaHOXA3gydF4y2Ba如果5个或更少的CpG位点被甲基化,则扩增子的截止点设置为口腔上皮,如果6个或更多的CpG位点被甲基化,则截止点设置为唾液白细胞(TPR = 0.969, FPR = 0.023)。为gydF4y2BaMAP4K1gydF4y2Ba如果3个或更少的CpGs被甲基化,则被分类为颊上皮细胞,如果4个或更多的CpGs被甲基化,则被分类为唾液白细胞(TPR = 0.969, FPR = 0.025)。gydF4y2Ba
唾液细胞成分的反褶积。gydF4y2Ba一个gydF4y2Ba- - - - - -gydF4y2BacgydF4y2Ba光镜下H&E染色图像gydF4y2Ba一个gydF4y2Ba唾液白细胞(唾液白细胞标记为i),gydF4y2BabgydF4y2Ba颊上皮细胞和gydF4y2BacgydF4y2Ba混合唾液(单个核细胞标记为ii,多态核细胞标记为iii)。gydF4y2BadgydF4y2Ba分离的唾液白细胞和口腔上皮的甲基化热图示例。每一行代表一个单独的读取,每列是扩增子内的一个CpG位点;列名表示CpG在读取中的位置。gydF4y2BaegydF4y2Ba密度图(如图。gydF4y2Ba3.gydF4y2Bac)为分离的口腔上皮(蓝色)和唾液白细胞(红色)样本中每次读取的甲基化CpG位点数量(带宽= 0.45)。gydF4y2BafgydF4y2Ba甲基化热图(如图所示)gydF4y2Ba3.gydF4y2BaB)用于三份混合唾液样本。gydF4y2BaggydF4y2Ba甲基化分析中观察到的读分类与唾液白细胞比例显微测量中预期读分类的散点图gydF4y2Ba
已经证实了甲基化之间的关系gydF4y2BaHOXA3gydF4y2Ba和gydF4y2BaMAPK4gydF4y2Ba在口腔白细胞和颊细胞中,我们试图量化它们在混合唾液样本中的比例。异质唾液样本中细胞的相对比例最初通过组织学检查进行评估,在100个细胞样本中,38%为唾液白细胞,62%为颊上皮(图2)。gydF4y2Ba4gydF4y2Baa).亚硫酸盐转化和PCR检测(图。gydF4y2Ba4gydF4y2Baf,图SgydF4y2Ba7gydF4y2Ba),我们发现预测的白细胞数为38.1% (sd = 2.5)和39.3% (sd = 1.5)gydF4y2BaHOXA3gydF4y2Ba和gydF4y2BaMAP4K1gydF4y2Ba,分别。线性回归模型显示了较高的准确度(RgydF4y2Ba2gydF4y2BaHOXA3gydF4y2Ba= 0.998, rgydF4y2Ba2gydF4y2BaMAP4K1gydF4y2Ba= 0.997)。gydF4y2Ba4gydF4y2Bag,图SgydF4y2Ba7gydF4y2Bac).这些发现表明gydF4y2BaHOXA3gydF4y2Ba和gydF4y2BaMAP4K1gydF4y2BaDNA甲基化模式可以准确地解旋白细胞和颊上皮细胞从混合唾液样本。gydF4y2Ba
ls-DMPs在炎症疾病中的独立验证gydF4y2Ba
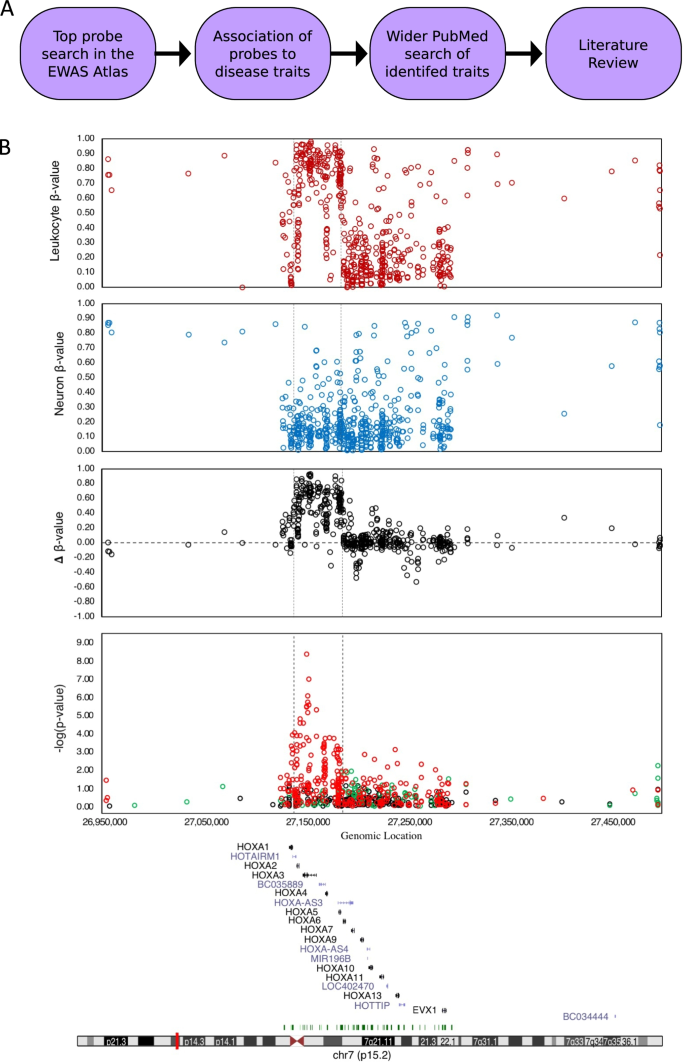
免疫细胞驱动炎症,因此,如果我们识别的ls-DMPs是有效的,与对照组相比,我们预计它们在以炎症疾病组织为特征的甲基化数据集中会被过度代表。利用网上数据库“EWAS Atlas”(gydF4y2Bahttps://ngdc.cncb.ac.cn/ewas/atlasgydF4y2Ba) [gydF4y2Ba20.gydF4y2Ba],我们从35篇论文中发现了28个相关性状(表SgydF4y2Ba2gydF4y2Ba),与我们的ls- dmp相关。CpG位点重叠数量最多的前三个特征是牛皮癣(Chandra等鉴定出33个DMPs)。gydF4y2Ba21gydF4y2Ba]),炎症性肠病(IBD) (Agliata等人确定的12个DMPs。[gydF4y2Ba22gydF4y2Ba]),以及阿尔茨海默病(AD)(在三份出版物中确定了12个dmp [gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba])。gydF4y2Ba
虽然EWAS地图集是一个很好的资源,但它受到数据库中出版物数量较少的限制(分析时为910份)。因此,我们在PubMed数据库中搜索了检测上述特征的病例与对照组DNA甲基化差异的研究gydF4y2Ba2gydF4y2Ba及表SgydF4y2Ba3.gydF4y2Ba).总的来说,我们发现了3篇关于牛皮癣的文章,2篇关于IBD的文章,7篇关于AD的文章。在使用Illumina Infinium HumanMethylation450 BeadChip阵列平台的11篇论文中,我们发现7篇论文至少有一个甲基化差异区域重叠于我们的ls-DMPs-cg00921266和cg08101036gydF4y2BaHOXA3gydF4y2Ba基因座分别在9个和4个数据集中存在,cg05258935和cg02798280来自于gydF4y2BaMAP4K1gydF4y2Ba位点分别出现在3个和4个数据集中。此外,gydF4y2BaHOXA3gydF4y2Ba和gydF4y2BaMAP4K1gydF4y2Ba基因座在每个疾病性状中至少出现一次(表SgydF4y2Ba4gydF4y2Ba及表SgydF4y2Ba5gydF4y2Ba).gydF4y2Ba
的gydF4y2BaHOXA3gydF4y2Ba在关注AD的出版物中,基因座的比例大大过高[gydF4y2Ba23gydF4y2Ba,gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba34gydF4y2Ba],高甲基化与AD严重程度相关的一致报告。例如,De Jager等人[gydF4y2Ba32gydF4y2Ba]和史密斯等人。[gydF4y2Ba30.gydF4y2Ba独立报告一个跨越HOXA集群的48 kb区域gydF4y2BaHOXA2gydF4y2Ba来gydF4y2BaHOXA6gydF4y2Ba在Braak期表现为高甲基化。我们假设,这种特征是由于白细胞浸润到AD大脑造成的,这种现象已知发生在衰老的人脑和AD小鼠模型中,因为血脑屏障变得渗漏[gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba].我们复制了Smith等人使用的DMP发现管道。gydF4y2Ba30.gydF4y2Ba]用于HOXA集群(底部面板,图。gydF4y2Ba5gydF4y2Bab)并将其与Moss数据集中的白细胞和皮层神经元甲基化进行比较(GEO登录:GSE122126)。叠加图清楚地表明,白细胞在HOXA位点具有高度的DNA甲基化,而皮层神经元则没有。这种DNA甲基化的差异反映了AD中高甲基化的确切基因组位置,表明两种观察结果之间存在高度相关性(图2)。gydF4y2Ba5gydF4y2Bab).的确,比较来自Moss数据集的皮层神经元和白细胞,可以发现两者中有5个ls- dmpgydF4y2BaHOXA3gydF4y2Ba之前在表中描述的cgigydF4y2Ba1gydF4y2Ba(chr7:27,153,187-27,153,647和chr7:27,154,999-27,155,426)的β值差异≥0.9(表SgydF4y2Ba6gydF4y2Ba).假设我们可以将亚硫酸氢盐测序测试的原理扩展到阵列数据,我们使用Braak 0期和Smith等人的6个样本进行了简单的免疫细胞定量。[gydF4y2Ba30.gydF4y2Ba]数据使用cg00921266。我们计算出,由于Braak 6期大脑的变化,总细胞中额外的13.9% (sd = 6.6%)是白细胞来源的。(图gydF4y2Ba11gydF4y2Ba).gydF4y2Ba
HOXA3和MAP4K1在炎性疾病中的作用。gydF4y2Ba一个gydF4y2Ba在线数据选择方法流程图。gydF4y2BabgydF4y2Ba甲基化用a表示gydF4y2BaβgydF4y2Ba为白细胞(第一张图,红色),皮质神经元(第二张图,蓝色)的值,以及gydF4y2BaβgydF4y2Ba-value之间的值(第三个面板,黑色)用于HOXA集群。数据来自Moss等人[gydF4y2Ba10gydF4y2Ba数据集GEO接入:(GSE122126)。下图是重建的迷你曼哈顿图,显示了与阿尔茨海默病Braak分期相关的HOXA聚类甲基化。gydF4y2BaPgydF4y2Ba-值是使用DNA甲基化与每个Braak分期(0-6)的线性回归模型生成的。红圈表示甲基化增加(gydF4y2BaβgydF4y2Ba-value)随Braak阶段的增加而降低≥0.1,绿色为降低≥0.1,黑色为变化< 0.1。数据来自Smith等人。[gydF4y2Ba30.gydF4y2Ba] (GEO加入:GSE80970)gydF4y2Ba
有趣的是,我们研究的许多出版物在寻找疾病生物标志物的过程中甚至没有提到白细胞浸润是一个混杂因素,尽管研究了炎症性疾病[gydF4y2Ba21gydF4y2Ba,gydF4y2Ba26gydF4y2Ba,gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba].其他人提到白细胞浸润的可能性或讨论了细胞异质性[gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,gydF4y2Ba28gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,但没有控制这种可变性。在某些情况下,这种情况发生在荟萃分析中,其中作者检查了先前收集的数据,并且不可能进行额外的实验控制[gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,gydF4y2Ba34gydF4y2Ba].只有一份出版物调查了他们样本中白细胞的比例;然而,这些数据并未显示[gydF4y2Ba27gydF4y2Ba].gydF4y2Ba
总之,在多种炎症疾病的甲基化数据集中存在许多ls-DMPs,进一步验证了它们作为泛白细胞生物标志物的有用性。我们的分析还强调了这些研究中对细胞组成的控制极少使用。最后,我们发现AD中先前无法解释的HOXA聚类的高甲基化与白细胞和脑组织之间的DNA甲基化差异之间存在很强的相关性。我们认为,白细胞浸润是甲基化变化的驱动因素,而不是常驻细胞类型甲基化状态的改变。gydF4y2Ba
与免疫细胞反褶积技术在癌症中的比较gydF4y2Ba
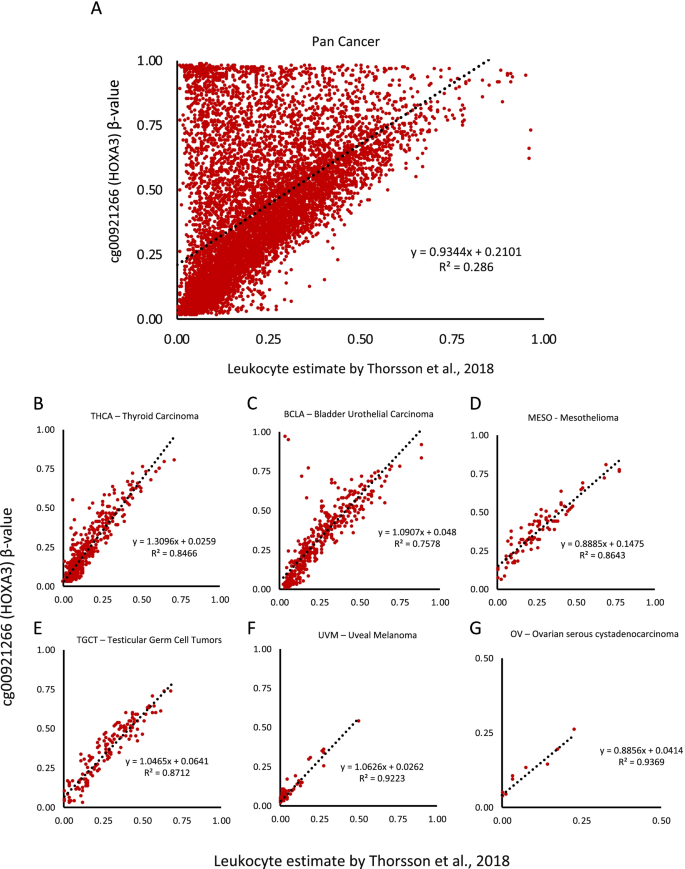
免疫系统在应对肿瘤方面具有复杂而关键的作用,在某些情况下,炎症可导致肿瘤发生[gydF4y2Ba13gydF4y2Ba,gydF4y2Ba37gydF4y2Ba].Thorsson等人的里程碑式研究[gydF4y2Ba13gydF4y2Ba量化了对大量不同类型癌症的免疫反应。Thorsson等人使用相对论复杂反褶积方法,要求Illumina Infinium HumanMethylation450 BeadChip阵列;我们询问在我们的研究中确定的ls-DMPs是否可以在不使用阵列技术的情况下预测非血液来源肿瘤中白细胞种群的比例。利用TCGA数据库,我们比较了cg00921266 (gydF4y2BaHOXA3gydF4y2Ba)与Thorsson等人估计的白细胞值相对应。[gydF4y2Ba13gydF4y2Ba]收集了30种不同肿瘤类型的8000多个肿瘤样本(图。gydF4y2Ba6gydF4y2Baa).泛癌症比较显示总体相关性较差(gydF4y2BaRgydF4y2Ba2gydF4y2Ba= 0.286);然而,白细胞估计值与cg00921266甲基化之间的许多数据点呈1:1的线性关系。其余数据位于这条线之上,表明肿瘤中白细胞的数量在该位点上设定了DNA甲基化的基线,在该位点上频繁发生突变gydF4y2BaHOXA3gydF4y2Ba这些实际上都是高甲基化事件。乳腺浸润性癌、大肠腺癌、肝细胞癌和皮肤黑色素瘤是这方面的个别例子(图SgydF4y2Ba12gydF4y2Ba).这与其他强调癌症中HOXA聚类基因高甲基化的报道一致[gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba,gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba].虽然cg00921266与Thorsson等人估计的白细胞之间的相关性在许多单个肿瘤中很难看到,但六种肿瘤类型(葡萄膜黑色素瘤(gydF4y2BapgydF4y2Ba-value = 9.70e−11),甲状腺癌(gydF4y2BapgydF4y2Ba-value = 2.32e−08),睾丸生殖细胞肿瘤(gydF4y2BapgydF4y2Ba-value = 5.12e−08),尿路上皮性膀胱癌(gydF4y2BapgydF4y2Ba-value = 9.5e−08),间皮瘤(gydF4y2BapgydF4y2Ba-value = 6.28e−21)和卵巢浆液性囊腺癌(gydF4y2BapgydF4y2Ba-value = 1.31e−03)为显著的1:1关系(图;gydF4y2Ba6gydF4y2Bab-g)。这些数据表明,至少对于某些癌症类型,单个CpG位点(cg00921266)的甲基化是准确测定白细胞浸润肿瘤所需要的全部。gydF4y2Ba
cg00921266甲基化与肿瘤中白细胞比例的关系gydF4y2Ba.gydF4y2Bacg00921266 DNA甲基化散点图(用a表示)gydF4y2BaβgydF4y2Ba-value和白细胞估计值gydF4y2Ba一个gydF4y2BaTCGA泛癌症数据集中的所有癌症,gydF4y2BabgydF4y2Ba甲状腺癌,gydF4y2BacgydF4y2Ba膀胱尿路上皮癌,gydF4y2BadgydF4y2Ba间皮瘤,gydF4y2BaegydF4y2Ba睾丸生殖细胞肿瘤,gydF4y2BafgydF4y2Ba葡萄膜黑色素瘤gydF4y2BaggydF4y2Ba卵巢浆液性囊腺癌。数据来源于TCGA泛癌症图集出版页面(gydF4y2Bahttps://gdc.cancer.gov/about-data/publications/pancanatlasgydF4y2Ba)gydF4y2Ba
讨论gydF4y2Ba
使用阵列数据,我们在gydF4y2BaHOXA3gydF4y2Ba和gydF4y2BaMAP4K1gydF4y2Ba在所有主要的血细胞种群中,位点都被特异性甲基化(图2)。gydF4y2Ba2gydF4y2Bab).我们使用pmcs和唾液白细胞进行验证实验;PBMCs主要由CD4组成gydF4y2Ba+gydF4y2Ba和CD8gydF4y2Ba+gydF4y2Bat细胞,以及在较小程度上的单核细胞、b细胞和自然杀伤细胞[gydF4y2Ba42gydF4y2Ba],而95%的唾液白细胞是中性粒细胞[gydF4y2Ba18gydF4y2Ba].这两种组织类型都在白细胞来源部分显示出高甲基化。因此,我们已经在大多数成熟的血液来源细胞类型中识别并验证了两个具有独特DNA甲基化模式的区域。gydF4y2Ba
HOXA3和MAP4K1 DNA甲基化在血源性细胞中的生物学意义gydF4y2Ba
虽然生物标记物不需要反映功能,但与生物功能的相关性可以提供对标记物的信心。HOX基因是一个高度保守的转录因子家族,参与造血,特别是骨髓细胞的造血[gydF4y2Ba43gydF4y2Ba,gydF4y2Ba44gydF4y2Ba,gydF4y2Ba45gydF4y2Ba].决定性造血细胞的发育发生在胎儿主动脉,由此发生从内皮细胞向造血干细胞的转变[gydF4y2Ba46gydF4y2Ba].在小鼠身上进行的研究表明,晚期HOXA基因的内侧gydF4y2BaHOXA5gydF4y2Ba,gydF4y2BaHOXA7gydF4y2Ba,gydF4y2BaHOXA9gydF4y2Ba对内皮细胞向造血干细胞转变至关重要[gydF4y2Ba47gydF4y2Ba),而gydF4y2BaHOXA10gydF4y2Ba负责向髓系和红系分化[gydF4y2Ba48gydF4y2Ba].gydF4y2BaHOXA3gydF4y2Ba与HOXA簇中的其他基因不同,该基因起着维持内皮表型的作用,其在造血干细胞中的表达可以使它们回到内皮谱系[gydF4y2Ba49gydF4y2Ba].总的来说,这些数据表明,虽然中间和晚期HOXA基因驱动造血干细胞的分化,但早期HOXA基因,gydF4y2BaHOXA3gydF4y2Ba,抑制这种分化。与中间和晚期基因相比,早期HOXA基因中观察到的DNA甲基化增加。gydF4y2Ba5gydF4y2BaB,顶部面板)可能反映的沉默gydF4y2BaHOXA3gydF4y2Ba在造血干细胞分化过程中。gydF4y2Ba
MAP4K1是一种丝氨酸/苏氨酸激酶,大量参与JNK信号通路和NF- κB调控[gydF4y2Ba50gydF4y2Ba].MAP4K1似乎通过磷酸化和抑制SLP-76或BLNK等重要信号分子的活性来抑制t细胞和b细胞受体的激活[gydF4y2Ba50gydF4y2Ba];MAP4K1在免疫细胞粘附中具有双重作用,抑制淋巴细胞粘附,增强中性粒细胞粘附[gydF4y2Ba50gydF4y2Ba].差异甲基化区域内gydF4y2BaMAP4K1gydF4y2Ba基因座在基因体中被发现,而启动子区域在不同类型的细胞中始终未甲基化。这与最近的一项研究一致,该研究表明大多数基因内cgi在转录过程中发生甲基化[gydF4y2Ba51gydF4y2Ba].组织表达数据来自人类蛋白质图谱[gydF4y2Ba52gydF4y2Ba) (gydF4y2Bahttp://www.proteinatlas.orggydF4y2Ba)gydF4y2BaMAP4K1gydF4y2Ba表明它在血液、淋巴组织和胃肠道组织中高度表达;单细胞表达数据显示胃肠道表达主要来自于常驻免疫细胞的结果。gydF4y2Ba
总的来说,有证据表明白细胞特异性DNA甲基化模式在gydF4y2BaHOXA3gydF4y2Ba和gydF4y2BaMAP4K1gydF4y2Ba甲基化在造血干细胞发育早期沉积于这两个位点,可能在骨髓控制造血之前,并在整个成熟到成年细胞的过程中保持(图2)。gydF4y2Ba2gydF4y2Bac)。gydF4y2Ba
唾液中细胞成分的反褶积gydF4y2Ba
唾液是一种广泛用于表观遗传流行病学研究的样本类型,因为它是非侵入性的,易于储存较长一段时间,并含有充足的人类DNA [gydF4y2Ba19gydF4y2Ba,gydF4y2Ba53gydF4y2Ba].然而,基于唾液的研究并非没有局限性。Langie等人最近的一篇综述[gydF4y2Ba53gydF4y2Ba]讨论了采集方法如何极大地影响样品的组成。此外,个体年龄对样本异质性也有显著影响[gydF4y2Ba18gydF4y2Ba].gydF4y2Ba
唾液的异质性是表观遗传学研究人员面临的一个重大障碍;DNA甲基化差异可能反映了细胞类型之间的典型生物学差异,而不是疾病生物标志物。目前的细胞反褶积方法试图从生物信息学上做到这一点;然而,大多数需要参考数据集进行比较。不幸的是,这些方法只能应用于全基因组数据,而参考数据的选择会显著影响估计的细胞群[gydF4y2Ba19gydF4y2Ba,gydF4y2Ba53gydF4y2Ba,gydF4y2Ba54gydF4y2Ba].使用我们的双指标,4引物PCR检测,我们已经证明有可能精确地确定唾液样本中白细胞与颊细胞的比例,有可能解决这一问题。gydF4y2Ba
HOXA3和MAP4K1 DNA甲基化在疾病中的应用gydF4y2Ba
我们已经证明了gydF4y2BaHOXA3gydF4y2Ba和gydF4y2BaMAP4K1gydF4y2Ba能够准确区分混合细胞系统中的白细胞(例如,肠类器官与pmcs;唾液白细胞vs口腔上皮)。我们的实验有效地证明了高通量亚硫酸氢盐扩增子测序在确定组织样本中白细胞比例方面具有极好的潜力,尽管它在更复杂的活组织检查或组织系统中的表现仍有待观察。然而,在研究一系列炎症性疾病的出版物中检测到大量的ls-DMPs在这方面是令人鼓舞的(图。gydF4y2Ba5gydF4y2Ba).gydF4y2Ba
当我们检查其他数据中ls-DMPs的存在时,每项研究报告了我们的ls-DMPs列表中至少一个DMP,或报告了包含我们的ls-DMPs之一的DMR(表S .)gydF4y2Ba4gydF4y2Ba及表SgydF4y2Ba5gydF4y2Ba).我们还表明,在Braak 6期阿尔茨海默病中,HOXA集群的高甲基化,这是常见的报道[gydF4y2Ba23gydF4y2Ba,gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba],与正常白细胞和神经元之间的DNA甲基化差异有惊人的重叠。同时发生的ls-DMPs,特别是gydF4y2BaHOXA3gydF4y2Ba,提示这些研究中观察到的DNA甲基化差异可能是由于白细胞的浸润,而不是受影响组织内源性细胞DNA甲基化的变化。我们建议在研究炎症性疾病的病例对照甲基化实验中使用额外的对照,以解释白细胞的存在。这可能包括(a)使用流式细胞仪或等效技术分类的纯细胞群,(b)在白细胞DNA中添加一定数量的对照样本,以模拟白细胞浸润,或(c)与健康组织和白细胞的甲基化数据进行比较,以确定DNA甲基化的细胞类型特异性差异。gydF4y2Ba
我们在检测IBD的甲基化数据集中观察到许多ls- dmp [gydF4y2Ba22gydF4y2Ba,gydF4y2Ba28gydF4y2Ba].基于数组数据[gydF4y2Ba10gydF4y2Ba(图。gydF4y2Ba2gydF4y2Baa),我们收集到的亚硫酸氢矿扩增子亮片数据(图;gydF4y2Ba3.gydF4y2Ba),以及小鼠全基因组测序数据[gydF4y2Ba5gydF4y2Ba) (gydF4y2BaHOXA3gydF4y2Ba图SgydF4y2Ba2gydF4y2Ba),以及gydF4y2BaHOXA3gydF4y2Ba和gydF4y2BaMAP4K1gydF4y2Ba结肠上皮中的基因座几乎没有DNA甲基化。因此,可以想象,这些位点也可以作为肠道炎症标志物应用于粪便样本,特别是作为IBD的诊断工具。临床医生将IBD归类为克罗恩病或溃疡性结肠炎;两者的特点都是肠道粘膜屏障的破坏导致慢性炎症[gydF4y2Ba55gydF4y2Ba].诊断IBD的金标准是结肠镜检查;然而,这是一个侵入性和耗时的过程。虽然仍有待测试,但这些生物标记物可能在这种情况下有用,完全开发的诊断测试可能是当前钙保护蛋白测定的替代方法[gydF4y2Ba55gydF4y2Ba,gydF4y2Ba56gydF4y2Ba].gydF4y2Ba
使用来自TCGA数据库的泛癌症数据(来自8000多个个体的30种癌症),我们已经证明了来自单个CpG位点cg00921266 (gydF4y2BaHOXA3gydF4y2Ba),与总白细胞估计数有很强的线性相关(先前由Thorsson等人发表。[gydF4y2Ba13gydF4y2Ba])六种不同的癌症类型:葡萄膜黑色素瘤、间皮瘤、睾丸生殖细胞肿瘤、膀胱尿路上皮癌、卵巢浆液性囊腺癌和甲状腺癌。尽管单一肿瘤内的癌细胞存在异质性,更不用说不同的肿瘤类型[gydF4y2Ba57gydF4y2Ba,gydF4y2Ba58gydF4y2Ba],我们的数据表明,差异甲基化模式在gydF4y2BaHOXA3gydF4y2Ba基因座在这些癌症的整个癌变过程中都保持不变[gydF4y2Ba59gydF4y2Ba].因此,我们在此使用的基于扩增子的方法测试,或仅来自单个CpG (cg00921266)的450 K Illumina阵列数据,应该能够提供白细胞比例的准确估计,而无需复杂的反褶积[gydF4y2Ba4gydF4y2Ba].gydF4y2Ba
结论gydF4y2Ba
总之,我们已经验证了两个基因座gydF4y2BaHOXA3gydF4y2Ba和gydF4y2BaMAP4K1gydF4y2Ba使用高通量扩增子-亚硫酸氢酶测序方法,准确测量了两种不同背景下的白细胞比例。这在临床和研究环境中都是有价值的,因为它以更低的成本和更高的吞吐量消除了基于阵列的反褶积的需要。重要的是,我们发现许多此类ls-DMPs在炎症性疾病中甲基化差异的研究中都有报道,这表明该信号是浸润白细胞的结果,而不是原生细胞的变化。最后,我们发现,在六种癌症类型的集合中,一个CpG位点(cg00921266)的甲基化发生在肿瘤的起始位点gydF4y2BaHOXA3gydF4y2Ba位点与Thorsson等人进行的白细胞估计高度相关。[gydF4y2Ba13gydF4y2Ba],这表明单个CpG位点或扩增子可能能够准确地确定癌症样本中白细胞的比例。gydF4y2Ba
方法gydF4y2Ba
数据采集gydF4y2Ba
我们使用了几个Illumina InfiniumHumanMethylation450 BeadChip数据集,这些数据集来自基因表达Omnibus和癌症基因组图谱计划(TCGA)。所有数据下载并导入R-Studio和Microsoft Excel进行分析。我们使用Moss数据集进行了初始生物标志物发现(GEO接入:GSE122126) [gydF4y2Ba10gydF4y2Ba]以及使用Reinius数据集验证血液来源的甲基化模式(GEO登录:GSE35069) [gydF4y2Ba11gydF4y2Ba].检查…的甲基化gydF4y2BaHOXA3gydF4y2Ba和gydF4y2BaMAP4K1gydF4y2Ba在不同的造血干细胞群中,我们使用Lessard数据集(GEO登录:GSE56491)。使用Smith等人制作的数据集重新分析了阿尔茨海默病HOXA聚类的DNA甲基化差异(GEO登录:GSE80970) [gydF4y2Ba30.gydF4y2Ba].我们从TCGA泛癌图谱发布页面(gydF4y2Bahttps://gdc.cancer.gov/about-data/publications/pancanatlasgydF4y2Ba),分别[gydF4y2Ba13gydF4y2Ba,gydF4y2Ba60gydF4y2Ba].gydF4y2Ba
生物标志物的发现gydF4y2Ba
我们使用Moss等人发表的数据集进行了初始生物标志物发现。[gydF4y2Ba10gydF4y2Ba] (GEO加入:GSE122126)。该数据集包含了423213个CpG位点的β值形式的甲基化数据。我们排除了所有cfDNA和体外混合样本,只检查了来自分类的健康组织样本的数据。总的来说,我们使用了数据集中101个样本中的23个。我们计算了白细胞之间的差异gydF4y2BaβgydF4y2Ba-值和平均值gydF4y2BaβgydF4y2Ba-所有其他细胞的值,以确定白细胞特异性甲基化模式。我们认为CpG站点是绝对的gydF4y2BaβgydF4y2Ba-值差异≥0.8作为ls-DMPs。在CpG岛(如Illumina Infinium所述)外的ls-DMPs被丢弃,以确保每次读取的高密度CpG位点。CpG探针位置用注释包‘illuminahumanmethylation450kano .ilmn12确定。hg19 ',在R-Studio。gydF4y2Ba
样品采集与处理gydF4y2Ba
PBMC分离及DNA纯化gydF4y2Ba
采用Ficoll-Paque密度离心法分离PBMCs。简单地说,血液被收集到EDTA收集管中。转移到50ml Falcon试管后,将血液与等量PBS混合,然后在干净的50ml Falcon试管中小心地分层到Ficoll-Paque溶液(室温)中。室温下800 g离心20 min,不制动,提取单个核细胞(Ficoll-Paque层顶部)。然后用红细胞裂解缓冲液(155 mM NH)清洗pbmc多次gydF4y2Ba4gydF4y2Ba氯,10毫米KHCOgydF4y2Ba3.gydF4y2Ba(0.1 mM EDTA)来去除受污染的红细胞。然后使用QIAAMP DNA血液迷你试剂盒(QIAGEN #QIAG51104)分离PBMC DNA。gydF4y2Ba
肠道类器官培养及DNA纯化gydF4y2Ba
按照制造商的说明,使用超纯苯酚:氯仿:异戊醇(25:24:1)(Invitrogen Cat# 15,593-031)从冷冻保存的肠类器官中提取DNA。分离的DNA重悬在无rnase的水中,用Thermo Scientific纳米滴分光光度计定量。肠道类器官按照既定方案培养[gydF4y2Ba61gydF4y2Ba]取自上文所述从直肠活检中分离的隐窝[gydF4y2Ba62gydF4y2Ba].参与者提供书面知情同意书。样本收集得到了悉尼儿童医院伦理审查委员会的批准(HREC/16/SCHN/120)。隐窝在细胞外基质(70%基质(生长因子减少,无酚;Corning 356,231)在24孔板中,密度为~ 10 - 30隐窝,每孔3 × 10 μ l液滴。每隔2天更换一次IntestiCult类器官生长培养基(STEMCELL Technologies),类器官生长7-10天,从Matrigel中收获,并在DNA提取前冷冻保存。gydF4y2Ba
唾液制备,细胞分离,DNA纯化gydF4y2Ba
我们收集5毫升未刺激的唾液样本,如前所述[gydF4y2Ba18gydF4y2Ba].在采集前30分钟,参与者只喝水。为了从口腔上皮中分离唾液白细胞,我们在400 RCF下对样本进行离心,并用0.01 Molar PBS清洗。使用荧光激活细胞分选(FACS)分离颊上皮细胞,仅使用正向散射和侧散射。采用序贯细胞过滤法分离唾液白细胞。样品首先通过40µm过滤,然后用20µm滤网过滤以排除颊部细胞。gydF4y2Ba
我们将每个样本的一部分涂抹在显微镜载玻片上,用苏木精和伊红染色,以评估分类后的细胞群和混合唾液的细胞组成。细胞计数手动进行,每张载玻片在显微镜下使用20 ×物镜观察。随机选择一个视野,其中有适当的细胞数量,视野中的所有细胞都可以充分识别(即细胞的聚集和重叠),并且有一个可接受的染色水平来识别每个细胞。我们在每张幻灯片的两个字段中计算了至少100个单元格。gydF4y2Ba
DNA是用炸弹提取的。bio TNA extraction from mammalian tissue protocol [63gydF4y2Ba].简而言之,细胞在1 × TNES和0.5µL的20 mg mL中裂解gydF4y2Ba−1gydF4y2Ba裂解液分别与6 M GITC、悬浮TE中羧基包被磁珠和绝对异丙醇按1:2:2:4的比例混合。孵育5分钟后,将样品放在磁铁上,用绝对异丙醇洗涤一次,用70%乙醇洗涤两次。我们用TE缓冲液从磁珠中洗脱DNA,并用高灵敏度dsDNA Qubit测量其浓度。gydF4y2Ba
亚硫酸盐转化和扩增子测序gydF4y2Ba
根据制造商指南,我们使用Zymo Research EZ-96 DNA甲基化MagPrep试剂盒对200 ng提取的DNA进行亚硫酸氢盐转换,并使用ssDNA Qubit测量DNA浓度。我们使用双指标、四引物PCR法扩增了至少75ng的转化DNA(图。gydF4y2Ba7gydF4y2Ba).我们进行了两个放大步骤的反应。在第一步中,用KAPA HiFi Uracil + ReadyMix和0.1 μ M或0.3 μ M的第一步引物扩增转化的DNAgydF4y2BaHOXA3gydF4y2Ba和gydF4y2BaMAP4K1gydF4y2Ba分别扩增子。每个反应加无核酸酶H 25µLgydF4y2Ba2gydF4y2BaO.扩增循环参数为95°C 2 min, 98°C 20 s, 59°C 10 s, 72°C 20 s,循环23次。最后一个延伸步骤在72°C下进行5分钟。将反应置于冰上,0.2µM的第二步引物(Illumina P5和P7适配器和TruSeq索引,添加连接子序列,表SgydF4y2Ba7gydF4y2Ba)。重复上述扩增5个周期。我们使用固相反向固定化聚乙二醇中羧基包被磁珠来为DNA片段选择扩增子的长度[gydF4y2Ba63gydF4y2Ba].我们在Illumina isq100系统上对扩增子进行了测序。gydF4y2Ba
引物序列见表SgydF4y2Ba7gydF4y2Ba.HOXA3扩增子包括cg00921266和cg08101036。MAP4K1扩增子的引物不能设计为cg05258935或cg02798280;然而,该扩增子针对两个位点的250个碱基对内的相同CpG岛。gydF4y2Ba
数据处理和统计分析gydF4y2Ba
我们使用Cutadapt和TrimGalore从每次读取中删除适配器序列[gydF4y2Ba64gydF4y2Ba].使用俾斯麦[gydF4y2Ba65gydF4y2Ba],我们将扩增子reads映射到只包含扩增子序列的自定义“基因组”。用于作图的序列来自UCSC基因组浏览器hg38 (gydF4y2BaHOXA3gydF4y2Ba: chr7:27,113,957 - 27114300,gydF4y2BaMAP4K1gydF4y2Ba: chr19:38,596,411 - 38596696)。Hon等人获得19种不同小鼠组织的全基因组亚硫酸氢盐测序数据。[gydF4y2Ba5gydF4y2Ba]使用Bismark将其映射到MGSCv37 (mm9)小鼠参考基因组,并使用SeqMonk进行可视化。gydF4y2Ba
我们在R-Studio中使用自定义r脚本制作了热图、密度图、细胞类型调用分析和线性回归。我们使用pROC包在R-Studio中进行logistic回归分析和ROC曲线构建。gydF4y2Ba
我们利用cg00921266的甲基化来估计阿尔茨海默病患者前额叶皮层浸润白细胞的比例。为此,我们使用了以下公式:gydF4y2Ba\ ({L} _ {est} = \压裂{{m} _ {6} - {m} _ {0}} {{m} _ {L}} \)gydF4y2Ba,这里的LgydF4y2Ba美国东部时间gydF4y2Ba白细胞估计数,mgydF4y2Ba6gydF4y2BaBraak第6期,mgydF4y2Ba0gydF4y2Ba甲基化在Braak阶段0gydF4y2BalgydF4y2Ba基于Moss数据集的白细胞甲基化。gydF4y2Ba
选择用于炎症疾病分析的在线数据集gydF4y2Ba
我们搜索了EWAS Atlas存储库中的所有ls- dmp (gydF4y2Bahttps://ngdc.cncb.ac.cn/ewas/atlasgydF4y2Ba)以确定与低甲基化或高甲基化相关的相关疾病特征。我们总结了与每种疾病特征相关的独特ls-DMPs的数量,并使用PubMed数据库对前三种疾病进行了更全面的文献搜索。我们的具体搜索词是“DNA甲基化”,以及“牛皮癣”、“炎症性肠病”或“阿尔茨海默病”。我们纳入了2010年以后的出版物,其中检查了受影响组织的分析(即皮肤、肠道或大脑),并排除了仅检查受影响患者免疫细胞群的出版物。我们记录了每篇文章中报道的低甲基化或高甲基化CpG位点和区域的数量(来自正文和补充数据),这些位点和区域与我们已经确定的ls-DMPs重叠。此外,我们还根据一些标准评估了每篇文章:(1)研究的组织类型是散装细胞还是分选细胞,(2)收集数据的方法,(3)使用对照或细胞反褶积技术来减轻多细胞对数据分析的影响,以及(4)对细胞异质性的总体讨论,特别是白细胞浸润到样本组织的问题,以及它与研究结果的关系。gydF4y2Ba
数据和材料的可用性gydF4y2Ba
在这项研究中产生或分析的所有数据都包含在这篇发表的文章[及其补充信息文件]中。gydF4y2Ba
缩写gydF4y2Ba
- CpG:gydF4y2Ba
-
胞嘧啶磷酸鸟嘌呤gydF4y2Ba
- cfDNA:gydF4y2Ba
-
无细胞脱氧核糖核酸gydF4y2Ba
- DMP:gydF4y2Ba
-
差异甲基化位置gydF4y2Ba
- ls-DMP:gydF4y2Ba
-
白细胞特异性差异甲基化位置gydF4y2Ba
- CGI:gydF4y2Ba
-
CpG岛gydF4y2Ba
- PBMC:gydF4y2Ba
-
外周血单个核细胞gydF4y2Ba
- 中华民国:gydF4y2Ba
-
接收机工作特性gydF4y2Ba
- TPR:gydF4y2Ba
-
真阳性率gydF4y2Ba
- 玻璃钢:gydF4y2Ba
-
假阳性率gydF4y2Ba
- 炎症性肠病:gydF4y2Ba
-
炎症性肠病gydF4y2Ba
- 广告:gydF4y2Ba
-
阿尔茨海默病gydF4y2Ba
- TCGA:gydF4y2Ba
-
癌症基因组图谱gydF4y2Ba
- 流式细胞仪:gydF4y2Ba
-
荧光激活细胞分选gydF4y2Ba
- PBS:gydF4y2Ba
-
磷酸盐gydF4y2Ba
- 种:gydF4y2Ba
-
额下回gydF4y2Ba
- MTG:gydF4y2Ba
-
颞中回gydF4y2Ba
- 电子商务:gydF4y2Ba
-
内嗅皮层gydF4y2Ba
- IEC:gydF4y2Ba
-
肠上皮细胞gydF4y2Ba
- PFC:gydF4y2Ba
-
前额叶皮层gydF4y2Ba
- STG:gydF4y2Ba
-
颞上回gydF4y2Ba
- UTR:gydF4y2Ba
-
翻译区gydF4y2Ba
- TSS:gydF4y2Ba
-
转录起始位点gydF4y2Ba
参考文献gydF4y2Ba
Goll MG, Bestor TH。真核胞嘧啶甲基转移酶。生物化学,2005;74:481-514。gydF4y2Ba
饶S,等。由于CpG甲基化导致的DNA形状变化的系统预测解释了蛋白质- DNA结合的表观遗传效应。表观遗传学染色质。2018;11(1):6。gydF4y2Ba
Du Q,等。甲基- cpg结合域蛋白:表观基因组的读者。表观基因组学,2015;7(6):1051 - 73。gydF4y2Ba
施密特M,等。基于单个CpG位点靶向DNA甲基化分析的人体组织细胞亚群反褶积。中国生物医学工程学报。2020;18(1):178。gydF4y2Ba
GC议员等。在成年小鼠组织DNA甲基化图中鉴定的胚胎增强子的表观遗传记忆。中国科学,2013;45(10):1198-206。gydF4y2Ba
Bendich A, Wilczok T, Borenfreund E.循环DNA可能是肿瘤发生的因素。科学。1965;148(3668):374 - 6。gydF4y2Ba
Stroun M,等。在癌症患者血浆中发现的DNA的肿瘤特征。肿瘤。1989;46(5):318 - 22所示。gydF4y2Ba
Lehmann-Werman R等。在无细胞循环DNA中使用肝细胞特异性甲基化标记监测肝损伤。JCI Insight, 2018;3(12):e120474。gydF4y2Ba
Lehmann-Werman R等。利用循环DNA的甲基化模式鉴定组织特异性细胞死亡。中国科学:自然科学,2016;29(2):344 - 344。gydF4y2Ba
Moss J,等。全面的人类细胞型甲基化图谱揭示了循环无细胞DNA在健康和疾病中的起源。地理学报。2018;9(1):5068。gydF4y2Ba
Reinius LE,等。纯化人血细胞中的差异DNA甲基化:对细胞谱系和疾病易感性研究的影响。PLoS ONE。7 (7): 2012; e41361。gydF4y2Ba
你C,等等。全血EWAS的细胞型反褶褶元分析揭示了血统特异性吸烟相关的DNA甲基化变化。网络学报。2020;11(1):4779。gydF4y2Ba
Thorsson V等。癌症的免疫图景。免疫力。2018;48 (4):812 - 830. - e14灯头。gydF4y2Ba
Titus AJ等。DNA甲基化的反褶积确定了乳腺癌亚型1p36上不同甲基化的基因区域。科学通报2017;7(1):11594。gydF4y2Ba
Lessard S,等。人类胎儿和成人红细胞祖细胞DNA甲基化谱的比较。基因组医学。2015;7(1):1。gydF4y2Ba
Lange V,等。MiSeq扩增子测序高通量HLA分型。BMC基因组学。2014;15(1):63。gydF4y2Ba
佐藤T,等。单个Lgr5干细胞在体外构建隐窝-绒毛结构,没有间充质小生境。大自然。2009;459(7244):262 - 5。gydF4y2Ba
Theda C,等。唾液和口腔拭子样本的细胞含量定量。科学通报2018;8(1):6944。gydF4y2Ba
Nishitani S,等。流行病学研究唾液样本DNA甲基化分析。表观遗传学。2018;13(4):352 - 62。gydF4y2Ba
李敏,等。EWAS图谱:表观基因组全关联研究的策划知识库。核酸研究进展2019;47(D1):D983-d988。gydF4y2Ba
钱德拉A等人。全表观基因组DNA甲基化调控银屑病的主要病理特征。临床表观遗传学。2018;10(1):108。gydF4y2Ba
Agliata I等人。炎症性肠病(IBD)的DNA甲基化组反映肠粘膜细胞的内源性和外源性因素。表观遗传学。2020;15(10):1068 - 82。gydF4y2Ba
张磊,等。前额叶皮层DNA甲基化差异的表观基因组范围元分析暗示了阿尔茨海默病的免疫过程。网络学报。2020;11(1):6114。gydF4y2Ba
Haertle L,等。甲基组学分析21三体识别认知和阿尔茨海默病相关的失调。临床表观遗传学。2019;11(1):195。gydF4y2Ba
Semick SA,等。整合DNA甲基化和基因表达谱跨多个大脑区域涉及阿尔茨海默病的新基因。神经病理学报,2019;137(4):557-69。gydF4y2Ba
周峰,等。表观基因组全关联分析确定了9个银屑病皮肤DNA甲基化位点。中华皮肤病学杂志,2016;36(4):779-87。gydF4y2Ba
Verma D,等。全基因组DNA甲基化分析鉴定了未受累性银屑病表皮的差异甲基化。中华皮肤病学杂志,2018;38(5):1088-93。gydF4y2Ba
Harris RA等。DNA甲基化相关的结肠粘膜免疫和防御反应treatment-naïve儿童溃疡性结肠炎。表观遗传学。2014;9(8):1131 - 7。gydF4y2Ba
Gasparoni G,等。DNA甲基化分析纯化神经元和神经胶质解剖年龄和阿尔茨海默病特异性变化在人类皮层。表观遗传学染色质。2018;11(1):41。gydF4y2Ba
史密斯RG,等。跨越HOXA基因簇的48kb区域的DNA甲基化升高与阿尔茨海默病神经病理学相关。老年痴呆症。2018;14(12):1580-8。gydF4y2Ba
李群生,孙勇,王涛。阿尔茨海默病全表观基因组关联研究复制了22个差异甲基化位点和30个差异甲基化区域。临床表观遗传学。2020;12(1):149。gydF4y2Ba
De Jager PL,等。阿尔茨海默病:ANK1, BIN1, RHBDF2和其他位点的早期脑DNA甲基化改变神经科学。2014;17(9):1156-63。gydF4y2Ba
Altuna M,等。阿尔茨海默病中海马的DNA甲基化特征与神经发生有关。临床表观遗传学。2019;11(1):91。gydF4y2Ba
史密斯RG,等。一项对阿尔茨海默病表观基因组全关联研究的荟萃分析突出了横跨皮层的新型差异甲基化位点。网络通信。2021;12(1):3517。gydF4y2Ba
阿尔茨海默病中白细胞转运的影像学研究。Front Immunol. 2016;7(33)。gydF4y2Ba
Gemechu JM, Bentivoglio M. T细胞招募在正常衰老的大脑。神经科学。2012;6:38。gydF4y2Ba
Coussens LM, Werb Z.炎症和癌症。大自然。2002;420(6917):860 - 7。gydF4y2Ba
达芬奇,等。HOXA3、7、9和10基因在胶质瘤中的甲基化定量分析:与肿瘤WHO分级和临床结局相关中国临床肿瘤学杂志,2012;29(1):457 - 457。gydF4y2Ba
Daugaard I等人。肺腺癌候选表观遗传生物标志物的鉴定和验证。科学通报2016;6:35807。gydF4y2Ba
Kuasne H,等。阴茎癌的全基因组甲基化和转录组分析:发现新的分子标记。临床表观遗传学。2015;7(1):46。gydF4y2Ba
甘BL,等。RT-qPCR、TCGA、硅晶分析HOXA3在肺腺癌中的下调及其分子机制。中国生物医学工程学报,2018;29(4):457 - 457。gydF4y2Ba
Kleiveland CR等。外周血单个核细胞。在:Verhoeckx K,等人,编辑。食品生物活性物质对健康的影响:体外和体外模型。湛:施普林格国际出版;2015.161 - 7页。gydF4y2Ba
Alharbi RA,等。HOX基因在正常造血和急性白血病中的作用。白血病。2013;27(5):1000 - 8。gydF4y2Ba
Ramos-Mejía V,等。HOXA9促进人类胚胎干细胞的造血承诺。血。2014;124(20):3065 - 75。gydF4y2Ba
Sauvageau G,等。同源盒基因在功能不同的CD34+人骨髓细胞亚群中的差异表达。中国科学(d辑:自然科学),2001;29(1):1 - 7。gydF4y2Ba
内皮到造血的转变:血液生成过程的最新进展。生物化学学报,2019;47(2):591-601。gydF4y2Ba
窦德华,等。内侧HOXA基因在人类发育过程中划定造血干细胞的命运。细胞生物学杂志,2016;18(6):595-606。gydF4y2Ba
Magnusson M,等。HOXA10是造血干细胞和红细胞/巨核细胞发育的关键调节因子。血。2007;109(9):3687 - 96。gydF4y2Ba
Iacovino M,等。HoxA3是一种致血内皮的顶端调节因子。细胞生物学杂志,2011;13(1):72-8。gydF4y2Ba
庄兴华,王旭,谭涛。MAP4K家族激酶在免疫和炎症中的作用。Adv immune . 2016; 129:277-314。gydF4y2Ba
Jeziorska DM,等。基因内CpG岛的DNA甲基化取决于它们在分化和疾病期间的转录活性。中国生物医学工程学报,2017;26(3):344 - 344。gydF4y2Ba
Uhlén M,等。蛋白质组学:基于组织的人类蛋白质组图谱。科学。2015;347(6220):1260419。gydF4y2Ba
Langie SAS,等。唾液DNA甲基化分析:生物标志物鉴定需要考虑的方面。《基础临床药理学杂志》2017;121(增刊3):93-101。gydF4y2Ba
黄依涛,等。用细胞学和DNA甲基化估计口腔样本上皮细胞含量的比较。表观遗传学。2022;17(3):327 - 34。gydF4y2Ba
粪钙保护蛋白在炎症性肠病中的应用。中华胃肠病学杂志。2017;13(1):53-6。gydF4y2Ba
林爱玲,郭国强。粪便钙保护素。临床化学杂志。2018;87:161-90。gydF4y2Ba
达戈戈-杰克一世,肖AT。肿瘤的异质性和对癌症治疗的耐药性。中华儿科杂志,2018;15(2):81-94。gydF4y2Ba
郭敏,等。癌症的表观遗传异质性。生物标志物res 2019;7(1):23。gydF4y2Ba
王志强,王志强,王志强,等。免疫系统在肿瘤中的作用:从肿瘤起始到转移进展。基因工程学报,2018;32(19-20):1267-84。gydF4y2Ba
Hoadley KA,等。来自33种癌症的10,000个肿瘤的分子分类中,细胞起源模式占主导地位。细胞。2018;173 (2):291 - 304. - e6。gydF4y2Ba
王世林,等。气道和肠道类器官的分子动力学和异型性揭示了R352Q-CFTR导通缺陷。bioRxiv。2021.2021.08.11.456003页。gydF4y2Ba
Berkers G,等。直肠类器官可实现囊性纤维化的个性化治疗。2019;26(7):1701-1708.e3。gydF4y2Ba
Oberacker P,等。Bio-On-Magnetic-Beads (BOMB):高通量核酸提取和操作的开放平台。《公共科学图书馆·生物学》2019;17(1):e3000107。gydF4y2Ba
Martin M. Cutadapt从高通量测序读取中移除适配器序列。财经学报,2011;17(1):10-2。gydF4y2Ba
Krueger F, Andrews SR. Bismark:用于亚硫酸氢盐- seq应用的灵活对准器和甲基化调用器。生物信息学。2011;27(11):1571 - 2。gydF4y2Ba
确认gydF4y2Ba
我们感谢A. Keith Ooi教授为悉尼儿童医院分子和综合囊性纤维化研究中心收集直肠活检。SAW由澳大利亚国家卫生和医学研究委员会资助(NHMRC_APP1188987)。我们感谢Michelle Wilson对流式细胞仪的帮助。我们感谢唐娜·邦德博士的技术支持和审阅手稿。gydF4y2Ba
资金gydF4y2Ba
这项研究由奥塔哥大学资助。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
RW进行PBMC分离和DNA纯化。SW培养肠道类器官,进行DNA纯化。MD完成了所有其他的实验工作。OO在统计分析方面提供了广泛的建议。MD撰写了初稿,并准备了数据。MD和OO负责本研究中使用的定制r脚本。TH和IM构思了这项研究。作者们阅读并批准了最终的手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
伦理批准并同意参与gydF4y2Ba
肠类器官细胞的收集和培养gydF4y2Ba
参与者提供直肠活检和随后肠类器官培养的书面知情同意。样本收集得到了悉尼儿童医院伦理审查委员会的批准(HREC/16/SCHN/120)。gydF4y2Ba
PBMC和唾液收集gydF4y2Ba
在知情同意的情况下,采集研究人员的外周血单个核细胞和唾液样本。gydF4y2Ba
发表同意书gydF4y2Ba
不适用。gydF4y2Ba
相互竞争的利益gydF4y2Ba
TH是Totovision/Totogen Ltd(一家小型农业和生物技术咨询公司)的股东兼董事。其他作者宣称没有利益竞争。gydF4y2Ba
补充信息gydF4y2Ba
附加文件1。表S1:gydF4y2Ba
在Moss等人,2018数据集(GSE122126)中,白细胞和所有其他组织之间Δ β值≥0.8的CpG位点。gydF4y2Ba表S2。gydF4y2Ba使用EWAS ATLAS鉴定的出版物和相关特征列表,报告表S1中的一个或多个CpG位点在其队列中甲基化不同。gydF4y2Ba表S3。gydF4y2Ba表2中列出的出版物的标题和doi。gydF4y2Ba表S4。gydF4y2Ba来自EWAS ATLAS或PubMed搜索的相关特征的发表表,报告了表S1中的一个或多个CpG位点在其队列中甲基化不同。gydF4y2Ba表S5。gydF4y2Ba来自EWAS ATLAS或PubMed搜索的相关性状的发表表,报告了不同甲基化区域,其中包含表S1中的一个CpG位点。gydF4y2Ba表S6。gydF4y2Ba在Moss等人,2018数据集(GSE122126)中,白细胞和皮层神经元之间Δ β值≥0.8的CpG位点。gydF4y2Ba表S7。gydF4y2Ba本研究中使用的引物列表。gydF4y2Ba
附加文件2:图S1。gydF4y2Ba
ls-DMPs在基因和CpG岛方面的分布。饼图分别显示了高甲基化(a - b)和低甲基化(c - d) ls-DMPs与最近基因和最近CpG岛的关系。gydF4y2Ba图S2。gydF4y2Ba19种不同小鼠组织HOXA3位点全基因组亚硫酸氢盐测序热图(chr6: 52125343-52127779, mm9;人类相当于chr7:27,113,454-27,115,864, hg38)。数据被分离到100个碱基对窗口中,用SeqMonk检测整体甲基化。gydF4y2Ba图S3。gydF4y2Ba基于MAP4K1 DNA甲基化的体外混合PBMC和肠道类器官DNA反褶积(a) pmcs(左)、50:50 mix(中)和肠道类器官(右)MAP4K1扩增子甲基化热图输出示例。每一行代表一个单独的读取,每列是扩增子内的一个CpG位点;列名表示CpG在读取中的位置。(b)纯肠道类器官(蓝色)和纯PBMC(红色)样品中每次读取的甲基化CpG位点数量的密度图。y轴表示x轴上每单位的概率,这样在特定区间内,曲线下的面积等于该区间内甲基化cpg数量的概率。带宽为0.8 (c)每个样本类型的分类比例的堆叠柱状图。读数分为肠类器官(蓝色)、PBMC(红色)和未分类(灰色)。(d)肠类器官和PBMC观察读数分类与预期读数分类的散点图。gydF4y2Ba图S4。gydF4y2BaHOXA3体外混合物的所有热图输出。第1行:肠类器官复制1-3个。第2行:肠类器官重复4-6个。第3行:体外混合99:1,95:5,50:50(如PBMC DNA:肠道类器官DNA)。第4行:体外混合5:95,1:99,PBMC复制1(作为PBMC DNA:肠类器官DNA)。第5行:PBMC复制2-3个。gydF4y2Ba图S5。gydF4y2BaMAP4K1体外混合物的所有热图输出。第1行:肠类器官复制1-3个。第2行:肠类器官重复4-6个。第3行:体外混合99:1,95:5,50:50(如PBMC DNA:肠道类器官DNA)。第4行:体外混合5:95,1:99,PBMC复制1(作为PBMC DNA:肠类器官DNA)。第5行:PBMC复制2-3个。gydF4y2Ba图S6。gydF4y2BaPBMC和肠类器官体外混合物的二项logistic回归。HOXA3和MAP4K1扩增子的二项逻辑回归(a和c)和受体工作特征(b和d)。gydF4y2Ba
附加文件3:图S7。gydF4y2Ba
基于MAP4K1 DNA甲基化模式的唾液反褶积。(a)唾液白细胞(左)、原始唾液(中)和颊上皮(右)MAP4K1扩增子甲基化热图输出示例。每一行代表一个单独的读取,每列是扩增子内的一个CpG位点;列名表示CpG在读取中的位置。(b)纯颊上皮(蓝色)和纯唾液白细胞(红色)样本中每次读取的甲基化CpG位点数量的密度图。y轴表示x轴上每单位的概率,这样在特定区间内,曲线下的面积等于该区间内甲基化cpg数量的概率。带宽为0.45 (c)三个混合唾液技术重复的观察读分类与预期读分类的散点图。基于手动细胞计数的预期百分比。gydF4y2Ba图S8。gydF4y2BaHOXA3唾液样本的所有热图输出。第1排:唾液白细胞1.1 - 1.3,唾液白细胞2.1。第二排:唾液白细胞2.2 - 2.3,唾液白细胞3.1 - 3.2。第三排:唾液白细胞3.3,生唾液1.1 - 1.3。第三行:口腔上皮1.1 - 1.3,口腔上皮2.1。第3行:颊上皮2.2 - 2.3,颊上皮3.1 - 3.2。第4行:颊上皮3.3gydF4y2Ba图S9。gydF4y2BaMAP4K1唾液样本的所有热图输出。第1排:唾液白细胞1.1 - 1.3,唾液白细胞2.1。第二排:唾液白细胞2.2 - 2.3,唾液白细胞3.1 - 3.2。第三排:唾液白细胞3.3,生唾液1.1 - 1.3。第三行:口腔上皮1.1 - 1.3,口腔上皮2.1。第3行:颊上皮2.2 - 2.3,颊上皮3.1 - 3.2。第4行:颊上皮3.3gydF4y2Ba图S10。gydF4y2Ba唾液样本的二项逻辑回归。HOXA3和MAP4K1扩增子的二项逻辑回归(a和c)和受体工作特征(b和d)。gydF4y2Ba图S11。gydF4y2Ba阿尔茨海默病大脑白细胞增加的估计。(a) Braak 0期和Braak 6期前额叶皮层中cg00921266位点(HOXA3)的DNA甲基化(以β值表示)。(b)根据cg00921266位点DNA甲基化的增加,估计Braak 6期大脑中白细胞的增加。阿尔茨海默病甲基化数据来自Smith等人,2018 (GSE80970)。gydF4y2Ba图S12。gydF4y2Ba个别癌症中cg00921266甲基化与白细胞比例的关系。TCGA泛癌症数据集中所有个体癌症的cg00921266 DNA甲基化散点图(表示为β值与白细胞估计数)。肾上腺皮质癌;乳腺癌;宫颈鳞癌和宫颈内腺癌;胆管癌;结肠腺癌;食管癌;多形性胶质母细胞瘤;头颈部鳞状细胞癌; KICH = Kidney Chromophobe; KIRC = Kidney renal clear cell carcinoma; KIRP = Kidney renal papillary cell carcinoma; LGG = Brain Lower Grade Glioma; LIHC = Liver hepatocellular carcinoma; LUAD = Lung adenocarcinoma; LUSC = Lung squamous cell carcinoma; PAAD = Pancreatic adenocarcinoma; PCPG = Pheochromocytoma and Paraganglioma; PRAD = Prostate adenocarcinoma; READ = Rectum adenocarcinoma; SARC = Sarcoma; SKCM = Skin Cutaneous Melanoma; STAD = Stomach adenocarcinoma; UCS = Uterine Carcinosarcoma; UCEC = Uterine Corpus Endometrial Carcinoma. Excludes thyroid carcinoma, bladder urothelial carcinoma, mesothelioma, testicular germ cell cancer, uveal melanoma, ovarian serous cystadenocarcinoma (found in Figure 5). Data was obtained from the TCGA Pan-cancer atlas publication page (https://gdc.cancer.gov/about-data/publications/pancanatlasgydF4y2Ba).gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.创作共用公共领域奉献弃权书(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本条所提供的资料,除非在资料的信用额度中另有说明。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
邓尼特,m.j.,奥尔特加-雷尔德,o.j.,沃特斯,S.A.gydF4y2Baet al。gydF4y2Ba白细胞特异性DNA甲基化生物标志物及其对病理表观遗传学分析的意义。gydF4y2Ba表观遗传学Commun。gydF4y2Ba2gydF4y2Ba, 5(2022)。https://doi.org/10.1186/s43682-022-00011-zgydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s43682-022-00011-zgydF4y2Ba
关键字gydF4y2Ba
- DNA甲基化gydF4y2Ba
- 生物标志物gydF4y2Ba
- 白细胞gydF4y2Ba
- 炎症gydF4y2Ba
- 癌症gydF4y2Ba
- 阿尔茨海默病gydF4y2Ba
- 唾液gydF4y2Ba
- 反褶积gydF4y2Ba
- HOXA3gydF4y2Ba
- MAP4K1gydF4y2Ba