摘要
背景
肾细胞癌(RCC)是世界上第14种最常见的癌症,约占所有癌症的4%。70%以上的RCC为透明细胞RCC (ccRCC)。到目前为止,还没有发现检测ccRCC的可靠生物标志物。本研究的目的是鉴定血液DNA甲基化(DNAm)标志物,用于早期发现和治疗ccRCC。
结果
为了识别ccRCC相关的DNAm标记,我们在发现阶段使用50名ccRCC患者和50名健康对照的全血DNA进行了靶向亚硫酸氢盐测序(TB-seq)和表观基因组全关联研究(EWAS)。EWAS采用线性回归模型进行。分析根据年龄、性别和估计的细胞类型组成进行了调整。在复制阶段,在48名独立的ccRCC患者和48名健康对照中验证了确定的ccRCC相关CpGs的准确性。我们鉴定了6种ccrcc相关的低甲基化cpgPCBD2 / MTND4P12在发现阶段(p< 1.75 × 10−8);在复制阶段有四个是可重复的(p< 2.96 × 10−8).在发现阶段(受试者工作特征曲线下面积[AUC-ROC] = 0.922)和复制阶段(AUC-ROC = 0.871), 6个cpg的DNAm水平之和都是ccRCC的有效指标。而且,结果独联体-表达定量甲基化分析表明,ccrcc相关CpGs的DNAm水平影响基因表达转录因子7(TCF7),电压依赖性阴离子选择通道1(VDAC1),它们与癌症的发展有关。
结论
在本研究中,我们鉴定了6种ccrcc相关的CpGsPCBD2 / MTND4P12EWAS使用基于血液的DNA我们发现六种cpg的DNAm水平PCBD2 / MTND4P12可能是早期ccRCC检测的潜在生物标志物,但作为生物标志物的价值需要在未来的研究中进行研究。
背景
肾细胞癌(RCC)涉及肾实质恶性细胞的发展。RCC占所有癌症的4%和肾癌的80%,是全球第14种最常见的癌症[1].大多数rcc是在健康检查和其他疾病(如高血压、糖尿病和肥胖症)检查时偶然发现的。RCC在男性和女性中的发病率之比为1.7-2.0:1 [2].70%以上的RCC诊断为透明细胞RCC (ccRCC) [3.].早期RCC的总体5年生存率约为93%,而转移性RCC患者的5年生存率仅为12% [4].因此,早期诊断RCC对患者生存至关重要。然而,由于RCC早期没有主要症状,也没有有效的生物标志物,早期检测依赖于计算机断层扫描和超声检查。
11种生殖系突变(在BAP1,FLCN,跳频,见过,PTEN,SDHB,SDHC,SDHD,TSC1,TSC2,VHL)均可导致遗传性肾癌[5,6].尽管在全基因组关联研究和荟萃分析中已经确定了18个rcc相关的单核苷酸多态性[7,8,9,10,11,12],只有3-5%的RCC可以用遗传背景解释,具有遗传易感性的人在年轻时就会发生RCC。相比之下,大多数rcc在50岁后发病频率增加,通常可归因于吸烟、饮酒和肥胖等非遗传因素。近年来,不同组织学定义的RCC亚型(例如,ccRCC和CpG岛甲基化表型-RCC (cmp -RCC))表现出特征性突变、染色体拷贝数变化以及mRNA、miRNA和lncRNA表达模式[13,14].
生物标记物检测必须简单且微创。从这个角度来看,那些使用在医学检查中收集的全血或血浆的人是最合适的。DNA甲基化(DNAm)是一种基因表达的调节因子,作为多种疾病(包括癌症)的潜在生物标志物而引起了人们的关注[15].虽然使用肾组织来源DNA的微阵列DNAm分析已经鉴定出与肾细胞癌相关的几种DNAm生物标志物[16,17,18],关于血液来源的DNAm生物标志物的报道很少。在全面的DNAm分析和表观基因组全关联研究(EWAS)中,微阵列因其高通量和低成本而得到广泛应用。癌症基因组图谱(TCGA)中总结的大部分DNAm数据[19]及EWAS数据中心[20.]来源于微阵列分析。然而,通过微阵列分析(如使用Infinium MethylationEPIC微阵列[21])在整个基因组中仅占CpGs的3-3.5%,这表明新的DNAm生物标志物可能隐藏在其他CpGs中,而没有在微阵列上检测到。因此,我们使用基于测序的方法探索了环境暴露和疾病相关的DNAm生物标志物[22,23,24,25,它们覆盖了大量的cpg。
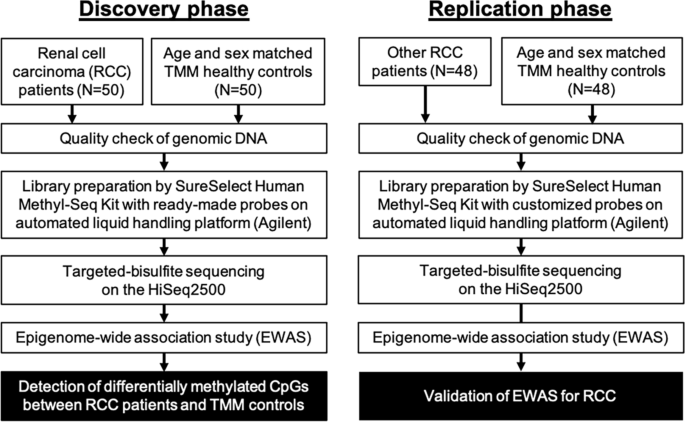
在本研究中,为了识别基于全血的新型DNAm生物标志物用于检测ccRCC,作为RCC的主要类型,我们进行了靶向亚硫酸氢盐测序(TB-seq),其成本低于全基因组亚硫酸氢盐测序(WGBS)。我们使用来自ccRCC患者的全血DNA和匹配的健康对照,并对ccRCC进行EWAS。此外,为了验证确定的全血DNAm生物标志物用于ccRCC的准确性,在另一组独立的ccRCC患者和对照组中使用全血DNA进行DNAm分析和EWAS。
结果
参与者特征
总体研究设计如图所示。1.本研究的个体特征如表所示1.在发现阶段,显著差异(pccRCC组与对照组糖化血红蛋白(HbA1c)、总胆固醇(TC)、血脂异常或糖尿病患者数量均< 0.05)差异均有统计学意义(p < 0.05)。相比之下,复制阶段显示出显著差异(p在平均估计肾小球滤过率(eGFR)、糖化血红蛋白(HbA1c)、高密度脂蛋白胆固醇(HDL)和慢性肾脏疾病患者人数方面,ccRCC组与对照组的差异< 0.05)。在本研究的参与者中,处于发现阶段的ccRCC患者在国家癌症中心(NCC)医院接受了手术;因此,病理信息,如癌症分期和肿瘤大小是可用的。然而,25%的ccRCC患者在复制期因在其他医院接受手术而难以获得病理信息(表2)1).发现组每个癌症阶段的肿瘤大小±SD平均值如下:I期,3.8±1.2 cm;II期,7.9±0.8 cm;III期,7.2±3.3 cm;IV期,7.7±3.7 cm。
表观全基因组关联研究
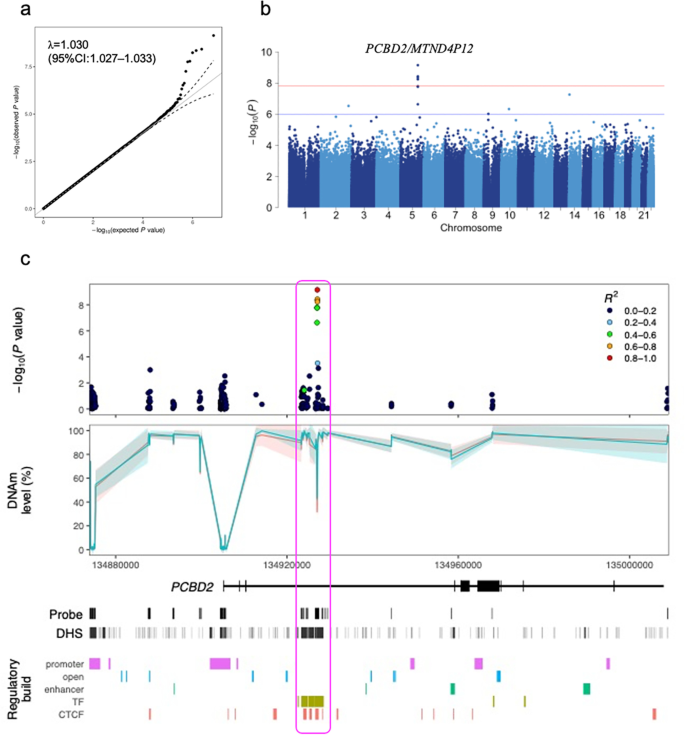
为了鉴定与ccRCC相关的差异甲基化CpGs,我们使用线性回归模型进行了表观基因组范围的关联分析。该模型对性别、年龄和细胞类型组成进行了很好的校正,膨胀因子(λ值)为1.030(95%置信区间(CI): 1.027-1.033)(图2)。2一个)。
发现阶段EWAS结果总结。一个分位数-分位数(QQ)图。观察(y-轴)与预期(x设在)p-值使用线性回归模型。的λ价值(通货膨胀因子)由观察值与预期值的中位数估算p值。b曼哈顿的阴谋。的负对数p值(日志10(p-value))和染色体位置。全基因组显著性阈值(红线):Bonferroni校正p< 1.59 × 10−8.建议阈值(蓝线):p< 1.00 × 10−6.cDNAm水平与基因调控元件的位点变焦图PCBD2 / MTND4P12基因。6个ccrcc相关DNAm标记周围的Chr5:134923256-134928594区域(洋红色线的平方区域)分布有DNase I超敏位点(DHS)、转录因子结合位点、CCCTC结合因子结合位点。CTCF, cctc结合因子;DHS, DNase I高敏感位点;TF,转录因子结合位点;EWAS,表观基因组全关联研究;DNAm, DNA甲基化
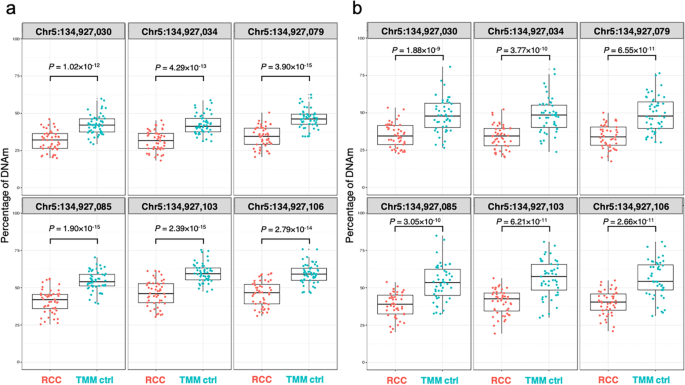
在发现阶段,EWAS结果显示有11个cpg与p-低于建议阈值的值(p< 1.00 × 10−6),其中6项低于显著性阈值(p< 1.59 × 10−8).6个cpg位于两个pterin-4 -甲醇胺脱水酶2而且MTND4P12(线粒体编码的NADH:泛醌氧化还原酶核心亚基4假基因12)5号染色体上的基因(图;2b和附加文件1:表S1)。6个ccrcc相关DNAm标记周围的Chr5:134923256-134928594区域密集分布着DNase I超敏位点(DHS)、转录因子结合位点(TFBS)和cctc -结合因子(CTCF)结合位点(图5)。2c).与Tohoku Medical Megabank (TMM)组(健康对照组)相比,ccRCC组的6个CpGs的甲基化程度约低10%,这表明这些CpGs与ccRCC相关(图2)。3.a).参考区间(RI),定义为个体间DNAm水平的变化[22],在TMM组中,所有6个cpg均大于30%,这表明cpg在个体之间的DNAm水平存在差异(附加文件1:表S1)。此外,为了确定EWAS的结果是否受到参与者特征的显著差异的影响(p< 0.05;HbA1c、TC、血脂异常和糖尿病),将这些特征添加到矫正期进行EWAS。结果表明,6种与ccrcc相关的CpGs在PCBD2 / MTNT4P12基因校正后仍与ccRCC相关(p< 0.05;额外的文件1:表S2)。
在复制阶段,p来自ccRCC EWAS的-值没有完全校正性别、年龄和细胞类型组成,λ值计算为1.109 (95% CI: 1.104-1.112),表明有轻微膨胀(附加文件)2:图S1a)。EWAS结果显示相同的6个cpgPCBD2 / MTND4P12低于提示阈值;6个cpg中有4个低于显著性阈值(p< 3.42 × 10−8)(附加文件1:表S3和附加文件2:图S1b)。与发现阶段一样,与TMM组相比,ccRCC组中6个CpGs的甲基化减少了约10%(图2)。3.b), RI大于30%(补充文件1:表S3)。此外,为了确定EWAS的结果是否受到参与者特征的显著差异的影响(p< 0.05;eGFR, HbA1c, HDL和慢性肾脏疾病),进行EWAS,将这些特征添加到校正期。结果表明,6种与ccrcc相关的CpGs在PCBD2 / MTNT4P12基因校正后仍与ccRCC相关(p< 0.05;额外的文件1:表S4)。
为了研究ccrc相关DNAm标记物中DNAm水平与癌症分期之间的趋势,我们进行了Jonckheere-Terpstra趋势检验。在对照组和所有阶段的ccRCC中,DNAm水平有显著差异(附加文件)2:无花果。S2和S3)。但在发现期和复制期,肿瘤分期间均无变化趋势。
独联体-表达数量性状甲基化(独联体-eQTM)分析
在基于血液的多组学数据库iMETHYL中,我们发现有56个注释基因位于其上游或下游的100万个碱基对范围内PCBD2 / MTND4P12.表达水平与6种CpGs中DNAm水平相关的基因PCBD2 / MTND4P12由独联体-eQTM分析;T细胞特异性转录因子7 (TCF7)和电压依赖性阴离子选择通道蛋白1 (VDAC1)均显著相关(表2).TCF7而且VDAC1分别位于上游80万和90万个碱基对PCBD2 / MTND4P12,分别。eQTM结果显示,5号染色体134927085位点CpG的高甲基化与基因表达增加显著相关TCF75号染色体134927106位点CpG的高甲基化与基因表达降低有关VDAC1(表2).换句话说,ccRCC中CpGs的低甲基化与基因表达的降低有关TCF7基因表达增加VDAC1.与此同时,独联体-eQTM分析表明,6种CpGs的DNAm水平对基因表达水平影响不大PCBD2/MTND4P12(附加文件1:表S5)。
受试者工作曲线下面积(AUC-ROC)分析
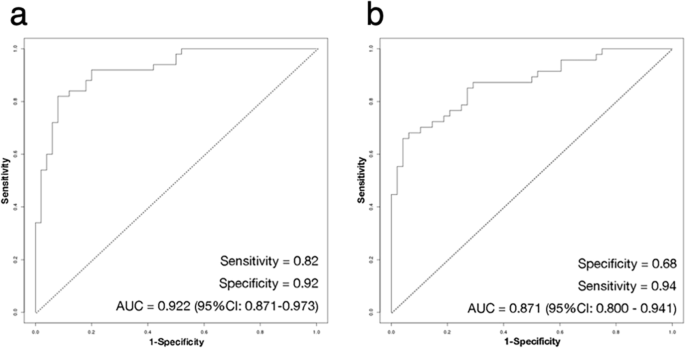
为了研究确定的六种CpGs是否是ccRCC检测的有效DNAm生物标志物,我们使用六种CpGs中的每一种的DNAm水平及其DNAm水平之和进行了ROC曲线分析。在发现阶段,6个CpGs的DNAm水平均高到足以检测出ccRCC,但6个CpGs的DNAm水平之和显示出最好的ccRCC检测(AUC-ROC = 0.922)(表3.和无花果。4a).此外,当考虑6个cpg的DNAm水平之和时,复制阶段的ccRCC检测能力略低于发现阶段,但足以可靠(AUC-ROC = 0.871)(表3.和无花果。4b)。
讨论
使用微创筛查早期发现ccRCC对患者的治疗和生存非常重要。在本研究中,我们确定了6个cpg位于PCBD2 / MTND4P12首次使用TB-seq和EWAS作为与ccRCC相关的全血DNAm生物标志物。我们的研究结果不仅有助于早期发现和治疗ccRCC,而且还证明了基于测序的DNAm分析在寻找新型癌症相关DNAm生物标志物方面的有效性。
本研究中发现的含有ccrcc相关CpGs的DNA区域之前没有被确定为具有ccrcc相关CpGs,因为它没有用于常见的商业DNAm微阵列。此外,因为在人类基因组中有多个区域与PCBD2 / MTND4P12该区域的特异性PCR引物设计较为困难。然而,通过使用特定的探针可以杂交到PCBD2 / MTND4P12TB-seq分析能够避免传统PCR无法扩增该区域的问题。大多数先前报道的全基因组DNAm分析都是使用微阵列技术进行的。微阵列分析可以检测加载在微阵列上的cpg的DNAm水平,但不能检测未加载的cpg。换句话说,很难考虑DNAm分析的结果,包括位于微阵列加载的cpg周围但未加载在微阵列上的cpg的DNAm水平。在之前报道的癌症DNAm生物标志物中,只有0.8%得到了验证,而通过微阵列分析识别的DNAm生物标志物的大量假阳性是有问题的[15].相比之下,基于测序的分析可以将DNAm标记作为“区域”检测,这与单个CpG位点相比,提高了生物标记的鉴别能力。未来基于测序的DNAm分析和EWAS在cccc以及各种其他疾病中的应用将允许识别新的DNAm标记,以早期发现这些疾病。
6个ccrcc相关CpGs所在区域的基因组结构包括TFBS和ctcf结合位点,提示该区域DNAm水平的变化会影响染色质结构,进而影响附近的基因表达。事实上,iMETHYL数据库的eQTM结果显示PCBD2 / MTND4P12影响基因表达VDAC1而且TCF7,它们位于上游的80 - 90万个碱基对。VDAC蛋白位于血浆和线粒体外膜上,与腺嘌呤核苷酸的转运有关[26]、钙离子[27],以及若干代谢产物[28]进出线粒体和细胞。在正常凋亡刺激下,VDAC1与促凋亡蛋白Bax和其他VDAC分子相互作用,通过促进细胞色素c从线粒体释放到细胞质来寡聚和抑制凋亡。过度的VDAC1已在几种癌细胞系中观察到,并被认为是癌细胞线粒体异常的重要促成因素,包括ccRCC [29,30.,31].目前的研究支持这些结果。然而,本研究并没有直接考察VDAC1基因在ccRCC患者血细胞中的表达,这应该在未来进行检查。
与此同时,TCF7本研究发现的另一个eQTM基因,编码转录因子T细胞因子-1 (TCF1),该基因具有DNA结合和组蛋白脱乙酰酶(HDAC)活性,在包括先天淋巴细胞和T细胞在内的多种淋巴系的发育中发挥重要作用。TCF1 (TCF7)与转录因子LEF1一起参与cd8阳性T细胞的命运[32,33].尽管过度表达TCF1(TCF7)已在几种癌症类型的肿瘤组织中报道,包括ccRCC [34,35,36的表达式配置文件TCF1(TCF7)基因在癌症患者的血细胞中很少有报道,目前细节尚不清楚。虽然这是一项数据驱动的eQTM分析,但这项研究为TCF1 (TCF7)提供了新的见解,因为据我们所知,这是第一次显示6个CpGs的低甲基化在中国PCBD2 / MTND4P12与减少有关TCF1(TCF7基因表达(表2及其他资料1:表S5)。但是,由于我们没有分析表达式TCF1(TCF7)基因在本研究中对ccRCC患者血液细胞中的基因进行了分析,今后有必要进行这样的分析。
本研究的主要局限性在于其主要针对日本患者。目前尚不清楚ccrcc相关的CpGs是否在其他种族群体中也可检测到,有必要验证已识别的ccrcc相关DNAm标记在其他种族群体中的有效性。此外,本研究仅调查了ccRCC;因此,不可能区分识别出的与ccrcc相关的cpg是ccrcc特异性的还是其他癌症常见的。为了澄清这一点,应该对其他类型的癌症进行基于测序的DNAm分析和EWAS。虽然本研究发现了一种基于血液的DNAm标志物用于检测ccRCC,但血细胞DNAm水平的变化并不能解释ccRCC的病因。因此,需要进一步的研究来揭示为什么低甲基化PCBD2 / MTND4P12发生在ccRCC患者的血细胞DNA中。
结论
我们鉴定出6个低甲基化cpgPCBD2 / MTND4P12作为ccRCC的DNAm生物标志物。此外,我们发现这6种cpg的DNAm水平之和可以更准确地检测ccRCC。鉴定出的CpGs是新的DNAm生物标志物,可能在ccRCC诊断中有用。本研究的结果为早期发现和治疗ccRCC以及ccRCC治疗方法的发展提供了新的选择。
方法
道德
本研究由国立癌症中心(日本东京)、庆应义塾大学和岩手医科大学伦理委员会批准(批准ID: HG H25-19)。所有实验均按照批准指南进行。所有参与者提供书面知情同意书。
研究参与者和样本收集
这项研究分为两个阶段:发现和复制。在发现阶段,50名ccRCC患者的全血DNA由NCC(东京,日本)提供,50名性别和年龄匹配的健康对照者的全血DNA由Tohoku Medical Megabank社区队列研究(TMM CommCohort)提供[37].在复制阶段,NCC和TMM CommCohort分别为ccRCC和对照样本提供了48个个体的全血DNA(与发现阶段无关)。所有血液样本均在EDTA采血管中采集,NCC样本使用Gentra Puregene blood Kit, TMM CommCohort样本使用QIAGEN Autopure LS纯化血源DNA。ccRCC通过影像学(MRI或CT)以及熟练的病理学家的显微和大体观察进行诊断。在两个阶段中,ccRCC组和TMM组样本在±2年内进行年龄匹配,并尽可能匹配身体质量指数(BMI)。使用个人健康检查和自我报告问卷来确定吸烟状况、饮酒状况和流行疾病(即慢性肾脏疾病、高血压、血脂异常和糖尿病)。使用配对t检验(如实验室值)和卡方检验(如疾病流行率)对参与者特征进行显著性检验。
测序文库和TB-seq的制备
基因组DNA等分(gDNA;1 μg),在50 μL TE缓冲液中洗脱,使用Covaris LE220聚焦超声仪(thermofisher Scientific, Waltham, MA, USA)剪切成150-200 bp片段。TB-seq测序文库使用Agilent SureSelect Human Methyl-Seq捕获文库和试剂盒在Agilent Bravo自动化文库制备系统(Agilent Technologies, Santa Clara, CA, USA)上按照制造商说明制备。在复制阶段,我们使用安捷伦SureSelect Human Methyl-Seq自定义捕获试剂盒和定制探针(即常见的DNA甲基化变异(CDMV) [22]探测集)。使用EZ DNA甲基化金试剂盒(Zymo Research, Irvine, CA, USA)对所有测序文库进行亚硫酸氢盐处理。将合并的17-pM文库加入20% PhiX Control v3 (Illumina Inc., San Diego, CA, USA),并在HiSeq 2500系统(Illumina)上进行配对端测序(2 × 125 bp)。
靶向CpGs的DNA甲基化分析
使用Illumina bcl2fastq2转换软件v2.20将原始测序数据转换为FASTQ格式。使用FastQC软件v0.11.5评估原始数据的测序质量,使用Trim Galore软件v0.4.2修剪适配器,删除短读(< 20 bp)。其余的读数与基因组参考联盟人类参考38 (GRCh38)构建一致,从UCSC基因组浏览器网站下载[38],使用Novoalign软件v3.6.5。如先前报道的那样,使用生物信息学工具处理对齐的数据[25].使用NovoMethyl软件v1.4检测甲基化CpGs,使用R软件v3.3.1计算目标CpGs的甲基化水平。
表观全基因组关联研究
使用线性回归模型进行EWAS,以确定与ccRCC相关的差异甲基化CpGs。分析根据年龄、性别和估计的细胞类型组成进行了调整。使用estimateCellCounts估算细胞类型组成。minfi Bioconductor封装中的R功能[39,40作了修改。具体来说,不是使用Illumina Infinium HumanMethylation450数据在flowsort . blood中实现的分类血细胞群。在Bioconductor的450 k包中,我们参考了来自6个分类的白细胞群体(B细胞,CD4细胞,CD4细胞)的DNAm数据+T淋巴细胞,CD8+T淋巴细胞,单核细胞,NK细胞和中性粒细胞)来自12个个体的全基因组亚硫酸氢盐测序[22,23],并选择每种细胞类型中显示高甲基化和低甲基化CpGs的前50个CpGs进行进一步分析。所有分析均在相同的条件下进行,全基因组提示阈值定义为p< 1.00 × 10−6;但是,bonferroni校正显著性阈值设置为p< 1.59 × 10−8(0.05/3145479)在发现阶段和p< 3.42 × 10−8(0.05/1460699)。本研究中使用的统计分析脚本可在我们的GitHub网站(https://github.com/H-Ohmomo/ccRCC_EWASscript_20220323).
通过绘制受试者工作特征(ROC)曲线,评估识别的CpGs作为全血DNAm生物标志物对ccRCC的预测准确性。特异性、敏感性和曲线下面积(AUC)根据DNAm水平使用ROCR计算[41]和pROC [42)包。
为了检查ccRCC和ccRCC癌症阶段显著不同甲基化CpGs的DNAm水平之间的趋势,我们使用DescTools包进行了Jonckheere-Terpstra趋势测试[43有统计学意义的差异定义为P趋势< 0.05。
定量性状甲基化分析
为了评估ccrcc相关的CpGs与基因表达之间的关系,我们进行了独联体-eQTM分析,采用简单线性回归模型,使用iMETHYL数据库[44].表达式级别[log .10(FPKM + 0.1)]为因变量,各ccrcc相关CpG位点的DNAm水平为自变量。邻近地区(独联体)定义为ccrcc相关cpg上游或下游100万碱基对内的区域。
数据和材料的可用性
为了保护参与者的隐私,每个人的DNA甲基化信息不能公开。不过,DNA甲基化信息的汇总统计数据将在iMETHYL数据库(http://imethyl.iwate-megabank.org)。
缩写
- AUC-ROC:
-
接收机工作特性曲线下面积
- ccRCC:
-
透明细胞肾细胞癌
- CTCF:
-
CCCTC-binding因素
- CDMV:
-
常见的DNA甲基化变异
- 装备:
-
染色体
- CIMP-RCC:
-
CpG岛甲基化表型- rcc
- 论文认定:
-
胞嘧啶,鸟嘌呤,核苷酸
- CT:
-
计算机断层扫描
- DNAm:
-
DNA甲基化
- 表皮生长因子受体:
-
估计肾小球滤过率
- eQTM:
-
表达数量性状甲基化
- ewa:
-
表观全基因组关联研究
- FPKM:
-
每百万外显子每千碱基的片段数
- gDNA:
-
基因组DNA
- GRCh38:
-
基因组参考联盟人类参考文献38
- 核磁共振成像:
-
磁共振成像
- 碾压混凝土:
-
肾细胞癌
- 国际扶轮:
-
参考区间
- TB-Seq:
-
Targeted-bisulfite测序
- TFBS:
-
转录因子结合位点
- TMM CommCohort:
-
东北医疗megank社区队列
- WGBS:
-
亚硫酸氢盐全基因组测序
参考文献
国际癌症研究机构。全球癌症观察(GCO)。http://gco.iarc.fr/.2021年11月10日访问。
Vincent T Jr, DeVita TS, SAR L. DeVita, Hellman和Rosenberg的癌症:肿瘤学原理与实践(肿瘤学原理与实践)。第11版。沃尔特斯·克鲁威;2019.
Moch H, Cubilla AL, Humphrey PA,路透VE, Ulbright TM。2016年世界卫生组织泌尿系统和男性生殖器官肿瘤分类——a部分:肾脏、阴茎和睾丸肿瘤。中国生物医学工程学报,2016;29(1):344 - 344。https://doi.org/10.1016/j.eururo.2016.02.029.
美国癌症协会。2017年癌症事实与数据。亚特兰大,乔治亚州。https://www.cancer.org/research/cancer-facts-statistics/all-cancer-facts-figures/cancer-facts-figures-2017.html.2021年11月10日访问。
李志刚,李志刚,李志刚。肾癌的遗传基础:一种代谢性疾病。自然科学进展。2010;7(5):277-85。https://doi.org/10.1038/nrurol.2010.47.
Haas NB, Nathanson KL.遗传性肾癌综合征。中华慢性肾病杂志2014;21(1):81-90。https://doi.org/10.1053/j.ackd.2013.10.001.
普渡议员,Johansson M, Zelenika D,等。肾细胞癌全基因组关联研究发现2p21和11q13上有两个易感位点。3.中国科学。2011;43(1):60-5。https://doi.org/10.1038/ng.723.
吴霞,谢志刚,普渡MP,等。一项全基因组关联研究发现了一种新的肾细胞易感性位点。胡晓明。2012;21(2):456-62。https://doi.org/10.1093/hmg/ddr479.
王志强,王志强,等。一个常见的8q24.21变异与肾细胞癌相关。Nat Commun. 2013;4:2776。https://doi.org/10.1038/ncomms3776.
李国强,李国强,等。2q22.3 (ZEB2)常见变异影响肾癌风险。中国生物医学工程学报。2013;22(4):825-31。https://doi.org/10.1093/hmg/dds489.
Henrion MYR, Purdue MP, Scelo G, Broderick P. 1q21 .1常见变异(ALDH9A1)是肾癌的潜在危险因素。公共科学学报。2015;10(3):e0122589。https://doi.org/10.1371/journal.pone.0122589.
张志强,李志强,张志强,等。12p12.1肾癌易感性位点的功能特征与BHLHE41有关。Nat Commun. 2016;7:12098。https://doi.org/10.1038/ncomms12098.
李国强,田勇,等。临床病理侵袭性CpG岛甲基化表型透明细胞肾细胞癌中梭形检查点通路的改变。中华肿瘤学杂志,2015;37(11):589 - 596。https://doi.org/10.1002/ijc.29630.
Ricketts CJ, De Cubas AA, Fan H,等。肾细胞癌的肿瘤基因组图谱综合分子特征。细胞代表2018;23:313-326.e5。https://doi.org/10.1016/j.celrep.2018.03.075.
张志强,张志强,张志强,等。癌症中DNA甲基化的分析:重新定位。中华儿科杂志,2018;15(7):459-66。https://doi.org/10.1038/s41571-018-0004-4.
李志刚,李志刚,李志刚,等。基于阵列比较基因组杂交的透明细胞肾细胞癌的遗传聚类:其与DNA甲基化改变和患者结局的关系。临床癌症杂志,2008;14(17):5531-9。https://doi.org/10.1158/1078-0432.CCR-08-0443.
李志强,李志强,李志强,等。单CpG分辨率甲基组分析鉴定临床病理侵袭性CpG岛甲基化表型透明细胞肾细胞癌。致癌作用。2012;33(8):1487 - 93。https://doi.org/10.1093/carcin/bgs177.
李志强,李志强,李志强,等。透明细胞肾细胞癌的综合分子特征。大自然。2013;499(7456):43-9。https://doi.org/10.1038/nature12222.
国家癌症研究所癌症基因组学中心。基因组数据共享(GDC)。https://gdc.cancer.gov/.2021年11月10日访问。
国家基因组数据中心数控B. EWAS数据中心。https://bigd.big.ac.cn/ewas/datahub/index.2021年11月10日访问。
李国强,李国强,等。Illumina MethylationEPIC串珠芯片微阵列用于全基因组DNA甲基化分析的关键评估。中国生物工程学报。2016;17(1):208。https://doi.org/10.1186/s13059-016-1066-1.
田八也,古川R,石和Y,等。个体间可变DNA甲基化位点的全基因组鉴定提高了表观遗传关联研究的有效性。NPJ基因医学。2017;2:11。https://doi.org/10.1038/s41525-017-0016-5.
李志强,李志强,李志强,等。一项基于细胞类型特异性全基因组亚硫酸氢盐测序的表观基因组范围关联研究:筛选与骨量相关的DNA甲基化特征。中华医学杂志,2017;4(5):1-7。https://doi.org/10.15761/imm.1000307.
李志强,李志强,等。表观基因组全关联研究在严重主动脉瓣狭窄患者中发现了一种新的DNA甲基化。中国生物医学杂志2020;13(1):e002649。https://doi.org/10.1161/CIRCGEN.119.002649.
王晓明,王志强,等。用于定向亚硫酸氢盐测序的临床福尔马林固定石蜡包埋组织质量评价。中华病毒学杂志,2011;21(2):344 - 344。https://doi.org/10.1111/pin.13054.
Rostovtseva T, Colombini M. ATP通量由线粒体外膜的电压门控通道控制。中国生物医学工程学报。1996;21(4):339 - 339。https://doi.org/10.1074/jbc.271.45.28006.
金泽华,李志强,李志强,等。钙离子离子通道在线粒体功能调控中的作用机制。中国生物化学杂志,2001;29(1):457 - 457。https://doi.org/10.1042/0264-6021:3580147.
霍奇T, Colombini .通过VDAC通道电压门控调节代谢物通量。中国生物医学工程学报。1997;37(3):379 - 379。https://doi.org/10.1007/s002329900235.
Shinohara Y, Ishida T, Hino M, Yamazaki N, Baba Y, Terada H.肿瘤细胞中孔蛋白异构体表达的特征。中国生物医学工程学报,2000;29(4):344 - 344。https://doi.org/10.1046/j.1432-1327.2000.01687.x.
Grills C, Jithesh PV, Blayney J, Zhang SD, Fennell DA。基因表达元分析发现VDAC1是早期非小细胞肺癌预后不良的预测因子。公共科学学报,2011;6(1):e14635。https://doi.org/10.1371/journal.pone.0014635.
王晓明,王晓明,王晓明,等。透明细胞肾细胞癌患者纤毛发生和VDAC1- Δ C缺失驱动的新侵袭轴的鉴定。开展。2020;10(6):2696 - 713。https://doi.org/10.7150/thno.41001.
邢松,李峰,曾忠,等。Tcf1和Lef1转录因子通过内在的HDAC活性建立CD8(+) T细胞身份。中华免疫杂志,2016;17(6):695-703。https://doi.org/10.1038/ni.3456.
拉古D,薛洪辉,米尔克LA。TCF-1对淋巴细胞命运、感染和肿瘤免疫的控制免疫杂志2019;40(12):1149-62。https://doi.org/10.1016/j.it.2019.10.006.
李志强,李志强,等。透明细胞肾细胞癌中分泌卷曲相关蛋白1、3、t细胞因子1和淋巴样增强因子1的表达病原生物学杂志,2013;19(3):545-51。https://doi.org/10.1007/s12253-013-9615-3.
刘徐X, Z,田F,徐J,陈y转录因子7的临床意义(TCF7)作为胃癌预后因素。医学科学,2019;25:39 - 63。https://doi.org/10.12659/MSM.913913.
詹艳,冯娟,陆娟,徐玲,王伟,樊松。LEF1和TCF1 (TCF7)蛋白表达与鼻咽癌临床进展相关。中华临床病理学杂志2019;72:425-30。https://doi.org/10.1136/jclinpath-2019-205698.
肖泽,田野,中谷,等。东北医疗megank社区队列研究的研究概况。中华流行病学杂志,2021;31(1):65-76。https://doi.org/10.2188/jea.JE20190271.
CAC基因组学研究所。UCSC基因组浏览器。https://hgdownload.soe.ucsc.edu/downloads.html.2021年11月10日访问。
Houseman EA, Accomando WP, Koestler DC,等。DNA甲基化阵列作为细胞混合物分布的替代测量方法。生物信息学。2012;13:86。https://doi.org/10.1186/1471-2105-13-86.
Link C, Jaffe AE, Irizarry RA。在全表观基因组关联研究中,解释细胞异质性至关重要哈佛社区公开了这篇文章。解释细胞异质性在表观基因组全关联研究中至关重要。中国生物医学工程学报。2014;15(2):R31。https://doi.org/10.1186/gb-2014-15-2-r31.
王志强,王志强,王志强,等。基于分类器的生物信息学研究。2005;21(20):366 - 366。https://doi.org/10.1093/bioinformatics/bti623.
Robin X, Turck N, Vutskits L等人pROC:用于R和S+分析和比较ROC曲线的开源包。BMC生物信息学。2011;12:77。https://doi.org/10.1186/1471-2105-12-77.
DescTools:描述性统计工具。https://cran.r-project.org/web/packages/DescTools/.2022年5月23日访问。
Komaki S, Shiwa Y, Furukawa R等。iMETHYL:人类DNA甲基化、基因表达和基因组变异的综合数据库。Hum Genome Var. 2018;5:18008。https://doi.org/10.1038/hgv.2018.8.
确认
作者感谢Kumi Furusawa和Miyuki Horie在本研究中对实验的帮助。我们要向东北医疗megank社区队列(TMM CommCohort)研究的所有参与者表示最深切的感谢。我们也要感谢岩手医科大学的岩手东北医学大银行组织成员和东北大学的东北医学大银行组织成员的支持和鼓励。
资金
这项研究得到了日本医学研究开发机构(AMED;授予号JP17km0105003和JP17km0105004)。
作者信息
作者及隶属关系
贡献
概念和设计:HO, EA, YK, AS。数据获取:HO, SK, KO。数据分析与解释:HO, SK, YS, TH, KA, AS。稿件的撰写、评审和/或修改:所有作者。行政、技术或物质支持:EA、HF、TY和YK。研究指导:MS和AS。作者阅读并批准最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
本研究由国立癌症中心(日本东京)、庆应义塾大学和岩手医科大学伦理委员会批准(批准ID: HG H25-19)。所有参与者提供书面知情同意书。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:表S1。
全血差异甲基化CpGs (p< 1.00 × 10−5)在发现阶段,ccRCC患者与健康对照组之间的差异。空空的,染色体;透明细胞肾细胞癌;TSS,转录起始位点;CR,拆借利率;RI,参考区间;系数,系数;SE,标准误差;RSQ平方。表S2。在发现阶段,ccRCC的CpGs显著甲基化差异受年龄、性别、细胞类型组成、HbA1c、总胆固醇、血脂异常和糖尿病的调整。空空的,染色体;透明细胞肾细胞癌;CR,拆借利率;RI,参考区间;系数,系数;SE,标准误差;RSQ平方;糖化血红蛋白。表S3。全血差异甲基化CpGs (p< 1.00 × 10−5)在复制阶段,ccRCC患者与健康对照之间的差异。空空的,染色体;透明细胞肾细胞癌;TSS,转录起始位点;TMM、控制;CR,拆借利率;CI,置信区间;RI,参考区间;系数,系数;SE,标准误差; RSQ, R-squared.表S4。在复制阶段,ccRCC的CpGs甲基化显著差异受年龄、性别、细胞类型组成、HbA1c、总胆固醇、血脂异常和糖尿病的调节。空空的,染色体;透明细胞肾细胞癌;CR,拆借利率;RI,参考区间;系数,系数;SE,标准误差;RSQ平方;糖化血红蛋白。表S5。基于血液的结果独联体-eQTM分析(p< 0.05)PCBD2 / MTND4P12使用iMETHYL数据库。
补充文件2:补充图S1。
复制阶段EWAS结果总结。A,观测值(y轴)与预期值(x轴)p-值使用线性回归模型。λ值(膨胀因子)由观测值与预期值的中位数估计p值。B,曼哈顿地块。的负对数p值(日志10(p-value))和染色体位置。全基因组显著性阈值(红线):Bonferroni校正p< 3.42 × 108.建议阈值(蓝线):p< 1.00 × 106.EWAS,表观基因组全关联研究。补充图S2。六种cpg的DNAm水平及其在发现阶段健康对照与ccRCC癌症分期之间的DNAm水平之和的比较。一个,染色体(Chr) 5,位置134927030;B, Chr 5,位置134927034;C, Chr5,位置134927079;D, Chr5,位置134927085;E, Chr5,位置134927103;F, Chr5,位置134927106;G,六个cpg中DNAm水平的总和。P趋势值采用Jonckheere-Terpstra趋势检验获得。透明细胞肾细胞癌;DNAm, DNA甲基化。补充图S3。比较六种cpg的DNAm水平及其在健康对照组和复制期ccRCC癌症分期之间的DNAm水平之和。A,染色体(Chr) 5,位置134927030;B, Chr 5,位置134927034;C, Chr 5,位置134927079;D, Chr 5,位置134927085;E, Chr 5,位置134927103;F, Chr 5,位置134927106;G,六个cpg中DNAm水平的总和。P趋势值采用Jonckheere-Terpstra趋势检验获得。透明细胞肾细胞癌;DNAm, DNA甲基化。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
大模,H.,小木,S.,须藤,Y.。et al。通过全血表观基因组全关联研究,发现透明细胞肾细胞癌检测的潜在DNA甲基化生物标志物。表观遗传学Commun。2, 2(2022)。https://doi.org/10.1186/s43682-022-00009-7
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s43682-022-00009-7
关键字
- 透明细胞肾细胞癌
- DNA甲基化生物标志物
- 亚硫酸氢盐定向测序
- 表观全基因组关联研究
- 整个blood-based