摘要
背景
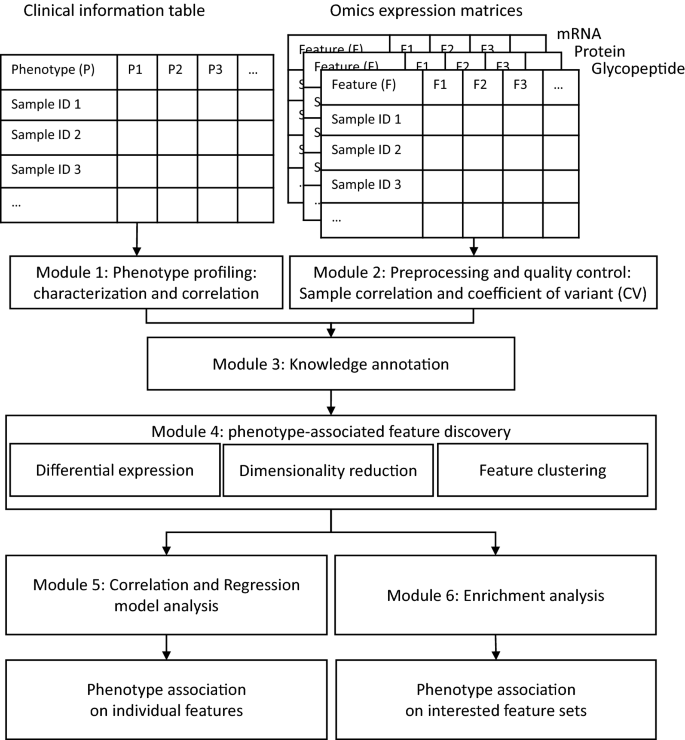
高通量“组学”技术的快速发展,在实验期间和实验结束后带来了大量的数据进行处理。多组学分析有助于对数据集进行更深入的查询,并发现有趣的基因、蛋白质、脂质、聚糖、代谢物或与研究中相应表型相关的途径。许多单独的软件工具已经开发用于数据分析和可视化。然而,目前还缺乏一种利用多组学数据研究表型的有效方法。在这里,我们将OmicsOne作为一个基于web的交互式框架,通过“一键式”集成质量控制、统计分析和交互式数据可视化,用于多组学数据的快速表型关联分析。
材料与方法
将OmicsOne应用于先前发表的高级别浆液性卵巢癌(HGSOC)的蛋白质组学和糖蛋白组学数据集以及肺鳞状细胞癌(LSCC)的蛋白质组学数据集,以证实其性能。通过OmicsOne实现的六个主要功能模块对数据进行分析:(1)表型分析,(2)数据预处理和质量控制,(3)知识标注,(4)表型相关特征发现,(5)个体特征表型关联分析的相关和回归模型分析,(6)感兴趣特征集表型关联分析的富集分析。
结果
我们开发了一个集成的软件解决方案,OmicsOne,用于多组学数据集的表型关联分析。OmicsOne在卵巢癌数据公开数据集上的应用表明,该软件可以一致地证实之前的观察结果,并发现了HNRNPU和HYOU1糖肽作为HGSOC数据集潜在生物标志物的新证据。在LSCC蛋白质组数据集的Tumor和NAT对比研究中进一步证明了OmicsOne的性能。
结论
OmicsOne可以有效简化数据分析,并在几分钟内揭示表型与潜在生物标志物(包括基因、蛋白质和糖肽)之间的显著关联,帮助用户了解异常的生物过程。
背景
表型可定义为由基因、环境、疾病、分子机制和机会相互作用而产生的生物体的任何可观察到的特征或状态[1].基因组学和蛋白质组学在疾病研究中的表型关联分析的目的是阐明蛋白质表达与临床表型之间的关系。随着高通量“组学”技术的进步,包括基因组学、表观基因组学、转录组学、蛋白质组学、蛋白质修饰、糖组学、脂质组学和代谢组学,产生了令人难以置信的大量数据[2,3.,4,5,6,7].可以预见的是,随着新技术的发展和现有方法的进步,生成大型数据集的趋势将继续下去。在这个组学数据爆炸的时代,多组学表型关联分析的自动化解决方案将显著增加疾病(如癌症)研究中大量数据的知识发现。
在过去的几十年里,人们在自动化组学数据分析和可视化的生物信息学工具开发方面做出了许多努力,包括匠心路径分析的商业解决方案[8] (Ingenuity Systems, QIAGEN Inc.)和ProteinCenter (Thermo Scientific/Proxeon)以及非商业工具,包括infernoRDN(前DanTE和DanteR) [9,10], protesign [11,12], GproX [13], DAPAR/ProStaR [14], GiaPronto [15],柏修斯[16], PANDA-view [17],和IOAT [18].开发这些工具是为了对定量发现蛋白质组学实验进行统计分析,其中包括进行数据处理、执行零假设显著性检验、生成定量蛋白质组学数据和其他组学数据的可视化以及以下基因本体论(GO) [19富集分析。然而,所有这些工具都是为组学数据的日常数据分析工作而设计的。自动化表型关联分析仍然存在一些问题。(1)缺乏实用的集成模式。功能模块被分隔在不同的页面中,并没有针对高效的自动化管道进行最佳组织。(2)这些工具缺乏表型研究的支持,以及表型与翻译后修饰(PTMs)之间的关联分析,特别是糖基化。(3)大多数工具仅为定制数据库和脚本以及静态数据可视化提供有限的可扩展性。
为了解决这些问题,我们在这里介绍了OmicsOne工具,这是一个基于Dash框架的Python开发的软件[20.可以在“一键式”模式下对多“组学”数据进行自动表型关联分析。定量表达矩阵和临床信息表是初始化表型关联分析所需的唯一输入。结果以tab分隔的text.txt或.csv文件格式报告,并通过简单的“一键式”按钮在web浏览器的交互式图形界面中可视化。此外,OmicsOne还支持完整糖肽的注释和表型关联分析。蛋白质翻译后修饰(PTMs)在蛋白质和基因表达以及各种细胞机制中起着至关重要的作用,增加了蛋白质组的复杂性和多样性[21,22,23].蛋白糖基化是PTMs最丰富的例子之一[24],因为它是多种生物功能的关键因素,如细胞-细胞识别、细胞-细胞粘附、决定蛋白质结构和参与人类疾病[25,26,27,28,29].由于其与人类疾病的联系,糖基化研究已经允许在改变的糖蛋白和丰富的癌细胞特征之间建立联系[30.].OmicsOne的所有功能模块都支持对符合条件的mRNA、蛋白质和完整糖肽的表达矩阵进行分析,以发现研究中与相应表型相关的有趣分子或通路。OmicsOne还支持交互式数据可视化和可扩展性,可与用户自定义的Python脚本和数据处理管道中的数据库集成,便于对数据集进行更深入的查询。OmicsOne在GitHub上免费提供(https://github.com/huizhanglab-jhu/OmicsOne),并可在Microsoft Windows的Python 3.8环境中本地安装和运行。硬件配置最低要求为2核CPU(如Intel i5-6300U)和12gb内存。
方法
输入文件格式
OmicsOne最初是为等压标记的定量蛋白质组学数据(例如,串联质量标记(TMT))设计的,但可以在无标记定量和数据独立采集(DIA)数据集中找到应用,以及其他“组学”数据,如果数据符合图中所示的输入格式。1.两个样例数据集包含在安装包中,随软件一起安装。在默认设置中,OmicsOne接受以' wide '格式保存的log2转换的表达矩阵,其中样本名称是行索引,特征名称(基因名称或糖肽)是列标签(图2所示)。1).为了兼容完整糖肽分析,完整糖肽的名称,也称为糖形,定义为GeneName_PeptideStartSite_PeptideSequence_GlycositeNumber_GlycositePosition_Glycancomposition。蛋白质的名称就是相应的基因名称。
OmicsOne安装中嵌入的示例数据集也可以在Github存储库中下载。OmicsOne还允许用户在示例文件夹中添加自定义的注释数据库,用于知识注释,路径数据库用于充实分析。
软件模块
我们在Python 3.8下开发了OmicsOne,用于自动化多组学数据分析,以发现与表型相关的分子变化和途径。OmicsOne集成了用于统计计算和数据可视化的科学Python包,包括NumPy(v1.21.4) [32], SciPy(v1.7.1) [33]用于统计计算,使用Pandas(v1.3.4) [34]用于数据表操作,Scikit-learn(v0.24.2) [35GSEApy(v0.10.5) [36,37,38],以及Plotly(v5.3.1)和Dash(v2.0.0) [20.]用于交互式数据可视化和仪表盘构造。经过模块化开发,将所有功能集成为一个高效的分析套件,该套件提供了六个主要功能模块:(1)表型分析,(2)数据预处理和质量控制,(3)知识标注,(4)表型相关特征发现,(5)个体特征表型关联分析的相关和回归模型分析,以及(6)感兴趣特征集表型关联分析的丰富分析(图2)。1).结果被可视化为基于网页的交互式图形。下面几节将详细介绍六个已实现模块。
模块1。表型分析
了解数据队列的组成始终是所有后续表型关联分析研究的第一步和关键步骤。OmicsOne支持对表型信息进行统计,计算不同表型组样本的总体。OmicsOne还将研究表型的成对相关性,以揭示表型之间的依赖关系。
模块2。数据预处理和质量控制
在数据分析之前,通常需要对原始数据进行预处理,以适应算法要求,控制数据质量。OmicsOne提供了几个基本的预处理功能,包括(1)Log-transformation算法,它支持将表达式值转换为log2值。OmicsOne默认接受log2转换的数据。(2)归一化算法。我们实现了常用的中值归一化方法,将所有样本中所有特征的中值调整为相同(默认为0),以减少潜在的批处理效应和测量误差。(3)噪声滤波算法。我们将表达小于50%(用户自定义)样本的特征去除为噪声特征,(4)Imputation算法。在OmicsOne中实现了三种基本的imputation方法,包括:GlobalMin:用缩放的全局最小值来赋值缺失值;SampleMin:在本行中赋值缩放最小值(本样本中所有特征的最小值);而且FeatureMin:在该行中赋值比例最小值(所有样本中该特征的最小值)。
质量控制样本的再现性评价是表型关联分析之前的另一个关键步骤。OmicsOne支持计算技术或生物重复的相关值和所选质量控制样本的变异系数(CV),以估计所测基因或蛋白质水平表达的再现性。
模块3。知识注释
基因注释功能有助于理解生物功能。快速注释工具对于自动化数据分析和人工调查至关重要。在OmicsOne中,特征被自动注释并链接到知识库(例如UniProtKB [39]用于基因和蛋白质注释及n -糖位点图谱[40数据库用于n链糖位点注释)。据我们所知,还没有一个包含糖位点与疾病表型之间联系的具体信息的大规模数据库。因此,该工具有助于将糖肽表型关联分析的结果直接链接到知识库。n -糖位点图谱是一个包含历史上发表的糖位点样本信息的数据库。OmicsOne既提供了基于糖苷图谱的数据库注释功能,也提供了对新发现糖肽的表型关联分析功能。用户还可以添加他们的定制数据库来扩展注释或导出他们与原始糖位点图谱的识别,以供将来的研究。
模块4。表型相关特征的发现
OmicsOne为表型相关的特征发现提供了三个子模块,包括差异表达分析、降维(也称为分解)和特征聚类。
差异表达分析是一种描述特征(如基因、蛋白质和PTMs)的改变表达谱的方法,它在比较研究(如肿瘤与非肿瘤)中提供了对异常生物学的最大见解。在OmicsOne中实现的假设检验(例如t检验和Wilcoxon)算法可以识别显著的差异表达特征,利用配对或独立组的多个统计检验。学生t检验是最常用的统计假设检验,其中检验统计量遵循学生t分布。Wilcoxon秩和检验是一种非参数统计假设检验,用于分别比较两个独立总体的位置[41].对于依赖组,OmicsOne支持相应的配对t检验和Wilcoxon符号秩检验进行比较。结果可以在交互式火山图中直接可视化,以探索测试中涉及的所有特征。在默认设置下,如果有超过1.5倍的变化和小于0.01的调整p值(由Benjamini-Hochberg程序调整],OmicsOne报告为显著变化的特征。42])。中间测试结果将存储为a.csv文件,并为回归和丰富分析提供候选特性。
降维方法是一种有价值和常用的方法,可以在没有先验知识的情况下根据驱动不同表型的最突出因素对样本进行分类,特别是对于具有数千个特征的样本。在一系列降维方法中,主成分分析[43是Python包支持的OmicsOne中实现的最广泛的方法之一:Scikit-learn [35],分离样本,识别与相应样本群相关联的特征基因群。前10个最突出的特征可以在每个主组件中可视化。根据贡献评分选择最显著的特征(默认为100)进行表型关联分析。贡献分数定义为\ (\ sum_ {i = 1} ^ {n} V {R} _{我}* \压裂{{abs (V} _ {ij})} {\ sum_ {j = 1} ^ {m} abs ({V} _ {ij})} \),在那里\ \(米)特征被分解为\ (n \)主部件(pc)、R \ (V{} _{我}\)所解释的方差比是\ (P {C} _{我}\),\ ({V} _ {ij} \)是特征的方差吗\ (j \)有助于\ (P {C} _{我}\).
特征聚类基于Python包Scipy支持的分层聚类[33]以发现在不同表型中具有相似改变模式的基因集。各特征的表达值为聚类前经过z-score变换的交叉样本。用户可自定义集群号。每个表型对应的聚类基因集被导出用于下面的分析。
模块5。个体特征表型关联分析的相关和回归模型分析
OmicsOne提供个体特征的表型关联分析。从差异表达分析、降维和特征聚类方法获得的基因集中涉及的特征可以单独研究表型关联。对与分类表型相关的个体特征进行了相关分析和逻辑回归分析。相关p值< 0.05的特征被认为是表型相关特征。应用于表型和特征表达的逻辑回归模型有助于证明单个特征是否可以被视为表型预测的潜在指标。
模块6对感兴趣的特征集进行表型关联分析的富集分析
基因集可以通过后续的富集分析,使用GSEApy的过度表征分析(ORA)进一步研究[36,37,38]以发现与不同表型状态相关的基因背后丰富的通路。GSEApy是一个用于基因集富集分析(GSEA)的python实现,以及用于富贵的包装器[36,37,38].OmicsOne可以自动回忆GSEApy的功能,对上游分析中选取的显著特征进行富集分析,揭示这些特征所涉及的通路和生物学功能。
结果的交互式数据可视化
OmicsOne以表(.csv或.txt)和所有数据分析的相应交互式图形的形式报告中间结果和最终结果。交互式图形是使用Plotly在Dash框架中生成的,用于直接检查。OmicsOne自动为每个处理步骤的表型关联结果在.csv或.txt(制表符分开)文件中生成中间表。
结果
高级浆液性卵巢癌(HGSOC)的公开蛋白质组学数据集[44]及肺鳞状细胞癌[45]来演示OmicsOne的功能。将表型临床信息表导出为宽格式文件(支持tab-separated.txt或Excel文件),样本名为行下标,表型名为列标签。在表型的每个列标签中添加‘(Categorical)’或‘(Numerical)’标签,以便OmicsOne识别表型的数据类型。
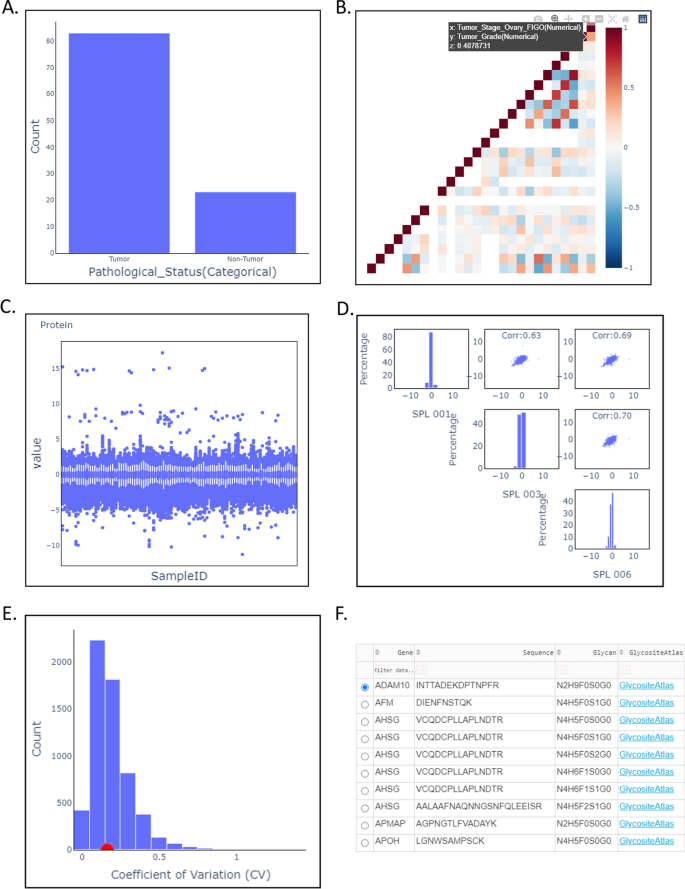
OmicsOne首次应用于HGSOC附加表中的公共蛋白质组学和糖蛋白组学数据集[44]来演示功能。结果显示在附加文件中2:蛋白质组学数据分析表S1及附加文件2:糖蛋白组数据分析见表S2。表型表包含来自正常输卵管的106个样本(83个肿瘤样本和23个非肿瘤样本),与9类表型(如病理状态、肿瘤细胞数和肿瘤分级)和3个样本簇信息相关。本研究将样本聚类结果作为分类表型处理。肿瘤和非肿瘤样本的表型病理状态表征如图所示。2A来演示表型分析的模块1。将分类表型自动转换为数值表型进行相关分析,如图所示。2B.我们发现Tumor_Stage_Ovary_FICO与Tumor_Grade评分(0.4)正相关。表型相关表还揭示了我们在接下来的研究中可能需要考虑的其他表型依赖信息。
本研究涉及蛋白质和完整糖肽的两个表达矩阵,分别包括5916个蛋白质和365个完整糖肽。在本研究中,我们将蛋白质或完整的糖肽作为描述样本的特征。这些特征在高维空间中描述了每个样本。虽然OmicsOne在数据预处理和质量控制的模块2中提供了预处理功能,但它也接受外部不同预处理方法预处理的数据。蛋白质和完整糖肽的表达矩阵经过log2转换,归一化,没有缺失值。各样本的表达式分布如图所示。2C.质控模块通过计算样本之间的相关性和特征交叉样本的变异系数来评估样本的方差。选取样本簇1中的三个样本(' SPL 001 '、' SPL 003 '和' SPL 006 ')的蛋白表达矩阵来演示功能。如图所示。2D和E,我们观察到平均相关性为0.67,中位CV为0.16,表明评价程序可以为质量控制提供有效信息。在知识库注释模块3中,两个知识库UniProtKB [39),N-Glycositeatlas [40],以注释蛋白质和完整糖肽的鉴定。注释表支持目标特征的快速查询,支持与数据库的链接,用于进一步的知识发现,支持特征选择,用于后续的表型-特征关联分析(图。2F)。
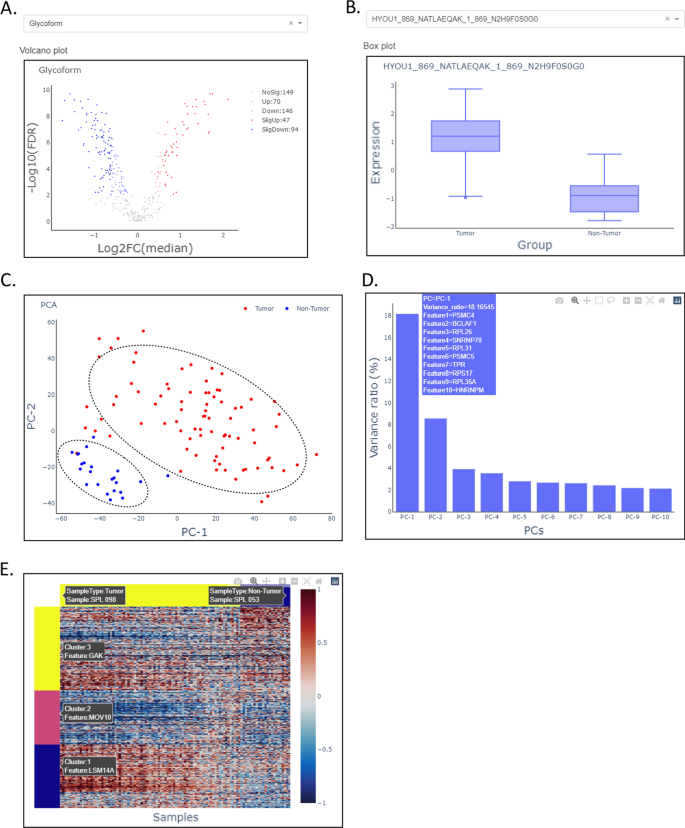
在表型相关特征发现模块4中,我们实现了三个功能:差异表达分析、降维和特征聚类。本模块的目的是寻找与特定表型相关的个体特征或特征集。采用Wilcoxon秩和检验,考虑Benjamini-Hochberg (BH)校正p值< 0.01,折变> 1.5,与非肿瘤样本相比,肿瘤样本中有47个n链完整糖肽表达显著上调,94个n链完整糖肽表达显著下调。3.A).将HYOU1_869_NATLAEQAK_1_869_N2H9在不同表型肿瘤和非肿瘤样本中的个体特征盒状图可视化,如图所示。3.B.在降维部分,OmicsOne显示肿瘤和非肿瘤样本基本是根据其蛋白表达数据进行分类的(图。3.C),并列出了按解释方差比降序排序的pc,以及对pc贡献最大的10个最突出的特征(图。3.D).在特征聚类部分,用户可以直观地看到不同表型下特征聚类的效果(图;3.E),选择合适的聚类号得到特征集,用于后续的富集分析,寻找这些特征聚类背后的路径。在此过程后,我们可以从三种上游分析方法中收集到有趣的特征,包括差异表达分析中的上调或下调特征,pc中用于解释样本方差的顶部贡献特征,以及与表型相关的聚类特征集。
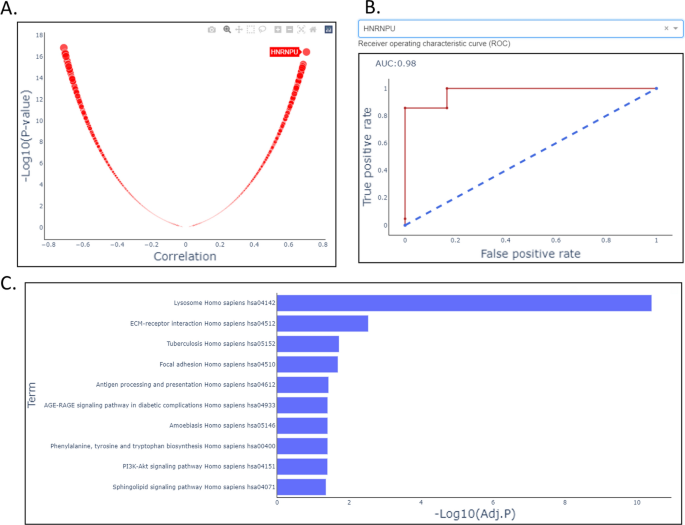
OmicsOne提供了相关和回归模型分析的功能模块(模块5),用于表型和个体特征关联的研究。如图所示。4A,我们发现HNRNPU蛋白与肿瘤病理状态的正相关程度最高(图;4A), logistic回归结果显示受试者工作特征曲线(ROC)下面积为0.98(图。4B).富集分析模块6提供了一种过度表示方法,用于发现由感兴趣的基因集富集的通路。例如,溶酶体途径富集在差异表达分析部分中鉴定的显著上调的完整糖肽的基因中(图2)。3.一个和4C).这一观察结果与先前出版物的结果一致[44].
我们还将OmicsOne应用于LSCC的蛋白质组数据集,以验证其性能[44].从原始附加文件中提取临床信息表和蛋白质组表达表2:表S1,附加文件4:表S3。PCA结果(附加文件1:图S1A)与原始观察结果一致,肿瘤与NAT样本分离清楚[44].根据LSCC出版物中的方法章节,在OmicsOne中使用几乎相同的设置对99对肿瘤和NAT样本进行差异表达分析,以找到显著的肿瘤相关蛋白(FDR < 0.01, fold change > 2)。结果也与原来的观察相一致(附加文件1:图S1B)。我们应用了基因本体(GO)生物过程(BP) (v.2021)的两个数据库[46]和MSigDB_HallMark (v.2020) [36]进行富集分析。我们一致观察到与细胞增殖和DNA修复相关的通路富集在肿瘤中显著上调的蛋白中,而与细胞粘附和急性免疫反应相关的通路富集在肿瘤中显著下调的蛋白中(附加文件)4:表S3)。
讨论
OmicsOne是一种高效的自动化工具,可以将特征的改变与表型联系起来。该软件使用经验设置,以“一键式”模式为标准关联分析构建健壮的工作管道,并允许对分析进行交互操作,以适应定制的需求。“一键式”模式可以加快感兴趣的特征和特征集的发现以及接下来的表型关联分析。但是,我们仍然强烈建议用户仔细研究每个模块的设置和结果,不要将OmicsOne用作黑盒。因此,我们在OmicsOne中开发了一个基于网页的仪表板,集成了结果的交互式数据可视化和相应的参数设置,使分析更清晰,更有效地验证。在整个数据分析的运行过程中,用户可以实时监控各个模块的结果。
OmicsOne支持表型分析、知识注释和完整的糖肽分析。它提供了一种方便的方法将完整的糖肽与临床表型联系起来(图。3.A和B)。完整糖肽的文献信息可以通过注释表中的链接轻松获取。OmicsOne还为分析结果提供直观和交互式的数据可视化。用户可以直接在图中选择感兴趣的数据点,获取详细信息,便于进一步调查。在这个示范调查中,我们报告了HGSOC的蛋白质和糖肽数据集中的两个观察结果。在蛋白质数据集中,HNRNPU蛋白与肿瘤病理状态的正相关程度最高(图2)。4A),采用logistic回归模型预测肿瘤,相应的ROC曲线下面积(AUC)评分为0.98。在肿瘤与非肿瘤样本的差异表达分析结果中,HNRNPU蛋白表达的中位数翻倍变化为1.67,校正后的p值< 0.01。这些测试可以在几分钟内在OmicsOne中有效地完成,并提示HNRNPU可能是HGSOC的潜在生物标志物,最近的研究支持了这一点[44,47].此外,我们还观察到NATLAEQAK的低聚糖聚糖N2H9的糖型,其中HYOU1基因最近被报道为上皮性卵巢癌中细胞生长和转移的启动子,通过激活PI3K/AKT信号通路,预测不良预后[48].研究糖基化在这一促进机制中的作用将是有趣的。
通过对LSCC蛋白质组数据的应用,进一步验证了OmicsOne的性能。肿瘤和NAT样品比较的PCA和差异表达分析结果(附加文件)1:图S1A和S1B)证实了先前观测结果的可重复性。OmicsOne还提供了更多分析结果的细节。肿瘤相关蛋白中的富集通路(附加文件)4:表S3)具有典型的组织学特征,包括氧化磷酸化和糖酵解相关通路的上调和免疫应答的下调。原始出版物中报道的新亚型' EMT- e '中的富集EMT通路[45]在肿瘤和NAT的比较中也发现了改变,这表明EMT通路中改变的蛋白质可以作为诊断和预后的潜在生物标志物进一步研究。
结论
OmicsOne集成了多个用于表型关联分析的基本模块,并提供了一个全面的分析,以在几分钟内发现有趣的表型相关特征(例如,基因、蛋白质或经PTMs修饰的多肽)。数据分析结果实时显示在交互式仪表板中。在本研究中,我们使用已发表的HGSOC和LSCC数据集演示了OmicsOne的性能,并相信它将是一种有效的生物信息学解决方案,用于调查和评估与个体特征或感兴趣的特征集的表型关联,以了解异常生物过程。
数据和材料的可用性
OmicsOne在GitHub上免费提供(https://github.com/huizhanglab-jhu/OmicsOne).HGSOC和LSCC的蛋白质组学和糖蛋白组学数据此前已发表[44,45,并与OmicsOne一起作为样本数据发布。
缩写
- 走:
-
基因本体论
- 铝:
-
翻译修饰
- 信使rna:
-
信使核糖核酸
- 台湾海陆运输公司:
-
Tandem-mass-tag
- 迪亚:
-
数据独立采集
- 简历:
-
变异系数
- 主成分分析:
-
主成分分析
- 奥拉:
-
代表比例分析
- BH:
-
Benjamini-Hochberg
- 中华民国:
-
受试者工作特征曲线
- AUC:
-
ROC曲线下的面积
参考文献
程kc,林艾,辛霞,丁勇。第四章全生物细胞病理学:表型组学的系统方法。进:Foulkes NS,编辑。遗传学的进步。学术出版社;2016.p . 89 - 115。
Hoadley KA, Yau C, Hinoue T, Wolf DM, Lazar AJ, Drill E, Shen R, Taylor AM, Cherniack AD, Thorsson V,等。来自33种癌症的10,000个肿瘤的分子分类中,细胞起源模式占主导地位。细胞。2018;173 (2):291 - 304. - e296。
Nawy T.泛癌症地图集。Nat Methods. 2018;15(6): 407-407。
Mertins P, Mani DR, Ruggles KV, Gillette MA, Clauser KR,王鹏,王霞,乔金伟,曹山,Petralia F,等。蛋白质基因组学将体细胞突变与乳腺癌信号通路联系起来。大自然。2016;534:55。
Vasaikar S, Huang C, Wang X, Petyuk VA, Savage SR, Wen B, Dou Y, Zhang Y, Shi Z, Arshad OA等。人类结肠癌的蛋白质基因组分析揭示了新的治疗机会。2019.(电子)(1097 - 4172)。
张华,刘涛,张震,Payne SH,张波,McDermott JE,周建勇,Petyuk VA,陈琳,Ray D,等。人高级别浆液性卵巢癌的综合蛋白质基因组学特征。细胞。2016;166(3):755 - 65。
张斌,王娟,王霞,朱娟,刘强,史志,Chambers MC, Zimmerman LJ, Shaddox KF, Kim S,等。人类结肠癌和直肠癌的蛋白质基因组学特征。大自然。2014;513(7518):382 - 7。
Krämer A, Green J, Pollard J Jr, Tugendreich S.匠心路径分析中的因果分析方法。生物信息学。2014;30(4):523 - 30。
Polpitiya AD, Qian W-J, Jaitly N, Petyuk VA, Adkins JN, Camp DG II, Anderson GA, Smith RD. DAnTE:组学数据定量分析的统计工具。生物信息学,2008;24(13):1556 - 8。
卡皮耶奇,斯坦利J, Taverner T,黄杰,Adkins JN,安松C, Heffron F, Metz TO,钱文杰,尹华,等。基于ms的自下而上蛋白质组学中蛋白质定量的统计框架。生物信息学。2009;25(16):2028 - 34。
Efstathiou G, Antonakis AN, Pavlopoulos GA, Theodosiou T, Divanach P, Trudgian DC, Thomas B, Papanikolaou N, Aivaliotis M, Acuto O,等。ProteoSign:最终用户在线差异蛋白质组统计分析平台。核酸研究,2017;45(W1):W300-w306。
Theodorakis E, Antonakis AN, Baltsavia I, Pavlopoulos Georgios A, Samiotaki M, Amoutzias Grigoris D, Theodosiou T, Acuto O, Efstathiou G, Iliopoulos I. ProteoSign v2:用于差异蛋白质组学统计分析的更快和用户友好的在线工具。Nucleic Acids Res. 2021;49(W1): W573-7。
Rigbolt KT, Vanselow JT, Blagoev B. GProX,一个用户友好的生物信息学分析和定量蛋白质组学数据可视化平台。Mol Cell Proteomics 2011;10 (8): O110.007450。
Wieczorek S, Combes F, Lazar C, Giai Gianetto Q, Gatto L, Dorffer A, Hesse A-M, Couté Y, Ferro M, Bruley C,等。DAPAR和ProStaR:在定量发现蛋白质组学中进行统计分析的软件。生物信息学。2017;33(1):135 - 6。
Weiner AK, Sidoli S, Diskin SJ, Garcia B. GiaPronto:蛋白质组学数据集的一键图形可视化软件。Mol细胞蛋白质组学2017:mcp.TIR117.000438。
Tyanova S, Temu T, Sinitcyn P, Carlson A, Hein MY, Geiger T, Mann M, Cox J.用于蛋白质组学数据综合分析的Perseus计算平台。Nat Methods. 2016;13(9): 731-40。
常超,徐坤,郭超,王娟,闫强,张娟,何峰,朱勇。PANDA-view:一种简单易用的定量蛋白质组学数据统计分析和可视化工具。生物信息学。2018;34(20):3594 - 6。
吴玲,刘峰,蔡宏。IOAT:组学数据与临床数据的交互式统计分析工具。生物信息学杂志。2021;22(1):326。
Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM, Davis AP, Dolinski K, Dwight SS, Eppig JT,等。基因本体论:生物学统一的工具。基因本体联盟。植物遗传学报,2000;25(1):25 - 9。
基于R的交互式数据可视化。查普曼和霍尔/CRC 2020。https://plotly-r.com.
陈志伟,陈志伟,陈志伟,等。RAS基因翻译后修饰的研究进展。中国生物医学工程学报。2012;13(1):39-51。
曼恩M,詹森ON。翻译后修饰的蛋白质组学分析。生物技术学报。2003;21(3):255-61。
李志强,李志强,李志强。基因转化后修饰对基因编码的影响。中国生物医学杂志,2012;4(6):565-83。
Khoury GA, Baliban RC, Floudas CA.蛋白质组翻译后修饰统计:瑞士-prot数据库的频率分析和管理。科学通报2011;1(1):90。
王志强,李志强,李志强,等。n -链蛋白糖基化的研究进展。中国生物医学工程杂志,2011;21(5):576-82。
糖基化在蛋白质抗原性质中的作用。细胞生命科学,2002;39(3):445-55。
Haltiwanger RS, Lowe JB。糖基化在发育中的作用。生物化学,2004;73:491-537。
糖基化对蛋白质折叠的影响:热力学稳定的近距离观察。自然科学进展,2008;26(3):344 - 344。
斯皮罗RG。蛋白质糖基化:糖肽键的性质、分布、酶的形成和疾病的意义。糖生物学。2002;12 (4):43 r-56r。
李志强,李志强。糖基化在癌症中的作用机制。中华癌症杂志,2015;15(9):540-55。
Anaconda软件发行公司。计算机软件版本2-240 2016。https://anaconda.com/.
Harris CR, Millman KJ, van der Walt SJ, Gommers R, Virtanen P, Cournapeau D, Wieser E, Taylor J, Berg S, Smith NJ,等。使用NumPy进行数组编程。自然。2020;585(7825):357 - 62。
Virtanen P, Gommers R, Oliphant TE, Haberland M, Reddy T, Cournapeau D, Burovski E, Peterson P, Weckesser W, Bright J,等。SciPy 1.0: Python科学计算的基本算法。Nat Methods. 2020;17(3): 261-72。
熊猫开发团队:pandas-dev/pandas:熊猫。Zenodo2020.https://doi.org/10.5281/zenodo.3509134
Pedregosa F, Varoquaux G, Gramfort A, Michel V, Thirion B, Grisel O, Blondel M, pretenhofer P, Weiss R, Dubourg V,等。Scikit-learn: python中的机器学习。J Mach Learn Res. 2011; 12:2825-30。
Subramanian A, Tamayo P, Mootha VK, Mukherjee S, Ebert BL, Gillette MA, Paulovich A, Pomeroy SL, Golub TR, Lander ES,等。基因集富集分析:解释全基因组表达谱的一种基于知识的方法。自然科学进展,2005;26(3):344 - 344。
Mootha VK, Lindgren CM, Eriksson K-F, Subramanian A, Sihag S, Lehar J, Puigserver P, Carlsson E, Ridderstråle M, Laurila E,等。参与氧化磷酸化的pgc -1α反应基因在人糖尿病中协调下调。中国科学。2003;34(3):267-73。
Kuleshov MV, Jones MR, Rouillard AD, Fernandez NF, Duan Q, Wang Z, Koplev S, Jenkins SL, Jagodnik KM, Lachmann A,等。enrichment:一个全面的基因集富集分析web服务器2016年更新。核酸研究,2016;44(W1): W90-7。
UniProt C. UniProt: 2021年的通用蛋白质知识库。核酸Res. 2021;49(D1): D480-9。
孙松,胡勇,敖敏,沙P,陈杰,杨伟,贾欣,田勇,Thomas S,张慧。n -糖蛋白位点图谱:基于质谱的人n -糖蛋白和糖基化位点图谱数据库资源。临床蛋白质组学。2019;16(1):35。
科诺菲尔WJ。实用非参数统计,第3版:约翰·威利父子公司;1999.
Benjamini Y, Hochberg Y.控制错误发现率:一种实用而强大的多重测试方法。J Roy Stat Soc: Ser B (Methodol)。1995; 57(1): 289 - 300。
皮尔逊。在空间中最接近点系统的直线和面上。伦敦,爱丁堡,都柏林哲学杂志J科学1901;1(11): 559 - 572。
胡勇,潘杰,Shah P, Ao M, Thomas SN,刘勇,陈林,Schnaubelt M, Clark DJ, Rodriguez H,等。人高级别浆液性卵巢癌的综合蛋白质组学和糖蛋白组学特征。Cell rep 2020;33(3):108276。
Satpathy S, Krug K, Jean Beltran PM, Savage SR, Petralia F, Kumar-Sinha C, Dou Y, Reva B, Kane MH, Avanessian SC,等。肺鳞状细胞癌的蛋白质基因组图谱。细胞。2021;184 (16):4348 - 4371. - e4340。
基因本体资源:丰富一座金矿。核酸Res 2021;49 (D1): D325-d334。
罗娟,乐玲,唐霞,付艳,冯艳,傅峰。HNRNPU过表达对人卵巢癌有预后价值,并促进卵巢癌细胞增殖。2020.
李霞,张娜霞,叶海燕,宋鹏,常伟,陈琳,王铮,张磊,王楠。HYOU1在上皮性卵巢癌中通过激活PI3K/AKT信号通路促进细胞生长和转移,预测不良预后。中国医药科学,2019;23(10):4126-35。
确认
作者要感谢David J. Clark博士在阅读手稿和有益讨论方面的帮助。
资金
这项工作得到了美国国家癌症研究所、临床蛋白质组学肿瘤分析联盟(CPTAC, Grant U24CA210985)和早期检测研究网络(EDRN, U01CA152813)的支持。
作者信息
作者及隶属关系
贡献
HZ提出并设计了该项目。MA和YH开发了该软件。AB设计了数据表结构。MS对软件进行单元测试。YH执行数据分析应用程序。AB, YH和HZ撰写了手稿。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
所有作者都同意发表这篇手稿。
相互竞争的利益
作者声明没有财务或商业利益冲突。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:图S1。
差异表达分析应用于肿瘤和NAT比较LSCC的蛋白质组数据。PCA图能清晰地区分肿瘤和NAT样本。B肿瘤和NAT样品中差异表达蛋白的火山图
附加文件2:表S1。
OmicsOne软件的分析结果应用于HGSOC的蛋白质组数据集
附加文件3:表S2。
OmicsOne软件的分析结果应用于HGSOC糖蛋白组数据集
附加文件4:表S3。
OmicsOne软件的分析结果应用于LSCC糖蛋白组数据集
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
张,H, Ao, M, Boja, A。et al。OmicsOne:一键关联组学数据和表型。中国Proteom18, 29(2021)。https://doi.org/10.1186/s12014-021-09334-w
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12014-021-09334-w
关键字
- 蛋白质组学
- Glycoproteomics
- 表现型协会
- 生物信息学
- 软件
- 卵巢癌