- 审查gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
纳米孔测序单细胞图谱时代的表观遗传肿瘤异质性gydF4y2Ba
临床实验胚胎学gydF4y2Ba体积gydF4y2Ba14gydF4y2Ba文章编号:gydF4y2Ba107gydF4y2Ba(gydF4y2Ba2022gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
纳米孔测序将该技术带入了测序科学的下一代。这是通过研究进展来实现的:孔隙效率,创建控制DNA易位的机制,提高信噪比,扩展到长读取范围。表观遗传学的异质性是广泛的,因为表观基因组的突变很容易引起癌症研究的新挑战。催化DNA甲基化和组蛋白修饰的表观遗传酶在癌细胞中失调,导致大量异质克隆进化。检测这些克隆的这种异质性在各种癌症的治疗中起着不可或缺的作用。通过单细胞分析,纳米孔测序技术可以提供长读数的简单序列,预计很快将用于床边或医生办公室。在此,我们综述了纳米孔测序及其在癌症表观遗传异质性检测中的应用进展。gydF4y2Ba
简介gydF4y2Ba
20世纪上半叶,DNA结构的发现带来了对其测序的需求[gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba].分别介绍了基于链终止反应和化学解理分析的两种最常用的方法Sanger和Maxam-Gilbert [gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba].Sanger方法依赖于用三磷酸二脱氧嘧啶(ddTTP)终止不断增长的核苷酸链,主导了传统的Maxam-Gilbert方法[gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba].它也被自动化和大规模应用于人类基因组计划(HGP) [gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba].由于技术的不足,人类基因组未能在2003年完全测序[gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba].最近,T2T联盟的下一代无间隙测序(NGS)使得处理全基因组部分成为可能。NGS是一种测序技术,使牛津纳米孔测序的超长读取能力成为可能[gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba].gydF4y2Ba
口袋大小的纳米孔测序仪不需要逆转录过程,也不需要高技能的数据输入方法,在2014年为商业目的推出后,这种测序仪越来越有需求[gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba].在西非偏远地区爆发埃博拉病毒和巴西森林深处爆发寨卡病毒期间,该技术使病毒基因组测序成为可能[gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba].目前,它在中国被用于对SARS-CoV-2进行测序和识别[gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba].gydF4y2Ba
基于纳米孔的测序方法的单分子直接测序特性看起来是为表观遗传学序列量身定制的,而表观遗传学在驱动癌症及其异质性方面具有重要作用[gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba].甲基- cpg结合蛋白是甲基胞嘧啶残基的标识符,以吸引转录抑制因子复合物,如组蛋白去乙酰化酶(HDAC)。那些连接甲基化和组蛋白修饰的蛋白质是表观遗传学的基础[gydF4y2Ba26gydF4y2Ba,gydF4y2Ba27gydF4y2Ba].在此,我们综述了基于纳米孔测序的NGS技术的发展及其在识别表观遗传肿瘤异质性方面的应用[gydF4y2Ba28gydF4y2Ba].我们还讨论了最受研究和更有影响的甲基化和相关的胞嘧啶修饰,以CpG岛的形式存在。gydF4y2Ba
纳米孔测序作为第四代测序仪的研究进展gydF4y2Ba
从Sanger到第四代NGS的测序gydF4y2Ba
从Sanger和Maxam-Gilbert到第四代NGS, DNA测序技术经历了半个世纪的进步,而纳米孔测序被标志着第四代基因测序技术的开始[gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba].gydF4y2Ba
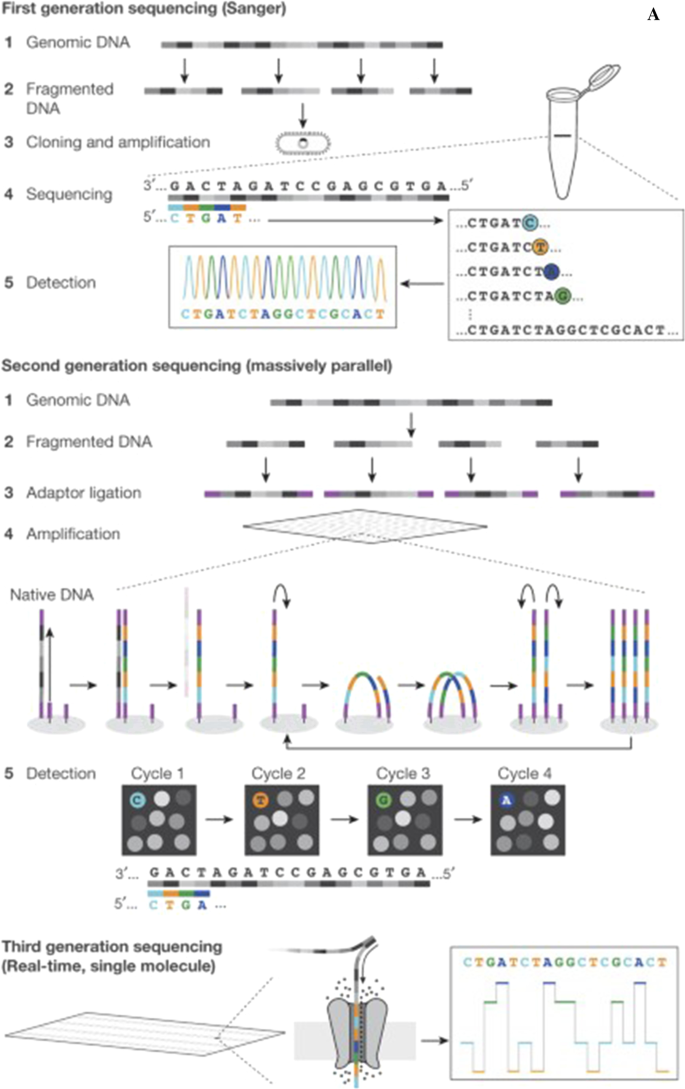
HGP在1990年启动时需要有一种完善的测序技术,使项目可行,因为测序技术的自动化和一些进展的扩大[gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba].HGP的最终确定带来了参考人类基因组序列,也带来了测序技术的进步[gydF4y2Ba10gydF4y2Ba,gydF4y2Ba31gydF4y2Ba].用于HGP的第一代测序需要更长的运行时间和高成本,且吞吐量有限。由于测序需要更高的通量和低成本技术,在20年代中期,从第一代转移到第二代,建立了第二代测序(SGS) [gydF4y2Ba31gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba].这一转变是通过设计一个大规模并行测序系统实现的,该系统始于罗氏454的焦磷酸测序[gydF4y2Ba39gydF4y2Ba,gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba].由于SGS仅限于短读区(35-1000碱基),需要像Sanger的方法一样进行PCR扩增,因此无法读取高/低G + C区、串联重复区、穿插重复区等区域,难以测序[gydF4y2Ba36gydF4y2Ba,gydF4y2Ba38gydF4y2Ba].这些SGS在解决高度碎片化组装的重复序列方面的困难导致了基因测序的下一个时代的发展,包括Illumina/Solexa和PacBio在内的第三代测序(TGSs) [gydF4y2Ba44gydF4y2Ba,gydF4y2Ba45gydF4y2Ba,gydF4y2Ba46gydF4y2Ba].TGS采用单分子实时(single-molecule real-time, SMRT)测序,阅读长度从几十个碱基提高到几万个碱基,测序时间从几天缩短到几小时,PCR消除了测序偏差[gydF4y2Ba44gydF4y2Ba,gydF4y2Ba47gydF4y2Ba(图。gydF4y2Ba1gydF4y2Ba).gydF4y2Ba
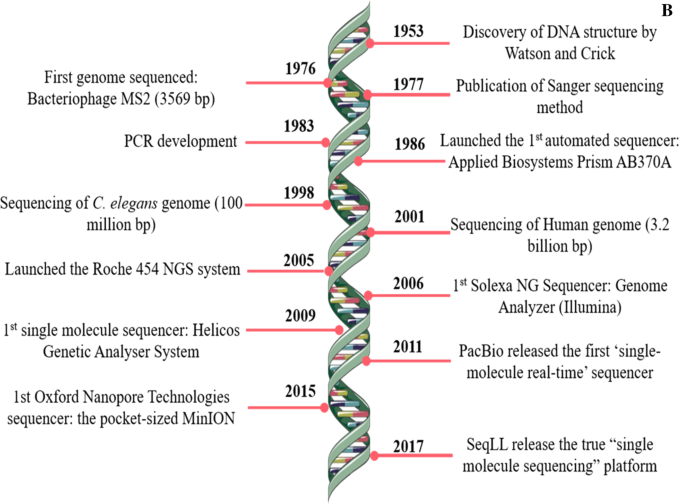
一个gydF4y2Ba第一代、第二代和第三代测序的图解例子。图像转载自[gydF4y2Ba48gydF4y2Ba,并获得出版商的许可(请求ID 600061564, 2021年11月25日)。gydF4y2BaBgydF4y2BaDNA测序时间表。DNA测序的里程碑事件。图像改编自[gydF4y2Ba49gydF4y2Ba]gydF4y2Ba
2007年,Illumina/Solexa推出了基因组分析仪平台的合成测序(SBS)方法,随后ABI的SOLID-Applied Biosystems仪器的连接系统进行测序[gydF4y2Ba42gydF4y2Ba,gydF4y2Ba43gydF4y2Ba].SBS与亚硫酸氢盐测序可用于鉴定胞嘧啶的甲基化。然而,它无法从5hmC区分C和5mC [gydF4y2Ba59gydF4y2Ba,gydF4y2Ba60gydF4y2Ba].gydF4y2Ba
PacBio RS II作为第一个商业化的第三代DNA测序仪,能够直接观察DNA合成,具有测序读取长度长、一致性精度高、偏置程度低、表观遗传表征能力同步等优点,有助于直接检测碱基修饰,如甲基化[gydF4y2Ba36gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba44gydF4y2Ba,gydF4y2Ba47gydF4y2Ba,gydF4y2Ba50gydF4y2Ba,gydF4y2Ba54gydF4y2Ba,gydF4y2Ba55gydF4y2Ba,gydF4y2Ba56gydF4y2Ba].一般来说,PacBio RS II是全基因组测序、靶向测序、复杂群体分析、RNA测序和表观遗传学表征的理想选择。PacBio RS II在没有PCR扩增的情况下工作,相比第一代和第二代平台,PacBio RS II具有提供长读取长度(> 20 kb)和最大读取长度(> 60 kb)的优点。PacBio系统还能够直接检测和识别表观遗传修饰[gydF4y2Ba28gydF4y2Ba,gydF4y2Ba54gydF4y2Ba].此外,许多混合测序策略已经被开发出来,并与PacBio结合,使其更经济和可扩展。PacBio的显著局限性包括吞吐量较低、错误率较高和每个基地成本较高[gydF4y2Ba51gydF4y2Ba,gydF4y2Ba52gydF4y2Ba,gydF4y2Ba53gydF4y2Ba].gydF4y2Ba
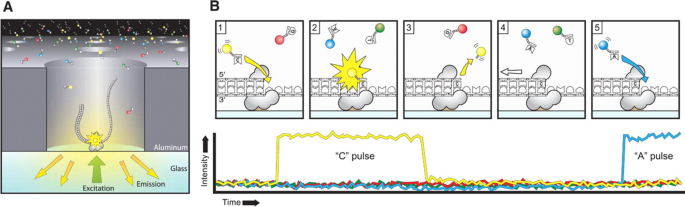
在PacBio单核苷酸测序中,在称为SMRT细胞的芯片上添加4个荧光标记的具有不同发射光谱的核苷酸,当添加碱基时捕获一个零模波长光脉冲(图1)。gydF4y2Ba2gydF4y2Ba).然后将脉冲解释为基序列[gydF4y2Ba38gydF4y2Ba,gydF4y2Ba54gydF4y2Ba].gydF4y2Ba
单分子实时测序原理。gydF4y2Ba一个gydF4y2Ba一个DNA模板结合的Phi 29单分子被固定在零模波导纳米光子结构的底部,并被激光照射。gydF4y2BaBgydF4y2Ba磷酸链dNTP关联循环的图解顺序。(1)磷链核苷酸与聚合酶活性位点的模板形成结合。(2)在类似颜色通道上超前荧光输出。(3)磷酸二酯键的形成释放染料粘合剂磷酸产物,零模波导纳米光子荧光脉冲结束。(4)聚合酶转位到模板链的下一个核苷酸上。(5)下一个同源核苷酸与聚合酶活性位点结合,继续循环[gydF4y2Ba57gydF4y2Ba,gydF4y2Ba58gydF4y2Ba].图像转载自[gydF4y2Ba57gydF4y2Ba(订单许可ID: 1164215- 1,2021年11月25日)gydF4y2Ba
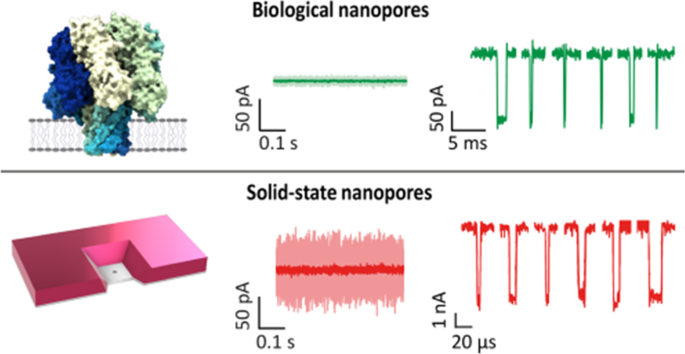
最新的(NGS)纳米孔测序技术(主要在这篇综述中讨论)有一个薄膜结构,容纳纳米级孔。当比纳米孔小的生物分子通过小孔时,它会检测到通过小孔的单个分子的潜在电荷[gydF4y2Ba31gydF4y2Ba,gydF4y2Ba61gydF4y2Ba,gydF4y2Ba62gydF4y2Ba].这四家公司正在以价格、方法和平均阅读长度为标准,争夺NGS市场的主导权gydF4y2Ba1gydF4y2Ba).gydF4y2Ba
牛津纳米孔测序技术的发展gydF4y2Ba
2012年,纳米孔技术开始应用于RNA的逆转录和扩增测序。随后,牛津纳米孔技术公司(ONT)开发了一种基于生物纳米孔阵列的设备,能够可靠解码长序列,错误率可接受,成本低,小型化更好[gydF4y2Ba64gydF4y2Ba,gydF4y2Ba94gydF4y2Ba].其长读测序能力使其成为测序史上的里程碑[gydF4y2Ba63gydF4y2Ba,gydF4y2Ba64gydF4y2Ba,gydF4y2Ba65gydF4y2Ba,gydF4y2Ba66gydF4y2Ba].测序是一种直接、高度并行、实时的单分子方法,可以提高核苷酸的阅读长度[gydF4y2Ba95gydF4y2Ba,gydF4y2Ba96gydF4y2Ba,gydF4y2Ba97gydF4y2Ba,gydF4y2Ba98gydF4y2Ba].gydF4y2Ba
NGS中的纳米孔可以减少样品扩增所需的时间,以及合成方法测序中使用的酶、试剂和光学器件。纳米孔传感器是纯电动的,可以穿透血液或唾液DNA样本[gydF4y2Ba67gydF4y2Ba,gydF4y2Ba68gydF4y2Ba,gydF4y2Ba69gydF4y2Ba,gydF4y2Ba70gydF4y2Ba].纳米孔是一种通过脂质膜模拟蛋白质通道的纳米级开放生物孔。孔可以通过离子轨迹蚀刻或直接平面光刻来制造。使用灵敏的膜片钳放大器,通过单个孔的离子电流可以用来分离标记为顺式和反式的两个腔。gydF4y2Ba71gydF4y2Ba,gydF4y2Ba72gydF4y2Ba,gydF4y2Ba73gydF4y2Ba].同时在膜上施加电压,使离子电流通过纳米孔[gydF4y2Ba67gydF4y2Ba,gydF4y2Ba72gydF4y2Ba].gydF4y2Ba
随着以往测序方法的强制性改变,纳米孔测序成为医学领域的重要工具,例如在癌症研究和诊断中[gydF4y2Ba73gydF4y2Ba,gydF4y2Ba74gydF4y2Ba,gydF4y2Ba75gydF4y2Ba,gydF4y2Ba76gydF4y2Ba].此外,基于孔的测序可用于测序、组装和分析结构变异,并检测表观遗传标记,以实现未来人类基因组学应用[gydF4y2Ba75gydF4y2Ba,gydF4y2Ba77gydF4y2Ba,gydF4y2Ba78gydF4y2Ba,gydF4y2Ba79gydF4y2Ba(图。gydF4y2Ba3.gydF4y2Ba).gydF4y2Ba
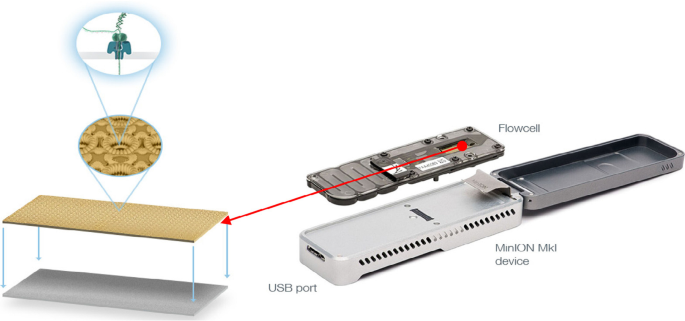
MinION测序装置- dna测序是通过在流细胞中添加一个样本来进行的。当DNA分子通过纳米孔时,传感器会测量纳米孔中电流的大小变化。数据流被传递到应用特定的集成电路ASIC和MinKnow,它们产生信号级数据。图像转载自[gydF4y2Ba63gydF4y2Ba得到出版商的许可(请求ID 600062077, 2021年12月1日)gydF4y2Ba
纳米孔测序技术是来自不同方向的循序渐进、长期、多学科努力的结果[gydF4y2Ba80gydF4y2Ba].第一次培养是在1976年完成的,当时Erwin Neher和Bert Sakmann开发了一种机制来记录和测量通过嵌入在生物膜中的单个离子通道的电流量[gydF4y2Ba81gydF4y2Ba,gydF4y2Ba82gydF4y2Ba].但是,1989年David Deamer提出了使用离子电流测量来通过嵌入膜的纳米孔进行测序的直接想法[gydF4y2Ba83gydF4y2Ba,gydF4y2Ba84gydF4y2Ba].gydF4y2Ba
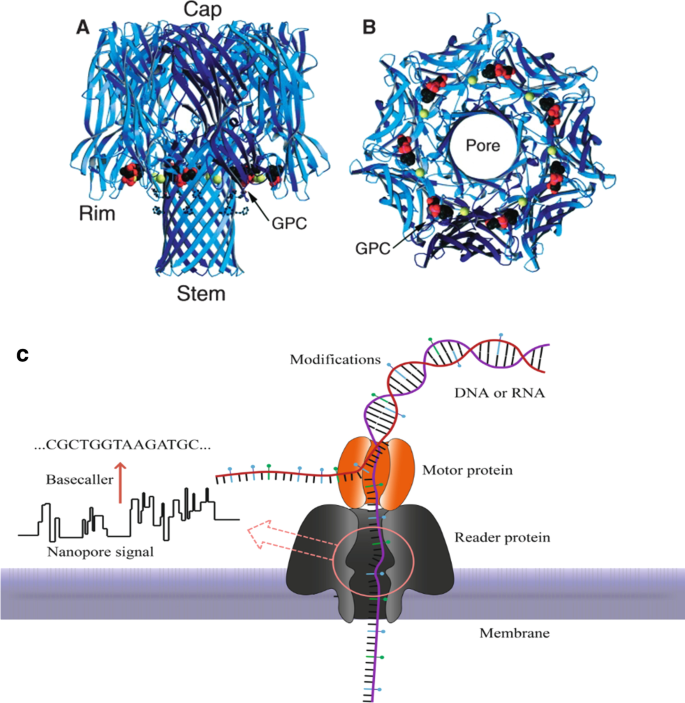
当Deamer在研究α-溶血素(一种金黄色葡萄球菌分泌的蛋白质毒素)时遇到John Kasianozicz时,他解决了核苷酸可能缺乏离子通道的问题。gydF4y2Ba4gydF4y2BaA、B) [gydF4y2Ba85gydF4y2Ba,gydF4y2Ba86gydF4y2Ba].嵌入生物溶血素纳米孔的磷脂双分子层被分离成两个腔室,充满KCl溶液。外加离子电流的电势(图。gydF4y2Ba5gydF4y2Ba)将带负电荷的DNA通过孔推至正极,直到它移位(图。gydF4y2Ba4gydF4y2BaC) (gydF4y2Ba87gydF4y2Ba].易位速度取决于施加的电势、使用的纳米孔以及DNA的单股或双股。最佳速度约为每毫秒2个核苷酸,一个10 × 10阵列的人类基因组测序可在8小时内完成[gydF4y2Ba88gydF4y2Ba].这四个核苷酸是由离子信号堵塞的易位所产生的各种电流扰动所分化的。堵塞的幅度和持续时间取决于易位聚合物的长度和宽度[gydF4y2Ba89gydF4y2Ba,gydF4y2Ba90gydF4y2Ba].gydF4y2Ba
金黄色葡萄球菌α-溶血素的表达。从[转载gydF4y2Ba91gydF4y2Ba,并获得了出版商的许可(CCC许可ID: 5196920847168, 2021年11月27日)。gydF4y2Ba一个gydF4y2Baα -溶血素七聚物的侧视图显示了磷脂双分子层的确切位置。gydF4y2BaBgydF4y2Baα -溶血素从顺式入口到毛孔的视图[gydF4y2Ba86gydF4y2Ba].gydF4y2BaCgydF4y2Ba嵌入磷脂双分子层的α-溶血素纳米孔的结构。在纳米孔测序中,马达蛋白引导DNA链通过孔。这导致电流通过薄膜波动。纳米孔信号随后被碱基调用者转换成核酸序列。DNA底物(紫色)通过外加电场插入孔中。图像改编自[gydF4y2Ba92gydF4y2Ba]gydF4y2Ba
具有代表性ssDNA和蛋白质结合DNA事件的生物纳米孔仪器[gydF4y2Ba67gydF4y2Ba,gydF4y2Ba93gydF4y2Ba].图像转载自[gydF4y2Ba67gydF4y2Ba,并获得了出版商的许可(CCC许可ID: 600061571, 2021年11月25日)。gydF4y2Ba一个gydF4y2Ba纳米孔仪器使用放大器施加命令电压Vc,并通过纳米孔通道测量离子电流Ip。gydF4y2BaBgydF4y2Ba在1 M缓冲的KCl溶液中,当ssDNA从顺式室捕获到通道中时,120毫伏的开通道电流在0.2毫秒内衰减到15毫伏,直到DNA通过孔。当Exol和ssDNA在顺式腔中时,也观察到电流漂移持续时间延长2 ms的结合事件[gydF4y2Ba67gydF4y2Ba]gydF4y2Ba
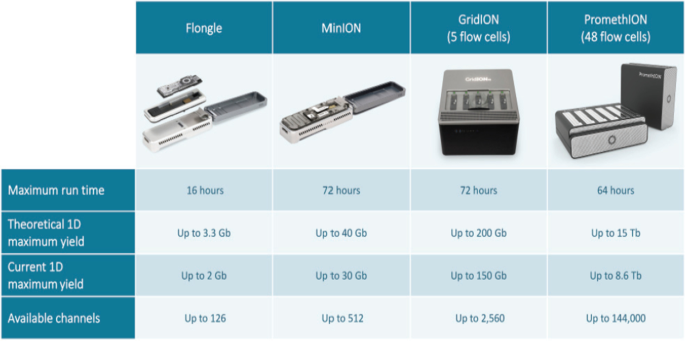
为了进入合成测序领域,ONT设计了一种更稳定的膜来支持纳米孔,纳米孔最初是由脂质制造的。由于脂质对pH值和温度极为敏感,因此被涂有脂质的特氟龙手工制作材料所取代[gydF4y2Ba99gydF4y2Ba].通常的薄膜只工作几秒钟到几分钟就会崩溃,需要一整天的时间才能生成半小时的数据。ONT转向合成膜材料,使其更有效。此外,为了克服这一挑战,在2012年2月,GridION, Flongle, MinION和PromethION平台展示了[gydF4y2BaOne hundred.gydF4y2Ba,gydF4y2Ba104gydF4y2Ba,gydF4y2Ba105gydF4y2Ba].MinION可能是最受关注的,因为它在6小时内破译了近10亿个DNA碱基,而定价为900美元。gydF4y2Ba6gydF4y2Ba) [gydF4y2Ba102gydF4y2Ba,gydF4y2Ba103gydF4y2Ba].gydF4y2Ba
牛津纳米孔技术的比较。改编自(gydF4y2Ba23gydF4y2Ba]gydF4y2Ba
生物纳米孔与固态纳米孔的对比gydF4y2Ba
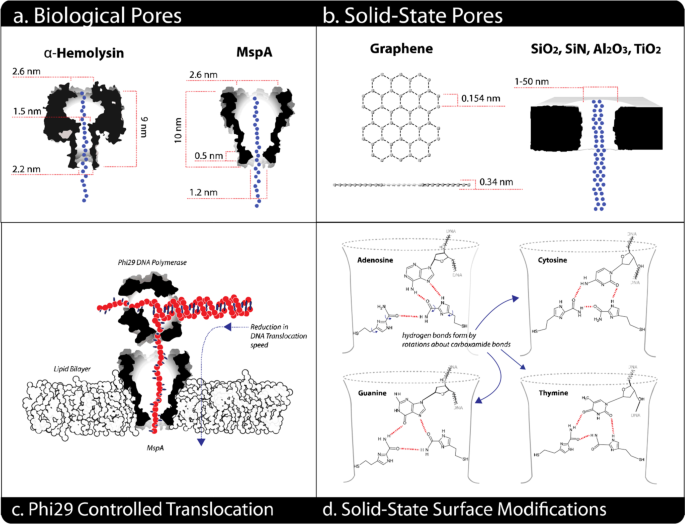
最初的生物纳米孔仍然产生了最好的结果,其结构易于制作、高度修改和可复制,允许重复的电流测量[gydF4y2Ba109gydF4y2Ba,gydF4y2Ba110gydF4y2Ba,gydF4y2Ba111gydF4y2Ba].无机纳米孔在温度、溶剂相容性、鲁棒性以及与半导体电子集成能力方面具有强度[gydF4y2Ba112gydF4y2Ba,gydF4y2Ba113gydF4y2Ba,gydF4y2Ba114gydF4y2Ba].与生物材料相比,固态纳米孔具有更强的热稳定性、机械稳定性和化学稳定性;易于修改;可调的孔径和形貌,易于集成到纳米流体或其他纳米器件中,以及可伸缩性的制造[gydF4y2Ba115gydF4y2Ba,gydF4y2Ba116gydF4y2Ba,gydF4y2Ba117gydF4y2Ba,gydF4y2Ba118gydF4y2Ba].最常见的固态纳米孔是SiOgydF4y2Ba2gydF4y2Ba低应力富硅氮化物SiNgydF4y2BaxgydF4y2Ba.除了在半导体微电子制造中很好地处理这些材料外,硅基纳米孔因其鲁棒性、良好的电阻率和介电强度而受到青睐[gydF4y2Ba119gydF4y2Ba,gydF4y2Ba120gydF4y2Ba,gydF4y2Ba121gydF4y2Ba].其他用于纳米孔的元素有铝gydF4y2Ba2gydF4y2BaOgydF4y2Ba3.gydF4y2Ba和高频振荡器gydF4y2Ba2gydF4y2Ba,提供独特的薄膜制作方法[gydF4y2Ba122gydF4y2Ba,gydF4y2Ba123gydF4y2Ba,gydF4y2Ba124gydF4y2Ba,gydF4y2Ba125gydF4y2Ba].gydF4y2Ba
固态孔隙最初通过离子束雕刻,后来通过透射电子显微镜(TEM)钻孔或介电击穿制成,其局限性在于无法达到膜稳定性所需的厚度[gydF4y2Ba107gydF4y2Ba].与生物孔相比,固态纳米孔由于其固有厚度和缺乏对表面电荷分布的控制,其单分子检出率较低[gydF4y2Ba126gydF4y2Ba].gydF4y2Ba
一种基于MoS的多功能纳米孔膜gydF4y2Ba2gydF4y2Ba其信号振幅是固态SigydF4y2Ba3.gydF4y2BaNgydF4y2Ba4gydF4y2Ba不同于石墨烯纳米孔,不需要特殊的表面处理来避免DNA和表面之间的强相互作用[gydF4y2Ba126gydF4y2Ba,gydF4y2Ba127gydF4y2Ba].单层二维材料,如石墨烯,MoSgydF4y2Ba2gydF4y2Ba, WSgydF4y2Ba2,gydF4y2Ba和六方氮化硼(h-BN)随着核苷酸之间的间距而变厚[gydF4y2Ba128gydF4y2Ba,gydF4y2Ba129gydF4y2Ba].与传统的固态纳米孔膜相比,单层二维膜具有较高的离子电流信噪比和较大的传感区域,是纳米孔器件的理想选择[gydF4y2Ba129gydF4y2Ba,gydF4y2Ba130gydF4y2Ba].固态纳米孔通道长约为DNA分子中两个碱基之间距离(0.5 nm)的100倍[gydF4y2Ba131gydF4y2Ba,gydF4y2Ba132gydF4y2Ba].尽管在易位过程中存在粘着效应,但将超薄石墨烯单层膜置于氮化硅上后,由电子束钻成的薄膜是较好的固态纳米孔技术[gydF4y2Ba131gydF4y2Ba,gydF4y2Ba133gydF4y2Ba].gydF4y2Ba
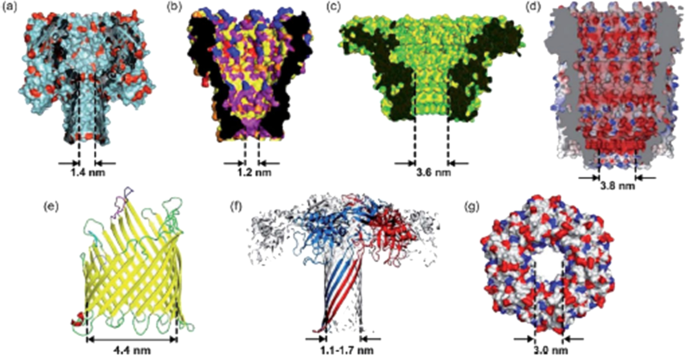
在将溶血素鉴定为生物孔后,需要稳定的膜纳米孔允许一次通过更少的核苷酸,以减少一次大量核苷酸的进入[gydF4y2Ba116gydF4y2Ba].因此,漏斗形耻垢分枝杆菌孔蛋白A (MspA)被引入作为溶血素的替代品[gydF4y2Ba116gydF4y2Ba,gydF4y2Ba134gydF4y2Ba].与蘑菇型α-溶血素不同,MspA在茎中的核苷酸通过数量减少[gydF4y2Ba135gydF4y2Ba].为了提高ONT纳米孔的读数,CsgG (curli特异性基因产物A-G)gydF4y2Ba大肠杆菌gydF4y2Ba外膜脂蛋白也被引入[gydF4y2Ba136gydF4y2Ba].在测试的数十个纳米孔和数千个突变体中,CsgG孔具有非常狭窄和明确的DNA链通道,并胜过ONT所尝试的所有孔[gydF4y2Ba137gydF4y2Ba,gydF4y2Ba138gydF4y2Ba].后来在CsgG孔中设计了读取头,提高了序列读取的信号和准确性[gydF4y2Ba139gydF4y2Ba,gydF4y2Ba140gydF4y2Ba].其他蛋白质纳米孔包括外膜蛋白F (OmpF),外膜蛋白G (OmpG), Aerolysin,诺卡菌farcinica肽A/B (NfpA/ nffb)和细胞溶解素A (ClyA)也被尝试过[gydF4y2Ba112gydF4y2Ba,gydF4y2Ba141gydF4y2Ba(图。gydF4y2Ba7gydF4y2Ba、表gydF4y2Ba2gydF4y2Ba).gydF4y2Ba
生物纳米孔的例子。gydF4y2Ba一个gydF4y2Baα霍奇金淋巴瘤,gydF4y2BabgydF4y2BaMspA,gydF4y2BacgydF4y2Baφ29日gydF4y2BadgydF4y2BaClyA,gydF4y2BaegydF4y2BaFhuA,gydF4y2BafgydF4y2Baaerolysin,gydF4y2BaggydF4y2BaSP1。从[转载gydF4y2Ba142gydF4y2Ba(CCC许可ID: 1164219- 1,2021年11月25日)gydF4y2Ba
从不同的建筑材料中获取多样化的纳米孔类型,以获得更高的精度、尺寸和化学性能,已经扩大了纳米孔的应用范围,超越了测序[gydF4y2Ba143gydF4y2Ba].自组装孔类型由多种材料产生,包括蛋白质、多肽、合成有机化合物和各种[gydF4y2Ba144gydF4y2Ba].Genia technologies(被罗氏以3亿美元收购,旨在将生物纳米孔与光学检测相结合)、量子孔、量子生物系统(由T Kawai教授将隧道电子探测器与纳米孔测序相结合)、Base4和Noblemen Biosciences等公司旨在将单核苷酸切割成水-油乳状液中的小滴,并通过化学级联反应检测它们的存在[gydF4y2Ba89gydF4y2Ba,gydF4y2Ba145gydF4y2Ba(图。gydF4y2Ba8gydF4y2Ba).gydF4y2Ba
各种类型和几何形状的纳米孔。从[转载gydF4y2Ba67gydF4y2Ba(CCC许可ID: 600061571, 2021年11月25日)gydF4y2Ba
通过纳米孔控制DNA易位gydF4y2Ba
纳米孔成功成为一种可靠的DNA分析工具的关键障碍之一是DNA易位的超快和随机性质,这需要加入马达蛋白,通过碱基和其他实验修饰来进行DNA易位[gydF4y2Ba107gydF4y2Ba].这个问题的起源是由随机扩散布朗运动引起的速度波动,它与有向运动结合,产生漂移扩散过程的事件[gydF4y2Ba146gydF4y2Ba].为了实现单核苷酸分辨率,DNA的易位速度预计为1-100 ms/nt [gydF4y2Ba107gydF4y2Ba,gydF4y2Ba147gydF4y2Ba].结合生物马达或纳米指令和通过调节孔隙几何形状和实验条件来调节驱动电压是已经尝试过的两种方法[gydF4y2Ba107gydF4y2Ba,gydF4y2Ba148gydF4y2Ba].研究人员试图通过修改溶剂粘度、离子浓度或温度等宏观特性来感知DNA链的每个核苷酸并以可控的方式将链送入纳米孔[gydF4y2Ba149gydF4y2Ba,gydF4y2Ba150gydF4y2Ba].提供一系列金属-介电层的分子动力学模拟也被提出作为一种额外的选择[gydF4y2Ba151gydF4y2Ba].gydF4y2Ba
生物马达或纳米马达的合并gydF4y2Ba
为了增强碱基识别,DNA外切酶(从gydF4y2Ba大肠杆菌gydF4y2Ba外切酶I (ExoI))和DNA聚合酶作为α-HL的马达[gydF4y2Ba152gydF4y2Ba].考虑到由于链的完全消化而无法进行多次读取,以及需要将核苷酸精确地送入孔隙等缺点,使外切酶在运动蛋白研究的早期就过时了[gydF4y2Ba153gydF4y2Ba,gydF4y2Ba154gydF4y2Ba].的Klenow片段(KF)是第一个被认为是a家族的聚合酶gydF4y2Ba大肠杆菌gydF4y2Ba带有α-HL孔的ddna I [gydF4y2Ba155gydF4y2Ba].但由于稳定性和连续性问题,a家族dna被b家族dna所取代,即Phi 29 [gydF4y2Ba156gydF4y2Ba].噬菌体phi29 DNA聚合酶(phi29 DNAP)对DNA底物具有很高的亲和力,并与α-HL和MspA孔工作良好[gydF4y2Ba157gydF4y2Ba].与聚合酶不同,具有结合单链核酸能力的解旋酶需要部分双工结构,其中新的核苷酸被添加到引物的3 '端[gydF4y2Ba158gydF4y2Ba].解旋酶也具有较好的亲和力,可以消除双读碱基和合成速率波动引起的跳跃性,表现出Phi 29- dap的校对特性[gydF4y2Ba159gydF4y2Ba,gydF4y2Ba160gydF4y2Ba(图。gydF4y2Ba9gydF4y2Ba).gydF4y2Ba
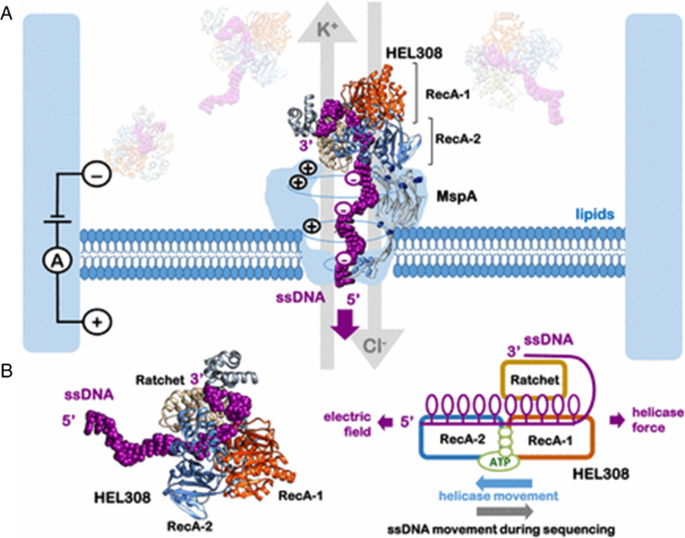
hel308 -解旋酶作为移动ssDNA的马达蛋白gydF4y2Ba一个gydF4y2Ba展示了机理和gydF4y2BaBgydF4y2BaHEL308的域组织与运动。两个(重组蛋白A) RecA 1和2结构域组成运动部分;在这里,ATP在它们之间结合并驱动或纠正机械化学循环,辅助棘轮结构域与ssDNA进行多次接触,并可能提供潜在序列特异性的决定因素[gydF4y2Ba159gydF4y2Ba,gydF4y2Ba160gydF4y2Ba].图像改编自[gydF4y2Ba159gydF4y2Ba]gydF4y2Ba
据报道,一种具有纳米串珠结构的集成纳米孔平台可以通过聚酰亚胺层以及纳米孔制造的可控介电击穿(CDB)过程来减缓DNA的移动,并降低噪声[gydF4y2Ba161gydF4y2Ba].第二种控制易位的方式依赖于前面提到的调节驱动电压,调节孔隙几何形状和实验条件是有帮助的[gydF4y2Ba162gydF4y2Ba,gydF4y2Ba163gydF4y2Ba,gydF4y2Ba164gydF4y2Ba].gydF4y2Ba
调整孔隙几何形状gydF4y2Ba
有限的孔隙几何形状是迫使研究扩展到固态纳米孔隙的因素,固态纳米孔隙可以提供孔隙形状的多样性。但是,由于膜稳定性所需的厚度,它们降低了空间分辨率[gydF4y2Ba107gydF4y2Ba,gydF4y2Ba119gydF4y2Ba,gydF4y2Ba165gydF4y2Ba].将纳米孔直径减小到几乎与ssDNA相同的尺寸,即1.4 nm,将易位速度降低到1.4微秒/基,使缩小纳米孔成为改善易位的有效方法[gydF4y2Ba166gydF4y2Ba].当孔径减小时,来自DNA的电流信号幅值增大。与连续统建模系统上的圆柱形纳米孔相比,锥形纳米孔从生物分子易位中产生更大的信号振幅[gydF4y2Ba167gydF4y2Ba].gydF4y2Ba
调整实验条件gydF4y2Ba
单链DNA (ssDNA)在固态纳米孔中的超快易位速度是其所面临的困境之一,有多种方法可以降低该速度[gydF4y2Ba161gydF4y2Ba,gydF4y2Ba166gydF4y2Ba,gydF4y2Ba168gydF4y2Ba一种是二价金属离子(尤其是钙)的可控介电击穿(CBD)gydF4y2Ba2 +gydF4y2Ba提供氮化硅纳米孔,每基减速100微秒[gydF4y2Ba169gydF4y2Ba].结果表明,改变电解质阳离子种类和溶液质量浓度可增加孔滞留时间。对于固态孔隙,当阳离子尺寸从KgydF4y2Ba+gydF4y2Ba以NagydF4y2Ba+gydF4y2Ba李gydF4y2Ba+gydF4y2Ba, dsDNA和ssDNA的易位时间都大幅增加,这是由于较小阳离子与DNA链的结合能力更强[gydF4y2Ba170gydF4y2Ba].利用LiCl盐梯度和纳米纤维网格减缓DNA易位速度,以保持DNA分子在纳米孔的传感时间内。与其他碱溶液相比,LiCl可以延长停留时间20 ms(比NaCl和KCl长5倍),当浓度增加时停留时间达到100 ms,纳米纤维网进一步延迟停留时间162 ~ 185 ms [gydF4y2Ba171gydF4y2Ba].通过将LiCl盐浓度提高15倍来降低ssDNA的易位速度,带来了反离子结合,有效降低了DNA的整体电荷,从而降低了系统的电泳驱动能力,减缓了易位速度。必须降低易位增强分辨率,直到允许在不使用甲基特异性标签的情况下将5'mC与C区分开来[gydF4y2Ba172gydF4y2Ba].另一方面,当KCl浓度从1 M降低到0.1 M时,通过纳米孔的时间缩短,而当MgCl浓度较低时,通过时间延长gydF4y2Ba2gydF4y2Ba在氮化硅纳米孔系统中[gydF4y2Ba173gydF4y2Ba].gydF4y2Ba
提高信噪比信噪比gydF4y2Ba
纳米孔技术发展的主要障碍是离子电流中的噪声,这限制了信噪比。固态纳米孔具有最高的信噪比,这是因为它们可以在较大的电流下工作,并且在高频下噪声相对较低。尽管如此,易位速度减缓发挥了主要作用,MspA被证明可以将信噪比>提高160倍[gydF4y2Ba174gydF4y2Ba(图。gydF4y2Ba10gydF4y2Ba).gydF4y2Ba
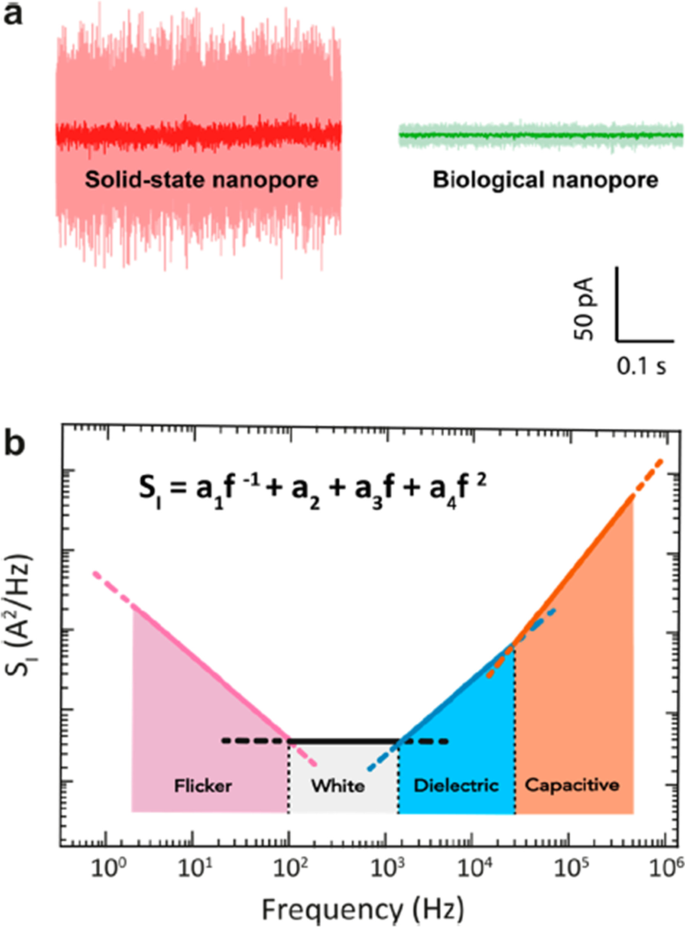
生物和固态纳米孔中的噪声。图像改编自[gydF4y2Ba174gydF4y2Ba]gydF4y2Ba
纳米孔噪声功率谱密度(PSD)由1/gydF4y2BafgydF4y2Ba噪声:白噪声、介电噪声和放大器噪声,每种噪声在不同的频率上占主导地位。当我们看到噪声的来源时,1/f的噪声是由于表面和体积效应;白噪声来自热效应和射击效应;来自介质膜电流泄漏的介质噪声和放大器噪声是由于芯片和放大器中的电容[gydF4y2Ba175gydF4y2Ba(图。gydF4y2Ba11gydF4y2Ba).gydF4y2Ba
固态SiN纳米孔中的离子电流噪声gydF4y2Ba2gydF4y2Ba纳米孔与生物α-HL (gydF4y2Ba一个gydF4y2Ba)在pH值为7、带宽为10 kHz (gydF4y2BabgydF4y2Ba).图像改编自[gydF4y2Ba174gydF4y2Ba]gydF4y2Ba
为了提高信噪比,纳米孔的直径受分子大小的限制,膜的厚度受材料性能的限制[gydF4y2Ba176gydF4y2Ba,gydF4y2Ba177gydF4y2Ba,gydF4y2Ba178gydF4y2Ba].利用非晶硅膜基纳米孔的理论厚度极限,正成为提高离子电导和为测序应用产生高信噪比的主要材料[gydF4y2Ba179gydF4y2Ba,gydF4y2Ba180gydF4y2Ba].为了克服噪声的限制,人们采用了多种方法,例如提高生物纳米孔的构象刚度和减小孔径[gydF4y2Ba174gydF4y2Ba, SiN的表面功能化gydF4y2BaxgydF4y2Ba具有亲水层的纳米孔,如铝gydF4y2Ba2gydF4y2BaOgydF4y2Ba3.gydF4y2Ba或SiOgydF4y2Ba2gydF4y2Ba[gydF4y2Ba124gydF4y2Ba,高电场对孔隙的应用[gydF4y2Ba181gydF4y2Ba],选择远离纳米孔材料等电点的pH值[gydF4y2Ba174gydF4y2Ba],它们被证明有助于降低固态纳米孔中的噪声[gydF4y2Ba178gydF4y2Ba].通过最小化芯片的电容和介电损耗来抑制介电噪声也是降低噪声的另一种方法[gydF4y2Ba174gydF4y2Ba,gydF4y2Ba175gydF4y2Ba].gydF4y2Ba
ONT的另一个改进领域是对更高信噪比和吞吐量的计算要求[gydF4y2Ba182gydF4y2Ba,gydF4y2Ba183gydF4y2Ba].这就需要更多的算法来实现基调用、映射和变量调用[gydF4y2Ba184gydF4y2Ba].由于纳米孔测序器的技术限制,其低信噪比使其无法读取和确定所需的核苷酸序列[gydF4y2Ba182gydF4y2Ba].gydF4y2Ba
扩大范围太长读数gydF4y2Ba
为了对基因组中重复的元素进行明确的测序,需要增加显著的长度[gydF4y2Ba187gydF4y2Ba,gydF4y2Ba188gydF4y2Ba].在文库制备过程中,尽可能慢地移液以减少剪切力并保存长DNA模板的方法被开发出来,称为snail(一种用于长序列的慢核酸仪器)[gydF4y2Ba187gydF4y2Ba].snail实现了文库制备试剂的慢移液自动化,以增加长读纳米孔测序的一致性和吞吐量[gydF4y2Ba187gydF4y2Ba,gydF4y2Ba189gydF4y2Ba].专注于DNA提取和酶促反应以进一步增加读取长度,有可能从50 - 70 kb的机械剪切转换为90 - 100 kb的转位酶介导的片段读取[gydF4y2Ba190gydF4y2Ba].gydF4y2Ba
在千禧年之初,人类基因组的初稿尚未完成,直到牛津纳米孔测序技术补充了PacBio测序[gydF4y2Ba191gydF4y2Ba].所以,我们看到了完整的人类基因组测序。由端粒到端粒(T2T)联盟处理的剩下8%的基因组包括:所有22个常染色体加上X染色体的无间隙组合、所有的着丝体卫星阵列和5个端粒染色体的短臂[gydF4y2Ba16gydF4y2Ba,gydF4y2Ba192gydF4y2Ba].长读测序进入基因组中难以进入的部分,如着丝粒[gydF4y2Ba101gydF4y2Ba,端粒和远端中心基因组区[gydF4y2Ba193gydF4y2Ba].在这些区域,大量串联重复序列在种系和体细胞中占主导地位,突变率最高[gydF4y2Ba194gydF4y2Ba].这些技术的鉴定允许进入这些区域是基因组分析研究和产业的福音[gydF4y2Ba101gydF4y2Ba].gydF4y2Ba
计算进步gydF4y2Ba
测序实验中的计算分析有各种工具[gydF4y2Ba104gydF4y2Ba,gydF4y2Ba105gydF4y2Ba].但是它们的选择需要明确,每个步骤需要单独的工具。管理和集成工具也很困难。将工具与管道相结合可能有助于绘制测序读数,计算甲基化水平,并区分不同的甲基化位置或区域[gydF4y2Ba106gydF4y2Ba].由于移动缓慢,无法识别单个核苷酸,另一个挑战是在孔中创建一个控制良好的核苷酸棘轮[gydF4y2Ba87gydF4y2Ba,gydF4y2Ba107gydF4y2Ba,gydF4y2Ba108gydF4y2Ba].gydF4y2Ba
由于结合了纳米孔化学和碱基呼叫软件的计算系统的发展,纳米孔测序器可以在短时间内生成大量数据[gydF4y2Ba182gydF4y2Ba,gydF4y2Ba184gydF4y2Ba].该软件执行核苷酸片段的测序和读取,随后采用两种方法:读取映射和从头组装[gydF4y2Ba345gydF4y2Ba].Read mapping是将reads与参考基因组进行比对,以识别测序基因组中的变异[gydF4y2Ba383gydF4y2Ba].在没有参考基因组的情况下,De novo组装用于组合reads以构建原始序列[gydF4y2Ba384gydF4y2Ba].2014年,牛津纳米孔技术公司(ONT)为MinION启动了beta测试项目,随后开发了新的计算方法,用于基调用、数据处理、读映射、从头组装和新一代数据的变体发现[gydF4y2Ba15gydF4y2Ba,gydF4y2Ba195gydF4y2Ba].这些方法改进了基因组从头测序,并使研究结构变异具有无与伦比的准确性和分辨率成为可能。这一进展还可以降低纳米孔测序技术较高的错误率[gydF4y2Ba196gydF4y2Ba].gydF4y2Ba
纳米孔化学软件gydF4y2Ba
MinION和GridION等测序器的测序化学变化在错误率方面取得了有价值的改善。在生产MinION之前,通过生物纳米孔测序允许在马达蛋白解开双链时对模板链进行1D测序[gydF4y2Ba182gydF4y2Ba,gydF4y2Ba387gydF4y2Ba].然而,早期的MinION模型提供了二维测序软件,它包含了两条链的校对(模板和补充),这是由于发夹结构连接到DNA链而实现的。2D读取的准确性已超过1D读取的5%(仅读取模板链)[gydF4y2Ba64gydF4y2Ba].最近,ONT开发了1DgydF4y2Ba2gydF4y2Ba测序软件,允许测序模板和互补链无需物理连接。由于这个变化,1DgydF4y2Ba2gydF4y2Ba比1D软件的精确度提高了7% [gydF4y2Ba182gydF4y2Ba,gydF4y2Ba385gydF4y2Ba,gydF4y2Ba386gydF4y2Ba].gydF4y2Ba
基地调用软件gydF4y2Ba
碱基调用涉及将获得的原始电流信号转换为核苷酸序列的计算过程,这对于表观遗传修饰的检测非常重要[gydF4y2Ba388gydF4y2Ba].因此,ONT经历了基站呼叫软件的各个发展阶段。基本调用是在开发早期阶段使用HMM从碎片化的当前数据中获得的,随后在2016年实现了一个循环神经网络[gydF4y2Ba389gydF4y2Ba].2017年已使用原始当前数据采集基站呼叫。随着精度需求的增加,2018年和2019年分别实践了更新的触发器和定制的基站呼叫模型[gydF4y2Ba184gydF4y2Ba,gydF4y2Ba390gydF4y2Ba].gydF4y2Ba
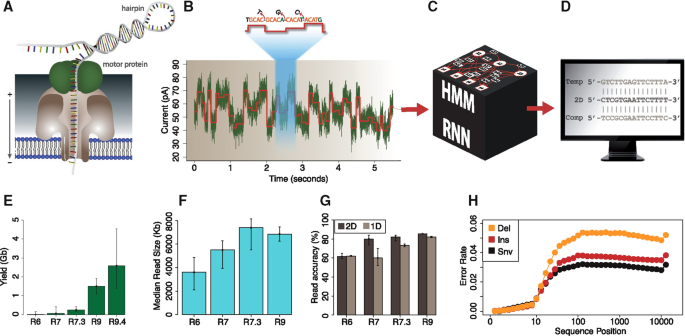
实时基调用可以简化,因为当前的格式如BAM/CRAM(二进制对齐映射/压缩引用对齐映射)无法完全达到超长读取[gydF4y2Ba77gydF4y2Ba].多达5个邻近碱基影响穿过MspA的单个DNA链的当前水平[gydF4y2Ba185gydF4y2Ba].这种限制激发了人们使用最动态的规划方法,如维特比算法[gydF4y2Ba186gydF4y2Ba].当然,基因分型的准确性正在与短读测序仪器竞争,这是由于杂合和纯合等位基因的区分不足。这就迫切需要用于长时间单分子测序的结构变异基因分型工具。gydF4y2Ba77gydF4y2Ba].MinION的计算程序具有识别步骤,可将碱基调用电子数据转换为所需的核苷酸序列[gydF4y2Ba63gydF4y2Ba].首先,在纳米孔上方发现的马达蛋白解开dsDNA,使ssDNA通过纳米孔(图1)。gydF4y2Ba12gydF4y2BaA).其次,将核苷酸读取得到的离子电流信号分离为平均值、标准差和长度(图1)。gydF4y2Ba12gydF4y2BaB).这些信号有恒定的采样频率为5000赫兹。第三,将分离出来的结果转移到机器学习方法框中,翻译成模板和附加信号(图1)。gydF4y2Ba12gydF4y2BaC).最后,信号序列的结果在计算机设备上显示(图1)。gydF4y2Ba12gydF4y2BaD).每个步骤的性能可以通过基于吞吐量、读取长度和准确性的图表来评估(图5)。gydF4y2Ba12gydF4y2BaE, f, g, h) [gydF4y2Ba195gydF4y2Ba].gydF4y2Ba
使用纳米孔进行DNA计算测序的步骤。图像转载自[gydF4y2Ba195gydF4y2Ba(许可证ID: 1164222-1, 2021年11月25日)gydF4y2Ba
纳米孔测序技术目前面临的挑战和机遇gydF4y2Ba
纳米孔测序需要解决的两个挑战是酶的周转和纳米孔电流释放的时间间隔[gydF4y2Ba67gydF4y2Ba,gydF4y2Ba186gydF4y2Ba].酶周转被用于识别随机序列中的连续碱基,给出一个不完美的棘轮,其中DNA每次前进的间隔是可变的[gydF4y2Ba197gydF4y2Ba].有些间隔可能太短,在系统噪声中被忽略了,或者相同碱基的重复序列在较长的间隔中可能无法识别。改进的棘轮机制用于精确的纳米孔测序可能解决这个问题[gydF4y2Ba186gydF4y2Ba].gydF4y2Ba
固体纳米孔的修饰和功能化模拟一些重要的生物孔的特性正在发展中。然而,纳米孔只能一次性使用,需要付出更多努力才能实现可逆功能化[gydF4y2Ba198gydF4y2Ba,gydF4y2Ba199gydF4y2Ba].因此,混合生物/人造纳米孔是结合鲁棒性和选择性的最有前途的策略[gydF4y2Ba200gydF4y2Ba,gydF4y2Ba201gydF4y2Ba,gydF4y2Ba202gydF4y2Ba].纳米孔技术在共识碱基调用精度方面无法与其他测序平台竞争[gydF4y2Ba203gydF4y2Ba,gydF4y2Ba204gydF4y2Ba].单分子测序(SMS)无法产生足够的信号,因此单个测序读取的错误率高于SBS测序数据[gydF4y2Ba205gydF4y2Ba,gydF4y2Ba206gydF4y2Ba].当然,在表观基因组学的这些碱基修饰之上,纳米孔使全基因组和转录组范围的分析成为可能。此外,由于纳米孔技术也被应用于蛋白质测序,对于蛋白质组学来说,这一机遇将多组学带到一个单一平台,这将是纳米孔测序,未来的测序应用包括人类健康和医学[gydF4y2Ba207gydF4y2Ba,gydF4y2Ba208gydF4y2Ba,gydF4y2Ba209gydF4y2Ba].gydF4y2Ba
与PacBio和最大的市场股东Illumina的竞争是巨大的。虽然在SMRT中需要高覆盖率测序,但在纳米孔测序中使用低覆盖率reads可以实现高精度检测[gydF4y2Ba209gydF4y2Ba,gydF4y2Ba210gydF4y2Ba].牛津纳米孔在法律诉讼的战场上击败Illumina和PacBio是很容易的;由于牛津纳米孔公司在十多年的生产、搜寻和申请过程中拥有超级多的专利,这种情况似乎还会继续下去。gydF4y2Ba211gydF4y2Ba,gydF4y2Ba212gydF4y2Ba,gydF4y2Ba213gydF4y2Ba].gydF4y2Ba
尽管许多解决方案都是针对上述挑战提出的。gydF4y2Ba3.gydF4y2Ba在美国,纳米孔测序技术长达十年之久的挑战仍然令人担忧的专家工作的技术。Daniel Branton曾在2008年的论文《纳米孔测序的潜力与挑战》中预测,类似的挑战仍然存在,但也取得了巨大的进展[gydF4y2Ba108gydF4y2Ba].gydF4y2Ba
纳米孔测序工作流程gydF4y2Ba
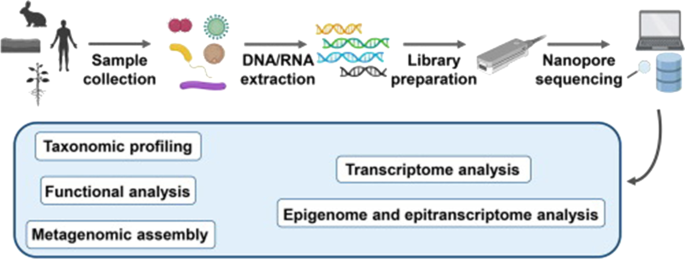
在进行纳米孔测序的样品和文库制备之前,应编制所有与人类受试者工作相关的规定[gydF4y2Ba214gydF4y2Ba].提取核酸,然后进行文库制备和碱基调用[gydF4y2Ba66gydF4y2Ba].在测序和组装来自短DNA寡核苷酸的大DNA片段之前,一般的步骤是增加小DNA扩增子的纳米孔测序通量[gydF4y2Ba214gydF4y2Ba,gydF4y2Ba215gydF4y2Ba(图。gydF4y2Ba13gydF4y2Ba).gydF4y2Ba
纳米孔测序工作流程。图像改编自[gydF4y2Ba216gydF4y2Ba]gydF4y2Ba
用Minimap2比对参考基因组完成纳米孔读数的定位。对于匹配已知基因的reads,使用Sicelore Add gene name Tag方法将基因名添加到相应的SAM记录中;在这里,基因用它们的纳米孔读取序列和读取质量注释[gydF4y2Ba217gydF4y2Ba(图。gydF4y2Ba14gydF4y2Ba).gydF4y2Ba
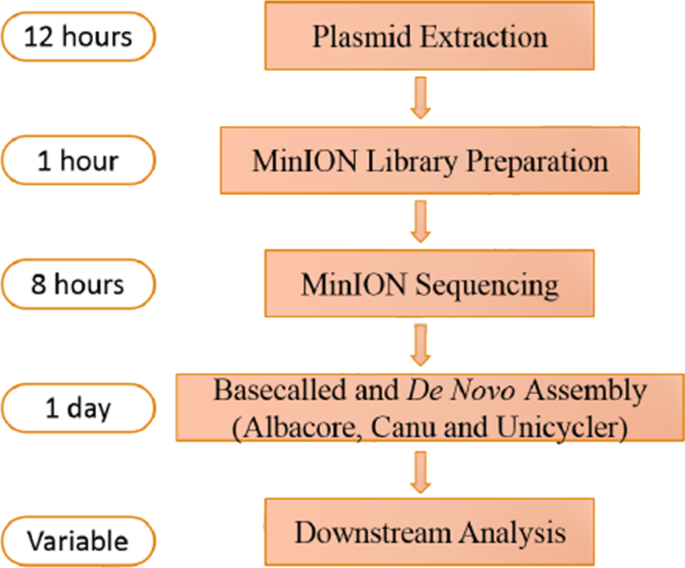
MinION纳米孔测序和组装过程的工作流程和周期。估计是基于一个快速条形码测序套件,它可以在一次运行中收集12个样本。基调用和从头组装取决于所使用的计算机的容量。图像改编自[gydF4y2Ba218gydF4y2Ba]gydF4y2Ba
表观遗传肿瘤异质性与测序技术gydF4y2Ba
表观遗传学与肿瘤异质性gydF4y2Ba
表观遗传学gydF4y2Ba
表观遗传成分可以被认为是作者,读者和橡皮擦;作者在组蛋白或DNA上添加化学基团(例如,组蛋白乙酰转移酶HATs,组蛋白甲基转移酶HMT,或DNA甲基转移酶)[gydF4y2Ba219gydF4y2Ba].组蛋白去乙酰化酶HDACs或组蛋白去甲基化酶HDMTs等清除添加的化学基团[gydF4y2Ba220gydF4y2Ba,gydF4y2Ba221gydF4y2Ba,gydF4y2Ba222gydF4y2Ba].一组通过附加到特定序列而充当效应蛋白的读本结构域,例如甲基结合结构域蛋白或溴化结构域和末端外(BETs)结构域蛋白,也为人所知[gydF4y2Ba220gydF4y2Ba,gydF4y2Ba223gydF4y2Ba,gydF4y2Ba224gydF4y2Ba].DNA甲基化是指修饰的核苷酸5-甲基胞嘧啶(5mC) [gydF4y2Ba225gydF4y2Ba是第一个被确定的表观遗传因素,也是这里的主要焦点。5mC在所有序列中都有发现,但在5 '到3 '方向上胞嘧啶紧跟着鸟嘌呤的序列中含量非常丰富[gydF4y2Ba226gydF4y2Ba].5mC被认为是一个CpG位点,而CpG位点高的区域被称为CpG岛,发现超过三分之二的基因启动子,可以作为表观遗传调节开关,在甲基化时限制基因表达[gydF4y2Ba227gydF4y2Ba,gydF4y2Ba228gydF4y2Ba].启动子区CpG岛对正常发育需求和肿瘤发生过程中的基因沉默[gydF4y2Ba229gydF4y2Ba,gydF4y2Ba230gydF4y2Ba].与组蛋白修饰的相对可塑性转录调控不同,通过DNA甲基化的基因沉默更持久持久[gydF4y2Ba231gydF4y2Ba].因此,甲基化是主要的表观遗传沉默机制,用于抑制体细胞中内源性转座子、印迹基因和多能相关基因[gydF4y2Ba232gydF4y2Ba,gydF4y2Ba233gydF4y2Ba(图。gydF4y2Ba15gydF4y2Ba).gydF4y2Ba
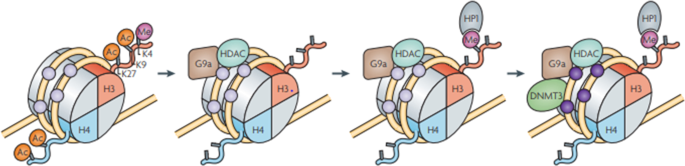
多能性基因中DNA甲基化与组蛋白修饰之间的联系。在胚胎干细胞中,多能性基因如Oct 3/4和Nanog有乙酰化(未甲基化)CpG岛。这些岛与乙酰化的Histone (Ac) H3和H4以及Histone H3的甲基化赖氨酸4(Me) (K4)结合。随着分化的开始,组蛋白甲基转移酶(G9a)和组蛋白去乙酰化酶(HDAc)结合到复合物上。结合导致H3和H4的去乙酰化。同时HDAc催化K4的去甲基化,G9a催化K9的甲基化。这一修饰为染色体域蛋白异染色质蛋白1(HP1)创造了一个结合位点。最后,G9a招募甲基化酶DNMT3A和DNMT3B(深紫色圆圈),这将介导脱乙酰DNA的重新甲基化[gydF4y2Ba232gydF4y2Ba,gydF4y2Ba234gydF4y2Ba].该过程有利于表观遗传沉默和甲基化,而阻碍异染色质化。图像转载自[gydF4y2Ba235gydF4y2Ba(请求ID 600061575 2021年11月25日)gydF4y2Ba
除了甲基化,还有其他具有潜在调节作用的二核苷酸修饰,如5-羟甲基胞嘧啶(5hmC)、5-甲酰胞嘧啶(5Fc)和5-羧基胞嘧啶(5CaC) [gydF4y2Ba236gydF4y2Ba].DNA在胞嘧啶第5位的甲基化形成5-甲基胞嘧啶(5mC),这是主要发生在哺乳动物CpG二核苷酸位点的DNA修饰。5mC可以被称为α-酮戊二酸依赖性双加氧酶的10 - 11个易位家族的酶转化为5hmC, 5Fc和5CaC [gydF4y2Ba237gydF4y2Ba,gydF4y2Ba238gydF4y2Ba].事实上,5hmC可能分布在蛋白质编码基因体和长链非编码rna (lncrna)上的启动子上。gydF4y2Ba16gydF4y2Ba) [gydF4y2Ba239gydF4y2Ba].gydF4y2Ba
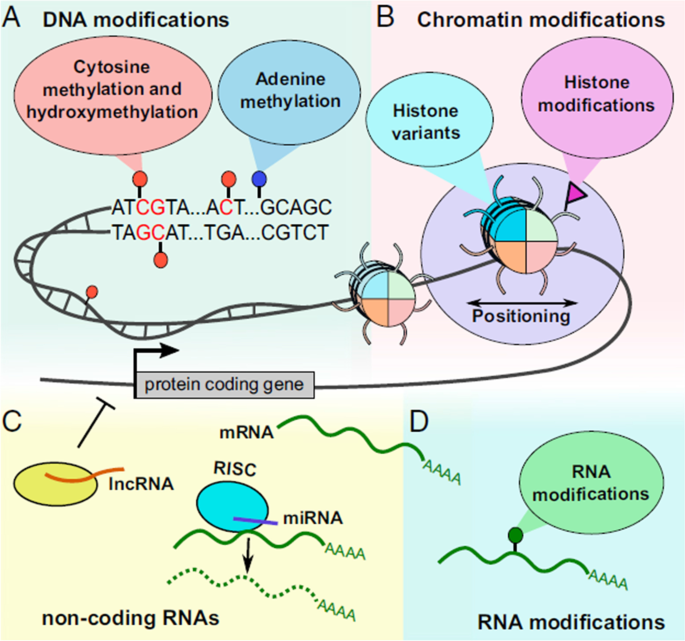
表观遗传机制的景观。gydF4y2Ba一个gydF4y2Ba胞嘧啶和腺嘌呤修饰,胞嘧啶通过甲基化、羟甲基化(hmC)、甲酰化(fC)和羧化(caC)修饰,腺嘌呤通过甲基化修饰。gydF4y2BaBgydF4y2Ba组蛋白修饰和具有不同组蛋白变体的核小体改变位置。gydF4y2BaCgydF4y2Ba非编码rna在转录调控中起着重要作用,有时被认为是表观遗传机制。gydF4y2BaDgydF4y2Ba所有的RNA修饰也可以被认为是表观遗传学的一部分。图像改编自[gydF4y2Ba240gydF4y2Ba]gydF4y2Ba
甲基化的调节功能,尤其是在高甲基化中,在于基因的启动子区域得到额外的甲基化后,辅助抑制因子的招募[gydF4y2Ba241gydF4y2Ba].这种额外的甲基化导致转录沉默。调控过程由DNA甲基转移酶(DNMT 1、2、3A和3B)和甲基- cpg结合蛋白指导,它们识别甲基胞嘧啶残基,吸引转录抑制因子复合物,如组蛋白去乙酰化酶(HDAC) [gydF4y2Ba27gydF4y2Ba,gydF4y2Ba242gydF4y2Ba,gydF4y2Ba243gydF4y2Ba].组蛋白乙酰化(HAT)和组蛋白去乙酰化(HDAC)作为调控因子最终影响基因转录[gydF4y2Ba27gydF4y2Ba].有一种小型rna管理互补的新生支架,但用作引导组蛋白和DNA甲基转移酶的试剂[gydF4y2Ba244gydF4y2Ba].除了小RNA外,染色质相关的长链非编码RNA支架发挥着独立但协同转录沉默作用,提供了一个系统来检测和沉默不适当的转录事件[gydF4y2Ba245gydF4y2Ba].该系统还允许注册记忆,这是自我强化的表观遗传循环[gydF4y2Ba246gydF4y2Ba)(表gydF4y2Ba3.gydF4y2Ba).gydF4y2Ba
氧化的5-甲基胞嘧啶的作用在很长一段时间内一直存在争议,但作为这些位点的读本的结合蛋白的发现开始显示出它们的作用[gydF4y2Ba248gydF4y2Ba,gydF4y2Ba249gydF4y2Ba].对于5hmC,像UHRF2 (PHD和无名指结构域的泛素样蛋白2)这样的解读蛋白被识别出来[gydF4y2Ba250gydF4y2Ba].但是,这种结合的下游生物效应尚未被确定[gydF4y2Ba248gydF4y2Ba,gydF4y2Ba251gydF4y2Ba].5fC和5caC以少量的数量存在,特别是在某些基因组位置,如增强子和启动子中,有针对性的研究已经确定了这些修饰核苷酸的结合蛋白[gydF4y2Ba252gydF4y2Ba].gydF4y2Ba
表观遗传失调与癌症和靶向治疗的关系gydF4y2Ba
分子测序技术在表征表观遗传方面的进步使其成为癌症的另一个标志之一[gydF4y2Ba253gydF4y2Ba,gydF4y2Ba254gydF4y2Ba].DNA甲基化谱可调节关键的细胞过程,如凋亡、脂肪生成和mapk通路的下游转录效应[gydF4y2Ba255gydF4y2Ba].这些基因区域的甲基化不受控制的调节导致结直肠癌(CRC)中肿瘤细胞的生长[gydF4y2Ba256gydF4y2Ba].此外,甲基化相关的表观遗传驱动基因已被确定参与CRC肿瘤发生的早期阶段。CRC肿瘤显示CpG岛甲基化表型(cmp)。这些表型与特定遗传变化、疾病危险因素和患者预后高度一致[gydF4y2Ba257gydF4y2Ba].因此,CpG岛区的高甲基化导致肿瘤抑制基因的沉默,从而导致肿瘤细胞的生长[gydF4y2Ba258gydF4y2Ba],而CpG岛的低甲基化促进转录癌基因[gydF4y2Ba259gydF4y2Ba].失调的表观遗传机制、甲基化和组蛋白修饰也与胶质母细胞瘤的发生高度相关[gydF4y2Ba260gydF4y2Ba].gydF4y2Ba
5hmC具有特定的特性,使其适合于生物学功能,主要是阻止5mc -seek蛋白与DNA的相互作用[gydF4y2Ba261gydF4y2Ba,gydF4y2Ba262gydF4y2Ba].作为一种短暂的中间体,它在生殖细胞和早期胚胎发育过程中具有促进DNA去甲基化的作用[gydF4y2Ba263gydF4y2Ba,gydF4y2Ba264gydF4y2Ba,gydF4y2Ba265gydF4y2Ba,gydF4y2Ba266gydF4y2Ba].在细胞分化和重编程过程中,tet介导的DNA去甲基化是从5mC氧化为5hmC开始的[gydF4y2Ba267gydF4y2Ba,gydF4y2Ba268gydF4y2Ba,gydF4y2Ba269gydF4y2Ba].随着进一步的氧化,5hmC转化为中间产物5caC,并最终在转化为胞嘧啶时完成DNA去甲基化[gydF4y2Ba266gydF4y2Ba,gydF4y2Ba267gydF4y2Ba,gydF4y2Ba268gydF4y2Ba,gydF4y2Ba269gydF4y2Ba,gydF4y2Ba270gydF4y2Ba].gydF4y2Ba
在基因体和启动子上,5-羟甲基胞嘧啶(5hmC)在癌症标志物中具有多种作用,不同的5hmC水平与结直肠癌(CRC)患者的临床结果和肿瘤状态相关[gydF4y2Ba239gydF4y2Ba].另一方面,5hmC在DNA功能的调节中发挥作用,使其成为未来早期癌症诊断和预后的标记之一[gydF4y2Ba271gydF4y2Ba,gydF4y2Ba272gydF4y2Ba].这一预期是在认识到5hmC作为过渡状态中间体在遗传调控的去甲基化过程中发挥作用之后产生的[gydF4y2Ba263gydF4y2Ba,gydF4y2Ba273gydF4y2Ba].gydF4y2Ba
一般来说,DNA甲基化、组蛋白修饰、染色质重构和微rna的表观遗传畸变可以显示癌症的发展和进展,并被用作患者分层的生物标志物[gydF4y2Ba274gydF4y2Ba,gydF4y2Ba275gydF4y2Ba].它们还被用作预测模型,允许在患者的诊断、预后和治疗中使用癌症表观遗传学[gydF4y2Ba274gydF4y2Ba,gydF4y2Ba276gydF4y2Ba)(表gydF4y2Ba4gydF4y2Ba).gydF4y2Ba
表观遗传学研究深入探索靶向表观遗传学畸变作为一种潜在的抗癌疗法,适合于表观遗传学变化的可逆性[gydF4y2Ba277gydF4y2Ba,gydF4y2Ba278gydF4y2Ba].几种表观遗传抑制剂已被开发并批准用于常规临床实践[gydF4y2Ba253gydF4y2Ba,gydF4y2Ba254gydF4y2Ba,gydF4y2Ba279gydF4y2Ba].表观遗传治疗的机制包括抑制DNA和组蛋白的甲基化或去甲基化以及乙酰化或去乙酰化[gydF4y2Ba253gydF4y2Ba,gydF4y2Ba280gydF4y2Ba,gydF4y2Ba281gydF4y2Ba,gydF4y2Ba282gydF4y2Ba].表观遗传调控机制的抑制剂包括腺苷、胞苷或脱氧胞苷的各种类似物,或DNMT的非核苷类小分子抑制剂和羟肟酸的抑制剂,如对HDAC的trichostatin A (TSA)和亚基苯胺比酰二羟酰胺(SAHA) [gydF4y2Ba27gydF4y2Ba,gydF4y2Ba283gydF4y2Ba].Epidrug设计的靶向HDAC抑制剂如SAHA和roidepsin用于治疗难治性皮肤T细胞淋巴瘤[gydF4y2Ba284gydF4y2Ba,gydF4y2Ba285gydF4y2Ba]、别立诺他治疗外周T细胞淋巴瘤[gydF4y2Ba286gydF4y2Ba,gydF4y2Ba287gydF4y2Ba],或用于多发性骨髓瘤的panobinostat,包括作为DNMT抑制剂的地西他滨用于血液系统恶性肿瘤,如骨髓增生异常综合征、急性髓系白血病和慢性髓单核细胞白血病[gydF4y2Ba220gydF4y2Ba,gydF4y2Ba288gydF4y2Ba,gydF4y2Ba289gydF4y2Ba].gydF4y2Ba
许多具有癌症检测、诊断和/或预后能力的表观遗传生物标志物已被确认[gydF4y2Ba290gydF4y2Ba,gydF4y2Ba291gydF4y2Ba].然而,它们的临床可用性很低。在多中心组中缺乏独立验证和变量实验设计阻碍了转化研究的进展,将标记物转化为临床有用的工具[gydF4y2Ba292gydF4y2Ba].缺乏验证也阻碍了方便和负担得起的癌症检测的可用性[gydF4y2Ba290gydF4y2Ba].gydF4y2Ba
肿瘤表观遗传异质性gydF4y2Ba
肿瘤的异质性可以发生在患者之间、同一患者的多个肿瘤的同源性或肿瘤亚群内,称为患者间异质性、患者内异质性或肿瘤内异质性[gydF4y2Ba23gydF4y2Ba,gydF4y2Ba293gydF4y2Ba].个体细胞间DNA修饰作为一种在各种环境条件下的生存机制,是调节表型异质性的重要表观遗传因子[gydF4y2Ba294gydF4y2Ba,gydF4y2Ba295gydF4y2Ba].即使在形态学上无法区分的细胞中也发现了表达的显著异质性,这些细胞在组织生物学和疾病状态(如癌症)中发挥着重要的功能作用[gydF4y2Ba233gydF4y2Ba].gydF4y2Ba
在人类癌症中,表观遗传畸变变化比基因突变发生得更频繁[gydF4y2Ba23gydF4y2Ba,gydF4y2Ba296gydF4y2Ba,gydF4y2Ba297gydF4y2Ba].然而,大多数癌症研究集中在遗传基础上,特别是致癌基因的突变激活或肿瘤抑制基因(TSG)的失活[gydF4y2Ba23gydF4y2Ba].在肿瘤细胞分化程序的几个谱系中,表观遗传机制是不可分割的部分,并在癌症、干细胞生物学和耐药性之间具有潜在的分子联系[gydF4y2Ba24gydF4y2Ba].gydF4y2Ba
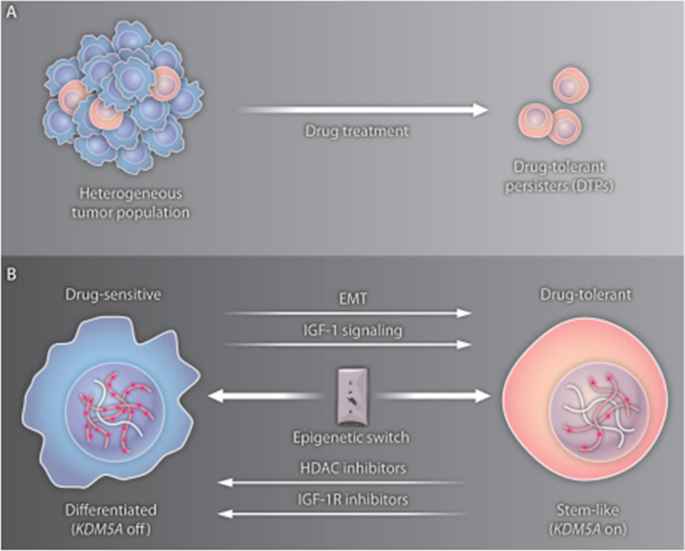
在79例肿瘤内结直肠肿瘤中发现甲基化异质性水平与无复发时间和总生存期相关[gydF4y2Ba298gydF4y2Ba,gydF4y2Ba299gydF4y2Ba].大量证据支持肿瘤通常由异质细胞类型组成,耐药性似乎与这些细胞类型有关[gydF4y2Ba300gydF4y2Ba,gydF4y2Ba301gydF4y2Ba]以及在癌症细胞亚群中介导耐药的表观遗传机制的作用有令人信服的证据[gydF4y2Ba24gydF4y2Ba,gydF4y2Ba302gydF4y2Ba(无花果。gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba).gydF4y2Ba
肿瘤细胞的异质性导致了肿瘤的耐药表型。gydF4y2Ba一个gydF4y2Ba选择治疗后dtp的子集。gydF4y2BaBgydF4y2Ba表观遗传变化调节药物敏感状态到耐药状态的过渡。图像改编自[gydF4y2Ba24gydF4y2Ba]gydF4y2Ba
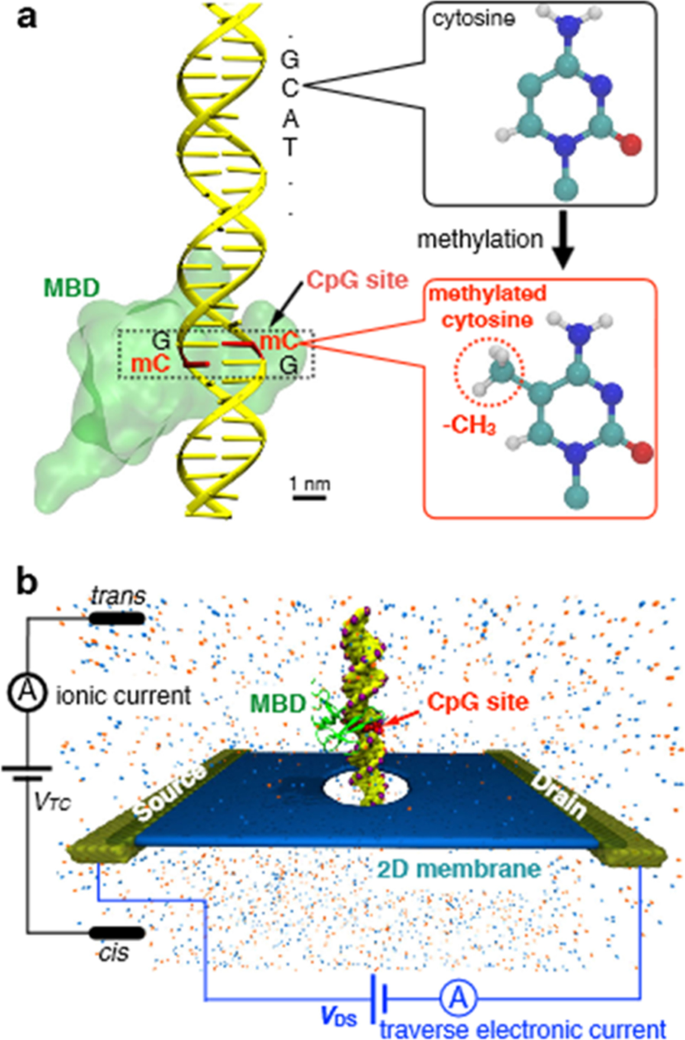
利用二维石墨烯或MoS合成的固体纳米孔鉴定甲基化胞嘧啶残基gydF4y2Ba2gydF4y2Ba.图像改编自[gydF4y2Ba351gydF4y2Ba]gydF4y2Ba.gydF4y2Ba一个gydF4y2Ba利用MBD1蛋白鉴别C和mC结构。甲基化发生在胞嘧啶环结构的第5个碳位,大部分的mC核苷酸出现在基因的CpG岛区。gydF4y2BabgydF4y2BaDNA纳米孔测序过程中mC的图解检测模型。识别是基于利用离子电流差异获得的应用所需的电压gydF4y2Ba
定位肿瘤的表观遗传异质性gydF4y2Ba
表观遗传测序在肿瘤异质性中的作用gydF4y2Ba
当我们观察TET蛋白的生理功能及其调节DNA甲基化和转录的机制时,发现在三个TET基因TET1和TET2中,肝细胞癌(HCC)组织中的表达水平较低[gydF4y2Ba303gydF4y2Ba,gydF4y2Ba304gydF4y2Ba].研究还发现,在HCC组织和细胞系中,全球基因组5hmC水平下调[gydF4y2Ba305gydF4y2Ba,gydF4y2Ba306gydF4y2Ba].对于设计早期检测和治疗策略,在HCC组织和循环无细胞DNA中发现的5hmC特征是重要的[gydF4y2Ba305gydF4y2Ba].利用从49例7种不同类型癌症患者中获得的无细胞5hmC序列,研究了5-羟甲基胞嘧啶(5hmC)在基因调控和癌症发病机制中的作用。这一发现表明,在预测癌症的类型和阶段方面,具有较高的准确性。该研究还表明,无细胞的5hmC信号可能被用于跟踪某些癌症类型的肿瘤分期[gydF4y2Ba307gydF4y2Ba].gydF4y2Ba
cfDNA中发现了与癌症相关的5hmC特征[gydF4y2Ba308gydF4y2Ba,gydF4y2Ba309gydF4y2Ba].这些特征是对结直肠癌、胃癌、肺癌和胰腺癌高度可预测的特定癌症类型的特征[gydF4y2Ba307gydF4y2Ba,gydF4y2Ba308gydF4y2Ba].通过分析血液样本,该标记对癌症的诊断和预后也有很大的潜力[gydF4y2Ba308gydF4y2Ba,gydF4y2Ba310gydF4y2Ba].因此,进一步需要优于5hmC的传统生物标志物。gydF4y2Ba
表观遗传学中的常规测序方法gydF4y2Ba
DNA甲基化可以通过以下方法进行评估:用化学转化(亚硫酸氢盐反应)消化DNA、甲基敏感限制性内切酶和甲基化DNA片段的亲和富集[gydF4y2Ba311gydF4y2Ba,gydF4y2Ba312gydF4y2Ba].一种能够从5-甲基胞嘧啶中区分5-羟甲基胞嘧啶、5-甲酰胞嘧啶和5-羧基胞嘧啶的策略很重要,许多策略都有各自的优点和局限性[gydF4y2Ba236gydF4y2Ba].甲基化测序和/或基于微阵列的分析策略与NGS技术一起工作[gydF4y2Ba313gydF4y2Ba].所有用于绘制5mC的表观遗传测序方法都需要与下一代测序协同工作,从而有机会对DNA和RNA进行长读取测序,并且可以立即直接读取修改[gydF4y2Ba314gydF4y2Ba].gydF4y2Ba
亚硫酸氢盐测序(BS-Seq)是基于亚硫酸氢盐处理带来的甲基胞嘧啶和未甲基胞嘧啶的反应性差异,亚硫酸氢盐处理使未甲基胞嘧啶脱胺为尿嘧啶(U),而甲基胞嘧啶保留自身[gydF4y2Ba315gydF4y2Ba],这样,在PCR扩增时,甲基化的胞嘧啶仍然是胞嘧啶,而未甲基化的胞嘧啶将被读出为T [gydF4y2Ba314gydF4y2Ba].gydF4y2Ba
虽然碱基分辨率亚硫酸氢盐法被作为金标准,但到目前为止,由于其化学处理性质苛刻,降解了大部分DNA,限制了生成的表观遗传测序文库[gydF4y2Ba316gydF4y2Ba].亚硫酸氢盐测序有许多完整的错误,从缺失到区分5mC和5hmC [gydF4y2Ba317gydF4y2Ba].亚硫酸氢盐测序还提供了组合信号,如降低序列复杂性导致低制图率、基因组覆盖不均和固有偏差[gydF4y2Ba314gydF4y2Ba,gydF4y2Ba318gydF4y2Ba].这些缺陷的出现是因为哺乳动物基因组中95%的胞嘧啶被转化为胸腺嘧啶[gydF4y2Ba314gydF4y2Ba].碱基分辨率测序中最严重的问题是在亚硫酸氢盐处理过程中,大部分DNA降解,转化效率低。亚硫酸氢盐的转化也是盲的,无法区分5mC和5hmc [gydF4y2Ba319gydF4y2Ba].gydF4y2Ba
除了亚硫酸氢盐技术,已经有了不含亚硫酸氢盐和基级分辨率测序方法,如tet辅助的吡啶硼烷测序(tap),这些方法可用于5mC和5hmC [gydF4y2Ba316gydF4y2Ba,gydF4y2Ba320gydF4y2Ba].tap结合TET氧化5mC和5hmC生成5-羧基胞嘧啶(5caC)与吡啶硼烷还原5caC生成二氢尿嘧啶(东华大学)[gydF4y2Ba321gydF4y2Ba].当PCR将东华大学转化为胸腺嘧啶时,C-to-T转化完成,tap以高灵敏度和特异性直接检测修饰,而不影响未修饰的胞嘧啶[gydF4y2Ba322gydF4y2Ba].该方法可保留长达10个碱基,使甲基组分析更便宜[gydF4y2Ba316gydF4y2Ba].gydF4y2Ba
另一种基于氧化亚硫酸氢盐测序(oxBS-Seq)的方法应用了过酸钾(KRuO)的氧化能力gydF4y2Ba4gydF4y2Ba)生成5fC,经亚硫酸氢盐处理后转化为U,转化率为94.5% [gydF4y2Ba314gydF4y2Ba].最后,通过BS-Seq减去oxBS-Seq得到5hmC水平和位置[gydF4y2Ba323gydF4y2Ba,gydF4y2Ba324gydF4y2Ba,gydF4y2Ba325gydF4y2Ba].过鲁特酸钾比鲁特酸钾更具破坏性,后者更有助于在有限的生物和临床样本中进行纳米级基因组制图[gydF4y2Ba320gydF4y2Ba].该方法据称能够检测健康供体和癌症患者的细胞游离DNA (cfDNA),首次显示人类cfDNA中的基分辨率羟甲基组[gydF4y2Ba314gydF4y2Ba,gydF4y2Ba326gydF4y2Ba].gydF4y2Ba
甲基化的数据分析需要一个具有亚硫酸氢盐测序数据集的高效工具,而最近开发的工具bsword(亚硫酸氢盐模式分析)已经去除了多个/成对序列比对方法,以快速比对序列读取。为了探索DNA甲基化的机制和调控,bspit总结并可视化了DNA甲基化共发生模式[gydF4y2Ba327gydF4y2Ba].gydF4y2Ba
提高方法的可及性和基因组覆盖率的成本是重要的,特别是对具有碱基对分辨率的无亚硫酸盐方法(现在是单分子和单细胞分析)[gydF4y2Ba328gydF4y2Ba].甲基组的大部分可以通过微阵列和全基因组水平的下一代测序技术进行定位,生成基因组样本中5mC及其氧化衍生物的基分辨率图[gydF4y2Ba329gydF4y2Ba,gydF4y2Ba330gydF4y2Ba].为此目的,以亚硫酸氢盐为基础的方法建立了定量方法,如经典的亚硫酸氢盐测序、热测序等[gydF4y2Ba331gydF4y2Ba,gydF4y2Ba332gydF4y2Ba,gydF4y2Ba333gydF4y2Ba].gydF4y2Ba
PCR扩增前,可通过甲基化敏感的限制性内切酶测定单碱基分辨率的CpG甲基化[gydF4y2Ba332gydF4y2Ba].基于亲和的方法也丰富了甲基化区域。但很难达到直接确定的确切地点。此外,亚硫酸氢盐法需要DNA变性,导致DNA降解,降低了其效率[gydF4y2Ba334gydF4y2Ba,gydF4y2Ba335gydF4y2Ba].也有聚合酶链反应导致的亚硫酸氢盐处理DNA的定位效率低和亚硫酸氢盐转化率需要考虑[gydF4y2Ba311gydF4y2Ba].gydF4y2Ba
由于使用亚硫酸氢盐将未甲基胞嘧啶转化为尿嘧啶,文库制备的复杂性和不完全的化学转化偏差增加了[gydF4y2Ba25gydF4y2Ba,gydF4y2Ba336gydF4y2Ba].基于illumina的测序失败的短读取长度,阻碍了等位基因特异性甲基化。另一方面,PacBio长读测序缺乏高序列覆盖率,限制了对甲基化核苷酸的测序。然而,牛津纳米孔测序正成为适应这种情况的最先进的方法[gydF4y2Ba25gydF4y2Ba].gydF4y2Ba
表观遗传肿瘤异质性的纳米孔测序gydF4y2Ba
纳米孔测序促进表观遗传定位gydF4y2Ba
DNA甲基化是最常见的表观遗传修饰之一,可用于表观遗传作图[gydF4y2Ba337gydF4y2Ba,gydF4y2Ba338gydF4y2Ba].甲基化在哺乳动物基因细胞表达中也起着至关重要的作用[gydF4y2Ba339gydF4y2Ba,gydF4y2Ba340gydF4y2Ba].这些作用包括细胞发育、衰老和调节肿瘤抑制基因[gydF4y2Ba341gydF4y2Ba,gydF4y2Ba342gydF4y2Ba,gydF4y2Ba343gydF4y2Ba].然而,大多数DNA测序技术无法区分DNA链中的甲基化和非甲基化核苷酸[gydF4y2Ba25gydF4y2Ba,gydF4y2Ba344gydF4y2Ba].然而,牛津纳米孔MinION测序器的发现使得甲基化调控标记的序列无需特殊的样品制备,且具有长读单分子性质[gydF4y2Ba345gydF4y2Ba].这一特性使得MinION更容易研究异质性癌症样本中的等位基因特异性甲基化[gydF4y2Ba25gydF4y2Ba,gydF4y2Ba54gydF4y2Ba,gydF4y2Ba346gydF4y2Ba].确定了限制,如由于一次有5个核苷酸进入孔而产生的多个核苷酸信号以及当前甲基化和未甲基化碱基的重叠[gydF4y2Ba186gydF4y2Ba].通过设计基本调用者计算隐马尔可夫模型(HMM)软件,可以解决这些缺陷[gydF4y2Ba64gydF4y2Ba,gydF4y2Ba347gydF4y2Ba].基于不同电流分布的可见性,该软件允许区分三种修饰的胞嘧啶(C, 5mC和5hmC)和两种修饰的腺嘌呤变体(A和6-mA) [gydF4y2Ba348gydF4y2Ba,gydF4y2Ba349gydF4y2Ba,gydF4y2Ba350gydF4y2Ba].尽管加入了HMM,但通过由二维(2D)石墨烯或二硫化钼构建的固体纳米孔传感器清晰地检测DNA甲基化也拓宽了该过程的有效性[gydF4y2Ba351gydF4y2Ba].此外,为了检测通过纳米孔的mC核苷酸,通过甲基- cpg结合结构域蛋白(MBD1)的适配器标记DNA甲基化位点也是必须的(图1)。gydF4y2Ba19gydF4y2Ba).gydF4y2Ba
通过纳米孔测序直接读取DNA甲基化。当单链DNA通过小孔时,离子电流发生变化;有一个甲基和由于甲基化而产生的微小变化可以用一套新的算法来解释。图像转载自[gydF4y2Ba25gydF4y2Ba(请求ID: 600061678, 2021年11月27日)gydF4y2Ba
为了研究DNA长片段上的CpG甲基化和染色质可及性,纳米孔测序可以检测测序困难区域,以表征基因组元素,如重复元素[gydF4y2Ba352gydF4y2Ba,gydF4y2Ba353gydF4y2Ba].寻找CCN1基因(结直肠癌中预后不良的相关基因),在三个增强子区域观察到甲基化异质性,增强子3活性最高,负责CCN1上调。破解这个问题的唯一方法是使用长读纳米孔技术[gydF4y2Ba346gydF4y2Ba].通过使用纳米孔测序数据,通过长读染色质可及性测量(nanoNOMe)与CUT和RUN数据配对,产生了最完整的人类甲基组[gydF4y2Ba354gydF4y2Ba,gydF4y2Ba355gydF4y2Ba].低甲基化区域非常难以到达,并与CENP-A/B结合[gydF4y2Ba354gydF4y2Ba].然而,存在于X染色体失活的复杂宏观卫星阵列中的长reads查询等位基因特异性的远程表观遗传模式是可以被破译的。这种基于表观遗传异质性和同质性甲基化状态的单分子测量簇读数为研究人类基因组中最模糊的区域提供了一个框架[gydF4y2Ba354gydF4y2Ba].gydF4y2Ba
用高通量测序技术增强DNA亚硫酸氢盐方法扩大了全基因组DNA甲基化的范围,而不仅仅局限于CpG位点和CpG岛[gydF4y2Ba356gydF4y2Ba,gydF4y2Ba357gydF4y2Ba].全基因组DNA甲基化研究表明,在启动子、cgi和各自的元素等基因组位点上存在差异甲基化[gydF4y2Ba358gydF4y2Ba].这些差异甲基化是产生异质性的各种克隆细胞群的来源[gydF4y2Ba359gydF4y2Ba,gydF4y2Ba360gydF4y2Ba].识别修饰的最简单的方法对表观遗传学有积极的影响,并与亚硫酸氢盐测序具有良好的重复性和相关性。有人建议说,纳米孔测序可能成为检测甲基化模式的金标准。由于短读亚硫酸氢盐测序需要差异甲基化评估,我们目前在长读测序中缺乏的统计方法甚至可以扩展到单倍型中的纳米孔测序修饰[gydF4y2Ba77gydF4y2Ba,gydF4y2Ba361gydF4y2Ba].gydF4y2Ba
methqa软件包解决了使用亚硫酸氢盐转化技术将未甲基化胞嘧啶转化为U和T时出现的故障[gydF4y2Ba360gydF4y2Ba,gydF4y2Ba362gydF4y2Ba].在该软件的帮助下,NGS技术可以输出存在质量问题的甲基化测序数据,如3 '端碱基测序低、PCR扩增偏倚和亚硫酸氢盐转化率低[gydF4y2Ba362gydF4y2Ba,gydF4y2Ba363gydF4y2Ba].gydF4y2Ba
5hmC检测局限性阻碍了对5hmC生理功能及其在去甲基化途径中的作用的评估[gydF4y2Ba364gydF4y2Ba].这种限制也影响了5hmC的深层识别作用:定位、转录调控、复制和表观遗传重编程[gydF4y2Ba365gydF4y2Ba].因此,这种5hmC功能的确定需要纳米孔测序最适合的单分子DNA测序技术的发展[gydF4y2Ba365gydF4y2Ba,gydF4y2Ba366gydF4y2Ba].gydF4y2Ba
通过纳米孔测序检测表观遗传修饰的准确性测量gydF4y2Ba
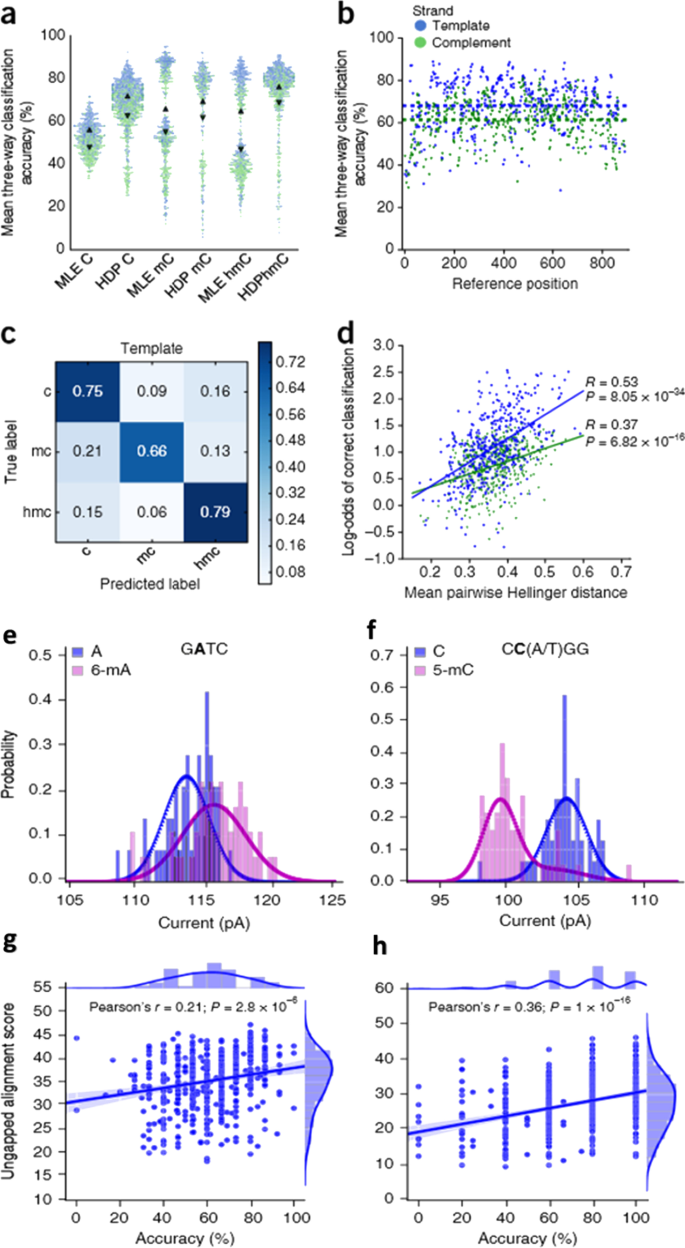
在上述讨论的方法之外,牛津MinION纳米孔测序模型和HMM(隐马尔可夫模型)据报道具有区分胞嘧啶所有修饰碱基的能力[gydF4y2Ba63gydF4y2Ba,gydF4y2Ba347gydF4y2Ba].在改进HMM的基础上,建立了HMM- hdp (hidden Markov model With hierarchical Dirichlet process)模型,结合MinION测序检测的修饰碱基的精度测量(图1)。gydF4y2Ba20.gydF4y2Ba模拟)[gydF4y2Ba64gydF4y2Ba,gydF4y2Ba348gydF4y2Ba,gydF4y2Ba367gydF4y2Ba].该模型区分所有五个CgydF4y2Ba5gydF4y2Ba基于低通量纳米孔传感器离子电流测量的胞嘧啶变体[gydF4y2Ba368gydF4y2Ba].在HMM-HDP中,碱基修饰被检测为ONT-MinION离子电流信号的变化。MinION经常记录离子电流,将它们分成称为事件的段。该设计将每个事件建模为一个长度为K-mer的核苷酸打击[gydF4y2Ba369gydF4y2Ba].每个K-mer都与Picomas (PgydF4y2Ba一个gydF4y2Ba).单独的C, mC和hmC碱基从合成的核苷酸区域分类,通过离子电流信号的变化来测量检测的准确性。在检测到模型的变化后,必须测量离子电流信号的分布,以确定偏析强度(图1)。gydF4y2Ba20.gydF4y2Ba情况)。该模型还包含了来自CC(A/T) GG基元的5mC映射和来自G的6ma映射gydF4y2Ba一个gydF4y2BaTC图案使用gydF4y2Ba大肠杆菌gydF4y2Ba基因组DNA (gydF4y2Ba367gydF4y2Ba].gydF4y2Ba
模拟gydF4y2BaMinION检测合成寡核苷酸中发现的胞嘧啶甲基化变异的准确性结果。6966 C、294 5mC和467 5hmC链的分类输出在类似的MinION流细胞中进行了测序。gydF4y2Ba一个gydF4y2Ba通过比较最大似然估计(MLE)和HDP模型分布的正态分布表示预读精度分布结果。分布用三角形表示。gydF4y2BabgydF4y2Ba在所有引用的三种分类(C, mC和hmC)的模板和共模板读取。gydF4y2BacgydF4y2Ba混淆矩阵显示HMM-HDP三向胞嘧啶分类对合成寡核苷酸模板读取的性能。gydF4y2BadgydF4y2Ba重叠在胞嘧啶上的6-聚体分布的甲基化状态的对数比值与平均两两海灵格距离之间的相关性。gydF4y2BaegydF4y2Ba- - - - - -gydF4y2BahgydF4y2Ba左(GATC为6 mA)和右(CC(A/T) GG为5mC)基序的离子电流分布变化与阅读质量的影响。A和6-mA之间的离子电流分布gydF4y2BaegydF4y2Ba)和C及5mC (gydF4y2BafgydF4y2Ba)显示出了差异。离子电流水平从100校准显示为直方图gydF4y2BaggydF4y2BaA和6-mAgydF4y2BahgydF4y2Ba学习到的HDP概率密度用曲线表示。图像转载自[gydF4y2Ba348gydF4y2Ba(请求ID 600061677, 2021年11月27日)gydF4y2Ba
利用纳米孔测序器进行单细胞肿瘤表观遗传定位gydF4y2Ba
单细胞表观基因组学领域正处于起步阶段。但是,由于随着技术的快速发展,肿瘤细胞间异质性的重要性越来越被认识到,预计在未来几年将取得巨大进展[gydF4y2Ba370gydF4y2Ba].单细胞表观基因组学将表观遗传学分析与单细胞的分离、条形码和分离细胞基因组的高通量测序结合在一起[gydF4y2Ba371gydF4y2Ba].由于表观基因修饰的基因出现在大多数癌细胞中,因此必须使用简单和低成本的方法来识别这些修饰[gydF4y2Ba372gydF4y2Ba].采用最新升级技术的纳米孔测序是检测各种器官特定癌症类型中发生的表观遗传修饰的更简单和更好的方法[gydF4y2Ba373gydF4y2Ba].gydF4y2Ba
DNA序列中核苷酸的缺失、扩增、倒置和易位是导致基因突变的四个与DNA复制相关的原因。纳米孔测序可用于检测这些变化导致的肿瘤异质性,这导致了表观遗传修饰过程中预期的改变[gydF4y2Ba391gydF4y2Ba].此外,纳米孔测序被认为是理解癌症异质性的下一代方法的主要重点领域之一[gydF4y2Ba392gydF4y2Ba].gydF4y2Ba
除了先前公认的遗传改变外,由表观遗传重编程产生的肿瘤异质性导致了肿瘤细胞的耐药亚群[gydF4y2Ba374gydF4y2Ba].这表明需要单细胞表观遗传技术的能力来装载药物诱导的肿瘤进化,以便及时调解治疗[gydF4y2Ba293gydF4y2Ba].在肝细胞癌中,使用纳米孔测序对肿瘤抑制基因的修饰状态进行鉴定,发现约有10个潜在的肿瘤抑制基因候选基因和葡萄糖激酶基因,更多被证实参与了HCC的发展[gydF4y2Ba375gydF4y2Ba].纳米孔测序可实现全基因组测序,并可能识别肺癌细胞系LC2/ad基因的表观遗传修饰[gydF4y2Ba376gydF4y2Ba].它还允许检测各种癌症类型的表观遗传修饰基因gydF4y2Ba5gydF4y2Ba).gydF4y2Ba
纳米孔技术在表观遗传学-癌症领域的主要成果gydF4y2Ba
纳米孔测序(NGS)作为一种癌症研究工具仍处于起步阶段,在分子癌症研究中的应用尤其缺乏。当然,NGS技术更适合用于植物科学和微生物学等领域的研究。然而,利用细胞系作为研究媒介正逐渐应用于人类样本[gydF4y2Ba395gydF4y2Ba,gydF4y2Ba396gydF4y2Ba,gydF4y2Ba397gydF4y2Ba].即使测序克服了一些障碍,在检测全基因组DNA改变方面仍有改进和对标计算机技术的机会[gydF4y2Ba398gydF4y2Ba].这表明,迫切需要一个能够预测CpG甲基化在多个基因组背景下的基准,特别是那些涉及肿瘤异质性和肿瘤抑制的基因。gydF4y2Ba
DNA片段拷贝上的表观基因组模式已被用作先兆,而纳米孔测序仍在与旧的标准方法同时使用。这些模式是通过纳米孔测序确定的,并允许分配单倍型的reads,以实现四种人类细胞系(GM12878, MCF-10°,MCF-7和MDA-MB-231)上CpG甲基化和染色质可及性的染色体水平等位基因特异性图谱,这是核小体定位和DNA可及性的决定因素。然后扩展纳米孔测序的应用,发现乳腺癌模型肿瘤的异质性[gydF4y2Ba346gydF4y2Ba].由于纳米孔测序能够识别核苷酸并对其进行测序,即使核苷酸发生了微小的变化,纳米孔测序正逐渐成为统治测序市场的标准[gydF4y2Ba399gydF4y2Ba].希望未来的甲基化映射完整软件,如NanoMetPhase,能够提供检测5mC和6ma的第二个深度信号。该软件采用2 ×覆盖率来寻找任何DNA碱基甲基化状态,这些状态是更准确检测肿瘤异质性的可靠标记[gydF4y2Ba400gydF4y2Ba].这种培养将支持基于纳米孔的计算和实验应用方法的并行实施。gydF4y2Ba
Oxford Nanopore可用于全基因组测序,以确定肝癌中的插入、缺失、倒置和染色体内易位,然后可用于表观基因组分析,因为该仪器允许并行基因组和表观基因组测序,以确定肿瘤细胞的复杂异质性和变异[gydF4y2Ba401gydF4y2Ba].这种口袋大小的纳米孔测序装置的神奇之处是,在同一天对低通全基因组的基因组和表观基因组进行同时测序,从同一次测序中生成诊断拷贝数(CN)和甲基化图谱。这是利用纳米孔进行癌症重要分子分类的开始,为临床提供更好的诊断、预后和治疗决策。gydF4y2Ba75gydF4y2Ba].另一项研究发现,纳米孔cas9靶向测序(nCATS)更有效地检测弥漫性胶质瘤中异柠檬酸脱氢酶1和2 (IDH1, IDH2)和o6 -甲基鸟嘌呤dna甲基转移酶(MGMT)的突变和36 h内甲基化状态[gydF4y2Ba402gydF4y2Ba].Cas9突变和测序文库创建的结合似乎是目前最有效的耦合,它可以帮助识别单核苷酸变异(SNVs)、结构变异(SVs)和CpG甲基化[gydF4y2Ba403gydF4y2Ba].为了实现远程扩增和纳米孔测序,使用cas9辅助靶向染色体片段(CATCH)方法从外周血细胞中分离出BRCA1乳腺癌基因体和侧翼区域。我们有理由认为,这项技术最终将在医务室和病人的口袋里使用[gydF4y2Ba404gydF4y2Ba].肿瘤特异性的LINE-1插入及其逆转录转座子标记的表观基因组测序是至关重要的,因为CpG甲基化控制着肿瘤生长进化过程中涉及的转座子元件(Tes)。[gydF4y2Ba405gydF4y2Ba]gydF4y2Ba
研究发现,用于术中神经病理分类的纳米孔全基因组测序可提高影响手术决策的实际术中诊断准确性[gydF4y2Ba406gydF4y2Ba],因此,随着之前使用化学方法和Illumina完成的全基因组分析中表观基因组肿瘤特征的数据积累,现在随着纳米孔测序的快速上升,将成为背景。gydF4y2Ba
为了对癌症表观遗传异质性的高水平识别,纳米孔测序一般都走在与纳米结构组分/材料(如玻璃纳米微管、纳米吸管、碳纳米管探针和其他纳米材料)连接的道路上[gydF4y2Ba381gydF4y2Ba].通过在细胞膜的胞内和胞外部分之间构建通道,这些纳米组件可通过单细胞采样促进测序[gydF4y2Ba382gydF4y2Ba].与这些纳米组分类似,亚硫酸氢盐测序在单细胞水平的应用解决了具有显著DNA降解的肿瘤细胞的内部或内部异质性[gydF4y2Ba382gydF4y2Ba].为了在未来的癌症治疗中准确识别基因的异质性,建议结合纳米孔测序、纳米结构组分和亚硫酸氢盐测序或直接测序进行研究。gydF4y2Ba
总结与未来展望gydF4y2Ba
表观遗传学是一种重要的基因调节剂,需要进行彻底的测序。如果没有表观遗传学的测序,未来基于多组学的医学将是不完整的,特别是在癌症生物学的背景下。此外,研究和个性化的循证医疗服务将受益于将表观遗传学作为诊断的生物标志物和作为药物靶点。癌症的异质性受表观遗传学的影响,这使得表观遗传学测序至关重要。到目前为止,测序都是采用传统的方法,但在未来,纳米孔测序将是一种更专业的方法。根据早期的研究,牛津纳米孔测序器是推进基因组和表观基因组测序的最佳方法,在多组学领域展示表观遗传学时,它比竞争对手的测序技术具有更多优势。此外,牛津纳米孔技术公司(Oxford Nanopore Technologies)允许直接测序而不需要大量试剂,比任何其他测序设备都更适合探索表观遗传学在癌症异质性中的作用。gydF4y2Ba
在多组学时代,牛津纳米孔测序技术将非常有效地展示表观遗传学的一个分支和基因组学的另一个分支。牛津纳米孔测序是一种快速发展的测序方法,对Illumina公司的测序技术构成了激烈的挑战。由于尺寸和价格的降低,Oxford Nanopore测序技术有望在几个方面超越Illumina测序技术。因此,一个单一的纳米孔测序平台可以进行表观基因组学、基因组学、转录组学和蛋白质组学。gydF4y2Ba
最后,未来的癌症医学研究将需要考虑将不同的纳米生物材料与纳米孔测序技术相结合,以便以更准确的方式检测癌症的表观遗传学。除了加入生物材料外,纳米组合测序程序还必须考虑到临床可行性和传递机制。gydF4y2Ba
数据可用性gydF4y2Ba
本审稿中的数据(数字和表格)已获得出版者的许可。gydF4y2Ba
缩写gydF4y2Ba
- 5 cac:gydF4y2Ba
-
5-CarboxylcytosinegydF4y2Ba
- 5 fc:gydF4y2Ba
-
5-FluorocytosinegydF4y2Ba
- 5 hmc:gydF4y2Ba
-
5-HydroxymethylcytosinegydF4y2Ba
- 主持人:5gydF4y2Ba
-
5-MethylcytosinegydF4y2Ba
- 国际清算银行/ oxBis:gydF4y2Ba
-
亚硫酸氢/氧化亚硫酸氢盐测序gydF4y2Ba
- BSPAT:gydF4y2Ba
-
亚硫酸氢模式分析gydF4y2Ba
- 国开行:gydF4y2Ba
-
控制介质击穿gydF4y2Ba
- CIMP:gydF4y2Ba
-
CpG岛甲基化表型gydF4y2Ba
- clyA:gydF4y2Ba
-
溶细胞素一gydF4y2Ba
- CpG岛:gydF4y2Ba
-
胞嘧啶后面是鸟嘌呤残基gydF4y2Ba
- 儿童权利公约:gydF4y2Ba
-
结肠直肠癌gydF4y2Ba
- CsgG:gydF4y2Ba
-
咖喱特异基因产物GgydF4y2Ba
- DNMT:gydF4y2Ba
-
DNA甲基转移酶gydF4y2Ba
- 帽子:gydF4y2Ba
-
组蛋白乙酰转移酶gydF4y2Ba
- 肝细胞癌:gydF4y2Ba
-
肝细胞癌gydF4y2Ba
- HDAC:gydF4y2Ba
-
组蛋白去乙酰酶抑制剂gydF4y2Ba
- HDMT:gydF4y2Ba
-
组蛋白demethylasesgydF4y2Ba
- 计画:gydF4y2Ba
-
人类基因组计划gydF4y2Ba
- hMeDIP-seq:gydF4y2Ba
-
Hydroxymethylation DNA免疫沉淀反应gydF4y2Ba
- HMT:gydF4y2Ba
-
组蛋白甲基转移酶gydF4y2Ba
- 嗯:gydF4y2Ba
-
隐马尔可夫模型gydF4y2Ba
- HMM-HDP:gydF4y2Ba
-
具有层次Dirichlet过程的隐马尔可夫模型gydF4y2Ba
- KF:gydF4y2Ba
-
大片段gydF4y2Ba
- LncRNAs:gydF4y2Ba
-
长非编码rnagydF4y2Ba
- MspA:gydF4y2Ba
-
耻垢分枝杆菌孔隙素AgydF4y2Ba
- 美国/ NfpB:gydF4y2Ba
-
法氏诺卡菌肽A/BgydF4y2Ba
- 门店:gydF4y2Ba
-
新一代测序gydF4y2Ba
- OmpF:gydF4y2Ba
-
外膜蛋白FgydF4y2Ba
- OmpG:gydF4y2Ba
-
外膜蛋白GgydF4y2Ba
- 游客:gydF4y2Ba
-
牛津纳米孔技术gydF4y2Ba
- 聚合酶链反应:gydF4y2Ba
-
聚合酶链反应gydF4y2Ba
- PSD:gydF4y2Ba
-
功率谱密度gydF4y2Ba
- 萨哈:gydF4y2Ba
-
Suberoyl苯胺bishydroxamidegydF4y2Ba
- SBS:gydF4y2Ba
-
序列的合成gydF4y2Ba
- SGS:gydF4y2Ba
-
第二代测序gydF4y2Ba
- SMRT:gydF4y2Ba
-
单分子实时gydF4y2Ba
- 短信:gydF4y2Ba
-
单分子测序gydF4y2Ba
- 蜗牛:gydF4y2Ba
-
用于长测序的慢速核酸仪器gydF4y2Ba
- 信噪比:gydF4y2Ba
-
信噪比gydF4y2Ba
- T2T:gydF4y2Ba
-
Telomere-to-telomeregydF4y2Ba
- TAB-Seq:gydF4y2Ba
-
TET-assisted重亚硫酸盐测序gydF4y2Ba
- 水龙头:gydF4y2Ba
-
tet辅助吡啶硼烷测序gydF4y2Ba
- 春节:gydF4y2Ba
-
十十一个易位家族蛋白质gydF4y2Ba
- 运输安全管理局:gydF4y2Ba
-
Trichostatin一gydF4y2Ba
- 次数:gydF4y2Ba
-
肿瘤抑制基因gydF4y2Ba
- αhl:gydF4y2Ba
-
α溶血素gydF4y2Ba
参考文献gydF4y2Ba
沃森JD,克里克FH。DNA的结构。见:冷泉港定量生物学专题讨论会,第18卷。冷泉港实验室出版社;1953.123 - 31页gydF4y2Bahttps://doi.org/10.1101/SQB.1953.018.01.020gydF4y2Ba.gydF4y2Ba
达姆·r·弗里德里希·米舍尔和DNA的发现。Dev杂志。2005;278:274 - 88。gydF4y2Bahttps://doi.org/10.1016/j.ydbio.2004.11.028gydF4y2Ba.gydF4y2Ba
克鲁格·a·罗莎琳德·富兰克林和DNA结构的发现。大自然。1968;219:808-10。gydF4y2Bahttps://doi.org/10.1038/219808a0gydF4y2Ba.gydF4y2Ba
男AE, Wilson P, Siemering K, Forrest S. Sanger DNA测序。In:下一代基因组测序:走向个性化医疗;2008.1 - 11页。gydF4y2Bahttps://doi.org/10.1002/9783527625130gydF4y2Ba.gydF4y2Ba
Baudhuin LM, Lagerstedt SA, Klee EW, Fadra N, Oglesbee D, Ferber MJ。通过Sanger测序确认下一代测序面板试验中的变体。中华医学杂志。2015;17:456-61。gydF4y2Bahttps://doi.org/10.1016/j.jmoldx.2015.03.004gydF4y2Ba.gydF4y2Ba
França LT, Carrilho E, Kist TB。DNA测序技术综述。生物物理学报2002;35:169-200。gydF4y2Bahttps://doi.org/10.1017/S0033583502003797gydF4y2Ba.gydF4y2Ba
一种DNA测序的新方法。科学通报1977;74:560-4。gydF4y2Bahttps://doi.org/10.1073/pnas.74.2.560gydF4y2Ba.gydF4y2Ba
王晓燕,王晓燕。DNA测序的进化。2015; 25:210-5。gydF4y2Ba
坂本芳,铃木E,藤井Y.用Sanger法有效测定DNA分裂位点的新方法。生物化学与生物物理方法。2002;52:97-109。gydF4y2Bahttps://doi.org/10.1016/s0165 - 022 x (02) 00053 - 2gydF4y2Ba.gydF4y2Ba
王晓燕,王晓燕。基因组测序。生物信息学》2016。gydF4y2Bahttps://doi.org/10.1007/978-1-4939-6622-6_1gydF4y2Ba.gydF4y2Ba
Collins FS, McKusick VA.人类基因组计划对医学科学的影响。《美国医学协会杂志》上。2001; 285:540-4。gydF4y2Bahttps://doi.org/10.1001/jama.285.5.540gydF4y2Ba.gydF4y2Ba
Lunshof JE, Bobe J, Aach J, Angrist M, Thakuria JV, Vorhaus DB, Hoehe MR, Church GM.个人基因组进展:从人类基因组计划到个人基因组计划。临床神经科学。2010;12:47。gydF4y2Bahttps://doi.org/10.31887/DCNS.2010.12.1/jlunshofgydF4y2Ba.gydF4y2Ba
Powledge TM。人类基因组计划完成。基因组医学杂志。2003;4:1-3。gydF4y2Bahttps://doi.org/10.1186/gb-spotlight-20030415-01gydF4y2Ba.gydF4y2Ba
Collins FS, Morgan M, Patrinos A.人类基因组计划:大规模生物学的教训。科学。2003;300:286 - 90。gydF4y2Bahttps://doi.org/10.1126/science.1084564gydF4y2Ba.gydF4y2Ba
胡涛,Chitnis N, Monos D, Dinh A.下一代测序技术综述。哼Immunol》2021。gydF4y2Bahttps://doi.org/10.1016/j.humimm.2021.02.012gydF4y2Ba.gydF4y2Ba
Miga KH, Koren S, Rhie A, Vollger MR, Gershman A, Bzikadze A, Brooks S, Howe E, Porubsky D, Logsdon GA, Schneider VA.完整的人类X染色体端粒到端粒的组装。大自然。2020;585:79 - 84。gydF4y2Bahttps://doi.org/10.1038/s41586-020-2547-7gydF4y2Ba.gydF4y2Ba
冯燕,张颖,应春,王栋,杜聪。基于纳米孔的第四代DNA测序技术。Genom Proteom Bioinform 2015; 13:4-16。gydF4y2Bahttps://doi.org/10.1016/j.gpb.2015.01.009gydF4y2Ba.gydF4y2Ba
Göpfrich K, K法官用口袋大小的测序仪解码DNA。健康杂志。2018;43:17-20。gydF4y2Ba
Hoenen T, Groseth A, Rosenke K, Fischer RJ, Hoenen A, Judson SD, Martellaro C, Falzarano D, Marzi A, Squires RB, Wollenberg KR.纳米孔测序作为一种快速部署的埃博拉爆发工具。《新兴感染杂志》2016;22:331。gydF4y2Bahttps://doi.org/10.3201/eid2202.151796gydF4y2Ba.gydF4y2Ba
Hall CL, Zascavage RR, Sedlazeck FJ, Planz JV。纳米孔测序在法医分析中的潜在应用。法医科学评论,2020;32:23-54。gydF4y2Ba
颜颖,吴凯,陈娟,刘辉,黄颖,张颖,熊娟,权伟,吴霞,梁颖,何坤。利用扩增子-牛津纳米孔测序快速获取高质量SARS-CoV-2基因组。性罪。2021。gydF4y2Bahttps://doi.org/10.1007/s12250-021-00378-8gydF4y2Ba.gydF4y2Ba
王敏,付安,胡斌,童勇,刘锐,刘铮,顾杰,项斌,刘杰,蒋伟,沈刚。纳米孔靶向测序技术用于SARS-CoV-2等呼吸道病毒的精确、全面检测。小。2020;16:2002169。gydF4y2Bahttps://doi.org/10.1002/smll.202002169gydF4y2Ba.gydF4y2Ba
郭敏,彭毅,高阿,杜超,赫尔曼金刚。癌症的表观遗传异质性。Biomark杂志2019;7:1-19。gydF4y2Bahttps://doi.org/10.1186/s40364-019-0174-ygydF4y2Ba.gydF4y2Ba
耐药、表观遗传学和肿瘤细胞异质性。中华医学杂志2010;2:19-28。gydF4y2Bahttps://doi.org/10.1126/scitranslmed.3001056gydF4y2Ba.gydF4y2Ba
纳米孔测序符合表观遗传学。Nat方法。2017;14:347-8。gydF4y2Bahttps://doi.org/10.1038/nmeth.4240gydF4y2Ba.gydF4y2Ba
Brero A, Easwaran HP, Nowak D, Grunewald I, Cremer T, Leonhardt H, Cardoso MC.甲基cpg结合蛋白在末端分化过程中诱导大规模染色质重组。细胞生物学杂志2005;169:733-74。gydF4y2Bahttps://doi.org/10.1083/jcb.200502062gydF4y2Ba.gydF4y2Ba
弗格森LR,塔瑟姆AL,林Z,丹尼WA。作为抗癌药物靶点的基因表达的表观遗传调控。《当代癌症药物靶点》2011;11:199-212。gydF4y2Ba
Beckmann ND, Karri S, Fang G, Bashir A.在低覆盖率和宏基因组设置中检测表观遗传基序。BMC Bioinform。2014;15:1-12。gydF4y2Bahttps://doi.org/10.1186/1471-2105-15-S9-S16gydF4y2Ba.gydF4y2Ba
Krishna BM, Khan MA, Khan ST.下一代测序(NGS)平台:基因组序列分析令人兴奋的时代。微生物基因支持农业生态系统,2019。gydF4y2Bahttps://doi.org/10.1007/978-981-32-9860-6_6gydF4y2Ba.gydF4y2Ba
古普塔AK古普塔UD。下一代测序及其应用。生物科技动物》。2020。gydF4y2Bahttps://doi.org/10.1016/B978-0-12-811710-1.00018-5gydF4y2Ba.gydF4y2Ba
林斌,惠娟,毛辉。纳米孔技术及其在基因测序中的应用。生物传感器。2021;11:214。gydF4y2Bahttps://doi.org/10.3390/bios11070214gydF4y2Ba.gydF4y2Ba
和记黄埔CA三世。DNA测序:从实验室到床边,甚至更远。核酸决议2007;35:6227-37。gydF4y2Bahttps://doi.org/10.1093/nar/gkm688gydF4y2Ba.gydF4y2Ba
沃森JD。人类基因组计划:过去、现在和未来。科学。1990;248:44-9。gydF4y2Bahttps://doi.org/10.1126/science.2181665gydF4y2Ba.gydF4y2Ba
建立大规模生物学:在人类基因组计划的早期建立一个管理体制。BioSocieties。2013; 8:397 - 416。gydF4y2Bahttps://doi.org/10.1057/biosoc.2013.31gydF4y2Ba.gydF4y2Ba
Herlihy W.人类基因组计划。肛门化学1991;63:416a - 423 a。gydF4y2Bahttps://doi.org/10.1021/ac00007a740gydF4y2Ba.gydF4y2Ba
古德温S,麦克弗森JD,麦库姆比WR。成熟:下一代测序技术的十年。Nat Rev Genet. 2016;17:33 - 51。gydF4y2Bahttps://doi.org/10.1038/nrg.2016.49gydF4y2Ba.gydF4y2Ba
Schloss JA, Gibbs RA, Makhijani VB, Marziali A.人类基因组工程后培育DNA测序技术。Annu Rev Genom Hum Genet。2020;21:117-38。gydF4y2Bahttps://doi.org/10.1146/annurev-genom-111919-082433gydF4y2Ba.gydF4y2Ba
中野K, Shiroma A, Shimoji M, Tamotsu H, Ashimine N,等。SMRT长读测序仪在医学领域基因组测序的优势。哼。2017;30:149 - 61。gydF4y2Bahttps://doi.org/10.1007/s13577-017-0168-8gydF4y2Ba.gydF4y2Ba
Shokralla S, Spall JL, Gibson JF, Hajibabaei M.环境DNA研究的下一代测序技术。摩尔生态。2012;21:1794 - 805。gydF4y2Bahttps://doi.org/10.1111/j.1365-294X.2012.05538.xgydF4y2Ba.gydF4y2Ba
Balzer S, Malde K, Lanzén A, Sharma A, Jonassen I.使用flowsim实现454焦磷酸测序数据的现实模拟的特征。生物信息学。2010;26:i420-5。gydF4y2Bahttps://doi.org/10.1093/bioinformatics/btr384gydF4y2Ba.gydF4y2Ba
Kchouk M, Gibrat JF, Elloumi M.一代一代的测序技术:从第一代到下一代。生物医学杂志》2017。gydF4y2Bahttps://doi.org/10.4172/0974-8369.1000395gydF4y2Ba.gydF4y2Ba
Shuikan A, Alharbi SA, Alkhalifah DHM, Hozzein WN。高通量测序和宏基因组数据分析。见:宏基因组学的基础、方法和应用。IntechOpen;2019.gydF4y2Bahttps://doi.org/10.5772/intechopen.78746gydF4y2Ba.gydF4y2Ba
Schatz MC, Delcher AL, Salzberg SL.使用第二代测序组装大基因组。基因组研究》2010;20:1165 - 73。gydF4y2Bahttps://doi.org/10.1101/gr.101360.109gydF4y2Ba.gydF4y2Ba
Schadt EE, Turner S, Kasarskis a . R2,第三代测序的窗口。中华医学杂志2010;19:R227-40。gydF4y2Bahttps://doi.org/10.1093/hmg/ddq481gydF4y2Ba.gydF4y2Ba
肖涛,周伟。第三代测序:遗传疾病的先进方法。Transl Pediatr。2020;9:163。gydF4y2Bahttps://doi.org/10.21037/tp.2020.03.06gydF4y2Ba.gydF4y2Ba
Niedringhaus TP, Milanova D, Kerby MB, Snyder MP, Barron AE。下一代测序技术的前景。肛门化学2011;83:4327-41。gydF4y2Bahttps://doi.org/10.1021/ac2010857gydF4y2Ba.gydF4y2Ba
第三代DNA测序:太平洋生物科学的单分子实时技术。化学杂志。2010;17:675-6。gydF4y2Bahttps://doi.org/10.1016/j.chembiol.2010.07.004gydF4y2Ba.gydF4y2Ba
Shendure J, Balasubramanian S, Church GM, Gilbert W, Rogers J, Schloss JA, Waterston RH。40岁的DNA测序:过去,现在和未来。大自然。2017;550:345-53。gydF4y2Bahttps://doi.org/10.1038/nature24286gydF4y2Ba.gydF4y2Ba
彭晓燕,李晓燕。下一代临床遗传学测序分析的生物信息学和计算工具。中华临床杂志。2020;9:132。gydF4y2Bahttps://doi.org/10.3390/jcm9010132gydF4y2Ba.gydF4y2Ba
介绍应用太平洋生物科学技术(Iso-Seq)的亚型测序。在:转录组学和基因调控。施普林格;2016.141 - 60页。gydF4y2Bahttps://doi.org/10.1007/978-94-017-7450-5_6gydF4y2Ba.gydF4y2Ba
Koren S, Schatz MC, Walenz BP, Martin J, Howard JT, Ganapathy G, Wang Z, Rasko DA, McCombie WR, Jarvis ED, Phillippy AM。混合误差校正和单分子测序读数从头组装。生物科技Nat》。2012;30:693 - 700。gydF4y2Bahttps://doi.org/10.1038/nbt.2280gydF4y2Ba.gydF4y2Ba
Schloss PD, Jenior ML, Koumpouras CC, Westcott SL, Highlander SK.使用PacBio SMRT DNA测序系统对16S rRNA基因片段进行测序。PeerJ。2016; 4:1869。gydF4y2Bahttps://doi.org/10.7717/peerj.1869gydF4y2Ba.gydF4y2Ba
阿里Ş,阿里坎M.下一代测序:优点,缺点和未来。见:植物组学:趋势和应用。可汗:施普林格;2016.109 - 35页。gydF4y2Bahttps://doi.org/10.1007/978-3-319-31703-8_5gydF4y2Ba.gydF4y2Ba
Rhoads A, Au KF。PacBio测序及其应用。Genom Proteom Bioinform. 2015; 13:278-89。gydF4y2Bahttps://doi.org/10.1016/j.gpb.2015.08.002gydF4y2Ba.gydF4y2Ba
Raley C, Munroe D, Jones K, Tsai YC, Guo Y, Tran B, Gowda S, Troyer JL, Soppet DR, Stewart C, Stephens R.从超低输入DNA制备下一代DNA测序文库:应用于太平洋生物科学RS II上的单分子实时(SMRT)测序。bioRxiv。2014.gydF4y2Bahttps://doi.org/10.1101/003566gydF4y2Ba.gydF4y2Ba
Ardui S, Ameur A, Vermeesch JR, Hestand MS.单分子实时(SMRT)测序成熟:医疗诊断的应用和实用。核酸决议2018;46:2159-68。gydF4y2Bahttps://doi.org/10.1093/nar/gky066gydF4y2Ba.gydF4y2Ba
Eid J, Fehr A, Gray J, Luong K, Lyle J, Otto G, Peluso P, Rank D, Baybayan P, Bettman B, Bibillo A.单聚合酶分子的实时DNA测序。科学。2009;323:133-8。gydF4y2Bahttps://doi.org/10.1126/science.1162986gydF4y2Ba.gydF4y2Ba
Niedringhaus TP, Milanova D, Kerby MB, Snyder MP, Barron AE。下一代测序技术的前景。肛门化学2011;83:4327-41。gydF4y2Bahttps://doi.org/10.1021/ac2010857gydF4y2Ba.gydF4y2Ba
李颖。生物学研究中的现代表观遗传学方法。方法。2021;187:104-13。gydF4y2Bahttps://doi.org/10.1016/j.ymeth.2020.06.022gydF4y2Ba.gydF4y2Ba
吕维希,王晓东,王晓东,等。单细胞水平的染色质修饰。发展。2019;146:170217。gydF4y2Bahttps://doi.org/10.1242/dev.170217gydF4y2Ba.gydF4y2Ba
Hagan JT, Sheetz BS, Bandara YND, Karawdeniya BI, Morris MA, Chevalier RB, Dwyer JR.用于单分子传感和糖基的化学定制纳米孔。肛门生物肛门化学。2020;412:6639-54。gydF4y2Bahttps://doi.org/10.1007/s00216-020-02717-2gydF4y2Ba.gydF4y2Ba
王勇,张敏,张敏。纳米孔技术在生物医学中的应用。生物医学微型装置。1999;2:11-40。gydF4y2Bahttps://doi.org/10.1023/A:1009903215959gydF4y2Ba.gydF4y2Ba
吕宏,Giordano F,宁卓。Oxford Nanopore MinION测序及基因组组装。Genom Proteom Bioinform. 2016; 14:65 - 79。gydF4y2Bahttps://doi.org/10.1016/j.gpb.2016.05.004gydF4y2Ba.gydF4y2Ba
Jain M, Olsen HE, Paten B, Akeson M.牛津纳米孔MinION:向基因组学社区提供纳米孔测序。基因组医学杂志。2016;17:1-11。gydF4y2Bahttps://doi.org/10.1186/s13059-016-1103-0gydF4y2Ba.gydF4y2Ba
Laver T, Harrison J, O 'Neill PA, Moore K, Farbos A, Paszkiewicz K, Studholme DJ。评估牛津纳米孔技术的小黄人的性能。生物生物检测量化。2015;3:1-8。gydF4y2Bahttps://doi.org/10.1016/j.bdq.2015.02.001gydF4y2Ba.gydF4y2Ba
Bowden R, Davies RW, Heger A, Pagnamenta AT, de Cesare M, Oikkonen LE, Parkes D, Freeman C, Dhalla F, Patel SY, Popitsch N.利用纳米孔技术对人类基因组进行测序。Nat Commun。2019;10:1-9。gydF4y2Bahttps://doi.org/10.1038/s41467-019-09637-5gydF4y2Ba.gydF4y2Ba
麦特拉RD,金J,邓巴WB。纳米孔测序的最新进展。电泳。2012;33:3418-28。gydF4y2Bahttps://doi.org/10.1002/elps.201200272gydF4y2Ba.gydF4y2Ba
王iy, Bhatia SN, Toner M.纳米技术:生物和医学的新兴工具。基因Dev。2013;27:2397 - 408。gydF4y2Bahttps://doi.org/10.1101/gad.226837.113gydF4y2Ba.gydF4y2Ba
Nehra A, Ahlawat S, Singh KP。纳米孔生物传感研究综述。Sens Actuators B Chem 2019; 284:595-622。gydF4y2Bahttps://doi.org/10.1016/j.snb.2018.12.143gydF4y2Ba.gydF4y2Ba
Karawdeniya BI, Bandara YND, Nichols JW, Chevalier RB, Hagan JT, Dwyer JR.具有分析范围和环境的纳米孔挑战。肛门试验杂志2019;3:61-79。gydF4y2Bahttps://doi.org/10.1007/s41664-019-00092-1gydF4y2Ba.gydF4y2Ba
Kowalczyk SW, brosser TR, Dekker C.仿生纳米孔:向自然学习。生物科技趋势》。2011;29:607-14。gydF4y2Bahttps://doi.org/10.1016/j.tibtech.2011.07.006gydF4y2Ba.gydF4y2Ba
人工分子机器:膜结合的。化学学报,2016;45:6118-29。gydF4y2Bahttps://doi.org/10.1039/C5CS00874CgydF4y2Ba.gydF4y2Ba
Norris AL, Workman RE, Fan Y, Eshleman JR, Timp W.纳米孔测序检测癌症的结构变异。癌症生物学杂志。2016;17:46 - 53。gydF4y2Bahttps://doi.org/10.1080/15384047.2016.1139236gydF4y2Ba.gydF4y2Ba
Ko J, Bhagwat N, Black T, Yee SS, Na YJ, Fisher S, Kim J, Carpenter EL, Stanger BZ, Issadore d磁纳米孔分离细胞外囊泡的miRNA谱分析在胰腺癌诊断中的应用可以研究》2018年;78:3688 - 97。gydF4y2Bahttps://doi.org/10.1158/0008-5472.CAN-17-3703gydF4y2Ba.gydF4y2Ba
Euskirchen P, Bielle F, Labreche K, Kloosterman WP, Rosenberg S, Daniau M, Schmitt C, Masliah-Planchon J, Bourdeaut F, Dehais C, Marie Y.使用实时纳米孔测序对脑肿瘤的当日基因组和表观基因组诊断。Acta Neuropathol。2017;134:691 - 703。gydF4y2Bahttps://doi.org/10.1007/s00401-017-1743-5gydF4y2Ba.gydF4y2Ba
洪敏,陶思,张磊,刁丽彤,黄霞,黄松,谢世杰,肖志东,张华。RNA测序:新技术及其在癌症研究中的应用。中华血液病杂志2020;13:1-16。gydF4y2Bahttps://doi.org/10.1186/s13045-020-01005-xgydF4y2Ba.gydF4y2Ba
Jain M, Koren S, Miga KH, Quick J, Rand AC, Sasani TA, Tyson JR, Beggs AD, Dilthey AT, Fiddes IT, Malla S超长reads人类基因组的纳米孔测序和组装。生物科技Nat》。2018;36:338-45。gydF4y2Bahttps://doi.org/10.1038/nbt.4060gydF4y2Ba.gydF4y2Ba
Schweiger MR, Kerick M, Timmermann B, Isau M. NGS技术在描述癌症基因组组织中的力量:从突变到结构变化和表观遗传改变。癌症转移,2011;30:199-210。gydF4y2Bahttps://doi.org/10.1007/s10555-011-9278-zgydF4y2Ba.gydF4y2Ba
库CS, Roukos DH。从下一代测序到纳米孔测序技术:为个性化基因组医学铺平道路。专家Rev Med Devices 2013; 10:1-6。gydF4y2Bahttps://doi.org/10.1586/erd.12.63gydF4y2Ba.gydF4y2Ba
Brown CG, Clarke J.牛津纳米孔的发展。生物科技Nat》。2016;34:810-1。gydF4y2Bahttps://doi.org/10.1038/nbt.3622gydF4y2Ba.gydF4y2Ba
来自失神经蛙肌纤维膜的单通道电流记录。大自然。1976;260:799 - 802。gydF4y2Bahttps://doi.org/10.1038/nbt.3622gydF4y2Ba.gydF4y2Ba
纳米孔:DNA测序之旅。《物理生活》2012;9:125-58。gydF4y2Bahttps://doi.org/10.1016/j.plrev.2012.05.010gydF4y2Ba.gydF4y2Ba
Deamer DW, Nichols JW。模型和生物膜中的质子通量机制。J会员。1989;107:91 - 103。gydF4y2Bahttps://doi.org/10.1007/BF01871715gydF4y2Ba.gydF4y2Ba
纳米孔测序:从想象到现实。中国化学2015;61:25-31。gydF4y2Bahttps://doi.org/10.1373/clinchem.2014.223016gydF4y2Ba.gydF4y2Ba
布兰登·D,迪默尔DW。纳米孔测序:介绍。世界科学。2019年。gydF4y2Ba
马丽萍,陈晓明。生物纳米孔在单分子生物物理中的应用。ChemBioChem。2010; 11:25-34。gydF4y2Bahttps://doi.org/10.1002/cbic.200900526gydF4y2Ba.gydF4y2Ba
王晓燕,王晓燕,王晓燕。纳米孔技术在DNA测序中的应用。应用物理学报。2016;49:413001。gydF4y2Bahttps://doi.org/10.1088/0022-3727/49/41/413001gydF4y2Ba.gydF4y2Ba
Schneider GF, Dekker C.用纳米孔测序DNA。生物科技Nat》。2012;30:326-8。gydF4y2Bahttps://doi.org/10.1038/nbt.2181gydF4y2Ba.gydF4y2Ba
王晓峰,王晓峰。纳米孔在下一代测序中的应用。纳米技术。2015;26:074003。gydF4y2Bahttps://doi.org/10.1088/0957-4484/26/7/074003gydF4y2Ba.gydF4y2Ba
纳米孔读取长基因组DNA。Nat方法。2014;11:887 - 887。gydF4y2Bahttps://doi.org/10.1038/nmeth.3085gydF4y2Ba.gydF4y2Ba
Galdiero S, Gouaux E. α-溶血素-磷脂配合物的高分辨率晶体学研究定义了七聚体-脂质头基团相互作用:理解蛋白质-脂质相互作用的意义。蛋白质科学。2004;13:1503-11。gydF4y2Bahttps://doi.org/10.1110/ps.03561104gydF4y2Ba.gydF4y2Ba
何敏,迟旭,任杰。牛津纳米孔测序在裂糖酵母中的应用。:酵母协议;2021.p . 97 - 116。gydF4y2Bahttps://doi.org/10.1007/978-1-0716-0868-5_9gydF4y2Ba.gydF4y2Ba
Celaya G, Perales-Calvo J, Muga A, Moro F, rodrigrodriguez - larrea D.具有纳米孔的蛋白质- dna复合体的无标签、复用、单分子分析。ACS Nano。2017;11:5815-25。gydF4y2Bahttps://doi.org/10.1021/acsnano.7b01434gydF4y2Ba.gydF4y2Ba
Mikheyev AS, Tin MM.牛津纳米孔MinION测序仪的第一眼。中国生态学报,2014;14:1097-102。gydF4y2Bahttps://doi.org/10.1111/1755-0998.12324gydF4y2Ba.gydF4y2Ba
谢松,梁安生,郑铮,张东,肖超,罗锐,罗敏,张松。纳米孔测序在(epi)基因组和(epi)转录组时代的应用和潜力。2021年创新。。gydF4y2Bahttps://doi.org/10.1016/j.xinn.2021.100153gydF4y2Ba.gydF4y2Ba
RNA测序:平台选择、实验设计和数据解释。核酸科学。2012;22:271-4。gydF4y2Bahttps://doi.org/10.1089/nat.2012.0367gydF4y2Ba.gydF4y2Ba
赵磊,张宏,Kohnen MV, Prasad KV,顾磊,Reddy AS。利用PacBio Iso-Seq和基于纳米孔的直接RNA测序分析植物的转录组和转录组。麝猫。2019;10:253。gydF4y2Bahttps://doi.org/10.3389/fgene.2019.00253gydF4y2Ba.gydF4y2Ba
Garalde DR, Snell EA, Jachimowicz D, Sipos B, Lloyd JH, Bruce M, Pantic N, Admassu T, James P, Warland A, Jordan M.纳米孔阵列上的高度平行直接RNA测序。Nat方法。2018;15:201-6。gydF4y2Bahttps://doi.org/10.1038/nmeth.4577gydF4y2Ba.gydF4y2Ba
姜旭,马alibakhshi MA, Wanunu M.提高双分子膜稳定性的纳米孔传感应用。Biophys j . 2019; 116:148a。gydF4y2Bahttps://doi.org/10.1016/j.bpj.2018.11.821gydF4y2Ba.gydF4y2Ba
Leggett RM, Clark MD。纳米孔测序的机遇世界。J Exp Bot. 2017; 68:5419-29。gydF4y2Bahttps://doi.org/10.1093/jxb/erx289gydF4y2Ba.gydF4y2Ba
Logsdon GA, Vollger MR, Eichler EE。长读人类基因组测序及其应用。Nat Rev Genet。2020;21:597-614。gydF4y2Bahttps://doi.org/10.1038/s41576-020-0236-xgydF4y2Ba.gydF4y2Ba
Loit K, Adamson K, Bahram M, Puusepp R, Anslan S, Kiiker R, Drenkhan R, Tedersoo L. MinION(牛津纳米孔技术公司)与Sequel(太平洋生物科学公司)第三代测序仪器在农业和森林真菌病原体鉴定中的相对性能。应用环境微生物学报2019;85:e01368-e1419。gydF4y2Bahttps://doi.org/10.1128/AEM.01368-19gydF4y2Ba.gydF4y2Ba
检查海登E.纳米孔基因组测序仪首次亮相。自然新闻。2012。gydF4y2Bahttps://doi.org/10.1038/nature.2012.10051gydF4y2Ba.gydF4y2Ba
Andrews TS, Kiselev VY, McCarthy D, Hemberg M.教程:单细胞RNA测序数据的计算分析指南。Nat Protoc。2021;16:1-9。gydF4y2Bahttps://doi.org/10.1038/s41596-020-00409-wgydF4y2Ba.gydF4y2Ba
公园PJ。ChIP-seq:成熟技术的优势和挑战。Nat Rev Genet 2009; 10:669-80。gydF4y2Bahttps://doi.org/10.1038/nrg2641gydF4y2Ba.gydF4y2Ba
Rauluseviciute I, Drabløs F, Rye MB.测序DNA甲基化数据:数据分析工具和管道的实验方法和建议。Epigenet。2019;11:1-13。gydF4y2Bahttps://doi.org/10.1186/s13148-019-0795-xgydF4y2Ba.gydF4y2Ba
利用纳米孔器件进行DNA运动控制和序列读出的挑战。纳米技术。2015;26:074004。gydF4y2Bahttps://doi.org/10.1088/0957-4484/26/7/074004gydF4y2Ba.gydF4y2Ba
Branton D, Deamer DW, Marziali A, Bayley H, Benner SA, Butler T, Di Ventra M, Garaj S, Hibbs A, Huang X, Jovanovich SB.纳米孔测序的潜力和挑战。纳米科学技术收集Rev Nat . 2010。gydF4y2Bahttps://doi.org/10.1142/9789814287005_0027gydF4y2Ba.gydF4y2Ba
Vogel R, Pal AK, Jambhrunkar S, Patel P, Thakur SS, Reátegui E, Parekh HS, Saá P, Stassinopoulos A, Broom MF。利用可调电阻脉冲传感对生物纳米颗粒的高分辨率单粒子zeta电位表征。Sci众议员2017;7:1-13。gydF4y2Bahttps://doi.org/10.1038/s41598-017-14981-xgydF4y2Ba.gydF4y2Ba
生物纳米颗粒及其对生物体的影响。生物技术,2014;28:69-74。gydF4y2Bahttps://doi.org/10.1016/j.copbio.2013.11.014gydF4y2Ba.gydF4y2Ba
明显的WJ。生物系统中的纳米粒子。Angew Chem Int Ed. 2011; 50:1242-58。gydF4y2Bahttps://doi.org/10.1002/anie.200906684gydF4y2Ba.gydF4y2Ba
生物纳米孔传感和测序的膜平台。Curr Opin生物技术,2016;39:17-27。gydF4y2Bahttps://doi.org/10.1016/j.copbio.2015.12.015gydF4y2Ba.gydF4y2Ba
王红梅,王慧敏。无机纳米粒子的加入对溶胶-凝胶复合环氧体系粘结强度的影响。中华医学杂志。2010;30:505-12。gydF4y2Bahttps://doi.org/10.1016/j.ijadhadh.2010.05.002gydF4y2Ba.gydF4y2Ba
Cumbal L, Greenleaf J, Leun D, SenGupta AK。聚合物负载无机纳米颗粒:表征和环境应用。反应功能Polym, 2003; 54:17 7 - 80。gydF4y2Bahttps://doi.org/10.1016/s1381 - 5148 (02) 00192 - xgydF4y2Ba.gydF4y2Ba
李mh, Kumar A, Park KB, Cho SY, Kim HM, Lim MC, Kim YR, Kim KB。基于高绝缘基板的低噪声固态纳米孔平台。Sci众议员2014;4:1-7。gydF4y2Bahttps://doi.org/10.1038/srep07448gydF4y2Ba.gydF4y2Ba
郝峰,李娟,吴洪昌,梁新军,郭鹏。用于单化学物质实时传感和DNA测序的固态生物纳米孔。今天Nano。2013;56 - 74。gydF4y2Bahttps://doi.org/10.1016/j.nantod.2012.12.008gydF4y2Ba.gydF4y2Ba
王晓燕,王晓燕,王晓燕,等。DNA选择性固态纳米孔通道的研究。Nat Nanotechnol。2007;2:243-8。gydF4y2Bahttps://doi.org/10.1038/nnano.2007.78gydF4y2Ba.gydF4y2Ba
陈强,刘铮。固态纳米孔的制备与应用。传感器。2019;19:1886。gydF4y2Bahttps://doi.org/10.3390/s19081886gydF4y2Ba.gydF4y2Ba
Miles BN, Ivanov AP, Wilson KA, dodogan F, Japrung D, Edel JB。固体纳米孔单分子传感:新材料、方法和应用。化学学报,2013;42:15-28。gydF4y2Bahttps://doi.org/10.1039/C2CS35286AgydF4y2Ba.gydF4y2Ba
Di Fiori N, Squires A, Bar D, Gilboa T, Moustakas TD, Meller A.固体纳米孔表面电荷和易位动力学的光电控制。Nat Nanotechnol。2013;8:946-51。gydF4y2Bahttps://doi.org/10.1038/nnano.2013.221gydF4y2Ba.gydF4y2Ba
罗玲,李建军,李建军,李建军。固体纳米孔的几何形状和稳定性对检测DNA单分子的影响。纳米技术。2015;26:044001。gydF4y2Bahttps://doi.org/10.1088/0957-4484/26/4/044001gydF4y2Ba.gydF4y2Ba
拉金J, Henley R, Bell DC, Cohen-Karni T, Rosenstein JK, Wanunu M. DNA在氧化铪膜纳米孔中的缓慢转运。ACS Nano。2013;7:10121-8。gydF4y2Bahttps://doi.org/10.1021/nn404326fgydF4y2Ba.gydF4y2Ba
李娟,于东,赵强。基于固态纳米孔的DNA单分子检测与测序。Microchim学报。2016;183:941-53。gydF4y2Bahttps://doi.org/10.1007/s00604-015-1542-4gydF4y2Ba.gydF4y2Ba
陈伟,刘春春,欧阳娟,高美明,刘斌,赵永元。石墨烯纳米孔用于DNA测序的实验研究进展。中国化学科学2017;60:721-9。gydF4y2Bahttps://doi.org/10.1007/s11426-016-9016-5gydF4y2Ba.gydF4y2Ba
库德J, Skalickova S, Nejdl L, Moulick A, ruttky - nedecky B, Adam V, Kizek R.固态纳米孔的制备及其展望。电泳。2015;36:2367 - 79。gydF4y2Bahttps://doi.org/10.1002/elps.201400612gydF4y2Ba.gydF4y2Ba
刘坤,冯杰,张晓燕,张晓燕。DNA易位的原子薄二硫化钼纳米孔。ACS Nano。2014;8:2504-11。gydF4y2Bahttps://doi.org/10.1021/nn406102hgydF4y2Ba.gydF4y2Ba
熊明,格拉芙,阿特瑞亚,拉德诺维奇,莱伯顿。MoS2膜纳米孔中单分子的显微检测分析。ACS Nano。2020;11(14):16131 - 9。gydF4y2Bahttps://doi.org/10.1021/acsnano.0c08382gydF4y2Ba.gydF4y2Ba
Boutilier MS, Jang D, Idrobo JC, Kidambi PR, Hadjiconstantinou NG, Karnik R.厘米级单层纳米多孔石墨烯膜的分子筛分。ACS Nano。2017;11:5726-36。gydF4y2Bahttps://doi.org/10.1021/acsnano.7b01231gydF4y2Ba.gydF4y2Ba
刘元Z, Y,戴M,彝族X,王c控制DNA通过固态纳米孔易位。纳米尺度研究,2020;15:1-9。gydF4y2Bahttps://doi.org/10.1186/s11671-020-03308-xgydF4y2Ba.gydF4y2Ba
Thakur M, Macha M, Chernev A, Graf M, Lihter M, Deen J, Tripathi M, Kis A, Radenovic A.利用MoS2制备单分子DNA生物传感纳米孔器件。小方法。2020;4:2000072。gydF4y2Bahttps://doi.org/10.1002/smtd.202000072gydF4y2Ba.gydF4y2Ba
Schneider GF, Kowalczyk SW, Calado VE, Pandraud G, Zandbergen HW, Vandersypen LM, Dekker C. DNA在石墨烯纳米孔中的易位。纳米列托人。2010;10:3163-7。gydF4y2Bahttps://doi.org/10.1021/nl102069zgydF4y2Ba.gydF4y2Ba
Kowalczyk SW, Hall AR, Dekker C.使用固态纳米孔沿DNA检测局部蛋白质结构。纳米列托人。2010;10:324-8。gydF4y2Bahttps://doi.org/10.1021/nl903631mgydF4y2Ba.gydF4y2Ba
Wasfi A, Awwad F, Ayesh AI。基于石墨烯的纳米孔DNA测序方法:文献综述。Biosens Bioelectron。2018;119:191 - 203。gydF4y2Bahttps://doi.org/10.1016/j.bios.2018.07.072gydF4y2Ba.gydF4y2Ba
曹军,贾伟,张军,徐旭,闫珊,王赟,张鹏,陈海燕,黄松。四氯酸盐(III)嵌入垢垢分枝杆菌孔蛋白a纳米孔中观察到的巨大单分子化学事件。Nat Commun。2019;10:1-11。gydF4y2Bahttps://doi.org/10.1038/s41467-019-13677-2gydF4y2Ba.gydF4y2Ba
Manrao EA, Derrington IM, Pavlenok M, Niederweis M, Gundlach JH。核苷酸鉴别与DNA固定在MspA纳米孔。PLoS ONE。2011; 6:25723。gydF4y2Bahttps://doi.org/10.1371/journal.pone.0025723gydF4y2Ba.gydF4y2Ba
曹斌,赵燕,寇燕,倪丹,张晓春,黄燕。非美洲细菌淀粉样蛋白分泌通道的结构。中国生物医学工程学报(英文版)2014;gydF4y2Bahttps://doi.org/10.1073/pnas.1411942111gydF4y2Ba.gydF4y2Ba
Carter JM, Hussain S.使用ONT CsgG纳米孔系统进行稳健的长读取原生DNA测序。Wellcome Open Res. 2017。gydF4y2Bahttps://doi.org/10.12688/wellcomeopenres.11246.1gydF4y2Ba.gydF4y2Ba
戈亚尔P,克拉斯特娃PV,范·热尔文N,古贝利尼F,范登·布罗克I, Troupiotis-Tsaïlaki A, Jonckheere W, Péhau-Arnaudet G,平克纳JS,查普曼MR, Hultgren SJ。细菌淀粉样蛋白分泌通道CsgG的结构和机制研究。大自然。2014;516:250-3。gydF4y2Bahttps://doi.org/10.1038/nature13768gydF4y2Ba.gydF4y2Ba
Stoddart D, Maglia G, Mikhailova E, Heron AJ, Bayley H.生物纳米孔中的多个碱基识别位点:两个头比一个头好。Angew化学2010;122:566-9。gydF4y2Bahttps://doi.org/10.1002/ange.200905483gydF4y2Ba.gydF4y2Ba
Van der Verren SE, Van Gerven N, Jonckheere W, Hambley R, Singh P, Kilgour J, Jordan M, Wallace EJ, Jayasinghe L, Remaut h。生物科技Nat》。2020;38:1415-20。gydF4y2Bahttps://doi.org/10.1038/s41587-020-0570-8gydF4y2Ba.gydF4y2Ba
王晓明,王晓明,王晓明。生物纳米孔的研究进展。工程需求。生活。2021;十一27。gydF4y2Bahttps://doi.org/10.3390/life11010027gydF4y2Ba.gydF4y2Ba
应YL,曹c,龙YT。生物纳米孔传感器的单分子分析。分析师。2014;139:3826-35。gydF4y2Bahttps://doi.org/10.1039/C4AN00706AgydF4y2Ba.gydF4y2Ba
柳H,赵h,吴SS.检测和超越:基于适配体的生物传感器的挑战和进展。板牙放置2020;1:2663 - 87。gydF4y2Bahttps://doi.org/10.1039/D0MA00639DgydF4y2Ba.gydF4y2Ba
建造膜纳米孔。Nat Nanotechnol。2017;12:619-30。gydF4y2Bahttps://doi.org/10.1038/nnano.2017.99gydF4y2Ba.gydF4y2Ba
将DNA穿过纳米孔用于生物传感应用。中国物理学报2015;27:273101。gydF4y2Bahttps://doi.org/10.1088/0953-8984/27/27/273101gydF4y2Ba.gydF4y2Ba
Lu B, Albertorio F, Hoogerheide DP, Golovchenko JA。DNA通过纳米孔时速度波动的起源和后果。Biophys j . 2011; 101:70-9。gydF4y2Bahttps://doi.org/10.1016/j.bpj.2011.05.034gydF4y2Ba.gydF4y2Ba
Tan CS。生物纳米孔检测DNA碱基修饰。盐湖城:犹他大学;2018.gydF4y2Ba
Kulkarni SK, Kulkarni SK.纳米技术:原理和实践。可汗:施普林格;2015.gydF4y2Bahttps://doi.org/10.1007/978-3-319-09171-6gydF4y2Ba.gydF4y2Ba
基于纳米孔的单分子DNA分析。未来医学》2007。gydF4y2Bahttps://doi.org/10.2217/17435889.2.4.459gydF4y2Ba.gydF4y2Ba
生物分子在固态纳米孔中的易位:理论与实验的结合。高分子学报,2011;49:985-1011。gydF4y2Bahttps://doi.org/10.1002/polb.22284gydF4y2Ba.gydF4y2Ba
王晓燕,王晓燕,王晓燕。固态纳米孔中DNA易位的研究进展。纳米级。2012;4:1068 - 77。gydF4y2Bahttps://doi.org/10.1039/C1NR11201EgydF4y2Ba.gydF4y2Ba
王静,杨静,应丽丽,龙依东。用于单分子分析的纳米孔受限空间。化学通报2019;14:389-97。gydF4y2Bahttps://doi.org/10.1002/asia.201801648gydF4y2Ba.gydF4y2Ba
Reiner JE, Balijepalli A, Robertson JW,溺水BS, Burden DL, Kasianowicz JJ。扩散对外切酶/纳米孔DNA测序引擎的影响。化学物理学报,2012;137:214903。gydF4y2Bahttps://doi.org/10.1063/1.4766363gydF4y2Ba.gydF4y2Ba
dicingham MS, Kowalczykowski SC. RecBCD酶与双链DNA断裂的修复。微生物Mol Biol Rev. 2008; 72:642-71。gydF4y2Bahttps://doi.org/10.1128/MMBR.00020-08gydF4y2Ba.gydF4y2Ba
Gyarfas B, Olasagasti F, Benner S, Garalde D, Lieberman KR, Akeson M.在5 Å分辨率下绘制DNA聚合酶结合DNA模板在纳米孔中的位置。ACS Nano。2009;3:1457 - 66。gydF4y2Bahttps://doi.org/10.1021/nn900303ggydF4y2Ba.gydF4y2Ba
Cherf GM, Lieberman KR, Rashid H, Lam CE, Karplus K, Akeson M.纳米孔中DNA在5-Å精度的自动正向和反向棘轮。生物科技Nat》。2012;30:344-8。gydF4y2Bahttps://doi.org/10.1038/nbt.2147gydF4y2Ba.gydF4y2Ba
李伯曼KR, Cherf GM, Doody MJ, Olasagasti F, Kolodji Y, Akeson M. phi29 DNA聚合酶催化单DNA分子在纳米孔中的过程复制。化学学报2010;132:17961-72。gydF4y2Bahttps://doi.org/10.1021/ja1087612gydF4y2Ba.gydF4y2Ba
Cho MW, Richards OC, Dmitrieva TM, Agol V, Ehrenfeld E.脊髓灰质炎病毒RNA依赖RNA聚合酶3Dpol的RNA双链展开活性。J微生物学报。1993;67:3010-8。gydF4y2Bahttps://doi.org/10.1128/jvi.67.6.3010-3018.1993gydF4y2Ba.gydF4y2Ba
考德威尔,密探m,解旋酶正在穿过纳米孔。中国生物医学工程学报2017;114:11809-11。gydF4y2Bahttps://doi.org/10.1073/pnas.1716866114gydF4y2Ba.gydF4y2Ba
Craig JM, Laszlo AH, Nova IC, Brinkerhoff H, Noakes MT, Baker KS, Bowman JL, Higinbotham HR, Mount JW, Gundlach JH。用纳米孔镊子测定DNA序列对Hel308解旋酶沿单链DNA易位的影响。核酸决议2019;47:2506-13。gydF4y2Bahttps://doi.org/10.1093/nar/gkz004gydF4y2Ba.gydF4y2Ba
后藤Y,柳木I,松井K,横井T,武田KI。集成固态纳米孔平台,通过介电击穿、dna速度减速和噪声降低制备纳米孔。Sci众议员2016;6:1-8。gydF4y2Bahttps://doi.org/10.1038/srep31324gydF4y2Ba.gydF4y2Ba
陈晓燕,陈晓燕,陈晓燕。合成纳米孔中DNA易位的微观动力学研究。Biophys j . 2004; 87:2086 - 97。gydF4y2Bahttps://doi.org/10.1529/biophysj.104.042960gydF4y2Ba.gydF4y2Ba
Avdoshenko SM, Nozaki D, Gomes da Rocha C, González JW, Lee MH, Gutierrez R, Cuniberti G. DNA在石墨烯纳米孔中易位的动力学和电子输运特性。纳米列托人。2013;13:1969 - 76。gydF4y2Bahttps://doi.org/10.1529/biophysj.104.042960gydF4y2Ba.gydF4y2Ba
Kawano R, Schibel AE, Cauley C, White HS。利用黏度控制单链DNA通过α-溶血素离子通道的易位。朗缪尔。2009;25:1233-7。gydF4y2Bahttps://doi.org/10.1021/la803556pgydF4y2Ba.gydF4y2Ba
Ali M, Yameen B, Cervera J, Ramirez P, Neumann R, Ensinger W, Knoll W, Azzaroni O.聚电解质逐层组装成离子电流整流固态纳米孔:理论和实验的见解。中国化学学报2010;132:8338-48。gydF4y2Bahttps://doi.org/10.1021/ja101014ygydF4y2Ba.gydF4y2Ba
Akahori R, Haga T, Hatano T, Yanagi I, Ohura T, Hamamura H, Iwasaki T, Yokoi T, Anazawa T通过减小纳米孔直径来减缓单链DNA在固态纳米孔中的易位。纳米技术。2014;25:2。gydF4y2Bahttps://doi.org/10.1088/0957-4484/25/27/275501gydF4y2Ba.gydF4y2Ba
涂斌,白松,吕斌,方强。在纳米孔DNA测序中,圆锥形状比圆柱形形状灵敏度更高。Sci众议员2018;8:1-11。gydF4y2Bahttps://doi.org/10.1038/s41598-018-27517-8gydF4y2Ba.gydF4y2Ba
后藤Y,赤堀R,柳木I,武田KI。固态纳米孔用于单分子DNA测序。J Hum Genet, 2020; 65:69-77。gydF4y2Bahttps://doi.org/10.1038/s10038-019-0655-8gydF4y2Ba.gydF4y2Ba
后藤Y,松井K,柳木I,武田KI。二价阳离子的介电击穿所产生的氮化硅纳米孔:易位速度的减慢和单核苷酸的识别。纳米级。2019;11:14426-33。gydF4y2Bahttps://doi.org/10.1039/C9NR03563JgydF4y2Ba.gydF4y2Ba
Kowalczyk SW, Wells DB, Aksimentiev A, Dekker C.通过氯化锂纳米孔减缓DNA易位。纳米列托人。2012;12:1038-44。gydF4y2Bahttps://doi.org/10.1021/nl204273hgydF4y2Ba.gydF4y2Ba
闫辉,周东,石斌,张铮,田辉,于磊,王赟,关旭,王铮,王东。利用LiCl盐梯度和纳米纤维网减缓DNA易位速度。中国生物学报,2019;gydF4y2Bahttps://doi.org/10.1007/s00249-019-01350-xgydF4y2Ba.gydF4y2Ba
Vu T, Borgesi J, Soyring J, D 'Alia M, Davidson SL, Shim J.在野生型α-溶血素纳米孔中使用LiCl盐梯度减缓DNA易位和检测甲基化胞嘧啶。纳米级。2019;11:10536-45。gydF4y2Bahttps://doi.org/10.1039/C9NR00502AgydF4y2Ba.gydF4y2Ba
李建军,李建民,李建民,李建民,李建民。氮化硅纳米孔中DNA易位的研究。电泳。2012;33:3448-57。gydF4y2Bahttps://doi.org/10.1002/elps.201200165gydF4y2Ba.gydF4y2Ba
王晓燕,王晓燕。固体纳米孔与生物纳米孔电流噪声的比较。ACS Nano。2020;14:1338-49。gydF4y2Bahttps://doi.org/10.1021/acsnano.9b09353gydF4y2Ba.gydF4y2Ba
梁松,向峰,唐铮,努里瑞,何旭,董明,关伟。纳米孔传感器噪声的来源、模型、降噪与标杆。纳米技术精密工程。2020;3:9-17。gydF4y2Bahttps://doi.org/10.1016/j.npe.2019.12.008gydF4y2Ba.gydF4y2Ba
王晓峰,王晓明。固体纳米孔中的光学传感与分析物操作。分析师。2015;140:4733-47。gydF4y2Bahttps://doi.org/10.1039/C4AN02388AgydF4y2Ba.gydF4y2Ba
王晓燕,王晓燕,王晓燕。纳米孔和离子通道记录的高带宽方法研究进展。化学学报2019;1061:13-27。gydF4y2Bahttps://doi.org/10.1016/j.aca.2019.01.034gydF4y2Ba.gydF4y2Ba
李凯,朴浩浩,金浩杰,余杰,蔡浩,金浩浩,金浩浩。固体纳米孔的研究进展。难以板牙。2018;30:1704680。gydF4y2Bahttps://doi.org/10.1002/adma.201704680gydF4y2Ba.gydF4y2Ba
Rodríguez-Manzo JA, Puster M, Nicolaï A, Meunier V, Drndic M. DNA在纳米厚硅纳米孔中的易位。ACS Nano。2015;9:6555 - 64。gydF4y2Bahttps://doi.org/10.1021/acsnano.5b02531gydF4y2Ba.gydF4y2Ba
张志强,张志强。离子和分子传输的二维纳米孔和纳米多孔膜。Curr Opin生物技术2019;55:124-33。gydF4y2Bahttps://doi.org/10.1016/j.copbio.2018.09.002gydF4y2Ba.gydF4y2Ba
郭h,郭海峰,郭丁。高电场对固态纳米孔尺寸和噪声的精确控制。纳米技术。2012;23:405301。gydF4y2Bahttps://doi.org/10.1088/0957-4484/23/40/405301gydF4y2Ba.gydF4y2Ba
王晓燕,王晓燕,王晓燕。从波形到碱基对:提高纳米孔测序读取精度的计算方法。基因组医学杂志。2018;19:1-11。gydF4y2Bahttps://doi.org/10.1186/s13059-018-1462-9gydF4y2Ba.gydF4y2Ba
在DNA计算中寡核苷酸的纳米孔解码。Biotechnol j . 2018; 13:1800091。gydF4y2Bahttps://doi.org/10.1002/biot.201800091gydF4y2Ba.gydF4y2Ba
Senol Cali D, Kim JS, Ghose S, Alkan C, Mutlu O.基因组组装的纳米孔测序技术和工具:现状、瓶颈和未来方向的计算分析。短暂的Bioinform。2019;20:1542-59。gydF4y2Bahttps://doi.org/10.1093/bib/bby017gydF4y2Ba.gydF4y2Ba
Manrao EA, Derrington IM, Laszlo AH, Langford KW, Hopper MK, Gillgren N, Pavlenok M, Niederweis M, Gundlach JH。用突变的MspA纳米孔和phi29 DNA聚合酶以单核苷酸分辨率读取DNA。生物科技Nat》。2012;30:349-53。gydF4y2Bahttps://doi.org/10.1038/nbt.2171gydF4y2Ba.gydF4y2Ba
Deamer D, Akeson M, Branton D.三十年的纳米孔测序。生物科技Nat》。2016;34:518-24。gydF4y2Bahttps://doi.org/10.1038/nbt.3423gydF4y2Ba.gydF4y2Ba
Prall TM, Neumann EK, Karl JA, Shortreed CG, Baker DA, Bussan HE, Wiseman RW, O 'Connor DH。一致的超长DNA测序与自动慢移液。BMC染色体组。2021;22:1-12。gydF4y2Bahttps://doi.org/10.1186/s12864-021-07500-wgydF4y2Ba.gydF4y2Ba
McCoy RC, Taylor RW, Blauwkamp TA, Kelley JL, Kertesz M, Pushkarev D, Petrov DA, Fiston-Lavier AS。Illumina TruSeq合成长读支持从头组装,解决复杂的高重复性转座子。PLoS ONE。2014; 9: e106689。gydF4y2Bahttps://doi.org/10.1371/journal.pone.0106689gydF4y2Ba.gydF4y2Ba
Mason CE, Afshinnekoo E, Tighe S, Wu S, Levy S基因组、转录组和宏基因组的国际标准。J生物科技JBT。2017; 28:8。gydF4y2Bahttps://doi.org/10.7171/jbt.17-2801-006gydF4y2Ba.gydF4y2Ba
龚琳、王青、偶像J、颜兆贤、魏丽麟。用于全基因组DNA分析的超长读取测序。JoVE J Vis Exp 2019;145:58954。gydF4y2Bahttps://doi.org/10.3791/58954gydF4y2Ba.gydF4y2Ba
韦拉瑟JL、德·切萨雷·M、王煜、皮亚扎·P、Sebastiano V、王旭杰、Buck D、Au KF。太平洋生物科学和牛津纳米孔技术的综合比较及其在转录组分析中的应用。F1000Research。2017.gydF4y2Bahttps://doi.org/10.12688/f1000research.10571.2gydF4y2Ba.gydF4y2Ba
Nurk S, Koren S, Rhie A, Rautiainen M, Bzikadze AV, Mikheenko A, Vollger MR, Altemose N, Uralsky L, Gershman A, Aganezov S.人类基因组的完整序列。bioRxiv。2021.gydF4y2Bahttps://doi.org/10.1101/2021.05.26.445798gydF4y2Ba.gydF4y2Ba
Levy-Sakin M, Pastor S, Mostovoy Y, Li L, Leung AK, McCaffrey J, Young E, Lam ET, Hastie AR, Wong KH, Chung CY. 26个人类群体的基因组图谱揭示了群体特异性的结构变异模式。Nat Commun。2019;10:1-14。gydF4y2Bahttps://doi.org/10.1038/s41467-019-08992-7gydF4y2Ba.gydF4y2Ba
Cooper DN, Bacolla A, Férec C, Vasquez KM, kehher - sawatzki H,陈建明。人类基因突变的序列导向性质:基因组结构和局部DNA序列环境在介导人类遗传疾病的基因突变中的作用。哼Mutat。2011;32:1075 - 99。gydF4y2Bahttps://doi.org/10.1002/humu.21557gydF4y2Ba.gydF4y2Ba
Magi A, Semeraro R, Mingrino A, Giusti B, D’aurizio R.纳米孔测序数据分析:技术现状,应用和挑战。短暂的Bioinform。2018;19:1256 - 72。gydF4y2Bahttps://doi.org/10.1093/bib/bbx062gydF4y2Ba.gydF4y2Ba
Silvestre-Ryan J, Holmes I.对共识解码提高神经网络基序调用器在纳米孔测序中的准确性。基因组医学杂志。2021;22:1-6。gydF4y2Bahttps://doi.org/10.1186/s13059-020-02255-1gydF4y2Ba.gydF4y2Ba
罗森斯坦JK,李梅SG,谢泼德KL.单分子生物电子学。纳米生物技术。2015;7:47 - 93。gydF4y2Bahttps://doi.org/10.1002/wnan.1323gydF4y2Ba.gydF4y2Ba
薛琳,山崎H,任瑞,瓦努努M,伊万诺夫AP, Edel JB。固态纳米孔传感器。2020; 5:931-51。gydF4y2Bahttps://doi.org/10.1038/s41578-020-0229-6gydF4y2Ba.gydF4y2Ba
马涛,杨建军,陈晓燕,等。轨道蚀刻纳米孔/膜的基本原理与应用。小方法。2020;4:2000366。gydF4y2Bahttps://doi.org/10.1002/smtd.202000366gydF4y2Ba.gydF4y2Ba
巴尔梅,李波伊特文,贝切拉尼,贾诺特。混合生物/人工纳米孔。纳米结构的物理、化学和应用:纳米国际会议论文集;2015.454 - 6页。gydF4y2Bahttps://doi.org/10.1142/9789814696524_0112gydF4y2Ba.gydF4y2Ba
贝切拉尼,巴尔梅,米埃勒,等。能源、环境和健康应用中生物基纳米结构界面的原子层沉积。纯应用化学2015;87:751-8。gydF4y2Bahttps://doi.org/10.1515/pac-2015-0102gydF4y2Ba.gydF4y2Ba
李波特文,马婷,王晓燕,王晓燕,王晓燕。单固态纳米孔功能化模拟生物离子通道的研究进展。Adv Coll Interface science 2017; 250:195-213。gydF4y2Bahttps://doi.org/10.1016/j.cis.2017.09.001gydF4y2Ba.gydF4y2Ba
Plesivkova D, Richards R, Harbison S.综述了MinION™单分子测序系统在法医应用中的潜力。威利跨学科司法科学,2019;1:e1323。gydF4y2Bahttps://doi.org/10.1002/wfs2.1323gydF4y2Ba.gydF4y2Ba
纳米孔测序技术在功能基因组学中的潜在应用。开发增长差异2019;61:316-26。gydF4y2Bahttps://doi.org/10.1111/dgd.12608gydF4y2Ba.gydF4y2Ba
Chaisson MJ, Tesler G.使用基本局部序列连续细化(BLASR)绘制单分子测序:应用和理论。BMC Bioinform。2012;13:1-18。gydF4y2Bahttps://doi.org/10.1186/1471-2105-13-238gydF4y2Ba.gydF4y2Ba
狂欢节。DNA测序技术:2006-2016年。Nat Protoc。2017;12:213-8。gydF4y2Bahttps://doi.org/10.1038/nprot.2016.182gydF4y2Ba.gydF4y2Ba
Petersen LM, Martin IW, Moschetti WE, Kershaw CM, Tsongalis GJ。第三代测序在临床实验室:探索纳米孔测序的优势和挑战。中国临床微生物学杂志2019;58:e01315-e1319。gydF4y2Bahttps://doi.org/10.1128/JCM.01315-19gydF4y2Ba.gydF4y2Ba
纳米孔测序技术和生物互联网:u -医疗的一大希望。适用于:用于运行状况监视的传感器。学术出版社;2019.p . 95 - 116。gydF4y2Bahttps://doi.org/10.1016/B978-0-12-819361-7.00005-1gydF4y2Ba.gydF4y2Ba
徐磊,Seki M.纳米孔测序仪检测碱基修饰的研究进展。J Hum Genet。2020;65:25-33。gydF4y2Bahttps://doi.org/10.1038/s10038-019-0679-0gydF4y2Ba.gydF4y2Ba
引用本文:Sedlazeck FJ, Rescheneder P, Smolka M, Fang H, Nattestad M, Von Haeseler A, Schatz MC.单分子测序技术用于复杂结构变异的精确检测。Nat方法。2018;15:461-8。gydF4y2Bahttps://doi.org/10.1038/s41592-018-0001-7gydF4y2Ba.gydF4y2Ba
Thayer A. Illumina起诉牛津Nano。化学工程学报。2016;94(9):16-16。gydF4y2Ba
牛津纳米孔最好的PacBio。生物科技Nat》。2019;37:333-9。gydF4y2Ba
萨顿JM,米尔伍德JD,菲斯特JL。与牛津纳米孔技术公司进行基因组测序和组装的优化实验设计。bioRxiv, p . 2020 - 05。2021.gydF4y2Bahttps://doi.org/10.46471/gigabyte.27gydF4y2Ba.gydF4y2Ba
Voelkerding KV, Dames SA, Durtschi JD。下一代测序:从基础研究到诊断。中国化学2009;55:641-58。gydF4y2Bahttps://doi.org/10.1373/clinchem.2008.112789gydF4y2Ba.gydF4y2Ba
Lopez R, Chen YJ, Ang SD, Yekhanin S, Makarychev K, Racz MZ, Seelig G, Strauss K, Ceze L.用于纳米孔数据存储读取的DNA组装。Nat Commun。2019;10:1-9。gydF4y2Bahttps://doi.org/10.1038/s41467-019-10978-4gydF4y2Ba.gydF4y2Ba
陈晓明,陈晓明,陈晓明,等。纳米孔测序及其在微生物群落研究中的应用。计算结构生物技术J. 2021。gydF4y2Bahttps://doi.org/10.1016/j.csbj.2021.02.020gydF4y2Ba.gydF4y2Ba
Lebrigand K, Magnone V, Barbry P, Waldmann R.高通量错校正纳米孔单细胞转录组测序。Nat Commun。2020;11:1-8。gydF4y2Bahttps://doi.org/10.1038/s41467-020-17800-6gydF4y2Ba.gydF4y2Ba
李锐,谢敏,董宁,林丹,杨旭,黄明海,陈ewc,陈松。快速组装MinION条形码测序数据高效生成mdr编码质粒完整序列。Gigascience。2018;7:132。gydF4y2Bahttps://doi.org/10.1093/gigascience/gix132gydF4y2Ba.gydF4y2Ba
Di Costanzo A, Del Gaudio N, Migliaccio A, Altucci L.抗癌症表观遗传药物:一个不断发展的景观。拱Toxicol。2014;88:1651 - 68。gydF4y2Bahttps://doi.org/10.1007/s00204-014-1315-6gydF4y2Ba.gydF4y2Ba
Cossío FP, Esteller M, Berdasco M.朝着癌症更精确的治疗:探索表观遗传复杂性。Curr Opin Chem Biol. 2020; 57:41-9。gydF4y2Bahttps://doi.org/10.1016/j.cbpa.2020.04.008gydF4y2Ba.gydF4y2Ba
Neganova ME, Klochkov SG, Aleksandrova YR, Aliev G.组蛋白修饰在癌症表观遗传调控中的研究进展。在:癌症生物学研讨会。学术出版社;2020.gydF4y2Bahttps://doi.org/10.1016/j.semcancer.2020.07.015gydF4y2Ba.gydF4y2Ba
由表观遗传基因突变引起的遗传综合征。哼麝猫。2013;132:359 - 83。gydF4y2Bahttps://doi.org/10.1007/s00439-013-1271-xgydF4y2Ba.gydF4y2Ba
引用本文赵亮,段东涛,陆平,张志军,郑晓坤,王建林,冯文伟。癌症治疗中的表观遗传靶点及其抑制剂。《当代医学化学》2018;18:2395-419。gydF4y2Bahttps://doi.org/10.2174/1568026619666181224095449gydF4y2Ba.gydF4y2Ba
陈晓燕,陈晓燕。表观遗传多药理学:表观药物发现的新前沿。2020; 40:190-244。gydF4y2Bahttps://doi.org/10.1002/med.21600gydF4y2Ba.gydF4y2Ba
Kumar S, Chinnusamy V, Mohapatra T.修饰DNA碱基的表观遗传学:5-甲基胞嘧啶及以上。麝猫。2018;9:640。gydF4y2Bahttps://doi.org/10.3389/fgene.2018.00640gydF4y2Ba.gydF4y2Ba
于敏、韩志强、Szulwach KE、宋春霞、张磊、金阿、李欣、戴强、沈燕、朴波、闵建华。哺乳动物基因组中5-羟甲基胞嘧啶的基分辨率分析。细胞。2012;149:1368 - 80。gydF4y2Bahttps://doi.org/10.1016/j.cell.2012.04.027gydF4y2Ba.gydF4y2Ba
王晓燕,王晓燕。DNA甲基化及其基本功能。神经精神药理学。2013;38:23-38。gydF4y2Bahttps://doi.org/10.1038/npp.2012.112gydF4y2Ba.gydF4y2Ba
Liyanage VR, Jarmasz JS, Murugeshan N, Del Bigio MR, Rastegar M, Davie JR. DNA修饰在正常和疾病状态下的功能及其应用。生物学。2014;3:670 - 723。gydF4y2Bahttps://doi.org/10.3390/biology3040670gydF4y2Ba.gydF4y2Ba
癌症中的DNA甲基化和基因沉默。2005;2: S4-11。gydF4y2Bahttps://doi.org/10.1038/ncponc0354gydF4y2Ba.gydF4y2Ba
外甥KP,黄天明。表观基因沉默在癌症起始和进展中的作用。癌症。2003;190:125-33。gydF4y2Bahttps://doi.org/10.1016/s0304 - 3835 (02) 00511 - 6gydF4y2Ba.gydF4y2Ba
Raynal NJM, Si J, Taby RF, Gharibyan V, Ahmed S, Jelinek J, Estécio MR, Issa JPJ。DNA甲基化并不稳定地锁定基因表达,而是作为基因沉默记忆的分子标记。可以研究》2012年;72:1170 - 81。gydF4y2Bahttps://doi.org/10.1158/0008-5472.CAN-11-3248gydF4y2Ba.gydF4y2Ba
杨文华,陈文华。DNA甲基化与组蛋白修饰的关系:模式与范式。Nat Rev Genet 2009; 10:295-304。gydF4y2Bahttps://doi.org/10.1038/nrg2540gydF4y2Ba.gydF4y2Ba
赵凯。细胞异质性的表观遗传基础。Nat Rev Genet 2021; 22:235-50。gydF4y2Bahttps://doi.org/10.1038/s41576-020-00300-0gydF4y2Ba.gydF4y2Ba
Feldman N, Gerson A, Fang J, Li E, Zhang Y, Shinkai Y, Cedar H, Bergman Y. g9a介导的10 -3/4在早期胚胎发生中的不可逆表观失活。细胞生物学杂志2006;8:188-94。gydF4y2Bahttps://doi.org/10.1038/ncb1353gydF4y2Ba.gydF4y2Ba
Epsztejn-Litman S, Feldman N, Abu-Remaileh M, Shufaro Y, Gerson A, Ueda J, Deplus R, Fuks F, Shinkai Y, Cedar H, Bergman Y. G9a促进的DNA甲基化抑制了胚胎沉默基因的重编程。生物学杂志。2008;15:1176-83。gydF4y2Bahttps://doi.org/10.1038/nsmb.1476gydF4y2Ba.gydF4y2Ba
甲基化和羟甲基化DNA生物标志物的发现和验证策略。癌症医学。2012;1:237-60。gydF4y2Bahttps://doi.org/10.1002/cam4.22gydF4y2Ba.gydF4y2Ba
Richa R, Sinha RP。DNA羟甲基化:表观遗传标记。EXCLI j . 2014; 13:592。gydF4y2Ba
边凯,Lenz SA,唐青,陈芳,祁瑞,Jost M, Drennan CL, Essigmann JM, Wetmore SD,李东。DNA修复酶ALKBH2、ALKBH3和AlkB在体外氧化5-甲基胞嘧啶为5-羟甲基胞嘧啶、5-甲酰胞嘧啶和5-羧基胞嘧啶。核酸决议2019;47:5522-9。gydF4y2Bahttps://doi.org/10.1093/nar/gkz395gydF4y2Ba.gydF4y2Ba
胡红,舒敏,何亮,余旭,刘旭,陆莹,陈莹,苗曦,陈旭。5-羟甲基胞嘧啶的表观基因组图谱揭示了其在结直肠癌中lncrna的转录调控作用。中华癌症杂志2017;116:658-68。gydF4y2Bahttps://doi.org/10.1038/bjc.2016.457gydF4y2Ba.gydF4y2Ba
Aristizabal MJ, Anreiter I, Halldorsdottir T, Odgers CL, McDade TW, Goldenberg A, Mostafavi S, Kobor MS, Binder EB, Sokolowski MB, O 'Donnell KJ。经验的生物嵌入:表观遗传学入门。中国生物医学工程学报2020;117:23261-9。gydF4y2Bahttps://doi.org/10.1073/pnas.1820838116gydF4y2Ba.gydF4y2Ba
DNA甲基化和组蛋白去乙酰化对基因表达的控制:人类发育和疾病的基础生物化学。基因表达与肝脏杂志2001;9:63-75。gydF4y2Bahttps://doi.org/10.3727/000000001783992731gydF4y2Ba.gydF4y2Ba
Majumder S, Kutay H, Datta J, Summers D, Jacob ST, Ghoshal K.金属硫蛋白i基因表达的表观遗传调控:DNA甲基转移酶和甲基CpG结合蛋白对甲基化和非甲基化启动子的差异调控。细胞生物化学杂志,2006;97:1300-16。gydF4y2Bahttps://doi.org/10.1002/jcb.20738gydF4y2Ba.gydF4y2Ba
Saito Y, Kanai Y,坂本M, Saito H, Ishii H, Hirohashi s人肝癌发生过程中CpG岛和周围熵点卫星区DNA甲基转移酶和甲基-CpG结合蛋白mRNA的表达及DNA甲基化状态。肝脏病学。2001;33(3):561 - 8。gydF4y2Bahttps://doi.org/10.1053/jhep.2001.22507gydF4y2Ba.gydF4y2Ba
李LC。1、哺乳动物细胞中小RNA机制的染色质重构。表观遗传学。2014;9:45-52。gydF4y2Bahttps://doi.org/10.4161/epi.26830gydF4y2Ba.gydF4y2Ba
李旭,付晓东。染色质相关rna作为功能基因组相互作用的促进者。Nat Rev Genet 2019; 20:503-19。gydF4y2Bahttps://doi.org/10.1038/s41576-019-0135-1gydF4y2Ba.gydF4y2Ba
rna介导的基因表达的表观遗传调控。Nat Rev Genet 2015; 16:71-84。gydF4y2Bahttps://doi.org/10.1038/nrg3863gydF4y2Ba.gydF4y2Ba
癌症表观遗传学:从机制到治疗。细胞。2012;150:12-27。gydF4y2Bahttps://doi.org/10.1016/j.cell.2012.06.013gydF4y2Ba.gydF4y2Ba
宋j, Pfeifer GP。氧化的5-甲基胞嘧啶碱有特定的读者群吗?BioEssays。2016; 38:1038-47。gydF4y2Bahttps://doi.org/10.1002/bies.201600126gydF4y2Ba.gydF4y2Ba
王晓燕,王晓燕,王晓燕。氧化5-甲基胞嘧啶碱基与蛋白质的相互作用。中华分子生物学杂志。2020;432:1718-30。gydF4y2Bahttps://doi.org/10.1016/j.jmb.2019.07.039gydF4y2Ba.gydF4y2Ba
周涛,熊娟,王敏,杨楠,王杰,朱斌,徐仁仁。UHRF2的SRA结构域识别羟甲基胞嘧啶的结构基础。摩尔细胞。2014;54:879 - 86。gydF4y2Bahttps://doi.org/10.1016/j.molcel.2014.04.003gydF4y2Ba.gydF4y2Ba
陈涛,陈晓燕,陈晓燕。人类疾病中DNA甲基化机制的遗传改变。表观基因组学。2015;7:247 - 65。gydF4y2Bahttps://doi.org/10.2217/epi.14.80gydF4y2Ba.gydF4y2Ba
Ong CT, Corces VG。CTCF:一种连接基因组拓扑结构和功能的结构蛋白。Nat Rev Genet。2014;15:234-46。gydF4y2Bahttps://doi.org/10.1038/nrg3663gydF4y2Ba.gydF4y2Ba
王晓燕,王晓燕。癌症表观遗传治疗的研究进展。Cureus》2020。gydF4y2Bahttps://doi.org/10.7759/cureus.11725gydF4y2Ba.gydF4y2Ba
王晓燕,王晓燕,王晓燕。表观遗传肿瘤治疗的研究进展。Cureus》2021。gydF4y2Bahttps://doi.org/10.7759/cureus.11725gydF4y2Ba.gydF4y2Ba
徐辉,刘亮,李伟,邹东,于娟,王磊,王成昌。大肠癌转录因子的分子机制及其治疗意义。致癌基因。2021;40:1555 - 69。gydF4y2Bahttps://doi.org/10.1038/s41388-020-01587-3gydF4y2Ba.gydF4y2Ba
Boot A, Oosting J, van Eendenburg JD, Kuppen PJ, Morreau H, van Wezel T.新型结直肠癌细胞系中ELOVL5的甲基化相关转录抑制。PLoS ONE。2017; 12:0184900。gydF4y2Bahttps://doi.org/10.1371/journal.pone.0184900gydF4y2Ba.gydF4y2Ba
魏森伯格DJ,梁G,伦茨HJ。DNA甲基化异常描述了结直肠癌的临床不同亚群,并为表观遗传治疗提供了新的靶点。致癌基因。2018;37:566 - 77。gydF4y2Bahttps://doi.org/10.1038/onc.2017.374gydF4y2Ba.gydF4y2Ba
黄刚,张娟,龚琳,刘东,王旭,陈艳,郭珊。基于DNA甲基化模式的特异性肺鳞癌预后亚型区分。27:e929524-1。gydF4y2Bahttps://doi.org/10.12659/MSM.929524gydF4y2Ba.gydF4y2Ba
严sw, Kim HK, Kim Y, Lee BB, Kim D, Han J, Kim H, Shim YM, Kim DH。支气管活检标本作为不能手术肺癌DNA甲基化分析的替代物。Epigenet。2017;9:1-10。gydF4y2Bahttps://doi.org/10.1186/s13148-017-0432-5gydF4y2Ba.gydF4y2Ba
董卓,崔辉。胶质母细胞瘤的表观遗传代谢调控。中国癌症杂志2019;57:45-51。gydF4y2Bahttps://doi.org/10.1016/j.semcancer.2018.09.002gydF4y2Ba.gydF4y2Ba
施劳德H,冯S,金尼SM,普拉丹S,雅各布森SE。5-羟甲基胞嘧啶与人类胚胎干细胞中的增强子和基因体有关。基因组医学杂志。2011;12:1-8。gydF4y2Bahttps://doi.org/10.1186/gb-2011-12-6-r54gydF4y2Ba.gydF4y2Ba
王晓燕,王晓燕。DNA去甲基化途径的研究进展。BioEssays。2017; 39:1-13。gydF4y2Bahttps://doi.org/10.1002/bies.201600178gydF4y2Ba.gydF4y2Ba
张勇,张勇。TET酶、TDG和DNA去甲基化动力学。大自然。2013;502:472-9。gydF4y2Bahttps://doi.org/10.1038/nature12750gydF4y2Ba.gydF4y2Ba
Hackett JA, Dietmann S,村上K, Down TA, Leitch HG, Surani MA。向基态多能性转变过程中DNA去甲基化的协同机制。干细胞的报道。2013; 1:518-31。gydF4y2Bahttps://doi.org/10.1016/j.stemcr.2013.11.010gydF4y2Ba.gydF4y2Ba
陈晓燕,陈晓燕。TET蛋白在DNA去甲基化和转录中的作用。细胞生物学杂志。2013;14:341-56。gydF4y2Bahttps://doi.org/10.1038/nrm3589gydF4y2Ba.gydF4y2Ba
吴sc,张颖。活性DNA去甲基化:条条大路通罗马。细胞生物学杂志2010;11:607-20。gydF4y2Bahttps://doi.org/10.1038/nrm2950gydF4y2Ba.gydF4y2Ba
徐国良,王娟。Tet酶介导的DNA氧化去甲基化。自然科学进展2015;2:318-28。gydF4y2Bahttps://doi.org/10.1093/nsr/nwv029gydF4y2Ba.gydF4y2Ba
胡旭,张磊,毛思琪,李铮,陈杰,张瑞仁,吴和平,高洁,郭峰,刘伟,徐光发。Tet和TDG介导的DNA去甲基化是体细胞重编程中间充质到上皮转变所必需的。细胞干细胞。2014;14:512-22。gydF4y2Bahttps://doi.org/10.1016/j.stem.2014.01.001gydF4y2Ba.gydF4y2Ba
Bagci H, Fisher AG。DNA去甲基化在多能性和重编程中的作用:tet蛋白和细胞分裂。细胞干细胞。2013;13:265-9。gydF4y2Bahttps://doi.org/10.1016/j.stem.2013.08.005gydF4y2Ba.gydF4y2Ba
Hahn MA, Szabó PE, Pfeifer GP。5-羟甲基胞嘧啶:一种稳定或短暂的DNA修饰?基因组学。2014;104:314-23。gydF4y2Bahttps://doi.org/10.1016/j.ygeno.2014.08.015gydF4y2Ba.gydF4y2Ba
Münzel M, Globisch D, Carell T. 5-羟甲基胞嘧啶,基因组的第6个碱基。Angew Chem Int Ed 2011; 50:6460-8。gydF4y2Bahttps://doi.org/10.1002/anie.201101547gydF4y2Ba.gydF4y2Ba
曾聪,Stroup EK,张卓,赵伯昌,张伟。迈向精准医疗:液体活检中5-羟甲基胞嘧啶癌症生物标志物的发现进展。癌症Commun。2019;39:1-9。gydF4y2Bahttps://doi.org/10.1186/s40880-019-0356-xgydF4y2Ba.gydF4y2Ba
许涛,高辉。羟甲基化与肿瘤:5-羟甲基化能否作为肿瘤诊断和治疗的标志?哼染色体组。2020;14:1-10。gydF4y2Bahttps://doi.org/10.1186/s40246-020-00265-5gydF4y2Ba.gydF4y2Ba
Nebbioso A, Tambaro FP, Dell ' avversana C, Altucci L.癌症表观遗传学:向前发展。公共科学图书馆麝猫。2018;14:1007362。gydF4y2Bahttps://doi.org/10.1371/journal.pgen.1007362gydF4y2Ba.gydF4y2Ba
Taby R, Issa JPJ。癌症表观遗传学。中华癌症杂志2010;60:376-92。gydF4y2Bahttps://doi.org/10.3322/caac.20085gydF4y2Ba.gydF4y2Ba
Joosten SC, Smits KM, Aarts MJ, Melotte V, Koch A, Tjan-Heijnen VC, van Engeland M.肾细胞癌的表观遗传学:机制和临床应用。Nat Rev urrol . 2018; 15:430-51。gydF4y2Bahttps://doi.org/10.1038/s41585-018-0023-zgydF4y2Ba.gydF4y2Ba
瓦尔德斯比诺五世,瓦尔德斯比诺首相。表观遗传疗法在实体瘤治疗中的潜力。Cancer Manag Res. 2015;7:241。gydF4y2Bahttps://doi.org/10.2147/CMAR.S70358gydF4y2Ba.gydF4y2Ba
冯生,De Carvalho DD.靶向表观遗传学在癌症治疗中的临床进展。2021年2月j。gydF4y2Bahttps://doi.org/10.1111/febs.15750gydF4y2Ba.gydF4y2Ba
临床表观遗传学:抓住转译的机会。Nat Rev Genet 2019; 20:109-27。gydF4y2Bahttps://doi.org/10.1038/s41576-018-0074-2gydF4y2Ba.gydF4y2Ba
Lakshmaiah KC, Jacob LA, Aparna S, Lokanatha D, Saldanha SC.组蛋白去乙酰化酶抑制剂对癌症的表观遗传治疗。中华癌症杂志2014;10:469。gydF4y2Bahttps://doi.org/10.4103/0973-1482.137937gydF4y2Ba.gydF4y2Ba
马立军,陈晓明。DNA去甲基化剂与癌症的表观遗传治疗。难以麝猫。2010;70:327-40。gydF4y2Bahttps://doi.org/10.1016/B978-0-12-380866-0.60012-5gydF4y2Ba.gydF4y2Ba
Yoo CB, Jones PA。癌症的表观遗传治疗:过去,现在和未来。药物发现,2006;5:37-50。gydF4y2Bahttps://doi.org/10.1038/nrd1930gydF4y2Ba.gydF4y2Ba
朱WG, Otterson GA。组蛋白去乙酰化酶抑制剂和DNA甲基转移酶抑制剂在人类癌细胞治疗中的相互作用。当代医学化学抗癌剂。2003;3:187-99。gydF4y2Bahttps://doi.org/10.2174/1568011033482440gydF4y2Ba.gydF4y2Ba
Ganesan A, Arimondo PB, Rots MG, Jeronimo C, Berdasco M.表观遗传药物发现的时间轴:从现实到梦想。Epigenet。2019;11:1-17。gydF4y2Bahttps://doi.org/10.1186/s13148-019-0776-0gydF4y2Ba.gydF4y2Ba
Ganai SA。基于不同分类的不同组HDAC抑制剂。在:组蛋白去乙酰化酶抑制剂-神经障碍的外用药物。施普林格;2019.33-38页。gydF4y2Bahttps://doi.org/10.1007/978-981-13-8019-8_5gydF4y2Ba.gydF4y2Ba
O 'Connor OA, Horwitz S, Masszi T, Van Hoof A, Brown P, Doorduijn J, Hess G, Jurczak W, Knoblauch P, Chawla S, Bhat G. Belinostat在复发或难治性周围T细胞淋巴瘤患者中的作用:关键II期believe (CLN-19)研究结果中华临床杂志2015;33:2492。gydF4y2Bahttps://doi.org/10.1200/JCO.2014.59.2782gydF4y2Ba.gydF4y2Ba
Lee HZ, Kwitkowski VE, Del Valle PL, Ricci MS, Saber H, Habtemariam BA, Bullock J, Bloomquist E, Shen YL, Chen XH, Brown J FDA批准:belinostat用于治疗复发或难治性外周t细胞淋巴瘤患者。clinin Cancer Res. 2015; 21:2666-70。gydF4y2Bahttps://doi.org/10.1158/1078-0432gydF4y2Ba.gydF4y2Ba
泛dac抑制剂panobinostat (LBH589)的开发:成功与挑战。癌症。2009;280:233-41。gydF4y2Bahttps://doi.org/10.1016/j.canlet.2009.02.019gydF4y2Ba.gydF4y2Ba
TOCRIS。C一个ncer research product guide (2015).
Costa-Pinheiro P, Montezuma D, Henrique R, Jerónimo C.癌症诊断和预后的表观遗传生物标志物。表观基因组学。2015;7:1003-15。gydF4y2Bahttps://doi.org/10.2217/epi.15.56gydF4y2Ba.gydF4y2Ba
基因和表观遗传改变作为癌症检测、诊断和预后的生物标志物。摩尔杂志。2007;1:26-41。gydF4y2Bahttps://doi.org/10.1016/j.molonc.2007.01.004gydF4y2Ba.gydF4y2Ba
Ziegler A, Koch A, Krockenberger K, Großhennig A.使用DNA生物标志物的个性化医疗:综述。哼麝猫。2012;131:1627-38。gydF4y2Bahttps://doi.org/10.1007/s00439-012-1188-9gydF4y2Ba.gydF4y2Ba
隐藏在光天化日之下:药物诱导肿瘤进化的表观遗传可塑性。Epigenet见解。2019。gydF4y2Bahttps://doi.org/10.1177/2516865719870760gydF4y2Ba.gydF4y2Ba
冯铮,李娟,张建军,张旭。qDNAmod:一种基于统计模型的工具,从SMRT测序数据揭示DNA修饰的细胞间异质性。核酸学报2014;42:13488-99。gydF4y2Bahttps://doi.org/10.1093/nar/gku1097gydF4y2Ba.gydF4y2Ba
冯铮,李娟,张建军,张旭。qDNAmod:一种基于统计模型的工具,从SMRT测序数据揭示DNA修饰的细胞间异质性。核酸学报2015;43:3406。gydF4y2Bahttps://doi.org/10.1093/nar/gkv067gydF4y2Ba.gydF4y2Ba
高东,Herman JG,郭明。人类癌症中DNA损伤修复基因异常表观遗传变化的临床价值。Oncotarget。2016;7:37331。gydF4y2Bahttps://doi.org/10.18632/oncotarget.7949gydF4y2Ba.gydF4y2Ba
范柏玲,王晓燕,王晓燕,等。人类癌症的表观遗传起源。Nat Rev Genet。2006;7:21-33。gydF4y2Bahttps://doi.org/10.1038/nrg1748gydF4y2Ba.gydF4y2Ba
Martínez-Cardús A, Moran S, Musulen E, Moutinho C, Manzano JL, Martinez-Balibrea E, Tierno M, Élez E, Landolfi S, Lorden P, Arribas C.结直肠肿瘤的表观遗传同一性预测局部癌患者的无复发和总生存时间较短。胃肠病学。2016;151:961 - 72。gydF4y2Bahttps://doi.org/10.1053/j.gastro.2016.08.001gydF4y2Ba.gydF4y2Ba
asenov Y, broks D, Gerhäuser C.表观遗传模式的肿瘤内异质性。中国癌症杂志2018;51:12-21。gydF4y2Bahttps://doi.org/10.1016/j.semcancer.2018.01.010gydF4y2Ba.gydF4y2Ba
Shibue T, Weinberg RA。EMT, CSCs和耐药性:机制联系和临床意义。2017; 14:611-29。gydF4y2Bahttps://doi.org/10.1038/nrclinonc.2017.44gydF4y2Ba.gydF4y2Ba
王旭,张宏,陈旭。耐药与癌症耐药的防治。癌症药物耐药。2019;2:141-60。gydF4y2Bahttps://doi.org/10.20517/cdr.2019.10gydF4y2Ba.gydF4y2Ba
Marusyk A, Almendro V, Polyak K.肿瘤内异质性:癌症的一面镜子?癌症杂志2012;12:323-34。gydF4y2Bahttps://doi.org/10.1038/nrc3261gydF4y2Ba.gydF4y2Ba
刘晨,刘玲,陈鑫,沈娟,单娟,徐莹,杨铮,吴玲,夏峰,别萍,崔莹。5-羟甲基胞嘧啶通过下调TET1与肝细胞癌进展相关。PLoS ONE。2013; 8: e62828。gydF4y2Bahttps://doi.org/10.1371/journal.pone.0062828gydF4y2Ba.gydF4y2Ba
刘静,蒋静,莫静,刘冬,曹冬,王红,何莹,王红。肝细胞癌早期全球DNA 5-羟甲基胞嘧啶和5-甲酰胞嘧啶含量降低。肝脏病学。2019;69:196 - 208。gydF4y2Bahttps://doi.org/10.1002/hep.30146gydF4y2Ba.gydF4y2Ba
王鹏,闫燕,于伟,张浩。10 - 11易位蛋白和5-羟甲基胞嘧啶在肝细胞癌中的作用。细胞Prolif。2019;52:12626。gydF4y2Bahttps://doi.org/10.1111/cpr.12626gydF4y2Ba.gydF4y2Ba
蒋娟,颜涛,郭峰。全球DNA 5hmC和CK195hmC+含量:预测小肝细胞癌预后的有希望的生物标志物。咕咕叫杂志。2021;28:3758 - 70。gydF4y2Bahttps://doi.org/10.3390/curroncol28050321gydF4y2Ba.gydF4y2Ba
宋春霞,尹松,马琳,Wheeler A,陈艳,张艳,刘斌,熊娟,张伟,胡娟,周铮。细胞游离DNA中5-羟甲基胞嘧啶的特征提供肿瘤类型和分期的信息。细胞研究》2017;27:1231-41。gydF4y2Bahttps://doi.org/10.1038/cr.2017.106gydF4y2Ba.gydF4y2Ba
李伟,张鑫,吕鑫,尤玲,宋颖,罗铮,张娟,聂娟,郑伟,徐东,王赟。循环游离DNA中5-羟甲基胞嘧啶信号作为人类癌症诊断生物标志物。细胞研究》2017;27:1243 - 2125。gydF4y2Bahttps://doi.org/10.1038/cr.2017.121gydF4y2Ba.gydF4y2Ba
Guler GD, Ning Y, Ku CJ, Phillips T, McCarthy E, Ellison CK, Bergamaschi A, Collin F, Lloyd P, Scott A, Antoine M.利用循环无细胞DNA中的5-羟甲基胞嘧啶特征检测早期胰腺癌。Nat Commun。2020;11:1-12。gydF4y2Bahttps://doi.org/10.1038/s41467-020-18965-wgydF4y2Ba.gydF4y2Ba
徐松,陈亮,沈强,于浩,裴松,杨庭珍,何鑫,王强,李东。循环游离DNA中5-羟甲基胞嘧啶标记作为迟发性阿尔茨海默病的诊断生物标志物。《老年痴呆症杂志》。gydF4y2Bahttps://doi.org/10.3233/JAD-215217gydF4y2Ba.gydF4y2Ba
Jankowska AM, Millward CL, Caldwell CW。DNA修饰作为肿瘤生物标志物和治疗靶点的潜力。专家Rev Mol Diagn. 2015; 15:1325-37。gydF4y2Bahttps://doi.org/10.1586/14737159.2015.1084229gydF4y2Ba.gydF4y2Ba
内斯特C,鲁佐夫A,米汉RR,杜肯DS。酶法和亚硫酸氢盐测序无法区分DNA中的5-甲基胞嘧啶和5-羟甲基胞嘧啶。生物学技术。2010;48:317-9。gydF4y2Bahttps://doi.org/10.2144/000113403gydF4y2Ba.gydF4y2Ba
黄玉伟,黄天明,王林生。从微阵列到基因组尺度测序的DNA甲基组分析。科技癌症治疗。2010;9:39 - 47。gydF4y2Bahttps://doi.org/10.1177/153303461000900203gydF4y2Ba.gydF4y2Ba
赵丽丽,宋娟,刘燕,宋春霞,易春。DNA和RNA表观遗传修饰的图谱。蛋白质细胞。2020;11:792 - 808。gydF4y2Bahttps://doi.org/10.1007/s13238-020-00733-7gydF4y2Ba.gydF4y2Ba
王涛,卢策,Kohli RM。胞嘧啶甲基化和羟甲基化的酶学分析方法。摩尔金属底座,2021。gydF4y2Bahttps://doi.org/10.1016/j.molmet.2021.101314gydF4y2Ba.gydF4y2Ba
刘宇,siejka - zielikowska P, Velikova G,毕宇,袁芳,Tomkova M,白超,陈磊,Schuster-Böckler B,宋昌祥。无亚硫酸氢盐直接检测5-甲基胞嘧啶和5-羟甲基胞嘧啶在碱分辨率。生物科技Nat》。2019;37:424-9。gydF4y2Bahttps://doi.org/10.1038/s41587-019-0041-2gydF4y2Ba.gydF4y2Ba
Cavalcante RG, Patil S, Park Y, Rozek LS, Sartor MA。将DNA甲基化和羟甲基化数据与造币厂管道相结合。可以研究》2017;77:e27-30。gydF4y2Bahttps://doi.org/10.1158/0008-5472gydF4y2Ba.gydF4y2Ba
曹敏,张超,周磊。DNA甲基化检测技术及基于血浆的甲基化生物标志物在胃肠道癌筛查中的应用。表观基因组学。2021;13:1329-42。gydF4y2Bahttps://doi.org/10.2217/epi-2021-0118gydF4y2Ba.gydF4y2Ba
Clark SJ, Smallwood SA, Lee HJ, Krueger F, Reik W, Kelsey G.使用单细胞亚硫酸氢盐测序(scBS-seq)对单细胞DNA甲基化的全基因组基分辨率定位。Nat Protoc。2017;12:534-47。gydF4y2Bahttps://doi.org/10.1038/nprot.2016.187gydF4y2Ba.gydF4y2Ba
刘勇,胡铮,程杰,siejka - zielizynska P,陈杰,Inoue M, Ahmed AA,宋昌祥。5-甲基胞嘧啶及其氧化衍生物的碱基分辨率无减法和无亚硫酸氢盐特异性测序。Nat Commun。2021;12:1-8。gydF4y2Bahttps://doi.org/10.1038/s41467-021-20920-2gydF4y2Ba.gydF4y2Ba
Jurkowski TP。5-羟甲基胞嘧啶的评价技术及应用。Epigenet方法。2020。gydF4y2Bahttps://doi.org/10.1016/B978-0-12-819414-0.00013-6gydF4y2Ba.gydF4y2Ba
刘铮,王铮,贾娥,欧阳涛,潘敏,卢杰,葛强,白岩。全基因组游离DNA甲基化分析的进展与展望。分析师。2019;144:5912-22。gydF4y2Bahttps://doi.org/10.1039/C9AN00935CgydF4y2Ba.gydF4y2Ba
Booth MJ, Branco MR, Ficz G, Oxley D, Krueger F, Reik W, Balasubramanian S. 5-甲基胞嘧啶和5-羟甲基胞嘧啶的单碱基分辨率定量测序。科学。2012;336:934-7。gydF4y2Bahttps://doi.org/10.1126/science.1220671gydF4y2Ba.gydF4y2Ba
Booth MJ, Ost TW, Beraldi D, Bell NM, Branco MR, Reik W, Balasubramanian S. 5-甲基胞嘧啶和5-羟甲基胞嘧啶的氧化亚硫酸氢盐测序。Nat Protoc。2013;8:1841-51。gydF4y2Bahttps://doi.org/10.1038/nprot.2013.115gydF4y2Ba.gydF4y2Ba
氧化亚硫酸氢盐测序:一种实验和计算方案。:修改DNA;2021.p . 333 - 348。gydF4y2Bahttps://doi.org/10.1007/978-1-0716-0876-0_26gydF4y2Ba.gydF4y2Ba
曾辉,何斌,夏斌,白东,吕旭,蔡娟,陈亮,周阿,朱超,高勇,5-羟甲基胞嘧啶在单碱基分辨率下的无亚硫酸盐纳米尺度分析。中国化学学报2018;140:13190-4gydF4y2Bahttps://doi.org/10.1021/jacs.8b08297gydF4y2Ba.gydF4y2Ba
胡凯,丁亚华,李娟。基于高通量亚硫酸氢盐测序数据的DNA甲基化共发生模式快速在线分析工具。BMC Bioinform。2015;16:1-9。gydF4y2Bahttps://doi.org/10.1186/s12859-015-0649-2gydF4y2Ba.gydF4y2Ba
王晓燕,王晓燕。DNA甲基化在肿瘤组织中的作用。学生物化学论文。2019;63:797 - 811。gydF4y2Bahttps://doi.org/10.1042/EBC20190037gydF4y2Ba.gydF4y2Ba
解码DNA中胞嘧啶修饰的化学方法。化学启2015;115:2240-54。gydF4y2Bahttps://doi.org/10.1021/cr5002904gydF4y2Ba.gydF4y2Ba
DNA甲基化:方法和应用的时间轴。麝猫。2011;2:74。gydF4y2Bahttps://doi.org/10.3389/fgene.2011.00074gydF4y2Ba.gydF4y2Ba
冯丽,娄杰。DNA甲基化分析。: Nanotoxicity;2019.p . 181 - 227。gydF4y2Bahttps://doi.org/10.1007/978-1-4939-8916-4_12gydF4y2Ba.gydF4y2Ba
DNA甲基化分析技术。Biogerontology。2003;4:233-50。gydF4y2Bahttps://doi.org/10.1023/A:1025103319328gydF4y2Ba.gydF4y2Ba
杨文华,陈文华,陈文华,等。DNA甲基化分析方法的选择。生物学。2016;5:3。gydF4y2Bahttps://doi.org/10.3390/biology5010003gydF4y2Ba.gydF4y2Ba
Dietrich D, Uhl B, Sailer V, Holmes EE, Jung M, Meller S, Kristiansen G.通过克服PCR抑制,改进了福尔林固定组织和石蜡包被组织的模板DNA的PCR性能。PLoS ONE。2013; 8: e77771。gydF4y2Bahttps://doi.org/10.1371/journal.pone.0077771gydF4y2Ba.gydF4y2Ba
Grunau C, Clark SJ, Rosenthal A.亚硫酸氢盐基因组测序:关键实验参数的系统研究。核酸条例2001;29:e65-e65。gydF4y2Bahttps://doi.org/10.1093/nar/29.13.e65gydF4y2Ba.gydF4y2Ba
Darst RP, Pardo CE, Ai L, Brown KD, Kladde MP。亚硫酸氢盐DNA测序。分子生物学杂志2010;91:7-9。gydF4y2Bahttps://doi.org/10.1002/0471142727.mb0709s91gydF4y2Ba.gydF4y2Ba
表观遗传修饰与人类疾病。生物科技Nat》。2010;28:1057 - 68。gydF4y2Bahttps://doi.org/10.1038/nbt.1685gydF4y2Ba.gydF4y2Ba
癌症表观基因组学:DNA甲基组和组蛋白修饰图谱。Nat Rev Genet 2007; 8:286-98。gydF4y2Bahttps://doi.org/10.1038/nrg2005gydF4y2Ba.gydF4y2Ba
Jones PA, Takai D. DNA甲基化在哺乳动物表观遗传学中的作用。科学。2001;239:1068 - 70。gydF4y2Bahttps://doi.org/10.1126/science.1063852gydF4y2Ba.gydF4y2Ba
信号R, Ginder GD。DNA甲基化。中华血液杂志1999;93:4059-70。gydF4y2Bahttps://doi.org/10.1182/blood.V93.12.4059gydF4y2Ba.gydF4y2Ba
理查森BC。DNA甲基化在调节细胞功能中的作用:自身免疫、衰老和癌症。J减轻。2002;132:2401s - 2405 s。gydF4y2Bahttps://doi.org/10.1093/jn/132.8.2401SgydF4y2Ba.gydF4y2Ba
Luczak MW, jagodzi夫斯基PP. DNA甲基化在癌症发展中的作用。细胞生物学杂志2006;44:43 - 54。gydF4y2Ba
Lee PP, Fitzpatrick DR, Beard C, Jessup HK, Lehar S, Makar KW, Pérez-Melgosa M, Sweetser MT, Schlissel MS, Nguyen S, Cherry SR. Dnmt1和DNA甲基化在T细胞发育、功能和存活中的关键作用。免疫力。2001;15:763 - 74。gydF4y2Bahttps://doi.org/10.1016/s1074 - 7613 (01) 00227 - 8gydF4y2Ba.gydF4y2Ba
未来基因组学研究的单分子DNA测序技术。生物科技趋势》。2008;26:602-11。gydF4y2Bahttps://doi.org/10.1016/j.tibtech.2008.07.003gydF4y2Ba.gydF4y2Ba
van Dijk EL, Jaszczyszyn Y, Naquin D, Thermes C.测序技术的第三次革命。趋势麝猫。2018;34:666 - 81。gydF4y2Bahttps://doi.org/10.1016/j.tig.2018.05.008gydF4y2Ba.gydF4y2Ba
Lee I, Razaghi R, Gilpatrick T, Molnar M, Gershman A, Sadowski N, Sedlazeck FJ, Hansen KD, Simpson JT, Timp W.利用纳米孔测序对人类细胞系染色质可及性和甲基化的同步分析。Nat方法。2020;17:1191-9。gydF4y2Bahttps://doi.org/10.1038/s41592-020-01000-7gydF4y2Ba.gydF4y2Ba
亨德森J,萨尔兹伯格S,法斯曼KH。用隐藏的马尔可夫模型在DNA中寻找基因。《计算生物学杂志》1997;4:127-41。gydF4y2Bahttps://doi.org/10.1089/cmb.1997.4.127gydF4y2Ba.gydF4y2Ba
兰德华,贾恩·M, Eizenga JM, Musselman-Brown A, Olsen HE, Akeson M, Paten B.利用高通量纳米孔测序技术定位DNA甲基化。Nat方法。2017;14:411-3。gydF4y2Bahttps://doi.org/10.1038/nmeth.4189gydF4y2Ba.gydF4y2Ba
Simpson JT, Workman RE, Zuzarte PC, Matei DM, Dursi LJ, Timp W.用纳米孔测序检测DNA胞嘧啶甲基化。Nat方法。2017;14:407-10。gydF4y2Bahttps://doi.org/10.1038/nmeth.4184gydF4y2Ba.gydF4y2Ba
吴KJ。DNA n6 -甲基腺嘌呤(6mA)修饰在真核生物中的表观遗传作用。癌症。2020;494:40-6。gydF4y2Bahttps://doi.org/10.1016/j.canlet.2020.08.025gydF4y2Ba.gydF4y2Ba
邱H, Sarathy A, Schulten K, Leburton JP。二维材料纳米孔对DNA甲基化的检测和定位。:母校呢。2017;1:1-8。gydF4y2Bahttps://doi.org/10.1038/s41699-017-0005-7gydF4y2Ba.gydF4y2Ba
Ameur A, Kloosterman WP, Hestand MS.单分子测序的临床应用。生物科技趋势》。2019;37:72 - 85。gydF4y2Bahttps://doi.org/10.1016/j.tibtech.2018.07.013gydF4y2Ba.gydF4y2Ba
小泰森,奥尼尔NJ, Jain M,奥尔森HE, Hieter P, Snutch TP。基于minion的长读测序和组装扩展了秀丽隐杆线虫参考基因组。基因组研究》2018;28:266 - 74。gydF4y2Bahttps://doi.org/10.1101/gr.221184.117gydF4y2Ba.gydF4y2Ba
Gershman A, Sauria ME, Hook PW, Hoyt SJ, Razaghi R, Koren S, Altemose N, Caldas GV, Vollger MR, Logsdon GA, Rhie A.完整人类基因组的表观遗传模式。bioRxiv。2021.gydF4y2Bahttps://doi.org/10.1101/2021.05.26.443420gydF4y2Ba.gydF4y2Ba
Minnoye L, Marinov GK, Krausgruber T, Pan L, Marand AP, Secchia S, Greenleaf WJ, Furlong EE, Zhao K, Schmitz RJ, Bock c。Nat Rev Methods引物2021;1:1-24。gydF4y2Bahttps://doi.org/10.1038/s43586-020-00008-9gydF4y2Ba.gydF4y2Ba
沈磊,华兰德公司。DNA甲基化分析方法。Curr Opin clininnutr Metab Care. 2007; 10:576-81。gydF4y2Bahttps://doi.org/10.1097/MCO.0b013e3282bf6f43gydF4y2Ba.gydF4y2Ba
王晓燕,王晓燕。DNA甲基化分析方法的研究进展。抗氧化氧化还原信号。2013;18:1972-86。gydF4y2Bahttps://doi.org/10.1089/ars.2012.4923gydF4y2Ba.gydF4y2Ba
周丽,沈锐,陈晓燕,陈晓燕。基于Infinium®检测技术的全基因组DNA甲基化分析。表观基因组学。2009;1:177 - 200。gydF4y2Bahttps://doi.org/10.2217/epi.09.14gydF4y2Ba.gydF4y2Ba
孙XX,于强。肿瘤细胞瘤内异质性及其对癌症治疗的意义。药理学学报2015;36:1219-27。gydF4y2Bahttps://doi.org/10.1038/aps.2015.92gydF4y2Ba.gydF4y2Ba
Adusumalli S, Mohd Omar MF, Soong R, Benoukraf T.全基因组亚硫酸氢盐测序分析方法。短暂的Bioinform。2015;16:369 - 79。gydF4y2Bahttps://doi.org/10.1093/bib/bbu016gydF4y2Ba.gydF4y2Ba
De Coster W, Weissensteiner MH, Sedlazeck FJ。面向人群规模的长读测序。Nat Rev Genet, 2021年。gydF4y2Bahttps://doi.org/10.1038/s41576-021-00367-3gydF4y2Ba.gydF4y2Ba
孙硕,Noviski A, Yu x。methylqa:一种亚硫酸盐处理的甲基化测序质量评估管道。BMC Bioinform。2013;14:1-9。gydF4y2Bahttps://doi.org/10.1186/1471-2105-14-259gydF4y2Ba.gydF4y2Ba
刘亮,李勇,李胜,胡楠,何莹,庞锐,林丹,陆林,罗明。新一代测序系统的比较。生物技术杂志,2012。gydF4y2Bahttps://doi.org/10.1155/2012/251364gydF4y2Ba.gydF4y2Ba
唐勇,郑淑娟,祁CB,冯永强,袁BF。化学衍生化-液相色谱-串联质谱法同时灵敏测定基因组DNA中5-甲基胞嘧啶及其氧化产物肛门化学2015;87:3445-52。gydF4y2Bahttps://doi.org/10.1021/ac504786rgydF4y2Ba.gydF4y2Ba
沈玲,张莹。5-羟甲基胞嘧啶:世代、命运与基因组分布。细胞生物学杂志2013;25:289-96。gydF4y2Bahttps://doi.org/10.1016/j.ceb.2013.02.017gydF4y2Ba.gydF4y2Ba
杜艳,王艳,胡旭,刘杰,刁杰。癌症基因组中5-甲基胞嘧啶和5-羟甲基胞嘧啶的单分子定量研究。视图。2020;1:e9。gydF4y2Bahttps://doi.org/10.1002/viw2.9gydF4y2Ba.gydF4y2Ba
Rand AC, Jain M, Eizenga J, Musselman-Brown A, Olsen HE, Akeson M, Paten B.胞嘧啶变异呼叫高通量纳米孔测序。bioRxiv。2016.gydF4y2Bahttps://doi.org/10.1101/047134gydF4y2Ba.gydF4y2Ba
Jain M.用牛津纳米孔MinION进行高覆盖长读DNA测序。加州大学;2017.gydF4y2Bahttps://escholarship.org/uc/item/11p602ctgydF4y2Ba.gydF4y2Ba
Kirk JM, Kim SO, Inoue K, Smola MJ, Lee DM, Schertzer MD, Wooten JS, Baker AR, Sprague D, Collins DW, Horning CR.基于K -mer含量的长链非编码rna功能分类。Nat麝猫。2018;50:1474 - 82。gydF4y2Bahttps://doi.org/10.1038/s41588-018-0207-8gydF4y2Ba.gydF4y2Ba
王燕,Navin NE。单细胞测序技术的进展和应用。摩尔细胞。2015;58:598 - 609。gydF4y2Bahttps://doi.org/10.1016/j.molcel.2015.05.005gydF4y2Ba.gydF4y2Ba
单细胞表观基因组学:技术和新应用。Nat Rev Genet 2015; 16:716-26。gydF4y2Bahttps://doi.org/10.1038/nrg3980gydF4y2Ba.gydF4y2Ba
Grønbaek K, Hother C, Jones PA。癌症的表观遗传变化。学院。2007; 115:1039-59。gydF4y2Bahttps://doi.org/10.1111/j.1600-0463.2007.apm_636.xml.xgydF4y2Ba.gydF4y2Ba
哈里斯TJ,麦考密克F.癌症分子病理学。中华医学杂志2010;7:251-65。gydF4y2Bahttps://doi.org/10.1038/nrclinonc.2010.41gydF4y2Ba.gydF4y2Ba
Chatterjee N, Bivona TG。多疗法和靶向癌症耐药性。趋势癌症。2019;5:170 - 82。gydF4y2Bahttps://doi.org/10.1016/j.trecan.2019.02.003gydF4y2Ba.gydF4y2Ba
达文波特CF, Scheithauer T, Dunst A, Bahr FS, Dorda M, Wiehlmann L, Tran DDH。使用纳米孔测序技术的全基因组甲基化定位在肝细胞癌中识别新的肿瘤抑制基因。国际分子科学,2021;22:3937。gydF4y2Bahttps://doi.org/10.3390/ijms22083937gydF4y2Ba.gydF4y2Ba
坂本勇,Sereewattanawoot S, Suzuki A.癌症基因组学长读测序的新时代。中华医学杂志。2020;65:3-10。gydF4y2Bahttps://doi.org/10.1038/s10038-019-0658-5gydF4y2Ba.gydF4y2Ba
庄健,霍青,杨芳,谢宁。组蛋白修饰在乳腺癌进展中的作用及转移表观遗传决定因素研究的先进技术工具。麝猫。2020。gydF4y2Bahttps://doi.org/10.3389/fgene.2020.603552gydF4y2Ba.gydF4y2Ba
Nattestad M, Goodwin S, Ng K, Baslan T, Sedlazeck FJ, Rescheneder P, Garvin T, Fang H, Gurtowski J, Hutton E, Tseng E乳腺癌细胞系长读DNA和RNA测序揭示的复合物重排和癌基因扩增。基因组研究》2018;28:1126-35。gydF4y2Bahttps://doi.org/10.1101/gr.231100.117gydF4y2Ba.gydF4y2Ba
Minervini CF、Cumbo C、Orsini P、Anelli L、Zagaria A、Impera L、Coccaro N、Brunetti C、Minervini A、Carcieri P、Tota g基于纳米孔MinION技术的BCR-ABL1阳性白血病基因突变分析中华医学杂志2017;103:33-7。gydF4y2Bahttps://doi.org/10.1016/j.yexmp.2017.06.007gydF4y2Ba.gydF4y2Ba
Tang AD, Soulette CM, van Baren MJ, Hart K, hrabetta - robinson E, Wu CJ, Brooks AN。慢性淋巴细胞白血病中SF3B1突变的全长转录物表征揭示了保留内含子的下调。Nat Commun。2020;11:1-12。gydF4y2Bahttps://doi.org/10.1038/s41467-020-15171-6gydF4y2Ba.gydF4y2Ba
徐旭,贾军,郭敏。纳米结构/纳米材料在单细胞采样中的应用研究进展。前化学》2020。gydF4y2Bahttps://doi.org/10.3389/fchem.2020.00718gydF4y2Ba.gydF4y2Ba
Kelsey G, Stegle O, Reik W.单细胞表观基因组学:记录过去和预测未来。科学。2017;358:69 - 75。gydF4y2Bahttps://doi.org/10.1126/science.aan6826gydF4y2Ba.gydF4y2Ba
如何将数十亿的短读映射到基因组上。生物科技Nat》。2009;27:455-7。gydF4y2Bahttps://doi.org/10.1038/nbt0509-455gydF4y2Ba.gydF4y2Ba
Steinberg KM, Schneider VA, Alkan C,等。建立和改进参考基因组组合。Proc IEEE。2017; 105:422-35。gydF4y2Bahttps://doi.org/10.1109/JPROC.2016.2645402gydF4y2Ba.gydF4y2Ba
牛津纳米孔技术公司。1Dsquared工具包可在商店:提高精度,简单的准备;2017.gydF4y2Bahttps://nanoporetech.com/about-us/news/1d-squared-kit-available-store-boost-accuracy-simple-prepgydF4y2Ba.已于2018年4月20日访问。gydF4y2Ba
棕色的CG。牛津纳米孔技术公司:GridION X5续作。gydF4y2Bahttps://www.youtube.com/results?search_query=Oxford+Nanopore+Technologies%3A+GridION+X5+The+Sequel+gydF4y2Ba.2017年3月直播。已于2018年5月29日访问。gydF4y2Ba
Cornelis S, Gansemans Y, Vander Plaetsen AS, Weymaere J, Willems S, Deforce D, Van Nieuwerburgh F.基于纳米孔测序的法医三等位基因SNP基因分型。国际法医科学2019;38:204-10。gydF4y2Bahttps://doi.org/10.1016/j.fsigen.2018.11.012gydF4y2Ba.gydF4y2Ba
王燕,赵燕,波拉斯·阿,王燕,欧凯夫。纳米孔测序技术、生物信息学及其应用。生物科技Nat》。2021;39(2021):1348 - 65。gydF4y2Bahttps://doi.org/10.1038/s41587-021-01108-xgydF4y2Ba.gydF4y2Ba
威克RR,贾德LM,霍尔特KE。牛津纳米孔测序的神经网络基本调用工具性能。基因组医学杂志。2019;20:1-10。gydF4y2Bahttps://doi.org/10.1186/s13059-019-1727-ygydF4y2Ba.gydF4y2Ba
阿玛拉辛河SL,苏S,董x, Zappia L, Ritchie ME, Gouil Q.长读测序数据分析的机遇和挑战。基因组医学杂志。2020;21:1-16。gydF4y2Bahttps://doi.org/10.1186/s13059-020-1935-5gydF4y2Ba.gydF4y2Ba
Vu T, Davidson SL, Borgesi J, Maksudul M, Jeon TJ, Shim J.拼接谜:纳米孔技术在癌症生物标志物的检测和量化。RSC放置2017;7:42653 - 66。gydF4y2Bahttps://doi.org/10.1039/C7RA08063HgydF4y2Ba.gydF4y2Ba
Khoo BL, Chaudhuri PK, Ramalingam N, Tan DS, Lim CT, Warkiani ME。单细胞分析方法探测肿瘤异质性。国际癌症杂志2016;139:243-55。gydF4y2Bahttps://doi.org/10.1002/ijc.30006gydF4y2Ba.gydF4y2Ba
Euskirchen P, Bielle F, Labreche K, Kloosterman WP, Rosenberg S, Daniau M,等。利用实时纳米孔测序对脑肿瘤的当日基因组和表观基因组诊断。Acta Neuropathol。2017;134:691 - 703。gydF4y2Bahttps://doi.org/10.1007/s00401-017-1743-5gydF4y2Ba.gydF4y2Ba
周亮,邱强,周强,李娟,于敏,李凯,徐亮,柯欣,徐红,陆波,王辉。Long-read测序揭示宫颈癌中高分辨率HPV整合及其致癌进展。Nat Commun。2022;13:1-18。gydF4y2Bahttps://doi.org/10.1038/s41467-022-30190-1gydF4y2Ba.gydF4y2Ba
下一代测序技术时代植物微生物相互作用的分析。植物学报(英文版)2014;gydF4y2Bahttps://doi.org/10.3389/fpls.2014.00216gydF4y2Ba.gydF4y2Ba
Malla MA, Dubey A, Kumar A, Yadav S, Hashem A, Abd Allah EF。探索人类微生物群:下一代测序在疾病诊断和治疗中的潜在未来作用。Immunol前面。2019;9:2868。gydF4y2Bahttps://doi.org/10.3389/fimmu.2018.02868gydF4y2Ba.gydF4y2Ba
倪萍,黄宁,聂芳,张军,张铮,吴斌,白亮,刘伟,肖春林,罗芳,王杰。基于深度学习的纳米孔数据全基因组检测植物胞嘧啶甲基化。Nat Commun。2021;12:1-11。gydF4y2Bahttps://doi.org/10.1038/s41467-021-26278-9gydF4y2Ba.gydF4y2Ba
刘颖,Rosikiewicz W, Pan Z, Jillette N, Wang P, Taghbalout A, Foox J, Mason C, Carroll M, Cheng A, Li S.牛津纳米孔测序的DNA甲基化调用工具:调查和人类表观基因组全基因组评估。基因组医学杂志。2021;22:1-33。gydF4y2Bahttps://doi.org/10.1186/s13059-021-02510-zgydF4y2Ba.gydF4y2Ba
库马尔KR,考利MJ,戴维斯RL。下一代测序和新兴技术。中国输血杂志2019;45:661-73。gydF4y2Bahttps://doi.org/10.1055/s-0039-1688446gydF4y2Ba.gydF4y2Ba
倪萍,黄宁,张铮,王德鹏,梁峰,苗艳,肖春林,罗芳,王娟。深度学习在纳米孔测序读取中检测DNA甲基化状态。生物信息学。2019;35:4586 - 95。gydF4y2Bahttps://doi.org/10.1093/bioinformatics/btz276gydF4y2Ba.gydF4y2Ba
Fujimoto A, Wong JH, Yoshii Y, Akiyama S, Tanaka A, Yagi H, Shigemizu D, Nakagawa H, Mizokami M, Shimada M.长reads全基因组测序揭示了人类癌症遗传变异和体细胞突变的复杂结构和结构变异的起源。基因组医学。2021;13:1-15。gydF4y2Bahttps://doi.org/10.1186/s13073-021-00883-1gydF4y2Ba.gydF4y2Ba
Wongsurawat T, Jenjaroenpun P, De Loose A, Alkam D, Ussery DW, Nookaew I, Leung YK, Ho SM, Day JD, Rodriguez A.一种新颖的cas9靶向长读检测同时检测弥漫性胶质瘤新鲜活检中IDH1/2突变和临床相关MGMT甲基化。神经病理学报。2020;8:1-13。gydF4y2Bahttps://doi.org/10.1186/s40478-020-00963-0gydF4y2Ba.gydF4y2Ba
Gilpatrick T, Lee I, Graham JE, Raimondeau E, Bowen R, Heron A, Downs B, Sukumar S, Sedlazeck FJ, Timp W. cas9引导适配器结扎靶向纳米孔测序。生物科技Nat》。2020;38:433-8。gydF4y2Bahttps://doi.org/10.1038/s41587-020-0407-5gydF4y2Ba.gydF4y2Ba
Gabrieli T, Sharim H, Fridman D, Arbib N, Michaeli Y, Ebenstein Y.利用cas9辅助靶向染色体片段(CATCH)对人BRCA1进行选择性纳米孔测序。核酸学报2018;46:e87-e87。gydF4y2Bahttps://doi.org/10.1093/nar/gky411gydF4y2Ba.gydF4y2Ba
尤因AD, Smits N, Sanchez-Luque FJ, Faivre J, Brennan PM, Richardson SR, Cheetham SW, Faulkner GJ。纳米孔测序可以实现全面的转座元素表观基因组分析。摩尔细胞。2020;80:915-28。gydF4y2Bahttps://doi.org/10.1016/j.molcel.2020.10.024gydF4y2Ba.gydF4y2Ba
Djirackor L, Halldorsson S, Niehusmann P, Leske H, Capper D, Kuschel LP, Pahnke J, due - t - nnessen BJ, Langmoen IA, Sandberg CJ, Euskirchen P脑肿瘤术中DNA甲基化分类对神经外科策略的影响。神经肿瘤学杂志。2021;3:149。gydF4y2Bahttps://doi.org/10.1093/noajnl/vdab149gydF4y2Ba.gydF4y2Ba
确认gydF4y2Ba
此外,我们感谢Tibebe Antonios博士对本文最终版本的修订和评论。gydF4y2Ba
资金gydF4y2Ba
这项研究没有从公共、商业或非营利部门的资助机构获得任何具体的资助。gydF4y2Ba
作者信息gydF4y2Ba
作者和联系gydF4y2Ba
贡献gydF4y2Ba
YW和BA对手稿进行构思、撰写、审稿,整理、构造表格和图表,并请求许可转载表格和图表;SA, ST, MF, EK, TK, MD, EY, TG, TN, EA, DO, AD选标题,审稿,对重要知识内容进行批判性修改和排版;BD对最终稿进行了审稿和评论。所有作者阅读并批准了最终稿件。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
伦理批准和同意参与gydF4y2Ba
不适用。gydF4y2Ba
同意出版gydF4y2Ba
不适用。gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有竞争利益。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba
伟德体育在线施普林格自然对出版的地图和机构附属的管辖权要求保持中立。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。欲查看此许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.创作共用公共领域奉献放弃书(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本文提供的数据,除非在数据的信用额度中另有说明。gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
Ahmed, y.w., Alemu, B.A, Bekele, s.agydF4y2Baet al。gydF4y2Ba纳米孔测序单细胞图谱时代的表观遗传肿瘤异质性。gydF4y2Ba中国EpigenetgydF4y2Ba14gydF4y2Ba107(2022)。https://doi.org/10.1186/s13148-022-01323-6gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s13148-022-01323-6gydF4y2Ba
关键字gydF4y2Ba
- 表观遗传学gydF4y2Ba
- 纳米孔测序gydF4y2Ba
- CpG岛gydF4y2Ba
- 肿瘤的异质性gydF4y2Ba
- 甲基化胞嘧啶gydF4y2Ba
- 牛津纳米孔gydF4y2Ba
- 奴才gydF4y2Ba