- 研究gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
诱导的肝干细胞通过TET1和CTCF共同调节Myc的高表达来维持自我更新gydF4y2Ba
细胞与生物科学gydF4y2Ba体积gydF4y2Ba12gydF4y2Ba,文章号:gydF4y2Ba143gydF4y2Ba(gydF4y2Ba2022gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
本研究以小鼠胚胎成纤维细胞(mef)为原料,通过谱系重编程产生具有自我更新和向肝细胞和胆管细胞双分化能力的诱导肝干细胞(iHepSCs)。然而,iHepSC自我更新的机制尚未阐明。Tet1调控的活性去甲基化在干细胞的自我更新中起着重要作用,包括多能干细胞和成体干细胞。在此,我们研究了tet1调节的去甲基化在iHepSCs自我更新中的作用和机制。gydF4y2Ba
方法gydF4y2Ba
采用免疫荧光染色、定量反转录PCR和western blotting分析iHepSCs和mef中Tet1的甲基化水平和表达情况。然后,通过CCK8、菌落形成和球形成实验分析Tet1下调对iHepSCs增殖和自我更新的影响。通过染色质免疫沉淀、亚硫酸氢盐序列PCR和甲基化敏感限制性内切酶PCR研究Tet1调控iHepSCs自我更新的机制。gydF4y2Ba
结果gydF4y2Ba
iHepSCs中5hmC的高表达和5mC的低表达均伴随Tet1的高表达。shRNA下调Tet1表达后,iHepSCs的增殖和自我更新能力受到抑制,Myc的表达也降低。Myc在iHepSCs中的高表达水平保持自我更新,并受到Tet1的调控,Tet1直接与Myc启动子的CBS-1和A位点区域结合,并使CpG胞嘧啶去甲基化。此外,CTCF还与Myc启动子的CBS-1和site A区域结合,并与TET1一起调节Myc的表达。gydF4y2Ba
结论gydF4y2Ba
iHepSCs的自我更新通过Myc的高表达来维持,而Myc的高表达由TET1和CTCF共同调控。这项研究可能为干细胞的自我更新提供新的见解,可以促进“重编程”干细胞的研究和应用。gydF4y2Ba
简介gydF4y2Ba
许多不同类型的组织特异性祖细胞/干细胞是由成纤维细胞通过直接谱系重编程产生的[gydF4y2Ba1gydF4y2Ba].在前期研究中,我们通过过表达Hnf1b和Foxa3这两种肝脏器官发生转录因子,建立了由mef直接转化的诱导肝干细胞(iHepSCs) [gydF4y2Ba2gydF4y2Ba].进一步研究表明,iHepSCs在体外可稳定扩增(超过50个传代),并保持正常染色体数和向肝细胞或胆管细胞分化的双电位能力,且无明显衰老[gydF4y2Ba3.gydF4y2Ba].显然,在体外长期培养过程中,iHepSCs保持了自我更新的能力,即维持未分化状态的分裂。然而,iHepSCs维持自我更新能力的确切机制尚未阐明。gydF4y2Ba
干细胞的自我更新不仅需要细胞周期的控制,而且还依赖于干细胞保持多能性或多能性,其中涉及DNA甲基化、RNA加工和组蛋白修饰等表观遗传调控机制[gydF4y2Ba4gydF4y2Ba].DNA甲基化通过甲基化和去甲基化之间的转换影响基因表达,这通常发生在CpG二核苷酸的背景下[gydF4y2Ba5gydF4y2Ba].DNA去甲基化既可以被动发生,也可以主动发生。被动的过程发生在DNA复制期间,而主动的DNA去甲基化需要10 - 11易位(TET)蛋白家族中的酶的作用,包括TET1, TET2和TET3 [gydF4y2Ba6gydF4y2Ba].TET酶,2-羟戊二酸盐,氧和铁依赖的双加氧酶,能够催化5-甲基胞嘧啶(5-mC)氧化成5-羟甲基胞嘧啶(5-hmC)。这个反应是最终导致DNA去甲基化的一系列事件的第一步[gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba].TET蛋白除了依赖于氧化活性外,还与靶基因的启动子区结合,以转录因子或与其他转录因子形成复合物的形式调控其表达[gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba].gydF4y2Ba
近年来,TET家族蛋白的功能在发育过程中的表观遗传重编程和干细胞研究中得到了越来越多的关注[gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba].在TET蛋白家族中,TET1不仅在胚胎干细胞和神经元中高度表达,而且在诱导多能干细胞的建立中也至关重要[gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba].此外,据报道TET1参与干细胞自我更新的维持,包括胚胎干细胞和组织特异性干细胞[gydF4y2Ba13gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba].在本研究中,我们比较了甲基化水平和Tet1在iHepSCs和mef中的表达水平。然后,通过shRNA下调Tet1在维持iHepSCs自我更新中的作用,并研究了iHepSCs自我更新的机制。本研究可能为干细胞的自我更新提供新的思路,促进干细胞的研究和应用。gydF4y2Ba
材料与方法gydF4y2Ba
细胞培养和细胞分化gydF4y2Ba
mef和293T细胞在添加10%胎牛血清、100 U/mL青霉素和100µg/mL链霉素的DMEM中培养。如先前报道,iHepSCs在SCM-A培养基中培养[gydF4y2Ba2gydF4y2Ba].如我们之前报道的程序所述,诱导iHepSCs的肝脏分化和胆管细胞分化[gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba].TET-IN-C35 (AOB11121)购自AOBIOUS, Inc.,溶解于DMSO中制备5 mM原液。对于低密度脂蛋白(LDL)摄取试验,将肝分化细胞与10µg/mL DiI AcLDL (Invitrogen)在37℃下孵育4小时,然后用荧光显微镜观察。糖原储存采用周期性酸性希夫(PAS)染色试剂盒(SJ1269,双健生物技术,上海),根据制造商的用户手册进行检测。gydF4y2Ba
慢病毒的产生和感染gydF4y2Ba
将Myc的开放阅读框(ORF)单独克隆到pLVX-IRES-ZsGreen1 (Clontech)慢病毒载体中。将靶向Tet1、Myc和Ctcf的短发夹rna (Short hairpin rna, shRNAs)分别克隆到pLKO中。1lentiviral vector (#10878, Addgene), which can induce stable and long-term gene silencing in mammalian cells [22gydF4y2Ba].shrna的目标序列在附加文件中显示gydF4y2Ba1gydF4y2Ba:表S1。慢病毒被包装在293T细胞中,并与慢病毒表达载体和慢病毒包装载体共转染,包括psPAX2 (#12260, Addgene)和pMD2。G (#12259, Addgene)。在转染后48和72小时收集病毒上清。慢病毒颗粒经45000倍超离心浓缩后gydF4y2BaggydF4y2Ba在PBS中重悬2小时,用TransLv™慢病毒qPCR滴定试剂盒(TransGen Biotech, China)测定滴度。病毒感染细胞的感染倍数(MOI)为10。gydF4y2Ba
定量反转录PCR (qRT-PCR)gydF4y2Ba
使用TRIzol试剂(TaKaRa)提取总RNA,并根据制造商的指南使用MMLV逆转录酶(Promega)合成第一个cdna。采用LighterCylcer®96 (Roche)和SYBR Premix Ex Taq试剂(TaKaRa)进行qRT-PCR分析。所有样本均作三份检查。GAPDH作为内部对照。引物在附加文件中列出gydF4y2Ba1gydF4y2Ba:表S2。gydF4y2Ba
细胞增殖试验gydF4y2Ba
细胞增殖试验如前所述[gydF4y2Ba3.gydF4y2Ba].简单地说,1 × 10gydF4y2Ba3.gydF4y2Ba每孔细胞镀于96孔板上,孵育24 h。然后,每孔加入10µL CCK8试剂(Dojindo, Japan), 37℃孵育4 h。每天在450 nm处测量光密度值,连续5天。gydF4y2Ba
细胞球形成试验gydF4y2Ba
消化并计数贴壁细胞,1 × 10gydF4y2Ba4gydF4y2Ba细胞被播种到超低附着6孔板(#3471,康宁)每孔。细胞在37℃下培养7天,形成细胞球。在显微镜下计数细胞球的数量。gydF4y2Ba
菌落形成试验gydF4y2Ba
将iHepSCs以每孔200个细胞的密度播种到6孔板中,在37℃的细胞培养箱中培养7天。然后用4% PFA固定细胞,用结晶紫染色液(#Y1232,育秀生物科技,中国)染色。计数含有50个以上细胞的菌落数量。gydF4y2Ba
Tet酶活性测定gydF4y2Ba
用核提取试剂盒(ab113474, Abcam)制备iHepSCs和mef中的核蛋白,用Optiblot Bradford Reagent (ab119216, Abcam)定量。采用Tet羟化酶活性定量试剂盒(比色法)(ab156912, Abcam),按照厂家说明书检测Tet家族5mC羟化酶的活性,包括TET1, TET2和TET3。每次测定的核提取物量为每个样品10µg。光学值用Molecular Devices Spectra MAX190在450nm处测量,可选参考波长为655nm。Tet酶活计算公式为:Tet酶活(OD/min/mg) =(样品OD -空白OD)/(蛋白量(µg) × 90(min)) × 1000。gydF4y2Ba
染色质免疫沉淀反应gydF4y2Ba
染色质免疫沉淀(ChIP)检测使用EZ-ChIP试剂盒(Millipore, USA)按照制造商的方案进行。简单地说,细胞首先用1%的甲醛固定,以产生dna -蛋白质交联。然后,将细胞裂解并超声处理,生成200 ~ 300 bp的染色质片段,并将细胞裂解液与靶蛋白抗体进行免疫沉淀。免疫沉淀后,分离基因组DNA,用蛋白酶k消化结合蛋白,使用附加文件中列出的引物进行qRT-PCRgydF4y2Ba1gydF4y2Ba:表S3。gydF4y2Ba
Immunocytofluorescence染色gydF4y2Ba
免疫细胞荧光染色既往报道[gydF4y2Ba2gydF4y2Ba].简单地说,用0.3% Triton X-100渗透固定细胞5分钟,然后用1% BSA阻断1小时。细胞用一抗在4℃孵育过夜,然后用二抗孵育。核用DAPI反染色。阴性对照,省略一抗,只使用二抗。gydF4y2Ba
Western blot和dot blot检测gydF4y2Ba
如前所述进行Western blotting分析[gydF4y2Ba23gydF4y2Ba].点印迹法如下:将样品DNA稀释至最终DNA浓度为1000 ng/µL、500 ng/µL和200 ng/µL。将2微升稀释后的DNA滴入醋酸纤维素膜上,6× SSC浸泡5分钟,70℃烘箱烘干,5%脱脂牛奶在TBS-T中堵塞2-4小时,用5hmC或5mC抗体检测后,用酶标山羊抗小鼠IgG孵育膜,用荧光剂(#T7103Q, Takara)暴露斑点标记。用于免疫细胞荧光染色,斑点杂交和western blot的一抗列在附加文件中gydF4y2Ba1gydF4y2Ba:表S4。gydF4y2Ba
亚硫酸氢盐序列PCR (BSP)gydF4y2Ba
使用QIAamp DNA Mini Kit (51304, QIAGEN)从细胞中提取基因组DNA,并使用methylleasy Xceed快速DNA亚硫酸氢修饰试剂盒(ME002, Human Genetic Signatures)将亚硫酸氢介导的胞嘧啶转化为尿嘧啶。扩增PCR在50µL的反应体积中进行,其中包含100 ng基因组DNA模板,每个引物0.4µM, 0.3 mM dNTP和1.25 U TaKaRa EpiTaq™HS (R110Q, TaKaRa),在ProFlex PCR系统上进行,程序如下:在98°C变性10 s, 55°C退火30 s, 72°C延伸30 s。的网站上使用Methprimer设计引物gydF4y2Bahttp://www.urogene.org/methprimer/index.htmlgydF4y2Ba.引物的序列列在附加文件中gydF4y2Ba1gydF4y2Ba:表S5。PCR产物用MinElute PCR纯化试剂盒(28004,QIAGEN)进行纯化。纯化的片段克隆到pMD™18-T载体(6011,TaKaRa)。从三个独立的扩增实验中选取10个克隆进行测序。序列结果由在线QUMA软件处理[gydF4y2Ba24gydF4y2Ba].gydF4y2Ba
甲基化敏感限制性内切酶pcr (MSRE-PCR)gydF4y2Ba
从细胞中提取的基因组DNA分别用AciI (R0551 V, New England BioLabs)和HpaII (R0171 V, New England BioLab)消化。以EcoRI酶切的基因组DNA作为对照。以MinElute Gel Extraction Kit (28604, QIAGEN)纯化消化后的基因组DNA为模板进行PCR扩增。MSRE-PCR引物序列在附加文件中列出gydF4y2Ba1gydF4y2Ba:表S6。PCR产物在2%琼脂糖凝胶中进行电泳。gydF4y2Ba
统计分析gydF4y2Ba
所有统计分析均使用GraphPad Prism (version 7.0)程序进行。数据显示为平均值和平均值的标准误差。统计方法见图例,以P < 0.05为差异有统计学意义。gydF4y2Ba
结果gydF4y2Ba
iHepSCs中5hmC和Tet1的表达水平高于mefgydF4y2Ba
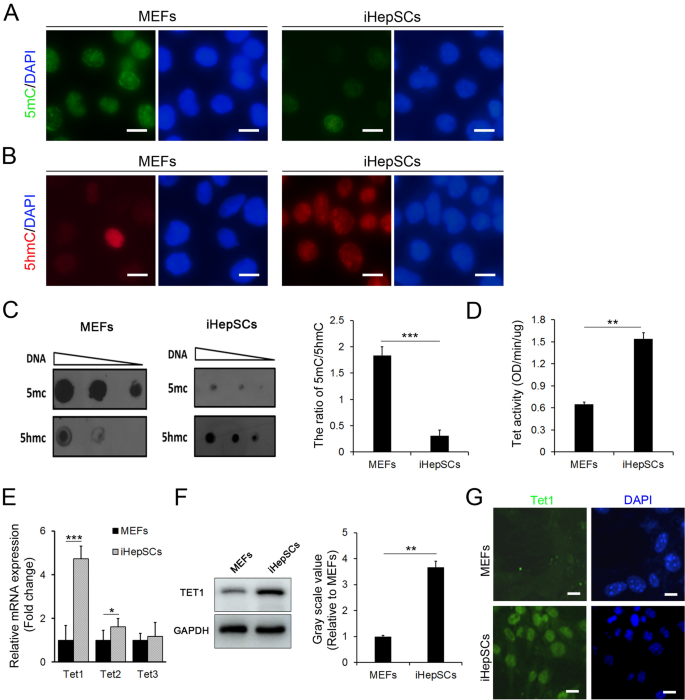
在之前的研究中,我们证明iHepSCs在大量扩张后仍保持自我更新的能力[gydF4y2Ba3.gydF4y2Ba].虽然DNA甲基化被报道参与了干细胞自我更新和干性的维持[gydF4y2Ba25gydF4y2Ba],检测iHepSCs和祖先细胞mef的DNA甲基化(5mC)和羟甲基化(5hmC)水平。免疫细胞荧光染色结果(图;gydF4y2Ba1gydF4y2BaA, B)和点印迹分析(图。gydF4y2Ba1gydF4y2BaC)显示iHepSCs中5mC的水平远低于mef,而5hmC的水平远高于mef。因为活性DNA去甲基化通常主要是由TET酶介导的,TET酶通过将5mc氧化为5hmC来修饰甲基[gydF4y2Ba26gydF4y2Ba],测定iHepSCs中TET酶活性。结果显示,iHepSCs中TET的酶活性明显高于MEFs(图。gydF4y2Ba1gydF4y2BaD).与这一发现一致的是,Tet1和Tet2在iHepSCs中的表达明显高于mef。具体而言,Tet1在iHepSCs中的表达明显高于Tet2和Tet3的表达(图2)。gydF4y2Ba1gydF4y2BaE)。我们进一步用western blot和免疫细胞荧光染色证实TET1在iHepSCs中的表达明显高于mef(图。gydF4y2Ba1gydF4y2BaF, G)。gydF4y2Ba
iHepSCs表现出较高水平的5hmC和Tet1表达。gydF4y2Ba一个gydF4y2Ba,gydF4y2BaBgydF4y2Ba免疫细胞荧光染色显示iHepSCs中5mC的水平远低于MEFs (gydF4y2Ba一个gydF4y2Ba),而iHepSCs中5hmC的水平远高于mef (gydF4y2BaBgydF4y2Ba).gydF4y2BaCgydF4y2Ba斑点杂交检测显示mef和iHepSCs中5mC和5hmC的基因组水平。gydF4y2BaDgydF4y2Ba与mef相比,iHepSCs中的Tet酶活性显著增加。gydF4y2BaEgydF4y2BaqRT-PCR结果显示,Tet1 mRNA在iHepSCs中的表达水平明显高于MEFs。gydF4y2BaFgydF4y2Ba,gydF4y2BaGgydF4y2BaWestern blot (gydF4y2BaFgydF4y2Ba)及免疫细胞荧光染色(gydF4y2BaGgydF4y2Ba)检测显示,Tet1在iHepSCs中的表达明显高于mef。数据显示为均值±SEM, n = 3,学生的t检验,* * * P < 0.05, P < 0.01, * * * P < 0.001gydF4y2Ba
Tet1在iHepSCs中保持高水平的5hmCgydF4y2Ba
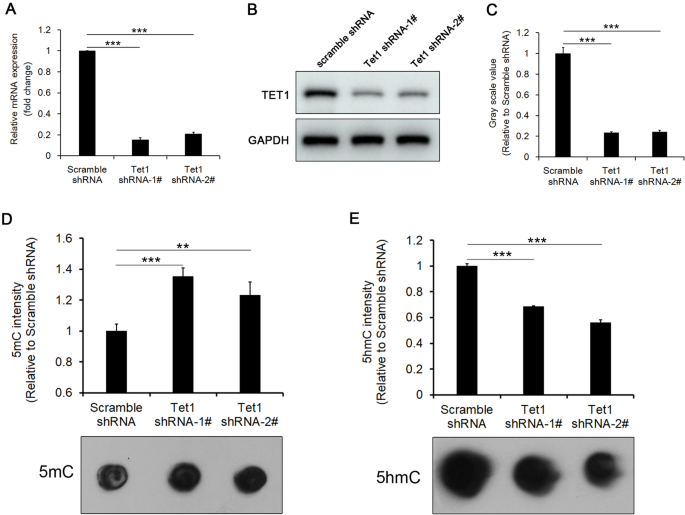
为了说明Tet1在iHepSCs中参与维持高水平的5hmC,首先,通过shRNA下调Tet1在iHepSCs中的表达(图2)。gydF4y2Ba2gydF4y2Baa - c)。在iHepSCs中抑制Tet1表达后,基因组5mC水平上调(图2)。gydF4y2Ba2gydF4y2BaD),而基因组5hmC水平下调(图;gydF4y2Ba2gydF4y2BaE)。此外,我们发现病毒感染后第三代(P3)和第十代(P10) Tet1的表达水平没有显著差异(附加文件gydF4y2Ba1gydF4y2Ba:图S1A-C),表明慢病毒介导的shRNA可以在iHepSCs中长时间维持Tet1的低表达。同时,斑点杂交结果也显示P3和p10 Tet1-KD iHepSCs的基因组5hmC整体水平无显著差异(附加文件gydF4y2Ba1gydF4y2Ba:图S1D, E)。gydF4y2Ba
Tet1在iHepSCs中保持高水平的5hmC。gydF4y2Ba一个gydF4y2BaqRT-PCR结果显示,shRNA抑制了Tet1 mRNA的表达。gydF4y2BaBgydF4y2BaWestern blot检测Tet1蛋白的表达。gydF4y2BaCgydF4y2Ba印迹相对强度的量化(gydF4y2BaBgydF4y2Ba)显示Tet1蛋白表达明显下调。gydF4y2BaDgydF4y2Ba斑点杂交结果显示,Tet1表达下调时,基因组5mC水平上调。每个样品装载500ng基因组DNA。gydF4y2BaEgydF4y2Ba斑点杂交结果表明,当Tet1的表达被抑制时,基因组5hmC的水平被下调。每个样品装载500ng基因组DNA。数据以均数±SEM表示,n = 3, Student 's t检验,**P < 0.01, ***P < 0.001gydF4y2Ba
Tet1维持iHepSCs的自我更新能力gydF4y2Ba
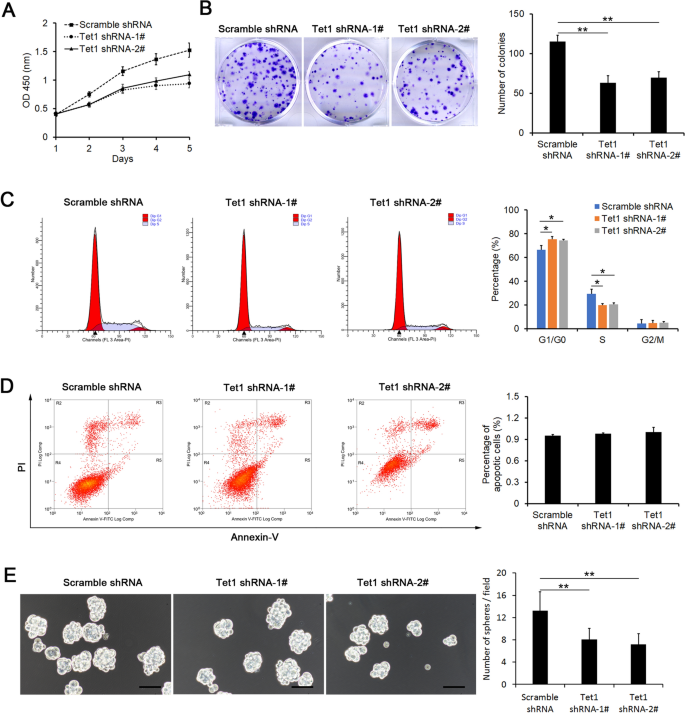
我们进一步检测了Tet1对iHepSCs表征的影响。在iHepSCs中shRNA下调Tet1表达后,细胞增殖和集落形成能力明显降低(图2)。gydF4y2Ba3.gydF4y2BaA, B)。细胞周期和凋亡实验显示,抑制Tet1表达可促进iHepSCs G1/G0期阻滞(图2)。gydF4y2Ba3.gydF4y2BaC),而凋亡细胞的百分比没有明显变化(图。gydF4y2Ba3.gydF4y2BaD).此外,在体外单细胞水平上,球形实验已被广泛用于评价干细胞的自我更新和分化[gydF4y2Ba27gydF4y2Ba].在本实验中,细胞形成球的实验结果显示,tet1下调的iHepSCs形成球的速率明显低于对照iHepSCs(图2)。gydF4y2Ba3.gydF4y2BaE).同时,iHepSCs中Tet1表达下调不影响向肝细胞分化的潜力(附加文件gydF4y2Ba1gydF4y2Ba:图S2)和胆管细胞(附加文件gydF4y2Ba1gydF4y2Ba:图S3)。总之,这些结果表明抑制Tet1的表达可以抑制iHepSCs的自我更新能力。gydF4y2Ba
Tet1维持iHepSCs的自我更新。gydF4y2Ba一个gydF4y2BaCCK 8实验显示,shRNA下调Tet1表达后iHepSCs的增殖能力受到抑制。gydF4y2BaBgydF4y2BaiHepSCs-scramble和Tet1- kd集落形成的代表性图像(左),下调Tet1表达后iHepSCs的集落数量明显减少(右)。gydF4y2BaCgydF4y2BaG .的百分比的代表图(左)和统计图(右)gydF4y2Ba0gydF4y2Ba/ GgydF4y2Ba1gydF4y2BaS和GgydF4y2Ba2gydF4y2BaiHepSCs-scramble和Tet1-KD iHepSCs的M细胞。Tet1-KD iHepSCs的细胞周期表现出明显的G阻滞gydF4y2Ba0gydF4y2Ba/ GgydF4y2Ba1gydF4y2Ba相比iHepSCs-scramble。gydF4y2BaDgydF4y2BaiHepSCs-scramble和Tet1-KD iHepSCs凋亡的代表图(左)和统计图(右)。gydF4y2BaEgydF4y2BaiHepSCs-scramble和Tet1- kd球的代表图像(左),下调Tet1表达后iHepSCs中球的数量明显减少(右)。数据以均数±SEM表示,n = 3, Student 's t检验,*P < 0.05, **P < 0.01gydF4y2Ba
Myc有助于iHepSCs的自我更新gydF4y2Ba
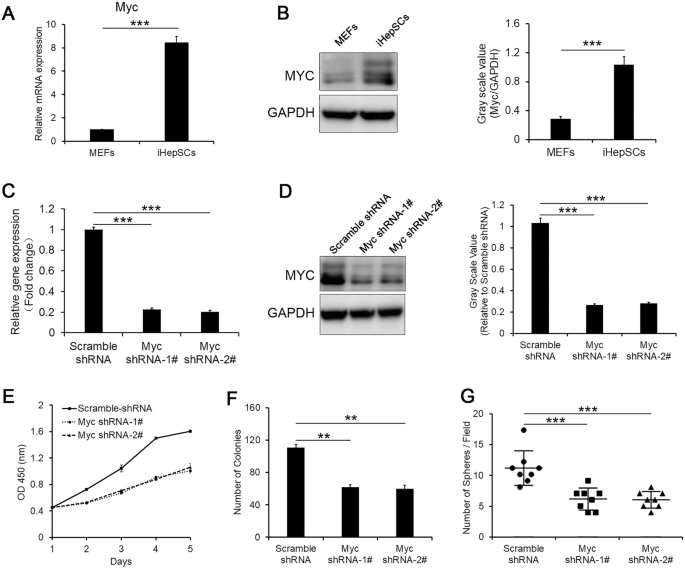
由于抑制Tet1表达可促进G0/G1期阻滞,因此Myc蛋白可促进细胞分裂,加速细胞从G0/G1期进入S期[gydF4y2Ba28gydF4y2Ba].我们推测Myc可能参与了iHepSCs的自我更新。首先,我们通过qRT-PCR和western blotting比较了Myc在iHepSCs和MEFs中的表达水平,结果显示Myc在iHepSCs中的表达水平明显高于MEFs(图。gydF4y2Ba4gydF4y2Ba为了进一步阐明Myc是否参与iHepSCs的自我更新,我们建立了Myc通过shRNA敲低iHepSCs的机制(图2)。gydF4y2Ba4gydF4y2BaC, D),观察细胞增殖(图;gydF4y2Ba4gydF4y2BaE),菌落形成(图;gydF4y2Ba4gydF4y2BaF)和细胞球的形成(图。gydF4y2Ba4gydF4y2BaG)由于Myc表达受到抑制,与对照iHepSCs相比均显著降低。gydF4y2Ba
Myc维持iHepSCs的自我更新。gydF4y2Ba一个gydF4y2Ba,gydF4y2BaBgydF4y2BaqRT-PCR检测Myc在iHepSCs中的表达高于MEFs (gydF4y2Ba一个gydF4y2Ba)和Western blotting (gydF4y2BaBgydF4y2Ba).gydF4y2BaCgydF4y2Ba,gydF4y2BaDgydF4y2Ba采用qRT-PCR检测Myc在iHepSCs中的表达及Myc敲除iHepSCs (gydF4y2BaCgydF4y2Ba)和Western blotting (gydF4y2BaDgydF4y2Ba).gydF4y2BaEgydF4y2BashRNA下调Myc表达后,iHepSCs的增殖能力受到抑制。gydF4y2BaFgydF4y2BashRNA敲除Myc表达后iHepSCs的菌落数量明显减少。gydF4y2BaGgydF4y2BashRNA敲除Myc表达后iHepSCs的球数减少。数据以均数±SEM表示,n = 3, Student 's t检验,**P < 0.01, ***P < 0.001gydF4y2Ba
Tet1在iHepSCs中通过靶向Myc维持自我更新gydF4y2Ba
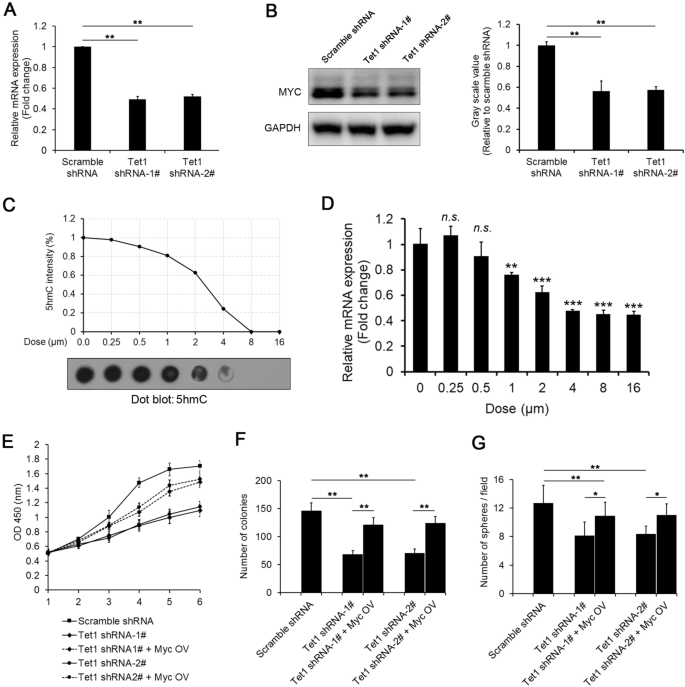
为了揭示Tet1与Myc在维持iHepSCs自我更新中的可能联系,我们研究了下调Tet1后Myc的表达水平。qRT-PCR和western blotting结果显示,shRNA抑制Tet1表达后,Myc的表达显著降低(图。gydF4y2Ba5gydF4y2BaA, B).接下来,我们使用了TET- in- c35,这是一种一流的TET抑制剂,可特异性阻断TET酶的催化活性[gydF4y2Ba29gydF4y2Ba],以治疗iHepSCs,结果显示基因组5hmC水平呈剂量依赖性下降(图;gydF4y2Ba5gydF4y2BaC).相应的,Myc的表达也减少。然而,当TET-IN-C35浓度超过4µM时,Myc的表达不再随着药物剂量的增加而降低(图。gydF4y2Ba5gydF4y2BaD).上述结果提示Tet1可能靶向Myc表达以维持iHepSCs的自我更新。此外,作为该假说的重要支持数据,慢病毒过表达Myc挽救了Tet1敲低iHepSCs中Myc的下调,并进一步恢复了增殖(图。gydF4y2Ba5gydF4y2BaE),菌落形成(图;gydF4y2Ba5gydF4y2BaF)和细胞球的形成(图。gydF4y2Ba5gydF4y2BaG)。总的来说,这些结果表明Tet1可以通过靶向Myc维持iHepSCs的自我更新。gydF4y2Ba
Tet1在iHepSCs中通过靶向Myc维持自我更新。gydF4y2Ba一个gydF4y2Ba,gydF4y2BaBgydF4y2Ba通过qRT-PCR检测,shRNA敲除Tet1表达后,Myc的表达降低(gydF4y2Ba一个gydF4y2Ba)和Western blotting (gydF4y2BaBgydF4y2Ba).gydF4y2BaCgydF4y2Ba采用斑点印迹法检测经指定剂量TET-IN-C35处理6 d后iHepSCs中5hmC的水平。每个样品装载500ng基因组DNA。gydF4y2BaDgydF4y2Ba采用qRT-PCR检测TET-IN-C35处理后iHepSCs中Myc的表达水平。gydF4y2BaEgydF4y2BaMyc的过表达恢复了Tet1-KD iHepSCs的增殖能力。gydF4y2BaFgydF4y2BaMyc的过表达增加了Tet1-KD iHepSCs的菌落数量。gydF4y2BaGgydF4y2BaMyc的过表达增加了Tet1-KD iHepSCs的球数。数据显示为均值±SEM, n = 3,学生t检验(gydF4y2Ba一个gydF4y2Ba,gydF4y2BaBgydF4y2Ba,gydF4y2BaFgydF4y2Ba而且gydF4y2BaGgydF4y2Ba)和邓尼特测验(gydF4y2BaDgydF4y2Ba), * p < 0.05, ** p < 0.01, *** p < 0.001,gydF4y2Ban。gydF4y2Ba,不显著gydF4y2Ba
Tet1通过直接结合Myc启动子的CBS-1和site A区域并去甲基化CpG胞嘧啶来调控Myc的表达gydF4y2Ba
众所周知,Myc的表达受到这些顺式元素的调控,包括CTCF结合位点(CBS-1和CBS-2)、CSL结合位点(site A)和TATA盒(图2)。gydF4y2Ba6gydF4y2Ba) (gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba].为了验证Tet1直接与启动子区域结合以调节Myc的表达,我们用抗Tet1抗体沉淀了基因组DNA片段。通过qRT-PCR定量了四种cis元素的含量,结果表明TET1更倾向于结合iHepSCs中的CBS-1和site A区域,而不是TATA box和CBS-2区域(图2)。gydF4y2Ba6gydF4y2BaB),然后用BSP检测CBS-1和site A区域胞嘧啶的甲基化,结果显示Tet1敲除iHepSCs中CBS-1区域和site A区域胞嘧啶的甲基化水平高于对照iHepSCs,但低于MEFs(图2)。gydF4y2Ba6gydF4y2BaC),提示在iHepSCs中,shRNA敲除Tet1表达后,CBS-1和site A区域的胞嘧啶可再次部分甲基化。甲基化敏感限制性内切核酸酶pcr (MSRE-PCR)的结果也显示,CBS1核心区域附近的CpG胞嘧啶中只有两个(Chr15:61,983,422-61,983,456)在mef中被甲基化,而在iHepSCs中未被甲基化gydF4y2Ba1gydF4y2Ba:图S4)。此外,anti-pol II ChIP-PCR结果显示,iHepSCs中A位点被更多的聚合酶II占据,这表明iHepSCs中Myc启动子具有更高的转录活性(图2)。gydF4y2Ba6gydF4y2BaD).此外,激活基因转录的H3K4me3的水平[gydF4y2Ba35gydF4y2Ba],在iHepSCs中A位点的表达也明显高于MEFs(图2)。gydF4y2Ba6gydF4y2BaE).而抑制转录的H3K27me3则无显著差异[gydF4y2Ba35gydF4y2Ba],在iHepSCs和mef之间(图;gydF4y2Ba6gydF4y2BaF)。gydF4y2Ba
Tet1通过直接结合Myc启动子的CBS-1和site A区域并去甲基化CpG胞嘧啶来调控Myc的表达。gydF4y2Ba一个gydF4y2Ba所识别的位置示意图gydF4y2Ba顺式gydF4y2Ba在小鼠15号染色体上参与Myc表达调控的元素。红色矩形代表CBS-1和CBS-2。蓝色箭头表示两个转录起始位点(P1, P2)。绿色矩形为a位点,蓝色双面箭头为Tet1-ChIP PCR引物组位点。gydF4y2BaBgydF4y2BaChIP-PCR结果显示,TET1富集于iHepSCs的CBS-1和site A区域,而非TATA box和CBS-2区域(n = 6, Student 's t检验,***P < 0.001)。gydF4y2BaCgydF4y2BaBSP检测mef、iHepSCs和Tet1敲除iHepSCs中CBS-1序列和A位点核心区CpG胞嘧啶甲基化示意图iHepSCs两个区域胞嘧啶甲基化水平均低于mef和tet1敲除的iHepSCs。圆圈代表CpG二核苷酸。填满的圆圈为甲基化胞嘧啶,空圆圈为未甲基化胞嘧啶。每个样品测序10个克隆。圆圈为深蓝色~ 100%阳性;圆形填充浅蓝色50-70%阳性,空圆形~≤30%阳性。gydF4y2BaDgydF4y2BaPol II ChIP-PCR结果显示,iHepSCs中A位点聚合酶II的富集程度高于MEFs。gydF4y2BaEgydF4y2BaChIP-PCR结果显示,iHepSCs中H3K4me3在A位点的富集程度高于mef (n = 6, Student’s t检验,**P < 0.01)。gydF4y2BaFgydF4y2BaChIP-PCR结果显示,H3K27me3在iHepSCs和mef中的富集程度无差异(n = 6, Student’s t检验)。gydF4y2Ba
CTCF和TET1共同调控Myc的表达gydF4y2Ba
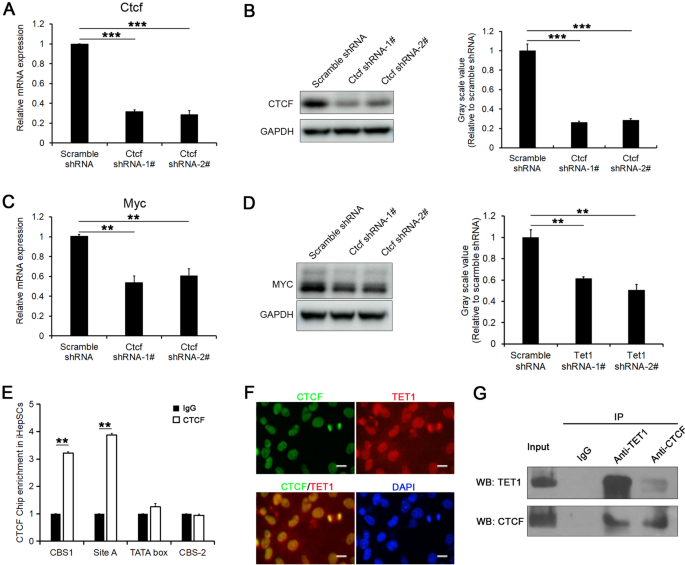
据报道,cctc - binning factor (CTCF)可介导遗传或表观遗传调节功能,包括启动子激活或抑制、基因沉默、绝缘和印迹[gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba].特别是,CTCF也被报道以细胞类型依赖的方式调节Myc作为启动子激活剂或抑制剂的表达[gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba39gydF4y2Ba].在这里,我们发现shRNA抑制CTCF在iHepSCs中的表达也降低了Myc的表达(图2)。gydF4y2Ba7gydF4y2BaA-D),提示CTCF可能促进Myc的表达。此外,我们通过anti-CTCF ChIP-PCR发现,CTCF与TET1一样,在iHepSCs中也优先与CBS-1和A位点区域直接结合,而不是与TATA盒区结合(图2)。gydF4y2Ba7gydF4y2BaE).为了进一步验证TET1和CTCF是否共同调节Myc的表达,我们进行了免疫荧光染色和免疫共沉淀(co-IP)实验。免疫荧光染色结果显示,TET1和CTCF在细胞核中共定位(图2)。gydF4y2Ba7gydF4y2BaF). co-IP的结果也表明TET1和CTCF可以形成复合物共同调节Myc的表达(图。gydF4y2Ba7gydF4y2BaG)。gydF4y2Ba
CTCF和Tet1共同调控Myc的表达。gydF4y2Ba一个gydF4y2Ba,gydF4y2BaBgydF4y2BaqRT-PCR检测到shRNA显著抑制了iHepSCs中CTCF的表达(gydF4y2Ba一个gydF4y2Ba)及Western blot (gydF4y2BaBgydF4y2Ba) (n = 3, Student 's t检验,***P < 0.001)。gydF4y2BaCgydF4y2Ba,gydF4y2BaDgydF4y2Ba通过qRT-PCR检测,CTCF-KD iHepSCs中Myc的表达降低(gydF4y2BaCgydF4y2Ba)和Western blotting (gydF4y2BaDgydF4y2Ba) (n = 3,学生t检验,**P < 0.01)。gydF4y2BaEgydF4y2BaChIP-PCR结果显示,CTCF在iHepSCs的CBS-1和site A区域富集,而在TATA box和CBS-2区域富集较少(n = 6, Student 's t检验,**P < 0.01)。gydF4y2BaFgydF4y2Ba抗CTCF抗体和抗TET1抗体的免疫细胞荧光染色显示CTCF蛋白和TET1蛋白在细胞核内共定位。gydF4y2BaGgydF4y2BaCo-IP实验表明,抗tet1抗体和抗ctcf抗体可以分别从iHepSCs核提取物中拉低CTCF1和TET蛋白gydF4y2Ba
讨论gydF4y2Ba
以往的研究表明,TET蛋白的主动去甲基化对于消除原有的DNA甲基化遗传,在体细胞重编程和谱系重编程过程中建立和维持新的DNA甲基化模式,以及维持细胞的新身份,包括干细胞的自我更新是不可或缺的[gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba].在本研究中,我们发现与mef相比,Tet1在iHepSCs中的表达和活性显著增加,细胞DNA甲基化水平下降,羟甲基化水平升高,这表明iHepSCs通过mef的重编程重新建立了新的甲基化模式。同时,目前的研究结果阐明了Tet1在维持iHepSCs自我更新中起着重要作用,因为下调Tet1会导致体外增殖和成球能力明显降低。gydF4y2Ba
为了阐明Tet1如何参与iHepSCs的自我更新维持,我们重点研究了维持iPSC自我更新的关键基因Myc对稳定iHepSCs身份的贡献。一般认为TET1蛋白可以以依赖脱甲基酶或不依赖脱甲基酶的方式完成转录控制活动,更多的是作为一种以环境依赖方式激活和抑制因子的一般促进者/招募者[gydF4y2Ba6gydF4y2Ba].一些研究报道了TET1参与胚胎干细胞的自我更新维持[gydF4y2Ba13gydF4y2Ba,gydF4y2Ba19gydF4y2Ba]和成体干细胞[gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba42gydF4y2Ba].例如,TET1和TET2被证明在成年小鼠大脑中神经干细胞(NSCs)的增殖中发挥重要作用,具体调节至少16个常见基因,其中Myc参与DNA复制和细胞周期[gydF4y2Ba20.gydF4y2Ba].然而,TET1/2调控Myc表达的确切机制仍有待研究。gydF4y2Ba
CTCF作为锌指转录因子,首次被鉴定为鸡Myc基因的转录抑制因子。然而,后来的研究发现CTCF通过多种机制参与Myc的转录调控,具有细胞类型特异性和细胞生理特异性机制[gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba43gydF4y2Ba,gydF4y2Ba44gydF4y2Ba].我们目前的研究表明,Tet1和CTCF之间的相互作用有助于通过特定的顺式元件“A位点”控制Myc的表达。A位点是Ensemble数据库中标记的启动子区保守的CSL结合位点,已被证明是Notch/Cdf1复合物的结合位点,直接调控淋巴母细胞白血病/淋巴瘤和乳腺肿瘤发生中的Myc表达[gydF4y2Ba45gydF4y2Ba,gydF4y2Ba46gydF4y2Ba,gydF4y2Ba47gydF4y2Ba].我们的结果为A位点参与Myc表达的控制提供了有力的证据。众所周知,真核生物Pol II酶转录所有蛋白质编码基因和非编码调控rna(例如,snRNA和microRNA) [gydF4y2Ba34gydF4y2Ba].在启动子上形成一个起始前复合物,由Pol II和几个一般的转录因子组成,启动转录。我们的ChIP-qPCR结果显示,Pol II和H3K4me3在A位点序列中富集,提示A位点可能是iHepSCs Myc启动子的转录激活位点,这与近期的一些报道一致[gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba45gydF4y2Ba].gydF4y2Ba
此外,我们的结果通过colocation assay和co-IP assay为Tet1和CTCF之间的相互作用提供了积极的证据,这与最近的报道一致[gydF4y2Ba48gydF4y2Ba,gydF4y2Ba49gydF4y2Ba].有趣的是,尽管在CBS-1的核心区域与A位点之间有大约400bp的距离,但在iHepSCs中,ChIP-PCR检测可以检测到TET1在这两个位点的富集,CTCF也是如此,这可能为TET1与CTCF之间的相互作用提供了额外的证据。此外,这种相互作用可能促进TET1与site A的结合,CTCF与CBS1的结合,以及Myc的转录。一方面,结合之前关于CTCF作为边界绝缘体的功能,将活性染色质与非活性染色质分开,以维持Myc转录的激活[gydF4y2Ba33gydF4y2Ba],我们推测TET1与CTCF之间的相互作用可能通过促进或稳定CTCF与CBS1区域的结合以及由于TET1氧化酶活性导致CpG胞嘧啶的去甲基化来提高其作为绝缘体的功能。另一方面,Tet1还促进了CpG胞嘧啶在A位点的去甲基化,这可能促进了其他转录因子在A位点的募集和Myc转录的启动。这些假设在未来需要更多的研究数据来支持。gydF4y2Ba
结论gydF4y2Ba
在这里,我们证明了iHepSCs的自我更新是通过Myc的高表达来维持的,这是由Tet1调控的,Tet1直接结合到CBS-1和Myc启动子的A位点区域,并使CpG胞嘧啶去甲基化。我们还证实CTCF与Myc启动子的CBS-1和site A区域结合,并与Tet1一起调控Myc的表达。这项研究可能为干细胞的自我更新提供新的见解,可以促进“重编程”干细胞的研究和应用。gydF4y2Ba
数据和材料的可用性gydF4y2Ba
在这项研究中产生或分析的所有数据都包含在这篇发表的文章及其附加文件中。gydF4y2Ba
缩写gydF4y2Ba
- Myc:gydF4y2Ba
-
Myelocytomatosis致癌基因gydF4y2Ba
- Tet1:gydF4y2Ba
-
二甲基胞嘧啶双加氧酶gydF4y2Ba
- CTCF:gydF4y2Ba
-
CCCTC-binding因素gydF4y2Ba
- iHepSCs:gydF4y2Ba
-
诱导肝干细胞gydF4y2Ba
- mef:gydF4y2Ba
-
小鼠胚胎成纤维细胞gydF4y2Ba
- 主持人:5gydF4y2Ba
-
5-MethylcytosinegydF4y2Ba
- 5 hmc:gydF4y2Ba
-
5-HydroxymethylcytosinegydF4y2Ba
- 芯片:gydF4y2Ba
-
染色质免疫沉淀反应gydF4y2Ba
- BSP:gydF4y2Ba
-
亚硫酸氢盐序列PCRgydF4y2Ba
- MSRE-PCR:gydF4y2Ba
-
甲基化敏感限制性内切酶pcrgydF4y2Ba
参考文献gydF4y2Ba
Horisawa K,铃木A.体细胞的直接细胞命运转换:朝向再生医学和工业。中国生物医学工程学报,2015;26(4):531 - 531。gydF4y2Ba
于波,何志智,游鹏,韩庆文,向东,陈峰,等。通过确定的因素将成纤维细胞重新编程为双潜能肝干细胞。细胞干细胞。2013;13(3):328-40。gydF4y2Ba
于波,李华,陈杰,何忠,孙华,杨刚,等。广泛扩张的小鼠诱导的肝干细胞保持高效的肝分化潜力,用于损伤肝脏的再生。肝脏杂志2020;40(9):2293-304。gydF4y2Ba
何s,中田D,莫里森SJ。干细胞自我更新的机制。细胞发育生物学,2009;25:37 - 406。gydF4y2Ba
李颖,陈霞,陆超。DNA与组蛋白甲基化的相互作用:分子机制与疾病意义。EMBO代表2021;22(5):e51803。gydF4y2Ba
王志强,王志强,王志强,等。TET酶的研究进展。生物化学学报,2019;47(3):875-85。gydF4y2Ba
何玉峰,李伯泽,李震,刘鹏,王勇,唐强,等。哺乳动物DNA中tet介导的5-羧基胞嘧啶的形成及其TDG的切除。科学。2011;333(6047):1303 - 7。gydF4y2Ba
伊藤生,沈亮,戴强,吴sc, Collins LB, Swenberg JA,等。Tet蛋白能将5-甲基胞嘧啶转化为5-甲酰胞嘧啶和5-羧基胞嘧啶。科学。2011;333(6047):1300 - 3。gydF4y2Ba
陈志强,陈志强,陈志强,陈志强,等。MLL伙伴TET1将哺乳动物DNA中的5-甲基胞嘧啶转化为5-羟甲基胞嘧啶。科学。2009;324(5929):930 - 5。gydF4y2Ba
胡霞,陈勇,赵志军。第17章TET家族蛋白的结构、调控与功能。进:Huang S, Litt MD, Blakey CA,编辑。表观遗传基因表达与调控。伦敦:学术出版社;2015.p . 379 - 95。gydF4y2Ba
吴华,D 'Alessio AC, Ito S,夏凯,王忠,崔凯,等。Tet1在小鼠胚胎干细胞转录调节中的双重功能。自然。2011;473(7347):389 - 93。gydF4y2Ba
Amouroux R, Nashun B, Shirane K, Nakagawa S, Hill PW, D 'Souza Z,等。从头DNA甲基化驱动小鼠受精卵中5hmC的积累。细胞生物学杂志,2016;18(2):225-33。gydF4y2Ba
杜丽丽,李志强,李志强,等。Tet酶的缺失会影响胚胎干细胞的正常分化。生物工程学报。2014;29(1):102-11。gydF4y2Ba
顾婷婷,郭峰,杨辉,吴惠鹏,徐广发,刘伟,等。Tet3 DNA双加氧酶在卵母细胞表观遗传重编程中的作用。大自然。2011;477(7366):606 - 10。gydF4y2Ba
李志强,李志强,李志强,等。Tet1和Tet2调节小鼠胚胎干细胞中5-羟甲基胞嘧啶的产生和细胞系的规范。细胞干细胞,2011;8(2):200-13。gydF4y2Ba
Khoueiry R, Sohni A, Thienpont B, Luo X, Velde JV, Bartoccetti M,等。TET1在小鼠胚胎移植后的谱系特异性功能。中国科学,2017;49(7):1061-72。gydF4y2Ba
高原,陈杰,李凯,吴涛,黄斌,刘伟,等。在iPSC诱导过程中,Tet1取代Oct4揭示了DNA甲基化和羟甲基化在重编程中的重要作用。细胞干细胞。2013;12(4):453-69。gydF4y2Ba
Cimmino L, Abdel-Wahab O, Levine RL, Aifantis I. TET家族蛋白及其在干细胞分化和转化中的作用。细胞干细胞,2011;9(3):193-204。gydF4y2Ba
Freudenberg JM, Ghosh S, Lackford BL, Yellaboina S,郑霞,李锐,等。tet1依赖的5-羟甲基胞嘧啶水平的急性缺失会损害LIF/Stat3信号通路,并导致胚胎干细胞身份的丧失。中国生物医学工程学报,2012;40(8):3364-77。gydF4y2Ba
Shimozaki K. Ten-eleven易位1和2为培养的成人神经干细胞增殖提供了重叠的转录程序。中国生物医学杂志,2017;37(6):995-1008。gydF4y2Ba
杨锐,于涛,寇旭,高旭,陈超,刘东,等。Tet1和Tet2通过P2rX7启动子的去甲基化维持间充质干细胞稳态。地理学报。2018;9(1):2143。gydF4y2Ba
王晓燕,王晓燕,王晓燕,等。短发夹rna的慢病毒表达研究。中国医药杂志,2009;29(9):732-45。gydF4y2Ba
李霞,陈伟,黄玲,朱明,张宏,司勇,等。青藤碱通过下调WNT10B抑制Wnt信号通路抑制乳腺癌干细胞干性。Pharmacol Res. 2022;179:106222。gydF4y2Ba
Kumaki Y, Oda M, Okano M. QUMA:甲基化分析的定量工具。核酸决议2008;36(Web服务器问题):W170-5。gydF4y2Ba
葛瑞芝LM, Mikkola HK。DNA甲基化是干细胞自我更新和多能性的守护者。植物学报。2009;41(11):1164-6。gydF4y2Ba
郭菊,苏勇,钟超,明葛亮,宋华。TET1诱导5-甲基胞嘧啶羟基化促进成人大脑DNA去甲基化。细胞。2011;145(3):423 - 34。gydF4y2Ba
Pastrana E, Silva-Vargas V, Doetsch F.睁大眼睛:球体形成作为干细胞检测的重要回顾。细胞干细胞。2011;8(5):486-98。gydF4y2Ba
赛斯A,古普塔S,戴维斯RJ。c-Myc转录激活域的细胞周期调控。分子生物学杂志,1993;13(7):4125-36。gydF4y2Ba
辛格阿克,赵波,刘霞,王霞,李红,秦红,吴霞,马勇,Horne D,余旭。TET催化结构域选择性靶向促进体细胞重编程。美国国家科学研究院。2020年,117(7):3621 - 6。gydF4y2Ba
彭文祥,何仁泽,张震,杨林,莫悠悠。LINC00346通过ctcf介导的Myc转录促进胰腺癌进展。致癌基因。2019;38(41):6770 - 80。gydF4y2Ba
切尔努钦I,沙姆苏丁S,姜世永,Bergström R,权耀文,余伟,等。CTCF与RNA聚合酶II的最大亚基相互作用并招募到CTCF全基因组靶点。分子生物学杂志,2007;27(5):1631-48。gydF4y2Ba
Gombert WM, Farris SD, Rubio ED, Morey-Rosler KM, Schubach WH, Krumm A. c-myc绝缘体元件和基质附着区域定义了c-myc染色体结构域。分子生物学杂志,2003;23(24):9338-48。gydF4y2Ba
陈红,田勇,舒伟,博新,王松。人类基因组中ctcf结合位点的综合鉴定与注释。PLoS ONE。7 (7): 2012; e41374。gydF4y2Ba
Schuijers J, Manteiga JC, Weintraub AS, Day DS, Zamudio AV, Hnisz D,等。MYC的转录异常揭示了共同的增强子-对接机制。Cell Rep. 2018;23(2): 349-60。gydF4y2Ba
李峰,万敏,张波,彭勇,周勇,皮成,等。二价组蛋白的修饰与发展。中国生物医学工程学报,2018;13(2):83-90。gydF4y2Ba
王华,毛拉诺,曲华,柯瓦雷,格茨J,保利F,等。CTCF占用的广泛可塑性与DNA甲基化有关。基因组学报,2012;22(9):1680-8。gydF4y2Ba
金秀玲,刘永平,姜波凯。CTCF在基因表达和基因调控中的作用。中华药物学杂志,2015;29(6):344 - 344。gydF4y2Ba
Ohlsson R, Renkawitz R, Lobanenkov V. CTCF是一种与表观遗传学和疾病相关的独特的多功能转录调控因子。植物学报,2001;17(9):520-7。gydF4y2Ba
Hyle J,张勇,Wright S,徐波,邵勇,Easton J,等。CTCF的急性损耗通过增强子-启动子环的丢失直接影响MYC的调节。中国生物医学工程学报,2019;47(13):6699-713。gydF4y2Ba
杨娟,Bashkenova N,臧瑞,黄霞,王娟。TET家族蛋白在发育和干细胞中的作用。发展。2020;147 (2):dev183129。gydF4y2Ba
潘志伟,陈志伟,陈志伟,等。DNA甲基化在细胞命运决定中的作用。科学通报。2021;22(1):59-66。gydF4y2Ba
金桂芳,孙志强,李志强,等。TET1/2蛋白在滋养层干细胞细胞周期进程中的关键作用。中国医学杂志,2018;10(4):1355-68。gydF4y2Ba
人C-MYC基因中多个CTCF结合元件的靶向缺失揭示了CTCF在C-MYC表达中的必要性。PLoS ONE。4 (7): 2009; e6109。gydF4y2Ba
霍龙,魏伟,吴松,赵旭,赵晨,赵华,等。双氢青蒿素联合靶向Notch1的siRNA对t细胞淋巴瘤Notch1/c-Myc信号通路的影响中华兽医杂志,2018;15(3):3059-65。gydF4y2Ba
夏尔玛VM, Calvo JA, Draheim KM, Cunningham LA, Hermance N, Beverly L,等。Notch1通过直接诱导c-myc的表达参与小鼠t细胞白血病。分子生物学杂志,2006;26(21):8022-31。gydF4y2Ba
翁ap, Millholland JM, Yashiro-Ohtani Y, Arcangeli ML, Lau A, Wai C,等。C - myc是Notch1在t细胞急性淋巴母细胞白血病/淋巴瘤中的重要直接靶点。基因开发,2006;20(15):2096-109。gydF4y2Ba
Klinakis A, Szabolcs M, Politi K, Kiaris H, Artavanis-Tsakonas S, Efstratiadis A. Myc是Notch1的转录靶点,也是Notch1诱导小鼠乳腺肿瘤发生的必要条件。美国国家科学研究院。103(24): 9262 - 2006; 7。gydF4y2Ba
Dubois-Chevalier J, Oger F, Dehondt H, Firmin FF, Gheeraert C, Staels B,等。动态CTCF染色质结合景观促进DNA羟甲基化和脂肪细胞分化的转录诱导。中国生物医学工程学报,2014;42(17):10943-59。gydF4y2Ba
Nanan KK, Sturgill DM, Prigge MF, Thenoz M, Dillman AA, Mandler MD,等。tet催化的5-羧基胞嘧啶促进CTCF与全基因组次优序列的结合。iScience。2019; 19:326-39。gydF4y2Ba
确认gydF4y2Ba
我们要感谢这项研究的参与者。gydF4y2Ba
资金gydF4y2Ba
本研究得到国家自然科学基金项目(32171387,82173369,31771511,31471284)和国家技术领域基金强化计划项目(2019-JCJQ-JJ-068)的资助。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
概念化:CW, XY, HZ;方法:CW、XY、SD、YL;形式分析和调查:CW, HZ和JF;写作-初稿准备:CW, XY, YL;编审:BY、HZ;资金获取:BY、HZ;资源:BY和HZ;监督:赫兹。所有作者都阅读并批准了最终的手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
伦理批准并同意参与gydF4y2Ba
不适用。gydF4y2Ba
发表同意书gydF4y2Ba
不适用。gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者宣称他们没有利益冲突。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
补充信息gydF4y2Ba
附加文件1:图S1。gydF4y2Ba
shRNA在iHepSCs中长期保持Tet1和DNA甲基化模式的低表达。gydF4y2Ba图S2。gydF4y2BaTet1表达下调对iHepSCs的肝分化无影响。gydF4y2Ba图S3。gydF4y2BaTet1表达下调不影响iHepSCs的胆管细胞分化。gydF4y2Ba图S4。gydF4y2Ba采用MSRE-PCR分析CBS-1区胞嘧啶甲基化状态。gydF4y2Ba表S1。gydF4y2Bashrna的目标序列。gydF4y2Ba表S2。gydF4y2BaqRT-PCR引物。gydF4y2Ba表S3。gydF4y2BaMyc基因顺式元件在第15染色体和ChIP引物组的确切位置。gydF4y2Ba表S4gydF4y2Ba.用于IF和Western blot检测的一抗和二抗。gydF4y2Ba表S5。gydF4y2Ba扩增CBS1和Site A区域BSP的引物。gydF4y2Ba表S6。gydF4y2BaMSRE检测引物组。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.创作共用公共领域奉献弃权书(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本条所提供的资料,除非在资料的信用额度中另有说明。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
王,C, Yu, X,丁,S。gydF4y2Baet al。gydF4y2Ba诱导的肝干细胞通过TET1和CTCF共同调节Myc的高表达来维持自我更新。gydF4y2Ba细胞BioscigydF4y2Ba12gydF4y2Ba143(2022)。https://doi.org/10.1186/s13578-022-00883-7gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s13578-022-00883-7gydF4y2Ba
关键字gydF4y2Ba
- Tet1gydF4y2Ba
- CtcfgydF4y2Ba
- MycgydF4y2Ba
- 诱导肝干细胞gydF4y2Ba
- 自我更新gydF4y2Ba