摘要
光敏色素B-4 #3的抑制因子(SUPPRESSOR OF PHYTOCHROME B-4 #3, SOB3)是AT-HOOK MOTIF CONTAINING NUCLEAR localization (AHL)家族的转录因子,参与光介导的植物生长拟南芥,影响下胚轴伸长等过程。大多数关于ahl的研究都是在连续光下进行的。然而,与持续光照条件相比,有一些独特的分子事件可以促进短日(SD)内的生长。因此,我们研究了AHLs如何影响SD下胚轴伸长。首先,我们观察到AHLs在SD中抑制下胚轴的生长,这与它们在恒定光照下的作用相似。接下来,我们通过对两个sd生长的幼苗进行RNA-seq鉴定了ahl调控基因sob3不同时间点的突变体。我们的转录组数据表明了这一点光敏色素相互作用因子(论坛s)4,5,7,8以及pif靶基因被SOB3和/或其他ahl抑制。我们还确定了PIF靶基因被抑制,以前没有被描述为ahl调节,包括Pre1 pil1 hfr1,CDF5,而且XTR7.有趣的是,我们的RNA-seq数据也表明,AHLs激活生长抑制因子的表达来控制下胚轴伸长,如HY5而且IAA17.值得注意的是,从RNA-seq实验中鉴定出的许多生长调节基因和其他基因在这两者之间被差异调节sob3测试时间点的变种人。令人惊讶的是,我们的ChIP-seq数据表明,SOB3在一天中主要与相似的基因结合。总的来说,这些数据表明AHLs以特定时间点的方式影响基因表达,而与整个SD中与DNA结合的变化无关。
简介
植物有一套复杂的信号通路,使它们能够解释和回应周围的环境。植物根据环境信号改变生长发育的一种机制是通过改变基因转录。参与这一过程的一组重要转录调控因子是AT-HOOK MOTIF NUCLEAR localization (AHL)家族的成员[1,2,3.].在拟南芥(拟南芥),AHL29/ SUPPRESSOR OF PHYTOCHROME B4-#3 (SOB3)具有调节生长发育的重要功能,以响应光,影响下胚轴生长、开花和衰老等过程[1,4,5,6,7,8,9,10].SOB3在长下胚轴表型抑制因子的激活标记筛选中被确定为下胚轴生长的抑制因子光敏色素B-4(phyB-4)白光下生长的感光突变体[1,11,12,13].进一步的研究表明,SOB3以光依赖的方式调节下胚轴的生长,特别是在昏暗的光线下影响生长[1].因此,SOB3-Dominant(SOB3-D)的幼苗,从上述活化标记实验中得到的SOB3与野生型Col-0植物相比,下胚轴较短[1,5].除SOB3外,其他ahl与该家族成员以半冗余的方式调节下胚轴生长[6].
的AHL基因家族的特征是两个对其功能重要的保守元件:一个植物和原核生物保守/未知功能域#296 (PPC/DUF296)和一个或两个AT-hook motif [6,14,15].SOB3通过PPC结构域与自身、其他ahl和非ahl转录因子相互作用[6].另一方面,AT-hook motif结合了富含腺嘌呤和胸腺嘧啶的DNA序列[4,6,16,17,18].值得注意的是,在AT-hook结构域的中心有一个错义突变SOB3在SOB3-D背景,称为sob3-6等位基因,产生极高的幼苗[1].这种表型归因于非功能性SOB3-6蛋白过表达的显性-负性效应,即SOB3-6蛋白可以像正常情况下那样参与蛋白-蛋白相互作用,但不能与DNA结合[6].这导致了不能抑制幼苗生长的非生产性复合物的形成。从这项研究中,我们发现AT-hook和PPC结构域对SOB3和其他ahl的功能都很重要。
AHLs在恒定光照和长日照条件下调节生长(LD;光照16小时/暗8小时),至少部分通过抑制生长素途径。在恒定光照下生长的幼苗中,SOB3直接与生长素生物合成基因的启动子结合YUCCA8(YUC8)和生长素信号基因属于小生长素上升RNA 19(SAUR19)亚家族并抑制其转录[5].然而,SAUR19的过表达只能部分恢复下胚轴短的表型SOB3-D这表明AHLs对生长的影响并不能完全通过其对生长素途径的影响来解释。在另一项使用在LD中生长的幼苗的研究中,Lee和Seo发现AHLs会抑制YUC9转录通过促进H2A的沉积。该基因启动子上的Z组蛋白变体[8].这可能会抑制生长素的生物合成,从而抑制老年幼苗的下胚轴生长。此外,另一项使用ld生长的植物的研究结果表明,AHLs主要通过作用于生长素途径来抑制叶柄生长[18].在这项研究中,发现AHLs通过拮抗促生长光敏色素相互作用因子(PIFs)来限制叶柄伸长。具体来说,AHLs抑制pif介导的基因激活,涉及激素介导的生长,如ACS8它参与乙烯的生物合成,NPY1,参与生长素运输,YUC8,SAUR24,从而解释了它们对叶柄生长的负面影响。
大多数关于AHLs的研究都是在持续的白光或LD中进行的。不仅持续的光不是自然生长环境的特征,而且可能有不同的机制调节短期内的生长(SD;8小时光照/16小时黑暗),与持续光照或LD相比[18].此外,尚不清楚ahl在一天中是否对基因表达有不同的影响。因此,为了研究这些问题,我们研究了AHLs是如何调节SD下胚轴伸长的,在这种情况下,我们对这个转录因子家族如何影响生长知之甚少。
结果
AHLs抑制SD下胚轴生长
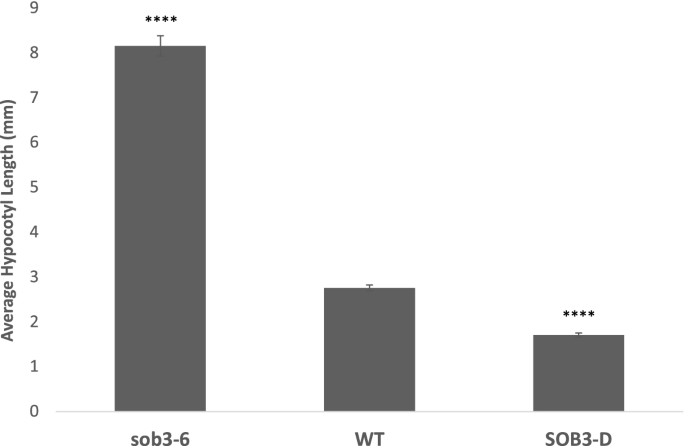
我们研究了AHLs如何在短昼(SD)(8小时光照/16小时黑暗)下调节下胚轴伸长,在这种情况下,我们对该转录因子家族如何影响生长知之甚少。突变体的表型分析SOB3透露,SOB3-D显示SD下胚轴生长减慢,而sob3-6幼苗非常高(图;1).这些表型表明SOB3和/或其他ahl抑制SD下胚轴的生长,这与已报道的在恒光或长时间下生长的幼苗相似[1,4,5,6,8,18].
AHLs在SD中以时间点特异性的方式调节许多基因的表达
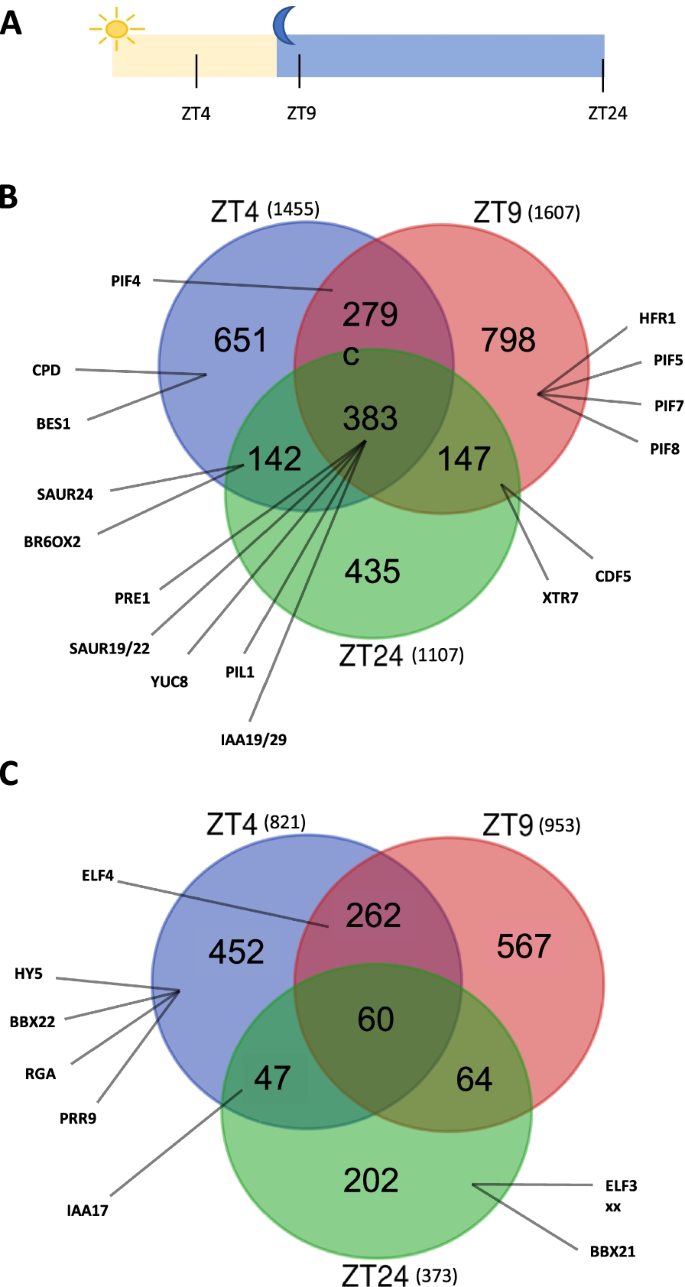
接下来,为了深入了解AHLs影响SD下胚轴生长的分子机制,我们确定了在两者之间差异表达的核编码基因SOB3-D而且sob3-6.具体来说,我们使用在三个不同时间点收获的幼苗进行RNA-seq:在ZT4(黎明后4小时)和ZT9(黄昏后1小时)收获的5天大的幼苗,以及在黎明收获的6天大的幼苗(ZT24)(图2)。2一个;表的年代1).ZT4、ZT9和ZT24的三个时间点是根据已知的下胚轴生长调控和PIF活性在SD中的具体选择的。在SD中,下胚轴的生长主要发生在夜间,此时高表达PIF4而且PIF5与最小的翻译后抑制相一致论坛S受光敏色素和其他抑制因子的影响[19,20.,21].因此,我们选择测试ZT24时间点,以研究AHLs是否可能通过结合和调节的表达来防止在这个时间点的过度生长论坛S和它们的靶基因。另一方面,我们选择了另外两个时间点,因为它们代表了白天(ZT4)或晚上(ZT9)中PIF活动和生长最小的时间。我们推断ahl可能在一个或两个时间点抑制生长,可能是通过抑制论坛转录和/或pif激活基因的表达。
我们RNA-seq数据的EdgeR分析SOB3-D而且sob3-6在ZT4、ZT9和ZT24分别发现了2276个、2560个和1480个差异表达基因。2B, C),表明SOB3和/或其他AHLs在白天中期和傍晚早期对基因表达的影响可能比在黎明时更大。在这些差异表达的基因中,发现有2,835个基因被AHLs抑制(图。2B) AHLs仅诱导1654例(图;2C).这表明AHLs在SD中可能更多地作为转录抑制因子而不是转录激活因子。此外,大多数基因只在一个时间点被错误调控。在ahl被抑制的基因中,651个在ZT4被抑制,798个在ZT9被抑制,435个在ZT24被抑制,分别占所有被抑制基因的23%,28%和15%(图)。2B).与ahl诱导的基因总体数量少于ahl抑制的基因一致,在所有时间点都有较少的唯一诱导基因:ZT4为452个,ZT9为567个,ZT24为202个(图2)。2C). ZT4和ZT9的唯一诱导基因分别占所有诱导基因的28%和26%,而ZT24仅占13%。有趣的是,在所有三个时间点都有相对较少的基因错误调控,特别是ahl诱导的基因。在诱导基因中,只有4%的基因在所有三个时间点都被错误调控,而在ahl抑制基因中,13%的基因在所有三个时间点都被错误调控(图。2B, C)。总的来说,这些数据表明,大多数ahl调控基因的表达只在一天中的特定时间受到这些转录因子的影响。
AHLs调节的表达论坛s及其靶基因控制SD下胚轴的生长
AHLs抑制生长的至少一种方式是通过减少pif激活基因的表达[18].因此,我们研究了RNA-seq数据中几个PIF靶基因的表达是如何受到影响的,重点研究了最近一篇综述中强调的PIF激活基因[21].事实上,我们的转录组数据表明,在sd生长的幼苗中,PIF靶基因一整天都被AHLs抑制(图2)。2B).一些pif激活基因在所有三个时间点都被鉴定为ahl抑制,如YUC8,IAA19,二阿富汗二月(图。2B),它们都是与生长素途径相关的促生长基因,这些基因之前也被确定为直接受ahl抑制的基因[5,18].值得注意的是,我们发现除了IAA19这是一种功能相似的pif靶基因IAA29[22,23,24]也在所有三个时间点被ahl抑制(图。2B) pif激活基因PIL1而且PRE1[25,26]也在所有三个时间点的ahl抑制基因中被发现。2B).值得注意的是,PRE1促进下胚轴生长,可能至少部分通过增强PIF蛋白活性[26,27,28,29,30.,31].其他PIF靶点仅在一两个时间点被ahl抑制。例如,促进生长的基因CDF5[32在ahl抑制的ZT9和ZT24基因中发现,但在ZT4基因中没有发现。2B).此外,一些基因之间的差异调节SOB3-D而且sob3-6在这三个时间点上,在不同时间点上的表达表现出不同的折叠变化,如SAUR22(ZT4处折叠变化最大),SAUR19(ZT9处褶皱变化最大),IAA19 (ZT24处褶皱变化最大),2).因此,AHLs似乎在一天中对一些pif调控基因的表达有不同的定量影响。
除了发现许多PIF靶标被AHLs抑制外,我们的数据还表明AHLs抑制的表达论坛在SD中。具体地说,PIF4是ahl在ZT4和ZT9的抑制基因之一,而PIF5,7,8只在ZT9被抑制(图;2B).这些数据表明AHLs调节的表达论坛在白天和傍晚的早些时候,但不要在深夜。值得注意的是,这与上一段所述的AHLs对PIF靶基因表达的影响相反,后者似乎持续了一整天。因此,AHLs可能通过降低pif激活基因的表达来抑制SD下胚轴的生长,这可能部分是AHLs抑制pif激活基因表达的下游结果论坛年代。
AHLs抑制油菜素类固醇生物合成基因,并激活生长抑制因子的表达,以调节SD下胚轴的生长
我们的RNA-seq数据还表明,AHLs可能抑制pif下游油菜素类固醇(BR)的生物合成以及BR信号。CPD而且BR6OX2,这两个pif4激活的基因都编码油菜素类固醇生物合成相关的酶[33,34],在ZT4的ahl抑制基因中发现BR6OX2在ZT24也属于这一组(图。2B)我们进一步发现喜欢CPD,BES1,该基因编码一个参与br介导的下胚轴生长的关键转录因子[35,36],在ZT4位点被AHLs特异性抑制(图。2B).综上所述,这些数据表明,在SD中,AHLs对下胚轴生长的抑制作用可能部分是由它们抑制BR通路引起的。
除了抑制SD中促进下胚轴生长的基因表达外,我们的RNA-seq数据还表明AHLs激活生长抑制因子的表达。具体来说,Favero等人强调了8种生长抑制因子.评论(21]是我们从RNA-seq实验中鉴定出的ahl诱导基因(图。2C).在这8个基因中,有6个被鉴定为仅在单一时间点上由ahl诱导的。四种生长抑制因子,包括HY5而且BBX22,是ahl在ZT4特异诱导的。值得注意的是,HY5和BBX22蛋白只有在光照下才不会受到cop1介导的降解的影响[37,38],因此ahl似乎被激活了HY5而且BBX22特别是当它们编码的蛋白质能够促进下胚轴生长抑制时,即在白天[39,40,41].BBX22的近亲,BBX21另一方面,它是我们发现的仅在ZT24上被ahl诱导的两个基因之一;然而,BBX21不太可能在夜晚结束时导致生长抑制,因为它像BBX22一样,在黑暗中以依赖cop1的方式大量降解[41,42].除了这六种生长抑制因子外,有两种被确定为在两个不同的时间点被ahl诱导:ELF4在ZT4和ZT9和IAA17在ZT4和ZT24。总的来说,我们的RNA-seq数据表明,AHLs不仅通过抑制pif激活的生长促进基因的表达,还通过增加生长抑制因子的表达,特别是在白天,来抑制SD下胚轴的生长。
SOB3结合的变化并不能解释SD中ZT4、ZT9和ZT24基因的差异调控
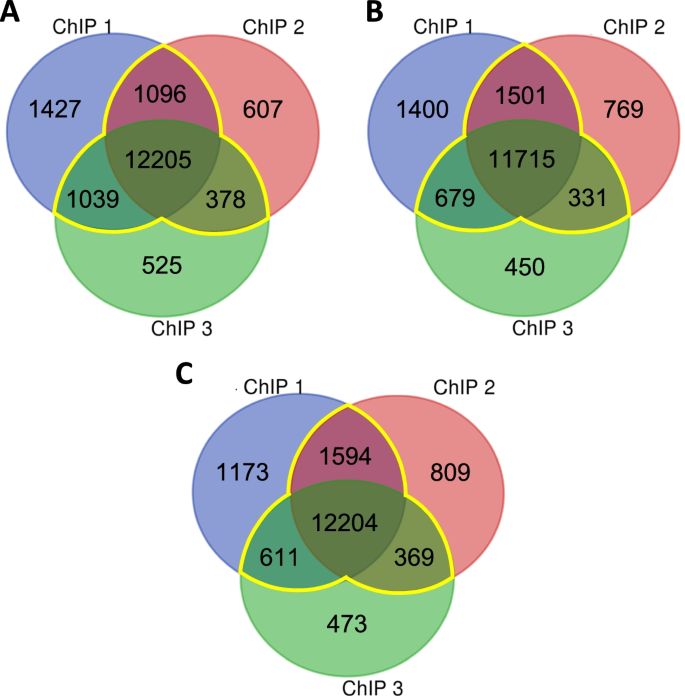
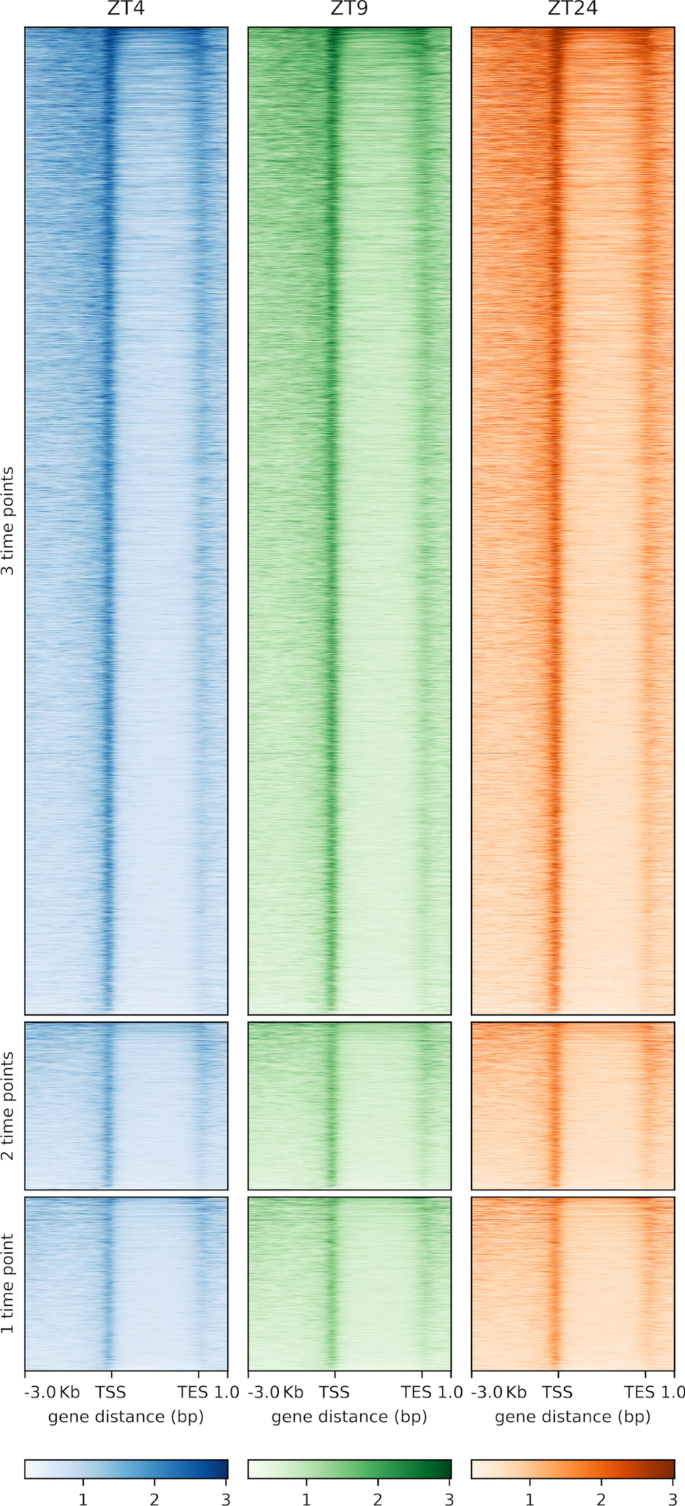
由于我们的RNA-seq数据显示AHLs在一天中的不同时间调节不同的基因集,我们接下来通过在RNA-seq相同的三个时间点执行ChIP-seq来研究SOB3与DNA的结合是否在SD中全天发生变化。2一个;表的年代3.).在给定时间点的三个重复中至少有两个被SOB3结合的基因被认为是后续分析的SOB3结合基因(图2)。3.(黄色轮廓线),结果显示ZT4基因14,718个,ZT9基因14,226个,ZT24基因14,778个。当这些基因相互比较时,我们发现大多数基因在所有三个时间点都与SOB3结合(图3)。4).有趣的是,有少数基因只在一个或两个时间点结合(图2)。4).这些观察结果表明,SOB3主要在这三个时间点与相似基因结合。
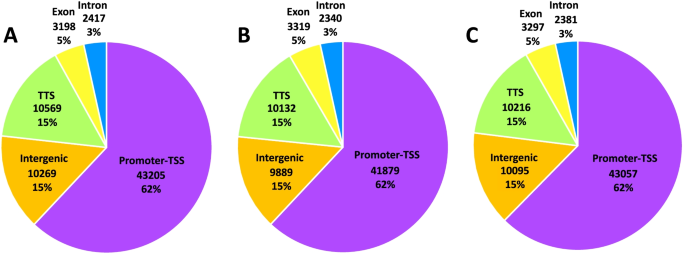
接下来,我们研究了SOB3结合位点的分布,以评估SOB3在基因组中的功能。我们观察到大多数结合位点都靠近转录起始位点(启动子- tss)(图。5).此外,SOB3在基因体内的结合事件较少,5%的结合事件发生在外显子,3%发生在内含子。Favero等人同样观察到SOB3主要结合位于基因体外的基因组区域,其中高比例的结合事件发生在启动子- tss区域[18].有趣的是,在这五个类别中,每个时间点都有相同的绑定事件百分比。在启动子- tss区域在所有三个时间点观察到的高比例结合事件进一步支持了先前的发现,即SOB3作为转录因子发挥作用[1,4,6,14,16,17,43,44,45,46,47,48,49,50,51],并进一步表明SOB3的这种作用在一天中保持一致。此外,当比较三个时间点的结合模式时,我们观察到SOB3在所有三个时间点的结合几乎相同(图2)。6),这表明这种转录因子在一天中很大程度上与相似的位点结合。因此,AHLs对ZT4、ZT9和ZT24基因表达的不同影响不太可能是通过与靶基因结合的改变来解释的。
与tcp和pif相关的基序在SOB3 ChIP-seq数据中高度丰富
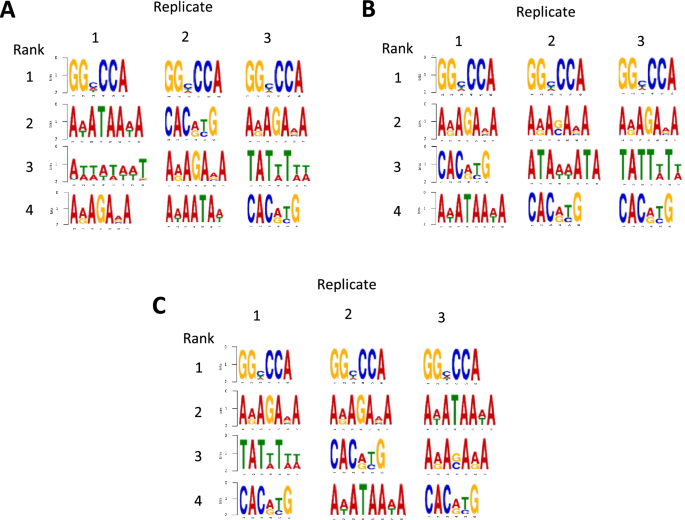
然后我们利用MEME-ChIP对ChIP-seq数据进行motif富集分析[52].该分析显示了类似于tcp结合序列的GGHCCA基序[53,54,55,56],在所有时间点的所有重复中都是最丰富的motif(图。7).在每个重复和每个时间点,该motif也比所有其他motif丰富得多,这表明在SD中ahl和tcp之间存在密切的关系。Favero等人最近的一篇论文研究了SOB3在长时间内对叶柄生长的影响,GGHCCA motif也被认为是最丰富的motif [18].正如他们在论文中所指出的那样,这个主题可能是这个数据集中最丰富的,因为tcp和ahl已经被证明具有物理相互作用[6,53,54,57].因此,该基序可能与SOB3蛋白- dna复合物中的tcp结合。我们的模因芯片分析也揭示了其他类型的基元在所有三个时间点上的富集(图。7).其中一个基序是carpg,它类似于由pif结合的序列[25,26,54,56,58,59,60].考虑到我们从RNA-seq数据中发现的几个pif激活基因的表达被ahl抑制,这是特别有趣的。因此,AHLs很可能直接结合到PIF靶标的启动子上,并调节其在SD中的表达,这似乎也发生在叶柄生长的背景下[18].各种at丰富的主题也在所有三个时间点排名很高。其中一些,包括AWATAAWA、AWAATAW、TATWTWW、ATAWWATA和TATTWTW,只含有腺嘌呤和胸腺嘧啶残基,并且与已在体外被证明可与其他ahl结合的motif相似,包括AHL12、AHL20和AHL25 [4,6,14,16,17,51,54,56].这些基序可能与SOB3本身和/或与SOB3复合物中发现的其他AHL家族成员结合。值得注意的是,ARAGAVA或ARASAVA motif也出现在所有时间点的所有重复中,并且可能与ahl结合,因为它们也富含at。然而,这种可能性似乎不太可能,因为从结构的角度来看,似乎需要四个连续的a /T碱基对来适应AT-hook的结合[61].
GO项富集显示AHLs在ZT4和ZT9直接诱导相关基因,但在ZT24不诱导
接下来,为了识别AHLs直接调控的基因,我们比较了RNA-seq和ChIP-seq结果。我们在ZT4位点鉴定出1042个被AHLs直接抑制的基因,在ZT9位点鉴定出1183个基因,在ZT24位点鉴定出771个基因。有趣的是,我们发现AHLs直接诱导的基因较少,在ZT4有595个,在ZT9有604个,在ZT24有301个(图4)。8).因此,AHLs在SD中可能同时扮演转录抑制因子和激活因子的角色。然而,ahl似乎也倾向于充当抑制因子,这与我们之前仅根据RNA-seq数据推断的结果一致。值得注意的是,AHLs作为转录抑制因子的这种偏向似乎在ZT24中特别高。比较各时间点sob3结合的ahl调控基因,我们发现其中64%在ZT4位点被抑制,66%在ZT9位点被抑制,72%在ZT24位点被抑制。8).
为了进一步研究基于时间点的AHLs直接调控基因的差异,我们比较了我们鉴定为AHLs直接靶点的基因在不同时间点的GO术语富集情况。在直接被ahl抑制的基因中,富集最多的GO项主要包括那些参与外部环境反应的基因,以及随后的内部反应(图2)。9).“对内源性刺激的反应”、“对有机物的反应”、“对刺激的反应”和“对激素刺激的反应”等术语在所有时间点上都是最受抑制的GO术语。然而,当ZT24与ZT4和ZT9进行比较时,有一些显著的差异。例如,“催化活性”在ZT24的直接抑制基因中是第二高的GO项,但在ZT4中要低得多,在ZT9中也不在前25名之内。还有其他有趣的时间点特定的发现,在GO方面的抑制基因,如“响应生长素刺激”,它在ZT4和ZT24高度富集,但在ZT9不富集。此外,“对油菜素类固醇刺激的反应”,“蛋白质磷酸化氨基酸结合”和“磷酸化蛋白结合在ZT4高度富集。相比之下,“光收获复合物”和“叶绿素结合”都在ZT9上高度富集。AHLs在ZT4和ZT9时间点直接诱导的基因中,排名靠前的是“叶绿体部分”、“质体部分”或“质体部分”和“叶绿体”,但在ZT24时间点,排名靠前的GO术语完全不同,包括“转录因子活性”、“转录调节活性”和“对激素刺激的反应”。此外,与ZT24位点直接诱导基因相关的与叶绿体功能无关的顶级功能,大多是此时点直接抑制靶标中也高度富集的功能。综合这些数据,在不同的时间点,ahl似乎有一些特定的过程被调节,但与ZT24相比,ZT4和ZT9有更多的相似之处。
SOB3与靶基因结合的变化并不能解释AHLs在ZT4、ZT9和ZT24基因调控的差异
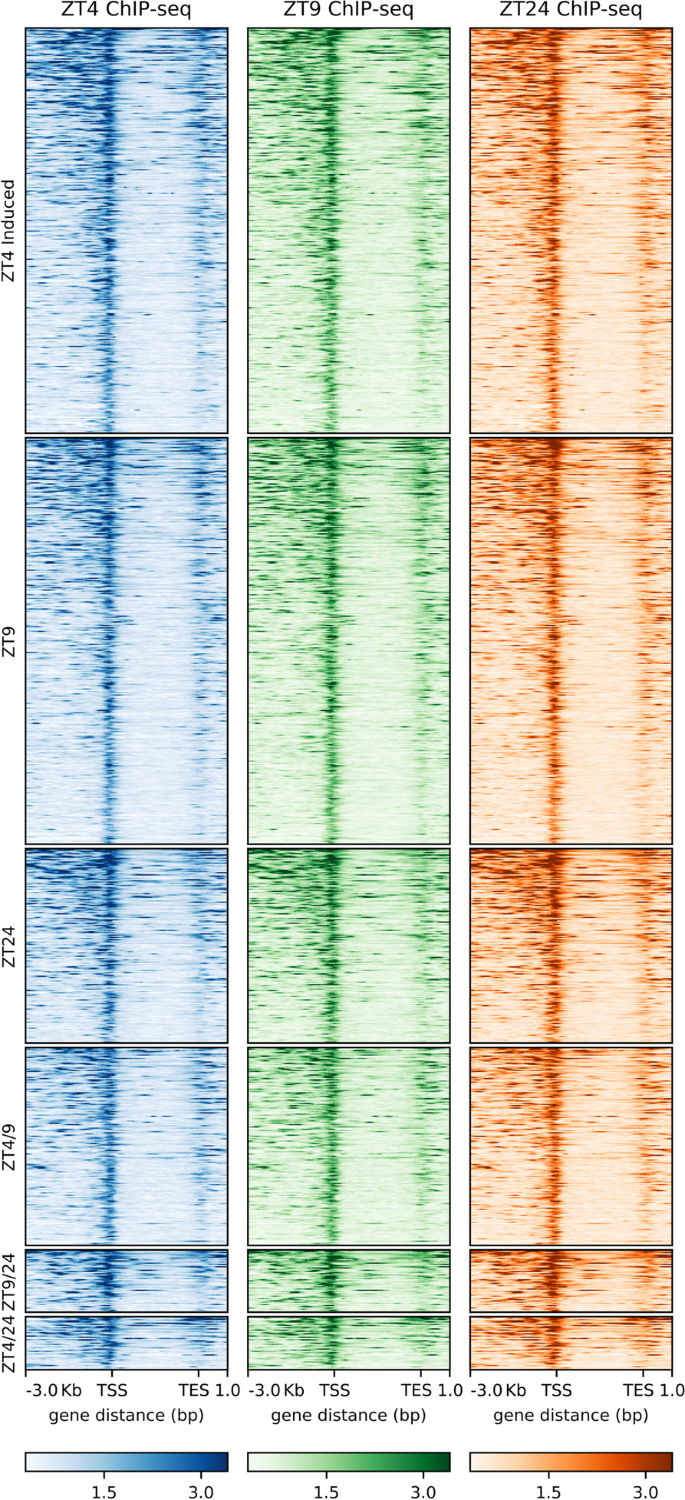
最后,我们回到SOB3在ZT4、ZT9和ZT24位点结合的差异是否可以解释AHLs对基因表达的时间点特异性影响的问题。首先,我们检查了从RNA-seq数据中识别出的ahl调控基因是否以及在哪个时间点识别出来,并在图中特别突出显示。2B和2C与SOB3结合。根据我们的ChIP-seq数据,我们发现几乎所有这些基因在所有三个时间点都与SOB3结合(表S4).重要的是,基因如CPD,BES1,HY5,BBX22在ZT4、ZT9和ZT24位点与SOB3结合,尽管它们只在三个时间点中的一个受到AHL的调控(图S1).这些数据表明AHLs对靶基因的时间点特异性调控并不是通过转录因子结合的差异来实现的。为了进一步验证这一假设,我们检查了SOB3特异性地与被鉴定为ahl抑制的基因的结合分布。10)或ahl诱导(图;11),只有一两个时间点。使用这种方法,我们再次观察到SOB3在所有三个时间点的相似绑定配置文件。最后,我们研究了目标基因上SOB3富集水平的变化是否可以解释AHLs对基因表达的时间点特异性效应(图S2和S3.).然而,我们没有观察到SOB3在三个时间点之间的富集变化,这可能解释了AHLs对靶基因表达的时间点特异性影响。这进一步表明,AHLs对基因表达的特定时间效应不是由这些转录因子与其靶基因结合的差异引起的。
讨论
AHLs在整个SD中调节生长以及特定基因的表达
SOB3和其他ahl已被证明参与连续白光和LD下光介导的下胚轴发育[1,4,5,6,8,10].首先,我们在这里证明了SOB3也能够影响SD下胚轴的生长(图2)。1),与先前报道的AHL22相同[4].接下来,我们在三个不同的时间点(ZT4, ZT9和ZT24)进行RNA-seq,以确定可能参与下胚轴生长和发育的错误调控基因(图2)。2).通过测试整个SD的三个不同时间点,我们希望发现仅测试一个时间点就可能忽略的基因调控错误。事实上,我们的数据表明,AHLs在白天中期和傍晚早期对基因表达的影响比在夜晚结束时更大(图2)。2B, C),尽管有可能只是在ZT24的幼苗中表达的基因比ZT4和ZT9少。此外,我们发现大多数基因只在一个时间点被错误调控。这些数据表明,ahl调控基因在一天中的特定时间受到很大程度的影响。
ahl分子调节进行通信和pif靶基因,包括新发现的Pre1 pil1 hfr1,CDF5,而且XTR7, SD格式
为了更多地了解SOB3和其他ahl可能对生长产生的具体影响,我们将重点放在论坛s和pif靶基因。我们发现已知的生长促进基因被PIFs激活并参与生长素生物合成(YUC8, (62])或信号(阿富汗二月年代19/22, (25,26,63,64),而国际宇航科学院年代19/29,[22,23,24])在所有时间点都被ahl抑制(图。2B).另一方面,论坛S本身(4,5,7,8),BES1,参与BR信号传递[35]只在一个或两个时间点被抑制(图;2B).值得注意的是,已知BES1和PIF4在激活生长促进基因表达的复合物中共同起作用[26,34],我们的数据表明,AHLs通过抑制两者的表达来降低该复合物的活性,特别是在ZT4BES1而且PIF4(图;2B).此外,我们还鉴定了pif调控的BR生物合成基因CPD而且BR6OX2[33,34]在ZT4处被直接抑制(图;2B、表S4),表明AHLs可能通过减少BR的生物合成进一步降低了BES1- pif4复合物的活性,从而减少了活性BES1的数量。
除了CPD而且BR6OX2,我们还确定了5个PIF靶基因被抑制,以前没有被描述为被ahl抑制:Pre1 pil1 hfr1,CDF5,而且XTR7(无花果。2B)。PRE1而且PIL1在所有时间点都被压抑。PRE1促进PIF-和br介导的细胞伸长调控[26,27,28,29,30.,31].另一方面,PIL1,最近更名为PIF2 [65],是一个以非典型方式工作的PIF。在负反馈回路中,PIL1抑制其他PIF家族成员基因表达的激活,从而抑制生长[66,67,68].相比之下PRE1而且PIL1,HFR1仅在ZT9处被抑制(图;2B).HFR1也抑制pif介导的基因表达和生长的激活,作为负反馈循环的一部分[69,70,71,72].CDF5而且XTR7在ZT9和ZT24位点均被SOB3抑制,但在ZT4位点未被SOB3抑制。2B). CDF5促进一些pif激活基因的表达,包括YUC8而且IAA19,并始终对下胚轴的生长起积极作用[32].XTR7(也称为XTH15)是一种细胞壁修饰酶,已被证明能积极调节叶柄伸长[73].鉴于调节下胚轴和叶柄生长的分子机制有许多相似之处[21],可能XTR7也促进下胚轴伸长;因此,AHLs对SD下胚轴生长的影响可能部分是由于其抑制作用所致XTR7.
此外,微管已被证明参与了拟南芥下胚轴的伸长[74,75].事实上,在我们的RNA-seq数据中发现几个编码微管相关蛋白(MAPs)的基因被SOB3抑制。例如,MAP65-6,其蛋白质定位于线粒体并结合微管[76]在ZT4和ZT9被抑制,并在所有时间点被SOB3结合。微管稳定基因,WDL4[77,78], ZT9抑制,SOB3在各时间点结合。事实上,最近人们发现WDL4通过调节根尖钩形成过程中的PIN运输来调节生长素最大值[79].因此,我们的数据表明,SOB3可以通过抑制SD下胚轴伸长部分控制地图年代。
我们的RNA-seq数据还表明,AHLs激活SD中生长抑制因子的表达。我们特别鉴定了Favero等人讨论过的生长抑制因子。21,例如HY5, BBX21-22,ELF4,而且IAA17,为ahl诱导(图;2C).然而,有趣的是,Favero et al. [18]观察到较高的表达BIN2在SOB3-D相比sob3-6使用在LD生长并在ZT4收获的14天大的植物,我们没有发现BIN2两种突变体在ZT4、ZT9或ZT24位点被差异调控。因此,虽然AHLs可能通过促进bin2介导的磷酸化以及随后PIF3和PIF4的降解来抑制生长[80,81通过加强…的表达BIN2在幼莲座中[18], AHLs可能不会通过这一途径抑制幼苗的生长,至少那些在SD中生长的幼苗。考虑到我们和Favero等人。18研究确定RGA由于在SOB3-D相比sob3-6在ZT4(图;2C), AHLs可能部分通过促进della介导的PIF降解和抑制PIF与靶基因结合来抑制SD下胚轴生长[82,83,84,特别是在这个时间点。
一个重要的问题是,PIF靶基因是否真的像我们的ChIP-seq数据和Favero等人的数据所表明的那样,由AHLs直接调控。[18)的研究。可以想象,PIF指标的下调是在SOB3-D相比sob3-6可能仅仅是PIF活性降低的下游结果SOB3-D,鉴于我们发现PIF4,5,7,8都受到ahl的抑制(图;2B)和AHLs可能促进della介导的PIFs降解,如上所述。然而,虽然这些机制可能会降低PIF活性,从而降低PIF靶基因在ZT4和ZT9上的激活,但它们似乎不能解释AHLs对这些基因在ZT24上表达的负面影响。AHLs理论上影响ZT24中PIF活性的另一种方式是增加、转录,从而增强了phyB介导的这些转录因子在夜间的降解,因为phyB在黄昏后才慢慢恢复到其不活跃的Pr形式[85,86,87,88,89,90,91,92].然而,在我们的RNA-seq数据中,唯一被发现有差异调节的光受体是UVR8。因此,至少在SD的ZT24中,AHLs可能直接抑制PIF靶基因的表达。
SOB3:一种转录因子和潜在的染色质重塑蛋白
在我们的数据中看到的SOB3结合模式可能表明SOB3是一种染色质重塑蛋白。AT-hook motif首次在高迁移率组(HMG)蛋白中被描述[93,94,95],是一个非组蛋白染色体蛋白质家族,在多种真核生物中都有发现[96,97].与SOB3类似,HMGA蛋白含有一个AT-hook基序,通过该基序结合AT-rich DNA [95,98,99].此外,HMGA蛋白也参与染色质的组织[97]和enhancesome的形成[One hundred.,101].HMGA蛋白与组蛋白H1竞争与连接体DNA的结合,导致染色质结构松动[95,102,103,104].此外,HMGA蛋白已被证明与H2A/H2B/H3/H4核小体结合[105],表明这些蛋白在转录调节过程中对核心组蛋白的驱逐和/或动员作用[95].HMGA蛋白也可能参与DNA环和染色质重排,将增强体/启动子聚集在一起,从而开始转录[95,One hundred.,101].我们的SOB3 ChIP-seq结合数据可能表明SOB3的作用类似于HMGA蛋白,其作用是通过修饰染色质被动地调节转录。SOB3也有可能作为转录因子或染色质重塑蛋白,这取决于结合发生的特定环境。
SOB3本身可能不是染色质重塑因子,而是通过其他机制影响染色质重塑,例如与染色质重塑复合物的成员相互作用。这些复合物在进化上是保守的,包含多个亚基。染色质重塑复合物可以通过改变核小体的组成来调节染色质结构[106,107].目前已知的染色质重塑atp酶有四个主要亚家族:INO80/SWR1、CHD、ISWI和SWI/SNF [108,109,110,111,112,113,114].SWR1将组蛋白H2A交换为H2A. z。这是特别有趣的,因为温暖的温度促进H2A的排出。从染色质中提取Z,增强了pif靶向基因的激活,如YUC8[18,62,115,116,117,118,119].此外,有一些证据表明SOB3抑制转录YUC9通过增加H2A来招聘。通过招募SWR1复合物在其启动子上沉积Z [8].有趣的是,肌动蛋白相关蛋白9 (ARP9)已被证明参与SWI/SNF染色质重塑[120,121],在我们的RNA-seq数据中发现,在所有时间点都是诱导的。需要进一步的实验来确定SOB3是否通过与染色质重塑复合物相互作用或影响染色质重塑蛋白的表达来影响染色质重塑。
AHLs在植物发育中的作用:农业意义
先前的研究表明过度表达SOB3而且AHL20拟南芥和亚麻荠延缓开花[9,10].进一步研究新发现的sob3调控基因,如CDF5,可能阐明了SOB3调节花期的其他机制。例如,丧失功能cdf5-1与野生型植物相比,突变体开花时间更早[122].改变或调节作物物种开花时间的能力有许多宝贵的优势,例如允许不同或增加种植季节。进一步的实验评估的作用CDF5在ahl调控的花期可能阐明操纵这一有利农艺性状的附加机制。
结论
总之,我们的数据显示,在sd生长的幼苗中,在三个不同的时间点,参与下胚轴生长的基因被抑制,而生长抑制因子被AHLs诱导(图2)。2).此外,我们发现尽管AHLs在不同时间点调控的基因组略有不同,但SOB3的结合模式在很大程度上保持一致(图2)。4,5,6,10,11).重要的是要注意,我们不能排除在基因表达的一些差异之间观察到的可能性SOB3-D而且sob3-6在我们的RNA-seq实验中,两种基因型之间下胚轴长度的表型差异是间接引起的,因此在本实验中使用的时间点不受AHLs的直接调控。然而,我们怀疑至少在本实验中鉴定的ahl对基因表达的某些时间点特异性影响可归因于含有ahl的蛋白复合物在ZT4、ZT9和ZT24的组成不同。AHL复合物的活性可能是通过与不同的非AHL转录因子相互作用而改变的,这些转录因子对调节基因表达和最终下胚轴在SD中特定时间点的生长很重要。或者,AHL复合物中特定AHL家族成员在不同时间点的变化可能解释了这些转录因子在三个时间点对基因表达的不同影响。AHLs也有可能通过作为染色质重塑蛋白调控SD中的基因表达,因为AT-hook motif也在染色质重塑HMGA蛋白中被发现。或者,在SD中ahl本身并不是染色质重塑者,它们可能参与染色质重塑复合物的成员来调节基因表达。未来的研究应重点验证这些假设,以阐明SOB3和其他ahl调控基因表达和下胚轴生长的精确分子机制。
方法
本研究的目的是揭示在SD条件下促进拟南芥在不同时间点生长的独特分子事件。更准确地说,我们的目标是识别与SOB3结合并在两者之间被错误调控的特定基因sob3可能参与拟南芥下胚轴生长发育的突变体。总体设计包含两个主要实验:ChIP-seq和RNA-seq,下面将详细描述。
植物材料和生长条件
拟南芥(拟南芥研究中使用的系都在Columbia (Col-0)背景中,之前已经描述过:SOB3-D而且sob3-6[1];ProSOB3: SOB3-GFP sob3-4[5].种子表面消毒,在含0.6% Gelzan CM (Sigma-Aldrich)和1%蔗糖(w/v)的全强度MS培养基上生长。种子在4°C黑暗中分层3天。植物在MLR-351植物生长室中生长。在22°C的温度和短日光周期(8小时亮/16小时暗)下保持5 - 6天。白天使用3LS作为光照设置,产生的白光荧光灯的通量率约为35 ~ 55 μmol/m2/秒。在RNA-seq和ChIP-seq实验中,幼苗在黎明后4小时(ZT4)、熄灯后1小时(ZT9)或第二天黎明时(ZT24)三个时间点之一收获,并立即在液氮中冷冻。
拟南芥基因座标识:
PIF4: AT2G43010
CPD: AT5G05690
BES1: AT1G19350
SAUR24: AT5G18080
BR6OX2: AT3G30180
PRE1: AT5G39860
SAUR19: AT5G18010
SAUR22: AT5G18050
YUC8: AT4G28720
PIL1: AT2G46970
IAA19: AT3G15540
IAA29: AT4G32280
HRF1: AT1G02340
PIF5: AT3G59060
PIF7: AT5G61270
PIF8: AT4G00050
CDF5: AT1G69570
XTR7: AT4G14130
ELF4: AT2G40080
HY5: AT5G11260
BBX22: AT1G78600
RGA: AT2G01570
PRR9: AT2G46790
IAA17: AT1G04250
ELF3: AT2G25930
BBX21: AT1G75540
下胚轴的测量
苗木用于定量sob3在上述相同的条件下培养突变表型。野生型种子为拟南芥(l)Col-0生态型。5天大的种子被转移到透明体中,用平板扫描仪对其进行数字化处理。这些透明度包括一个用于测量1毫米长度的尺子,以设置ImageJ (NIH)中测量的参数。用分段线仪从下胚轴顶部到根部开始测量下胚轴。测量结果被转移到Excel电子表格中进行分析。采用双尾韦尔奇t检验检验突变体和野生型下胚轴长度是否存在显著差异。
RNA测序(RNA-seq)和数据分析
在指定的时间点收获20-35株完整的幼苗,在不同的培养皿上生长不同的生物重复。同一生物复制组中不同基因型的样本从同一培养皿中收获。这种实验设计是为了尽量减少板间变异对基因表达的影响。根据制造商的说明,使用RNeasy Plant Mini Kit (QIAGEN)提取总RNA,包括柱上DNase消化以消除基因组DNA。分离出的RNA使用KAPA链RNA- seq试剂盒(KAPA Biosystems)进行文库制备,使用NEBNext Multiplex Oligos for Illumina (New England Biolabs)作为接头,使用Agencourt AMPure XP (Beckman Coulter)珠而不是KAPA Pure珠。使用Illumina NextSeq500测序仪获得84或86 bp的单读序列。原始数据文件(bcl格式)通过bcl2fastq (Illumina)转换为fastq文件。利用Bowtie方法将50%以上的reads定位到拟南芥TAIR10 cDNA参考位点。123使用以下参数:' -all -best -strata '。每个样本映射的总reads数为600 - 1200万。使用R/Bioconductor的edgeR包识别成对样品之间差异表达的转录本[124]而FDR的临界值为0.05。ahl调控基因被定义为至少有一个转录本在sob3-6和sob3- 3之间有差异调控的核编码基因。维恩图是使用在线工具生成的http://bioinformatics.psb.ugent.be/webtools/Venn/.
染色质免疫沉淀测序(ChIP-seq)
ChIP程序包括每个时间点3个生物重复。ChIP-seq的染色质免疫沉淀基本上如前所述[18].1.5克ProSOB3: SOB3-GFP sob3-4[5整个幼苗在ZT4收获,立即用液氮冷冻。使用MB1200多珠震荡器(Yasui Kikai)将样品研磨成细粉末,在1%甲醛中交联10分钟,之后分离核部分。染色质悬浮在SDS裂解缓冲(50毫米Tris-HCl (pH值7.8),1% SDS, 10毫米EDTA)与芯片稀释大约5倍稀释缓冲(50毫米Tris-HCl pH值7.8,0.167 M氯化钠,特里同x - 100 1.1%, 0.11%钠脱氧胆酸盐,1罗氏完整EDTA-free平板/ 50毫升溶液),在十二5°C 15分钟使用Covaris S2 Focused-ultrasonicator milliTUBE 1毫升阿发纤维管(Covaris)和以下设置:责任周期5%,强度4,周期每200年破灭。这产生的平均碎片大小约为100-300 bp。超声染色质在4℃下使用抗gfp抗体(ab290, Abcam)和Dynabeads蛋白A(赛默飞世尔科学公司)进行免疫沉淀。用抗体孵卵后,用低盐RIPA缓冲液(50mMTris-HCl [pH 7.8], 150mMNaCl, 1mMEDTA, 0.1% SDS, 1% Triton X-100, 0.1%脱氧胆酸钠,1个Roche cOmplete Ultra片/50 mL溶液)洗涤1次,用高盐RIPA缓冲液(50mMTris-HCl [pH 7.8], 500mMNaCl, 1mMEDTA, 0.1% SDS, 1% Triton X-100, 0.1%脱氧胆酸钠,1个Roche cOmplete Ultra片/50 mL溶液)洗涤2次,用LNDET (10mm Tris-HCl [pH 7.8], 250 mM LiCl, 1% IGEPAL CA-630清洗1次,1%脱氧胆酸钠,1mm EDTA)和一次TE缓冲液。染色质从Dynabeads中洗脱,并在含有10 mM Tris-HCl [pH 7.8], 300 mM NaCl, 5 mM EDTA和0.5% SDS的溶液中在65°C交联。采用苯酚-氯仿萃取-乙醇沉淀法分离DNA。测序库基本上按照Rymen等人的描述制备.(2019)。使用Qubit dsDNA HS测定试剂盒和Qubit 2.0荧光计(赛默飞世尔科学公司)对分离的DNA进行定量,并对3.65 ng ChIPed或输入(用于对照文库)DNA进行文库制备。使用KAPA Hyper Prep Kit for Illumina (KAPA Biosystems)和Illumina兼容适配器(New England Biolabs)制备文库。使用Illumina NextSeq500测序仪获得86 bp的单读序列。
染色质免疫沉淀测序(ChIP-seq)数据分析
原始数据文件(bcl格式)通过bcl2fastq (Illumina)转换为fastq文件。超过70%的reads使用Bowtie方法唯一映射到拟南芥TAIR10参考文献[123,设置为“-m”。“每个样本的唯一映射reads总数为1100万到2600万。通过使用MACS2中的" callpeak "命令将ChIP样本与输入进行比较来调用峰[125].折叠富集bdg格式峰值文件是通过使用处理堆积生成的,控制lambda输出文件由“callpeak”生成,作为MACS2“bdgcmp”命令的输入,设置为“-m FE”。然后,这些折叠富集bdg文件被转换为bigWig文件,bigWig文件又被用于在deepTools中分别通过computeMatrix和plotHeatmap或plotProfile命令生成显示SOB3结合分布或靶基因相对结合的图形[126].使用整合基因组学查看器(IGV) 2.3.88版本对峰值进行可视化[127,128].Motif分析采用模因芯片[129]拟南芥PBM基序被选为已知基序[56].以SOB3-GFP ChIP-seq峰峰为中心的300 bp核苷酸序列作为主要输入序列进行MEME-ChIP分析。山峰用HOMER标注[130].维恩图是使用在线工具生成的http://bioinformatics.psb.ugent.be/webtools/Venn/.
量化和统计分析
在指示的地方,在哪里进行了双尾韦尔奇t检验P≤0.05 = *;P≤0.005 = **;P≤0.0005 = ***;而且P≤0.00005 = ****
数据和材料的可用性
在当前研究中生成和/或分析的数据集可在基因表达综合库中获得,https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE189265.
参考文献
Street IH, Shah PK, Smith AM, Avery N, Neff MM.含有at -hook的蛋白SOB3/AHL29和ESC/AHL27是拟南芥下胚轴生长的负调控因子。植物学报,2008;32(1):1 - 14。https://doi.org/10.1111/j.1365-313x.2007.03393.x.
周玲,刘震,刘颖,孔东,李涛,余松,梅红,徐旭,刘红,陈玲,罗磊。水稻一种新型基因OsAHL1在提高水稻抗旱能力和抗旱性方面具有重要意义。科学通报2006;6:30264。
黄MM, Bhaskara GB,文TN,林卫东,阮TT, Chong GL, Verslues PE。拟南芥高度aba诱导的磷酸化蛋白组学研究发现AT-Hook-Like10磷酸化对胁迫生长调控至关重要。美国国家科学研究院。2019; 116:2354 - 63。https://doi.org/10.1073/pnas.1819971116.
肖c,陈峰,于X,林c,付艳芳。在拟南芥中,AT-hook基因AHL22的过表达延迟开花并抑制下胚轴的伸长。植物化学学报,2009;https://doi.org/10.1007/s11103-009-9507-9.
张晓明,李志刚,李志刚,李志刚。光敏色素B4- 3抑制因子对下胚轴生长的调控作用。中国生物医学工程学报,2016;29(4):344 - 344。https://doi.org/10.1104/pp.16.00405.
赵娟,范维罗,彭华,Neff MM.拟南芥AHL家族通过PPC/DUF296结构域相互作用冗余调控下胚轴生长。美国国家科学研究院。2013年,110年(48)。https://doi.org/10.1073/pnas.1219277110
彭华,赵娟,Neff MM. ATAF2整合拟南芥油菜素类固醇失活与幼苗光形态建成。发展。2015;142(23):4129 - 38。https://doi.org/10.1242/dev.124347.
拟南芥基质附着和atp依赖性染色质重塑的协调调节生长素生物合成和下胚轴伸长。Plos One. 2017;12(7)。https://doi.org/10.1371/journal.pone.0181804
Tayengwa R, Koirala PS, Pierce CF, Werner BE, Neff MM. AtAHL20过表达通过抑制FT表达导致拟南芥开花延迟。BMC植物生物学2020;20:559。https://doi.org/10.1186/s12870-020-02733-5.
通过过表达Atsob3-6基因变异改善亚麻荠种子大小、种子重量和出苗率。转基因Res. 2020;29(4): 409-18。https://doi.org/10.1007/s11248-020-00208-9.
李志强,李志强,李志强,等。拟南芥光敏色素B全蛋白的生物学特性研究。植物细胞学报。1997;9(12):2271。https://doi.org/10.2307/3870584.
Weigel D, Ahn JH, Blázquez MA, Borevitz JO, Christensen SK, Fankhauser C, Ferrandiz C, Kardailsky I, Malancharuvil EJ, Neff, MM, Nguyen JT, Sato S, Wang ZY, Xia Y, Dixon RA, Harrison MJ, Lamb CJ, Yanofsky MF, Chory J.拟南芥的激活标记。植物科学进展。2000;22(4):344 - 344。https://doi.org/10.1104/pp.122.4.1003.
Neff MM, Fankhauser C, Chory J.光:时间和地点的指示器。基因工程,2000;14:257-71。
Fujimoto S, Matsunaga S, Yonemura M, Uchiyama S, Azuma T, Fukui K.定位于染色体表面的一种新的植物MAR DNA结合蛋白的鉴定。植物分子生物学杂志,2004;26(2):344 - 344。https://doi.org/10.1007/s11103-004-3249-5.
赵j, Favero DS,邱j, Roalson EH, Neff MM.陆生植物AT-hook Motif核定位基因家族的进化与多样性。中国生物医学工程学报,2014;26(3):342 - 342。
Lim PO, Kim Y, Breeze E, Koo JC, Woo HR, Ryu JS, Park DH, Beynon J, Tabrett A, Buchanan-Wollaston V, Nam HG.一种染色质结构控制AT-hook蛋白的过表达延长了植物叶片寿命和收获后贮藏寿命。植物学报,2007;26(6):344 - 344。https://doi.org/10.1111/j.1365-313x.2007.03317.x.
贾秋生,朱娟,徐晓峰,娄勇,张志林,张志平,杨忠。拟南芥AT-hook蛋白TEK正向调控阿拉伯半乳聚糖蛋白的表达以形成Nexine。植物学报。2015;8(2):251-60。https://doi.org/10.1016/j.molp.2014.10.001.
Favero DS, Kawamura A, Shibata M, Takebayashi A, Jung JH, Suzuki T, Jaeger KE, Ishida T, Iwase A, Wigge PA, Neff MM, Sugimoto K. AT-Hook转录因子通过拮抗PIFs限制叶柄生长Curr Biol 2020;30(8)。https://doi.org/10.1016/j.cub.2020.02.017
Nozue K, Covington MF, Duek PD, Lorrain S, Fankhauser C, Harmer SL, Maloof JN。由内部和外部线索的巧合所解释的有节奏的生长。自然。2007;448(7151):358 - 61。
Box MS, Huang BE, Domijan M, Jaeger KE, Khattak AK, Yoo SJ, Sedivy EL, Jones DM, Hearn TJ, Webb AA, Grant A, Locke JC, Wigge PA。ELF3控制拟南芥的热响应性生长。中国生物医学杂志,2015;25(2):194-9。
Favero DS, Lambolez A, Sugimoto K.调节空中植物器官伸长的分子途径:光,生物钟和温度的焦点。植物学报(自然科学版);2011;26(2):369 - 369。https://doi.org/10.1111/tpj.14996.
Kunihiro A, Yamashino T, Nakamichi N, Niwa Y, Nakanishi H, Mizuno T.光敏色素相互作用因子4和5 (PIF4和PIF5)激活同源异型箱ATHB2和生长素诱导的IAA29基因,这是拟南芥光周期控制植物生长的符合机制。中国生物医学工程学报,2011;
孙娟,齐琳,李艳,翟强,李晨。PIF4和PIF5转录因子连接蓝光和生长素调控拟南芥的向光反应。中国生物医学工程学报,2013;
Pucciariello O, Legris M, Rojas CC, Iglesias MJ, Hernando CE, Dezar C, Vazquez M, Yanofsky MJ, Finlayson SA, Prat S, Casal JJ。持续荫凉下生长素信号的重新布线。中国生物医学工程学报,2018;29(4):344 - 344。https://doi.org/10.1073/pnas.1721110115.
Hornitschek P, Kohnen MV, Lorrain S, Rougemont J, Ljung K, Lopez-Vidriero I, Franco-Zorrilla JM, Solano R, Trevisan M, Pradervand S, Xenarios I, Fankhauser C.光敏色素相互作用因子4和5通过直接控制生长素信号通路控制幼苗在光照条件变化下的生长。植物学报,2012;
吴娥,朱建勇,王志智。BZR1和PIF4的相互作用整合了油菜素类固醇和环境反应。中国生物医学工程学报,2012;
Lee S, Lee S, Yang KY, Kim YM, Park SY, Kim SY, Soh MS. PRE1及其同源基因的过表达在拟南芥中激活赤霉素依赖反应。植物细胞物理学报。2006;47:591-600。
白my,范M,吴娥,王志智。在拟南芥中,三重螺旋-环-螺旋/基本螺旋-环-螺旋级联控制多种激素和环境信号通路下游的细胞伸长。中国生物医学工程学报,2012;
白美美,尚建新,吴娥,范敏,白勇,Zentella R,孙TP,王志智。油菜素内酯、赤霉素和光敏色素影响拟南芥的一个共同转录模块。中国生物医学工程学报,2012;
郝艳,吴娥,崔庚,梁志,王哲。植物光调节发育中HLH与bHLH因子的相互作用。植物学报,2012;5(3):688-97。https://doi.org/10.1093/mp/sss011.
Ikeda M, Fujiwara S, Mitsuda N, Ohme-Takagi M.一种三拮抗基本螺旋-环-螺旋系统调节拟南芥细胞伸长。中国生物医学工程学报,2012;
Martin G, Rovira A, Veciana N, Soy J, tolado - ortiz G, Gommers CMM, Boix M, Henriques R, Minguet EG, Alabadi D, Halliday KJ, Leivar P, Monte E.拟南芥转录抑制昼夜波形成pif调控的光周期响应生长。中国生物医学杂志,2018;28(2):311-8。https://doi.org/10.1016/j.cub.2017.12.021.
魏震,袁涛,Tarkowská D,金俊,南HG, Novák O,何凯,勾小鹏,李娟。油菜素类固醇素合成的转录因子级联调控研究。中国生物医学工程学报,2017;29(2):379 - 379。https://doi.org/10.1104/pp.16.01778.
Martínez C, Espinosa‐Ruíz A, de Lucas M, Bernardo‐García S, Franco‐Zorrilla JM, Prat S. PIF4‐诱导BR合成对日生和热生生长至关重要。管理学报,2018;37(23)。doi:https://doi.org/10.15252/embj.201899552
尹艳,张志刚,张志刚,张志刚。拟南芥油菜素激素调控基因表达的转录因子研究。细胞。2005;120(2):249 - 59。https://doi.org/10.1016/j.cell.2004.11.044.
Saito M, Kondo Y, Fukuda H. BES1和BZR1冗余促进韧皮部和木质部分化。中国生物医学工程学报,2018;29(3):379 - 379。https://doi.org/10.1093/pcp/pcy012.
张佳杰,吴松。cop1介导的BBX22/LZF1降解对拟南芥幼苗发育的影响。中国生物医学工程学报。2011;26(1):344 - 344。https://doi.org/10.1104/pp.111.175042.
杨晓东,李志强,李志强,李志强。拟南芥轻度调控发育过程中HY5的靶向失稳。自然,2000;405:462-6。https://doi.org/10.1038/35013076.
王丽华,李志强,王志强,王志强,王志强,王志强,王志强,王志强。COP1和HY5之间的分子相互作用确定了拟南芥发育中光控制的调节开关。化学学报,1998;1:213-22。https://doi.org/10.1016/s1097 - 2765 (00) 80022 - 2.
Burko Y, Seluzicki A, Zander M, Pedmale UV, Ecker JR, Chory J.嵌合激活物和阻遏物定义HY5活性并揭示光调节反馈机制。植物细胞。2020;32:967-83。https://doi.org/10.1105/tpc.19.00772.
Bursch K, tolato - ortiz G, Pireyre M, Lohr M, Braatz C, Johansson H. BBX蛋白作为HY5限速辅因子的鉴定。植物学报。2020;6(8):921-8。https://doi.org/10.1038/s41477-020-0725-0.
徐东,姜勇,李娟,林峰,霍尔姆,邓小文。BBX21是拟南芥B-box蛋白,可直接激活HY5,被COP1靶向降解26S蛋白酶体。美国国家科学研究院。2016; 113:7655-60。
加拉沃蒂A,马尔康伯S,盖恩斯C,斯坦菲尔德S,惠普尔C,凯洛格E,施密特RJ。贫瘠秸秆FASTIGIATE1是玉米穗形成所必需的AT-Hook蛋白。植物学报,2011;23(5):1756-71。https://doi.org/10.1105/tpc.111.084590.
金勇,罗强,童红,王安,程忠,唐杰,李东,赵霞,李霞,万杰,焦勇,褚超,朱林。水稻中一个AT-hook基因是水稻胚芽形成和花器官数量控制的必需基因。中国生物医学工程学报。2011;39(2):357 - 357。https://doi.org/10.1016/j.ydbio.2011.08.023.
楼艳,徐旭,朱娟,顾娟,Blackmore S,杨忠。绒毡层AHL家族蛋白TEK决定花粉壁外环蛋白的形成。自然通讯,2014;5(1)。doi:https://doi.org/10.1038/ncomms4855
Matsushita, Furumoto T, Ishida S, Takahashi Y. AGF1是AtGA3ox1编码GA3 -氧化酶负反馈所必需的AT-Hook蛋白。植物科学进展。2007;43(3):344 - 344。https://doi.org/10.1104/pp.106.093542.
Vom Endt D, Soares e Silva M, Kijne JW, Pasquali G, Memelink J.蔷薇ORCA3转录因子基因中茉莉酸响应启动子元件的鉴定,该启动子与AT-Hook dna结合蛋白特异性相互作用。植物科学进展。2007;44(3):344 - 344。
吴凯,于海华,伊藤涛。AGAMOUS控制GIANT KILLER,一种多功能染色质修饰物在生殖器官模式和分化中的作用。科学通报,2009;7(11)。doi:https://doi.org/10.1371/journal.pbio.1000251
吴坤,伊藤。AT-hook/PPC结构域蛋白在拟南芥中的作用。植物信号行为研究。2010;5(2):200-1。https://doi.org/10.4161/psb.5.2.11111.
徐颖,甘娥,伊藤涛。AT-hook/PPC结构域蛋白TEK负调控花抑制因子包括MAF4和MAF5。植物信号行为研究。2013;8(8)。doi:https://doi.org/10.4161/psb.25006
尹俊,金玉生,郑志华,徐普杰,朴明哲。含AT-hook motif的蛋白AHL22通过修饰开花位点T染色质调控拟南芥开花起始。中国生物医学工程学报,2012;https://doi.org/10.1074/jbc.m111.318477.
Bailey TL, Johnson J, Grant CE, Noble WS。MEME套房。核酸研究,2015;43(W1): W39-W49。
Kosugi S, Ohashi Y. TCP蛋白家族的DNA结合和二聚化特异性和潜在靶点。植物学报2002;30:337-48。
李志强,李志强,李志强。量化主题之间的相似性。中国生物工程学报。2007;8:R24。
Schommer C, Palatnik JF, Aggarwal P, Chetelat A, Cubas P, Farmer EE, Nath U, Weigel D. miR319靶点对茉莉酸盐生物合成和衰老的控制。公共科学图书馆。2008;6:e230。
Franco-Zorrilla JM, Lopez-Vidriero I, Carrasco JL, Godoy M, Vera P, Solano R.植物转录因子的dna结合特异性及其定义靶基因的潜力。中国科学院学报(自然科学版),2014;
尹东,刘霞,石志,李东,朱林。水稻AT-hook蛋白PALEA1与TCP家族转录因子RETARDED PALEA1的物理相互作用。生物化学与物理学报,2018;49:487 - 92。
Oh E, Yamaguchi S, Hu J, Yusuke J, Jung B, Paik I, Lee HS, Sun TP, Kamiya Y, Choi G. PIL5是一种与光敏色素相互作用的bHLH蛋白,通过直接与GAI和RGA启动子结合来调控赤霉素的反应性。植物学报。2007;19:1192-208。
Kim DH, Yamaguchi S, Lim S, Oh E, Park J, Hanada A, Kamiya Y, Choi G. SOMNUS是拟南芥ccch型锌指蛋白,对PIL5下游光依赖种子萌发具有负向调控作用。植物学报,2008;20:1260-77。
张颖,Mayba O, Pfeiffer A, Shi H, Tepperman JM, Speed TP, Quail PH.拟南芥中PIF bHLH因子通过共享靶基因的差异表达模式调控幼苗形态发生,提供了一个转录中心信号枢纽。PLoS Genet, 2013;9: e1003244。
Huth JR, Bewley CA, Nissen MS, Evans JN, Reeves R, Gronenborn AM, Clore GM. HMG-I(Y)-DNA复合物的溶液结构定义了一种新的结构小槽结合基序。生物工程学报。1997;4:657-65。
孙娟,齐玲,李艳,褚娟,李晨。pif4介导的YUCCA8表达激活与温度调控拟南芥下胚轴生长的生长素通路整合。PLoS Genet, 2012;8: e1002594。
Spartz AK, Lee SH, Wenger JP, Gonzalez N, Itoh H, Inze D, Peer WA, Murphy AS, Overvoorde PJ, Gray WM。SMALL生长素UP RNA基因SAUR19亚家族促进细胞扩张。植物学报(英文版);2012;
Spartz AK, Ren H, Park MY, Grandt KN, Lee SH, Murphy AS, Sussman MR, Overvoorde PJ, Gray WM。SAUR抑制PP2C-D磷酸酶激活质膜H+- atp酶促进拟南芥细胞扩张《植物科学》2014;26:2129-42。
范维恩,王志强,王志强,等。光敏色素及其相互作用因子。植物学报。2018;176:1025-38。
罗伊-维拉诺瓦I,鲍J,索林C,德夫林PF,马丁内斯-加西亚JF。光敏色素信号主要靶基因的鉴定。拟南芥避荫反应的早期转录控制。植物物理学报。2006;41:85 - 96。
李玲,张强,Pedmale UV, Nito K,付伟,林玲,Hazen SP, Chory J. PIL1参与了一个负反馈循环,调节自身基因表达对阴影的响应。植物学报。2014;7:1582-5。
罗强,连海林,何素生,李林,贾平,杨海峰。拟南芥COP1和phyB与PIL1物理相互作用调控其稳定性和光形态发生发育《植物科学》2014;26:2441-56。
Fairchild CD, Schumaker MA, Quail PH. HFR1编码非典型bHLH蛋白,在光敏色素A信号转导中起作用。基因工程,2000;14:2377-91。
Fankhauser C, Chory J. RSF1,一个涉及光敏色素A信号的拟南芥基因座。植物学报。2000;124:39-45。
Duek PD, Fankhauser C. HFR1,一个假定的bHLH转录因子,介导光敏色素a和隐色素信号。植物学报(英文版);2003;
Hornitschek P, Lorrain S, Zoete V, Michielin O, Fankhauser C.通过形成非dna结合的bHLH异二聚体抑制遮阴反应。杨晓明,2009;
Sasidharan R, Chinnappa CC, Staal M, Elzenga JT, Yokoyama R, Nishitani K, Voesenek LA, Pierik R.拟南芥避荫过程中光质介导的叶柄伸长涉及木葡聚糖内转葡萄糖酶/水解酶对细胞壁的修饰。植物科学进展。2010;
李文杰,李文杰,李文杰,李文杰。拟南芥下胚轴细胞伸长和微管行为对乙烯和生长素的响应。植物生长学报,2005;24(3):166-78。https://doi.org/10.1007/s00344-005-0044-8.
Crowell EF, Timpano H, Desprez T, Franssen-Verheijen T, Emons A, Höfte H, Vernhettes S.拟南芥下胚轴表皮细胞内外表面纤维素取向的差异调控。植物学报。2011;23(7):2592-605。https://doi.org/10.1105/tpc.111.087338.
毛涛,金亮,李红,刘波,袁敏。拟南芥MAP65家族两种微管相关蛋白在微管上的不同功能。植物科学进展。2005;38(2):344 - 344。https://doi.org/10.1104/pp.104.052456.
袁春春,李志刚,刘志刚,刘志刚,刘志刚。拟南芥WVD2和WDL1基因对螺旋器官生长和各向异性细胞扩张的调控作用。植物科学学报,2003;
王勇,阮春华,王志强,马森PH. WVD2是拟南芥中一种新的微管相关蛋白。植物J. 2007; 49:961-71。
邓娟,王霞,刘忠,毛涛。拟南芥微管相关蛋白WDL4调控生长素分布促进根尖钩开放。植物学报。2021;33(6):1927-44。https://doi.org/10.1093/plcell/koab080.
Bernardo-Garcia S, de Lucas M, Martinez C, Espinosa-Ruiz A, Daviere JM, Prat S. br依赖性磷酸化调节PIF4转录活性并影响下胚轴的日生长。基因开发,2014;28:1681-94。
凌俊杰,李娟,朱东,邓小伟。拟南芥COP1/SPA复合物在抑制bin2介导的PIF3磷酸化和黑暗降解中的非典型作用中国科学院学报(自然科学版),2017;
阿拉巴迪D,加列戈-巴托洛梅J,奥兰多L,加西亚-卡塞尔L,卢比奥V,马丁内斯C,弗里基利奥M,伊格莱西亚斯-佩德拉兹JM,埃斯皮诺萨A,邓XW,布拉斯克斯MA。赤霉素调节光信号通路防止拟南芥幼苗在黑暗中去黄化。植物J. 2008; 53:324-35。
de Lucas M, Daviere JM, Rodriguez-Falcon M, Pontin M, iglesia - pedraz JM, Lorrain S, Fankhauser C, Blazquez MA, Titarenko E, Prat S.光和赤霉素控制细胞伸长的分子框架。大自然。2008;451:480-4。
李凯,于睿,范丽敏,魏宁,陈宏,邓小伟。在拟南芥中,della介导的PIF降解有助于光和赤霉素信号的协调。Nat Commun. 2016;7:11868。
杨晓明,张晓明,张晓明,等。拟南芥下胚轴伸长的研究进展。足底。1999;208:257 - 63。
李华,刘涛,刘涛,张志刚,张志刚。植物光敏色素介导的bHLH转录因子对幼苗光形态发育的抑制作用。当代生物学,2008;18:1815-23。
罗森伯格,胡松A,柯彻S, Kirchenbauer D, Timmer J, Nagy F, Schafer E, Fleck C.光敏色素B介导光形态建成的综合模型:从蛋白质动力学到生理学。PLoS ONE。2010; 5: e10721。
大豆J, Leivar P, gonzalz - schain N, Sentandreu M, Prat S, Quail PH, Monte E.光敏色素对拟南芥下胚轴生长的影响。植物J. 2012;
大豆J, Leivar P, Monte E. PIF1与PIF3, PIF4, PIF5共同促进拟南芥光周期条件下光敏色素调控生长。中国科学(d辑),2014;
陈敏。光敏色素B的光体定位与下胚轴伸长的延长和光依赖抑制密切相关。植物科学进展。2014;
Jung JH, Domijan M, Klose C, Biswas S, Ezer D, Gao M, Khattak AK, Box MS, Charoensawan V, Cortijo S, Kumar M, Grant A, Locke JC, Schafer E, Jaeger KE, Wigge PA。光敏色素在拟南芥中起着热传感器的作用。科学。2016;354:886-9。
Legris M, Klose C, Burgie ES, Rojas CC, Neme M, Hiltbrunner A, Wigge PA, Schafer E, Vierstra RD, Casal JJ。光敏色素B在拟南芥中整合光和温度信号。科学。2016;354:897 - 900。
高迁移率组(HMG)染色体蛋白的修订命名法。生物化学,2001;26(3):152-3。https://doi.org/10.1016/s0968 - 0004 (00) 01777 - 1.
黄志刚,黄志刚。HMG蛋白在染色质上的结合和相互作用:来自活细胞成像的经验教训。生物化学学报。2010;1799:15-27。https://doi.org/10.1016/j.bbagrm.2009.11.001.
Ozturk N, Singh I, Mehta A, Braun T, Barreto G. HMGA蛋白在转录激活过程中作为染色质结构的调节剂。前沿细胞发展生物学。2014;2:5。https://doi.org/10.3389/fcell.2014.00005.
Aravind L, Landsman D.在多种dna结合蛋白中鉴定的AT-hook基序。核酸研究,1998;26(19):4413-21。https://doi.org/10.1093/nar/26.19.4413.
HMGA蛋白的分子生物学:核功能中心。基因。2001;277(2):63 - 81。https://doi.org/10.1016/s0378 - 1119 (01) 00689 - 8.
崔天杰,冷锋。哺乳动物高迁移率群蛋白AT-hook 2特异性识别AT-Rich DNA序列:SELEX研究。生物化学,2007;46:13059 - 66。https://doi.org/10.1021/Bi701269s.
温冬,Nimzyk R, Bosche C, Meyer A, Bullerdiek J.染色质免疫沉淀分析HMGA2的DNA结合位点。PLoS ONE。2011; 6: e18837。https://doi.org/10.1371/journal.pone.0018837.
Bouallaga I, Massicard S, Yaniv M, Thierry F.一个包含Jun B/ fr -2异二聚体和HMG-I(Y)结构蛋白的增强体控制HPV18的转录。EMBO代表2000;1:422-7。https://doi.org/10.1093/embo-reports/kvd091.
Bouallaga I, Teissier S, Yaniv M, Thierry F. HMG-I(Y)和CBP/p300共激活子对于人乳头瘤病毒18型增强体转录活性至关重要。生物化学学报,2003;23:2329-40。https://doi.org/10.1128/Mcb.23.7.2329-2340.2003.
赵K, Kas E, Gonzalez E, Laemmli英国。HMG-I/Y在体外对组蛋白H1的sar依赖动员:HMG-I/Y富集在H1缺失的染色质中。安波j . 1993;12: 337 - 47。
杨海燕,李晓霞,李志强,李志强。组蛋白H1与染色质高迁移基蛋白的动态相互作用网络。分子生物学杂志,2004;24:4321-8。https://doi.org/10.1128/MCB.24.10.4321-4328.2004.
Kishi Y, Fujii Y, Hirabayashi Y, Gotoh Y. HMGA调节新皮层前体细胞的整体染色质状态和神经源性潜能。神经科学。2012;15:1127-33。https://doi.org/10.1038/Nn.3165.
李涛,李建平,李建平,等。DNA结构因子HMGA2在人胚胎干细胞中的高表达及其与核小体的关系。《创世纪》,2006;44:523-9。https://doi.org/10.1002/dvg.20242.
王志强,王志强,王志强,等。染色质结构与转录的关系。细胞。2002;108(4):475 - 87。https://doi.org/10.1016/s0092 - 8674 (02) 00654 - 2.
Ojolo SP,曹s, Priyadarshani SVGN, Li W, Yan M, Aslam M,赵宏,秦勇。植物生长发育调控:染色质重塑视角的研究进展。植物科学,2018;9:1232。https://doi.org/10.3389/fpls.2018.01232.
李志强,李志强,李志强,等。植物Snf2蛋白的基因沉默研究。植物科学进展,2008;13(10):557-65。https://doi.org/10.1016/j.tplants.2008.08.004.
凯恩斯BR。启动子上染色质结构和重塑的逻辑。大自然。2009;461:193-8。
克拉皮尔CR,凯恩斯BR。染色质重塑复合物的生物学。生物化学,2009;78:273-304。
Hargreaves DC, Crabtree GR. atp依赖性染色质重塑:遗传学,基因组学和机制。Cell Res. 2011; 21:396-420。
陈晓明,陈晓明,陈晓明,等。atp依赖性染色质重塑酶的研究进展。细胞。2013;154:490 - 503。
胡艳,朱楠,王霞,易强,朱东,赖艳,赵艳。水稻Snf2家族蛋白及其表观遗传调控作用分析。植物化学学报,2013;
韩雪芹,吴文明,崔胜,魏华,等。植物染色质重塑atp酶的作用及活性。中国植物学报,2015;29(1):344 - 344。https://doi.org/10.1111/tpj.12877.
Kumar SV, Wigge PA。H2A。含z的核小体介导拟南芥的热感觉反应。细胞。2010;140:136-47。
Coleman-Derr, D. Zilberman, D.组蛋白变体H2A沉积。基因体内的Z调控反应性基因。PLoS Genet, 2012;8:e1002988。
Cortijo S, Charoensawan V, Brestovitsky A, Buning R, Ravarani C, Rhodes D, van Noort J, Jaeger KE, Wigge PA。H2A对环境温度响应的转录调控。拟南芥Z核小体与HSF1转录因子。分子植物学报,2017;10(10):1258-73。https://doi.org/10.1016/j.molp.2017.08.014.
Tasset C, Yadav AS, Sureshkumar S, Singh R, van der Woude L, Nekrasov M, Tremethick D, van Zanten M, Balasubramanian S. powerdress介导的组蛋白去乙酰化在拟南芥热形态发生中至关重要。PLOS Genetics. 2018;14(3)。https://doi.org/10.1371/journal.pgen.1007280
van der Woude LC, Perrella G, Snoek BL, van Hoogdalem M, Novák O, van Verk MC, van Kooten HN, Zorn LE, Tonckens R, Dongus JA, Praat M, Stouten EA, Proveniers MCG, Vellutini E, Patitaki E, Shpulatov U, Kohlen W, Balasubramanian S, Ljung K, van der Krol AR, Smeekens S, Kaiserli E, van Zanten M。Z损耗。美国国家科学研究院。2019; 116(50): 25343 - 54。https://doi.org/10.1073/pnas.1911694116.
Cairns BR, erdjumant - bromage H, Tempst P, Winston F, Kornberg RD.两种肌动蛋白相关蛋白是染色质重塑复合物RSC和SWI/SNF的共同功能成分。生物化学学报,1998;2(5):639-51。https://doi.org/10.1016/s1097 - 2765 (00) 80162 - 8.
米格尔RB,交易RB,坎达萨米MK,麦金尼EC。核肌动蛋白相关蛋白作为发育的表观遗传调控因子。植物科学进展。2005;39(4):366 - 366。https://doi.org/10.1104/pp.105.072447.
Fornara F, Panigrahi KC, Gissot L, Sauerbrunn, Rühl M, Jarillo JA, Coupland G.拟南芥DOF转录因子冗余降低CONSTANS表达并对光周期开花响应至关重要。生物技术学报。2009;17(1):75-86。https://doi.org/10.1016/j.devcel.2009.06.015.
Langmead B, Trapnell C, Pop M, Salzberg SL.人类基因组短DNA序列的超快和内存高效对齐。中国生物工程学报。2009;10(3)。https://doi.org/10.1186/gb-2009-10-3-r25.
Robinson MD, McCarthy DJ, Smyth GK。edgeR:用于数字基因表达数据差异表达分析的Bioconductor包。生物信息学,2010,26(1):139 - 40。
张勇,刘涛,梅耶CA, Eeckhoute J, Johnson DS, Bernstein BE, Liu XS。基于模型的芯片序列分析(MACS)。基因组生物学,2008;9(9)。https://doi.org/10.1186/gb-2008-9-9-r137
Ramirez F, Ryan DP, Gruning B, Bhardwaj V, Kilpert F, Richter AS, Heyne S, Dundar F, Manke T. deepTools2:用于深度测序数据分析的下一代web服务器。核酸研究,2016;44:W160-5。
Robinson JT, Thorvaldsdóttir H, Winckler W, Guttman M, Lander ES, Getz G, Mesirov JP。整合基因组学查看器。中国生物工程学报。2011;29(1):24-6。https://doi.org/10.1038/nbt.1754.
Thorvaldsdottir H, Robinson JT, Mesirov JP。整合基因组Viewer (IGV):高性能基因组数据可视化和探索。生物信息学报。2013;14(2):178-92。https://doi.org/10.1093/bib/bbs017.
王志强,王志强。基于模因芯片的大型DNA数据集基序分析。生物信息学。2011;27:1696-7。
Heinz S, Benner C, Spann N, Bertolino E, Lin YC, Laslo P, Cheng JX, Murre C, Singh H, Glass CK。谱系决定转录因子的简单组合巨噬细胞和B细胞鉴定所需的主要顺式调节元件。生物化学学报,2010;38(4):576-89。https://doi.org/10.1016/j.molcel.2010.05.004.
Maere S, Heymans K, Kuiper M. BiNGO:一个评估生物网络中基因本体类别过度表示的Cytoscape插件。生物信息学,2005;21(16):3448 - 9。https://doi.org/10.1093/bioinformatics/bti551.
王志强,王志强,王志强,等。MeV基因序列分析。生物信息学。2011;27(22):3209 - 10。https://doi.org/10.1093/bioinformatics/btr490.
确认
我们感谢Doi纪子的技术支持和杉本实验室成员的有益评论和讨论。
资金
这项工作得到了美国国家科学基金会项目#1656265(给M.M.N.),美国农业部国家食品和农业研究所,Hatch Umbrella项目#1015621(给M.M.N.),以及日本科学促进协会(JSPS)研究员#19F19781(给D.S.F.)的资助。C.N.J.和D.S.F.都得到了jsp奖学金的支持。
作者信息
作者及隶属关系
贡献
C.N.J.和D.S.F.进行了大部分实验,分析了数据,创建了图表,并撰写了手稿。A.K.进行RNA提取,并为RNA-seq和ChIP-seq样品制备文库。T.S.对全基因组数据集进行了测序和初步分析。K.S.和M.M.N.与C.N.J.和D.S.F.一起构思了这项研究,并提供了监督,并对论文的写作和修改做出了重大贡献。所有作者都审阅了手稿。
相应的作者
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:
补充表1。来自于ZT4 (A)、ZT9 (B)和ZT24 (C)收获的sob3-6和sob3- 3幼苗的RNA-seq数据的CPM值和edgeR结果(为每个转录本提供)。
附加文件2:补充表2。
pif -靶基因的折叠变化2.Logfc表示sob3-6和sob3- 3幼苗基因表达差异为log2。高亮值表示在给定的时间点上,两种基因型之间差异调节的转录本,如p值(FDR-adjusted)所示p-value) < 0.05
附加文件3:补充表3。
使用MACS2从SOB3-GFP ChIP-seq数据中鉴定出的峰值,用于ZT4 (A-C)、ZT9 (D-F)和ZT24 (G-I)收获的幼苗。每个时间点来自三个不同生物重复的数据分别呈现。
附加文件4:补充表4。
基于ChIP-seq数据,SOB3分别与图2B或2C中所示的ahl抑制或ahl诱导的基因结合。* = SOB3绑定在一个代表;** =两组SOB3约束;*** = 3次被SOB3结合。蓝色高亮表示该基因在该时间点被算作SOB3结合基因(即至少2次ChIP-seq结合)。“binding uORF”表示尽管使用HOMER的注释没有检测到该基因与SOB3结合(即该基因的TSS不是与任何SOB3 ChIP-seq峰最接近的TSS),但位于该基因上游的uORF被识别为SOB3结合。
附加文件5:补充图1。
SOB3与BES1 (A)、CPD (B)、BBX22 (C)或PIF4 (D)相关的基因座结合,基于来自于ZT4、ZT9或ZT24收获的ProSOB3::SOB3- gfp SOB3- 4幼苗的ChIP-seq数据。图显示ChIP样品相对于各自输入对照的相对富集倍数。
附加文件6:补充图2。
根据SOB3::SOB3- gfp SOB3- 4幼苗在ZT4、ZT9或ZT24收获的ChIP-seq数据,SOB3与SOB3- 3和SOB3- 6的RNA-seq数据中仅在一个时间点被AHLs诱导或抑制的基因相对结合。(A) SOB3与仅在ZT4被AHLs抑制(左)或诱导(右)的基因的相对结合。(B) SOB3与仅在ZT9被AHLs抑制(左)或诱导(右)的基因相对结合。(C) SOB3与仅在ZT24被AHLs抑制(左)或诱导(右)的基因相对结合。
附加文件7:补充图3。
根据SOB3::SOB3- gfp SOB3- 4幼苗在ZT4、ZT9或ZT24收获的ChIP-seq数据,SOB3的相对结合与SOB3- 3和SOB3- 6的RNA-seq数据中仅在两个时间点被AHLs诱导或抑制的基因相结合。(A) SOB3与基因的相对结合被确定为在ZT4和ZT9被AHLs抑制(左)或诱导(右),但在ZT24没有。(B) SOB3与基因的相对结合被确定为在ZT4和ZT24被AHLs抑制(左)或诱导(右),但在ZT9没有。(C) SOB3与在ZT9和ZT24被AHLs抑制(左)或诱导(右)的基因相对结合,但在ZT4没有。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
雅克,c.n.,法弗罗,d.s.,川村,A。et al。抑制光敏色素B-4 #3减少pif激活基因的表达,增加生长抑制因子的表达,以调节下胚轴伸长。BMC植物生物学22, 399(2022)。https://doi.org/10.1186/s12870-022-03737-z
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-022-03737-z
关键字
- SOB3
- AHL
- 下胚轴
- 论坛
- SD
- 拟南芥