- 方法论的文章gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
用于植物多重基因组编辑的CRISPR/Cas9工具包gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba14gydF4y2Ba、物品编号:gydF4y2Ba327gydF4y2Ba(gydF4y2Ba2014gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
为了加速CRISPR/Cas9(聚集规律间隔短回文重复序列/ CRISPR相关蛋白9)系统在各种植物物种中的应用,需要一个包含额外植物可选择标记、更多gRNA模块和更容易组装一个或多个gRNA表达盒的方法的工具包。gydF4y2Ba
结果gydF4y2Ba
我们开发了基于pGreen或pCAMBIA主干的CRISPR/Cas9二元载体集,以及gRNA(向导RNA)模块载体集,作为植物多重基因组编辑的工具包。除此之外,这个工具包不需要限制性内切酶gydF4y2BaBsagydF4y2Ba生成包含玉米密码子优化的最终结构gydF4y2BaCas9gydF4y2Ba并且在一个克隆步骤中高效地克隆一个或多个grna。该工具包使用玉米原生质体、转基因玉米品系和转基因玉米进行了验证gydF4y2Ba拟南芥gydF4y2Ba并显示出高效率和特异性。更重要的是,使用这个工具箱,针对三个突变gydF4y2Ba拟南芥gydF4y2Ba在T1代转基因幼苗中检测到基因。此外,这种多基因突变可以遗传给下一代。gydF4y2Ba
结论gydF4y2Ba
我们开发了一个工具包,可以促进CRISPR/Cas9系统在多种植物物种中的瞬时或稳定表达,这将促进植物研究,因为它可以高效地产生携带多个基因突变的突变体。gydF4y2Ba
背景gydF4y2Ba
精确、高效的基因靶向或基因组编辑方法对于植物的功能基因组分析和基因工程作物的生产非常重要。对于大多数研究人员来说,转移DNA (T-DNA)和转座子插入突变仍然是模式植物(如dicot)中感兴趣的基因突变的主要来源gydF4y2Ba拟南芥gydF4y2Ba而单子稻(gydF4y2Ba栽培稻gydF4y2Ba) [gydF4y2Ba1gydF4y2Ba]、[gydF4y2Ba2gydF4y2Ba]。为了解剖具有冗余功能的基因家族成员的功能和分析遗传途径中的上位性关系,对多基因突变植物的需求日益增加。然而,目前产生携带多突变基因的植物的方法需要对单突变植物进行耗时且费力的遗传杂交。此外,T-DNA插入突变体不能为每个感兴趣的基因获得。因此,需要经济、高效、用户友好的新技术来实现植物基因组定位。gydF4y2Ba
特定基因组位点的双链断裂(DSBs)可以通过易出错的非同源末端连接(NHEJ)途径在DNA断裂位点引入突变。DSBs还可通过HR途径导致染色体DNA与外源供体DNA之间的同源重组(homologous recombination, HR) [gydF4y2Ba3.gydF4y2Ba]。基于靶座上的dsb,序列特异性核酸酶,包括归巢巨核酶、锌指核酸酶和转录激活因子样效应(TALE)核酸酶已经成为真核生物中靶向基因组编辑的强大技术[gydF4y2Ba3.gydF4y2Ba]。gydF4y2Ba
最近,另一项基于dsb的基因组编辑突破性技术CRISPR/Cas系统被开发出来[gydF4y2Ba4gydF4y2Ba]、[gydF4y2Ba5gydF4y2Ba]。该系统是基于细菌和古细菌聚集规律间隔短回文重复(CRISPR)适应性免疫系统,用于清除入侵的病毒和质粒DNA,其依赖于CRISPR相关(Cas)蛋白的内切酶活性,由CRISPR rna (crrna)指导序列特异性[gydF4y2Ba6gydF4y2Ba]-[gydF4y2Ba11gydF4y2Ba]。CRISPR/Cas系统,被应用于多种生物体,是源自于基因gydF4y2Ba酿脓链球菌gydF4y2BaII型CRISPR系统由三个基因组成,包括一个编码Cas9核酸酶的基因和两个非编码RNA基因:反式激活crRNA (tracrRNA)和前体crRNA (pre-crRNA)。可编程的pre-crRNA包含由相同的直接重复序列间隔的核酸酶引导序列(间隔序列),与tracrRNA结合加工成成熟的crRNA。这两个RNA基因可以被一个RNA基因取代,使用一个工程化的单导RNA (gRNA),其中包含一个设计的发夹,模仿crRNA-tracrRNA复合物。Cas9与靶DNA的结合特异性是由gRNA-DNA碱基配对和靶区下游邻近原间隔基序(PAM, sequence: NGG)决定的。Cas9的两个核酸酶结构域(HNH和RuvC-like)在同一位点(距离PAM 3个核苷酸[nt])切割一条双链DNA,导致DSB [gydF4y2Ba8gydF4y2Ba]-[gydF4y2Ba11gydF4y2Ba]。CRISPR/Cas系统已被用于在多种生物中实现高效的基因组编辑,包括细菌、酵母、植物和动物,以及人类细胞系[gydF4y2Ba12gydF4y2Ba]-[gydF4y2Ba27gydF4y2Ba]。更重要的是,利用这种rna引导的核酸内切酶技术,已经实现了多个基因突变及其种系传播[gydF4y2Ba28gydF4y2Ba]-[gydF4y2Ba30.gydF4y2Ba]。gydF4y2Ba
在斑马鱼、小鼠、大鼠和猴子等脊椎动物中,将体外转录的gRNA和编码cas9的mRNA共同注射到单细胞期胚胎中,可以高效地产生具有多个双等位基因突变的动物,这些突变可以高效地传递给下一代[gydF4y2Ba18gydF4y2Ba]、[gydF4y2Ba28gydF4y2Ba]-[gydF4y2Ba32gydF4y2Ba]。然而,这种方法在植物中是不可行的,在植物中,需要稳定表达CRISPR/Cas9系统的转基因系来产生具有一个或多个基因突变的植物。gydF4y2Ba农杆菌属gydF4y2Ba-介导转化是产生转基因植物的常规方法,目前已经开发出一些二元载体,通过这种方法将CRISPR/Cas9系统导入植物基因组[gydF4y2Ba15gydF4y2Ba]、[gydF4y2Ba20.gydF4y2Ba]、[gydF4y2Ba23gydF4y2Ba]、[gydF4y2Ba24gydF4y2Ba]、[gydF4y2Ba33gydF4y2Ba]-[gydF4y2Ba40gydF4y2Ba]。然而,为了加速该系统在正常或复杂条件下(如在T-DNA插入突变背景下基因的靶向突变)的各种植物物种中的应用,通常需要一个具有额外植物可选择标记的工具包,更多的gRNA模块,以及更容易组装一个或多个gRNA表达盒的方法,特别是对于多个基因的靶向突变。我们报告了这种用于植物多重基因组编辑的工具包的开发。gydF4y2Ba
结果gydF4y2Ba
植物多重基因组编辑的CRISPR/Cas9二元载体集和gRNA模块载体集gydF4y2Ba
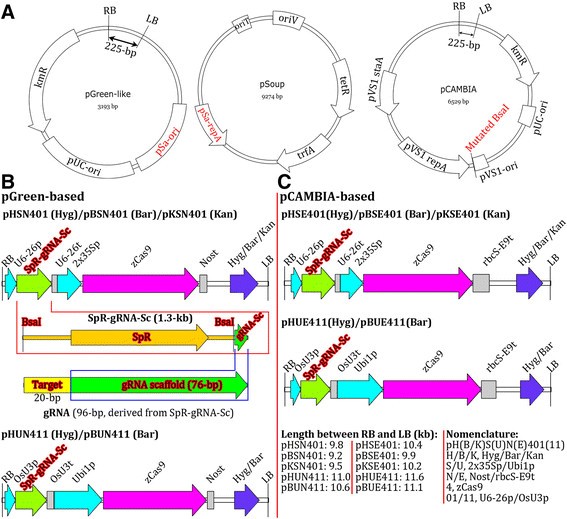
利用具有两种类型主干的二值向量;一种基于pGreen,另一种基于pCAMBIA。pGreen二值向量是基于先前报道的策略构建的[gydF4y2Ba41gydF4y2Ba]。pGreen-like载体的优势在于其相对较小的尺寸,允许它们用于原生质体中Cas9和gRNA的瞬时表达,以测试目标位点的有效性。由于这些载体在原生质体中验证后可直接用于产生转基因植株,因此使用这种基于单一载体的策略来实现CRISPR/Cas9的瞬时和稳定表达可以节省时间、精力和金钱。在gydF4y2Ba农杆菌属gydF4y2Ba在美国,pgreen样载体依靠其pSa来源进行繁殖,它们需要一个辅助质粒来提供复制蛋白(RepA)。gydF4y2Ba农杆菌属gydF4y2Ba含有pSoup辅助质粒可作为pgreen样载体的宿主[gydF4y2Ba41gydF4y2Ba]。在pcambia衍生的二元载体中,以潮霉素耐药基因为选择标记的载体来源于pCAMBIA1300,卡那霉素耐药基因来源于pCAMBIA2300, basta耐药基因来源于pCAMBIA3300。载体pCAMBIA1300/2300/3300及其衍生物(包括gateway兼容的pMDC系列)是一些最广泛使用的用于各种植物物种的二进制载体[gydF4y2Ba42gydF4y2Ba]、[gydF4y2Ba43gydF4y2Ba],一些植物转化方案已经根据这些载体进行了专门优化。因此,基于pcambia的CRISPR/Cas9二元载体的生成增强了这些载体与一些优化的植物转化方案和/或一些研究人员的习惯或偏好的兼容性。每个pcambia衍生载体的一个重要改进是gydF4y2BaBsagydF4y2BapVS1区的I位点(这是质粒在gydF4y2Ba农杆菌属gydF4y2Ba)被中断,以便能够使用gydF4y2BaBsagydF4y2Ba1个组装gRNA表达盒的位点(图2)gydF4y2Ba1gydF4y2Ba)。gydF4y2Ba
CRISPR/Cas9二元载体的物理图谱和结构。(一)gydF4y2Ba导出CRISPR/Cas9二值载体的pGreen和pCAMBIA的主干物理图谱。pGreen在细胞中繁殖所需的辅助质粒图gydF4y2Ba农杆菌属gydF4y2Ba而变异的gydF4y2BaBsagydF4y2BapCAMBIA骨干网上的1个位点。LB/RB, T-DNA左/右边界;中的复制所需的pSa-origydF4y2Ba农杆菌属gydF4y2Ba用相应的复制蛋白(pSa-repA)工程化;KmR,卡那霉素抗性基因;pu -ori,复制所需的复制源gydF4y2Ba大肠杆菌gydF4y2Ba;pVS1-staA, pVS1-ori和pVS1-rep是在细胞内复制所需的DNA元件gydF4y2Ba农杆菌属gydF4y2Ba。只留下LB和RB之间的225 bp片段,用于比较pGreen和pCAMBIA主干的大小。gydF4y2Ba(B, C)gydF4y2BaRB和LB之间区域的物理图谱。T-DNA区域的大小以及pr -gRNA- sc和最终工作gRNA的结构。gydF4y2BazCas9gydF4y2Ba,gydF4y2Ba玉米gydF4y2Bacodon-optimizedgydF4y2BaCas9gydF4y2Ba;U6-26p,gydF4y2Ba拟南芥U6gydF4y2Ba基因启动子;U6-26t,gydF4y2BaU6-26gydF4y2Ba终止与下游序列;OsU3p、大米gydF4y2BaU3gydF4y2Ba启动子;OsU3t、大米gydF4y2BaU3gydF4y2Ba终止与下游序列;SpR,大霉素耐药基因;gRNA- sc, gRNA支架。gydF4y2Ba
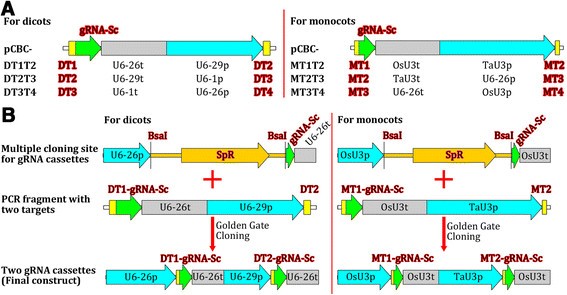
为了将多个gRNA整合到一个二元载体中进行多重基因组编辑,我们构建了6个gRNA模块载体,其中3个为双子叶植物设计,3个为单子叶植物设计(图2)gydF4y2Ba2gydF4y2Ba)。利用这些gRNA模块载体,利用金门克隆法可以很容易地组装两个或多个gRNA表达盒[gydF4y2Ba44gydF4y2Ba]、[gydF4y2Ba45gydF4y2Ba]或吉布森组装法[gydF4y2Ba46gydF4y2Ba]。通过使用更多合适的Pol III启动子,可以构建额外的gRNA模块,以组装更多的gRNA表达盒。因此,gRNA模块向量集具有可扩展性,易于更新。gydF4y2Ba
预制gRNA模块,用于装配2 ~ 4个gRNA表达盒。(一)gydF4y2Ba双子叶和单子叶的grna表达模块。U6-29p, U6-26p和U6-1p是三个gydF4y2Ba拟南芥U6gydF4y2Ba基因启动子;对应U6-29t、U6-26t和U6-1tgydF4y2Ba拟南芥U6gydF4y2Ba具有下游序列的基因终止子;OsU3p和TaU3p,水稻和小麦gydF4y2BaU3gydF4y2Ba分别启动子;OsU3t和TaU3t,水稻和小麦gydF4y2BaU3gydF4y2Ba分别具有下游序列的终止子;gRNA- sc, gRNA支架;DT1/2/3/4, dicot目标-1/2/3/4;MT1/2/3/4,单片目标-1/2/3/4。载体pCBC是分别插入gRNA模块的克隆载体。gydF4y2Ba(B)gydF4y2Ba使用gRNA模块组装双gRNA表达盒的例子。注:每个PCR片段两侧有两个gydF4y2BaBsagydF4y2BaI个站点(未显示)。gydF4y2Ba
CRISPR/Cas9工具箱在玉米原生质体中的验证gydF4y2Ba
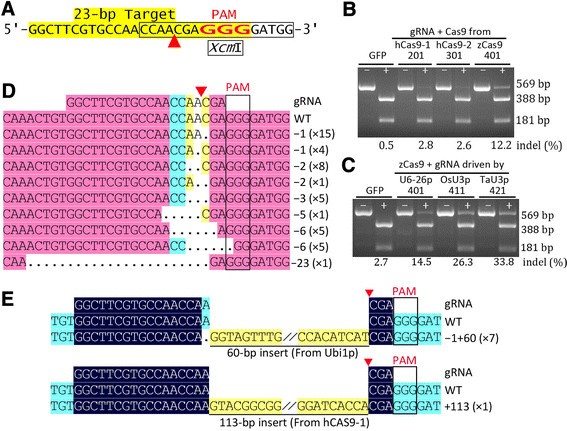
验证该工具箱并比较不同方法的突变效率gydF4y2BaCas9gydF4y2Ba或用于驱动gRNAs的Pol III启动子,我们生成了两组针对同一玉米基因组DNA位点(gydF4y2BaZmHKT1gydF4y2Ba)。pBUN201-ZT1、pBUN301-ZT1、pBUN401-ZT1为一组,三个型号不同gydF4y2BaCas9gydF4y2Ba序列,包括gydF4y2BahCas9-NLS-3 × FLAGgydF4y2Ba在pBUN201-ZT1,gydF4y2Ba3 × FLAG-NLS-hCas9-NLSgydF4y2BapBUN301-ZT1和gydF4y2Ba3 × FLAG-NLS-zCas9-NLSgydF4y2Ba在pBUN401-ZT1。的gydF4y2BahCas9gydF4y2Ba和gydF4y2BazCas9gydF4y2Ba序列是人类密码子和gydF4y2Ba玉米gydF4y2Ba密码子优化gydF4y2BaCas9,gydF4y2Ba分别。另一组包括pBUN401-ZT1、pBUN411-ZT1和pBUN421-ZT1。这些载体根据用于驱动gRNA的Pol III启动子的不同而不同:pBUN401-ZT1中的AtU6-26p, pBUN411-ZT1中的OsU3p和pBUN421-ZT1中的TaU3p。gydF4y2Ba
对于目标站点gydF4y2BaZT1gydF4y2Ba,突变等位基因通过gydF4y2BaXcmgydF4y2Ba对假定的裂解位点周围的PCR片段进行酶切(图2)gydF4y2Ba3.gydF4y2Ba一个)。gydF4y2BaXcmgydF4y2BaI分析表明玉米密码子优化gydF4y2BaCas9gydF4y2Ba性能明显优于两个人类密码子优化gydF4y2BaCas9gydF4y2Ba基因(图gydF4y2Ba3.gydF4y2BaB)。gydF4y2BaTaU3gydF4y2Ba推动者的表现似乎比gydF4y2BaOsU3gydF4y2Ba发起人,以及gydF4y2BaOsU3gydF4y2Ba启动器的性能比gydF4y2BaAtU6-26gydF4y2Ba启动子(图gydF4y2Ba3.gydF4y2BaC)。gydF4y2Ba
玉米密码子优化的Cas9和三个Pol-III启动子在玉米原生质体中驱动gRNA表达的验证(一)gydF4y2Ba来自Zm的目标位点序列gydF4y2BaHKT1gydF4y2Ba轨迹。PAM,假定的裂解位点(红色箭头),和gydF4y2BaXcmgydF4y2Ba1个站点(方框)被指示。gydF4y2Ba(B, C)gydF4y2Ba突变分析gydF4y2BaXcmgydF4y2Ba1 . PCR片段的酶切。绿色荧光,2011,301,401gydF4y2Ba(B)gydF4y2Ba:从分别转染了pcu - gfp(对照)、pBUN201-ZT1、pBUN301-ZT1和pBUN401-ZT1的玉米原生质体基因组DNA中扩增出PCR片段。这三种CRISPR/Cas9载体具有相同但不同的gRNAgydF4y2BaCas9gydF4y2Ba:gydF4y2BahCas9-1gydF4y2Ba/gydF4y2Ba2gydF4y2Ba两种类型的人类密码子优化gydF4y2BaCas9gydF4y2Ba;gydF4y2BazCas9gydF4y2Ba,gydF4y2Ba玉米gydF4y2Bacodon-optimizedgydF4y2BaCas9gydF4y2Ba。绿色荧光,401,411,421gydF4y2Ba(C)gydF4y2Ba: pucc - gfp、pBUN401-ZT1、pBUN411-ZT1和pBUN421-ZT1分别转染的PCR片段;三个CRISPR/Cas9载体具有相同的zgydF4y2BaCas9gydF4y2Ba和gRNA,但gRNA是由三种不同的Pol-III启动子驱动的。−和+表示PCR片段是否被酶切gydF4y2BaXcmgydF4y2BaI.突变效率(% indel)基于剩余未消化PCR片段(+ lanes: 569 bp)与总PCR产物(−lanes)的百分比比率计算;WT indel值应被视为背景电平。gydF4y2Ba(D, E)gydF4y2Ba抗性克隆PCR片段突变等位基因序列比对gydF4y2BaXcmgydF4y2Ba我消化。突变的等位基因包括缺失gydF4y2Ba(D)gydF4y2Ba和插入gydF4y2Ba(E)gydF4y2Ba。点,删除的碱基。高亮表示对齐片段的同源程度,只显示感兴趣的对齐区域。显示索引的类型和相同类型的索引的数量。gydF4y2Ba
为了验证突变事件,对PCR产物进行克隆,并对所得到的菌落进行集落PCR和筛选gydF4y2BaXcmgydF4y2Ba菌落PCR产物的消化。从其菌落PCR产物具有抗性的克隆中提取DNAgydF4y2BaXcmgydF4y2BaI酶解测序(图2)gydF4y2Ba3.gydF4y2Ba有趣的是,我们获得了8个插入突变,其中一个来自于gydF4y2BahCas9gydF4y2Ba7个来自于泛素启动子,这可能是来自于降解的载体而不是玉米基因组(图2)gydF4y2Ba3.gydF4y2BaE).这些结果表明,当提供供体基因时,靶向整合的效率相对较高。gydF4y2Ba
CRISPR/Cas9工具箱在转基因玉米中的验证gydF4y2Ba
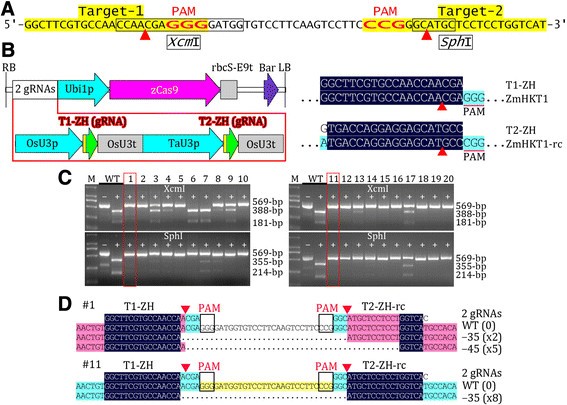
为了测试该工具包在单子房中的靶向突变效率,我们生成了一个pcambia衍生的CRISPR/Cas9二元载体,该载体具有两个gRNA表达盒,靶向同一玉米基因的两个相邻位点。gydF4y2BaZmHKT1gydF4y2Ba(图gydF4y2Ba4gydF4y2BaA, B)。我们通过限制性内切酶酶切跨越两个目标位点的PCR片段,分析了20个T0转基因系,发现超过60%的转基因系对两个目标位点的突变效率都接近100%(图2)gydF4y2Ba4gydF4y2BaC).我们克隆了两条序列的PCR片段,并对其进行了测序,突变效率约为100%,发现两个目标位点之间的序列被删除,如图所示gydF4y2Ba4gydF4y2BaD.这些结果表明,该工具箱可以用于玉米和其他作物的高效靶向突变。gydF4y2Ba
通过玉米基因的靶向突变验证该工具包。(一)gydF4y2Ba玉米区域序列gydF4y2BaZmHKT1gydF4y2Ba有两个目标地点。gydF4y2Ba(B)gydF4y2Ba携带两个grna表达磁带的T-DNA物理图谱。图中显示了gRNA的靶基因与靶基因的排列关系。只显示感兴趣的对齐区域。rc,反向补码。gydF4y2Ba(C)gydF4y2Ba20个T0转基因系的突变分析gydF4y2BaXcmgydF4y2Ba我或gydF4y2BaSphgydF4y2Ba1 . PCR片段的酶切。用于测序分析的细胞系用方框表示。gydF4y2Ba(D)gydF4y2Ba从两个具有代表性的T0转基因株系克隆PCR片段中鉴定的突变等位基因序列比对。高亮表示对齐片段的同源程度,只显示感兴趣的对齐区域。显示相同类型的索引的数量。gydF4y2Ba
CRISPR/Cas9工具箱的验证gydF4y2Ba拟南芥gydF4y2Ba用于产生具有多个基因突变的突变体gydF4y2Ba
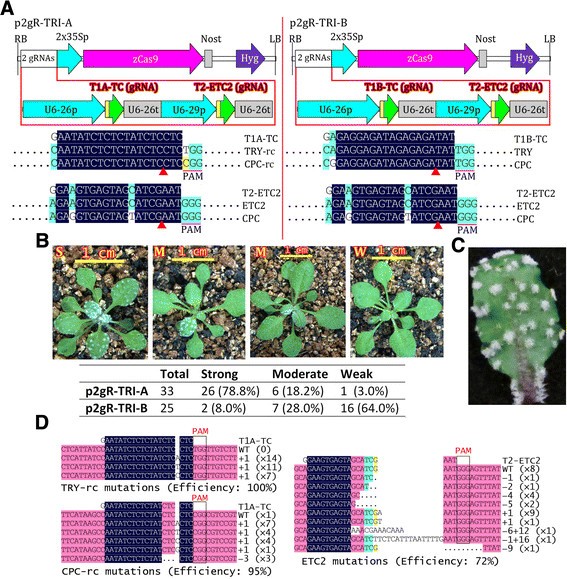
两个向量,p2gR-TRI-A和p2gR-TRI-B(图gydF4y2Ba5gydF4y2BaA),每个携带两个grna靶向三个与毛状体发育相关的基因,用于转化gydF4y2Ba拟南芥gydF4y2Ba。两种载体含有相同的gRNA (T2-ETC2),其目标是gydF4y2BaETC2gydF4y2Ba并有可能gydF4y2Ba中国共产党gydF4y2Ba这是一个不太有利的目标(图1)gydF4y2Ba5gydF4y2BaA).载体也含有不同的grna (T1A-TC或T1B-TC)。T1A-TC的18 bp靶序列与T1B-TC的18 bp靶序列呈反向互补关系。T1A-TC和T1B-TC都针对相同的两个基因:gydF4y2Ba试一试gydF4y2Ba和gydF4y2Ba中国共产党gydF4y2Ba(图gydF4y2Ba5gydF4y2BaA). T1A-TC gRNA的20-nt靶标与gydF4y2Ba试一试gydF4y2Ba或gydF4y2Ba中国共产党gydF4y2Ba介于T1B-TC和gydF4y2Ba试一试gydF4y2Ba,而T1B-TC与gydF4y2Ba中国共产党gydF4y2Ba(图gydF4y2Ba5gydF4y2BaA)。对于p2gR-TRI-A,超过70%的T1转基因植株显示出高度聚集的毛状体(图2)gydF4y2Ba5gydF4y2BaB,C,正如所预料的gydF4y2Ba试着中国共产党gydF4y2Ba两倍或gydF4y2Ba尝试CPC等gydF4y2Ba三突变体植物[gydF4y2Ba47gydF4y2Ba]。对于p2gR-TRI-B,只有不到10%的植株表现出预期的表型,这表明T1B-TC的性能水平远不如T1A-TC。对p2gR-TRI-B T1转基因品系突变等位基因的测序结果显示,尽管p2gR-TRI-B T1转基因品系的突变效率较高gydF4y2Ba试一试gydF4y2Ba等位基因占90%以上gydF4y2Ba中国共产党gydF4y2Ba由相同的T1B-TC gRNA导致的死亡率仅为42%(附加文件)gydF4y2Ba1gydF4y2Ba:图S1)。相比之下,两者gydF4y2Ba中国共产党gydF4y2Ba和gydF4y2Ba试一试gydF4y2Ba尽管两个靶点之间存在不同的PAMs,但同一T1A-TC gRNA靶向的突变频率相似(大于90%)(图2)gydF4y2Ba5gydF4y2BaA, D)。这些结果表明,这两个错配可能解释了T1B-TC性能差的原因,尽管这两个错配位于gRNA的5 '端。此外,当T2-ETC2 gRNA的20-nt靶序列与靶基因存在3次错配时gydF4y2Ba中国共产党gydF4y2Ba(图gydF4y2Ba5gydF4y2BaA),在p2gr - tra转基因系的100多个克隆中未检测到突变。相比之下,gydF4y2BaETC2gydF4y2Ba来自同一T2-ETC2 gRNA的突变效率为72%(图2)gydF4y2Ba5gydF4y2BaD,这些结果表明gydF4y2Ba在足底gydF4y2BaCRISPR/Cas9系统具有较高的序列特异性,两次或两次以上的错配会大大降低靶向效率和脱靶效应,特别是当错配位于gRNA的20 nt靶标的3 '端附近时。gydF4y2Ba
CRISPR/Cas工具箱的验证gydF4y2Ba拟南芥gydF4y2Ba。(一)gydF4y2Ba两个pgreen衍生的CRISPR/Cas9二元载体的t - dna物理图谱,每个载体携带两个靶向三个的grnagydF4y2Ba拟南芥gydF4y2Ba基因(gydF4y2Ba试一试gydF4y2Ba,gydF4y2Ba中国共产党gydF4y2Ba和gydF4y2BaETC2gydF4y2Ba)。图中显示了gRNA与其靶基因的排列。只显示感兴趣的对齐区域。rc,反向补码。gydF4y2Ba(B)gydF4y2Bap2gr - tra - T1转基因系的代表性表型。S,强表型相似gydF4y2Ba尝试CPC等gydF4y2Ba三突变体,具高度簇生的毛状体在叶片和叶柄上;M,中度表型,部分叶片或部分叶片显示表型gydF4y2Ba试着中国共产党gydF4y2Ba双突变体或三突变体;W,表型弱或无突变的植株。T1转基因植株的总数,T1转基因植株表现出强、中、弱表型的数量,以及占总数的百分比(括号内)。T0种子在湿霉素MS板上筛选13天,在土壤中培养10天,然后拍照。gydF4y2Ba(C)gydF4y2Ba在叶柄上显示高度聚集的毛状体的离体叶片的放大图像,这是相似的表型gydF4y2Ba尝试CPC等gydF4y2Ba三突变体。gydF4y2Ba(D)gydF4y2Ba典型p2gr - tra系靶基因突变的测序分析。点,删除的碱基。高亮表示对齐片段的同源程度。显示索引的类型和相同类型的索引的数量。gydF4y2Ba
在一株转基因植物中有许多不同的突变等位基因(图2)gydF4y2Ba5gydF4y2BaD),这表明CRISPR/Cas9在受精卵分裂后起作用。为了证实突变在T2植株中的种系传播是可能的,我们检测了5个具有强表型的p2gr - tra T1转基因株系的突变传播。由于Cas9和gRNA在T2转基因细胞系中组成性表达,因此有时很难确定突变的来源,这些突变可能来自生殖细胞或体细胞。相反,从分离的T2非转基因植株中产生的突变必须是由T1植株的突变的种系传播引起的。因此,我们将重点放在分离的非转基因T2植株上,以简化突变的种系传播分析。用PCR反选择方法鉴定了非转基因植株,其中两对引物为潮霉素抗性基因,一对引物为潮霉素抗性基因gydF4y2BaCas9gydF4y2Ba。我们确定了两者的双等位基因突变gydF4y2Ba试一试gydF4y2Ba和gydF4y2Ba中国共产党gydF4y2Ba基于它们的簇状毛状体表型,发现gydF4y2Ba试一试gydF4y2Ba和gydF4y2Ba中国共产党gydF4y2Ba突变高效传递到T2植株;来自5个T1系的非转基因T2植株分别为46.2%、100%、82.6%、100%和100%的双等位突变体gydF4y2Ba试一试gydF4y2Ba和gydF4y2Ba中国共产党gydF4y2Ba(表gydF4y2Ba1gydF4y2Ba)。我们首先分析gydF4y2BaETC2gydF4y2Ba通过直接测序PCR产物或测序不同克隆的PCR产物,分析非转基因T2双突变植株的基因突变gydF4y2Ba试一试gydF4y2Ba和gydF4y2Ba中国共产党gydF4y2Ba突变的gydF4y2Baetc2gydF4y2Ba在第一步分析中验证的突变体(表1)gydF4y2Ba2gydF4y2Ba)。我们发现这是经过验证的gydF4y2Ba尝试CPC等gydF4y2Ba三突变体很容易被区分gydF4y2Ba试着中国共产党gydF4y2Ba双突变体;前者比后者短,叶片向上卷曲(图1)gydF4y2Ba6gydF4y2Ba)。双等位T2突变体gydF4y2BaETC2gydF4y2Ba在检测的5个品系中,仅从2个T1品系中分离(表gydF4y2Ba1gydF4y2Ba),证明该病毒的种系传播频率gydF4y2BaETC2gydF4y2Ba突变进入T2植株的比例远低于gydF4y2Ba试一试gydF4y2Ba和gydF4y2Ba中国共产党gydF4y2Ba突变。这一结果可以用突变频率较低来解释gydF4y2BaETC2gydF4y2Ba在T1植株中gydF4y2Ba
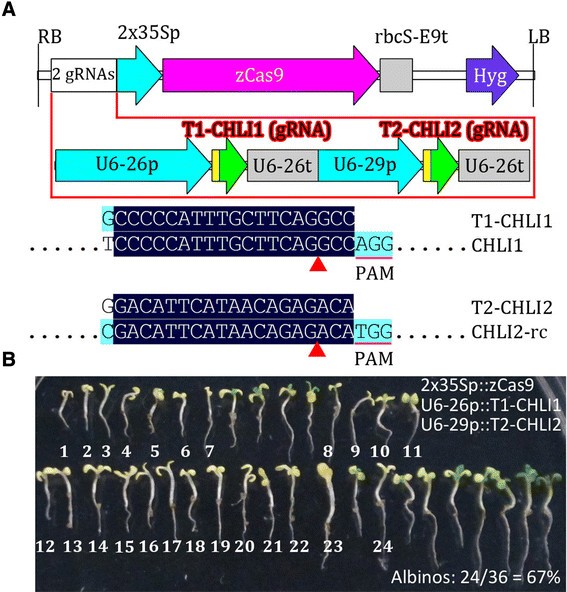
中进一步验证工具包gydF4y2Ba拟南芥gydF4y2Ba,我们构建了一个基于pcambia的载体pHSE-2gR-CHLI,携带两个靶向gydF4y2BaCHLI1gydF4y2Ba和gydF4y2BaCHLI2gydF4y2Ba(图gydF4y2Ba7gydF4y2Ba),与先前研究中使用的grna相同[gydF4y2Ba22gydF4y2Ba]。同时中断gydF4y2BaCHLI1gydF4y2Ba和gydF4y2BaCHLI2gydF4y2Ba导致白化表型,而gydF4y2Bachli1gydF4y2Ba或gydF4y2Bachli2gydF4y2Ba单突变体表现为淡绿色表型[gydF4y2Ba48gydF4y2Ba]。T1转基因比例较高gydF4y2Ba拟南芥gydF4y2Ba幼苗显示白化表型(24/36 = 67%)比之前报道的(23/60 = 38%),进一步证明该工具包适用于gydF4y2Ba拟南芥gydF4y2Ba。通过使用两个或更多grna靶向同一基因的两个或更多靶点,可以进一步提高突变频率。随着突变效率的提高,体细胞突变可以更有效地传递给下一代。因此,本研究开发的工具包可用于生成gydF4y2Ba拟南芥gydF4y2Ba具有高效率和特异性的突变体。gydF4y2Ba
讨论gydF4y2Ba
解剖具有冗余功能的基因家族成员的功能和分析遗传途径中的上位性关系,往往需要植物突变体携带多个基因突变。最近开发的CRISPR/Cas9系统为基因组编辑提供了一种极好的方法[gydF4y2Ba4gydF4y2Ba]、[gydF4y2Ba9gydF4y2Ba]、[gydF4y2Ba21gydF4y2Ba]。然而,为了在植物中产生多个基因突变,往往需要多个gRNA表达盒组装的资源和方法。在本报告中,我们描述了用于生成gRNA模块和使用预制gRNA模块组装多个gRNA表达盒的方法。这些资源包括二进制载体和gRNA模块载体,能够满足在正常或复杂条件下多种植物中使用的大部分要求。这些方法还允许研究人员定制自己的gRNA模块,并组装多个gRNA表达盒进行多重基因组编辑。使用该试剂盒,我们发现CRISPR/Cas9可以同时敲除多个植物基因,并且根据经济学中的“桶效应”理论,多基因突变的效率取决于目标基因的最低突变效率。gydF4y2Ba
在植物中使用CRISPR/Cas9需要二元载体。为了将20bp的目标序列融合到gRNA支架的5 '端,最好使用IIs型限制性内切酶。尽管一些ii型限制性内切酶,比如gydF4y2BaAragydF4y2Ba我,gydF4y2Ba论坛gydF4y2Ba我/gydF4y2BaBpigydF4y2Ba我,gydF4y2BaBsagydF4y2Ba我/gydF4y2Ba生态gydF4y2Ba31日我gydF4y2BaBsmgydF4y2BaBI /gydF4y2BaEspgydF4y2Ba3,我gydF4y2BaBspgydF4y2BaMI /gydF4y2BaBfugydF4y2BaAI /gydF4y2BaBvegydF4y2Ba我和gydF4y2BaBtggydF4y2BaZI是市售的,很少有这样的酶可以用于线性化常用的二元载体,如pCAMBIA系列和pPZP系列载体[gydF4y2Ba43gydF4y2Ba]、[gydF4y2Ba49gydF4y2Ba],因为在这些载体的主干中存在一个或多个站点。例如,不包括T-DNA区域,pCAMBIA主干包含一个gydF4y2BaBsagydF4y2Ba我,两个gydF4y2Ba论坛gydF4y2Ba我,两个gydF4y2BaBsmgydF4y2BaBI,两个gydF4y2BaBspgydF4y2BaMI和4gydF4y2BaBtggydF4y2Ba子站点。虽然没有gydF4y2BaAargydF4y2Ba我可以在pCAMBIA骨干网中找到一个位点gydF4y2BaAargydF4y2Ba我的网站gydF4y2Ba酒吧gydF4y2BapCAMBIA3300 T-DNA区域的选择性标记基因。幸运的是,尽管存在gydF4y2BaBsagydF4y2BaI位点位于pVS1复制区,这是质粒在gydF4y2Ba农杆菌属gydF4y2Ba,没有。gydF4y2BaBsagydF4y2BaI网站中常用的元素包括双启动子等gydF4y2BaCaMV 35 sgydF4y2Ba发起人和gydF4y2BaUbi1gydF4y2Ba启动子,或可选择的标记,包括gydF4y2Ba菅直人gydF4y2Ba,gydF4y2BaHyggydF4y2Ba和gydF4y2Ba酒吧gydF4y2Ba。此外,gydF4y2BaBsagydF4y2BaI是常用的I型限制性内切酶中最便宜的。例如,每活动单位的价格gydF4y2BaBsagydF4y2Ba我/gydF4y2Ba生态gydF4y2Ba大约是的1/50gydF4y2BaAargydF4y2Ba(赛默飞世尔科学和新英格兰生物实验室)。利用gydF4y2BaBsagydF4y2Ba为了将gRNA表达盒组装成pCAMBIA二元载体,我们破坏了gydF4y2BaBsagydF4y2BapVS1区的I位点。因此,对于我们开发的二元载体集,没有限制性内切酶,但gydF4y2BaBsagydF4y2BaI是一个或多个grna组装所必需的。gydF4y2Ba
该工具包提供了生成植物CRISPR/Cas9二进制载体的最简单方法。当构建携带一个或两个grna的二元载体时,只需要两个23-nt合成寡核苷酸(退火为插入物)或PCR片段,以及本报告中描述的任何二元载体,就可以建立金门反应。当构建携带多个grna的二元载体时,需要两个或多个PCR片段。基于金门克隆法[gydF4y2Ba45gydF4y2Ba]或Gibson Assembly [gydF4y2Ba46gydF4y2Ba],两个或多个PCR片段可以很容易地组装成多个gRNA表达盒gydF4y2BaBsagydF4y2Bai -线性化的二进制矢量,仅在一个克隆步骤。装配四个以上的gRNA表达盒可采用两种策略,即生成更多带有额外经过验证的Pol III启动子的gRNA模块,以及(第二次)将含有大霉素耐药基因的gRNA表达盒插入已经包含四个gRNA的二元载体中,然后将额外的gRNA组装到二进制载体中gydF4y2BaBsagydF4y2BaI-linearized向量。因此,二元载体集结合gRNA模块载体集组成了一个高效、廉价、省时、用户友好、多方面、可扩展的工具包,用于生成携带一个或多个gRNA的CRISPR/Cas9二元载体,用于多个基因的靶向突变。gydF4y2Ba
结论gydF4y2Ba
我们开发了一个基于CRISPR/ cas9的二进制载体集和一个gRNA模块载体集,作为植物多重基因组编辑的工具包。我们用玉米原生质体、玉米转基因株系和转基因玉米株系验证了该试剂盒gydF4y2Ba拟南芥gydF4y2Ba结果表明,该方法具有较高的效率和特异性。该二元载体集与gRNA模块载体集相结合,构成了一个高效、廉价、省时、用户友好、多方面、可扩展的工具包,用于生成携带一种或多种gRNA的CRISPR/Cas9二元载体,用于多种植物基因的靶向突变。该工具包可促进CRISPR/Cas9在多种植物系统中的瞬时或稳定表达,可应用于多种植物,尤其适用于高效生成携带多基因突变的突变体。gydF4y2Ba
方法gydF4y2Ba
向量构造gydF4y2Ba
矢量构造的详细描述在附加文件中提供gydF4y2Ba2gydF4y2Ba方法:本报告中使用的所有引物都列在附加文件中gydF4y2Ba1gydF4y2Ba表S1。gydF4y2Ba
金门方法构建表达一个或两个grna的载体gydF4y2Ba
为了组装一个gRNA,将等体积的100 μmol/L寡核苷酸1和寡核苷酸2混合,在65℃下孵育5分钟,然后缓慢冷却至室温,得到两端有4-nt 5 '悬垂的双链插入物。为了组装两个grna,将两个目标位点分别纳入PCR正向和反向引物中。PCR片段以pCBC-DT1T2为双子型靶扩增,以pCBC-MT1T2为单子型靶扩增,分别用2条长引物或4条短引物扩增,其中2条正向引物或2条反向引物部分重叠。插入物或纯化的PCR片段(T1T2-PCR)与本报告中描述的任何二元载体一起用于建立限制性结扎反应,如其他地方所述[gydF4y2Ba44gydF4y2Ba),用gydF4y2BaBsagydF4y2BaI和T4连接酶(新英格兰生物实验室)。反应在热循环器中37°C孵育5小时,50°C孵育5分钟,80°C孵育10分钟。详细信息包括gRNA模块序列,PCR引物,菌落PCR引物和测序引物可以在附加文件中找到gydF4y2Ba3.gydF4y2Ba方法:gydF4y2Ba
金门克隆法或吉布森组装法生成表达3或4个grna的载体gydF4y2Ba
有两种方法可以组装3个以上的grna: Golden Gate Cloning [gydF4y2Ba45gydF4y2Ba]及Gibson Assembly [gydF4y2Ba46gydF4y2Ba]。金门克隆时,纯化两条(T1-PCR和T2T3-PCR2)或三条(T1-PCR、T2-PCR和T3T4-PCR2) PCR片段,与任一CRISPR/Cas9二元载体混合,建立如上所述的金门反应。对于Gibson Assembly,纯化两个(T1T2-PCR和T2T3-PCR)或三个(T1T2-PCR, T2T3-PCR和T3T4-PCR) PCR片段,并与Gibson Assembly Master Mix (New England Biolabs)混合,根据制造商的方案建立反应。将所需大小的片段进行凝胶纯化,并用作第二轮PCR扩增的PCR模板。将第二轮PCR的产物纯化,并与本报告中描述的任何二元载体混合,建立如上所述的金门反应。详细信息包括gRNA模块序列,PCR引物,菌落PCR引物和测序引物可以在附加文件中找到gydF4y2Ba4gydF4y2Ba方法S3和附加文件gydF4y2Ba5gydF4y2Ba方法:融合PCR方法也用于组装三个以上的grna;然而,由于持续的非特异性扩增,第二轮PCR的效率有时会大大降低。gydF4y2Ba
玉米原生质体的分离与转染gydF4y2Ba
B73玉米种子在无菌水中浸泡过夜,播种于土壤中,在生长室内22°C光照16 h /黑暗8 h,生长4-6天。根据先前描述的方法,将20-30株幼苗的茎和鞘组织用于原生质体分离[gydF4y2Ba50gydF4y2Ba],进行一次修饰,即在100 ×离心下收集原生质体微球gydF4y2BaggydF4y2Ba3分钟。peg介导的转染按照描述[gydF4y2Ba50gydF4y2Ba]。每个样品10 - 15 μg质粒DNA与200 μL原生质体混合(约2 × 10gydF4y2Ba5gydF4y2Ba细胞)。加入新鲜配制的PEG溶液(200 μL),室温黑暗孵育10-20 min。随后,加入800 μL W5溶液混合,在100 ×离心下将原生质体成球gydF4y2BaggydF4y2Ba将原生质体重悬于1.5 mL W5溶液中,100 ×离心成球gydF4y2BaggydF4y2Ba将原生质体在800 μL W5溶液中重悬,22℃暗室培养14-16 h。每个质粒转染3次。gydF4y2Ba
玉米原生质体突变的验证gydF4y2Ba
将来自同一载体的3个转染原生质体样本合并,提取基因组DNA。利用两对基因特异性引物ZT-IDF0/-IDR0和ZT-IDF/-IDR(附加文件),通过巢式PCR从基因组DNA中扩增出包含CRISPR靶位点的DNA片段gydF4y2Ba1gydF4y2Ba:表1)。对于突变的限制性内切酶酶切分析,每个PCR产物建立两个限制性内切酶反应:在一个反应中加入相应的限制性内切酶;在另一个反应中,酶被水代替作为阴性对照。每个反应纯化的PCR产物约500 ng,在20 μ l反应中消化过夜。与对照组一起,用2.0%溴化乙啶染色琼脂糖凝胶分离消化的DNA。将纯化后的PCR产物克隆到克隆载体pCBC中,进行集落PCR鉴定,并进行限制性内切酶酶切分析。一些限制性内切酶,比如gydF4y2BaXcmgydF4y2Ba我和gydF4y2BaSphgydF4y2Ba我有活动gydF4y2BaTaqgydF4y2Ba聚合酶链反应混合物。PCR结束后,将酶加入到PCR混合物中进行隔夜消化,然后进行琼脂糖凝胶电泳分析。利用T7引物对抗消化片段进行测序。gydF4y2Ba
转基因玉米的产生及突变分析gydF4y2Ba
将CRISPR/Cas9二进制载体pBUE-2gRNA-ZH转化为gydF4y2Ba农杆菌属gydF4y2Ba菌株EHA105gydF4y2Ba农杆菌属gydF4y2Ba在中国农业大学转基因设施中心,采用-介导法转化B73玉米未成熟胚。从20株转基因苗中提取基因组DNA,得到PCR片段,引物及反应与前文相同。为了进行限制性内切酶酶切分析,每个反应纯化的PCR产物约500 ng用gydF4y2BaXcmgydF4y2Ba我或gydF4y2BaSphgydF4y2BaI在20 μ l反应体积下。测序分析,将2个代表性转基因苗的PCR产物克隆到克隆载体pCBC中,用T7引物对阳性克隆进行测序。gydF4y2Ba
转基因的产生gydF4y2Ba拟南芥gydF4y2Ba植物与突变分析gydF4y2Ba
将p2gR-TRI-A和p2gR-TRI-B载体转化为gydF4y2Ba农杆菌属gydF4y2Ba菌株GV3101/pSoup采用冻融法,pHSE-2gR-CHLI转化gydF4y2Ba农杆菌属gydF4y2Ba应变GV3101。gydF4y2Ba拟南芥gydF4y2Ba以Col-0野生型植株为材料,采用花浸法进行转化。收集的种子在含25 mg/L潮霉素的MS板上筛选。从T1转基因植株中提取基因组DNA。利用基因特异性引物TRY-IDF/R、CPC-IDF/R和ETC2-IDF/R对目标位点周围的片段进行PCR扩增gydF4y2Ba1gydF4y2Ba:表1)。将纯化后的PCR产物克隆到克隆载体pCBC中,利用T7引物对每个PCR片段的阳性克隆DNA进行测序,鉴定突变。为了筛选分离的非转基因T2植株,从土壤中培养的T2植株中提取基因组DNA。以野生型基因组DNA为阴性对照,T1转基因植株基因组DNA为阳性对照,对潮霉素抗性基因Hyg-IDF/R和Hyg-IDF2/R2以及潮霉素抗性基因zCas9-IDF/R三对引物进行反选择PCRgydF4y2BazCas9gydF4y2Ba(附加文件gydF4y2Ba1gydF4y2Ba:表1)。为了分析非转基因T2植株的突变,研究了围绕靶点的片段gydF4y2Ba试一试gydF4y2Ba,gydF4y2Ba中国共产党gydF4y2Ba或gydF4y2BaETC2gydF4y2Ba采用基因特异性引物TRY-IDF0/R0、CPC-IDF0/R0和ETC2-IDF0/R0(附加文件gydF4y2Ba1gydF4y2Ba:表1)。纯化的PCR产物提交测序,引物(TRY/CPC/ETC2-seqF)位于PCR片段(附加文件)内gydF4y2Ba1gydF4y2Ba:表1)。将测序差的PCR产物克隆到克隆载体pCBC上,用T7引物对阳性克隆的DNA进行测序。gydF4y2Ba
附加文件gydF4y2Ba
参考文献gydF4y2Ba
杨建军,杨建军,刘建军。拟南芥中T-DNA的插入诱变。植物学报,1999,31(2):387 - 398。10.1105 / tpc.11.12.2283。gydF4y2Ba
荣格KH全JS,李年代,小君SH, Jeong DH,李J,金正日C,张成泽年代,杨K,南J, K,韩寒MJ, Sung RJ崔HS, JH,崔JH,曹SY, Cha党卫军,Kim SI G:本文分别插入突变在水稻功能基因组学。植物学报,2000,22(2):561-570。10.1046 / j.1365 - 313 x.2000.00767.x。gydF4y2Ba
Gaj T, Gersbach CA, Barbas CF: ZFN, TALEN,基于CRISPR/ cas的基因组工程方法。生物医学进展,2013,31(3):397-405。10.1016 / j.tibtech.2013.04.004。gydF4y2Ba
彭尼西·E: CRISPR热潮。科学通报,2013,34(1):833-836。10.1126 / science.341.6148.833。gydF4y2Ba
Segal DJ:细菌预示着基因编辑的新时代。电子学报,2013,2:00563-10.7554/电子学报,00563。gydF4y2Ba
Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P: CRISPR在原核生物中提供获得性病毒抗性。科学,2007,31(3):1709-1712。10.1126 / science.1138140。gydF4y2Ba
霍瓦特P, Barrangou R: CRISPR/Cas,细菌和古细菌的免疫系统。科学通报,2010,32(3):397 - 397。10.1126 / science.1179555。gydF4y2Ba
李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军。科学通报,2012,37(3):816-821。10.1126 / science.1225829。gydF4y2Ba
Jinek M, Jiang F, Taylor DW, Sternberg SH, Kaya E, Ma E, Anders C, Hauer M, Zhou K, Lin S, Kaplan M, Iavarone AT, Charpentier E, Nogales E, Doudna JA: Cas9内切酶在rna介导的构象激活中的结构。科学,2014,34 (3):1247997-10.1126/ Science. 1247997。gydF4y2Ba
Nishimasu H, Ran FA, Hsu PD, Konermann S, Shehata SI, Dohmae N, Ishitani R, Zhang F, Nureki O: Cas9与引导RNA和靶DNA复合物的晶体结构。Cell. 2014, 56(6): 935-949。10.1016 / j.cell.2014.02.001。gydF4y2Ba
Sternberg SH, Redding S, Jinek M, Greene EC, Doudna JA: CRISPR rna引导的Cas9内切酶的DNA询问。自然科学,2014,37(7):62-67。10.1038 / nature13011。gydF4y2Ba
赵世文,金世文,金建民,金钟生:Cas9 rna引导的人类细胞靶向基因组工程。生物医学工程学报,2013,31(1):393 - 398。10.1038 / nbt.2507。gydF4y2Ba
丛玲,冉芳,Cox D,林森,Barretto R, Habib N,徐佩德,吴晓,姜文,Marraffini LA,张飞:CRISPR/Cas系统的多重基因组工程。科学通报,2013,39(3):819-823。10.1126 / science.1231143。gydF4y2Ba
DiCarlo JE, Norville JE, Mali P, Rios X, Aach J, Church GM:基于CRISPR-Cas系统的酿酒酵母基因组工程。中国生物医学工程学报,2013,41(1):436 - 443。10.1093 / nar / gkt135。gydF4y2Ba
冯志,张斌,丁伟,刘霞,杨道林,魏鹏,曹峰,朱松,张峰,毛勇,朱建军:基于CRISPR/Cas系统的植物高效基因组编辑。生物医学工程学报,2013,23:1229-1232。10.1038 / cr.2013.114。gydF4y2Ba
Friedland AE, Tzur YB, Esvelt KM, Colaiacovo MP, Church GM, Calarco JA:基于CRISPR-Cas9系统的秀丽隐门线虫遗传基因组编辑。地理学报,2013,32(1):444 - 444。10.1038 / nmeth.2532。gydF4y2Ba
Hsu PD, Scott DA, Weinstein JA, Ran FA, Konermann S, Agarwala V, Li Y, Fine EJ, Wu X, Shalem O, Cradick TJ, Marraffini LA,包刚,张飞:rna引导Cas9核酸酶的DNA靶向特异性。生物工程学报,2013,31(1):827-832。10.1038 / nbt.2647。gydF4y2Ba
黄文勇,付勇,Reyon D, Maeder ML,蔡sq, Sander JD, Peterson RT, Yeh JR, young JK:基于CRISPR-Cas系统的斑马鱼基因组编辑。生物工程学报,2013,31(1):229 -229。10.1038 / nbt.2501。gydF4y2Ba
蒋伟,周辉,毕辉,Fromm M,杨波,Weeks DP: CRISPR/Cas9/ sgrna介导的拟南芥、烟草、高粱和水稻靶向基因修饰的研究。核酸学报,2013,41 (1):188-10.1093/nar/gkt780。gydF4y2Ba
李金峰,Norville JE, Aach J, McCormack M, Zhang D, Bush J, Church GM, Sheen J:利用引导RNA和Cas9介导拟南芥和拟南芥基因组编辑。生物工程学报,2013,31(1):688-691。10.1038 / nbt.2654。gydF4y2Ba
Mali P, Yang L, Esvelt KM, Aach J, Guell M, DiCarlo JE, Norville JE, Church GM: rna引导的Cas9人类基因组工程。科学通报,2013,33(3):823-826。10.1126 / science.1232033。gydF4y2Ba
毛燕,张华,徐宁,张波,高峰,朱建军:CRISPR-Cas系统在植物基因组工程中的应用。植物学报,2013,6(6):2008-2011。10.1093 / mp / sst121。gydF4y2Ba
Nekrasov V, Staskawicz B, Weigel D, Jones JD, Kamoun S:利用Cas9 rna引导的内切酶靶向诱变模式植物烟叶。生物工程学报,2013,31(1):691-693。10.1038 / nbt.2655。gydF4y2Ba
单强,王勇,李军,张勇,陈凯,梁震,张凯,刘健,奚建军,邱建林,高超:基于CRISPR-Cas系统的作物基因组靶向修饰。生物工程学报,2013,31(1):686-688。10.1038 / nbt.2650。gydF4y2Ba
沈波,张健,吴辉,王健,马凯,李铮,张翔,张鹏,黄翔:Cas9/ rna介导的基因靶向基因修饰小鼠的产生。中国生物医学工程学报,2013,23(3):779 - 779。10.1038 / cr.2013.46。gydF4y2Ba
谢凯,杨勇:基于CRISPR-Cas系统的rna引导植物基因组编辑。植物学报,2013,6(6):1975-1983。10.1093 / mp / sst119。gydF4y2Ba
杨慧,王慧,Shivalila CS,程安华,石磊,Jaenisch R: CRISPR/ cas介导的基因工程一步生成携带报告基因和条件等位基因的小鼠。Cell. 2013, 54(4): 1370-1379。10.1016 / j.cell.2013.08.022。gydF4y2Ba
李丹,邱忠,邵毅,陈毅,关毅,刘敏,李毅,高宁,王玲,陆霞,赵燕,刘敏:基于CRISPR-Cas系统的小鼠和大鼠遗传基因靶向。生物工程学报,2013,31(1):681-683。10.1038 / nbt.2661。gydF4y2Ba
李伟,滕峰,李涛,周强:基于CRISPR-Cas系统的大鼠多基因突变同时产生及种系传递。生物工程学报,2013,31(1):684-686。10.1038 / nbt.2652。gydF4y2Ba
王慧,杨慧,Shivalila CS, Dawlaty MM,程文华,张峰,Jaenisch R: CRISPR/ cas介导的基因组工程一步生成多基因突变小鼠。细胞学报,2013,32(3):990 -918。10.1016 / j.cell.2013.04.025。gydF4y2Ba
饶乐,文德生,陈伟:利用CRISPR核酸酶系统对斑马鱼基因组进行高效多等位基因编辑。美国国家科学促进会。中国机械工程,2013,31(2):391 - 391。10.1073 / pnas.1308335110。gydF4y2Ba
牛燕,沈波,崔燕,陈燕,王健,王磊,康勇,赵鑫,司伟,李伟,向爱普,周健,郭鑫,毕宇,司诚,胡斌,董刚,王宏,周铮,李涛,谭涛,蒲晓,王峰,季森,周强,黄翔,季伟,沙杰:单细胞胚胎Cas9/ rna介导的基因靶向产生基因修饰食蟹猴。Cell. 2014, 56(6): 836-843。10.1016 / j.cell.2014.01.027。gydF4y2Ba
苗军,郭东,张军,黄强,秦刚,张旭,万军,顾华,曲丽娟:利用CRISPR-Cas系统实现水稻靶向诱变。生物医学工程学报,2013,23:1233-1236。10.1038 / cr.2013.123。gydF4y2Ba
Belhaj K, Chaparro-Garcia A, Kamoun S, Nekrasov V:利用CRISPR/Cas系统对模型和作物植物进行靶向诱变,使植物基因组编辑变得容易。植物学报,2013,9:39-10.1186/1746-4811-9-39。gydF4y2Ba
Fauser F, Schiml S, Puchta H:基于CRISPR/ cas的核酸酶和缺口酶在拟南芥基因组工程中的应用。植物学报,2014,37(4):348-359。10.1111 / tpj.12554。gydF4y2Ba
冯志,毛燕,徐宁,张波,魏鹏,杨道林,王志,张志,郑瑞,杨丽,曾丽,刘鑫,朱建军:拟南芥CRISPR/ cas诱导基因修饰的多代分析揭示了基因修饰的遗传、特异性和模式。美国国家科学促进会。[j] .中国科学:自然科学。10.1073 / pnas.1400822111。gydF4y2Ba
贾红,王宁:利用Cas9/sgRNA靶向编辑甜橙基因组。科学通报,2014,9:e93806-10.1371/journal.pone.0093806。gydF4y2Ba
蒋伟,杨波,周DP: CRISPR/ cas9介导的拟南芥高效基因编辑及修饰基因在T2和T3代中的遗传。科学通报,2014,9:e99225-10.1371/journal.pone.0099225。gydF4y2Ba
谢凯,张军,杨勇:模式植物和主要作物crispr - cas9介导的基因组编辑中高特异性引导RNA间隔物的全基因组预测。植物学报,2014,27(7):923-926。10.1093 / mp / ssu009。gydF4y2Ba
张辉,张军,魏平,张斌,苟峰,冯志,毛燕,杨玲,张宏,徐宁,朱建军:CRISPR/Cas9系统在水稻一代中产生特异性和纯合子靶向基因编辑。植物学报,2014,32(2):797-807。10.1111 / pbi.12200。gydF4y2Ba
刘建军,刘建军,刘建军,等。一种农业细菌介导植物转化的Ti载体。植物生理学报,2004,22(2):819-832。10.1023 /: 1006496308160。gydF4y2Ba
Curtis MD, Grossniklaus U:一种用于植物基因高通量功能分析的门户克隆载体。植物生理学报,2009,33(4):462-469。10.1104 / pp.103.027979。gydF4y2Ba
李丽娟,李志强,李志强:T-DNA二元载体及其系统。植物生理学报,2009,35(4):357 - 357。10.1104 / pp.107.113001。gydF4y2Ba
张建军,张建军,张建军,张建军,等。多基因克隆技术的研究进展。科学通报,2011,6:e16765-10.1371/journal.pone.0016765。gydF4y2Ba
Engler C, Kandzia R, Marillonnet S:一种高通量的一锅一步精确克隆方法。科学通报,2008,31 (3):e3647-10.1371/journal.pone.0003647。gydF4y2Ba
Gibson DG, Young L, Chuang RY, Venter JC, Hutchison CA, Smith HO: DNA分子的酶组装高达数百千碱基。地理学报,2009,6(6):343-345。10.1038 / nmeth.1318。gydF4y2Ba
Kirik V, Simon M, Wester K, Schiefelbein J, Hulskamp M:拟南芥毛状体发育区域特异性控制中TRY和CPC 2 (ETC2)增强子的冗余。植物生理学报,2004,25(5):389-398。10.1007 / s11103 - 004 - 0893 - 8。gydF4y2Ba
黄玉英,李慧敏:拟南芥CHLI2可以替代CHLI1。植物生理学报,2009,35(2):636-645。10.1104 / pp.109.135368。gydF4y2Ba
李建军,李建军,李建军,等。农杆菌pPZP家族基因的克隆与应用。植物生理学报,1994,25(4):989-994。10.1007 / BF00014672。gydF4y2Ba
张勇,苏军,段松,敖勇,戴军,刘军,王鹏,李勇,刘波,冯丹,王军,王辉:水稻绿色组织原生质体系统瞬时基因表达及光/叶绿体相关过程研究。植物学报,2011,(7):30-10.1186/1746-4811-7-30。gydF4y2Ba
致谢gydF4y2Ba
我们感谢张锋的pX3300, Keith young的pJDS246,王国良的pXSN和pXUN, Roger Hellens的pSoup, M. Curtis的pMDC99/100/123,杨淑华在玉米原生质体转染方面的帮助,以及中国农业大学转基因设施中心的同事们在转基因玉米的产生方面的帮助。我们要感谢以英语为母语的Elixigen公司的科学家编辑我们的手稿。国家基础研究计划项目(2012CB114200)、国家自然科学基金项目(31070329)和国家转基因研究项目(2011ZX08009)资助。gydF4y2Ba
作者信息gydF4y2Ba
作者及单位gydF4y2Ba
相应的作者gydF4y2Ba
额外的信息gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者宣称他们没有竞争利益。gydF4y2Ba
作者的贡献gydF4y2Ba
HLX, LD, ZPW, HYZ, CYH和BL进行了实验并分析了数据。本研究由CQJ和WXC构思,参与设计和协调,并起草了稿件。所有作者都阅读并认可了稿件。gydF4y2Ba
电子补充资料gydF4y2Ba
12870 _2014_327_moesm1_esm.pdfgydF4y2Ba
附加文件1:图S1。:Sequencing analysis of target gene mutations of a representative p2gR-TRI-B line. Table S1. Primers used in this study. (PDF 945 KB)
作者提交的原始图片文件gydF4y2Ba
下面是作者提交的原始图片文件的链接。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。gydF4y2Ba
本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接获得版权所有者的许可。gydF4y2Ba
如欲查阅本许可证副本,请浏览gydF4y2Bahttps://creativecommons.org/licenses/by/4.0/gydF4y2Ba。gydF4y2Ba
创作共用公共领域免责声明(gydF4y2Bahttps://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本文中提供的数据,除非在数据的信用额度中另有说明。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
兴,霍奇金淋巴瘤。,Dong, L., Wang, ZP.et al。gydF4y2Ba用于植物多重基因组编辑的CRISPR/Cas9工具包。gydF4y2BaBMC Plant BiolgydF4y2Ba14gydF4y2Ba, 327(2014)。https://doi.org/10.1186/s12870-014-0327-ygydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-014-0327-ygydF4y2Ba
关键字gydF4y2Ba
- CRISPR / Cas9gydF4y2Ba
- 基因组编辑gydF4y2Ba
- 多基因突变gydF4y2Ba
- 多个grna的组装gydF4y2Ba