摘要
背景
动物及其微生物群适应饥饿并在重新进食后恢复体内平衡的能力是其持续生存和共生的基础。肠道是营养吸收和微生物相互作用的主要场所,然而,我们对肠道适应饥饿和再喂养的了解仍然有限。在这里,我们使用RNA测序和16S rRNA基因测序来揭示长期饥饿和再喂食的斑马鱼与连续喂食的对照组相比,肠道转录组和微生物组的变化。
结果
与对照组相比,21天的饥饿导致肠道微生物群的多样性增加和组成改变,包括相对增加弧菌减少邻单胞菌属细菌。饥饿也导致肠道中宿主基因表达的显著改变,在饥饿的早期和晚期有不同的途径受到影响。这包括在饥饿早期核糖体生物发生基因的表达增加,随后在后期参与抗病毒免疫和脂质转运的基因表达减少。这些饥饿对宿主转录组和微生物组的影响在重新喂食后3天内几乎完全恢复。与已发表的数据集进行比较,确定了对饥饿、高脂肪摄食或微生物定殖有反应的宿主基因,并预测了可能参与饥饿反应的宿主转录因子。
结论
长期饥饿诱导斑马鱼肠道微生物组组成和宿主基因表达的进行性变化,这些变化在重新喂食后迅速逆转。我们对参与这一反应的细菌分类群、宿主基因和宿主途径的鉴定,为未来研究肠道对食物限制适应的生理和生态机制提供了框架。
介绍
饥饿是一种严格限制热量的状态,许多动物和很大一部分人类经常经历这种状态。对人类来说,饥饿可能是环境或社会经济条件的结果,包括战争、饥荒和贫困[1].它也可能与神经性厌食症和癌症等疾病一起发生。2].对动物来说,绝对饥饿或相对饥饿的时期可能是由季节变化(如干旱和严寒)或筑巢、哺乳、迁徙和冬眠等行为造成的。3.].动物王国中导致饥饿的各种各样的情况,唤起了不同物种对饥饿的一系列渐进的生理适应。事实上,以前的研究已经报告了动物类群在饥饿生理学上的相似性和差异性,如人类、啮齿动物、北极熊、企鹅、爬行动物、两栖动物、鱼类和昆虫[4].然而,先前的研究主要集中在与饥饿相关的组织病理学上,而对潜在生理过程的影响仍然不完全清楚。
在许多动物物种中,饥饿会导致代谢率的逐渐降低[5].作为糖异生前体的血甘油增加,在饥饿的动物中也很常见,游离脂肪酸的波动也是如此[3.,4].能量储存的整体消耗导致体重下降,与变温动物相比,恒温动物的体重下降幅度通常更大[6].饥饿还与肝脏、骨骼肌和肠道等重要器官的质量逐渐减少有关[4,7].这些影响需要从饥饿中恢复,以恢复这些器官的最佳功能。与饥饿有内在联系,饥饿后恢复体内平衡是由再进食反应促进的,该反应逐渐逆转饥饿引起的适应并恢复能量平衡。对饥饿和随后的再进食的生理反应是动态和复杂的,涉及主要器官系统之间通过营养和激素信号的协调。这些饥饿反应的最终结果往往是保持瘦体重,同时有利于消耗能量储存,如糖原和脂肪[8,9].然而,尽管有这些影响,饥饿经常在重新进食后很长时间内对骨密度、胰腺功能和智力发育造成持久的缺陷[10,11,12].因此,提高对这些动态生理过程的理解可能会导致新的方法来减少人类和其他动物与饥饿相关的发病率和死亡率[13].
先前对饥饿后再进食影响的研究主要集中在肝脏、骨骼肌、大脑和胰岛等组织上[14,15,16,17,18].我们对肠道中的转录饥饿和再喂养反应的理解相对较差。肠道是膳食营养感知和吸收的主要部位,也是复杂微生物群落(微生物组)的栖息地。先前对人类和啮齿动物模型的研究表明,肠道微生物组的组成会随着饥饿和饮食组成的变化而变化,对宿主的营养生理有不同的贡献[19,20.,21,22,23,24,25,26].这些发现为最近研究微生物组靶向治疗减轻饥饿及其相关发育缺陷提供了依据[27,28,29].然而,肠道微生物对饥饿的反应在很大程度上仅限于哺乳动物,我们对任何动物对饥饿和摄食的肠道生理反应的了解仍然非常有限。
动物模型为研究脊椎动物饥饿和再进食反应背后的过程提供了机会,从而产生了可能转化为人类的一般理解[30.].像鲤科鱼类这样的变温脊椎动物特别有趣,因为它们有能力忍受长时间的饥饿。在长时间饥饿的反应中,鲤类如鲤鱼表现出肠道厚度和重量的减少,肠细胞形态的改变,体重和肝脏大小的减少,与小鼠的饥饿反应相似[17,31,32,33,34,35].斑马鱼(鲐鱼类)在饥饿的情况下,成年后能存活4周,而一套基因组和遗传资源有助于研究它们的生理[36].使用体内成像来监测白色脂肪组织作为能量储存的测量,我们之前表明,成年斑马鱼的长期饥饿会导致储存在白色脂肪组织中的脂肪逐步动员,并在重新喂食时得到补充。37,38,39].由于脂肪组织在幼鱼和成鱼阶段逐渐发育,完全动员脂肪脂所需的饥饿时间随着动物年龄的增长而增加(例如,从幼鱼的1周到成鱼的3周)[37,38,39].然而,长期饥饿和重新进食对斑马鱼肠道的影响尚未被探索。
斑马鱼的肠道在细胞和生理上与哺乳动物的肠道具有广泛的同源性,其微生物群的组成随年龄和饮食组成的变化而变化[40,41,42,43,44,45].斑马鱼肠道微生物群的存在和组成通过调节膳食营养吸收、上皮细胞更新和炎症来影响宿主[41,43,46,47,48,49,50].通过比较斑马鱼、小鼠和人类肠上皮细胞的基因表达模式和可接近的染色质,我们最近发现了一个保守的转录调控网络,该网络在4.2亿年的脊椎动物进化中被保存下来。51].在此基础上,我们定义了长时间饥饿和再摄食对成年斑马鱼肠道基因表达的影响,并利用这些结果来预测对饥饿和再摄食反应的生理过程和转录调控途径。我们还展示了成年斑马鱼肠道微生物组的分类组成是如何在相同的长期饥饿和重新喂食方案中改变的。
结果
饥饿伴随着肠道微生物组成的显著变化,而这种变化在重新喂食期间被逆转
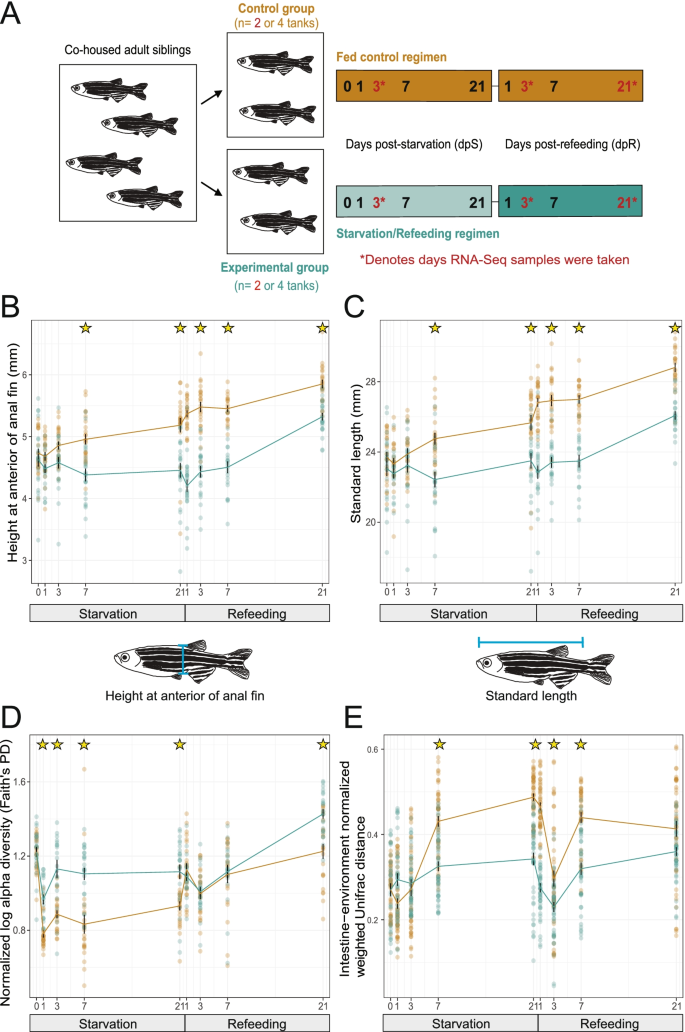
为了确定饥饿对斑马鱼微生物组组成和肠道基因表达的影响,在常规条件下使用标准饮食饲养斑马鱼至成年早期(受精后60天或dpf)。然后将动物转移到干净的水箱中,随机分为两个治疗组:一组禁食21天,然后再喂食21天,参照对照组在相同的42天时间内持续喂食(图2)。1A).之所以选择21天的饥饿方案,是因为成年斑马鱼的饥饿方案足以完全消耗脂肪组织中储存的脂质,从而降低体重和肝脏大小,而随后的再喂食在14天内基本恢复了脂肪组织的总脂质、体重和肝脏大小[37,38,52,53].我们在饥饿后0、1、3、7和21天对斑马鱼的全肠样本进行了16S rRNA基因测序,然后在标准饮食重新喂食后1、3、7和21天对斑马鱼的全肠样本进行了16S rRNA基因测序。1A).年龄相匹配的兄弟姐妹每天喂食相同的标准饮食作为参考对照,并在同一时间点取样。
饥饿和再喂养会影响斑马鱼的体细胞生长以及肠道和环境微生物群的多样性。一个研究设计示意图。被关在一起的成年兄弟姐妹被分成对照(喂食)和实验(饥饿)两组。然后在饥饿后第0、1、3、7和21天,以及再喂后第1、3、7和21天,从每个水箱中取样,进行16S rRNA基因测序。在3 dpS、21 dpS和3 dpR时采集RNA-seq样本。B饲喂和饥饿斑马鱼在相应时间点的肛鳍前高度(HAA),单位为mm。C饥饿和喂食斑马鱼的标准长度(毫米)。D费思的PD α多样性,饲养和控制斑马鱼。数值通过第0天的分数进行对数转换和规范化。E肠道与相关环境样本之间的加权UniFrac距离。图B-E中的星号表示显著差异(pTukey’s HSD检验< 0.05)
在这些早期成年阶段,正常喂养的斑马鱼继续表现出预期的体细胞生长。对动物尺寸的标准长度(SL)和前肛鳍(HAA)高度的测量显示,与对照鱼相比,饥饿鱼的躯体生长在很大程度上被阻止(图2)。1c)。饥饿后第7天,饥饿鱼明显小于喂食鱼,这一趋势持续到饥饿期结束后。饥饿的动物在重新喂食后恢复了生长,尽管在整个实验过程中它们仍然明显小于喂食的鱼(p< 0.05,经Bonferroni校正的双向方差分析)。1c)。在这些情况下,我们没有观察到死亡率,这与之前的研究一致[38,53].因此,饥饿导致体细胞生长的普遍停滞,在重新喂食后恢复。
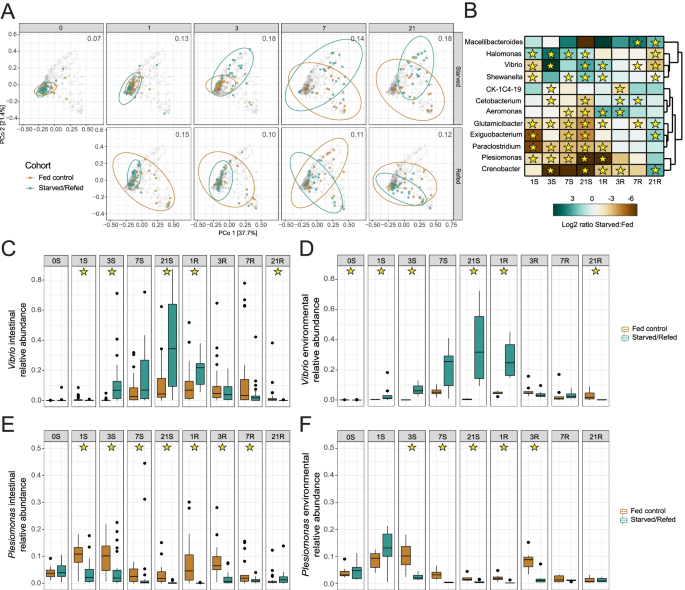
对肠道样本16S rRNA基因测序数据的分析揭示了长期饥饿和再摄食对肠道微生物组组成的影响。总体而言,与喂食对照组相比,饥饿斑马鱼的肠道微生物群保持了更高的Faith 's PD多样性。这两种情况都显示了多样性在1dpS时的初始损失,可能是由于实验开始时坦克在0dpS时转移造成的压力。然而,饥饿的群落从1dpS到21dpS和21dpR的饥饿结束都保持着更高的多样性。p< 0.05, ANOVA和Tukey HSD)。1D)。利用加权UniFrac距离对群落组成进行Beta多样性分析,结果表明,饥饿和进食群落开始出现1 dpS的差异,在3dpS和21 dpS时质心距离最大(图2)。2A).在重新喂食期间,饥饿的鱼样本迅速恢复到与喂食对照组更相似的成分(图2)。2A)。PERMANOVA进一步证实饥饿、实验时间点和相互作用是显著因素(p< 0.05,R2= 0.017, 0.17和0.07),影响肠道微生物组组成。因此,长时间的饥饿导致肠道细菌群落的整体组成发生可检测的变化,这种变化在重新喂食后迅速逆转。
饥饿和再喂食动态地改变了成年斑马鱼肠道微生物群的组成。一个饲喂和饥饿斑马鱼加权UniFrac多样性的主坐标分析。两个队列在相应时间点的质心之间的距离显示在每个图的右上方。灰点代表研究中的每个样本,而来自给定时间点的样本则根据其队列在各自的面板中进行标记(绿色:饥饿/喂食,金色:喂食对照)。B饥饿对照和喂食对照之间细菌属相对丰度的log2比值热图。星星表示由LEfSe确定的有意义的日子。C的相对丰度弧菌在饥饿和控制斑马鱼肠的白天。D的相对丰度弧菌在饥饿和控制环境水箱水样的白天。E的相对丰度邻单胞菌属在饥饿和控制斑马鱼肠的白天。F的相对丰度邻单胞菌属在饥饿和控制环境水箱水样的白天。C-F栏中的星号表示重要性(p< 0.05),经双相Wilcoxon检验并进行BH校正
饥饿增加了斑马鱼肠道微生物群与其周围水环境之间的相似性
居住在斑马鱼肠道中的微生物群与周围水环境的微生物群是连续存在的,然而这些群落通常表现出不同的组成[42,43,44].造成这些差异的生态过程尚不清楚,但可能包括非中性过程,如宿主选择[54,55]或肠道与周围环境之间扩散的程度[56].为了测试饥饿和重新喂食是否会改变肠道和环境微生物组之间的关系,我们比较了饥饿/冷藏和对照鱼的匹配肠道和环境样本之间的加权UniFrac距离,这些样本按第0天的值归一化。肠道和环境样本之间的距离在0 dpS和21 dpS之间增加(图2)。1E),可能反映了在0 dpS时转入新坦克的压力后的体内平衡的恢复。然而,从7 dpS到7 dpR,饥饿组的肠道和环境样本之间的距离比喂食组大。这表明,在这些阶段,与饥饿的鱼相比,喂食对照组的肠道微生物组成更接近环境。在21 dpR时,这些距离在治疗组之间无统计学差异。这些结果表明,饥饿增加了肠道和环境群落之间的相似性,而再摄食将这些群落之间的差异恢复到恒定摄食条件下的水平。
弧菌在饥饿期间,肠道内的细菌显著增加
接下来,我们试图用LEfSe识别受饥饿和再喂养显著影响的特定细菌分类群[57].LEfSe鉴定出120个属级分类群的对数LDA评分达到2.0(表5)1).该类群包括12个丰度属(所有样本的中位数相对丰度> 0.1%);如图所示。2B)包括饥饿引起的枯竭邻单胞菌属和丰富弧菌。引人注目的是,弧菌在21dpS时,饥饿斑马鱼肠道中的最大相对丰度达到87%(中位数34%),显著高于同一时间点的喂食对照组(最大62%,中位数4%)(图2)。2C).相反,饥饿导致食物相对丰富度的降低邻单胞菌属1dpS持续至7dpR(图2)。2E)饥饿对……的影响弧菌和邻单胞菌属在饥饿的鱼的环境样本中反映出饥饿的肠道中的sp.。2D-F)。重要的是,经过Bonferroni校正后,没有一个显着耗尽或富集的门或目与SL显著相关(见表S)2).这表明它们的消耗和富集是由于饮食处理,而不仅仅是在饥饿动物中观察到的生长停滞(图2)。1c - d)。这些结果表明,长期饥饿导致肠道微生物组组成的显著变化,包括显著富集弧菌属成员,并且这些微生物组组成的变化在重新喂食的1-3天内基本正常化[41,42,48].
饥饿和再喂养导致肠道基因表达的明显变化,这种变化随饥饿的持续时间而变化
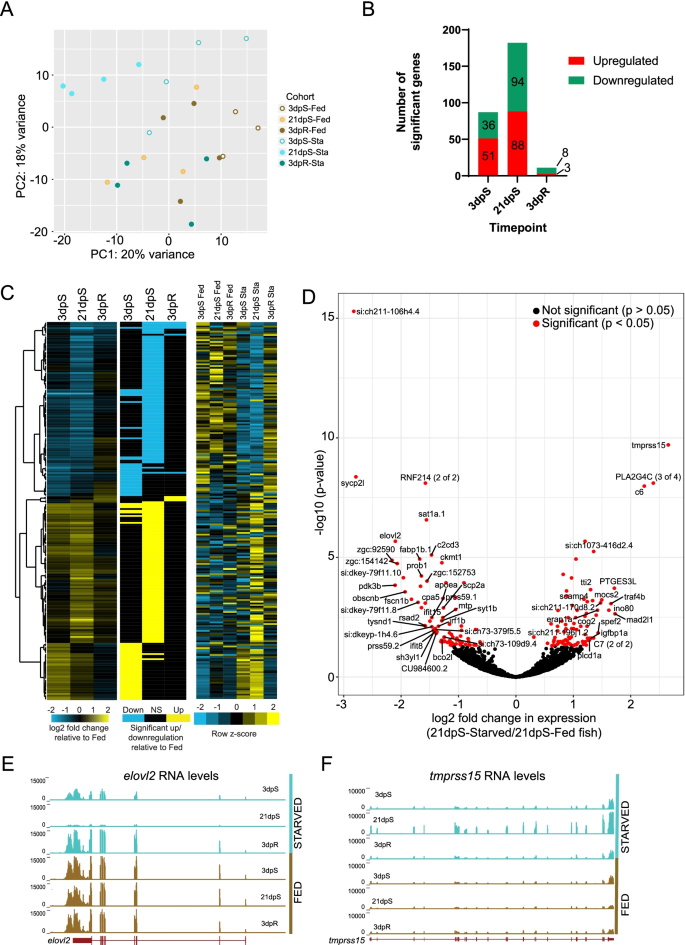
先前对脊椎动物的研究表明,饥饿可以显著影响宿主多个器官的基因表达[4,7,17,33,52].我们对饥饿和再摄食期间肠道微生物组的分析显示了不同的阶段——最初观察到微生物组效应时的早期饥饿(即3 dpS),微生物组变化最大时的后期饥饿(即21 dpS),以及微生物组组成基本正常化时的早期再摄食(即3 dpR)。因此,我们解剖了3dpS, 21dpS和3dpR成年斑马鱼及其喂养年龄匹配的对照组的整个肠道进行RNA-seq分析(3-4个生物重复样本/条件;无花果。1A).这些数据的主成分分析(PCA)揭示了生物重复之间的相似性(图2)。3.A)。与观察到的对肠道微生物组的影响相似,PCA表明饥饿在21dpS时对肠道基因表达的影响大于3dpS或3dpR。然后,我们使用DEseq2分析来鉴定在每个时间点,与喂食对照相比,饥饿/冷藏鱼中表达差异的基因(表5)3.A)。根据我们的PCA分析,显著差异表达基因的数量从3dpS时的87个基因增加到21dpS时的182个基因,而3dpR后减少到11个基因(图2)。3.B).这进一步支持了饥饿通过3dpS和21dpS对肠道转录组的进行性影响,而这些影响在很大程度上是由3dpR标准化的。
与喂食的斑马鱼相比,饥饿的斑马鱼对肠道基因表达的调节存在差异。一个饥饿/冷藏和饲喂对照斑马鱼肠道在3dpS、21dpS和3dpR时RNA-Seq文库的主成分分析(PCA)。B定量饥饿/冷藏斑马鱼肠道中各时间点显著上调和下调的基因数量。请注意,图B中的这些数字包括在我们的饲料对照比较中也有显著差异的基因。C饥饿斑马鱼肠道中基因表达变化的log2倍的分层聚类,以及显示基因表达显著变化的扁平值,以及基于每个基因归一化计数的z分数。D饥饿斑马鱼21dpS肠道中基因表达的变化是21dpS喂食鱼的Log2倍,根据它们的-log绘制10调整p值。请注意,图C和图D中绘制的数据不包括饲料鱼中显著差异的基因(见表S)3.B和图S2).E代表性重复的UCSC轨迹表明elovl2编码脂肪酸延长酶的mRNA在饥饿的斑马鱼肠道中下调,并在重新喂食后恢复到与喂食组相当的水平。F代表性重复的UCSC轨迹表明tmprss15编码肠肽酶的mRNA在饥饿的斑马鱼肠道中上调,并在重新喂食后恢复到与喂食组相当的水平
作为对照,为了估计实验期间所覆盖的发育时间的影响,我们比较了饲喂时间点之间的差异基因表达。在这些比较中有135个基因被发现是显著的,但其中只有30个基因在我们的饥饿与饥饿比较中具有显著性(在3dpS时18个,在21dpS时15个,在3dpR时2个;如表5所示3.B,如图S所示1B),并且从我们随后的饥饿效应分析中删除了它们(见图S)2对于那些保留在分析中的基因的结果)。日志的分层聚类2转录物丰度的折叠变化揭示了不同的基因组在饥饿反应中上调或下调,包括在第3dpS和21dpS对饥饿的反应之间的显著差异(图2)。3.C)。尽管3dpR在很大程度上恢复了饥饿/冷藏鱼与喂食对照鱼之间的基因表达差异,但即使在3dpR下,仍有一小组9个基因继续差异表达(图2)。3.B;表的年代3.A).尽管这一持久基因列表太小,无法进行功能富集分析,但它确实表明,可能有一小部分肠道功能在恢复摄食后仍然改变,或者对饥饿和再摄食都有反应。这些持续不同的基因将在下文中讨论。
在已知的饥饿反应基因中,我们首先检查了转录水平elovl2一种脂肪酸延长酶在斑马鱼饥饿时被下调[58,59,60].Elovl2还与小鼠葡萄糖诱导胰岛素分泌有关,脂肪酸延长酶在鱼类中被广泛研究,因为它们在长链多不饱和脂肪酸的生物合成中起作用,这在鱼类养殖中具有重要的商业意义[58,61,62].的协议,elovl2被3dpS显著下调,是21dpS时饥饿鱼中下调最显著的基因之一,并且在对照组中也一致表达(图2)。3.D-E,图51A).这种下调表明饥饿期间肠道脂肪酸合成减少。
为了了解哪些生物过程受到饥饿的影响,我们对数据集中的四组基因进行了基因本体(GO)术语搜索;基因在3dpS或21dpS时显著上调,而基因在3dpS或21dpS时显著下调(图2)。3.C,年代1C-D和S3.).我们首先确定了不同的、不重叠的功能,这些功能在饥饿早期(即在3d时)和饥饿后期(即在21dpS时)富集。例如,仅在3dpS上调基因中富集的功能,而不是21dpS,包括“核糖体”和“核糖体大亚基生物发生”(图5)1E).这些包括核糖体生物发生因子nsa2和gtpbp4这与60S核糖体亚基的生物发生有关,在3ps时显著上调,而21dpS时则没有上调。然而,“核糖体大亚基组装”功能在21dpS而不是3dpS上调的基因中富集。这包括一些仅在21dpS时显著增加的基因,例如ruvbl1和srfbp1,以及其他在3d和21dpS时显著增加的,例如rsl24d1,ptges3l,gltscr2(表3.A)总的来说,与21dpS相比,参与核糖体生物发生的基因在3dpS时被诱导得更强烈,这表明它是对饥饿的相对早期反应,其方面持续到21dpS(图5)3.CD,表S3.一个)。
在3dpS特异性上调的基因中还有热休克蛋白hsp90ab1,一种分子伴侣先前被证明在成年斑马鱼的肝脏中对饥饿的反应是上调的[17].在我们的数据集中,最显著的上调基因是肠肽酶/肠激酶tmprss15,将胰蛋白酶原转化为活性胰蛋白酶,进而激活胰酶,并可能激活肠道内的抗菌蛋白(图2)。3.D-F) [63,64].肠内表达tmprss15不受饥饿的影响,但在21dpS时上调了6倍(图2)。3.D-F)。值得注意的是,缺乏TMPRSS15已被证明在人类中具有瘦削、饥饿样的表型,这与它在营养消化和吸收中的已知关键作用相一致[65].Upregulation的tmprss15在饥饿的斑马鱼肠道中,表明营养消化程序的潜在适应性增加,以从肠腔中挽救营养物质,或者对改变的和潜在的促炎微生物群进行抗菌防御。
同样,在3dpS而非21dpS下调基因中特异性富集的功能包括“脂质代谢”、“细胞增殖调节”和“泛素依赖的蛋白质分解代谢过程”(图5)1E).这包括酰基转移酶的下调lclat1和脂肪酸结合蛋白fabp1b.13d而不是21dpS。而在21dpS下调基因中,“甘油磷脂代谢”、“脂质转运”和“脂质代谢过程”等相关功能得到了增强,而在3dpS下调基因中则没有。其中包括磷脂酶pla2g12b即脂肪酸去饱和酶fads2即脂质转移蛋白scp2a,以及多种载脂蛋白,包括apoa1a,apoa4b.1,apobb.1(无花果。3.D,年代1E,年代3.AB) [66].值得注意的是,载脂蛋白基因已被证明在饥饿的虹鳟肝脏中下调[67].尽管这些基因仅在21dpS时显著下调,但大多数基因在3dpS时开始趋于下调。其他参与这些功能的基因包括elovl2和fabp1b.1在3dpS和21dpS时均显著下调,在21dpS时差异更大(图2)。3.D和S1A、表S3.A,值得注意的是,传送器slc31a1/ctr1在这两个时间点,参与的饮食铜摄取也显著下调。因此,虽然较短的饥饿时间(如3天)会导致一些代谢功能下调,但大多数与脂质代谢有关的基因直到饥饿21天才显著下调。因此,长期饥饿导致参与脂质生物合成和运输的基因表达减少,这可能代表了对长期缺乏膳食脂肪和其他营养物质的适应。然而,这与较短的饥饿时间形成对比,例如48小时,其他斑马鱼研究已经观察到脂质分解代谢增加,可能增加可用能量并提高抗寒能力[68].
被饥饿显著下调的基因在宿主免疫功能上也得到了富集。例如,信号换能器stat1b,这是肠道炎症反应和斑马鱼骨髓发育所必需的[69,70],以及干扰素反应基因ifit8被3dpS下调,并持续到21dpS。由21个dps,ifit15抗病毒蛋白rsad2也被显著下调。与此一致的是,在21dpS处下调的基因在“病毒防御反应”相关功能上富集。最后是羧基肽酶cpa5它被认为是斑马鱼肥大细胞的标记物[71]在21dpS时也显著下调,表明肠道组织中肥大细胞数量或活性可能减少。尽管在饥饿的鱼类中,一些免疫相关基因被下调,但补体蛋白c6和c7在饥饿的鱼中均显著上调(表53.).因此,我们对饥饿期间下调的基因的分析表明,饥饿期间肠道免疫功能和炎症张力的减少或损害,以及脂质代谢和脂蛋白生成的显著减少。饥饿期间免疫功能降低可能是导致在这些时间点观察到的微生物群落变化的一种机制。
虽然在3dpR后再喂养的显著基因太少,无法进行功能富集分析,但其中一些基因暗示了潜在的肠道功能。这包括编码串联复制胰蛋白酶样丝氨酸蛋白酶的基因在3dpR上的表达增加prss59.1和prss59.2。这一小组基因还包括三种线粒体酶-胡萝卜素双加氧酶样基因bco2l,参与膳食类胡萝卜素裂解为类维生素A合成所需的类维生素A;二甲基甘氨酸脱氢酶dmgdh在胆碱氧化途径中参与甘氨酸的合成和肌氨酸的产生。值得注意的是,Dmgdh先前的研究表明,在禁食的小鼠肝脏中会被诱导,而在准备冬眠的地松鼠肝脏中则会减少[72,73].与饲料对照相比,3dpR鱼的11个基因中有6个在21dpS时也有差异表达,包括prss59.1,prss59.2,bco2l。这些可能代表在恢复摄食后仍然改变的饥饿适应,或者对饥饿和再摄食都有反应。
尽管我们已经从该分析中删除了饲喂控制时间点之间差异表达的任何基因(表5)3.B,图52),我们希望进一步评估是否存在更广泛的生物过程,这些生物过程可能在喂食对照样本之间存在差异,从而影响我们与饥饿/喂食动物的比较。因此,我们对饲料控制时间点之间存在显著差异的基因进行了氧化石墨烯项分析。相较于3dp饲料,21dpS饲料显著下调的基因中,GO术语“脂质代谢过程”显著富集。相反,在21dpS喂养的鱼中,相对于3dpS喂养的鱼,显著上调的基因中,GO术语“脂质定位”和“脂质反应”显著富集(表5)3.重要的是,GO术语“脂质代谢过程”也富含相对于21dpS喂养的鱼而言,在21dpS饥饿中显著下调的基因,即使在我们的对照分析中显着的基因被删除后也是如此(图5)1E).这就提出了一种可能性,即我们观察到的饥饿对脂质代谢基因的影响,可能部分是由于21dpS喂养的鱼中某些脂质代谢基因的表达异常低,而其他相关的脂质代谢功能可能在21dpS喂养的鱼中相对于其他喂食时间点异常高。因此,我们评估了该对照分析中基因的log2倍变化,以及饥饿和喂食鱼之间显着差异的基因,以确定这些差异中的一些是否可能是由喂食21dpS的对照鱼驱动的(图5)3.).我们发现GO术语“核糖体”和“核糖体大亚基生物发生”下的基因在饥饿鱼中没有差异表达,这受到喂食鱼中异常基因表达的影响(图5)3.c - d)。相比之下,基因的子集,如Pdk3b, syt1b, apoa1a, apoa4b.1,和fads2这些基因在21dpS的饥饿鱼中相对于21dpS喂食的鱼显著下调,部分原因可能是这些基因在21dpS喂食的鱼中异常高的表达(图5)3.a - b)。然而,这里强调的大多数基因,比如Elovl2, pl2g12b, slc31a1,还有许多其他的,不受喂食鱼群异常的影响,很可能是饥饿治疗的真正生物效应。为了证实这一点,我们建立了一个独立的成年斑马鱼队列,分别接受21天的饥饿和正常喂养,并使用定量RT-PCR来评估肠道脂质代谢基因的表达。根据我们原始实验队列的RNA-seq数据(表S)3.),饥饿鱼在21dpS时的表达显著降低elovl2 pla2g12b,和apoa4b.2与21dpS喂养对照组相比(图5)4).
一些对饥饿有反应的基因也对高脂肪喂养有反应
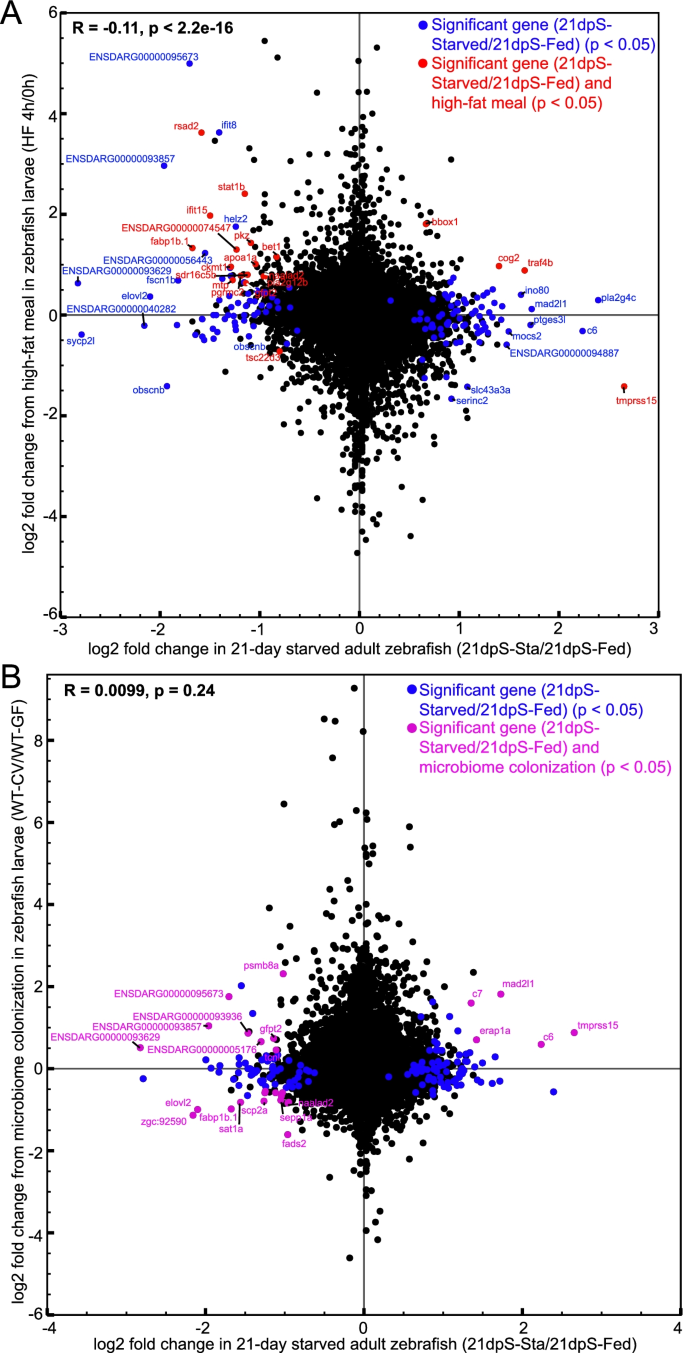
为了解释我们数据集中哪些饥饿反应基因在广泛的营养可用性范围内转录反应,以及哪些基因可能构成饥饿特异性反应,我们将我们的肠道RNA-seq结果与先前发表的RNA-seq数据进行了比较,这些数据比较了未喂食或喂食高脂肪食物(蛋黄)的斑马鱼幼虫的消化道[59].这揭示了显著差异表达基因的大量重叠(图2)。4A),特别是参与脂质转运和代谢的基因,如fabp1b.1和pla2g12b在斑马鱼中,在饥饿期间下调的基因在高脂肪喂养期间上调,强调了这些基因对斑马鱼营养作出反应的能力。一些与免疫功能有关的基因,如rsad2 stat1b,和ifit15在饥饿期间下调,在高脂肪喂养后上调。重叠基因中也有肠肽酶tmprss15哪一种在饥饿时上调,而在高脂肪喂养时下调,以及γ丁甜菜碱羟化酶bbox1而饥饿和高脂肪喂养都会导致这种情况的发生。
肠道中一些对饥饿有反应的基因也对高脂肪喂养和微生物定植有反应。一个来自21dpS的基因的Log2倍变化(x轴)是根据饲喂蛋黄的斑马鱼幼虫与未饲喂对照(y轴)的Log2倍变化绘制的,Zeituni等人[59].只有在饥饿的斑马鱼中才有显著差异的基因用蓝色表示,而在两个数据集中都有显著差异的基因用红色表示。皮尔逊相关性揭示了两个数据集之间的显著相关性(p< 0.05)。B来自21dpS的基因的Log2倍变化(x轴)根据与无菌对照(y轴)相比,带有微生物组定植的斑马鱼幼虫的Log2倍变化绘制,Davison等人[74].仅在饥饿的斑马鱼中具有显著log2倍变化的基因用蓝色绘制,而在两个数据集中都具有显著变化的基因用洋红色绘制。皮尔逊相关并没有显示两个数据集之间有显著的相关性(p> 0.05)
虽然上述数据集中的基因之间存在重叠,暗示它们与肠道营养反应有关,但一些受饥饿显著影响的基因并未受到高脂肪喂养的显著影响。这些基因包括补体因子c6即脂肪酸延长酶elovl2,还有磷脂酶pla2g4c。这些发现表明,参与脂质转运或炎症的某些基因类别可能受到与饥饿相关的因素的差异调节,而不是高脂肪喂养固有的营养过剩。或者,这些差异可能归因于斑马鱼生命阶段(成鱼与幼虫)或器官(肠道与包括肠道、肝脏、胰腺和鱼鳔在内的完整消化道)特有的转录反应,或者是与营养无关的高脂肪蛋黄喂养的间接影响。
一小部分对饥饿有反应的基因也对微生物定植有反应
我们和其他人已经表明,肠道基因表达在一定程度上受到肠道微生物群的存在和组成的调节[55,74,75,76].我们的16S rRNA序列数据显示饥饿诱导了肠道微生物组组成的显著和可逆的改变,包括富集弧菌Sp .,其成员已被证明具有促炎作用,并且饥饿的鱼与其环境样本之间微生物组组成的相似性降低[48].尽管这些结果表明饥饿期间肠道微生物生态发生了改变,但我们的研究设计不允许我们将观察到的肠道转录组和微生物组的变化联系起来。因此,为了确定对饥饿的转录反应可能也对微生物组敏感,我们将我们的RNA-seq数据与先前研究微生物定殖对斑马鱼幼虫消化道影响的研究进行了比较[74].我们发现两个数据集之间没有相关性,这意味着在这些时间点上,微生物定植和饥饿的转录反应之间可能没有广泛的重叠。4B).这种适度的重叠可能是由于斑马鱼生命阶段(成鱼与幼虫)或器官(肠道与完整的消化道,包括肠道、肝脏、胰腺和鱼鳔)特有的转录反应。然而,我们确实发现了几个重叠的基因,这些基因在两个数据集中都很重要。两个补体因子,c6和c7、它们在斑马鱼中被上调,在饥饿时也被上调。此外,两个基因参与处理主要组织相容性复合体,mad2l和erap1,也在重叠基因中。参与脂质代谢和细胞内胆固醇转运的一小组基因,如elovl2,fads2,fabp1b.1和scp2a被殖民和饥饿显著下调。肠激酶的tmprss15在饥饿和高脂肪摄食期间差异表达,微生物定殖也显著上调。总的来说,这种比较确定了对饥饿和微生物定植都有反应的候选基因。
饥饿反应基因也可能受到转录因子肝细胞核因子4 α (HNF4A)的控制。
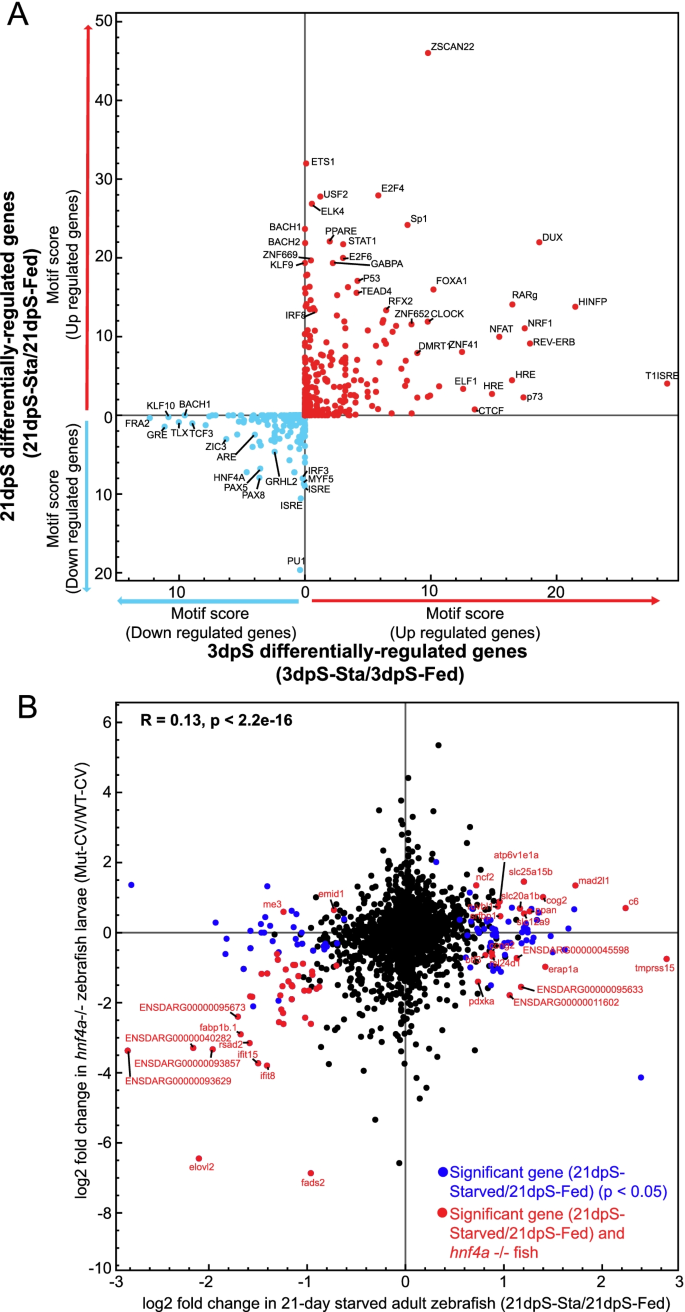
接下来,我们试图确定与饥饿反应调节有关的转录因子。使用荷马[77],我们查询了所有被确定为因饥饿而显著上调或下调的基因附近的基因组区域。我们将搜索范围限制在基因体内以及我们之前确定为斑马鱼肠道中可接近染色质的上游和下游侧翼10kb区域[51].这表明脊椎动物转录因子基序在饥饿反应基因附近显著富集在3dpS或21dpS(图2)。5一个;表的年代3.C).在3dpS和21dpS时,PAX5基序在下调基因附近显著富集,而FOXA1基序在上调基因附近显著富集。PAX5和FOXA1都与小鼠肠道发育有关[78,79].此外,在3dpS和21dpS时,HNF4A基序在下调基因附近显著富集。我们之前的研究表明,核受体HNF4A介导了斑马鱼对微生物定植的宿主转录反应[74].此外,在其他动物中,HNF4A是肠道特异性基因调控所必需的,并且在葡萄糖稳态、糖异生和脂质代谢中具有保守作用,这表明饥饿相关基因可能受HNF4A的控制[80,81,82,83].这一观察结果表明,HNF4转录因子可能促进对饥饿的反应和宿主微生物组的变化。
转录因子hnf4a可能调控与饥饿有关的基因子集。一个根据每个时间点转录起始位点上游或下游10kb内的转录因子结合位点(基于这些位点是否位于可达的染色区域)的HOMER分析,在3dpS (x轴)和21dpS (y轴)显著上调或下调的基因的基序富集分数的散点图。母题得分反映了log10p-值,以同一时间点相反方向调控的基因为背景,比较在指定时间点上调或下调的基因。HNF4A是在3dpS和21dpS下调的基因上结合位点富集的转录因子之一。B来自21dpS的基因的Log2倍变化(x轴)根据其消化道的Log2倍变化绘制hnf4a突变斑马鱼幼鱼与野生型对照(mutv - cv /WT-CV) (y轴)的比较,Davison等人[74].仅在饥饿斑马鱼中具有显著差异基因表达(21dpsSta/Fed)变化的基因用蓝色表示,而在两个数据集中均具有显著差异的基因用红色表示。皮尔逊相关性揭示了两个数据集之间的显著相关性(p< 0.05)
基于这些先前的发现,将我们的饥饿数据集与来自hnf4a突变斑马鱼[74证明了基因被认为是由hnf4a在我们的数据集中被显著下调(图2)。5B). GO Term分析显示,在这些重叠基因中,“脂质代谢过程”、“病毒反应”等多种代谢功能显著富集。具体来说,基因pla2g12b和elovl2,其他与脂质代谢有关的基因在饥饿的鱼体内被显著下调,在饥饿的鱼体内也被下调hnf4a−−/这意味着饥饿时的脂质代谢反应可能受到Hnf4a的积极调节。同样,在饥饿的鱼类中,几种免疫反应基因的调节也存在差异,比如Ifit15, ifit8, c6,和erap1a也受到了不同的监管/ hnf4a−−这表明对饥饿的免疫反应也可能部分受到Hnf4a功能的影响。
讨论
微生物组和宿主代谢之间的相互作用已知发生在多种病理生理背景下,包括饥饿和营养不良。然而,很少有先前的研究同时探讨宿主基因表达和微生物组组成的变化作为饥饿的功能[84].我们在这里关注的是肠道,它是动物与肠道微生物群和饮食营养物质的主要界面。我们的RNA-Seq数据表明,斑马鱼肠中细胞质翻译、核糖体基因和核糖体合成基因在饥饿早期上调,而DNA修复、维生素和辅助因子代谢基因在21dpS时上调。同样,21dpS时显著下调的一些通路与3dpS时明显下调的通路不同,其中21dpS包括参与抗病毒反应、精氨酸和脯氨酸代谢、甘油磷脂代谢等的基因。在3dpS和21dpS编码的不同功能表明饥饿的不同阶段,如先前在斑马鱼肝脏中描述的[52].与以往在其他器官中的研究形成鲜明对比的是,斑马鱼肠道中只有少数基因在再摄食后出现差异表达,这表明肠道在长时间饥饿期间表现出的适应性生理在再摄食后可以迅速逆转。在饥饿和冷冻的斑马鱼肝脏中,表达上调的基因在TCA循环、蛋白酶体组装、氧化磷酸化、DNA复制和修复等功能中富集[52].在牛的肝脏以及鲑鱼和鳟鱼的肌肉中也观察到类似的补偿机制[85,86,87].这些结果表明,与肌肉和肝脏等其他器官相比,肠道在适应饥饿和再进食方面可能具有特别的可塑性。
为了探索肠道对饥饿反应的基因调控机制,我们提供的证据表明hnf4a可能调控大量与饥饿相关的变化,扩大与该基因相关的大量生理功能。考虑到hnf4a斑马鱼和小鼠体内的微生物组抑制活性[74],hnf4a可能与饥饿期间宿主微生物组和转录组的改变有关。未来的研究可以使用ChIP-Seq测试饥饿对Hnf4a占用的影响,或对肠上皮中染色质可及性或组蛋白修饰的影响,以确定参与协调饥饿反应的顺式调控区域。我们的数据还提供了许多候选基因,这些基因可用于未来的实验,以探索斑马鱼肠道饥饿反应的特异性、区域性和调控。
尽管我们的RNA-Seq数据表明斑马鱼和其他脊椎动物的饥饿反应有许多共同点,但它也强调了斑马鱼肠道适应长期饥饿的独特方式。例如,我们观察到补体蛋白、核糖体蛋白的诱导,以及饥饿期间抗病毒反应的下调。这与虹鳟鱼肝脏形成对比,据报道,饥饿会降低核糖体蛋白的表达[67].同时,在21dpS时显著下调的基因包括几个参与抗病毒反应的基因。在其他动物饥饿的情况下,这些途径尚未被报道过,因此可能代表了斑马鱼特有的适应机制。
饥饿的斑马鱼表现出明显的生长减少,在21天的重新喂食时间内没有完全恢复。这表明身体从饥饿中完全恢复可能需要更多的时间,或者存在与饥饿相关的永久性身体变化。相反,我们发现饥饿诱导的斑马鱼肠道转录组和微生物组的变化在重新喂食后迅速正常化。与喂食对照组相比,饥饿显著影响了肠道中200多种独特基因的表达,而仅仅重新喂食3天,除了9种基因外,其他基因的表达都恢复了正常水平。3.B-C,图52D)。同样,饥饿后肠道微生物群落的多样性也显著增加(图2)。1D)和改变的成分(图2)。2A)与喂食对照组相比,但这些差异在重新喂食后1至3天内基本正常化。相比之下,另一种经历长期饥饿的动物,冬眠的地松鼠,在冬季冬眠的早期阶段保持肠道微生物群多样性的基线水平,在冬季后期减少多样性,然后在春季重新进食时增加多样性[88].重新喂食后多样性的增加归因于与引入食物相关的新细菌分类群。由于本研究没有采集食物相关细菌分类群的样本,因此我们无法区分这两种可能的解释。无论如何,饥饿和再摄食对斑马鱼和地松鼠肠道微生物群多样性的不同影响强调了研究不同动物宿主长期饥饿和再摄食的生态学和生理学的重要性。
我们的研究结果还提供了对斑马鱼肠道中对饥饿和重新进食最敏感的特定细菌谱系的见解。我们之前证明过弧菌和邻单胞菌属属是斑马鱼核心肠道微生物群的一部分[89].我们推测,这两个分类群相对丰度的相反变化可能反映了在饥饿肠道改变的环境中生存的不同能力。2B, c, e)。弧菌Sp.是斑马鱼肠道微生物群的常见成员,它们的相对丰度与肠道炎症呈正相关[41,42,48].这表明,饥饿引起的相对丰度的变化弧菌Sp.和斑马鱼肠道中的其他细菌可能与肠道基因表达的改变有关。在鸡的饥饿过程中,肠道粘液的数量和厚度增加,可能为黏液降解细菌创造了竞争优势[90,91].弧菌细菌可以降解肠道粘液,这可能就是为什么观察到肠道粘液增加的原因弧菌在饥饿期间[92,93].相反,邻单胞菌属可能不太适合在肠道内长时间的饥饿中生存。目前尚不清楚这些相对丰度的变化是否伴随着微生物群落密度的变化,这可以在未来的研究中进行探讨。令人惊讶的是,这些和其他饥饿引起的肠道微生物组组成的扰动,类似于肠道中宿主基因的表达,在重新喂食后的1到3天内基本恢复。这强调了肠道生理和微生物生态对饥饿和再喂养的显著可塑性。
方法
畜牧业
除非另有说明,所有鱼在28°C下保持14小时光照周期。用于这些实验的所有斑马鱼都是在同一天出生的,来自同一个兄弟姐妹的1对(RNA-Seq)或3对(16S rRNA基因扩增子测序和qRT-PCR测定)育种对。
用于RNA-Seq和16S rRNA基因扩增子测序的动物在杜克大学产生如下。将受精卵转移到含有蛋水(6 g海盐,1.5 g硫酸钙,0.75 g碳酸氢钠,10 - 12滴亚甲蓝,10 L水)的培养皿中,密度为50个/皿,28.5℃孵育。受精后1天(dpf),将胚胎转移到含有500 mL水的3 L水箱中,这些水来自循循环的斑马鱼养殖系统(系统水)。每个培养皿含有10个(16S rRNA基因测序)或30个(RNA-Seq)胚胎。然后在一个循循环的斑马鱼养殖系统中维持鱼的标准饲养,直到实验开始时为60dpf。然后将斑马鱼随机转移到4个(RNA-Seq)或8个(16S rRNA基因测序)清洁的10 L鱼缸中,每个鱼缸的密度为44条(RNA-Seq)或67条(16S rRNA基因测序),其中一半的鱼缸在接下来的21天内没有食物(图5)。1A).饥饿21 d后,允许所有水箱按照标准饲养方式进行饲养:每天两次投喂活Artemia,穿插两次投喂Gemma 300 (Skretting)。在21天的饥饿和21天的重新喂食中,所有鱼都留在循环水斑马鱼养殖系统中,我们在任何条件或实验中都没有观察到死亡。
用于qRT-PCR验证的动物在卡内基研究所产生如下。将受精卵以5dpf的速度转移到10 L的培养皿中,每个培养皿中约有70只幼虫。饲喂Gemma Micro (Skretting)饲料,逐渐增加颗粒大小。饲养前2周,幼虫纯饲Gemma Micro - 75,然后过渡到饲粮中添加活青蒿(www.artemia-international.com),在随后的3周内,将鱼过渡到成鱼饲料Gemma Micro 300,仅偶尔补充活盐水。以这种方式饲养的成虫被转移到12个密度和性别比例平衡的3升实验箱中,其中一半的箱在接下来的21天内不给食物。
所有在特定日期取样的鱼都是在鱼设施的第一次每日喂食之前收集的。在饥饿后0天(0dpS)、1dps、3dpS、7dpS、21dpS、再喂后1天(dpR)、3dpR、7dpR和21dpR采集16S rRNA基因扩增子测序样本(图2)。1A)在每个鱼缸的每个时间点随机选择6条鱼,用过量的三卡因(0.83 mg/ml三卡因)安乐死。鱼在解剖镜上成像,以方便随后的标准长度(SL)和肛鳍前高度(HAA)测量[94].然后从每条鱼身上解剖肠道,分别置于裂解缓冲液(20 mM Tris-HCl (pH 8.0), 2 mM EDTA (pH 8.0), 1% Triton X-100)中,在干冰/乙醇浴中快速冷冻,并在- 80°C保存,直到提取DNA。
RNA-Seq样品于3dpS、21dpS和3dpR采集。在每个时间点,每个鱼缸随机选择3只鱼,使用过量三卡因(0.83 mg/ml三卡因)实施安乐死。鱼在解剖镜上成像,以方便随后的标准长度(SL) [94].然后从每条鱼身上解剖肠道,单独放置在2ml充满TRIzol试剂(Thermo Fisher, 15,596,026)的冷冻瓶中,在干冰-乙醇浴中快速冷冻,并在- 80°C保存,直到RNA提取。
在21dpS时收集qRT-PCR样本。用过量的三卡因对鱼实施安乐死,解剖鱼肠,分别保存在0.5 mL RNAlater中。
16S rRNA基因测序
基因组DNA从斑马鱼个体的肠道中提取,使用Qiagen DNA血液和组织试剂盒(Qiagen,如前所述,经头部跳动修饰)[42].随后以基因组DNA为模板,对16S rRNA基因的v4区进行PCR扩增,在Illumina HiSeq 2000测序系统上进行150 bp/cycle的配对端测序(见表5)4(引物)在俄勒冈大学基因组学和细胞表征核心设施。
16S rRNA基因序列生物信息学及统计学分析
使用QIIME (v1.9.1)按样本ID对FASTQ文件进行解复用和拆分。在RStudio 3.4.1版本中,这些文件被进行了质量过滤、裁剪、去噪、合并、嵌合体检查,并使用DADA2进行分类。使用Silva v132数据库进行分类分配。数据分析使用R软件包vegan和phyloseq。LEFsE是通过胡佛星系网站访问的:https://huttenhower.sph.harvard.edu/galaxy/。以下软件包用于分析和图形的创建:猿,picante, vegan, ggtree, SEPP, ployly, heatmap和pairwiseadonis [95,96,97,98,99,One hundred.,101,102].
RNA提取与测序
在- 80°C下保存的冷冻全肠样品,使用氧化锆珠(Biospec, 11079107)和配有Vortex Adapter (Scientific Industries, 1311-V)的Vortex Genie2 (Scientific Industries, 13000 - v1 - 24)在三次45 s间隔内均质。样品在均质过程中放置在冰上以防止过热。均质后,每个样品加入200ul氯仿进行相分离,并通过强力反转混合15次。然后样品在室温下孵育3分钟,在4℃下在12000rcf下离心15分钟。然后将每个样品的500ul上相水转移到一个新的Eppendorf管中,在DEPC水中加入500ul 70%乙醇并进行涡流。相分离后,样品进行DNase处理,根据制造商的说明,使用PureLink DNase Set (Thermo Fisher, 12,185,010)和PureLink RNA Mini kit (Thermo Fisher, 12,183,025)通过柱纯化提取总RNA。通过分光光度法评估最终样品的质量和浓度,并将样品保存在-80°C,直到提交给杜克测序和基因组技术核心。RNA-seq文库由Duke Sequencing和Genomic Technologies Core在Illumina HiSeq 2500上进行测序,生成50 bp的单端reads (SR)。
RNA-seq生物信息学
所有原始的斑马鱼RNA-seq数据都在Galaxy服务器上处理[103].使用Trim Galore对原始fastq文件进行裁剪。104].然后使用STAR将修剪后的fastq文件映射到斑马鱼基因组(GRCz10),使用默认设置生成BAM文件,使用HTSeq (v.0.9.1)将其转换为计数。在UCSC基因组浏览器上可视化之前,使用bamCompare工具(v2.5.0)将BAM文件转换为bigWig文件。105,106,107].
通过Salmon获得转录本的TPM表达值[108].利用HTSeq生成的计数文件,用DESeq2进行两两差异基因表达检测[108,109].对于饥饿和喂食鱼之间的比较,默认的显著性阈值调整p每次比较均采用- 0.05。对于饲料鱼对照的比较,显著性阈值定义为基因具有绝对对数2折叠变化大于1.0或ap-值小于0.05(见图。年代1,年代3.,表53.B)。包含通过该过滤步骤去除的基因的分析版本见图S2。
日志的分层聚类2使用Cluster 3.0计算基因的折叠变化值,使用Java Treeview生成热图[110,111].
HOMER软件(http://homer.ucsd.edu/homer/motif/)对3dpS和21dpS(分别为3dpS饥饿/3dpS喂养和21dpS饥饿/21dpS喂养)中显著上调和下调的基因进行了分析,使用基因体内的区域以及我们之前确定为斑马鱼肠道中可接近染色质的上游和下游侧翼10kb区域[51使用findMotifs.pl命令。通过取-log得到motif分数10的值p-由HOMER指定的值。在每个时间点(3dpS或21dpS),一个基序在上调或下调的基因中被认为是“富集”的,这取决于它在上调或下调的基因集中是否具有更高的基序评分。
为了与斑马鱼幼虫卵黄喂养数据集进行比较,log221dpS喂食的鱼相对于21dpS饥饿的鱼的变化是log的两倍2饲喂蛋黄后4h斑马鱼幼虫消化道的折线变化(即“HF 4h logFC”),数据来自补充表1在[59],其原始数据可在GSE87704上获得。为了比较hnf4a突变体和微生物定植数据集,数据来自补充表2在[74],使用日志2消化道的折叠变化hnf4a在常规无菌条件下饲养的纯合突变型和野生型6dpf斑马鱼幼鱼(“MutCV/WTCV”)和在无菌或无菌常规条件下饲养的野生型6dpf斑马鱼幼鱼(“WTGF/WTCV”)。原始数据来自[74]的网址为GSE90462。
定量RT-PCR检测
16条饥饿鱼和15条喂食5 - 6个月大的鱼的解剖肠被储存在RNAlater,并运往罗尔斯实验室。组织用头部加热管(Bertin Corp, P000912-LYSK0-A)均质,用氯仿提取RNA,用异丙醇沉淀,用乙醇洗涤,用DNase (Invitrogen, AM1906)处理。使用Qubit RNA BR Assay Kit (ThermoFisher, Q10210)检测RNA浓度。然后使用iScript cDNA合成试剂盒(Bio-Rad, 1708891)逆转录800 ng RNA。采用基因特异性引物在quantibio Studio6 (Applied Biosystems)上进行实时荧光定量PCRpla2g12b(f 5 ' - cagtaccgctgcagatggt -3 ', r 5 ' - attcggtacctgagccaag -3 '),elovl2(f 5 ' - cagttttcctgtcccgt -3 ', r 5 ' - cagttttcctgtcccgt -3 ')apoa4b.2(f5 ' - ttgtggtctttgcttgct -3 ', r5 ' - TCATCTTGACGGTTTCCTCTG-3 ')和ef1a(5 ' - cttctcaggctgactgtgc-3 ', r 5 ' - ccgctaggattaccctcc-3 ')。每组随机抽取6份样本(男3名,女3名),用2X SYBR Green SuperMix (PerfeCTa, Hi Rox, Quanta Biosciences, 95055)进行重复25 μl反应分析,归一化至表达ef1a作为一个管家基因。采用∆∆CT法测定表达谱及相关统计参数,并在Prism (GraphPad)中作图。
数据和材料的可用性
生成序列变体的所有质量过滤参数、16S rRNA分析的ASV表和图可在以下网站找到:https://github.com/alexmccumber/fishguts。原始16S rRNA基因扩增子FASTQ文件可从欧洲核苷酸档案馆获取,项目访问号为PRJEB31503。原始和处理的RNA-Seq数据可在NCBI GEO上获得,登录号为GSE140821。
参考文献
Watts, M. J. & Bohle, H. G.脆弱性空间:饥饿与饥荒的因果结构:人文地理进展(2016)doi:https://doi.org/10.1177/030913259301700103。
费伦·K,等。癌症恶病质的定义和分类:国际共识。中华医学杂志,2011;12:489-95。
野生动物的食物需求:自由生活的哺乳动物、爬行动物和鸟类的预测方程;2018.
饥饿生理学:回顾动物在共同挑战中生存的不同策略。中国生物医学工程学报,2010;32(1):1 - 8。
层KB,层JM。厌氧症的代谢率降低和生化适应。生物学报。1990;65:145-74。
Nagy KA, Girard IA, Brown TK。自由放养的哺乳动物、爬行动物和鸟类的能量学。神经网络学报。1999;19:247-77。
衰老过程中骨骼肌质量的减少:饥饿、肌肉减少症和恶病质之间关系的研究。中华医学杂志,2007;26(3):389 - 99。
爱啊。营养不良的代谢反应:与肠内喂养的相关性。肠道。1986;27:9-13。
摩尔FD。能量和身体细胞质量的维持。[J]父母进入营养学。1980;4:228-60。
麦克法兰H,等。免疫、转铁蛋白和夸希奥尔科患者的存活率。[j] .中华医学杂志,2007;4:368 - 368。
Champakam S, Srikantia SG, Gopalan C. Kwashiorkor与心理发展。[J] .中华医学杂志。1998;21(1):444 - 444。
艾奇逊RM,麦金泰尔MN。急性感染和急性饥饿对骨骼发育的影响。[J]中华检验医学杂志,1998;39(3):337 - 345。
巴勒斯坦JA, Dudrick SJ。饥饿和再喂养的金凤花模式。中华临床医学杂志,2006;21:147-54。
卡希尔的女朋友。人类的饥饿。中华医学杂志。1970;28(2):668 - 75。
Felig P, Owen OE, Wahren J, Cahill GF。长时间饥饿时的氨基酸代谢。[J]中华医学杂志,2009;48(4):584 - 594。
dsamset C等。鸡肝脏从摄食到禁食转变的转录组分析。生物医学工程学报。2008;9:611。
Drew RE等人。饥饿对成年雌性斑马鱼脑和肝脏转录组的影响。中国生物医学工程学报。2008;35(5):393 - 394。
哈克沃特,t.b.m.等。小鼠对禁食适应性反应的器官间协调。生物。化学。jbc.M110.216986 (2011) doi:https://doi.org/10.1074/jbc.M110.216986。
Crawford PA,等。营养剥夺时肠道菌群对心肌酮体代谢的调节。《美国国家科学院学报》上。2009; 106:11276 - 81。
David LA等人。饮食会迅速且可重复地改变人体肠道微生物群。大自然。2014;505:559 - 63。
Hildebrandt, m.a.等。高脂肪饮食决定了小鼠肠道微生物群的组成,与肥胖无关。胃肠病学137,1716-1724。e1-2(2009)。
特恩博PJ, Bäckhed F,富尔顿L,戈登JI。饮食引起的肥胖与小鼠远端肠道微生物群的显著但可逆的改变有关。中国生物医学工程学报。2008;3(3):213 - 223。
Basolo, A.等。摄食不足和口服万古霉素对人体肠道微生物群和营养吸收的影响。自然医学1-10 (2020)doi:https://doi.org/10.1038/s41591-020-0801-z。
Backhed, F. et al.肠道微生物群作为调节脂肪储存的环境因素。美国国家科学院院刊,2004年。
Smith MI,等。马拉维双胞胎的肠道微生物组不一致的夸希奥克尔病。科学。2013;339:548-54。
Ridaura VK等。肥胖双胞胎肠道菌群调节小鼠代谢。科学。2013;341:1241214。
Cowardin CA等。唾液化乳寡糖影响婴儿营养不良小鼠骨生物学的机制。《美国国家科学院学报》上。2019; 116:11988 - 96。
希伯德,m.c.等。微量营养素缺乏对人类肠道菌群细菌种类的影响。中国科学(英文版)2017(9):444 - 444。
Gehrig, j.l.等。微生物导向食物对非生动物和营养不良儿童的影响。Science 2019 365, eaau4732。
Leulier F等。综合生理学:处于营养学、微生物群、动物生理学和人类健康的十字路口。中华医学杂志,2017;25:522 - 534。
Gas N, noailliak - depeyre J.长时间禁食对肠上皮内化的影响。[J]中国生物医学工程学报,2006;22(6):557 - 557。
sener, H. & Braunbeck, T.肝脏细胞对极端营养条件的适应;黑斑柳l .(鲤科)。形态功能分析。水生生物学报,1998,18(5):559 - 557。
Lenaerts K等人。饥饿诱导小鼠小肠蛋白质组的阶段性变化。[J]中国生物医学工程学报,2006;5(5):1145 - 1145。
saomome I, Curto M, McClatchey AI。在发育中的肠中,Ezrin对上皮组织和绒毛形态发生至关重要。Dev Cell. 2004; 6:855-64。
李建军,李建军,李建军。禁食和复食大鼠肠道细胞凋亡变化与代谢状态的关系。中国生物医学工程学报。2006;31(1):559 - 559。
刘建军,刘建军,刘建军,等。斑马鱼尾鳍发育过程中生长控制的研究进展。开发生物学。2006;29:450 - 7。
弗林EJ,特伦特CM,罗尔斯JF。斑马鱼脂肪生成的个体发生与营养调控。[J]中国生物医学工程学报,2009;30(5):591 - 591。
McMenamin SK, Minchin JEN, Gordon TN, Rawls JF, Parichy DM. gh1突变斑马鱼的侏儒症和肥胖增加。内分泌学。2013;154:1476 - 87。
任明钦,罗尔斯。斑马鱼脂肪组织的分类系统。Dis Model Mech. 2017; 10:797-809。
吴安等。。斑马鱼消化系统的形成:三。肠上皮的形态发生。中国生物医学工程学报,2005,28(6):1145 - 1145。
罗尔斯JF,塞缪尔BS,戈登JI。白生斑马鱼揭示了对肠道微生物群的进化保守反应。《美国国家科学院学报》上。2004; 101:4596 - 601。
Stephens WZ等。斑马鱼肠道微生物群落的组成随着发育而变化。[j] . 2016; 10:644-54。
Semova I等。微生物群调节斑马鱼肠道对脂肪酸的吸收和代谢。细胞与宿主微生物。2012;12:277-88。
黄。et al。饮食脂肪的个体发生差异影响斑马鱼肠道微生物群的聚集。mBio 2015; 6: e00687-e00615。
斑马鱼肠道发育的独特和保守方面。开发生物学。2003;25(5):12 - 29。
Cheesman SE, Neal JT, Mittge E, Seredick BM, Guillemin K.发育中的斑马鱼肠上皮细胞增殖受Wnt通路和Myd88微生物信号的调控。科学通报,2011;33(增刊1):1 - 7。
Bates JM,等。来自微生物群的不同信号促进了斑马鱼肠道分化的不同方面。发展生物学。2006;29:374 - 86。
Rolig AS, Parthasarathy R, Burns AR, Bohannan BJM, Guillemin K.微生物群的个体成员不比例地调节宿主先天免疫反应。细胞与宿主微生物。2015;18:613-20。
默多克CC,等。肠道血清淀粉样蛋白a抑制系统中性粒细胞的激活和杀菌活性,以响应微生物群定植。中华医学杂志,2019;15:e1007381。
Kanther M,等。微生物定植诱导斑马鱼消化道NF-κB激活的动态时空格局。胃肠病学。2011;141:197 - 207。
Lickwar CR等。肠上皮细胞保守转录调控的基因组解剖。生物科学进展,2017;15:e2002054。
贾杰,等。斑马鱼(Danio rerio)对禁食和随后再喂养的肝脏反应的微阵列和代谢组分析。生物医学工程学报,2019;20:19 19。
Minchin JEN, Scahill CM, Staudt N, Busch-Nentwich EM, Rawls JF。斑马鱼的深层表型揭示了遗传和饮食诱导的肥胖变化,这些变化可能会通知疾病风险。[J]中国生物医学工程学报,2018;39(5):536 - 536。
Burns AR等。中性过程对斑马鱼宿主发育过程中肠道微生物群落组装的贡献。[j] . 2016;10:65 - 64。
罗尔斯JF,马霍瓦尔德MA,莱利RE,戈登JI。从斑马鱼和小鼠到无菌接受者的互惠肠道微生物群移植揭示了宿主栖息地选择。细胞。2006;127:423-33。
基于“增大化现实”技术,B。et al。在实验斑马鱼模型中,宿主间扩散改变了微生物组的组装,并可以压倒宿主的先天免疫。《美国国家科学院院刊》第114卷http://pubmed.ncbi.nlm.nih.gov/28973938/(2017)。
Segata N,等。宏基因组生物标志物的发现与解释。中国生物医学工程学报,2011;16(2):564 - 564。
Morais S, Monroig O, Zheng X, Leaver MJ, Tocher DR.大西洋鲑鱼高不饱和脂肪酸合成:ELOVL5-和elovl2样延长酶的表征。生物工程学报,2009;11:627-39。
Zeituni, e。M。et al。内质网脂质通量影响肠细胞核形态和脂质依赖性转录反应。生物。化学。jbc.M116.749358 (2016) doi:https://doi.org/10.1074/jbc.M116.749358。
王莹,等。大鼠脂肪酸延长酶的组织特异性、营养和发育调控。[J]中国生物医学工程学报,2005;16(6):559 - 564。
crucani - guglielmacci C等。代谢刺激下的多个小鼠品系的分子表型揭示了Elovl2在葡萄糖诱导的胰岛素分泌中的作用。分子代谢学报。2017;6:340-51。
Alimuddin, Kiron, V., Satoh, S., Takeuchi, T.和Yoshizaki, G.克隆和masu三文鱼的过表达(雄鱼masou)脂肪酸延长酶样基因在斑马鱼。水产养殖2008;十三至十八,282。
Antalis TM, Shea-Donohue T, Vogel SN, Sears C, Fasano A.肠道黏膜蛋白酶的病理机制。中华胃肠病杂志,2007;4:393-402。
Mukherjee S, Hooper LV。肠道的抗菌防御。免疫力。2015;42:28-39。
Holzinger A,等。肠肽酶基因突变是先天性肠肽酶缺乏的分子原因。[J] .中国生物医学工程学报。2002;37(1):1 - 5。
刘建军,刘建军,刘建军。LipoGlo报告系统用于敏感和特异性监测致动脉粥样硬化脂蛋白。学报。2019;10:34 . 26。
王晓明,王晓明,王晓明,等。饥饿对虹鳟鱼基因表达和蛋白水解的影响。生物医学工程学报。2007;8:328。
卢德龙,等。禁食通过刺激脂质分解代谢和自噬增强鱼类的抗寒能力。中国生物医学工程学报(英文版);2019;37(5):585 - 593。
宋辉,严勇,Titus T,何欣,Postlethwait JH。stat1b在斑马鱼造血中的作用。机甲发展。2011;128:442-56。
Richmond CA等。JAK/STAT-1信号是急性炎症后肠道再生过程中储备肠干细胞激活所必需的。干细胞报告。2017;10:17-26。
Dobson JT等。羧基肽酶A5在斑马鱼中鉴定出一种新的肥大细胞谱系,为肥大细胞命运的决定提供了新的见解。血。2008;112:2969 - 72。
刘建军,刘建军,李建军,等。长冬禁食对13列地松鼠肝脏蛋白质组的影响。生物工程学报。2014;46(6):348 - 61。
2D DIGE蛋白质组学分析揭示了小鼠通过器官特异性转录因子(S)禁食诱导的蛋白质重塑。生物学报,2018;8:1524-43。
Davison JM等。微生物群通过抑制转录因子肝细胞核因子4 α调控肠上皮基因表达。基因工程学报,2017;27:1195-206。
j·G·坎普。et al。微生物群调节肠上皮的转录而不重塑可接近的染色质景观。Genome Res. gr.165845.113 (2014) doi:https://doi.org/10.1101/gr.165845.113。
Larsson E,等。通过MyD88分析肠道微生物沿肠道长度方向对宿主基因表达的调控及肠道微生物生态的调控。肠道。2012;61:1124-31。
Heinz S等。决定谱系的转录因子的简单组合是巨噬细胞和B细胞身份所需的顺式调节元件。生物医学工程学报。2010;38:576 - 589。
徐海,等。糖皮质激素对肠道NHE8表达的转录抑制涉及Pax5。[J] .中国生物医学工程学报。2010;31(2):555 - 557。
Kaestner KH。肝脏的形成:转录调控因子HNF4α的众多靶标。肝脏病学。2010;51:376-7。
Roman AKS, Aronson BE, Krasinski SD, Shivdasani RA, Verzi MP。转录因子GATA4和HNF4A与转录因子CDX2一起控制肠道内稳态的不同方面。中国生物医学工程学报,2015;29(1):1 - 6。
张国强,张国强,张国强。果蝇HNF4调节脂质动员和β-氧化。中华医学杂志。2009;9:228-39。
Frochot V等人。转录因子HNF-4α:小鼠肠道脂肪酸摄取的关键因子。[J] .中国生物医学工程学报,2012;31(2):563 - 563。
Barry WE, Thummel CS。果蝇HNF4核受体促进成人葡萄糖刺激的胰岛素分泌和线粒体功能。Elife。2016;5。
夏建辉,等。饥饿状态下鱼类的肠道微生物群。生物医学工程学报。2014;15:266。
重新扫描P-Y等。禁食-再摄食计划诱导鱼类肌肉恢复生长过程中的动态基因表达。生物医学工程学报,2007;8:438。
Connor EE,等。线粒体复合体基因功能的增强和肝脏体积的减小可能介导了肉牛代偿生长期间饲料效率的提高。功能集成基因组学。2010;10:39-51。
李春华,李春华,李春华,等。鲑鱼肌肉组织中肌纤维的表达变化及其对肌肉纤维生成的影响。生物医学工程学报,2017;18:447。
Carey HV, Walters WA, Knight R.在每年的冬眠周期中,地鼠肠道微生物群的季节性重组。[J] .中国生物医学工程学报,2013;33(4):563 - 567。
罗泽尔,等。斑马鱼体内存在核心肠道微生物群的证据。[j] . 2011; 5:1595-608。
施罗德BO。要么对抗它们,要么喂养它们:肠道黏液层如何管理肠道微生物群。Gastroenterol Rep (Oxf)。2019; 7:3-12。
Smirnov A, Sklan D, Uni Z.饥饿改变了小鸡小肠中的粘蛋白动力学。[J]中华医学杂志。2004;34(4):736 - 742。
Szabady RL, Yanta JH, Halladin DK, Schofield MJ, Welch RA。TagA是霍乱弧菌分泌的一种蛋白酶,能特异性地裂解粘蛋白糖蛋白。微生物学。2011;157:516-25。
Bhowmick R,等。霍乱弧菌的肠道粘附涉及定植因子GbpA和粘蛋白之间的协调相互作用。感染免疫。2008;76:468 - 477。
Parichy DM, Elizondo MR, Mills MG, Gordon TN, Engeszer RE.胚胎后斑马鱼发育的正常表:通过活鱼的外部可见解剖分期。开发应用。2009;38:2975 - 3015。
张建军,张建军,张建军,等。汉语R语言的进化与系统发育。生物信息学。2004;20:289 - 90。
Sievert, C. et al. plotly:通过plotly创建交互式Web图形。js”。R包版本2017;4,110。
Galili T, O 'Callaghan A. Sidi, J. & Sievert, C. heatmap:一个用于创建在线发布交互式集群热图的R包。生物信息学。2018;34:1600-2。
Kembel SW等人。整合系统发生学和生态学的R工具。生物信息学。2010;26:1463-4。
Oksanen J,等。素食包装。社区生态包。2007; 10:631-7。
于刚,史密斯达克,朱华,关勇,林天勇。Ggtree:一个可视化和注释系统发育树及其协变量和其他相关数据的软件包。方法生态学杂志,2017;8:28-36。
詹森,S。et al。精确扩增子序列的系统发育定位改善了与临床信息的关联。mSystems 2018; 3: e00021-e00018。
McMurdie P. J. & Holmes S. phyloseq:一个可重复交互分析和微生物组普查数据图形的R包。科学通报,2013;8:e61217。
阿富汗E,等。可获取、可重复和协作的生物医学分析银河平台:2018年更新。中国生物医学工程学报,2018;46:537 - 544。
F.克鲁格。一个围绕Cutadapt和FastQC的包装工具,可以始终如一地对FastQ文件进行质量和适配器修剪。
Casper J,等。UCSC基因组浏览器数据库:2018年更新。中国生物医学工程学报,2018;33(4):762 - 769。
Dobin A等。STAR:超快速通用rna序列比对仪。生物信息学。2013;29:15-21。
Ramírez F等人deepTools2:用于深度测序数据分析的下一代web服务器。中国生物医学工程学报,2016;44(4):564 - 564。
Patro R, Duggal G, Love MI, Irizarry RA, Kingsford C. Salmon提供了快速和偏差感知的转录表达定量。Nat Methods. 2017; 14:417-9。
Love MI, Huber W, Anders S.使用DESeq2对RNA-seq数据的折叠变化和离散度进行调节估计。基因组生物学。2014;15:550。
J.基于树视图的微阵列数据可视化。生物信息学。2004;20:3246-8。
de Hoon MJL, Imoto S, Nolan J, Miyano S.开源集群软件。生物信息学。2004;20:1453-4。
致谢
我们感谢Joshua Granek博士,他作为IBIEM的一部分提供指导,没有他,我们就无法完成这项工作;我们感谢Doug Turnbull博士,他提供了有益的建议。
资金
这项工作得到了NIH (R01-GM095385)和Gordon and Betty Moore Foundation对b.j.m.b.、k.g.和J.F.R的资助;以及美国国立卫生研究院的J.F.R (R01-DK093399, R01-DK111857和R01-DK081426)。J.J.得到了美国心脏协会博士预科奖学金(825770)的支持。A.W.M.和C.A.获得了美国国家科学基金会研究培训计划“微生物组研究和工程的综合生物信息学”或IBIEM(1545220)的支持。A.W.M.也得到了生物分子和组织工程中心(NIH T32-GM008555)的支持。这项工作还得到了卡内基科学基金会和g·哈罗德和莱拉·y·马瑟斯慈善基金会对S.A.F.的资助。
作者信息
作者及单位
贡献
j.r., k.g., b.b., s.w.和S.G.构思并策划了这项研究。s.w., s.g., j.t.和M.M.进行了动物实验并生成了数据。a.m.、j.j.、c.l.、c.a.和M.M.分析了数据。a.m.、J.J.和M.M.起草了手稿,并制作了图表。部件点,j。,。,南卡罗来纳州,是以,其它,jr编辑了手稿。所有作者都审阅了手稿。j。r。和c。l。监督这个项目。j.r., k.g., b.b.和S.F.获得了资助。作者阅读并批准了最后的手稿。
相应的作者
道德声明
伦理批准并同意参与
所有斑马鱼实验均按照杜克大学机构动物护理和使用委员会(IACUC)批准的A165-13-06和A115-16-05协议,以及卡内基科学研究所IACUC批准的#139协议,按照公共卫生服务政策对实验动物进行人道关怀和使用。本研究是按照ARRIVE指南完成的。
发表同意书
不适用。
相互竞争的利益
作者宣称他们没有竞争利益。
额外的信息
出版商的注意
伟德体育在线施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。
补充信息
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接获得版权所有者的许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非在数据的信用额度中另有说明。
关于本文
引用本文
贾瓦哈尔,J.,麦坎伯,a.w.,利克瓦尔,C.R.et al。饥饿引起肠道转录组和微生物组的变化,而这些变化在重新进食时被逆转。BMC基因组学23, 225(2022)。https://doi.org/10.1186/s12864-022-08447-2
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12864-022-08447-2