- 研究文章gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
帕金森病相关肠道微生物组的改变预测了代谢功能的疾病相关变化gydF4y2Ba
BMC生物学gydF4y2Ba体积gydF4y2Ba18gydF4y2Ba、物品编号:gydF4y2Ba62gydF4y2Ba(gydF4y2Ba2020gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
帕金森病(PD)是一种全身性疾病,临床上以大脑多巴胺能神经元变性为特征。虽然PD中已经报道了肠道微生物组组成的改变,但其功能后果尚不清楚。在此,我们通过对卢森堡帕金森研究(gydF4y2BangydF4y2Ba= 147例典型PD病例;gydF4y2BangydF4y2Ba= 162个对照)。gydF4y2Ba
结果gydF4y2Ba
所有人都进行了详细的临床评估,包括神经学检查和神经心理学测试,然后进行自我报告问卷调查。首先对这些人的粪便样本进行16S rRNA基因测序分析。其次,我们利用微生物组数据和人类肠道微生物基因组尺度的代谢重建,通过个性化代谢模型预测了129种微生物代谢物的潜在分泌。我们的主要结果包括以下内容。PD患者与健康对照间8属和7种相对丰度发生显著变化。pd相关的微生物模式在统计学上取决于性别、年龄、BMI和便秘。特别是,相对丰度gydF4y2BaBilophilagydF4y2Ba和gydF4y2BaParaprevotellagydF4y2Ba在控制病程后与Hoehn和Yahr分期显著相关。此外,肠道微生物组的个性化代谢模型揭示了PD中9种微生物代谢物的预测分泌潜力与PD相关的代谢模式,包括蛋氨酸和半胱氨酸的增加。预测的微生物泛酸生产潜力与特定的非运动症状的存在有关。gydF4y2Ba
结论gydF4y2Ba
我们的研究结果表明,pd相关的肠道微生物组改变可以转化为影响宿主代谢和疾病表型的实质性功能差异。gydF4y2Ba
背景gydF4y2Ba
帕金森病(PD)是一种复杂的多因素疾病,遗传和环境因素共同影响疾病的演变和进展[gydF4y2Ba1gydF4y2Ba]。虽然一些研究已经阐明了遗传因素在疾病发病机制中的作用[gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba],各种环境和生活方式因素的作用和贡献仍未完全了解[gydF4y2Ba6gydF4y2Ba]。重要的是,大约60%的PD患者患有便秘[gydF4y2Ba7gydF4y2Ba],可在诊断前20年开始,是前驱症状之一[gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba]。gydF4y2Ba
认识到宿主与微生物之间复杂的相互作用,人类被认为是超级有机体[gydF4y2Ba10gydF4y2Ba]。例如,人类肠道微生物群已被证明具有补充宿主的基本功能(营养、代谢和保护),并通过肠-脑轴调节神经通路和氨基丁酸能和血清素能信号系统影响宿主的中枢神经系统(CNS) [gydF4y2Ba11gydF4y2Ba]。gydF4y2Ba
最近的研究报道了PD患者肠道成分的改变[gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba]。这些研究表明,与年龄匹配的对照组相比,PD患者的微生物组组成发生了变化。然而,改变的微生物组的功能影响仍有待阐明,例如使用动物模型[gydF4y2Ba21gydF4y2Ba]。一种补充方法是计算建模,或基于约束的重建和分析(COBRA) [gydF4y2Ba22gydF4y2Ba],微生物水平的代谢。在这种方法中,数百种肠道微生物的代谢重建[gydF4y2Ba23gydF4y2Ba]根据微生物组数据进行组合[gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba]。通量平衡分析[gydF4y2Ba22gydF4y2Ba]然后用于计算,例如,每种微生物组模型的可能代谢物摄取或分泌通量率(微生物组代谢谱)[gydF4y2Ba25gydF4y2Ba]或研究微生物代谢相互作用(交叉喂养)[gydF4y2Ba27gydF4y2Ba,gydF4y2Ba28gydF4y2Ba]。该方法已应用于各种微生物组数据集,以获得功能见解[gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba29gydF4y2Ba],包括PD,我们提出微生物硫代谢可能有助于PD患者血液代谢组的观察变化[gydF4y2Ba29gydF4y2Ba]。gydF4y2Ba
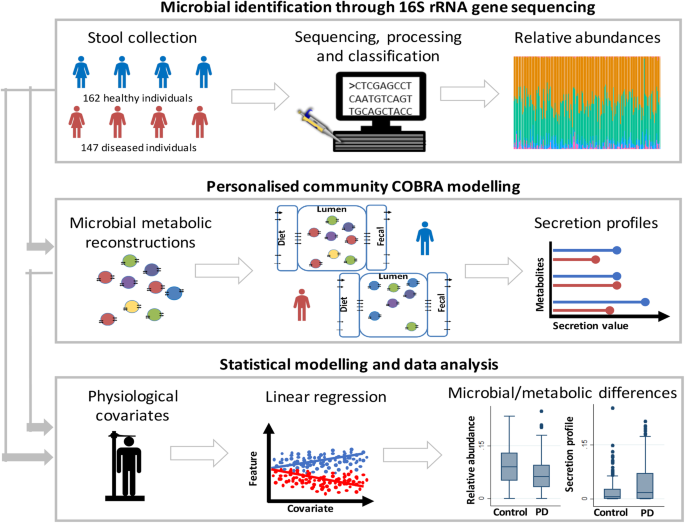
在本研究中,我们旨在研究与PD相关的微生物变化,同时关注影响微生物组成的可能协变量,并提出微生物组变化引起的功能,即代谢后果。首先,我们分析了来自卢森堡帕金森研究的PD患者和对照组的粪便微生物组成[gydF4y2Ba30.gydF4y2Ba(图。gydF4y2Ba1gydF4y2Ba).其次,基于观察到的PD患者和对照组之间微生物群落组成的显著差异,我们创建并询问了代表每个个体微生物群落代谢的个性化计算模型。我们证明,结合微生物组成和功能代谢物分析为PD和疾病严重程度相关的微生物变化提供了新的假设,为未来基于机制的实验提供了可能。gydF4y2Ba
概述研究方法和使用的主要方法。相对丰度来源于16S rRNA基因序列(gydF4y2Ba16S rRNA基因测序分析微生物组成gydF4y2Ba”部分),并用作个性化社区建模的输入,以模拟代谢物分泌概况。相对丰度和分泌谱进行统计分析,以确定PD患者和对照组之间的微生物或代谢差异gydF4y2Ba
结果gydF4y2Ba
卢森堡帕金森氏病研究包括来自卢森堡及其邻近地区的典型帕金森氏病、非典型帕金森氏病和任何疾病阶段的继发性帕金森氏病患者,以及年龄/性别匹配的健康对照受试者,年龄范围广泛[gydF4y2Ba30.gydF4y2Ba]。在本研究中,我们关注的是典型的PD患者和50岁以上的健康对照(表1)gydF4y2Ba1gydF4y2Ba,”gydF4y2Ba方法gydF4y2Ba”一节)。利用16S rRNA基因序列对147名PD患者和162名对照者的粪便样本进行了分析。gydF4y2Ba16S rRNA基因测序分析微生物组成gydF4y2Ba”一节)。在这309个人中,有一个人因为缺少身体质量指数(BMI)而被排除在分析之外,结果308个人被纳入统计分析。请注意,由于其他变量(如临床评估)的缺失值,个别统计分析的病例数可能会较低(见表)gydF4y2Ba1gydF4y2Ba).gydF4y2Ba
β多样性在PD微生物群落中发生改变gydF4y2Ba
我们分析了健康对照和PD微生物组的α和β多样性指数。这些分析是通过线性回归对308名具有完整协变量数据的个体进行的。在alpha多样性方面,我们计算了物种丰富度、Shannon熵和均匀度Pielou指数[gydF4y2Ba31gydF4y2Ba]。PD病例和对照组之间的Shannon熵没有显著差异,这与早期的研究一致[gydF4y2Ba12gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba]但与两项不同的PD研究不一致[gydF4y2Ba13gydF4y2Ba,gydF4y2Ba16gydF4y2Ba]。物种丰富度随PD(回归系数)的增加而略有增加gydF4y2BabgydF4y2Ba= 4.76, 95%可信区间(CI) 0.44;9.08,gydF4y2BapgydF4y2Ba= 0.03)(附加文件gydF4y2Ba1gydF4y2Ba:图S1)。重要的是,我们发现在Pielou指数中存在显著的性别群体相互作用项,PD的效应符号被逆转。女性PD患者的Pielou指数降低,而男性PD患者的Pielou指数升高gydF4y2Ba1gydF4y2Ba:图S1)。然而,最值得注意的是男性Pielou指数的差异增加gydF4y2Ba1gydF4y2Ba:图S1)。这些结果表明了α多样性的性别依赖性,尽管这一孤立的结果需要验证。然而,疾病背景下性别依赖的微生物组变化至少在炎症性肠病的小鼠模型中得到了描述,并在肠-脑轴的背景下进行了讨论[gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba]。因此,性别-微生物组在健康和疾病中的相互作用值得进一步研究。gydF4y2Ba
在β多样性方面,对布雷-柯蒂斯差异进行的ANOSIM分析表明,PD与健康微生物群落之间存在微小但显著的差异(ANOSIM统计)gydF4y2BaRgydF4y2Ba= 0.04,gydF4y2BapgydF4y2Ba= 0.001)。因此,β多样性证实了先前的结果[gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba]),健康和帕金森病微生物群落之间存在差异(附加文件gydF4y2Ba1gydF4y2Ba:图S2)。gydF4y2Ba
PD微生物组的种属水平变化gydF4y2Ba
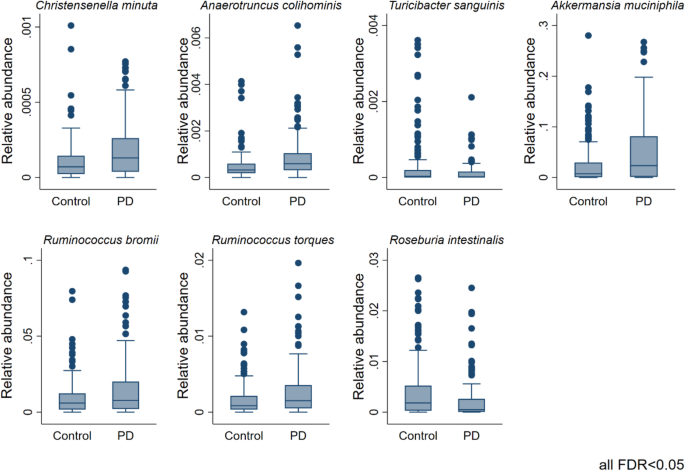
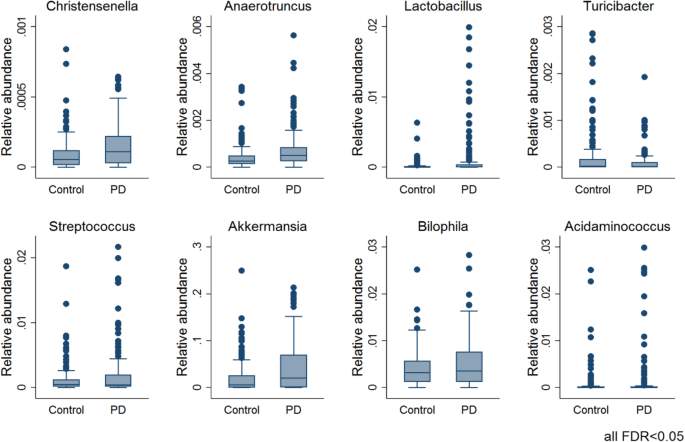
我们在物种水平上研究了与疾病相关的微生物变化。7个物种的PD显著改变(FDR < 0.05)。gydF4y2Ba2gydF4y2Ba),在308个具有完整协变量数据的个体的多变量分数回归中。注意,在比较不同分类水平的结果时,观察到的变化为gydF4y2Ba瘤胃球菌属gydF4y2Ba和gydF4y2BaRoseburiagydF4y2Ba物种在属水平上不显著,但在种水平上显著,表明物种水平分辨率的重要性。在物种水平上的最高效应值与gydF4y2BaAkkermansia muciniphilagydF4y2Ba(优势比(OR) = 1.80, 95% CI = (1.29, 2.51);gydF4y2BapgydF4y2Ba= 6.02e - 04, FDR < 0.05),与先前报道的较高丰度一致gydF4y2Ba答:muciniphilagydF4y2BaPD患者[gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba])。比值比为1.8意味着某个序列读取被分配的概率gydF4y2Ba答:muciniphilagydF4y2Ba估计PD病例比对照组高80%。这些比值比和后来的估计是从分数数据(例如相对丰度数据)中计算出来的。随后,我们在调整年龄、性别、体重指数(BMI)、批次和总阅读数的同时,通过执行半参数分数回归来检验属水平上可能存在的差异。我们发现有8个属的PD显著增加(FDR < 0.05;无花果。gydF4y2Ba3.gydF4y2Ba、表gydF4y2Ba2gydF4y2Ba),gydF4y2Ba乳酸菌gydF4y2Ba显示出最高的效应量(优势比(OR) = 5.75, 95% CI = (2.29, 14.45),gydF4y2BapgydF4y2Ba= 1.96e−04,FDR < 0.05,图gydF4y2Ba3.gydF4y2Ba).相比之下,属gydF4y2BaTuricibactergydF4y2BaPD组显著降低(FDR < 0.05)。我们重复了这些分析,对便秘进行了额外调整,以解释敏感性分析中潜在的混杂因素。所有属和种均保持显著性gydF4y2Ba瘤胃球菌属gydF4y2Ba物种。综上所述,在种和属水平上可以观察到显著的变化。gydF4y2Ba
PD改变了基本协变量对微生物组的影响gydF4y2Ba
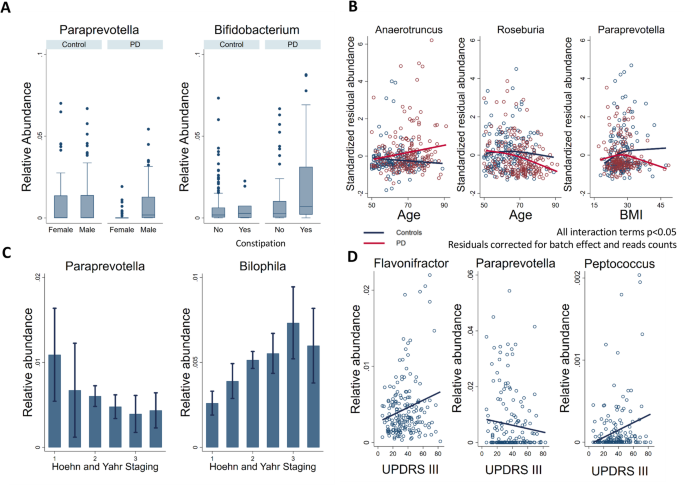
此外,我们基于308个具有完整协变量数据的个体的数据,使用多变量分数回归研究了PD的属级改变是否受到基本混杂因素的影响。这种相互作用分析揭示了丰富的效应修饰,揭示了PD中微生物组的变化应该在年龄、BMI和性别的背景下考虑。我们的分析表明,PD的影响在重要的亚组患者中并不均匀。例如,gydF4y2BaParaprevotellagydF4y2Ba在女性患者中,与男性受试者相比,完全减少,而在女性对照组中则没有减少(图2)。gydF4y2Ba4gydF4y2Baa),强调PD中微生物群落的性别依赖性变化。此外,PD病例中BMI和年龄的影响也有所改变。PD病例有所增加gydF4y2BaAnaerotruncusgydF4y2Ba丰度随年龄的增长呈非线性变化,总体丰度呈递减趋势gydF4y2BaRoseburiagydF4y2Ba和gydF4y2BaParaprevotellagydF4y2Ba分别随年龄和BMI进行观察(图2)。gydF4y2Ba4gydF4y2Bab).综上所述,这些分析表明PD病例中的微生物丰度发生了变化,PD中重要协变量的影响也发生了变化,反映了PD的系统性和复杂性。gydF4y2Ba
PD患者的微生物丰度、药物摄入和便秘gydF4y2Ba
卢森堡帕金森研究纳入了所有阶段的帕金森患者[gydF4y2Ba30.gydF4y2Ba]。因此,患者在pd相关特征上存在较大的个体间差异,如便秘和药物摄入(表1)gydF4y2Ba1gydF4y2Ba).我们通过对所有308名具有完整协变量数据的研究参与者的数据进行多变量分数回归分析了这些特征是否对PD中微生物组组成有影响。gydF4y2Ba1gydF4y2Ba表2)。值得注意的是,由于病例较少,我们无法调查COMT抑制剂的作用(gydF4y2BangydF4y2Ba= 6)。相比之下,便秘是PD患者常见的非运动症状[gydF4y2Ba37gydF4y2Ba],与增加的gydF4y2Ba双歧杆菌属gydF4y2Ba,在便秘的PD病例中效果明显(图2)。gydF4y2Ba4gydF4y2Baa)。然而,由于只有10个便秘对照组(表2)gydF4y2Ba2gydF4y2Ba),这些结果必须在更大的队列中得到证实。gydF4y2Ba
属与疾病严重程度有关gydF4y2Ba
接下来,我们研究了疾病的分期,即由Hoehn和Yahr分期、非运动症状量表(NMS-PD)和运动障碍学会统一帕金森病评定量表(MDS-UPDRS);进一步简称为UPDRS)评分及其亚量表,与属丰度的改变有关。由于缺少数据,纳入统计分析的病例数在这些变量之间变化gydF4y2BangydF4y2BaHoehn和Yahr分期= 146;gydF4y2BangydF4y2BaNMS-PD = 133;gydF4y2BangydF4y2Ba= 145的UPDRS量表gydF4y2BangydF4y2Ba= 138为疾病持续时间。对于霍恩和雅尔的舞台,gydF4y2BaParaprevotellagydF4y2Ba表现出负相关gydF4y2BaBilophilagydF4y2Ba呈正相关,经多次检验均具有显著性(图2)。gydF4y2Ba4gydF4y2Bac). UPDRS III子量表评分(即运动症状,见表gydF4y2Ba2gydF4y2Ba),三个属,是gydF4y2BaPeptococcusgydF4y2Ba,gydF4y2BaFlavonifractorgydF4y2Ba,gydF4y2BaParaprevotellagydF4y2Ba,经多次检验后存活校正(图2)。gydF4y2Ba4gydF4y2Bad).相比之下,经多次检验校正后,其他UPDRS子量表和NMS-PD与微生物变化无显著相关性。注意,这些分析是在调整疾病持续时间的情况下进行的。在分析疾病持续时间的关联模式时,我们发现gydF4y2Ba乳酸菌gydF4y2Ba与病程呈正相关(FDR < 0.05,附加文件)gydF4y2Ba1gydF4y2Ba:图S3)。总之,我们的数据表明微生物组成可能被用作疾病严重程度的相关因素。gydF4y2Ba
代谢模型揭示了PD微生物组独特的代谢分泌能力gydF4y2Ba
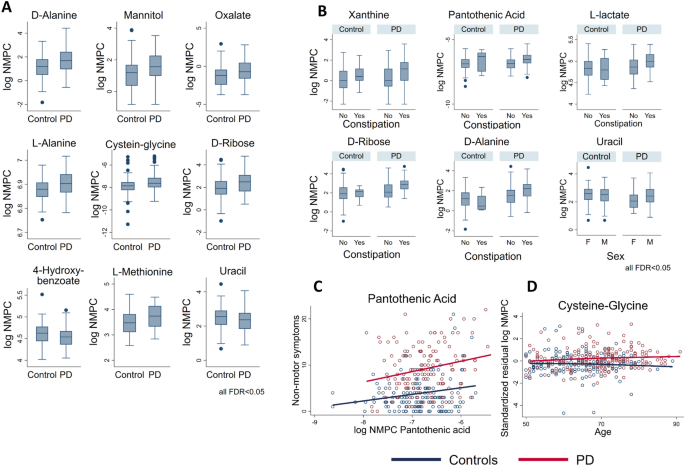
为了深入了解观察到的PD中微生物组变化可能带来的功能后果,我们使用了代谢模型(参见“gydF4y2Ba方法gydF4y2Ba节和附加文件gydF4y2Ba1gydF4y2Ba).简单地说,我们在由819个肠道微生物重建组成的通用微生物群落模型上绘制了308个微生物组样本的完整协变量数据[gydF4y2Ba23gydF4y2Ba,gydF4y2Ba25gydF4y2Ba]建立个人化的微生物群模型[gydF4y2Ba24gydF4y2Ba]。然后,我们计算了每种微生物群落模型可以分泌的129种不同代谢物的净最大生产能力(NMPC),或最大分泌通量势(cf)。”gydF4y2Ba方法gydF4y2Ba”部分),从而提供了pd和对照中不同微生物代谢能力的特征。对于一个个体,由于应用饮食限制导致不可行的社区模型,计算失败。通过多元混合效应线性回归对307个群落模型的计算建模结果进行统计分析。9种代谢物的预测NMPCs在PD中是不同的(图2)。gydF4y2Ba5gydF4y2Baa,所有FDR均< 0.05)。此外,尽管与丰度数据相比,PD-协变量相互作用不那么占主导地位,但PD-协变量相互作用也很普遍,预测尿嘧啶NMPC显示出性别特异性效应,半胱氨酸-甘氨酸显示出年龄依赖性PD效应(图2)。gydF4y2Ba5gydF4y2Bab, d)。在随后的分析中,我们测试了NMPCs与便秘、用药、病程、Hoehn-Yahr分期、NMS和UPDRS III评分的相关性,从而补充了丰度水平的分析。值得注意的是,我们发现了黄嘌呤的NMPCs,gydF4y2BadgydF4y2Ba丙氨酸,gydF4y2BalgydF4y2Ba乳酸酸,gydF4y2BadgydF4y2Ba-核糖和泛酸与便秘呈正相关(图2)。gydF4y2Ba5gydF4y2Bab),而NMPC与药物或疾病持续时间无关。然而,泛酸的NMPC与更高的NMS评分呈正相关,有趣的是,在PD和对照组中都是如此(图2)。gydF4y2Ba5gydF4y2Bac).没有NMPC在与UPDRS III评分和Hoehn-Yahr分期相关的多重测试中存活。总之,这些结果表明PD中微生物组成的改变可能导致代谢能力的广泛变化,这在非运动症状和便秘中也表现出来。gydF4y2Ba
在基本协变量和临床变量的背景下pd属的改变。gydF4y2Ba一个gydF4y2Ba的箱线图gydF4y2BaParaprevotellagydF4y2Ba和gydF4y2Ba双歧杆菌属gydF4y2Ba性依赖和便秘分别为病例和对照组。在这两种情况下,平均丰度的差异FDR < 0.05。gydF4y2BabgydF4y2Ba属丰度的病例和对照的散点图和非线性回归线gydF4y2BaAnaerotruncusgydF4y2Ba,gydF4y2BaRoseburiagydF4y2Ba,gydF4y2BaParaprevotellagydF4y2Ba与年龄和体重指数的相关性整体检验(Wald检验,同时在零上检验所有相互作用项)在所有三种情况下的FDR均< 0.05。对于相互作用项的图形化评估gydF4y2BazgydF4y2Ba-转换后的剩余丰度显示技术协变量(批和读取计数)校正后。gydF4y2BacgydF4y2Ba的误差柱图gydF4y2BaParaprevotellagydF4y2Ba和gydF4y2BaBilophilagydF4y2Ba疾病分期依赖性的丰度。属与疾病分期相关,呈相对丰度下降gydF4y2BaParaprevotellagydF4y2Ba增加了gydF4y2BaBilophilagydF4y2Ba随着Hoehn和Yahr尺度值的增加(FDR < 0.05)。误差条代表95%的置信区间。gydF4y2BadgydF4y2Ba运动症状散点图(UPDRS-part III)与…呈正相关gydF4y2BaFlavonifractorgydF4y2Ba和gydF4y2BaPeptococcusgydF4y2Ba丰度和负的gydF4y2BaParaprevotellagydF4y2Ba丰度(FDR < 0.05)。统一帕金森评定量表;BMI,身体质量指数;罗斯福,错误发现率gydF4y2Ba
由于群落结构和物种丰度的变化,pd特异性分泌谱发生了变化gydF4y2Ba
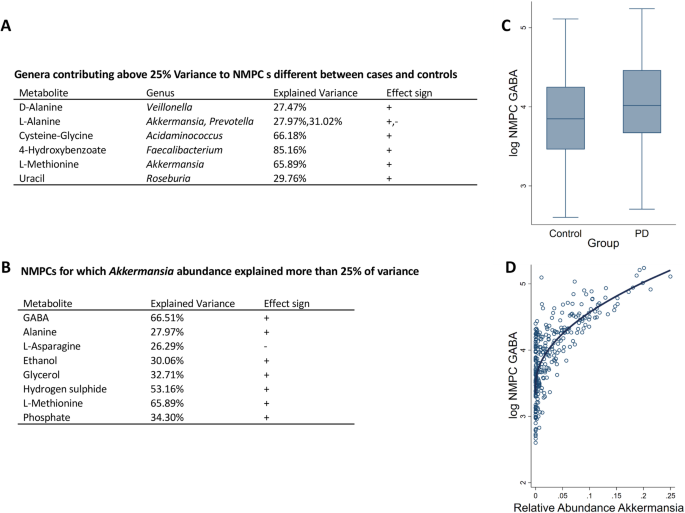
接下来,我们通过将微生物与丰度数据相关联,分析了哪些微生物导致了预测的NMPCs差异(图2)。gydF4y2Ba6gydF4y2Ba).采用完整协变量数据和可行的社区模型对307例病例进行线性回归分析。对于6个NMPCs,大部分观察到的方差可以用单个属来解释(图2)。gydF4y2Ba6gydF4y2Baa)其他4种NMPCs未发现单一优势属。此外,我们计算了每个属对每种分泌代谢物的预测NMPCs的解释方差。仅来自pd相关属gydF4y2BaAkkermansiagydF4y2Ba,gydF4y2Ba氨基酸球菌属gydF4y2Ba,gydF4y2BaRoseburiagydF4y2Ba解释了超过25%的NMPCs差异。gydF4y2Ba氨基酸球菌属gydF4y2Ba是造成64%的半胱氨酸-甘氨酸产量差异的原因gydF4y2BaRoseburiagydF4y2Ba尿嘧啶产生潜能变异的30%gydF4y2BaAkkermansiagydF4y2Ba通过实质上解释9种代谢物预测NMPCs的差异,对预测的NMPCs影响最大(图2)。gydF4y2Ba6gydF4y2Bab),包括神经递质γ -氨基丁酸(GABA),图。gydF4y2Ba6gydF4y2BaD)和两种硫,即硫化氢和蛋氨酸。在PD和对照组之间,GABA在名义水平上也发生了显著变化,略低于fdr校正的显著性(gydF4y2BabgydF4y2Ba= 0.18, 95% ci 0.06;0.30,gydF4y2BapgydF4y2Ba= 0.003, FDR = 0.0501,图gydF4y2Ba6gydF4y2Bac).请注意,微生物丰度与NMPC之间的关系并不一定是线性的,如GABA和的情况所示gydF4y2BaAkkermansiagydF4y2Ba(无花果。gydF4y2Ba6gydF4y2Bad).这些分析证明了代谢模型在研究整个微生物组成的代谢功能改变方面的附加价值。gydF4y2Ba
PD患者和健康个体微生物群落预测净代谢物生产潜力(NMPCs)的显著差异综述gydF4y2Ba一个gydF4y2Ba当FDR < 0.05时,病例与对照组NMPC差异的箱形图。gydF4y2BabgydF4y2Ba具有性别特异性PD特征或便秘效应的NMPCs的箱形图(所有FDR < 0.05)。gydF4y2BacgydF4y2Ba非运动症状评分和泛酸NMPC的散点图显示了病例和对照组的回归线。斜率显著大于零(FDR < 0.05)。gydF4y2BadgydF4y2Ba病例和对照组的半胱氨酸-甘氨酸NMPC与年龄的散点图显示相应的回归线。回归线斜率差异有统计学意义(FDR < 0.05)。罗斯福,错误发现率gydF4y2Ba
讨论gydF4y2Ba
在这项研究中,我们旨在阐明PD患者粪便微生物组的组成和功能变化。因此,我们分析了一组典型PD患者的16S rRNA基因测序数据(gydF4y2BangydF4y2Ba= 147)和对照组(gydF4y2BangydF4y2Ba= 162)并进行个性化微生物计算模型。我们发现(i) PD患者和健康对照者之间的相对丰度发生显著变化的8个属和7个种;(ii) pd相关微生物模式依赖于性别、年龄、BMI和便秘;(iii)在PD患者中,使用微生物群落代谢模型预测的代谢物分泌电位发生了变化,特别是硫代谢。总的来说,我们的工作证明了帕金森病患者肠道微生物群落的组成和预测功能差异,为帕金森病的发病机制提供了新的实验可验证的假设。gydF4y2Ba
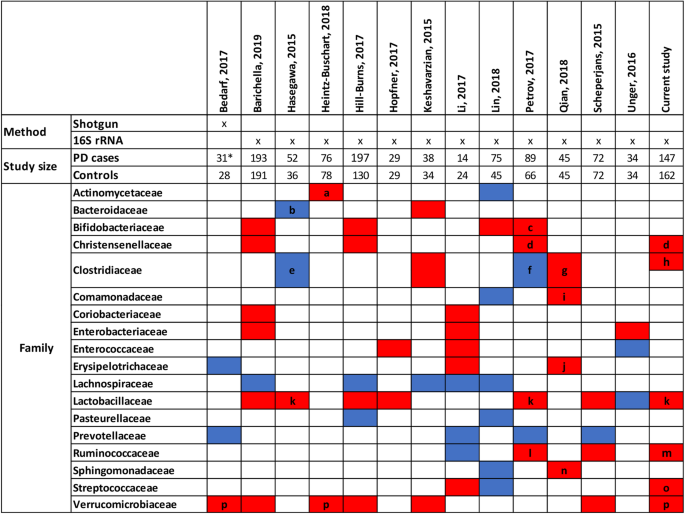
我们的队列微生物组成分析发现PD患者和健康对照之间的微生物丰度分布显著不同(表1)gydF4y2Ba2gydF4y2Ba).越来越多的研究描述了与PD相关的结肠微生物组成的改变,并开始出现一个整体的图景[gydF4y2Ba38gydF4y2Ba(图。gydF4y2Ba7gydF4y2Ba).例如,微生物家族gydF4y2BaVerrucomicrobiaceaegydF4y2Ba和gydF4y2Ba乳杆菌科gydF4y2Ba一直被发现在PD中有增加的丰度(图2)。gydF4y2Ba7gydF4y2Ba).因此,我们的研究也报告了PD的丰度增加gydF4y2BaAkkermansiagydF4y2Ba,gydF4y2BaChristensenellagydF4y2Ba,gydF4y2Ba乳酸菌gydF4y2Ba。同样的,gydF4y2Ba双歧杆菌gydF4y2Ba也多次与PD相关(图2)。gydF4y2Ba7gydF4y2Ba),但在我们的研究中,我们可以证明gydF4y2Ba双歧杆菌gydF4y2Ba这种关联依赖于便秘(图2)。gydF4y2Ba4gydF4y2BaA)强调将疾病特异性表型作为协变量纳入统计设计的必要性。gydF4y2Ba
同时,研究之间的不一致性仍然存在,这可能是由于研究设计、纳入标准、粪便采样、全基因组测序使用等方面的差异[gydF4y2Ba12gydF4y2Ba]、待定位的16S标记基因区域、DNA提取方案、统计方法等。例如,我们使用了一个相对较大的PD队列,而Bedarf和同事[gydF4y2Ba12gydF4y2Ba]研究了一个小的、定义明确的队列,其中包括未用药的男性PD患者和男性对照组(图2)。gydF4y2Ba7gydF4y2Ba).三项研究包括华人后裔[gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba],其中一项采用qPCR方法进行,其中包括来自日本的个体[gydF4y2Ba19gydF4y2Ba],而其他研究则集中在高加索人身上。研究表明,微生物组成与种族背景、地理位置和饮食习惯有关[gydF4y2Ba39gydF4y2Ba,gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba],这或许可以解释其中的一些差异。因此,这些研究之间的差异突出了进行荟萃分析以确定全球微生物特征的重要性,就像对结直肠癌等研究所做的那样[gydF4y2Ba42gydF4y2Ba]。这样的荟萃分析也允许调查PD的亚组,因为病例和对照的数量将大大增加,从而提供更高的统计能力。例如,我们观察到以前未在人类中报道的各种效应调节剂(表1)gydF4y2Ba2gydF4y2Ba),例如gydF4y2BaParaprevotellagydF4y2Ba丰度减少是女性特有的。这一结果显然与Bedarf及其同事的研究结果相矛盾[gydF4y2Ba12gydF4y2Ba他们报告的维生素d水平有所下降gydF4y2BaPrevotellaceaegydF4y2Ba在只有男性PD患者的队列中然而,由于属水平上的变化可以补偿在科分类水平上发现的变化(例如降低)gydF4y2BaParaprevotellagydF4y2Ba但更高的gydF4y2Ba普氏菌gydF4y2Ba),属水平上描述的结果与门水平上的结果不能直接比较。此外,如上所述,差异可能是由于性别特异性效应造成的,如图2所示。gydF4y2Ba4gydF4y2Baa).因此,一项研究报告了更高的丰度gydF4y2BaParaprevotellagydF4y2Ba雄性小鼠与雌性小鼠相比gydF4y2Ba43gydF4y2Ba]。尽管缺乏对微生物组组成的性别差异的广泛研究,但我们建议微生物组数据的机器学习程序应该以性别分层的方式进行。更大的队列,例如通过对已发表队列的荟萃分析,将允许识别PD患者中普遍的微生物差异,以及与某些特征和生理特征相关的特定微生物变化,正如我们的数据所表明的那样。gydF4y2Ba
经过多次测试校正后,我们没有发现任何证据表明多巴胺能、pd特异性药物对微生物组组成有影响。值得注意的是,在我们的研究设计中,我们不能排除多巴胺能药物对微生物组的影响。为了检测多巴胺能药物的小效应,需要比我们的队列更大的样本量。同样值得注意的是,PD药物通常与其他药物联合使用,同样需要比我们研究中使用的更大的样本量,以允许调查所有可能的药物组合。例如,由于缺少样本量,本研究无法分析COMT抑制剂的潜在作用。尽管如此,在之前的研究中,gydF4y2BaDoreagydF4y2Ba和gydF4y2BaPhascolarctobacteriumgydF4y2Ba与左旋多巴当量剂量呈负相关[gydF4y2Ba36gydF4y2Ba],以及…的家庭成员gydF4y2Ba芽胞杆菌科gydF4y2Ba与左旋多巴治疗相关[gydF4y2Ba13gydF4y2Ba]。也可以认为左旋多巴是在小肠上部吸收的[gydF4y2Ba44gydF4y2Ba],因此,小肠微生物而不是大肠微生物可能在左旋多巴的生物利用度中发挥更重要的作用。最近的一项研究一致表明,细菌酪氨酸脱羧酶限制了左旋多巴的生物利用度[gydF4y2Ba45gydF4y2Ba]。有趣的是,818个肠道微生物中有193个(24%)具有基因组规模的代谢重建[gydF4y2Ba23gydF4y2Ba,gydF4y2Ba46gydF4y2Ba携带必需的基因,编码将左旋多巴转化为多巴胺的蛋白质。左旋多巴总是与脱羧酶抑制剂一起给药,如卡比多巴或苯塞拉肼,靶向人类脱羧酶,但不能排除它们也作用于微生物的对应物。然而,van Kessel等人的研究表明,卡比多巴和苯塞拉肼只是微生物酪氨酸脱羧酶的弱抑制剂[gydF4y2Ba45gydF4y2Ba]。gydF4y2Ba
我们发现了一个正相关的gydF4y2BaBilophilagydF4y2Ba与Hoehn和Yahr分期一致,该分期捕捉运动损伤和残疾,与疾病持续时间无关。的确,丰饶gydF4y2BaBilophilagydF4y2Ba与病程无关,表明主要依赖于症状的进展。这一发现与实验小鼠研究表明的促炎作用相一致gydF4y2BaBilophilagydF4y2Ba增生(gydF4y2Ba47gydF4y2Ba,gydF4y2Ba48gydF4y2Ba]。gydF4y2BaBilophilagydF4y2Ba在AGORA覆盖的微生物中具有独特的能力[gydF4y2Ba23gydF4y2Ba]使用牛磺酸,一种具有神经保护作用的抑制性神经递质[gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba],作为能源[gydF4y2Ba51gydF4y2Ba]。该途径涉及丙酮酸转氨酶[gydF4y2Ba51gydF4y2Ba],将丙酮酸和牛磺酸转化为gydF4y2BalgydF4y2Ba-丙氨酸和磺乙醛。作为gydF4y2BaBilophilagydF4y2Bap < 0.05, p < 0.05gydF4y2BaBilophilagydF4y2Ba代表了唯一能够利用牛磺酸生成ATP的属,因此PD微生物组丰富了这种特定的代谢功能(从牛磺酸生成ATP)。在之前的一项研究中[gydF4y2Ba29gydF4y2Ba],我们已经证明血牛磺酸结合胆汁酸与运动症状呈正相关。我们已经提出gydF4y2BaBilophilagydF4y2Ba可能是PD疾病进展的标志,并且可以通过其牛磺酸降解能力调节人体硫代谢[gydF4y2Ba29gydF4y2Ba]。因此,我们报道了使用微生物组计算模型时硫代谢的变化[gydF4y2Ba29gydF4y2Ba]来自一组早期诊断和未使用左旋多巴的PD患者[gydF4y2Ba12gydF4y2Ba]以及血液样本中蛋氨酸及其衍生代谢物浓度的增加[gydF4y2Ba29gydF4y2Ba]。此外,我们和其他人报道了PD患者胆汁酸和牛磺酸共轭胆汁酸的变化[gydF4y2Ba29gydF4y2Ba,gydF4y2Ba52gydF4y2Ba]。根据,我们目前的研究发现gydF4y2BaBilophilagydF4y2Ba与疾病严重程度有关,加强了两者之间的联系gydF4y2BaBilophilagydF4y2Ba牛磺酸和帕金森氏症gydF4y2Ba
有趣的是,越来越多的gydF4y2BaBilophila wadsworthiagydF4y2Ba与便秘有关[gydF4y2Ba53gydF4y2Ba],另一项针对慢性便秘患者的研究报告显示,这种情况有所减少gydF4y2Ba双歧杆菌gydF4y2Ba丰度(gydF4y2Ba54gydF4y2Ba]。我们发现增加了gydF4y2Ba双歧杆菌gydF4y2Ba便秘个体的丰度,特别是便秘的PD患者;然而,在我们的研究中,便秘的对照组人数非常少(gydF4y2BangydF4y2Ba= 10)。相比之下,我们没有发现丰度之间的关联有统计学上的显著变化gydF4y2Bab . wadsworthiagydF4y2Ba个体便秘PD患者(图2)gydF4y2Ba4gydF4y2Bac).总体而言,现有数据表明,微生物组成的复杂变化与便秘有关,但在不同疾病之间可能有所不同。因此,是否gydF4y2Bab . wadsworthiagydF4y2Ba在PD患者便秘中的作用有待进一步研究。gydF4y2Ba
黏液降解微生物,gydF4y2Ba答:muciniphilagydF4y2Ba,约占人体粪便微生物群的1-4% [gydF4y2Ba55gydF4y2Ba]。许多疾病都与维生素d的减少有关gydF4y2Ba答:muciniphilagydF4y2Ba丰度(gydF4y2Ba56gydF4y2Ba,gydF4y2Ba57gydF4y2Ba],而PD患者中一直有增加的报道(图2)。gydF4y2Ba7gydF4y2Ba).的gydF4y2Ba答:muciniphilagydF4y2Ba丰度对代谢物分泌谱的显著改变贡献最大(图2)。gydF4y2Ba6gydF4y2Bab),包括神经递质γ -氨基丁酸(GABA)。虽然在本研究中,其预测的分泌潜力在PD患者中只是名义上的增加,但基于早期左旋多巴未发作PD患者的微生物组数据,也预测了更高的GABA分泌率[gydF4y2Ba29gydF4y2Ba]。重要的是,在肠神经系统、肠肌肉、肠上皮层和内分泌样细胞中都发现了GABA受体[gydF4y2Ba58gydF4y2Ba],其肠道受体被认为与胃运动(蠕动)、胃排空和胃酸分泌有关[gydF4y2Ba58gydF4y2Ba]。GABA的实验gydF4y2BabgydF4y2Ba受体激动剂巴氯芬显示GABAgydF4y2BabgydF4y2Ba受体通过胆碱能调节降低家兔结肠胃蠕动[j]。gydF4y2Ba59gydF4y2Ba]。GABA可以作为一种亲脂化合物通过血液到达中枢神经系统,能够通过血脑屏障。此外,微生物GABA可以通过贡献人类GABA池来影响脑肠轴,特别是有研究表明微生物组可以通过迷走神经影响中枢神经系统中GABA受体的密度[gydF4y2Ba60gydF4y2Ba]。有趣的是,gydF4y2Ba答:muciniphilagydF4y2Ba已被证明与胃肠道转运时间呈正相关[gydF4y2Ba61gydF4y2Ba,gydF4y2Ba62gydF4y2Ba],所以人们可以假设这种影响可能是由于它的GABA生产能力。确定是否和哪个角色gydF4y2Ba答:muciniphilagydF4y2BaGABA可能在PD前驱中发挥作用,需要进一步的实验研究。gydF4y2Ba
物种对NMPCs的贡献分析综述。gydF4y2Ba一个gydF4y2Ba解释病例和对照组之间代谢物NMPCs差异超过25%的属表。gydF4y2BabgydF4y2Banmpc表,其中gydF4y2BaAkkermansiagydF4y2Ba解释总生产能力与社区净生产能力之间至少25%的差异。gydF4y2BacgydF4y2BaPD病例和对照组γ -氨基丁酸(GABA)净生产能力的箱形图(gydF4y2BabgydF4y2Ba= 0.18, 95% ci 0.06, 0.30,gydF4y2BapgydF4y2Ba= 0.003, fdr = 0.0501)。gydF4y2BadgydF4y2BaGABA净产能与Akkermansia丰度关系的非线性回归散点图。净最大生产能力;GABA, gamma-aminobutyrate;罗斯福,错误发现率。效应符号“-”:负相关。效应符号“+”:表示正相关gydF4y2Ba
为了超越仅仅对与疾病相关的微生物变化进行编目,基于途径的工具(例如:gydF4y2Ba63gydF4y2Ba])已经开发出来,其中微生物序列(或读取)被映射,例如到KEGG数据库中存在的京都基因和基因组百科全书(KEGG)本体[gydF4y2Ba64gydF4y2Ba]。使用这些工具,Bedarf等人报道了葡萄糖酸盐降解减少,色氨酸降解和甲酸转化增加[gydF4y2Ba12gydF4y2Ba]。类似地,Heinz-Buschart等人报道了PD微生物组中26条KEGG通路的改变[gydF4y2Ba13gydF4y2Ba]。在我们的研究中,我们用计算模型补充了成分分析,以深入了解PD中微生物丰度变化的潜在功能,即代谢后果。我们的方法的优势在于,功能分配可能比更规范的方法(如KEGG本体论)更全面,因为(1)潜在的基因组规模的代谢重建是基于精细的基因组注释组装的,并且已经被人工整理,以确保反应和基因内容与当前关于微生物生理学的知识一致[gydF4y2Ba65gydF4y2Ba],并且(2)这些重建中的每一个,单独或组合,都适合代谢模型,因此,变化的环境(例如模型中的营养物质或其他微生物)的功能和代谢后果可以计算出来[gydF4y2Ba22gydF4y2Ba]。因此,这些模拟可以预测功能结果,而不仅仅是途径或反应富集,就像通常所做的那样。gydF4y2Ba
优势与局限gydF4y2Ba
在这里,我们介绍了一项大型单中心PD纵向研究(包括病例和对照)的微生物组分析,该研究在卢森堡和邻近的边境地区(大区)进行了全国范围的推广,其中包括所有疾病阶段的临床谱[gydF4y2Ba30.gydF4y2Ba]。我们证明,微生物组成不仅在PD中发生改变,而且观察到PD与微生物组成变化的关联应该在年龄、性别、BMI和便秘的背景下进行解释。这一信息对于临床翻译非常重要,强调需要(i)个性化和(ii)整体方法,以了解微生物群落在PD发病机制中的作用。在针对pd相关微生物组的潜在功能变化的第二步中,我们基于AGORA收集进行了代谢建模[gydF4y2Ba23gydF4y2Ba]的基因组尺度代谢重建,允许代谢物分泌谱的预测。因此,我们的分析促进了肠道中pd相关微生物群落代谢改变的详细调查,指出了已知促炎物种的作用gydF4y2Bab . wadsworthiagydF4y2Ba与寄主的硫代谢相互作用。因此,代谢模型为破译PD中微生物群落的代谢活性提供了一个有价值的工具。gydF4y2Ba
然而,尽管我们的研究部分证实了之前的结果(图2)。gydF4y2Ba7gydF4y2Ba),但应注意几个限制。首先,某些协变量没有被调查,比如饮食、运动和吸烟。这些协变量是否会改变pd特异性特征还有待分析。虽然我们的研究属于对PD进行的三个最大的研究,但我们的样本量仍然太小,无法提供对药物组合的见解,因为由于样本量减少,检测药物效果的统计能力低于检测病例和对照组之间差异的统计能力。此外,在我们的研究中使用的16S rRNA基因测序不允许对菌株水平进行分析,因为它可能导致错误分类[gydF4y2Ba66gydF4y2Ba]。此外,SPINGO分类器没有排除排序错误的策略,但作者已经表明,这一缺点对SPINGO的准确性影响很小[gydF4y2Ba67gydF4y2Ba]。使用16S rRNA基因测序的另一个限制在于缺少菌株水平的分辨率,迫使我们在物种水平上对菌株的代谢能力进行分组。由于同一物种的不同菌株可能具有不同的代谢能力,因此必须谨慎对待基于物种水平的计算模型。因此,需要基于霰弹枪测序的后续研究进一步证实我们的结果和其他16S RNA基因测序研究的发现(图2)。gydF4y2Ba7gydF4y2Ba).在这方面,值得注意的是,AGORA对主要在微生物组中检测到的物种具有很高的覆盖率(附加文件)gydF4y2Ba1gydF4y2Ba:表1)。然而,AGORA并没有显示完全的覆盖,这对本研究有一定的局限性。因此,微生物组中存在的某些物种和属可能被排除在分析之外,因为它们没有包括在AGORA收集中。因此,需要进一步扩大微生物代谢重建收集。此外,我们只能从测序样本中提取物种(而不是菌株)丰度,这对代谢模型有重要影响。由于缺乏菌株解决的分类信息,我们需要生成物种水平的代谢模型,其中我们将多个菌株的代谢能力分组到相应物种的一个代谢模型中。这种方法可能高估了代谢能力,因为物种代谢模型中包含的所有生化反应可能存在于单个菌株中。gydF4y2Ba
不同研究中与PD相关的家族水平的微生物变化。只有那些细菌科被显示出来,至少有两项比较患者和对照组粪便样本的研究报告了与物种或属的显著关联。PD呈红色增高;PD患者蓝色减少。答:gydF4y2Ba放线菌目gydF4y2Ba;b:gydF4y2Ba脆弱拟杆菌gydF4y2Ba;c:gydF4y2Ba双歧杆菌属gydF4y2Ba;d:gydF4y2BaChristensenellagydF4y2Ba;艾凡:gydF4y2Ba球菌样的梭状芽胞杆菌gydF4y2Ba/gydF4y2BaleptumgydF4y2Ba;f:gydF4y2BaFaecalibacteriumgydF4y2Ba和gydF4y2BaDoreagydF4y2Ba;旅客:gydF4y2Ba梭状芽胞杆菌gydF4y2BaIV /十八,gydF4y2BaButyricicoccusgydF4y2Ba和gydF4y2BaAnaerotruncusgydF4y2Ba;h:gydF4y2BaAnaerotruncusgydF4y2Ba;我:gydF4y2BaAquabacteriumgydF4y2Ba;珍:gydF4y2BaHoldemaniagydF4y2Ba;凯西:gydF4y2Ba乳酸菌gydF4y2Ba;李:gydF4y2BaOscillospiragydF4y2Ba;m:gydF4y2Ba瘤胃球菌属romiigydF4y2Ba和gydF4y2Ba瘤胃球菌属扭矩gydF4y2Ba;护士:gydF4y2BaSphingomonasgydF4y2Ba;o:gydF4y2Ba链球菌gydF4y2Ba;病人:gydF4y2BaAkkermansiagydF4y2Ba。*仅限新发PD患者,基于[gydF4y2Ba14gydF4y2Ba]gydF4y2Ba
因为本质上是横断面的,所以因果推理是不可能的。因此,尽管代谢模型已多次被证明能够正确地预测生命系统的属性[gydF4y2Ba68gydF4y2Ba,gydF4y2Ba69gydF4y2Ba,gydF4y2Ba70gydF4y2Ba,我们对……的作用的假设gydF4y2Bab . wadsworthiagydF4y2BaPD中硫代谢与疾病严重程度之间的联系需要实验验证。此外,分泌谱的计算预测需要通过与粪便和血液代谢组数据的整合来验证。此外,到目前为止,基于约束的代谢模型群体的统计特性尚未得到详细描述。基本上,本研究使用预测的净生产能力作为组学数据的进一步层,在此基础上,通过顺序回归进行的统计筛选可以类似于“全基因组关联研究”。此外,目前的研究还没有考虑到宿主的代谢,但这种分析在计算上是可能的(例如[gydF4y2Ba27gydF4y2Ba,gydF4y2Ba71gydF4y2Ba,gydF4y2Ba72gydF4y2Ba,gydF4y2Ba73gydF4y2Ba,gydF4y2Ba74gydF4y2Ba]。为此,性别特异性全身代谢模型[gydF4y2Ba26gydF4y2Ba]已经开发出来,其中包括人类生理限制,并且可以通过微生物组模型扩展以研究潜在的宿主-微生物组代谢相互作用,但此类分析超出了本研究的范围。gydF4y2Ba
结论gydF4y2Ba
总的来说,这项研究代表了向微生物组中pd相关改变的代谢后果的系统生物学描述迈出的一步,但需要进一步开发将组学数据与建模技术(如基于约束的建模)相结合的统计和计算工具。gydF4y2Ba
方法gydF4y2Ba
卢森堡帕金森研究的描述gydF4y2Ba
数据和生物标本来自卢森堡帕金森研究队列[gydF4y2Ba30.gydF4y2Ba]。卢森堡帕金森氏症研究包括一组患有典型帕金森氏症、非典型帕金森氏症和继发性帕金森氏症的患者,以及来自卢森堡及其邻近地区(地理上被定义为大区)的健康对照组[gydF4y2Ba30.gydF4y2Ba]。在队列中,从选定患者的配偶、志愿者和来自卢森堡其他独立研究的个人中选择健康对照[gydF4y2Ba75gydF4y2Ba,gydF4y2Ba76gydF4y2Ba]。但是,没有关于对照和病例之间家庭关系的相应资料。癌症诊断并持续治疗、妊娠和中度脑积水框架下的继发性帕金森病是入选患者或健康对照组的排除标准。454人(对照):gydF4y2BangydF4y2Ba= 248, pd:gydF4y2BangydF4y2Ba= 206),粪便样本可用于16S RNA基因测序数据(见下文)。由于我们的目标是典型的PD,我们排除了所有年龄低于50岁的个体(对照组:gydF4y2BangydF4y2Ba= 47, pd:gydF4y2BangydF4y2Ba= 9)以及所有PD诊断状态不明确或PD诊断不典型的个体(PD:gydF4y2BangydF4y2Ba= 47)。根据英国帕金森病学会脑库临床诊断标准将PD患者定义为典型PD [gydF4y2Ba77gydF4y2Ba]。此外,我们排除了UPDRS III评分高于10分的对照患者,除了一名由手臂损伤引起的高UPDRS III评分的对照患者。此外,我们还排除了服用多巴胺能药物的对照组(gydF4y2BangydF4y2Ba= 5;服用不同于抗pd指征的抗生素)和报告在过去6个月内服用抗生素的个体(对照组:gydF4y2BangydF4y2Ba= 20, pd:gydF4y2BangydF4y2Ba= 13)。请注意,由于排除标准之间的重叠(即年龄在50岁以下且服用抗生素的个体),排除的观察结果表现为次相加性。最后,从309个人(对照)中:gydF4y2BangydF4y2Ba= 162例:gydF4y2BangydF4y2Ba= 147例)符合纳入标准,308例因BMI值缺失1例纳入统计分析。gydF4y2Ba
所有研究参与者均给予书面知情同意,研究按照赫尔辛基宣言进行。卢森堡帕金森研究[gydF4y2Ba30.gydF4y2Ba]已获得国家伦理委员会(CNER Ref: 201407/13)和数据保护委员会(CNPD Ref: 446/2017)的批准。gydF4y2Ba
测量和神经精神测试gydF4y2Ba
所有患者和健康对照在临床综合评估期间由神经科医生、神经心理学家或训练有素的研究护士进行评估。嗅觉测试使用嗅探棒(Sniffin’Sticks)的16项版本(SS)进行,产生“嗅得分”。抗生素使用定义为收集粪便前6个月内的抗生素摄入量。便秘是由研究神经学家根据每个人的个人临床故事来评估的。对于便秘的定义,考虑了两个标准:(i)大便困难包括紧张、排便困难感、排便不完全、大便坚硬/块状、排便时间延长或需要手动排便[gydF4y2Ba78gydF4y2Ba],以及(ii)基于症状的,包括每周少于三次大便,大便形式多为坚硬或块状,大便通过困难(需要拉稀或不完全排便)超过6个月[gydF4y2Ba79gydF4y2Ba]。为了评估pd相关的运动和非运动症状,使用UPDRS评定量表I-IV [gydF4y2Ba80gydF4y2Ba]。疾病的严重程度可以通过Hoehn和Yahr分期来反映[gydF4y2Ba81gydF4y2Ba]。非运动症状采用相应的问卷NMS-PD进行测量[gydF4y2Ba82gydF4y2Ba]。gydF4y2Ba
收集和处理粪便样本gydF4y2Ba
所有样本均按标准操作程序处理[gydF4y2Ba83gydF4y2Ba,gydF4y2Ba84gydF4y2Ba]:患者使用OMNIgene在家中收集粪便样本。GUT® kit (DNA Genotek) and sent to the Integrated Biobank Luxembourg (IBBL) where one aliquot of 1 ml was used for DNA extraction. The mean delay from sample collection to sample reception at IBBL was on average of 3.8 days. The microbiome profiles were obtained from stabilised samples within the OMNIgene®-GUT kit, which has been shown to be comparable for downstream 16S rRNA gene sequencing, robustness, and sample stability to the snap-frozen samples [84gydF4y2Ba,gydF4y2Ba85gydF4y2Ba]。将经OMNIgene®- gut稳定的样品在室温下放置2周不会对微生物组谱产生任何显著影响[gydF4y2Ba86gydF4y2Ba,gydF4y2Ba87gydF4y2Ba]。对于DNA提取,使用改良的Chemagic DNA血液方案,使用MSM I仪器(PerkinElmer), Chemagic血液试剂盒专用4ml(参考编号:CMG-1074),带有粪便样品的裂解缓冲液,以及MSM I软件。使用SEB裂解缓冲液(包含在试剂盒中)裂解样品,并旋转以获得均匀的悬浮液,在70°C下孵育10分钟,然后在95°C下孵育5分钟。裂解物(1.5 ml)在10000℃下离心5分钟gydF4y2BaggydF4y2Ba上清液转移至24XL深孔板。用MSM I自动程序处理板材。gydF4y2Ba
16S rRNA基因测序分析微生物组成gydF4y2Ba
使用基因特异性引物靶向16S rRNA基因的V3-V4区域,并使用Illumina平台(Illumina MiSeq)在IBBL上使用2 × 300 bp的对端reads进行测序[gydF4y2Ba30.gydF4y2Ba]。引物采用Illumina悬挑接头设计,用于扩增基因组DNA模板。扩增子根据illumina演示的16S宏基因组测序文库制备方案进行生成、清洗、索引和测序,并进行一定的修改。简而言之,最初的PCR反应至少包含12.5 ng的DNA。随后进行限环扩增步骤,添加多路复用索引和Illumina测序适配器。文库归一化,汇集,并在Illumina MiSeq系统上使用2 × 300 bp对端reads进行测序。双索引条形码采用Illumina Nextera XT索引引物。gydF4y2Ba
解复用后的样本经合并正读和反读处理,并使用专用管道“合并和滤波工具(MeFit)”进行质量滤波[gydF4y2Ba88gydF4y2Ba使用默认参数。为了获得可靠的微生物鉴定,使用SPINGO (species level identification of metaGenOmic amplicons)分类器进行属和种两个分类水平的鉴定[gydF4y2Ba67gydF4y2Ba使用默认参数。SPINGO分类器被设计用于物种分类分配[gydF4y2Ba67gydF4y2Ba]。计算每个样本的相对丰度,使用R (R Foundation for Statistical Computing, Vienna, Austria)解析SPINGO分类器的分类结果[gydF4y2Ba89gydF4y2Ba自定义脚本。简单地说,对于每个样本,检索每个属/种的计数,然后将所有属/种的计数之和归一化为每个属/种计数的总数为1。有关读取计数的信息可以在附加文件中找到gydF4y2Ba1gydF4y2Ba:表S3。gydF4y2Ba
基因组尺度的代谢重建、通量平衡分析(FBA)和社区代谢模型gydF4y2Ba
代谢重建包括已知在生物体中发生的所有代谢反应的列表,并描述每个反应的化学计量和方向性[gydF4y2Ba22gydF4y2Ba,gydF4y2Ba65gydF4y2Ba]。这种重建通常来自生物体的基因组、相应的基因组注释以及对生物体特异性、生化和生理学文献的广泛回顾[gydF4y2Ba65gydF4y2Ba]。这些代谢重建可以可视化为网络(每个反应都是连接相关代谢物的拱门),并且可以以计算格式转换。在转换为计算格式时,称为化学计量矩阵(gydF4y2Ba年代gydF4y2Ba)由每个反应的化学计量系数生成。在S矩阵中,每一行代表网络中不同的代谢物,每一列代表不同的反应[gydF4y2Ba22gydF4y2Ba]。根据定义,反应的每个底物得到一个负号,而产物得到一个正号。如果代谢物不参与反应,则化学计量系数,因此gydF4y2Ba年代gydF4y2Ba矩阵的入口是0。每种代谢物随时间变化(即gydF4y2BadxgydF4y2Ba/gydF4y2BadtgydF4y2Ba)将因此由的乘法得到gydF4y2Ba年代gydF4y2Ba对于包含每个反应发生的速度(通量)的矢量V,一个线性方程组可以写成[gydF4y2Ba22gydF4y2Ba]。gydF4y2Ba
通量平衡分析[gydF4y2Ba22gydF4y2Ba是一种用于研究代谢重建特性的方法。FBA基于三个假设:(i)稳态:在此假设下,没有代谢物可以积累,每种代谢物的浓度随时间变化为零,因此;gydF4y2Ba年代gydF4y2Ba×gydF4y2BavgydF4y2Ba=gydF4y2BadxgydF4y2Ba/gydF4y2BadtgydF4y2Ba= 0)。(二)能力限制:存在用上界和下界表示的反应所允许的最小通量和最大通量。这些约束可以从实验数据(例如:gydF4y2BavgydF4y2Ba马克斯gydF4y2Ba酶(膳食摄食量)。(iii)目标函数:通过一个反应的通量,最常见的是生物量(生长)反应[gydF4y2Ba90gydF4y2Ba],是最优的。这些假设允许检索一个可能的,但不一定是唯一的,通过网络的通量分布(编码在通量向量中)gydF4y2BavgydF4y2Ba),与所有应用的约束一致。相反,目标函数的目标值是唯一的[gydF4y2Ba22gydF4y2Ba]。gydF4y2Ba
代谢重建可以提供在特定条件下单个生物体代谢的机制。然而,微生物群落是复杂的系统,其最终结果是由不同微生物的相互作用给出的[gydF4y2Ba91gydF4y2Ba]。因此,开发了一种称为区隔化的多物种微生物群落代谢建模方法[gydF4y2Ba24gydF4y2Ba,gydF4y2Ba73gydF4y2Ba]。在区隔化方法中,不同的重建通过一个共同的区隔连接起来,允许它们进行代谢相互作用(共享代谢物)。微生物组建模工具箱[gydF4y2Ba24gydF4y2Ba,gydF4y2Ba92gydF4y2Ba]允许创建个性化的微生物群模型,其中数百种微生物重建是根据它们在相对样品中的存在/不存在而连接起来的。对于每个微生物群模型,相对丰度被整合到一个群落目标函数中,加入了每个生物对群落的生物量贡献。这种方法是专门为成分数据开发的,因为所有系数的总和必须等于1。然后,计算每个群落分泌的代谢物,然后这些代谢物可以被人类宿主吸收或以其他方式随粪便排出。gydF4y2Ba
在肠道微生物重建收集上绘制检测到的物种gydF4y2Ba
目前,已经发表了819种肠道微生物的菌株特异性代谢重建,命名为AGORA集合[gydF4y2Ba23gydF4y2Ba,gydF4y2Ba25gydF4y2Ba,gydF4y2Ba46gydF4y2Ba]相当于646种。在当前研究的分析数据集中(gydF4y2BangydF4y2Ba= 309),其中515种至少在5%的粪便样本中检出,243种与AGORA重叠。在至少50%的样本中共检测到125种,其中有87种AGORA物种重叠。因此,70%的鉴定物种被AGORA选择所覆盖(附加文件)gydF4y2Ba1gydF4y2Ba:表1)。我们的结论是,我们的AGORA收集涵盖了我们数据集中最常见的物种。gydF4y2Ba
生成个性化模型gydF4y2Ba
下一步,我们生成了一个由257个微生物代谢重建组成的通用微生物组代谢重建,这些微生物代谢重建在我们的数据集中具有相对丰富度,高于1e−4,并且存在于AGORA集合中。然后,通过消除样本中低于该阈值(为1e−4)的所有物种,并根据SPINGO获得的归一化相对丰度数据调整群落生物量反应系数,对每个样本进行个性化的通用微生物组重建[gydF4y2Ba67gydF4y2Ba]。在缺乏个人营养信息的情况下,使用平均欧洲饮食来约束每个微生物组模型[gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba[附加文件gydF4y2Ba1gydF4y2Ba:表S4)。平均而言,个性化微生物组模型包含67种,77,390(非独特)反应和69,265(非独特)代谢物(附加文件)gydF4y2Ba1gydF4y2Ba表2)。此外,个性化微生物组模型平均涵盖2727种独特的反应(附加文件)gydF4y2Ba1gydF4y2Ba表2)。与对照组相比,PD组的独特反应数、总反应数和总代谢物数略高。gydF4y2Ba
多样性指数分析gydF4y2Ba
所有的数值生态学分析都用R计算属和种的分类分辨率。丰富度计算为检测到的生物体总数,而α多样性是使用Shannon指数计算的,该指数在2.5-2 R纯素包装的“多样性”函数中实现[gydF4y2Ba93gydF4y2Ba]。用2.5-2 R纯素包装的“多样性”函数计算Pielou均匀度[gydF4y2Ba93gydF4y2Ba使用辛普森指数。多样性是使用2.5-2 R纯素包装的“素食主义者”函数计算的[gydF4y2Ba93gydF4y2Ba使用布雷-柯蒂斯不相似指数。使用线性混合模型评估PD患者与对照组在丰富度、Shannon多样性和Pielou指数方面可能存在的差异,其中批次变量为随机效应变量,包括年龄、BMI、性别和阅读计数变量为协变量,而组变量为感兴趣的预测因子。在探索性事后分析中,我们进一步测试了是否有任何基本混杂因素(年龄、性别和BMI)与影响各种多样性指数的群体变量相互作用。对于beta多样性,我们进行了ANOSIM和ADONIS分析,在2.5-2 R纯素包中实施[gydF4y2Ba93gydF4y2Ba使用函数“Adonis”和“Anosim”使用默认参数。gydF4y2Ba
通过分数回归分析相对丰度gydF4y2Ba
对于描述性统计,度量变量用均值和标准差来描述,而名义变量用比例来描述。缺失值未被估算,缺失值的模式无法通过ADA平台评估[gydF4y2Ba30.gydF4y2Ba]。每个宏基因组特征(如属和种)的读取计数除以总读取计数,以便检索相对丰度。对相对丰度进行异常值检查。与平均值有超过4个标准差的观测结果被排除在分析之外。只有在所有样本中检测到50%以上的属和种被纳入分析,结果是62属和127种。gydF4y2Ba
宏基因组数据使用分数回归进行分析,该回归由[gydF4y2Ba94gydF4y2Ba]。分数回归是在计量经济学领域发展起来的,是广义线性模型家族的一部分,专门用于分析分数数据,如相对丰度数据。相对丰度是这里的响应变量,然后在预测向量上进行回归。分数回归是一种半参数方法,设计用于对分数数据进行建模,而无需指定响应变量的分布。此外,分数回归对异方差和过度分散具有固有的鲁棒性。这些特点使得该方法非常适合分析微生物组数据,因为不同的物种可能不会从同一类分布中取样。一般来说,准对数似然lngydF4y2BalgydF4y2Ba由伯努利对数似然函数给出:gydF4y2Ba
与gydF4y2BaGgydF4y2Ba(gydF4y2Ba∗gydF4y2Ba)为满足0≤的函数gydF4y2BaGgydF4y2Ba(gydF4y2BazgydF4y2Ba)≤1gydF4y2BazgydF4y2Ba∈gydF4y2BaℝgydF4y2Ba,在那里gydF4y2BaxgydF4y2BajgydF4y2Ba表示个体的预测因子gydF4y2BajgydF4y2Ba,gydF4y2BaygydF4y2BajgydF4y2Ba个体的分数响应变量gydF4y2BajgydF4y2Ba,gydF4y2BaNgydF4y2Ba样本数目(见[gydF4y2Ba94gydF4y2Ba]查阅详情)。最规范的选择gydF4y2BaGgydF4y2Ba(gydF4y2Ba∗gydF4y2Ba)是逻辑函数,它允许将回归系数解释为比值比(OR)。在微生物组数据的情况下,OR指的是特定序列被分配给特定物种的机会。例如,考虑一个病例对照设计,在这些病例中,某一物种的平均相对丰度为gydF4y2Ba\({\overline{y}}_{\mathrm{cases}}=0.04 \)gydF4y2Ba在控制中,gydF4y2Ba\({\overline{y}}_{\mathrm{controls}}=0.02 \)gydF4y2Ba。然后,使用logit参数化的分数回归,将物种的相对丰度作为响应变量,将组变量作为预测变量,将得到:gydF4y2Ba
因此,在这个例子中,我们可以说,在这种情况下,特定序列被分配给这个物种的几率是对照组的2.04倍。现在,就像在其他回归分析中一样,我们可以包括协变量,以我们想要控制的一组变量为条件,得出估计。请注意,我们在此将相对丰度理解为对某一物种被分配到某一物种的概率的估计。gydF4y2Ba
所有分数回归都包括技术协变量,我们指的是批(测序运行)、总读取计数和未分类序列读取计数(不可能独立于所使用的任何置信度估计值阈值进行分类分配的读取)。读计数变量被包含在统计模型中,因为已经表明,如果应用除法所隐含的某些统计假设不满足,则通过除法进行的归一化可能会引入偏差[gydF4y2Ba95gydF4y2Ba]。在宏基因组数据的情况下,如果观察结果从多项分布中采样,则读取计数的影响将通过分割来消除。然而,这不是给定的,因为种和属之间相互关联,违反了构建多项分布所需的假设。因此,通过分割的读计数归一化很容易在宏基因组数据中引入偏差;作为潜在的偏差,我们通过将读取计数作为协变量纳入模型来纠正。gydF4y2Ba
在拟合最终的统计模型之前,我们使用上述的分数回归探讨了基本协变量(即年龄、性别和BMI)与宏基因组特征的关联,以避免统计模型的错误规范。由于数据显示年龄和BMI的范围很广,我们通过将这些变量作为受限三次样条纳入模型来测试潜在的非线性关联[gydF4y2Ba96gydF4y2Ba用5%百分位数、中位数和95%百分位数来定义三个结点。就像年龄的情况一样,我们发现物种与年龄有非线性的联系gydF4y2BapgydF4y2Ba< 0.01;在所有的分析中,年龄都是通过限制三次样条来建模的。gydF4y2Ba
所有gydF4y2BapgydF4y2Ba值是双尾报告的。在STATA 14/MP (College Station, TX, USA)中进行统计分析。在附加文件中给出了执行分析的汇总统计gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba。gydF4y2Ba
PD组与对照组微生物组成差异及协变量影响gydF4y2Ba
为了分析PD与对照组之间属丰度的差异,以属的相对丰度作为响应变量进行分数回归,同时将技术协变量、年龄(限制三次样条)、性别和BMI纳入统计模型。感兴趣的预测因子是研究组指标变量。我们使用Benjamini-Hochberg程序对多重测试进行了修正[gydF4y2Ba97gydF4y2Ba通过将错误发现率(FDR)设置为0.05。因此,我们在报告属结果时纠正了62个测试。这些分析在物种的分类水平上类似地重复,同时通过FDR纠正多重测试。gydF4y2Ba
接下来,我们探讨了基本协变量(年龄、性别和BMI)与群体指标之间统计相互作用的可能性。对于这些分析,我们再次通过允许非线性相互作用项的受限三次样条对年龄和BMI进行建模。我们只测试了双向互动。所有相互作用项同时引入统计模型,并通过Wald检验检验显著性[gydF4y2Ba96gydF4y2Ba],通过罗斯福修正多重测试。对于全局显著性检验,研究了单个相互作用项,以探索哪些协变量-组相互作用有助于整体显著性。为了解释,通过绘制以技术协变量为条件的预测来直观地检查相互作用项。然后用物种丰度代替属丰度作为响应变量重新进行这些分析。gydF4y2Ba
我们评估了便秘对微生物组成的影响。我们将二进制预测便秘(是/否)作为一个额外的预测因子引入模型和相应的群体便秘相互作用项。用Wald检验同时在零上检验两项。分析再次调整了技术协变量、年龄(限制三次样条)、性别和BMI,并通过FDR对多重检验进行了校正。gydF4y2Ba
PD内表型与微生物组成的关系分析gydF4y2Ba
我们调查了有关微生物组成的药物与临床特征的关联模式。这些分析仅在IPD病例中进行,而对照组被排除在分析之外。首先,我们分析了粪便采样日期和诊断年份之间以年为单位测量的疾病持续时间。分析与之前一样,通过分数回归进行,属丰度作为响应变量,同时调整技术协变量,年龄(限制三次样条),性别和BMI。然后,我们在单独的分析中评估UPDRS III评分作为运动症状的指标,NMS测量的非运动症状,疾病的Hoehn-Yahr分期作为疾病进展的整体衡量标准,以及嗅觉评分。所有这些分析都校正了技术协变量、年龄(受限三次样条)、性别、BMI和疾病持续时间。这些回归序列中的每一个都代表了62个测试,这些测试使用FDR进行了解释。通过检查三种药物来分析药物的影响:(a)左旋多巴,(b)单氨基氧化酶/儿茶酚-gydF4y2BaOgydF4y2Ba甲基转移酶抑制剂和(c)多巴胺受体激动剂。我们生成了三种相应的二元表型(摄入/不摄入),并将这三个变量同时添加到统计模型中,通过Wald检验确定该附加项的显著性。然后,我们在单独的分析中测试了每个药物类别,严格纠正了通过FDR进行的多次测试(总共186次测试)。对技术协变量、年龄(受限三次样条)、性别、BMI和疾病持续时间进行校正。gydF4y2Ba
微生物群落的个性化约束建模gydF4y2Ba
AGORA由一组818个菌株特异性基因组尺度的代谢重建组成,这些代谢重建是针对人类肠道中常见的微生物的[gydF4y2Ba23gydF4y2Ba,gydF4y2Ba46gydF4y2Ba]。为了匹配物种分类学分辨率,我们使用createPanModels函数将同一物种的菌株特异性代谢重建中的代谢物和生化反应结合在一个泛物种重建(“panSpeciesModel.m”)中。微生物组建模工具箱的M " [gydF4y2Ba24gydF4y2Ba]。对所有菌株特异性生物量反应进行平均,建立泛生物量反应。以这种方式共组装了646个物种特异性代谢重建。随后,我们在SPINGO物种分类分配和panSpecies名称之间进行了名称自动匹配。请注意,我们必须忽略所有不存在于这646个代谢重建中但在微生物组数据中确定的物种(附加文件)gydF4y2Ba1gydF4y2Ba:表1)。采用相对丰度为0.0001的细菌存在评估阈值来减少计算时间,同时将化学计量系数模拟结果的数量级限制为10。当考虑到至少在10%的微生物组样本中发现的物种时,我们的物种模型和SPINGO物种分配之间共有259种重叠(附加文件)gydF4y2Ba1gydF4y2Ba表1)。下一步,将每个样品的微生物丰度信息整合到群落建模设置中,使用微生物组建模工具箱的自动化模块获得个性化的微生物组模型[gydF4y2Ba24gydF4y2Ba在COBRA工具箱中调用mgPipe [gydF4y2Ba92gydF4y2Ba[commit: b097185b641fc783fa6fea4900bdd303643a6a7e]。简而言之,群落成员的代谢模型通过一个共同的隔间连接起来,每个模型都可以分泌/摄取代谢物。每个微生物组模型中的代谢物(饮食)更新反应采用欧洲平均饮食作为输入约束[gydF4y2Ba46gydF4y2Ba]。欧洲人的平均饮食(参见附加文件)gydF4y2Ba1gydF4y2Ba:表S4)是根据奥地利的一项调查推断出来的,该调查包括1002名不同年龄的参与者[gydF4y2Ba98gydF4y2Ba]。根据各微生物模型目标函数之和,制定群落目标函数,约束下限为0.4 / d,上限为1 / d,对应于每2.5天排便一次至每天排便一次。一组交换反应将共享隔间与环境连接起来,从而能够预测代谢物的摄取(从规定的饮食中)和分泌通量率(代谢谱/NMPCs)与应用限制相一致。在从群落模型中去除未检测到的物种后,通过调整群落生物量对每个样品相对微生物丰度的化学计量系数来实现每个微生物组模型的个性化。gydF4y2Ba
相对反应丰度是通过将微生物组模型中发生反应的物种数量相加并按各自物种相对丰度对总和进行缩放来计算的。利用交换反应的通量变异性分析评估了这些微生物群落的代谢谱[gydF4y2Ba99gydF4y2Ba]。用于构建群落模型的AGORA微生物代谢重建图从VMH (gydF4y2Bawww.vmh.lifegydF4y2Ba, (gydF4y2Ba46gydF4y2Ba])。所有计算均在MATLAB版本2018a (Mathworks, Inc.)中进行,通过Tomlab (Tomlab, Inc.)接口使用IBM CPLEX (IBM, Inc.)求解器。gydF4y2Ba
通量的统计分析gydF4y2Ba
nmpc经对数变换,使分布的偏度减至最低[gydF4y2BaOne hundred.gydF4y2Ba]。应用这种类型的转换是因为单个NMPCs的倾斜分布非常不同。然后,与之前一样,使用4-SD异常值规则排除异常值。分析中只保留非零值超过50%的通量。此外,排除了分布不适合统计分析的NMPCs(例如,具有完全相同数值的大量观测值的分布),导致129个NMPCs被纳入分析。gydF4y2Ba
对NMPCs进行混合线性回归分析,包括批次作为随机效应。将批变量作为随机效应与固定效应方法相比具有更高的统计能力,但依赖于更严格的假设。我们用Hausman规格检验了相应的随机效应假设,没有发现违反随机效应假设的迹象。请注意,这种通过随机效应来解释批效应的可能性在通过固定效应来纠正批效应的分数回归中是不可用的。gydF4y2Ba
我们进行了与宏基因组数据相同的分析,唯一的例外是用线性混合模型代替了分数回归模型。在所有其他方面,分析都遵循同样的方案。使用FDR进行多次测试校正,校正了129个测试。gydF4y2Ba
物种对通量贡献的分析gydF4y2Ba
为了研究种和属的贡献,我们计算了所有纳入的属和所有分析的通量、两两相关和相应的方差贡献(平方相关)。根据效应大小的经典分类,我们将高于0.5(等于方差贡献的25%)的每个相关性分类为强相关性[gydF4y2Ba101gydF4y2Ba]。gydF4y2Ba
数据和材料的可用性gydF4y2Ba
本文的数据集(16S rRNA序列)不公开,因为它们与卢森堡帕金森研究及其内部规定有关。访问数据集的请求应直接发送给Rejko Krueger教授,联系方式为:gydF4y2Barejko.krueger@uni.lugydF4y2Ba。mgPipe管道可在COBRA工具箱(gydF4y2Bahttps://github.com/opencobra/cobratoolboxgydF4y2Ba),自定义脚本和相关文档可在GitHub存储库中获得:gydF4y2Bahttps://github.com/ThieleLab/CodeBase/tree/master/ND_collectgydF4y2Ba。gydF4y2Ba
参考文献gydF4y2Ba
Kalia LV, Lang AE, Hazrati LN, Fujioka S, Wszolek ZK, Dickson DW, Ross OA, Van Deerlin VM, Trojanowski JQ, Hurtig HI,等。lrrk2相关帕金森病与路易体病理的临床相关性中华神经科杂志,2015;32(1):1 - 5。gydF4y2Ba
Bonifati V, Rizzu P, Squitieri F, Krieger E, Vanacore N, van sweeten JC, Brice A, van Duijn CM, Oostra B, Meco G,等。j -1(PARK7),常染色体隐性遗传,早发性帕金森病的新基因。神经科学进展。2003;24(3):159-60。gydF4y2Ba
Di Fonzo A, Dekker MC, Montagna P, Baruzzi A, Yonova EH, Correia Guedes L, Szczerbinska A, Zhao T, Dubbel-Hulsman LO, Wouters CH,等。FBXO7突变导致常染色体隐性,早发性帕金森锥体综合征。神经学。2009;72(3):240 - 5。gydF4y2Ba
Kitada T, Asakawa S, Hattori N, Matsumine H, Yamamura Y, Minoshima S, Yokochi M, Mizuno Y, Shimizu N. parkin基因突变导致常染色体隐性青少年帕金森病。大自然。1998;392(6676):605 - 8。gydF4y2Ba
Paisan-Ruiz C, Jain S, Evans EW, Gilks WP, Simon J, van der Brug M, Lopez de Munain A, Aparicio S, Gil AM, Khan N,等。克隆含有导致park8相关帕金森病突变的基因。神经元。2004;44(4):595 - 600。gydF4y2Ba
Gatto NM, Rhodes SL, Manthripragada AD, Bronstein J, Cockburn M, Farrer M, Ritz B. α-Synuclein基因可能与环境因素相互作用增加帕金森病的风险。神经。2010;35(3):191 - 5。gydF4y2Ba
刘立文,刘立文,王爱华,王志强。帕金森病的胃肠功能障碍。中华神经科杂志,2015;14(6):625-39。gydF4y2Ba
Savica R, Carlin JM, Grossardt BR, Bower JH, Ahlskog JE, Maraganore DM, Bharucha AE, Rocca WA。帕金森病前便秘的医疗记录:一项病例对照研究神经学。2009;73(21):1752 - 8。gydF4y2Ba
Cersosimo MG, Raina GB, Pecci C, Pellene A, Calandra CR, Gutierrez C, Micheli FE, Benarroch EE。帕金森病的胃肠道表现:运动症状出现前的患病率和发生率。中国生物医学工程学报,2013;32(5):563 - 568。gydF4y2Ba
人类的超个体——由微生物和人组成。医学进展与展望。2010;32(2):391 - 391。gydF4y2Ba
马建军,李建军,李建军,等。肠脑轴:肠道微生物群与中枢神经系统的相互作用。胃肠病学杂志,2015;28(2):203-9。gydF4y2Ba
Bedarf JR, Hildebrand F, Coelho LP, Sunagawa S, Bahram M, Goeser F, Bork P, wulner U.早期左旋多巴初始化帕金森病患者肠道微生物和病毒宏基因组变化的功能意义。中华医学杂志,2017;9(1):39。gydF4y2Ba
Heintz-Buschart A, Pandey U, Wicke T, Sixel-Doring F, Janzen A, Sittig-Wiegand E, Trenkwalder C, Oertel WH, Mollenhauer B, Wilmes P.帕金森病和特发快速眼动睡眠行为障碍的鼻和肠道微生物群。数学学报,2018;33(1):88-98。gydF4y2Ba
Barichella M, Severgnini M, Cilia R, Cassani E, Bolliri C, Caronni S, Ferri V, Cancello R, Ceccarani C, Faierman S等。揭示帕金森病和非典型帕金森病的肠道微生物群。力学学报,2019;34(3):396-405。gydF4y2Ba
Scheperjans F, ho V, Pereira PA, Koskinen K, Paulin L, Pekkonen E, Haapaniemi E, Kaakkola S, eerola - ratio J, Pohja M,等。肠道菌群与帕金森病和临床表型相关。运动失调,2015;30(3):350-8。gydF4y2Ba
Keshavarzian A, Green SJ, Engen PA, Voigt RM, Naqib A, Forsyth CB, Mutlu E, Shannon KM。帕金森病的结肠细菌组成。运动障碍,2015;30(10):1351-60。gydF4y2Ba
Hill-Burns EM, Debelius JW, Morton JT, Wissemann WT, Lewis MR, Wallen ZD, pedada SD, Factor SA, Molho E, Zabetian CP等。帕金森氏症和帕金森氏症药物具有明显的肠道微生物特征。Mov disorder . 2017;32(5): 739-49。gydF4y2Ba
Petrov VA, Saltykova IV, Zhukova IA, Alifirova VM, Zhukova NG, Dorofeeva YB, Tyakht AV, Kovarsky BA, Alekseev DG, Kostryukova ES,等。帕金森病患者肠道菌群分析。中国生物医学工程学报,2017;32(6):734 - 737。gydF4y2Ba
长谷川S,后藤S, Tsuji H, Okuno T, Asahara T,野本K,柴田A,藤泽Y, Minato T,冈本A等。帕金森病的肠道失调和血清脂多糖结合蛋白降低。科学通报,2015;10(11):e0142164。gydF4y2Ba
Hopfner F, Kunstner A, Muller SH, Kunzel S, Zeuner KE, Margraf NG, Deuschl G, Baines JF, Kuhlenbaumer G.德国北部帕金森病的肠道微生物群。中国生物医学工程学报,2017;17(6):391 - 391。gydF4y2Ba
Sampson TR, Debelius JW, Thron T, Janssen S, Shastri GG, Ilhan ZE, Challis C, Schretter CE, Rocha S, Gradinaru V,等。肠道微生物群调节帕金森病模型中的运动缺陷和神经炎症。细胞学报,2016;16(6):1469-80。gydF4y2Ba
north JD, Thiele I, Palsson BO。什么是通量平衡分析?生物工程学报,2010;28(3):245-8。gydF4y2Ba
Magnusdottir S, Heinken A, Kutt L, Ravcheev DA, Bauer E, Noronha A, Greenhalgh K, Jager C, Baginska J, Wilmes P,等。人类肠道菌群773个成员的基因组级代谢重建。生物工程学报,2017;35(1):81-9。gydF4y2Ba
李建军,李建军,李建军,李建军,李建军。微生物群落模型的研究进展。生物信息学》2018。gydF4y2Ba
Heinken A, Ravcheev DA, Baldini F, Heirendt L, Fleming RMT, Thiele I.人类肠道微生物组的个性化建模揭示了健康和IBD个体不同的胆汁酸解结和生物转化潜力。微生物。2019;7:75。gydF4y2Ba
李建军,李建军,李建军,李建军,李建军,李建军。个性化的全身模型整合了新陈代谢、生理学和肠道微生物组。中国生物医学工程学报,2016;16(5):889 - 882。gydF4y2Bahttps://doi.org/10.15252/msb.20198982gydF4y2Ba。gydF4y2Ba
李建平,李建平,李建平,等。缺氧条件下肠道微生物间的相互作用。环境微生物学报,2015;81(12):4049-61。gydF4y2Ba
李建平,李建平。微生物合成生态系统的研究进展。科学通报,2010;6(11):e1001002。gydF4y2Ba
Hertel J, Harms AC, Heinken A, Baldini F, Thinnes CC, Glaab E, Vasco DA, Pietzner M, Stewart ID, Wareham NJ等。微生物组和纵向代谢组数据的综合分析揭示了微生物-宿主相互作用对帕金森病硫代谢的影响。中国生物医学工程学报,2019;29(7):1767-77 e1768。gydF4y2Ba
Hipp G, Vaillant M, Diederich NJ, Roomp K, Satagopam VP, Banda P, Sandt E, Mommaerts K, Schmitz SK, Longhino L,等。卢森堡帕金森研究:分层和早期诊断的综合方法。神经科学进展,2018;10:326。gydF4y2Ba
张建军,李建军,李建军,等。一种微生物生态系统功能均匀度指标。中华微生物学杂志,2001;7(4):283-96。gydF4y2Ba
孙海杰,金宁,宋昌,南荣,崔思,金三思,李德生。炎症性肠病C57BL/6小鼠模型中肠道微生物群的性别相关改变中华癌症杂志,2019;24(3):173-82。gydF4y2Ba
贾格尔M,雷凯,Spichak S,狄南TG, Cryan JF。你有男性:性别和贯穿一生的微生物-肠道-大脑轴。中华神经内分泌杂志,2019;100815。gydF4y2Ba
李伟,吴鑫,胡鑫,王涛,梁松,段勇,金峰,秦波。帕金森病肠道菌群结构变化及其与临床特征的相关性。中国科学:生命科学,2017;60(11):1223-33。gydF4y2Ba
林安,郑伟,何勇,唐伟,魏鑫,何锐,黄伟,苏勇,黄勇,周宏,等。中国南方帕金森病患者的肠道微生物群帕金森氏症相关障碍。2018;53:82-8。gydF4y2Ba
钱燕,杨霞,徐生,吴超,宋燕,秦宁,陈世德,肖强。中国帕金森病患者粪便微生物群的变化。中国生物医学工程学报,2018;37(2):444 - 444。gydF4y2Ba
胃肠蠕动频率与帕金森病的未来风险。神经病学杂志,2002;58(5):838作者回复838-839。gydF4y2Ba
Boertien JM, Pereira PAB, Aho VTE, Scheperjans F.帕金森病肠道微生物组研究的可比性和实用性:系统综述。[J] .植物学报,2019;9(2):997 - 992。gydF4y2Ba
De Filippo C, Cavalieri D, Di Paola M, Ramazzotti M, Poullet JB, Massart S, Collini S, Pieraccini G, Lionetti P.饮食对欧洲和非洲农村儿童肠道微生物群形成的影响的比较研究。中国科学:自然科学学报,2010;37(3):14691-6。gydF4y2Ba
谭博PJ, Backhed F, Fulton L, Gordon JI。饮食引起的肥胖与小鼠远端肠道微生物群的显著但可逆的改变有关。细胞与宿主微生物。2008;3(4):213-23。gydF4y2Ba
Yatsunenko T, Rey FE, Manary MJ, Trehan I, Dominguez-Bello MG, Contreras M, Magris M, Hidalgo G, Baldassano RN, Anokhin AP,等。不同年龄和地域的人类肠道微生物组。大自然。2012;486(7402):222 - 7。gydF4y2Ba
Wirbel J, Pyl PT, Kartal E, Zych K, Kashani A, Milanese A, Fleck JS, Voigt AY, Palleja A, Ponnudurai R,等。粪便宏基因组的荟萃分析揭示了结直肠癌特异性的全球微生物特征。中华医学杂志,2019;25(4):679-89。gydF4y2Ba
黄锐,李婷,倪军,白霞,高燕,李燕,张鹏,龚莹。肝脏特异性tsc1基因敲除小鼠肝癌发生过程中肠道微生物群的性别差异反应。中华微生物学杂志,2018;9:1008。gydF4y2Ba
李建军,李建军,李建军,等。胃保留技术在小肠上窗给药中的应用。时药学杂志,2006;6(5):501-8。gydF4y2Ba
van Kessel SP, Frye AK, El- gendy AO, Castejon M, Keshavarzian A, van Dijk G, El Aidy S.肠道细菌酪氨酸脱羧酶限制左旋多巴治疗帕金森病的水平。生物医学工程学报,2019;10(1):310。gydF4y2Ba
Noronha A, Modamio J, Jarosz Y, guard E, Sompairac N, Preciat G, Danielsdottir AD, Krecke M, Merten D, Haraldsdottir HS,等。虚拟人体代谢数据库:将人体和肠道微生物代谢与营养和疾病相结合。核酸学报,2019;47(D1): D614-24。gydF4y2Ba
Devkota S, Wang Y, Musch MW, Leone V, Fehlner-Peach H, Nadimpalli A, Antonopoulos DA, Jabri B, Chang EB。饮食脂肪诱导的牛磺胆酸促进il - 10-/-小鼠的病原体扩张和结肠炎。大自然。2012;487(7405):104 - 8。gydF4y2Ba
Natividad JM, Lamas B, Pham HP, Michel ML, Rainteau D, Bridonneau C, da Costa G, van Hylckama VJ, Sovran B, Chamignon C,等。华氏双歧杆菌加重高脂饮食诱导的小鼠代谢功能障碍。生态学报,2018;9(1):2802。gydF4y2Ba
细胞损伤条件下小鼠脑干切片中牛磺酸的释放。氨基酸学报,2007;32(3):439-46。gydF4y2Ba
吴建勇,吴海,金勇,魏军,沙东,普伦蒂斯,李海红,林超,李永华,杨丽丽。牛磺酸神经保护作用的机制。中华医学杂志,2009;43(3):379 - 379。gydF4y2Ba
劳·H,库克上午。厌氧菌华氏Bilophila wadsworthia牛磺酸:丙酮酸转氨酶的生化和分子表征。中国生物医学工程学报,2009;26(3):444 - 444。gydF4y2Ba
Graham SF, Rey NL, Ugur Z, Yilmaz A, Sherman E, Maddens M, Bahado-Singh RO, Becker K, Schulz E, Meyerdirk LK,等。前驱帕金森病实验模型胆汁酸代谢组学分析。中国生物医学工程学报,2018;8(4):1145 - 1145。gydF4y2Ba
Vandeputte D, Falony G, Vieira-Silva S, Wang J, Sailer M, Theis S, Verbeke K, Raes J.益生元胰岛素型果聚糖诱导人类肠道微生物群的特异性变化。66。2017;(11):1968 - 74。gydF4y2Ba
Khalif IL, Quigley EM, Konovitch EA, Maximova ID。慢性便秘患者结肠菌群和肠道通透性的改变和免疫激活的证据。中华肝病杂志,2005;37(11):838-49。gydF4y2Ba
李建军,李建军,李建军,等。一种新一代有益微生物:嗜粘杆菌。临床生化杂志,2018;63(1):33 - 35。gydF4y2Ba
Schneeberger M, Everard A, Gomez-Valades AG, Matamoros S, Ramirez S, Delzenne NM, Gomis R, Claret M, Cani PD。嗜粘杆菌与小鼠肥胖期间炎症的发生、脂肪组织代谢的改变和代谢紊乱呈负相关。科学通报,2015;5:663。gydF4y2Ba
Grander C, Adolph TE, Wieser V, Lowe P, Wrzosek L, Gyongyosi B, Ward DV, Grabherr F, Gerner RR, Pfister A,等。恢复乙醇诱导的嗜粘阿克曼氏菌耗竭可改善酒精性肝病。肠道。2018;67(5):891 - 901。gydF4y2Ba
Hyland NP, Cryan JF。对GABA的直觉:关注GABA(B)受体。中华医学杂志,2010;1(1):124。gydF4y2Ba
Tonini M, Crema A, Frigo GM, Rizzi CA, Manzo L, Candura SM, Onori L.兔远端结肠GABA受体功能与推进运动关系的体外研究。中国生物医学工程杂志,2009;31(4):359 - 361。gydF4y2Ba
Bravo JA, Forsythe P, Chew MV, Escaravage E, Savignac HM, Dinan TG, Bienenstock J, Cryan JF。摄入乳酸杆菌菌株通过迷走神经调节小鼠的情绪行为和中枢GABA受体的表达。中国科学:自然科学版,2011;38(6):1049 - 1049。gydF4y2Ba
Vandeputte D, Falony G, Vieira-Silva S, Tito RY, Joossens M, Raes J.粪便一致性与肠道微生物群丰富度和组成、肠道类型和细菌生长速度密切相关。肠道。2016;65(1):57 - 62。gydF4y2Ba
Gobert AP, Sagrestani G, Delmas E, Wilson KT, Verriere TG, Dapoigny M, Del 'homme C, Bernalier-Donadille A.便秘型肠易激综合征患者肠道微生物群具有抗炎作用。科学通报2016;6:39 - 39。gydF4y2Ba
Abubucker S, Segata N, Goll J, Schubert AM, Izard J, Cantarel BL, Rodriguez-Mueller B, Zucker J, Thiagarajan M, Henrissat B,等。宏基因组数据的代谢重建及其在人类微生物组中的应用。科学通报,2012;8(6):e1002358。gydF4y2Ba
金久久,古道明,田边,陈志强,陈志强,陈志强,陈志强。核酸学报,2017;45(D1): D353-61。gydF4y2Ba
Thiele I, Palsson BØ。生成高质量基因组级代谢重建的方案。地理学报,2010;5(1):93-121。gydF4y2Ba
Janda JM, Abbott SL. 16S rRNA基因测序在诊断实验室中的细菌鉴定:优点,危险和陷阱。中华临床微生物学杂志,2007;45(9):2761-4。gydF4y2Ba
Allard G, Ryan FJ, Jeffery IB, claessmj。微生物扩增子序列的快速物种分类器。BMC生物信息学。2015; 16:324。gydF4y2Ba
李建平,李建平。人体代谢的计算模型及其在系统生物医学中的应用。方法:中华医学杂志。2016;36(1):253 - 81。gydF4y2Ba
Oberhardt MA, Palsson BO, Papin JA。基因组尺度代谢重建的应用。生物化学学报。2009;5:20 20。gydF4y2Ba
代谢系统生物学:发展个体化和精准医疗的驱动因素。细胞医学杂志,2017;25(3):572-9。gydF4y2Ba
李建平,李建平。微生物代谢组学的研究进展。中国生物医学工程学报,2015;7(4):195-219。gydF4y2Ba
李建平,李建平。基于遗传算法的人-微生物协同代谢研究进展。肠道微生物,2015;6(2):120-30。gydF4y2Ba
Thiele I, Heinken A, Fleming RM。研究微生物在人类健康中的作用的系统生物学方法。生物工程学报,2013;24(1):4-12。gydF4y2Ba
Thiele I, Clancy CM, Heinken A, Fleming RMT。定量系统药理学和个性化药物-微生物-饮食轴。生物医学工程学报,2017;4:43-52。gydF4y2Ba
Crichton GE, Alkerwi A.久坐行为时间与理想心血管健康的关系:ORISCAV-LUX研究。科学通报,2014;9(6):e99829。gydF4y2Ba
刘建军,张建军,张建军,张建军,张建军,张建军,张建军,张建军,张建军,张建军。2013 - 2015年欧洲地区高血压风险因素分析。地中海(巴尔的摩)。2016; 95 (36): e4758。gydF4y2Ba
Hughes AJ, Daniel SE, Kilford L, Lees AJ。特发性帕金森病临床诊断的准确性:100例临床病理研究中华神经外科杂志。1992;21(3):391 - 391。gydF4y2Ba
美国胃肠病学会慢性便秘任务f:北美慢性便秘管理的循证方法。中华胃肠病杂志。2005;21(增刊1):1 - 4。gydF4y2Ba
Pare P, Bridges R, Champion MC, Ganguli SC, Gray JR, Irvine EJ, Plourde V, Poitras P, Turnbull GK, Moayyedi P,等。关于慢性便秘(包括与肠易激综合征相关的便秘)治疗的建议。中华胃肠病杂志。2007;21(增刊B): 3b - 3b。gydF4y2Ba
Goetz CG, Tilley BC, Shaftman SR, Stebbins GT, Fahn S, Martinez-Martin P, Poewe W, Sampaio C, Stern MB, Dodel R等。运动障碍学会赞助的统一帕金森病评定量表(MDS-UPDRS)的修订:量表的呈现和临床测试结果。力学学报,2008;23(15):2129-70。gydF4y2Ba
Hoehn MM, Yahr MD。帕金森病:发病、进展和死亡率。神经学。1967;17(5):427 - 42。gydF4y2Ba
罗梅纳斯,伍夫森,盖拉塔斯,佩尔蒂埃,阿尔特曼,Wadup L, Postuma RB。非运动症状问卷(NMS-Quest)的验证。帕金森病相关障碍[j] . 2012;18(1): 54-8。gydF4y2Ba
Lehmann S, Guadagni F, Moore H, Ashton G, Barnes M, Benson E, Clements J, Koppandi I, Coppola D, demmiroglu SY,等。生物标本的标准预分析编码:样品预分析代码(SPREC)的审查和实施生物资源学报,2012;10(4):366-74。gydF4y2Ba
matay C, Hamot G, Henry E, Georges L, Bellora C, Lebrun L, de Witt B, Ammerlaan W, Buschart A, Wilmes P,等。粪便样本采集及粪便DNA提取方法优化。生物储备,2015;13(2):79-93。gydF4y2Ba
刘建军,刘建军,刘建军,等。人类粪便DNA提取方法的研究进展[j]。生物储备生物银行,2020。gydF4y2Bahttps://doi.org/10.1089/bio.2019.0112gydF4y2Ba。gydF4y2Ba
Anderson EL, Li W, Klitgord N, Highlander SK, Dayrit M, Seguritan V, youseph S, Biggs W, Venter JC, Nelson KE,等。一个强大的环境温度收集和稳定策略:实现人类微生物组的全球功能研究。科学通报2016;6:31731。gydF4y2Ba
Ilett EE, Jorgensen M, Noguera-Julian M, Daugaard G, Murray DD, Helleberg M, Paredes R, Lundgren J, Sengelov H, MacPherson C.利用16S rRNA基因和shotgun宏基因组测序分析新鲜冷冻和稳定冷冻住院患者肠道微生物组的比较。科学通报,2019;9(1):1 - 3。gydF4y2Ba
Parikh HI, Koparde VN, Bradley SP, Buck GA, Sheth NU。MeFiT:用于16S rRNA扩增子测序的illumina配对末端读段合并和过滤工具。BMC生物信息学。2016; 17(1): 491。gydF4y2Ba
Ihaka R, Gentleman R. R:一种数据分析和图形语言。计算机图形统计。1996;5(3):299-314。gydF4y2Ba
费斯特是我,帕尔森是我。生物量目标函数。微生物学杂志,2010;13(3):344-9。gydF4y2Ba
从宏基因组数据到个性化的计算机微生物群:预测克罗恩病的膳食补充剂。中国生物医学工程学报,2018;4:27。gydF4y2Ba
herendt L, Arreckx S, Pfau T, Mendoza SN, Richelle A, Heinken A, Haraldsdottir HS, Wachowiak J, Keating SM, Vlasov V,等。使用COBRA工具箱v.3.0创建和分析基于生化约束的模型。自然科学学报,2019;14(3):639-702。gydF4y2Ba
Oksanen J, Blanchet FG, Friendly M, Kindt R, Legendre P, mcglin D, Minchin PR, O 'Hara RB, Simpson GL, Solymos P等:素食:群落生态包(R包版本2.5-2,2018)。https://CRAN.R-project.org/package=vegan;2018.gydF4y2Ba
帕克LE,伍尔德里奇JM。分数响应变量的计量经济学方法与401(k)计划参与率的应用。应用经济学报。1996;11(6):619-32。gydF4y2Ba
Hertel J, Rotter M, Frenzel S, Zacharias HU, Krumsiek J, Rathkolb B, Hrabe de Angelis M, Rabstein S, Pallapies D, Bruning T,等。动态影响尿液分析物数据的稀释校正。植物学报,2018;32(2):18 - 31。gydF4y2Ba
哈勒尔菲。回归建模策略。编辑:Harrell FE。纽约:斯普林格出版社;2001.gydF4y2Ba
benjamin Y.发现错误发现率。[J]中国农业大学学报(自然科学版);2010;32(4):444 - 444。gydF4y2Ba
Elmadfa I. Österreichischer Ernährungsbericht 2012。第1版,维也纳;2012.gydF4y2Bahttps://ernaehrungsbericht.univie.ac.at/fileadmin/user_upload/dep_ernaehrung/forschung/ernaehrungsberichte/oesterr_ernaehrungsbericht_2012.pdfgydF4y2Ba。gydF4y2Ba
李建平,李建平。基于计算效率的通量变率分析。BMC生物信息学。2010; 11:489。gydF4y2Ba
Box GEP, Cox博士,转型分析。统计方法系列B. 1964;26(2): 211-43。gydF4y2Ba
J.行为科学的统计能力分析,第1卷。第2版。纽约:劳特利奇出版社;1988.gydF4y2Ba
致谢gydF4y2Ba
我们要特别感谢所有参与研究的患者。此外,我们非常感谢所有收到的资金和私人捐款,使我们能够开展这个项目。此外,我们感谢NCER-PD联盟成员对卢森堡帕金森研究的共同努力,如下所列:Aguayo, Gloria;艾伦,多米尼克;Ammerlann Wim;Aurich,迈科;球磨机,鲁迪;班达,彼得;博蒙特,凯蒂;贝克尔,Regina; Berg, Daniela; Binck, Sylvia; Bisdorff, Alexandre; Bobbili, Dheeraj; Brockmann, Kathrin; Calmes, Jessica; Castillo, Lorieza; Diederich, Nico; Dondelinger, Rene; Esteves, Daniela; Ferrand, Jean-Yves; Fleming, Ronan; Gantenbein, Manon; Gasser, Thomas; Gawron, Piotr; Geffers, Lars; Giarmana, Virginie; Glaab, Enrico; Gomes, Clarissa P.C.; Goncharenko, Nikolai; Graas, Jérôme; Graziano, Mariela; Groues, Valentin; Grünewald, Anne; Gu, Wei; Hammot, Gaël; Hanff, Anne-Marie; Hansen, Linda; Hansen, Maxime; Haraldsdöttir, Hulda; Heirendt, Laurent; Herbrink, Sylvia; Herzinger, Sascha; Heymann, Michael; Hiller, Karsten; Hipp, Geraldine; Hu, Michele; Huiart, Laetitia; Hundt, Alexander; Jacoby, Nadine; Jarosław, Jacek; Jaroz, Yohan; Kolber, Pierre; Kutzera, Joachim; Landoulsi, Zied; Larue, Catherine; Lentz, Roseline; Liepelt, Inga; Liszka, Robert; Longhino, Laura; Lorentz, Victoria; Mackay, Clare; Maetzler, Walter; Marcus, Katrin; Marques, Guilherme; Martens, Jan; Mathay, Conny; Matyjaszczyk, Piotr; May, Patrick; Meisch, Francoise; Menster, Myriam; Minelli, Maura, Mittelbronn, Michel; Mollenhauer, Brit; Mommaerts, Kathleen; Moreno, Carlos; Mühlschlegel, Friedrich; Nati, Romain; Nehrbass, Ulf; Nickels, Sarah; Nicolai, Beatrice; Nicolay, Jean-Paul; Noronha, Alberto; Oertel, Wolfgang; Ostaszewski, Marek; Pachchek, Sinthuja; Pauly, Claire; Perquin, Magali; Reiter, Dorothea; Rosety, Isabel; Rump, Kirsten; Satagopam, Venkata; Schlesser, Marc; Schmitz, Sabine; Schmitz, Susanne; Schneider, Reinhard; Schwamborn, Jens; Schweicher, Alexandra; Simons, Janine; Stute, Lara; Trefois, Christophe; Trezzi, Jean-Pierre; Vaillant, Michel; Vasco, Daniel; Vyas, Maharshi; Wade-Martins, Richard; and Wilmes, Paul.
资金gydF4y2Ba
本研究由欧洲研究委员会(ERC)根据欧盟地平线2020研究与创新计划(资助协议编号757922)向IT提供资助,由卢森堡国家研究基金(FNR)在国家卓越研究中心(NCER)内资助帕金森病(FNR/ NCER13/BM/11264123)和PEARL计划(FNR/P13/6682797给RK),以及欧盟地平线2020研究与创新计划资助协议编号692320给RK。gydF4y2Ba
作者信息gydF4y2Ba
作者及单位gydF4y2Ba
财团gydF4y2Ba
贡献gydF4y2Ba
IT和RK构思了这项研究。FB, JH和IT起草了手稿。JH和FB分析了数据。FB, JH, IT, RK和LP解释数据。ES, LN-C和FB进行测序。CCT、LP和RK对卢森堡帕金森研究的研究设计做出了贡献。所有作者都阅读并批准了最终的手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
伦理批准并同意参与gydF4y2Ba
所有研究参与者均给予书面知情同意,研究按照赫尔辛基宣言进行。卢森堡帕金森研究[gydF4y2Ba30.gydF4y2Ba]已获得国家伦理委员会(CNER Ref: 201407/13)和数据保护委员会(CNPD Ref: 446/2017)的批准。gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者宣称他们没有竞争利益。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba
伟德体育在线施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。gydF4y2Ba
补充信息gydF4y2Ba
附加文件1。gydF4y2Ba
微生物丰度分析的扩展结果(图S1、S2和S3)以及关于群落模型特征和读取计数的背景信息(表S1、S2、S3和S4)。gydF4y2Ba
附加文件2。gydF4y2Ba
物种表型关联分析的汇总统计。gydF4y2Ba
附加文件3。gydF4y2Ba
属表型关联分析的汇总统计。gydF4y2Ba
附加文件4。gydF4y2Ba
对净生产能力的差异贡献的汇总统计。gydF4y2Ba
附加文件5。gydF4y2Ba
净生产能力表型关联分析汇总统计。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接获得版权所有者的许可。如欲查阅本许可证副本,请浏览gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba。创作共用公共领域免责声明(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本文中提供的数据,除非在数据的信用额度中另有说明。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
Baldini, F., Hertel, J., Sandt, E.。gydF4y2Baet al。gydF4y2Ba帕金森病相关肠道微生物组的改变预测了代谢功能的疾病相关变化。gydF4y2BaBMC医学杂志gydF4y2Ba18gydF4y2Ba, 62(2020)。https://doi.org/10.1186/s12915-020-00775-7gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12915-020-00775-7gydF4y2Ba
关键字gydF4y2Ba
- 帕金森病gydF4y2Ba
- 肠道微生物组gydF4y2Ba
- 计算模型gydF4y2Ba
- 代谢模型gydF4y2Ba
- Transsulfuration通路gydF4y2Ba