摘要
宏基因组学允许探索微生物群落的各个方面,这些方面是基于培养的针对单个微生物的方法无法达到的。利用宏基因组学已经发现了许多新的微生物分类群和基因,但不同种类的“未知”仍然存在于微生物组实验中。我们在这里讨论是否可能以及如何可能处理这些问题。
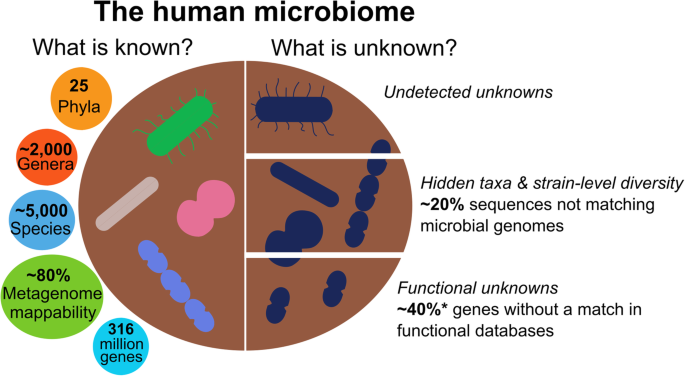
在过去的十年中,由于宏基因组领域的生物技术和计算技术的进步,我们对居住在人体和其他环境中的微生物群落的理解有了很大的提高[1].特别值得注意的是,在鉴定和遗传描述新微生物物种方面的成功努力,这些新微生物物种以前是一组未知微生物的一部分,有时被称为“微生物暗物质”。然而,在典型的微生物组实验中,微生物群落的几个方面仍然无法获得。这种无法充分探索样品中微生物组多样性的情况发生在多个不同的水平上。1),并应予以承认,以避免误解和过度解读。
在隐藏多样性的最深层,有一些群体成员根本没有被实验捕捉到,他们就是未被发现的未知。这些包括低丰度但可能至关重要的分类群,其遗传物质由于低于检测水平而无法通过测序技术进行采样。这个阈值的确切位置部分取决于实验选择和具体技术;例如,宿主细胞和DNA在样本中的优势地位(例如,肠粘膜活检)使得微生物分类群更难被发现,这是宏基因组学实验中的一个常见问题。与基于测序的方法相比,培养对样品中微生物浓度的敏感性较低,并且在表征低丰度分类群方面做出了重大贡献,特别是在高通量环境中应用时[4].然而,现有的分离方案不可避免地偏向于某些类别的微生物,并且仅对微生物组生物多样性的一小部分成功。噬菌体由于其短基因组和生化特性(例如,具有RNA或单链DNA基因组)通常不被标准样品制备方案考虑,因此特别容易取样不足。虽然病毒体富集方案已经开发和应用,病毒仍然可能是微生物群落中最被忽视的一类成员。
微生物组分类群的DNA至少在微生物组实验中被部分测序,但以前没有被描述过,并且在系统发育上与存放在公共数据库中的基因组相差甚远,这代表了另一种未表征的多样性。这是为了如此难以刻画隐藏的分类单元受物理学启发,“微生物暗物质”一词最初被创造出来[5,6].然而,这种类比受到了质疑。7],因为在物理学中,暗物质被认为是一种不同形式的物质,而在微生物学中,未发现的微生物与已知的微生物具有相同的分子基础。这种类型的微生物隐藏多样性是大规模分离测序和宏基因组组装工作的有效目标,最近发现了许多以前未被探索的分类群[2,8].由于将新分类群整合到参考基因组中,微生物组可以得到更全面的分析,因为霰弹枪测序实验中与已编目的微生物基因组相匹配的部分读数(即:宏基因组的可绘制性增加。通过大规模宏基因组组装工作,我们对人类肠道微生物组整体多样性的了解确实大大提高了。例如在我们的研究中[2],肠道宏基因组的可绘制率平均达到85%以上(中位数接近95%),而以前的可绘制率在50-70%之间。基于宏基因组学的独立努力[9,10]和大规模种植[8证实了这一趋势。除了肠道以外的人体部位,如皮肤和口腔,宏基因组的可绘制性也同样增加[2],而且对于更多样化的非人类环境,这些方法已被证明是有效和有前途的[11].然而,从未表征的分类群中组织大量的草图基因组是具有挑战性的,尽管对细菌表现良好,但基于组装的宏基因组工具在针对新的真核微生物和病毒时效果较差。
在细菌和古细菌中,种内基因组多样性可能是广泛的。事实上,几项关于(潜在)病原体的分离测序研究强调,存在于某一特定物种的某些菌株(但不是所有菌株)中的一组基因(即附属或可变基因组)可能比存在于该物种所有菌株中的一组“核心”基因大十倍以上。由于大多数微生物组物种的可用基因组很少(如果有的话),因此许多物种的辅助基因组代表性不足,因此微生物组中属于核心基因组以外区域的不可测绘遗传物质的比例可能很大。当肠道宏基因组与所有> 154,000个新恢复的宏基因组组装的基因组比对时,而不是与每个候选物种(包括已知和新定义的)的4930个单基因组代表比对时,观察到的约8%的映射性增加了这一点。非西化人群的阴道样本增加1.7%至粪便样本增加23.8% [2].在揭露方面取得进一步进展隐藏的菌株水平多样性因此,从分析的宏基因组中重建样本特异性组装并在参考数据库中包括尽可能多的每个物种的基因组是至关重要的。由于物种的泛基因组可能是“开放的”(即,没有附件基因组大小的上限),这主要是由于广泛的水平基因转移,从技术上讲,似乎不可能在样品中恢复物种的所有菌株水平多样性,但继续努力编目菌株变异对于深入了解微生物组的功能潜力仍然至关重要。
在整个微生物组及其单一微生物成分中编码的功能电位是理解微生物群落的关键。的功能未知然而,微生物组的研究范围要比它们的分类学对应物广泛得多,也更难解决。这种功能的不可接近性源于我们对微生物基因组中基因和途径的有限理解,特别是对非模式生物,以及微生物组成员广泛的系统发育多样性,导致序列同源性只能部分捕获功能相似性。以功能和基因为中心描述宏基因组的努力包括创建人类肠道微生物组的综合基因目录(IGC),其中包含近1000万个基因[3.].这是一种非冗余资源,以≥95%的身份阈值和≥90%的重叠将基因分组,从而将人类微生物组中极其庞大的独特基因(超过3.16亿个)分解为基因家族[2].有趣的是,IGC目录中存在的39.6%的基因未映射到功能数据库中。然而,将基因与功能数据库中的目标基因相匹配的能力,只是对其功能进行注释的部分步骤;例如,在IGC中注释的60.4%基因中,有15-20%是以前观察到的基因,但被标记为“功能未知”[3.].这些数字表明,对于微生物群落中存在的基因及其功能,我们所知甚少。而对于分类学和系统发育多样性,最新的高通量技术正在迅速减少难以接近的分类群的比例,基因的实验功能表征本身就难以在高通量和成本效益的系统中进行扩展,并且没有得到足够的研究投资。虽然对不同条件下宏基因组的功能潜力进行比较分析可以帮助确定实验功能表征中基因的优先级,但很可能对微生物组的功能理解在短期内无法得到实质性改善,这似乎是该领域的主要限制因素之一。
当前和未来的努力将揭示微生物群未被探索的方面,这将对几个应用产生直接影响。粪便微生物组移植就是这样一个例子,因为更完整的肠道微生物组样本分析可以更好、更安全地选择供体样本,并提高对哪种分类群对这种医疗实践的成功贡献最大的理解。揭示目前无法获得的微生物组成员对于扩展疾病预测分类和功能微生物组特征也至关重要[12],并更好地描述研究较少的种群和环境,从而显示出更大比例的未开发多样性。最近用宏基因组学描述了在整个生命之树中具有有趣的系统发育位置的几个新门。13],而这种已编目的微生物多样性的不断扩大也可能有助于我们对若干生物学方面的理解,例如真核发生的过程、真核细胞的起源[14].
微生物组领域已经准备好接受新的和改进的技术,以继续减少微生物组实验中不同程度的未知因素的影响。这些范围从高通量培养[4]到单细胞测序[6],但也需要改进的计算方法来更深入地探索宏基因组数据集,特别是在大规模的情况下。然而,对微生物组的功能理解仍然是最大的挑战,尽管针对特定基因的低通量实验是不可替代的,但技术可以再次提供补充解决方案。这些包括微生物转录组、代谢组和蛋白质组的综合高通量分析,以及基于培养的分析的自动化,以扩大对感兴趣表型的多个分类群和基因的筛选。因此,有条件实质性地揭示目前难以接近的微生物组,但具体的差异和挑战与这里概述的每种不同类型的未知有关。
数据和材料的可用性
不适用。
参考文献
王志强,王志强,王志强,等。散弹枪宏基因组学的研究进展。生物工程学报。2017;35:833 - 844。
李建军,李建军,李建军,等。来自宏基因组的超过15万个基因组揭示了广泛未开发的人类微生物组多样性,这些基因组跨越年龄、地理和生活方式。Cell. 2019; 176:649-62 [j]。
李江,贾H, Cai X,钟H,冯Q, Sunagawa年代,et al。人类肠道微生物组中参考基因的综合目录。生物工程学报,2014;32:834-41。
Lagier J-C, armogom F, Million M, Hugon P, Pagnier I, Robert C,等。微生物培养组学:人类肠道微生物组研究的范式转变。中华临床微生物学杂志,2012;18(6):1185 - 993。
Rinke C, Schwientek P, Sczyrba A, Ivanova NN, Anderson IJ,程建峰,等。微生物暗物质的系统发育和编码潜力。大自然。2013;499:431-7。
Marcy Y, Ouverney C, Bik EM, Lösekann T, Ivanova N, Martin HG,等。通过对人类口腔中罕见和未培养的TM7微生物的单细胞遗传分析,解剖生物学“暗物质”。科学通报,2007;44(4):389 - 394。
Murat。微生物暗物质:微生物生态学的鲻鱼微生物暗物质:微生物生态学的鲻鱼http://merenlab.org/2017/06/22/microbial-dark-matter/。2019年5月13日查阅。
邹勇,薛伟,罗刚,邓忠,秦鹏,郭睿,等。人类肠道细菌1520个参考基因组的功能微生物组分析。生物工程学报。2019;37:179-85。
Almeida A, Mitchell AL, Boland M, Forster SC, Gloor GB, Tarkowska A,等。人类肠道微生物群的新基因组蓝图。大自然。2019;https://doi.org/10.1038/s41586-019-0965-1。
Nayfach S, Shi ZJ, Seshadri R, Pollard KS, Kyrpides NC。来自未培养的全球人类肠道微生物组基因组的新见解。大自然。2019;https://doi.org/10.1038/s41586-019-1058-x。
Parks DH, Rinke C, Chuvochina M, Chaumeil P-A, Woodcroft BJ, Evans PN,等。近8000个宏基因组组装基因组的恢复大大扩展了生命之树。微生物学报,2017;2:1533-42。
Thomas AM, Manghi P, Asnicar F, Pasolli E, Armanini F, Zolfo M,等。结直肠癌数据集的宏基因组分析确定了跨队列微生物诊断特征和与胆碱降解的联系。中华医学杂志,2019;25:667-78。
Brown CT, Hug LA, Thomas BC, Sharon I, Castelle CJ, Singh A,等。不寻常的生物学跨越一组超过15%的领域细菌。大自然。2015;523:208-11。
李建军,李建军,李建军,李建军,李建军,等。探索微生物暗物质以解决真核生物的深层古细菌祖先。[j] .中国生物医学工程学报,2015;37(2):444 - 444。
致谢
我们要感谢莫伦佐·佐尔福和阿德里安·泰特就本评论的主题进行了深刻的讨论。
资金
这项工作得到了欧洲H2020计划(ONCOBIOME-825410项目)、欧洲研究理事会(MetaPG-716575 ERC-STG项目)、MIUR(“Ricerca的未来”RBFR13EWWI_001)、欧洲H2020计划(MASTER-818368项目)和利奥制药研究奖(LEO Pharma Research award)的支持
作者信息
作者及单位
贡献
AMT和NS构思了评注的内容并撰写了正文。两位作者都阅读并认可了最终版本。
相应的作者
道德声明
相互竞争的利益
作者宣称他们没有竞争利益。
额外的信息
出版商的注意
伟德体育在线施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上不受限制地使用、分发和复制,前提是您要适当地注明原作者和来源,提供到知识共享许可协议的链接,并注明是否进行了修改。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有说明。
关于本文
引用本文
Thomas, a.m., Segata, N.微生物组研究中未知的多个层次。BMC医学杂志17, 48(2019)。https://doi.org/10.1186/s12915-019-0667-z
发表:
DOI:https://doi.org/10.1186/s12915-019-0667-z