摘要
背景
在哺乳动物配子中建立的表观遗传修饰在早期发育期间大部分被重新编程,然而,部分由胚胎继承以支持其发育。在这项研究中,我们检查了CpG岛(CGI)序列,以预测小鼠囊胚CGI是否继承了来自母体基因组的卵母细胞来源的DNA甲基化。递归神经网络(rnn),包括基于门控递归单元(gru)的递归神经网络,最近被用于分类和回归分析中的变长输入。这种策略的一个优点是rnn能够通过学习模型参数自动学习嵌入在输入中的潜在特征。然而,现有的用于预测卵母细胞来源的DNA甲基化遗传的CGI数据集不够大,无法训练神经网络。
结果
我们提出了一个基于gru的模型,称为CMIC (CGI甲基化继承分类器),通过将CGI序列转换为变长来增强它k-mers,其中长度k是从范围中随机选择的\ (k_{\分钟}\)来\ (k_{马克斯}\ \),N时间,然后作为神经网络的输入。N在默认设置中设置为1000。此外,我们提出了一种新的嵌入向量发生器k-mers叫做splitDNA2vec。该过程的随机性高于之前的工作dna2vec。
结论
我们发现CMIC可以以高F-measure(0.93)预测囊胚母基因组cgi上卵母细胞来源的DNA甲基化的遗传。我们还表明,f测度可以通过增加参数来改善N,即变长序列的个数k-从单个CGI序列派生的mers。这意味着通过将DNA序列转换为输入数据来增加输入数据的有效性N可变长度的序列k即。这种方法可以应用于不同的DNA序列分类和回归分析,特别是涉及少量数据的分析。
背景
DNA甲基化是一种表观遗传修饰,主要发生在CpG位点,并调节基因表达。因此,DNA甲基化显著影响各种哺乳动物的生物学过程,包括胚胎发育、基因组印迹、x染色体失活、转座因子抑制、衰老和致癌。全局表观遗传重编程是一种与发生在原始生殖细胞和早期胚胎中的表观遗传修饰重塑相关的擦除过程,确保每一代开始一个新的发育周期[1].早期胚胎全局重编程的一个显著例外是基因组印迹,在这种情况下,亲代配子中建立的差异DNA甲基化被传递到合子,并调节亲代起源特异性基因的表达[2].然而,最近的研究表明,环境因素诱导的异位甲基化也可能逃脱重编程,在后代中引起疾病易感性[3.].
CpG岛(CpG islands, CGIs)是基因组中CpG密集的区域,通常与基因启动子重叠[4].在小鼠基因组中已鉴定出约16,000-22,000个cgi,其中大多数在发育过程中保持未甲基化。另外,某些cgi在特定组织中甲基化或在特定条件下易于甲基化,如细胞培养和癌变。cgi也可以在基因组印迹和x染色体失活时甲基化[4].然而,在所有情况下,无论甲基化状态如何,CGI中的大多数CpG位点表现相似,其甲基化强烈下调相关基因[4].
我们之前研究了在cgi中引入的DNA甲基化,包括印迹控制区,是如何从卵母细胞传递到胚胎的[5,6,7].在小鼠卵母细胞中,大约1100个cgi通过转录偶联从头甲基化而甲基化[8].受精后,通过重编程去除CGI甲基化;然而,有一部分(高达15%)的cgi,包括母系甲基化印迹控制区,可在囊胚内保持甲基化[8].这种CGI甲基化遗传部分依赖于序列特异性的甲基化dna结合蛋白Zfp57,它招募其他对甲基化维持必不可少的蛋白质[9].因此,我们专注于确定CGI序列是否包含足够的信息来预测通过受精和早期发育的甲基化遗传。先前的研究报道了基于序列和其他特征预测各种细胞类型和组织的CGI甲基化状态或甲基化倾向的方法[10,11,12,13,14,15,16].值得注意的是,郑等.报道了一种基于支持向量机的模型,该模型涉及组蛋白修饰数据(甲基化和乙酰化),具有高特异性和准确性[15].然而,由于样本稀缺和伦理问题(特别是涉及人类时),很难从卵母细胞和囊胚中获得高质量的组蛋白修饰信息。
基于递归神经网络(RNN)的方法在基于序列的甲基化预测方面比上述方法有优势。首先,神经网络可以在没有先验生物学知识的情况下自动学习输入数据的潜在特征表示。相比之下,以前的研究使用决策树、支持向量机或逻辑回归,这些都需要先前设计的特征向量作为输入。第二,rnn以可变长度的数据作为输入,包括不同大小的cgi的序列。基于rnn的分类器已广泛应用于生物信息学,例如在KEGRU中,用于预测转录因子结合位点[17], DNA序列功能[18],以及染色质可及性[19].KEGRU将DNA序列分为k-mer序列,具有指定的长度和步幅,并考虑每个k-mer作为单词,使用word2vec算法将它们转换为预先训练好的嵌入向量[20.].然后构建双向门控循环单元(BiGRU)神经网络进行分类。虽然KEGRU适用于我们的实验目标,但仍然存在一个挑战。从卵母细胞中甲基化的cgi库中,我们选择了那些可以使用单核苷酸多态性(SNP)信息在囊胚母基因组中以甲基化或非甲基化形式进行评估的cgi。然而,这个过程只产生了272个cgi,这不足以训练rnn。
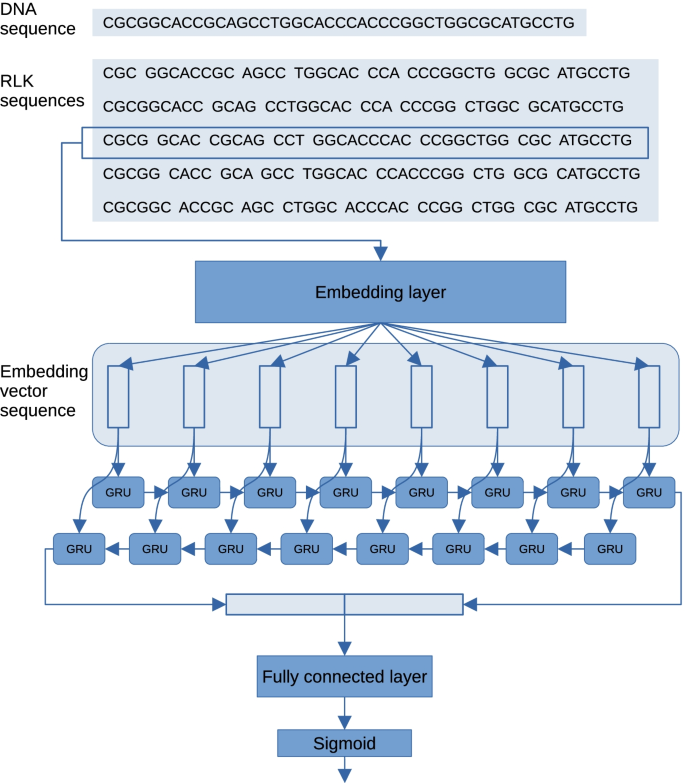
因此,我们设计了CMIC (CGI甲基化继承分类器)作为一种将CGI序列转换为基因的新方法k- m序列。一个CGI序列被划分为可变长度k-mers这样的邻居k-mers不重叠,重复N次了。的变长k-mer序列作为类标签共享CGI的甲基化状态。因此,我们增加了给GRU网络的输入数据量。此外,可变长度k使用word2vec算法从整个信息性cgi中提取-mer序列来创建预训练的嵌入向量,这种新的嵌入分配方法称为splitDNA2vec。将嵌入向量的序列传递给BiGRU层来预测输入序列的DNA甲基化状态,我们将其命名为CGI甲基化分类方法CMIC。
我们表明,CMIC达到了0.93的高f测量值。我们还讨论了如何的价值N影响CMIC的预测结果。总的来说,这项工作演示了如何将一个CGI序列转换为多个可变长度的序列k-mer序列可有效预测其DNA甲基化状态。
方法
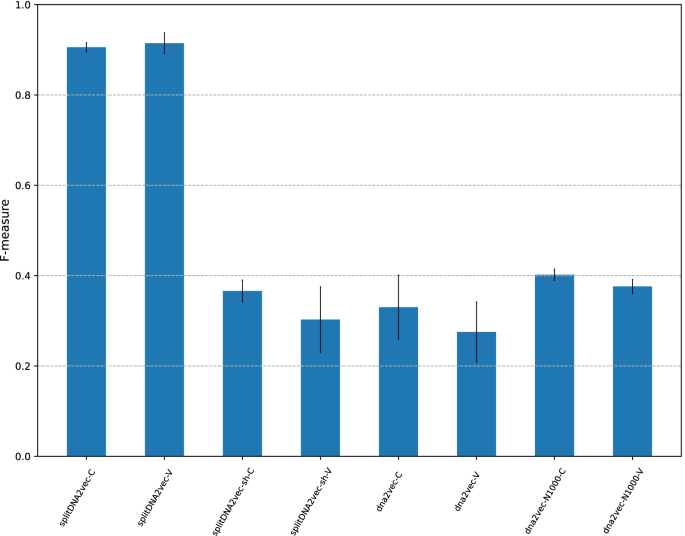
f -测度与不同方案的嵌入向量见表3..x轴表示要比较的8种方法:splitDNA2vec-C、splitDNA2vec-V、splitDNA2vec-sh-C、splitDNA2vec-sh-V、dna2vec-V、dna2vec-N1000-C和dna2vec-N1000-V。y轴表示测试数据集上的f测度
我们所提出的CMIC方法概要如下(图1)。1).当CMIC提取DNA序列时年代输入是2N变长k-mer序列通过分裂生成年代它的逆补变成k-mers哪里有这样的长度k是根据间隔上的离散均匀分布随机确定的。每一个变长k-mer序列作为CMIC的BiGRU神经网络的输入。的k-指定变量长度的参数k-mer序列转换为嵌入层中相应的嵌入向量。接下来,将得到的嵌入向量序列交给BiGRU层。输出到输入的变长k的概率解释为年代unmethylated。
生成随机长度的序列k-mers来自DNA序列
在CMIC的第一步中,输入DNA序列年代是否拆分为非重叠k-聚合物,其长度根据在最小到最大长度区间上的离散均匀分布随机确定,\ (k_{\分钟}\)而且\ (k_{马克斯}\ \),分别。重复这个步骤N各时段年代还有它的逆补。因此,2N变长k-mer序列由相同的序列生成年代序列。这些序列被称为同源序列,因为源DNA序列是相同的。这些变长k-mer序列作为CMIC的BiGRU神经网络的输入。随着输入实例数量的增加,此过程是一种数据增强方法(参见[21])。
此外,这种多重同源变长k-mer序列是原始DNA序列的另一种视图。因此,多重同源变长k-mer序列有望使训练后的神经网络更加健壮。
神经网络架构
本文介绍了CMIC的BiGRU神经网络的设计。如前一节所述,输入层的长度是可变的k-mer序列作为输入。网络的第二层是嵌入层,其中每个k将给定序列的-mer转换为相应的实数向量D.我们用嵌入向量生成方法splitDNA2vec生成的嵌入向量初始化层的嵌入向量,splitDNA2vec将在后续小节中描述。生成的嵌入向量序列被传递到BiGRU层,在那里它们被向前和向后处理。GRU在每个方向上的每个时间都持有一个由a表示的隐藏状态H维向量。向前和向后方向的最后一个隐藏状态被连接到单个向量中。这个向量被输入到一个全连接层中,然后是一个sigmoid激活函数。函数的输出是该网络的总输出,并被解释为输入未甲基化的概率。
训练数据集是一对可变长度的数据集k- m序列\ (x_i \)以及它的甲基化状态\(y_i \in \{0,1\}\)为\(i = 1, \ldots, M\),在那里\(y_i = 1\)(0,应答)表示未甲基化(甲基化,应答)。我们用CMIC神经网络的参数表示\ ({\ mathbf {w}} \).让\({\mathbf {y}} = (y_1, \ldots, y_M)\)而且\ \(帽子{{\ mathbf {y}}} =({\帽子{y}} _1, \ ldots{\帽子{y}} _M) \)是观察到的(真实的)和预测的甲基化状态\(x_1, \ldots, x_M\).在训练模型的过程中,模型参数\ ({\ mathbf {w}} \)通过最小化损失函数来优化训练数据集
在哪里\α(\ \)是权值衰减超参数,\(\Vert w\Vert _2\)是\ (l2 \)规范,\(E(\hat{{\mathbf {y}}}, {\mathbf {y}})\)交叉熵函数定义为
除了向量的嵌入维数D, GRU隐向量维数H,权值衰减率\α(\ \)时,学习过程中使用了CMIC的BiGRU方面的几个超参数,即epoch,e,学习率,r、批量大小、b,辍学率,d.BiGRU层采用dropout技术[22].它们的默认值在表中给出1.我们通过自适应矩估计(Adam)来训练网络[23]的随机梯度下降优化算法。
splitDNA2vec:随机长度的嵌入向量生成器k即
在这里,我们制定了新的嵌入向量创建方法,称为splitDNA2vec.用该方法的输出初始化CMIC嵌入层中的嵌入向量。首先,变长k-mer序列的生成过程与CMIC with中描述的第一步相同\ \ (N = 1000)从所有可用的cgi,这相当于训练和测试数据。生成的变长k-mer序列用连续词袋模型(continuous bag-of-words, CBOW)输入word2vec算法。word2vec算法的参数配置如下。上下文窗口大小设置为10。一个单词的最小计数,即。,k- m,\(c_{\min} = 1\).也就是说,所有发生的k-mer被计数。嵌入向量的维数设置为\ (D = 20 \).
生成2的过程N变长kDNA序列中的-mer序列灵感来自dna2vec [24]在这种方法中,给定的DNA序列被反复分割成重叠的部分k-mers,从跨步滑动窗口中提取,其长度根据离散均匀分布随机选择。dna2vec和我们的splitDNA2vec方法之间的主要区别在于我们的可变长度k-mer序列比dna2vec序列具有更高的随机性,这是由于提取序列的长度和起始位置k-mer在我们的方法中都是随机的,而只有的长度k-mers在dna2vec中随机化。此外,splitDNA2vec生成N可变长度的序列k-mers来自同一DNA序列,尽管dna2vec只产生一个可变长度的序列k-mers的DNA序列。
代替splitDNA2vec,我们还评估了用dna2vec算法输出初始化嵌入层的情况。此外,我们考虑了splitDNA2vec的洗牌版本,由splitDNA2vec-sh表示,其中的映射k-mers到splitDNA2vec生成的嵌入向量随机洗牌。最后,我们研究了这些嵌入向量初始化方法对CMIC性能的影响。
门控循环装置
在本节中,我们将解释用于CMIC的BiGRU神经网络。“GRU”是赵处长制定的et al。[25],类似于长短期记忆[26],但更简单,因为它不使用输出门。我们将在结果部分讨论CMIC变体与标准RNN和LSTM架构的性能。
我们在这里解释一下GRU的架构。复位门将时间步长的输入作为输入t,\ ({\ mathbf {x}} ^ t \),和时间步长的输出(隐藏)向量\ (t - 1 \),\ ({\ mathbf {h}} ^ {t - 1} \).这个门输出一个复位门矢量,r \ ({\ mathbf {}} _t \),表示为
在哪里\ ({\ mathbf {W}} _r \)而且\ ({\ mathbf{你}}_ {r} \)的权重矩阵\ ({\ mathbf {x}} ^ t \)而且\ ({\ mathbf {h}} ^ {t - 1} \)分别\ ({\ mathbf {b}} _r \)是一种偏见,和σ\ (\ \)是一个s型函数。类似地,更新门输出一个更新门向量,\ ({\ mathbf {z}} _t \),定义为
\ ({\ mathbf {W}} _z \)而且\ ({\ mathbf{你}}_ {z} \)的权重矩阵\ ({\ mathbf {x}} ^ t \)而且\ ({\ mathbf {h}} ^ {t - 1} \),以及\ ({\ mathbf {b}} _z \)是一种偏见。
使用r \ ({\ mathbf {}} ^ t \)作为监管者,GRU产生了\ \(波浪号{{\ mathbf {h}}} ^ t \)时间步上的候选激活向量t,计算方法为
在哪里\ ({\ mathbf {W}} \)而且\ ({\ mathbf{你}}\)都是权重矩阵,\ ({\ mathbf {b}} _h \)是一种偏见,和\ \ odot \ ()是阿达玛乘积。请注意,\(双曲正切\)双曲正切函数,值域是什么\ ([1] \).进一步,复位门矢量r \ ({\ mathbf {}} ^ t \)用作表示输出向量,\ ({\ mathbf {h}} ^ {t - 1} \),应该忘记使候选激活向量,\ \(波浪号{{\ mathbf {h}}} ^ t \).
时间步长的输出向量t,\ ({\ mathbf {h}} ^ t \)的仿射组合\ ({\ mathbf {h}} ^ {t - 1} \)而且\ \(波浪号{{\ mathbf {h}}} ^ {t} \)比例为\ ({\ mathbf {z}} ^ t: ({\ mathbf {1}} - {\ mathbf {z}} ^ t) \);也就是说,
在哪里\ ({\ mathbf {1}} \)向量中是否都是相同维数的向量\ ({\ mathbf {z}} ^ t \).
为了增强CMIC的可预测性,我们使用了BiGRU模型,这是BiRNN模型的GRU版本[27].即它有正向GRU网络和向后GRU网络,其中输入分别在正向和向后方向上进行处理,我们称之为BiGRU层。BiGRU层的输出是通过将两个方向的最后一个隐藏状态向量合并成适当的模式来表示的,比如拼接、求和、平均和乘法。本工作采用串联。
材料
我们使用小鼠发育完全的卵母细胞(FGOs)全基因组亚硫酸氢盐测序(WGBS)获得的数据[6,7,28,29和囊胚(GSE174311)。FGOs来自小鼠株系C57BL/6J (kyyudo Co, Japan),囊胚来自于C57BL/6J卵母细胞与JF1/Ms小鼠(遗传资源中心,National Institute of Genetics, Japan)精子体外受精。在实验进行时,这些小鼠至少有10周大。cgi的位置基于小鼠基因组的mm10组装,并从UCSC基因组注释数据库获得[30.].使用Bismark将FGO和囊胚的WGBS读数映射到小鼠基因组[31].
对于囊胚的等位基因特异性甲基化分析,reads被映射到基于JF1发表的SNP数据生成的n -mask基因组序列[32].利用SNPsplit方法选择等位基因[33].
cgi的甲基化状态
对于每个CGI,我们计算CGI中覆盖CpG位点的WGBS读取数以及表示为甲基化的读取数。我们将一个CGI的DNA甲基化比率定义为甲基化对整个计数的计数。如果CGI的整个读计数小于阈值T,它不用于任何进一步的分析。然后,标记一个CGI米(甲基化)如果其甲基化率大于或等于阈值\(β_ {M} \ \),而它是标签U(未甲基化)如果其甲基化率低于阈值,\(β_{你}\ \).表格2为FGO和囊胚母细胞基因组参数的设置。在FGOs中甲基化的cgi的小鼠囊胚母基因组中甲基化比例的分布在附加文件中给出1:图S1。
我们的目标数据也是甲基化状态米或U,属于类的cgi米在FGOs中,在囊胚的母体基因组中。这些类分别表示为M2M(DNA甲基化遗传)和M2U(DNA甲基化损失)。的数量M2Mcgi是182,而M2Ucgi是90。其中,有150人M2Mcgi和60M2U长度不超过500 bp的cgi。这些cgi适用于学习模型,因为较长的序列需要更多的时间来训练模型。我们在三层交叉验证中使用它们。
性能指标
CMIC对可变长度输入的输出k-mer序列是输入未甲基化的概率。如果概率大于0.5,我们将预测的类别标签确定为“未甲基化”,否则为“甲基化”。
作为性能指标,我们使用平衡精度、f测量、MCC(马修斯相关系数)和AUC。我们在3倍分层交叉验证中计算这些性能指标,并显示了标准误差的平均值。
结果
求出最佳的一对变量长度的上界和下界k即
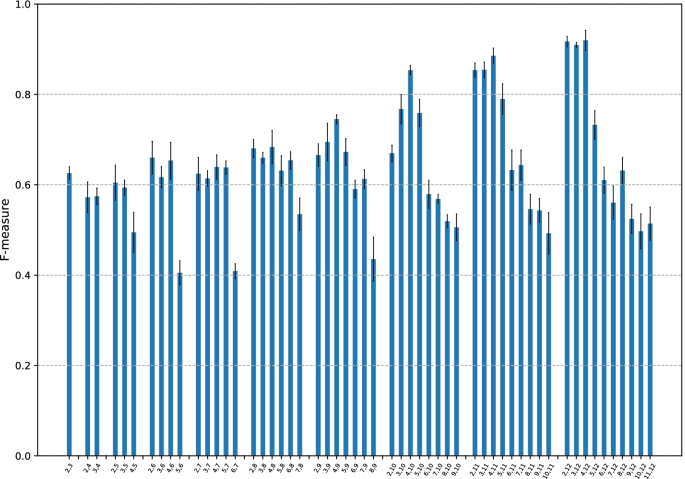
回想一下,\ (k_{\分钟}\)而且\ (k_{马克斯}\ \)变量长度的下界和上界是什么k-一个给定的DNA序列被分裂成的mers。我们在这里找到最好的一双\ (k_{\分钟}\)而且\ (k_{马克斯}\ \)使用其他默认参数。我们设置了对的搜索空间\ (k_{\分钟}\)而且\ (k_{马克斯}\ \)这样\(k_{\min} = 2, \ldots, 11\)而且\(k_{\max} = 3, \ldots, 12\)这样\(k_{\max} - k_{\min} \ge 1\).
基于3倍交叉验证的f -测度总结在图中的柱状图中。2酒吧按什么分类\ (k_{马克斯}\ \).结果表明:\ (k_{马克斯}\ \)增加,所以f测量值与一个小\ (k_{\分钟}\)取值约为2、3和4。假设splitDNA2vec指定了这样的短和长的嵌入向量k-mers同时,这些嵌入向量可能协同作用来表征CGI序列。其中,\((k_{\min}, k_{\max}) = (4,12)\)f值最高,为0.93。在随后的分析中,这对值被用作默认值。平衡精度、MCC和AUC图与图中相似。2(附加文件1:无花果。S2 S3 S4)。
最后,在……的情况下\ (k_{马克斯}\ \)的值大于12,考虑到CMIC的f值最高\(k_{\max} = 12\),我们运行splitDNA2vec\(k_{\max} = 13\);然而,这个过程非常耗时,特别是在执行word2vec时;因此,我们没有深入研究这个案例。
splitDNA2vec的评估
接下来,我们分析了嵌入层中使用的嵌入向量对CGI甲基化状态预测的影响程度。为此,我们考虑了三种不同的生成嵌入向量的方法,如表所示3..第一个方法是我们的默认方案splitDNA2vec,第二个方法是splitDNA2vec-sh,它随机打乱的映射k-mers到splitDNA2vec制作的嵌入向量,最终的方法为dna2vec,如上所述。回想一下,dna2vec生成了一个可变长度的序列k-mers的DNA序列。这里我们扩展了dna2vec,它可以生成1000个可变长度的序列k-mers来自同一序列。这个扩展名为dna2vec-N1000。
这个嵌入向量的赋值k-mers是一个无监督的学习过程。因此,我们的数据集(包括训练、测试和长CGI数据集)的DNA序列在没有它们的类标签的情况下被应用。注意,每个方法都会生成\ \ (N = 1000)变长k-mer序列从输入的CGI序列,保存原始的dna2vec。
从CMIC神经网络部分的训练过程中嵌入层中的嵌入向量是否更新的角度出发,我们进一步从上述四种方法中分别推导出更具体的嵌入向量方法,如[34].更具体地说,这些向量被视为网络的可变权值(V)或常量向量(C)。对于splitDNA2vec,我们将这两个版本分别表示为splitDNA2vec-V和splitDNA2vec,其余版本也是如此。因此,我们将得到的8个方法与其余参数作为默认值进行比较。
上图所示为8种不同方案在测试数据集上的f测度。3..这个结果有一些有趣的含义。
首先,发现最好的方法是splitDNA2vec-V, F-measure为0.914,其次是splitdna2vec, F-measure为0.906。这意味着即使嵌入层被冻结,由于splitDNA2vec方法构建的嵌入向量的有效性,CMIC也能达到相同的性能水平。此外,splitDNA2vec方法在CMIC神经网络端到端训练过程中不需要进一步更新splitDNA2vec方法给出的初始嵌入向量。因此,我们在进一步分析中使用splitdna2vec作为默认设置。
其次,我们可以看到dna2vec-N1000-C和dna2vec-N1000-V优于dna2vec和dna2vec-V,突出了生成多个变长序列的好处k-mers来自相同的CGI序列。dna2vec-N1000-C得到的最高f值为0.40,显著低于splitDNA2vec的0.91。考虑到splitDNA2vec和dna2vec-N1000之间的主要区别是相邻提取之间是否存在重叠k-mers,此结果暗示重叠邻接k-mer的信息比不重叠的要少。
第三,在F-measure方面,dna2vec和dna2vec-V的结果分别比随机洗牌的splitDNA2vec、splitDNA2vec-sh- c和splitDNA2vec-sh- v的结果更差。word2vec的选项“window”,指定连续的长度k-mers用作训练word2vec模型的实例,在所有8种嵌入向量的方法中都设置为10。dna2vec的一个可能的缺点是大小为10的滑动窗口所覆盖的基因组区域由于相邻区域之间的重叠而比splitDNA2vec要短k即。
平衡精度、MCC、AUC曲线图如图所示。3.(详见附加文件1:无花果。S5, S6和S7)。
变长数k-mer序列强烈影响DNA甲基化遗传预测
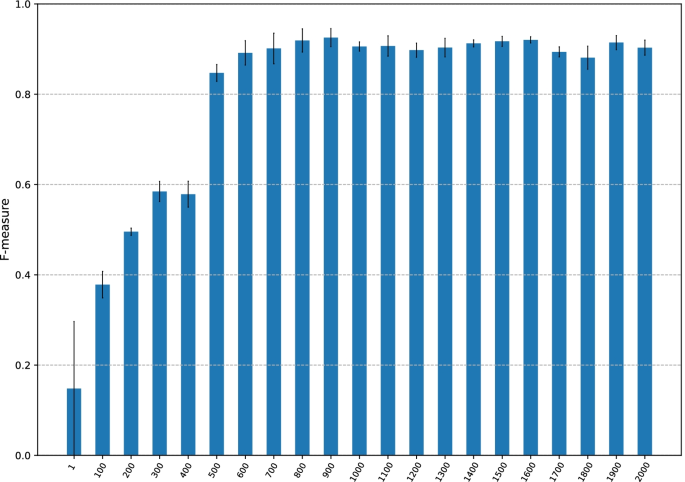
类的总数M2Ucgi为60,可能不足以充分训练CMIC。然后我们增加N,为变长序列的个数k-由输入CGI序列生成的mers,直接作为网络的输入。结果如图所示。4.
作为N增加到900时,F-measure逐渐改善,在F-measure达到0.93\ \ (N = 900).随后,曲线趋于平稳。这个结果清楚地表明了多个变长序列k-mers应该用来训练模型。
平衡精度、MCC、AUC曲线图如图所示。4(详见附加文件1:无花果。S8, S9和S10)。
长cgi训练模型的可预测性
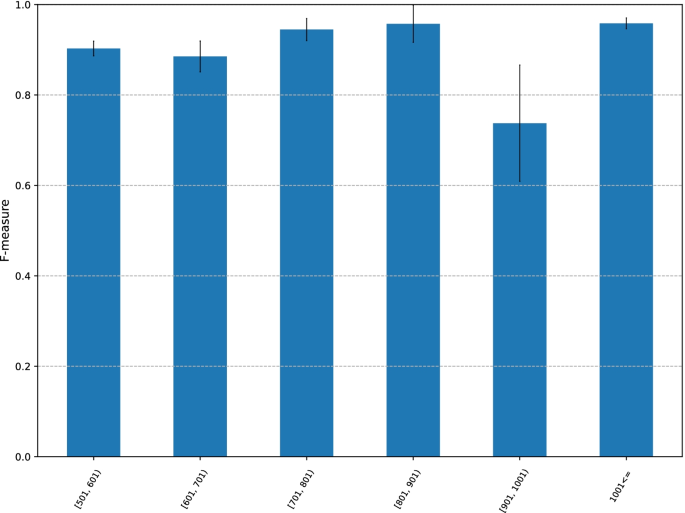
在这项工作中,我们将训练数据集中的cgi的长度限制在500 bp,因为较长的序列会导致CMIC神经网络的展开形式更深,使学习过程更加困难。如果决定CGI甲基化遗传的关键特征编码在一个相对较短的区域内,使用短CGI训练的网络可以学习该特征,并以相同水平的可预测性准确预测长度超过500 bp的CGI甲基化遗传。然后,我们检查了训练后的CMIC的F-measure,用短序列代替长序列。
长度分布较长M2M而且M2Ucgi在表中给出4.这些序列预测的f测度如图所示。5.
CGI长度间隔[501,600]、[601,700]、[701,800]、[701,800]、[801,900]、1000及以上的平均f -测度分别为0.90、0.89、0.94、0.96、0.74和0.96。回想一下,到目前为止,使用默认参数值的最佳f测量值是0.93。出乎意料的是,有三个区间的f测量值均高于0.93。这一结果表明,甲基化遗传的一些基因组特征嵌入在500 bp或更短的区域内,并且被许多长cgi共享。
平衡精度、MCC、AUC曲线图如图所示。5(详见附加文件1:无花果。S11, S12和S13)。
不同循环神经网络架构的性能
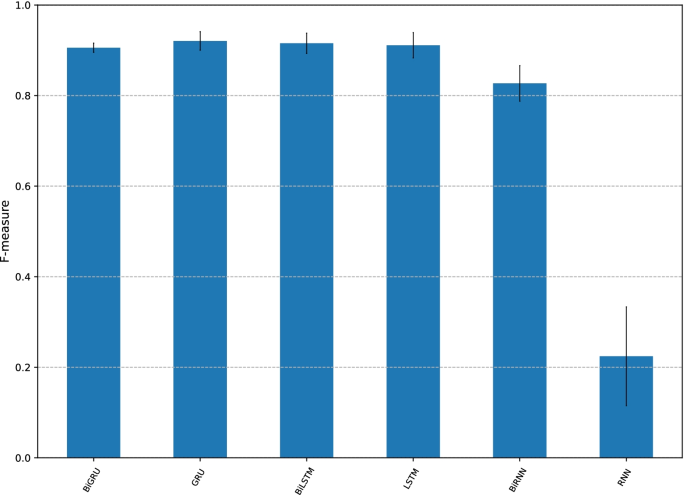
虽然CMIC采用的循环单元架构默认为BiGRU,但也有其他知名的架构,包括标准的RNN单元[35]和LSTM单元[26].然后我们考虑了CMIC的变体,其中BiGRU网络被BiRNN网络和双向LSTM (BiLSTM)网络所取代。此外,我们考虑了它们的单向版本,用GRU、RNN和LSTM表示。
它们的f值如图所示。6.首先,BiGRU、GRU、BiLSTM和LSTM具有相似的f -测度,比BiRNN和RNN的f -测度高。因此,GRU和LSTM架构中的双向性是不必要的。这意味着基因组特征的DNA甲基化状态可以在一个方向上学习。同时,RNN表现出较差的性能,但双向版本BiRNN在一定程度上弥补了RNN的不足。
平衡精度、MCC、AUC曲线图如图所示。6(详见附加文件1:无花果。S14, S15和S16)。
KEGRU的性能
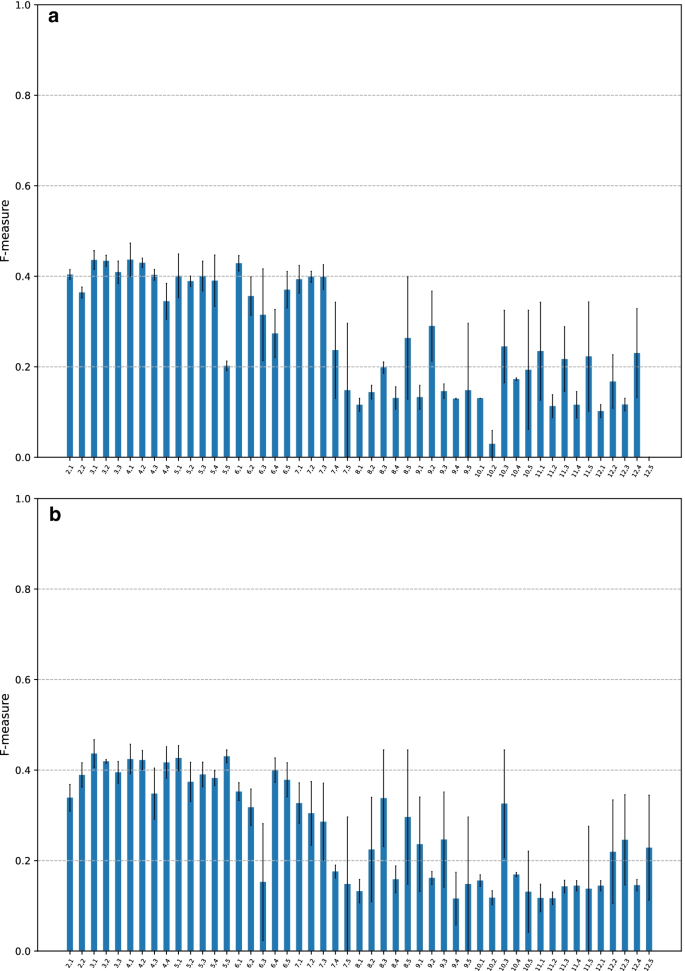
在众多基于神经网络的变长DNA序列分类器中,与CMIC最相似的模型是KEGRU [17]其中有一个固定的k而一个固定的步幅,一个单一的序列k-mers由DNA序列生成。事实上,它们的结构本质上与dna2vec相同。这与CMIC不同,CMIC生成多个变长序列k-mers由splitDNA2vec执行。
然后,我们用表中所示的超参数值对KEGRU进行了全面的计算实验5.我们扩大了的范围k-mer长度从原来的长度,4,5,6,在[17)\(2, \ldots, 12\).我们还拓宽了跨步范围\(2, \ldots, 5\)来\(1, \ldots, 5\).最后给出了嵌入向量的维数,表示为D分别从50、100、150、200改为20和50。在[17],这些情况\(d = 50,100,150 \)对200名学生进行了比较,结果显示,这些学生的得分与\(d = 50,100 \)150。因此,我们采用了\ (D = 50 \)除了我们的默认值\ (D = 20 \).
无花果。7a, b为KEGRU的f -测度图\ (D = 20 \)和50个。一般来说,结果与左轴上的条形图相似,显示f -测度约为0.4,而右轴上的条形图显示约为0.2,其中一些条形图具有较大的标准误差。将这些结果与图中所示的结果进行比较。2说明了多序列生成的有效性k- splitDNA2vec的mers。
平衡精度、MCC、AUC曲线图如图所示。7(详见附加文件1:无花果。S17, S18, S19, S20, S21和S22)。
12-mers的嵌入载体特征
当我们生成一个变长序列时k-从CGI序列使用splitDNA2vec,最大长度设置为\(k_{\max} = 12\).然而,例如,关于染色质可及性的工作[19]采用6-mers, KEGRU采用\(k= 4,5 \),以及上文所述的[17].这些原因比较短k-mers的选择是为了避免由于长而过拟合k即。然而,k从训练CGI序列中提取的-mer可能永远不会出现在任何测试CGI序列中,包括反向补。因此,这样的长k-mer将成为训练模型的一个障碍。
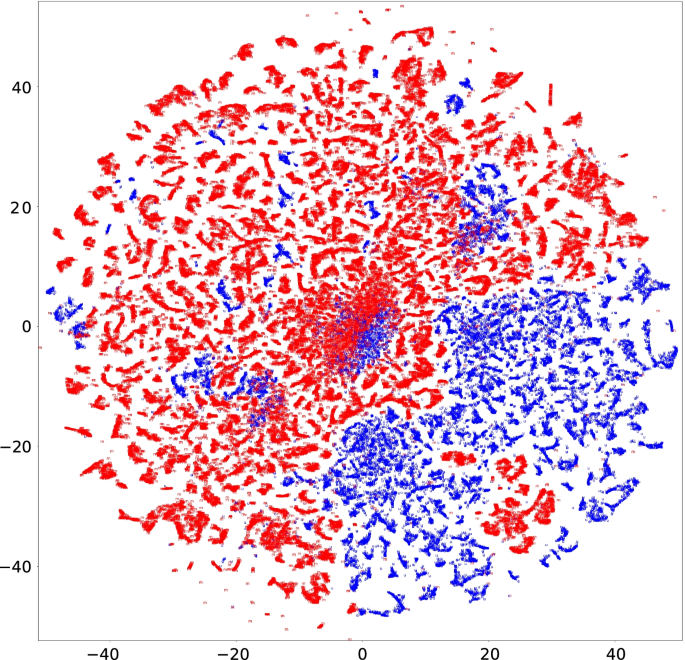
然而,提取k-mers被转化为预训练的DCMIC中的-维嵌入向量。就算两个人长了k-mers不同,但它们的子字符串相似或相同,即longk-mer可以被映射成彼此接近的嵌入向量。如果这个假设是在甲基化的cgi和/或非甲基化的cgi中实现的,那么长时间k-mers有助于提高CGI甲基化遗传的可预测性。
为了验证这一假设,我们应用了t-分布式随机邻居嵌入(t-SNE),这是一种基于数据点之间的距离将高维数据映射到低维空间的算法[36],到CMIC默认参数生成的12-mers。考虑到有太多的12元是由CGI序列生成的\ le 500 (\ \),我们删除了出现在甲基化和非甲基化CGI序列中的12-mers,因为它们不能区分CGI甲基化遗传。总共有107129名12-mers患者。由t-SNE生成的默认困惑度为30的图如图所示。8.大多数未甲基化和甲基化的12-mer根据其甲基化遗传分别分布在不同的区域。困惑度为10和50的图显示了类似的观点(附加文件1:无花果。S23和S24)。这意味着存在许多具有甲基化遗传特征的嵌入载体的12-mers。
预测人淋巴细胞中cgi的甲基化状态
除了我们最初的DNA甲基化遗传预测研究目标外,我们还将CMIC应用于预测人淋巴细胞中cgi的DNA甲基化状态[37用在博克语中et al。[12],以评价CMIC的通用性。该数据集包含21号染色体上29个甲基化和103个未甲基化的cgi。假设这个数据集小于包含60的数据集M2U和150年M2Mcgi,这个研究问题对CMIC来说更具挑战性。
由于测试了各种属性集,我们选择了准确率最高的0.919进行分析。属性集是第1类(DNA序列属性和模式)和第2类(重复频率和分布)的组合。他们报告了在重复20次的10倍分层交叉验证中真阳性(TPs)、假阴性(FNs)、真阴性(TNs)和假阳性(FPs)的总数2在[12])。根据这些数据,我们计算了平衡精度、F-measure和MCC,如表所示6.
同样,我们从CMIC三次分层交叉验证测试数据集上的结果中统计TPs、FNs、TNs和FPs,并在表中给出性能指标6.可以看出,CMIC的平衡准确度和f值提高了14%,MCC提高了15%。
注意博克等.设计了1184个基于序列的特征向量,并使用支持向量机进行预测。同时,给出了该方法的关键向量,即变长嵌入向量k-mer和BiGRU层的隐藏向量,通过指定超参数自动学习。这一结果表明了CMIC的优越性。
结论
在本文中,我们讨论了是否有可能根据cgi的序列来预测胚泡母基因组中是否维持卵母细胞来源的甲基化,并提出了一种方法CMIC来进行预测。出现的一个关键问题是可用的cgi数量不足,无法训练神经网络。因此,我们设计了一种随机数据增强方法,将输入的单个DNA序列转换为提取的可变长度嵌入向量的多个序列k-mers的DNA序列。此外,这样的变长k从我们的CGI数据集中得到的-mers序列作为输入,生成的嵌入向量k即。这种新的DNA序列嵌入载体生成方法为splitDNA2vec。CMIC的嵌入层默认使用这些嵌入向量。总的来说,CMIC获取一个输入CGI序列,将其转换为多个变长k-mer序列,并进一步转换每个序列k-mer到指定的嵌入向量。BiGRU网络给出了输入的CGI遗传囊胚母基因组DNA甲基化的概率。
在本研究中,我们发现splitDNA2vec比dna2vec更适合甲基化遗传分类。进一步,我们证明了生成的大量变长k来自DNA序列的-mer序列在增加输入数据方面是有效的,因为DNA序列的分割为可变长度k-mer序列提供原始DNA序列的不同表示形式。
所提出的CMIC方法的设计不依赖于DNA甲基化。因此,该方法应适用于其他DNA序列分类问题,包括染色质可达性预测[19].
数据和材料的可用性
CMIC可通过GitHub访问https://github.com/maruyama-lab-design/CMIC根据GNU通用公共许可证v3。FGO(为本研究重新处理)和囊胚(在本研究期间生成)的WGBS数据集可在NCBI的基因表达Omnibus和DDBJ序列读取档案中获得,登录号为GSE112320, GSE174311, DRA000570, DRA005849和DRA011758。
缩写
- CGI:
-
CpG岛
- 格勒乌:
-
门控循环装置
- RNN:
-
循环神经网络
- LSTM:
-
长短期记忆
- FGO:
-
成熟卵母细胞
- WGBS:
-
亚硫酸氢盐全基因组测序
参考文献
Seisenberger S, Peat JR, Reik W.早期胚胎和原始生殖细胞中DNA甲基化重编程之间的概念联系。中国生物医学工程学报。2013;25:1 - 8。
Tucci V, Isles AR, Kelsey G, Ferguson-Smith AC, Tucci V, Bartolomei MS, Benvenisty N, Bourc 'his D, Charalambous M, Dulac C, Feil R, Glaser J, Huelsmann L, John RM, McNamara GI, Moorwood K, Muscatelli F, Sasaki H, Strassmann BI, Vincenz C, Wilkins J, Isles AR, Kelsey G, Ferguson-Smith AC.哺乳动物的基因组印迹和生理过程。细胞。2019;176:952 - 65。
拉卡尔I,文图拉R.表观遗传:概念,机制和观点。11 .《分子神经科学》2018
迪顿,鸟,A. CpG岛和转录调控。Gene Dev. 2011;25:10 - 22。
Hirasawa R, Chiba H, Kaneda M, Tajima S, Li E, Jaenisch R, Sasaki H.在着床前发育过程中,母系和合子的Dnmt1对于维持DNA甲基化印迹是必要和充分的。基因开发,2008;22:1607-16。
Maenohara S, Unoki M, Toh H, Ohishi H, Sharif J, Koseki H, Sasaki H. UHRF1在卵母细胞新生DNA甲基化和着床前胚胎维持甲基化中的作用。PLoS Genet, 2017;13: e1007042。
Au Yeung WK, Brind Amour J, Hatano Y, Yamagata K, Feil R, Lorincz MC, Tachibana M, Shinkai Y, Sasaki H.卵母细胞中的组蛋白H3K9甲基转移酶G9a对着床前发育至关重要,但对CG甲基化保护是不必要的。2019; 27:82 - 93。
Smallwood SA, Tomizawa S- i, Krueger F, Ruf N, Carli N, segonts - pichon A, Sato S, Hata K, Andrews SR, Kelsey G.卵母细胞和着泡前胚胎的动态CpG岛甲基化景观。Nat Genet. 2011; 43:811-4。
Strogantsev R, Krueger F, Yamazawa K, Shi H, Gould P, Goldman-Roberts M, McEwen K, Sun B, Pedersen R, Ferguson-Smith AC. ZFP57在印迹和非印迹单等位基因表达表观遗传调控中的等位基因特异性结合。中国生物医学工程学报。2015;16:12 2。
Feltus FA, Lee EK, Costello JF, Plass C, Vertino PM。预测异常CpG岛甲基化。美国国家科学研究院。2003; 100:12253-8。
Feltus FA, Lee EK, Costello JF, Plass C, Vertino PM。与异常CpG岛甲基化相关的DNA基序。基因组学。2006;87:572-9。
Bock C, Paulsen M, Tierling S, Mikeska T, Lengauer T, Walter J.人淋巴细胞CpG岛甲基化与DNA序列、重复序列和预测DNA结构高度相关。PLoS Genet, 2006;2: e26。
方飞,范S,张X,张MQ。预测人脑中CpG岛的甲基化状态。生物信息学。2006;22:2204-9。
杨毅,孙伟文,金世峰。一种新的K- mer混合逻辑回归用于人类基因启动子CpG二核苷酸甲基化易感性建模。BMC bioinfo . 2012;13:S15。
郑辉,吴辉,李娟,姜世伟。CpGIMethPred:预测人类基因组中CpG岛甲基化状态的计算模型。BMC医学基因组学杂志2013;6(增刊1):S13。
雅素D,欧图HH。一种无偏预测模型来检测人类基因组中CpG岛的DNA甲基化倾向。《生物信息》,2021;16:179-96。
沈忠,鲍伟,黄德生。预测转录因子结合位点的递归神经网络。科学通报2018;8:1-10。
广东,谢晓丹q:一种用于量化DNA序列功能的卷积和递归混合深度神经网络。核酸研究,2016;44:e107。
闵晓霞,曾伟,陈宁,陈涛,蒋蓉。基于卷积长短期记忆网络的染色质可及性预测\ (k \)- m嵌入。生物信息学。2017;33:i92 - 101。
乔杜里GG,自然语言处理。科学通报2003;37:51-89。
陈松,杜布里班,李俊华。数据增强的群理论框架。J Mach Learn res 2020;21(245): 1-71。
Srivastava N, Hinton G, Krizhevsky A, Sutskever I, Salakhutdinov R. Dropout:一种防止神经网络过拟合的简单方法。J Mach Learn res 2014; 15:29 - 58。
金玛DP, Ba J. Adam。一种随机优化方法。预印arXiv: 1412.6980.2014.
Ng P. dna2vec:变长k-mers的一致向量表示。2017.
赵k, Van Merriënboer B, Bahdanau D, Bengio Y.关于神经机器翻译的性质:编码器-解码器方法。预印arXiv: 1409.1259.2014.
张志刚,张志刚。长短期记忆。神经计算,1997;9:1735-80。
舒斯特尔。双向循环神经网络。电子工程学报。1997;29(2):366 - 366。
Shirane K, Toh H, Kobayashi H, Miura F, Chiba H, Ito T, Kono T, Sasaki H小鼠卵母细胞甲基化在碱基分辨率下揭示了非cpg甲基化的全基因组积累和DNA甲基转移酶的作用。PLoS Genet, 2013;9: e1003439。
Kibe K, Shirane K, Ohishi H, Uemura S, Toh H, Sasaki H. DNMT3A PWWP结构域对小鼠体细胞和卵母细胞正常DNA甲基化景观至关重要。PLoS Genet, 2021;17: e1009570。
Karolchik D, Baertsch R, Diekhans M, Furey TS, Hinrichs A, Lu Y, Roskin KM, Schwartz M, Sugnet CW, Thomas DJ,等。UCSC基因组浏览器数据库。核酸决议2003;31:51-4。
Krueger F, Andrews SR. Bismark:用于亚硫酸氢盐序列应用的灵活对准器和甲基化调用器。生物信息学。2011;27:1571-2。
高田T, Ebata T,野口H, Keane TM, Adams DJ,成田T, Shin T,藤泽H, Toyoda A, Abe K,等。现存日本花式鼠的祖先为经典近交系的镶嵌基因组做出了贡献。基因组Res. 2013; 23:1329-38。
Krueger F, Andrews SR. SNPsplit:已知SNP基因型的基因组之间对齐的等位基因特异性分裂。F1000Research。2016; 5:1479。
沈忠,鲍伟,黄德生。预测转录因子结合位点的递归神经网络。科学通报2018;8:15270。
鲁默哈特德,辛顿GE,威廉姆斯RJ。通过错误传播学习内部表示。技术报告,加州大学圣地亚哥拉霍亚认知科学研究所,1985年。
范德玛滕,辛顿。用t-SNE可视化数据。J Mach Learn res 2008; 9:2579-605。
Yamada Y, Watanabe H, Miura F, Soejima H, Uchiyama M, Iwasaka T, Mukai T, Sakaki Y, Ito T.人类21q染色体CpG岛等位基因甲基化状态的综合分析。基因组学报,2004;14(2):247-66。https://doi.org/10.1101/gr.1351604.
确认
我们要感谢亮清水(九州大学)的技术援助。
资金
这项工作得到了KAKENHI Grant (JP18H05214)对H.S.和O.M.以及KAKENHI Grant (JP21H03544)对O.M.的支持
作者信息
作者及隶属关系
贡献
OM和YL制定了数学模型,并撰写了本文的初稿。YL编写了该方法第一版代码的大部分,并运行计算。OM和HN编写了该方法的修订版本,OM用该代码进行了计算实验。HT和WKAY对输入数据进行预处理。OM、YL、WKAY、HS进行数据分析。OM、WKAY和HS对项目进行了概念化和监督,并撰写了最终版本的手稿。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
所有的动物实验都是在九州大学的伦理指导方针下进行的,并且议定书得到了机构动物护理和使用委员会的批准。在这项研究中使用的所有小鼠都被二氧化碳窒息安乐死。本研究根据arrival指南进行报道。
相互竞争的利益
作者宣称他们之间没有利益冲突。
发表同意书
不适用。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1。图S1。
在FGOs中甲基化的cgi的小鼠囊胚母基因组中甲基化比例的分布。fgo的M类(甲基化)的下界设置为β米= 0.8如表2所示。图S2。不同对CMIC的平衡精度k最小值而且k马克斯.的搜索空间k最小值而且k马克斯被设定为k最小值= 2,…, 11和k马克斯= 3,…, 12和k马克斯−k最小值≥1。这些条形图是按k马克斯.图S3。MCC的CMIC与不同的对k最小值而且k马克斯.的搜索空间k最小值而且k马克斯被设定为k最小值2、…, 11和k马克斯3、……,12与k马克斯−k最小值≥1。这些条形图是按k马克斯.图S4。不同对CMIC的AUCk最小值而且k马克斯.的搜索空间k最小值而且k马克斯被设定为k最小值2、…, 11和k马克斯3、……,12与k马克斯−k最小值≥1。这些条形图是按k马克斯.图S5。用表3所示的不同嵌入向量方案平衡精度。x轴表示要比较的8种方法:splitDNA2vec-C、splitDNA2vec-V、splitDNA2vec-sh-C、splitDNA2vec-sh-V、dna2vec-V、dna2vec-N1000-C和dna2vec-N1000-V。y轴表示测试数据集上的f测度。图S6。采用表3所示的不同嵌入向量方案的MCC。x轴表示要比较的8种方法:splitDNA2vec-C、splitDNA2vec-V、splitDNA2vec-sh-C、splitDNA2vec-sh-V、dna2vec-V、dna2vec-N1000-C和dna2vec-N1000-V。y轴表示测试数据集上的f测度。图S7。不同嵌入向量方案的AUC如表3所示。x轴表示要比较的8种方法:splitDNA2vec-C、splitDNA2vec-V、splitDNA2vec-sh-C、splitDNA2vec-sh-V、dna2vec-V、dna2vec-N1000-C和dna2vec-N1000-V。y轴表示测试数据集上的f测度。图S8。不同数量变长CMIC的平衡精度k-mer序列由输入的CGI序列生成,N.x轴表示的值N.y轴表示f值N.图S9。CMIC的MCC具有不同数量的变长k-mer序列由输入的CGI序列生成,N.x轴表示的值N.y轴表示f值N.图S10。不同数量变长CMIC的AUCk-mer序列由输入的CGI序列生成,N.x轴表示的值N.y轴表示f值N.图S11。用短cgi训练的CMIC对长cgi的平衡精度。x轴表示CGI序列长度的范围。y轴表示f测度。图S12。CMIC的MCC以短cgi训练长cgi。x轴表示CGI序列长度的范围。y轴表示f测度。图向。CMIC的AUC以短cgi训练为长cgi。x轴表示CGI序列长度的范围。y轴表示f测度。图S14系列。采用RNN、BiRNN、GRU、BiGRU、LSTM和BiLSTM等备选循环单元平衡CMIC的精度。图S15。CMIC的MCC与交替循环单元,RNN, BiRNN, GRU, BiGRU, LSTM和BiLSTM。图S16。CMIC的可选循环单位,RNN, BiRNN, GRU, BiGRU, LSTM和BiLSTM的AUC。图肌力。不同长度的KEGRU平衡精度k-mers和stride。向量大小设置为20。x轴表示一对k和步伐。S18无花果。美国。不同长度的KEGRU的MCCk-mers和stride。向量大小设置为20。x轴表示一对k和步伐。图S19。不同长度的KEGRU的AUCk-mers和stride。向量大小设置为20。x轴表示一对k和步伐。图S20。不同长度的KEGRU平衡精度k-mers和stride。向量大小设置为50。x轴表示一对k和步伐。图S21。不同长度的KEGRU的MCCk-mers和stride。向量大小设置为50。x轴表示一对k和步伐。图S22。不同长度的KEGRU的AUCk-mers和stride。向量大小设置为50。x轴表示一对k和步伐。图S23。t-SNE生成的困惑度为10的图。图S24。t-SNE生成的图,困惑度为50。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
丸山O,李Y,成田H。et al。CMIC:用变长包埋载体预测CpG岛的DNA甲基化遗传k即。BMC生物信息学23, 371(2022)。https://doi.org/10.1186/s12859-022-04916-3
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12859-022-04916-3
关键字
- 循环神经网络
- 门控循环装置
- 分类
- 卵母细胞
- 胚泡
- 胚胎
- 表观遗传修饰
- 重新编程
- 发展