摘要
背景
视网膜是一个复杂的组织,包含多种对视觉至关重要的细胞类型。了解各种视网膜细胞类型的基因表达模式在再生医学中有潜在的应用。来自多能干细胞的视网膜类器官(视神经囊泡)已经开始通过单细胞rna测序研究,对人类视网膜细胞类型发育的转录组学进行深入研究。以前的基因报告方法依赖于使用微阵列数据的体内技术,或用于计算分析单细胞rna测序数据的相关和降维方法。我们的目标是开发一种最先进的布尔方法,过滤掉噪音,可以应用于各种各样的数据集,并深入了解基因表达的差异。
结果
在这里,我们提出了一种生物信息学方法,利用布尔蕴涵来发现视网膜细胞类型特异性或参与视网膜细胞命运的基因。我们将这种方法应用于先前发表的视网膜和视网膜类器官数据集,并改进了先前发表的相关方法。我们的方法提高了视网膜细胞类型标记基因的预测精度,并发现了几个新的高置信度锥体和杆状特异性基因。
结论
这项研究的结果证明了考虑非对称关系的布尔方法的好处。在视网膜细胞类型特异性基因的预测准确性方面,我们已经从相关的对称方法中显示了统计上的显著改进。
此外,我们的方法不包含细胞或组织特异性调整,因此可能会影响癌症和其他人类疾病中基因表达分析的其他领域。
简介
视网膜细胞类型的表征是一个重要的研究领域,广泛应用于眼科和再生医学。随着单细胞rna测序(scRNA-seq)的出现,基因报告的计算方法可以对决定细胞命运的重要基因产生有价值的见解[1].人类多能干细胞(hPSCs)可用于体外生成视网膜细胞类型,具有治疗老年性黄斑变性、色素性视网膜炎和其他视网膜相关致盲的潜在应用。然而,这些细胞类型的基因报告和表征是困难的,因为它们在复杂的培养中异步分化[2].此外,与人类或类器官模型相比,存在更多的小鼠模型数据集。我们建议使用布尔蕴涵分析来提高现有基因报告相关方法的预测精度。
以前的方法在体内和体外
研究关键基因对视网膜发育影响的最常用方法之一是使用转基因“敲除”小鼠模型,该模型经常用于从微阵列数据验证差异表达基因[3.,4,5,6,7,8,9,10,11,12,13,14,15,16,17,18,19,20.].荧光基因报告系广泛用于检测单细胞或单细胞类型的纯化群体中的基因表达[2,21,22,23,24,25].大体积RNA测序(RNA-seq)已经帮助定义了更大群体的视网膜细胞类型的转录组[3.,9,14,17,21,24,26,27,28,29,30.,31,32,33,34,35].流式细胞术是研究分离细胞或液滴特性的主要方法[36,37].单细胞RNA测序(scRNA-seq)如今越来越普遍,是分析视网膜细胞类型和亚型转录组的最详细的方法之一[2,8,13,22,38,39,40,41,42,43,44,45,46,47,48].
大多数关于视网膜细胞类型的研究都依赖于小鼠模型,但许多研究越来越多地研究人类供体视网膜[6,30.,31,48,49,50],特别是为了分析视网膜疾病[31,43,50,51,52,53].青光眼、老年性黄斑变性和视网膜光损伤也在小鼠模型中进行了研究[7,14,29,34,35,54,55].一些研究从胎儿视网膜体外培养细胞系[49,56],而其他人则使用人类多能性、诱导多能性或胚胎干细胞来产生纯化的细胞群或视网膜类器官[2,3.,8,28,38,57,58,59].为了研究视网膜细胞类型随时间的发展,干细胞子代的谱系[58]和不同时间点的时间过程数据(使用PCR和RNA-seq)进行了研究[39,41,54].
以前的计算方法
差异表达分析是从微阵列、RNA-seq和scRNA-seq数据中识别视网膜细胞类型特异性基因和生物标志物的最常用方法[10,13,14,17,24,29,30.,31,39,41,46,47,53,56,59].在单细胞分析中,通常在分层聚类识别细胞簇之前,通过主成分分析降维以减少数据的大小并实现可视化[2,7,30.,41,42,49,56,60].细胞簇可以根据关键标记基因的表达来划分为不同的细胞类型或亚型[48].人工智能引导的细胞簇识别最近已被研究[61].
scRNA-seq数据为深入分析单个细胞的转录组,以及随后对细胞类型、亚型和视网膜区域的描述提供了机会。但是,scRNA-seq数据噪声较大,且包含大量的0,其中真假阴性难以区分。这些零中有许多是由于未能捕获或放大文本而导致的辍学。因此,scRNA-seq数据生成的稀疏阵列具有较低的误漏率和较高的负预测值[62].
迄今为止,大多数研究都高度依赖于细胞聚类,这并不总是可以实现的,特别是在包含未成熟或发育中的细胞的数据集中[1].伪时间分析,沿着发育过程绘制单细胞轨迹,已应用于视网膜类器官,并考虑到短暂状态,而不是离散的簇[38].然而,这些方法受到视网膜细胞类型的异步分化和聚类算法的对称性质的阻碍[63].对基因表达进行排序的相关方法也被广泛使用,绕过了发现细胞集群和识别复杂培养中的共表达基因的需要,包括开发视网膜类器官[2,8,23,27,49,64].
确定基因之间的关系已经朝着更广泛的目标[47,60]和基于网络的分析[9,10,17,25,27,31,60,65].基因表达网络可用于识别表型和疾病状态之间的转变,为临床靶标识别铺平道路。相关分析传统上用于推导共表达网络,敲除小鼠模型被用于直接研究一个基因缺失的影响。然而,相关的对称性质可能导致有价值的信息的损失,并不能提供深入了解随着时间的推移基因的表达。视网膜基因调控和表达的贝叶斯网络主要识别转录因子及其靶标[60,66].因此,我们工作的动机是开发一种普遍适用的最先进的方法,过滤掉噪音,可以应用于各种各样的数据集,并深入了解基因表达对分化的影响。
布尔方法
布尔逻辑是两个值之间的简单数学关系,如高/低或1/0。我们建议使用布尔蕴涵(“if-then”关系)来研究来自scRNA-seq数据的基因之间的依赖性。Sahoo等人的研究表明,布尔蕴涵关系的分析在过滤噪声方面比相关方法更好[67].对布尔蕴涵的分析有助于洞察被相关性忽略的非对称关系。
布尔蕴涵,像相关性一样,并不意味着因果关系,非对称布尔关系可以用子集来考虑。比如,基因A高的关系⇒基因B高表明所有基因B高的细胞都是基因a高的细胞的子集。这允许使用布尔暗示分析发育调控基因,Sahoo等人首次在MiDReG工具中发表。[68].
在之前的研究中,布尔方法已经发现了膀胱癌和结肠癌的预后生物标志物[69,70,71].这些方法也导致了造血干细胞的特征和B细胞和T细胞前体的鉴定[72,73].我们的方法以前没有应用于干细胞来源的视网膜细胞类型,但已经对健康视网膜和视网膜母细胞瘤的转录谱变化有了深入的了解[74].StepMiner和BooleanNet算法是Sahoo等人为微阵列数据开发的,用于识别基因之间的布尔含义关系,但已被应用于各种各样的高通量数据,如RNA-seq, scRNA-seq和微生物组数据[68,75,76,77].
以前的少量研究使用单细胞RNA-seq数据构建基因调控网络,该网络使用布尔关系(如AND, OR和NOT)来模拟造血等过程[78].这些研究还从数据二值化开始,以构建动态可执行模型(有内存的顺序逻辑),这与组合逻辑(无内存)之后的布尔蕴涵关系有典型的不同。Qiu 2020认为,通过使用数据中的零值来表征细胞类型,二值化基因表达值可以在单细胞数据中“包含dropout”[79].然而,通过简单地将任何非零值替换为1,数据被二值化,失去了基因表达的定量信息。在这项工作中,StepMiner算法计算一个阈值,该阈值在将定量表达式值二值化为低或高之前考虑它们。这种方法侧重于布尔暗示关系,因为它们可以根据两个基因之间的关系识别细胞群,并阐明分化过程中的基因表达。
方法
数据规范化和注释
我们应用了log2(v + 1)转换为来自Phillips 2018数据集(GSE98556, n = 546)和Macosko 2015数据集(GSE63472, n = 49,300)的TPM值。使用对数转换CPM值对GSE84859 (n = 14)、GSE98838 (n = 22)、GSE130636 (n = 8217)、GSE148077 (n = 86,253)和Lu 2020数据集(GSE138002 GSE11606 GSE122970, n = 118,555)进行分析。
细胞被标记为临床特征,数据被上传到hegemon.ucsd.edu/eye,在那里他们是公开的。在霸权在线工具中,生成基因之间的散点图,每个点代表单个细胞中基因的表达水平[67,68,69,70,71].
发现布尔含义
StepMiner算法
StepMiner算法通过对排序值拟合步进函数来确定将连续表达式值转换为离散值的阈值。阶跃可以定义为在一段时间内排序基因表达值的最大增加。确定了阈值之后t,基因表达值大于t+ 0.5被认为是高的,低于0.5的被认为是高的t−0.5为低。之间的t+ 0.5和t−0.5被认为是中间值,其中0.5是一个误差范围,相当于从高值到低值的两倍以上变化。在确定布尔关系类型时,中间区域中的点被忽略,因为由于随机错误,它们很可能出现在阈值的错误一侧。这些阈值用于将图划分为四个象限(参见附加文件)1:图S1G) [67,80].
BooleanNet算法
BooleanNet算法通过使用统计数据识别稀疏象限来识别布尔蕴涵关系的类型年代以及似然错误率p.布尔蕴涵关系有六种类型:高⇒高,低⇒低,高⇒低,低⇒高,等价和相反。前四个是不对称的,只有一个稀疏象限。后两者是对称的,有两个稀疏象限。更多信息可以在附加文件中找到1:图S1 [67,80].
错误发现率
对于我们分析的每个scRNA-seq数据集,我们计算了错误发现率(FDR)来评估发现的布尔蕴涵关系的重要性。我们将每个微生物的计数随机排列5次,并使用上述方法识别该随机数据集中的所有布尔关系[81].FDR是随机数据集中布尔关系的平均数量与原始数据集的比率。
视网膜细胞类型基因报告的布尔方法
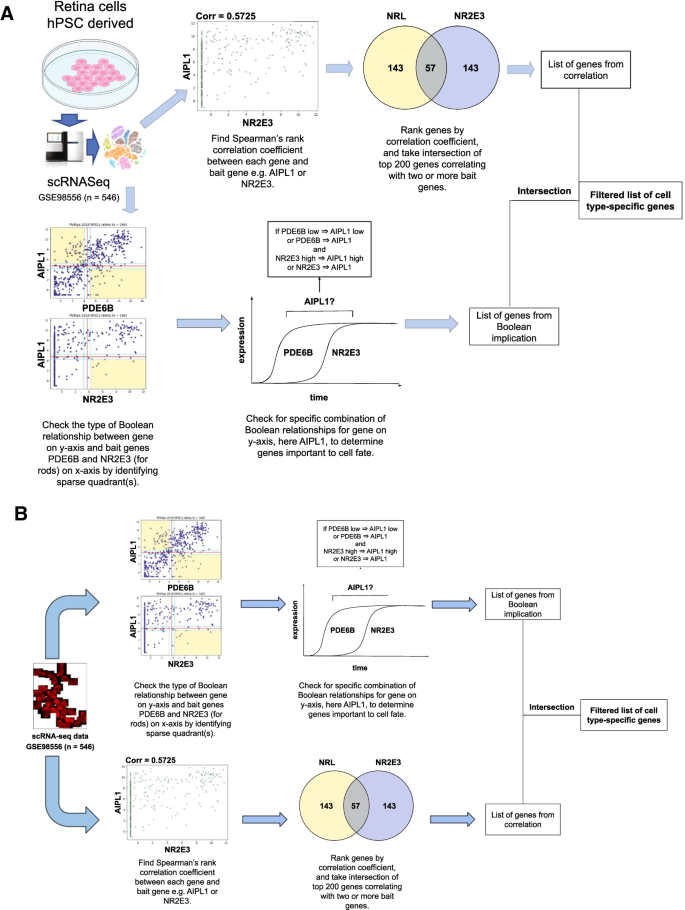
我们建议使用图中描述的方法。1从单细胞数据集中识别视网膜细胞类型的特定标记,如GSE98556 (n = 546)和GSE63472 (n = 49,300)。每一种细胞类型都需要两个或两个以上的已知基因,称为“诱饵基因”。我们寻找低的基因⇒低或等价布尔关系与第一诱饵基因和高⇒与第二诱饵基因具有较高或相当的布尔关系。
这种布尔关系的特殊组合类似于寻找对细胞命运有影响的基因。如果某个基因通过了这个分析,那么基因X低的那组细胞就是第一个诱饵基因低的那组细胞的子集,而基因X高的那组细胞就是第二个诱饵基因高的那组细胞的子集。该方法可以推断出在发育过程中,哪些基因在第一个诱饵基因之后表达,哪些基因在第二个诱饵基因之前表达。因此,诱饵基因的选择对结果起着重要的决定作用。我们选择了诱饵基因,与相关分析相比,可以得到更短的基因列表,这样就可以用布尔分析来过滤相关结果。我们还选择了产生这四种视网膜细胞类型的更多已知标记的诱饵基因。这些是从以前的杆状和锥状光感受器的文献中选择和验证的[6,82,83]、视网膜神经节细胞[23,24]及视网膜色素上皮[84,85,86,87,88].这些基因显示在附加文件中3.:表S2,其中可与CellMarker等现有数据库进行比较[83和我们的研究结果。
两个以上的诱饵基因可以考虑通过搜索高⇒高,低⇒三分之二的诱饵基因存在低或等价的布尔关系,而不是二分之一。这允许在分析中结合多个细胞类型特异性标记基因。
我们在GSE98556中发现了35,389,605个布尔蕴涵关系,FDR为0.000958。我们在GSE63472中发现了3,772,614条关系,FDR为0.0466。附加文件中以直方图的形式显示了六种类型布尔蕴涵关系的分布1:图S1K。在本研究使用的数据集中,四种最常见的关系类型都很高⇒低,低⇒低,高⇒高,等价。相反,低⇒高关系在数据中非常罕见,因此没有被考虑。高⇒基因之间的低关系对我们的目的没有帮助,因为这些基因可能不会同时高度表达,而可能在发育过程中短暂表达。因此,我们关注低价⇒低,高⇒高,等价的关系,因为它们可以识别在特定细胞类型中保持高度表达的标记基因。
斯皮尔曼等级相关系数
斯皮尔曼秩相关系数(SRCC)是非参数度量两个排序变量之间的关联。我们回顾并复制了Phillips等人2018年的方法,称为斯皮尔曼等级相关系数分析(SRCCA)。找到饵基因与所有其他基因的相关系数,并进行排序。然后取前200个相关基因与每个诱饵基因的交集[2].
我们将两种方法结合起来,将两种方法得到的基因列表相互作用,利用布尔蕴涵过滤出相关基因列表,如图所示。1.所有的分析都是使用霸权网站进行的,在Python 3中使用霸权util和ScanPy库,在R版本4.0.1中进行的。
分析阈值
为了考虑scRNA-seq数据中的噪声,应用S和p的阈值来调整布尔网算法的灵敏度。为了确定最合适的阈值,我们考虑了错误发现率(FDR)和获得的基因数量。在之前的工作中,S > 3和p< 0.1通常被认为是微阵列数据,其中FDR < 0.001是首选[67].然而,单单元数据由于有dropout,比微阵列数据有更多的噪声,S > 3和p< 0.1可能不会产生任何布尔关系。降低S阈值可以放松阈值,因为象限中仍然可以认为有更多的点是稀疏的。增加p阈值还可以通过增加似然错误率来放松阈值。放宽算法的阈值会增加发现的布尔蕴涵关系的数量,但也会增加FDR。
我们选择了导致FDR小于0.001的阈值,以及比SRCCA更短的非空基因列表。因此,单细胞数据的阈值确定为S > 2.5和p< 0.35。虽然GSE63472中的FDR大于0.001,但我们使用这些阈值来获得大量的基因,然后对其进行验证。为了确保来自布尔分析的基因列表比来自SRCCA的基因列表更短,我们将GSE98556中的棒的p增加到0.25。
结果量化
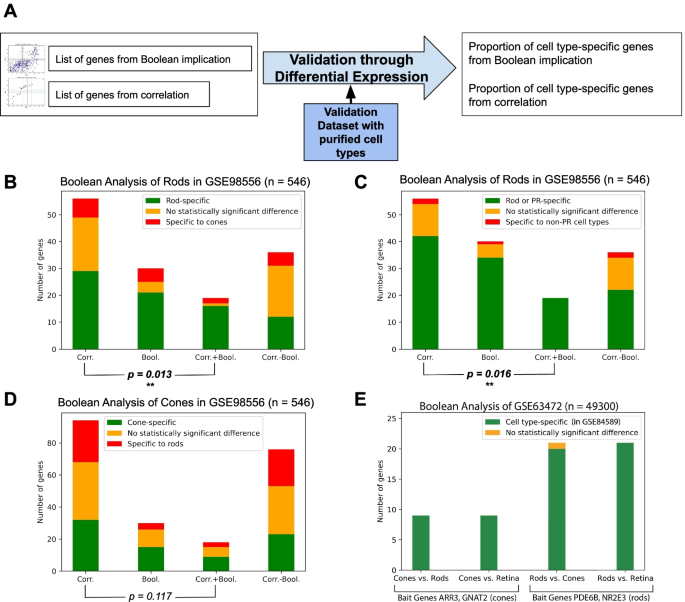
结果通过差异表达独立验证。我们评估了视杆细胞和视锥细胞之间以及感光细胞和非感光细胞类型之间的基因表达是否存在差异。
我们选择并处理了几个验证数据集。两个是包含小家鼠纯化视网膜细胞类型的大量RNA-seq数据集:Hartl 2017 (GSE84589, n = 14)和Sarin 2018 (GSE98838, n = 22) [40,46].第三个是类似的人类视网膜scRNA-seq数据集,Voigt 2020 (GSE130636, n = 20,797) [48].
使用纯化细胞类型的验证数据集,我们通过在细胞组之间执行单尾韦尔奇t检验来检查视网膜细胞类型之间的差异表达,以确定两组之间的均值是否有统计学上的显著差异。使用这种方法,我们可以评估特定于所讨论的细胞类型的基因的比例,在整个视网膜中平等地表达,并在不同的非靶细胞类型中表达。
小提琴图由使用ScanPy[的对数标准化CPM值生成。89].附加文件中的假体分析1:图S2是通过对每种细胞类型的原始计数求和来聚合每个簇中的测量结果。然后进行CPM归一化,排除高表达基因,以近似每种细胞类型的表达水平。
结果
布尔暗示能够识别细胞类型的特定基因,如SRCCA
布尔蕴涵分析同时探讨了基因之间的对称关系和非对称关系,而SRCCA只关注对称关系。我们假设,应用非对称布尔蕴涵关系可以提高细胞类型特异性基因鉴定的准确性(图2)。1).
与SRCCA相比,布尔蕴涵分析的应用导致了更短的基因列表(图。2).饵基因的选择对于SRCCA和布尔分析都是至关重要的。对于布尔分析,一般标记和更具体的标记是理想的候选者。然而,SRCCA只依赖于特定的诱饵基因。由于这些特异性的差异,我们从每种视网膜细胞类型的已知标记基因中选择了不同的诱饵基因进行布尔分析。在对GSE98556和GSE63472的布尔分析中使用了杆状和锥状感光细胞的相同诱饵基因。
结果。Phillips 2018 scRNA-seq数据集GSE98556 (n = 546)使用两个或多个诱饵基因对5种视网膜细胞类型进行SRCCA结果和布尔含义分析。用于分析的诱饵基因显示在顶部。杆状和锥状pr显示布尔蕴涵分析的基因,然后与SRCCA相交。对于rgc,只显示布尔蕴涵与SRCCA相交的基因。对于RPE,布尔蕴涵基因显示为没有交集基因。还列出了Macosko 2015 scRNA-seq数据集GSE63472 (n = 49,300)对2种视网膜细胞类型的布尔含义分析结果。缩写:SRCCA,斯皮尔曼秩相关系数分析;公关,光感受器;RGC,视网膜神经节细胞; RPE, retinal pigment epithelium
光感受器基因报告的布尔分析的应用导致了比其他类型细胞更长的基因列表。在杆状光感受器中观察到SRCCA和布尔蕴涵的最大交集。从GSE63472获得的基因序列比从GSE98556获得的基因序列短。其他视网膜细胞类型如RGCs和RPE中布尔蕴涵基因的数量远低于光感受器。
对于RPE,由于从两个诱饵基因获得的基因数量过少,所以选择了三个诱饵基因。这可能是由于我们的数据集(GSE98556)中来自这些类型的细胞数量较少,与光感受器相比。RPE中SRCCA和布尔基因完全没有交集,这也可以用Phillips等人使用的方法培养的视神经囊泡中存在非常少的RPE细胞来解释。[2]
使用布尔蕴涵过滤SRCCA可以提高预测精度
我们使用纯化的视网膜细胞类型的大量RNA-seq数据集独立验证了SRCCA和布尔含义的基因(图2)。3.A).在图中。3.B,当SRCCA和布尔暗示的交集时,杆特异性基因的比例有明显的改善。同样,在布尔蕴涵中缺失的SRCCA基因,大多数既不特异性于杆状细胞,也不特异性于球果。通过使用布尔暗示过滤相关基因,我们能够在棒状pr特异性基因的比例上显示出统计上显著的改善。通过布尔蕴涵过滤,SRCCA中棒特异性基因的比例从56个(0.517)中29个提高到19个(0.842)中16个。通过执行双比例z检验,该比例显示出统计学意义,返回的p值为0.013。
结果的独立验证。一个使用包含纯化小家鼠棒状和球果的GSE84589等海量RNA-seq数据集,通过差异表达验证棒状和球果基因列表。B杆状细胞基因的类型特异性分为布尔暗示、SRCCA、用布尔暗示过滤的SRCCA和不含布尔暗示的SRCCA 4种方法。C杆状基因的光感受器特异性从4种方法中列出。D杆状基因锥细胞类型特异性从4种方法中列出。E从大型单细胞数据集GSE63472的布尔分析验证锥型和杆型基因。在批量数据集GSE84589中评估了基因特异性的验证。“视锥与视杆”表示假定的视锥基因与视杆基因相比的特异性,反之亦然。“Vs.视网膜”表示假定的锥/杆基因与整个视网膜相比的特异性。缩写:Corr.,相关性;保龄球,Boolean; SRCCA, Spearman’s rank correlation coefficient analysis.请注意:P值来自SRCCA列表中细胞类型特异性基因的比例与使用布尔暗示过滤的SRCCA之间的双比例z检验
同理,如图所示。3.C,我们能够使用相关和布尔方法组合显示杆基因的光感受器特异性在统计上有显著改善。通过布尔隐式筛选SRCCA得到的19个基因均为光感受器特异性基因,双比例z检验p值为0.016。
如图所示。3.D, SRCCA和布尔分析在锥型光感受器中的预测精度都较低。锥特异性基因的比例为15 / 30(0.500),仍然是布尔暗示最高的。在这里,布尔方法单独的预测精度并没有通过与SRCCA的交集来提高。然而,由于SRCCA的总基因数量较多,这一结果在统计学上并不显著。因此,我们在一个更大、更全面的数据集中评估布尔分析的性能。
我们还在GSE63472 (Macosko P14小鼠视网膜)中使用相同的诱饵基因对杆状和锥状光感受器进行了布尔含义分析。GSE63472和GSE98556都包含视网膜的发育阶段,尽管GSE63472来自小鼠视网膜的Drop-seq。由于GSE63472的体积更大(49,300个细胞vs. 546个细胞),因此它更全面,并且可能包含比GSE98556更多可识别的视网膜细胞类型[42].
如图所示。3.E,来自GSE63472的9个锥基因的列表是高度特异性的。与视杆细胞和整个视网膜相比,所有基因都在视锥细胞中富集。21个杆状基因也高度集中,只有1个基因在球果和杆状基因之间表达无统计学差异。从GSE63472的布尔分析中,细胞类型特异性基因的总体比例高于GSE98556。3.罪犯)。
布尔蕴涵提高了新型高置信度基因的预测精度
考虑到通过布尔蕴涵分析预测精度的整体提高,我们还通过该方法调查了几个新发现的具体例子。
新型高置信度基因是基因报告计算方法的重要贡献。单独使用SRCCA识别视网膜细胞类型的高置信标记可能是任意的,但我们表明布尔暗示可以更深入地了解细胞类型特异性基因。
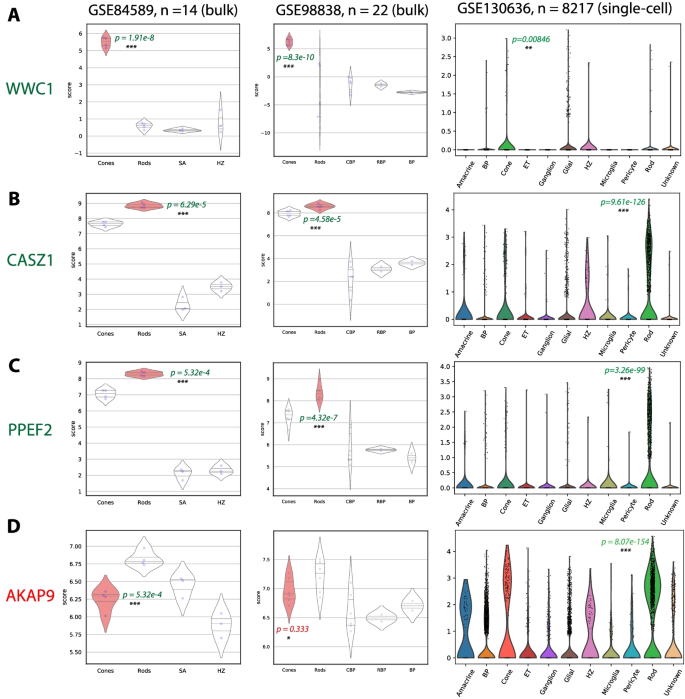
布尔含义分析表明WWC1 (WW结构域含蛋白-1)是一个新的高置信锥感光基因。这在GSE84589和GSE98838中得到独立验证,在锥感光细胞中有统计学意义上的过表达,如图中的小提琴图所示。4A.在人类成人视网膜scRNA-seq数据集GSE130636 (n = 20,797)中,WWC1在锥体光感受器中略有富集,但也在其他几种视网膜细胞类型中表达。这表明WWC1可能参与了视锥细胞的命运,并在早期发育过程中作为一个标记。WWC1在之前的研究中被描述为在大脑和记忆中具有广泛的功能[90,91].
对蓖麻的布尔暗示分析还发现了两个新的蓖麻特异性基因:CASZ1(蓖麻锌指1)(图2)。4B)和PPEF2 (Protein Phosphatase with EF-Hand Domain 2)(图。4C).这些在两个验证数据集中都显示了棒的特异性。已知CASZ1在细胞分化中发挥作用,因此可能在影响棒状细胞命运中发挥重要作用[92].PPEF2以前曾在杆状细胞中被记录过,但关于它在杆状细胞中的重要性有几项相互矛盾的研究[93,94].这证明了它在人或hpsc来源的视网膜中的杆状特异性功能。布尔蕴涵分析揭示了锥体和杆状光感受器的潜在新标记。
附加文件1:图S2,这些基因也在大型和近期的scRNA-seq数据集中得到验证,如Peng 2020 (GSE148077, n = 86,253) [95]和Lu 2020 (GSE138002 GSE122970 GSE116106, n = 118,555) [41].GSE148077附加文件1:图S2A)显示PPEF2和CASZ1的棒特异性表达。WWC1在锥细胞中富集,但在Muller胶质细胞中也有表达。在Lu 2020数据集中,GSE138002(附加文件1:图S2B)显示了WWC1的锥特异性表达和PPEF2和CASZ1的棒特异性表达。CASZ1也在视网膜祖细胞中表达,这表明它在祖细胞发育为杆状细胞时继续表达。GSE122970附加文件1:图S2C)显示PPEF2和CASZ1在棒状细胞中特异性表达。WWC1似乎仅在杆状细胞中表达,但这可能是由于数据集中锥状细胞的数量非常少(20个)。Lu 2020数据集(GSE116106)中的人类胚胎细胞表现出不同的表达模式,因为它们属于更早期的发育阶段,分化不那么高级。GSE138002仅含有成人视网膜细胞,而GSE122970由人类新生儿细胞组成,因此显示出所识别的基因的差异表达。
根据SRCCA的结果,Phillips et al. 2018鉴定出AKAP9 (a -激酶锚定蛋白-9)是一个高置信锥感光基因。数字4D表明,根据GSE84589和GSE130636,它在球果中没有差异表达,并且更具有杆状特异性。
讨论
布尔方法在相关方法的基础上进行了改进,滤除了噪声,识别了非对称关系,从而深入了解了基因的特异性。筛选相关基因可显著提高杆状和光感受器基因的杆状和光感受器基因的特异性,以及锥状光感受器基因的重复性。GSE98556的布尔分析允许比较布尔方法和相关方法,GSE63472的分析允许我们展示布尔分析在大型单细胞数据集中的有效性。因此,我们已经证明布尔蕴涵分析和SRCCA的组合提高了视网膜细胞类型标记的预测准确性。
布尔暗示分析提供了对高置信度基因的更准确的见解,并导致了WWC1作为锥体光感受器的新标记基因的鉴定。从文献中可知,ARR3在球果第一次出现后60天表达> [8].ARR3等效WWC1和GNAT2等效WWC1的布尔关系使我们得出结论,这些基因在大约相似的时间表达,并在球果发育和成熟的后期继续保持高表达。以前仅通过SRCCA从广泛的基因列表中识别高置信度基因的尝试无法区分噪声和真正的细胞类型特异性基因。布尔关系的不对称性质使我们能够确定一个基因是更普遍地表达还是更具体地表达,这在相关性中并不存在。
布尔蕴涵的另一个优点是分析总是可以在整个数据集上执行。当细胞类型(包括不表达该基因的细胞类型)多样性更大时,基因之间的布尔蕴涵关系最明显。然而,SRCCA通常要求操作者根据所讨论的细胞类型是否存在于该发育阶段,选择数据的特定子集(例如第70天)进行分析。这种选择对SRCCA的结果有重大影响,子集的不恰当选择可能导致错误的关联,不能在更大的数据集上泛化。这个问题可以使用布尔蕴涵来解决。
然而,布尔蕴涵分析也不是完全没有错误。误差的主要来源似乎是dropout,它导致象限a中的点密度更大10,一个00和一个01在很多情况下。随着时间的推移或在分化过程中,这些人为的零可能导致对基因表达的不正确解释。与适用于scRNA-seq的轻微放松阈值一起,这增加了错误发现率,特别是在GSE63472中。这个问题可能降低了锥型光感受器分析质量的提高。即便如此,在GSE98556中结合相关分析和布尔蕴涵分析在某些情况下可以导致完全无错误的结果(图98556)。3.C). GSE63472中的布尔分析导致在锥和杆中几乎没有错误的结果(图。3.E),可能是由于数据集的更全面的性质。为了过滤掉像退学这样的噪音,我们过滤了我们的列表并调整了阈值以产生更少的基因,特别是在GSE63472中,这可能会导致假阴性。然而,我们方法的目的是提供一种更具体而不是更敏感的方法。对于本研究的目的而言,相对于相关表达和差异表达等其他方法,少量具有高度特异性和保守性的标记更有用,这些方法产生的基因更多,但也不能滤除噪声。
我们的方法在不同细胞类型之间的性能存在差异。在视网膜中数量较少的细胞类型中,我们可以观察到布尔分析单独和结合SRCCA的基因数量也较小。在杆状细胞(数量最多的神经视网膜细胞类型)中分析效果最好,其次是锥状细胞[96].获得的RGCs基因数量也较低,一些基因如EBF1、ELAVL2和ELAVL4也在无分泌细胞中表达。Phillips等人描述了他们的类器官生成方案的结果,与光感受器相比,其中含有更低比例的RGCs。他们估计rgc的数目随着发展而减少[2].在RPE中,Phillips等人2018年采用的视囊泡培养方案中很少出现RPE,布尔分析和SRCCA之间没有交集,这表明在这种情况下的结果可能包含许多假阳性。此外,rpe相关基因如ADH1A3 [97]和RLBP1 [98]不在列表中,因为这些基因与GSE98556中的诱饵基因没有布尔关系。这可能是因为adh1a3 -高细胞(3.6%)和rlbp1 -高细胞(7.3%)的百分比非常小,在高-高象限没有足够的值。比较不同细胞类型的结果,所获得的假定标记基因的质量取决于数据集的全面性。然而,从SRCCA中获得的基因数量与细胞类型的群体之间没有联系,因为考虑的是固定数量的顶级相关基因。因此,布尔分析可以深入了解数据足够全面的细胞类型,从而提供准确的分辨率。
独立验证方法考虑了多个数据集来评估特异性和再现性。这些高质量的海量RNA-seq数据集为大多数基因提供了可靠的结果,因为我们对hpsc来源的视网膜分析中的许多基因在人类和小鼠视网膜中是保守的。然而,由于物种之间的微小差异,这也不是绝对正确的。对来自GSE63472(家鼠)的基因列表进行验证,发现了比GSE98556(人类)更多的特异性基因,这除了GSE63472分析的更高特异性外,还可以归因于物种变异。更大、更全面的单细胞数据集,如GSE130636、GSE148077和Lu 2020数据集,验证了来自体内源性人类视网膜的细胞类型中的基因。这些共享的细胞类型标记证明了干细胞衍生的类器官模拟体内样本的实用性。即便如此,当使用体内来源的视网膜数据集进行验证时,一些细胞类型(如RGCs和RPE)的结果并不那么具体。体内来源的视网膜含有数百万个RPE,这个数字与杆状细胞和锥状细胞的数量成正比。然而,在GSE98556 (hscs来源的视网膜类器官)中显然不是这样[99].在这方面,我们的分析强调了hPSC和体内来源的视网膜细胞群之间的差异,以及它如何影响结果。
布尔隐含分析提供了SRCCA之前提供的所有优势,包括效率,组合多个诱饵基因的能力,以及与早期方法相比提高的预测精度。我们的方法可以让研究人员分析单细胞数据,即使无法识别细胞集群,这是包含发育细胞的数据集中的一个常见问题。结合这两种方法在基因的特异性和重现性方面提供了统计学上的显著改善。布尔含义可以很容易地从霸权在线工具上的散点图中推断出来,使其成为生物学家和计算机科学家的直观选择[76].
结论
在这项工作中,我们开发了一种基于布尔蕴涵的scRNA-seq数据分析的新方法。与早期仅基于相关性的方法相比,我们在视网膜细胞类型特异性基因的预测准确性方面有统计学上的显著改善。我们的方法应用于视网膜类器官数据集,识别出新的高置信细胞类型特异性基因,如锥体的WWC1和杆状的CASZ1和PPEF2。这种布尔方法允许在复杂培养中分析和描述细胞类型,即使在无法实现细胞聚类的情况下。考虑不对称关系使我们能够有效地过滤掉噪音,从而深入了解在再生医学中具有潜在重要性的基因。
数据和材料的可用性
所有数据均可在公共存储库中获得,相关的加入编号载于文本和补充材料中。分析数据集GSE98556和GSE63472,并使用GSE84859、GSE98838、GSE130636、GSE148077、GSE138002和GSE122970进行验证。数据集可以在hegemon.ucsd.edu/eye上查询。要复制基因间的霸权散点图,输入基因名称后点击“探索”,然后点击“下载”下载高分辨率PDF文件。代码,以重现所有其他数字可在Jupyter笔记本在https://github.com/RohanS14/Boolean-lab/blob/main/Boolean-Retina-Analysis-Revised.ipynb.
参考文献
Zerti D,等。用单细胞RNA测序了解视网膜和多能干细胞来源的视网膜类器官的复杂性:目前的进展,仍然存在的挑战和未来的展望。Curr Eye res 2020;45(3): 385-96。
Phillips MJ,等。一种单细胞rna序列分析的新方法促进了人类多能干细胞来源的视网膜细胞类型的硅基因报告。中国生物医学杂志,2018;36(3):313-24。
布鲁克斯MJ,等。通过比较转录组分析显示,通过调节信号通路改善视网膜类器官分化。中国医学杂志2019;13(5):891-905。
布鲁克斯MJ,等。下一代测序有助于对野生型和Nrl(-/-)视网膜转录组进行定量分析。Mol Vis 2011; 17:3034-54。
程华,等。孤儿核受体NR2E3在哺乳动物视网膜发育过程中建立光受体身份的体内功能。胡文杰,2006;15(17):2588-602。
Corbo JC等。小鼠中感光基因表达模式的类型学。美国国家科学研究院。2007; 104(29): 12069 - 74。
Howell GR,等。分子聚类鉴定补体和内皮素诱导为青光眼小鼠模型的早期事件。中国科学(d辑),2011;
Kallman A,等。利用患者来源的NRL缺失视网膜类器官研究锥型感光器的发育。中国生物医学工程学报。2020;3(1):82。
Kim JW,等。nrl调节的棒状光感受器发育的转录组动力学。Cell Rep. 2016;17(9): 2460-73。
Ma H,等。锥体环核苷酸门控通道的缺失导致光响应调节系统和细胞应激反应途径的改变:基因表达谱研究。中国生物医学工程学报。2013;22(19):3906-19。
米泽拉卡K,德玛索CR, Cepko CL。在新的有丝分裂后细胞中,Notch1是抑制杆状光感受器命运所必需的。发展。2013;140(15):3188 - 97。
Montana CL,等。成人视杆感光器的重编程可以防止视网膜变性。美国国家科学研究院。2013年,110(5):1732 - 7。
穆斯塔菲D,等。转录组分析揭示了哺乳动物视网膜中杆状/锥状感光细胞的特异性特征。中国生物科学。2016;25(20):4376-88。
穆斯塔菲D,等。增强s锥综合征小鼠模型中的感光细胞吞噬缺陷导致进行性视网膜变性。中国科学(d辑). 2011;25(9):3157-76。
大西A,等。孤核激素受体ERRbeta控制杆状感光细胞的存活。美国国家科学研究院。2010; 107(25): 11579 - 84。
Palczewska G,等。受体MER酪氨酸激酶原癌基因(MERTK)在双类视黄酮向视网膜色素上皮细胞的转移中是不需要的。中国生物医学工程学报,2016;
佩雷斯-塞万提斯C等人。增强子转录识别光感受器细胞类型的顺式调节元件。发展。2020;147(3):1。
Roger JE,等。在仅视锥细胞的Nrl-/-小鼠视网膜中快速而短暂的变性和重塑后的视锥光感受器的保存。神经科学杂志,2012;32(2):528-41。
Sundermeier TR,等。DICER1对小鼠有丝分裂后视杆感光细胞的存活至关重要。中国科学进展。2014;28(8):3780-91。
吉田,等。发育和成熟的Nrl-/-小鼠视网膜的表达谱:视网膜疾病候选者的鉴定和Nrl的转录调控靶点。胡文杰,2004;13(14):1487-503。
早期发育的小鼠锥体光感受器中富集表达基因的鉴定。中华眼科杂志2019;60(8):2787-99。
Cherry TJ,等。单细胞分辨率下视网膜无分泌中间神经元的发育和多样化。美国国家科学研究院。2009; 106(23): 9495 - 500。
兰格KB,等。人多能干细胞视网膜神经节细胞的多样性和亚型规范。中国医学杂志,2018;10(4):1282-93。
Sajgo S,等。Brn3视网膜神经节细胞中细胞类型规范的分子编码。美国国家科学研究院。2017; 114 (20): e3974 - 83。
Siegert S,等。成人视网膜细胞类型的转录编码和疾病图谱。神经科学。2012;15(3):487。
Cherry TJ,等。绘制人类视网膜的顺式调节结构揭示了疾病的非编码遗传变异。美国国家科学研究院。117(16): 9001 - 2020; 12。
Dorrell MI,等。发育中的小鼠视网膜整体基因表达分析。中华眼科杂志2004;45(3):1009-19。
吉尔KP,等。来源于人类胚胎干细胞的视网膜神经节细胞。科学通报2016;6:30552。
更难的JM,等。Jnk2缺乏增加了高眼压DBA/2J小鼠青光眼神经退行性变的发生率。细胞死亡,2018;9(6):705。
李敏,等。人视网膜及支持组织基因表达的综合分析。中国科学,2014;23(15):4001-14。
纽曼AM,等。年龄相关性黄斑变性的系统级分析揭示了全球生物标志物和表型特异性功能网络。基因组医学,2012;4(2):16。
Ratnapriya R,等。视网膜转录组和eQTL分析鉴定出与年龄相关性黄斑变性相关的基因。植物学报。2019;51(4):606-10。
Sugino K,等。绘制小鼠大脑中遗传和解剖学定义的细胞群的转录多样性。Elife。2019;8:1。
Williams PA等人。烟酰胺和WLD(S)共同作用预防青光眼神经退行性变。前沿神经科学。2017;11:232。
Williams PA等人。维生素B3调节线粒体脆弱性,预防老年小鼠青光眼。科学。2017;355(6326):756 - 60。
卡特DA,迪克AD,梅尔EJ。CD133+成人视网膜细胞在白血病抑制因子(LIF)中仍未分化。眼科杂志2009;9:1。
Portillo JA,等。原代视网膜细胞的鉴定和体外促炎分子的流式细胞仪检测。生物学报。2009;15:1383-9。
科林J,等。解构视网膜类器官:单细胞RNA-Seq揭示了人类多能干细胞来源的视网膜的细胞成分。中国生物医学工程学报。2019;37(5):593-8。
Daum JM,等。锥状光感受器的光感室的形成与转录开关一致。Elife。2017;6:1。
Hartl D,等。视网膜四种细胞类型的顺式调节景观。中国生物医学工程学报,2017;45(20):11607-21。
卢Y,等。人类视网膜的单细胞分析确定了进化上保守的和物种特有的控制发育的机制。生物工程学报。2015;36 (4):473-491.e9。
Macosko EZ,等。使用纳米升液滴对单个细胞进行高度平行的全基因组表达分析。细胞。2015;161(5):1202 - 14所示。
Orozco LD,等。在人眼中整合eQTL和单细胞图谱确定年龄相关性黄斑变性的致病基因。Cell rep 2020;30(4):1246-1259.e6。
Rheaume BA,等。视网膜神经节细胞的单细胞转录组谱鉴定细胞亚型。地理学报。2018;9(1):2759。
罗旭东,马志强,李志强。色素性视网膜炎Müller胶质细胞内的基因表达变化。生物学报。2012;18:1197-214。
沙林S,等。Wnt信号在视网膜神经瘤发育中的作用:通过RNA-Seq和体内体细胞CRISPR突变分析。神经元。2018;98 (1):109 - 126. - e8。
Shekhar K,等。视网膜双极神经元的单细胞转录组综合分类。细胞。2016;166 (5):1308 - 1323. - e30。
福格特美联社,等。用单细胞RNA测序研究人视网膜中央凹与周围的分子特征。Exp Eye res 2019; 184:234-42。
崔喆,等。原生视网膜和视网膜类器官发育异同的转录组分析。中国眼科杂志2020;61(3):6。
Kirwan RP,等。正常和青光眼人横纹板细胞的整体和细胞外基质聚焦基因表达模式差异。生物学报,2009;15:76-88。
Bennis A,等。小鼠和人视网膜色素上皮细胞基因表达谱的比较:年龄相关性黄斑变性的潜在意义。PLoS ONE。2015; 10 (10): e0141597。
Charish J,等。新原素中和可防止遗传性视网膜变性中光感受器的丧失。中华临床杂志。2020;130(4):2054-68。
Galvao J,等。kruppel样因子基因靶点Dusp14调控轴突生长和再生。中国眼科杂志,2018;29(7):337 - 337。
Agudo M,等。视神经横断和视神经挤压后视网膜转录组的时间进程谱。生物学报。2008;14:1050-63。
Hadziahmetovic M,等。小鼠视网膜光损伤的微阵列分析揭示了神经感觉视网膜和分离的RPE中铁调节、补体和抗氧化基因的变化。中华眼科杂志,2012;29(9):531 - 531。
Strunnikova NV,等。人视网膜色素上皮细胞的转录组分析及分子特征。中国生物工程学报。2010;19(12):2468-86。
Kuroda T,等。鉴定编码慢骨骼肌肌钙蛋白T的基因作为视网膜色素上皮细胞永生化的新标记。科学通报2017;7(1):8163。
Hafler BP,等。转录因子Olig2定义视网膜祖细胞亚群偏向于特定的细胞命运。美国国家科学研究院。2012; 109(20): 7882 - 7。
庄俊华,等。人诱导多能干细胞分化为视网膜谱系过程中细胞内在调控因子的表达谱。中国生物医学工程杂志,2018;9(1):140。
胡静,等。组织特异性基因网络的计算分析:应用于小鼠视网膜功能研究。生物信息学,2010,26(18):2289 - 97。
陈琳,等。将深度监督、自监督和无监督学习集成到单细胞RNA-seq聚类和注释中。基因(巴塞尔)。2020; 11(7): 792。
Haque A,等。生物医学研究和临床应用的单细胞rna测序实用指南。中国医学杂志,2017;9(1):75。
Saelens W,等。单细胞轨迹推断方法的比较。中国生物工程学报。2019;37(5):547-54。
张世生,等。小鼠视网膜发育过程中基因表达的双相模式。BMC发展生物学。2006;6:48。
Howell GR,等。Datgan,一个可重复使用的软件系统,用于复杂转录分析数据的简便查询和可视化。BMC基因组学,2011;12:429。
钱杰,等。组织特异性转录因子调控靶点的鉴定:应用于视网膜特异性基因调控。核酸研究,2005;33(11):3479-91。
Sahoo D,等。布尔蕴涵网络源自大规模全基因组微阵列数据集。中国生物工程学报。2008;9(10):R157。
Sahoo D,等。MiDReG:一种利用布尔暗示挖掘发育调控基因的方法。美国国家科学研究院。2010; 107(13): 5732 - 7。
Dalerba P,等。人类结肠肿瘤转录异质性的单细胞解剖。中国生物工程学报。2011;29(12):1120-7。
Dalerba P,等。CDX2作为II期和III期结肠癌的预后生物标志物。中华实用外科杂志,2016;36(3):344 - 344。
Volkmer JP,等。三种分化状态将膀胱癌风险分层为不同的亚型。美国国家科学研究院。2012, 109(6): 2078 - 83。
镶嵌MA,等。Ly6d标志着b细胞规格的最早阶段,并确定了b细胞和t细胞发育之间的分支点。基因开发,2009;23(20):2376-81。
庞文文,等。随着年龄的增长,人骨髓造血干细胞的频率和髓性偏向性增加。美国国家科学研究院。2011; 108(50): 20012 - 7。
Rajasekaran S,等。非编码和编码转录谱在儿童视网膜母细胞瘤肿瘤中显著改变。Front Oncol, 2019; 9:21 21。
王晓燕,王晓燕,王晓燕,等。基于神经网络的细胞周期基因表达分析。分子生物学杂志,2019;30(14):1770-9。
王晓明,张晓明,张晓明,等。拟南芥基因表达逻辑不变量的鉴定。植物学报,2019;3(3):e00123。
Vo D,等。布尔蕴涵分析揭示了微生物组数据中的候选普遍关系。中国生物医学工程学报。2021;22(1):49。
Schwab JD等。从单细胞数据重建布尔网络集合,以揭示人类造血干细胞衰老的动态。中国生物工程学报,2017;19(5):531 - 532。
邱鹏。单细胞rna序列分析中的缺失。网络学报。2020;11(1):1169。
Dang D,等。识别通用巨噬细胞生物标志物的计算方法。前沿物理。2020;11(275):1。
王志强,王志强,王志强,等。全基因组研究的统计学意义。美国国家科学研究院。2003; 100(16): 9440 - 5。
de Melo J,等。Spalt家族转录因子Sall3调控视锥感光细胞和视网膜水平中间神经元的发育。发展。2011;138(11):2325 - 36。
张旭,等。细胞标记:一种人工管理的人类和小鼠细胞标记资源。核酸res 2019;47(D1): D721-8。
Bennis A,等。干细胞来源的视网膜色素上皮:色素沉着作为成熟标记的作用和与人内源性视网膜色素上皮的基因表达谱比较。干细胞研究进展,2017;13(5):659-69。
Brandl C等。从人诱导多能干细胞(hiPSC)中提取的视网膜色素上皮(RPE)细胞的深入研究。神经医学,2014;16(3):551-64。
廖建林,等。原代视网膜色素上皮细胞和干细胞来源的RPE细胞的分子特征。中国生物医学工程学报。2010;19(21):4229-38。
Plaza Reyes A,等。细胞表面标记物的鉴定及视网膜色素上皮细胞单层分化的建立。自然科学进展。2020;11(1):1609。
刘b,等。人胎儿视网膜色素上皮基因表达的遗传分析提示眼病机制。《社区生物学》2019;2:186。
狼FA,愤怒者P,泰斯FJ。SCANPY:大规模单细胞基因表达数据分析。中国生物医学杂志,2018;19(1):15。
Kremerskothen J,等。一种新型含WW结构域蛋白KIBRA的表征。生物化学学报,2003;30(4):344 - 344。
Papassotiropoulos A等。常见的Kibra等位基因与人类的记忆表现有关。科学。2006;314(5798):475 - 8。
刘智,等。蓖麻基因是一种在细胞分化过程中上调的新基因。生物化学与生物物理学报。2006;39(3):334 - 44。
Ramulu P,等。缺乏EF手蛋白磷酸酶(PPEF-1和PPEF-2)的小鼠正常光反应,光感受器完整性和视紫红质去磷酸化。分子生物学杂志2001;21(24):8605-14。
谢尔曼PM等。果蝇视网膜变性蛋白丝氨酸/苏氨酸磷酸酶保守家族的鉴定和鉴定。1997; 94(21): 11639 - 44。
闫伟,等。人类中央凹和周围视网膜的细胞图谱。科学通报2020;10(1):9802。
Reese BE, Keeley PW。视网膜神经元人口学的基因组控制。Prog Retin Eye res 2016; 55:246-59。
巴特勒JM,等。老化人类视网膜色素上皮细胞的RNA-seq分析:视觉周期基因转录的意外上调。中国生物医学杂志,2016;25(12):567 - 567。
Lidgerwood GE,等。人类多能干细胞来源的视网膜色素上皮随时间的转录组分析。生物信息学。2021;19(2):223-42。
Panda-Jonas S, Jonas JB, Jakobczyk-Zmija M.正常人眼视网膜色素上皮细胞计数、分布和相关性。中华眼科杂志。1996;21(2):319 - 319。
确认
作者要感谢加州大学圣地亚哥分校布尔实验室的成员,他们对我们的工作进行了审查并提供了建设性的批评。
资金
这项工作得到了美国国立卫生研究院(NIH)资助r00ca151673, R01-GM138385, UG3 TR003355, R01-AI155696(到DS), UCOP-RGPO (R00RG2628 & R00RG2642到DS),加州大学圣地亚哥分校桑福德干细胞临床中心(到DS), Padres Pedal病因/Rady儿童医院转化性儿童癌症研究奖(Padres Pedal病因/Rady #PTC2017)到DS, 2017,Padres Pedal原因/C3协作转化癌症研究奖(圣地亚哥NCI癌症中心委员会(C3) #PTC2017)给DS。
作者信息
作者及隶属关系
贡献
DS对项目进行概念化、监督和管理。DS为该项目获得了资金。RS和DS开发软件和方法,策划和可视化数据,并执行形式化分析和计算建模。RS和DS撰写、编辑并审阅了手稿。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者声明没有利益竞争。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1。图S1:
在单细胞RNA测序数据中发现和应用布尔蕴涵关系的方法。(A-F):散点图上可见六种类型的布尔蕴涵关系。两个是对称的,有两个稀疏象限(A,B),四个是不对称的,有一个稀疏象限(C-F)。(G):根据StepMiner算法识别的阈值,将该图分为四个象限。(H-I):布尔网算法识别稀疏象限使用统计量S和似然错误率p和应用阈值分别为2.5和0.35。(J):利用布尔蕴涵关系分析,寻找利用诱饵基因(A和B)决定细胞命运的基因。(K):单细胞数据集GSE98556中六种类型布尔蕴涵关系的分布(见A- f)。对数-对数图显示了GSE98556中每种关系的数量和显示该关系的基因数量的直方图。图S2.单细胞验证(A): Peng 2020数据集(GSE148077)中的小提琴图。(B): Lu 2020数据集(GSE138002)成人视网膜细胞的小提琴图。(C): Lu 2020数据集(GSE122970)中人类新生儿视网膜的小提琴图。小提琴图(左x轴)由对数归一化CPM值生成。折线图(右x轴)表示每种细胞类型的伪体表达式值,使用CPM标准化。WWC1是提出的锥状感光基因,CASZ1和PPEF2是提出的杆状感光基因。
附加文件2。
单细胞验证数据集中的高置信标记。
附加文件3。
布尔蕴涵分析与已知视网膜细胞类型标记物的比较。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
Subramanian, R., Sahoo, D.单细胞数据布尔蕴涵分析预测视网膜细胞类型标记。BMC生物信息学23, 378(2022)。https://doi.org/10.1186/s12859-022-04915-4
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12859-022-04915-4
关键字
- 视网膜
- 单细胞RNA测序
- 多能干细胞
- 布尔分析
- 生物信息学