- 研究gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
Struct2Graph:基于结构预测蛋白质-蛋白质相互作用的图注意网络gydF4y2Ba
BMC生物信息学gydF4y2Ba体积gydF4y2Ba23gydF4y2Ba,文章号:gydF4y2Ba370gydF4y2Ba(gydF4y2Ba2022gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
在分子和纳米尺度上分析蛋白质-蛋白质相互作用(PPIs)的新方法的发展,有助于深入了解细胞内信号通路,并将提高对蛋白质功能以及生物和非生物起源的其他纳米级结构的理解。计算工具的最新进展,特别是涉及现代深度学习算法的工具,已被证明是对描述和合理化PPIs的实验方法的补充。然而,现有的PPI预测工作大多使用蛋白质序列信息,因此在解释蛋白质链的三维组织方面存在困难。gydF4y2Ba
结果gydF4y2Ba
在本研究中,我们解决了这个问题,并描述了一个基于图注意网络的PPI分析gydF4y2BaStruct2GraphgydF4y2Ba,用于直接从折叠蛋白球的结构数据识别PPIs。我们的方法能够在由相等数量的正负对组成的平衡集上预测PPI,准确度为98.89%。在正负对比例为1:10的不平衡集上,Struct2Graph实现了五倍交叉验证的平均准确率为99.42%。此外,Struct2Graph可以潜在地识别可能有助于蛋白质-蛋白质复合物形成的残基。对两种不同的相互作用类型进行了重要残基的识别测试:(a)具有多个配体的蛋白质竞争相同的结合区域,(b)动态蛋白质-蛋白质粘附相互作用。Struct2Graph识别相互作用残基的敏感性为30%,特异性为89%,准确率为87%。gydF4y2Ba
结论gydF4y2Ba
在这篇手稿中,我们解决了PPIs的预测问题,使用了第一个同类的,基于3d结构的图注意网络(代码可在gydF4y2Bahttps://github.com/baranwa2/Struct2GraphgydF4y2Ba).此外,新的相互关注机制通过其无监督的知识选择过程提供了可能的交互位点。这项研究表明,从单个蛋白质的图结构中学习的相对低维特征嵌入优于其他基于全局蛋白质特征的现代机器学习分类器。此外,通过对单氨基酸变异的分析,该关注机制对致病残基变异的偏好高于良性多态性,表明其不局限于界面残基。gydF4y2Ba
简介gydF4y2Ba
蛋白质-蛋白质相互作用(PPIs)是许多生物过程的基础。对人类蛋白质组的分析表明,大多数蛋白质不是单独发挥作用,而是作为多单位复合体的一部分[gydF4y2Ba1gydF4y2Ba].事实上,PPIs是信号转导、代谢调节、环境感知和细胞组织的核心部分[gydF4y2Ba2gydF4y2Ba].在这些过程中,PPIs可以改变酶动力学,促进底物通道,形成新的结合位点,使蛋白质失活,或修改蛋白质相对于底物的特异性[gydF4y2Ba3.gydF4y2Ba].由于PPIs在生命系统中无处不在,能够描述这些相互作用有望进一步加深我们对细胞过程的理解[gydF4y2Ba4gydF4y2Ba]并为疾病治疗和药物发现提供了不可或缺的工具[gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba].PPI及其数学描述对于从其他纳米级构建块(包括但不限于脂质)中创建蛋白质类似物也是必不可少的[gydF4y2Ba7gydF4y2Ba]、糖[gydF4y2Ba8gydF4y2Ba],聚合物[gydF4y2Ba9gydF4y2Ba],纳米级缀合物[gydF4y2Ba10gydF4y2Ba],以及无机纳米颗粒[gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba].gydF4y2Ba
许多策略已被用于解码PPIs,主要针对分子尺度数据和氨基酸序列[gydF4y2Ba14gydF4y2Ba].传统上,高通量实验技术,如双混合屏幕[gydF4y2Ba15gydF4y2Ba],串联亲和纯化[gydF4y2Ba16gydF4y2Ba],以及质谱分析[gydF4y2Ba17gydF4y2Ba]已被应用于创建蛋白质相互作用网络。对准确性不足的关注[gydF4y2Ba18gydF4y2Ba],实验吞吐量低[gydF4y2Ba19gydF4y2Ba]和高成本[gydF4y2Ba20.gydF4y2Ba然而,这些方法中的大多数已经激发了计算方法,可以补充传统的和机器人的实验协议。计算方法可以根据蛋白质的遗传背景、氨基酸序列或结构信息的数据来预测蛋白质是否会相互作用。基因组学分析考虑基因融合等因素[gydF4y2Ba21gydF4y2Ba]、常见物种的保护(系统发育分析)[gydF4y2Ba22gydF4y2Ba],以及进化史[gydF4y2Ba23gydF4y2Ba]来确定一对蛋白质是否相互作用。gydF4y2Ba
PPI分析的典型计算技术使用两种蛋白质的氨基酸序列来确定是否发生相互作用[gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba].若干特征,如共有子序列的频率[gydF4y2Ba26gydF4y2Ba]和auto-covariance [gydF4y2Ba27gydF4y2Ba]已经提出将不同长度的序列转换为统一大小的表示。基于序列的方法最近能够利用蛋白质数据库和机器学习技术进行高精度预测。通过从已解决的复杂结构数据库中识别蛋白质复合物模板的CO-threading算法(COTH)可以从序列中预测蛋白质-蛋白质复合物的三维结构。COTH利用评分函数和结构信息校准氨基酸链序列[gydF4y2Ba28gydF4y2Ba].DeepPPI模型[gydF4y2Ba29gydF4y2Ba]使用人工神经网络预测相互作用,该网络将捕获序列的组成、分布和顺序的特征向量作为输入。DeepFE [gydF4y2Ba30.gydF4y2Ba]在氨基酸序列上使用自然语言处理算法来创建适合作为神经网络分析输入的序列的低维嵌入。特别是DeepFE,已经被证明是相当有效的,预测精度达到94.78%和98.77%gydF4y2Ba酿酒酵母gydF4y2Ba和人类数据集。事实上,大多数基于深度学习的方法已被证明可以实现较高的PPI预测精度[gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba]因为他们的代表权要大得多。除了纯粹依赖基于序列的信息外,现代机器学习方法通常包含网络级信息用于PPI预测。在PPI网络中,每个节点代表一个蛋白质,而它们之间的边代表相互作用。因此,预测任意两个节点之间的相互作用是一个伪装的链接预测问题。最近的方法利用网络结构,以及使用氨基酸序列的向量化表示,以获得更强的预测性能[gydF4y2Ba13gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba].gydF4y2Ba
尽管取得了成功,但上述基于序列的方法并不能推广到与蛋白质类似规模的更广泛的化合物类别,这些化合物同样能够与不基于氨基酸的蛋白质形成复合物,因此缺乏等效的基于序列的表示。虽然蛋白质与DNA的相互作用可以被准确预测[gydF4y2Ba38gydF4y2Ba],基于机器学习的高分子量脂质蛋白质复合物预测方法[gydF4y2Ba7gydF4y2Ba]、糖[gydF4y2Ba8gydF4y2Ba],聚合物[gydF4y2Ba9gydF4y2Ba],树状分子[gydF4y2Ba39gydF4y2Ba]和无机纳米颗粒[gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba]在纳米医学和纳米诊断学中受到广泛关注[gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba],在实验主义者中并不广为人知[gydF4y2Ba42gydF4y2Ba,gydF4y2Ba43gydF4y2Ba,gydF4y2Ba44gydF4y2Ba,gydF4y2Ba45gydF4y2Ba,gydF4y2Ba46gydF4y2Ba,gydF4y2Ba47gydF4y2Ba,gydF4y2Ba48gydF4y2Ba],尽管随着蛋白质和纳米颗粒的统一结构描述符的开发,在这一方向上取得了重大进展[gydF4y2Ba13gydF4y2Ba].因此,考虑到蛋白质的结构及其可变的非蛋白质性、仿生和非生物对应物的预测计算方法成为可能。一些方法利用蛋白质的3D结构预测相互作用[gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba]使用基于知识的方法来评估候选蛋白与模板蛋白复合物的结构相似性。由于这种方法需要更复杂的、无模板的对接方法的详细信息[gydF4y2Ba51gydF4y2Ba]分析未结合的蛋白质成分,并从大量潜在的相互作用位点中识别出最有希望的相互作用。虽然对接方法已证明对某些蛋白质成功,但它们面临着蛋白质在相互作用过程中发生构象变化的困难[gydF4y2Ba52gydF4y2Ba].这些结构方法中的许多也作为机器学习模型的基础。Zhang等开发了PrePPI [gydF4y2Ba53gydF4y2Ba它使用氨基酸序列和系统发育特征作为朴素贝叶斯分类器的输入。Northey等人开发了IntPred [gydF4y2Ba54gydF4y2Ba它将蛋白质分割成一组补丁,将3D结构信息整合到一个特征集中,以预测与多层感知网络的相互作用。这些模型是在精心策划的相互作用数据库上训练的,这些数据库描述了蛋白质之间的二元相互作用,以及相应的接口位点或原子。gydF4y2Ba
在这项工作中,我们迈出了第一步,朝着一个广义的方法来评估蛋白质与其他纳米结构的超分子相互作用。所提出的方法从晶体学数据确定蛋白质纳米级表示上蛋白质-蛋白质复合物的形成概率,与氨基酸-氨基酸序列信息形成对比。我们开发了互图注意网络和相应的计算工具,gydF4y2BaStruct2GraphgydF4y2Ba,仅从三维结构信息预测PPIs。而不是利用蛋白质的几个特定特性,如,疏水性,溶剂可及表面积(SASA),电荷,频率gydF4y2BangramsgydF4y2Ba等,Struct2Graph使用基于图的蛋白球表示gydF4y2Ba只有gydF4y2Ba原子的三维位置。这种基于图的解释允许神经信息传递[gydF4y2Ba55gydF4y2Ba]用于有效地学习蛋白质的表示。Struct2Graph建立在我们之前关于代谢途径预测的工作之上[gydF4y2Ba56gydF4y2Ba],其中表明,小分子和多肽的等效基于图的结构表示加上图卷积网络,显著优于其他涉及计算各种生化特征作为输入的分类器。该方法还利用图论的泛化来描述类似于PPI的复杂纳米级组件[gydF4y2Ba57gydF4y2Ba].gydF4y2Ba
除了其PPI预测的高精度之外,Struct2Graph还提供了许多优点。与利用几何仿生学思想的ML算法类似,Struct2Graph只需要单个蛋白质的3D结构。此外,虽然在本文中我们专注于蛋白质相互作用,但通过在我们的分析中仅使用原子的位置,该框架可以推广到其他具有3D信息的分子结构。此外,Struct2Graph还能够洞察蛋白质相互作用的本质。通过其注意机制,该模型可以潜在地识别可能有助于蛋白质-蛋白质复合物形成的残基。与其他模型不同,Struct2Graph能够以无监督的方式产生这些数据,因此不需要蛋白质复合物信息,而蛋白质复合物信息通常是不可用的[gydF4y2Ba58gydF4y2Ba].gydF4y2Ba
拟议工作的主要贡献可归纳为:gydF4y2Ba
用于PPI预测的图卷积网络gydF4y2BaStruct2Graph使用多层图卷积网络(GCN)从折叠蛋白球的结构数据进行PPI预测。该方法具有通用性,可应用于其他具有三维信息的纳米结构。gydF4y2Ba
PPI数据库的管理gydF4y2Ba:大型PPI数据库仅包含直接/物理交互gydF4y2Ba异源gydF4y2Ba蛋白质对gydF4y2Ba脚注gydF4y2Ba1gydF4y2Ba以及相应PDB文件的信息。特别强调的是基于链ID的长度和每个PDB文件中的最高分辨率的PDB文件的管理,以确保捕获感兴趣蛋白质的最完整的结构信息。gydF4y2Ba
最先进的预测性能gydF4y2Ba:我们的方法能够正确预测PPIs,在由相等数量的正对和负对组成的平衡集上的准确度为98.89%。在正负对比例为1:10的不平衡集上,Struct2Graph实现了五倍交叉验证的平均准确率为99.42%。Struct2Graph不仅优于经典的基于特征的机器学习方法,而且优于其他现代深度学习方法,如Deep-PPI和DeepFE-PPI,它们使用序列信息和特征选择进行PPI预测。gydF4y2Ba
重要残差的无监督预测gydF4y2Ba:新的相互注意机制可以潜在地识别蛋白质-蛋白质复合物形成的重要残基。这种重要性可以源于直接参与相互作用过程(即结合位点),也可以间接通过对适当的蛋白质折叠的贡献,从而形成正确的结合位点几何结构。对于两种不同的相互作用类型(都不是训练集的一部分),测试了重要残基的识别:(a)具有多个配体的蛋白质竞争相同的结合区域,(b)动态蛋白质-蛋白质粘附相互作用。Struct2Graph识别相互作用残基的敏感性为30%,特异性为89%,准确率为87%。gydF4y2Ba
单氨基酸变异(SAV)数据集分析gydF4y2Ba:已知致病突变优先位于界面核心,而不是边缘。在已知的2724个致病SAVs和1364个多态性中,我们的注意机制识别出33.55%的致病SAVs为重要的(注意权重在前20%内),而85.30%的多态性被识别为重要的gydF4y2Ba联合国gydF4y2Ba这表明先前建立的SAV研究与所提出的注意机制确定的重要残差之间存在显著重叠。gydF4y2Ba
材料与方法gydF4y2Ba
PPI数据库gydF4y2Ba
Struct2Graph侧重于基于结构的蛋白质对预测和相互作用位点。因此,我们的PPI数据库仅基于蛋白质的直接/物理相互作用,不包括弱相互作用和松散关联的纳米级生物分子。构建一个大型物理交互数据库,仅由gydF4y2Ba不齐的gydF4y2Ba对,我们搜索所有可能的数据库可用(STRING, BioGRID,完整,薄荷,BIND, DIP, HPRD, APID, OpenWetWare)。并非所有PPI数据库都使用相同的出版物和相同的本体来报告交互。因此,每个数据库报告不同的PPI也就不足为奇了。因此,所有PPI数据库之间的一致性最高可达75% [gydF4y2Ba59gydF4y2Ba].对于Struct2Graph,两个最大的编译数据库,完整的[gydF4y2Ba60gydF4y2Ba和STRING [gydF4y2Ba61gydF4y2Ba]进行进一步分析,并将结果相互比较,以找到真正的相互作用。只选择这两个数据库之间的一致匹配。Struct2Graph数据库是根据常用的研究生物体(gydF4y2Ba酿酒酵母gydF4y2Ba,gydF4y2Ba智人gydF4y2Ba,gydF4y2Ba大肠杆菌gydF4y2Ba,gydF4y2Ba秀丽隐杆线虫gydF4y2Ba而且gydF4y2Ba金黄色葡萄球菌gydF4y2Ba质子泵抑制剂)。对于这些生物,完整的提供了427,503个PPIs,而STRING提供了852,327个PPIs。gydF4y2Ba
STRING将相互作用的类型区分为“激活”、“结合”、“催化”、“表达”、“抑制”和“反应”。另一方面,完整的将交互类型描述为“关联”、“物理关联”、“直接关联/交互”和“共定位”。只有来自完整的“直接关联/相互作用”和来自STRING的“绑定”被认为是物理相互作用。我们只从两个数据库中选择一致的物理交互对。因此,只从其余的交互中提取物理交互数据将ppi的实际数量减少到完整的12,676对和STRING的446,548对。负PPI是从大规模双混合实验中得出负相互作用的工作中提取出来的[gydF4y2Ba62gydF4y2Ba].将来自双杂交系统的阴性蛋白-蛋白对与由STRING和完整构造的数据库进行进一步比较,只选择完全不涉及任何相互作用的蛋白对。我们进一步排除了共定位蛋白对在我们的分析。Struct2Graph的结构信息是从PDB文件中获得的。因此,我们只使用与PDB文件相关联的对。这使得配对总数减少到117,933对(4698对阳性,112,353对阴性)。有些蛋白质研究得很好,因为它们在当前医学和生物技术的兴趣范围内。因此,对PDB文件的交叉引用不止一个,因为这些蛋白质的各种结构都是可访问的。为了找到与PDB文件匹配的蛋白质,数据库中的所有蛋白质都与UniProt登录号(UniProt Acc)匹配,并与UniProt中的PDB文件进行映射[gydF4y2Ba63gydF4y2Ba].不幸的是,并不是所有的蛋白质都在每个PDB文件中完全结晶,随机选择PDB文件可能会导致蛋白质结合位点信息的不完整。因此,我们根据链ID的长度和每个PDB文件中的最高分辨率来策划PDB文件,以确保我们捕获感兴趣蛋白质的最完整的结构信息。每个蛋白质晶体结构的链长和分辨率从RCSB网站[gydF4y2Ba64gydF4y2Ba].完整的负对集被减少到5036对,以创建一个相当平衡的训练样本,其中正对和负对的数量大约相等。对于这个仅由异源对组成的数据库,我们定义了两类,“0”表示非相互作用(阴性:不形成复合体)对,“1”表示相互作用(阳性:形成复合体)对。gydF4y2Ba
蛋白质-蛋白质对的互图注意网络gydF4y2Ba
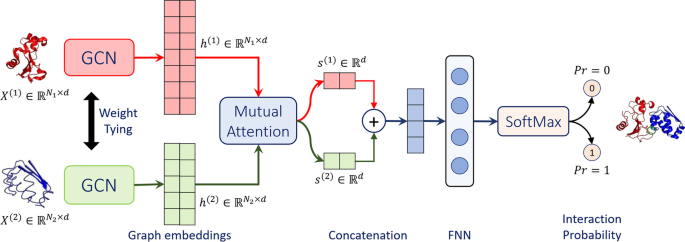
我们提出了一种新的基于多层互图注意网络(GAT)的PPI预测任务架构,如图所示。gydF4y2Ba1gydF4y2Ba.我们将这种体系结构称为gydF4y2BaStruct2GraphgydF4y2Ba,因为所提议的GAT的输入是查询蛋白质-蛋白质对的粗粒度结构描述符。Struct2Graph输出查询蛋白之间交互的概率。Struct2Graph使用两个权值共享的图卷积网络(GCNs)和一个相互注意网络来提取与查询蛋白质对相关的几何特征。然后将这些提取的特征连接起来,并将其馈送到一个前馈神经网络(FNN)gydF4y2BaSoftMaxgydF4y2Ba函数,它最终输出两个类的概率——' 0 '(负对)和' 1 '(正对)。本节首先描述预处理和指纹识别过程,指定如何将蛋白质对的空间信息转换为相应的蛋白质图,然后详细说明Struct2Graph深度学习架构的不同组件。gydF4y2Ba
蛋白质结构图gydF4y2Ba
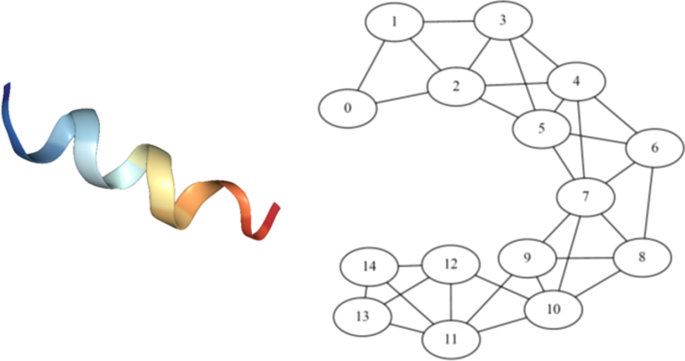
图构建步骤的目的是以一种可由神经网络进一步降维的方式捕获蛋白质的显著几何结构。从单个原子的空间坐标构建图形有许多可能的方法,每一种方法都捕获了蛋白质几何结构的不同细节水平[gydF4y2Ba13gydF4y2Ba].例如,在[中描述的蛋白质接触图gydF4y2Ba65gydF4y2Ba]在三个最近的邻居之间添加边,并将节点标识为螺旋、薄片和转弯。拉莱沃拉等人[gydF4y2Ba66gydF4y2Ba]使用分子指纹来绘制化合物的接触图。皮雷等人[gydF4y2Ba67gydF4y2Ba]采用了一种基于距离的方法,通过编码组成原子之间的距离模式来构建蛋白质图。Cha等人。[gydF4y2Ba13gydF4y2Ba]开创了嵌入化学、几何和图理论描述符的多维蛋白质图。我们构建蛋白质图的方法受到后者的启发,然而,它也可以推广到其他非蛋白质结构。我们首先将原子聚集到它们所组成的氨基酸中,并将氨基酸的位置定义为其组成原子位置的平均值。这些氨基酸构成了蛋白质图的顶点。如果两个顶点之间的距离小于某个阈值,则在两个顶点之间放置一条边。与之前使用7Å阈值的研究不同[gydF4y2Ba13gydF4y2Ba],在这项工作中,我们使用阈值9.5Å从氨基酸的平均位置创建蛋白质图。这个阈值是根据经验获得的,以便使底层图完全连通,同时简化了图的表示。请注意,当我们使用氨基酸作为蛋白质图的组成顶点时,这种方法可以很容易地扩展到多分辨率表示,其中一个顶点表示两个或多个氨基酸。粗粒度表示为研究蛋白质复合物的其他纳米级成分(如脂质和多糖)开辟了新的可能性,因为降低从全原子到亚分子的表示水平可以很容易地推广到其他非蛋白质实体。用官能团作为氨基酸也可以得到结构更精细的图。此外,蛋白质图的这种几何结构确保了显著的几何特征,如沿多肽链的非相邻氨基酸的空间接近性被捕获。基于序列的蛋白质表示可能无法捕获这种几何结构(见图)。gydF4y2Ba2gydF4y2Ba).gydF4y2Ba
图构造方法将与蛋白质相关的空间信息转换为等效的蛋白质图对象gydF4y2Ba\ (\ mathcal {G} = (\ mathcal {V} \ mathcal {E}) \)gydF4y2Ba,在那里gydF4y2Ba\ (\ mathcal {V} \)gydF4y2Ba顶点的集合和gydF4y2Ba\ (\ mathcal {E} \)gydF4y2Ba是它们之间的边的集合。在图中蛋白质图的背景下。gydF4y2Ba2gydF4y2Ba,gydF4y2Ba在\ \ (v_i \ mathcal {V} \)gydF4y2Ba是gydF4y2Ba我\ \ ()gydF4y2BaTh氨基酸和gydF4y2Ba在\ \ (e_ {ij} \ mathcal {E} \)gydF4y2Ba表示之间的边gydF4y2Ba我\ \ ()gydF4y2Bath和gydF4y2Ba\ (j \)gydF4y2BaTh氨基酸,满足它们在9.5Å的指定阈值内的接近性。为了使用我们的机器学习框架,这些图对象必须嵌入到实值向量空间中。我们使用1邻域子图[gydF4y2Ba56gydF4y2Ba]由距离顶点1跳距离的相邻顶点和边引起。通过扫描训练数据库中的所有蛋白质图来构造一个包含所有唯一子图的字典。因此,蛋白质中的每个顶点都等效地由字典中的一个元素表示。gydF4y2Ba
作用于蛋白质图的图卷积网络gydF4y2Ba
图卷积网络(GCN)将图映射到实值gydF4y2Ba嵌入向量gydF4y2Ba以这样一种方式,嵌入向量的几何反映了图之间的相似性。GCN的嵌入部分如下所示。到每个顶点gydF4y2Ba在\ \ (v_i \ mathcal {V} \)gydF4y2Ba,我们会联想到gydF4y2BadgydF4y2Ba-维特征向量,对距离顶点1跳距离的相邻顶点和边诱导的1邻域子图进行编码。这与氨基酸特异性的显式包含形成对比,如疏水性、溶剂可及表面积(SASA)、电荷等。在我们的编码中,类似于其他研究[gydF4y2Ba56gydF4y2Ba,gydF4y2Ba68gydF4y2Ba],子图字典的每个元素都被分配一个随机的单位范数向量。gydF4y2Ba
GCN的每一层更新所有顶点特征,首先将每个顶点特征替换为所有1跳相邻顶点特征的标准化平均值。接着是训练的权重矩阵和偏差参数给出的仿射变换。为了赋予GCN架构表达性,产生的仿射转换嵌入向量的每个坐标都通过非线性激活函数传递,如校正线性单元(ReLU)或sigmoid激活。这一过程对所有后续层重复,最后一层的输出是新转换的嵌入(特征)向量,该向量进一步传播到相互注意网络。在这里,层数是一个超参数,而权重矩阵是从训练数据中学习的,以便优化整个系统在交互预测任务上的性能。gydF4y2Ba
更简单地说,给定输入蛋白质图gydF4y2Ba\ (\ mathcal {G} ^ {(1)}, \ mathcal {G} ^ {(2)} \)gydF4y2Ba用邻接矩阵gydF4y2Ba\(^{(1)},一个^ {(2)}\)gydF4y2Ba组成的gydF4y2Ba\ (N_1、甲烷、\)gydF4y2Ba顶点(氨基酸)和数量gydF4y2Ba\(X_0^{(1)}\in \mathbb {R}^{N_1\乘d}\)gydF4y2Ba,gydF4y2Ba\(X_0^{(2)}\in \mathbb {R}^{N_2\乘d}\)gydF4y2Ba代表gydF4y2BadgydF4y2Ba查询蛋白-蛋白对的顶点子图的-维嵌入,分别为gydF4y2BalgydF4y2Ba-layer GCN使用以下更新规则更新顶点嵌入:gydF4y2Ba
在哪里gydF4y2Ba\(\波浪号{一}^ {(m)} = \离开帽子(\ D {} ^ {(m)} \右)^{- \压裂{1}{2}}\帽子{一}^ {(m)} \离开帽子(\ D {} ^ {(m)} \右)^{- \压裂{1}{2}}\)gydF4y2Ba表示归一化邻接矩阵,和gydF4y2Ba\ (m \ \ {1,2 \} \)gydF4y2Ba.在这里,gydF4y2Ba\(\帽子{一}^ {(m)} = ^ {(m)} + I \)gydF4y2Ba而且gydF4y2Ba帽子\ (\ D {} ^ {(m)} \)gydF4y2Ba的度矩阵是gydF4y2Ba\ \(帽子{一}^ {(m)} \)gydF4y2Ba.参数gydF4y2Ba\ (W_t \)gydF4y2Ba表示与之相关的权重矩阵gydF4y2Ba\ \ (t)gydF4y2BaGCN的第3层。特征嵌入gydF4y2Ba\(X_{l}^{(1)}\in \mathbb {R}^{N_1\ * d}\)gydF4y2Ba而且gydF4y2Ba\(X_{l}^{(2)}\in \mathbb {R}^{N_2\乘d}\)gydF4y2Ba由GCN的最后一层产生的信号被馈送到相互注意网络,以下记为gydF4y2Ba\ (h ^ {(1)} \)gydF4y2Ba而且gydF4y2Ba\ (h ^ {(2)} \)gydF4y2Ba,为便于标记。gydF4y2Ba
相互注意网络用于PPI预测gydF4y2Ba
提出的相互关注网络的目的有两个:(a)提取相关特征用于查询蛋白质-蛋白质对gydF4y2Ba相互gydF4y2Ba有助于预测蛋白质的物理相互作用,(b)结合不同尺寸的嵌入矩阵gydF4y2Bad \ \ (N_1 \倍)gydF4y2Ba而且gydF4y2Bad \ \(甲烷\倍)gydF4y2Ba生成一个具有代表性的单输出嵌入向量,其维数为(2gydF4y2BadgydF4y2Ba).注意机制最初是为了解释序列到序列的翻译模型而引入的,它允许模型以不同的方式关注编码输入的不同部分。从那时起,它已被应用于其他深度学习领域,如计算机视觉[gydF4y2Ba69gydF4y2Ba],以及生物信息学[gydF4y2Ba68gydF4y2Ba].gydF4y2Ba
在这项工作中提出的相互注意机制计算注意权重gydF4y2Ba\(左\[正确\α_ {ij} \] \ \ mathbb {R} ^ {N_1 \ *甲烷}\)gydF4y2Ba和上下文向量gydF4y2Ba\(s^{(1)}\in \mathbb {R}^d\)gydF4y2Ba,gydF4y2Ba\(s^{(2)}\in \mathbb {R}^d\)gydF4y2Ba从gcn转换隐藏嵌入gydF4y2Ba\ (h ^ {(1)} \)gydF4y2Ba而且gydF4y2Ba\ (h ^ {(2)} \)gydF4y2Ba(如图。gydF4y2Ba1gydF4y2Ba).这些gcn嵌入的大小为gydF4y2Bad \ \ (N_1 \倍)gydF4y2Ba而且gydF4y2Bad \ \(甲烷\倍)gydF4y2Ba,分别。对于每个余项gydF4y2Ba我gydF4y2Ba在第一个蛋白中,gcn包埋表示为gydF4y2Ba\ (h_i ^ {(1)} \)gydF4y2Ba,即gydF4y2BadgydF4y2Ba维。类似地,嵌入的gydF4y2Ba\ (j \)gydF4y2Ba第二种蛋白质中的残基表示为gydF4y2Ba\ (h_j ^ {(2)} \)gydF4y2Ba.根据输入蛋白质的大小,gydF4y2Ba\ (N_1 \)gydF4y2Ba而且gydF4y2Ba\(甲烷\)gydF4y2Ba可以是任意的,我们希望我们的Struct2Graph模型是不变的大小gydF4y2Ba\ (N_1 \)gydF4y2Ba而且gydF4y2Ba\(甲烷\)gydF4y2Ba.这是通过使用可学习权重来实现的gydF4y2BaUgydF4y2Ba而且gydF4y2BaVgydF4y2Ba的大小gydF4y2Bad \ \ (d \倍)gydF4y2Ba每一个,和一个权向量gydF4y2Ba\(w\in \mathbb {R}^d\)gydF4y2Ba.特别地,注意权重计算为:gydF4y2Ba
在这里gydF4y2BaUgydF4y2Ba,gydF4y2BaVgydF4y2Ba而且gydF4y2BawgydF4y2Ba与GCN的权重一起以端到端方式进行训练。然后将这些注意力权重转换为上下文向量gydF4y2Ba\ (^ {(1)}, s ^ {(2)} \)gydF4y2Ba(见图。gydF4y2Ba1gydF4y2Ba),使用以下的知识选择程序:gydF4y2Ba
从第一个蛋白质的角度来看gydF4y2Ba\ (N_1 \)gydF4y2Ba残基,提出的知识选择过程(gydF4y2Ba3.gydF4y2Ba)取注意权重矩阵的列平均,得到gydF4y2Ba\ (N_1 \)gydF4y2Ba份向量gydF4y2Ba\ \(η^ {(1)}\)gydF4y2Ba.然后我们执行gydF4y2BaSoftMaxgydF4y2Ba运算,输出概率向量gydF4y2Ba\ (p ^ {(1)} \)gydF4y2Ba从中间嵌入gydF4y2Ba\ \(η^ {(1)}\)gydF4y2Ba.最后,一个gydF4y2BadgydF4y2Ba维嵌入,gydF4y2Ba\ (^ {(1)} \)gydF4y2Ba,作为第一个蛋白质的GCN-embedding的加权平均值,使用残基概率作为权重。对第二个蛋白质重复类似的过程以获得另一个蛋白质gydF4y2BadgydF4y2Ba维嵌入。因此,虽然形成前馈网络(FFN)输入的最终嵌入是上下文向量的连接gydF4y2Ba\ (^ {(1)} \)gydF4y2Ba而且gydF4y2Ba\ (^ {(2)} \)gydF4y2Ba,对应的概率向量gydF4y2Ba\ (p ^ {(1)} \)gydF4y2Ba而且gydF4y2Ba\ (p ^ {(2)} \)gydF4y2Ba分别捕获两种蛋白质中个体残基的相对显著性。那些学习注意权重较大的顶点可能代表直接或间接参与形成蛋白质-蛋白质复合体的残基。gydF4y2Ba
上下文向量gydF4y2Ba\ (^ {(1)} \)gydF4y2Ba而且gydF4y2Ba\ (^ {(2)} \)gydF4y2Ba然后连接到维度2的单个上下文向量gydF4y2BadgydF4y2Ba,作为单层全连接前馈神经网络(FNN)的输入gydF4y2Ba\ (f (\ cdot) \)gydF4y2Ba生成二维输出向量。FNN由另一个权重矩阵参数化,以端到端方式学习。最后一个gydF4y2BaSoftMaxgydF4y2Ba层用于产生一个概率,每个可能的类:0或1,如Eq. (gydF4y2Ba4gydF4y2Ba).这个输出表示分类器对两个蛋白质相互作用的概率的预测。gydF4y2Ba
下面的伪代码(模型细节)总结了提议的Struct2Graph模型的细节。gydF4y2Ba

结果gydF4y2Ba
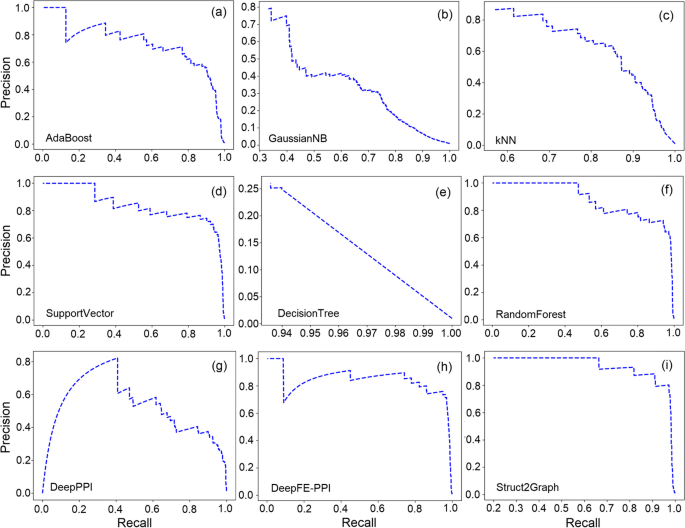
作为评估的一部分,我们将Struct2Graph的PPI预测性能与一些最新的机器学习模型进行了比较。这些方法包括:(a) DeepFE模型[gydF4y2Ba30.gydF4y2Ba],我们在原始出版物中使用的同一数据库上训练自然语言处理网络,并将嵌入馈送到完全连接的前馈神经网络中。(b)深ppi [gydF4y2Ba29gydF4y2Ba],提取了1164个与氨基酸组成、分布和顺序相关的序列特征。一个单独的神经网络用于蛋白质-蛋白质对中的每个蛋白质,它们的输出被连接到一个最终的网络中进行分类。此外,正如原出版物中所做的那样[gydF4y2Ba29gydF4y2Ba],我们将这些特征实现到一些传统的机器学习模型中[gydF4y2Ba70gydF4y2Ba],如(c)高斯朴素贝叶斯(GaussianNB)分类器,(d)二次判别分析(QDA), (e)gydF4y2BakgydF4y2Ba-最近邻(gydF4y2BakgydF4y2Ba(f)决策树(DT)分类器,(g)随机森林(RF)分类器,(h) Adaboost分类器,(i)支持向量分类器(SVC) [gydF4y2Ba71gydF4y2Ba].所有模型都是在Intel i7-7700HQ CPU和2.8 GHz x64处理器上使用Python 3.6.5实现的。对于常见的机器学习分类器,如GaussianNB, QDA, SVC, RF, DT,gydF4y2BakgydF4y2Ba-NN和Adaboost,我们使用scikit-learn中现成的实现[gydF4y2Ba70gydF4y2Ba)模块。深度学习分类器,特别是DeepPPI [gydF4y2Ba72gydF4y2Ba]和DeepFE-PPI [gydF4y2Ba73gydF4y2Ba]在Keras中实现[gydF4y2Ba74gydF4y2Ba],而Struct2Graph在PyTorch中实现[gydF4y2Ba75gydF4y2Ba].gydF4y2Ba
对于Struct2Graph,模型的超参数被调优,以实现报告的准确性。通过对可能的超参数设置集执行网格搜索来获得调优。我们的Struct2Graph实现的超参数如下:gydF4y2Ba优化器gydF4y2Ba: Adam optimizer [gydF4y2Ba76gydF4y2Ba与学习率gydF4y2Ba\ \(λ= 10 ^ {3}\)gydF4y2Ba每10个周期衰减0.5个;gydF4y2Ba总时代gydF4y2Ba: 50;gydF4y2BaGCN层数gydF4y2Ba:gydF4y2Ba\ (l = 2 \)gydF4y2Ba;gydF4y2BaGCN嵌入维数gydF4y2Ba:gydF4y2Ba\ (d = 20 \)gydF4y2Ba;gydF4y2Ba损失函数gydF4y2Ba:二进制交叉熵。对于其他竞争方法,我们使用从原始出版物中采用的调优超参数。gydF4y2Ba
平衡数据库上的性能gydF4y2Ba
表格gydF4y2Ba1gydF4y2Ba总结了Struct2Graph和各种机器学习模型对PPI预测的比较,进行了五次分层交叉验证研究。在交叉验证中,10004对(4698个阳性,5036个阴性)随机分成5个大小相等的子样本。在这五个子样本中,保留一个子样本作为测试各种机器学习模型的验证数据,其余四个子样本用作训练数据。为了减少我们的Struct2Graph模型的训练时间,在每个epoch的8003对(80%)中随机抽取800对进行替换,并使用随机选择的800对上的性能来更新神经网络的参数。这种修改不仅大大减少了训练时间,而且还在训练数据中注入了噪声,以避免任何潜在的过拟合。gydF4y2Ba
性能报告的各种措施,如,准确性,精密度,召回,特异性或真负率,马修斯相关系数(MCC), FgydF4y2Ba\ (_1 \)gydF4y2Ba-评分、受试者工作特征曲线下面积(ROC-AUC)和负预测值(NPV)(见表gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba而且gydF4y2Ba10gydF4y2Ba).对于一个平衡的训练集(表gydF4y2Ba1gydF4y2Ba),在所有测量中,Struct2Graph都优于文献中任何其他现有的机器学习模型(召回率、NPV和ROC-AUC评分除外),平均准确度和精密度分别为98.89%和99.50%。尽管在提出的Struct2Graph模型的训练过程中,我们显著降低了每个epoch中对的数量。gydF4y2Ba
表注gydF4y2Ba1gydF4y2Ba虽然QDA在召回率和NPV分数方面优于Struct2Graph,但在其他测量方面表现非常差,这表明QDA分类器高估了积极的相互作用,导致高假阳性计数。另一个观察结果是,对于这个平衡的训练集,Struct2Graph的性能仅略好于另一个深度学习PPI模型DeepFE-PPI。然而,正如下面所讨论的,DeepFE-PPI在不平衡训练集的情况下表现不佳,在不平衡训练集中,所有的交互作用中积极的交互作用未被充分代表,这种情况在实践中经常出现。gydF4y2Ba
的主要目的gydF4y2BakgydF4y2Ba-fold交叉验证研究是衡量一个模型的泛化能力。另一方面,Bootstrap重采样主要用于为广泛的统计数据建立经验分布函数。它的工作原理是对原始数据集进行采样和替换,同时假设未被选择的数据点是测试数据集。我们重复这个过程几次,并计算平均分数作为各种分类器性能的估计。表格gydF4y2Ba2gydF4y2Ba总结了Struct2Graph和各种机器学习模型在平衡数据集上的比较,用于PPI预测的自举重采样方法重复超过五次。和以前一样,我们在Struct2Graph的训练过程中降低了每个epoch的对数量,以加快计算速度并避免任何潜在的过拟合。使用自举重采样的Struct2Graph方法的性能统计数据与使用五倍交叉验证研究获得的性能统计数据非常相似。结果显示,Struct2Graph在所有测量(召回率和NPV分数除外)方面都优于其他现有机器学习模型,平均准确度和精密度分别为98.96%和99.40%。有趣的是,DeepPPI和DeepFE-PPI方法在平衡集上的性能略差于随机森林分类器。然而,随着类不平衡的增加,DeepFE-PPI被证明优于随机森林分类器。因此,我们还包括了随机森林分类器,用于对不平衡数据集进行比较分析。gydF4y2Ba
在不平衡数据库上的性能gydF4y2Ba
在大多数实际情况下,由于只有一小部分蛋白质对在所有可能的蛋白质对中相互作用,因此预期负对的数量要大于正对。因此,我们评估了深度学习模型deep - ppi和DeepFE-PPI与所提出的Struct2Graph模型在各种不平衡训练集上的性能,其中负对的数量超过正对。这些结果总结在表中gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba而且gydF4y2Ba6gydF4y2Ba对于几个阳性和阴性对比例不同的数据库:(a) 1:2(2518阳性和5036阴性),(b) 1:3(1679阳性和5036阴性),(c) 1:5(1007阳性和5036阴性),和(d) 1:10(504阳性和5036阴性)。注意,不平衡数据库的正对是从策划的正对集中随机选择的。在这种不平衡的情况下,Struct2Graph的表现再次优于深度学习。当正对和负对之间的比例变得越来越倾斜时,Struct2Graph的改善就会增加。例如,当正负比为1:10时,Struct2Graph模型的精度和召回统计量分别为97.54%和96.43%,分别比第二好的深度学习模型DeepFE-PPI的性能高0.98%和2.14%。gydF4y2Ba
Bootstrap重采样产生了非常相似的结论,其中Struct2Graph再次显示出优于其深度学习对应对象以及随机森林分类器,对于几个不平衡的情况一致(见表gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba而且gydF4y2Ba10gydF4y2Ba).当正负对比为1:10时,Struct2Graph模型的准确率、精密度和召回统计量分别为99.26%、97.04%和95.59%,分别比第二优深度学习模型DeepFE-PPI的性能提高了0.76%、0.58%和7.73%。gydF4y2Ba
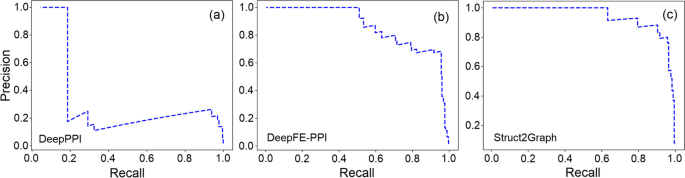
虽然1:10的比例反映了正面和负面例子之间的显著类不平衡,但蛋白质相互作用组中的类不平衡可能是1:100的数量级,甚至更大。在缺乏PPI数据库(包含3d结构信息)且类别不平衡严重的情况下,采用了流行度修正的Precision-Recall Curves (PRCs) [gydF4y2Ba77gydF4y2Ba以牺牲真阳性率为代价来降低假阳性率。数字gydF4y2Ba3.gydF4y2Ba描述了几个PPI分类器在平衡(1:1)上的流行度校正的prc。对精度的计算进行了适当的修正gydF4y2Ba\ \ (r = 100)gydF4y2Ba[gydF4y2Ba77gydF4y2Ba]在实际数据中,正样本与负样本的预期比例为1:100。prc最好地总结了使用不同概率阈值的分类器的真阳性率和阳性预测值(PPV)之间的权衡。图中曲线下面积(AUC)gydF4y2Ba3.gydF4y2Bai接近于统一,从而保证了所提出的Struct2Graph体系结构具有良好的识别能力。数字gydF4y2Ba4gydF4y2Ba描述了非平衡(1:10)数据集上深度学习分类器的流行度校正prc。与前面一样,Struct2Graph体系结构的AUC几乎接近统一。gydF4y2Ba
平衡数据库的流行率校正精度-召回曲线。gydF4y2Ba一个gydF4y2Ba演算法分类器,gydF4y2BabgydF4y2BaGaussianNB分类器,gydF4y2BacgydF4y2BakgydF4y2Ba神经网络分类器,gydF4y2BadgydF4y2BaSVC,gydF4y2BaegydF4y2Ba决策树分类器,gydF4y2BafgydF4y2Ba随机森林分类器,gydF4y2BaggydF4y2BaDeepPPI分类器,gydF4y2BahgydF4y2BaDeepFE-PPI分类器,gydF4y2Ba我gydF4y2BaStruct2Graph(我们的)分类器gydF4y2Ba
PPI预测算法比较的统计检验gydF4y2Ba
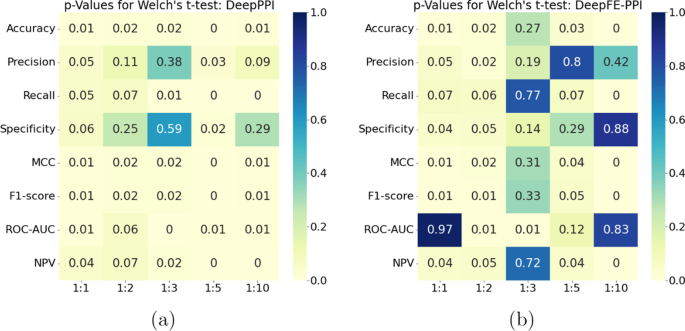
表gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba而且gydF4y2Ba10gydF4y2Ba描述了Struct2Graph在(几乎)所有指标上优于其他竞争分类器。另一方面,其他基于深度学习的分类器,如DeepPPI和DeepFE-PPI,似乎没有Struct2Graph分类器表现得那么好,但它们仍然设法在一些性能度量上合理地接近Struct2Graph。因此,我们将这两个分类器与使用Welch 's的Struct2Graph分类器进行比较gydF4y2BatgydF4y2Ba-test来阐明有利于Struct2Graph分类器的统计显著证据。特别是,我们使用单边韦尔奇在每个指标的交叉验证集的所有折叠上,分别比较了所提出的Struct2Graph模型与DeepPPI和DeepFE-PPI模型的均值gydF4y2BatgydF4y2Ba以及。数字gydF4y2Ba5gydF4y2Ba描述了gydF4y2BapgydF4y2Ba-value拒绝原假设的统计学意义,即相等。在这里,行表示几种性能度量,而列描述了从平衡(1:1)到不平衡(1:2,1:3,1:5,1:10)数据集训练模型的场景。回想一下gydF4y2BapgydF4y2Ba-value小于0.05通常被认为具有统计学意义。可以看到,有统计上显著的证据支持Struct2Graph分类器的优越性能。gydF4y2Ba
PPI数据库的异构性gydF4y2Ba
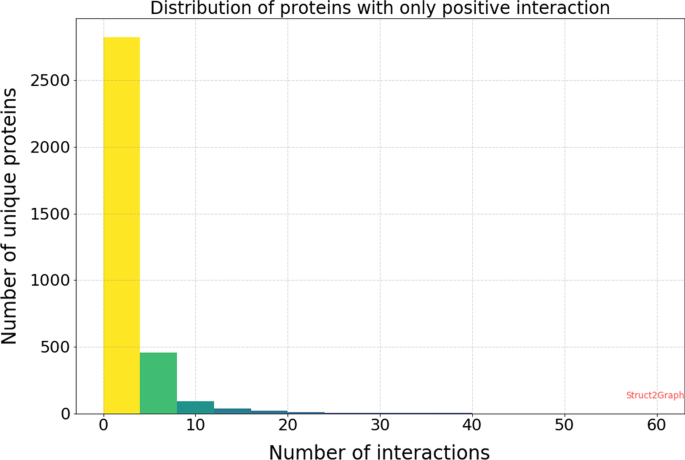
任何机器学习算法的成功都是基于它所提供的训练数据的质量。在横跨3677个独特蛋白质的4698个正面例子和5036个反面例子中,我们首先要确保学习算法不偏向于记忆训练数据[gydF4y2Ba78gydF4y2Ba],因为数据库中的一些蛋白质只参与积极或消极的相互作用。数字gydF4y2Ba6gydF4y2Ba显示每个蛋白质的相互作用数量的分布,这些相互作用只涉及积极的相互作用。可以看出,近82%的独特蛋白质只参与四种或更少的正相互作用。因此,对于分类器来说,如果没有每个蛋白质出现在相对大量的PPI实例中,那么记住训练数据而不学习预测正向相互作用将是极其困难的。gydF4y2Ba
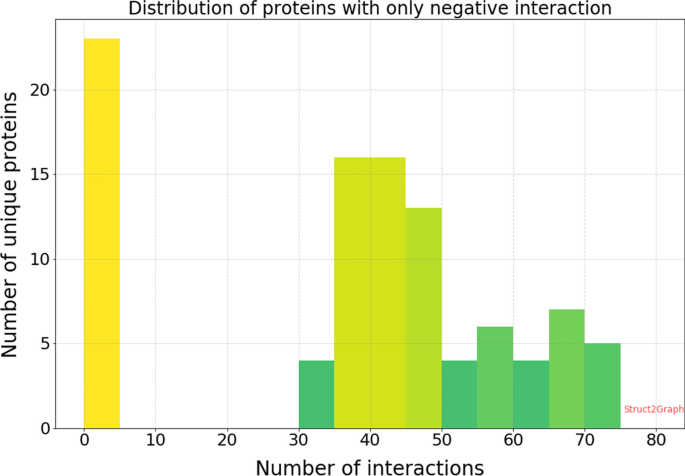
同样,在3677种独特的蛋白质中,104种独特的蛋白质只参与负相互作用(不形成复合体)。数字gydF4y2Ba7gydF4y2Ba显示每个蛋白质只涉及负相互作用的相互作用数量的分布。如直方图所示,在超过5个PPI例子中只出现负相互作用的蛋白质的总数非常少(81个),仅占我们工作中所考虑的整个PDB数据库的2.2%。因此,数据的分布使得学习算法不太可能仅仅通过记忆训练数据来表现良好。该数据集还包括120种参与积极和消极相互作用的独特蛋白质。这120种独特的蛋白质出现在我们数据库中的6335个PPI实例中。因此,任何分类器都几乎不可能简单地记住训练数据,并且仍然能够几乎准确地预测测试或验证集上的相互作用。gydF4y2Ba
我们通过构建一个随机森林分类器来进一步验证这一点,该分类器提供一个长度为3677的输入向量。输入向量长度的具体选择与数据库中唯一蛋白质的数量直接相关。我们首先创建一个包含所有独特蛋白质的字典,并记下这些独特蛋白质在字典中出现的顺序。然后,每个唯一的蛋白质都用一个3677长的单位向量表示,除了一个坐标外,其他坐标都为零。字典中蛋白质顺序对应的坐标标记为1。为了预测两个蛋白质(比如蛋白质A和B)的相互作用,单位向量被求和并作为输入提供给随机森林分类器。回想一下,求和运算是排列不变的,因此对(蛋白质A,蛋白质B)的相互作用预测与对(蛋白质B,蛋白质A)的相互作用预测是相同的gydF4y2Ba11gydF4y2Ba总结了随机森林分类器在平衡和不平衡数据集上的性能,仅使用蛋白质对的标签进行训练,忽略任何结构信息。在平衡的情况下,分类器可以以合理的精度训练gydF4y2Ba\ (91 \ % \ \ sim)gydF4y2Ba在训练集和测试集上。这仍然明显小于使用Struct2Graph和其他基于深度学习的分类器在平衡集上获得的精度。然而,随着训练数据库变得更加真实(即偏向显著丰富的负例),训练集上的性能下降,而测试集上的性能完全随机(gydF4y2Ba\ \ sim 50 \ % \)gydF4y2Ba),即,随机森林分类器就像一个随机预测器。在极端情况下(正例和反例之间的比例为1:10),训练精度似乎得到了提高,这主要是因为每当分类器预测一个负相互作用时,它很可能是正确的,因为训练集有大量的负例。然而,在由大约相等数量的正面或负面示例组成的测试集上,预测精度仍然在50%左右,表明零学习。gydF4y2Ba
讨论gydF4y2Ba
Struct2Graph的成功归因于对嵌入在图形式中的结构3D信息的深入分析,它比基于序列的方法更好地预测相互作用[gydF4y2Ba13gydF4y2Ba].此外,Struct2Graph可以潜在地识别可能有助于蛋白质-蛋白质复合物形成的残基。这是通过考虑概率元组实现的gydF4y2Ba\ (\ {(p_i p_j) \} \)gydF4y2Ba(式中所述的知识选择过程中,gydF4y2Ba3.gydF4y2Ba).这些概率捕捉了氨基酸的相对重要性,从而反映了不同氨基酸对相互作用预测的贡献。具有较大相对概率(前20%)的氨基酸被认为对蛋白质-蛋白质复合物的形成很重要。这种重要性可以源于直接参与相互作用过程(即结合位点),也可以间接通过对适当的蛋白质折叠的贡献,从而形成正确的结合位点几何结构。gydF4y2Ba
对Struct2Graph识别特定相互作用位点的潜力进行了演示,在两个示例案例中(都不是训练集的一部分),在文献中有来自蛋白质对的良好描述的相互作用残基。具体来说,我们研究了两种不同的相互作用类型:(1)多个配体竞争同一结合区域的蛋白质[gydF4y2Ba79gydF4y2Ba];(2)动态蛋白-蛋白黏附相互作用[gydF4y2Ba80gydF4y2Ba].使用标准2x2混淆矩阵将这些复合物中报告的相互作用残基与Struct2Graph的最高概率残基(前20%)进行比较。总的来说(即,两个案例,总共有三个相互作用对),Struct2Graph识别相互作用残基的敏感性为30%,特异性为89%,准确性为87%。需要注意的是,这些蛋白质对示例不在训练集中,Struct2Graph通过其知识选择过程以完全无监督的方式识别这些残基。此外,如上所述,所识别的残基对于确保正确的蛋白质折叠构象至关重要,因此gydF4y2Ba间接gydF4y2Ba对于预测结合很重要,但传统分析仅关注文献中确定的特定相互作用残基,无法捕获。下面描述了每个示例的详细结果:gydF4y2Ba
- (1)gydF4y2Ba
HMGB1和PSMgydF4y2Ba\ \(α_1 \)gydF4y2Ba争夺绑定TLR4gydF4y2Ba:酚溶性调制素(psm),短的、两性的螺旋多肽[gydF4y2Ba81gydF4y2Ba起着至关重要的作用gydF4y2Ba金黄色葡萄球菌gydF4y2Ba毒性,全球人类细菌感染的最常见原因之一[gydF4y2Ba82gydF4y2Ba].gydF4y2Ba金黄色葡萄球菌gydF4y2Ba有七个PSM (PSMgydF4y2Ba\(\alpha _1-\alpha _4\)gydF4y2Ba, PSMgydF4y2Ba\(\beta _1-\beta _2\)gydF4y2Ba,gydF4y2Ba\三角洲(\ \)gydF4y2Ba-毒素)具有多种功能,包括细胞溶解,生物膜结构,以及通过细胞因子释放和趋化作用激活炎症。psm特别触发高迁移率基盒-1蛋白(HMGB1)的释放。toll样受体-4 (TLR4)与HMGB1活化核因子相互作用gydF4y2Ba文本\ (\ {NF -} {B} \ \ upkappa \文本)gydF4y2Ba以及促炎细胞因子的产生[gydF4y2Ba83gydF4y2Ba].然而,gydF4y2Ba金黄色葡萄球菌gydF4y2BapsmgydF4y2Ba\(\alpha _1-\alpha _3\)gydF4y2Ba显著抑制hmgb1介导的磷酸化gydF4y2Ba文本\ (\ {NF -} {B} \ \ upkappa \文本)gydF4y2Ba通过与TLR4结构域相同残基的相互作用与HMGB1竞争[gydF4y2Ba79gydF4y2Ba].因此,HMGB1:TLR4 (2LY4: 3FXI)和PSM的特异性相互作用残基gydF4y2Ba\ \(α_1 \)gydF4y2Ba:TLR4 (5KHB: 3FXI)已得到很好的描述[gydF4y2Ba79gydF4y2Ba].gydF4y2Ba
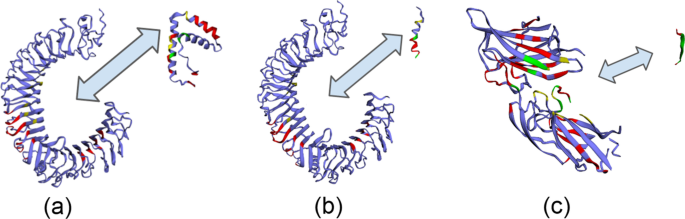
Struct2Graph以90%的准确率识别HMGB1:TLR4对的相互作用残基,其中TLR4的前9个预测残基落在报告的活性腔内(残基排名336-477)。此外,HMGB1预测残基前20%为特异性相互作用残基TyrgydF4y2Ba16gydF4y2Ba和赖氨酸gydF4y2Ba68gydF4y2Ba.对于PSMgydF4y2Ba\ \(α_1 \)gydF4y2Ba:TLR4对,Struct2Graph识别相互作用残基的准确率为92%。同样,预测最多的残基位于先前确定的TLR4活性腔内(336-477)。为PSMgydF4y2Ba\ \(α_1 \)gydF4y2Ba,相互作用残差GlygydF4y2Ba2gydF4y2Ba和瓦尔gydF4y2Ba10gydF4y2Ba被正确地识别出来了。而检测相互作用残留物的总体灵敏度为gydF4y2Ba\ (20 \ % \ \ sim)gydF4y2Ba对于这个例子,Struct2Graph能够预测PSMgydF4y2Ba\ \(α_1 \)gydF4y2Ba在HMGB1结合位点的同一区域与TLR4相互作用。更具体地说,两者在TLR4上的预测结合位点具有94%的一致性。数字gydF4y2Ba8gydF4y2Baa和b显示了预测的基本残基,并强调了Struct2Graph如何预测两个相互作用的相似位点。gydF4y2Ba
- (2)gydF4y2Ba
SdrG-Fibrinogen附着力gydF4y2Ba微生物附着在宿主组织上是大多数细菌感染的关键步骤。革兰氏阳性病原体,如葡萄球菌、链球菌和肠球菌,含有多种细胞壁锚定蛋白,作为粘附蛋白介导细菌附着到宿主组织。这些黏着素介导的相互作用被称为m冲压件(识别黏着基质分子的微生物表面成分)[gydF4y2Ba84gydF4y2Ba].SdrG是gydF4y2Ba葡萄球菌epidermidisgydF4y2Ba它与gydF4y2Baβ\ B \ \)gydF4y2Ba人纤维蛋白原(Fg)链通过动态“dock - lock - latch”机制[gydF4y2Ba80gydF4y2Ba].gydF4y2Ba
利用Struct2Graph评价SdrG (PDB:r19A)与Fg (PDB:r17C)中与其结合位点同源序列的合成肽之间的相互作用。SdrG与合成的Fg肽同源物相互作用残基的预测准确率为75%。在SdrG中鉴定出的高概率残基中,有9个与文献中的残基完全匹配[gydF4y2Ba80gydF4y2Ba].这包括,ProgydF4y2Ba337gydF4y2Ba先生,gydF4y2Ba338gydF4y2Ba,低浓缩铀gydF4y2Ba340gydF4y2Ba,板式换热器gydF4y2Ba344gydF4y2Ba、GlngydF4y2Ba425gydF4y2Ba先生,gydF4y2Ba437gydF4y2Ba,酪氨酸gydF4y2Ba577gydF4y2Ba, AspgydF4y2Ba578gydF4y2Ba,和AsngydF4y2Ba579gydF4y2Ba.数字gydF4y2Ba8gydF4y2BaC表示预测的相互作用所必需的残差。gydF4y2Ba
Struct2Graph对三个示例场景进行重要的剩余预测。gydF4y2Ba一个gydF4y2BaTLR4与HMGB1,gydF4y2BabgydF4y2BaTLR4与PSMgydF4y2Ba\ \(α_1 \)gydF4y2Ba,gydF4y2BacgydF4y2BaSdrG和纤维蛋白原粘连。不同颜色的残基编码不同的信息:(i)红色:Struct2Graph认为重要的前20%残基,(ii)黄色:Struct2Graph认为不重要的实际结合位点,(iii)绿色:与Struct2Graph认为重要的残基重叠的真实结合位点,(iV)紫色:既不重要,也不是实际的相互作用位点。回想HMGB1和PSMgydF4y2Ba\ \(α_1 \)gydF4y2Ba已知在TLR4上竞争相同的结合位点,这也反映在Struct2Graph预测分析中gydF4y2Ba
这些结果表明,Struct2Graph可以深入了解蛋白质-蛋白质相互作用中涉及的关键残基,而无需对这些相互作用的具体性质进行任何训练数据。由Struct2Graph为前面的例子识别的残差的完整摘要包含在补充材料中(参见附加文件)gydF4y2Ba1gydF4y2Ba).任何被识别但未被确认为直接相互作用的高概率残基都可能通过维持蛋白质的适当3D构象而产生间接影响。gydF4y2Ba
除了这些特定的绑定示例之外,我们还考虑了我们的注意力机制在更广泛的数据集中预测有用残差的能力。我们的注意力机制不一定能预测相互作用的位点,但可以预测对蛋白质相互作用很重要的残基,不管它们是否靠近界面。据观察,贯穿整个肽链的残基可以驱动相互作用[gydF4y2Ba85gydF4y2Ba].因此,无论位置如何,注意机制都会识别显著改变相互作用倾向的残基。为了证明这一点,我们分析了单氨基酸变异(SAV)数据集[gydF4y2Ba86gydF4y2Ba](请参阅支持信息附加文件gydF4y2Ba2gydF4y2Ba人类SAV数据集)。作者在[gydF4y2Ba86gydF4y2Ba]对人类单氨基酸变异(SAVs)进行了大规模的结构分析,并证明致病突变优先位于界面核心,而不是边缘。他们的工作共分析了3282种致病SAVs和发生在705种蛋白质中的1699种良性多态性。已确定致病SAVs发生在界面核心的可能性比发生在边缘的可能性高49%,发生在界面核心的可能性比发生在非相互作用蛋白表面的可能性高72%,从而清楚地证明了核心区域和边缘区域对人类疾病的不同贡献。另一方面,78.7%的多态性位于表面可接近残基(241个在界面残基,1096个在表面非界面残基),即多态性不太可能位于界面核心,而不太可能位于边缘。gydF4y2Ba
自从在[gydF4y2Ba86gydF4y2Ba]主要处理人类数据库,他们的数据集和我们手稿中使用的PPI数据库之间有足够的重叠。在重叠的2724个致病SAVs(横跨342个独特蛋白质)和1364个多态性(横跨528个独特蛋白质)中,我们的注意机制确定了33.55%的致病SAVs为重要的(注意权重在前20%内),而85.30%的所有多态性被确定为重要的gydF4y2Ba联合国gydF4y2Ba这表明先前建立的SAV研究与所提出的注意机制确定的重要残差之间存在显著重叠。gydF4y2Ba
结论gydF4y2Ba
提出了一种基于gcn的相互注意分类器Struct2Graph,用于仅从三维结构数据准确预测查询蛋白之间的相互作用。由于先前的研究表明,几何和图形理论描述符可能足以描述PPI [gydF4y2Ba13gydF4y2Ba], Struct2Graph不直接使用描述符,如序列信息,疏水性,表面电荷和溶剂可及表面积,因此可以推广到更广泛的一类纳米级结构,可以以类似的方式表示。这项研究表明,从单个蛋白质的图结构中学习的相对低维特征嵌入优于其他基于全局蛋白质特征的现代机器学习分类器。我们基于gcn的分类器在平衡和不平衡数据集上都达到了最先进的性能。gydF4y2Ba
此外,相互关注机制提供了可能通过直接或间接参与促进相互作用的重要残基的见解。这是通过完全无监督的方式进行知识选择过程来实现的。对两种不同的相互作用类型进行了重要残基的识别测试:(a)多个配体竞争同一结合区域的蛋白质,(b)动态蛋白质-蛋白质粘附相互作用。Struct2Graph识别相互作用残基的敏感性为30%,特异性为89%,准确率为87%。最后,通过对单氨基酸变异的分析,该关注机制对致病残基变异的偏好大于良性残基变异,表明其不局限于界面残基。这种相互作用位点的无监督发现和蛋白质的图形表示之间的联系是可能的,这要归功于原子类型和通常发生在此类分子中的键模式,这使得有可能表征局部原子排列的性质。总的来说,所提出的框架是一般的,而根据相应的训练数据的可用性,可以预测蛋白质和不同化学成分的纳米级物种之间的集体超分子相互作用的其他类型的复杂集。gydF4y2Ba
数据和材料的可用性gydF4y2Ba
源代码和PPI数据库可在gydF4y2Bahttps://github.com/baranwa2/Struct2GraphgydF4y2Ba.gydF4y2Ba
笔记gydF4y2Ba
-
基于对数据库中3677个独特蛋白质的配对同源性分析,只有0.3%的蛋白质被发现具有BLAST e-valuegydF4y2Ba\ (< 0.05 \)gydF4y2Ba0.26%的人gydF4y2Ba\ (< 0.001 \)gydF4y2Ba,表明同源关系在统计学上不显著。gydF4y2Ba
缩写gydF4y2Ba
- PPI:gydF4y2Ba
-
蛋白质相互作用gydF4y2Ba
- 得到:gydF4y2Ba
-
图注意网络gydF4y2Ba
- 政府通讯:gydF4y2Ba
-
图卷积网络gydF4y2Ba
参考文献gydF4y2Ba
Berggård T, Linse S, James P.蛋白质相互作用的检测和分析方法。蛋白质组学,2007;7(16):2833 - 42。gydF4y2Bahttps://doi.org/10.1002/pmic.200700131gydF4y2Ba.gydF4y2Ba
Braun P, Gingras A-C。蛋白质-蛋白质相互作用的历史:从蛋白到复杂网络。蛋白质组学。2012;12(10):1478 - 98。gydF4y2Bahttps://doi.org/10.1002/pmic.201100563gydF4y2Ba.gydF4y2Ba
phzicky EM, Fields S.蛋白质相互作用:检测和分析方法。中国生物医学工程学报,2001;29(1):344 - 344。gydF4y2Bahttps://doi.org/10.1128/MMBR.59.1.94-123.1995gydF4y2Ba.gydF4y2Ba
Ito T, Chiba T, Ozawa R, Yoshida M, Hattori M, Sakaki Y.酵母蛋白相互作用组的综合双杂交分析。自然科学进展,2001;29(4):489 - 497。gydF4y2Bahttps://doi.org/10.1073/pnas.061034498gydF4y2Ba.gydF4y2Ba
弗莱。蛋白质-蛋白质相互作用作为小分子药物发现的靶点。生物聚合物。2006;84(6):535 - 52。gydF4y2Bahttps://doi.org/10.1002/bip.20608gydF4y2Ba.gydF4y2Ba
Coelho ED, Arrais JP, Luis-Oliveira J.从蛋白质-蛋白质相互作用到合理药物设计:计算方法能够迎接挑战吗?中国医学杂志,2013;13(5):602-18。gydF4y2Bahttps://doi.org/10.2174/1568026611313050005gydF4y2Ba.gydF4y2Ba
马沙吉,李志强,李志强。脂质纳米技术。中华分子化学杂志,2013;14(2):4242-82。gydF4y2Ba
黄艳。纳米级粘接相互作用技术。中国医药科学,2004;36(11):1675-87。gydF4y2Ba
来自刺激反应性聚合物和脂质体的智能纳米级药物传递平台。大分子。2013;46(23):9169 - 80。gydF4y2Ba
孟H, Nel AE。利用纳米工程方法克服胰腺癌的间质屏障。Adv药物交付Rev. 2018; 130:50-7。gydF4y2Ba
Kotov NA。无机纳米颗粒作为蛋白质模拟物。科学。2010;330(6001):188 - 9。gydF4y2Ba
Bhandari S, Mondal D, Nataraj S, Balakrishna RG。用于可持续光电子学的生物分子衍生量子点。纳米学报,2019;1(3):913-36。gydF4y2Ba
Cha M, Emre EST, Xiao X, Kim J-Y, Bogdan P, VanEpps JS, Violi A, Kotov NA。统一生物和受生物启发的纳米级复合物的结构描述符。自然科学进展,2018;26(4):344 - 344。gydF4y2Ba
胡林,王旭,黄永安,胡萍,尤志华。预测蛋白质-蛋白质相互作用的计算模型综述。生物信息学报。2021;22(5):036。gydF4y2Ba
Uetz P, Giot L, Cagney G, Mansfield TA, Judson RS, Knight JR, Lockshon D, Narayan V, Srinivasan M, Pochart P, Qureshi-Emili A, Li Y, Godwin B, Conover D, Kalbfleisch T, Vijayadamodar G, Yang M, Johnston M, Fields S, Rothberg JM。蛋白质-蛋白质相互作用的综合分析gydF4y2Ba酿酒酵母gydF4y2Ba.大自然。2000;403(6770):623 - 7。gydF4y2Bahttps://doi.org/10.1038/35001009gydF4y2Ba.gydF4y2Ba
加文A-C, Bösche M, Krause R, Grandi P, Marzioch M, Bauer A, Schultz J, Rick JM, Michon A-M, Cruciat C-M, Remor M, Höfert C, Schelder M, Brajenovic M, Ruffner H, Merino A, Klein K, Hudak M, Dickson D, Rudi T, Gnau V, Bauch A, Bastuck S, Huhse B, Leutwein C, Heurtier M-A, Copley RR, Edelmann A, Querfurth E, Rybin V, Drewes G, Raida M, Bouwmeester T, Bork P, Seraphin B, Kuster B, Neubauer G,Superti-Furga G.蛋白质复合物系统分析酵母蛋白质组的功能组织。大自然。2002;415(6868):141 - 7。gydF4y2Bahttps://doi.org/10.1038/415141agydF4y2Ba.gydF4y2Ba
...Ho Y, Gruhler, Heilbut巴德GD,摩尔L,亚当斯的,米勒,泰勒P,贝内特K, Boutilier K, L,杨Wolting C,唐纳森,Schandorff年代,Shewnarane J,签证官M, Taggart J, Goudreault M, Muskat B, Alfarano C,杜瓦D,林Z, Michalickova K, Willems AR,天使H,尼尔森PA,拉斯穆森KJ,安徒生JR,约翰森LE,汉森LH, Jespersen H, Podtelejnikov,尼尔森E,克劳福德J,保尔森V,索伦森BD, Matthiesen J, Hendrickson RC,格里森F, Pawson T,莫兰MF, Durocher D,曼M,霍格CWV,张志刚,张志刚,张志刚,等gydF4y2Ba酿酒酵母gydF4y2Ba通过质谱分析。大自然。2002;415:4。gydF4y2Ba
spinzak E, Sattath S, Margalit H.实验蛋白质相互作用数据的可靠性如何?中华分子生物学杂志,2003;327(5):919-23。gydF4y2Bahttps://doi.org/10.1016/s0022 - 2836 (03) 00239 - 0gydF4y2Ba.gydF4y2Ba
Skrabanek L, Saini HK, Bader GD, Enright AJ。蛋白质-蛋白质相互作用的计算预测。生物工程学报。2008;38(1):1 - 17。gydF4y2Bahttps://doi.org/10.1007/s12033-007-0069-2gydF4y2Ba.gydF4y2Ba
王旭,黄磊,王晓明。基于亲和纯化和定量质谱的蛋白质复合物相互作用网络分析。蛋白质组学学报。2010;9(8):1650-65。gydF4y2Ba
Marcotte EM, Pellegrini M, Ng H-L, Rice DW, Yeates TO, Eisenberg D.从基因组序列检测蛋白质功能和蛋白质相互作用。科学。1999;285(5428):751 - 3。gydF4y2Bahttps://doi.org/10.1126/science.285.5428.751gydF4y2Ba.gydF4y2Ba
孙杰,李勇,赵忠。蛋白质-蛋白质相互作用预测的系统发育谱:如何选择参考生物?生物化学学报。2007;36(4):489 - 497。gydF4y2Bahttps://doi.org/10.1016/j.bbrc.2006.12.146gydF4y2Ba.gydF4y2Ba
王志刚,王志刚。蛋白质-蛋白质相互作用的系统发育树相似性研究。蛋白质工程学报,2001;14(9):609-14。gydF4y2Bahttps://doi.org/10.1093/protein/14.9.609gydF4y2Ba.gydF4y2Ba
徐杰,许杰。基于序列深度学习的蛋白质相互作用预测方法。生物信息学。2018;34(17):802 - 10。gydF4y2Ba
张峰,宋宏,曾敏,李勇,Kurgan L,李敏。Deepfunc:一种基于蛋白质序列和相互作用的深度学习框架。蛋白质组学。2019;19(12):1900019。gydF4y2Ba
沈娟,张娟,罗霞,朱伟,余凯,陈凯,李勇,姜华。基于序列信息的蛋白质相互作用预测。自然科学进展,2007;26(4):437 - 437。gydF4y2Bahttps://doi.org/10.1073/pnas.0607879104gydF4y2Ba.gydF4y2Ba
郭勇,于磊,文哲,李敏。基于支持向量机和自协方差的蛋白质序列相互作用预测。中国生物医学工程学报,2008;36(9):3025-30。gydF4y2Bahttps://doi.org/10.1093/nar/gkn159gydF4y2Ba.gydF4y2Ba
张勇,张志刚,张志刚。基于模板重组的蛋白质复合物结构预测。结构。2011;19(7):955 - 66。gydF4y2Ba
杜旭,孙松,胡超,姚勇,闫艳,张艳。深度pppi:利用深度神经网络促进蛋白质相互作用的预测。中国化学学报。2017;35(6):379 - 379。gydF4y2Bahttps://doi.org/10.1021/acs.jcim.7b00028gydF4y2Ba.gydF4y2Ba
姚勇,杜旭,刁旸,朱慧。深度学习与特征嵌入相结合的蛋白质相互作用预测。PeerJ。2019; 7:7126。gydF4y2Bahttps://doi.org/10.7717/peerj.7126gydF4y2Ba.gydF4y2Ba
史强,陈伟,黄松,王勇,薛哲。基于深度学习的蛋白质数据挖掘。生物工程学报。2021;22(1):194-218。gydF4y2Ba
Humphreys IR, Pei J, Baek M, Krishnakumar A, Anishchenko I, Ovchinnikov S, Zhang J, Ness TJ, Banjade S, Bagde SR,等。核心真核蛋白复合物的计算结构。科学。2021;374(6573):4805。gydF4y2Ba
刘玲,马艳,朱霞,杨艳,郝霞,王玲,彭娟。利用图卷积网络整合序列和网络信息,增强蛋白质相互作用预测。参加:2019年IEEE生物信息学和生物医学国际会议。IEEE;2019.1762 - 8页。gydF4y2Ba
张东,张晓明,张晓明。多模态深度表示学习在蛋白质相互作用识别和蛋白质家族分类中的应用。生物工程学报。2019;20(16):1-14。gydF4y2Ba
岳霞,王铮,黄娟,Parthasarathy S, Moosavinasab S,黄艳,林森明,张伟,张鹏,孙慧。生物医学网络中的图嵌入:方法、应用与评价。生物信息学。2020;36(4):1241 - 51。gydF4y2Ba
杨峰,范凯,宋东,林华。基于图的蛋白质相互作用预测方法。中国生物医学工程学报。2020;21(1):1 - 16。gydF4y2Ba
黄凯,肖晨,孙杰,孙杰。基于跳图网络的分子间相互作用预测。科学通报2020;10(1):1 - 16。gydF4y2Ba
Rastogi C, Rube HT, Kribelbauer JF, Crocker J, Loker RE, Martini GD, Laptenko O, Freed-Pastor WA, Prives C, Stern DL,等。准确、灵敏地定量蛋白质- dna结合亲和力。中国生物工程学报,2018;29(4):529 - 529。gydF4y2Ba
刘志强,李志强,李志强,刘志强。新型枝状聚合物在纳米医学中的应用。化学学报,2012;41(7):2824-48。gydF4y2Ba
Pelaz B, Alexiou C, Alvarez-Puebla RA, Alves F, Andrews AM, Ashraf S, Balogh LP, Ballerini L, Bestetti A, Brendel C,等。纳米医学的多种应用。中国生物医学工程学报,2017;11(3):2313-81。gydF4y2Ba
徐玲,王旭,王伟,孙敏,崔文杰,金建勇,郝超,李松,曲安,陆敏,等。手性纳米颗粒对映体依赖的免疫反应。自然。2022;601(7893):366 - 73。gydF4y2Ba
查S-H,洪杰,M McGuffie, Yeom B, VanEpps JS, Kotov NA。纳米颗粒对酶的形状依赖仿生抑制及其抑菌活性。中国生物医学工程学报。2015;9(9):9097-105。gydF4y2Ba
Kadiyala U, Turali-Emre ES, Bahng JH, Kotov NA, VanEpps JS。氧化锌纳米颗粒对甲氧西林耐药抗菌活性的意外发现gydF4y2Ba金黄色葡萄球菌gydF4y2Ba(耐甲氧西林金黄色葡萄球菌)。纳米级,2018;10(10):4927 - 39。gydF4y2Ba
Patra JK, Das G, fracto LF, Campos EVR, del Pilar Rodriguez-Torres M, Acosta-Torres LS, Diaz-Torres LA, Grillo R, Swamy MK, Sharma S等。纳米给药系统的最新进展及展望。纳米生物技术,2018;16(1):71。gydF4y2Ba
聚合物缀合物作为抗癌纳米药物。癌症杂志。2006;6(9):688-701。gydF4y2Ba
Bouffard E, El Cheikh K, Gallud A, Da Silva A, Maynadier M, Basile I, Gary-Bobo M, Morere A, Garcia M.为什么抗癌纳米药物需要糖?高等医学杂志,2015;22(26):3014-24。gydF4y2Ba
托里斯·m。纳米药物有输送问题吗?ACS的出版物,2016。gydF4y2Ba
Zamboni WC, Torchilin V, Patri AK, Hrkach J, Stern S, Lee R, Nel A, Panaro NJ, Grodzinski P.癌症纳米技术的最佳实践:来自NCI纳米技术联盟的观点。临床癌症杂志,2012;18(12):3229-41。gydF4y2Ba
福原,川端文。HOMCOS:通过复杂结构的同源建模来预测相互作用蛋白质对和相互作用位点的服务器。核酸决议2008;36:185-9。gydF4y2Bahttps://doi.org/10.1093/nar/gkn218gydF4y2Ba.gydF4y2Ba
胡拉AW,德维尼斯M- d, Smaïl-Tabbone M,里奇DW。基于模板的蛋白质对接的蛋白质结合位点空间聚类。生物信息学。2011;27(20):2820 - 7。gydF4y2Bahttps://doi.org/10.1093/bioinformatics/btr493gydF4y2Ba.gydF4y2Ba
Ohue M, Matsuzaki Y, Uchikoga N, Ishida T, Akiyama Y. MEGADOCK:基于三级结构数据的全对全蛋白质相互作用预测系统。蛋白质肽杂志,2013;21(8):766-78。gydF4y2Bahttps://doi.org/10.2174/09298665113209990050gydF4y2Ba.gydF4y2Ba
张勇,张志勇,张志勇。基于模板的蛋白质-蛋白质相互作用结构建模。中国生物医学杂志,2014;24:10-23。gydF4y2Bahttps://doi.org/10.1016/j.sbi.2013.11.005gydF4y2Ba.gydF4y2Ba
张qc, Petrey D,邓L,强L, Shi Y, Thu CA, Bisikirska B, Lefebvre C, Accili D, Hunter T, Maniatis T, Califano A, Honig B.基于结构的全基因组蛋白质相互作用预测。大自然。2012;490(7421):556 - 60。gydF4y2Bahttps://doi.org/10.1038/nature11503gydF4y2Ba.gydF4y2Ba
Northey TC, bareiich A, Martin ACR。IntPred:一种基于结构的蛋白质-蛋白质相互作用位点预测器。生物信息学。2018;34(2):223 - 9。gydF4y2Bahttps://doi.org/10.1093/bioinformatics/btx585gydF4y2Ba.gydF4y2Ba
Gilmer J, Schoenholz SS, Riley PF, Vinyals O, Dahl GE。量子化学的神经信息传递。见:第34届机器学习国际会议论文集第70卷;2017。JMLR。org。p . 1263 - 72gydF4y2Ba
Baranwal M, Magner A, Elvati P, Saldinger J, Violi A, Hero AO。代谢途径预测的深度学习架构。生物信息学》2019。gydF4y2Ba
姜伟,曲志斌,Kumar P, Vecchio D,王勇,马勇,邦俊华,Bernardino K, Gomes WR, Colombari FM,等。层次结构手性粒子复杂性的出现。科学》2020。gydF4y2Ba
朱红,杜旭,姚艳。基于特征图的集成卷积神经网络识别蛋白质-蛋白质相互作用位点。中国生物科学。2020;15(4):368-78。gydF4y2Ba
Lehne B, Schlitt T.蛋白质相互作用数据库:跟上不断增长的相互作用体。中国生物医学工程学报。2009;3(3):291。gydF4y2Ba
Orchard S, Ammari M, Aranda B, Breuza L, Briganti L, Broackes-Carter F, Campbell NH, Chavali G, Chen C, Del-Toro N,等。MIntAct项目——完整地作为11个分子相互作用数据库的公共管理平台。中国生物医学工程学报,2014;42(D1): 358-63。gydF4y2Ba
Szklarczyk D, Gable AL, Lyon D, Junge A, Wyder S, Huerta-Cepas J, Simonovic M, Doncheva NT, Morris JH, Bork P,等。String v11:覆盖范围增加的蛋白质-蛋白质关联网络,支持全基因组实验数据集中的功能发现。核酸工程学报2019;47(D1): 607-13。gydF4y2Ba
Trabuco LG, Betts MJ, Russell RB。来自大规模双杂交实验的蛋白质-蛋白质相互作用负数据集。方法。2012;58(4):343 - 8。gydF4y2Ba
贝特曼a . Uniprot:蛋白质知识的通用中心。见:蛋白质科学,第28卷。美国新泽西州霍博肯河街111号Wiley邮编07030-5774;2019.32页。gydF4y2Ba
Burley SK, Berman HM, Bhikadiya C, Bi C, Chen L, Di Costanzo L, Christie C, Dalenberg K, Duarte JM, Dutta S,等。RCSB蛋白质数据库:生物大分子结构,使基础生物学,生物医学,生物技术和能源的研究和教育。核酸工程学报,2019;47(D1): 464-74。gydF4y2Ba
Borgwardt KM, Ong CS, Schönauer S, Vishwanathan S, Smola AJ, Kriegel惠普。基于图核的蛋白质功能预测。生物信息学。2005;21(1):47-56。gydF4y2Ba
李志强,李志强,李志强。化学信息学的图形核。神经网络,2005;18(8):1093-110。gydF4y2Ba
Pires DE, Ascher DB, Blundell TL. MCSM:利用基于图的特征预测蛋白质突变的影响。生物信息学。2014;30(3):335 - 42。gydF4y2Ba
张晓明,李志强,张晓明。基于神经网络的蛋白质相互作用预测方法研究。生物信息学,2018,35(2):309 - 18。gydF4y2Ba
Xu K, Ba J, Kiros R, Cho K, Courville A, Salakhudinov R, Zemel R, Bengio Y. Show, attend and tell:视觉注意力的神经图像标题生成。正确的做法:机器学习国际会议。2015.2048 - 57页。gydF4y2Ba
Pedregosa F, Varoquaux G, Gramfort A, Michel V, Thirion B, Grisel O, Blondel M, pretenhofer P, Weiss R, Dubourg V,等。Scikit-learn: python中的机器学习。J Mach Learn Res. 2011; 12:2825-30。gydF4y2Ba
本虚A, Ong CS, Sonnenburg S, Schölkopf B, Rätsch G.支持向量机和计算生物学的内核。计算机科学进展。2008;4(10):1000173。gydF4y2Ba
杜旭,孙松,胡超,姚勇,闫艳,张艳。深度pppi:利用深度神经网络促进蛋白质相互作用的预测。GitHub;2017.gydF4y2Ba
姚勇,杜旭,刁旸,朱慧。深度学习与特征嵌入相结合的蛋白质相互作用预测。GitHub;2019.gydF4y2Ba
Ketkar N. Keras介绍。Apress,伯克利,加州;2017.p . 97 - 111。gydF4y2Bahttps://doi.org/10.1007/978-1-4842-2766-4_7gydF4y2Ba
Ketkar N. PyTorch简介Apress,伯克利,加州;2017.p . 195 - 208。gydF4y2Bahttps://doi.org/10.1007/978-1-4842-2766-4_12gydF4y2Ba
金玛。DP . Ba J. Adam:随机优化方法2014。gydF4y2BaarXiv: 1412.6980gydF4y2Ba
Dick K, Green JR.改进蛋白质-蛋白质相互作用预测的互惠视角。科学通报2018;8(1):1 - 12。gydF4y2Ba
Brown G, Bun M, Feldman V, Smith A, Talwar K.什么时候记忆无关的训练数据对高精度学习是必要的?在:第53届ACM SIGACT计算理论研讨会论文集。2021.123 - 32页。gydF4y2Ba
褚敏,周敏,蒋超,陈霞,郭玲,张敏,褚震,王颖。金黄色葡萄球菌酚溶性调素α1-α3作为新型toll样受体(TLR) 4拮抗剂抑制hmgb1/tlr4/nf-κb信号通路。前沿免疫,2018;9:862。gydF4y2Ba
Ponnuraj K, Bowden MG, Davis S, Gurusiddappa S, Moore D, Choe D, Xu Y, Hook M, Narayana SV。葡萄球菌粘连蛋白与纤维蛋白原结合的“码头、锁和闩”结构模型。细胞。2003;115(2):217 - 28。gydF4y2Ba
梅林C, Headley CM, Klebanoff SJ。表皮葡萄球菌炎症多肽复合物:分离和鉴定。中华检验医学杂志,1999;29(6):357 - 357。gydF4y2Ba
taeb - fligelman E, Tabachnikov O, Moshe A, Goldshmidt-Tran O, Sawaya MR, Coquelle N, Colletier J-P, Landau M.细胞毒性金黄色葡萄球菌PSMα3显示交叉-α淀粉样原纤维。科学。2017;355(6327):831 - 3。gydF4y2Ba
王勇,翁宏,宋金峰,邓玉华,李松,刘海波。HMGB1-TLR4-NF-κB通路的激活可能发生在特应性湿疹患者中。Mol Med 2017;16(3): 2714-20。gydF4y2Ba
Patti JM, Höök M.识别细胞外基质大分子的微生物粘连素。中国生物医学工程学报。1994;6(5):752-8。gydF4y2Ba
傅霞,王勇,宋霞,石霞,邵红,刘颖,张敏,常忠。多寡聚蛋白在活细胞中“非界面”残基介导亚基相互作用。生物化学学报。2019;51(1):100-5。gydF4y2Bahttps://doi.org/10.1016/j.bbrc.2019.03.004gydF4y2Ba.gydF4y2Ba
David A, Sternberg MJ。蛋白质-蛋白质界面核心和边缘残基错义突变对人类疾病的贡献。中国生物医学杂志,2015;gydF4y2Ba
确认gydF4y2Ba
我们感谢匿名推荐人提供的有用建议。gydF4y2Ba
资金gydF4y2Ba
AV, AOH, JSV, PE, MB, AM和SK感谢来自密歇根大学工程学院蓝天计划的支持。AOH感谢ARO W911NF-19-1-0269和ARO W911NF-14-1-0359的支持。AV和JSV承认来自DARPA HR00111720067的支持。NAK感谢Vannewar Bush DoD奖学金。资助者在研究设计、数据收集和分析、发表决定或手稿准备中没有任何作用。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
MB:方法论,软件,分析,写作-初稿。AM:方法论,软件,分析。JS: PPI结果的验证,与现有方法的比较。EST-E:数据管理、验证、写ppi数据库和交互站点预测部分。SK:数据管理。PE:方法学-分子的表现和性质,概念化,写作-评论和编辑。JSV:编写-评审和编辑,监督- ppi数据库。NAK:方法学-蛋白质和其他纳米结构的图形表示,纳米生物结构中几何相互作用的概念,写作-评审和编辑,监督- ppi数据库。AV:概念化,写作评审和编辑,监督。AOH:概念化,写作评审和编辑,监督。 All authors read and approved the final manuscript.
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
伦理批准并同意参与gydF4y2Ba
不适用。gydF4y2Ba
发表同意书gydF4y2Ba
不适用。gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者宣称他们之间没有利益冲突。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba
伟德体育在线施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
补充信息gydF4y2Ba
附加文件1:gydF4y2Ba
由Struct2Graph预测的重要残基列表。gydF4y2Ba
附加文件2:gydF4y2Ba
UniProtKB/Swiss-Prot人工条目中注释的错误含义变量列表。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.创作共用公共领域奉献弃权书(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本条所提供的资料,除非在资料的信用额度中另有说明。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
巴兰瓦尔,M.,马格纳,A.,萨尔丁格,J.。gydF4y2Baet al。gydF4y2BaStruct2Graph:基于结构预测蛋白质-蛋白质相互作用的图注意网络。gydF4y2BaBMC生物信息学gydF4y2Ba23gydF4y2Ba, 370(2022)。https://doi.org/10.1186/s12859-022-04910-9gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12859-022-04910-9gydF4y2Ba
关键字gydF4y2Ba
- 蛋白质相互作用gydF4y2Ba
- 深度学习gydF4y2Ba
- 基于结构的预测gydF4y2Ba
- 图注意网络gydF4y2Ba