摘要
背景
在多组学网络中,通过不同组学层表达不同类型的生物分子之间复杂的相互作用,对深入理解基因调控和疾病具有巨大的潜力。然而,多组学网络很容易发展成巨大的毛球结构,阻碍了生物学的解释。模块检测方法可以将这些网络分解为更小的可解释模块。然而,这些方法不适合处理多组学数据,也不考虑拓扑特征。当导出非常大的模块或忽略更广泛的网络上下文时,可解释性仍然是有限的。为了解决这些问题,我们开发了一个基于子图的多组学聚类框架(SUBATOMIC),它可以推断具有特定拓扑结构的小型可解释模块,同时跟踪与其他模块和调节器的连接。
结果
SUBATOMIC将特定的分子相互作用分组在两个节点和三个节点的复合网络子图中,并将它们聚类成拓扑模块。这些模块通过函数注释、可视化和表达式配置文件覆盖,从静态模块过渡到动态模块。为了保持更大的网络环境,SUBATOMIC从统计上调查了模块之间的连接,以及模块与调控因子(如miRNAs和转录因子)之间的连接。我们应用SUBATOMIC分析了一种复合材料智人网络包含转录因子-靶基因、mirna -靶基因、蛋白质-蛋白质、来自不同数据库的同源和共功能相互作用。我们推导并注释了5586个具有不同拓扑、功能和规则属性的模块。我们为未注释的基因创造了新的功能假设。此外,我们将模块与条件特异性表达数据集成,研究缺氧对三种癌细胞系的影响。我们开发了两种优先排序策略,以确定在特定生物环境中最相关的模块:一种考虑氧化石墨烯术语富集,另一种计算反映差异表达程度的活动评分。两种策略都产生了专门应对低氧水平的模块。
结论
我们开发了SUBATOMIC框架,从集成的多组学网络生成可解释模块,并将其应用于癌症的缺氧。SUBATOMIC可以推断和上下文化模块,探索特定的条件或疾病模块,识别调控器和功能相关模块,并为未特征基因推导新的基因功能。该软件可在https://github.com/CBIGR/SUBATOMIC.
简介
真核生物基因调控涉及不同类型生物分子之间复杂的相互作用,以保证基因在空间和时间上的正确表达。转录因子(tf)与DNA中的特定序列结合,如启动子和增强子区域,以激活或抑制基因表达[1,2,3.].辅助因子与转录因子结合并与转录机制相互作用[2,4].在表观遗传水平上,染色质的可及性是指tf、rna聚合酶或染色质组织蛋白等分子能够通过启动子、增强子和绝缘子区域与底层DNA建立物理接触的程度[5].可及性是动态的,对外界刺激和发育信号的响应的变化导致了不同细胞类型之间表达的显著差异[5,6].几种非编码RNA (ncRNA)对基因调控也有影响。微RNA (miRNA)抑制蛋白质翻译或诱导信使RNA (mRNA)降解,主要是通过与靶信使RNA的3 ' -UTR结合[7,8].此外,它们受DNA甲基化、组蛋白修饰和超过140种形式的RNA修饰的调控[9].反过来,miRNAs本身靶向表观遗传相关酶,如DNA甲基转移酶、10 - 11易位基因和组蛋白去乙酰化酶[9,10].长链非编码rna (lncrna)作为介导下游基因转录的信号分子,作为诱骗分子通过结合tf并阻断其调节活性来抑制生物过程和途径[11,12],或与mrna竞争miRNA结合[13,14].此外,一些基因,特别是调控蛋白如tf和miRNAs,在进化过程中经历了复制事件,导致基因冗余和/或随着时间的推移获得新的生物学功能[15,16].
RNA-seq、ChIP-seq和质谱等高通量技术在基因调控方面产生了大量高质量的数据。通过添加和集成新的数据类型和数据集,这些数据可以在不断增长的数据库中获得。一个例子是资源“判别正则表达式分析”(DORothEA) [17,18].它包含基于文献策划资源、ChIP-seq峰值、基于基因表达的推断和TF结合位点信息的签名TF-target相互作用[17,18].DORotheEA被嵌入到OmniPath数据库中,其中包括许多其他的相互作用类型,如mirna -靶标相互作用、lncrna -靶标相互作用、配体-受体结合和蛋白质-蛋白质相互作用[19,20.].HumanNet是一种捕获共功能和物理结合相互作用的人类基因网络资源:共功能网络(COF)包括共本质性和共表达相互作用,而蛋白质-蛋白质相互作用网络包含文献策划的和高通量的物理结合蛋白质相互作用[21].更多的数据库存在于不同类型的分子相互作用,它们的大小和数量不断增长。
为了深入了解基因调控,我们需要理解不同分子之间的相互作用如何协调表型特异性基因表达。事实上,一些研究表明,考虑互补的分子相互作用增加了我们对调控过程的理解。共表达基因和编码物理相互作用蛋白的基因通常由同一组tf调控[22,23].编码控制miRNA表达的tf的基因有更高的机会被miRNA转录后抑制[24].与tf调节的基因相比,miRNAs共同调节的基因显示出较弱的功能链接[25].基因调控中几个生物分子之间复杂多样的相互作用可以在基因调控网络(GRNs)的系统水平上建模。基于相关的高通量数据,grn绘制调控因子(主要是tf)及其靶基因之间的分子相互作用图谱,无论是否使用计算推理[26,27].整合grn考虑到基因调控中涉及的不同类型的分子相互作用[28].目前,还缺乏将多组学数据集成到这些grn中的熟练方法,以及对复杂的综合网络进行分析和生物学解释的方法。
生物网络通常很难作为一个整体来解释。它们拥有大量的节点和边缘,合并成一个巨大的“毛球”结构,这使得有意义的可视化和它们的功能解释极具挑战性[29,30.].为了解决这个问题,已经开发了许多方法。它们的共同原则是将这些毛球状结构分解为更小的可解释子网络,通常称为模块或社区。方法可以是基于共表达的、基于拓扑的、基于泛样本的和基于多边的,包括WGCNA、SimMod、ModulOmics和LemonTree等工具[31,32,33,34,35,36,37,38,39,40,41].WGCNA聚类具有高表达相关性的基因,并利用其模块本征基因对模块进行总结[31,32].SimMod使用混合整数非线性规划模型,将具有物理和遗传交互作用的基于wgcna的共表达网络集成到多组学社区[33].模块组学通过整合蛋白质-蛋白质相互作用、突变和拷贝数变化(CNVs)的互斥性、转录协同调控和共表达来识别新生癌症驱动途径和模块[40].其他方法通过基于共享GO注释的基因派生模块,将tf -靶基因相互作用和蛋白质-蛋白质相互作用与基于“功能到结构”的方法结合在一起[41].LemonTree在多次运行中推断共表达模块,将这些模块合并到共识模块中,最后使用多组学数据将这些模块连接到调控器,如tf、mirna或cnv [36].虽然现有的方法大大增加了大型多组学网络的可解释性,但仍存在一些挑战。一个常见的限制是派生模块的数量通常非常少,它们包含很多基因。大多数大型模块与生物学特性或表型相关良好,但缺乏详细的因果解释。此外,模块被解释为完全独立的实体,不共享任何基因。然而,当考虑多边网络的拓扑结构时,基因可能出现在不同的拓扑环境中,从而出现在不同的模块中。另一方面,考虑到存在许多可解释的小模块,跟踪模块之间的关系是至关重要的,以免错过更广泛的网络解释。因此,一种考虑网络拓扑、边缘因果关系和特定于条件的数据(例如表达式)的方法,同时在特定的网络上下文中生成小型和可解释的模块,可以在很大程度上补充现有的推理方法。
我们以前为蠕虫提出了一个数据集成框架秀丽隐杆线虫和植物拟南芥它将特定的分子相互作用分组在复合网络子图中,然后将这些聚类到与生物学相关的拓扑模块中,突出模块和调控器之间的连接,最后用基因表达谱覆盖这些模块,从静态模块到动态模块[28].我们了解到不同的分子相互作用在不同的拓扑模块中与特定的生物功能相互关联,从而在基因调控中产生协调的反应。在这里,我们将该数据集成框架扩展到基于子图的多组学聚类(SUBATOMIC)。SUBATOMIC从不同的交互数据库和基因表达谱推断基于复合子图的模块,并在更新的、通用的和自动化的分析框架中分析它们。在将复合网络分解为可解释的小模块后,我们在superview中跟踪调控器与模块之间以及模块之间的相互作用,以保存它们更大的网络环境,并促进生物解释。为了使静态网络模块具有动态性,并评估它们在特定条件下的作用,我们实现了模块活动评分。该分数可用于对模块在条件改变时的失调程度进行排名。该方法适用于任何用户定义的具有重叠节点的网络集合。
凭借其独特的方法,SUBATOMIC解决了网络模块化和多组学数据分析中的几个空白。超边缘聚类能够对网络中的特定拓扑特征进行选择,同时生成小的、易于解释的子网络。此外,还可以研究all模块设置中的所有拓扑特征。在分析模块之间以及模块与调节器之间的交互时,用户可以跟踪可能参与类似生物过程的其他模块和/或调节器,从而将模块嵌入到其全球网络环境中。自动化管道还有助于模块的功能注释及其在Cytoscape中的可视化探索。SUBATOMIC的这些特性极大地促进了多组学数据的生物学解释。虽然许多工具专注于特定的相互作用,如蛋白质-蛋白质相互作用网络,但我们的方法可以包含任何类型的独立于物种的定向和无定向相互作用。此外,SUBATOMIC还可以在整合特定条件数据(如转录组)的基础上探索动态网络,我们提供了几个指标来量化模块的特定条件活动。因此,利用多组学数据来研究特定的生物学问题是一个全面和多功能的网络分析工具。
我们应用SUBATOMIC集成了六个网络智人,分别基于TF-target相互作用、miRNA-mRNA相互作用、蛋白质-蛋白质相互作用、功能相互作用以及蛋白质和miRNAs的同源连接。推断出的模块使我们能够对注释不足的蛋白质提出功能假设。作为概念的证明,我们进一步将模块与低氧条件下癌细胞系的表达数据相结合。当细胞或组织没有足够的氧气供应以维持其内稳态时,就会发生缺氧(Gaspar和Velloso, 2018;Hiraga, 2018)。这种状态经常出现在肿瘤微环境中,导致细胞反应,增加转移风险,降低治疗的成功率[42,43].我们使用基于活度和GO项的特征来识别对缺氧条件敏感的模块。在我们的superview分析中,与随机对照相比,这些响应模块是高度连接的。我们强调了几个关于如何使用SUBATOMIC获得生物学见解的示例和指南。SUBATOMIC管道可在GitHub (https://github.com/CBIGR/SUBATOMIC).
结果
SUBATOMIC:基于子图的多组学聚类框架
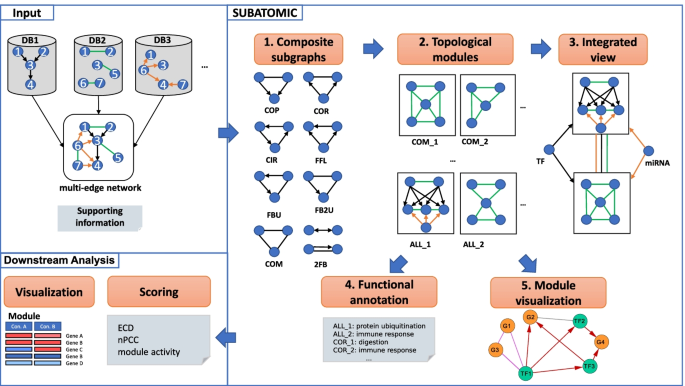
我们开发了SUBATOMIC,一种基于子图的多组学聚类框架,用于构建和分析多边缘网络。1).SUBATOMIC将由不同交互类型组成的网络作为输入。相互作用可以是定向的,如tf -靶标相互作用和mirna -靶标相互作用,也可以是无定向的,如蛋白质-蛋白质相互作用。网络需要有部分重叠的节点集,以允许在不同的交互类型上进行集成。对于多边网络,SUBATOMIC首先使用子图枚举算法ISMAGS将其分解为一组3节点的复合子图[44].此外,我们集成了一个自己的脚本来查找特定的2节点子图。子图是根据它们所包含的边的类型和方向进行分类的。通过在2节点和3节点复合子图中集成有向边和无向边,我们区分了8种不同的子图类型[28].在共指向子图(COP)中,一条无向边连接两个调节器,它们一起调节一个目标。共调控子图(COR)包含一个调控因子控制两个相互作用的靶基因。前馈循环(FFL)有一个直接调控靶基因的调控器和另一个同样调控靶基因的调控器。在循环反馈子图(CIR)中,监管机构通过反馈循环相互作用。反馈-无向子图(FBU)由级联中的两个有向相互作用组成,它们之间由一个无向相互作用连接。反馈二无向子图(FB2U)结合了两个无向相互作用和一个有向相互作用。复子图(COM)只包含无向边。最后,双节点反馈子图(2FB)将有向边与无向边耦合。
SUBATOMIC工作流概述。输入:SUBATOMIC将由不同交互类型的有向和/或无向交互组成的多边网络作为输入。支持信息可能包含额外的输入文件,如GO术语、基因注释和ISMAGS可用于专门筛选这些子图(方法)的子图定义列表。SUBATOMIC:(1)利用ISMAGS将多边缘网络分解为复合子图,分别为共指向(COP)、共调节(COR)、圆形(CIR)、前馈循环(FFL)、反馈无向循环(FBU)、反馈2无向循环(FB2U)、复数(COM)和2反馈(2FB)子图。(2)基于子图,SCHype生成每个子图类型以及所有子图类型(all)的拓扑模块。(3)模块之间相互连接,并与调节器连接,形成集成视图(进一步称为superview)。(4)模块使用GOATOOLS进行功能注释。(5)我们生成了可以导入到Cytoscape中进行模块可视化的文件。如果用这种方式分析的网络不包含任何特定的条件信息,则被认为是静态的。下游分析:这里的模块与特定于条件的表达式数据集成。 Several scores reflect the condition-specific activity of modules: the expression dynamicity score (ECD), the average Pearson correlation of expression values in a module (nPCC) and the module activity score
我们遵循ISMAGS命名法,通过特定的交互类型和边缘符号来表示子图[44].每一个特定相互作用类型的输入网络都被赋予一个特定的字母:R代表TF-gene, M代表miRNA-mRNA, P代表protein-protein, C代表co-functional, H代表同源相互作用。然后每个3节点子图根据其边的特定交互类型得到一个三字母表示。例如,PPP子图包含三条来自蛋白质-蛋白质相互作用网络的无向边,因此属于COM类型。RRP包含两个来自调节的tf -基因网络的边和一个来自蛋白质-蛋白质相互作用网络的边,因此属于COR类型。随后,通过基于超图的光谱聚类算法“超图中的光谱聚类”(SCHype)对所有子图进行聚类[45].SCHype在聚类过程中优化表示3节点和2节点子图的超边上的边节点比。由此产生的模块具有共同的拓扑特征,并具有特定的生物功能[28,45].SUBATOMIC使用SCHype首先在每种类型的子图(COM, CIR, FFL,…)中生成集群。此外,所有子图都聚集在一个名为“all”的模块类型中。我们进一步筛选包含5到50个基因的子图,与我们之前的方法相似[28].接下来,SUBATOMIC应用GOATOOLS对基于基因本体的模块进行功能注释[46].在这一点上,已经获得了小的和生物学上可解释的模块,但它们的网络环境还没有考虑。为了解决这个问题,我们将所有模块安排在superview中,superview将模块彼此连接起来,并找到连接到每个模块的调节器。SUBATOMIC还计算可导入Cytoscape的输出,用于模块网络可视化[47].作为后处理步骤,将拓扑模块与表达数据集成,研究不同实验条件下基因调控的动态。在这一步中,可以计算几个指标,以进一步表征特定条件下的模块并对其进行优先级排序。关于管道的更多细节可以在方法一节中找到。
综合人力调节网络
多组学数据整合有助于理解复杂疾病中的失调。我们的数据集成框架SUBATOMIC不仅利用了多组学网络,还连接了拓扑和功能信息,以利用它们的解释。在本研究中,我们旨在构建和分析多组学网络智人亚原子。因此,我们整合了来自公共资源的tf -靶基因、miRNA-mRNA、同源蛋白、蛋白-蛋白和协同功能相互作用,最终添加了来自肿瘤细胞系在缺氧条件下的表达数据。我们提供了一个如何使用SUBATOMIC来研究人类疾病环境中受干扰的基因调控的布局。
我们在分析中包括了五个不同的网络,涵盖了不同的相互作用类型,它们都影响基因调控(见表)1、方法和附加文件1).两个网络是有向的,并模拟调控关系:tf -靶基因网络(R)和miRNA-mRNA网络(M)。三个网络是无向的:同源网络(H),蛋白质-蛋白相互作用网络(P)和共功能网络(C)。R网络包括来自OmniPath三个不同子数据库的53,232个tf -靶基因相互作用:DoRothEA(级别A-C), tf -靶(管理评分> 1)和TF-miRNA相互作用[19,20.].M网络包括来自OmniPath的11085个miRNA-mRNA相互作用。为了包括基因同源性,我们包括了来自Ensemble档案的基因与miRbase中具有相同种子序列的同源miRNAs之间的10,847个同源相互作用[48,49].为了包含一层功能信息,我们从HumanNet v2中选择了两个互斥的网络[21,50].共功能边包括共本质、共表达和蛋白结构域关联边。24,773个物理蛋白质-蛋白质相互作用网络包含了高通量分析的边缘,如酵母双杂交和亲和纯化,以及文献策划的蛋白质-蛋白质相互作用。
超过一半的检测子图是特定于交互类型的
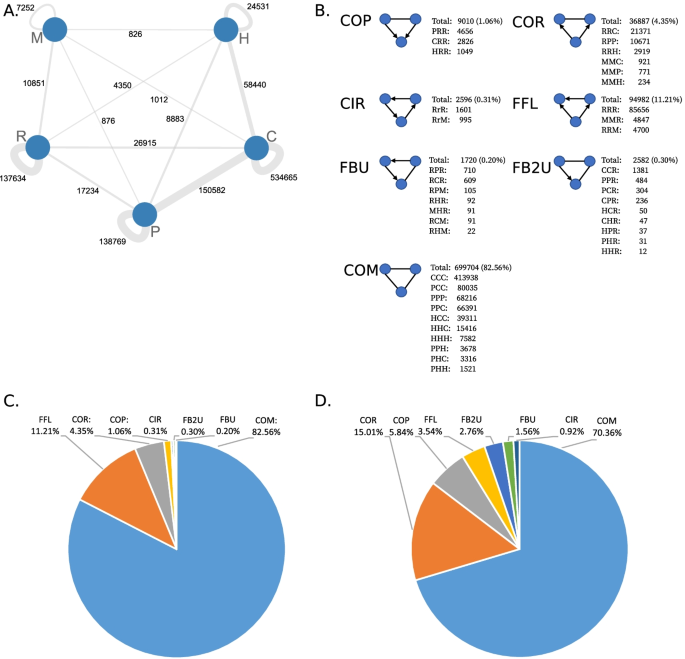
ISMAGS共检测到787,347个3节点子图。2).复合子图最为丰富,占复合子图的82.56%,其中以CCC(413,938 - 48.77%)和RRR(85,435 - 10.07%)最多,其次是PCC(800,035 - 9.43%)。虽然非com类型子图的总体比例可能很小,但罕见的子类型可以揭示有趣的机制见解。不同交互类型之间共享的子图比在一种交互类型中检测到的子图更不常见。共有534,665个子图包含至少两个共功能相互作用,而150,582个子图包含至少一个共功能和一个蛋白质-蛋白质相互作用。共功能网络和调节性tf -基因相互作用网络的边缘子图有26,915个出现。两边同类型的子图数量最少为7252,来自miRNA-mRNA相互作用,与同源网络共享的子图数量也最少(826)。而deforort等人所使用的交互类型和数量答:芥而且秀丽隐杆线虫虽然略有不同,但我们得到了类似的结果,其中复杂子图最为丰富,而只包含蛋白质-蛋白质和同源相互作用的子图的子图数量最高[28].
接下来,SUBATOMIC使用SCHype将复合子图聚类为7个模块类型。为我们的智人综合网络,共5586个模块(2762 ALL, 1987 COM, 424 COR, 165 COP, 100 FFL, 78 FB2U, 44 FBU, 26 CIR)。COM、COR、COP、FFL、FB2U、FBU和CIR是在互斥的子图类型上生成的,而ALL包含了所有类型的联合聚类,并允许发现不同拓扑模块之间的相互作用。因此,ALL模块是最丰富的,其次是COM和COR模块。CIR模块是最少的。关于基因调控的背景,COR、COP、CIR和FFL模块是最有趣的,包含定向调控相互作用。
大多数模块紧密相连,并受多种转录因子和mirna调控
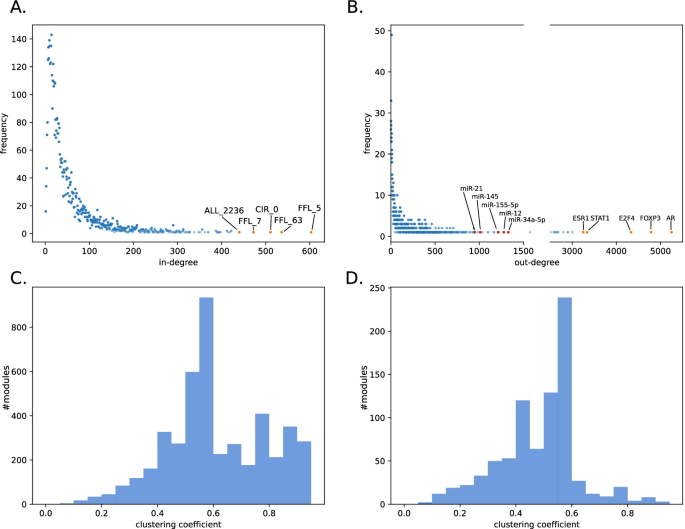
在计算模块时,我们在所谓的“superview”分析中跟踪它们更大的网络上下文。这包括模块之间的相互作用,以及与调控因子如mirna和tf的相互作用。我们首先分析了调节器的特异性,看看它们针对多少模块。这可以让我们深入了解监管者是可以被视为大师监管者还是特定监管者。在我们的分析中,我们总共包含了526个tf和850个miRNAs。
平均而言,TF靶向6%的模块,miRNA靶向2%的模块。共有25个tf调节5个或更少的模块,而5个tf只针对一个模块。在miRNA调控因子中,90种调控5个或更少的模块,19种仅调控一个模块。平均每个模块被33个tf和18个miRNAs靶向。然后,我们绘制了调节器-模块相互作用的输入度和输出度的度分布(图。3.A和B)。大部分模块和调节器的度数较低。五个最高度模块是FFL, CIR和ALL类型。对于监管者来说,高学位可以被理解为是监管者大师的标志。进一步,我们绘制了所有模块的聚类系数的分布(图。3.C).我们检测到聚类系数分布的模式为0.55-0.6,大部分模块的连通性高于该模式。由于模块中除了COM模块和ALL模块外,COM模块占据了强大的优势,所以现在大多数模块的连通性都低于模式(图5)。3.D).由于共功能网络和蛋白质-蛋白质相互作用网络高度互联,其聚类系数也很高。
调节器-模块网络的特定网络属性。一个入度和B调节器与模块之间相互作用的出度分布。前5个模块、转录因子和miRNAs都有标签显示。在B我们打乱了1500到2500之间的轴线。C各模块的聚类系数分布。模态分布在0.55 ~ 0.6之间。大多数模块的连通性高于模式。D除COM和all模块外,所有模块的聚类系数分布。大多数模块的连通性低于模式。在附加文件中给出了各个独立模块的聚类系数的可视化6
模块功能富集分析揭示未知基因功能
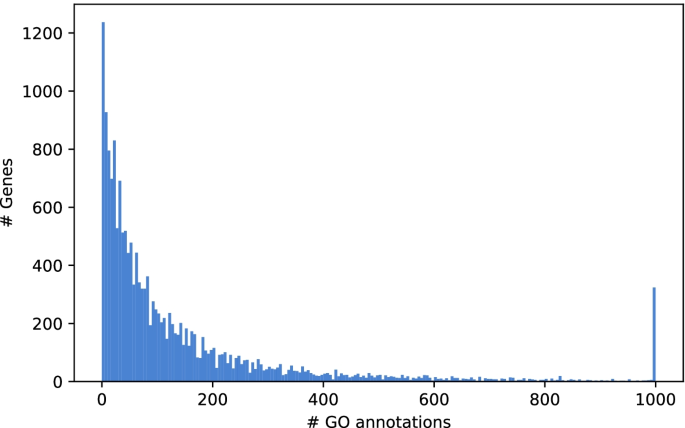
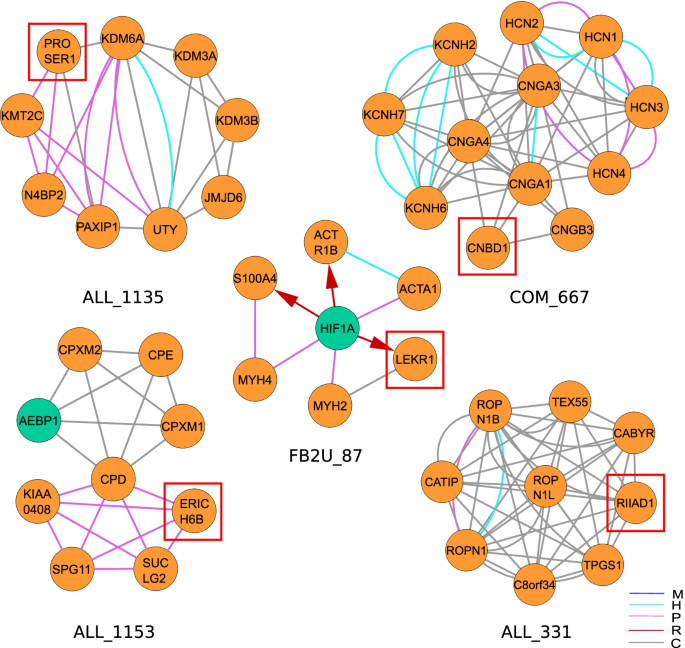
通过对功能GO本体的丰富分析,3805个模块获得了丰富的GO术语列表。我们可以利用这些功能注释,为那些基于“因关联而内疚”或“因重新连接而公会”原则未被很好描述的基因生成关于基因功能的假设[51,52].虽然我们的大多数基因智人有1404个基因的GO术语少于5个,345个基因根本没有GO术语(图1)。4).将我们的研究局限于蛋白质编码基因,我们发现模块中有53个基因只标注了两个或更少的GO术语,进一步被称为“弱特征基因”,其中25个基因根本没有GO术语。(未注释基因及其模块上下文的完整列表见补充)。这些基因中有几个存在于注释良好的模块中,我们可以预测它们的生物学功能与GO注释和模块结构的关系。我们选择了5个目前没有GO注释的基因进行进一步分析:富含脯氨酸和丝氨酸的蛋白1 (PROSER1)、含有环核苷酸结合结构域的蛋白1 (CNBD1)、富含亮氨酸、谷氨酸和赖氨酸的蛋白1 (LEKR1)、含有RIIa结构域的蛋白1 (RIIAD1)和富含谷氨酸的蛋白6B (ERICH6B)(图1)。5).PROSER1基因(ENSG00000120685)出现在ALL_1135、ALL_1880和ALL_2888模块中。后者由八个基因组成的蛋白质-蛋白质复合体。富集前5名分别是MLL3/4复合体、组蛋白甲基转移酶活性(H3-K4特异性)、Set1C/COMPASS复合体、组蛋白H3-K4甲基化和组蛋白甲基转移酶复合体。组蛋白甲基转移酶复合体GO术语由8个模块基因中的6个共享。PROSER1与组蛋白甲基转移酶KMT2和已知参与组蛋白H3-K4甲基化的PAXIP1直接连接[53].因此,我们假设PROSER1是组蛋白甲基化复合体的一部分。经过我们的分析,最近发表的一项工作证实了PROSER1s参与调节各种染色质相关蛋白[54].接下来,我们分析了CNBD1 (ENSG00000176571)。该基因出现在ALL_654和COM_667模块中。COM_667中富集前5位的项分别为:HCN通道复合物、细胞内camp激活的阳离子通道活性、细胞内环核苷酸激活的阳离子通道活性、细胞内环核苷酸激活的阳离子通道活性和环核苷酸门控的离子通道活性。它通过共功能边与CNGB1、CNGA1、CNGA3和CNGA4连接。这四个都是环核苷酸门控通道(CNGA)的亚基,除了HCN通道复合物外,出现在所有富集的前5个术语中。因此,我们假设CNBD1也是阳离子通道复合体的一部分,具有阳离子通道活性。LEKR1 (ENSG00000197980)基因出现在ALL_3385和FB2U_87模块中。而ALL_3385在FBU_87中富集前5位的词分别为细胞成分肌凝蛋白复合体、肌动蛋白复合体、肌凝蛋白丝、肌凝蛋白II复合体和肌节。它与MYH2连接,MYH2也被注释了除dynactin复合体以外的所有重要术语。 While we cannot derive a specific function for LEKR1, we can hypothesize that it plays a role in the myosin complex. The ERICH6B (ENSG00000165837) gene appears in ALL_1153 and COM_1252. The top 5 enriched terms are the molecular functions metallocarboxypeptidase activity, carboxypeptidase activity, metalloexopeptidase activity, exopeptidase activity, as well as the biological process protein processing. It has protein–protein interactions with four proteins, of which the carboxypeptidase D (CPD) is part of all enriched GO terms in this module, and the succinate–CoA ligase SUCLG2 is part of the cellular amide metabolic process. Although the function remains rather broad, we can hypothesize that this gene is involved in the amide metabolic process. Finally, the RIIAD1 (ENSG00000178796) gene appears in the modules ALL_331 and COM_380. The top 5 enriched terms in ALL_331 are sperm capacitation, sperm motility, flagellated sperm motility, cilium movement involved in cell motility and cilium or flagellum-dependent cell motility. Five out of nine genes are annotated with a cellular component of the motile cilium. We can hypothesize that RIIAD1 is involved in sperm motility. In fact, a recent paper mentioned RIIAD1 as co-expressed with the a-kinase anchor protein 3AKAP3, a gene whose knockdown was shown to induce infertility in male mice [55,56].在附加文件中可以看到前30个丰富的术语的可视化和GO术语的概述2而且6.
在三种癌细胞系中,动态模块与缺氧有关
缺氧可导致各种不同的反应,其中细胞可对严重的组织损伤产生耐受性,进而可能促进侵袭性癌症表型[57,58].这种受损细胞可嵌入肿瘤微环境中,影响治疗效果[58,59].为了将SUBATOMIC获得的结果背景化,我们选择了一项研究,研究了循环和慢性缺氧对黑色素瘤(WM793B)、卵巢癌(SK-OV-3)和前列腺癌细胞系(PC-3)基因表达的影响[58].慢性缺氧的特征是永久性的氧消耗,在研究中模拟的环境氧浓度为1%。在循环缺氧条件下,氧气的可用性在1 - 21%之间变化,在6个不同的时间点有一个开关。恒氧浓度为21%作为对照条件。我们选择了两种主要的优先排序方案来选择可能涉及缺氧的模块:一种基于丰富的氧化石墨烯术语,另一种基于表达数据。
我们首先根据至少一个包含“缺氧”关键字的GO术语的浓缩(附加文件3.).因此,我们确定了78个模块,进一步引用为“缺氧GO集”。然后,我们研究了这组模块在父视图分析中的紧密连接程度2).我们使用所选集合中模块之间的交互数量,并将其与1000个随机选择的相同大小的模块集合的背景进行比较。这让我们可以看到“缺氧GO集”中的模块是否比预期的连接得更紧密。我们计算了随机集的均值和标准差的95%置信区间的上边界,以及“缺氧GO集”与该上边界之间相互作用的折叠变化。我们观察到,“缺氧GO集合”的模块在该集合内的模块对之间的边大约是随机模块的18倍。特别是这些模块之间的调控边缘,如TF-target相互作用和miRNA-mRNA相互作用,被观察到的概率分别是预期的21倍和19倍。这揭示了这些模块之间的强烈联系,表明缺氧背后有复杂的调节机制。接下来,我们研究了在慢性缺氧中,与其他模块相比,“缺氧氧化石墨烯组”是否富集了显著差异表达(DE)基因,且最小变化大于2倍。我们没有将此应用于循环缺氧,因为DE基因的数量相当低。用超几何检验将出现在“缺氧氧化石墨烯组”中的DE基因与出现在所有其他模块中的DE基因进行比较。 We found a significant overexpression of DE genes in all the cell lines: WB793B (fold change 16.99,p-value 3.54E-38), PC3 (fold change 9.58,p-值1.47E-36)和SK-OV-3(倍数变化7.74,p价值1.43 e-30)。这些结果突出表明,通过结合superview分析和函数注释,即使不需要表达式数据集成,我们也可以筛选特定于条件的模块。
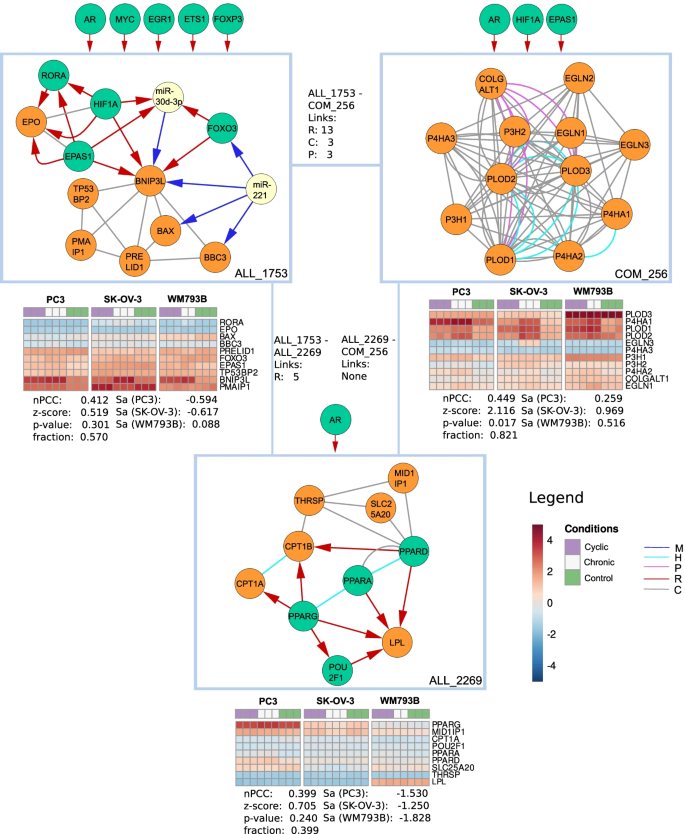
随后,我们仔细观察了“缺氧氧化石墨烯组”中的三个选定模块。6).例如,模块COM_256类似于一个共功能蛋白质复合体,它有68个富集的氧化石墨烯项,包括最富集的羟赖氨酸代谢过程、肽基脯氨酸4-双加氧酶活性以及l-抗坏血酸结合(参见完整的富集氧化石墨烯项列表的补充)。12个基因中的5个参与了对缺氧的反应以及对氧含量降低的反应。该模块主要由三种转录因子调控:雄激素受体(AR)、缺氧诱导因子1亚基α (HIF1A)和内皮PAS结构域蛋白1 (EPAS1),也称为缺氧诱导因子2- α。已知HIF1A和EPAS1可促进细胞对缺氧的适应,并调节多种组织中许多与缺氧相关的基因[60,61,62,63,64].此外,研究表明AR作为配体依赖的tf,在缺氧条件下对AR靶向的癌症治疗产生耐药性[65,66].许多模块基因差异表达,即前胶原赖氨酸,2-氧戊二酸5-双加氧酶1和2 (PLOD1, PLOD2),脯氨酰羟化酶(EGLN3),以及脯氨酰4-羟化酶亚基α 1和2 (P4HA1)和(P4HA2)。研究表明,PLOD1和PLOD2参与了缺氧诱导的转移和胶质母细胞瘤的肿瘤进展[63,67].SK-OV-3细胞系在慢性状态下上调EGLN3,催化缺氧诱导因子(HIF)的氧依赖性羟基化[68,69].其他基因,如脯氨酸3-羟化酶P4HA1和P4HA2,已知羟化HIF α-亚基中的564-脯氨酸残基[70].因此,我们得出结论,COM_256强烈参与了对缺氧的反应,并在三个细胞系中表现出一致但略有不同的表达。
然后我们研究了ALL_1753模块。该模块包含177个显著的氧化石墨烯长期富集,包括6个基因的细胞对缺氧的反应和细胞对低氧水平的反应。它以BCL2相互作用蛋白3样基因(BNIP3L)为中心,该基因被HIF1A和EPAS1差异表达并靶向。该模块包含三个有趣的前馈循环,其中HIF1A、EPAS和叉头盒O3a FOXO3靶向BNIPL3和miR-30d-3p,反过来也调节BNIP3L [73].此外,已知miR-30d-3p参与缺氧并直接调节AR [71].FOXO3在应对缺氧应激时被激活[72].该模块中的另一个DE基因是视黄酸受体相关孤儿受体(RORA),受HIF1A和EPAS1调控。已知RORA是由HIF1A诱导的,并在HIF1A的核积累中发挥作用[74].最后,miR-221调节FOXO3、BNIPL3、bcl-2结合成分3 (BBC3)和凋亡调节因子BAX,并在缺氧-再氧损伤中发挥细胞保护作用[75].因此,我们得出结论,ALL_1753强烈参与了对缺氧的反应,BNIPL3的缺氧反应可能是由三个调节前馈循环的参与驱动的。
最后对ALL_2269模块进行了研究。该模块富集了85个氧化围棋术语,包括两个和三个基因的最富集术语肉碱穿梭和肉碱o -棕榈酰转移酶活性,以及涉及一半模块基因的脂肪酸代谢过程的正调控。由于三个模块基因的存在,对缺氧和氧含量降低的反应增强了。该模块的中心是三个同源过氧化物酶体增殖物激活受体,PPARG, PPARA和PPARD。特别是在肺癌和肝细胞癌中,PPARG在低氧条件下被激活,与HIF1A相关[76,77].它调节差异表达基因肉碱棕榈酰转移酶1A (CPT1A)及其同源物CPT1B,被证明在低氧条件下调节前列腺癌的生长[78].虽然该模块在三种被分析的癌症类型中没有表现出强烈的失调,但它包含了与缺氧反应高度相关的基因和相互作用,如其他研究所证实的那样。
所有三个模块都由父视图中的许多边连接,并共享一组类似的调节器。我们证明,基于GO术语的方法发现的模块与缺氧环境高度相关,这得到了缺氧特异性DE基因数量增加的支持。在模块中,我们确定了前馈循环等调节结构,包括来自互补组学层的相互作用,这有助于解释和解释观察到的表达,并允许生成缺氧诱导机制的机制假设。
在第二种优先排序方法中,我们希望使用基因对刺激或条件的动态响应作为选择标准(附加文件4).我们实现了一个“模块活动”\ ({} _ {} \)可以捕捉模块对变化条件的响应的方法,例如基于两个条件之间的差异表达式数据[79(请参见方法)。为了找到一组高度缺氧相关的模块,我们过滤了具有积极活动评分的模块\ ({} _ {} \)在所有三个细胞系中。这导致了一组52个模块,我们进一步称为“缺氧活动集”。接下来,我们用与“缺氧GO集”相同的方法分析ALL模块中的superview连接(表3.).我们观察到,与相同大小的随机集合相比,活动模块之间的连通性要高28倍。虽然m型和r型相互作用的数量与“缺氧GO组”相似,但无向相互作用类型H、C和P要丰富得多,分别有90、69和88倍的连接数量。此外,“低氧GO套装”和“低氧活动套装”有12个共同模块。
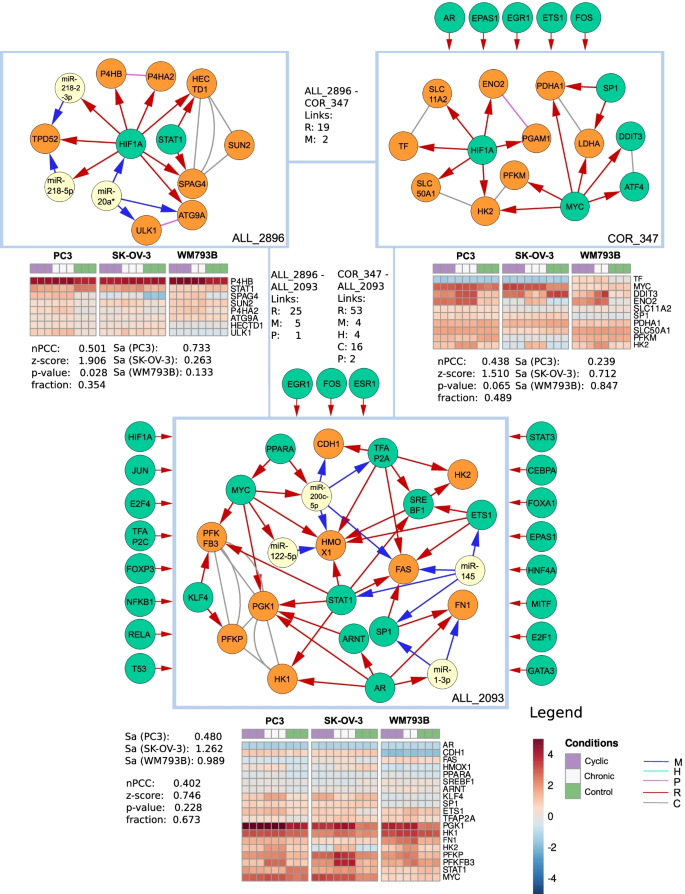
然后,我们检查了活动缺氧集中确定的三个模块,它们与缺氧的相关性(图。7).
COR_347模块以HIF1A和MYC为主,各调节6个靶点。该模块包含159个显著的氧化石墨烯长期富集,分别包括4个和3个基因的细胞对缺氧的反应和细胞对低氧水平的反应。除了HIF1A和MYC原癌基因(MYC)外,该模块还受”丛书原癌基因(FOS)与六种调控相互作用。MYC是所有模块的46%的主调节器之一。它在癌症的发展中发挥重要作用,调节缺氧诱导因子蛋白家族成员[80].己糖激酶2基因(HK2)受MYC和HIF1A调控,在表达数据中表现出失调。它最近被证明是HIF1A在氧还原环境中的一个重要靶点[81].
ALL_2896主要包含以HIF1A为主的COR和FFL子图。富集了11个GO注释,包括前胶原脯氨酸4-双加氧酶活性和肽基脯氨酸4-双加氧酶活性。HIF1A通过与脯氨酰基4-羟化酶β多肽基因(P4HB)共同调控差异表达基因P4HA1,该基因也发生在“缺氧氧化石墨烯组”的COM_256中。该模块中的另一个调控因子是转录1基因(STAT1)的信号换能器和激活器。它与HIF1A一起调控精子相关抗原4 (SPAG4)。虽然SPAG4的差异表达没有超过2倍的变化,但我们仍然观察到所有三个细胞系的表达一致减少。
ALL_2093富集了331个GO项。最丰富的词条包括后肾帽间充质细胞增殖调控和白细胞与动脉内皮细胞粘附负调控,但最丰富的15个词条仅包含一个基因。对缺氧和氧含量降低的反应包括6个基因。该模块主要由COR和FFL子图组成。许多与缺氧相关的基因已经出现在上述模块中,如AR、MYC、HK2、STAT1和PPARA。另一个有趣的基因是krüppel-like因子4 (KLF4)。它与靶标6-磷酸果糖-2-激酶/果糖-2,6-双磷酸酶3 (PFKFB3)一起差异表达,PFKFB3反过来也受STAT和MYC调控。KLF4参与缺氧诱导的血管平滑肌细胞迁移[82].然而,PFKFB3在PC-3和SK-OV-3细胞系中表达差异,而KLF4仅在PC-3中显著过表达。除了通过STAT1和MYC调控外,PFKFB3启动子还包含HIF1A结合位点,可能不依赖KLF4作为激活子在缺氧条件下过表达[83].此外,纤维连接蛋白1 (FN1)过表达,并受AR、SP1和miR-1-3p两种ffl调控。后者在小鼠肺组织缺氧条件下被发现下调[84].特异性蛋白1 (SP1)也被描述为直接受HIF1A调控,是其他下游基因缺氧诱导转录所必需的[85].
模块的稳定性分析
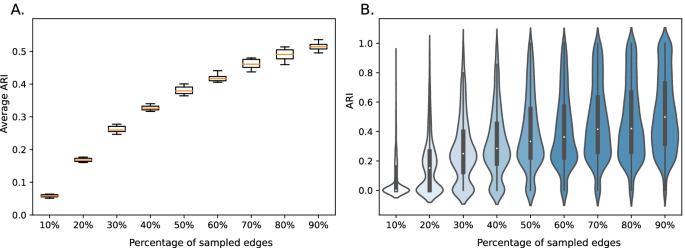
为了评估模块关于缺失边缘的稳定性,我们设计了一种重新采样方法[86].假设以整个网络为基础,我们从交互集合中采样90%、80%、……和10%的边,不进行替换,并在每个减少的集合上重新运行SUBATOMIC管道。然后,我们从全网络的模块中去除未采样的边缘,创建一组地真值模块,并根据减少的网络计算模块重叠。这允许我们量化模块的稳定性与缺失的边有关。为了解释抽样过程的随机性,我们将每个抽样重复十次。我们观察到,在这样的重采样量下,我们得到了一个关于方差的稳定输出。我们使用了三种不同的指标来比较集群与它们的地面真相:Jaccard指数(JI)、调整后的Rand指数(ARI)和调整后的互信息(AMI) [87,88,89].对于我们的下采样集中的每个模块,我们检索了与基础事实相比较的最大分数,并在每个集的箱线图中可视化了平均值(图1)。8一个额外的文件8).这样,与全网相比,在合并或拆分模块时,只考虑评分最高的模块。虽然分数在保持90%、80%和70%边缘之间相对稳定,但边缘被删除的越多,分数下降得越快。在所有三个评分指标中都观察到了这种趋势。由于方差在运行之间是稳定的,然后我们为每个向下采样选择10个运行中的一个,并在小提琴图中可视化分数的分布(图1)。8B).我们观察到,当添加更多的边时,高分的数量普遍增加。与箱线图类似,我们可以看到90%、80%和70%边缘的小提琴彼此非常相似。当使用AMI或JI作为度量进行比较时,我们观察到类似的结果。综上所述,我们的方法表明,在不考虑相互作用的情况下,模块是相对稳定的,并且在这个过程中会形成类似的模块。我们进一步分析了为缺氧数据集计算的nPCC值(附加文件8).所有簇中约24%的模块间基因表达存在显著相关性,进一步表明衍生的SUBATOMIC模块分离良好,且有正交表达数据支持。
讨论
我们开发了SUBATOMIC,这是一种集成管道,可以将多组学网络分解为拓扑模块及其相互作用,使用复合子图聚类、统计和泛函分析。获得的模块被进一步嵌入到它们的网络和调控器上下文中,并在superview分析中进行功能注释和可视化。在后处理步骤中,我们将获得的模块与三种缺氧细胞系的特定条件数据相结合,并计算活性和表达相关性评分。与我们之前的集成框架相比,SUBATOMIC包含了主要改进[28].最重要的是,我们自动化了工作流,并将其集成到一个通用的Snakemake管道中。虽然前一个版本仅为一个用例生成,但我们现在可以在一次执行中执行从分解复合网络到分析模块的分析的分析工作流。此外,它适用于任何物种的多边网络。我们调整了superview分析,以输出模块和调节器之间连接的摘要。我们还通过自动化的GO术语充实来改进模块的功能描述。由于在大型交互网络上运行时可能是一个问题,我们并行化了管道中时间关键的步骤,提高了可伸缩性和可计算性。此外,我们添加了脚本来支持Cytoscape中模块的可视化。为了计算模块的特定条件活性,我们开发了一个计算生物活性评分的后处理步骤。
我们将SUBATOMIC应用于由人tf靶基因、miRNA-mRNA、蛋白质-蛋白质、协同功能和同源相互作用组成的复合网络。我们的方法获得了5586个模块。大部分模块的GO项得到了丰富,说明模块拓扑与生物功能密切相关,因为对拓扑特征进行了聚类。得到的大部分模块是COM模块,由无向边组成。这是意料之中的,因为我们包含了更多的非定向交互。此外,我们观察到,在大多数情况下,三节点子图中的交互更多地来自同一输入网络,而不是来自不同的网络。这是由于输入网络并不拥有完全相同的节点集,也可以有不同数量的交互。我们没有使用完整的HumanNet数据库,而是只包含对数可能性评分大于3分的高质量交互。这通过设置质量值的临界值来平衡交互的数量。
给出模块及其功能注释,我们演示了如何使用SUBATOMIC预测未注释基因的功能。在推测的模块中也出现了53个弱注释基因,我们选择PROSER1, CNBD1, LEK1, RIIAD1和ERICH6B进行深入分析。虽然我们的一些预测,如PROSER1的染色质修饰作用是非常新颖的,但其他的预测得到了最近的出版物的支持,如RIIAD1可能参与精子运动。然而,基于关联行行原则的功能表征产生的指导性功能假设仍然需要实验构象。
在上下文化模块的情况下,我们提出了两种优先排序策略:通过丰富相关GO术语或通过模块活动评分。我们发现,共享氧化石墨烯术语的模块,如缺氧,是紧密相连的,并积累了相关条件的氧化石墨烯基因。此外,由于一个模块内部距离近,没有标注特定GO术语但与该条件相关的基因也可以被检测到。然而,当基因没有很好地描述或确实缺乏特定的GO关键词将它们与某个条件联系起来时,基于GO术语的优先排序策略可能会错过重要的模块。通过实现基于差异表达式数据的模块活动,我们进一步开发了一个更受数据驱动的优先级排序方案。使用活动评分,我们发现了一小组与缺氧环境相关的模块。这组基因与基于GO术语的基因组紧密相关,并有部分重叠。虽然模块本身显示了静态视图,并给出了生物体中可能发生什么的见解,但上下文化增加了特定于条件的动态性。例如,尽管在缺氧研究的表达阵列的最新探针注释中,主要驱动缺氧的基因之一HIF1A并不存在,但基于GO术语注释和活性,它在许多模块中被检测到。特别是HIF1A靶向的基因在含有HIF1A的模块中表现出高差异表达。 Our results indicate that we can prioritize the large amount of modules in different ways to end up with sets of modules highly relevant for a condition or disease context. With the activity prioritization method, we were able to find modules with different topologies strongly connected in the superview. We showed that many genes in these modules are already known to play important roles in hypoxia. Moreover, contextualizing the modules with expression data from three different cell lines revealed that the activation of response mechanisms can differ and that different parts of a module can be active in different tissues. For example, EGLN3 is a known hypoxia-induced factor and showed dysregulation only in the SK-OV-3 cell line (see COM_256). In ALL_2093, KLF4 is only weakly expressed in WM793B while its target PFKB3 is strongly expressed in all three cell lines under hypoxic conditions; thus other regulators such as MYC and STAT also targeting PFKB3 might have a stronger regulatory influence. Overall, the combination of annotated SUBATOMIC modules based on different topologies, their superview connections, their contextualization with expression data and their visualization among different conditions and cell lines delivers a versatile tool to deeply investigate multi-edge networks.
SUBTATOMIC不局限于本研究中描述的数据类型,还提供了额外的分析机会。虽然我们的分析仅限于基因和miRNAs,但只要输入网络共享一组用于交叉的公共节点,就可以添加任何类型的节点和相互作用,如代谢产物相互作用、lncRNA相互作用或siRNA相互作用。此外,虽然我们使用的静态先验网络在后面的步骤中被上下文化以增加动态性,但通过包括从上下文特定的高通量数据(如转录组或蛋白质组)推断出的条件或患者特定的关联网络,例如在大块或单细胞水平上的共表达网络,可以直接分析动态复合网络。此外,还可以采用混合方法,将某些交互类型的公共数据库与其他交互类型的特定于条件的交互结合起来。
为了从聚类的角度评估我们的模块,我们设计了一种重新采样的方法,在10次重复中随机删除固定数量的边,用于9个不同的采样分数(10-90%)。我们表明,当删除少量的边缘时,模块保持稳定,但当删除更多时,模块变得越来越不稳定。当将抽样网络的模块与完整网络进行比较时,使用三种不同的度量方法证实了这一趋势。由于模块受到输入先验网络的大小和完整性的强烈影响,分析还表明,当添加更多的边时,稳定性存在饱和效应,因为相邻采样点之间的平均ARI差异随着添加更多的边而减小。然而,并不是每个模块都可以在次采样模块中找到。这是意料之中的,因为有些边可能是连接模块的两个部分所必需的,当删除模块时,模块可能分裂成两个。由于聚类是在双节点和三节点超图上完成的,所以节点可以出现在几个模块中。为了避免比较不相关的模块,我们决定只包含ground truth和subsampling模块之间的最佳成对ARI来评估聚类稳定性。此外,大量nPCC值显著的模块以及大量GO项显著丰富的模块支持了稳定性。因此,我们可以得出这样的结论:SUBATOMIC以稳定的方式预测模块。
宽泛地说,当涉及到模块推断时,可以做出两个主要区别:一方面,方法直接从实验读出(如表达式数据)生成模块,或者使用先前的网络作为推断的基础。另一方面,聚类可以只基于一种数据模式,也可以包含多种数据模式。WGCNA、lmQCM、MiBiOmics和TPSC等方法是生成基于表达的共表达集群的方法[38,90,91,92].虽然它们被广泛使用,并显示出与许多生物特征相关的模块,但这些模块通常非常大,没有考虑因果调节相互作用,或没有多组学数据集成。其他方法则更进一步,额外整合了蛋白质-蛋白质相互作用[41,93,94].Dittrich等人的方法将表达数据的聚类与蛋白质-蛋白质相互作用网络相结合,从而衍生出代表合并、重叠和独立的群落的模块[95].虽然在独立、重叠和合并的社区中对模块的分类已经为集群提供了一些网络上下文,但它在输入数据方面没有那么灵活,而且没有考虑交互的方向性,集群只产生了少量的模块。多组学模块分析方法(MOMA)使用深度学习方法派生特定于组学的模块表示,这些模块在注意层中进一步集成,以寻找疾病预测的相关模块[96].稀疏多典型相关网络分析(SmCCNet)派生出一些大型模块,将组学测量与特定表型联系起来[97].另一类方法尝试使用静态先验网络作为基础,并在上下文化步骤中使用表达式数据来查找活动子网络[97,98,99].例如,连接分离连接组件(C3)通过迭代连接由少量疾病相关蛋白组成的子网络,将网络模块化为与疾病相关的模块[One hundred.].解除管制网络将先前的管制网络与组学丰度测量相结合,以识别最大限度解除管制的子网络[101].其他作者对这些方法和策略进行了进一步的评述[39,102,103,104].与现有方法相比,SUBATOMIC尝试解决开放问题,并创建了一个全面的分析框架,涵盖模块推断的各个方面(参见附加文件)8进一步显示了这里提到的模块推理方法和SUBATOMIC之间的特性的表格比较。)
它基于一种拓扑聚类方法,允许解释基因之间的子句关系,并强调不同的调节机制。它引入了灵活性,允许对文献定义的先验网络和组学派生的关联网络进行操作。此外,它可以以综合的方式包括所有类型的节点和生物相互作用。网络被划分为大量具有区域拓扑属性的可解释的小模块,同时仍然跟踪它们的全局网络上下文和调节器。此外,静态网络模块可以通过使用ECD、nPCC和模块活动评分来推断和上下文化表达数据。这种结合使其成为复合网络聚类领域中一种独特而杰出的方法。
虽然SUBATOMIC被证明能够回答许多生物学问题,但它也有一些局限性。输入先验网络通常是不完整的,可能会使生物学解释复杂化。然而,我们预计交互数据库将不断增长,从而多边缘网络将变得越来越完整,我们的稳定性分析进一步证明,当遇到缺少交互时,模块仍然是稳定的。此外,如果网络对于一定数量的节点没有重叠,派生的子图将主要由分离的交互类型组成模块。这个问题也会随着数据库数量的增加而得到解决。另一个限制是可计算性。我们使用SCHype作为我们的聚类算法,并证明它能够在足够的时间内处理超过750,000个子图。然而,随着网络的增大,检测到的子图数量会超线性增长。因此,网络规模存在一个可计算的上限。此外,当我们并行化GOATOOLS来一次注释几个模块时,计算速度的提高伴随着空间消耗的增加。 This limits the number of cores that can be used for parallelization. This will be addressed in a future version integrating a more space-efficient annotation tool.
结论
总之,我们开发了一个自动化子图聚类框架,它采用交互的基本构建块,并将它们聚为模块。通过超视图计算、调节器分析、GO术语丰富和模块活动评分,进一步对模块进行特征描述和上下文化。SUBATOMIC可以用于研究条件和疾病,发现功能相关模块之间的相互作用,并为未特征基因推导新的基因功能。主要的限制因素是互联网络的可用性。我们相信,随着越来越多的相互作用被发现,这个问题将及时得到解决。我们的方法区别于其他模块推理方法,它基于拓扑特征聚类,创建大量具有不同规则属性的小型、容易解释的模块,同时仍然考虑到整个网络上下文。
方法
主要的分析工作流程已经集成到一个Snakemake管道中。附加文件中提供了Snakemake工作流图7,其示意图概述见图。1.所有软件,包括一些用于预处理和后处理分析的脚本,以及Docker版本都可以在GitHub (https://github.com/CBIGR/SUBATOMIC).
子图检测
对于子图检测,我们使用了“基于索引的子图算法”(ISMAGS) [44,105].我们遵循了ISMAGS中使用的子图表示,其中三个节点的子图表示为三个字母的代码,它指定给定的边源自某一组输入交互。一个输入网络中的交互需要是全有向的或全无向的(例如,一个TF-target网络中的所有交互都应该是有向的;蛋白质-蛋白质网络中的所有相互作用都应该是无定向的……)每个输入网络都有一个独特的单字母表示,用户可以自由选择。例如,子图RRP将包含来自网络R的一条有向边,来自网络R的另一条有向边,以及来自蛋白质-蛋白质相互作用网络的一条无向边。有向网络的字母可以设置为小写,表示方向相反(参见ISMAGS论文[44])。由于对称,一些子图彼此之间是冗余的(例如,PPC, PCP和CPP代表同一个子图)。一个定制脚本基于提供的有向和无向网络字母列表计算非冗余子图表示集。然后,管道使用这个集合作为搜索子图的指南,用户可以进一步对其进行微调,以删除额外不需要的子图。然后用ISMAGS识别三节点子图[44].ISMAGS每次迭代都使用三个字母的子图表示和复合网络中包含该子图的交互的部分。它输出满足定义表示的所有三节点子图。除了三节点子图,我们还搜索了具有特殊属性的双节点子图:即,所有节点对,其中每个节点包含指向另一个节点的有向边(DD-type),以及所有由一个无向边和一个有向边(DU-type)连接的节点对。
子图聚类与模块推理
ISMAGS生成的子图随后被分组为以下子图类型之一:复子图(COM)、前馈回路(FFL)、共指向子图(COP)、共调节子图(COR)、循环反馈子图(CIR)、反馈无向子图(FBU)和反馈2无向子图(FB2U)和双节点反馈子图(2FB) [28].每一种子图类型都由有向和无向边的独特组合所表征,如图所示。1.例如,COM类型包含所有专门由无向边组成的子图。给定无向网络字母C, P和H,这三个字母的任何组合,导致一组非冗余子图被分组到COM类型。每个模块类型都由特定的子图类型组成,并作为以下集群的输入。
我们使用SCHype算法分别推断上述每种定义的子图类型的聚类,以及所有类(all)的并集[45].该算法基于Perron-Frobenius定理,对超图进行聚类,通过在网络的每个聚类中最大化边节点比来解决优化问题[106].输入是一个超图,每个超节点代表一个由ISMAGS计算的三节点子图或一个双节点子图。SCHype在默认设置下运行(p= 1),并为8类模块中的每一类输出几个模块,并将所有子图聚类在一起。对这些模块进行进一步过滤,以备后续分析:保留含有5-50个基因的聚类,排除含有90%以上同源边的模块。
父视图的计算
父视图步骤描述模块之间以及调节器和模块之间的交互。通过计算两个模块共享的每个输入网络的边数,将每个模块与其他模块进行比较。将该值与随机抽样中的共享边缘计数进行比较。在抽样中,我们生成了1000倍的两个随机模块,这两个模块的节点数量与被调查的两个模块相同。导出的分布被用于z分数变换与\(z = \frac{x - \mu}{\sigma}\)考虑到的意思是\μ(\ \)而标准差σ\ (\ \)从随机分布。的\ (z \)-score通过计算ap-value(显著性截止:0.05),对该分布进行右尾检验\(1 - CDF\左(z \右)\)在哪里\ (CDF \)为累积分布函数。输出由每个模块类型(ALL, COM, COR,…)的一个文件组成,其中包含该类型的每个模块与每个输入网络的所有其他模块之间的交互,并显示共享交互的计数、z得分和p价值。如果两个模块之间不存在交互,则将z分数设置为0p-value设置为1。
superview计算了另外三个输出,表征了模块和调节器之间的关系。对于每个调控因子(TF或miRNA),我们计算了每个rf -模块对和每种相互作用类型的rf -模块连接强度\ \(压裂{1}{\左| N \右|}\ mathop \总和\ limits_ {{n_{我}\ N}} n_{我}\)N是所有模块基因\ (n_{我}\)= 1表示边存在,否则\ (x = 0 \).这给出了一个TF和一个模块之间存在多少交互的部分,并用于查找与一个模块或一组模块强连接的调节器。另一项分析显示了针对每个模块的特定模块有多少不同的tf或mirna\ \(压裂{没有。调节器-模块交互}{{调节器总数}}\).这提供了一个模块的专一性,并允许调查一个模块是被少数还是许多监管者针对。另一项分析显示了某一调节器的目标模块数量,并显示了与派生模块总数相比的一小部分。对于每个调节器,我们计算调节器特异性通过\ \(压裂{没有。目标模块}{{模块总数}}\)这使得我们可以研究哪些tf和mirna针对的是广泛的模块,而不是一些特定于单个模块或少量模块。
功能富集分析
对于每个模块,SUBATOMIC都进行了功能富集,以了解其生物学相关性。我们使用GOATOOLS的Python实现来计算每个模块的GO术语的丰富程度[46].我们提供了三种选择作为富集背景:所有基因都存在于一种特定类型的模块中,所有基因都存在于输入网络中,或者所有基因都写在用户指定的文件中。在我们的分析中,我们使用了一个用户定义的输入,其中包含所有根据ensemble注释的人类基因作为输入。结果汇总在每个模块的一个文件中。只有用修正的结果p-value > 0.05根据Benjamini和Hochberg FDR校正而保留[107].除了标准GOATOOLS输出之外,我们还为上行排序提供了一个秩p-每个模块的值,sincep-值在模块之间可能有很大差异,丰富的功能取决于哪些进程注释得很好,以及有多少GO术语可用。允许筛选每个模块的前n个条目。我们进一步报告了每个重要GO项的log2折叠变化。
可视化
为了在Cytoscape中可视化模块,我们提供了许多可以导入的文件。最重要的文件是包含模块的网络表示的nnf文件。此外,我们生成了一个noa文件,用它的类型(TF、基因或miRNA)、基因名称和一个简短的可选功能描述来注释每个节点。管道的每次运行还会生成xml格式的Cytoscape样式表,以与nnf和noa文件格式提供的信息一致的方式格式化网络,也可以导入。xml文件还可以适应更多的自定义样式选择。
运行时注意事项
对图的操作通常会带来很高的计算成本。流水线中的几个步骤已经并行化了,但是仍然存在一些瓶颈。子图检测算法效率高,可在不到一分钟的时间内发现数百万个子图。SCHype可以聚集成千上万的子图,但它的运行时间超级线性地增加。此外,各模块之间的超视图比较计算和功能注释是最耗时的步骤。由于这些步骤一次处理一个模块,我们以一种可以由不同核心处理每个模块的方式并行处理它们。原则上,有多少内核,就可以并行处理多少模块。但是,由于每个单独的进程都需要一定数量的内存,因此必须在可用的内核数量和内存数量之间谨慎地保持平衡,以便为并行化找到合适的内核数量。对于我们在a上的应用智人在复合网络中,我们在2 × 18核Intel Xeon Gold 6240 (Cascade Lake @ 2.6 GHz)处理器上运行了限制在8核和70 GB RAM的SUBATOMIC,这导致了两天的运行时间。
智人复合网络的构建
我们将来自不同来源的五种不同类型的交互统一为一个复合网络表示(表1).在OmniPath中,我们纳入了53232个由526个调控因子和14488个靶基因形成的tf -靶基因相互作用(访问17.01.2022)[19,20.].我们将来自DoRothEA的所有相互作用纳入证据类A、B和C,以及tf -靶基因和TF-miRNA相互作用。从同一数据库中,我们包含了850个mirna和3210个靶基因之间的11085个mirna -靶标相互作用(访问17.01.2022)。基于GeneTree管道从ensemble中检索基因之间的同源性[49].该管道在简单情况下采用互惠最佳BLAST方法,但也通过解决一对多和多对多关系考虑更复杂的本体。我们应用最小倒数序列识别率50%作为阈值,以包括一对基因之间的同源相互作用。我们只考虑了该分析中至少一个其他数据集涉及的基因。此外,我们添加了具有相同种子序列的同源miRNAs。这总共为4862个基因或miRNAs提供了10,847个相互作用。我们从HumanNet v2中获得蛋白质-蛋白质相互作用和协同功能相互作用[21,50].我们使用HumanNet提供的log- likelihood score (LLS)来筛选LLS≤3.0的交互。由于HumanNet中相互作用的数量远远大于调控相互作用的数量,这些过滤步骤试图平衡相互作用的数量,而不丢失许多包含的基因。我们选择了由10,950个基因组成的24,773个蛋白质-蛋白质相互作用和10,683个基因组成的66,373个共功能相互作用。对于无向交互集,将重复项合并(例如,对于无向交互集,A-B和B-A等价)。这总共删除了6个蛋白质-蛋白质相互作用,28个共功能相互作用和39个同源相互作用。我们将所有基因映射到ensemble标识符,使它们在网络之间具有可比性。对于miRNAs,我们保持了标准命名约定(例如hsa-miR-600e),这避免了与ensemble基因标识符的潜在重叠。我们忽略了不能被映射到一个ensemble ID的基因。分析中包含的基因是基于第38版人类基因组。 We included annotated genes on chromosomes 1–22 as well as X and Y. All interactions were merged into one file to create the composite network. In this file, each interaction was represented by the two interacting nodes as well as their edge color. The edge color was represented by a network specific letter (TF-target gene interactions: R, miRNA-target gene interactions: M, homologous interactions: H, protein–protein interactions: P, Co-functional interactions: C, Additional file1).
缺氧表达数据集
我们使用了来自三种不同细胞系在周期性和慢性缺氧条件下的表达数据来对模块进行背景化(GEO: GSE53012) [58].使用单通道阵列归一化(SCAN)对Affymetrix微阵列数据进行处理和归一化[108].它通过应用混合建模方法纠正了技术偏差的影响,如GC含量[108].为了计算生物活性,我们使用p-来自原始发布的差异表达式分析的值。
语境化
我们实现了几种方法将来自摄动实验或病例和对照样本实验的表达式数据与模块相关联。我们计算了模块中每对基因之间的平均皮尔逊相关系数nPCC,并将其与1000个模块的样本的nPCC进行比较,以测量模块内的共表达。接下来,我们为每个模块nPCC推导出z分数,并将其转换为ap-值通过标准正态分布的CDF。我们还添加了表达式相关差异评分(ECD评分)的实现,与控制条件相比,它突出显示特定于实验条件的模块[28].在那里,我们从条件样本的皮尔逊相关性中减去模块中的每条边的案例样本的皮尔逊相关性,并在每个模块的所有边上取平均值。我们对1000个随机生成的模块重复此操作,以获得背景分布,然后用于计算z分数,并将其转换为p-值使用标准正常的cd。只要有足够的表达式值和条件,我们就可以通过关联来处理边的动态,通过重新布线原则来处理边的动态。
与之前的框架相比,我们实现了一个额外的度量,以捕获响应条件变化的模块。此模块活动评分(\ ({} _ {} \))使用p-不同条件之间的比较值(例如,从病例和对照样本之间的差异表达式),并将其转换为z分数\ (z_{我}= \θ^{- 1}\离开({1 - p_{我}}\)\)与\ \(θ^ {- 1}\)为逆正规CDF [79].对于每个模块,我们计算了总z分数\ (z_{一}= \压裂{1}{\√6 k} \ mathop \总和\ limits_{所有模块基因}z_{我}\)对于每个模块大小,我们抽取了1000个相同大小的随机模块,并将其作为背景分布来计算归一化活动得分\ (s_{一}= \压裂{{\离开({z_{一}- \ mu_ {k}} \右)}}{{\ sigma_ {k}}} \).高的值\ (s_{一}\)表明在特定的实验条件下,该模块可以解释为一个活动子网。
数据和材料的可用性
GitHub:https://github.com/CBIGR/SUBATOMIC,以及Docker版本的链接。Zenodo:https://doi.org/10.5281/zenodo.6556413,输入网络和SUBATOMIC输出的原始数据。
缩写
- 2 fb:
-
两节点反馈子图
- 背景:
-
循环反馈子图
- COM:
-
复杂的子图
- 警察:
-
Co-pointing子图
- 林后:
-
粘住子图
- FB2U:
-
反馈2无向子图
- FBU:
-
反馈无向子图
- 该组织:
-
前馈回路
- 射频:
-
调节因子
- TF:
-
转录因子
- 咖啡:
-
Co-functional交互
- 德:
-
差异表达
参考文献
基因表达的统一理论。细胞。2002;108:439-51。
斯皮兹F,弗隆EEM。转录因子:从增强子结合到发育控制。Nat Rev Genet。2012;13:613-26。
Venters BJ, Pugh BF。真核基因是如何转录的。危重生物化学与分子生物学2009;44:17 - 41。
王晓燕,王晓燕。转录因子及其辅助因子的组合功能。Curr Opin Genet Dev. 2017; 43:73-81。
克莱姆·SL, Shipony Z,格林利夫WJ。染色质可及性和调控表观基因组。Nat Rev Genet 2019; 20:207-20。
Hawkins RD, Hon GC, Lee LK, Ngo Q, Lister R, Pelizzola M,等。多能性和谱系承诺的人类细胞的独特表观基因组景观。细胞干细胞。2010;6:479-91。
Bartel DP。MicroRNAs:目标识别和调节功能。细胞。2009;136:215-33。
Morozova N, Zinovyev A, Nonne N, Pritchard L-L, Gorban AN, harrel - bellan A. microRNA作用模式的动力学特征。RNA。2012; 18:1635-55。
姚强,陈燕,周旭。microRNAs在表观遗传调控中的作用。Curr Opin chemistry Biol 2019; 51:11-7。
张智,曹勇,翟勇,马旭,安旭,张松,等。MicroRNA-29b在猪早期胚胎发育中通过靶向Dnmt3a/3b和Tet1/2/3调控DNA甲基化。2018; 60:197-204。
Bhat SA, Ahmad SM, Mumtaz PT, Malik AA, Dar MA, Urwat U,等。长链非编码rna:作用机制和功能用途。非编码RNA Res. 2016; 1:43-50。
李高N, Y,李江,高Z,杨Z,李Y, et al。长非编码rna:癌症的调控机制、研究策略和未来方向。肿瘤防治杂志。2020;10:2903。
Cesana M, Cacchiarelli D, Legnini I, Santini T, Sthandier O, Chinappi M,等。长链非编码RNA通过作为内源性竞争RNA的功能来控制肌肉分化。细胞。2011;147:358 - 69。
周勇,孟X,陈思,李伟,李丹,辛格R,等。IMP1通过促进UCA1的衰减和减少UCA1对miR-122-5p的海绵效应来调节UCA1介导的细胞侵袭。乳腺癌Res. 2018;20:32。
康拉德·B,安东纳拉基斯SE。基因复制:表现型多样性的驱动和人类疾病的原因。Annu Rev Genomics Hum Genet. 2007; 8:17-35。
Lan X, Pritchard JK。串联复制基因的协同调控减缓了哺乳动物亚功能化的进化。科学。2016;352:1009-13。
Garcia-Alonso L, Holland CH, Ibrahim MM, Turei D, Saez-Rodriguez J.人类转录因子活性评估的基准和资源整合。基因组研究》2019;29:1363 - 75。
Garcia-Alonso L, Iorio F, Matchan A, Fonseca N, Jaaks P, Peat G,等。转录因子活性增强肿瘤中药物敏感性标记物。实用癌症杂志2018;78:769 - 80。
Türei D, Valdeolivas A, Gul L, Palacio-Escat N, Klein M, Ivanova O,等。多细胞组学分析的细胞内和细胞间信号的综合知识。Mol sys Biol. 2021;17: e9923。
Türei D, Korcsmáros T . OmniPath:文献策划的信号通路资源的指南和网关。Nat方法。2016;13:966-7。
黄硕,金春春,杨硕,金娥,哈特,马珂特,等。HumanNet v2:用于疾病研究的人类基因网络。核酸决议2019;47:D573-80。
王晓燕,王晓燕。蛋白质- dna相互作用网络的研究进展。中华分子生物学杂志2003;333:75-85。
张晨,李硕,Mardinoglu A,华强。生物网络对基因共表达的组合效应研究。杂志。2016;7:160。
Martinez NJ, Ow MC, Barrasa MI, Hammell M, Sequerra R, doucte - stamm L,等。秀丽隐杆线虫基因组尺度的microRNA网络包含具有高通量容量的复合反馈基序。基因Dev。2008;22:2535-49。
Guo Y, Alexander K, Clark AG, Grimson A, Yu H.综合网络分析揭示了转录因子和microRNAs的不同调控作用。RNA。2016; 22:1663 - 72。
张晓燕,张晓燕,张晓燕,等。基于遗传算法的基因调控网络研究。Current Opin Syst bio . 2020; 23:38-46。
Williams RM, Candido-Ferreira I, Repapi E, Gavriouchkina D, Senanayake U, Ling ITC,等。全神经嵴基因在体调控网络的重建。Dev细胞。2019;51:255 - 276. e7。
李晓燕,王晓燕。植物和蠕虫综合基因调控网络中网络基序模块的功能、动态和演化。核酸决议2018;46:6480-503。
多林斯基K, Chatr-aryamontri A,泰尔斯M.可计算生物学中蛋白质和基因相互作用数据的系统策展。BMC医学杂志。2013;43。
着陆器广告。理解的边缘。BMC医学杂志。2010;首度。
Azad AKM。整合异构数据集进行癌症模块识别。正确的做法:Keith JM,编辑。生物信息学:卷二:结构,功能和应用。纽约:施普林格;2017.119 - 37页。
Azad AKM, Lee H.结合拓扑和数据驱动属性的基于投票的癌症模块识别。PLoS ONE。2013; 8: e70498。
Bennett L, Kittas A, Muirhead G, Papageorgiou LG, Tsoka s多重生物网络中复合群落的检测。Sci众议员2015;5:10345。
李晓燕,李晓燕,李晓燕,李晓燕。基于网络的多组学纵向数据整合方法研究。核酸学报。2021;18(9):551。
A牧师,A牧师,A律师,Lê曹K-A。整合微生物组纵向研究与其他数据类型的通用多变量框架。麝猫。2019;10:963。
李志刚,李志刚,李志刚。基于柠檬树的多组学模块网络推理。科学通报,2015;11:e1003983。
Durmaz A, Henderson TAD, Bebek G.跨多种癌症功能交互模式的频繁子图挖掘。Pac Symp生物计算。2021;26:61 - 72。
Langfelder P, Horvath S. WGCNA:加权相关网络分析的R包。BMC Bioinform。2008;9:559。
Saelens W, can面条R, Saeys Y.基因表达数据模块检测方法的综合评价。Nat Commun。2018;9:1090。
Silverbush D, Cristea S, Yanovich-Arad G, Geiger T, Beerenwinkel N, Sharan R.同时整合多组学数据提高癌症驱动模块的识别。细胞系统。2019;8:456 e5——466.。
吴超,张峰,李旭,张松,李娟,苏芳,等。复合功能模块推断:通过mantel试验检测转录调控与蛋白质相互作用的协同作用。BMC系统生物学。2010;4:82。
低氧微环境与转移性骨病。国际分子生物学杂志2018;19:E3523。
Todd VM, Vecchi LA, Clements ME, Snow KP, Ontko CD, Himmel L,等。乳腺肿瘤中的低氧诱导因子信号通过部位特异性的方式控制肿瘤的自发扩散。Commun杂志。2021;4:1-18。
Houbraken M, Demeyer S, Michoel T, Audenaert P, Colle D, Pickavet M.基于索引的一般对称子图匹配算法(ISMAGS):利用对称实现更快的子图枚举。PLoS ONE。2014; 9: e97896。
基于超图谱聚类的复杂网络对齐与整合。物理学报2012;86:056111。
Klopfenstein DV, Zhang L, Pedersen BS, Ramírez F, Warwick Vesztrocy A, Naldi A,等。GOATOOLS:用于基因本体分析的python库。Sci众议员2018;8:10872。
Shannon P, Markiel A, Ozier O, Baliga NS, Wang JT, Ramage D,等。Cytoscape:生物分子相互作用网络集成模型的软件环境。基因组研究》2003;13:2498 - 504。
李文华,李文华,李文华,等。miRBase:从microRNA序列到miRBase的功能。核酸决议2019;47:D155-62。
Vilella AJ, Severin J, Ureta-Vidal A, Heng L, Durbin R, Birney e。基因组研究》2009;19:327-35。
李毅,布卢姆,王皮,沈杰,Marcotte EM.基于网络增强全基因组关联数据的候选疾病基因优先排序。基因组研究》2011;21:1109-21。
侯亮,陈敏,张长强,赵杰,赵华。重组内疚:全基因组关联研究中通过网络重组的基因优先排序。中华医学杂志。2014;23:2780-90。
田伟,张绿,tazan M, Gibbons FD, King OD, Park J,等。结合联想罪恶感和剖面罪恶感预测酿酒酵母菌的基因功能。基因组医学杂志。2008;9:S7。
Shinsky SA, Monteith KE, Viggiano S, Cosgrove MS.参与组蛋白甲基化的人类SET1家族核心复合物的生化重建和系统发育比较。中国生物化学杂志2015;29:6361 - 75。
王曦,Rosikiewicz W, Sedkov Y, Martinez T, Hansen BS, Schreiner P,等。PROSER1介导TET2 O-GlcNAcylation,调节utx依赖增强子和CpG岛上的DNA去甲基化。生命科学联盟2021;5:e202101228。
Urizar-Arenaza I, Osinalde N, Akimov V, Puglia M, Candenas L, Pinto FM,等。磷蛋白组学和功能分析揭示了人类精子kappa阿片受体下游的精子特异性蛋白变化*。分子细胞蛋白质组学。2019;18:S118-31。
徐坤,杨磊,张磊,齐浩。AKAP3缺失破坏小鼠精子亚细胞结构和蛋白质组的完整性,导致雄性不育。发展。2020;147:dev181057。
陈培生,赵文涛,徐培良,林淑春,彭宜春,王春燕,等。人类疾病中缺氧的病理生理学意义。中国生物医学杂志2020;27:63。
Olbryt M, Habryka A,学生S, jarzibb M, Tyszkiewicz T, Lisowska KM。三种肿瘤细胞系在实验循环和慢性缺氧条件下的整体基因表达谱。PLoS ONE。2014; 9: e105104。
炎症和缺氧:HIF和PHD亚型选择性。2019; 25:33-46。
Depoix CL, de Selliers I, Hubinont C, Debieve F. HIF1A和EPAS1在体外增强缺氧诱导的人足月细胞滋养层细胞抑制素α链表达上调。Mol Hum red . 2017; 23:199-209。
李建伟,柯杰,朱超,Eltzschig HK。人类疾病中的缺氧信号及其治疗靶点。Exp Mol Med. 2019; 51:1-13。
王桂莲,王晓峰,王晓峰。低氧诱导因子1的纯化及鉴定(∗).生物化学杂志,1995;270:1230-7。
王铮,石勇,应春,蒋艳,胡娟。缺氧诱导的PLOD1过表达通过NF-κB信号通路参与胶质母细胞瘤的恶性表型。致癌基因。2021;40:1458 - 75。
黄燕。缺氧诱导因子(HIF)-1调节通路及其在恶性肿瘤和缺血治疗干预中的潜力。耶鲁生物医学杂志2007;80:51-60。
耿宏,薛晨,Mendonca J,孙晓霞,刘强,Reardon PN,等。缺氧和雄激素之间的相互作用控制了一个代谢开关,使其对雄激素/ ar靶向治疗产生耐药性。Nat Commun。2018;9:4972。
Mitani T, Yamaji R, Higashimura Y, Harada N, Nakano Y, Inui H.在低雄激素环境下,缺氧通过缺氧诱导因子-1α增强雄激素受体的转录活性。中国生物化学杂志,2011;123:58-64。
Gilkes DM, Bajpai S, Wong CC, Chaturvedi P, Hubbi ME, Wirtz D,等。前胶原赖氨酸羟化酶2在缺氧诱导的乳腺癌转移中至关重要。Mol Cancer Res. 2013; 11:456-66。
brick RK, McKnight SL.修饰HIF的脯氨酰基-4-羟基化酶的保守家族。科学。2001;294:1337-40。
献给KKW,黄乐。HIF脯氨酰羟化酶EGLN1对缺氧诱导因子1 α (HIF-1 α)转录活性的抑制作用生物化学杂志2005;280:38102-7。
Shah R, Smith P, Purdie C, Quinlan P, Baker L, Aman P,等。脯氨酰3-羟化酶P3H2和P3H3是乳腺癌表观遗传沉默的新靶点。中华癌症杂志2009;100:1687-96。
Kumar B, Khaleghzadegan S, Mears B, Hatano K, Kudrolli TA, Chowdhury WH,等。通过补充功能microRNA文库筛选,确定miR-30b-3p和miR-30d-5p作为前列腺癌雄激素受体信号的直接调控因子。Oncotarget。2016;7:72593 - 607。
贝克WJ,哈里斯IS,麦TW。FOXO3a在缺氧应激下被激活,通过调控CITED2抑制hif1诱导的细胞凋亡。摩尔细胞。2007;28:941-53。
Chaanine AH, Kohlbrenner E, Gamb SI, Guenzel AJ, Klaus K, Fayyaz AU,等。FOXO3a调节BNIP3,并调节线粒体钙、动力学和心脏应激功能。中华医学杂志2016;311:H1540-59。
维甲酸受体相关孤儿受体RORα在缺氧条件下调节角质形成细胞分化和存活。细胞物理学报。2018;233:641-50。
陈强,周莹,Richards AM,王鹏。miRNA-221上调通过DDIT4/mTORC1和Tp53inp1/p62途径抑制缺氧/再氧诱导的自噬。生物化学与生物物理学报。2016;474:168-74。
徐锐,罗旭,叶旭,李辉,刘辉,杜强,等。SIRT1/PGC-1α/PPAR-γ与缺氧诱导的非小细胞肺癌化疗耐药相关。肿瘤防治杂志。2021;11:2791。
赵永忠,刘学林,沈国明,马永宁,张凤玲,陈明涛,等。缺氧通过hif -1依赖机制诱导HepG2细胞系过氧化物酶体增殖物激活受体γ的表达。生物化学。2014;543:40-7。
Rios-Colon L, Kumar P, Kim S, Sharma M, Su Y, Kumar A,等。肉碱棕榈酰转移酶1在缺氧条件下调节前列腺癌生长。癌症(巴塞尔)。2021; 13:6302。
艾德克·T, Ozier O, Schwikowski B, Siegel AF.发现分子相互作用网络中的调节和信号通路。生物信息学,2002;18 (suppl_1): S233-40。
李燕,孙秀霞,钱德泽,戴明生。癌症中MYC和HIF之间的分子串扰。前沿细胞发展生物学。2020;8:590576。
Menendez MT, Teygong C, Wade K, Florimond C, blade IJ。siRNA筛选发现宿主己糖激酶2 (HK2)基因是弓形虫感染细胞中重要的缺氧诱导转录因子1 (HIF-1)靶基因。MBio。2015; 6: e00462。
单芳,黄铮,熊瑞,黄秋燕,李娟。缺氧条件下hif1 α诱导的KLF4上调促进人血管平滑肌细胞迁移。细胞物理杂志,2020;235:141-50。
Obach M, Navarro-Sabaté A, Caro J, Kong X, Duran J, Gómez M,等。6-磷酸果糖-2激酶(pfkfb3)基因启动子包含缺氧诱导因子-1结合位点,对缺氧反应的transactivation必不可少。生物化学杂志2004;279:53562-70。
Sysol JR, Chen J, Singla S,赵s, Comhair S, Natarajan V,等。缺氧使Micro-RNA-1减少,并通过调节鞘氨醇激酶1促进肺血管重构的发展。中华医学杂志。2018;314:L461-72。
禹sk,权MS,耿卓,陈卓,Ivanov A, Bhatta S,等。缺氧诱导因子1和特异性蛋白1的连续激活是缺氧诱导Abcc8转录刺激所必需的。中华脑血管病杂志2012;32:525-36。
李文峰,李文峰。无监督聚类有效性估计的重采样方法。神经第一版。2001;13:2573 - 93。
Hubert L, Arabie P.比较分区。J Classif。1985;2:193 - 218。
杨永平,李志刚,李志刚。聚类比较的信息理论度量:变量、属性、归一化和概率校正。J Mach Learn Res. 2010; 11:2837-54。
高寒地区植物区系的分布。新植醇。1912;11:37-50。
刘勇,叶鑫,于春春,邵伟,侯杰,冯伟,等。TPSC:加权网络中基于拓扑电位和谱聚类的模块检测方法及其在基因共表达模块发现中的应用。BMC Bioinform。2021;22:111。
张军,黄凯。归一化ImQCM:加权图弱拟团检测算法及其在癌症基因共表达模块发现中的应用。癌症告知。2014;13 s3: CIN.S14021。
Zoppi J, Guillaume J- f, Neunlist M, Chaffron S. MiBiOmics:用于多组学数据探索和集成的交互式web应用程序。BMC Bioinform。2021;22:6。
陆霞,刘芳,苗青,刘鹏,高原,何凯。一种识别基因相互作用模式的新方法。BMC基因组学。2021;22:436。
卢鑫,朱铮,彭鑫,苗奇,罗赟,陈欣。InFun:一种生物网络中重叠基因群落的检测方法。SIViP。2021; 15:681-6。
Dittrich MT, Klau GW, Rosenwald A, Dandekar T, Müller T.识别蛋白质-蛋白质相互作用网络中的功能模块:一种综合的精确方法。生物信息学。2008;24:i223 - 231。
Moon S, Lee H. MOMA:用于多组学数据解释和分类的多任务注意力学习算法。生物信息学。2022;38:2287 - 96。
史文杰,庄勇,Russell PH, Hobbs BD, Parker MM, Castaldi PJ,等。表型特异性多组学网络的无监督发现。生物信息学。2019;35:4336-43。
贾思安,王晓燕,王晓燕,Barabási。疾病模块检测(DIAMOnD)算法源自对人类交互组中疾病蛋白连接模式的系统分析。科学通报,2015;11:e1004120。
Glaab E, Baudot A, Krasnogor N, Schneider R, Valencia A. richnet:基于网络的基因集富集分析。生物信息学。2012;28:i451-7。
王波,胡娟,王勇,张超,周勇,于磊,等。将独立的连接部件连接起来,形成一个简洁的疾病模块。BMC Bioinform。2020;21:433。
Winkler S, Winkler I, Figaschewski M, Tiede T, Nordheim A, Kohlbacher O.基于多组学数据的最大去管制子网络的全新识别。BMC Bioinform。2022;23:139。
阮浩,Shrestha S, Tran D, Shafi A, Draghici S, Nguyen t。主动子网识别工具和软件的综合研究。麝猫。2019;10:155。
吴松,陈东,Snyder议员。网络生物学在定量遗传学和多组学之间架起桥梁,绘制复杂疾病图谱。《生物化学评论》2022;66:102101。
Alcalá-Corona SA, sandovar - motta S, Espinal-Enríquez J, Hernández-Lemus E.生物网络的模块化。麝猫。2021;12:701331。
Demeyer S, Michoel T, Fostier J, Audenaert P, Pickavet M, Demeester P.基于索引的子图匹配算法(ISMA):基于优化搜索树的大型网络快速子图枚举。PLoS ONE。2013; 8: e61183。
霍恩RA,约翰逊CR,矩阵分析。第二版。剑桥;纽约:剑桥大学出版社;2012.
控制错误发现率——一种实用而有效的多重测试方法。中国科学(d辑)1995;
短毛罗,孙Y,坎贝尔JD,伦伯格ME, Bild AH,约翰逊WE。一种促进个性化医疗工作流程的单样本微阵列归一化方法。基因组学。2012;100:337-44。
确认
我们要感谢Kenneth Stoop和Pieter Audenaert对运行ISMAGS算法的帮助。此外,我们还要感谢Hayoung Kim、Heesoo Song和Jietse Verweirder对Snakemake管道原型开发的支持。
资金
J.L.得到了根特大学BOF博士奖学金的支持,这项工作也得到了BOF启动拨款BOF/STA/201909/030“多组学数据集成以阐明复杂疾病的原因”的资助。
作者信息
作者和联系
贡献
根据征信制度的贡献(https://casrai.org/credit/, JL = Jens Uwe Loers, VV = Vanessa Vermeirssen):概念化:JL, VV,数据策划:JL,形式分析:JL,资金获取:JL, VV,调查:JL,方法:JL, VV,项目管理:VV,软件:JL,监督:VV,验证:JL,可视化:JL,写作-初稿:JL, VV,写作-重写和编辑JL, VV。两位作者都阅读并批准了最终稿。
相应的作者
道德声明
伦理批准和同意参与
本研究分析的数据来自公共数据库,因此不需要伦理批准和同意参与。
同意出版
不适用。
相互竞争的利益
作者声明他们没有竞争利益。
额外的信息
出版商的注意
伟德体育在线施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
权利和权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。欲查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
Loers, J.U., Vermeirssen, V. SUBATOMIC:基于子图的多组学聚类框架,用于分析集成的多边缘网络。BMC生物信息学23363(2022)。https://doi.org/10.1186/s12859-022-04908-3
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12859-022-04908-3
关键字
- 复合子图
- Multi-edge网络
- 网络分析
- Multi-omics
- 模块
- 基因调控
- 拓扑结构
- 缺氧
- 基因功能预测
- 基因调控网络